一种基于捕获测序的COL1A1-PDGFB融合基因检测方法
文献发布时间:2023-06-19 12:27:31
技术领域
本发明涉及融合基因检测领域,具体地说,特别涉及一种基于捕获测序的COL1A1-PDGFB融合基因检测方法。
背景技术
皮肤隆突性纤维肉瘤(dermatofibrosarcoma protuberans,DFSP)是一种发生于皮肤的缓慢生长的纤维肉瘤,常见于中年男性、好发于躯干(包括胸壁、腹壁和背部)、四肢近段和头颈部,但极少发生于四肢远端。肿瘤虽然生长缓慢,极少发生远处转移,但由于其可以侵袭性生长,侵犯皮下组织,病灶边缘往往边界不清,因此具有较高的局部复发率。
皮肤和软组织肿瘤中已报道存在大量融合基因,由于DFSP经常会从真皮延伸到皮下组织,仅依靠组织学很难区分DFSP与其他纤维瘤。目前临床上主要通过遗传分析方法检测COL1A1与PDGFB的融合基因加以鉴别,其主要由t(17;22)(q22;q13)的染色体易位所引起,已被公认为是一种可靠而又有价值的分子诊断工具。除此之外,有研究发现具有以上融合基因的DFSP可以应用伊马替尼进行治疗。
目前常规融合基因检测方法主要有细胞核型分析、反转录-聚合酶链反应(RT-PCR)、荧光原位杂交(FISH)3种。细胞核型分析是基于较大范围染色体异常而进行的初步分析,其灵敏度取决于可供检测的中期细胞数,成功率相对降低只能检测中期细胞。RT-PCR主要提取肿瘤组织中的mRNA,在反转录酶的作用下,合成cDNA,再以此为模板进行聚合酶链式反应。与提取肿瘤组织的DNA进行常规PCR相比,此法简单、快速,组织中只需要少量的肿瘤即可被检测,能显著提高肿瘤检测的特异性,但由于检测受RNA质量的影响,在石蜡等研究组织中敏感度较低。FISH技术最早由Pinkel等人报道,系采用标记有荧光分子的一小段DNA序列(探针),在合适的湿度和盐离子浓度下,与组织切片或细胞涂片上的组织DNA杂交,在荧光显微镜下显示与之相应的染色体某个区域或整条染色体。该技术可以探查有丝分裂期间细胞核内染色体数目和结构的异常,尤其适用于证实染色体易位、缺失和基因扩增。其灵敏度高于传统的细胞核型分析且对检测样本的制备无特殊要求,既可用新鲜组织也可用于4%甲醛溶液固定或石蜡包埋的组织标本。尽管该方法已经被认为是临床上检测融合基因的金标准,然而其局限性在于样本的判读受制片和染色质量的影响,且不同厂家的探针往往质量差异较大,不易形成成熟的检测规范。最近,也有人报道采用RNA高通量测序法检测融合基因,其在检测已知融合基因改变的同时可以发现未知的融合基因,有利于新致病基因的发掘。然而由于其建测序文库,对RNA的质量要求较高,因此不利于方法的持续开展。
发明内容
为了解决现有技术的问题,本发明实施例提供了一种基于捕获测序的COL1A1-PDGFB融合基因检测方法。所述技术方案如下:
一方面,提供了一种基于捕获测序的COL1A1-PDGFB融合基因检测方法,包括:
(1)合成COL1A1与PDGFB捕获探针;
(2)采用标准方法提取DNA和构建DNA测序文库;
(3)扩增DNA测序文库并进行质检;
(4)扩增的DNA文库探针杂交捕获;
(5)扩增捕获后的DNA测序文库并进行质检;
(6)高通量测序和生物信息分析。
进一步地,所述步骤(1)具体为:
根据COL1A1-PDGFB高发染色体断裂区设计捕获探针,探针捕获区域设计包含COL1A1区域,长度12,211bp和PDGFB区域,长度8,026bp;
其中,探针为DNA探针,长度100bp,重叠分布在捕获区域。
进一步地,所述步骤(2)具体为:
采用组织DNA提取试剂盒提取肿瘤组织的基因组DNA;
用500ng的基因组DNA构建DNA测序文库;基因组DNA与片段化酶在37℃反应20分钟,将DNA片段化成200-300bp大小的片段;
采用末端修复酶65℃反应30分钟补平DNA片段末端,同时利用加A酶在DNA片段的平末端连接一个A碱基;
最后,应用DNA连接酶20℃反应15分钟将测序的接头连接在DNA片段上;同时采用AMPure XP Beads对DNA片段进行纯化,去除未结合的接头。
进一步地,所述步骤(3)具体为:
采用KAPA HiFi HotStart ReadyMix扩增DNA文库;其中,扩增条件:98℃45秒;然后98℃15秒、60℃30秒、72℃30秒,如此反复11个循环;最后72℃1分钟;
PCR产物扩增后同样采用AMPure XP Beads进行产物纯化;
采用nano测定比值1.7~20之间,采用Qubit进行定量文库DNA要大于1.0微克;
最后用安捷伦2100进行片段分析,片段大小应该在150-500bp之间,片段峰应在320bp左右。
进一步地,所述步骤(4)具体为:
采用AMPure XP Beads纯化DNA文库;
将纯化产物与合成的核酸探针进行杂交47℃16-20小时,探针都采用生物素标记,用磁珠进行洗脱。
进一步地,所述步骤(5)具体为:
采用KAPA HiFi HotStart ReadyMix扩增DNA文库;其中,扩增条件:98℃45秒;然后(98℃15秒、60℃30秒、72℃30秒,如此反复13个循环;最后72℃1分钟;
PCR产物扩增后同样采用AMPure XP Beads进行产物纯化;
采用nano测定比值1.7~20之间,采用Qubit进行定量文库DNA要大于500纳克;
最后用安捷伦2100进行片段分析,片段大小应该在150-500bp之间,片段峰应在320bp左右。
进一步地,所述步骤(6)具体为:
采用Illumina高通量测序技术对序列进行检测,测序方法采用2*150Pair-End测序,NovaSeq检测;
测序数据下机后应用Base calling方法从图片信息中提取序列信息;生物信息分析:去除序列中的接头和低质量的数据;
采用BWA将检测到的序列与人的基因组数据库进行比对,并进行注释,找出其中横跨不同染色体的序列;
通过统计和筛选,找出其中比较确定的染色体融合基因变异。
本发明实施例提供的技术方案带来的有益效果是:
本发明提供的一种基于捕获测序的COL1A1-PDGFB融合基因检测方法,前期根据COL1A1与PDGFB经常发生融合基因断裂的位置设计DNA探针,通过外显子捕获技术,捕获肿瘤组织样本中发生融合的基因组DNA,再采用高通量测序技术对其中的融合基因DNA进行测序检测。该方法已完成30余例临床样本的检测,准确率超过目前临床上常用的荧光原位杂交技术。该技术一旦应用,形成商业化试剂盒可以发现融合基因的断裂位点,有效检测临床组织中极微量的肿瘤变异,而且不受样品种类和肿瘤细胞比例的影响,大大提高临床检测的灵敏度和特异度。
附图说明
为了更清楚地说明本发明实施例中的技术方案,下面将对实施例描述中所需要使用的附图作简单地介绍。显而易见地,下面描述中的附图仅仅是本发明的一部分实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
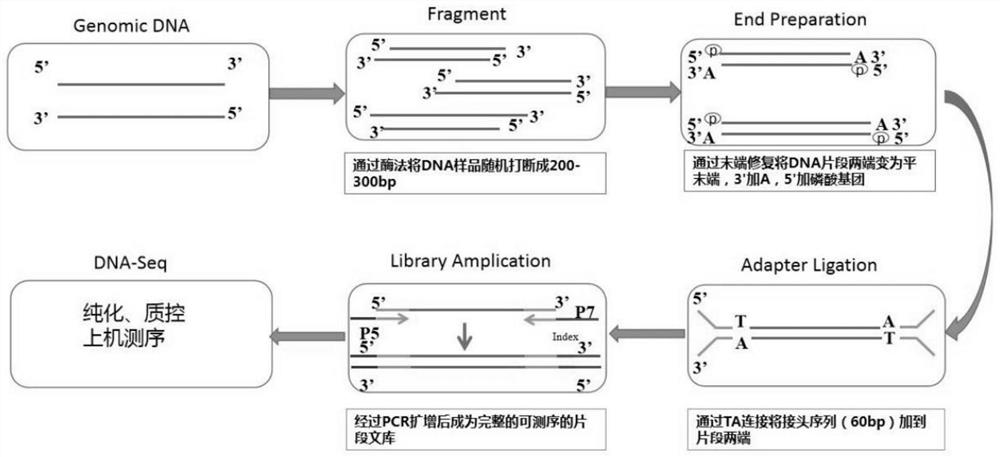
图1是本发明实施例的测序文库的构建与扩增的流程图;
图2是本发明实施例的探针捕获DNA的过程的流程图;
图3是本发明实施例的外显子捕获检测COL1A1-PDGFB融合基因与FISH的横向比较的示意图;
图4是本发明实施例的外显子捕获检测COL1A1-PDGFB融合基因的一代测序验证的示意图。
具体实施方式
为使本发明的目的、技术方案和优点更加清楚,下面将结合附图对本发明实施方式作进一步地详细描述。
本发明提供了一种基于捕获测序的COL1A1-PDGFB融合基因检测方法,包括:
1.合成捕获探针。根据COL1A1-PDGFB高发染色体断裂区设计捕获探针,探针捕获区域设计包含COL1A1(Exon 6-47)区域,长度12,211bp和PDGFB(Intron 2)区域,长度8,026bp。探针为DNA探针,长度100bp,重叠分布在捕获区域。
2.采用标准方法提取DNA和构建DNA测序文库。采用组织DNA提取试剂盒提取肿瘤组织的基因组DNA。用100ng的基因组DNA构建DNA测序文库,如图1所示。基因组DNA与片段化酶(KAPAFrag Enzyme)在37℃反应20分钟,将DNA片段化成200-300bp大小的片段。采用末端修复酶和加A酶(KAPA End Repair&A-tailing Enzyme)使DNA片段补平和加A(65℃反应30分钟)。最后,应用DNA连接酶(KAPADNA Ligase)20℃反应15分钟将测序的接头连接在DNA片段上。同时采用AMPure XP Beads对DNA片段进行纯化,去除未结合的接头。
3.扩增DNA测序文库并进行质检。采用KAPA HiFi HotStart ReadyMix扩增DNA文库(图1所示)。扩增条件:98℃45秒,(98℃15秒,60℃30秒,72℃30秒)11个循环,72℃1分钟。PCR产物扩增后同样采用AMPure XP Beads进行产物纯化。采用nano测定比值1.7~20之间,采用Qubit进行定量文库DNA要大于1.0微克。最后用安捷伦2100进行片段分析,片段大小应该在150-500bp之间,片段峰应在320bp左右。
4.扩增的DNA文库探针杂交捕获。如图2所示,采用AMPure XP Beads纯化DNA文库。将纯化产物与合成的核酸探针进行杂交47℃16-20小时,探针都采用生物素标记,用磁珠进行洗脱。
5.扩增捕获后的DNA测序文库并进行质检。采用KAPAHiFi HotStart ReadyMix扩增DNA文库。扩增条件:98℃45秒,(98℃15秒,60℃30秒,72℃30秒)13个循环,72℃1分钟。PCR产物扩增后同样采用AMPure XP Beads进行产物纯化。采用nano测定比值1.7~20之间,采用Qubit进行定量文库DNA要大于500纳克。最后用安捷伦2100进行片段分析,片段大小应该在150-500bp之间,片段峰应在320bp左右。
6.高通量测序和生物信息分析。采用Illumina高通量测序技术对序列进行检测,测序方法采用2*150Pair-End测序,NovaSeq检测。测序数据下机后应用Base calling方法从图片信息中提取序列信息。生物信息分析:去除序列中的接头和低质量的数据。采用BWA将检测到的序列与人的基因组数据库进行比对,并进行注释,找出其中横跨不同染色体的序列。通过统计和筛选,找出其中比较确定的染色体融合基因变异。
在本实施例中,对30例样本中的9例进行展示。根据COL1A1与PDGFB经常发生融合基因断裂的位置设计DNA探针,通过外显子捕获技术,对这9例隆突性皮肤纤维肉瘤的样本进行检测,发现其中7例具有COL1A1-PDGFB融合基因。进一步采用FISH检测,发现其中6例与FISH的检测结果相一致(如图3)。只有一个样本捕获法检测为阳性而FISH检测却为阴性。采用一代测序技术对该样本的融合位点进行验证,结果如图4所示,该样本的确存在COL1A1-PDGFB融合基因,从而证明了捕获测序的敏感性优于传统FISH检测方法。
以上仅为本发明的较佳实施例,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 一种基于捕获测序的COL1A1-PDGFB融合基因检测方法
- 一种基于单端测序的融合基因检测方法及其应用
