缩小碱基编辑器的编辑窗口的方法、碱基编辑器及用途
文献发布时间:2023-06-19 18:32:25
技术领域
本公开属于生物技术和基因编辑技术领域,具体涉及一种gRNA突变体的构建方法、gRNA突变体、缩小碱基编辑器的编辑窗口的方法、碱基编辑器、分离的多核苷酸、重组表达载体、重组宿主细胞、碱基编辑器、组合物及用途,以及在细胞或受试者中进行基因编辑的方法、治疗或预防疾病的方法。
背景技术
CRISPR/Cas系统(clustered regularly interspaced short palindromicrepeats/CRISPR-associated)是一种由RNA指导的Cas核酸酶对靶向基因进行特定DNA修饰的技术,它是细菌和古生菌为了应对噬菌体和外源质粒的不断攻击而演化出的获得性免疫防御机制。CRISPR/Cas系统自发现以来,已被成功运用到多个物种的基因编辑中,在基因编辑领域获得了广泛的应用。
目前已发现的CRISPR/Cas系统可以分为两类(第1类和第2类),并进一步分为六种类型(I型~Ⅵ型)。其中,第1类系统包括I型、III型和IV型,第2类系统包括II型、V型以及VI型。在常用的CRISPR/Cas系统中,向导RNA(guide RNA,gRNA)引导Cas蛋白在基因组的靶向位点进行精准切割造成DNA双链断裂(double strand break,DSB),宿主细胞利用自身的非同源末端连接(non-homologous end-joining,NHEJ)或基于同源末端重组(homologousend recombination repair,HDR)进行修复,但难以实现针对单碱基的特定编辑。由于DNA的双链断裂具有很多的不确定性,HDR发生的概率很低,而NHEJ会引起碱基的随机插入或缺失,因此,传统的CRISPR/Cas技术在针对单碱基进行基因编辑时存在一定弊端。
碱基编辑器(base editing,BE)的出现弥补了传统CRISPR/Cas技术在单碱基编辑中的缺陷,碱基编辑器无需产生DNA双链断裂,也无需供体DNA的参与,可实现靶位点的精准点突变,在基因突变遗传疾病治疗上具有重大应用前景。现有的碱基编辑器主要包括:胞嘧啶碱基编辑器(Cytosine base editor,CBE),可将目标序列的编辑窗口内的胞嘧啶核苷酸转化为胸腺嘧啶核苷酸(C>T);腺嘌呤碱基编辑器(Adenine base editor,ABE),可将编辑窗口内的腺嘌呤核苷酸转化为鸟嘌呤核苷酸(A>G);以及新型糖基化酶碱基编辑器(Glycosylase base editor,GBE),在大肠杆菌中可将胞嘧啶核苷酸编辑成腺嘌呤核苷酸、在哺乳动物细胞中可将胞嘧啶核苷酸特异性编辑成鸟嘌呤核苷酸。
目前,除了GBE在哺乳动物细胞中可以精确编辑C6位点外,其余基于CRISPR/Cas的碱基编辑器都存在多个核苷酸的编辑窗口,例如,经典的CBE的编辑窗口是4-5个碱基、ABE的编辑窗口也达到了4个碱基,编辑窗口内所有的C或者A都可能被编辑。然而,在已知的致病性突变中,约一半为点突变(也称为单核苷酸多态性,single nucleotidepolymorphism,SNP),对于致病性点突变的修正,往往需要精确地修正一个碱基,额外的碱基修饰反而会带来副作用。因此,缩小CBE和ABE的编辑窗口,甚至将编辑窗口精确到一个碱基显得尤为必要。
然而,目前尚缺少一种通用性的缩小碱基编辑窗口的方法,能够将CBE、ABE
针对CBE,通过选择不同来源的胞嘧啶脱氨酶、不同的CRISPR系统、不同的linker,以及对胞嘧啶脱氨酶的突变可以一定程度缩小CBE碱基编辑窗口。对于ABE而言,除了进化后来源于细菌的腺嘌呤脱氨酶,还未发现其它腺嘌呤脱氨酶与CRISPR系统融合后能有效的实现A到G突变,缺少有效的缩小ABE编辑窗口的方法。迄今为止,尚缺少一种通用性的缩小碱基编辑器的编辑窗口的方法,可以将CBE和ABE精确到一个碱基的方法,极大限制了碱基编辑系统在精准碱基编辑中的应用。
发明内容
鉴于现有技术中存在的问题,例如,缺少通用性的缩小碱基编辑器的编辑窗口的方法,CBE、ABE难以实现对单个碱基的精确编辑的缺陷。为此,本公开提供了一种gRNA突变体的构建方法,通过对gRNA中的向导序列区进行突变,得到gRNA突变体,gRNA突变体应用于碱基编辑器中,能够通用性地缩小碱基编辑窗口,实现对1个碱基的精确编辑。
第一方面,本公开提供了一种gRNA突变体的构建方法,其中,所述方法包括:
突变步骤:对gRNA中与目标核酸的靶序列杂交的向导序列区进行突变,使所述向导序列区的一个或多个位置处具有一个或多个碱基的替换、缺失或插入,形成包含突变核苷酸的突变序列区;
筛选步骤:筛选与未突变的gRNA相比,用于碱基编辑器的编辑窗口缩小的突变体,得到所述gRNA突变体。
在一些实施方式中,根据本公开所述的方法,其中,所述筛选步骤包括:
筛选用于碱基编辑器的编辑窗口为单个碱基的突变体,得到所述gRNA突变体。
在一些实施方式中,根据本公开所述的方法,其中,所述向导序列区具有靠近所述目标核酸的PAM序列的第一端,和远离所述PAM序列的第二端;
所述向导序列区具有m个核苷酸,任意一个所述突变核苷酸位于以所述第二端为起始的第n个核苷酸的位置,1≤n≤m,m、n取正整数;优选1≤n≤m/2,更优选1≤n≤m/3。
在一些实施方式中,根据本公开所述的方法,其中,m取15-30的任意整数,优选15-25的任意整数;
可选地,任意一个所述突变核苷酸位于以所述第二端为起始的第1-10个核苷酸的位置,优选第2-10个核苷酸的位置,更优选第2-7个核苷酸的位置,更优选第2-6个核苷酸的位置。
在一些实施方式中,根据本公开所述的方法,其中,所述突变核苷酸包含1-10个碱基的替换、缺失或插入;优选1-5个碱基的替换、缺失或插入,更优选1-3个碱基的替换、缺失或插入。
第二方面,本公开提供了一种缩小碱基编辑器的编辑窗口的方法,其中,所述方法包括使用如第一方面所述的方法构建gRNA突变体;优选地,所述碱基编辑器的编辑窗口为1个碱基。
第三方面,本公开提供了一种gRNA突变体,其中,所述gRNA突变体由第一方面所述的方法构建得到;优选地,所述gRNA突变体用于碱基编辑器的编辑窗口为单核苷酸位点;
优选地,所述gRNA突变体包括如下任一项所示的结构:5’-向导序列区-重复序列区-3’或5’-重复序列区-向导序列区-3’。
在一些实施方式中,根据本公开所述的gRNA突变体,其中,所述向导序列区具有靠近所述目标核酸的PAM序列的第一端,和远离所述PAM序列的第二端;
所述向导序列区具有m个核苷酸,任意一个所述突变核苷酸位于以所述第二端为起始的第n个核苷酸的位置,1≤n≤m,m、n取正整数;优选1≤n≤m/2,更优选1≤n≤m/3。
在一些实施方式中,根据本公开所述的gRNA突变体,其中,m取15-30的任意整数,优选15-25的任意整数;
可选地,任意一个所述突变核苷酸位于以所述第二端为起始的第1-12个核苷酸的位置,优选第2-10个核苷酸的位置,更优选第2-7个核苷酸的位置,更优选第2-6个核苷酸的位置。
在一些实施方式中,根据本公开所述的gRNA突变体,其中,所述突变核苷酸包含1-10个碱基的替换、缺失或插入;优选1-5个碱基的替换、缺失或插入,更优选1-3个碱基的替换、缺失或插入。
第四方面,本公开提供了一种分离的多核苷酸,其中,所述分离的多核苷酸编码如第三方面所述的gRNA突变体。
第五方面,本公开提供了一种重组表达载体,其中,所述重组表达包含如权利要求11所述的分离的多核苷酸。
第六方面,本公开提供了一种重组宿主细胞,其中,所述重组宿主细胞包含如权利要求12所述的重组表达载体。
第七方面,本公开提供了一种碱基编辑器,其中,所述碱基编辑器包括如下(i)-(ii)中的任一项,以及如下(iii)-(iv)中的任一项:
(i)如第三方面所述的gRNA突变体,
(ii)表达如(i)所述的gRNA突变体的多核苷酸、重组表达载体或重组宿主细胞;
(iii)融合蛋白,其中,所述融合蛋白包含与所述gRNA结合的第一结构域,和具有碱基修饰活性的第二结构域;
(iv)表达(iii)中所示融合蛋白的多核苷酸、重组表达载体或重组宿主细胞;
优选地,所述第一结构域为丧失或降低核酸酶活性的Cas蛋白突变体、同系物或多肽片段;
可选地,所述所述第一结构域选自如下的至少一种:丧失或降低核酸酶活性的Cas9蛋白突变体、同系物或多肽片段,丧失或降低核酸酶活性的Cas12a蛋白突变体、同系物或多肽片段;优选地,所述第一结构域为SpdCas9、SpnCas9、SadCas9、SanCas9或LbdCpf1。
在一些实施方式中,根据本公开所述的碱基编辑器,其中,所述第二结构域为具有脱氨酶活性的多肽;可选地,所述第二结构域为腺嘌呤脱氨酶,具有或部分具有其腺嘌呤脱氨酶活性的突变体、同系物或多肽片段;可选地,所述第二结构域为胞嘧啶脱氨酶,具有或部分具有其胞嘧啶脱氨酶活性的突变体、同系物或多肽片段;
可选地,所述第二结构域为具有腺嘌呤脱氨酶活性的酶,所述具有腺嘌呤脱氨酶活性的酶选自如下(c
(c
(c
可选地,所述第二结构域为具有胞嘧啶脱氨酶活性的酶,所述具有胞嘧啶脱氨酶活性的酶选自如下(d
(d
(d
第八方面,本公开提供了一种组合物,其中,所述组合物包含如第三方面所述的gRNA突变体,如第四方面所述的分离的多核苷酸,如第五方面所述的重组表达载体,如第六方面所述的重组宿主细胞,或如第七方面所述的碱基编辑器;
可选地,所述组合物还包括一种或多种药学上可接受的载体。
第九方面,本公开提供了根据第三方面所述的gRNA突变体,根据第四方面所述的分离的多核苷酸,根据第五方面所述的重组表达载体,或根据第六方面所述的重组宿主细胞,根据第七方面所述的碱基编辑器,或根据第八方面所述的组合物在如下(a)-(b)至少一种中的用途:
(a)作为或制备单碱基编辑的试剂或试剂盒;
(b)作为或制备基因治疗的药物。
第十方面,本公开提供了一种在细胞或受试者中进行基因编辑的方法,其中,所述方法包括使细胞或受试者与如下任一项接触:根据第三方面所述的gRNA突变体,根据第四方面所述的分离的多核苷酸,根据第五方面所述的重组表达载体,根据第六方面所述的重组宿主细胞,根据第七方面所述的碱基编辑器,或根据第八方面所述的组合物;
优选地,所述基因编辑为单个碱基的编辑;更优选地,所述基因编辑为1个碱基替换。
第十一方面,本公开提供了一种治疗或预防疾病的方法,其中,所述方法包括向受试者施用根据第三方面所述的gRNA突变体,根据第四方面所述的分离的多核苷酸,根据第五方面所述的重组表达载体,根据第六方面所述的重组宿主细胞,根据第七方面所述的碱基编辑器,或根据第八方面所述的组合物;
可选地,所述施用的方式包括:静脉内施用、腹膜内施用、冠状动脉内施用、动脉内施用、皮内施用、皮下施用、透皮递送、气管内施用、关节内施用、心室内施用、吸入、脑内、经脐、口服、眼内、肺部施用、导管注入、通过栓剂、病毒载体、脂质纳米材料以及直接注射到组织中。
在一些实施方式中,本公开提供的gRNA突变体的构建方法,通过对gRNA中的向导序列区进行突变,得到与目标核酸的靶序列不完全匹配的gRNA突变体。gRNA突变体应用于碱基编辑器中,能够显著缩小碱基编辑器的编辑窗口,提高了基因编辑的特异性,在基因治疗、药物筛选、动植物模型构建等方面具有广阔的应用前景。此外,以本公开的构建方法得到的gRNA突变体,还具有提高的碱基编辑效率,优化碱基编辑效果。
在一些实施方式中,本公开提供的gRNA突变体的构建方法,能够构建得到将碱基编辑器的编辑窗口精确到1个碱基的gRNA突变体,具有高的碱基编辑特异性和编辑效率,特别适合用于对由单碱基突变导致的遗传疾病的治疗。
在一些实施方式中,本公开提供的缩小碱基编辑器的编辑窗口的方法,通过使用本公开的gRNA突变体的构建方法对gRNA进行改造,能够在不应基因编辑效率的情况下,得到碱基编辑窗口明显缩小的碱基编辑器,在动物模型构建、功能基因组学研究、分子育种、临床医学、转化医学等领域应用前景广阔。
在一些实施方式中,本公开提供的碱基编辑器,其具有缩小的碱基编辑窗口,甚至可将编辑窗口缩小至1个碱基,能够高效、准确地用于纠正单个位点突变,为遗传疾病等的治疗提供了高效、准确的编辑工具。
附图说明
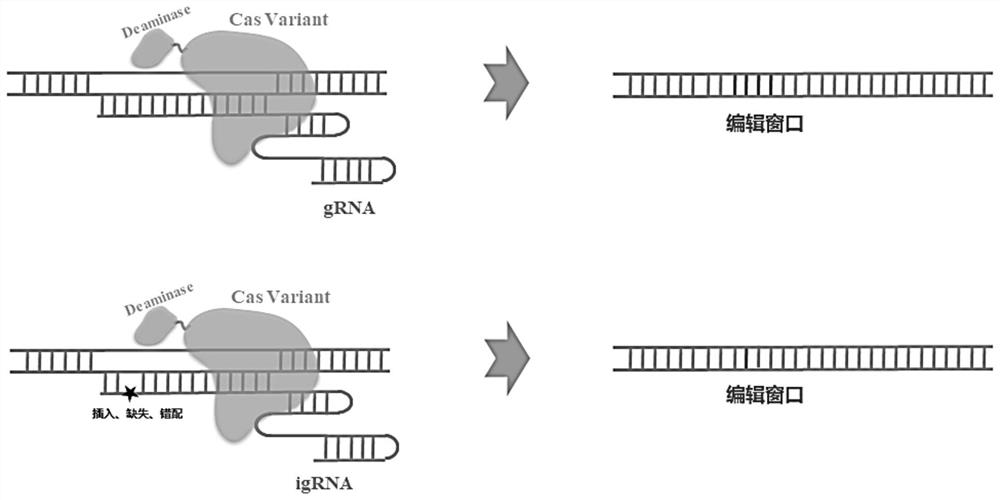
图1示出了本公开构建的包含gRNA突变体的碱基编辑器对1个碱基进行精准编辑的示意图。
图2示出了实施例3中通过改变PAM框,使碱基编辑器对任意位点碱基精准编辑的示意图。其中,Y轴为编辑效率。
具体实施方式
当在权利要求和/或说明书中与术语“包含”联用时,词语“一(a)”或“一(an)”可以指“一个”,但也可以指“一个或多个”、“至少一个”以及“一个或多于一个”。
如在权利要求和说明书中所使用的,词语“包含”、“具有”、“包括”或“含有”是指包括在内的或开放式的,并不排除额外的、未引述的元件或方法步骤。
在整个申请文件中,术语“约”表示:一个值包括测定该值所使用的装置或方法的误差的标准偏差。
虽然所公开的内容支持术语“或”的定义仅为替代物以及“和/或”,但除非明确表示仅为替代物或替代物之间相互排斥外,权利要求中的术语“或”是指“和/或”。
如本公开所使用的,术语“多肽”、“肽”和“蛋白质”在本文中互换地使用并且为任意长度的氨基酸聚合物。该聚合物可以是线形或分支的,它可以包含修饰的氨基酸,并且它可以由非氨基酸隔断。该术语也包括已经被修饰(例如,二硫键形成、糖基化、脂质化、乙酰化、磷酸化或任何其他操作,如以标记组分缀合)的氨基酸聚合物。
如本公开所使用的,术语“编辑窗口”是指向位于gRNA的向导序列区内,编辑效率大于所有可编辑碱基平均编辑效率的可编辑碱基所覆盖区域。示例性的,腺嘌呤碱基编辑器对向导序列区内存在的腺嘌呤(A)进行编辑,腺嘌呤(A)构成向导序列区内的可编辑碱基。若向导序列区内存在i(i≥1)个A,其中A
如本公开所使用的,术语“CRISPR”是指成簇、规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeats),其来自微生物的免疫系统。
如本公开所使用的,术语“Cas蛋白突变体”包括与野生型Cas蛋白相比核酸内切酶活性丧失或降低的Cas蛋白突变体、同系物或其多肽片段。
如本公开所使用的,术语“野生型Cas蛋白”是指CRISPR-associated蛋白,Cas蛋白与CRISPR序列共同构成CRISPR/Cas系统Cas蛋白具有与核酸酶相关的功能结构域,通过识别PAM(protospacer adjacent motif)在特定位置切割靶序列。
如本公开所使用的,术语“融合蛋白”是指包含来自至少两种不同蛋白质的蛋白质结构域的杂合多肽。融合蛋白可以通过最初编码单独蛋白质的两个或更多个基因的连接产生的嵌合蛋白。融合基因的翻译产生具有来源于每种原始蛋白质的功能特性的单一多肽。
如本公开所使用的,所述的“gRNA”又称为guide RNA或向导RNA,并且具有本领域技术人员通常理解的含义。一般而言,向导RNA可以包含同向(direct)重复序列和向导序列(guide sequence),或者基本上由或由同向重复序列和向导序列(在内源性CRISPR系统背景下也称为间隔序列(spacer))组成。gRNA在不同的CRISPR系统中,依据其所依赖的Cas蛋白的不同,可以包括crRNA和tracrRNA,也可以只含有crRNA。crRNA和tracrRNA可以经过人工改造融合形成single guide RNA(sgRNA)。
如本公开所使用的,原型间隔序列毗邻区(protospacer adjacent motifs,PAM)是指位于Cas蛋白识别的靶序列附近的序列,其可以位于靶序列的3’端(例如,CRISPR/Cpf1系统),也可以位于靶序列的5’端(例如,CRISPR/Cas9系统)。
如本公开所使用的,术语“靶序列”是指目标核酸中与gRNA互补或至少部分互补的核苷酸序列,在本公开中,“靶序列”与“靶核酸”可以互换地使用。
如本公开所使用的,术语“靶标链”(target strand)是指目标核酸中与gRNA杂交的核苷酸链;术语“非靶标链”(non-target strand)是指目标核酸中与gRNA不发生杂交配对的核苷酸链。
如本公开所使用的,术语“多核苷酸”指由核苷酸组成的聚合物。多核苷酸可以是单独片段的形式,也可以是更大的核苷酸序列结构的一个组成部分,其是从至少在数量或浓度上分离一次的核苷酸序列衍生而来的,能够通过标准分子生物学方法(例如,使用克隆载体)识别、操纵以及恢复序列及其组分核苷酸序列。当一个核苷酸序列通过一个DNA序列(即A、T、G、C)表示时,这也包括一个RNA序列(即A、U、G、C),其中“U”取代“T”。换句话说,“多核苷酸”指从其他核苷酸(单独的片段或整个片段)中去除的核苷酸聚合物,或者可以是一个较大核苷酸结构的组成部分或成分,如表达载体或多顺反子序列。多核苷酸包括DNA、RNA和cDNA序列。“重组多核苷酸”、“重组核酸分子”属于“多核苷酸”中的一种。
如本文所用,术语“杂交”是指使用核酸链通过碱基配对与互补链结合以形成杂交复合物的任何过程来配对互补核酸。
如本公开所使用的,术语“突变体”是指相对于“野生型”,或者“相比较的”多核苷酸或多肽,在一个或多个(例如,若干个)位置处包含改变(即,替换、插入和/或缺失的多核苷酸,其中,取代是指用不同的核苷酸置换占用一个位置的核苷酸。缺失是指去除占据某一位置的核苷酸。插入是指在邻接并且紧随占据位置的核苷酸之后添加核苷酸。在本公开中,核苷酸的改变也对应为碱基的改变,替换、插入或缺失的核苷酸对应为替换、插入或缺失的碱基。
如本公开所使用的,术语“突变核苷酸”或“核苷酸突变”,包括“替换、缺失或添加一个或多个核苷酸”。在本公开中,术语“突变”是指核苷酸序列的改变。
如本公开所使用的,术语“序列同一性”和“同一性百分比”指两个或更多个多核苷酸或多肽之间相同(即同一)的核苷酸或氨基酸的百分比。两个或更多个多核苷酸或多肽之间的序列同一性可通过以下方法测定:将多核苷酸或多肽的核苷酸或氨基酸序列对准且经对准的多核苷酸或多肽中含有相同核苷酸或氨基酸残基的位置数目进行评分,且将其与经对准的多核苷酸或多肽中含有不同核苷酸或氨基酸残基的位置数目进行比较。多核苷酸可例如通过含有不同核苷酸(即取代或突变)或缺失核苷酸(即一个或两个多核苷酸中的核苷酸插入或核苷酸缺失)而在一个位置处不同。多肽可例如通过含有不同氨基酸(即取代或突变)或缺失氨基酸(即一个或两个多肽中的氨基酸插入或氨基酸缺失)而在一个位置处不同。序列同一性可通过用含有相同核苷酸或氨基酸残基的位置数目除以多核苷酸或多肽中氨基酸残基的总数来计算。举例而言,可通过用含有相同核苷酸或氨基酸残基的位置数目除以多核苷酸或多肽中核苷酸或氨基酸残基的总数且乘以100来计算同一性百分比。
示例性的,当使用序列比较算法或通过目视检查测量以最大的对应性进行比较和比对时,两个或多个序列或子序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%核苷酸的“序列同一性”或“同一性百分比”。在某些实施方式中,所述序列在任一或两个相比较的生物聚合物(例如,多核苷酸)的整个长度上基本相同。
如本公开所使用的,术语“个体”和“受试者”可互换地使用,是指哺乳动物。哺乳动物包括但不限于驯化动物(例如,奶牛、绵羊、猫、犬和马)、灵长类(例如,人和非人灵长类如猴)、兔和啮齿类(例如,小鼠和大鼠)。特别地,个体是人。
如本公开所使用的,术语“载体”指的是DNA构建体,其含有与合适的控制序列可操作地连接的DNA序列,从而在合适的宿主中表达目的基因。
如本公开所使用的,术语“重组表达载体”指用于表达例如编码所需多肽的多核苷酸的DNA结构。重组表达载体可包括,例如包含i)对基因表达具有调控作用的遗传元素的集合,例如启动子和增强子;ii)转录成mRNA并翻译成蛋白质的结构或编码序列;以及iii)适当的转录和翻译起始和终止序列的转录亚单位。重组表达载体以任何合适的方式构建。载体的性质并不重要,并可以使用任何载体,包括质粒、病毒、噬菌体和转座子。用于本公开的可能载体包括但不限于染色体、非染色体和合成DNA序列,例如病毒质粒、细菌质粒、噬菌体DNA、酵母质粒以及从质粒和噬菌体DNA的组合中衍生的载体,来自如慢病毒、腺相关病毒、逆转录病毒、牛痘、腺病毒、鸡痘、杆状病毒、SV40和伪狂犬病等病毒的DNA。
如本公开所使用的,术语“宿主细胞”指已经向其中引入外源多核苷酸的细胞,包括这类细胞的子代。宿主细胞包括“转化体”和“转化的细胞”,这包括原代转化的细胞和从其衍生的子代。宿主细胞是可以用来产生本发明抗体分子的任何类型的细胞系统,包括真核细胞,例如,哺乳动物细胞、昆虫细胞、酵母细胞;和原核细胞,例如,大肠杆菌细胞。宿主细胞包括培养的细胞,也包括转基因动物、转基因植物或培养的植物组织或动物组织内部的细胞。
如本公开所使用的,“治疗”是指:在罹患疾病之后,使受试者接触(例如给药)本发明的环状RNA、线状RNA、重组核酸分子、重组表达载体、组合物,从而与不接触时相比使该疾病的症状减轻,并不意味着必需完全抑制疾病的症状。罹患疾病是指:身体出现了疾病症状。
如本公开所使用的,“预防”是指:在罹患疾病之前,通过使受试者接触(例如给药)本发明的环状RNA、线状RNA、重组核酸分子、重组表达载体、组合物等,从而与不接触时相比减轻罹患疾病后的症状,并不意味着必需完全抑制患病。
除非另外定义或由背景清楚指示,否则在本公开中的全部技术与科学术语具有如本公开所属领域的普通技术人员通常理解的相同含义。
本公开提供了gRNA突变体的构建方法,其包括如下步骤:
突变步骤:对gRNA中与目标核酸的靶序列杂交的向导序列区进行突变,使所述向导序列区的一个或多个位置处具有一个或多个碱基的替换、缺失或插入,形成包含突变核苷酸的突变序列区;
筛选步骤:筛选与未突变的gRNA相比,用于碱基编辑器的编辑窗口缩小的突变体,得到所述gRNA突变体。
本公开的gRNA突变体的构建方法,通过对gRNA中的向导序列区进行突变,得到与目标核酸的靶序列不完全互补配对的gRNA突变体,gRNA突变体又可称为不完美gRNA(Imperfect guide-RNA,igRNA)。与gRNA相比,igRNA具有缩小的用于碱基编辑器的编辑窗口,能够有效提高碱基编辑特异性,并一定程度上提高了碱基编辑效率,解决了目前碱基编辑器的编辑窗口内存在的碱基编辑位点多,难以针对特定位点实现高特异性的基因编辑的缺陷。
在一些优选地实施方式中,筛选步骤包括:筛选用于碱基编辑器的编辑窗口为1个碱基的突变体,得到所述gRNA突变体。将gRNA突变体应用于碱基编辑器后,能够实现对特定单个碱基的精确修饰,为疾病的基因治疗、动物模型构建、分子育种等提供了高特异性的碱基编辑工具,特别适合于对遗传疾病中致病性单碱基突变的纠正。
在一些实施方式中,所述向导序列区具有靠近所述目标核酸的PAM序列的第一端,和远离所述PAM序列的第二端,向导序列区具有m个核苷酸,任意一个所述突变核苷酸位于以所述第二端为起始的第n个核苷酸的位置,1≤n≤m,m、n取正整数。
本公开发现,gRNA碱基突变的位置对于编辑窗口的大小具有重要影响,其具体受突变核苷酸与目标核酸上PAM序列之间所间隔的核苷酸的个数影响。
在一些优选地实施方式中,2≤n≤m/2。在一些更为优选的实施方式中,2≤n≤m/3。通过在上述位置处引入突变核苷酸,能够有效缩小gRNA突变体应用于碱基编辑器的编辑窗口,为gRNA的改造提供了明确、可实施的改造位置。
在本公开中,对于向导序列区的核苷酸数量不进行具体限定,其具体数量可结合碱基编辑器的Cas蛋白的种类以及所述结合的靶序列进行设计。
在一些可选地实施方式中,向导序列区的核苷酸数量m取15-30的任意整数。在一些优选的实施方式中,向导序列区的核苷酸数量m取15-25的任意整数。在一些优选的实施方式中,gRNA突变体应用于CRISPR/Cas9系统,向导序列区的核苷酸数量m取19-21的任意整数。进一步地,任意一个所述突变核苷酸位于以所述第二端为起始的第1-12个核苷酸的位置,优选第2-10个核苷酸的位置,更优选第2-6个核苷酸的位置,更优选第2-6个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有20个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-10个核苷酸的位置,优选第2-10个核苷酸的位置,更优选第2-6个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8、9或10个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有21个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-10个核苷酸的位置,优选第2-10个核苷酸的位置,更优选第2-6个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8、9或10个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有22个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-11个核苷酸的位置,优选第2-11个核苷酸的位置,更优选第2-6个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8、9、10或11个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有23个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-11个核苷酸的位置,优选第2-11个核苷酸的位置,更优选第2-6个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8、9、10或11个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有24个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-12个核苷酸的位置,优选第2-12个核苷酸的位置,更优选第2-7个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8、9、10、11或12个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有25个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-12个核苷酸的位置,优选第2-12个核苷酸的位置,更优选第2-7个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8、9、10、11或12个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有19个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-9个核苷酸的位置,优选第2-9个核苷酸的位置,更优选第2-5个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8或9个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有18个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-9个核苷酸的位置,优选第2-9个核苷酸的位置,更优选第2-5个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7、8或9个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有17个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-8个核苷酸的位置,优选第2-8个核苷酸的位置,更优选第2-5个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7或8个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有16个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-8个核苷酸的位置,优选第2-8个核苷酸的位置,更优选第2-5个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6、7或8个核苷酸的位置。
示例性的,gRNA突变体应用于CRISPR/Cas9系统,向导序列区具有15个核苷酸,对应地任意一个突变核苷酸的位置位于以第二端为起始的第1-7个核苷酸的位置,优选第2-7个核苷酸的位置,更优选第2-4个核苷酸的位置。示例性地,任意一个突变核苷酸的位置位于以第二端为起始的第1、2、3、4、5、6或7个核苷酸的位置。
在另外一些实施方式中,m、n还可以有其它的取值,本公开对此不进行穷举。
在一些实施方式中,突变核苷酸包含1-10个碱基的替换、缺失或插入,优选1-5个碱基的替换、缺失或插入,更优选1-3个碱基的替换、缺失或插入。示例性地,突变核苷酸包含1、2、3、4、5、6、7、8、9、10个碱基的替换、缺失或插入。本公开发现,突变核苷酸包含的碱基的个数直接影响gRNA突变体用于碱基编辑的特异性和效率,当突变碱基的个数在上述范围内时,可得到高特异性、编辑窗口最优缩小为1个碱基的gRNA突变体。
需要说明的是,突变的碱基可以位于向导序列区以第二端为起始的第1个核苷酸至第n个核苷酸的任意地一个或多个位置,对于任意一个位置处的碱基的突变方式,均可独立地选自替换、缺失、插入中的任一种,只要使突变核苷酸所包含的突变碱基的总数量落入本公开所提供的范围内,即可优化gRNA用于碱基编辑的特异性。
进一步地,突变核苷酸所包含的突变碱基的数量,还依据碱基编辑器的类型决定:
在一些优选地实施方式中,碱基编辑器为腺嘌呤碱基编辑器(adenine baseeditors,ABEs),用于腺嘌呤碱基编辑器的gRNA突变体的突变核苷酸包含1-5个碱基的替换、缺失或插入,优选1-3个碱基的替换、缺失或插入,更优选1个碱基的替换、缺失或插入。示例性地,对于用于腺嘌呤碱基编辑器的gRNA突变体,其突变核苷酸包含1、2、3、4、5个等等的碱基的替换、缺失或插入。本公开在实验中发现,当将针对某些基因的gRNA突变体中突变碱基的个数设置为1时,得到了能够针对特定1个位点处的腺嘌呤进行突变的腺嘌呤碱基编辑器。
在一些优选地实施方式中,碱基编辑器为胞嘧啶碱基编辑器(cytosine baseeditors,CBEs),用于胞嘧啶碱基编辑器的gRNA突变体的突变核苷酸包含1-5个碱基的替换、缺失或插入,优选1-3个碱基的替换、缺失或插入,更优选2-3个碱基的替换、缺失或插入。示例性地,对于用于胞嘧啶碱基编辑器的gRNA突变体,其突变核苷酸包含1、2、3、4、5个等等的碱基的替换、缺失或插入。本公开在实验中发现,当将针对某些基因的gRNA突变体中突变碱基的个数设置为2或3时,得到了能够针对特定1个位点处的胞嘧啶进行编辑的胞嘧啶碱基编辑器。
本公开提供了gRNA突变体,其根据本公开所述的构建方法得到。gRNA突变体用于碱基编辑器,能够有效高碱基编辑的特异性,缩小碱基编辑器的编辑窗口,最终能够实现对特定地单个碱基的高效、特异性编辑,为疾病治疗、动植物模型的构建提供了积极、有效的基因编辑策略。
进一步地,gRNA突变体中还包括用于结合Cas蛋白的重复序列区。重复序列区可折叠形成特定结构(例如,茎环结构),供Cas蛋白识别。Cas蛋白与gRNA突变体结合后,通过识别目标核酸的靶序列附近的PAM(protospacer adjacent motif)序列对靶序列进行识别。gRNA突变体的突变序列区与靶序列杂交后,目标核酸的双链打开形成R-loop区,得到Cas蛋白-gRNA-靶序列的三元复合物,并进一步对R-loop区的单链核酸链进行编辑。根据gRNA突变体应用的CRISPR/Cas系统的不同,重复序列区可以连接于向导序列区的5’端或3’端。在一些可选的实施方式中,gRNA突变体应用于CRISPR/Cas9系统,gRNA突变体包括5’-向导序列区-重复序列区-3’的核苷酸序列。在一些可选的实施方式中,gRNA突变体应用于CRISPR/Cpf1系统,gRNA突变体包括5’-重复序列区-向导序列区-3’的核苷酸序列。
在本公开中,对于gRNA突变体的突变序列区的核苷酸序列不进行具体限定,只要其突变区序列能够使gRNA突变体与靶序列杂交形成双链,将目标核酸打开形成R-loop区;并且能够缩小gRNA突变体用于碱基编辑器的编辑窗口即可。
在一些可选地实施方式中,gRNA突变体用于将HIRA基因的特定位点的碱基C突变为碱基T,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:1所示。gRNA突变体(以下简称为igRNA)的突变序列区的核苷酸序列如SEQ ID NO:2-5任一项所示。其中,SEQ ID NO:2所示的序列对应SEQ ID NO:1所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQID NO:3所示的序列对应SEQ ID NO:1所示序列的第5个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:4所示的序列对应SEQ ID NO:1所示序列的第3个核苷酸的位置处具有1个碱基的缺失,SEQ ID NO:5所示的序列对应SEQ ID NO:1所示序列的第3个核苷酸的位置处具有2个碱基的插入。与向导序列区为SEQ ID NO:1所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对HIRA基因进行编辑时,具有普遍性提高的针对C6位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将DNMT3B基因的特定位点的碱基C突变为碱基T,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:6所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:7-10任一项所示。其中,SEQ ID NO:7所示的序列对应SEQ ID NO:6所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:8所示的序列对应SEQ ID NO:1所示序列的第3个和第4个核苷酸的位置处具有2个碱基的替换,SEQID NO:9所示的序列对应SEQ ID NO:6所示序列的第2个核苷酸的位置处具有1个碱基的缺失,SEQ ID NO:10所示的序列对应SEQ ID NO:6所示序列的第3个核苷酸的位置处具有2个碱基的插入。与向导序列区为SEQ ID NO:6所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对DNMT3B基因进行编辑时,具有普遍性提高的针对C8位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将RNF2基因的特定位点的碱基C突变为碱基T,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:11所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:12-14任一项所示。其中,SEQ ID NO:12所示的序列对应SEQ ID NO:11所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:13所示的序列对应SEQ ID NO:11所示序列的第2个核苷酸的位置处具有1个碱基的缺失,SEQ IDNO:14所示的序列对应SEQ ID NO:11所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:11所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对RNF2基因进行编辑时,具有普遍性提高的针对C6位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将NSD1基因的特定位点的碱基C突变为碱基T,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:19所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:20-22任一项所示。其中,SEQ ID NO:20所示的序列对应SEQ ID NO:19所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:21所示的序列对应SEQ ID NO:19所示序列的第2个和第3个核苷酸的位置处具有2个碱基的缺失,SEQ ID NO:22所示的序列对应SEQ ID NO:19所示序列的第2个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:19所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对NSD1基因进行编辑时,具有普遍性提高的针对C6位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将PSMB2基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:23所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:24-27任一项所示。其中,SEQ ID NO:24所示的序列对应SEQ ID NO:23所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:25所示的序列对应SEQ ID NO:23所示序列的第5个核苷酸的位置处具有1个碱基的替换,SEQ IDNO:26所示的序列对应SEQ ID NO:23所示序列的第3个核苷酸的位置处具有1个碱基的缺失,SEQ ID NO:27所示的序列对应SEQ ID NO:23所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:23所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对PSMB2基因进行编辑时,具有普遍性提高的针对A5位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将ABCA3基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:28所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:29-31任一项所示。其中,SEQ ID NO:29所示的序列对应SEQ ID NO:28所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:30所示的序列对应SEQ ID NO:28所示序列的第3个核苷酸的位置处具有1个碱基的缺失,SEQ IDNO:31所示的序列对应SEQ ID NO:28所示序列的第2个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:28所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对ABCA3基因进行编辑时,具有普遍性提高的针对A5位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将EMX1-SITE3基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:32所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:33-36任一项所示。其中,SEQ ID NO:33所示的序列对应SEQ ID NO:32所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:34所示的序列对应SEQ ID NO:32所示序列的第4个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:35所示的序列对应SEQ ID NO:32所示序列的第2个核苷酸的位置处具有1个碱基的缺失,SEQ ID NO:36所示的序列对应SEQ ID NO:32所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:32所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对EMX1-SITE3基因进行编辑时,具有普遍性提高的针对A6位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将VISTAHS267基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:37所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:38-41任一项所示。其中,SEQ ID NO:38所示的序列对应SEQ ID NO:37所示序列的第2个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:39所示的序列对应SEQ ID NO:37所示序列的第4个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:40所示的序列对应SEQ ID NO:37所示序列的第4个核苷酸的位置处具有1个碱基的缺失,SEQ ID NO:41所示的序列对应SEQ ID NO:37所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:37所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对VISTA HS267基因进行编辑时,具有普遍性提高的针对A5位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将SNCA基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:42所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:43-46任一项所示。其中,SEQ ID NO:43所示的序列对应SEQ ID NO:42所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:44所示的序列对应SEQ ID NO:42所示序列的第5个核苷酸的位置处具有1个碱基的替换,SEQ IDNO:45所示的序列对应SEQ ID NO:42所示序列的第3个核苷酸的位置处具有1个碱基的缺失,SEQ ID NO:46所示的序列对应SEQ ID NO:42所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:42所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对SNCA基因进行编辑时,具有普遍性提高的针对A5位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将ANO5基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:47所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:48-51任一项所示。其中,SEQ ID NO:48所示的序列对应SEQ ID NO:47所示序列的第2个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:49所示的序列对应SEQ ID NO:47所示序列的第4个核苷酸的位置处具有1个碱基的替换,SEQ IDNO:50所示的序列对应SEQ ID NO:47所示序列的第2个核苷酸的位置处具有1个碱基的缺失,SEQ ID NO:51所示的序列对应SEQ ID NO:47所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:47所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对ANO5基因进行编辑时,具有普遍性提高的针对A7位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将KCNQ2基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:52所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:53-55任一项所示。其中,SEQ ID NO:53所示的序列对应SEQ ID NO:52所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:54所示的序列对应SEQ ID NO:52所示序列的第3个核苷酸的位置处具有1个碱基的缺失,SEQ IDNO:55所示的序列对应SEQ ID NO:52所示序列的第2个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:52所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对KCNQ2基因进行编辑时,具有普遍性提高的针对A5位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将NOTCH2基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:56所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:57-59任一项所示。其中,SEQ ID NO:57所示的序列对应SEQ ID NO:56所示序列的第6个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:58所示的序列对应SEQ ID NO:56所示序列的第4个核苷酸的位置处具有1个碱基的缺失,SEQ IDNO:59所示的序列对应SEQ ID NO:56所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:56所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对NOTCH2基因进行编辑时,具有普遍性提高的针对A5位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将GFI1基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:60所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:61-63任一项所示。其中,SEQ ID NO:61所示的序列对应SEQ ID NO:60所示序列的第2个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:62所示的序列对应SEQ ID NO:60所示序列的第4个核苷酸的位置处具有1个碱基的缺失,SEQ IDNO:63所示的序列对应SEQ ID NO:60所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:60所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对GFI1基因进行编辑时,具有普遍性提高的针对A5位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将CFAP61基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:71所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:72-75任一项所示。其中,SEQ ID NO:72所示的序列对应SEQ ID NO:71所示序列的第3个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:73所示的序列对应SEQ ID NO:71所示序列的第2-3个核苷酸的位置处具有2个碱基的替换,SEQID NO:74所示的序列对应SEQ ID NO:71所示序列的第6个核苷酸的位置处具有1个碱基的确实,SEQ ID NO:75所示的序列对应SEQ ID NO:71所示序列的第3个核苷酸的位置处具有1个碱基的插入。与向导序列区为SEQ ID NO:71所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对CFAP61基因进行编辑时,具有普遍性提高的针对A11位点的碱基编辑特异性,且编辑效率也获得提升。
在一些可选地实施方式中,gRNA突变体用于将Query_55451基因的特定位点的碱基A突变为碱基G,未突变的gRNA的向导序列区的核苷酸序列如SEQ ID NO:76所示。gRNA突变体的突变序列区的核苷酸序列如SEQ ID NO:77-80任一项所示。其中,SEQ ID NO:77所示的序列对应SEQ ID NO:76所示序列的第2-3个核苷酸的位置处具有2个碱基的替换,SEQ IDNO:78所示的序列对应SEQ ID NO:76所示序列的第2个核苷酸的位置处具有1个碱基的替换,SEQ ID NO:79所示的序列对应SEQ ID NO:76所示序列的第3-4个核苷酸的位置处具有2个碱基的缺失,SEQ ID NO:80所示的序列对应SEQ ID NO:76所示序列的第3个核苷酸的位置处具有2个碱基的插入。与向导序列区为SEQ ID NO:76所示序列的gRNA相比,不同突变方式的igRNA应用于胞嘧啶碱基编辑器中对Query_55451基因进行编辑时,具有普遍性提高的针对A9位点的碱基编辑特异性,且编辑效率也获得提升。
据此可知,本公开的构建方法得到的gRNA突变体,能够通用性地提高胞嘧啶碱基编辑器、腺嘌呤碱基编辑器的编辑特异性,为缩小碱基编辑器的编辑窗口提供了一种通用、有效的优化策略。
在本公开中,对于gRNA突变体的重复序列区的核苷酸序列不进行具体限定,只要其突变区序列能够使gRNA突变体与Cas蛋白结合,引导Cas蛋白靶向编辑目标核酸的靶序列即可。
在一些可选地实施方式中,gRNA突变体应用于CRISPR/spCas9系统,gRNA突变体的重复序列区包含如下(v)-(vi)任一项所示的核苷酸序列:
(v)如SEQ ID NO:81所示的核苷酸序列;
(vi)与SEQ ID NO:81所示的核苷酸序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%的序列同一性的序列。
在一些可选地实施方式中,gRNA突变体应用于CRISPR/saCas9系统,gRNA突变体的重复序列区包含如下(vii)-(viii)任一项所示的核苷酸序列:
(vii)如SEQ ID NO:82所示的核苷酸序列;
(viii)与SEQ ID NO:82所示的核苷酸序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%的序列同一性的序列。
在一些实施方式中,本公开提供了分离的多核苷酸,其编码根据本公开所述的gRNA突变体,能够用于gRNA突变体的大量制备。
在一些实施方式中,本公开提供了重组表达载体,其包含根据本公开所述的分离的多核苷酸。其中,连接多核苷酸的载体可以是本领域常用的各类载体,适于体内或体外的细胞转导。
在一些实施方式中,本公开提供了重组宿主细胞,包含根据本公开所述的重组表达载体。通过重组表达载体在重组宿主细胞内的复制、表达,能够得到根据本公开所述的gRNA突变体。
本公开提供的缩小碱基编辑器的方法,包括使用根据本公开所述的gRNA突变体的构建方法,构建目标的gRNA突变体。
本公开提供的缩小碱基编辑器的方法,能够在不影响碱基编辑效率的情况下,提高碱基编辑特异性,有效缩小碱基编辑窗口;且对编辑窗口的缩小具有通用性,适合对胞嘧啶碱基编辑器、腺嘌呤碱基编辑器进行优化,拓展碱基编辑器在动物模型构建、功能基因组学研究、分子育种、临床医学、转化医学等领域的应用范围。
在一些优选地实施方式中,缩小碱基编辑器的方法将碱基编辑器的编辑窗口缩小为1个碱基,由于多数遗传性疾病是由单个碱基突变导致的,应用本公开的方法缩小碱基编辑器的窗口至1个碱基,能够针对性纠正致病性点突变,并且避免在基因编辑过程中发生不必要的碱基编辑,为遗传疾病的治疗提供了一种积极、有效的治疗工具。
本公开提供的碱基编辑器,包括如下(i)-(ii)中的任一项,以及如下(iii)-(iv)中的任一项:
(i)gRNA突变体,
(ii)表达gRNA突变体的多核苷酸、重组表达载体或重组宿主细胞;
(iii)融合蛋白,其中,所述融合蛋白包含与所述gRNA结合的第一结构域,和具有碱基修饰活性的第二结构域;
(iv)表达(iii)中所示融合蛋白的多核苷酸、重组表达载体或重组宿主细胞。
对于表达gRNA突变体的多核苷酸,可以是转录gRNA突变体的DNA核酸分子,或包含gRNA突变体的RNA核酸分子。对于重组表达载体,可以是由编码gRNA突变体的核苷酸序列与任意类型的载体重组连接形成,例如,重组表达载体是包含编码gRNA突变体的核苷酸序列的病毒载体等等。
对于表达融合蛋白的多核苷酸,可以是翻译产生融合蛋白的RNA核酸分子,或转录产生上述RNA核酸分子的DNA核酸分子。对于重组表达载体,可以是由编码融合蛋白的核苷酸序列与任意类型的载体重组连接形成,例如,重组表达载体是包含编码融合蛋白的开放阅读框的病毒载体等等。
在本公开中,融合蛋白的第一结构域和第二结构域可以直接相连,也可以是通过连接肽间接相连。
对于第一结构域,是具有丧失或降低核酸酶活性的Cas蛋白。在一些实施方式中,第一结构域是丧失核酸酶活性的Cas蛋白突变体、同系物或其多肽片段,融合蛋白通过第一结构域与gRNA结合,并识别目标核酸的PAM序列附近的靶序列,形成融合蛋白-gRNA突变体-靶序列的三元复合物。由于第一结构域丧失Cas蛋白的核酸酶活性,在三元复合物形成后,不会对靶标链或非靶标链进行切割,仅依靠第二结构域的碱基修饰活性对靶序列的编辑窗口内的碱基进行突变。在一些实施方式中,第一结构域是核酸酶活性降低的Cas蛋白突变体、同系物或其多肽片段,在三元复合物形成后,第一结构域对靶标链或非靶标链中的一条链进行切割。在一些实施方式中,第一结构域是PAM框拓展的Cas蛋白突变体、同系物或其多肽片段,示例性的,图2示出了一种腺嘌呤碱基编辑器,在以NGG为PAM框,使用未突变的gRNA时,碱基A1与碱基A2都位于腺嘌呤碱基编辑器的编辑窗口内;若使用本公开提供的gRNA突变体,其编辑窗口缩小到A2碱基,但无法实现对A1碱基的特异性编辑。此时,使用PAM框拓展的Cas蛋白突变体作为第一结构域,使碱基编辑器的编辑窗口由A2移动为A1,从而实现对A1碱基的特异性编辑。
在本公开中,Cas蛋白具有多种不同选择,示例性地,Cas蛋白选自Cas9蛋白(WP_032462936,WP_165886160,WP_002460848,WP_002807152)、Cas12a蛋白(也即Cpf1蛋白,PDB:6KLB_A,PDB:6KLB_D,UniProtKB/Swiss-Prot:U2UMQ6)、Cas12b蛋白(WP_217021837,WP_163299037,WP_027726362)、Cas13a蛋白(UniProtKB/Swiss-Prot:P0DPB8)等等。
在一些具体的实施方式中,Cas蛋白为化脓性链球菌Cas9(SpCas9)、金黄色葡萄球菌Cas9(SaCas9)、毛螺菌科Cpf1(LbCpf1)等。
在一些可选地实施方式中,第一结构域为SpCas9蛋白突变体。进一步地,SpCas9蛋白突变体选自核酸酶活性丧失的SpCas9蛋白突变体或核酸酶活性降低的SpCas9蛋白突变体(SpnCas9)。示例性地,SpdCas9的氨基酸序列如SEQ ID NO:85所示,或与SEQ ID NO:85所示序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%的序列同一性的序列。SpnCas9的氨基酸序列如SEQ ID NO:86所示,或与SEQ ID NO:86所示序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%的序列同一性的序列。
在一些可选地实施方式中,第一结构域为SaCas9蛋白突变体。进一步地,SaCas9蛋白突变体选自核酸酶活性丧失的SaCas9蛋白突变体(SadCas9)或核酸酶活性降低的SaCas9蛋白突变体(SanCas9)。示例性地,SadCas9的氨基酸序列如SEQ ID NO:87所示,或与SEQ IDNO:87所示序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%的序列同一性的序列。SanCas9的氨基酸序列如SEQ ID NO:88所示,或与SEQ ID NO:88所示序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%的序列同一性的序列。
在一些可选地实施方式中,第一结构域为LbCpf1蛋白突变体。进一步地,LbCpf1蛋白突变体为核酸酶活性丧失的LbCpf1蛋白突变体(LbdCpf1)。示例性地,LbdCpf1的氨基酸序列如SEQ ID NO:89所示,或与SEQ ID NO:89所示序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%的序列同一性的序列。
在一些实施方式中,通过选择合适的PAM序列,以及识别该PAM序列的Cas蛋白,本公开中的碱基编辑器能够实现对任意位点的1个碱基的编辑。
对于第二结构域,其具有碱基修饰活性。在一些实施方式中,碱基修饰活性为脱氨酶活性,第二结构域为脱氨酶,具有或至少部分具有脱氨酶活性的突变体、同系物、多肽。在形成融合蛋白-gRNA突变体-靶序列的三元复合物,第二结构域通过脱去编辑窗口内的碱基的氨基,对编辑窗口内的碱基进行突变。
在一些实施方式中,第二结构域为腺嘌呤脱氨酶,具有或部分具有其腺嘌呤脱氨酶活性的突变体、同系物或多肽片段。腺嘌呤脱氨酶会将编辑窗口内的A突变为G。由于本公开的碱基编辑器具有最优为1个碱基的编辑窗口,因此,利用本公开的碱基编辑器可以实现特定的1个A向G的碱基突变。
本公开对腺嘌呤脱氨酶或其突变体、多肽、同系物的氨基酸序列不进行具体限定,只要其能够具有腺嘌呤脱氨酶的酶活性,实现A向G的突变即可。
在一些可选地实施方式中,第二结构域为具有腺嘌呤脱氨酶活性的酶,其选自如下(c
(c
(c
在一些可选地实施方式中,第二结构域为大肠杆菌tRNA腺苷脱氨酶TadA,或保持其腺苷脱氨酶活性的突变体、同系物或多肽。示例性的,TadA的氨基酸序列可参见NP_417054。
在一些可选地实施方式中,第二结构域为具有腺苷脱氨酶活性的TadA的突变体TadA*。示例性地,TadA*的氨基酸序列如SEQ ID NO:83或SEQ ID NO:84所示。大肠杆菌来源的TadA或TadA的突变体TadA*通过使编辑窗口内的腺嘌呤脱氨,能够实现由腺嘌呤A向鸟嘌呤G的突变。
在一些实施方式中,第二结构域为胞嘧啶脱氨酶,具有或部分具有其胞嘧啶脱氨酶活性的突变体、同系物或多肽片段。胞嘧啶脱氨酶会将编辑窗口内的C突变为T。由于本公开的碱基编辑器具有最优为1个碱基的编辑窗口,因此,利用本公开的碱基编辑器可以实现对特定的1个C向T的碱基突变。
本公开对胞嘧啶脱氨酶或其突变体、多肽、同系物的氨基酸序列不进行具体限定,只要其能够具有胞嘧啶脱氨酶的酶活性,实现C向T的突变即可。
在一些可选地实施方式中,第二结构域为具有腺嘌呤脱氨酶活性的酶,其选自如下(d
(d
(d
示例性地,第二结构域可选自如下任意一种的胞嘧啶脱氨酶,或其突变体、同系物或多肽:AID(氨基酸序列可参见NP_001317272,NP_065712),APOBEC3A(如:AKE33285,AEH96362,ACH92046,CAK54680),APOBEC3G(如:人APOBEC3G,氨基酸序列可参见NP_068594,NP_001336365,NP_001336366,NP_001336367),APOBEC1(如:APOBEC1,氨基酸序列可参见NP_001127863,NP_112436;人APOBEC1,氨基酸序列可参见NP_001291495,NP_001635,NP_005880),CDA1(如:七鳃鳗CDA1,ABO15149,ABO15150)。
本公开提供的组合物包括根据本公开所述的gRNA突变体、分离的多核苷酸、重组表达载体、重组宿主细胞或碱基编辑器。本公开的碱基编辑器由于包含了优化的gRNA,其对目标核酸进行基因编辑的特异性高,能够实现针对1个碱基的高效编辑,在基因治疗、动植物模型构建等方面具有重要应用潜力。
在一些实施方式中,组合物中还包括一种或多种药学上可接受的载体。在本公开中,使用组合物的目的在于促进针对生物体的给药,有利于活性成分的吸收,进而发挥生物活性。本发明的组合物可以通过任何形式给药,包括注射(动脉内、静脉内、肌肉内、腹膜内、皮下)、粘膜、口服(口服固体制剂、口服液体制剂)、直肠、吸入、植入、局部(例如眼部)给药等。口服固体制剂的非限制性实例包括但不限于散剂、胶囊剂、锭剂、颗粒剂、片剂等。口服或粘膜给药的液体制剂的非限制性实例包括但不限于混悬剂、酊剂、酏剂、溶液剂等。局部给药制剂的非限制性实例包括但不限于乳剂、凝胶剂、软膏剂、乳膏剂、贴剂、糊剂、泡沫剂、洗剂、滴剂或血清制剂。胃肠外给药制剂的非限制性实例包括但不限于注射用溶液剂、注射用干粉剂、注射用悬浮液、注射用乳剂等。本发明的组合物还可以制成控制释放或延迟释放剂型(例如脂质体或微球)。
在本公开中,施用途经能够以任何适用的方式进行变化或调整,以满足药物的性质、患者和医务人员的便利以及其它相关因素的需求。
在一些实施方式中,本公开提供了gRNA突变体、碱基编辑器作为或制备单碱基编辑的试剂或试剂盒的用途。本公开的gRNA突变体、碱基编辑器能够实现更可控、更特异性地1个碱基的编辑,克服了传统碱基编辑器由于编辑窗口包含多个碱基编辑位点,无法针对特定1个碱基进行编辑的缺陷,特别适合用于作为基因治疗、动植物模型构建等领域中的单碱基编辑工具。
在一些实施方式中,本公开提供了gRNA突变体、碱基编辑器作为或制备基因治疗的药物。由于gRNA突变体、碱基编辑器具有缩小的编辑窗口,能够实现针对1个碱基的特定编辑,避免了由于额外基因编辑导致的副作用,适合作为疾病的基因治疗药物,具有重要医药应用前景。
本公开的其他目的、特征和优点将从以下详细描述中变得明显。但是,应当理解的是,详细描述和具体实施例(虽然表示本公开的具体实施方式)仅为解释性目的而给出,因为在阅读该详细说明后,在本公开的精神和范围内所作出的各种改变和修饰,对于本领域技术人员来说将变得显而易见。
以下结合具体的实施例进行说明,其中试剂以及样品等均有商品供应或以别的途径能为公众所得,它们仅作举例,对本发明不是唯一的,可分别用其它适合的工具和生物材料来代替。所涉及的实验操作可按照《分子克隆实验指南(第三版)》(科学出版社,2002)中所述条件和方法进行,并可按照商业化的酶、试剂盒的厂商说明来进行。其它未详细描述的试验方法如无特殊说明,均为本领域的技术人员所熟知的常规方法。下述实施例所述测序、基因合成于苏州金唯智生物科技有限公司完成。
BE4max(Addgene:112093)、hyBE4max(Addgene:157942)碱基编辑器质粒
NG-ABEmax(Addgene:124163)碱基编辑器质粒SaABEmax(Addgene:119814)
实施例1CBE gRNA改造编辑
在哺乳动物细胞中,不同的编辑位点利用igRNA进行胞嘧啶碱基编辑器编辑,其单窗口的碱基编辑占比相对于利用未改造的gRNA均有所提高,最高可以提高38.50倍。
实验过程:将HEK293T或者Hela细胞按5×10
实验结果:选择NSD1、DNMT3B、RNF2、RNF216、NSD1五个不同的基因组位点,每个位点选择与编辑位点完全匹配的gRNA以及与编辑位点存在插入、缺失、错配等错配的3~5种igRNA进行编辑。结果显示在HEK293T的NSD1位点,当使用BE4max和igRNA(缺失第三位C)时,编辑位点第六位单C(C6)的C到T的编辑效率由3.34±0.29%提高到34.89±0.23%、编辑特异性(指第六位单C的编辑占所有种类编辑的比率)由8.40±0.66%提高到71.70±0.85%;,当使用hyBE4max和igRNA(缺失第三位C)时,编辑位点第六位单C(C6)的C到T的编辑效率和编辑特异性均提高。使用不同的igRNA,DNMT3B的C8、RNF2的C6、RNF216的C5以及NSD1的C6,其单C编辑特异性和编辑效率均有一定程度提高,具体如表1-表4所示。
表1胞嘧啶编辑器(BE4max)C到T编辑效率(%)
表2胞嘧啶编辑器(hyBE4max)C到T编辑效率(%)
表3胞嘧啶编辑器(BE4max)C到T编辑特异性(%)
表4胞嘧啶编辑器(hyBE4max)C到T编辑特异性(%)
实施例2ABE gRNA的改造编辑
在哺乳动物细胞中,不同的编辑位点利用改造后gRNA进行腺嘌呤碱基编辑器编辑,其单窗口的碱基编辑占比相对于利用未改造的gRNA均有所提高,最高可以提高10.15倍。
实验过程:将HEK293T或者Hela细胞按5×10
实验结果:选择PSMB2、ABCA3、EMX1-SITE3、VISTA hs267、SNCA、ANO5、KCNQ2、NOTCH2、GFI1九个不同的基因组位点,每个位点选择与编辑位点完全匹配的gRNA以及与编辑位点存在插入、缺失、错配等错配的3~5种igRNA进行编辑。
结果显示在HEK293T的ANO5位点,当使用NG-ABEmax和igRNA(第三位插入碱基A)时,编辑位点第七位单A(A7)的A到G的编辑特异性由27.75±0.03%提高到91.34±0.94%,编辑效率由15.46±0.66%提高到24.94±0.59%。使用不同的igRNA,PSMB2的A5、ABCA3的A5、EMX1-SITE3的A6、VISTA hs267的A5、SNCA的A5、KCNQ2的A5、NOTCH2的A5、GFI1的A5,其单A编辑特异性和编辑效率也相应提高,具体如表5和表6所示。
表5腺嘌呤编辑器A到G编辑效率(%)
表6腺嘌呤编辑器A到G编辑特异性(%)
实施例3PAM拓展实现任意位点碱基精确编辑
在哺乳动物细胞中,选择PAM框拓展的碱基编辑器,通过改变PAM框,可以实现任意位点碱基精准编辑,如图2所示,以ABE编辑器为例,要编辑的位点为A1,但是此时A1附近有A2,以NGG为PAM框,当使用普通gRNA时A1和A2均被编辑,当使用igRNA时主要是A2被编辑,此时并不能实现只编辑A1的目的;若将PAM进一步拓展,以NNN为PAM时,选择合适的N20,当使用gRNA时,A1和A2也均被编辑,当使用igRNA时主要是A1被编辑。因此结合igRNA和PAM框的拓展,可以实现任意位点碱基精准编辑。
实验过程:将目前常用的代表性腺嘌呤碱基编辑器NG-ABEmax质粒中的nCas9替换成可以其识别PAM序列为NRN或者NYN(R为A/G、Y为C/T)的SpRYnCas9,构建可以识别任意PAM框的腺嘌呤碱基编辑器SpRY-ABEmax。将HEK293T细胞按5×105个铺24孔板,等每个孔细胞长至40%-60%时,将SpRY-ABEmax编辑器质粒分别与靶向不同编辑位点的gRNA质粒以及包含插入、缺失、错配的改造igRNA质粒,按照600ng编辑器质粒、300ng gRNA/igRNA质粒的量,利用Lipofectamine 2000(Life,Invitrogen,11668019)试剂转染到HEK293T,每种质粒组合转染设3个重复,转染24小时后添加5ug/ml嘌呤霉素(Merck,USA)到培养基中。转染120小时后使用快速提取DNA提取液(Epicentre,USA)提取基因组DNA,对被编辑位点附近200bp~300bp区域利用Taq DNA聚合酶(康为世纪,中国)PCR,将PCR产物进行高通量测序计算编辑效率(金唯智,中国)。
实验结果:选择EMX1-SITE3、NOTCH2两个不同的基因组位点,在EMX1-SITE3位点,当选择N20为5’-GGAGCACACATGCCCAGGTG-3’PAM序列为NGG,使用NG-ABEmax编辑时,第六位的A(A6)编辑特异性为48.20±0.31%,第八位的A(A8)编辑特异性为0.90±0.01%,使用igRNA后,第六位的A(A6)编辑特异性为72.37±0.40%,第八位的A(A8)编辑特异性为3.18±0.16%,A8的编辑占比仍然较少;当选择N20为5’-GCACACATGCCCAGGTGTGG-3’PAM序列为NAG,使用SpRY-ABEmax编辑时,原来的第六位A变成了第三位A,原来的第八位A变成了第五位A,此时使用对应gRNA编辑时,第三位A编辑特异性为0.91±0.09%,第五位A编辑特异性为36.06±0.47%,使用igRNA(第二位插入碱基T)后,第三位A编辑特异性为0.67±0.32%,第五位A编辑特异性为72.24±1.45%,明显提高第五位A(原第七位A)的编辑占比。NOTCH2位点也获得类似效果,具体如表7和表8所示。
表7腺嘌呤碱基编辑器拓展PAM框A到G编辑效率(%)
表8腺嘌呤碱基编辑器拓展PAM框A到G编辑特异性(%)
实施例4SaCas9 ABE gRNA的改造编辑
在哺乳动物细胞中,不同的编辑位点利用改造后gRNA进行SaCas9腺嘌呤碱基编辑器编辑,其单窗口的碱基编辑占比相对于利用未改造的gRNA均有所提高。
实验过程:将HEK293T细胞按5×10
实验结果:选择CFAP61、Query_55451两个不同的基因组位点,每个位点选择与编辑位点完全匹配的gRNA以及与编辑位点存在插入、缺失、错配等错配的3~5种igRNA进行编辑。
结果显示在HEK293T的CFAP61位点,当使用SaABEmax和igRNA(第三位插入碱基G)时,编辑位点第十一位单A(A11)的A到G的编辑特异性由32.88±0.35%提高到54.05±0.46%,编辑效率由12.83±0.27%提高到15.65±0.46%。使用不同的igRNA,Query_55451的A9其单A编辑特异性和编辑效率也相应提高,具体如表9和表10所示。
表9腺嘌呤编辑器SaABEmax A到G编辑效率(%)
表10腺嘌呤编辑器SaABEmax A到G编辑特异性(%)
本公开的上述实施例仅是为清楚地说明本公开所作的举例,而并非是对本公开的实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。凡在本公开的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本公开权利要求的保护范围之内。
序列表
<110> 中国科学院天津工业生物技术研究所
<120> 缩小碱基编辑器的编辑窗口的方法、碱基编辑器及用途
<130> 6A17-2163489I
<160> 89
<170> SIPOSequenceListing 1.0
<210> 1
<211> 15
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 1
gcacacccca gagcg 15
<210> 2
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 2
gttatcttta ccccagagcg 20
<210> 3
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 3
gtcatgttta ccccagagcg 20
<210> 4
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 4
gtatctttac cccagagcg 19
<210> 5
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 5
gtgacatctt taccccagag cg 22
<210> 6
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 6
gacacgtctg tgtagtgcac 20
<210> 7
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 7
gatacgtctg tgtagtgcac 20
<210> 8
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 8
gttacgtctg tgtagtgcac 20
<210> 9
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 9
gcacgtctgt gtagtgcac 19
<210> 10
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 10
gagtcacgtc tgtgtagtgc ac 22
<210> 11
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 11
gtcatcttag tcattacctg 20
<210> 12
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 12
gttatcttag tcattacctg 20
<210> 13
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 13
gcatcttagt cattacctg 19
<210> 14
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 14
gtacatctta gtcattacct g 21
<210> 15
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 15
gtgtcctttg agctcgtgca 20
<210> 16
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 16
gtgtcttttg agctcgtgca 20
<210> 17
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 17
gttcctttga gctcgtgca 19
<210> 18
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 18
gttgtccttt gagctcgtgc a 21
<210> 19
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 19
ggcatcagtg tgacatctgc 20
<210> 20
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 20
ggtatcagtg tgacatctgc 20
<210> 21
<211> 18
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 21
gatcagtgtg acatctgc 18
<210> 22
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 22
gtgcatcagt gtgacatctg c 21
<210> 23
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 23
gtaaacaaag catagactga 20
<210> 24
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 24
gttaacaaag catagactga 20
<210> 25
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 25
gtaatcaaag catagactga 20
<210> 26
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 26
gtaacaaagc atagactga 19
<210> 27
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 27
gtgaaacaaa gcatagactg a 21
<210> 28
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 28
gaagagcagg gtcatgaagg 20
<210> 29
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 29
gatgagcagg gtcatgaagg 20
<210> 30
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 30
gagagcaggg tcatgaagg 19
<210> 31
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 31
gcaagagcag ggtcatgaag g 21
<210> 32
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 32
ggagcacaca tgcccaggtg 20
<210> 33
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 33
ggtgcacaca tgcccaggtg 20
<210> 34
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 34
ggaccacaca tgcccaggtg 20
<210> 35
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 35
gagcacacat gcccaggtg 19
<210> 36
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 36
ggtagcacac atgcccaggt g 21
<210> 37
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 37
gaacacaaag catagactgc 20
<210> 38
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 38
gtacacaaag catagactgc 20
<210> 39
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 39
gaagacaaag catagactgc 20
<210> 40
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 40
gaaacaaagc atagactgc 19
<210> 41
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 41
gatacacaaa gcatagactg c 21
<210> 42
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 42
gaacacaatg catagattgc 20
<210> 43
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 43
gatcacaatg catagattgc 20
<210> 44
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 44
gaactcaatg catagattgc 20
<210> 45
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 45
gacacaatgc atagattgc 19
<210> 46
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 46
gacacacaat gcatagattg c 21
<210> 47
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 47
tcacacactt gatcacagag 20
<210> 48
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 48
tgacacactt gatcacagag 20
<210> 49
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 49
tcatacactt gatcacagag 20
<210> 50
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 50
tacacacttg atcacagag 19
<210> 51
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 51
tcaacacact tgatcacaga g 21
<210> 52
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 52
gaagaaggag acaccgatga 20
<210> 53
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 53
gacgaaggag acaccgatga 20
<210> 54
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 54
gagaaggaga caccgatga 19
<210> 55
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 55
gcaagaagga gacaccgatg a 21
<210> 56
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 56
tgacacagga gacctgtcac 20
<210> 57
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 57
tgacatagga gacctgtcac 20
<210> 58
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 58
tgaacaggag acctgtcac 19
<210> 59
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 59
tgaacacagg agacctgtca c 21
<210> 60
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 60
tgggaagggt ttccagagga 20
<210> 61
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 61
taggaagggt ttccagagga 20
<210> 62
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 62
tggaagggtt tccagagga 19
<210> 63
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 63
tgtggaaggg tttccagagg a 21
<210> 64
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 64
atacaggaga cctgtcacag 20
<210> 65
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 65
aacaggagac ctgtcacag 19
<210> 66
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 66
acgacaggag acctgtcaca g 21
<210> 67
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 67
gcacacatgc ccaggtgtgg 20
<210> 68
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 68
ggacacatgc ccaggtgtgg 20
<210> 69
<211> 19
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 69
gccacatgcc caggtgtgg 19
<210> 70
<211> 21
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 70
gtcacacatg cccaggtgtg g 21
<210> 71
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 71
ggagagaaag agaagttgat tg 22
<210> 72
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 72
ggcgagaaag agaagttgat tg 22
<210> 73
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 73
gttgagaaag agaagttgat tg 22
<210> 74
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 74
ggagaaagag aagttgattg 20
<210> 75
<211> 23
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 75
gggagagaaa gagaagttga ttg 23
<210> 76
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 76
gctgttgcat gaggaaaggg ac 22
<210> 77
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 77
gtagttgcat gaggaaaggg ac 22
<210> 78
<211> 22
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 78
ggtgttgcat gaggaaaggg ac 22
<210> 79
<211> 20
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 79
gcttgcatga ggaaagggac 20
<210> 80
<211> 24
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 80
gctatgttgc atgaggaaag ggac 24
<210> 81
<211> 76
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 81
gttttagagc tagaaatagc aagttaaaat aaggctagtc cgttatcaac ttgaaaaagt 60
ggcaccgagt cggtgc 76
<210> 82
<211> 76
<212> DNA/RNA
<213> Artificial Sequence
<220>
<223> 向导序列
<400> 82
gttttagtac tctggaaaca gaatctacta aaacaaggca aaatgccgtg tttatctcgt 60
caacttgttg gcgaga 76
<210> 83
<211> 166
<212> PRT
<213> Artificial Sequence
<220>
<223> TadA突变体序列
<400> 83
Ser Glu Val Glu Phe Ser His Glu Tyr Trp Met Arg His Ala Leu Thr
1 5 10 15
Leu Ala Lys Arg Ala Arg Asp Glu Arg Glu Val Pro Val Gly Ala Val
20 25 30
Leu Val Leu Asn Asn Arg Val Ile Gly Glu Gly Trp Asn Arg Ala Ile
35 40 45
Gly Leu His Asp Pro Thr Ala His Ala Glu Ile Met Ala Leu Arg Gln
50 55 60
Gly Gly Leu Val Met Gln Asn Tyr Arg Leu Ile Asp Ala Thr Leu Tyr
65 70 75 80
Val Thr Phe Glu Pro Cys Val Met Cys Ala Gly Ala Met Ile His Ser
85 90 95
Arg Ile Gly Arg Val Val Phe Gly Val Arg Asn Ala Lys Thr Gly Ala
100 105 110
Ala Gly Ser Leu Met Asp Val Leu His Tyr Pro Gly Met Asn His Arg
115 120 125
Val Glu Ile Thr Glu Gly Ile Leu Ala Asp Glu Cys Ala Ala Leu Leu
130 135 140
Cys Tyr Phe Phe Arg Met Pro Arg Gln Val Phe Asn Ala Gln Lys Lys
145 150 155 160
Ala Gln Ser Ser Thr Asp
165
<210> 84
<211> 166
<212> PRT
<213> Artificial Sequence
<220>
<223> TadA突变体序列
<400> 84
Ser Glu Val Glu Phe Ser His Glu Tyr Trp Met Arg His Ala Leu Thr
1 5 10 15
Leu Ala Lys Arg Ala Arg Asp Glu Arg Glu Val Pro Val Gly Ala Val
20 25 30
Leu Val Leu Asn Asn Arg Val Ile Gly Glu Gly Trp Asn Arg Ala Ile
35 40 45
Gly Leu His Asp Pro Thr Ala His Ala Glu Ile Met Ala Leu Arg Gln
50 55 60
Gly Gly Leu Val Met Gln Asn Tyr Arg Leu Ile Asp Ala Thr Leu Tyr
65 70 75 80
Val Thr Phe Glu Pro Cys Val Met Cys Ala Gly Ala Met Ile His Ser
85 90 95
Arg Ile Gly Arg Val Val Phe Gly Val Arg Asn Ser Lys Arg Gly Ala
100 105 110
Ala Gly Ser Leu Met Asn Val Leu Asn Tyr Pro Gly Met Asn His Arg
115 120 125
Val Glu Ile Thr Glu Gly Ile Leu Ala Asp Glu Cys Ala Ala Leu Leu
130 135 140
Cys Asp Phe Tyr Arg Met Pro Arg Gln Val Phe Asn Ala Gln Lys Lys
145 150 155 160
Ala Gln Ser Ser Ile Asn
165
<210> 85
<211> 1368
<212> PRT
<213> Artificial Sequence
<220>
<223> SpdCas9序列
<400> 85
Met Asp Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp Ala Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys
1010 1015 1020
Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser
1025 1030 1035 1040
Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1045 1050 1055
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile
1060 1065 1070
Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser
1075 1080 1085
Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1090 1095 1100
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile
1105 1110 1115 1120
Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser
1125 1130 1135
Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly
1140 1145 1150
Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile
1155 1160 1165
Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala
1170 1175 1180
Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1185 1190 1195 1200
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser
1205 1210 1215
Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1220 1225 1230
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His
1250 1255 1260
Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val
1265 1270 1275 1280
Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1285 1290 1295
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu
1300 1305 1310
Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp
1315 1320 1325
Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1330 1335 1340
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile
1345 1350 1355 1360
Asp Leu Ser Gln Leu Gly Gly Asp
1365
<210> 86
<211> 1368
<212> PRT
<213> Artificial Sequence
<220>
<223> SpnCas9序列
<400> 86
Met Asp Lys Lys Tyr Ser Ile Gly Leu Ala Ile Gly Thr Asn Ser Val
1 5 10 15
Gly Trp Ala Val Ile Thr Asp Glu Tyr Lys Val Pro Ser Lys Lys Phe
20 25 30
Lys Val Leu Gly Asn Thr Asp Arg His Ser Ile Lys Lys Asn Leu Ile
35 40 45
Gly Ala Leu Leu Phe Asp Ser Gly Glu Thr Ala Glu Ala Thr Arg Leu
50 55 60
Lys Arg Thr Ala Arg Arg Arg Tyr Thr Arg Arg Lys Asn Arg Ile Cys
65 70 75 80
Tyr Leu Gln Glu Ile Phe Ser Asn Glu Met Ala Lys Val Asp Asp Ser
85 90 95
Phe Phe His Arg Leu Glu Glu Ser Phe Leu Val Glu Glu Asp Lys Lys
100 105 110
His Glu Arg His Pro Ile Phe Gly Asn Ile Val Asp Glu Val Ala Tyr
115 120 125
His Glu Lys Tyr Pro Thr Ile Tyr His Leu Arg Lys Lys Leu Val Asp
130 135 140
Ser Thr Asp Lys Ala Asp Leu Arg Leu Ile Tyr Leu Ala Leu Ala His
145 150 155 160
Met Ile Lys Phe Arg Gly His Phe Leu Ile Glu Gly Asp Leu Asn Pro
165 170 175
Asp Asn Ser Asp Val Asp Lys Leu Phe Ile Gln Leu Val Gln Thr Tyr
180 185 190
Asn Gln Leu Phe Glu Glu Asn Pro Ile Asn Ala Ser Gly Val Asp Ala
195 200 205
Lys Ala Ile Leu Ser Ala Arg Leu Ser Lys Ser Arg Arg Leu Glu Asn
210 215 220
Leu Ile Ala Gln Leu Pro Gly Glu Lys Lys Asn Gly Leu Phe Gly Asn
225 230 235 240
Leu Ile Ala Leu Ser Leu Gly Leu Thr Pro Asn Phe Lys Ser Asn Phe
245 250 255
Asp Leu Ala Glu Asp Ala Lys Leu Gln Leu Ser Lys Asp Thr Tyr Asp
260 265 270
Asp Asp Leu Asp Asn Leu Leu Ala Gln Ile Gly Asp Gln Tyr Ala Asp
275 280 285
Leu Phe Leu Ala Ala Lys Asn Leu Ser Asp Ala Ile Leu Leu Ser Asp
290 295 300
Ile Leu Arg Val Asn Thr Glu Ile Thr Lys Ala Pro Leu Ser Ala Ser
305 310 315 320
Met Ile Lys Arg Tyr Asp Glu His His Gln Asp Leu Thr Leu Leu Lys
325 330 335
Ala Leu Val Arg Gln Gln Leu Pro Glu Lys Tyr Lys Glu Ile Phe Phe
340 345 350
Asp Gln Ser Lys Asn Gly Tyr Ala Gly Tyr Ile Asp Gly Gly Ala Ser
355 360 365
Gln Glu Glu Phe Tyr Lys Phe Ile Lys Pro Ile Leu Glu Lys Met Asp
370 375 380
Gly Thr Glu Glu Leu Leu Val Lys Leu Asn Arg Glu Asp Leu Leu Arg
385 390 395 400
Lys Gln Arg Thr Phe Asp Asn Gly Ser Ile Pro His Gln Ile His Leu
405 410 415
Gly Glu Leu His Ala Ile Leu Arg Arg Gln Glu Asp Phe Tyr Pro Phe
420 425 430
Leu Lys Asp Asn Arg Glu Lys Ile Glu Lys Ile Leu Thr Phe Arg Ile
435 440 445
Pro Tyr Tyr Val Gly Pro Leu Ala Arg Gly Asn Ser Arg Phe Ala Trp
450 455 460
Met Thr Arg Lys Ser Glu Glu Thr Ile Thr Pro Trp Asn Phe Glu Glu
465 470 475 480
Val Val Asp Lys Gly Ala Ser Ala Gln Ser Phe Ile Glu Arg Met Thr
485 490 495
Asn Phe Asp Lys Asn Leu Pro Asn Glu Lys Val Leu Pro Lys His Ser
500 505 510
Leu Leu Tyr Glu Tyr Phe Thr Val Tyr Asn Glu Leu Thr Lys Val Lys
515 520 525
Tyr Val Thr Glu Gly Met Arg Lys Pro Ala Phe Leu Ser Gly Glu Gln
530 535 540
Lys Lys Ala Ile Val Asp Leu Leu Phe Lys Thr Asn Arg Lys Val Thr
545 550 555 560
Val Lys Gln Leu Lys Glu Asp Tyr Phe Lys Lys Ile Glu Cys Phe Asp
565 570 575
Ser Val Glu Ile Ser Gly Val Glu Asp Arg Phe Asn Ala Ser Leu Gly
580 585 590
Thr Tyr His Asp Leu Leu Lys Ile Ile Lys Asp Lys Asp Phe Leu Asp
595 600 605
Asn Glu Glu Asn Glu Asp Ile Leu Glu Asp Ile Val Leu Thr Leu Thr
610 615 620
Leu Phe Glu Asp Arg Glu Met Ile Glu Glu Arg Leu Lys Thr Tyr Ala
625 630 635 640
His Leu Phe Asp Asp Lys Val Met Lys Gln Leu Lys Arg Arg Arg Tyr
645 650 655
Thr Gly Trp Gly Arg Leu Ser Arg Lys Leu Ile Asn Gly Ile Arg Asp
660 665 670
Lys Gln Ser Gly Lys Thr Ile Leu Asp Phe Leu Lys Ser Asp Gly Phe
675 680 685
Ala Asn Arg Asn Phe Met Gln Leu Ile His Asp Asp Ser Leu Thr Phe
690 695 700
Lys Glu Asp Ile Gln Lys Ala Gln Val Ser Gly Gln Gly Asp Ser Leu
705 710 715 720
His Glu His Ile Ala Asn Leu Ala Gly Ser Pro Ala Ile Lys Lys Gly
725 730 735
Ile Leu Gln Thr Val Lys Val Val Asp Glu Leu Val Lys Val Met Gly
740 745 750
Arg His Lys Pro Glu Asn Ile Val Ile Glu Met Ala Arg Glu Asn Gln
755 760 765
Thr Thr Gln Lys Gly Gln Lys Asn Ser Arg Glu Arg Met Lys Arg Ile
770 775 780
Glu Glu Gly Ile Lys Glu Leu Gly Ser Gln Ile Leu Lys Glu His Pro
785 790 795 800
Val Glu Asn Thr Gln Leu Gln Asn Glu Lys Leu Tyr Leu Tyr Tyr Leu
805 810 815
Gln Asn Gly Arg Asp Met Tyr Val Asp Gln Glu Leu Asp Ile Asn Arg
820 825 830
Leu Ser Asp Tyr Asp Val Asp His Ile Val Pro Gln Ser Phe Leu Lys
835 840 845
Asp Asp Ser Ile Asp Asn Lys Val Leu Thr Arg Ser Asp Lys Asn Arg
850 855 860
Gly Lys Ser Asp Asn Val Pro Ser Glu Glu Val Val Lys Lys Met Lys
865 870 875 880
Asn Tyr Trp Arg Gln Leu Leu Asn Ala Lys Leu Ile Thr Gln Arg Lys
885 890 895
Phe Asp Asn Leu Thr Lys Ala Glu Arg Gly Gly Leu Ser Glu Leu Asp
900 905 910
Lys Ala Gly Phe Ile Lys Arg Gln Leu Val Glu Thr Arg Gln Ile Thr
915 920 925
Lys His Val Ala Gln Ile Leu Asp Ser Arg Met Asn Thr Lys Tyr Asp
930 935 940
Glu Asn Asp Lys Leu Ile Arg Glu Val Lys Val Ile Thr Leu Lys Ser
945 950 955 960
Lys Leu Val Ser Asp Phe Arg Lys Asp Phe Gln Phe Tyr Lys Val Arg
965 970 975
Glu Ile Asn Asn Tyr His His Ala His Asp Ala Tyr Leu Asn Ala Val
980 985 990
Val Gly Thr Ala Leu Ile Lys Lys Tyr Pro Lys Leu Glu Ser Glu Phe
995 1000 1005
Val Tyr Gly Asp Tyr Lys Val Tyr Asp Val Arg Lys Met Ile Ala Lys
1010 1015 1020
Ser Glu Gln Glu Ile Gly Lys Ala Thr Ala Lys Tyr Phe Phe Tyr Ser
1025 1030 1035 1040
Asn Ile Met Asn Phe Phe Lys Thr Glu Ile Thr Leu Ala Asn Gly Glu
1045 1050 1055
Ile Arg Lys Arg Pro Leu Ile Glu Thr Asn Gly Glu Thr Gly Glu Ile
1060 1065 1070
Val Trp Asp Lys Gly Arg Asp Phe Ala Thr Val Arg Lys Val Leu Ser
1075 1080 1085
Met Pro Gln Val Asn Ile Val Lys Lys Thr Glu Val Gln Thr Gly Gly
1090 1095 1100
Phe Ser Lys Glu Ser Ile Leu Pro Lys Arg Asn Ser Asp Lys Leu Ile
1105 1110 1115 1120
Ala Arg Lys Lys Asp Trp Asp Pro Lys Lys Tyr Gly Gly Phe Asp Ser
1125 1130 1135
Pro Thr Val Ala Tyr Ser Val Leu Val Val Ala Lys Val Glu Lys Gly
1140 1145 1150
Lys Ser Lys Lys Leu Lys Ser Val Lys Glu Leu Leu Gly Ile Thr Ile
1155 1160 1165
Met Glu Arg Ser Ser Phe Glu Lys Asn Pro Ile Asp Phe Leu Glu Ala
1170 1175 1180
Lys Gly Tyr Lys Glu Val Lys Lys Asp Leu Ile Ile Lys Leu Pro Lys
1185 1190 1195 1200
Tyr Ser Leu Phe Glu Leu Glu Asn Gly Arg Lys Arg Met Leu Ala Ser
1205 1210 1215
Ala Gly Glu Leu Gln Lys Gly Asn Glu Leu Ala Leu Pro Ser Lys Tyr
1220 1225 1230
Val Asn Phe Leu Tyr Leu Ala Ser His Tyr Glu Lys Leu Lys Gly Ser
1235 1240 1245
Pro Glu Asp Asn Glu Gln Lys Gln Leu Phe Val Glu Gln His Lys His
1250 1255 1260
Tyr Leu Asp Glu Ile Ile Glu Gln Ile Ser Glu Phe Ser Lys Arg Val
1265 1270 1275 1280
Ile Leu Ala Asp Ala Asn Leu Asp Lys Val Leu Ser Ala Tyr Asn Lys
1285 1290 1295
His Arg Asp Lys Pro Ile Arg Glu Gln Ala Glu Asn Ile Ile His Leu
1300 1305 1310
Phe Thr Leu Thr Asn Leu Gly Ala Pro Ala Ala Phe Lys Tyr Phe Asp
1315 1320 1325
Thr Thr Ile Asp Arg Lys Arg Tyr Thr Ser Thr Lys Glu Val Leu Asp
1330 1335 1340
Ala Thr Leu Ile His Gln Ser Ile Thr Gly Leu Tyr Glu Thr Arg Ile
1345 1350 1355 1360
Asp Leu Ser Gln Leu Gly Gly Asp
1365
<210> 87
<211> 1053
<212> PRT
<213> Artificial Sequence
<220>
<223> SadCas9序列
<400> 87
Met Lys Arg Asn Tyr Ile Leu Gly Leu Ala Ile Gly Ile Thr Ser Val
1 5 10 15
Gly Tyr Gly Ile Ile Asp Tyr Glu Thr Arg Asp Val Ile Asp Ala Gly
20 25 30
Val Arg Leu Phe Lys Glu Ala Asn Val Glu Asn Asn Glu Gly Arg Arg
35 40 45
Ser Lys Arg Gly Ala Arg Arg Leu Lys Arg Arg Arg Arg His Arg Ile
50 55 60
Gln Arg Val Lys Lys Leu Leu Phe Asp Tyr Asn Leu Leu Thr Asp His
65 70 75 80
Ser Glu Leu Ser Gly Ile Asn Pro Tyr Glu Ala Arg Val Lys Gly Leu
85 90 95
Ser Gln Lys Leu Ser Glu Glu Glu Phe Ser Ala Ala Leu Leu His Leu
100 105 110
Ala Lys Arg Arg Gly Val His Asn Val Asn Glu Val Glu Glu Asp Thr
115 120 125
Gly Asn Glu Leu Ser Thr Lys Glu Gln Ile Ser Arg Asn Ser Lys Ala
130 135 140
Leu Glu Glu Lys Tyr Val Ala Glu Leu Gln Leu Glu Arg Leu Lys Lys
145 150 155 160
Asp Gly Glu Val Arg Gly Ser Ile Asn Arg Phe Lys Thr Ser Asp Tyr
165 170 175
Val Lys Glu Ala Lys Gln Leu Leu Lys Val Gln Lys Ala Tyr His Gln
180 185 190
Leu Asp Gln Ser Phe Ile Asp Thr Tyr Ile Asp Leu Leu Glu Thr Arg
195 200 205
Arg Thr Tyr Tyr Glu Gly Pro Gly Glu Gly Ser Pro Phe Gly Trp Lys
210 215 220
Asp Ile Lys Glu Trp Tyr Glu Met Leu Met Gly His Cys Thr Tyr Phe
225 230 235 240
Pro Glu Glu Leu Arg Ser Val Lys Tyr Ala Tyr Asn Ala Asp Leu Tyr
245 250 255
Asn Ala Leu Asn Asp Leu Asn Asn Leu Val Ile Thr Arg Asp Glu Asn
260 265 270
Glu Lys Leu Glu Tyr Tyr Glu Lys Phe Gln Ile Ile Glu Asn Val Phe
275 280 285
Lys Gln Lys Lys Lys Pro Thr Leu Lys Gln Ile Ala Lys Glu Ile Leu
290 295 300
Val Asn Glu Glu Asp Ile Lys Gly Tyr Arg Val Thr Ser Thr Gly Lys
305 310 315 320
Pro Glu Phe Thr Asn Leu Lys Val Tyr His Asp Ile Lys Asp Ile Thr
325 330 335
Ala Arg Lys Glu Ile Ile Glu Asn Ala Glu Leu Leu Asp Gln Ile Ala
340 345 350
Lys Ile Leu Thr Ile Tyr Gln Ser Ser Glu Asp Ile Gln Glu Glu Leu
355 360 365
Thr Asn Leu Asn Ser Glu Leu Thr Gln Glu Glu Ile Glu Gln Ile Ser
370 375 380
Asn Leu Lys Gly Tyr Thr Gly Thr His Asn Leu Ser Leu Lys Ala Ile
385 390 395 400
Asn Leu Ile Leu Asp Glu Leu Trp His Thr Asn Asp Asn Gln Ile Ala
405 410 415
Ile Phe Asn Arg Leu Lys Leu Val Pro Lys Lys Val Asp Leu Ser Gln
420 425 430
Gln Lys Glu Ile Pro Thr Thr Leu Val Asp Asp Phe Ile Leu Ser Pro
435 440 445
Val Val Lys Arg Ser Phe Ile Gln Ser Ile Lys Val Ile Asn Ala Ile
450 455 460
Ile Lys Lys Tyr Gly Leu Pro Asn Asp Ile Ile Ile Glu Leu Ala Arg
465 470 475 480
Glu Lys Asn Ser Lys Asp Ala Gln Lys Met Ile Asn Glu Met Gln Lys
485 490 495
Arg Asn Arg Gln Thr Asn Glu Arg Ile Glu Glu Ile Ile Arg Thr Thr
500 505 510
Gly Lys Glu Asn Ala Lys Tyr Leu Ile Glu Lys Ile Lys Leu His Asp
515 520 525
Met Gln Glu Gly Lys Cys Leu Tyr Ser Leu Glu Ala Ile Pro Leu Glu
530 535 540
Asp Leu Leu Asn Asn Pro Phe Asn Tyr Glu Val Asp His Ile Ile Pro
545 550 555 560
Arg Ser Val Ser Phe Asp Asn Ser Phe Asn Asn Lys Val Leu Val Lys
565 570 575
Gln Glu Glu Ala Ser Lys Lys Gly Asn Arg Thr Pro Phe Gln Tyr Leu
580 585 590
Ser Ser Ser Asp Ser Lys Ile Ser Tyr Glu Thr Phe Lys Lys His Ile
595 600 605
Leu Asn Leu Ala Lys Gly Lys Gly Arg Ile Ser Lys Thr Lys Lys Glu
610 615 620
Tyr Leu Leu Glu Glu Arg Asp Ile Asn Arg Phe Ser Val Gln Lys Asp
625 630 635 640
Phe Ile Asn Arg Asn Leu Val Asp Thr Arg Tyr Ala Thr Arg Gly Leu
645 650 655
Met Asn Leu Leu Arg Ser Tyr Phe Arg Val Asn Asn Leu Asp Val Lys
660 665 670
Val Lys Ser Ile Asn Gly Gly Phe Thr Ser Phe Leu Arg Arg Lys Trp
675 680 685
Lys Phe Lys Lys Glu Arg Asn Lys Gly Tyr Lys His His Ala Glu Asp
690 695 700
Ala Leu Ile Ile Ala Asn Ala Asp Phe Ile Phe Lys Glu Trp Lys Lys
705 710 715 720
Leu Asp Lys Ala Lys Lys Val Met Glu Asn Gln Met Phe Glu Glu Lys
725 730 735
Gln Ala Glu Ser Met Pro Glu Ile Glu Thr Glu Gln Glu Tyr Lys Glu
740 745 750
Ile Phe Ile Thr Pro His Gln Ile Lys His Ile Lys Asp Phe Lys Asp
755 760 765
Tyr Lys Tyr Ser His Arg Val Asp Lys Lys Pro Asn Arg Glu Leu Ile
770 775 780
Asn Asp Thr Leu Tyr Ser Thr Arg Lys Asp Asp Lys Gly Asn Thr Leu
785 790 795 800
Ile Val Asn Asn Leu Asn Gly Leu Tyr Asp Lys Asp Asn Asp Lys Leu
805 810 815
Lys Lys Leu Ile Asn Lys Ser Pro Glu Lys Leu Leu Met Tyr His His
820 825 830
Asp Pro Gln Thr Tyr Gln Lys Leu Lys Leu Ile Met Glu Gln Tyr Gly
835 840 845
Asp Glu Lys Asn Pro Leu Tyr Lys Tyr Tyr Glu Glu Thr Gly Asn Tyr
850 855 860
Leu Thr Lys Tyr Ser Lys Lys Asp Asn Gly Pro Val Ile Lys Lys Ile
865 870 875 880
Lys Tyr Tyr Gly Asn Lys Leu Asn Ala His Leu Asp Ile Thr Asp Asp
885 890 895
Tyr Pro Asn Ser Arg Asn Lys Val Val Lys Leu Ser Leu Lys Pro Tyr
900 905 910
Arg Phe Asp Val Tyr Leu Asp Asn Gly Val Tyr Lys Phe Val Thr Val
915 920 925
Lys Asn Leu Asp Val Ile Lys Lys Glu Asn Tyr Tyr Glu Val Asn Ser
930 935 940
Lys Cys Tyr Glu Glu Ala Lys Lys Leu Lys Lys Ile Ser Asn Gln Ala
945 950 955 960
Glu Phe Ile Ala Ser Phe Tyr Asn Asn Asp Leu Ile Lys Ile Asn Gly
965 970 975
Glu Leu Tyr Arg Val Ile Gly Val Asn Asn Asp Leu Leu Asn Arg Ile
980 985 990
Glu Val Asn Met Ile Asp Ile Thr Tyr Arg Glu Tyr Leu Glu Asn Met
995 1000 1005
Asn Asp Lys Arg Pro Pro Arg Ile Ile Lys Thr Ile Ala Ser Lys Thr
1010 1015 1020
Gln Ser Ile Lys Lys Tyr Ser Thr Asp Ile Leu Gly Asn Leu Tyr Glu
1025 1030 1035 1040
Val Lys Ser Lys Lys His Pro Gln Ile Ile Lys Lys Gly
1045 1050
<210> 88
<211> 1053
<212> PRT
<213> Artificial Sequence
<220>
<223> SanCas9序列
<400> 88
Met Lys Arg Asn Tyr Ile Leu Gly Leu Ala Ile Gly Ile Thr Ser Val
1 5 10 15
Gly Tyr Gly Ile Ile Asp Tyr Glu Thr Arg Asp Val Ile Asp Ala Gly
20 25 30
Val Arg Leu Phe Lys Glu Ala Asn Val Glu Asn Asn Glu Gly Arg Arg
35 40 45
Ser Lys Arg Gly Ala Arg Arg Leu Lys Arg Arg Arg Arg His Arg Ile
50 55 60
Gln Arg Val Lys Lys Leu Leu Phe Asp Tyr Asn Leu Leu Thr Asp His
65 70 75 80
Ser Glu Leu Ser Gly Ile Asn Pro Tyr Glu Ala Arg Val Lys Gly Leu
85 90 95
Ser Gln Lys Leu Ser Glu Glu Glu Phe Ser Ala Ala Leu Leu His Leu
100 105 110
Ala Lys Arg Arg Gly Val His Asn Val Asn Glu Val Glu Glu Asp Thr
115 120 125
Gly Asn Glu Leu Ser Thr Lys Glu Gln Ile Ser Arg Asn Ser Lys Ala
130 135 140
Leu Glu Glu Lys Tyr Val Ala Glu Leu Gln Leu Glu Arg Leu Lys Lys
145 150 155 160
Asp Gly Glu Val Arg Gly Ser Ile Asn Arg Phe Lys Thr Ser Asp Tyr
165 170 175
Val Lys Glu Ala Lys Gln Leu Leu Lys Val Gln Lys Ala Tyr His Gln
180 185 190
Leu Asp Gln Ser Phe Ile Asp Thr Tyr Ile Asp Leu Leu Glu Thr Arg
195 200 205
Arg Thr Tyr Tyr Glu Gly Pro Gly Glu Gly Ser Pro Phe Gly Trp Lys
210 215 220
Asp Ile Lys Glu Trp Tyr Glu Met Leu Met Gly His Cys Thr Tyr Phe
225 230 235 240
Pro Glu Glu Leu Arg Ser Val Lys Tyr Ala Tyr Asn Ala Asp Leu Tyr
245 250 255
Asn Ala Leu Asn Asp Leu Asn Asn Leu Val Ile Thr Arg Asp Glu Asn
260 265 270
Glu Lys Leu Glu Tyr Tyr Glu Lys Phe Gln Ile Ile Glu Asn Val Phe
275 280 285
Lys Gln Lys Lys Lys Pro Thr Leu Lys Gln Ile Ala Lys Glu Ile Leu
290 295 300
Val Asn Glu Glu Asp Ile Lys Gly Tyr Arg Val Thr Ser Thr Gly Lys
305 310 315 320
Pro Glu Phe Thr Asn Leu Lys Val Tyr His Asp Ile Lys Asp Ile Thr
325 330 335
Ala Arg Lys Glu Ile Ile Glu Asn Ala Glu Leu Leu Asp Gln Ile Ala
340 345 350
Lys Ile Leu Thr Ile Tyr Gln Ser Ser Glu Asp Ile Gln Glu Glu Leu
355 360 365
Thr Asn Leu Asn Ser Glu Leu Thr Gln Glu Glu Ile Glu Gln Ile Ser
370 375 380
Asn Leu Lys Gly Tyr Thr Gly Thr His Asn Leu Ser Leu Lys Ala Ile
385 390 395 400
Asn Leu Ile Leu Asp Glu Leu Trp His Thr Asn Asp Asn Gln Ile Ala
405 410 415
Ile Phe Asn Arg Leu Lys Leu Val Pro Lys Lys Val Asp Leu Ser Gln
420 425 430
Gln Lys Glu Ile Pro Thr Thr Leu Val Asp Asp Phe Ile Leu Ser Pro
435 440 445
Val Val Lys Arg Ser Phe Ile Gln Ser Ile Lys Val Ile Asn Ala Ile
450 455 460
Ile Lys Lys Tyr Gly Leu Pro Asn Asp Ile Ile Ile Glu Leu Ala Arg
465 470 475 480
Glu Lys Asn Ser Lys Asp Ala Gln Lys Met Ile Asn Glu Met Gln Lys
485 490 495
Arg Asn Arg Gln Thr Asn Glu Arg Ile Glu Glu Ile Ile Arg Thr Thr
500 505 510
Gly Lys Glu Asn Ala Lys Tyr Leu Ile Glu Lys Ile Lys Leu His Asp
515 520 525
Met Gln Glu Gly Lys Cys Leu Tyr Ser Leu Glu Ala Ile Pro Leu Glu
530 535 540
Asp Leu Leu Asn Asn Pro Phe Asn Tyr Glu Val Asp His Ile Ile Pro
545 550 555 560
Arg Ser Val Ser Phe Asp Asn Ser Phe Asn Asn Lys Val Leu Val Lys
565 570 575
Gln Glu Glu Asn Ser Lys Lys Gly Asn Arg Thr Pro Phe Gln Tyr Leu
580 585 590
Ser Ser Ser Asp Ser Lys Ile Ser Tyr Glu Thr Phe Lys Lys His Ile
595 600 605
Leu Asn Leu Ala Lys Gly Lys Gly Arg Ile Ser Lys Thr Lys Lys Glu
610 615 620
Tyr Leu Leu Glu Glu Arg Asp Ile Asn Arg Phe Ser Val Gln Lys Asp
625 630 635 640
Phe Ile Asn Arg Asn Leu Val Asp Thr Arg Tyr Ala Thr Arg Gly Leu
645 650 655
Met Asn Leu Leu Arg Ser Tyr Phe Arg Val Asn Asn Leu Asp Val Lys
660 665 670
Val Lys Ser Ile Asn Gly Gly Phe Thr Ser Phe Leu Arg Arg Lys Trp
675 680 685
Lys Phe Lys Lys Glu Arg Asn Lys Gly Tyr Lys His His Ala Glu Asp
690 695 700
Ala Leu Ile Ile Ala Asn Ala Asp Phe Ile Phe Lys Glu Trp Lys Lys
705 710 715 720
Leu Asp Lys Ala Lys Lys Val Met Glu Asn Gln Met Phe Glu Glu Lys
725 730 735
Gln Ala Glu Ser Met Pro Glu Ile Glu Thr Glu Gln Glu Tyr Lys Glu
740 745 750
Ile Phe Ile Thr Pro His Gln Ile Lys His Ile Lys Asp Phe Lys Asp
755 760 765
Tyr Lys Tyr Ser His Arg Val Asp Lys Lys Pro Asn Arg Glu Leu Ile
770 775 780
Asn Asp Thr Leu Tyr Ser Thr Arg Lys Asp Asp Lys Gly Asn Thr Leu
785 790 795 800
Ile Val Asn Asn Leu Asn Gly Leu Tyr Asp Lys Asp Asn Asp Lys Leu
805 810 815
Lys Lys Leu Ile Asn Lys Ser Pro Glu Lys Leu Leu Met Tyr His His
820 825 830
Asp Pro Gln Thr Tyr Gln Lys Leu Lys Leu Ile Met Glu Gln Tyr Gly
835 840 845
Asp Glu Lys Asn Pro Leu Tyr Lys Tyr Tyr Glu Glu Thr Gly Asn Tyr
850 855 860
Leu Thr Lys Tyr Ser Lys Lys Asp Asn Gly Pro Val Ile Lys Lys Ile
865 870 875 880
Lys Tyr Tyr Gly Asn Lys Leu Asn Ala His Leu Asp Ile Thr Asp Asp
885 890 895
Tyr Pro Asn Ser Arg Asn Lys Val Val Lys Leu Ser Leu Lys Pro Tyr
900 905 910
Arg Phe Asp Val Tyr Leu Asp Asn Gly Val Tyr Lys Phe Val Thr Val
915 920 925
Lys Asn Leu Asp Val Ile Lys Lys Glu Asn Tyr Tyr Glu Val Asn Ser
930 935 940
Lys Cys Tyr Glu Glu Ala Lys Lys Leu Lys Lys Ile Ser Asn Gln Ala
945 950 955 960
Glu Phe Ile Ala Ser Phe Tyr Asn Asn Asp Leu Ile Lys Ile Asn Gly
965 970 975
Glu Leu Tyr Arg Val Ile Gly Val Asn Asn Asp Leu Leu Asn Arg Ile
980 985 990
Glu Val Asn Met Ile Asp Ile Thr Tyr Arg Glu Tyr Leu Glu Asn Met
995 1000 1005
Asn Asp Lys Arg Pro Pro Arg Ile Ile Lys Thr Ile Ala Ser Lys Thr
1010 1015 1020
Gln Ser Ile Lys Lys Tyr Ser Thr Asp Ile Leu Gly Asn Leu Tyr Glu
1025 1030 1035 1040
Val Lys Ser Lys Lys His Pro Gln Ile Ile Lys Lys Gly
1045 1050
<210> 89
<211> 1228
<212> PRT
<213> Artificial Sequence
<220>
<223> LbdCpf1序列
<400> 89
Met Ser Lys Leu Glu Lys Phe Thr Asn Cys Tyr Ser Leu Ser Lys Thr
1 5 10 15
Leu Arg Phe Lys Ala Ile Pro Val Gly Lys Thr Gln Glu Asn Ile Asp
20 25 30
Asn Lys Arg Leu Leu Val Glu Asp Glu Lys Arg Ala Glu Asp Tyr Lys
35 40 45
Gly Val Lys Lys Leu Leu Asp Arg Tyr Tyr Leu Ser Phe Ile Asn Asp
50 55 60
Val Leu His Ser Ile Lys Leu Lys Asn Leu Asn Asn Tyr Ile Ser Leu
65 70 75 80
Phe Arg Lys Lys Thr Arg Thr Glu Lys Glu Asn Lys Glu Leu Glu Asn
85 90 95
Leu Glu Ile Asn Leu Arg Lys Glu Ile Ala Lys Ala Phe Lys Gly Asn
100 105 110
Glu Gly Tyr Lys Ser Leu Phe Lys Lys Asp Ile Ile Glu Thr Ile Leu
115 120 125
Pro Glu Phe Leu Asp Asp Lys Asp Glu Ile Ala Leu Val Asn Ser Phe
130 135 140
Asn Gly Phe Thr Thr Ala Phe Thr Gly Phe Phe Asp Asn Arg Glu Asn
145 150 155 160
Met Phe Ser Glu Glu Ala Lys Ser Thr Ser Ile Ala Phe Arg Cys Ile
165 170 175
Asn Glu Asn Leu Thr Arg Tyr Ile Ser Asn Met Asp Ile Phe Glu Lys
180 185 190
Val Asp Ala Ile Phe Asp Lys His Glu Val Gln Glu Ile Lys Glu Lys
195 200 205
Ile Leu Asn Ser Asp Tyr Asp Val Glu Asp Phe Phe Glu Gly Glu Phe
210 215 220
Phe Asn Phe Val Leu Thr Gln Glu Gly Ile Asp Val Tyr Asn Ala Ile
225 230 235 240
Ile Gly Gly Phe Val Thr Glu Ser Gly Glu Lys Ile Lys Gly Leu Asn
245 250 255
Glu Tyr Ile Asn Leu Tyr Asn Gln Lys Thr Lys Gln Lys Leu Pro Lys
260 265 270
Phe Lys Pro Leu Tyr Lys Gln Val Leu Ser Asp Arg Glu Ser Leu Ser
275 280 285
Phe Tyr Gly Glu Gly Tyr Thr Ser Asp Glu Glu Val Leu Glu Val Phe
290 295 300
Arg Asn Thr Leu Asn Lys Asn Ser Glu Ile Phe Ser Ser Ile Lys Lys
305 310 315 320
Leu Glu Lys Leu Phe Lys Asn Phe Asp Glu Tyr Ser Ser Ala Gly Ile
325 330 335
Phe Val Lys Asn Gly Pro Ala Ile Ser Thr Ile Ser Lys Asp Ile Phe
340 345 350
Gly Glu Trp Asn Val Ile Arg Asp Lys Trp Asn Ala Glu Tyr Asp Asp
355 360 365
Ile His Leu Lys Lys Lys Ala Val Val Thr Glu Lys Tyr Glu Asp Asp
370 375 380
Arg Arg Lys Ser Phe Lys Lys Ile Gly Ser Phe Ser Leu Glu Gln Leu
385 390 395 400
Gln Glu Tyr Ala Asp Ala Asp Leu Ser Val Val Glu Lys Leu Lys Glu
405 410 415
Ile Ile Ile Gln Lys Val Asp Glu Ile Tyr Lys Val Tyr Gly Ser Ser
420 425 430
Glu Lys Leu Phe Asp Ala Asp Phe Val Leu Glu Lys Ser Leu Lys Lys
435 440 445
Asn Asp Ala Val Val Ala Ile Met Lys Asp Leu Leu Asp Ser Val Lys
450 455 460
Ser Phe Glu Asn Tyr Ile Lys Ala Phe Phe Gly Glu Gly Lys Glu Thr
465 470 475 480
Asn Arg Asp Glu Ser Phe Tyr Gly Asp Phe Val Leu Ala Tyr Asp Ile
485 490 495
Leu Leu Lys Val Asp His Ile Tyr Asp Ala Ile Arg Asn Tyr Val Thr
500 505 510
Gln Lys Pro Tyr Ser Lys Asp Lys Phe Lys Leu Tyr Phe Gln Asn Pro
515 520 525
Gln Phe Met Gly Gly Trp Asp Lys Asp Lys Glu Thr Asp Tyr Arg Ala
530 535 540
Thr Ile Leu Arg Tyr Gly Ser Lys Tyr Tyr Leu Ala Ile Met Asp Lys
545 550 555 560
Lys Tyr Ala Lys Cys Leu Gln Lys Ile Asp Lys Asp Asp Val Asn Gly
565 570 575
Asn Tyr Glu Lys Ile Asn Tyr Lys Leu Leu Pro Gly Pro Asn Lys Met
580 585 590
Leu Pro Lys Val Phe Phe Ser Lys Lys Trp Met Ala Tyr Tyr Asn Pro
595 600 605
Ser Glu Asp Ile Gln Lys Ile Tyr Lys Asn Gly Thr Phe Lys Lys Gly
610 615 620
Asp Met Phe Asn Leu Asn Asp Cys His Lys Leu Ile Asp Phe Phe Lys
625 630 635 640
Asp Ser Ile Ser Arg Tyr Pro Lys Trp Ser Asn Ala Tyr Asp Phe Asn
645 650 655
Phe Ser Glu Thr Glu Lys Tyr Lys Asp Ile Ala Gly Phe Tyr Arg Glu
660 665 670
Val Glu Glu Gln Gly Tyr Lys Val Ser Phe Glu Ser Ala Ser Lys Lys
675 680 685
Glu Val Asp Lys Leu Val Glu Glu Gly Lys Leu Tyr Met Phe Gln Ile
690 695 700
Tyr Asn Lys Asp Phe Ser Asp Lys Ser His Gly Thr Pro Asn Leu His
705 710 715 720
Thr Met Tyr Phe Lys Leu Leu Phe Asp Glu Asn Asn His Gly Gln Ile
725 730 735
Arg Leu Ser Gly Gly Ala Glu Leu Phe Met Arg Arg Ala Ser Leu Lys
740 745 750
Lys Glu Glu Leu Val Val His Pro Ala Asn Ser Pro Ile Ala Asn Lys
755 760 765
Asn Pro Asp Asn Pro Lys Lys Thr Thr Thr Leu Ser Tyr Asp Val Tyr
770 775 780
Lys Asp Lys Arg Phe Ser Glu Asp Gln Tyr Glu Leu His Ile Pro Ile
785 790 795 800
Ala Ile Asn Lys Cys Pro Lys Asn Ile Phe Lys Ile Asn Thr Glu Val
805 810 815
Arg Val Leu Leu Lys His Asp Asp Asn Pro Tyr Val Ile Gly Ile Ala
820 825 830
Arg Gly Glu Arg Asn Leu Leu Tyr Ile Val Val Val Asp Gly Lys Gly
835 840 845
Asn Ile Val Glu Gln Tyr Ser Leu Asn Glu Ile Ile Asn Asn Phe Asn
850 855 860
Gly Ile Arg Ile Lys Thr Asp Tyr His Ser Leu Leu Asp Lys Lys Glu
865 870 875 880
Lys Glu Arg Phe Glu Ala Arg Gln Asn Trp Thr Ser Ile Glu Asn Ile
885 890 895
Lys Glu Leu Lys Ala Gly Tyr Ile Ser Gln Val Val His Lys Ile Cys
900 905 910
Glu Leu Val Glu Lys Tyr Asp Ala Val Ile Ala Leu Ala Asp Leu Asn
915 920 925
Ser Gly Phe Lys Asn Ser Arg Val Lys Val Glu Lys Gln Val Tyr Gln
930 935 940
Lys Phe Glu Lys Met Leu Ile Asp Lys Leu Asn Tyr Met Val Asp Lys
945 950 955 960
Lys Ser Asn Pro Cys Ala Thr Gly Gly Ala Leu Lys Gly Tyr Gln Ile
965 970 975
Thr Asn Lys Phe Glu Ser Phe Lys Ser Met Ser Thr Gln Asn Gly Phe
980 985 990
Ile Phe Tyr Ile Pro Ala Trp Leu Thr Ser Lys Ile Asp Pro Ser Thr
995 1000 1005
Gly Phe Val Asn Leu Leu Lys Thr Lys Tyr Thr Ser Ile Ala Asp Ser
1010 1015 1020
Lys Lys Phe Ile Ser Ser Phe Asp Arg Ile Met Tyr Val Pro Glu Glu
1025 1030 1035 1040
Asp Leu Phe Glu Phe Ala Leu Asp Tyr Lys Asn Phe Ser Arg Thr Asp
1045 1050 1055
Ala Asp Tyr Ile Lys Lys Trp Lys Leu Tyr Ser Tyr Gly Asn Arg Ile
1060 1065 1070
Arg Ile Phe Arg Asn Pro Lys Lys Asn Asn Val Phe Asp Trp Glu Glu
1075 1080 1085
Val Cys Leu Thr Ser Ala Tyr Lys Glu Leu Phe Asn Lys Tyr Gly Ile
1090 1095 1100
Asn Tyr Gln Gln Gly Asp Ile Arg Ala Leu Leu Cys Glu Gln Ser Asp
1105 1110 1115 1120
Lys Ala Phe Tyr Ser Ser Phe Met Ala Leu Met Ser Leu Met Leu Gln
1125 1130 1135
Met Arg Asn Ser Ile Thr Gly Arg Thr Asp Val Ala Phe Leu Ile Ser
1140 1145 1150
Pro Val Lys Asn Ser Asp Gly Ile Phe Tyr Asp Ser Arg Asn Tyr Glu
1155 1160 1165
Ala Gln Glu Asn Ala Ile Leu Pro Lys Asn Ala Asp Ala Asn Gly Ala
1170 1175 1180
Tyr Asn Ile Ala Arg Lys Val Leu Trp Ala Ile Gly Gln Phe Lys Lys
1185 1190 1195 1200
Ala Glu Asp Glu Lys Leu Asp Lys Val Lys Ile Ala Ile Ser Asn Lys
1205 1210 1215
Glu Trp Leu Glu Tyr Ala Gln Thr Ser Val Lys His
1220 1225
- 腺苷脱氨酶、碱基编辑器融合蛋白、碱基编辑器系统及用途
- 非靶脱氨反应减低的核碱基编辑器和用于定性核碱基编辑器的测定
