糖基转移酶、基因、载体、宿主细胞和糖基化方法
文献发布时间:2023-06-19 18:32:25
技术领域
本发明涉及一种糖基转移酶及其编码基因、载体和宿主细胞,还涉及一种芳香醇类化合物的糖基化方法。
背景技术
芳香类化合物具有抗氧化、降血脂、抗骨质疏松、抗肿瘤、抗病毒、抗菌等广泛的药理活性,在天然药物研究中占重要地位。其中,简单芳香类化合物如单环或多环芳香醇类化合物,除本身为药用活性化合物外,也往往作为重要的结构修饰前体或化学合成中间体,参与到结构多样性芳香类化合物的合成中,对于药物先导化合物的发现及结构修饰具有重要作用。例如,单环芳香醇类化合物酪醇除可抗炎、改善心脑血管疾病外,也是要用活性化合物红景天苷的合成前体。
糖基化修饰是天然产物结构修饰中重要的一种。糖基基团的引入能够增加天然产物的结构和功能的多样性,还能够提高不易溶解的化合物的水溶性,进而增强其生物利用度和成药性。
芳香醇类化合物的糖基化修饰方法主要有化学法和生物法。化学法的合成步骤较为繁琐,在合成过程中不可避免地涉及到糖链引入位点、数目、连接方式及保护和脱保护等问题,反应条件苛刻,成本较高,无法应用于大量生产。生物法具有反应选择性强、催化效率高、反应条件温和、副产物少、环境友好、后处理简单等优点,是对芳香醇类化合物进行糖基化修饰的理想方法。
CN104774815A公开了一种催化天麻素或红景天苷合成的糖基转移酶。但是,该糖基转移酶除了将芳香醇侧链醇羟基糖苷化外,还能催化相同底物在芳环酚羟基发生葡萄糖基化生成天麻素。因此,该糖基转移酶催化位置的特异性一般,催化反应中有副产物的生成,也未表现出显著的底物与糖基供体选择宽泛性。
发明内容
有鉴于此,本发明的一个目的在于提供一种糖基转移酶,该糖基转移酶的催化位点具有特异性,其催化位点特异性地发生在芳香醇类化合物的侧链醇羟基上,且具有催化底物和糖基供体的选择杂泛性。本发明的另一个目的在于提供一种编码上述糖基转移酶的核苷酸序列。本发明的再一个目的在于提供一种含有上述核苷酸序列的重组表达载体。本发明的又一个目的在于提供一种含有核苷酸序列或重组表达载体的重组宿主细胞。本发明的又一个目的在于提供一种芳香醇类化合物的糖基化方法,该方法糖基化催化位点特异性强,催化底物适用广泛。
本发明采用如下技术方案实现上述目的。
一方面,本发明提供了一种糖基转移酶,所述糖基转移酶的氨基酸序列如SEQ IDNO:1所示,或者该序列经替换、缺失或添加1~20个氨基酸形成的具有同等功能的氨基酸序列。
本发明还提供了一种糖基转移酶,所述糖基转移酶的氨基酸序列如SEQ ID NO:4或SEQ ID NO:5所示。
另一方面,本发明提供了一种编码上述糖基转移酶的核苷酸序列。
根据本发明的核苷酸序列,优选地,所述氨基酸序列如SEQ IDNO:1所示,所述核苷酸序列如SEQ ID NO:2或SEQ ID NO:3所示。
再一方面,本发明提供了一种重组表达载体,所述重组表达载体含有上述核苷酸序列。
根据本发明的重组表达载体,优选地,所使用的表达载体为pET-28a。
又一方面,本发明提供了一种重组宿主细胞,所述重组宿主细胞含有上述核苷酸序列或上述重组表达载体。
根据本发明的重组宿主细胞,优选地,所述重组宿主细胞为重组的大肠杆菌细胞。
又一方面,本发明提供了一种芳香醇类化合物的糖基化方法,包括使包含上述糖基转移酶、芳香醇类糖基受体和糖基供体的反应体系发生糖基化反应。
根据本发明的糖基化方法,优选地,所述糖基供体为核苷二磷酸化的含糖基化合物;芳香醇类糖基受体的芳香环选自苯、联苯、苄基苯、1,2,3,4-四氢萘、苯并环戊烷、苊、芴、蒽、芘中的一种。
本发明从管花肉苁蓉中克隆得到了糖基转移酶(SEQ ID NO:1),该糖基转移酶能够特异性地催化单环芳香醇类化合物的侧链醇羟基发生糖基化反应,且能够催化不同底物和糖基受体的糖基化反应。上述糖基转移酶N端截短12个氨基酸形成的糖基转移酶(SEQ IDNO:4)具有更高的催化效率。本发明还发现,在N端截短12个氨基酸形成的序列的基础上将159位的酪氨酸突变为缬氨酸形成的糖基转移酶(SEQ ID NO:5),进一步拓宽了催化底物,其不但能催化单环芳香醇类化合物还能够催化多环及稠环芳香醇类化合物。本发明还公开了优化后的核苷酸序列(SEQ ID NO:3),其蛋白表达量明显增加。
附图说明
图1为管花肉苁蓉总RNA琼脂糖凝胶电泳图。
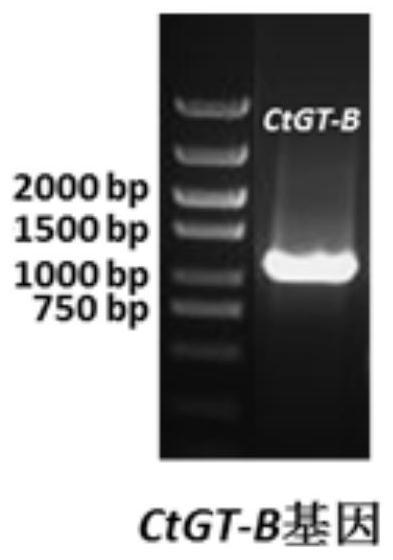
图2为糖基转移酶CtGT-B基因编码区琼脂糖凝胶电泳图。
图3为异源表达得到的糖基转移酶CtGT-B的SDS-PAGE电泳图。分别以20mM、30mM、70mM、110mM、160mM、250mM、300mM咪唑进行梯度洗脱,每2个柱体积接作一个流分,每个梯度冲10个柱体积。其中,a-b中a表示咪唑浓度,b表示该浓度洗脱流分。b部分为1时,表示a咪唑浓度下洗脱第1-2个柱体积时所接作的流分;b部分为2时,表示a咪唑浓度下洗脱第3-4个柱体积时所接作的流分,以此类推。例如,30-3表示30mM咪唑浓度下洗脱第5-6个柱体积时所接作的流分。
图4为糖基转移酶CtGT-Bopt-12aa和CtGT-Bopt-12aa-Y159V的SDS-PAGE电泳图。
图5A为糖基转移酶CtGT-B催化酪醇和UDP-葡萄糖糖基化反应的HPLC分析图,小图为红景天苷的DAD紫外特征吸收光谱图。
图5B为糖基转移酶CtGT-B催化酪醇和UDP-葡萄糖糖基化反应所得产物的分子离子质谱图。
图5C为糖基转移酶CtGT-B催化酪醇和UDP-葡萄糖糖基化反应所得产物的
图6A为糖基转移酶CtGT-Bopt催化酪醇和UDP-葡萄糖糖基化反应的HPLC分析图。
图6B为糖基转移酶CtGT-Bopt-12aa催化酪醇和UDP-葡萄糖糖基化反应的HPLC分析图。
图7为糖基转移酶CtGT-Bopt-12aa-Y159V催化1-萘乙醇不同糖基供体糖基化反应的HPLC-MS分析图;图a,b,c为HPLC-MS分析图,其中图a糖基供体为UDP-葡萄糖,图b糖基供体为UDP-N-乙酰葡糖胺,图c糖基供体为UDP-半乳糖,图d为1-萘乙醇的结构式及HPLC分析图。
图8为糖基转移酶CtGT-Bopt催化2-甲基苯乙醇葡萄糖基化产物核磁图谱(表2序号7-2);其中,图8A为以2-甲基苯乙醇为受体,以UDP-葡萄糖为供体,糖基化产物
图9为糖基转移酶CtGT-Bopt-12aa-Y159V以1-(2-萘基)乙醇为葡萄糖基化产物核磁图谱(表2序号7-22);图9A为以1-(2-萘基)乙醇为受体,以UDP-葡萄糖为供体,糖基化产物
具体实施方式
下面结合具体实施例对本发明作进一步的说明,但本发明的保护范围并不限于此。
<糖基转移酶及核苷酸序列>
本发明首次从管花肉苁蓉中克隆获得一种具有糖基转移催化功能的酶,并进一步筛选、鉴定及优化,得到本发明的糖基转移酶及编码本发明的糖基转移酶的核苷酸序列。
本发明首次从植物管花肉苁蓉(学名:Cistanche tubulosa(Schenk)Wight)中筛选鉴定得到一种糖基转移酶。本发明的糖基转移酶可为天然蛋白酶或包含突变且仍具有催化糖基转移活性的酶。优选地,本发明的糖基转移酶包含SEQ ID NO:1所示的氨基酸序列,或者该序列经替换、缺失或添加一个或几个氨基酸形成的具有同等功能的氨基酸序列。在某些实施方案中,糖基转移酶的氨基酸序列与SEQ ID NO:1所示序列具有90%以上,优选92%以上,更优选95%以上,进一步优选98%以上,还优选99%以上的同源性且来源于相同的物种管花肉苁蓉。本申请中“同源性”是指两个序列之间的相似性,可通过本领域已知的任何算法来确定。例如,两个氨基酸序列之间的同一性程度可使用Needleman-Wunsch算法确定。优选地,本发明的糖基转移酶的分子量为53.89kDa。理论等电点为5.39,表明其为酸性蛋白。不稳定系数(instability index)为45.73,表明该蛋白可能不稳定。疏水性平均值(Grand average of hydropathicity,GRAVY)为-0.145,表明其为亲水性蛋白,蛋白由482个氨基酸组成。氨基酸序列SEQ ID NO:1以下有时简称为“CtGT-B”或“CtGT-Bopt”。
根据本发明一个优选的技术方案,将N端截短12个氨基酸得到氨基酸序列SEQ IDNO:4(以下有时简称为“CtGT-Bopt-12aa”)。由SEQ ID NO:4氨基酸序列编码的糖基转移酶具有更高的催化效率。
根据本发明另一个优选的技术方案,将SEQ ID NO:4氨基酸序列159位的酪氨酸突变为缬氨酸,得到氨基酸序列SEQ ID NO:5(以下有时简称为“CtGT-Bopt-12aa-Y159V”)。由氨基酸序列SEQ ID NO:5编码的糖基转移酶进一步拓展了催化底物的范围,其能够催化单环、多环及稠环芳香醇类化合物发生多种糖基化反应。
一种编码上述糖基转移酶的核苷酸序列。所述糖基转移酶的氨基酸序列可以如SEQ ID NO:1所示。所述核苷酸序列可以如SEQ IDNO:2或SEQ ID NO:3所示。核苷酸序列SEQID NO:3是在SEQ IDNO:2核苷酸序列的基础上通过大肠杆菌密码子偏爱性优化后得到的核苷酸序列,其蛋白表达量较SEQ ID NO:2明显增加。核苷酸序列SEQID NO:3和核苷酸序列SEQ ID NO:2所编码的氨基酸序列相同。由SEQ ID NO:2编码的核苷酸序列得到的糖基转移酶以下有时简称为“CtGT-B”。由SEQ ID NO:3编码的核苷酸序列得到的糖基转移酶以下有时简称为“CtGT-Bopt”。
<重组表达载体及重组宿主细胞>
将编码上述糖基转移酶的核苷酸序列克隆到表达载体中,构建重组表达载体。所使用的表达载体可以为pET-28a。
将编码上述糖基转移酶的核苷酸序列或上述重组表达载体用于在宿主细胞中表达,形成重组宿主细胞。所使用的宿主细胞可以为大肠杆菌。
<糖基化方法>
一种芳香醇类化合物的糖基化方法,包括使上述糖基转移酶、芳香醇类糖基受体和糖基供体的反应体系发生糖基化反应。本发明的反应体系中还可以包含缓冲液。
糖基供体可以为核苷二磷酸化的含糖基化合物;优选地,糖基供给选自UDP-葡萄糖、UDP-N-乙酰葡糖胺或UDP-半乳糖中的一种或多种;更优选地,糖基供体为UDP-葡萄糖。
当糖基转移酶的氨基酸序列为SEQ ID NO:1(亦即“CtGT-B”或“CtGT-Bopt”)或SEQID NO:4(亦即“CtGT-Bopt-12aa”)时,芳香醇类糖基受体的芳香环为苯环。苯环经脂肪族羟基烷基或脂肪族羟基烯基取代形成芳香醇。脂肪族羟基烷基可以为直链脂肪族羟基烷基,也可以为支链脂肪族羟基烷基。脂肪族羟基烷基可以具有1~10个碳原子,优选为1~6个碳原子,更优选为1~3个碳原子。脂肪族羟基烯基优选为直链脂肪族羟基烯基。脂肪族羟基烯基优选为脂肪族羟基单烯基。脂肪族羟基烯基可以具有3~10个碳原子,优选地,具有3~6个碳原子,更优选地,具有3~4个碳原子。除羟烷基或羟基烯基外苯环上还可以具有一个或多个取代基。取代基的位置可以在苯环的2、3或4位上。苯环上的取代基可以选自羟基、C1~C3的烷基、卤素、C1~C3的烷氧基、硝基、氨基。C1~C3的烷基的实例包括但不限于甲基、乙基、丙基。C1~C3的烷氧基的实例包括但不限于甲氧基、乙氧基、丙氧基。卤素的实例包括但不限于氟、氯、溴。
在某些实施方式中,芳香醇类糖基受体选自具有如下结构的化合物之一:
(A)
其中,R选自羟基、甲氧基、甲基、硝基、氨基、氟、氯、溴、氢,n选自1~10的整数,优选为1~6的整数,更优选为1~3的整数;
(B)
根据本发明的一个实施方式,芳香醇类糖基受体选自如下所示的化合物之一:
当糖基转移酶的氨基酸序列为SEQ ID NO:5(亦即“CtGT-Bopt-12aa-Y159V”)时,芳香醇类糖基受体除上述单环芳香醇类化合物外,还可以为多环及稠环芳香醇类化合物。多环及稠环芳香醇类化合物的芳香环可以选自联苯、苄基苯、1,2,3,4-四氢萘、苯并环戊烷、苊、芴、蒽、芘中的一种。
根据本发明的一个实施方式,多环及稠环芳香醇类化合物具有如下所示的结构之一:
在某些实施方案中,本发明反应体系还可以包含缓冲液。缓冲液包含30~70mMTris-HCl,0.5~3mM DTT,0.5~3vol%甘油,70~120mM NaCl。根据本发明的一个实施方式,缓冲液由50mM Tris-HCl,1mM DTT,1.5vol%甘油,100mM NaCl组成。缓冲液中各物质的用量可以按照上述比例放大或缩小。
在某些实施方案中,反应体系以150μL计,包含0.01~0.15μM芳香醇类化合物、3~50μg糖基转移酶和0.05~0.20μM糖基供体,缓冲液补足至150μL,及按等比例放大的反应体系。
根据本发明一个优选的实施方式,反应体系以150μL计,包含0.04μM芳香醇类化合物、10μg糖基转移酶和0.08μM糖基供体,缓冲液补足至150μL,及按等比例放大的反应体系。
根据本发明另一个优选的实施方式,反应体系以150μL计,包含0.04μM芳香醇类化合物、20μg糖基转移酶和0.08μM糖基供体,缓冲液补足至150μL,及按等比例放大的反应体系。
在某些实施方案中,反应体系以75mL计,包含0.01~0.15mM芳香醇类化合物、3~50mg糖基转移酶和0.05~0.20mM糖基供体,缓冲液补足至75mL,及按等比例放大的反应体系。
根据本发明一个优选的实施方式,反应体系以75mL计,包含0.06mM芳香醇类化合物、30mg糖基转移酶和0.08mM糖基供体,缓冲液补足至75mL,及按等比例放大的反应体系。
本发明在能够使芳香醇类化合物、糖基转移酶和糖基供体糖基化反应的条件下进行。反应温度可以为20~40℃,优选为25~35℃,更优选为30℃。反应时间可以为10~50小时,优选为11~48小时,更优选为12小时。
以下实施例所使用的缓冲液除另有提及外,其组成为50mM Tris-HCl,1mM DTT,1.5vol%甘油,100mM NaCl。
选取管花肉苁蓉新鲜肉质茎,采用液氮速冻研磨法,利用Omega植物RNA提取试剂盒进行总RNA的提取。利用ClonTechSMARTer RACE 5′/3′Kit分别反转录获得5′-RACE-cDNA,3′-RACE-cDNA及总cDNA。
CtGT-B基因5′端和3′端序列的克隆使用Clontech SMARTer
PCR反应程序为:94℃预变性2min,然后94℃变性15s;起始65℃退火30s,每个循环降低0.5℃,68℃延伸50s,共30个循环;紧接着94℃变性15s,54℃退火30s,68℃延伸50s,共25个循环;最后72℃延伸1min。
将测序所得的5′RACE和3′RACE序列进行拼接,得到全长cDNA序列。利用NCBI OpenReading Frame(ORF)Finder分析基因的开放读码框,并分析设计一对带有酶切位点的特异性引物,序列如SEQ ID NO:8,9所示,其中SEQ ID NO:8通过引物设计引入BamH I酶切位点,SEQ ID NO:9通过引物设计引入Not I酶切位点。利用特异性引物SEQ ID NO:8和SEQ IDNO:9扩增得到约1449bp的编码区序列全长,序列如SEQ ID NO:2所示,编码氨基酸序列如SEQ IDNO:1所示,琼脂糖凝胶电泳如附图2所示。
SEQ ID NO:1利用Expasy进行蛋白的理化性质预测,显示其分子量为53.89KDa;理论等电点为5.39,表明其为酸性蛋白;不稳定系数(instability index)为45.73,表明该蛋白可能不稳定;疏水性平均值(Grand average of hydropathicity,GRAVY)为-0.145,表明其为亲水性蛋白,蛋白由482个氨基酸组成。
将实施例1中扩增获得的基因片段进行胶回收,胶回收产物用BamH I和Not I进行双酶切,使用Takara限制性内切酶,37℃酶切2~3h后,胶回收酶切片段,与同样方法双酶切后的pET-28a表达载体进行T4连接。利用NEB T4 DNA Ligase将目的基因连接至pET28a上。连接产物转化感受态细胞,选择E.coli BL21(DE3)作为表达菌株。通过菌落PCR筛选阳性克隆,取测序正确的菌株接种于含有Kana 50μg/mL,Chl 50μg/mL的LB液体培养基中,在37℃,200rpm的恒温摇床中震荡培养至菌液OD600值为0.6,加入诱导剂IPTG至终浓度为0.15mM,23℃,180rpm条件下培养20h,低温诱导目的蛋白表达。
利用镍离子亲和层析柱(Ni Sepharose 6Fast Flow,GE Healthcare)纯化CtGT-B蛋白表达,纯化结果如图3所示,目的蛋白大小在50~55kDa,与其理论大小53.89kDa相一致,160~250mM咪唑浓度下目的蛋白大量洗脱获得单一条带蛋白。合并目的蛋白流份于PD-10column中进行蛋白浓缩。利用Transgene公司Easy Protein QuantitativeKit使用Bradford方法测定。蛋白表达量为6.0mg/L菌体。
CtGT-Bopt基因序列是基于CtGT-B核苷酸序列的基础上,通过大肠杆菌密码子偏爱性优化后的序列(SEQ ID NO:3),其编码氨基酸具有与CtGT-B蛋白相同的序列,其表达载体的构建和原核表达流程同实施例2。利用Transgene公司Easy Protein QuantitativeKit使用Bradford方法测定。蛋白表达量为10.0mg/L菌体。
截短序列CtGT-Bopt-12aa的扩增以CtGT-Bopt基因为模板,截短后突变体CtGT-Bopt-12aa-Y159V的构建使用Transgene mutagenesis试剂盒,使用引物序列如SEQ ID NO:10,11所示。
截短后突变体CtGT-Bopt-12aa-Y159V蛋白表达纯化结果如附图4所示,其蛋白大小在52.6kDa,蛋白表达量相比CtGT-B蛋白有了明显的提高,可达13mg/L菌体。蛋白浓度利用Transgene公司Easy ProteinQuantitative Kit使用Bradford方法测定。
利用镍离子亲和层析纯化的CtGT-B蛋白进行酶反应实验。酶反应体系为:10μgCtGT-B蛋白,0.08μM UDP-葡萄糖,0.04μM酪醇;缓冲液补足至150μL。轻弹混匀,30℃水浴反应12h,于体系中加入300μL甲醇终止反应。12000×g离心除去沉淀,上清取出减压蒸干溶剂,残渣溶于150μL甲醇中,0.2μm滤膜过滤后用于HPLC-HR-MS分析。
Agilent 1260液相色谱检测条件为:色谱柱SHESHIDO CAPCELLPAK MG-IIICOLUMN(4.6mml.D.×250mm,5μm),流速1.0mL/min,DAD检测器全波长扫描,流动相为乙腈-0.1%甲酸水梯度洗脱:0~10min,5%~20%乙腈;10~15min,20%~25%乙腈;15~25min,25%~35%乙腈;25~30min,35%~95%乙腈;30~35min,100%乙腈。质谱条件为:LCMS-IT-TOF(Shimadzu,Japan)检测,正、负离子模式;雾化气体(nebulizing gas)为N
利用镍离子亲和层析纯化的CtGT-Bopt蛋白和N端截短12个氨基酸的CtGT-Bopt-12aa蛋白分别进行酶反应实验。
CtGT-Bopt蛋白酶反应体系为:20μg CtGT-Bopt蛋白,0.08μMUDP-葡萄糖,0.04μM酪醇;缓冲液补足至150μL。30℃水浴反应12h,于体系中加入300μL甲醇终止反应。12000×g离心除去沉淀,上清取出减压蒸干溶剂,残渣溶于150μL甲醇中,0.2μm滤膜过滤后用于HPLC-HR-MS分析。HPLC-HR-MS分析条件同实施例4。HPLC-HR-MS检测数据如附图6A所示。红景天苷的转化率为62.85%。
CtGT-Bopt-12aa蛋白酶反应体系为:20μgCtGT-Bopt-12aa蛋白,0.08μM UDP-葡萄糖,0.04μM酪醇;缓冲液补足至150μL。30℃水浴反应12h,于体系中加入300μL甲醇终止反应。12000×g离心除去沉淀,上清取出减压蒸干溶剂,残渣溶于150μL甲醇中,0.2μm滤膜过滤后用于HPLC-HR-MS分析。HPLC-HR-MS分析条件同实施例4。HPLC-HR-MS检测数据如附图6B所示。红景天苷的转化率为89.19%。
上述结果表明CtGT-Bopt-12aa蛋白的催化活性及催化反应类型与CtGT-Bopt蛋白相同,但催化效率较CtGT-Bopt蛋白具有显著提高。
酶反应体系为:20μg蛋白、0.08μM糖基供体、0.04μM 1-萘乙醇;缓冲液补足至150μL。30℃水浴反应12h,于体系中加入300μL甲醇终止反应。12000×g离心除去沉淀,上清取出减压蒸干溶剂,残渣溶于150μL甲醇中,0.2μm滤膜过滤后用HPLC-HR-MS分析。HPLC-HR-MS分析条件同实施例4。实施例6a-6c中蛋白和糖基供体如表1所示。HPLC-HR-MS分析结果如7所示。
表1
反应体系为:0.06mM糖基受体(溶于DMSO),0.08mM UDP-葡萄糖,30mg蛋白;反应缓冲液(50mM Tris-HCl,1mM DTT,1.5%甘油,100mM NaCl)补充至75mL。30℃反应12小时,反应液过0.45μm滤膜,滤液以<1mL/min的流速上样于MCI GELCHP系列树脂(1cm×15cm),20倍柱体积水洗脱杂质,依次以10倍体积20%、40%、60%、80%甲醇-水溶液梯度洗脱,最后以100%甲醇冲柱。各流份取样进行HPLC检测,合并目标产物流份,减压蒸干溶剂,残渣溶于甲醇,过0.45μm滤膜。采用岛津半制备液相色谱仪进一步纯化富集产物,制备柱YMC-PackODS-AHPLC COLUMN(10mml.D.×250mm,S-5μm,12nm),洗脱系统采用乙腈-水梯度洗脱:0~5min,5%~15%乙腈;5~10min,15%乙腈;10~15min,15%~18%乙腈;15~30min,18%~20%乙腈;30~35min,20%~75%乙腈;35~40min 90%~100%乙腈,手动进样,流速3mL/min。制备产物采用Varian 500核磁共振仪进行结构鉴定(图8A-9B)。糖基受体及蛋白如表2所示。
表2
本发明并不限于上述实施方式,在不背离本发明的实质内容的情况下,本领域技术人员可以想到的任何变形、改进、替换均落入本发明的范围。
序列表
<110> 北京中医药大学
<120> 糖基转移酶、基因、载体、宿主细胞和糖基化方法
<160> 11
<170> SIPOSequenceListing 1.0
<210> 1
<211> 482
<212> PRT
<213> 管花肉苁蓉( Cistanche tubulosa)
<400> 1
Met Gly Ser Leu Thr Lys Ile Ala Ala Thr Asn Glu Lys Pro His Ala
1 5 10 15
Val Cys Ile Pro Tyr Pro Ala Gln Gly His Ile Asn Pro Met Leu Lys
20 25 30
Leu Ala Lys Ile Leu His Ser Arg Gly Phe His Ile Thr Phe Val Asn
35 40 45
Thr Glu Tyr Asn His Asn Arg Leu Leu Arg Ser Arg Gly Pro Gly Ala
50 55 60
Leu Gln Gly Leu Pro Ser Phe Arg Phe Glu Thr Ile Thr Asp Gly Leu
65 70 75 80
Pro Pro Thr Asp Ala Asp Ala Thr Gln Ser Ile Pro Glu Leu Cys Arg
85 90 95
Ser Thr Glu Leu His Ser Leu Gly Pro Phe Arg Asp Leu Leu Arg Arg
100 105 110
Leu Asn Asp Ser Gly Val Val Pro Pro Val Ser Cys Ile Val Ser Asp
115 120 125
Ser Ala Met Phe Phe Thr Leu Asp Ala Ala Glu Glu Leu Gly Val Pro
130 135 140
Glu Val Leu Leu Trp Thr Ala Ser Ala Cys Gly Phe Leu Gly Tyr Thr
145 150 155 160
Gln Tyr Asp Gln Leu Val Glu Leu Gly Leu Thr Pro Phe Lys Asp Thr
165 170 175
Asn Phe Leu Thr Asn Gly Asp Leu Asp Lys Val Leu Asp Trp Val Pro
180 185 190
Ala Met Lys Gly Ile Arg Leu Arg Asp Ile Pro Ser Phe Ile Arg Thr
195 200 205
Thr Asp Pro Asp Glu Phe Met Val Lys Tyr Val Arg Arg Leu Val Tyr
210 215 220
Gln Ser Lys Arg Ala Ser Ala Ile Leu Phe Asn Thr Phe Asp Ala Leu
225 230 235 240
Glu Gly Asp Val Leu Gln Ser Leu Ser Ser Asn Phe Pro Arg Val Tyr
245 250 255
Ser Leu Gly Pro Leu Gln Leu Leu Leu Asp Pro Ile Asp Lys Glu Thr
260 265 270
Lys Ser Ile Gly Ser Asn Leu Trp Lys Glu Asp Gln His Cys Ile Asp
275 280 285
Trp Leu Asp Ser His Gly Pro Asn Ser Val Val Tyr Val Asn Phe Gly
290 295 300
Ser Ile Thr Val Met Ser Asn Asp Gln Leu Val Glu Phe Ala Trp Gly
305 310 315 320
Leu Ala Asn Ser Gly Arg Pro Phe Leu Trp Ile Ala Arg Pro Asp Leu
325 330 335
Val Ile Gly Asp Ser Ala Val Leu Pro Pro Glu Phe Leu Glu Glu Thr
340 345 350
Arg Ser Arg Gly Leu Ile Ala Ser Trp Cys Asp Gln Glu Arg Ile Leu
355 360 365
Ala His Pro Ala Ile Gly Gly Phe Leu Thr His Cys Gly Trp Asn Ser
370 375 380
Ile Val Glu Ser Ile Cys Asn Gly Val Pro Val Val Cys Trp Pro Phe
385 390 395 400
Phe Ala Glu Gln Gln Thr Asn Cys Trp Tyr Ser Cys Thr Lys Trp Gly
405 410 415
Ile Gly Met Glu Ile Asp Pro Asn Val Lys Arg Glu Val Val Glu Arg
420 425 430
Gln Val Arg Glu Leu Met Leu Gly Glu Glu Gly Lys Glu Met Lys Arg
435 440 445
Lys Ala Met Glu Trp Lys Ala Leu Ala Gln Glu Ala Thr Thr Ser Ser
450 455 460
His Gly Ser Ser Tyr Ser Asn Met Asp Asn Leu Met Thr Arg Val Leu
465 470 475 480
Ser Pro
<210> 2
<211> 1449
<212> DNA/RNA
<213> 管花肉苁蓉( Cistanche tubulosa)
<400> 2
atgggttctt tgacaaagat agcagccaca aatgaaaaac cacatgcggt gtgtatccca 60
tacccagcac agggccacat aaaccccatg ctgaaactag ccaaaatcct ccattctcga 120
ggcttccata tcacctttgt aaacactgag tataatcaca accgtttgct gcggtctcgg 180
ggtcctggtg ctctccaagg cctccccagt ttccgtttcg agacgattac cgatggtctc 240
ccccctactg acgctgatgc cacccagagt atccctgagc tttgtcgctc caccgagctt 300
cactctctcg gtcccttccg tgacctcctt aggcgtctca atgacagcgg tgttgtgccc 360
cctgtgagct gcatcgtgtc cgattcggcc atgtttttca cccttgatgc tgctgaggag 420
cttggagtcc ctgaagtcct gctttggact gctagcgcct gtggtttctt gggttatact 480
cagtatgatc agcttgttga gttggggctt actcccttca aagatacaaa cttcctcaca 540
aacggtgatc tagacaaggt cttggattgg gttccggcta tgaagggcat ccggttgagg 600
gacatcccaa gcttcataag aaccactgac cctgacgagt ttatggttaa atatgttcga 660
cgactggttt accaatccaa gcgggcatcg gccatccttt tcaacacatt tgatgctttg 720
gagggcgacg ttctccaatc cctttcctcc aatttcccac gtgtctactc cctcggtcca 780
ctgcagttgc tgcttgaccc catcgacaag gagaccaaat caatcggatc gaatctttgg 840
aaagaggacc aacattgcat tgactggctt gattctcatg gccccaactc cgttgtgtat 900
gtcaatttcg ggagtattac agtcatgtct aatgatcagc tcgtggaatt tgcttgggga 960
ctcgctaata gcgggcgacc cttcctatgg atcgctcgac ctgatttggt gattggggat 1020
tctgcagtgc tcccacccga attcttggag gagaccagaa gcagaggact cattgctagc 1080
tggtgtgacc aggagcggat tttggcccac ccggcaatcg ggggattctt gacgcattgt 1140
gggtggaatt caatagtaga aagcatctgt aatggagtgc cagtggtatg ctggcctttc 1200
ttcgcggagc aacagaccaa ttgttggtat tcttgcacca agtgggggat tggtatggag 1260
attgatccca acgtgaagag ggaagtggtt gagaggcagg tgagggagct gatgttggga 1320
gaagaaggca aagagatgaa gagaaaggca atggagtgga aagccttggc tcaagaggcc 1380
accacttctt ctcatggctc ttcttactcc aacatggata atctcatgac cagagtactt 1440
agtccctaa 1497
<210> 3
<211> 1449
<212> DNA/RNA
<213> 人工序列( Artifical sequence)
<400> 3
atgggtagcc tgaccaaaat tgcagcaacc aatgaaaaac cgcatgcagt ttgtattccg 60
tatccggcac agggtcatat taatccgatg ctgaaactgg caaaaattct gcatagccgt 120
ggttttcata tcacctttgt taacaccgag tataaccata atcgtctgct gcgtagtcgt 180
ggtccgggtg cactgcaggg tctgccgagc tttcgttttg aaaccattac cgatggtctg 240
cctccgaccg atgcagatgc aacccagagc attccggaac tgtgtcgtag caccgaactg 300
catagtctgg gtccgtttcg tgacctgctg cgtcgtctga atgatagcgg tgttgttccg 360
cctgttagct gtattgttag cgatagcgca atgtttttta ccctggatgc agccgaagaa 420
ctgggtgttc cggaagttct gctgtggacc gcaagcgcat gtggttttct gggttatacc 480
cagtatgatc agctggttga actgggtctg accccgttta aagataccaa ttttctgacc 540
aatggcgacc tggataaagt tctggattgg gttccggcaa tgaaaggtat tcgtctgcgt 600
gatattccga gctttattcg taccaccgat ccggatgaat ttatggttaa atatgttcgt 660
cgtctggtgt atcagagcaa acgtgcaagc gcaattctgt ttaatacctt tgatgcactg 720
gaaggtgatg ttctgcagag cctgagcagc aattttccgc gtgtttattc actgggtccg 780
ctgcagctgc tgctggaccc gattgataaa gaaaccaaaa gcattggtag caacctgtgg 840
aaagaagatc agcattgtat tgattggctg gatagtcatg gtccgaatag cgttgtttat 900
gtgaattttg gtagcatcac cgtgatgagc aatgatcaac tggtggaatt tgcatggggt 960
ctggcaaata gcggtcgtcc gtttctgtgg attgcacgtc cggatctggt tattggtgat 1020
agcgcagttc tgccaccgga atttctggaa gaaacccgtt cacgtggtct gattgcaagc 1080
tggtgtgatc aagaacgtat tctggcacat ccggcaattg gtggctttct gacccattgt 1140
ggttggaata gcattgttga aagcatttgt aatggtgtgc cggttgtttg ttggccgttt 1200
tttgcagaac agcagaccaa ttgttggtat agctgcacca aatggggtat tggtatggaa 1260
attgatccga atgttaaacg cgaagttgtt gaacgtcagg ttcgtgaact gatgctgggt 1320
gaagagggta aagaaatgaa acgtaaagca atggaatgga aagcactggc acaagaagca 1380
accaccagtt cacatggtag cagctattca aatatggata atctgatgac ccgtgttctg 1440
agcccgtaa 1497
<210> 4
<211> 470
<212> PRT
<213> 人工序列( Artifical sequence)
<400> 4
Lys Pro His Ala Val Cys Ile Pro Tyr Pro Ala Gln Gly His Ile Asn
1 5 10 15
Pro Met Leu Lys Leu Ala Lys Ile Leu His Ser Arg Gly Phe His Ile
20 25 30
Thr Phe Val Asn Thr Glu Tyr Asn His Asn Arg Leu Leu Arg Ser Arg
35 40 45
Gly Pro Gly Ala Leu Gln Gly Leu Pro Ser Phe Arg Phe Glu Thr Ile
50 55 60
Thr Asp Gly Leu Pro Pro Thr Asp Ala Asp Ala Thr Gln Ser Ile Pro
65 70 75 80
Glu Leu Cys Arg Ser Thr Glu Leu His Ser Leu Gly Pro Phe Arg Asp
85 90 95
Leu Leu Arg Arg Leu Asn Asp Ser Gly Val Val Pro Pro Val Ser Cys
100 105 110
Ile Val Ser Asp Ser Ala Met Phe Phe Thr Leu Asp Ala Ala Glu Glu
115 120 125
Leu Gly Val Pro Glu Val Leu Leu Trp Thr Ala Ser Ala Cys Gly Phe
130 135 140
Leu Gly Tyr Thr Gln Tyr Asp Gln Leu Val Glu Leu Gly Leu Thr Pro
145 150 155 160
Phe Lys Asp Thr Asn Phe Leu Thr Asn Gly Asp Leu Asp Lys Val Leu
165 170 175
Asp Trp Val Pro Ala Met Lys Gly Ile Arg Leu Arg Asp Ile Pro Ser
180 185 190
Phe Ile Arg Thr Thr Asp Pro Asp Glu Phe Met Val Lys Tyr Val Arg
195 200 205
Arg Leu Val Tyr Gln Ser Lys Arg Ala Ser Ala Ile Leu Phe Asn Thr
210 215 220
Phe Asp Ala Leu Glu Gly Asp Val Leu Gln Ser Leu Ser Ser Asn Phe
225 230 235 240
Pro Arg Val Tyr Ser Leu Gly Pro Leu Gln Leu Leu Leu Asp Pro Ile
245 250 255
Asp Lys Glu Thr Lys Ser Ile Gly Ser Asn Leu Trp Lys Glu Asp Gln
260 265 270
His Cys Ile Asp Trp Leu Asp Ser His Gly Pro Asn Ser Val Val Tyr
275 280 285
Val Asn Phe Gly Ser Ile Thr Val Met Ser Asn Asp Gln Leu Val Glu
290 295 300
Phe Ala Trp Gly Leu Ala Asn Ser Gly Arg Pro Phe Leu Trp Ile Ala
305 310 315 320
Arg Pro Asp Leu Val Ile Gly Asp Ser Ala Val Leu Pro Pro Glu Phe
325 330 335
Leu Glu Glu Thr Arg Ser Arg Gly Leu Ile Ala Ser Trp Cys Asp Gln
340 345 350
Glu Arg Ile Leu Ala His Pro Ala Ile Gly Gly Phe Leu Thr His Cys
355 360 365
Gly Trp Asn Ser Ile Val Glu Ser Ile Cys Asn Gly Val Pro Val Val
370 375 380
Cys Trp Pro Phe Phe Ala Glu Gln Gln Thr Asn Cys Trp Tyr Ser Cys
385 390 395 400
Thr Lys Trp Gly Ile Gly Met Glu Ile Asp Pro Asn Val Lys Arg Glu
405 410 415
Val Val Glu Arg Gln Val Arg Glu Leu Met Leu Gly Glu Glu Gly Lys
420 425 430
Glu Met Lys Arg Lys Ala Met Glu Trp Lys Ala Leu Ala Gln Glu Ala
435 440 445
Thr Thr Ser Ser His Gly Ser Ser Tyr Ser Asn Met Asp Asn Leu Met
450 455 460
Thr Arg Val Leu Ser Pro
465 470
<210> 5
<211> 470
<212> PRT
<213> 人工序列( Artifical sequence)
<400> 5
Lys Pro His Ala Val Cys Ile Pro Tyr Pro Ala Gln Gly His Ile Asn
1 5 10 15
Pro Met Leu Lys Leu Ala Lys Ile Leu His Ser Arg Gly Phe His Ile
20 25 30
Thr Phe Val Asn Thr Glu Tyr Asn His Asn Arg Leu Leu Arg Ser Arg
35 40 45
Gly Pro Gly Ala Leu Gln Gly Leu Pro Ser Phe Arg Phe Glu Thr Ile
50 55 60
Thr Asp Gly Leu Pro Pro Thr Asp Ala Asp Ala Thr Gln Ser Ile Pro
65 70 75 80
Glu Leu Cys Arg Ser Thr Glu Leu His Ser Leu Gly Pro Phe Arg Asp
85 90 95
Leu Leu Arg Arg Leu Asn Asp Ser Gly Val Val Pro Pro Val Ser Cys
100 105 110
Ile Val Ser Asp Ser Ala Met Phe Phe Thr Leu Asp Ala Ala Glu Glu
115 120 125
Leu Gly Val Pro Glu Val Leu Leu Trp Thr Ala Ser Ala Cys Gly Phe
130 135 140
Leu Gly Val Thr Gln Tyr Asp Gln Leu Val Glu Leu Gly Leu Thr Pro
145 150 155 160
Phe Lys Asp Thr Asn Phe Leu Thr Asn Gly Asp Leu Asp Lys Val Leu
165 170 175
Asp Trp Val Pro Ala Met Lys Gly Ile Arg Leu Arg Asp Ile Pro Ser
180 185 190
Phe Ile Arg Thr Thr Asp Pro Asp Glu Phe Met Val Lys Tyr Val Arg
195 200 205
Arg Leu Val Tyr Gln Ser Lys Arg Ala Ser Ala Ile Leu Phe Asn Thr
210 215 220
Phe Asp Ala Leu Glu Gly Asp Val Leu Gln Ser Leu Ser Ser Asn Phe
225 230 235 240
Pro Arg Val Tyr Ser Leu Gly Pro Leu Gln Leu Leu Leu Asp Pro Ile
245 250 255
Asp Lys Glu Thr Lys Ser Ile Gly Ser Asn Leu Trp Lys Glu Asp Gln
260 265 270
His Cys Ile Asp Trp Leu Asp Ser His Gly Pro Asn Ser Val Val Tyr
275 280 285
Val Asn Phe Gly Ser Ile Thr Val Met Ser Asn Asp Gln Leu Val Glu
290 295 300
Phe Ala Trp Gly Leu Ala Asn Ser Gly Arg Pro Phe Leu Trp Ile Ala
305 310 315 320
Arg Pro Asp Leu Val Ile Gly Asp Ser Ala Val Leu Pro Pro Glu Phe
325 330 335
Leu Glu Glu Thr Arg Ser Arg Gly Leu Ile Ala Ser Trp Cys Asp Gln
340 345 350
Glu Arg Ile Leu Ala His Pro Ala Ile Gly Gly Phe Leu Thr His Cys
355 360 365
Gly Trp Asn Ser Ile Val Glu Ser Ile Cys Asn Gly Val Pro Val Val
370 375 380
Cys Trp Pro Phe Phe Ala Glu Gln Gln Thr Asn Cys Trp Tyr Ser Cys
385 390 395 400
Thr Lys Trp Gly Ile Gly Met Glu Ile Asp Pro Asn Val Lys Arg Glu
405 410 415
Val Val Glu Arg Gln Val Arg Glu Leu Met Leu Gly Glu Glu Gly Lys
420 425 430
Glu Met Lys Arg Lys Ala Met Glu Trp Lys Ala Leu Ala Gln Glu Ala
435 440 445
Thr Thr Ser Ser His Gly Ser Ser Tyr Ser Asn Met Asp Asn Leu Met
450 455 460
Thr Arg Val Leu Ser Pro
465 470
<210> 6
<211> 21
<212> DNA/RNA
<213> 人工序列( Artifical sequence)
<400> 6
gacycattgy ggatggaayt c 21
<210> 7
<211> 28
<212> DNA/RNA
<213> 人工序列( Artifical sequence)
<400> 7
ggagtaagaa gagccatgcg aagaagtg 28
<210> 8
<211> 29
<212> DNA/RNA
<213> 人工序列( Artifical sequence)
<400> 8
ggatccatgg gttctttgac aaagatagc 29
<210> 9
<211> 30
<212> DNA/RNA
<213> 人工序列(Artifical sequence)
<400> 9
gcggccgcgg gactaagtac tctggtcatg 30
<210> 10
<211> 36
<212> DNA/RNA
<213> 人工序列(Artifical sequence)
<400> 10
gcatgtggtt ttctgggtgt tacccagtat gatcag 36
<210> 11
<211> 36
<212> DNA/RNA
<213> 人工序列( Artifical sequence)
<400> 11
aacacccaga aaaccacatg cgcttgcggt ccacag 36
- 人工合成的内含子、哺乳动物细胞重组表达载体、哺乳动物宿主细胞、表达方法及其应用
- 人工合成的polyA、哺乳动物细胞重组表达载体、哺乳动物宿主细胞、表达方法及应用
- 源自谷氨酸棒杆菌的新型启动子核酸分子、包括所述启动子的重组载体、包括所述重组载体的宿主细胞和利用所述宿主细胞表达基因的方法
- 源自谷氨酸棒杆菌的新型启动子核酸分子、包括所述启动子的重组载体、包括所述重组载体的宿主细胞和利用所述宿主细胞表达基因的方法
