一种miRNA、检测及调控红树植物次生生长速度的方法、检测试剂盒及其应用
文献发布时间:2023-06-19 19:30:30
技术领域
本发明属于分子遗传学技术领域,具体涉及一种miRNA、检测及调控红树植物次生生长速度的方法、检测试剂盒及其应用。
背景技术
红树作为当今海岸湿地生态系统中惟一的一类木本植物,在净化海水、防风消浪、控制海岸侵蚀等方面发挥着极为重要的作用。然而,红树林不断受到人类活动的直接或间接破坏,导致红树林面积急剧减少。虽然近年来红树林保护修复取得积极进展,但红树总体面积偏小、生境退化等问题还比较突出。树木的生长发育包括纵向延伸的初生生长以及径向增粗的次生生长。树木次生生长即次生木质部细胞不断形成和增加的过程。阐明红树植物次生生长的分子遗传机制,通过进一步选育出新型速生红树新品种来增加红树种质资源供应,对实现红树林湿地生态系统的恢复具有重要的现实意义。
miRNA是一类长度为21nt左右的内源性非编码小分子RNA,能够通过碱基互补配对方式与靶基因mRNA分子结合负调控基因的表达。研究发现,miRNA在个体生长发育、细胞分裂分化、应激反应等各种生物活动中起着重要调节作用。尤其是,相关研究证实,miRNA同样是树木次生生长过程的重要调控因子。如Ptr-miR397a在杨树中过表达后,会下调其靶基因漆酶基因的表达,导致木质素合成受阻。近些年随着基因组测序技术的发展,RNA测序技术被广泛用于一些与重要性状相关的关键miRNAs的寻找与鉴定。尤其是,第三代测序技术的实现,为基因组信息尚未完善的物种进行生长过程重要基因鉴定和功能挖掘提供了有效手段。红海榄是红树科红树属植物,为常绿灌木或小乔木,高7~8m,生长于热带及亚热带的沿海滩涂湿地,主要分布于广西、香港、广东和海南等地,是沿海红树林生态恢复的优选树种。然而,目前关于调控红海榄次生生长的miRNA相关研究工作尚未开展。
发明内容
针对上述存在的技术问题,本发明提供了一种miRNA、检测红树植物次生生长速度的方法、检测试剂盒及其应用、调控红树植物次生生长速度的方法,发现了如SEQ ID NO.1所示的novel-Rst-miR26与红树植物次生生长速度相关,其可用于开发次生生长的辅助选择标记或提高红树植物次生生长速度,也可用于制备验证红树植物次生生长快慢的检测试剂盒。
一方面,本发明提供了一种与红树植物次生生长相关的miRNA,所述红树植物为红海榄,所述miRNA为novel-Rst-miR26,其前体结构的核苷酸序列如SEQ ID NO.1所示,成熟区序列为前体序列第71-91位碱基。
再一方面,本发明提供了一种检测红树植物次生生长速度的方法,其方法包括检测红树植物次生木质部中SEQ ID NO.1所示的miRNA的表达量,SEQ IDNO.1所示的miRNA的表达量与红树植物次生生长速度呈负相关,以验证红树植物次生生长速度的快慢。
进一步的,红树植物次生木质部中SEQ ID NO.1所示的miRNA的表达量通过Stem-loop实时荧光定量PCR检测得到。
再一方面,本发明提供了一种检测试剂盒,所述检测试剂盒中包括用于检测如SEQID NO.1所示的novel-Rst-miR26表达量的检测试剂。
进一步的,所述检测试剂盒包括如SEQ ID NO.2所示的novel-Rst-miR26茎环反转录引物,反转录扩增缓冲液,PrimeScript RT Enzyme Mix I,无RNAase水构成的反转录PCR扩增试剂。
进一步的,所述检测试剂盒还包括如SEQ ID NO.3所示的novel-Rst-miR26正向扩增引物,如SEQ ID NO.4所示的反向通用引物,如SEQ ID NO.5和SEQ ID NO.6所示的内参U6的实时荧光定量PCR扩增引物,以及模板cDNA,SYBR
再一方面,本发明提供了如上所述检测试剂盒在验证红树植物生长速度中的应用。
进一步的,提取红树植物RNA,采用检测试剂盒检测SEQ ID NO.1所示的miRNA表达量检测,实时荧光定量PCR的反应程序设置为:预变性95℃,30s;95℃/5s→60℃/30s,共计40个循环。
再一方面,本发明提供了一种调控红树植物次生生长速度的方法,其方法包括增加或降低红树植物的novel-Rst-miR26表达量。
本发明以红海榄快—慢植株生长模型为基础,联合使用二代RNA和三代全长转录组测序技术及相关生物信息学手段,首次在红树植物中鉴定获得影响次生生长过程的miRNA,即novel-Rst-miR26,可用于miRNA介导的红树植物次生生长调控机制的研究,同时用于红树植物次生生长性状辅助选择标记的开发;此外,基于该miRNA进一步研发获得了一种检测试剂盒,可用于判定novel-Rst-miR26在红树植物次生木质部中的表达量水平,检测方法简单快捷,以此验证其次生生长的快慢。本发明还可利用对于novel-Rst-miR26的表达量的调控以控制红树植物次生生长的快慢,在实际滨海湿地生态建设中提高红树植物的生长速度。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
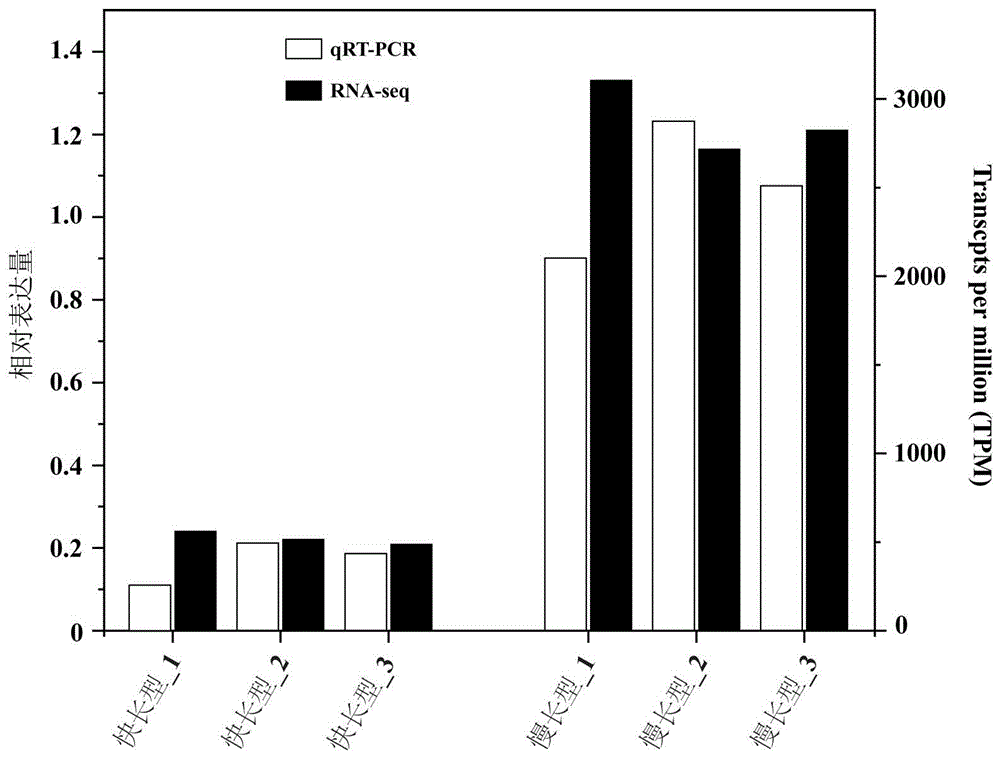
图1为快长型红海榄和慢长型红海榄的novel-Rst-miR26表达水平的RNA-seq和qRT-PCR验证结果。
具体实施方式
下面将结合本发明中的附图,对本发明中的技术方案进行清楚、完整地描述,显然,所描述的仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动的前提下所获得的所有其他实施例,都属于本发明保护的范围。
本发明一实施例提供了一种与红树植物次生生长相关的miRNA,所述红树植物为红海榄,所述miRNA为novel-Rst-miR26,其前体结构的核苷酸序列如SEQ ID NO.1所示,成熟区序列为前体序列第71-91位碱基。
具体的,序列SEQ ID NO.1如下所示:
acucauaucu cuaguuuguu cguggaucug accccaucac accguucaug ucacuaccau
ccaauggcgu uagauucacg cacaaacucg cgaucuguc
本发明另一实施例提供了一种检测红树植物次生生长速度的方法,其方法包括检测红树植物次生木质部中SEQ ID NO.1所示的miRNA的表达量,SEQ IDNO.1所示的miRNA的表达量与红树植物次生生长速度呈负相关,以验证红树植物次生生长速度的快慢。
在一些实施例中,红树植物次生木质部中SEQ ID NO.1所示的miRNA的表达量通过Stem-loop实时荧光定量PCR检测得到。
本发明另一实施例提供了一种检测试剂盒,所述检测试剂盒中包括用于检测如SEQ ID NO.1所示的novel-Rst-miR26表达量的检测试剂。
在一些实施例中,所述检测试剂盒包括如SEQ ID NO.2所示的novel-Rst-miR26茎环反转录引物,反转录扩增缓冲液,PrimeScript RT Enzyme Mix I,无RNAase水构成的反转录PCR扩增试剂。
在一些实施例中,所述检测试剂盒还包括如SEQ ID NO.3所示的novel-Rst-miR26正向扩增引物,如SEQ ID NO.4所示的反向通用引物,如SEQ ID NO.5和SEQ ID NO.6所示的内参U6的实时荧光定量PCR扩增引物,以及模板cDNA,SYBR
具体的,序列SEQ ID NO.2-SEQ ID NO.6如下所示:
本发明另一实施例提供了如上所述检测试剂盒在验证红树植物生长速度中的应用。
在一些实施例中,提取红树植物RNA,采用检测试剂盒检测SEQ ID NO.1所示的miRNA表达量检测,实时荧光定量PCR的反应程序设置为:预变性95℃,30s;95℃/5s→60℃/30s,共计40个循环。
本发明另一实施例提供了一种调控红树植物次生生长速度的方法,其方法包括增加或降低红树植物的novel-Rst-miR26表达量。
本发明以红海榄快—慢植株生长模型为基础,联合使用二代RNA和三代全长转录组测序技术及相关生物信息学手段,首次在红树植物中鉴定获得影响次生生长过程的miRNA,即novel-Rst-miR26,可用于miRNA介导的红树植物次生生长调控机制的研究,同时用于红树植物次生生长性状辅助选择标记的开发;此外,基于该miRNA进一步研发获得了一种检测试剂盒,可用于判定novel-Rst-miR26在红树植物次生木质部中的表达量水平,检测方法简单快捷,以此验证其次生生长的快慢。本发明还可利用对于novel-Rst-miR26的表达量的调控以控制红树植物次生生长的快慢,在实际滨海湿地生态建设中提高红树植物的生长速度。
以下通过具体实施例对本发明进行进一步说明:
1.样本采集及测序
测序样本原材料采集于广东省湛江市特呈岛红树林自然保护区。采集成熟的红海榄胚轴插入装有营养土的育苗袋中培养,三年后选取长势一致的快、慢型红海榄植株各3株,分别采集次生木质部组织于液氮速冻,提取RNA,质检合格后构建三代PacBio测序文库和sRNA测序文库,并进行测序及分析。其中,sRNA分析工作以PacBio单分子实时测序技术(SMRT)获得的红海榄全长转录本基因集作为数据库。测序工作由深圳华大基因科技有限公司完成。采集个体相关性状测定情况如表1所示。
表1快、慢红海榄植株生长指标
2.差异表达miRNA的筛选和靶基因预测
在对sRNA结果进行质量控制后,将快、慢两组植株miRNA的表达量数据进行比对,以|log2FC|≥3,显著性水平P<0.001,Q<0.01为标准进行差异表达miRNA的筛选,共获得17个差异表达的miRNA,包括10个保守miRNA和7个新发现miRNA。进一步利用psRNATarget方法对差异miRNA的靶基因进行预测,对获得的miRNA靶基因进行KEGG代谢通路富集分析,显示,在富集最显著的前20个代谢通路中,novel-Rst-miR26靶基因处于多个重要的通路,具体富集的通路如表2所示:
表2novel-Rst-miR26靶基因显著富集的通路
3.差异miRNA和靶基因调控网络构建
利用Cytoscape软件对获得的差异miRNA和靶基因进行调控网络构建,显示4个miRNA处于调控网络的核心,其中包括novel-Rst-miR26。进一步利用Cytoscape软件的Cytohubba插件,以及马修斯相关系数(Matthews correlation coefficient,MCC)方法,寻找靶基因中的关键基因(hub-genes),发现在前20个关键基因有15个受novel-Rst-miR26靶向调控。
表3novel-Rst-miR26的15个关键靶基因
/>
4.novel-Rst-miR26表达水平的qRT-PCR验证
提取快、慢红海榄植株次生木质部组织总RNA,采用Stem-loop实时荧光定量PCR试剂盒,经反转录扩增反应获得cDNA后,按照常规反应体系和扩增程序对novel-Rst-miR26的表达量进行测定。
该试剂盒由包括novel-Rst-miR26的茎环反转录引物(stem-loop RTprimer),反转录扩增缓冲液(5×PrimeScript Buffer),PrimeScript RT Enzyme Mix I和无RNAase水构成的反转录PCR扩增试剂,以及由包括novel-Rst-miR26的正向扩增引物,内参U6的引物对,模板cDNA,SYBR
SEQ ID NO.1
acucauaucu cuaguuuguu cguggaucug accccaucac accguucaugucacuaccauccaauggcgu uagauucacg cacaaacucg cgaucuguc
表4引物序列
荧光定量反应体系为20μl:SYBR
以U6为内参,运用2
以上内容是结合具体的优选实施方式对本发明所作的进一步详细说明,不能认定本发明的具体实施只局限于这些说明。对于本发明所属技术领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干简单推演或替换,都应当视为属于本发明的保护范围。
