使用可编程DNA结合蛋白增强靶向基因组修饰
文献发布时间:2024-04-18 20:01:23
技术领域
本公开内容涉及用于提高靶向基因组修饰的效率和/或特异性的组合物和方法。
背景技术
可编程核酸内切酶越来越成为真核生物中靶基因组工程或修饰的重要工具。最近,RNA引导的成簇规律间隔的短回文重复(CRISPR)/CRISPR-相关的(Cas)(CRISPR/Cas)系统已作为新一代基因组修饰工具出现。与前几代核酸酶,例如锌指核酸酶(ZFN)和转录激活因子样效应核酸酶(TALEN)相比,这些新的可编程核酸内切酶大大提高了基因组编辑能力。
然而,并非所有基因组靶标都可通过这些可编程核酸内切酶进行有效修饰。事实上,一些CRISPR-Cas核酸内切酶似乎在人类细胞中几乎没有或没有活性。染色质结构尤其可能成为这些可编程核酸内切酶的障碍并阻止它们与靶序列结合。因此,需要改进这些可编程核酸内切酶对靶序列的可及性和/或提高靶向基因组修饰的效率。此外,需要通过降低脱靶效应来增加靶向基因组修饰的特异性。
发明内容
在本公开内容的多个方面中包括一种组合物,其包含(a)可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸和(b)至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸。通常,可编程DNA修饰蛋白具有核酸酶活性(即切割双链序列的两条链)或非核酸酶活性(例如表观遗传修饰活性或转录调节活性)和所述至少一种可编程DNA结合蛋白缺乏核酸酶活性。
在可编程DNA修饰蛋白具有核酸酶活性的实施方案中,例如可编程DNA修饰蛋白可选自RNA引导的成簇规律间隔的短回文重复(CRISPR)/CRISPR
在可编程DNA修饰蛋白具有非核酸酶活性的实施方案中,例如可编程DNA修饰蛋白可以是融合蛋白,其包含与非核酸酶修饰结构域连接的可编程DNA结合结构域。在某些实施方案中,融合蛋白的可编程DNA结合结构域可以是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白或转录激活因子样效应子,并且融合蛋白的非核酸酶修饰结构域可具有乙酰转移酶活性、脱乙酰酶活性、甲基转移酶活性、去甲基酶活性、激酶活性、磷酸酶活性、泛素连接酶活性、去泛素化活性、腺苷酸化活性、去腺苷酸化活性、SUMO化活性、去SUMO化活性、核糖基化活性、去核糖基化活性、豆蔻酰化活性、去豆蔻酰化活性、瓜氨酸化活性、解旋酶活性、氨基化活性、脱氨基化活性、烷基化活性、脱烷基化活性、氧化活性、转录激活活性或转录阻遏活性。在具体的实施方案中,融合蛋白的非核酸酶修饰结构域具有胞嘧啶脱氨酶活性、组蛋白乙酰转移酶活性、转录激活活性或转录阻遏活性。
根据本文公开的组合物的某些实施方案,所述至少一种可编程DNA结合蛋白可以是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白、转录激活因子样效应子、CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。
通常,编码可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白的核酸是mRNA或DNA。在一些实施方案中,编码可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白的核酸是载体的一部分,例如质粒载体、慢病毒载体、腺伴随病毒载体、或腺病毒载体。
在具体的实施方案中,可编程DNA修饰蛋白包含CRISPR/Cas核酸酶系统、CRISPR/Cas双切口酶系统、或与非核酸酶结构域连接的无催化活性的CRISPR/Cas系统,并且至少一种可编程DNA结合蛋白包含无催化活性的CRISPR/Cas系统,其中每种CRISPR/Cas系统包含CRISPR/Cas蛋白和引导RNA。在多种实施方案中,每种CRISPR/Cas核酸酶系统可以是I型CRISPR/Cas系统、II型CRISPR/Cas系统、III型CRISPR/Cas系统或V型CRISPR/Cas系统。在一些实施方案中,每种引导RNA可以至少部分化学合成。在其它实施方案中,每种引导RNA可以至少部分酶促合成。在进一步的实施方案中,编码每种CRISPR/Cas蛋白的核酸可以是mRNA,并且编码每种引导RNA的核酸可以是DNA。在其它的实施方案中,编码每种CRISPR/Cas蛋白的核酸可以是mRNA、并且编码每种引导RNA的核酸可以是DNA。在某些方面中,编码CRISPR/Cas蛋白的核酸和/或编码引导RNA的核酸可以是载体的一部分,例如质粒载体、慢病毒载体、腺伴随病毒载体或腺病毒载体。
本公开内容的另一方面包括试剂盒,其包含上文详述的任何一种或多种组合物。
本公开内容的另一方面提供了用于提高真核细胞中靶向基因组修饰效率和/或特异性的方法。所述方法包括向真核细胞中引入(a)可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸和(b)至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸。可编程DNA修饰蛋白靶向于靶染色体序列,并且所述至少一种可编程DNA结合蛋白中的每种都靶向于靶染色体序列附近的位点。所述至少一种可编程DNA结合蛋白与靶染色体序列附近位点的结合提高了可编程DNA修饰蛋白对靶染色体序列的可及性,从而提高靶向基因组修饰效率和/或特异性。由至少一种可编程DNA结合蛋白中的每种结合的附近位点都位于例如靶染色体序列任一侧的约250个碱基对内。在一些实施方案中,附近结合位点位于靶染色体序列的任一侧的小于约200bp或小于约100bp。
所述方法中使用的可编程DNA修饰蛋白可以是CRISPR/Cas核酸酶系统、CRISPR/Cas双切口酶系统、锌指核酸酶(ZFN)、转录激活因子样效应核酸酶(TALEN)、大范围核酸酶、包含与核酸酶结构域连接的可编程DNA结合结构域的融合蛋白,或包含与非核酸酶结构域连接的可编程DNA结合结构域的融合蛋白。所述融合蛋白的可编程DNA结合结构域可以是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白或转录激活因子样效应子,并且融合蛋白的非核酸酶修饰结构域可具有乙酰转移酶活性、脱乙酰酶活性、甲基转移酶活性、去甲基酶活性、激酶活性、磷酸酶活性、泛素连接酶活性、去泛素化活性、腺苷酸化活性、去腺苷酸化活性、SUMO化活性、去SUMO化活性、核糖基化活性、去核糖基化活性、豆蔻酰化活性、去豆蔻酰化活性、瓜氨酸化活性、解旋酶活性、氨基化活性、脱氨基化活性、烷基化活性、脱烷基化活性、氧化活性、转录激活活性或转录阻遏活性。在具体的实施方案中,融合蛋白的非核酸酶修饰结构域具有胞嘧啶脱氨酶活性、组蛋白乙酰转移酶活性、转录激活活性或转录阻遏活性。
所述方法中使用的所述至少一种可编程DNA结合蛋白结合DNA但缺乏核酸酶活性(即双链切割活性)。在某些实施方案中,所述至少一种可编程DNA结合蛋白可以是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白、转录激活因子样效应子、CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。
在具体的实施方案中,可编程DNA修饰蛋白包含CRISPR/Cas核酸酶系统、CRISPR/Cas双切口酶系统、或与非核酸酶结构域连接的无催化活性的CRISPR/Cas系统,并且所述至少一种可编程DNA结合蛋白包含无催化活性的CRISPR/Cas系统,其中每种CRISPR/Cas系统包含CRISPR/Cas蛋白和引导RNA。
在多种实施方案中,将至少两种、至少三种或多于三种可编程DNA结合蛋白引入真核细胞中。在具体的实施方案中,所述真核细胞是哺乳动物细胞或人类细胞。
本公开内容的另一方面包括用于检测真核细胞中的染色体序列或基因组基因座的方法。所述方法包括向真核细胞中引入(a)包含至少一种可检测标记结构域的可编程DNA结合蛋白或编码包含至少一种可检测标记结构域的可编程DNA结合蛋白的核酸,和(b)至少一种可编程DNA结合蛋白或编码所述至少一种可编程DNA结合蛋白的核酸,其中包含至少一种可检测标记结构域的可编程DNA结合蛋白靶向靶染色体序列,并且所述至少一种可编程DNA结合蛋白中的每种都靶向于靶染色体序列附近的位点,其中所述至少一种可编程DNA结合蛋白与靶染色体序列附近的位点的结合提高包含至少一种可检测标记结构域的可编程DNA结合蛋白对靶染色体序列的可及性。所述方法可以进一步包括检测与靶染色体序列结合的包含至少一种可检测标记结构域的可编程DNA结合蛋白。检测步骤可以在活细胞或固定细胞中进行,并且可以涉及例如动态活细胞成像、荧光显微术、共聚焦显微术、免疫荧光、免疫检测、RNA-蛋白结合或蛋白-蛋白结合。
在检测方法中使用的包含至少一种可检测标记结构域的可编程DNA结合蛋白包含可编程DNA结合结构域,其可以是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白或转录激活因子样效应子。所述包含至少一种可检测标记结构域的可编程DNA结合蛋白的至少一种可检测标记结构域可以是例如荧光蛋白、荧光标签、表位标签、或可编程DNA结合蛋白内的天然存在的表位。在一些实施方案中,所述包含至少一种可检测标记结构域的可编程DNA结合蛋白可以进一步包含非核酸酶修饰。所述至少一种可编程DNA结合蛋白结合DNA但缺乏核酸酶活性(即双链切割活性)。在一些实施方案中,所述可编程DNA结合蛋白可以是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白、转录激活因子样效应子、CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。在具体的实施方案中,所述包含至少一种可检测标记结构域的可编程DNA结合蛋白可以是与至少一种可检测标记结构域连接的无催化活性的CRISPR/Cas系统,并且所述至少一种可编程DNA结合蛋白可以是无催化活性的CRISPR/Cas系统。
以下详述本公开内容的其它方面和特征。
附图简述
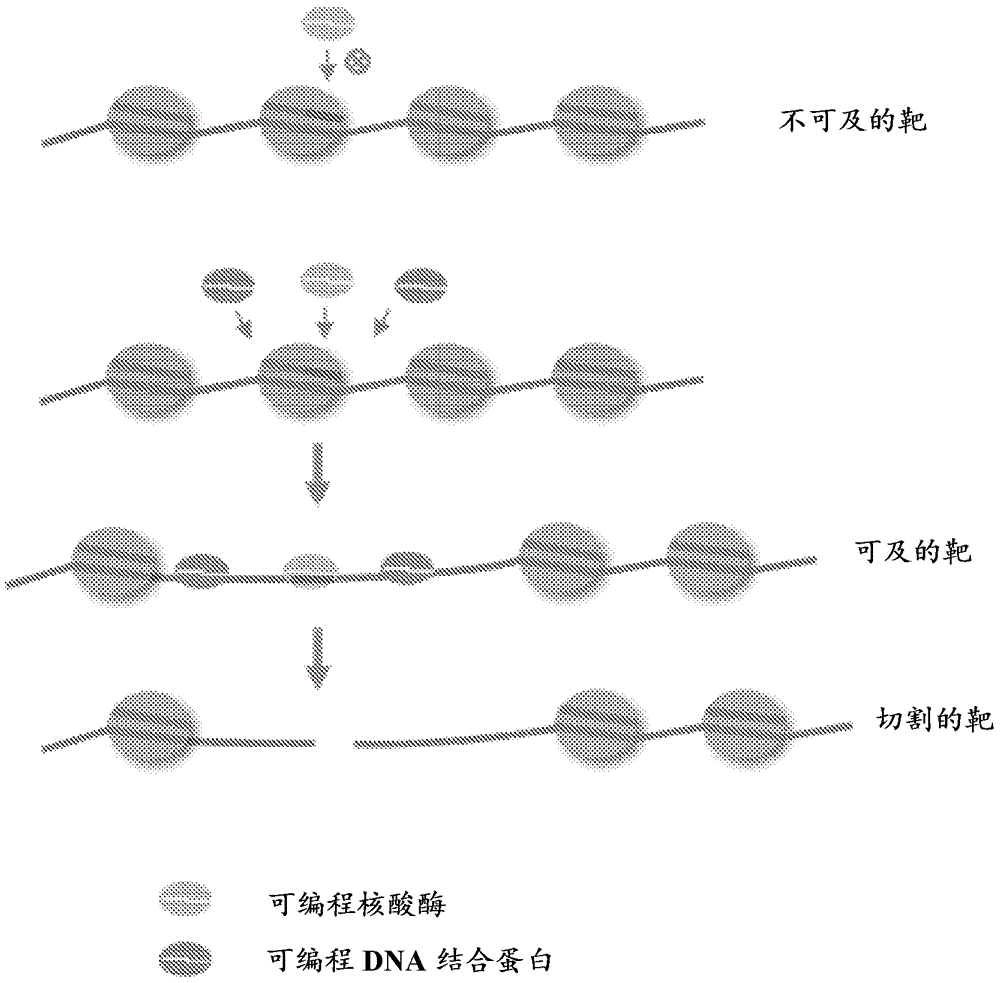
图1提供了本文公开的方法的一个实施方案的图示。可编程DNA结合蛋白的附近结合提高了靶位点对可编程核酸酶的可及性,从而提高了靶位点处的切割效率。
图2示出了无催化活性的SpCas9(SpdCas9)与附近位点的结合提高了FnCas9的切割效率。顶部呈现的序列显示了POR基因座中FnCas9靶位点和SpdCas9结合位点的相对位置。底部显示Cel-I核酸酶测定的结果。
图3A示出了实验设计以确定结合无催化活性的SpCas9(SpdCas9)是否增加带表位标签的(即带FLAG
图3B提供了染色质免疫沉淀结合测定的图示,该测定用于检测带表位标签的CjdCas9与POR和AAVS1基因座中的靶标位点的结合。
图3C示出了SpdCas9与附近位点的结合提高了带表位标签的CjCas9与POR基因座中先前不可及的位点的结合。
图4示出了无催化活性的SpCas9(SpdCas9)与附近位点的结合提高了CjCas9的切割效率。顶部呈现的序列显示了POR基因座中CjCas9靶位点与SpdCas9结合位点的相对位置。Cel-I核酸酶测定的结果显示在底部。
图5示出了无催化活性的SpCas9(SpdCas9)与附近位点的结合提高了FnCpf1的切割效率。顶部示出了POR基因座中FnCpf1靶位点与SpdCas9结合位点的相对位置,并且在底部示出了Cel-I核酸酶测定的结果。
图6示出了无催化活性的SpCas9(SpdCas9)与附近位点的结合提高了CjCas9的特异性切割。在顶部显示HBD和HBB基因座中CjCas9的靶位点以及HBB基因座中SpdCas9的结合位点。Cel-I核酸酶测定的结果显示在底部。
图7示出了无催化活性的FnCas9(FndCas9)与附近位点的结合提高了SpCas9的特异性切割。顶部显示了POR基因座中SpoSas9靶位点与FndCas9结合位点的相对位置。底部显示Cel-I核酸酶测定的结果。
图8示出了ssDNA寡核苷酸介导的基因编辑的增强。顶部显示了POR基因座中的靶位点和ssDNA寡核苷酸序列的相对位置。底部显示EcoRI位点靶向整合的结果。通过ImageJ确定EcoRI位点整合效率(%)。M: 宽范围的DNA标志物。ND:未测定。
发明详述
本公开内容提供了用于提高染色体DNA对靶向核酸内切酶和其它可编程DNA修饰蛋白的可及性的组合物和方法,其中提高的可及性使得靶向基因组修饰或表观遗传修饰的效率和/或特异性提高。已经发现一些CRISPR/Cas核酸内切酶在人细胞中活性降低或没有活性。核小体占用率、定位以及DNA序列如何包裹在组蛋白八聚体周围可决定该序列对DNA结合蛋白的可及性程度(Chereji et al., Briefing Functional Genomics, 2014, 14:506-60)。因此,在人细胞的许多CRISPR/Cas核酸内切酶的明显失活中,由局部染色质构型所施加的阻碍可能起作用。如本文详述的,已发现DNA结合蛋白与靶向DNA修饰蛋白的靶位点附近(即在约250个碱基对内)的位点结合提高了靶向DNA修饰蛋白对于所述靶位点的可及性,从而提高靶向基因组修饰或靶向表观遗传修饰的效率和/或特异性。因此,本文公开的组合物和方法使得能够使用先前认为在人细胞中失活的CRISPR/Cas核酸内切酶进行有效的靶向基因组修饰/表观遗传修饰。此外,本文公开的组合物和方法还改善了近似相同的靶位点之间的选择性基因组修饰,从而减少了脱靶效应。
组合物
本公开内容的一个方面提供了组合物,其包含(a)可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸和(b)至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸。可编程DNA修饰蛋白在以下(I)(a)部分中详述,可编程DNA结合蛋白在以下(I)(b)部分中详述,并且编码这些蛋白的核酸在以下(I)(c)部分中详述。
可编程DNA修饰蛋白
可编程DNA修饰蛋白是与染色体DNA中的特定靶序列结合并在靶序列处或附近修饰DNA或与DNA相关蛋白的蛋白。因此,可编程DNA修饰蛋白包含DNA结合结构域和催化活性修饰结构域。
所述DNA结合结构域是可编程的,因为其可以被设计或工程化以识别和结合不同的DNA序列。在一些实施方案中,例如DNA结合是由蛋白和靶DNA之间的相互作用介导的。因此,DNA结合结构域可以通过蛋白质工程编程以结合目的DNA序列。在其它实施方案中,例如DNA结合由引导RNA介导,所述引导RNA与蛋白的可编程DNA结合结构域和靶DNA相互作用。在这种情况下,通过设计合适的引导RNA,可编程DNA结合结构域可以靶向目的DNA序列。
可编程DNA修饰蛋白中可包含多种修饰结构域。在一些实施方案中,所述修饰结构域是核酸酶结构域,其具有核酸酶活性并切割双链DNA序列的两条链(即产生双链断裂)。然后可以通过细胞DNA修复过程来修复双链断裂,所述细胞DNA修复过程例如非同源末端连接(NHEJ)或同源定向修复(HDR)。因此,可以通过至少一个碱基对直至例如数千个碱基对的缺失、插入和/或取代来修饰DNA序列。包含核酸酶结构域的可编程DNA修饰蛋白的实例包括但不限于CRISPR/Cas核酸酶系统、CRISPR/Cas双切口酶系统、锌指核酸酶、转录激活因子样效应核酸酶、大范围核酸酶、包含与可编程DNA结合结构域连接的核酸酶结构域的融合蛋白及其组合。包含核酸酶结构域的可编程DNA修饰蛋白在以下(I)(a)(i)-(vi)部分中详述。
在其它实施方案中,所述可编程DNA修饰蛋白的修饰结构域具有非核酸酶活性(例如表观遗传修饰活性或转录调节活性),使得可编程DNA修饰蛋白修饰DNA和/或与DNA有关蛋白的结构和/或活性。因此,所述可编程DNA修饰蛋白是包含与可编程DNA结合结构域连接的非核酸酶修饰结构域的融合蛋白。这些蛋白在以下(I)(a)(vii)部分中详述。
所述可编程DNA修饰蛋白可包含野生型或天然存在的DNA结合和/或修饰结构域,天然存在的DNA结合和/或修饰结构域的修饰形式、合成或人工DNA结合和/或修饰结构域,及其组合。
(i)CRISPR/Cas核酸酶系统
在一些实施方案中,所述可编程DNA修饰蛋白可以是RNA指导的CRISPR/Cas核酸酶系统,其在DNA中引入双链断裂。CRISPR/Cas核酸酶系统包含CRISPR/Cas核酸酶和引导RNA。
CRISPR/Cas核酸酶。在某些实施方案中,CRISPR/Cas核酸酶可源自存在于各种细菌和古生菌中的I型(即IA、IB、IC、ID、IE或IF)、II型(即IIA、IIB或IIC)、III型(即IIIA或IIIB)、或V型CRISPR系统。例如,CRISPR/Cas系统可以来自链球菌属(Streptococcus sp.)(例如,酿脓链球菌(Streptococcus pyogenes))、弯曲杆菌属(Campylobacter sp.)(例如空肠弯曲杆菌(Campylobacter jejuni))、弗朗西斯菌属(Francisella sp.)(例如新凶手弗朗西斯菌(Francisella novicida))、Acaryochloris sp.、醋盐杆菌属(Acetohalobiumsp.)、氨基酸球菌属(Acidaminococcus sp.)、嗜酸硫杆菌属(Acidithiobacillus sp.)、脂环酸芽孢杆菌属(Alicyclobacillus sp.)、着色菌属(Allochromatium sp.)、制氨菌属(Ammonifex sp.)、鱼腥藻属(Anabaena sp.)、节螺藻属(Arthrospira sp.)、芽孢杆菌属(Bacillus sp.)、伯克氏菌属(Burkholderiales sp.)、Caldicelulosiruptor sp.、念珠菌属(Candidatus sp.)、梭菌属(Clostridium sp.)、鳄球藻属(Crocosphaera sp.)、蓝丝菌属(Cyanothece sp.)、微小杆菌属(Exiguobacterium sp.)、芬戈尔德菌属(Finegoldiasp.)、纤线杆菌属(Ktedonobacter sp.)、毛螺菌属(Lachnospiraceae sp.)、乳杆菌属(Lactobacillus sp.)、鞘丝藻属(Lyngbya sp.)、海洋杆菌属(Marinobacter sp.)、甲烷盐菌属(Methanohalobium sp.)、微颤菌属(Microscilla sp.)、微鞘菌属(Microcoleussp.)、微囊藻属(Microcystis sp.)、盐碱厌氧菌属(Natranaerobius sp.)、奈瑟球菌属(Neisseria sp.)、硝化球菌属(Nitrosococcus sp.)、拟诺卡氏菌属(Nocardiopsis sp.)、节球藻属(Nodularia sp.)、念珠藻属(Nostoc sp.)、颤藻属(Oscillatoria sp.)、极地单胞菌属(Polaromonas sp.)、Pelotomaculum sp.、假交替单胞菌属(Pseudoalteromonassp.)、Petrotoga sp.、普雷沃氏菌属(Prevotella sp.)、葡萄球菌属(Staphylococcussp.)、链霉菌属(Streptomyces sp.)、孢囊链霉菌属(Streptosporangium sp.)、Synechococcus sp.、热袍菌属(Thermosipho sp.)、或疣微菌属(Verrucomicrobia sp .)。在其它实施方案中,CRISPR/Cas核酸酶可源自古生菌CRISPR系统、CRISPR-CasX系统或CRISPR-CasY系统(Burstein et al., Nature, 2017, 542(7640):237-241)。
在一个具体的实施方案中,所述CRISPR/Cas核酸酶可源自I型CRISPR/Cas系统。在另一个具体的实施方案中,所述CRISPR/Cas核酸酶可源自II型CRISPR/Cas系统。在另一个具体的实施方案中,所述CRISPR/Cas核酸酶可源自III型CRISPR/Cas系统。在另一个具体的实施方案中,所述CRISPR/Cas核酸酶可源自V型CRISPR/Cas系统。
合适的CRISPR蛋白的非限制性实例包括Cas蛋白、Cpf蛋白、C2c蛋白(例如C2c1、C2c2、Cdc3)、Cmr蛋白、Csa蛋白、Csb蛋白、Csc蛋白、Cse蛋白、Csf蛋白、Csm蛋白、Csn蛋白、Csx蛋白、Csy蛋白、Csz蛋白及其衍生物或变体。在具体的实施方案中,CRISPR/Cas核酸酶可以是II型Cas9蛋白、V型Cpf1蛋白或其衍生物。
在一些实施方案中,CRISPR/Cas核酸酶可以是酿脓链球菌Cas9(SpCas9)或嗜热链球菌Cas9(StCas9)。在另一些实施方案中,CRISPR/Cas核酸酶可以是空肠弯曲杆菌Cas9(CjCas9)。在另一些实施方案中,CRISPR/Cas核酸酶可以是新凶手弗朗西斯菌Cas9(FnCas9)。在另一些实施方案中,CRISPR/Cas核酸酶可以是灰色奈瑟菌(
通常,所述CRISPR/Cas核酸酶包含RNA识别和/或RNA结合结构域,其与引导RNA相互作用。所述CRISPR/Cas核酸酶还包含至少一种具有核酸内切酶活性的核酸酶结构域。例如,Cas9蛋白包含RuvC样核酸酶结构域和HNH样核酸酶结构域,并且Cpf1蛋白包含RuvC样结构域。CRISPR/Cas核酸酶还可以包含DNA结合结构域、解旋酶结构域、RNA酶结构域、蛋白-蛋白相互作用结构域、二聚化结构域以及其它结构域。
所述CRISPR/Cas核酸酶可进一步包含至少一种核定位信号、细胞穿透结构域和/或标记结构域。核定位信号的非限制性实例包括PKKKRKV (SEQ ID NO:1)、PKKKRRV (SEQID NO:2)、KRPAATKKAGQAKKKK (SEQ ID NO:3)、 YGRKKRRQRRR (SEQ ID NO:28)、RKKRRQRRR(SEQ ID NO:29)、PAAKRVKLD (SEQ ID NO:30)、RQRRNELKRSP (SEQ ID NO:31)、VSRKRPRP(SEQ ID NO:32)、PPKKARED (SEQ ID NO:33)、PQPKKKPL (SEQ ID NO:34)、SALIKKKKKMAP(SEQ ID NO:35)、PKQKKRK (SEQ ID NO:36)、RKLKKKIKKL (SEQ ID NO:37)、REKKKFLKRR(SEQ ID NO:38)、KRKGDEVDGVDEVAKKKSKK (SEQ ID NO:39)、RKCLQAGMNLEARKTKK (SEQ IDNO:40)、NQSSNFGPMKGGNFGGRSSGPYGGGGQYFAKPRNQGGY (SEQ ID NO:41)和 RMRIZFKNKGKDTAELRRRRVEVSVELRKAKKDEQILKRRNV (SEQ ID NO:42)。合适的细胞穿透结构域的非限制性实例包括GRKKRRQRRRPPQPKKKRKV (SEQ ID NO:4)、PLSSIFSRIGDPPKKKRKV (SEQ ID NO:5)、GALFLGWLGAAGSTMGAPKKKRKV (SEQ ID NO:6)、GALFLGFLGAAGSTMGAWSQPKKKRKV (SEQ IDNO: 7)、KETWWETWWTEWSQPKKKRKV (SEQ ID NO: 8)、YARAAARQARA (SEQ ID NO:43)、THRLPRRRRRR (SEQ ID NO:44)、GGRRARRRRRR (SEQ ID NO:45)、RRQRRTSKLMKR (SEQ IDNO:46)、GWTLNSAGYLLGKINLKALAALAKKIL (SEQ ID NO:47)、KALAWEAKLAKALAKALAKHLAKALAKALKCEA (SEQ ID NO:48)和RQIKIWFQNRRMKWKK (SEQ ID NO:49)。标记结构域包括荧光蛋白和纯化或表位标签。合适的荧光蛋白包括但不限于绿色荧光蛋白(例如GFP、eGFP、GFP-2、tagGFP、turboGFP、Emerald、Azami Green、Monomeric Azami Green、CopGFP、AceGFP,ZsGreen1)、黄色荧光蛋白(例如YFP、EYFP、柠檬黄、金星、YPet、PhiYFP、ZsYellow1)、蓝色荧光蛋白(如BFP、EBFP、EBFP2、蓝铜矿、mKalama1、GFPuv、蓝宝石、T-蓝宝石)、青色荧光蛋白(例如ECFP、Cerulean、CyPet、 AmCyan1、Midoriishi-Cyan)、红色荧光蛋白(例如mKate、mKate2、mPlum、DsRed单体、mCherry、mRFP1、DsRed-Express、DsRed2、DsRed-单体、HcRed-Tandem、HcRed1、AsRed2、eqFP611、mRasberry、mStrawberry、Jred)和橙色荧光蛋白(例如mOrange、mKO、Kusabira-Orange、MonomericKusabira-Orange、mTangerine、tdTomato)。合适的纯化或表位标签的非限制性实例包括6xHis、FLAG
所述核定位信号、细胞穿透结构域和/或标记结构域可位于蛋白的N-末端、C-末端或内部位置。在一些实施方案中,所述CRISPR/Cas核酸酶还可包含至少一种可检测标记。可检测标记可以是荧光团(例如FAM、TMR、Cy3、Cy5、德克萨斯红、俄勒冈绿、Alexa Fluors、Halo标签或合适的荧光标签/染料)、发色团(例如生物素、地高辛等)、量子点或金颗粒。可检测的标记可以通过常规方法连接到蛋白的任何氨基酸上。
引导RNA。所述CRISPR/Cas核酸酶系统还包含引导RNA(gRNA)。所述引导RNA与CRISPR/Cas核酸酶和靶位点相互作用以将CRISPR/Cas核酸酶引导至染色体序列中的靶位点。所述靶位点没有序列限制,除了所述序列毗邻前间隔序列邻近基序(PAM)。例如,Cas9蛋白的PAM序列包括3'-NGG, 3'-NGGNG、3'-NNAGAAW和3'-ACAY,并且Cpf1的PAM序列包括5'-TTN(其中N定义为任何核苷酸,W定义为A或T,且Y定义为C或T)。
每种引导RNA可以包含三个区域:与染色体DNA序列中的靶位点互补的5'末端的第一区域,在内部并形成茎环结构的第二区域,和保持基本上是单链的位于3’末端的第三区域。第二和第三区域形成与CRISPR/Cas蛋白相互作用的二级结构。每种引导RNA的第一个区域是不同的(即是序列特异性的)。第二和第三区域在引导RNA中可以是相同的,所述引导RNA与特定CRISPR/Cas蛋白形成复合体。
引导RNA的第一区域与靶位点处的序列(即前间隔序列)具有互补性,使得引导RNA的第一区域可与靶序列碱基配对。例如SpCas9引导RNA的第一区域可包含GN
所述引导RNA还包含形成二级结构的第二区域。在一些实施方案中,所述二级结构包含至少一种茎(或发夹)和环。每个环和茎的长度可以不同。例如,环的长度可以为约3至约10个核苷酸,并且茎的长度可以为约6至约20个碱基对。所述茎可包含1至约10个核苷酸的一个或多个凸起。因此,第二区域的总长度可以为约16至约60个核苷酸的长度。引导RNA还在3'末端包含第三区域,其基本上保持单链。因此,第三区域与目标细胞中的任何核酸序列没有互补性,并且与引导RNA的其余部分没有互补性。所述第三区域的长度可以不同。通常,第三区域的长度大于约4个核苷酸。例如,第三区域的长度可以为约5至约60个核苷酸的长度。
引导RNA的第二和第三区域(也称为通用或支架区域)的组合长度可以为约30至约120个核苷酸长度。在一个方面中,引导RNA的第二和第三区域的组合长度为约70至约100个核苷酸长度。
在其它实施方案中,引导RNA的第二和第三区域可包含一个或多个另外的茎-环区域,其中茎-环区域包含适体序列(Konermann et al., Nature3, 2015, 517(7536):583-588; Zalatan et al., Cell, 2015, 160(1-2):339-50)。合适的适体序列包括结合选自以下的衔接蛋白的序列:MS2、PP7、COM、Qβ、F2、GA、fr、JP501、M12、R17、BZ13、JP34、JP500、KU1、M11、MX1、TW18、VK、SP、FI、ID2、NL95、TW19、AP205、φCb5、φCb8r、φCb12r、φCb23r、7s、PRR1、HSF1、AID、APOBEC1、p300、TET1/2/3、VP64、GFP、Rta、p65、MyoD1或VP160。在这些实施方案中,引导RNA的第二和第三区域的总长度可以在高达约125个核苷酸、高达约150个核苷酸、高达约175个核苷酸、高达约200个核苷酸、高达约225个核苷酸、高达约250个核苷酸、高达约275个核苷酸,或高达约300个核苷酸的范围。
在一些实施方案中,引导RNA可以是包含所有三个区域的单分子。在另一些实施方案中,引导RNA可包含两个独立的分子。第一RNA分子(即crRNA)可以包含引导RNA的第一区域和引导RNA的第二区域的“茎”的一半。第二RNA分子(即tracrRNA)可以包含引导RNA的第二区域的“茎”的另一半和引导RNA的第三区域。因此,在该实施方案中,第一和第二RNA分子的每个都含有彼此互补的核苷酸序列。例如,在一个实施方案中,crRNA和tracrRNA RNA分子各自包含与另一序列碱基配对的序列(约6至约20个核苷酸),以形成功能性引导RNA。例如,II型CRISPR/Cas系统的引导RNA可包含crRNA和tracrRNA。在一些方面中,可以化学合成II型CRISPR/Cas系统的crRNA,并且可以在体外合成tracrRNA II型CRISPR/Cas系统(参见以下(I)(c)部分)。在其它实施方案中,V型CRISPR/Cas系统的引导RNA可仅包含crRNA。
引导RNA可包含标准核糖核苷酸、修饰的核糖核苷酸(例如假尿苷)、核糖核苷酸异构体和/或核糖核苷酸类似物。在一些实施方案中,所述引导RNA还可包含至少一种可检测标记。可检测标记可以是荧光团(例如FAM、TMR、Cy3、Cy5、德克萨斯红、俄勒冈绿、AlexaFluors、Halo标签或合适的荧光染料)、发色团(例如生物素、地高辛等)、量子点或金颗粒。本领域技术人员熟悉gRNA设计和构建,例如gRNA设计工具可从因特网或商业来源获得。
引导RNA可以化学合成、酶促合成或其组合方法合成。例如,可以使用基于标准亚磷酰胺的固相合成方法合成引导RNA。可选的,可以通过将编码引导RNA的DNA可操作地连接到由噬菌体RNA聚合酶识别的启动子控制序列来体外合成引导RNA。合适的噬菌体启动子序列的实例包括T7、T3、SP6启动子序列或其变体。在引导RNA包含两个分开的分子(即crRNA和tracrRNA)的实施方案中,可以化学合成crRNA并且可以酶促合成tracrRNA。编码引导RNA的核酸可以是质粒载体的一部分,其可以进一步包含其它的表达控制序列(例如增强子序列、Kozak序列、多腺苷酸化序列、转录终止序列等)、选择标记序列(例如抗生素抗性基因)、复制起点等。如下文(I)(c)部分中详述的,编码引导RNA的核酸可以与RNA聚合酶III(PolIII)识别的启动子控制序列可操作地连接,用于在真核细胞中表达。
(ii)CRISPR/Cas双切口酶系统
在其它实施方案中,所述可编程DNA修饰蛋白可以是CRISPR/Cas双切口酶系统。CRISPR/Cas双切口酶系统与上文(I)(a)(i)部分中描述的CRISPR/Cas核酸酶系统类似,不同之处在于将CRISPR/Cas核酸酶修饰以便仅切割一条DNA链。因此,单个CRISPR/Cas切口酶系统在双链DNA中产生单链断裂或切口,并且包含成对补偿性(offset)引导RNA的成对CRISPR/Cas双切口酶系统在DNA中产生双链断裂。
可以通过一个或多个突变和/或缺失将CRISPR/Cas核酸酶转化为切口酶。例如,Cas9切口酶可以在核酸酶结构域之一(例如RuvC样结构域或HNH样结构域)中包含一个或多个突变。例如,所述一个或多个突变可以是RuvC样结构域中的D10A、D8A、E762A和/或D986A,或者所述一个或多个突变可以是HNH样结构域中的H840A、H559A、N854A、N856A和/或N863A。
(iii)锌指核酸酶
在其它实施方案中,所述可编程DNA修饰蛋白可以是锌指核酸酶(ZFN)。ZFN包含DNA结合锌指区域和核酸酶结构域。所述锌指区域可包括约二至七个锌指,例如约四至六个锌指,其中每个锌指结合三个核苷酸。可以将锌指区域改造为识别并结合任何DNA序列。锌指设计工具或算法可从因特网或商业来源获得。锌指可以使用合适的接头序列连接在一起。
ZFN还包含核酸酶结构域,其可以从任何核酸内切酶或核酸外切酶获得。可以衍生核酸酶结构域的核酸内切酶的非限制性实例包括但不限于限制性核酸内切酶和归巢核酸内切酶。在一些实施方案中,核酸酶结构域可以衍生自II-S型限制性核酸内切酶。II-S型核酸内切酶通常在距识别/结合位点几个碱基对的位点切割DNA,因此具有可分离的结合和切割结构域。这些酶通常是单体,其可瞬时缔合以形成二聚体,以在交错位置切割每条DNA链。合适的II-S核酸内切酶的非限制性实例包括BfiI、BpmI、BsaI、BsgI、BsmBI、BsmI、BspMI、FokI、MboII和SapI。在一些实施方案中,核酸酶结构域可以是FokI核酸酶结构域或其衍生物。可以对II-S型核酸酶结构域进行修饰以促进两个不同核酸酶结构域的二聚化。例如,可以通过突变某些氨基酸残基来修饰FokI的切割结构域。作为非限制性实例,位于FokI核酸酶结构域的446、447、479、483、484、486、487、490、491、496、498、499、500、531、534、537和538位处的氨基酸残基是修饰的靶标。例如,一个修饰的FokI结构域可以包含Q486E、I499L和/或N496D突变,并且其它修饰的FokI结构域可以包含E490K、I538K和/或H537R突变。
所述ZFN可进一步包含至少一种核定位信号、细胞穿透结构域和/或标记结构域,如上文(I)(a)(i)部分中所描述。
(iv)转录激活因子样效应核酸酶
在备选实施方案中,所述可编程DNA修饰蛋白可以是转录激活因子样效应核酸酶(TALEN)。TALEN包含DNA结合结构域,其由高度保守的重复序列组成,所述重复序列衍生自与核酸酶结构域连接的转录激活因子样效应子(TALE)。TALE是植物病原体黄单胞菌(Xanthomonas)分泌的蛋白,以改变宿主植物细胞中基因的转录。可以通过模块化蛋白设计来改造TALE重复阵列以靶向任何目标DNA序列。TALEN的核酸酶结构域可以是如上文(I)(a)(iii)部分中所述的任何核酸酶结构域。在具体的实施方案中,核酸酶结构域衍生自FokI(Sanjana
TALEN还可以包含至少一种核定位信号、细胞穿透结构域、标记结构域和/或可检测标记,如上文(I)(a)(i)部分中所描述。
(v)大范围核酸酶或稀切核酸内切酶
在其它实施方案中,所述可编程DNA修饰蛋白可以是大范围核酸酶或其衍生物。大范围核酸酶是内切脱氧核糖核酸酶,其特征在于长识别序列,即识别序列通常为约12个碱基对至约45个碱基对。由于该要求,识别序列通常在任何给定的基因组中仅出现一次。在大范围核酸酶中,命名为LAGLIDADG的归巢核酸内切酶家族已成为基因组和基因组工程研究的有价值工具。在一些实施方案中,大范围核酸酶可以是I-SceI、I-TevI或其变体。通过使用本领域技术人员熟知的技术修饰其识别序列,可以将大范围核酸酶靶向于特定的染色体序列。
在备选的实施方案中,所述可编程DNA修饰蛋白可以是稀切核酸酶内切核或其衍生物。稀切核酸内切酶是位点特异性核酸内切酶,其识别序列在基因组中很少出现,优选在基因组中仅出现一次。稀切核酸内切酶可识别7-核苷酸序列、8-核苷酸序列或更长的识别序列。稀切核酸内切酶的非限制性实例包括NotI、AscI、PacI、AsiSI、SbfI和FseI。
所述大范围核酸酶或稀切核酸内切酶还可以包含至少一种核定位信号、细胞穿透结构域、标记结构域和/或可检测标记,如上文(I)(a)(i)部分中所描述。
(vi)包含核酸酶结构域的可编程融合蛋白
在另外的实施方案中,所述可编程DNA修饰蛋白可以是融合蛋白,其包含与(双链切割)核酸酶结构域连接的可编程DNA结合结构域。所述融合蛋白的核酸酶结构域可以是上文(I)(a)(iii)部分中所描述的任何核酸酶结构域、源自CRISPR/Cas核酸酶的核酸酶结构域(例如Cas9的RuvC样或HNH样核酸酶结构域或Cpf1的核酸酶结构域)、或源自大范围核酸酶或稀切核酸内切酶的核酸酶结构域。
所述融合蛋白的可编程DNA结合结构域可以是经修饰而缺乏所有核酸酶活性的可编程核酸内切酶(即CRISPR/Cas核酸酶或大范围核酸酶)。因此,所述融合蛋白的DNA结合结构域可以是无催化活性的CRISPR/Cas系统或无催化活性的大范围核酸酶。或者,所述融合蛋白的可编程DNA结合结构域可以是可编程DNA结合蛋白,例如锌指蛋白或转录激活因子样效应子。在一些实施方案中,所述可编程DNA结合结构域可以是无催化活性的CRISPR/Cas核酸酶,其中通过突变和/或缺失去除核酸酶活性。例如,所述无催化活性的CRISPR/Cas蛋白可以是无催化活性的(死)Cas9(dCas9),其中RuvC样结构域包含D10A、D8A、E762A和/或D986A突变,并且HNH样结构域包含H840A、H559A、N854A、N865A和/或N863A突变。或者,所述无催化活性的CRISPR/Cas蛋白可以是无催化活性的(死)Cpf1蛋白,其在核酸酶结构域中包含可比较的突变。在其它实施方案中,所述可编程DNA结合结构域可以是无催化活性的大范围核酸酶,其中通过突变和/或缺失去除核酸酶活性,例如无催化活性的大范围核酸酶可以包含C-末端截短。
所述包含核酸酶活性的融合蛋白还可以包含至少一种核定位信号、细胞穿透结构域、标记结构域和/或可检测标记,如上文(I)(a)(i)部分中所描述。
(vii)包含非核酸酶结构域的可编程融合蛋白/复合体
在备选实施方案中,所述可编程DNA修饰蛋白可以是融合蛋白,其包含与非核酸酶修饰结构域连接的可编程DNA结合结构域。合适的可编程DNA结合结构域在上文(I)(a)(vi)部分中描述。
在一些实施方案中,所述非核酸酶修饰结构域可以是表观遗传修饰结构域,其改变DNA或染色质结构(并且可能改变或可能不改变DNA序列)。合适的表观遗传修饰结构域的非限制性实例包括具有DNA甲基转移酶活性(例如,胞嘧啶甲基转移酶)、DNA去甲基化酶活性、DNA脱氨基(例如胞嘧啶脱氨酶、腺苷脱氨酶、鸟嘌呤脱氨酶)、DNA氨基化、DNA解旋酶活性、组蛋白乙酰转移酶(HAT)活性(例如,源自E1A结合蛋白p300的HAT结构域)、组蛋白脱乙酰酶活性、组蛋白甲基转移酶活性、组蛋白去甲基化酶活性、组蛋白激酶活性、组蛋白磷酸酶活性、组蛋白泛素连接酶活性、组蛋白去泛素化活性、组蛋白腺苷酸化活性、组蛋白去乙酰化活性、组蛋白SUMO化活性,组蛋白去SUMO化活性、组蛋白核糖基化活性、组蛋白去核糖基化活性、组蛋白豆蔻酰化活性、组蛋白去豆蔻酰化活性、组蛋白瓜氨酸化活性、组蛋白烷基化活性、组蛋白脱烷基化活性或组蛋白氧化活性的表观遗传修饰结构域。在具体的实施方案中,所述表观遗传修饰结构域可包含胞嘧啶脱氨酶活性、组蛋白乙酰转移酶活性或DNA甲基转移酶活性。
在其它实施方案中,所述非核酸酶修饰结构域可以是转录激活结构域或转录阻遏结构域。合适的转录激活结构域包括但不限于单纯疱疹病毒VP16结构域、VP64(其为VP16的四聚体衍生物)、VP160、NFκB p65激活结构域、p53激活结构域1和2、CREB(cAMP反应元件结合蛋白)激活结构域、E2A激活结构域、来自人类热休克因子1(HSF1)的激活结构域或NFAT(活化T细胞的核因子)激活结构域。合适的转录阻遏结构域的非限制性实例包括诱导型cAMP早期阻遏物(ICER)结构域、Kruppel相关盒A(KRAB-A)阻遏结构域、YY1富含甘氨酸的阻遏结构域、Sp1样阻遏物、E(spl)阻遏物、IκB阻遏物或MeCP2。转录激活或转录阻遏结构域可以与DNA结合蛋白在基因上融合或通过非共价蛋白-蛋白、蛋白-RNA或蛋白-DNA相互作用结合。
在可编程DNA修饰蛋白包含CRISPR/Cas系统的实施方案中,CRISPR/Cas系统的引导RNA可包含结合转录激活因子、转录阻遏物或表观遗传修饰蛋白的适体序列(Konermannet al., Nature, 2015, 517(7536):583-588; Zalatan et al., Cell, 2015, 160(1-2):339-50)。
所述包含非核酸酶活性的融合蛋白还可以包含至少一种核定位信号、细胞穿透结构域、标记结构域和/或可检测标记,如上文(I)(a)(i)部分中所描述。
(b)可编程DNA结合蛋白
所述组合物还包含至少一种可编程DNA结合蛋白。可编程DNA结合蛋白是与特定DNA序列结合但不修饰DNA或DNA相关蛋白的蛋白。
在一些实施方案中,所述至少一种可编程DNA结合蛋白可以是经修饰而缺乏核酸酶活性的CRISPR/Cas核酸酶。例如,所述可编程DNA结合蛋白可以是无催化活性的CRISPR/Cas系统。为此,可以通过突变和/或缺失修饰CRISPR/Cas核酸酶以去除所有核酸酶活性。在一个实施方案中,RuvC样结构域和HNH样结构域均包含一个或多个突变和/或缺失以去除核酸酶活性。例如,所述无催化活性的CRISPR/Cas蛋白可以是无催化活性的(死)Cas9(dCas9),其中RuvC样结构域包含D10A、D8A、E762A和/或D986A突变,并且HNH样结构域包含H840A、H559A、N854A、N856A和/或N863A突变。或者,所述无催化活性的CRISPR/Cas蛋白可以是无催化活性的(死)Cpf1蛋白,其包含核酸酶结构域中的可比较的突变。在其它方面中,所述可编程DNA结合蛋白可以是CRISPR/Cas蛋白,其经修饰以切割双链序列的一条链(即是切口酶),如上文(I)(a)(ii)部分中所详述。
在其它实施方案中,所述至少一种可编程DNA结合蛋白可以是无催化活性的大范围核酸酶,其中通过突变和/或缺失去除核酸酶活性,例如无催化活性的大范围核酸酶可以包含C-末端截短。在其它实施方案中,所述至少一种可编程DNA结合蛋白可以是锌指蛋白或转录激活因子样效应子(TALE)。在另外的实施方案中,所述至少一种可编程DNA结合蛋白可以是CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。ZFN、TALEN和大范围核酸酶切口酶在核酸酶结构域或半结构域之一中包含突变和/或缺失,使得切口酶仅切割双链序列中的一条链。
所述可编程DNA结合蛋白还可以包含至少一种核定位信号、细胞穿透结构域、标记结构域和/或可检测标记,如上文(I)(a)(i)部分中所描述。
(c)编码可编程DNA修饰蛋白或可编程DNA结合蛋白的核酸
编码上文(I)(a)部分中所描述的可编程DNA修饰蛋白,或上文(I)(b)部分中所描述的可编程DNA结合蛋白的核酸,可以是DNA或RNA,可以是线性的或环状的,可以是单链的或双链的。可以对RNA或DNA进行密码子优化以在目的真核细胞中有效翻译成蛋白。密码子优化程序可作为免费软件或从商业来源获得。
在一些实施方案中,编码所述可编程DNA修饰蛋白或所述至少一种可编程DNA结合蛋白的核酸可以是mRNA。mRNA可以在体外合成。为此,编码DNA修饰蛋白或至少一种DNA结合蛋白的DNA可以与噬菌体RNA聚合酶识别的启动子序列可操作地连接,用于mRNA的体外合成。例如,启动子序列可以是T7、T3或SP6启动子序列或T7、T3或SP6启动子序列的变体。在这些实施方案中,体外转录的RNA可以被纯化、加帽和/或多腺苷酸化。如下详述的,所述编码DNA修饰蛋白或DNA结合蛋白的DNA可以是载体的一部分。
在其它实施方案中,所述编码可编程DNA修饰蛋白或至少一种可编程DNA结合蛋白的核酸可以是DNA。所述编码可编程DNA修饰蛋白或至少一种可编程DNA结合蛋白的DNA序列可以与至少一种启动子控制序列可操作地连接,用于在目的细胞中表达。在一些实施方案中,所述DNA编码序列还可以与多腺苷酸化信号(例如SV40 polyA信号、牛生长激素(BGH)polyA信号等)和/或至少一种转录终止序列连接。
在某些实施方案中,DNA编码序列可以与启动子序列可操作地连接,用于在细菌(例如大肠杆菌)细胞或真核生物(例如酵母、昆虫或哺乳动物)细胞中表达DNA修饰蛋白或DNA结合蛋白。合适的细菌启动子包括但不限于T7启动子、lac操纵子启动子、trp启动子、tac启动子(其是trp和lac启动子的杂合体)、任何前述的变体以及任何前述的组合。合适的真核启动子的非限制性实例包括组成型、调节型、或细胞或组织特异性启动子。合适的真核组成型启动子控制序列包括但不限于巨细胞病毒立即早期启动子(CMV)、猿猴病毒(SV40)启动子、腺病毒主要晚期启动子、劳氏肉瘤病毒(RSV)启动子、小鼠乳腺肿瘤病毒(MMTV)启动子、磷酸甘油激酶(PGK)启动子、延伸因子(ED1)-α启动子、遍在蛋白启动子、肌动蛋白启动子、微管蛋白启动子、免疫球蛋白启动子、其片段或任何前述的组合。合适的真核调节型启动子控制序列的实例包括但不限于由热休克、金属、类固醇、抗生素或醇调节的启动子控制序列。组织特异性启动子的非限制性实例包括B29启动子、CD14启动子、CD43启动子、CD45启动子、CD68启动子、结蛋白(desmin)启动子、弹性蛋白酶-1启动子、内皮糖蛋白(endoglin)启动子、纤连蛋白启动子、Flt-1启动子、GFAP启动子、GPIIb启动子、ICAM-2启动子、INF-β启动子、Mb启动子、NphsI启动子、OG-2启动子、SP-B启动子、SYN1启动子和WASP启动子。启动子序列可以是野生型的,或者其可以进行修饰以更有效率或更有效地表达。
在多个实施方案中,编码可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白的核酸可以在载体中存在。合适的载体包括质粒载体、噬菌粒、粘粒、人工/微型染色体、转座子和病毒载体(例如慢病毒载体、腺伴随病毒载体、腺病毒载体等)。在一个实施方案中,编码可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白的DNA可以在质粒载体中存在。合适的质粒载体的非限制性实例包括pUC、pBR322、pET、pBluescript及其变体。在其它实施方案中,编码可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白的核酸可以在病毒载体中存在。所述质粒或病毒载体可包含另外的表达控制序列(例如增强子序列、Kozak序列、多腺苷酸化序列、转录终止序列等)、选择标记序列(例如抗生素抗性基因)、复制起点等。其它信息可以在“Current Protocols in Molecular Biology” Ausubel
在可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白包含CRISPR/Cas蛋白或其变体的实施方案中,包含编码可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白的核酸的表达载体可进一步包含编码一个或多个引导RNA的序列。所述编码引导RNA的序列通常与至少一种转录控制序列可操作地连接,用于在目的真核细胞中表达引导RNA。例如,编码引导RNA的核酸可以与RNA聚合酶III(Pol III)识别的启动子序列可操作地连接。合适的Pol III启动子的实例包括但不限于哺乳动物U6、U3、H1和7SL RNA启动子。
(d)具体组合物
在一些实施方案中,所述可编程DNA修饰蛋白和一种或多种可编程DNA结合蛋白作为蛋白(或在某些情况下,作为蛋白-RNA复合物)提供。所述可编程DNA修饰蛋白和所述可编程DNA结合蛋白可以在细菌或真核细胞中表达,并使用本领域熟知的方法纯化。在其它实施方案中,所述可编程DNA修饰蛋白和一种或多种可编程DNA结合蛋白作为编码核酸提供。
在一些实施方案中,所述组合物可包含一种可编程DNA结合蛋白/系统或编码核酸。在其它实施方案中,所述组合物可包含两种可编程DNA结合蛋白/系统或编码核酸。在其它实施方案中,所述组合物可包含三种可编程DNA结合蛋白/系统或编码核酸。在进一步的实施方案中,所述组合物可包含四种可编程DNA结合蛋白/系统或编码核酸。在其它实施方案中,所述组合物可包含五种或更多种可编程DNA结合蛋白/系统或编码核酸。
在具体的实施方案中,所述可编程DNA修饰蛋白可包含CRISPR/Cas系统(例如CRISPR/Cas核酸酶、CRISPR/Cas双切口酶或与非核酸酶修饰结构域连接的无催化活性的(死)CRISPR/Cas蛋白),并且所述可编程DNA结合蛋白可以是缺乏核酸酶活性的CRISPR/Cas系统。例如,所述可所述编程DNA结合蛋白可以是无催化活性的CRISPR/Cas系统。通常,每种CRISPR/Cas蛋白包含至少一种核定位信号。在一些迭代中,所述组合物可以包含CRISPR/Cas系统作为CRISPR/Cas蛋白和引导RNA,其中所述蛋白和RNA可以是分开的实体或者所述蛋白和RNA可以复合在一起。所述引导RNA可以至少部分化学合成。所述引导RNA可以酶促合成。在其它迭代中,所述组合物可包含CRISPR/Cas蛋白和编码引导RNA的DNA。在其它迭代中,所述组合物可包含编码CRISPR/Cas蛋白的mRNA和编码引导RNA的DNA。在其它迭代中,所述组合物可包含编码CRISPR/Cas蛋白和/或引导RNA的质粒或病毒载体。在某些实施方案中,催化活性CRISPR/Cas蛋白和催化失活(死)CRISPR/Cas蛋白是Cas9蛋白。编码CRISPR/Cas蛋白的核酸通常经密码子优化以在目的真核细胞中最佳表达。
试剂盒
本公开内容的另一方面提供了包含以上在(I)部分中详述的组合物的试剂盒。如上详述的,所述试剂盒可以提供可编程DNA修饰蛋白和至少一种可编程DNA结合蛋白作为蛋白、蛋白-RNA复合物、或编码各种组分的核酸。所述试剂盒可进一步包含转染试剂、细胞生长培养基、选择培养基、体外转录试剂、核酸纯化试剂、蛋白纯化试剂、缓冲液等。本文提供的试剂盒通常包括用于实施下文详述的方法的说明书。所述试剂盒中所包含的说明书可以贴在包装材料上,或可以作为包装说明书包含在内。虽然说明书通常是书面或印刷材料,但它们不限于此。本公开内容预期能够存储这些说明书并将它们传送给最终用户的任何介质。这种介质包括但不限于电子存储介质(例如磁盘、磁带、盒带(cartridge)、芯片)、光学介质(例如CD ROM)等。如这里所使用的,术语“说明书”可以包括提供说明书的因特网站点的地址。
在一些实施方案中,试剂盒的可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白可包含II型CRISPR/Cas系统。在某些实施方案中,II型CRISPR/Cas系统的引导RNA可包含crRNA和tracrRNA。因此,所述试剂盒可以提供通用tracrRNA(s),并且试剂盒的最终用户可以提供序列特异性crRNA(s)。在一些方面中,所述试剂盒可包含II型CRISPR/Cas蛋白和tracrRNA(s)。在其它方面中,所述试剂盒可包含编码II型CRISPR/Cas蛋白的mRNA或DNA以及编码tracrRNA的DNA。
在其它实施方案中,试剂盒的可编程DNA修饰蛋白和/或至少一种可编程DNA结合蛋白可包含V型CRISPR/Cas系统。如上所述,所述V型CRISPR/Cas系统的引导RNA仅包含crRNA。在一些方面中,所述试剂盒可包含V型CRISPR/Cas蛋白和crRNA(s),或所述试剂盒可包含编码V型CRISPR/Cas蛋白的mRNA或DNA以及编码crRNA(s)的DNA。在其它方面中,所述试剂盒可仅包含V型CRISPR/Cas蛋白或编码V型CRISPR/Cas蛋白的核酸,其中所述试剂盒的最终用户提供crRNA(s)。
(III)提高靶向染色体位点可及性的方法
本公开内容的另一方面包括通过提高可编程DNA修饰蛋白对其染色体DNA中的靶序列的可及性来提高真核细胞中靶向基因组/表观遗传修饰的效率和/或特异性的方法。所述方法包含向目的真核细胞中引入(a)可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸和(a)至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸。将所述可编程DNA修饰蛋白工程化为识别并结合染色体DNA中的靶序列,在该位点上DNA修饰蛋白可以修饰DNA或相关蛋白。将一种或多种可编程DNA结合蛋白中的每种都工程化以识别和结合DNA修饰蛋白的靶染色体序列附近的序列。可编程DNA修饰蛋白和可编程DNA结合蛋白在以上部分(I)中详述。
通常,靶染色体序列附近的序列位于靶染色体序列任一侧(即上游或下游)的约250个碱基对内。附近位点可位于双链DNA的任一链上。在一些实施方案中,靶染色体序列附近的序列可位于与所述DNA修饰蛋白的靶染色体序列相距小于约250bp、小于约200bp、小于约150bp、小于约100bp、小于约75bp、小于约50bp、小于约25bp、小于约20bp、小于约15bp、小于约10bp或小于约5bp的位置。在某些实施方案中,靶染色体序列附近的序列可位于靶染色体序列任一侧的约1bp至约10bp、约11bp至约20bp、约21bp至约30bp、约31bp至约40bp、约41bp至约50bp、约51bp至约60bp、约61bp至约70bp、约71bp至约80bp、约81bp至约90bp、约91bp至约100bp、约101bp至约150bp、约151bp至约200bp、或约201bp至约250bp的位置。在其它实施方案中,靶染色体序列附近的序列可位于靶染色体序列任一侧的约5bp至约75bp、约10bp至约50bp、或约15bp至约25bp的位置。
在一些实施方案中,所述方法包括向细胞中引入至少一种可编程DNA结合蛋白,其结合序列位于靶染色体序列的上游或下游。在其它实施方案中,所述方法包括向细胞中引入至少两种可编程DNA结合蛋白,其中一种的结合序列位于靶染色体序列的上游,另一种的结合序列位于靶染色体序列的下游。在进一步的实施方案中,所述方法包括向细胞中引入至少三种可编程DNA结合蛋白,其结合序列位于靶染色体序列的上游或下游。在其它的实施方案中,所述方法包括向细胞中引入四种或更多种可编程DNA结合蛋白,其结合序列位于靶染色体序列的上游或下游。在这些实施方案中,例如,所述方法可包括引入一、二、三、四、五、六、七、八、九、十种或超过十种的可编程DNA结合蛋白,其结合序列位于靶染色体序列任一侧(即上游或下游)的约250bp内。
一种或多种可编程DNA结合蛋白中的每种与靶染色体序列附近位点的结合改变了局部染色质构型,导致可编程DNA修饰蛋白对(先前不可及的)靶染色体序列的可及性提高(参见图1)。结果,DNA修饰蛋白的修饰效率提高(参见例如实施例1-3)。换言之,与将DNA修饰蛋白单独引入细胞时相比,当将DNA修饰蛋白与一种或多种可编程DNA结合蛋白组合引入细胞时,DNA修饰蛋白的修饰效率提高。
此外,本文公开的方法提高了靶向基因组修饰的特异性。虽然将可编程DNA修饰蛋白工程化以识别和结合特定染色体基因座中的靶序列,但是相同或接近相同的序列可存在于其它的染色体位置(导致脱靶效应)。然而在可编程DNA修饰蛋白与靶染色体序列的结合很大程度上取决于一种或多种可编程DNA结合蛋白与靶染色体序列附近序列的结合的实施方案中,所述一种或多种可编程DNA结合蛋白与目的染色体基因座中接近靶序列的位点的结合提供了对修饰事件的额外特异性(参见实施例4)。
因此,本文公开的方法可以提高靶向基因组编辑(例如基因校正、基因敲除、基因敲入等)、靶向表观遗传修饰和靶向转录调节的效率和/或特异性。
(a)引入细胞
如所述的,所述方法包括向细胞中引入(a)可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸和(b)至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸。可编程DNA修饰蛋白在以上(I)(a)部分中详述,可编程DNA结合蛋白在以上(I)(b)部分中详述,并且编码DNA修饰蛋白或可编程DNA结合蛋白的核酸在以上(I)(c)部分中描述。
所述可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸和至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸可通过多种方法引入目的细胞中。
在一些实施方案中,可以用合适的分子(即蛋白、DNA和/或RNA)转染细胞。合适的转染方法包括核转染(或电穿孔)、磷酸钙介导的转染、阳离子聚合物转染(例如DEAE-葡聚糖或聚乙烯亚胺)、病毒转导、病毒体转染、病毒粒子转染、脂质体转染、阳离子脂质体转染、免疫脂质体转染、非脂质体脂质转染、树枝状大分子转染、热休克转染、磁转染、脂质转染(lipofection)、基因枪递送、穿刺转染(impalefection)、细胞超声(sonoporation)、光学转染和核酸的专有试剂增强摄取。转染方法是本领域熟知的(参见,例如“CurrentProtocols in Molecular Biology” Ausubel
可以将多种分子同时或依次引入细胞中。例如,可以同时引入可编程DNA修饰蛋白(或其编码核酸)和至少一种可编程DNA结合蛋白(或其编码核酸)。或者,可以先引入一种,然后再将另一种引入细胞中。
通常,细胞在适于细胞生长和/或维持的条件下维持。合适的细胞培养条件是本领域熟知的,并且描述于例如Santiago
(b)靶向基因组修饰
所述一种或多种可编程DNA结合蛋白与靶染色体序列附近序列的结合改变局部染色质构型,例如可以改变核小体结构和/或可以置换组蛋白。因此,与单独使用可编程DNA修饰蛋白时相比,可编程DNA修饰蛋白能够更好地接近靶染色体序列。提高的可及性导致靶向基因组修饰的效率和/或特异性提高。靶向基因组/表观遗传修饰可以由具有核酸酶活性或非核酸酶活性的DNA修饰蛋白介导。
在可编程DNA修饰蛋白具有核酸酶活性的实施方案中,DNA修饰蛋白可在靶向染色体序列处引入双链断裂。染色体序列中的双链断裂可以通过非同源末端连接(NHEJ)修复过程来修复。因为NHEJ易于出错,所以在断裂修复期间可发生至少一个核苷酸的缺失、至少一个核苷酸的插入、至少一个核苷酸的取代或其组合。因此,可以修饰或灭活靶向染色体序列。例如,编码序列的阅读框移位中的缺失、插入或取代可导致蛋白产物改变或不产生蛋白产物(称为“敲除”)。在一些迭代中,所述方法可以进一步包括向细胞中引入供体多核苷酸(参见下文),所述供体多核苷酸包含供体序列,所述供体序列侧接与位于靶染色体序列任一侧的序列具有基本序列同一性的序列,使得在通过同源定向修复过程(HDR)修复双链断裂期间,供体多核苷酸中的供体序列可以与靶染色体序列处的染色体序列交换或整合到所述染色体序列中。外源序列的整合被称为“敲入”。如上详述的,本文公开的方法还降低脱靶效应,从而提高靶向基因组修饰的特异性。
因此,在多种迭代中,相对于单独使用具有核酸酶活性的可编程DNA修饰蛋白时,靶向基因组修饰的效率和/或特异性可以提高至少约0.1倍、至少约0.5倍、至少约1倍、至少约2倍、至少约2倍、至少约5倍、至少约10倍、或至少约20倍、至少约50倍、至少约100倍或超过约100倍。例如,当单独使用具有核酸酶活性的可编程DNA修饰蛋白时,可以没有可检测的indel或整合事件。然而,当所述具有核酸酶活性的可编程DNA修饰蛋白与至少一种可编程DNA结合蛋白组合使用时,可检测到indel和整合事件(例如至少约1% indel/整合、至少约5% indel/整合、至少约10% indel/整合、至少约20% indel/整合、至少约30% indel/整合、至少约40% indel/整合、至少约50% indel/整合或超过约50% indel/整合)。
在可编程DNA修饰蛋白具有非核酸酶活性的实施方案中,所述DNA修饰蛋白可修饰靶染色体序列处的DNA或相关蛋白或修饰靶染色体序列的表达。例如,当所述可编程DNA修饰蛋白包含表观遗传修饰活性时,组蛋白的乙酰化、甲基化、磷酸化、腺苷酸化等状态可以被修饰,或者DNA甲基化、氨基化等状态可以被修饰。例如,在可编程DNA修饰蛋白包含胞嘧啶脱氨酶活性的实施方案中,靶染色体序列上的一个或多个胞嘧啶残基可以转化为尿嘧啶残基。或者,当可编程DNA修饰蛋白包含转录激活或阻遏活性时,可以提高或降低靶染色体序列处的转录。相对于单独使用具有非核酸酶活性的可编程DNA修饰蛋白时,所得的表观遗传修饰或转录调节可以提高至少约0.1倍、至少约0.5倍、至少约1倍、至少约2倍、至少约5倍、至少约10倍、或至少约20倍、至少约50倍、至少约100倍或超过约100倍。
上文详述的靶向基因组修饰/表观遗传修饰可以单独进行或多重进行(即可以同时靶向两种或更多种染色体序列)。
(c)可选的供体多核苷酸
在所述可编程DNA修饰蛋白包含核酸酶活性的实施方案中,所述方法可以进一步包括将至少一种供体多核苷酸引入细胞中。所述供体多核苷酸可以是单链或双链的,可以是线性或环状的,并且/或可以是RNA或DNA。在一些实施方案中,所述供体多核苷酸可以是载体,例如质粒载体。
所述供体多核苷酸包含至少一种供体序列。在一些方面中,所述供体多核苷酸的供体序列可以是内源或天然染色体序列的修饰形式。例如,所述供体序列可以与DNA修饰蛋白靶向的序列处或附近的染色体序列的一部分基本相同,但其包含至少一个核苷酸变化。因此,在与天然序列整合或交换后,靶向染色体位置处的序列包含至少一个核苷酸变化。例如,所述改变可以是一个或多个核苷酸的插入、一个或多个核苷酸的缺失、一个或多个核苷酸的取代,或其组合。作为修饰序列的“基因校正”整合的结果,细胞可以从靶向染色体序列产生修饰的基因产物。
在其它方面中,所述供体多核苷酸的供体序列可以是外源序列。如本文所用,“外源”序列是指对于细胞不是天然的序列,或其天然位置在细胞基因组中的不同位置的序列。例如,所述外源序列可以包含蛋白编码序列,其可以与外源启动子控制序列可操作地连接,使其在整合到基因组中时,细胞能够表达由整合序列编码的蛋白。或者,所述外源序列可以整合到染色体序列中,使得其表达受内源启动子控制序列的调节。在其它迭代中,所述外源序列可以是转录控制序列、其它表达控制序列、RNA编码序列等。如上所述,将外源序列整合到染色体序列中称为“敲入”。
如本领域技术人员可以理解的,供体序列的长度可以并将会变化。例如,所述供体序列的长度可以从几个核苷酸到数百个核苷酸到数十万个核苷酸变化。
通常,所述供体多核苷酸中的供体序列侧接上游序列和下游序列,其分别与位于可编程DNA修饰蛋白靶向的序列的上游和下游的序列具有基本序列同一性。由于这些序列的相似性,所述供体多核苷酸的上游和下游序列允许供体多核苷酸和靶向染色体序列之间的同源重组,使得供体序列可以整合到染色体序列中(或与染色体序列交换)。
如本文所用,上游序列是指与可编程DNA修饰蛋白靶向的序列上游的染色体序列具有基本序列同一性的核酸序列。类似地,下游序列是指与可编程DNA修饰蛋白靶向的序列下游的染色体序列具有基本序列同一性的核酸序列。如本文所用,短语“基本序列同一性”是指具有至少约75%序列同一性的序列。因此,供体多核苷酸中的上游和下游序列可与靶序列上游或下游序列具有约75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列同一性。在一个示例性实施方案中,供体多核苷酸中的上游和下游序列可与可编程DNA修饰蛋白靶向的序列上游或下游的染色体序列具有约95%或100%的序列同一性。
在一些实施方案中,上游序列与位于可编程DNA修饰蛋白靶向的序列的紧邻上游的染色体序列具有基本序列同一性。在其它实施方案中,上游序列与位于靶序列上游约一百(100)个核苷酸内的染色体序列具有基本序列同一性。因此,例如,上游序列可以与位于靶序列上游的约1至约20、约21至约40、约41至约60、约61至约80、或约81至约100个核苷酸的染色体序列具有基本序列同一性。在一些实施方案中,下游序列与位于可编程DNA修饰蛋白靶向的序列的紧邻下游的染色体序列具有基本序列同一性。在其它实施方案中,下游序列与位于靶序列下游约一百(100)个核苷酸内的染色体序列具有基本序列同一性。因此,例如,下游序列可以与位于靶序列下游的约1至约20、约21至约40、约41至约60、约61至约80、或约81至约100个核苷酸的染色体序列具有基本序列同一性。
每个上游或下游序列的长度范围可为约20个核苷酸至约5000个核苷酸。在一些实施方案中,上游和下游序列可包含约50、100、200、300、400、500、600、700、800、900、1000、1100、1200、1300、1400、1500、1600、1700、1800、1900、2000、2100、2200、2300、2400、2500、2600、2800、3000、3200、3400、3600、3800、4000、4200、4400、4600、4800或5000个核苷酸。在具体的实施方案中,上游或下游序列的长度范围可为约50至约1500个核苷酸。
(e)细胞类型
多种细胞都适用于本文公开的方法。通常,所述细胞是真核细胞。例如,所述细胞可以是人类哺乳动物细胞、非人类哺乳动物细胞、非哺乳动物脊椎动物细胞、无脊椎动物细胞、昆虫细胞、植物细胞、酵母细胞或单细胞真核生物。在其它实施方案中,所述细胞也可以是单细胞胚胎。例如,非人类哺乳动物胚胎,包括大鼠、仓鼠、啮齿动物、兔、猫、犬、绵羊、猪、牛、马和灵长类动物胚胎。在其它实施方案中,所述细胞可以是干细胞,例如胚胎干细胞、ES样干细胞、胎儿干细胞、成体干细胞等。在一个实施方案中,所述干细胞不是人类胚胎干细胞。此外,所述干细胞可以包括通过WO2003/046141(以其整体并入本文)或Chung等人(CellStem Cell, 2008, 2:113-117)中公开的技术制备的干细胞。所述细胞可以是体外的或体内的(即在生物体内)。在示例性实施方案中,所述细胞是哺乳动物细胞。在具体的实施方案中,所述细胞是人类细胞。
合适的哺乳动物细胞的非限制性实例包括人胚肾细胞(HEK293、HEK293T);人宫颈癌细胞(HELA);人肺细胞(W138);人肝细胞(Hep G2);人U2-OS骨肉瘤细胞;人A549细胞;人A-431细胞和人K562细胞;中国仓鼠卵巢(CHO)细胞;幼仓鼠肾(BHK)细胞;小鼠骨髓瘤NS0细胞;小鼠胚胎成纤维细胞3T3细胞(NIH3T3);小鼠B淋巴瘤A20细胞;小鼠黑色素瘤B16细胞;小鼠成肌细胞C2C12细胞;小鼠骨髓瘤SP2/0细胞;小鼠胚胎间充质C3H-10T1/2细胞;小鼠癌CT26细胞;小鼠前列腺DuCuP细胞;小鼠乳腺EMT6细胞;小鼠肝细胞瘤Hepa1c1c7细胞;小鼠骨髓瘤J5582细胞;小鼠上皮MTD-1A细胞;小鼠心肌MyEnd细胞;小鼠肾脏RenCa细胞;小鼠胰腺RIN-5F细胞;小鼠黑色素瘤X64细胞;小鼠淋巴瘤YAC-1细胞;大鼠胶质母细胞瘤9L细胞;大鼠B淋巴瘤RBL细胞;大鼠神经母细胞瘤B35细胞;大鼠肝细胞瘤细胞(HTC);buffalo大鼠肝脏BRL 3A细胞;犬肾细胞(MDCK);犬乳腺(CMT)细胞;大鼠骨肉瘤D17细胞;大鼠单核细胞/巨噬细胞DH82细胞;猴肾SV-40转化成纤维细胞(COS7)细胞;猴肾CVI-76细胞;非洲绿猴肾(VERO-76)细胞。可以在美国典型培养物保藏中心目录(ATCC,Manassas,VA)中找到广泛的哺乳动物细胞系列表。
(IV)检测特定基因组基因座的方法
本文还提供了用于检测或可视化真核细胞中特定基因组基因座的方法。由于一种或多种可编程DNA结合蛋白的邻近结合改变了染色质结构并提高了可编程DNA修饰蛋白对先前不可及的染色体基因座的可及性,因此可以修改上文在(III)部分中描述的方法以增强检测特定基因组基因座或靶向染色体序列。所述方法包括向真核细胞中引入(a)包含至少一种可检测标记结构域的可编程DNA结合蛋白或编码包含至少一种可检测标记结构域的可编程DNA结合蛋白的核酸,和(b)至少一种可编程DNA结合蛋白或编码所述至少一种可编程DNA结合蛋白的核酸,其中包含至少一种可检测标记结构域的可编程DNA结合蛋白靶向靶染色体序列,并且所述一种或多种可编程DNA结合蛋白中的每种都靶向于靶染色体序列附近的位点。将至少一种可编程DNA结合蛋白结合到靶染色体序列附近的位点提高了包含至少一种可检测标记结构域的可编程DNA结合蛋白对靶染色体序列的可及性。所述方法进一步包括检测与靶染色体序列结合的包含至少一种可检测标记结构域的可编程DNA结合蛋白。
所述包含至少一种可检测标记结构域的可编程DNA结合蛋白包含可编程DNA结合结构域。合适的可编程DNA结合结构域在上文(I)(a)(vi)部分中描述。在具体的实施方案中,所述可编程DNA结合结构域可以是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白或转录激活因子样效应子。所述可编程DNA结合蛋白的至少一种可检测标记结构域可以是荧光蛋白(例如GFP、eGFP、RFP等)、荧光标签或表位标签(其在上文(I)(a)(i)部分中描述)。在某些实施方案中,所述可编程DNA结合蛋白的至少一种可检测标记结构域可以是可编程DNA结合蛋白内的天然存在的表位,使得可编程DNA结合蛋白可以通过针对可编程DNA结合蛋白的抗体检测。所述包含至少一种可检测标记结构域的可编程DNA结合蛋白可以进一步包含至少一种核定位信号和/或细胞穿透结构域,如上文(I)(a)(i)部分中所述。在一些实施方案中,所述包含至少一种可检测标记结构域的可编程DNA结合蛋白可以进一步包含非核酸酶修饰结构域(如上文(I)(a)(vi)部分中所述)。
所述一种或多种可编程DNA结合蛋白在上文(I)(b)部分中描述。通常,所述至少一种可编程DNA结合蛋白可以是无催化活性的CRISPR/Cas蛋白、无催化活性的大范围核酸酶、锌指蛋白、转录激活因子样效应子、CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。
所述方法进一步包括检测与靶染色体序列结合的包含可检测标记结构域的可编程DNA结合蛋白,其中所述检测可以通过动态活细胞成像、荧光显微术、共聚焦显微术、免疫荧光、免疫检测、RNA-蛋白结合、蛋白-蛋白结合等进行。所述检测步骤可以在活细胞或固定细胞中进行。
在所述方法包括检测活细胞中染色质结构动力学的实施方案中,包含可检测标记结构域的可编程DNA结合蛋白和一种或多种可编程DNA结合蛋白可以作为蛋白或核酸引入细胞中,基本上如上(III)(a)部分所述。在所述方法包括检测固定细胞中的靶向染色体序列的实施方案中,可将包含可检测标记结构域的可编程DNA结合蛋白和可编程DNA结合蛋白作为蛋白(或RNA-蛋白复合物)引入细胞中。固定和透化细胞的方法是本领域熟知的。在一些实施方案中,可以对固定细胞进行化学和/或热变性过程以将双链染色体DNA转化为单链DNA。在其它实施方案中,固定细胞不经历化学和/或热变性过程。
在具体的实施方案中,所述包含可检测标记结构域的可编程DNA结合蛋白是包含无催化活性(或死)CRISPR/Cas蛋白和荧光蛋白标记结构域的融合蛋白,并且所述至少一种可编程DNA结合蛋白是无催化活性的(或死)CRISPR/Cas蛋白。
在其中至少一种可编程DNA修饰或DNA结合蛋白包含CRISPR/CAS蛋白的实施方案中,引导RNA可进一步包含用于原位检测的可检测标记(例如FISH或CISH)。可检测标记在以上(I)(a)(i)部分中详述。在一些实施方案中,可编程DNA修饰和DNA结合蛋白各自包含CRISPR/Cas蛋白并且各引导RNA包含至少一种可检测标记,由此提高待检测信号的量或强度。
在其它实施方案中,可以通过邻近连接测定法检测邻近结合的可编程DNA修饰蛋白和一种或多种可编程DNA结合蛋白。例如,所述可编程DNA修饰蛋白可以被第一抗体结合,并且至少一种可编程DNA结合蛋白可以被第二抗体结合,其中每种都可以直接或间接(例如通过第二抗体)连接单链邻近检测寡核苷酸。在其它实施方案中,单链邻近检测寡核苷酸可以直接或间接连接至引导RNA。在其它实施方案中,单链邻近检测寡核苷酸可以直接或间接地连接至可编程DNA修饰或可编程DNA结合蛋白。可以通过原位邻近依赖性扩增反应,检测与位于邻近的染色体结合蛋白复合的邻近检测寡核苷酸。所述原位邻近依赖性扩增反应可以是邻近连接测定法(PLA,参见Söderberg, et al., Nature Methods, 2006, 3(12):995-1000)或杂交链反应的邻近依赖性引发(proxHCR,参见Koos et al., NatureCommunications, 2015, 6:7294, 10 pp.)。
(V)应用
本文公开的组合物和方法可用于多种治疗、诊断、工业和研究应用。在一些实施方案中,本公开内容可用于修饰细胞、动物或植物中的任何目的染色体序列,以模拟和/或研究基因的功能、研究感兴趣的遗传或表观遗传条件、或研究涉及多种疾病或病症的生物化学途径。例如,可以产生模拟疾病或病症的转基因生物,其中改变与疾病或病症相关的一种或多种核酸序列的表达。所述疾病模型可用于研究突变对生物体的影响,研究疾病的发展和/或进展,研究药学活性化合物对疾病的影响,和/或评估潜在基因疗法策略的功效。
在其它实施方案中,所述组合物和方法可用于进行有效且成本有效的功能基因组筛选,其可用于研究参与特定生物过程的基因的功能以及基因表达的任何改变如何影响生物过程,或者与细胞表型一起进行基因组基因座的饱和或深度扫描诱变。饱和或深度扫描诱变可用于确定例如基因表达、抗药性和疾病逆转所需的功能元件的关键最小特征和离散脆弱性。
在进一步的实施方案中,本文公开的组合物和方法可用于诊断测试以确定疾病或病症的存在和/或用于确定治疗选择。合适的诊断测试的实例包括检测癌细胞中的特定突变(例如EGFR、HER2等中的特定突变)、检测与特定疾病相关的特定突变(例如三核苷酸重复、与镰状细胞疾病相关的β-珠蛋白突变、特定SNP等)、肝炎的检测、病毒的检测(例如寨卡)等。
在另外的实施方案中,本文公开的组合物和方法可用于校正与特定疾病或病症相关的基因突变,例如校正与镰状细胞病或地中海贫血相关的珠蛋白基因突变,校正与严重联合免疫缺陷(SCID)相关的腺苷脱氨酶基因的突变,降低亨廷顿氏病的致病基因HTT的表达,或校正视紫红质基因的突变用于治疗色素性视网膜炎。这些修饰可以在离体细胞中进行。
在其它实施方案中,本文公开的组合物和方法可用于产生具有改善的性状或增加的对环境胁迫的抗性的作物植物。本公开内容还可用于产生具有改良性状的农场动物或生产动物。例如,猪具有许多特征使其作为具有吸引力的生物医学模型,特别是在再生医学或异种移植中。
定义
除非另外指明,本文使用的所有技术和科学术语具有与本发明所属领域的普通技术人员通常理解的相同含义。以下参考文献为本领域技术人员提供了本发明中使用的许多术语的一般定义:Singleton
当介绍本公开内容的要素或其优选实施方案时,冠词“一”、“一个”、“该”和“所述”意指有一个或多个该要素。术语“包含”、“包括”和“具有”旨在包含性的,并且意味着可能存在除所列要素之外的其它要素。
术语“约”当关于数值例如x使用时,表示x±5%。
如本文所用,术语“互补”或“互补性”是指双链核酸通过特定氢键的碱基配对的结合。碱基配对可以是标准的Watson-Crick碱基配对(例如5’-A G T C-3’与互补序列3’-T CA G-5’配对)。碱基配对也可以是Hoogsteen或反向Hoogsteen氢键键合。通常相对于双链体区域测量互补性,因此例如排除突出。双链区的两条链之间的互补性可以是部分的,并且如果仅有部分(例如70%)碱基是互补的,则表示为百分比(例如70%)。不互补的碱基是“错配的”。如果双链体区域中的所有碱基都是互补的,则互补性也可以是完全的(即100%)。
如本文所用,术语“CRISPR/Cas系统”是指包含CRISPR/Cas蛋白(即核酸酶、切口酶或催化死蛋白)和引导RNA的复合物。
如本文所用,术语“内源序列”是指对细胞天然的染色体序列。
如本文所用,术语“外源的”是指对细胞不是天然的序列,或其在细胞基因组中的天然位置处于不同染色体位置的染色体序列。
如本文所用,“基因”是指编码基因产物的DNA区域(包括外显子和内含子),以及调节基因产物产生的所有DNA区域,无论这些调节序列是否与编码和/或转录序列相邻。因此,基因包括但不必然限于启动子序列、终止子、翻译调节序列如核糖体结合位点和内部核糖体进入位点、增强子、沉默子、绝缘子、边界元件、复制起点、基质附着位点和基因座控制区域。
术语“异源的”是指对目的细胞而言不是内源的或天然的实体。例如,异源蛋白是指衍生自或最初源自外源的蛋白的蛋白,例如外源引入的核酸序列。在某些情况下,所述异源蛋白通常不是由目的细胞产生的。
如本文所用,术语“局部染色质结构”或“局部染色质构型”是指核小体结构和/或组蛋白蛋白间隔,并且通常不是指核小体压缩成染色质纤维和异染色质。
术语“切口酶”是指切割双链核酸序列中的一条链(即切割双链序列)的酶。例如,具有双链切割活性的核酸酶可以通过突变和/或缺失进行修饰以起到切口酶的作用,并且仅切割双链序列的一条链。
如本文所用,术语“核酸酶”是指切割双链核酸序列的两条链的酶。
术语“核酸”和“多核苷酸”是指脱氧核糖核苷酸或核糖核苷酸聚合物,其呈线状或环状构象,并且呈单链或双链形式。出于本公开的目的,这些术语不应被解释为关于聚合物长度的限制。所述术语可包括天然核苷酸的已知类似物以及在碱基、糖和/或磷酸酯部分(例如硫代磷酸酯骨架)中被修饰的核苷酸。通常,特定核苷酸的类似物具有相同的碱基配对特异性;即A的类似物将与T碱基配对。
术语“核苷酸”是指脱氧核糖核苷酸或核糖核苷酸。核苷酸可以是标准核苷酸(即腺苷、鸟苷、胞苷、胸苷和尿苷)、核苷酸异构体或核苷酸类似物。核苷酸类似物是指具有修饰的嘌呤或嘧啶碱基或修饰的核糖部分的核苷酸。核苷酸类似物可以是天然存在的核苷酸(例如肌苷、假尿苷等)或非天然存在的核苷酸。对核苷酸的糖或碱基部分的修饰的非限制性实例包括添加(或除去)乙酰基、氨基、羧基、羧甲基、羟基、甲基、磷酰基和硫醇基团,以及用其它原子(例如7-脱氮嘌呤)取代碱基的碳和氮原子。核苷酸类似物还包括双脱氧核苷酸、2'-O-甲基核苷酸、锁核酸(LNA)、肽核酸(PNA)和吗啉基核酸(morpholino)。
术语“多肽”和“蛋白”可互换使用,指氨基酸残基的聚合物。
如本文所用,术语“附近位点”是指位于染色体DNA中靶序列任一侧的约250个碱基对内的结合位点或核苷酸序列。
如本文所用,术语“可编程DNA修饰蛋白”是指经工程改造以结合染色体DNA中的特定靶序列并且在靶序列处或附近修饰DNA或DNA相关蛋白的蛋白。
如本文所用,术语“可编程DNA结合蛋白”是指经工程改造以结合染色体DNA中的特定靶序列的蛋白,但所述蛋白不会在靶序列处或附近修饰DNA或与DNA相关的蛋白。
术语“靶序列”、“靶染色体序列”和“靶位点”可互换使用,指可编程DNA修饰蛋白靶向的染色体DNA中的特定序列,以及可编程DNA修饰蛋白修饰DNA或DNA相关蛋白的位点。
用于确定核酸和氨基酸序列同一性的技术是本领域已知的。通常,此类技术包括确定基因的mRNA的核苷酸序列和/或确定由其编码的氨基酸序列,并将这些序列与第二核苷酸或氨基酸序列进行比较。也可以用这种方式确定和比较基因组序列。通常,同一性分别指两个多核苷酸或多肽序列的精确核苷酸与核苷酸或氨基酸与氨基酸的对应性。可以通过确定它们的百分比同一性来比较两个或更多个序列(多核苷酸或氨基酸)。无论是核酸还是氨基酸序列的两个序列的同一性百分比是两个比对序列之间的精确匹配数除以较短序列的长度并乘以100。Smith和Waterman, Advances in Applied Mathematics 2:482-489(1981)的局部同源性算法提供了核酸序列的近似比对。可以通过使用由Dayhoff, Atlasof Protein Sequences and Structure, M. O. Dayhoff ed., 5 suppl. 3:353-358,National Biomedical Research Foundation, Washington, D.C., USA开发的并由Gribskov, Nucl. Acids Res. 14(6):6745-6763 (1986)归一化的评分矩阵将所述算法应用于氨基酸序列。确定序列的百分比同一性的该算法的示例性实施方式由GeneticsComputer Group(Madison, Wis.)在“BestFit”实用应用中提供。用于计算序列之间的百分比同一性或相似性的其它合适程序通常是本领域已知的,例如另一个比对程序是BLAST,采用默认参数使用。例如,BLASTN和BLASTP可以采用以下默认参数使用:遗传密码=标准;过滤器=无;链=两条;截留值= 60;期望= 10;矩阵= BLOSUM62;描述= 50个序列;排序依据=HIGH SCORE;数据库=非冗余,GenBank+EMBL+DDBJ+PDB+GenBank CDS translations+Swissprotein+Spupdate+PIR。这些程序的详细信息可以在GenBank网站上找到。
由于可以在不脱离本发明的范围的情况下对上述细胞和方法进行各种改变,所以包含在以上描述和下面给出的实施例中的所有内容应当旨在被解释为说明性的而不是限制性意义。
列举的实施方案
提供以下列举的实施方案以说明本发明的某些方面,并且不旨在限制其范围。
1.一种组合物,包含:(a)可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸和(b)至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸。
2.实施方案1的组合物,其中所述可编程DNA修饰蛋白是RNA引导的成簇规律间隔的短回文重复(CRISPR)/CRISPR相关(Cas)(CRISPR/Cas)核酸酶系统、CRISPR/Cas双切口酶系统、锌指核酸酶(ZFN)、转录激活因子样效应核酸酶(TALEN)、大范围核酸酶、包含与核酸酶结构域连接的可编程DNA结合结构域的融合蛋白,或包含与非核酸酶结构域连接的可编程DNA结合结构域的融合蛋白。
3.实施方案2的组合物,其中所述融合蛋白的可编程DNA结合结构域是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白或转录激活因子样效应子。
4.实施方案2或3的组合物,其中所述融合蛋白的非核酸酶结构域具有乙酰转移酶活性、脱乙酰酶活性、甲基转移酶活性、去甲基酶活性、激酶活性、磷酸酶活性、泛素连接酶活性、去泛素化活性、腺苷酸化活性、去腺苷酸化活性、SUMO化活性、去SUMO化活性、核糖基化活性、去核糖基化活性、豆蔻酰化活性、去豆蔻酰化活性、瓜氨酸化活性、解旋酶活性、氨基化活性、脱氨基化活性、烷基化活性、脱烷基化活性、氧化活性、转录激活活性或转录阻遏活性。
5.实施方案4的组合物,其中所述融合蛋白的非核酸酶结构域具有胞嘧啶脱氨酶活性、组蛋白乙酰转移酶活性、转录激活活性或转录阻遏活性。
6.实施方案1至5中任一项的组合物,其中所述至少一种可编程DNA结合蛋白是无催化活性的CRISPR/Cas蛋白、无催化活性的大范围核酸酶、锌指蛋白、转录激活因子样效应子、CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。
7.实施方案1至6中任一项的组合物,其中编码所述可编程DNA修饰蛋白和至少一种可编程DNA结合蛋白的核酸是RNA或DNA,和/或其中所述核酸是质粒载体或病毒载体的一部分。
8.实施方案1至6中任一项的组合物,其中所述可编程DNA修饰蛋白是CRISPR/Cas核酸酶系统、CRISPR/Cas双切口酶系统、或与非核酸酶结构域连接的无催化活性的CRISPR/Cas系统,并且所述至少一种可编程DNA结合蛋白是无催化活性的CRISPR/Cas系统,其中每种CRISPR/Cas系统包含CRISPR/Cas蛋白和引导RNA。
9.实施方案8的组合物,其中每种CRISPR/Cas核酸酶系统是I型CRISPR/Cas系统、II型CRISPR/Cas系统、III型CRISPR/Cas系统或V型CRISPR/Cas系统。
10.实施方案9的组合物,其中每种CRISPR/Cas核酸酶系统是II型CRISPR/Cas系统或V型CRISPR/Cas系统。
11.实施方案8至10中任一项的组合物,其中编码每种CRISPR/Cas蛋白的核酸是mRNA或DNA。
12.实施方案8至11中任一项的组合物,其中编码每种CRISPR/Cas蛋白的核酸和/或编码每种引导RNA的核酸是质粒载体或病毒载体的一部分。
13.实施方案8至11中任一项的组合物,其中每种CRISPR/Cas系统的引导RNA是酶促合成的。
14.实施方案8至11中任一项的组合物,其中每种CRISPR/Cas系统的引导RNA至少部分是化学合成的。
15.一种试剂盒,其包含实施方案1至14中任一项的组合物。
16.一种用于提高真核细胞中靶向基因组修饰效率和/或特异性的方法,所述方法包括向真核细胞中引入:
可编程DNA修饰蛋白或编码可编程DNA修饰蛋白的核酸;和
至少一种可编程DNA结合蛋白或编码至少一种可编程DNA结合蛋白的核酸;
其中所述可编程DNA修饰蛋白靶向于靶染色体序列,并且至少一种可编程DNA结合蛋白中的每种都靶向于靶染色体序列附近的位点,并且所述至少一种可编程DNA结合蛋白与靶染色体序列附近位点的结合提高可编程DNA修饰蛋白对靶染色体序列的可及性,从而提高靶向基因组修饰效率和/或特异性。
17.实施方案16的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约250个碱基对内。
18.实施方案17的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约100个碱基对内。
19.实施方案18的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约75个碱基对内。
20.实施方案19的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约50个碱基对内。
21.实施方案20的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约25个碱基对内。
22.实施方案16至21中任一项的方法,其中所述可编程DNA修饰蛋白是CRISPR/Cas核酸酶系统、CRISPR/Cas双切口酶系统、锌指核酸酶(ZFN)、转录激活因子样效应核酸酶(TALEN)、大范围核酸酶、包含与核酸酶结构域连接的可编程DNA结合结构域的融合蛋白,或包含与非核酸酶结构域连接的可编程DNA结合结构域的融合蛋白。
23.实施方案22的方法,其中所述融合蛋白的可编程DNA结合结构域是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白或转录激活因子样效应子。
24.实施方案22或23的方法,其中所述融合蛋白的非核酸酶修饰结构域具有乙酰转移酶活性、脱乙酰酶活性、甲基转移酶活性、去甲基酶活性、激酶活性、磷酸酶活性、泛素连接酶活性、去泛素化活性、腺苷酸化活性、去腺苷酸化活性、SUMO化活性、去SUMO化活性、核糖基化活性、去核糖基化活性、豆蔻酰化活性、去豆蔻酰化活性、瓜氨酸化活性、解旋酶活性、氨基化活性、脱氨基化活性、烷基化活性、脱烷基化活性、氧化活性、转录激活活性或转录阻遏活性。
25.实施方案24的方法,其中所述融合蛋白的非核酸酶结构域具有胞嘧啶脱氨酶活性、组蛋白乙酰转移酶活性、转录激活活性或转录阻遏活性。
26.实施方案16至25中任一项的方法,其中所述至少一种可编程DNA结合蛋白是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白、转录激活因子样效应子、CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。
27.实施方案16至26中任一项的方法,其中所述可编程DNA修饰蛋白是CRISPR/Cas核酸酶系统、CRISPR/Cas双切口酶系统、或与非核酸酶结构域连接的无催化活性的CRISPR/Cas系统、并且所述至少一种可编程DNA结合蛋白是无催化活性的CRISPR/Cas系统,其中每种CRISPR/Cas系统包含CRISPR/Cas蛋白和引导RNA。
28.实施方案27的方法,其中每种CRISPR/Cas系统的引导RNA至少部分是化学合成的。
29.实施方案27的方法,其中每种CRISPR/Cas系统的引导RNA是酶促合成的。
30.实施方案16至29中任一项的方法,其中所述真核细胞是体外的。
31.实施方案16至29中任一项的方法,其中所述真核细胞是体内的。
32.实施方案16至31中任一项的方法,其中所述真核细胞是哺乳动物细胞。
33.实施方案32的方法,其中所述哺乳动物细胞是人类细胞。
34.实施方案32的方法,其中所述哺乳动物细胞是非人类细胞。
35.一种检测真核细胞中染色体序列的方法,所述方法包括:
I. 向真核细胞中引入(a)包含至少一种可检测标记结构域的可编程DNA结合蛋白或编码包含至少一种可检测标记结构域的可编程DNA结合蛋白的核酸;和(b)至少一种可编程DNA结合蛋白或编码所述至少一种可编程DNA结合蛋白的核酸,其中包含至少一种可检测标记结构域的可编程DNA结合蛋白靶向靶染色体序列,并且所述至少一种可编程DNA结合蛋白中的每种都靶向于靶染色体序列附近的位点,并且所述至少一种可编程DNA结合蛋白与靶染色体序列附近的位点的结合提高了包含至少一种可检测标记结构域的可编程DNA结合蛋白对靶染色体序列的可及性;并且
II. 检测与靶染色体序列结合的包含至少一种可检测标记结构域的可编程DNA结合蛋白。
36.实施方案35的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约250个碱基对内。
37.实施方案36的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约100个碱基对内。
38.实施方案37的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约75个碱基对内。
39.实施方案38的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约50个碱基对内。
40.实施方案39的方法,其中所述靶染色体序列附近的位点位于所述靶染色体序列任一侧的约25个碱基对内。
41.实施方案35至40中任一项的方法,其中所述包含至少一种可检测标记结构域的可编程DNA结合蛋白的至少一种可检测标记结构域是荧光蛋白、荧光标签、表位标签、或可编程DNA结合蛋白内的天然存在的表位。
42.实施方案35至41中任一项的方法,其中所述包含至少一种可检测标记结构域的可编程DNA结合蛋白是与至少一种可检测标记结构域连接的无催化活性的CRISPR/Cas系统、与至少一种可检测标记结构域连接的无催化活性的大范围核酸酶、与至少一种可检测标记结构域连接的锌指蛋白、或与至少一种可检测标记结构域连接的转录激活因子样效应子。
43.实施方案35至42中任一项的方法,其中所述至少一种可编程DNA结合蛋白是无催化活性的CRISPR/Cas系统、无催化活性的大范围核酸酶、锌指蛋白、转录激活因子样效应子、CRISPR/Cas切口酶、ZFN切口酶、TALEN切口酶或大范围核酸酶切口酶。
44.实施方案35至43中任一项的方法,其中所述包含至少一种可检测标记结构域的可编程DNA结合蛋白是与至少一种可检测标记结构域连接的无催化活性的CRISPR/Cas系统,并且所述至少一种可编程DNA结合蛋白是无催化活性的CRISPR/Cas系统,其中每种CRISPR/Cas系统包含CRISPR/Cas蛋白和引导RNA。
45.实施方案44的方法,其中每种CRISPR/Cas系统的引导RNA至少部分是化学合成的。
46.实施方案44的方法,其中每种CRISPR/Cas系统的引导RNA是酶促合成的。
47.实施方案35至46中任一项的方法,其中所述真核细胞是哺乳动物细胞。
48.实施方案47的方法,其中所述哺乳动物细胞是人类细胞。
49.实施方案47的方法,其中所述哺乳动物细胞是非人类细胞。
50.实施方案35至49中任一项的方法,其中所述真核细胞是活的或固定的。
51.实施方案35至50中任一项的方法,其中所述检测包括动态活细胞成像、荧光显微术、共聚焦显微术、免疫荧光、免疫检测、RNA-蛋白结合或蛋白-蛋白结合。
实施例
以下实施例说明了本公开内容的某些方面。
实施例1.新凶手弗朗西斯菌(Francisella novicida) CRISPR-Cas9(FnCas9)基因编辑增强
FnCas9是IIB型CRISPR-Cas9。它比广泛使用的SpCas9具有更高的内在特异性,但已发现其在人类细胞中的稳健性低于SpCas9。为了确定可编程DNA结合蛋白与附近位点的结合是否能够使核酸酶切割人类细胞中原本难以接近的靶标(即POR基因座),用每百万细胞中5.6μg的FnCas9质粒DNA、5μg的催化死SpCas9(SpdCas9)质粒DNA,以及每种sgRNA的3μg质粒DNA转染K562细胞(参见图2)。转染后3天收获基因组DNA,并使用正向引物5’-CTCCCCTGCTTCTTGTCGTAT-3’(SEQ ID NO:9)和反向引物5’-ACAGGTCGTGGACACTCACA-3’(SEQID NO:10)通过PCR扩增靶区域。通过Cel-I核酸酶消化和聚丙烯酰胺凝胶分析确定靶标上FnCas9的靶向插入/缺失(indel)。
如图2所示,单独转染时FnCas9不能切割靶标。但是,当其与SpdCas9组合转染以帮助破坏局部染色质构型时,当SpdCas9用于结合一个附近位点时,FnCas9能够以稳健的水平,以10-11%的indel切割靶标。当SpdCas9用于结合两个附近位点时,FnCas9活性进一步增加至28%的indel。这些结果证明,本文公开的方法能够使核酸内切酶有效地裂解原本难以接近的靶标,并且在用于破坏局部染色质构型的两个位点之间存在协同效应。
实施例2. 空肠弯曲杆菌(Campylobacter jejuni) CRISPR-Cas9(CjCas9)基因编辑增强
CjCas9是IIC型CRISPR-Cas9。它是迄今为止表征的最小Cas9,具有独特的ACAYPAM要求。但已发现该核酸酶对人类细胞中的大多数靶标无活性。为了确定本文公开的方法是否能够使CjCas9蛋白结合人类细胞中难以接近的靶标,用每百万细胞中4.2μg的带Flag标签的催化死CjCas9(CjdCas9)质粒DNA、5μg的催化死SpCas9(SpdCas9)质粒DNA和每种sgRNA的3μg质粒DNA转染K562细胞(参见图3A)。转染后16小时将细胞在甲醛中固定,并使用抗flag抗体进行染色质免疫沉淀(ChIP)。通过微滴式数字PCR(ddPCR)测定Flag-CjdCas9的靶标结合。
如图3C所示,Flag-CjdCas9能够结合AAVS1基因座中先前已知的可及靶标,但当单独转染时不能结合POR基因座中的不可及靶标。然而,当其与SpdCas9组合转染以破坏局部染色质构型时,Flag-CjdCas9能够比其与AAVS1靶标的结合更有效地结合POR靶标。
为了检查对靶DNA切割的影响,用每百万细胞中4.2μg的CjCas9质粒DNA、5μg的SpdCas9质粒DNA和每种sgRNA的3μg质粒DNA转染K562细胞。转染后3天收获基因组DNA,并使用正向引物5’- CTCCCCTGCTTCTTGTCGTAT-3’ (SEQ ID NO:9)和反向引物5’-ACAGGTCGTGGACACTCACA-3’ (SEQ ID NO:10)通过PCR扩增靶区域。通过Cel-I核酸酶消化和聚丙烯酰胺凝胶分析测定POR靶上CjCas9的切割活性。如图4所示,无SpdCas9时CjCas9无法切割靶标。但是,当其与SpdCas9组合转染时,CjCas9能够以34.1-37.9%的indel有效切割靶标。这些结果证明,本文公开的方法能够使核酸酶有效地结合和切割原本不可及的靶标。
实施例3. 新凶手弗朗西斯菌(Francisella novicida) Cpf1(FnCpf1)基因编辑增强
FnCpf1是V型CRISPR-Cas系统。Cpf1系统与II型CRISPR-Cas9系统显著不同。与Cas9系统不同,Cpf1系统使用富含5'T的PAM和单个RNA引导进行靶向而无需tracrRNA(Zetsche et al., Cell, 2015, 163:1-13)。这些“较新的”的CRISPR系统有可能使基因编辑实践更简单,但已发现许多Cpf1系统在人类细胞中无活性。为了确定本文公开的方法是否能够使不同的“无活性”Cpf1核酸酶切割人细胞中的内源性靶标,用每百万细胞中5μg的FnCpf1质粒DNA、5μg的SpdCas9质粒DNA和每种sgRNA的3μg质粒DNA转染K562细胞(参见图5)。转染后3天收获基因组DNA,并使用正向引物5’- CTCCCCTGCTTCTTGTCGTAT-3’ (SEQ IDNO:9)和反向引物5’-ACAGGTCGTGGACACTCACA-3’ (SEQ ID NO:10)通过PCR扩增靶区域。通过Cel-I核酸酶消化和聚丙烯酰胺凝胶分析测定POR靶上FnCpf1的切割活性。
如图5所示,FnCpf1在单独转染时不能切割靶标,但在与SpdCas9组合转染时能够有效切割靶标。这些结果表明本文公开的方法适用于不同的V型CRISPR-Cas系统。
实施例4. 在人类HBB和HBD中相同靶标之间的选择性编辑
使用人类的两个相同靶标(即HBB和HBD)来确定本文公开的方法是否可以促进不同基因中相同位点之间的选择性编辑。用每百万细胞中4.2μg的CjCas9质粒DNA、5μg的SpdCas9质粒DNA和每种sgRNA的3μg质粒DNA转染K562细胞(参见图6)。转染后3天收获基因组DNA,通过PCR扩增两个靶区域,HBB用正向引物5’-CGGCTGTCATCACTTAGACCTCA-3’ (SEQID NO:11)和反向引物5’-GCAGCCTAAGGGTGGGAAAATAGA-3’ (SEQ ID NO:12),HBD用正向引物5’-AGGGCAAGTTAAGGGAATAGTGGAA-3’ (SEQ ID NO:13)和反向引物5’-CCAAGGGTAGACCACCAGTAATCTG-3’ (SEQ ID NO:14)。通过Cel-I核酸酶消化和聚丙烯酰胺凝胶分析测定HBB和HBD靶上CjCas9的切割活性。
如图6所示,当单独转染时,CjCas9不能切割任一靶标。但是,当它与靶向HBB邻近位点的SpdCas9组合转染时,CjCas9有效切割HBB靶标,但仍不能切割相同的HBD靶标。前两个泳道中的两个Cel-I核酸酶消化带是由K562细胞群中存在的SNP引起的。这些结果证明了所公开方法提高基因编辑选择性的独特能力。
实施例5. 酿脓链球菌(
SpCas9是IIA型CRISPR-Cas9,并且由于其在真核细胞中的稳健活性已被广泛用于基因组修饰。但是,其活性也可能因靶标而异。为了确定本文公开的方法是否也能增强该核酸酶,用每百万细胞中5μg的SpCas9质粒DNA、5.6μg的催化死FnCas9(FndCas9)和每种sgRNA的3μg质粒DNA转染K562细胞(参见图7)。转染后3天收获基因组DNA,并使用正向引物5’-CTCCCCTGCTTCTTGTCGTAT-3’ (SEQ ID NO:9)和反向引物5’- ACAGGTCGTGGACACTCACA-3’(SEQ ID NO:10)通过PCR扩增靶区域。通过Cel-I核酸酶消化和聚丙烯酰胺凝胶分析测定POR靶上SpCas9的切割活性。
如图7所示,与单独转染时相比,当与FndCas9组合转染时,SpCas9的切割活性显著提高。这些结果表明本文公开的方法也可以应用于稳健的核酸内切酶。
实施例6. 使用ssDNA寡聚体供体增强基因编辑
用每百万细胞中4.2μg的CjCas9质粒DNA、5μg的SpdCas9质粒DNA、每种sgRNA的3μg质粒DNA和300pmol 88-nt ssDNA寡聚体供体转染K562细胞,用于靶向整合EcoRI限制性位点。转染后3天收获基因组DNA,并使用正向引物5’- CTCCCCTGCTTCTTGTCGTAT-3’ (SEQ IDNO:9)和反向引物5’-ACAGGTCGTGGACACTCACA-3’ (SEQ ID NO:10)通过PCR扩增靶区域。通过用EcoRI限制酶消化和聚丙烯酰胺凝胶分析测定EcoRI限制性位点的靶向整合。如图8所示,当与CjCas9和SpdCas9一起转染ssDNA寡聚体供体时,限制性位点在POR基因座中有效整合(28-37%),而当寡核苷酸供体单独转染或在没有SpdCas9而与CjCas9组合转染时未检测到整合。这些结果表明,本文公开的方法可以促进在原本不可及的靶标上使用ssDNA寡聚体供体进行有效的基因编辑。
实施例7. 在活细胞和固定细胞中增强序列特异性基因组DNA的检测
Cas9蛋白与荧光蛋白的融合使得能够检测活细胞中的染色体动力学(Chen etal., Cell, 2013, 155:1479-91)。因此认为染色质结构动力学将影响CRISPR/Cas系统复合物接近各种基因组基因座的能力。因此,认为邻近具有dCas9-GFP的那些的CRISPR(dCas9)复合体的放置增强了染色体动力学的检测,其程度类似于实施例2中对于染色质免疫沉淀观察到的程度。例如,CjdCas9可以与GFP融合并靶向具有染色质状态的区域,其阻止可检测的CjdCas9-GFP结合。然后可以邻近CjdCas9-GFP靶标设计基于SpdCas9的系统以产生可检测的信号。对于抵抗SpdCas9-GFP结合和检测的染色质区域,邻近FndCas9分子可用于增强检测,其程度与实施例5中对于SpCas9和FndCas9邻近靶向和双链断裂活性增强所示的相似。此外,鉴于先前的研究已经表明与对于双链切割相比,CRISPR引导RNA和基因组DNA之间的杂交要求的程度对于结合可能较低(Wu
基于CRISPR的类似检测方法已经应用于固定细胞(Deng et al., Proc. Natl.Acad, Sci. USA, 2015, 112(38):11870-75)。因此,认为邻近CRISPR靶向将以类似于上述对于活细胞的方式增强固定DNA的检测。由于固定细胞中的基因组DNA链是化学交联的,因此通过核酸探针的杂交来审查序列信息通常需要使用加热或化学处理的预处理步骤来充分分离链。因此,邻近CRISPR靶向可能使固定DNA更可及并且降低固定细胞的热或化学处理的程度(或要求)。消除热或化学处理将在诊断方案简化和细胞内分子结构维持方面提供优势,其更好地反映活细胞生物学并因此更了解诊断结果。
实施例8. 在真核细胞中增强基于CRISPR的基因激活和阻遏
Cas9蛋白与转录调节结构域的融合已经实现靶向基因激活和阻遏(Konermann etal., Nature, 2014; 517(7536):583-8; Gilbert et al., Cell, 2014, 159(3):547-661)。认为染色质结构动力学将影响CRISPR复合物接近各种基因组基因座并诱导激活或阻遏的能力。因此,认为邻近具有与转录调节结构域融合的dCas9的那些的CRISPR(dCas9)复合体的放置增强了靶向基因调节,其程度类似于实施例2中对于染色质免疫沉淀观察到的程度。对于抵抗通过SpdCas9-转录-调节子的结合和修饰的染色质区域,邻近FndCas9分子可用于增强基因激活或阻遏,其程度与实施例5中对于SpCas9和FndCas9邻近靶向和双链断裂活性增强所示的相似。
实施例9. 在真核细胞中增强基于CRISPR的表观遗传修饰
Cas9蛋白与表观遗传修饰结构域的融合已经实现了靶向表观遗传染色体修饰,例如通过p300的组蛋白乙酰化或通过胞嘧啶脱氨酶的胞嘧啶脱氨作用(Hilton et al.,Nat. Biotechnol; 2015, 33(5):510-7; Komor et al., Nature, 2016, 533(7603):420-4)。认为染色质结构动力学将影响CRISPR复合物接近各种基因组基因座的能力。因此,邻近具有与表观遗传修饰物融合的dCas9的CRISPR(dCas9)复合物的那些的放置应该增强染色体DNA、局部蛋白或局部RNA的靶向表观遗传修饰,其程度与实施例2中对于染色质免疫沉淀观察到的程度相似。对于抵抗通过SpdCas9-epi-修饰剂结合和修饰的染色质区域,邻近FndCas9分子可用于增强检测,其程度与实施例5中对于SpCas9和FndCas9邻近靶向和双链断裂活性增强所示的相似。
