制备环形RNA的构建体、方法及其用途
文献发布时间:2024-04-18 20:01:30
本申请要求2021年5月28日提交的中国申请号202110594352.4的权益,将其通过引用以其整体特此并入。
对电子提交的序列表的引用
本申请通过引用并入了随同本申请提交的作为2022年5月27日创建并且大小为101,197字节的标题为“S2901CCD33CN_Sequence_Listing.txt”的文本文件的序列表。
1.技术领域
本发明涉及分子生物学领域,具体来说涉及用于制备环形RNA的构建体、方法及其应用。所述环形RNA可用于在真核细胞中表达目标蛋白或以非编码RNA的形式行使相应的功能。
2.背景技术
环形RNA(circRNA)是一类通过头尾相连形成的环形RNA分子。近年来有文献报道环形RNA可以调控基因转录、中和miRNA活性以及RNA结合蛋白的结合,也可以作为模板翻译生成蛋白质(Yang,Y.等人,“Extensive translation of circular RNAs driven by N(6)-methyladenosine,”Cell Research,27(5):626-641(2017);Abe,N.等人,“RollingCircle Translation of Circular RNA in Living Human Cells”,Scientific Reports,5:16435(2015);Gao,X.等人,“Circular RNA-encoded oncogenic E-cadherin variantpromotes glioblastoma tumorigenicity through activation of EGFR-STAT3signalling,”Nature Cell Biology,23(3):278-291(2021);Pamudurti,NR.等人,“Translation of CircRNAs,”Molecular Cell,66(1):9-21(2017))。与线性RNA相比,环形RNA因其头尾共价闭环结构不易被RNA降解系统识别,因而具有更强的稳定性,有成为新一代RNA药物平台的潜力和前景。
目前体外制备环形RNA的方法主要有三种。一种方法是通过核酸连接酶催化的RNA连接反应,将线性RNA的5’端和3’端首尾相连,得到环形RNA。其中的RNA连接酶是外源蛋白质,如T4 RNA连接酶。一种方法是化学连接法,通过氰化溴及吗啉基衍生物的催化将RNA的5’端和3’端连接。另一种更为领先的方法是通过核酶催化的RNA剪接反应得到首尾相连的环形RNA。这种方法通过设计带有自剪接功能的含有核酶序列的表达框架来表达环形RNA。
目前可进行RNA自剪接的核酶通常被分为两大类,分别称为I型和II型内含子。据文献报道这两类内含子均可以在适当的反应条件下进行自剪接,将两个RNA片段连接到一起。虽然两类核酶剪接产物相似,但核酶本身的结构和剪接机制大相径庭。
I型内含子为9螺旋结构,催化剪接时需要外在的磷酸鸟苷中的羟基(pG-OH)触发反应,并且对位于I型内含子两端的外显子序列有较大的依赖性。
II型内含子依靠核酸序列内部自身的羟基触发剪接。这种剪接机制更接近通过剪接体介导的剪接反应,即可以更好地模拟高等生物剪接。
上述结构差异决定了I型内含子自剪接需要较长的原始外显子序列,也被称为疤痕序列(scar sequence)。
已有研究表明分别采用这两类内含子核酶均可以在体外制备环形RNA,但效率较低(Puttaraju,M.和Been,MD.,“Group I permuted intron-exon(PIE)sequences self-splice to produce circular exons,”Nucleic Acids Research,20(20):5357-64(1992);Mikheeva,S.等人,“Use of an engineered ribozyme to produce a circularhuman exon,”Nucleic Acids Research,25(24):5085-94(1997))。
Wesselhoeft等人的文章报道了通过优化包含I型内含子的构建体提高RNA环化效率的方法(Wesselhoeft,RA.等人,“Engineering circular RNA for potent and stabletranslation in eukaryotic cells,”Nature Communications,9(1):2629(2018)),相关专利申请(WO 2019/236673 A1)公开了用于形成环形编码RNA的含有I型内含子的构建体。Wesselhoeft等人对I型内含子和其两端的外显子进行重排,并将带有核糖体进入位点(IRES)的目标蛋白(POI)构建到此框架中,然后在GTP存在下,通过自剪接反应得到可以翻译目标蛋白的环形编码RNA。通过选择不同的I型内含子,并进行设计改造,提高了RNA环化效率。具体来说,该技术首先将T4噬菌体的Td基因进行一些删除,保留了可以进行正确折叠从而保持核酶活性的序列,包括内含子和一部分外显子,然后将其一分为二,将3’端内含子和外显子片段2(E2)构建到IRES-POI的5’端,将外显子片段1(E1)和5’端内含子构建到IRES-POI的3’端,在GTP和镁离子的存在下,自剪接得到环形RNA。然而,Wesselhoeft等人发现5’端和3’端剪接位点因为目标基因的插入而无法有效地进行剪接。为了解决这个问题,Wesselhoeft等人在剪接位点附近插入了互补配对的“同源臂”,从而提高了剪接效率。还根据已有文献(Mikheeva,S.等人,(1997),同上)选择了另一种I型内含子Anabaena,发现其剪接效率高于Td内含子,并对其进行类似的设计改造,进一步提高了剪接效率。文章最后验证了该表达框架可以有效翻译目标蛋白。
但是,Wesselhoeft等人的设计存在以下缺点:
1.使用I型内含子时必须含有较长的原始外显子序列,因此在表达产物中会包含一段原始外源序列(疤痕序列),在将目标序列制备成环形RNA的过程中通常希望将这段不属于目标序列的序列去除,以便于后续应用;
2.I型内含子在进行自剪接时需要GTP参与供能。
另一方面,以往文献中的II型内含子的剪接效率较低(约10%)(Mikheeva,S.等人,(1997),同上)。因此,本领域仍需要改进的构建体和方法来制备环形RNA。
3.发明内容
本申请的发明人通过筛选和设计优化,创造了一种通过II型内含子的自剪接制备环形RNA的方法学,克服了上述问题。
因此,本发明提供了一种在体外具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)目标序列;
(d)外显子片段1(E1);
(e)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
本发明还提供了一种在体外具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)接头序列;
(d)目标序列;
(e)接头序列;
(f)外显子片段1(E1);
(g)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
本发明还提供了一种在体外具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)目标序列;
(e)外显子片段1(E1);
(f)5’内含子片段;
(g)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
本发明还提供了一种在体外具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)接头序列;
(e)目标序列;
(f)接头序列;
(g)外显子片段1(E1);
(h)5’内含子片段;
(i)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为分割成两个片段的II型内含子的片段,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
在一些实施方案中,所述多核苷酸构建体为RNA多核苷酸构建体。
在一些实施方案中,所述多核苷酸构建体能够在体外形成目标序列的环形RNA。
在一些实施方案中,所述多核苷酸构建体能够在体内形成目标序列的环形RNA。
本发明提供了通过本发明的多核苷酸构建体产生的环形RNA。在一些实施方案中,所述环形RNA的长度为至少500个核苷酸、长度为至少1,000个核苷酸、长度为至少1,500个核苷酸。
本发明提供了一种使用本发明的多核苷酸构建体制造环形RNA的方法。
本发明提供了一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)目标序列;
(d)外显子片段1(E1);
(e)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
本发明还提供了一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)接头序列;
(d)目标序列;
(e)接头序列;
(f)外显子片段1(E1);
(g)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
本发明还提供了一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)目标序列;
(e)外显子片段1(E1);
(f)5’内含子片段;
(g)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
本发明还提供了一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)接头序列;
(e)目标序列;
(f)接头序列;
(g)外显子片段1(E1);
(h)5’内含子片段;
(i)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为分割成两个片段的II型内含子的片段,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
本发明提供了一种在细胞中表达蛋白质的方法,包括将本发明的环形RNA转染到所述细胞中。
本发明提供了一种在细胞中表达蛋白质的方法,包括(a)将本发明的环形RNA转染到所述细胞中,或者(b)使本发明的多核苷酸构建体发生自剪接环化反应形成环形RNA,并将所述环形RNA转染到所述细胞中;其中,优选所述细胞为真核细胞。
本发明提供了一种使用II型内含子生成具有自剪接活性的序列的方法,所述方法包括以下步骤:
(a)定义所述II型内含子的序列;任选地使用剪接测定(线性剪接)检查所述II型内含子的体外自剪接活性;
(b)将所述II型内含子劈分成两个片段,
(c)将所述两个内含子片段的顺序颠倒,
(d)使用剪接测定确认RNA的体外环化。
本发明的构建体、方法和应用至少具有以下优点:
1.产生不含疤痕序列的环形RNA,这种环形RNA更有利于有序应用;
2.多核苷酸的自剪接反应形成环形RNA不需要GTP(例如,在一些实施方案中,仅需要Mg离子、Na离子);和/或
3.大幅提高了II型内含子的剪接效率,可从10%提高至50%左右,甚至实现最高高达98%的剪接效率。
在具体的实施方案中,所述E1和/或E2的长度为0-20个核苷酸,优选0-10个核苷酸,如0、1、2、3、4、5、6、7、8、9、10个核苷酸。
在具体的实施方案中,所述5’内含子片段和3’内含子片段将II型内含子从非配对区进行分割成两个片段。在具体的实施方案中,所述非配对区选自II型内含子两个相邻结构域之间的线性区或结构域4茎环结构的环区。
在具体的实施方案中,所述II型内含子相对于其野生型形式包含一个或多个核苷酸的修饰,所述修饰选自删除、取代、添加中的一种或多种。
在具体的实施方案中,所述5’内含子片段和3’内含子片段中分别包含一对或多对彼此互补的配对序列。在优选的实施方案中,所述互补配对序列的长度大于20个核苷酸。
在具体的实施方案中,所述5’内含子片段和/或3’内含子片段中包含一个或者多个亲和标签序列,所述亲和标签序列选自下组中的一种或多种:探针结合序列、MS2结合位点、PP7结合位点、链霉亲和素结合位点。
在具体的实施方案中,其中所述E1和E2为0,并且所述修饰包括对所述II型内含子的一个或多个EBS序列进行修饰,使所述EBS序列分别与目标序列中相应长度的一个或多个区域在至少60%的核苷酸位置上互补配对。所述EBS序列选自EBS1、EBS2、EBS3中的一个或多个,优选任意两个,更优选EBS1和EBS3。在优选的实施方案中,所述修饰为对所述II型内含子的两个EBS序列,优选EBS1和EBS3,进行修饰,使所述EBS序列分别与目标序列中相应长度的两个区域在至少60%的核苷酸位置上互补配对。在优选的实施方案中,所述修饰为对所述II型内含子的两个EBS序列,优选EBS1’和EBS3’,进行修饰,使所述EBS序列分别与目标序列中相应长度的两个区域在至少60%的核苷酸位置上互补配对。在优选的实施方案中,所述修饰为对所述II型内含子的两个EBS序列,优选EBS1”和EBS3”,进行修饰,使所述EBS序列分别与目标序列中相应长度的两个区域在至少60%的核苷酸位置上互补配对。在另一个优选的实施方案中,所述修饰为对所述II型内含子的δ或δ"序列进行修饰,其中所述δ或δ"序列分别与目标序列中相应长度的区域在至少60%的核苷酸位置上互补配对;优选所述区域位于所述目标序列的一端。
在优选的实施方案中,所述目标序列中相应长度的两个区域分别位于所述目标序列的两端。
在具体的实施方案中,其中所述修饰是删除结构域4的部分或全部,例如删除结构域4中的IEP序列,优选删除全部结构域4。
在具体的实施方案中,所述II型内含子为源自微生物的II型内含子。优选地,所述II型内含子具有体外自剪接活性。在具体的实施方案中,所述II型内含子是来自梭菌属(Clostridium)如破伤风梭菌(Clostridium tetani)或芽孢杆菌属(Bacillus)如苏云金芽孢杆菌(Bacillus thuringiensis)的II型内含子。在具体的实施方案中,所述II型内含子为SEQ ID NO:1或2的核苷酸序列中包含的II型内含子。
在具体的实施方案中,所述蛋白非编码序列选自下组中的一种或多种:间隔序列如SEQ ID NO:4-6中的任一个、富含A和/或T的序列、polyA序列、polyA-C序列、polyC序列、polyU序列、IRES、核糖体结合位点、适配体序列、RNA支架、核糖开关、除自剪接核酶之外的核酶、小RNA、翻译调控序列、蛋白结合位点。
在具体的实施方案中,所述多核苷酸构建体能够在体外形成目标序列的环形RNA。
在具体的实施方案中,所述多核苷酸构建体能够在体内形成目标序列的环形RNA。
在第二方面,本发明提供了通过第一方面的构建体产生的环形RNA。优选所述环形RNA不含任何不属于所述目标序列的其他序列,如不含E2、E1序列。
在具体的实施方案中,如目标序列为蛋白编码序列的技术方案中,所述环形RNA的长度为至少500个核苷酸,优选至少1,000个核苷酸,优选至少1,500个核苷酸。在目标序列为非编码RNA的技术方案中,目标序列可以更短。
在第三方面,本发明提供了一种在细胞中表达蛋白质的方法,包括将第二方面的环形RNA转染到所述细胞中。
在第四方面,本发明提供了一种在细胞中表达蛋白质的方法,包括使第一方面的构建体发生自剪接环化反应形成环形RNA,并将所述环形RNA转染到所述细胞中。
在第三方面和第四方面的具体实施方案中,所述细胞为真核细胞。
本发明的构建体、方法和应用至少具有以下优点:
1.在优选的技术方案中,可以产生不含疤痕序列的环形RNA,这种环形RNA更有利于有序应用;
2.在形成环形RNA的自剪接反应过程中不需要GTP参与,仅需要提供Mg离子、Na离子即可;
3.大幅提高了II型内含子的剪接效率,可从10%提高至50%左右,甚至实现最高高达98%的剪接效率。
4.说明性实施方案
第1组
1.一种在体外具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(1)3’内含子片段;
(2)外显子片段2(E2);
(3)目标序列;
(4)外显子片段1(E1);
(5)5’内含子片段,
其中所述5’内含子片段和3’内含子片段通过将II型内含子分割成两个片段获得,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列和/或非编码序列。
2.条款1的多核苷酸构建体,其中所述E1和/或E2的长度为0-20个核苷酸,优选0-10个核苷酸,如0、1、2、3、4、5、6、7、8、9、10个核苷酸。
3.条款1的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从非配对区进行分割成两个片段获得,所述非配对区优选选自II型内含子两个相邻结构域之间的线性区或结构域4茎环结构的环区。
4.条款1的多核苷酸构建体,其中所述II型内含子相对于其野生型形式包含一个或多个核苷酸的修饰,所述修饰选自删除、取代、添加中的一种或多种。
5.条款4的多核苷酸构建体,其中所述E1和E2为0,并且所述修饰包括对所述II型内含子的一个或多个EBS序列进行修饰,使所述EBS序列分别与目标序列中相应长度的一个或多个区域在至少60%的核苷酸位置上互补配对。
6.条款5的多核苷酸构建体,其中所述修饰为对所述II型内含子的两个EBS序列,如EBS1和EBS3,进行修饰,使所述EBS序列分别与目标序列中相应长度的两个区域在至少60%的核苷酸位置上互补配对;优选所述两个区域分别位于所述目标序列的两端。
7.条款4的多核苷酸构建体,其中所述修饰包括删除结构域4的部分或全部,例如删除结构域4中的IEP序列,优选删除全部结构域4。
8.条款1的多核苷酸构建体,所述II型内含子为源自微生物的II型内含子。
9.条款1的多核苷酸构建体,其中所述非编码序列选自下组的序列:间隔序列SEQID NO:4-6中的任一个、polyA序列、polyA-C序列、polyC序列、polyU序列、IRES、核糖体结合位点、适配体序列、RNA支架、核糖开关、除自剪接核酶之外的核酶、小RNA结合位点、翻译调控序列、蛋白结合位点。
10.通过条款1-9中任一项的多核苷酸构建体产生的环形RNA。
11.条款10的环形RNA,其不含任何不属于所述目标序列的其他序列,如不含E2、E1序列的全部或部分。
12.一种在细胞中表达蛋白质的方法,包括(a)将条款10或11的环形RNA转染到所述细胞中,或者(b)使条款1-9中任一项的构建体发生自剪接环化反应形成环形RNA,并将所述环形RNA转染到所述细胞中;
其中优选所述细胞为真核细胞。
第2组
实施方案1
一种具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)目标序列;
(d)外显子片段1(E1);
(e)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案2
一种具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)接头序列;
(d)目标序列;
(e)接头序列;
(f)外显子片段1(E1);
(g)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案3
一种具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)目标序列;
(e)外显子片段1(E1);
(f)5’内含子片段;
(g)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案4
一种具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)接头序列;
(e)目标序列;
(f)接头序列;
(g)外显子片段1(E1);
(h)5’内含子片段;
(i)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为分割成两个片段的II型内含子的片段,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案5
实施方案1-4中任一项的多核苷酸构建体,其中所述多核苷酸构建体在体外具有自剪接活性。
实施方案6
实施方案1-5中任一项的多核苷酸构建体,其中所述E1和/或E2的长度为0-20个核苷酸,优选长度为0-10个核苷酸,如长度为0、1、2、3、4、5、6、7、8、9、10个核苷酸。
实施方案7
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从非配对区,例如为II型内含子两个相邻结构域之间的线性区的非配对区,进行分割成两个片段获得。
实施方案8
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域1茎环结构的环区进行分割获得。
实施方案9
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域2茎环结构的环区进行分割获得。
实施方案10
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域3茎环结构的环区进行分割获得。
实施方案11
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域4茎环结构的环区进行分割获得。
实施方案12
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域5茎环结构的环区进行分割获得。
实施方案13
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域6茎环结构的环区进行分割获得。
实施方案14
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域1与结构域2之间的线性区进行分割获得。
实施方案15
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域2与结构域3之间的线性区进行分割获得。
实施方案16
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域3与结构域4之间的线性区进行分割获得。
实施方案17
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域4与结构域5之间的线性区进行分割获得。
实施方案18
实施方案1-6中任一项的多核苷酸构建体,其中所述5’内含子片段和3’内含子片段通过将II型内含子从结构域5与结构域6之间的线性区进行分割获得。
实施方案19
实施方案1-18中任一项的多核苷酸构建体,其中所述II型内含子相对于其野生型形式包含一个或多个核苷酸的修饰,所述修饰选自删除、取代、添加中的一种或多种。
实施方案20
实施方案19的多核苷酸构建体,其中所述修饰包括对所述II型内含子的一个或多个EBS序列进行修饰,其中所述EBS序列分别与目标序列中相应长度的一个或多个区域在至少60%的核苷酸位置上互补配对。
实施方案21
实施方案19的多核苷酸构建体,其中所述修饰为对所述II型内含子的两个EBS序列,如EBS1和EBS3,进行修饰,其中所述EBS序列分别与目标序列中相应长度的两个区域在至少60%的核苷酸位置上互补配对;优选所述两个区域分别位于所述目标序列的两端。
实施方案19的多核苷酸构建体,其中所述修饰为对所述II型内含子的EBS1和/或δ序列进行修饰或对EBS1'和/或δ”序列进行修饰,其中所述EBS1和/或δ序列与目标序列中相应长度的区域在至少60%的核苷酸上互补配对,任选地所述修饰为对EBS1和/或δ序列及其上游序列进行修饰,其中所述EBS1和/或δ序列及其上游序列与目标序列中相应长度的区域在至少60%的核苷酸上互补配对。在一些实施方案中,所述目标序列中相应长度的区域为IBS3、IBS3'、具有下游序列的IBS3、具有下游序列的IBS3'。在一些实施方案中,所述δ序列及其上游包括选自下组的核酸序列:(a)其中所述修饰为对所述II型内含子的δ或δ"序列进行修饰,其中所述δ或δ"序列分别与目标序列中相应长度的区域在至少60%的核苷酸位置上互补配对;优选所述区域位于所述目标序列的一端。在一些实施方案中,所述δ序列及其上游包含SEQ ID NO:127、(b)SEQ ID NO:128、(c)SEQ ID NO:129、(d)SEQ ID NO:130。在一些实施方案中,所述IBS3及其下游包含选自下组的核酸序列:(a)SEQ ID NO:131、(b)SEQID NO:132、(c)SEQ ID NO:133、(d)SEQ ID NO:134。
实施方案22
实施方案19的多核苷酸构建体,其中所述修饰包括删除结构域4的部分或全部,例如删除结构域4中的内含子编码蛋白(IEP)序列,优选删除全部结构域4。
实施方案23
实施方案19的多核苷酸构建体,其中所述修饰包括删除开放阅读框(ORF)。
实施方案24
实施方案1-23中任一项的多核苷酸构建体,其中所述多核苷酸构建体能够形成所述目标序列的近无痕环形RNA。
实施方案25
实施方案24的多核苷酸构建体,其中所述近无痕环形RNA具有长度等于或小于1个核苷酸、2个核苷酸、3个核苷酸、4个核苷酸、5个核苷酸、6个核苷酸、7个核苷酸、8个核苷酸、9个核苷酸、10个核苷酸、11个核苷酸、12个核苷酸、13个核苷酸、14个核苷酸、15个核苷酸、16个核苷酸、17个核苷酸、18个核苷酸、19个核苷酸、20个核苷酸的疤痕区。
实施方案26
实施方案1-23中任一项的多核苷酸构建体,其中所述多核苷酸构建体能够形成所述目标序列的无痕环形RNA。
实施方案27
实施方案1-26中任一项的多核苷酸构建体,其中E1和E2各自的长度为0个核苷酸。
实施方案28
实施方案1-26中任一项的多核苷酸构建体,其中所述E1的长度为0个核苷酸。
实施方案29
实施方案1-26中任一项的多核苷酸构建体,其中所述E2的长度为0个核苷酸。
实施方案30
实施方案1-29中任一项的多核苷酸构建体,其中所述II型内含子是源自微生物(如破伤风梭菌或芽孢杆菌属如苏云金芽孢杆菌)的II型内含子。
实施方案31
实施方案1-30中任一项的多核苷酸构建体,其中所述非编码序列选自下组:间隔序列SEQ ID NO:4-6、polyA序列、polyA-C序列、polyC序列、polyU序列、IRES、核糖体结合位点、适配体序列、RNA支架、核糖开关、除自剪接核酶之外的核酶、反义寡核苷酸(ASO)、支架、小RNA结合位点、翻译调控序列、蛋白结合位点。
实施方案32
实施方案1-31中任一项的多核苷酸构建体,其中所述II型内含子包含选自下组的核酸序列:
(a)SEQ ID NO:33;
(b)SEQ ID NO:34;
(c)SEQ ID NO:35;
(d)SEQ ID NO:36;
(e)SEQ ID NO:37;
(f)SEQ ID NO:38;
(g)SEQ ID NO:39;
(h)SEQ ID NO:40;
(i)SEQ ID NO:41。
实施方案32-1
实施方案32的多核苷酸构建体,其中所述II型内含子基本上由选自下组的核酸序列组成:SEQ ID NO:33-SEQ ID NO:41。
实施方案32-2
实施方案32的多核苷酸构建体,其中所述II型内含子由选自下组的核酸序列组成:SEQ ID NO:33-SEQ ID NO:41。
实施方案33
实施方案1-32中任一项的多核苷酸构建体,其中所述多核苷酸构建体为RNA多核苷酸构建体。
实施方案34
实施方案33的多核苷酸构建体,其中所述3’内含子片段包含选自下组的核酸序列:
(a)与SEQ ID NO:42具有95%的同一性的核酸序列;
(b)与SEQ ID NO:42具有98%的同一性的核酸序列;
(c)与SEQ ID NO:42具有99%的同一性的核酸序列;
(d)SEQ ID NO:42;
(e)与SEQ ID NO:43具有95%的同一性的核酸序列;
(f)与SEQ ID NO:43具有98%的同一性的核酸序列;
(g)与SEQ ID NO:43具有99%的同一性的核酸序列;
(h)SEQ ID NO:43;
(i)与SEQ ID NO:44具有95%的同一性的核酸序列;
(j)与SEQ ID NO:44具有98%的同一性的核酸序列;
(k)与SEQ ID NO:44具有99%的同一性的核酸序列;
(l)SEQ ID NO:44;
(m)与SEQ ID NO:45具有95%的同一性的核酸序列;
(n)与SEQ ID NO:45具有98%的同一性的核酸序列;
(o)与SEQ ID NO:45具有99%的同一性的核酸序列;
(p)SEQ ID NO:45;
(q)与SEQ ID NO:46具有95%的同一性的核酸序列;
(r)与SEQ ID NO:46具有98%的同一性的核酸序列;
(s)与SEQ ID NO:46具有99%的同一性的核酸序列;
(t)SEQ ID NO:46;
(u)与SEQ ID NO:47具有95%的同一性的核酸序列;
(v)与SEQ ID NO:47具有98%的同一性的核酸序列;
(w)与SEQ ID NO:47具有99%的同一性的核酸序列;
(x)SEQ ID NO:47;
(y)与SEQ ID NO:48具有95%的同一性的核酸序列;
(z)与SEQ ID NO:48具有98%的同一性的核酸序列;
(aa)与SEQ ID NO:48具有99%的同一性的核酸序列;
(bb)核酸序列SEQ ID NO:48;
(cc)与SEQ ID NO:49具有95%的同一性的核酸序列;
(dd)与SEQ ID NO:49具有98%的同一性的核酸序列;
(ee)与SEQ ID NO:49具有99%的同一性的核酸序列;
(ff)SEQ ID NO:49;
(gg)与SEQ ID NO:50具有95%的同一性的核酸序列;
(hh)与SEQ ID NO:50具有98%的同一性的核酸序列;
(ii)与SEQ ID NO:50具有99%的同一性的核酸序列;
(jj)SEQ ID NO:50;
(kk)与SEQ ID NO:51具有95%的同一性的核酸序列;
(ll)与SEQ ID NO:51具有98%的同一性的核酸序列;
(mm)与SEQ ID NO:51具有99%的同一性的核酸序列;
(nn)SEQ ID NO:51;
(oo)与SEQ ID NO:52具有95%的同一性的核酸序列;
(pp)与SEQ ID NO:52具有98%的同一性的核酸序列;
(qq)与SEQ ID NO:52具有99%的同一性的核酸序列;
(rr)SEQ ID NO:52。
实施方案34-1
实施方案34的多核苷酸构建体,其中所述3’内含子片段基本上由选自下组的核酸序列组成:与SEQ ID NO:42-SEQ ID NO:52中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:42-SEQ ID NO:52中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:42-SEQ ID NO:52中的任一个具有99%的同一性的核酸序列、SEQ ID NO:42-SEQ ID NO:52中的任一个。
实施方案34-2
实施方案34的多核苷酸构建体,其中所述3’内含子片段由选自下组的核酸序列组成:与SEQ ID NO:42-SEQ ID NO:52中的任一个具有95%的同一性的核酸序列、与SEQ IDNO:42-SEQ ID NO:52中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:42-SEQ IDNO:52中的任一个具有99%的同一性的核酸序列、SEQ ID NO:42-SEQ ID NO:52中的任一个。
实施方案35
实施方案33或34的多核苷酸构建体,其中所述E2包含选自下组的核酸序列:
(a)SEQ ID NO:53;
(b)SEQ ID NO:54;
(c)SEQ ID NO:55;
(d)SEQ ID NO:56;
(e)SEQ ID NO:57;
(f)SEQ ID NO:58;
(g)SEQ ID NO:59;
(h)SEQ ID NO:60;
(i)SEQ ID NO:61;
(j)SEQ ID NO:62;
(k)SEQ ID NO:63。
实施方案35-1
实施方案35的多核苷酸构建体,其中所述E2基本上由选自下组的核酸序列组成:SEQ ID NO:53-SEQ ID NO:63。
实施方案35-2
实施方案35的多核苷酸构建体,其中所述E2由选自下组的核酸序列组成:SEQ IDNO:53-SEQ ID NO:63。
实施方案36
实施方案33-35中任一项的多核苷酸构建体,其中所述E1包含选自下组的核酸序列:
(a)SEQ ID NO:64;
(b)SEQ ID NO:65;
(c)SEQ ID NO:66;
(d)SEQ ID NO:67;
(e)SEQ ID NO:68;
(f)SEQ ID NO:69;
(g)SEQ ID NO:70;
(h)SEQ ID NO:71;
(i)SEQ ID NO:72;
(j)SEQ ID NO:73;
(k)SEQ ID NO:74。
实施方案36-1
实施方案36的多核苷酸构建体,其中所述E1基本上由选自下组的核酸序列组成:SEQ ID NO:64-SEQ ID NO:74。
实施方案36-2
实施方案36的多核苷酸构建体,其中所述E1由选自下组的核酸序列组成:SEQ IDNO:64-SEQ ID NO:74。
实施方案37
实施方案33-36中任一项的多核苷酸构建体,其中所述5’内含子片段包含选自下组的核酸序列:
(a)与SEQ ID NO:75具有95%的同一性的核酸序列;
(b)与SEQ ID NO:75具有98%的同一性的核酸序列;
(c)与SEQ ID NO:75具有99%的同一性的核酸序列;
(d)SEQ ID NO:75;
(e)与SEQ ID NO:76具有95%的同一性的核酸序列;
(f)与SEQ ID NO:76具有98%的同一性的核酸序列;
(g)与SEQ ID NO:76具有99%的同一性的核酸序列;
(h)SEQ ID NO:76;
(i)与SEQ ID NO:77具有95%的同一性的核酸序列;
(j)与SEQ ID NO:77具有98%的同一性的核酸序列;
(k)与SEQ ID NO:77具有99%的同一性的核酸序列;
(l)SEQ ID NO:77;
(m)与SEQ ID NO:78具有95%的同一性的核酸序列;
(n)与SEQ ID NO:78具有98%的同一性的核酸序列;
(o)与SEQ ID NO:78具有99%的同一性的核酸序列;
(p)SEQ ID NO:78;
(q)与SEQ ID NO:79具有95%的同一性的核酸序列;
(r)与SEQ ID NO:79具有98%的同一性的核酸序列;
(s)与SEQ ID NO:79具有99%的同一性的核酸序列;
(t)SEQ ID NO:79;
(u)与SEQ ID NO:80具有95%的同一性的核酸序列;
(v)与SEQ ID NO:80具有98%的同一性的核酸序列;
(w)与SEQ ID NO:80具有99%的同一性的核酸序列;
(x)SEQ ID NO:80;
(y)与SEQ ID NO:81具有95%的同一性的核酸序列;
(z)与SEQ ID NO:81具有98%的同一性的核酸序列;
(aa)与SEQ ID NO:81具有99%的同一性的核酸序列;
(bb)SEQ ID NO:81;
(cc)与SEQ ID NO:82具有95%的同一性的核酸序列;
(dd)与SEQ ID NO:82具有98%的同一性的核酸序列;
(ee)与SEQ ID NO:82具有99%的同一性的核酸序列;
(ff)SEQ ID NO:82;
(gg)与SEQ ID NO:83具有95%的同一性的核酸序列;
(hh)与SEQ ID NO:83具有98%的同一性的核酸序列;
(ii)与SEQ ID NO:83具有99%的同一性的核酸序列;
(jj)SEQ ID NO:83;
(kk)与SEQ ID NO:84具有95%的同一性的核酸序列;
(ll)与SEQ ID NO:84具有98%的同一性的核酸序列;
(mm)与SEQ ID NO:84具有99%的同一性的核酸序列;
(nn)SEQ ID NO:84;
(oo)与SEQ ID NO:85具有95%的同一性的核酸序列;
(pp)与SEQ ID NO:85具有98%的同一性的核酸序列;
(qq)与SEQ ID NO:85具有99%的同一性的核酸序列;
(rr)SEQ ID NO:85;
(ss)与SEQ ID NO:86具有95%的同一性的核酸序列;
(tt)与SEQ ID NO:86具有98%的同一性的核酸序列;
(uu)与SEQ ID NO:86具有99%的同一性的核酸序列;
(vv)SEQ ID NO:86;
(ww)与SEQ ID NO:87具有95%的同一性的核酸序列;
(xx)与SEQ ID NO:87具有98%的同一性的核酸序列;
(yy)与SEQ ID NO:87具有99%的同一性的核酸序列;
(zz)SEQ ID NO:87;
(aaa)与SEQ ID NO:88具有95%的同一性的核酸序列;
(bbb)与SEQ ID NO:88具有98%的同一性的核酸序列;
(ccc)与SEQ ID NO:88具有99%的同一性的核酸序列;
(ddd)SEQ ID NO:88。
实施方案37-1
实施方案37的多核苷酸构建体,其中所述5’内含子片段基本上由选自下组的核酸序列组成:与SEQ ID NO:75-SEQ ID NO:88中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:75-SEQ ID NO:88中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:75-SEQ ID NO:88中的任一个具有99%的同一性的核酸序列、SEQ ID NO:75-SEQ ID NO:88中的任一个。
实施方案37-2
实施方案37的多核苷酸构建体,其中所述5’内含子片段由选自下组的核酸序列组成:与SEQ ID NO:75-SEQ ID NO:88中的任一个具有95%的同一性的核酸序列、与SEQ IDNO:75-SEQ ID NO:88中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:75-SEQ IDNO:88中的任一个具有99%的同一性的核酸序列、SEQ ID NO:75-SEQ ID NO:88中的任一个。
实施方案38
实施方案3-37中任一项的多核苷酸构建体,其中所述5’同源臂包含SEQ ID NO:105的核酸序列。
实施方案38-1
实施方案38的多核苷酸构建体,其中所述5’同源臂基本上由SEQ ID NO:105的核酸序列组成。
实施方案38-2
实施方案38的多核苷酸构建体,其中所述5’同源臂由SEQ ID NO:105的核酸序列组成。
实施方案39
实施方案3-38中任一项的多核苷酸构建体,其中所述3’同源臂包含SEQ ID NO:106的核酸序列。
实施方案39-1
实施方案39的多核苷酸构建体,其中所述3’同源臂基本上由SEQ ID NO:106的核酸序列组成。
实施方案39-2
实施方案39的多核苷酸构建体,其中所述3’同源臂由SEQ ID NO:106的核酸序列组成。
实施方案40
实施方案3-39中任一项的多核苷酸构建体,其中所述5’同源臂或3’同源臂的长度为15-60个核苷酸。
实施方案41
实施方案3-40中任一项的多核苷酸构建体,其中所述5’同源臂或3’同源臂序列具有至多10%的碱基错配。
实施方案42
实施方案1-41中任一项的多核苷酸构建体,其中所述目标序列包含选自下组的5’臂序列:
(a)SEQ ID NO:89;
(b)SEQ ID NO:90;
(c)SEQ ID NO:91;
(d)SEQ ID NO:92;
(e)SEQ ID NO:93;
(f)SEQ ID NO:94;
(g)SEQ ID NO:95;
(h)SEQ ID NO:96。
实施方案43
实施方案1-42中任一项的多核苷酸构建体,其中所述目标序列包含选自下组的3’臂序列:
(a)SEQ ID NO:97;
(b)SEQ ID NO:98;
(c)SEQ ID NO:99;
(d)SEQ ID NO:100;
(e)SEQ ID NO:101;
(f)SEQ ID NO:102;
(g)SEQ ID NO:103;
(h)SEQ ID NO:104。
实施方案44
实施方案1-43中任一项的多核苷酸构建体,其中所述目标序列包含式I:
TI-(L)
其中:
TI为包含内部核糖体进入位点(IRES)样多核苷酸序列或天然IRES序列的改造过的翻译起始元件,
Z1为编码治疗产物的表达序列;
L为接头序列;
A1和B1为能够使所述RNA多核苷酸环化的一对序列;
n为选自0-2的整数。
实施方案45
实施方案44的多核苷酸构建体,其中Z1包括选自下组的核酸序列:
(a)与SEQ ID NO:107具有95%的同一性的核酸序列;
(b)与SEQ ID NO:107具有98%的同一性的核酸序列;
(c)与SEQ ID NO:107具有99%的同一性的核酸序列;
(d)SEQ ID NO:107;
(e)与SEQ ID NO:108具有95%的同一性的核酸序列;
(f)与SEQ ID NO:108具有98%的同一性的核酸序列;
(g)与SEQ ID NO:108具有99%的同一性的核酸序列;
(h)SEQ ID NO:108;
(i)与SEQ ID NO:109具有95%的同一性的核酸序列;
(j)与SEQ ID NO:109具有98%的同一性的核酸序列;
(k)与SEQ ID NO:109具有99%的同一性的核酸序列;
(l)SEQ ID NO:109;
(m)与SEQ ID NO:110具有95%的同一性的核酸序列;
(n)与SEQ ID NO:110具有98%的同一性的核酸序列;
(o)与SEQ ID NO:110具有99%的同一性的核酸序列;
(p)SEQ ID NO:110;
(q)与SEQ ID NO:111具有95%的同一性的核酸序列;
(r)与SEQ ID NO:111具有98%的同一性的核酸序列;
(s)与SEQ ID NO:111具有99%的同一性的核酸序列;
(t)SEQ ID NO:111;
(u)与SEQ ID NO:112具有95%的同一性的核酸序列;
(v)与SEQ ID NO:112具有98%的同一性的核酸序列;
(w)与SEQ ID NO:112具有99%的同一性的核酸序列;
(x)SEQ ID NO:112。
实施方案45-1
实施方案45的多核苷酸构建体,其中Z1基本上由选自下组的核酸序列组成:与SEQID NO:107-SEQ ID NO:112中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:107-SEQ ID NO:112中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:107-SEQ ID NO:112中的任一个具有99%的同一性的核酸序列、SEQ ID NO:107-SEQ ID NO:112中的任一个。
实施方案45-2
实施方案45的多核苷酸构建体,其中Z1由选自下组的核酸序列组成:与SEQ IDNO:107-SEQ ID NO:112中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:107-SEQID NO:112中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:107-SEQ ID NO:112中的任一个具有99%的同一性的核酸序列、SEQ ID NO:107-SEQ ID NO:112中的任一个。
实施方案46
实施方案44的多核苷酸构建体,其中Z1包括编码选自下组的氨基酸序列的核酸序列:
(a)SEQ ID NO:113;
(b)SEQ ID NO:114;
(c)SEQ ID NO:115;
(d)SEQ ID NO:116;
(e)SEQ ID NO:117;
(f)SEQ ID NO:118;
实施方案46-1
实施方案46的多核苷酸构建体,其中所述Z1基本上由编码选自下组的氨基酸序列的核酸序列组成:SEQ ID NO:113-SEQ ID NO:118。
实施方案46-2
实施方案46的多核苷酸构建体,其中所述Z1由编码选自下组的氨基酸序列的核酸序列组成:SEQ ID NO:113-SEQ ID NO:118。
实施方案47
实施方案1-46中任一项的多核苷酸构建体,其包含经修饰的RNA核苷酸和/或经修饰的核苷。
实施方案48
实施方案1-47中任一项的多核苷酸构建体,其包含10%-100%的经修饰的RNA核苷酸和/或经修饰的核苷。
实施方案49
实施方案47-48中任一项的多核苷酸构建体,其中所述经修饰的RNA核苷酸和/或经修饰的核苷中的至少一种为m5C(5-甲基胞苷)。
实施方案50
实施方案47-48中任一项的多核苷酸构建体,其中所述经修饰的RNA核苷酸和/或经修饰的核苷中的至少一种为m5U(5-甲基尿苷)。
实施方案51
实施方案47-48中任一项的多核苷酸构建体,其中所述经修饰的RNA核苷酸和/或经修饰的核苷中的至少一种为m6A(N6-甲基腺苷)。
实施方案52
实施方案47-48中任一项的多核苷酸构建体,其中所述经修饰的RNA核苷酸和/或经修饰的核苷中的至少一种为Y(假尿苷)。
实施方案53
实施方案47-48中任一项的多核苷酸构建体,其中所述经修饰的RNA核苷酸和/或经修饰的核苷中的至少一种为m1A(1-甲基腺苷)。
实施方案54
实施方案47-53中任一项的多核苷酸构建体,其中在体外转录(IVT)时引入所述经修饰的RNA核苷酸和/或经修饰的核苷中的至少一种。
实施方案55
实施方案47-48中任一项的多核苷酸构建体,其中所述经修饰的核苷选自下组:m5C(5-甲基胞苷)、m5U(5-甲基尿苷)、m6A(N6-甲基腺苷)、s2U(2-硫代尿苷)、Y(假尿苷)、Um(2'-O-甲基尿苷)、m1A(1-甲基腺苷)、m2A(2-甲基腺苷)、Am(2’-0-甲基腺苷)、ms2 m6A(2-甲基硫代-N6-甲基腺苷)、i6A(N6-异戊烯基腺苷)、ms2i6A(2-甲基硫代-N6异戊烯基腺苷)、io6A(N6-(顺式-羟基异戊烯基)腺苷)、ms2io6A(2-甲基硫代-N6-(顺式-羟基异戊烯基)腺苷)、g6A(N6-甘氨酰基氨基甲酰基腺苷)、t6A(N6-苏氨酰基氨基甲酰基腺苷)、ms2t6A(2-甲基硫代-N6-苏氨酰基氨基甲酰基腺苷)、m6t6A(N6-甲基-N6-苏氨酰基氨基甲酰基腺苷)、hn6A(N6-羟基正缬氨酰基氨基甲酰基腺苷)、ms2hn6A(2-甲基硫代-N6-羟基正缬氨酰基氨基甲酰基腺苷)、Ar(p)(2’-0-核糖基腺苷(磷酸))、I(肌苷)、m1I(1-甲基肌苷)、m1hn(1,2’-O-二甲基肌苷)、m3C(3-甲基胞苷)、Cm(2’-0-甲基胞苷)、s2C(2-硫代胞苷)、ac4C(N4-乙酰基胞苷)、(5-甲酰基胞苷)、m5Cm(5,2'-O-二甲基胞苷)、ac4Cm(N4-乙酰基-2’-O-甲基胞苷)、k2C(赖胞苷)、m!G(1-甲基鸟苷)、m2G(N2-甲基鸟苷)、m7G(7-甲基鸟苷)、Gm(2'-0-甲基鸟苷)、m2 2G(N2,N2-二甲基鸟苷)、m2Gm(N2,2’-O-二甲基鸟苷)、m2 aGm(N2,N2,2’-O-三甲基鸟苷)、Gr(p)(2’-0-核糖基鸟苷(磷酸))、yW(怀丁苷)、oayW(过氧怀丁苷)、OHyW(羟基怀丁苷)、OHyW*(修饰不足的羟基怀丁苷)、imG(怀俄苷)、mimG(甲基怀俄苷)、Q(辫苷)、oQ(环氧辫苷)、galQ(半乳糖基-辫苷)、manQ(甘露糖基-辫苷)、preQo(7-氰基-7-去氮鸟苷)、preQi(7-氨基甲基-7-去氮鸟苷)、G+(古嘌苷)、D(二氢尿苷)、m5Um(5,2’-0-二甲基尿苷)、s4U(4-硫代尿苷)、m5s2U(5-甲基-2-硫代尿苷)、s2Um(2-硫代-2’-0-甲基尿苷)、acp3U(3-(3-氨基-3-羧基丙基)尿苷)、ho5U(5-羟基尿苷)、mo5U(5-甲氧基尿苷)、cmo5U(尿苷5-氧基乙酸)、mcmo5U(尿苷5-氧基乙酸甲基酯)、chm5U(5-(羧基羟基甲基)尿苷)、mchm5U(5-(羧基羟基甲基)尿苷甲基酯)、mcm5U(5-甲氧基羰基甲基尿苷)、mcm5Um(5-甲氧基羰基甲基-2’-0-甲基尿苷)、mcm5s2U(5-甲氧基羰基甲基-2-硫代尿苷)、nm5S2U(5-氨基甲基-2-硫代尿苷)、mnm5U(5-甲基氨基甲基尿苷)、mnm5s2U(5-甲基氨基甲基-2-硫代尿苷)、mnm5se2U(5-甲基氨基甲基-2-硒基尿苷)、ncm5U(5-氨基甲酰基甲基尿苷)、ncm5Um(5-氨基甲酰基甲基-2'-O-甲基尿苷)、cmnm5U(5-羧基甲基氨基甲基尿苷)、cmnm5Um(5-羧基甲基氨基甲基-2'-0-甲基尿苷)、cmnm5s2U(5-羧基甲基氨基甲基-2-硫代尿苷)、m6 2A(N6,N6-二甲基腺苷)、Im(2’-0-甲基肌苷)、m4C(N4-甲基胞苷)、m4Cm(N4,2’-0-二甲基胞苷)、hm5C(5-羟基甲基胞苷)、m3U(3-甲基尿苷)、cm5U(5-羧基甲基尿苷)、m6Am(N6,2’-O-二甲基腺苷)、m6 2Am(N6,N6,0-2’-三甲基腺苷)、m2,7G(N2,7-二甲基鸟苷)、m2,2,7G(N2,N2,7-三甲基鸟苷)、m3Um(3,2’-0-二甲基尿苷)、m5D(5-甲基二氢尿苷)、f5Cm(5-甲酰基-2’-0-甲基胞苷)、m'Gm(l,2’-0-二甲基鸟苷)、m'Am(l,2’-0-二甲基腺苷)、rm 5U(5-牛磺酸甲基尿苷)、τm5s2U(5-牛磺酸甲基-2-硫代尿苷))、imG-14(4-去甲基怀俄苷)、imG2(异怀俄苷)、ac6A(N6-乙酰基腺苷)、吡啶-4-酮核糖核苷、5-氮杂-尿苷、2-硫代-5-氮杂-尿苷、2-硫代尿苷、4-硫代-假尿苷、2-硫代-假尿苷、5-羟基尿苷、3-甲基尿苷、5-羧基甲基-尿苷、1-羧基甲基-假尿苷、5-丙炔基-尿苷、1-丙炔基-假尿苷、5-牛磺酸甲基尿苷、1-牛磺酸甲基-假尿苷、5-牛磺酸甲基-2-硫代-尿苷、l-牛磺酸甲基-4-硫代-尿苷、5-甲基-尿苷、1-甲基-假尿苷、4-硫代-1-甲基-假尿苷、2-硫代-1-甲基-假尿苷、1-甲基-1-去氮-假尿苷、2-硫代-1-甲基-1-去氮-假尿苷、二氢尿苷、二氢假尿苷、2-硫代-二氢尿苷、2-硫代-二氢假尿苷、2-甲氧基尿苷、2-甲氧基-4-硫代-尿苷、4-甲氧基-假尿苷、4-甲氧基-2-硫代-假尿苷、5-氮杂-胞苷、假异胞苷、3-甲基-胞苷、N4-乙酰基胞苷、5-甲酰基胞苷、N4-甲基胞苷、5-羟基甲基胞苷、1-甲基-假异胞苷、吡咯并-胞苷、吡咯并-假异胞苷、2-硫代-胞苷、2-硫代-5-甲基-胞苷、4-硫代-假异胞苷、4-硫代-1-甲基-假异胞苷、4-硫代-1-甲基-1-去氮-假异胞苷、1-甲基-1-去氮-假异胞苷、泽布拉林(zebularine)、5-氮杂-泽布拉林、5-甲基-泽布拉林、5-氮杂-2-硫代-泽布拉林、2-硫代-泽布拉林、2-甲氧基-胞苷、2-甲氧基-5-甲基-胞苷、4-甲氧基-假异胞苷、4-甲氧基-1-甲基-假异胞苷、2-氨基嘌呤、2,6-二氨基嘌呤、7-去氮-腺嘌呤、7-去氮-8-氮杂-腺嘌呤、7-去氮-2-氨基嘌呤、7-去氮-8-氮杂-2-氨基嘌呤、7-去氮-2,6-二氨基嘌呤、7-去氮-8-氮杂-2,6-二氨基嘌呤、1-甲基腺苷、N6-甲基腺苷、N6-异戊烯基腺苷、N6-(顺式-羟基异戊烯基)腺苷、2-甲基硫代-N6-(顺式-羟基异戊烯基)腺苷、N6-甘氨酰基氨基甲酰基腺苷、N6-苏氨酰基氨基甲酰基腺苷、2-甲基硫代-N6-苏氨酰基氨基甲酰基腺苷、N6,N6-二甲基腺苷、7-甲基腺嘌呤、2-甲基硫代-腺嘌呤、2-甲氧基-腺嘌呤、肌苷、1-甲基-肌苷、怀俄苷、怀丁苷、7-去氮-鸟苷、7-去氮-8-氮杂-鸟苷、6-硫代-鸟苷、6-硫代-7-去氮-鸟苷、6-硫代-7-去氮-8-氮杂-鸟苷、7-甲基-鸟苷、6-硫代-7-甲基-鸟苷、7-甲基肌苷、6-甲氧基-鸟苷、1-甲基鸟苷、N2-甲基鸟苷、N2,N2-二甲基鸟苷、8-氧代-鸟苷、7-甲基-8-氧代-鸟苷、1-甲基-6-硫代-鸟苷、N2-甲基-6-硫代-鸟苷、N2,N2-二甲基-6-硫代-鸟苷、5-甲基胞嘧啶、假尿苷、1-甲基假尿苷。
实施方案56
通过实施方案1-55中任一项的多核苷酸构建体产生的环形RNA,例如,所述环形RNA的长度为至少500个核苷酸、长度为至少1,000个核苷酸、长度为至少1,500个核苷酸。
实施方案57
实施方案56的环形RNA,其不含任何不属于所述目标序列的其他序列,如不含E2、E1序列的全部或部分。
实施方案58
一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)目标序列;
(d)外显子片段1(E1);
(e)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案59
一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)3’内含子片段;
(b)外显子片段2(E2);
(c)接头序列;
(d)目标序列;
(e)接头序列;
(f)外显子片段1(E1);
(g)5’内含子片段,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案60
一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)目标序列;
(e)外显子片段1(E1);
(f)5’内含子片段;
(g)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案61
一种制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:
(a)5’同源臂;
(b)3’内含子片段;
(c)外显子片段2(E2);
(d)接头序列;
(e)目标序列;
(f)接头序列;
(g)外显子片段1(E1);
(h)5’内含子片段;
(i)3’同源臂,
其中:
所述5’内含子片段和3’内含子片段各自为II型内含子的片段,其中所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧,
所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸,
所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,
所述目标序列为空,或为蛋白编码序列、非编码序列或二者的组合。
实施方案62
一种在细胞中表达蛋白质的方法,包括(a)将实施方案58-61中任一项的环形RNA转染到所述细胞中,或者(b)使实施方案1-57中任一项的多核苷酸构建体发生自剪接环化反应形成环形RNA,并将所述环形RNA转染到所述细胞中;其中,优选所述细胞为真核细胞。
实施方案63
一种在细胞中表达蛋白质的方法,包括(a)将实施方案58-61中任一项的环形RNA转染到所述细胞中,或者(b)使实施方案1-57中任一项的构建体发生自剪接环化反应形成环形RNA,并将所述环形RNA转染到所述细胞中;其中,优选所述细胞为肝细胞、上皮细胞、造血细胞、上皮细胞、内皮细胞、肺细胞、骨细胞、干细胞、间充质细胞、神经细胞(例如,脑膜细胞、星形胶质细胞、运动神经元、背根神经节细胞、前角运动神经元)、感光细胞(例如,视杆细胞、视锥细胞)、视网膜色素上皮细胞、分泌细胞、心脏细胞、脂肪细胞、血管平滑肌细胞、心肌细胞、骨骼肌细胞、β细胞、垂体细胞、滑膜衬里细胞、卵巢细胞、睾丸细胞、成纤维细胞、B细胞、T细胞、树突细胞、巨噬细胞、网织红细胞、白细胞、粒细胞、肿瘤细胞、NK细胞、肝星形细胞(starlet cell)、HEK293、HEK293T、HeLa、MCF7、PC3、A549、NCI-H727、HCT-116、MCF10A、HPReC、FHC、永生化细胞系、原代细胞、酵母细胞、酿酒酵母(Saccharomyces cerevisiae)、毕赤酵母(Pichia pastoris)、细菌细胞、大肠杆菌(Escherichia coli)、昆虫细胞、草地贪夜蛾(Spodoptera frugiperda)sf9、Mimic Sf9、sf21、果蝇(Drosophila)S2。
实施方案64
一种使用II型内含子生成具有自剪接活性的序列的方法,所述方法包括以下步骤:
(a)定义所述II型内含子的序列;任选地使用剪接测定(线性剪接)检查所述II型内含子的体外自剪接活性;
(b)将所述II型内含子劈分成两个片段,
(c)将所述两个内含子片段的顺序颠倒,
(d)使用剪接测定确认RNA的体外环化。
本发明还包括以下实施方案。
实施方案65
前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述5’内含子片段和3’内含子片段中分别包含一对或多对彼此互补的配对序列。在优选的实施方案中,所述互补配对序列的长度大于20个核苷酸。
实施方案66
前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述5’内含子片段和/或3’内含子片段中包含一个或者多个亲和标签序列,所述亲和标签序列选自下组:探针结合序列、MS2结合位点、PP7结合位点、链霉亲和素结合位点。
实施方案67
前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述EBS序列选自EBS1、EBS2、EBS3中的一个或多个,优选两个,更优选EBS1和EBS3。
实施方案68
前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述II型内含子的一个或多个EBS序列,优选EBS1和EBS3,被修饰,其中所述EBS序列分别与目标序列中相应长度的两个区域在至少60%的核苷酸位置上互补配对。在优选的实施方案中,所述目标序列中相应长度的两个区域分别位于所述目标序列的两端。
实施方案69
前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述多核苷酸构建体能够在体外形成目标序列的环形RNA。
实施方案70
前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述多核苷酸构建体能够在体内形成目标序列的环形RNA。
5.附图说明
参考各个附图来说明本发明的实施方案。
图1是介绍本发明的方法的流程图,展现了从天然的自剪接核酶开始,经过设计、改造,最终反应获得环形RNA的过程。
图2A-B展现了实施例1的II型内含子的筛选过程。(A)制备包含Gluc编码序列片段和E1-II型内含子(自剪接核酶)-E2的DNA构建体,以该DNA构建体为模板,通过体外转录制备线性RNA并纯化,如果线性RNA通过体外自剪接反应产生大小不同的两个片段(被切下的内含子,以及构建体的剩余部分),则证明了体外自剪接活性,II型内含子及其侧翼E1、E2序列可作为设计cRNAzyme构建体的cRNAzyme前体。(B)用于筛选cRNAzyme前体的体外自剪接反应条件。
图3是根据实施例1的方法确认具有自剪接活性的2种II型内含子的凝胶电泳图。II型内含子的名称在各自电泳图上以3字母代码标出。
图4A-C是以Cte为例设计cRNAzyme构建体的方案和不同方案之间的比较性实验结果。(A)cRNAzyme构建体设计;(B)在不同位置分割IICte内含子并得到构建体之后,通过凝胶电泳确定的环化效率;(C)通过不同方法验证环形RNA成功形成的实验的结果图。
图5A-C显示了优化过程中,不同条件下获得的结果。(A)通过优化反应条件和改造序列,cRNAzyme构建体环化效率得到了升高;(B)以Cte作为示例性自剪接核酶,在不同反应条件下环化产物的凝胶电泳结果图,下部的柱状图显示了量化的环化效率PC%(环化效率(PC%)=环形/(环形+线性)×100%);(C)以Cte作为示例性核酶,以海肾萤光素酶(Renilla Luciferase)(Rluc)作为插入片段,在加入不同间隔序列的情况下,通过三种构建体产生的环化产物的凝胶电泳图,下部的柱状图显示了量化的环化效率PC%。
图6A-B涉及实施例4中制备的能够消除疤痕序列的改进构建体。(A)构建体结构示意图。(B)不同镁离子浓度下,三种目标序列的环化产物的凝胶电泳图以及测序结果。
图7显示了插入不同长度目标序列时生成的环形RNA的凝胶电泳结果。
图8A-B是使用本发明的构建体和方法生成的不同目标序列的环形RNA在胞内表达的结果。(A)使用“无痕”构建体并以GFP为目标序列形成环形RNA,在转染细胞后通过Western印迹检测GFP表达的结果;(B)使用“无痕”构建体并以Gluc为目标序列形成环形RNA,在转染细胞后通过酶标仪检测Gluc表达的结果。
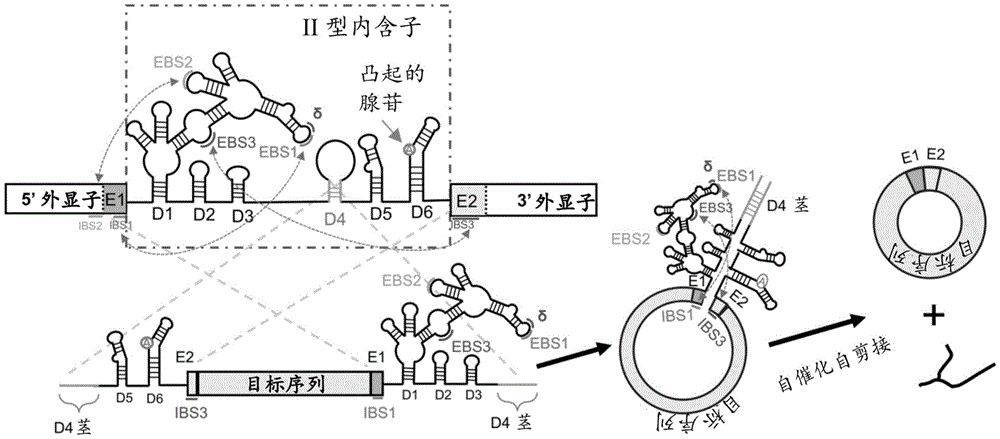
图9是II型内含子结构示意图。
图10显示了II型内含子的(a.)分支途径、(b.)水解途径。
图11显示了I型、II型内含子的剪接机制。
图12A是基于IBS1与EBS1、IBS2与EBS2、IBS3与EBS3之间的相互作用而设计的近无痕系统的示意图。自催化自剪接II型内含子从D4结构域被劈分成两个片段,并在劈分的内含子之间插入包含E1、E2、目标序列的定制外显子。箭头指示IBS1与EBS1、IBS2与EBS2、IBS3与EBS3之间的相互作用。
图12B是基于δ与IBS3之间的相互作用而设计的近无痕系统的设计的示意图。自催化自剪接II型内含子从D4结构域被劈分成两个片段,并在劈分的内含子之间插入包含E1、E2、目标序列的定制外显子。箭头指示IBS1与EBS1、IBS2与EBS2、IBS3与δ之间的相互作用。
图12C是基于IBS1’与EBS1之间的相互作用而设计的无痕系统的示意图。自催化自剪接II型内含子从D4结构域被劈分成两个片段,并在劈分的内含子之间插入目标序列。箭头指示IBS1’与EBS1、IBS3’与EBS3之间的相互作用。IBS1’是目标序列上功能与IBS1相似的区域。IBS3’是目标序列上功能与IBS3相似的区域。
图12D是基于δ与IBS3’之间的相互作用而设计的无痕系统的示意图。自催化自剪接II型内含子从D4结构域被劈分成两个片段,并在劈分的内含子之间插入目标序列。箭头指示IBS1’与EBS1、IBS3’与δ之间的相互作用。
图13是体外合成并用琼脂糖凝胶分析的circRNA的结果。IBS1被突变以无法自剪接。IVT后,通过Poly A加尾和RNase R处理确认环化的RNA。
图14是图4C的更新图,其显示了通过不同方法验证环形RNA成功形成的实验结果以及线性、环形RNA构建体的图。
图15显示了跨越图14的泳道1、泳道3中描绘的CircRNA样品的剪接接头(junction)的RT-PCR的sanger测序输出。
图16是图6B的更新图,其显示了构建体的图、三种目标序列的环化产物在不同镁离子浓度下的凝胶电泳图以及测序结果。
图17显示了在CVB3 IRES前使用不同间隔区的环化效率。
图18显示了来自源自不同cRNAzyme变体的环形RNA的萤光素酶蛋白表达的发光信号。
图19显示了不同剂量的circRNA的转染、翻译。对包含两种不同间隔子的circRNA进行凝胶纯化并以不同剂量转染到在24孔板中培养的三个细胞系中,转染后24小时测量萤光素酶的活性。
图20显示了包含不同基因的cRNAzyme变体CV4的环化效率。将不同的RNA在体外转录并在体外转录反应中环化。基因1:Gluc,基因2:EGFP,基因3:RBD,基因4:Rluc,基因5:Fluc,基因6:saCAS9。
图21显示了circRNA翻译的时间过程实验的结果。将编码Rluc基因的circRNA转染到293T细胞中(在每次转染中使用500ng circRNA),并在转染后6、12、24小时测量萤光素酶活性。
图22显示了转染到细胞中后来自线性mRNA、circRNA的产生。
图23是来自线性mRNA与使用PIE方案或新的CirCode系统产生的circRNA的蛋白质产生的比较。
图24显示了在IVT后来自旋转柱纯化的样品的CVB3-Gluc circRNA的HPLC纯化。上部小图是分别指示前体、环形、内含子RNA的峰的HPLC色谱图。下部小图展示了输入级分、收集的级分的琼脂糖凝胶。
图25显示了与模拟转染相比,由未纯化的circRNA、纯化的circRNA的转染引起的细胞死亡量。
图26显示了通过诱导RIG-I、IFN-B1而刺激先天免疫反应的未纯化的circRNA与纯化的circRNA的比较。
图27展示了circRNA的产生的放大。
图28展示了使用采用Agilent 2100生物分析仪的毛细管电泳分析从HPLC纯化的四批CVB3-Gluc circRNA。
图29是CircRNA-LNP复合物的示意图和CircRNAGluc-LNP的粒度。
图30显示了注射采用不同配制品的CircRNAGluc-LNP后24小时从小鼠血清中测定的Gaussia萤光素酶活性。
图31显示了通过肌内(i.m.)途径施用采用两种配制品的20ug CircRNAGluc-LNP的BALB/c小鼠的代表性IVIS图像。显示了相对发光图,并指示了发光的标度。
图32显示了从HPLC纯化的circRNA-RBD、circRNA-RBD二聚体,将它们使用采用Agilent 2100生物分析仪的毛细管电泳(左下)和琼脂糖凝胶电泳进行分析。
图33显示了CircRNARBD-LNP复合物的粒度、包封效率。
图34显示了BALB/c小鼠中的CircRNARBD-LNP疫苗接种过程和血清收集时间表的示意图。
图35显示了RBD结合B细胞的结果。设计流式细胞术抗体组,以鉴定幼稚B细胞(CD19+IgD+CD27-)、总记忆B细胞(CD19+CD27+)(包括未转换的IgD+群体、转换的IgD-群体)、浆细胞(CD19+IgD-CD38+CD27+)、移行性B细胞(CD19+IgD暗CD38+)。为了确定LNP-circRNA-RBP疫苗接种是否诱导抗原特异性B细胞的激活、扩增,我们使用Alexa 647标记的RBD(RBD-Alexa 647)测量RBD结合B细胞的频率。我们发现在记忆B细胞中检测到很大一部分的RBD特异性淋巴细胞(即,CD19+CD27+B淋巴细胞),包括转换的RBD特异性B细胞群体(CD19+CD27+IgD-RBD+)、未转换的RBD特异性记忆B细胞群体(CD19+CD27+IgD+RBD+)。
图36显示了CircRNARBD-LNP疫苗接种引起的抗体反应。加强后2周收集血清,并通过ELISA评估RBD特异性IgG1、IgG2a、IgG2c。
图37显示了RBD与hACE2过表达细胞系的结合的抑制。
图38显示了IgG2a/IgG1与IgG2c/IgG1之间的比率。
图39显示了CircRNARBD-LNP疫苗接种引起的中和抗体反应。获得加强后2周收集的血清的假病毒中和滴度。
图40显示了具有IBS1、IBS2、IBS3、EBS1、EBS2、EBS3(以粗线显示)的II型内含子的示意图。
图41是具有IBS1、IBS2、IBS3、EBS1、EBS2、EBS3、δ(以粗体显示)的II型内含子的结构示意图。
6.具体实施方式
6.1.定义
如本文所用,关于指定组分的“基本上不含”在本文中用于意指没有指定组分已经被有目的地配制到组合物中和/或仅作为污染物或以痕量存在。因此,由组合物的任何意外污染产生的指定组分的总量远低于0.1%,优选低于0.05%,更优选低于0.01%。最优选的是这样的组合物,其中用标准分析方法不能检测到指定组分的量。
如本文在说明书中所用,“一个/一种(a)”或“一个/一种(an)”可以意指一个或多个/一种或多种。如本文在一项或多项权利要求中所用,当与词语“包含”结合使用时,词语“一个/一种(a)”或“一个/一种(an)”可以意指一个或多于一个/一种或多于一种。
如本文所用,在权利要求中的术语“或”用于意指“和/或”,除非明确指出仅指代替代方案或替代方案是相互排斥的,尽管本公开文本支持仅指代替代方案和“和/或”的定义。如本文所用,“另一个/另一种”或“另外的”可以意指至少第二个或更多个/至少第二种或更多种。
如本文所用,术语“约”用于指示值包括用于确定值的装置、方法的误差的固有变化或研究对象之间存在的变化。在一些实施方案中,“约”意指变化是“约”所指代的值的±5%、±4%、±3%、±2%、±1%、±0.5%、±0.2%或±0.1%。在一些实施方案中,“约”意指变化是“约”所指代的值的±1%、±0.5%、±0.2%或±0.1%。
术语“cRNAzyme”在本文中用于指代能够经由自催化的反向剪接反应产生环形RNA的线性核糖核酸(RNA)。
术语“cRNAzyme构建体”是具有cRNAzyme活性的线性RNA构建体。
术语“EBS”在本文中用于指代外显子结合序列,其与外显子区域中的内含子结合序列(IBS)相互作用(例如形成互补配对),依靠EBS核酸序列内部自身的羟基触发剪接。
术语“EBS1”在本文中用于指代外显子结合序列1。参见图9和41。
术语“EBS2”在本文中用于指代外显子结合序列2。参见图9和41。
术语“EBS3”在本文中用于指代外显子结合序列3。参见图9和41。
术语“EBS1’”在本文中用于指代与IBS1’相互作用的经修饰的EBS1序列。EBS1’与IBS1’之间的相互作用和EBS1与IBS1之间的相互作用相似。参见图12C和12D。
术语“EBS3’”在本文中用于指代与IBS3’相互作用的经修饰的EBS3序列。EBS3’与IBS3’之间的相互作用和EBS3与IBS3之间的相互作用相似。参见图12C。
术语“结构域1”或“D1”在本文中用于指代II型内含子的结构域1的茎环结构。术语“结构域2”或“D2”在本文中用于指代II型内含子的结构域2的茎环结构。术语“结构域3”或“D3”在本文中用于指代II型内含子的结构域3的茎环结构。术语“结构域4”或“D4”在本文中用于指代II型内含子的结构域4的茎环结构。术语“结构域5”或“D5”在本文中用于指代II型内含子的结构域5的茎环结构。术语“结构域6”或“D6”在本文中用于指代II型内含子的结构域6的茎环结构。茎环结构是一种类型的RNA二级结构,其可以通过任何合适的多核苷酸折叠算法来确定。一些程序是基于最小吉布斯自由能的计算。一种这样的算法的一个例子是mFold,并且由Zuker和Stiegler(Nucleic Acids Res.9(1981),133-148)描述。另一种示例性折叠算法是由维也纳大学理论化学研究所使用质心结构预测算法(例如,AR Gruber等人,2008,Cell106).(1):23-24;以及PA Carr和GM Church,2009,Nature Biotechnology27(12):1151-62)开发的在线网络服务器RNAfold。另外的算法可以在美国临时专利申请号61/836,080(代理人案卷号44790.11.2022;泛参考编号(Broad reference number)BI-2013/004A)中找到,将其通过引用并入本文。II型内含子主要包括6个茎环结构,称为结构域1-6(D1-D6),6个结构域顺序排列,其中含有多个外显子结合序列(EBS),如EBS1、EBS2、EBS3。这些EBS序列与外显子区域中的内含子结合序列(IBS)进行相互作用如互补配对,依靠EBS核酸序列内部自身的羟基触发剪接。
如本文所用,术语“II型内含子”在本文中用于指代具有相似的二级和三级结构的由II型内含子编码的RNA分子。II型内含子RNA分子通常具有六个结构域。参见图9和图41。II型内含子RNA的结构域4(也称为结构域IV)包含编码“II型内含子编码蛋白”的核苷酸序列。
术语“IBS”在本文中用于指代内含子结合序列,其与外显子结合序列(EBS)相互作用以定位剪接位点。
术语“IBS1”在本文中用于指代内含子结合序列1,其与外显子结合序列1(EBS1)相互作用以定位剪接位点。
术语“IBS1’”在本文中用于指代目标序列上功能与IBS1相似的区域。
术语“IBS2”在本文中用于指代内含子结合序列2,其与外显子结合序列2(EBS2)相互作用以定位剪接位点。
术语“IBS3”在本文中用于指代内含子结合序列3,其与外显子结合序列3(EBS3)相互作用以定位剪接位点。
术语“IBS3’”在本文中用于指代目标序列上功能与IBS3相似的区域。
术语“δ”(delta)在本文中用于指代II型内含子的结构域1上的区域,其是EBS1的直接上游的单个核苷酸。δ与IBS3配对并且δ与IBS3之间的相互作用称为δ-IBS3配对。参见图41、12B。
术语“δ””(delta”)在本文中用于指代II型内含子的结构域1上的区域,其是EBS1’的直接上游的单个核苷酸。δ”与IBS3’配对并且δ”与IBS3'之间的相互作用称为δ”-IBS3’配对。参见图41、12D。
术语“IVT”在本文中用于指代体外转录,其是使用RNA聚合酶、核糖核苷酸和适当的缓冲条件从DNA模板合成RNA以在体外产生RNA的通用方法。
如本文所用,术语“部分”当关于多肽或肽使用时是指多肽或肽的片段。在一些实施方案中,多肽或肽的“部分”保留衍生出它的全长多肽或肽的至少一种功能和/或活性。例如,在一些实施方案中,如果全长多肽结合给定的配体,则该全长多肽的一部分也结合相同的配体。
术语“蛋白质”和“多肽”在本文中可互换地使用。
当关于细胞或生物体中的蛋白质、基因、核酸或多核苷酸使用时,术语“外源”是指已经通过人工或天然手段引入细胞或生物体中的蛋白质、基因、核酸或多核苷酸;或者当关于细胞使用时,所述术语是指通过人工或天然手段分离并且随后引入细胞群体或生物体中的细胞。外源核酸可以来自不同的生物体或细胞,或者它可以是在生物体或细胞内天然存在的核酸的一个或多个另外的拷贝。外源细胞可以来自不同的生物体,或者它可以来自相同的生物体。作为非限制性例子,外源核酸是处于与其在天然细胞中的位置不同的染色体位置中的核酸,或者是以其他方式侧翼于与在自然界中发现的核酸序列不同的核酸序列的核酸。术语“外源”可与术语“异源”互换使用。
“表达构建体”或“表达盒”用于意指能够指导转录的核酸分子。表达构建体至少包括一种或多种转录控制元件(如启动子、增强子或其功能上等同的结构),其指导在一种或多种所需细胞类型、组织或器官中的基因表达。还可以包括另外的元件,如转录终止信号。
“载体”或“构建体”(有时称为基因递送系统或基因转移“媒介物”)是指包含待在体外或体内递送到宿主细胞的多核苷酸或由所述多核苷酸表达的蛋白质的大分子或分子复合物。
“质粒”(一种常见类型的载体)是能够独立于染色体DNA复制的与染色体DNA分开的染色体外DNA分子。在某些情况下,它是环形双链的。
术语“核酸序列”、“多核苷酸”和“寡核苷酸”在本文中可互换地使用,并且是指嘧啶和/或嘌呤碱基(如分别地胞嘧啶、胸腺嘧啶和尿嘧啶,腺嘌呤和鸟嘌呤)的聚合物或低聚物(参见Albert L.Lehninger,Principles of Biochemistry,793-800(WorthPub.1982)),除非另有说明或上下文指示相反的内容。所述术语涵盖任何脱氧核糖核苷酸、核糖核苷酸或肽核酸组分及其任何化学变体,如这些碱基的甲基化、羟甲基化或糖基化形式。聚合物或低聚物的组成可以是异质的或同质的,可以从天然存在的来源中分离,或者可以人工或合成产生。此外,核酸可以是DNA或RNA或其混合物,并且可以以单链或双链形式(包括同源双链、异源双链和杂交状态)永久或过渡地存在。核酸或核酸序列可以包含其他种类的核酸结构,例如像DNA/RNA螺旋、肽核酸(PNA)、吗啉代核酸(参见例如,Braasch和Corey,Biochemistry,4/(14):4503-4510(2002)和美国专利5,034,506)、锁核酸(LNA;参见Wahlestedt等人,Proc.Natl.Acad.Sci.U.S.A.,97:5633-5638(2000))、环己烯基核酸(参见Wang,Am.Chem.Soc.,122:8595-8602(2000))和/或核酶。术语“核酸”、“核酸序列”、“多核苷酸”和“寡核苷酸”还可以涵盖包含可以展现出与天然核苷酸相同的功能的非天然核苷酸、经修饰的核苷酸和/或非核苷酸结构单元(例如,“核苷酸类似物”)的链。术语“DNA序列”在本文中用于指代包含一系列DNA碱基的核酸。
术语“多肽”和“蛋白质”在本文中可互换地使用,并且是指包含至少两个或更多个连续氨基酸化学或生物化学修饰或衍生的氨基酸的聚合形式的氨基酸。如本文所用的术语“肽”是指一类短多肽。术语肽可以指代具有多达约100个氨基酸的长度的氨基酸(天然或非天然存在的)的聚合物。例如,肽的长度可以为约1至约10、约10至约25、约25至约50、约50至约75、约75至约100个氨基酸残基。在一些实施方案中,肽的长度可以为约100、约200、约300、约400、约500、约600、约700、约800、约900、约1000、约1250、约1500、约1750、约2000、约2250、约2500、约2750、约3000、约3250、约3500、约3750、约4000、约4250、约4500、约4750、约5000个氨基酸残基。
本文使用的核苷酸、核酸、核苷和氨基酸的命名法与国际纯粹与应用化学联合会(IUPAC)标准(参见例如,bioinformatics.org/smsylupac.html)一致。
当指代核酸序列或蛋白质序列时,术语“同一性”用于表示两个序列之间的相似性。序列相似性或同一性可以使用本领域已知的标准技术来确定,所述标准技术包括但不限于Smith和Waterman,Adv.Appl.Math.2,482(1981)的局部序列同一性算法、Needleman和Wunsc h,J Mol.Biol.48,443(1970)的序列同一性比对算法、Pearson和Lipma n,Proc.Natl.Acad.Sci.USA85,2444(1988)的相似性搜索方法、这些算法的计算机化实现(威斯康星州麦迪逊科学大道575号(575Science Drive)的Genetics Computer Group的威斯康星遗传学软件包(Wisconsi n Genetics Software Package)中的GAP、BESTFIT、FASTA和TFAST A)、Devereux等人,Nucl.Acid Res.12,387-395(1984)描述的Best Fi t程序或检查。另一种算法是Altschul等人,J Mol.Biol.215,403-410,(1990)和Karlin等人,Proc.Natl.Acad.Sci.USA 90,5873-5787(1993)中所描述的BLAST算法。特别有用的BLAST程序是WU-BLAST-2程序,其获自Altschul等人,Methods in Enzymology,266,460-480(1996);bl ast.wustl/edu/blast/README.html.。WU-BLAST-2使用几个搜索参数,其任选地设置为默认值。参数是动态值,并且由程序本身根据特定序列的组成和正在搜索的目标序列所针对的特定数据库的组成来建立;然而,可以调整值以增加灵敏度。进一步地,另外的有用的算法是gapped BLA ST,如Altschul等人,(1997)Nucleic Acids Res.25,3389-3402所报道的。除非另有指示,否则在本文中使用可在如下因特网地址获得的算法确定同一性百分比:blast.ncbi.nlm.nih.gov/Blast.cgi。
术语“内部核糖体进入位点”、“内部核糖体进入位点序列”、“IRES”和“IRES序列区”在本文中可互换地使用,并且是指病毒或人细胞RNA(例如,信使RNA(mRNA)和/或circRNA)的绕过经典真核帽依赖性翻译起始步骤的顺式元件。绝大多数真核mRNA使用的经典帽依赖性机制需要在mRNA的5'端的m
术语“IRES样序列”或“内部核糖体进入位点样序列”是指显示天然IRES功能的合成核苷酸序列。在一些实施方案中,IRES样序列可以募集核糖体组分以介导帽非依赖性翻译。
当指代核酸序列时,术语“编码序列”、“编码序列区”、“编码区”和“CDS”在本文中可以可互换地使用,以指代例如被或可以被翻译成蛋白质的DNA或RNA序列部分。术语“阅读框”、“开放阅读框”和“ORF”在本文中可以可互换地使用,以指代以起始密码子(例如,ATG)开始且在一些实施方案中以终止密码子(例如,TAA、TAG或TGA)结束的核苷酸序列。开放阅读框可以包含内含子和外显子,因此,所有CDS均是ORF,但是并非所有ORF均是CDS。
术语“互补的”和“互补性”是指两个核酸序列或核酸单体之间的关系,所述核酸序列或核酸单体具有通过传统的沃森-克里克碱基配对或其他非传统类型的配对彼此形成一个或多个氢键的能力。两个核酸序列之间的互补性程度可以通过核酸序列中可以与第二核酸序列形成氢键(例如,沃森-克里克碱基配对)(例如,约50%、约60%、约70%、约80%、约90%和100%互补)的核苷酸的百分比来指示。如果核酸序列的所有连续核苷酸均将与第二核酸序列中相同数量的连续核苷酸形成氢键,则这两个核酸序列是“完全互补的”。如果在至少8个核苷酸(例如,至少9、至少10、至少11、至少12、至少13、至少14、至少15、至少16、至少17、至少18、至少19、至少20、至少21、至少22、至少23、至少24、至少25、至少30、至少35、至少40、至少45、至少50个或更多个核苷酸)的区域上两个核酸序列之间的互补性程度是至少60%(例如,至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少97%、至少98%、至少99%或100%),或者如果两个核酸序列在至少中等或在一些实施方案中高严格性条件下杂交,则这两个核酸序列是“基本上互补的”。示例性中等严格性条件包括在包含20%甲酰胺、5%SSC(150mM NaCl、15mM柠檬酸三钠)、50mM磷酸钠(pH 7.6)、5xDenhardt溶液、10%硫酸葡聚糖和20mg/ml变性剪切的鲑鱼精DNA的溶液中在37℃下过夜孵育,然后将过滤器在1*SSC中在约37℃-50℃下洗涤,或基本上相似的条件,例如Sambrook,J.,Molecular Cloning:A Laboratory Manual,Cold Spring Harbor Laboratory Press;第4版(2012年6月15日)中所描述的中等严格条件。高严格性条件是这样的条件,其使用例如(1)低离子强度和高温用于洗涤,如在50℃下的0.015M氯化钠/0.0015M柠檬酸钠/0.1%十二烷基硫酸钠(SDS);(2)在杂交期间在42℃下采用变性剂,如甲酰胺,例如具有0.1%牛血清白蛋白(BSA)/0.1%Ficoll/0.1%聚乙烯吡咯烷酮(PVP)/含750mM氯化钠和75mM柠檬酸钠的pH 6.5的50mM磷酸钠缓冲液的50%(v/v)甲酰胺;或者(3)在42℃下采用50%甲酰胺、5xSSC(0.75M NaCl、0.075M柠檬酸钠)、50mM磷酸钠(pH 6.8)、0.1%焦磷酸钠、5xDenhardt溶液、超声处理的鲑鱼精DNA(50pg/ml)、0.1%SDS和10%硫酸葡聚糖,并且(i)在42℃下在0.2*SSC中、(ii)在55℃下在50%甲酰胺中以及(iii)在55℃下在0.1*SSC(任选地与EDTA组合)中洗涤。在例如Sambrook,同上和Ausubel等人编辑,Short Protocols inMolecular Biology,第5版,John Wiley&Sons,Inc.,新泽西州霍博肯(2002)中提供了杂交反应严格性的另外的细节和解释。
当指代核酸序列时,术语“杂交”或“杂交的”是在具有互补性的序列之间(between和/或among)形成的缔合。
术语“控制元件”统指启动子区、多腺苷酸化信号、转录终止序列、上游调控结构域、复制起点、内部核糖体进入位点(IRES)、增强子、剪接接头等,它们共同提供编码序列在受体细胞中的复制、转录、转录后加工和翻译。只要所选择的编码序列能够在适当的宿主细胞中复制、转录和翻译,并非所有这些控制元件均需要存在。
术语“启动子”在本文中用于指代包含DNA调控序列的核苷酸区域,其中调控序列源自能够与RNA聚合酶结合并且允许启动下游(3'方向)编码序列的转录的基因。它可以包含调控蛋白和分子可以结合的遗传元件,如RNA聚合酶和其他转录因子,以启动核酸序列的特异性转录。短语“可操作性地定位”、“可操作性地连接”、“在控制之下”和“在转录控制之下”意指启动子相对于核酸序列而言处于正确的功能位置和/或取向,以控制该序列的转录起始和/或表达。
“增强子”意指当接近启动子定位时,相对于在不存在增强子结构域的情况下由启动子产生的转录活性,赋予增加的转录活性的核酸序列。
关于核酸分子的“可操作地连接”意指两个或更多个核酸分子(例如,待转录的核酸分子、启动子和功能性效应子元件)以允许核酸分子转录的方式连接。
术语“同源性”是指两种多核苷酸的核酸残基或两种多肽的氨基酸残基之间的同一性百分比。一个序列与另一个序列之间的对应关系可以通过本领域已知的技术来确定。例如,可以通过比对序列信息并且使用容易获得的计算机程序来直接比较两种多肽之间的序列信息来确定同源性。如使用上述方法确定的,当至少约80%、优选至少约90%、最优选至少约95%的核苷酸或氨基酸在限定长度的分子上分别匹配时,两个多核苷酸(例如,DNA)或两个多肽序列彼此“基本上同源”。
术语“疤痕”是指环形产物中不包括目标序列的一定长度的区域。无痕cirRNA包含0个核苷酸的疤痕序列。近无痕cirRNA包含长度等于或小于20个核苷酸的疤痕序列。
“治疗”或“疾病或病症的治疗”是指执行方案或治疗计划,其可以包括向患者施用一种或多种药物或活性剂,以努力缓解疾病的体征或症状或者疾病的复发。令人希望的治疗效果包括降低疾病进展速率、改善或缓和疾病状态、以及消退、增加的存活期、改进的生活质量或预后的改进。缓解或预防可以在疾病或病症的体征或症状出现之前以及出现之后发生。此外,“治疗(treating)”或“治疗(treatment)”不需要完全缓解体征或症状,并且不需要治愈。
如贯穿本申请使用的术语“治疗益处”或“治疗有效的”是指关于此病症的医学治疗,促进或增强受试者的健康的任何事物。这包括但不限于疾病的体征或症状的频率、严重程度或进展速度的降低。例如,癌症的治疗可以涉及例如肿瘤大小的减小、肿瘤侵袭性的降低、癌症生长速率的降低或者转移或复发率的降低。癌症的治疗还可以指代延长患有癌症的受试者的存活期。
短语“药学上或药理学上可接受的”是指当施用至动物(如人)时分子实体和组合物不会产生不利、过敏或其他不良反应,视情况而定。对于动物(例如,人)施用,将理解制剂应当满足如例如FDA的生物标准局(Office of Biological Standards)所要求的无菌度、产热原性、一般安全性和纯度标准。
如本文所用,“药学上可接受的载体”包括任何和所有水性生物相容性溶剂(例如,盐水溶液,磷酸盐缓冲盐水,肠胃外媒介物如氯化钠,林格氏右旋糖等)、抗氧化剂、防腐剂(例如,抗细菌剂或抗真菌剂、抗氧化剂、螯合剂和惰性气体)、等渗剂、这样的类似材料及其组合,如本领域普通技术人员已知的。根据熟知的参数调整药物组合物中各种组分的pH和精确浓度。
如本文所用并且除非另有说明,否则术语“约”意指在给定值或范围的±10%内。在某些实施方案中,术语“约”涵盖所列举的确切数字。
6.2.核酶和II型内含子
核酶本身是一段RNA核酸分子。由于这样的核酸序列具有酶活性,因此它们被称为核酶。如一些线粒体或者细菌中的某些内含子序列,可以不依赖于剪接体直接催化剪接的发生,将它们称为“具有自剪接活性的核酶”、“自剪接核酶”或“自剪接内含子”。无需任何蛋白质即可完成剪接的自剪接内含子包括I型和II型两种。如上文所述,两种内含子在结构上和自剪接反应机理上均有明显区别。参见图10。本发明具体涉及II型自剪接内含子,或简称为“II型内含子”。
使用自剪接核酶制备环形RNA的方法具有以下优势:
1)减少生物和化学试剂使用。核酶的使用可以有效减少制备过程中外源生物制品(如连接酶)的污染以及其他化学试剂的污染。在用核酶进行催化自剪接时,反应体系中只需要使用少数几种试剂,如Tris-HCl缓冲液、Mg离子、钠离子和GTP。在本发明的情况下,由于使用了II型内含子,GTP也可以被省略。相比之下,使用连接酶进行连接反应制备环形RNA时,除了连接酶自身外,一方面酶的保存需要用到相应的化学试剂,例如Tris-HCl缓冲液、KCl、DTT、EDTA、甘油等,另一方面反应体系也需要化学试剂参与,如Mg离子、DTT、ATP等。试剂种类的减少可节约成本、简化操作。
2)操作简便。如上所述,由于反应所需的试剂种类少,只需要在RNA中加入含有GTP(仅就I型自剪接内含子而言)和离子的缓冲液,即可在PCR仪上一步完成环化反应。相比之下,使用RNA连接酶进行连接反应时,至少要加入额外的连接酶。
3)设计简便。对于分子量较大的环形RNA,例如含有编码序列的环形RNA,直接连接的效率很低,通常还需要引入外源的夹板DNA(DNA splint),这需要RNA和DNA的精确配对,增加了设计和操作的复杂性。
图9中展示了II型内含子的二级结构示意图。如图9所示,II型内含子主要包括6个茎环结构,称为结构域1-6(D1-D6),6个结构域顺序排列,其中含有多个外显子结合序列(EBS),如EBS1、EBS2、EBS3。这些EBS序列与外显子区域中的内含子结合序列(IBS)进行相互作用如互补配对,依靠EBS核酸序列内部自身的羟基触发剪接。这种剪接机制更接近通过剪接体介导的剪接反应,更类似于高等生物的剪接。
在优选的实施方案中,II型内含子源自微生物界(细菌域)。在具体的实施方案中,II型内含子源自梭菌属如破伤风梭菌或芽孢杆菌属如苏云金芽孢杆菌。本领域技术人员能够理解,本发明的关键在于构建体和方法的设计,这种设计适用于各种II型内含子。本发明的实施不局限于具体的II型内含子类型,只要该II型内含子在体外具有自剪接环化活性即可,这种活性是本领域技术人员通过常规手段能够确认的。
在本发明的一些实施方案中,II型内含子可以是野生型II型内含子或经修饰的II型内含子。所述经修饰的II型内含子包含一个或多个核苷酸的取代、缺失和/或添加。优选所述修饰不影响II型内含子的自剪接活性,特别是体外自剪接活性。
6.3.本发明的构建体
在本发明的上下文中,可以将天然自剪接核酶称为自剪接核酶或cRNAzyme前体,将重排并改造后的自剪接核酶称为cRNAzyme。进一步地,将与目标序列,如蛋白编码序列或蛋白非编码序列,连接起来的cRNAzyme称为cRNAzyme构建体,即本发明的多核苷酸构建体。
具体来说,通过将由天然II型内含子及其两个侧翼的外显子片段(E1、E2)组成的一段序列(E1-内含子-E2)一分为二,形成两个片段,即结构为E1-5’内含子片段的第一片段,和结构为3’内含子片段-E2的第二片段。其中5’内含子片段原先位于3’内含子片段的5’端,并且彼此紧邻。在构建cRNAzyme时,将所述第一片段和第二片段位置互换并重新连接。重排后的序列结构为“3’内含子片段-E2-E1-5’内含子片段”。将具有这种结构并且具有自剪接活性的序列称为cRNAzyme。所述自剪接活性优选是发生自剪接并使插入其中的目标蛋白序列形成环形RNA的活性。所述自剪接活性优选是在体外发生自剪接的活性。
在将cRNAzyme用于催化目标蛋白形成环形RNA时,将目标蛋白序列,包括目标蛋白编码序列和/或非编码序列,构建到cRNAzyme的E2和E1之间的位置,由此形成cRNAzyme构建体。cRNAzyme构建体可以通过转录成为RNA,然后通过其中含有的cRNAzyme结构元件发生自剪接,使其中含有的目标蛋白序列形成环形RNA。
总体而言,基于II型内含子(cRNAzyme前体)设计cRNAzyme构建体的原则是在整体长度尽量短的情况下保留最大的环化效率。发生自剪接环化反应后,内含子部分会被切出,如图1所示。获得的环形RNA产物中不再包含内含子部分。因此环形RNA产物要比未发生剪接反应的线性cRNAzyme构建体结构的总核苷酸数少。基于这点,可以通过琼脂糖凝胶电泳来区分环形RNA产物和cRNAzyme构建体。在本发明的上下文中,将环化效率(PC)定义为环形RNA占线性RNA和环形RNA总和的百分比。具体的定量方式采用本领域常用的半定量方式,根据凝胶电泳图中条带的强度来确定。
基于上述原则,E1和/或E2的长度优选不超过20个核苷酸,例如不超过10个核苷酸,如1、2、3、4、5、6、7、8、9或10个核苷酸。在特殊的实施方案中,E1和E2可为0。
同样基于上述原则,cRNAzyme构建体中的内含子序列,如5’内含子片段和/或3’内含子片段,和/或外显子序列,如E1和/或E2,相对于其天然存在的野生型序列,可以包含一个或多个核苷酸的修饰,例如一个或多个核苷酸的添加、删除、取代。
茎环结构是一种类型的RNA二级结构,其可以通过任何合适的多核苷酸折叠算法来确定。一些程序是基于最小吉布斯自由能的计算。一种这样的算法的一个例子是mFold,并且由Zuker和Stiegler(Nucleic Acids Res.9(1981),133-148)描述。另一种示例性折叠算法是由维也纳大学理论化学研究所使用质心结构预测算法(例如,AR Gruber等人,2008,Cell 106).(1):23-24;以及PA Carr和GM Church,2009,Nature Biotechnology 27(12):1151-62)开发的在线网络服务器RNAfold。另外的算法可以在美国临时专利申请号61/836,080(代理人案卷号44790.11.2022;泛参考编号BI-2013/004A)中找到,将其通过引用并入本文。
在一个实施方案中,为了使序列缩短,可以在不影响活性的情况下,删除一部分序列或核苷酸。例如,可以删除II型内含子结构域4中的内含子编码蛋白(IEP)序列。结构域4中的IEP序列或类似的结构存在于全部II型内含子中,其编码具有逆转录酶活性的蛋白质,该蛋白质可催化内含子充当逆转录因子,并通过RNA中间体在其基因组中移动。这一功能是天然II型内含子在基因组中进行逆转录转座所需的,但是在体外转录中不需要此功能,因此在本发明的构建体中可以删除结构域4中的该段序列的部分或全部。
E1和E2通常需要包括IBS序列,从而与内含子中所含的EBS序列进行相互作用以实现自剪接。在本发明的一个实施方案中,E1和E2序列可以为0,这样的好处是在最终环化的RNA中除了目标序列不再包含任何其他序列。在这种情况下,为了保证内含子中的EBS仍然能够有与之配对的“IBS”序列,需要对内含子的EBS序列进行修饰,使其与目标序列中的一段序列互补配对,从而发生相互作用。换言之,将目标序列中的一段序列视作“IBS”,与内含子中经过修饰的EBS序列发生相互作用,以确保自剪接的完成。
因此,在本发明的一个实施方案中,所述II型内含子是经修饰的II型内含子,具体来说是EBS区经修饰的II型内含子。所述修饰可以是一个或多个核苷酸的取代,具体来说取代一个或多个EBS区的核苷酸,从而使得经修饰的EBS区与目标序列中的一段相应长度的区域互补配对。表述“互补配对”意指两段序列在转录成RNA之后,能够互补配对,所述配对涵盖RNA中的G和U的配对方式。所述经修饰的EBS的长度可为3-20个核苷酸,优选5-15个核苷酸,更优选6-10个核苷酸,例如6个、7个、8个、9个或10个核苷酸。
与修饰的EBS互补配对的目标序列区域可以存在于目标序列的任意位置,只要能够实现与EBS配对,并由此形成能够促成自剪接的二级结构即可。通常而言,可以使用目标序列两端的序列作为修饰的EBS设计的基础,这是因为两端的序列在构建体中位于原先E1和E2所处的位置,而E1和E2的位置也是原先与EBS进行相互作用的IBS序列的位置。因此,在具体的实施方案中,所述经修饰的EBS区是经修饰的EBS1和EBS3区。在具体的实施方案中,与经修饰的EBS区互补配对的目标序列中的区域位于目标序列的3’和/或5’端。
由于这种互补配对的目的是为了确保EBS和充当IBS的目标序列片段之间的相互作用,因此只要相互作用存在,可以允许一定程度的错配。在一些实施方案中,所述经修饰的EBS区与目标序列中的一段相应长度的区域在至少60%,例如至少70%、至少80%、至少90%、至少95%或100%的核苷酸位置上互补配对,或与目标序列中的一段相应长度的区域的互补配对序列具有至少60%的同一性,例如至少70%、至少80%、至少90%、至少95%或100%的同一性。
在另一个实施方案中,所述5’内含子片段和3’内含子片段中可以包含一对或多对彼此互补的配对序列。这样的配对序列使5’内含子片段和3’内含子片段在空间上的距离缩短,从而促进环化反应发生。在优选的实施方案中,所述互补配对序列的长度至少为约20个核苷酸。
构建体中的目标序列可以包含任意希望制备成环形RNA的序列。目标序列可以是蛋白编码序列,或蛋白非编码序列,或二者的组合。换言之,目标序列中可以包含多种元件。蛋白编码序列可以编码任意蛋白,例如选自功能性蛋白、抗原蛋白、信号肽、标签蛋白等。
例如,目标序列中包括的蛋白非编码序列可以是间隔序列,例如富含AT的序列,其可以调节序列的柔性。这种间隔序列可以位于目标序列中的任意位置,例如位于目标序列的一端,紧邻E1和/或E2的位置。
例如,目标序列中包括的蛋白非编码序列可以是翻译调控序列,如内部核糖体进入位点(IRES)。可用于本发明的IRES可以来自任何来源。
本发明的cRNAzyme和cRNAzyme构建体以DNA的形式完整制备,然后通过转录和自剪接形成期望的环形RNA。
6.4.自剪接反应体系
II型内含子的自剪接需要在高盐度条件下完成,并且与I型内含子相比,不需要引入GTP。
在本发明的具体实施方案中,自剪接反应使用的自剪接缓冲液含有10mM-100mM,如10mM、20mM、30mM、40mM、50mM、60mM、70mM、80mM、90mM、100mM的二价镁离子,如MgCl2。所述自剪接缓冲液可以含有10mM-100mM,如10mM、20mM、30mM、40mM、50mM、60mM、70mM、80mM、90mM、100mM的NaCl。
在优选的实施方案中,本发明的自剪接反应在体外进行约5min至约1h,如约5min、约10min、约15min、约20min、约25min、约30min、约35min、约40min、约45min、约50min、约55min、约1h。
在优选的实施方案中,本发明的构建体能够实现至少30%的环化率,如至少50%、至少60%、至少70%、至少80%、至少90%、至少95%的环化率。
6.5.目标序列
在一些实施方案中,所述目标序列为空。在一些实施方案中,所述目标序列为蛋白编码序列。在一些实施方案中,所述目标序列为非编码序列。
在一些实施方案中,所述目标序列编码治疗产物。
在具体的实施方案中,所述治疗产物是多肽、蛋白质、酶或抗体。在具体的实施方案中,所述治疗产物包含一种或多种多肽、蛋白质、酶、抗体或其组合。
在具体的实施方案中,所述蛋白质或酶与具有病理表现的疾病相关,所述病理表现可追溯到遗传改变和/或蛋白质失调。
在具体的实施方案中,所述多肽或蛋白质类似于弱化或死亡形式的致病剂,其可以是微生物,如细菌、病毒、真菌、寄生生物,或这种微生物的一种或多种毒素和/或一种或多种蛋白质(例如,表面蛋白(即,抗原))。在具体的实施方案中,所述治疗产物是这样的抗原或药剂,其可以刺激机体的免疫系统以将所述药剂识别为外来入侵者,生成针对所述药剂的抗体,破坏所述药剂并且产生对所述药剂的记忆。在具体的实施方案中,所述治疗产物是这样的抗原或药剂,其可以诱导疫苗诱导的记忆和/或使免疫系统能够迅速起作用,以保护机体免于以后遇到这些药剂中的任一种。
在一些实施方案中,所述治疗产物源自感染原。在一些实施方案中,所述感染原选自下组的成员:病毒株和细菌株。
在本文提供的任何实施方案中,所述感染原是选自下组的病毒株:腺病毒;单纯疱疹病毒1型;单纯疱疹病毒2型;脑炎病毒、乳头瘤病毒、水痘-带状疱疹病毒;EB病毒;人巨细胞病毒;人疱疹病毒8型;人乳头瘤病毒;BK病毒;JC病毒;天花病毒;脊髓灰质炎病毒(poliovirus);乙型肝炎病毒;人博卡病毒;细小病毒B19;人星状病毒;诺沃克病毒;柯萨奇病毒;甲型肝炎病毒;脊髓灰质炎病毒(poliovirus);鼻病毒;严重急性呼吸综合征病毒;丙型肝炎病毒;黄热病毒;登革病毒;西尼罗病毒;风疹病毒;戊型肝炎病毒;人类免疫缺陷病毒(HIV);流感病毒;瓜纳瑞托病毒;胡宁病毒;拉沙病毒;马秋波病毒;萨比亚病毒;克里米亚-刚果出血热病毒;埃博拉病毒;马尔堡病毒;麻疹病毒;腮腺炎病毒;副流感病毒;呼吸道合胞病毒(RSV);人偏肺病毒;亨德拉病毒;尼帕病毒;狂犬病病毒;丁型肝炎病毒;轮状病毒;环状病毒;科罗拉多壁虱热病毒;版纳病毒;人肠道病毒;汉坦病毒;西尼罗病毒;冠状病毒、严重急性呼吸综合征(SARS)相关冠状病毒(SARS-CoV)、SARS-CoV-2病毒(COVID-19相关);中东呼吸综合征冠状病毒;日本脑炎病毒;水疱疹病毒;东方马脑炎病毒;和流感病毒。在一些实施方案中,所述感染原是选自以下的细菌株:结核(结核分枝杆菌(Mycobacteriumtuberculosis))、耐克林霉素的艰难梭状芽孢杆菌(Cl ostridium difficile)、耐氟喹诺酮的艰难梭状芽孢杆菌、耐甲氧西林的金黄色葡萄球菌(Staphylococcus aureus)(MRSA)、耐多药的粪肠球菌(Enterococcus faecalis)、耐多药的屎肠球菌(Enterococcusfaecium)、耐多药的铜绿假单胞杆菌(Pseudomonas aeruginosa)、耐多药的鲍曼不动杆菌(Acinetobacter baumannii)和耐万古霉素的金黄色葡萄球菌(VR SA)。
在一些实施方案中,所述感染原与鸟、猪、马、狗、人或非人灵长类动物相关。
在一些实施方案中,所述抗体包括但不限于单克隆抗体、多克隆抗体、重组产生的抗体、人抗体、人源化抗体、嵌合抗体、合成抗体、包含两个重链和两个轻链分子的四聚体抗体、抗体轻链单体、抗体重链单体、抗体轻链二聚体、抗体重链、抗体重链二聚体、抗体轻链-重链对、胞内抗体、杂缀合抗体、单价抗体、全长抗体的抗原结合片段以及上述的融合蛋白。这样的抗原结合片段包括但不限于单结构域抗体(重链抗体(VHH)或纳米抗体的可变结构域)、Fab、F(ab’)2和scFv(单链可变片段)。
在具体的实施方案中,本文公开的核酸(例如,多核苷酸)和核酸序列可以例如经由本领域技术人员已知的任何密码子优化技术(参见例如,Quax等人,2015,Mol Cell 59:149-161的综述)进行密码子优化。
在一些实施方案中,所述目标序列编码适配体序列。在一些实施方案中,所述目标序列编码可以与特定目标(包括蛋白质、肽、碳水化合物、小分子、毒素、甚至活细胞)选择性地结合的单链DNA或RNA(ssDNA或ssRNA)分子。
在一些实施方案中,所述目标序列编码核酶,其是可以催化化学反应的核糖核酸(RNA)酶。
在一些实施方案中,所述目标序列编码反义寡核苷酸(ASO),其与目标RNA序列特异性地结合并且通过几种不同的机制调节蛋白质表达。
在一些实施方案中,所述目标序列编码Decoy,其是与内源性目标中的miRNA结合位点或蛋白结合位点相同或具有同源性的一段短序列。
在一些实施方案中,所述目标序列编码RNA支架,其是经设计以在体内通过支架的蛋白对接结构域与其亲和蛋白-酶融合体之间的相互作用共定位改造过的生物途径中的酶的RNA序列。
6.6.载体、线性RNA、前体RNA和环形RNA
在一些实施方案中,本文提供的RNA多核苷酸是单链RNA。在一些实施方案中,所述多核苷酸是线性RNA。在一些实施方案中,本文提供了前体RNA。在一些实施方案中,所提供的RNA多核苷酸由载体编码。在一些实施方案中,所述前体RNA是通过本文提供的载体的体外转录产生的线性RNA。
在一些实施方案中,所述RNA多核苷酸是环形RNA或可用于制造环形RNA多核苷酸。在一些实施方案中,本文提供了环形RNA。在一些实施方案中,所述环形RNA是由本文提供的载体产生的环形RNA。在一些实施方案中,所述环形RNA是通过本文提供的前体RNA的环化产生的环形RNA。
环形RNA
环形RNA(也称为“circRNA”或“cRNA”)是头尾相连的单链RNA。circRNA一直被认为是真核细胞中普遍存在的一类非编码RNA。发现通常通过反向剪接生成的circRNA非常稳定。
在一些实施方案中,夹板连接可用于生成环形RNA。夹板连接涉及使用寡核苷酸夹板,其与线性RNA的两端杂交以将线性RNA的末端聚在一起进行连接。夹板(其可以是脱氧核糖寡核苷酸或核糖寡核苷酸)的杂交定向RNA末端的5-磷酸和3-OH进行连接。如上所述,可以使用化学或酶促技术进行后续连接。可以例如用T4 DNA连接酶(需要DNA夹板)、T4 RNA连接酶1(需要RNA夹板)或T4 RNA连接酶2(DNA或RNA夹板)进行酶促连接。如果杂交的夹板-RNA复合物的结构干扰酶活性,则化学连接(如用BrCN或EDC)在一些情况下比酶促连接更有效(参见例如,Dolinnaya等人Nucleic Acids Res,2/(23):5403-5407(1993);Petkovic等人,Nucleic Acids Res,43(4):2454-2465(2015))。
在一些实施方案中,所述RNA多核苷酸(例如,环形RNA)可以具有任何长度或大小。在一些实施方案中,所述RNA多核苷酸的长度在300与10000、400与9000、500与8000、600与7000、700与6000、800与5000、900与5000、1000与5000、1100与5000、1200与5000、1300与5000、1400与5000和/或1500与5000个核苷酸之间。
在一些实施方案中,所述RNA多核苷酸(例如,环形RNA)的长度为至少300nt、400nt、500nt、600nt、700nt、800nt、900nt、1000nt、1100nt、1200nt、1300nt、1400nt、1500nt、2000nt、2500nt、3000nt、3500nt、4000nt、4500nt或5000nt。在一些实施方案中,所述RNA多核苷酸的长度不超过3000nt、3500nt、4000nt、4500nt、5000nt、6000nt、7000nt、8000nt、9000nt或10000nt。
在一些实施方案中,所述RNA多核苷酸(例如,环形RNA)的长度为约300nt、400nt、500nt、600nt、700nt、800nt、900nt、1000nt、1100nt、1200nt、1300nt、1400nt、1500nt、2000nt、2500nt、3000nt、3500nt、4000nt、4500nt、5000nt、6000nt、7000nt、8000nt、9000nt或10000nt。
在一些实施方案中,所述RNA多核苷酸(例如,环形RNA)的长度为至少500、600、700、800、900、1000、1500、2000、2500、3000、3500、4000、4500、5000、5500、6000、6500、7000、7500、8000、8500、9000、9500或10000nt。所述RNA多核苷酸(例如,环形RNA)可以是未经修饰的、经部分修饰的或经完全修饰的。
在一些实施方案中,本文提供的环形RNA比包含相同的表达序列的mRNA具有更高的功能稳定性。在一些实施方案中,本文提供的环形RNA比包含相同的表达序列、5moU修饰、经优化的UTR、帽和/或polyA尾的mRNA具有更高的功能稳定性。
在一些实施方案中,本文提供的环形RNA多核苷酸具有至少5小时、10小时、15小时、20小时、30小时、40小时、50小时、60小时、70小时或80小时的功能半衰期。在一些实施方案中,本文提供的环形RNA多核苷酸具有5-80、10-70、15-60和/或20-50小时的功能半衰期。在一些实施方案中,本文提供的环形RNA多核苷酸的功能半衰期大于编码相同蛋白质的等同的线性RNA多核苷酸的功能半衰期(例如,是至少1.5倍、是至少2倍)。在一些实施方案中,可以通过检测功能性蛋白合成来评估功能半衰期。
在一些实施方案中,本文提供的环形RNA多核苷酸具有至少5小时、10小时、15小时、20小时、30小时、40小时、50小时、60小时、70小时或80小时的半衰期。在一些实施方案中,本文提供的环形RNA多核苷酸具有5-80、10-70、15-60和/或20-50小时的半衰期。在一些实施方案中,本文提供的环形RNA多核苷酸的半衰期大于编码相同蛋白质的等同的线性RNA多核苷酸的半衰期(例如,是至少1.5倍、是至少2倍)。
在一些实施方案中,本文提供的环形RNA可以具有比等同的线性mRNA更高的表达幅度,例如在将RNA施用至细胞后24小时具有更高的表达幅度。在一些实施方案中,本文提供的环形RNA具有比包含相同的表达序列、5moU修饰、经优化的UTR、帽和/或polyA尾的mRNA更高的表达幅度。在一些实施方案中,本文提供的环形RNA可以具有比等同的线性mRNA更高的稳定性。在一些实施方案中,这可以通过电穿孔后经1周测量的时间点在体外或体内测量受体的存在和密度来显示。在一些实施方案中,这可以通过经由qPCR或ISH测量RNA的存在来显示。
在一些实施方案中,本文提供的环形RNA多核苷酸包含经修饰的RNA核苷酸和/或经修饰的核苷。在一些实施方案中,所述经修饰的核苷是m
在一些实施方案中,所述经修饰的核苷可以包括选自下组的化合物:吡啶-4-酮核糖核苷、5-氮杂-尿苷、2-硫代-5-氮杂-尿苷、2-硫代尿苷、4-硫代-假尿苷、2-硫代-假尿苷、5-羟基尿苷、3-甲基尿苷、5-羧基甲基-尿苷、1-羧基甲基-假尿苷、5-丙炔基-尿苷、1-丙炔基-假尿苷、5-牛磺酸甲基尿苷、1-牛磺酸甲基-假尿苷、5-牛磺酸甲基-2-硫代-尿苷、l-牛磺酸甲基-4-硫代-尿苷、5-甲基-尿苷、1-甲基-假尿苷、4-硫代-1-甲基-假尿苷、2-硫代-1-甲基-假尿苷、1-甲基-1-去氮-假尿苷、2-硫代-1-甲基-1-去氮-假尿苷、二氢尿苷、二氢假尿苷、2-硫代-二氢尿苷、2-硫代-二氢假尿苷、2-甲氧基尿苷、2-甲氧基-4-硫代-尿苷、4-甲氧基-假尿苷、4-甲氧基-2-硫代-假尿苷、5-氮杂-胞苷、假异胞苷、3-甲基-胞苷、N4-乙酰基胞苷、5-甲酰基胞苷、N4-甲基胞苷、5-羟基甲基胞苷、1-甲基-假异胞苷、吡咯并-胞苷、吡咯并-假异胞苷、2-硫代-胞苷、2-硫代-5-甲基-胞苷、4-硫代-假异胞苷、4-硫代-1-甲基-假异胞苷、4-硫代-1-甲基-1-去氮-假异胞苷、1-甲基-1-去氮-假异胞苷、泽布拉林、5-氮杂-泽布拉林、5-甲基-泽布拉林、5-氮杂-2-硫代-泽布拉林、2-硫代-泽布拉林、2-甲氧基-胞苷、2-甲氧基-5-甲基-胞苷、4-甲氧基-假异胞苷、4-甲氧基-1-甲基-假异胞苷、2-氨基嘌呤、2,6-二氨基嘌呤、7-去氮-腺嘌呤、7-去氮-8-氮杂-腺嘌呤、7-去氮-2-氨基嘌呤、7-去氮-8-氮杂-2-氨基嘌呤、7-去氮-2,6-二氨基嘌呤、7-去氮-8-氮杂-2,6-二氨基嘌呤、1-甲基腺苷、N6-甲基腺苷、N6-异戊烯基腺苷、N6-(顺式-羟基异戊烯基)腺苷、2-甲基硫代-N6-(顺式-羟基异戊烯基)腺苷、N6-甘氨酰基氨基甲酰基腺苷、N6-苏氨酰基氨基甲酰基腺苷、2-甲基硫代-N6-苏氨酰基氨基甲酰基腺苷、N6,N6-二甲基腺苷、7-甲基腺嘌呤、2-甲基硫代-腺嘌呤、2-甲氧基-腺嘌呤、肌苷、1-甲基-肌苷、怀俄苷、怀丁苷、7-去氮-鸟苷、7-去氮-8-氮杂-鸟苷、6-硫代-鸟苷、6-硫代-7-去氮-鸟苷、6-硫代-7-去氮-8-氮杂-鸟苷、7-甲基-鸟苷、6-硫代-7-甲基-鸟苷、7-甲基肌苷、6-甲氧基-鸟苷、1-甲基鸟苷、N2-甲基鸟苷、N2,N2-二甲基鸟苷、8-氧代-鸟苷、7-甲基-8-氧代-鸟苷、1-甲基-6-硫代-鸟苷、N2-甲基-6-硫代-鸟苷和N2,N2-二甲基-6-硫代-鸟苷。在另一个实施方案中,所述修饰独立地选自下组:5-甲基胞嘧啶、假尿苷和1-甲基假尿苷。
在一些实施方案中,可以对多核苷酸进行密码子优化。经密码子优化的序列可以是编码治疗产物的多核苷酸中的密码子已经被取代以增加治疗产物的表达、稳定性和/或活性的序列。影响密码子优化的因素包括但不限于以下中的一种或多种:(i)两种或更多种生物体或基因之间的密码子偏倚或合成构建的偏倚表的变化;(ii)生物体、基因或基因集内的密码子偏倚程度的变化;(iii)包括背景在内的密码子的系统变化;(iv)根据其解码tRNA的密码子的变化;(v)根据总体上或在三联体的一个位置中的GC%的密码子的变化;(vi)与参考序列(例如天然存在的序列)的相似性程度的变化;(vii)密码子频率截止的变化;(viii)从DNA序列转录的mRNA的结构特性;(ix)关于密码子取代集的设计所基于的DNA序列的功能的先前知识;和/或(x)每种氨基酸的密码子集的系统变化。在一些实施方案中,经密码子优化的多核苷酸可以最小化核酶碰撞和/或限制表达序列与IRES之间的结构干扰。
在一些实施方案中,一种具有自剪接活性的多核苷酸构建体,从5'到3'包含以下可操作地连接的元件:(a)3'内含子片段;(b)外显子片段2(E2);(c)目标序列;(d)外显子片段1(E1);(d)5'内含子片段。在一个实施方案中,所述5’内含子片段和3’内含子片段各自为II型内含子的片段。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,一种具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:(a)3’内含子片段;
(b)外显子片段2(E2);(c)接头序列;(d)目标序列;(e)接头序列;(f)外显子片段1(E1);(g)5’内含子片段。在一个实施方案中,所述5’内含子片段和3’内含子片段各自为II型内含子的片段。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,一种具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:(a)5’同源臂;(b)3’内含子片段;(c)外显子片段2(E2);(d)目标序列;(e)外显子片段1(E1);(f)5’内含子片段;(g)3’同源臂。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,一种具有自剪接活性的多核苷酸构建体,其从5’到3’包含以下可操作地连接的元件:(a)5’同源臂;(b)
3’内含子片段;(c)外显子片段2(E2);(d)接头序列;(e)目标序列;(f)接头序列;(g)外显子片段1(E1);(h)5’内含子片段;(i)3’同源臂。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,所述多核苷酸构建体在体外具有自剪接活性。
在一些实施方案中,所述E1和/或E2的长度为0-20个核苷酸。在优选的实施方案中,所述E1和/或E2的长度为0-10个核苷酸。在一个实施方案中,所述E1和/或E2的长度为0、1、2、3、4、5、6、7、8、9、10个核苷酸。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从非配对区,例如为II型内含子两个相邻结构域之间的线性区的非配对区,进行分割成两个片段获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域1茎环结构的环区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域2茎环结构的环区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域3茎环结构的环区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域4茎环结构的环区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域5茎环结构的环区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域6茎环结构的环区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域1与结构域2之间的线性区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域2与结构域3之间的线性区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域3与结构域4之间的线性区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域4与结构域5之间的线性区进行分割获得。
在一些实施方案中,所述5’内含子片段和3’内含子片段通过将II型内含子从结构域5与结构域6之间的线性区进行分割获得。
在一些实施方案中,所述II型内含子相对于其野生型形式包含一个或多个核苷酸的修饰,所述修饰选自删除、取代、添加中的一种或多种。
在一些实施方案中,所述修饰包括对所述II型内含子的一个或多个EBS序列进行修饰,其中所述EBS序列分别与目标序列中相应长度的一个或多个区域在至少60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、100%的核苷酸位置上互补配对。
在一些实施方案中,所述修饰为对所述II型内含子的两个EBS序列,如EBS1和EBS3,进行修饰,其中所述EBS序列分别与目标序列中相应长度的两个区域在至少60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、100%的核苷酸位置上互补配对;优选所述两个区域分别位于所述目标序列的两端。
在一些实施方案中,所述修饰为对所述II型内含子的两个EBS序列,如EBS1’和EBS3’,进行修饰,其中所述EBS序列分别与目标序列中相应长度的两个区域在至少60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、100%的核苷酸位置上互补配对;优选所述两个区域分别位于所述目标序列的两端。
在一些实施方案中,所述修饰为对所述II型内含子的EBS1和/或δ序列进行修饰或对EBS1’和/或δ”序列进行修饰,其中所述EBS1和/或δ序列与目标序列中相应长度的区域在至少60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、100%的核苷酸上互补配对,任选地所述修饰为对EBS1和/或δ序列及其上游序列进行修饰,其中所述EBS1和/或δ序列及其上游序列与目标序列中相应长度的区域在至少60%的核苷酸上互补配对。在一些实施方案中,所述目标序列中相应长度的区域为IBS3、IBS3'、具有下游序列的IBS3、具有下游序列的IBS3'。在一些实施方案中,所述δ序列及其上游包含选自下组的核酸序列:(a)SEQID NO:127、(b)SEQ ID NO:128、(c)SEQ ID NO:129、(d)SEQ ID NO:130。在一些实施方案中,所述IBS3及其下游包含选自下组的核酸序列:(a)SEQ ID NO:131、(b)SEQ ID NO:132、(c)SEQ ID NO:133、(d)SEQ ID NO:134。参见图6和16。
在一些实施方案中,所述修饰包括删除结构域4的部分或全部,例如删除结构域4中的内含子编码蛋白(IEP)序列,优选删除全部结构域4。
在一些实施方案中,所述修饰包括删除开放阅读框(ORF)。
在一些实施方案中,所述多核苷酸构建体能够形成所述目标序列的近无痕环形RNA。
在一些实施方案中,所述近无痕环形RNA具有长度等于或小于1个核苷酸、2个核苷酸、3个核苷酸、4个核苷酸、5个核苷酸、6个核苷酸、7个核苷酸、8个核苷酸、9个核苷酸、10个核苷酸、11个核苷酸、12个核苷酸、13个核苷酸、14个核苷酸、15个核苷酸、16个核苷酸、17个核苷酸、18个核苷酸、19个核苷酸、20个核苷酸的疤痕区。
在一些实施方案中,所述多核苷酸构建体能够形成所述目标序列的无痕环形RNA。
在一些实施方案中,E1和E2各自的长度为0个核苷酸。在一些实施方案中,E1的长度为0个核苷酸。在一些实施方案中,E2的长度为0个核苷酸。
在一些实施方案中,所述II型内含子是源自微生物(如破伤风梭菌或芽孢杆菌属如苏云金芽孢杆菌)的II型内含子。
在一些实施方案中,所述非编码序列选自下组:间隔序列SEQ ID NO:4-6、polyA序列、polyA-C序列、polyC序列、polyU序列、IRES、核糖体结合位点、适配体序列、RNA支架、核糖开关、除自剪接核酶之外的核酶、反义寡核苷酸(ASO)、支架、小RNA结合位点、翻译调控序列、蛋白结合位点。
在一些实施方案中,所述II型内含子包含选自下组的核酸序列:SEQ ID NO:33;SEQ ID NO:34;SEQ ID NO:35;SEQ ID NO:36;SEQ ID NO:37;SEQ ID NO:38;SEQ ID NO:39;SEQ ID NO:40;SEQ ID NO:41。
在一些实施方案中,所述II型内含子包含选自下组的核酸序列:与SEQ ID NO:33具有95%的同一性的核酸序列;与SEQ ID NO:34具有95%的同一性的核酸序列;与SEQ IDNO:35具有95%的同一性的核酸序列;与SEQ ID NO:36具有95%的同一性的核酸序列;与SEQ ID NO:37具有95%的同一性的核酸序列;与SEQ ID NO:38具有95%的同一性的核酸序列;与SEQ ID NO:39具有95%的同一性的核酸序列;与SEQ ID NO:40具有95%的同一性的核酸序列;与SEQ ID NO:41具有95%的同一性的核酸序列。
在一些实施方案中,所述II型内含子包含选自下组的核酸序列:与SEQ ID NO:33具有98%的同一性的核酸序列;与SEQ ID NO:34具有98%的同一性的核酸序列;与SEQ IDNO:35具有98%的同一性的核酸序列;与SEQ ID NO:36具有98%的同一性的核酸序列;与SEQ ID NO:37具有98%的同一性的核酸序列;与SEQ ID NO:38具有98%的同一性的核酸序列;与SEQ ID NO:39具有98%的同一性的核酸序列;与SEQ ID NO:40具有98%的同一性的核酸序列;与SEQ ID NO:41具有98%的同一性的核酸序列。
在一些实施方案中,所述II型内含子包含选自下组的核酸序列:与SEQ ID NO:33具有99%的同一性的核酸序列;与SEQ ID NO:34具有99%的同一性的核酸序列;与SEQ IDNO:35具有99%的同一性的核酸序列;与SEQ ID NO:36具有99%的同一性的核酸序列;与SEQ ID NO:37具有99%的同一性的核酸序列;与SEQ ID NO:38具有99%的同一性的核酸序列;与SEQ ID NO:39具有99%的同一性的核酸序列;与SEQ ID NO:40具有99%的同一性的核酸序列;与SEQ ID NO:41具有99%的同一性的核酸序列。
在一些实施方案中,所述II型内含子包含选自表16-24的核酸序列。
在一些实施方案中,所述II型内含子基本上由选自下组的核酸序列组成:SEQ IDNO:33-SEQ ID NO:41。
在一些实施方案中,所述II型内含子由选自下组的核酸序列组成:SEQ ID NO:33-SEQ ID NO:41。
在一些实施方案中,所述II型内含子由选自下组的核酸序列组成:SEQ ID NO:33-SEQ ID NO:41。
在一些实施方案中,所述多核苷酸构建体为RNA多核苷酸构建体。
在一些实施方案中,所述3’内含子片段包含选自下组的核酸序列:(a)与SEQ IDNO:42具有95%的同一性的核酸序列;(b)与SEQ ID NO:42具有98%的同一性的核酸序列;(c)与SEQ ID NO:42具有99%的同一性的核酸序列;(d)SEQ ID NO:42;(e)与SEQ ID NO:43具有95%的同一性的核酸序列;(f)与SEQ ID NO:43具有98%的同一性的核酸序列;(g)与SEQ ID NO:43具有99%的同一性的核酸序列;(h)SEQ ID NO:43;(i)与SEQ ID NO:44具有95%的同一性的核酸序列;(j)与SEQ ID NO:44具有98%的同一性的核酸序列;(k)与SEQID NO:44具有99%的同一性的核酸序列;(l)SEQ ID NO:44;(m)与SEQ ID NO:45具有95%的同一性的核酸序列;(n)与SEQ ID NO:45具有98%的同一性的核酸序列;(o)与SEQ IDNO:45具有99%的同一性的核酸序列;(p)SEQ ID NO:45;(q)与SEQ ID NO:46具有95%的同一性的核酸序列;(r)与SEQ ID NO:46具有98%的同一性的核酸序列;(s)与SEQ ID NO:46具有99%的同一性的核酸序列;(t)SEQ ID NO:46;(u)与SEQ ID NO:47具有95%的同一性的核酸序列;(v)与SEQ ID NO:47具有98%的同一性的核酸序列;(w)与SEQ ID NO:47具有99%的同一性的核酸序列;(x)SEQ ID NO:47;(y)与SEQ ID NO:48具有95%的同一性的核酸序列;(z)与SEQ ID NO:48具有98%的同一性的核酸序列;(aa)与SEQ ID NO:48具有99%的同一性的核酸序列;(bb)核酸序列SEQ ID NO:48;(cc)与SEQ ID NO:49具有95%的同一性的核酸序列;(dd)与SEQ ID NO:49具有98%的同一性的核酸序列;(ee)与SEQ ID NO:49具有99%的同一性的核酸序列;(ff)SEQ ID NO:49;(gg)与SEQ ID NO:50具有95%的同一性的核酸序列;(hh)与SEQ ID NO:50具有98%的同一性的核酸序列;(ii)与SEQ ID NO:50具有99%的同一性的核酸序列;(jj)SEQ ID NO:50;(kk)与SEQ ID NO:51具有95%的同一性的核酸序列;(ll)与SEQ ID NO:51具有98%的同一性的核酸序列;(mm)与SEQ ID NO:51具有99%的同一性的核酸序列;(nn)SEQ ID NO:51;(oo)与SEQ ID NO:52具有95%的同一性的核酸序列;(pp)与SEQ ID NO:52具有98%的同一性的核酸序列;(qq)与SEQ ID NO:52具有99%的同一性的核酸序列;(rr)SEQ ID NO:52。
在一些实施方案中,所述3’内含子片段基本上由选自下组的核酸序列组成:与SEQID NO:42-SEQ ID NO:52中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:42-SEQID NO:52中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:42-SEQ ID NO:52中的任一个具有99%的同一性的核酸序列、SEQ ID NO:42-SEQ ID NO:52中的任一个。
在一些实施方案中,所述3’内含子片段由选自下组的核酸序列组成:与SEQ IDNO:42-SEQ ID NO:52中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:42-SEQ IDNO:52中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:42-SEQ ID NO:52中的任一个具有99%的同一性的核酸序列、SEQ ID NO:42-SEQ ID NO:52中的任一个。
在一些实施方案中,所述E2包含选自下组的核酸序列:(a)SEQ ID NO:53;(b)SEQID NO:54;(c)SEQ ID NO:55;(d)SEQ ID NO:56;(e)SEQ ID NO:57;(f)SEQ ID NO:58;(g)SEQ ID NO:59;(h)SEQ ID NO:60;(i)SEQ ID NO:61;(j)SEQ ID NO:62;(k)SEQ ID NO:63。
在一些实施方案中,所述E2基本上由选自下组的核酸序列组成:SEQ ID NO:53-SEQ ID NO:63。
在一些实施方案中,所述E2由选自下组的核酸序列组成:SEQ ID NO:53-SEQ IDNO:63。
在一些实施方案中,所述E1包含选自下组的核酸序列:SEQ ID NO:64;SEQ ID NO:65;SEQ ID NO:66;SEQ ID NO:67;SEQ ID NO:68;SEQ ID NO:69;SEQ ID NO:70;SEQ IDNO:71;SEQ ID NO:72;SEQ ID NO:73;SEQ ID NO:74。
在一些实施方案中,其中所述E1基本上由选自下组的核酸序列组成:SEQ ID NO:64-SEQ ID NO:74。
在一些实施方案中,所述E1由选自下组的核酸序列组成:SEQ ID NO:64-SEQ IDNO:74。
在一些实施方案中,所述5’内含子片段包含选自下组的核酸序列:(a)与SEQ IDNO:75具有95%的同一性的核酸序列;(b)与SEQ ID NO:75具有98%的同一性的核酸序列;
(c)与SEQ ID NO:75具有99%的同一性的核酸序列;(d)SEQ ID NO:75;(e)与SEQID NO:76具有95%的同一性的核酸序列;(f)与SEQ ID NO:76具有98%的同一性的核酸序列;(g)与SEQ ID NO:76具有99%的同一性的核酸序列;(h)SEQ ID NO:76;(i)与SEQ IDNO:77具有95%的同一性的核酸序列;(j)与SEQ ID NO:77具有98%的同一性的核酸序列;(k)与SEQ ID NO:77具有99%的同一性的核酸序列;(l)SEQ ID NO:77;(m)与SEQ ID NO:78具有95%的同一性的核酸序列;(n)与SEQ ID NO:78具有98%的同一性的核酸序列;(o)与SEQ ID NO:78具有99%的同一性的核酸序列;(p)SEQ ID NO:78;(q)与SEQ ID NO:79具有95%的同一性的核酸序列;(r)与SEQ ID NO:79具有98%的同一性的核酸序列;(s)与SEQID NO:79具有99%的同一性的核酸序列;(t)SEQ ID NO:79;(u)与SEQ ID NO:80具有95%的同一性的核酸序列;(v)与SEQ ID NO:80具有98%的同一性的核酸序列;(w)与SEQ IDNO:80具有99%的同一性的核酸序列;(x)SEQ ID NO:80;(y)与SEQ ID NO:81具有95%的同一性的核酸序列;(z)与SEQ ID NO:81具有98%的同一性的核酸序列;(aa)与SEQ ID NO:81具有99%的同一性的核酸序列;(bb)SEQ ID NO:81;(cc)与SEQ ID NO:82具有95%的同一性的核酸序列;(dd)与SEQ ID NO:82具有98%的同一性的核酸序列;(ee)与SEQ ID NO:82具有99%的同一性的核酸序列;(ff)SEQ ID NO:82;(gg)与SEQ ID NO:83具有95%的同一性的核酸序列;(hh)与SEQ ID NO:83具有98%的同一性的核酸序列;(ii)与SEQ ID NO:83具有99%的同一性的核酸序列;(jj)SEQ ID NO:83;(kk)与SEQ ID NO:84具有95%的同一性的核酸序列;(ll)与SEQ ID NO:84具有98%的同一性的核酸序列;(mm)与SEQ ID NO:84具有99%的同一性的核酸序列;(nn)SEQ ID NO:84;(oo)与SEQ ID NO:85具有95%的同一性的核酸序列;(pp)与SEQ ID NO:85具有98%的同一性的核酸序列;(qq)与SEQ ID NO:85具有99%的同一性的核酸序列;(rr)SEQ ID NO:85;(ss)与具SEQ ID NO:86具有95%的同一性的核酸序列;(tt)与SEQ ID NO:86具有98%的同一性的核酸序列;(uu)与SEQ ID NO:86具有99%的同一性的核酸序列;(vv)SEQ ID NO:86;(ww)与SEQ ID NO:87具有95%的同一性的核酸序列;(xx)与SEQ ID NO:87具有98%的同一性的核酸序列;(yy)与SEQ ID NO:87具有99%的同一性的核酸序列;(zz)SEQ ID NO:87;(aaa)与SEQ ID NO:88具有95%的同一性的核酸序列;(bbb)与SEQ ID NO:88具有98%的同一性的核酸序列;(ccc)与SEQ IDNO:88具有99%的同一性的核酸序列;(ddd)SEQ ID NO:88。
在一些实施方案中,所述5’内含子片段基本上由选自下组的核酸序列组成:与SEQID NO:75-SEQ ID NO:88中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:75-SEQID NO:88中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:75-SEQ ID NO:88中的任一个具有99%的同一性的核酸序列、SEQ ID NO:75-SEQ ID NO:88中的任一个。
在一些实施方案中,所述5’内含子片段由选自下组的核酸序列组成:与SEQ IDNO:75-SEQ ID NO:88中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:75-SEQ IDNO:88中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:75-SEQ ID NO:88中的任一个具有99%的同一性的核酸序列、SEQ ID NO:75-SEQ ID NO:88中的任一个。
在一些实施方案中,所述5’同源臂包含SEQ ID NO:105的核酸序列。在一些实施方案中,所述5’同源臂包含与SEQ ID NO:105具有95%的同一性的核酸序列。在一些实施方案中,所述5’同源臂包含与SEQ ID NO:105具有98%的同一性的核酸序列。在一些实施方案中,所述5’同源臂包含与SEQ ID NO:105具有99%的同一性的核酸序列。
在一些实施方案中,所述5’同源臂基本上由SEQ ID NO:105的核酸序列组成。
在一些实施方案中,所述5’同源臂由SEQ ID NO:105的核酸序列组成。
在一些实施方案中,所述3’同源臂包含SEQ ID NO:106的核酸序列。在一些实施方案中,所述3’同源臂包含与SEQ ID NO:106具有95%的同一性的核酸序列。在一些实施方案中,所述3’同源臂包含与SEQ ID NO:106具有98%的同一性的核酸序列。在一些实施方案中,所述3’同源臂包含与SEQ ID NO:106具有99%的同一性的核酸序列。
在一些实施方案中,所述3’同源臂基本上由SEQ ID NO:106的核酸序列组成。
在一些实施方案中,所述3’同源臂由SEQ ID NO:106的核酸序列组成。
在一些实施方案中,所述5’同源臂或3’同源臂的长度为15-60个核苷酸。在一些实施方案中,所述5’同源臂或3’同源臂的长度为15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60个核苷酸。
在一些实施方案中,所述5’同源臂或3’同源臂序列具有至多1%、2%、3%、4%、5%、6%、7%、8%、9%、10%的碱基错配。
在一些实施方案中,所述目标序列包含选自下组的5’臂序列:(a)SEQ ID NO:89;(b)SEQ ID NO:90;(c)SEQ ID NO:91;(d)SEQ ID NO:92;(e)SEQ ID NO:93;(f)SEQ ID NO:94;(g)SEQ ID NO:95;(h)SEQ ID NO:96。
在一些实施方案中,所述目标序列包含选自下组的3’臂序列:(a)SEQ ID NO:97;(b)SEQ ID NO:98;(c)SEQ ID NO:99;(d)SEQ ID NO:100;(e)SEQ ID NO:101;(f)SEQ IDNO:102;(g)SEQ ID NO:103;(h)SEQ ID NO:104。
在一些实施方案中,所述目标序列包含式I:
TI-(L)n-Z1(I),其中:TI为包含内部核糖体进入位点(IRES)样多核苷酸序列或天然IRES序列的改造过的翻译起始元件,
Z1为编码治疗产物的表达序列;L为接头序列;A1和B1为能够使所述RNA多核苷酸环化的一对序列;n为选自0-2的整数。
在一些实施方案中,Z1包括选自下组的核酸序列:与SEQ ID NO:107具有95%的同一性的核酸序列;与SEQ ID NO:107具有98%的同一性的核酸序列;与SEQ ID NO:107具有99%的同一性的核酸序列;SEQ ID NO:107;与SEQ ID NO:108具有95%的同一性的核酸序列;与SEQ ID NO:108具有98%的同一性的核酸序列;与SEQ ID NO:108具有99%的同一性的核酸序列;SEQ ID NO:108;与SEQ ID NO:109具有95%的同一性的核酸序列;与SEQ IDNO:109具有98%的同一性的核酸序列;与SEQ ID NO:109具有99%的同一性的核酸序列;SEQ ID NO:109;与SEQ ID NO:110具有95%的同一性的核酸序列;与SEQ ID NO:110具有98%的同一性的核酸序列;与SEQ ID NO:110具有99%的同一性的核酸序列;SEQ ID NO:110;与SEQ ID NO:111具有95%的同一性的核酸序列;与SEQ ID NO:111具有98%的同一性的核酸序列;与SEQ ID NO:111具有99%的同一性的核酸序列;SEQ ID NO:111;与SEQ IDNO:112具有95%的同一性的核酸序列;与SEQ ID NO:112具有98%的同一性的核酸序列;与SEQ ID NO:112具有99%的同一性的核酸序列;SEQ ID NO:112。
在一些实施方案中,Z1基本上由选自下组的核酸序列组成:与SEQ ID NO:107-SEQID NO:112中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:107-SEQ ID NO:112中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:107-SEQ ID NO:112中的任一个具有99%的同一性的核酸序列、SEQ ID NO:107-SEQ ID NO:112中的任一个。
在一些实施方案中,Z1由选自下组的核酸序列组成:与SEQ ID NO:107-SEQ IDNO:112中的任一个具有95%的同一性的核酸序列、与SEQ ID NO:107-SEQ ID NO:112中的任一个具有98%的同一性的核酸序列、与SEQ ID NO:107-SEQ ID NO:112中的任一个具有99%的同一性的核酸序列、SEQ ID NO:107-SEQ ID NO:112中的任一个。
在一些实施方案中,Z1包括编码选自下组的氨基酸序列的核酸序列:(a)SEQ IDNO:113;(b)SEQ ID NO:114;(c)SEQ ID NO:115;(d)SEQ ID NO:116;(e)SEQ ID NO:117;(f)SEQ ID NO:118。
在一些实施方案中,Z1基本上由编码选自下组的氨基酸序列的核酸序列组成:SEQID NO:113-SEQ ID NO:118。
在一些实施方案中,Z1由编码选自下组的氨基酸序列的核酸序列组成:SEQ IDNO:113-SEQ ID NO:118。
在一些实施方案中,所述多核苷酸构建体包含经修饰的RNA核苷酸和/或经修饰的核苷。
在一些实施方案中,所述多核苷酸构建体包含10%-100%的经修饰的RNA核苷酸和/或经修饰的核苷。在一些实施方案中,所述多核苷酸构建体包含10%、11%、12%、13%、14%、15%、16%、17%、18%、19%、20%、21%、22%、23%、24%、25%、26%、27%、28%、29%、30%、31%、32%、33%、34%、35%、36%、37%、38%、39%、40%、31%、32%、33%、34%、35%、36%、37%、38%、39%、40%、41%、42%、43%、44%、45%、46%、47%、48%、49%、50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%、100%的经修饰的RNA核苷酸和/或经修饰的核苷。
在一些实施方案中,所述经修饰的RNA核苷酸和/或经修饰的核苷为m5C(5-甲基胞苷)。在一些实施方案中,实施方案47-48中任一项的多核苷酸构建体,其中所述经修饰的RNA核苷酸和/或经修饰的核苷中的至少一种为m5U(5-甲基尿苷)。
在一些实施方案中,所述经修饰的RNA核苷酸和/或经修饰的核苷为m6A(N6-甲基腺苷)。
在一些实施方案中,所述经修饰的RNA核苷酸和/或经修饰的核苷为Y(假尿苷)。
在一些实施方案中,所述经修饰的RNA核苷酸和/或经修饰的核苷为m1A(1-甲基腺苷)。
在一些实施方案中,在体外转录(IVT)时引入所述经修饰的RNA核苷酸和/或经修饰的核苷。
在一些实施方案中,所述经修饰的核苷选自下组:m5C(5-甲基胞苷)、m5U(5-甲基尿苷)、m6A(N6-甲基腺苷)、s2U(2-硫代尿苷)、Y(假尿苷)、Um(2'-O-甲基尿苷)、m1A(1-甲基腺苷)、m2A(2-甲基腺苷)、Am(2’-0-甲基腺苷)、ms2 m6A(2-甲基硫代-N6-甲基腺苷)、i6A(N6-异戊烯基腺苷)、ms2i6A(2-甲基硫代-N6异戊烯基腺苷)、io6A(N6-(顺式-羟基异戊烯基)腺苷)、ms2io6A(2-甲基硫代-N6-(顺式-羟基异戊烯基)腺苷)、g6A(N6-甘氨酰基氨基甲酰基腺苷)、t6A(N6-苏氨酰基氨基甲酰基腺苷)、ms2t6A(2-甲基硫代-N6-苏氨酰基氨基甲酰基腺苷)、m6t6A(N6-甲基-N6-苏氨酰基氨基甲酰基腺苷)、hn6A(N6-羟基正缬氨酰基氨基甲酰基腺苷)、ms2hn6A(2-甲基硫代-N6-羟基正缬氨酰基氨基甲酰基腺苷)、Ar(p)(2’-0-核糖基腺苷(磷酸))、I(肌苷)、m1I(1-甲基肌苷)、m1hn(1,2’-O-二甲基肌苷)、m3C(3-甲基胞苷)、Cm(2’-0-甲基胞苷)、s2C(2-硫代胞苷)、ac4C(N4-乙酰基胞苷)、(5-甲酰基胞苷)、m5Cm(5,2'-O-二甲基胞苷)、ac4Cm(N4-乙酰基-2’-O-甲基胞苷)、k2C(赖胞苷)、m!G(1-甲基鸟苷)、m2G(N2-甲基鸟苷)、m7G(7-甲基鸟苷)、Gm(2'-0-甲基鸟苷)、m2 2G(N2,N2-二甲基鸟苷)、m2Gm(N2,2’-O-二甲基鸟苷)、m2 aGm(N2,N2,2’-O-三甲基鸟苷)、Gr(p)(2’-0-核糖基鸟苷(磷酸))、yW(怀丁苷)、oayW(过氧怀丁苷)、OHyW(羟基怀丁苷)、OHyW*(修饰不足的羟基怀丁苷)、imG(怀俄苷)、mimG(甲基怀俄苷)、Q(辫苷)、oQ(环氧辫苷)、galQ(半乳糖基-辫苷)、manQ(甘露糖基-辫苷)、preQo(7-氰基-7-去氮鸟苷)、preQi(7-氨基甲基-7-去氮鸟苷)、G+(古嘌苷)、D(二氢尿苷)、m5Um(5,2’-0-二甲基尿苷)、s4U(4-硫代尿苷)、m5s2U(5-甲基-2-硫代尿苷)、s2Um(2-硫代-2’-0-甲基尿苷)、acp3U(3-(3-氨基-3-羧基丙基)尿苷)、ho5U(5-羟基尿苷)、mo5U(5-甲氧基尿苷)、cmo5U(尿苷5-氧基乙酸)、mcmo5U(尿苷5-氧基乙酸甲基酯)、chm5U(5-(羧基羟基甲基)尿苷)、mchm5U(5-(羧基羟基甲基)尿苷甲基酯)、mcm5U(5-甲氧基羰基甲基尿苷)、mcm5Um(5-甲氧基羰基甲基-2’-0-甲基尿苷)、mcm5s2U(5-甲氧基羰基甲基-2-硫代尿苷)、nm5S2U(5-氨基甲基-2-硫代尿苷)、mnm5U(5-甲基氨基甲基尿苷)、mnm5s2U(5-甲基氨基甲基-2-硫代尿苷)、mnm5se2U(5-甲基氨基甲基-2-硒基尿苷)、ncm5U(5-氨基甲酰基甲基尿苷)、ncm5Um(5-氨基甲酰基甲基-2'-O-甲基尿苷)、cmnm5U(5-羧基甲基氨基甲基尿苷)、cmnm5Um(5-羧基甲基氨基甲基-2'-0-甲基尿苷)、cmnm5s2U(5-羧基甲基氨基甲基-2-硫代尿苷)、m6 2A(N6,N6-二甲基腺苷)、Im(2’-0-甲基肌苷)、m4C(N4-甲基胞苷)、m4Cm(N4,2’-0-二甲基胞苷)、hm5C(5-羟基甲基胞苷)、m3U(3-甲基尿苷)、cm5U(5-羧基甲基尿苷)、m6Am(N6,2’-O-二甲基腺苷)、m6 2Am(N6,N6,0-2’-三甲基腺苷)、m2,7G(N2,7-二甲基鸟苷)、m2,2,7G(N2,N2,7-三甲基鸟苷)、m3Um(3,2’-0-二甲基尿苷)、m5D(5-甲基二氢尿苷)、f5Cm(5-甲酰基-2’-0-甲基胞苷)、m'Gm(l,2’-0-二甲基鸟苷)、m'Am(l,2’-0-二甲基腺苷)、rm 5U(5-牛磺酸甲基尿苷)、τm5s2U(5-牛磺酸甲基-2-硫代尿苷))、imG-14(4-去甲基怀俄苷)、imG2(异怀俄苷)、ac6A(N6-乙酰基腺苷)、吡啶-4-酮核糖核苷、5-氮杂-尿苷、2-硫代-5-氮杂-尿苷、2-硫代尿苷、4-硫代-假尿苷、2-硫代-假尿苷、5-羟基尿苷、3-甲基尿苷、5-羧基甲基-尿苷、1-羧基甲基-假尿苷、5-丙炔基-尿苷、1-丙炔基-假尿苷、5-牛磺酸甲基尿苷、1-牛磺酸甲基-假尿苷、5-牛磺酸甲基-2-硫代-尿苷、l-牛磺酸甲基-4-硫代-尿苷、5-甲基-尿苷、1-甲基-假尿苷、4-硫代-1-甲基-假尿苷、2-硫代-1-甲基-假尿苷、1-甲基-1-去氮-假尿苷、2-硫代-1-甲基-1-去氮-假尿苷、二氢尿苷、二氢假尿苷、2-硫代-二氢尿苷、2-硫代-二氢假尿苷、2-甲氧基尿苷、2-甲氧基-4-硫代-尿苷、4-甲氧基-假尿苷、4-甲氧基-2-硫代-假尿苷、5-氮杂-胞苷、假异胞苷、3-甲基-胞苷、N4-乙酰基胞苷、5-甲酰基胞苷、N4-甲基胞苷、5-羟基甲基胞苷、1-甲基-假异胞苷、吡咯并-胞苷、吡咯并-假异胞苷、2-硫代-胞苷、2-硫代-5-甲基-胞苷、4-硫代-假异胞苷、4-硫代-1-甲基-假异胞苷、4-硫代-1-甲基-1-去氮-假异胞苷、1-甲基-1-去氮-假异胞苷、泽布拉林、5-氮杂-泽布拉林、5-甲基-泽布拉林、5-氮杂-2-硫代-泽布拉林、2-硫代-泽布拉林、2-甲氧基-胞苷、2-甲氧基-5-甲基-胞苷、4-甲氧基-假异胞苷、4-甲氧基-1-甲基-假异胞苷、2-氨基嘌呤、2,6-二氨基嘌呤、7-去氮-腺嘌呤、7-去氮-8-氮杂-腺嘌呤、7-去氮-2-氨基嘌呤、7-去氮-8-氮杂-2-氨基嘌呤、7-去氮-2,6-二氨基嘌呤、7-去氮-8-氮杂-2,6-二氨基嘌呤、1-甲基腺苷、N6-甲基腺苷、N6-异戊烯基腺苷、N6-(顺式-羟基异戊烯基)腺苷、2-甲基硫代-N6-(顺式-羟基异戊烯基)腺苷、N6-甘氨酰基氨基甲酰基腺苷、N6-苏氨酰基氨基甲酰基腺苷、2-甲基硫代-N6-苏氨酰基氨基甲酰基腺苷、N6,N6-二甲基腺苷、7-甲基腺嘌呤、2-甲基硫代-腺嘌呤、2-甲氧基-腺嘌呤、肌苷、1-甲基-肌苷、怀俄苷、怀丁苷、7-去氮-鸟苷、7-去氮-8-氮杂-鸟苷、6-硫代-鸟苷、6-硫代-7-去氮-鸟苷、6-硫代-7-去氮-8-氮杂-鸟苷、7-甲基-鸟苷、6-硫代-7-甲基-鸟苷、7-甲基肌苷、6-甲氧基-鸟苷、1-甲基鸟苷、N2-甲基鸟苷、N2,N2-二甲基鸟苷、8-氧代-鸟苷、7-甲基-8-氧代-鸟苷、1-甲基-6-硫代-鸟苷、N2-甲基-6-硫代-鸟苷、N2,N2-二甲基-6-硫代-鸟苷、5-甲基胞嘧啶、假尿苷、1-甲基假尿苷。
在一些实施方案中,所述环形RNA的长度为至少500个核苷酸、长度为至少1,000个核苷酸、长度为至少1,500个核苷酸。
在一些实施方案中,所述环形RNA不含任何不属于所述目标序列的其他序列,如不含E2、E1序列的全部或部分。
在一些实施方案中,本文提供了制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:(a)3’内含子片段;(b)外显子片段2(E2);(c)目标序列;(d)外显子片段1(E1);(d)5’内含子片段。在一个实施方案中,所述5’内含子片段和3’内含子片段各自为II型内含子的片段。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,本文提供了制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:(a)3’内含子片段;(b)外显子片段2(E2);(c)接头序列;(d)目标序列;(e)接头序列;(f)外显子片段1(E1);(g)5’内含子片段。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,本文提供了制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:(a)5’同源臂;(b)3’内含子片段;(c)外显子片段2(E2);(d)目标序列;(e)外显子片段1(E1);(f)5’内含子片段;(g)3’同源臂。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,本文提供了制造环形RNA的方法,所述方法包括:制备从5’到3’包含以下可操作地连接的元件的载体:(a)5’同源臂;(b)3’内含子片段;(c)外显子片段2(E2);(d)接头序列;(e)目标序列;(f)接头序列;(g)外显子片段1(E1);(h)5’内含子片段;(i)3’同源臂。在一个实施方案中,所述5’内含子片段在所述II型内含子中位于3’内含子片段的5’侧。在一个实施方案中,所述E1为所述II型内含子的5’紧邻外显子片段,其长度≥0个核苷酸。在一个实施方案中,所述E2为所述II型内含子的3’紧邻外显子片段,其长度≥0个核苷酸,所述目标序列为空,或为蛋白编码序列、非编码序列或其组合。
在一些实施方案中,本文提供了在细胞中表达蛋白质的方法,包括(a)将实施方案58-61中任一项的环形RNA转染到所述细胞中,或者(b)使实施方案1-57中任一项的多核苷酸构建体发生自剪接环化反应形成环形RNA,并将所述环形RNA转染到所述细胞中;其中,优选所述细胞为真核细胞。
在一些实施方案中,本文提供了在细胞中表达蛋白质的方法,包括(a)将实施方案58-61中任一项的环形RNA转染到所述细胞中,或者(b)使实施方案1-57中任一项的构建体发生自剪接环化反应形成环形RNA,并将所述环形RNA转染到所述细胞中;其中,优选所述细胞为肝细胞、上皮细胞、造血细胞、上皮细胞、内皮细胞、肺细胞、骨细胞、干细胞、间充质细胞、神经细胞(例如,脑膜细胞、星形胶质细胞、运动神经元、背根神经节细胞、前角运动神经元)、感光细胞(例如,视杆细胞、视锥细胞)、视网膜色素上皮细胞、分泌细胞、心脏细胞、脂肪细胞、血管平滑肌细胞、心肌细胞、骨骼肌细胞、β细胞、垂体细胞、滑膜衬里细胞、卵巢细胞、睾丸细胞、成纤维细胞、B细胞、T细胞、树突细胞、巨噬细胞、网织红细胞、白细胞、粒细胞、肿瘤细胞、NK细胞、肝星形细胞、HEK293、HEK293T、HeLa、MCF7、PC3、A549、NCI-H727、HCT-116、MCF10A、HPReC、FHC、永生化细胞系、原代细胞、酵母细胞、酿酒酵母、毕赤酵母、细菌细胞、大肠杆菌、昆虫细胞、草地贪夜蛾sf9、Mimic Sf9、sf21、果蝇S2。
在一些实施方案中,本文提供了使用II型内含子生成具有自剪接活性的序列的方法,所述方法包括以下步骤:定义所述II型内含子的序列;任选地使用剪接测定(线性剪接)检查所述II型内含子的体外自剪接活性;将所述II型内含子劈分成两个片段,将所述两个内含子片段的顺序颠倒,使用剪接测定确认RNA的体外环化。
在一些实施方案中,前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述5’内含子片段和3’内含子片段中分别包含一对或多对彼此互补的配对序列。在优选的实施方案中,所述互补配对序列的长度大于20个核苷酸。
在一些实施方案中,前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述5’内含子片段和/或3’内含子片段中包含一个或者多个亲和标签序列,所述亲和标签序列选自下组:探针结合序列、MS2结合位点、PP7结合位点、链霉亲和素结合位点。
在一些实施方案中,前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述EBS序列选自EBS1、EBS2、EBS3中的一个或多个,优选两个,更优选EBS1和EBS3。
在一些实施方案中,前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述II型内含子的一个或多个EBS序列,优选EBS1和EBS3,被修饰,其中所述EBS序列分别与目标序列中相应长度的两个区域在至少60%的核苷酸位置上互补配对。
在优选的实施方案中,所述目标序列中相应长度的两个区域分别位于所述目标序列的两端。
在优选的实施方案中,前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述多核苷酸构建体能够在体外形成目标序列的环形RNA。
在优选的实施方案中,前述实施方案中任一项的多核苷酸构建体、环形RNA或方法,其中所述多核苷酸构建体能够在体内形成目标序列的环形RNA。
在一些实施方案中,所述II型内含子包含选自表16-24的核酸序列。
6.6.1.纯化
在一些实施方案中,所述多核苷酸包含纯化标签。在优选的实施方案中,所述纯化标签是与缀合到纯化基质的寡核苷酸退火的15-40nt的多核苷酸。纯化基质包括但不限于磁性树脂或珠、硅树脂、Sephadex树脂、亲和树脂、纳米颗粒和纳米材料表面或包被的表面。
在一些实施方案中,所述纯化标签是内含子标签。在一些实施方案中,所述纯化标签是5’内含子标签。在一些实施方案中,所述纯化标签是3’内含子标签。
可以对通过本发明的构建体或方法生成的环形RNA进行纯化。例如,所述纯化方式选自下组中的一种或多种:酶处理;色谱,包括但不限于亲和柱色谱、反向硅胶柱液相色谱、凝胶排阻液相色谱;电泳,包括但不限于凝胶电泳如琼脂糖凝胶电泳,和毛细管电泳。
在将环形RNA产物转染进入细胞之前,优选通过纯化处理尽可能地去除未环化的线性RNA、dsRNA以及其他不想要的组分。线性RNA和一些dsRNA两端的磷酸基团会激活RIG-1信号通路,在细胞中引起强烈的免疫反应,导致外源RNA的降解,影响环形RNA在细胞中发挥功能。用于去除线性RNA的方法包括酶处理,如用RNase R处理;色谱,如高效液相色谱(HPLC)。用于去除末端磷酸基团的方法包括用碱性磷酸酶处理,如来源于牛小肠的碱性磷酸酶(calf intestinal alkaline phosphatase;CIP)。施用和递送
通过本发明的构建体或方法生成的环形RNA可以使用多种递送系统中的任一种递送到细胞内或动物体内。例如,所述递送系统选自下组中的一种或多种:脂质体、聚乙烯亚胺(PEI)、有机金属框架材料(MOF)、脂质体纳米材料(LNP)、聚阳离子、血液糖蛋白、红细胞运输媒介物、金纳米颗粒(AuNP)媒介物、磁性纳米材料媒介物、碳纳米管、石墨烯分子媒介物、量子点材料媒介物、上转换纳米晶、层状氢氧化物材料媒介物、硅晶纳米材料、磷酸钙。在一些实施方案中,可以使用例如脂质体转染或电穿孔将环形RNA转染到细胞中。
6.6.2.目标细胞
在一些实施方案中,所述目标细胞缺乏目的蛋白或酶。例如,在希望将核酸递送到肝细胞的情况下,肝细胞代表目标细胞。在一些实施方案中,本公开文本的组合物在鉴别的基础上转染目标细胞(即,不转染非目标细胞)。本公开文本的组合物还可以制备成优先靶向多种目标细胞和/或在多种目标细胞中表达,所述目标细胞包括但不限于肝细胞、上皮细胞、造血细胞、上皮细胞、内皮细胞、肺细胞、骨细胞、干细胞、间充质细胞、神经细胞(例如,脑膜细胞、星形胶质细胞、运动神经元、背根神经节细胞、前角运动神经元)、感光细胞(例如,视杆细胞、视锥细胞)、视网膜色素上皮细胞、分泌细胞、心脏细胞、脂肪细胞、血管平滑肌细胞、心肌细胞、骨骼肌细胞、β细胞、垂体细胞、滑膜衬里细胞、卵巢细胞、睾丸细胞、成纤维细胞、B细胞、T细胞、树突细胞、巨噬细胞、网织红细胞、白细胞、粒细胞、肿瘤细胞、NK细胞、肝星形细胞、HEK293、HEK293T、HeLa、MCF7、PC3、A549、NCI-H727、HCT-116、MCF10A、HPReC、FHC、其他永生化细胞系、原代细胞系。
在一些实施方案中,本公开文本的组合物还可以针对多种酵母细胞进行优化,所述酵母细胞包括但不限于酿酒酵母、毕赤酵母。
在一些实施方案中,本公开文本的组合物还可以针对多种细菌细胞进行优化,所述细菌细胞包括但不限于大肠杆菌。
在一些实施方案中,本公开文本的组合物还可以针对多种昆虫细胞进行优化,所述昆虫细胞包括但不限于草地贪夜蛾sf9、Mimic Sf9、sf21、果蝇S2。
本公开文本的组合物可以制备成优先分布到目标细胞中和/或针对目标细胞进行优化,所述目标细胞如在心脏、肺、肾脏、肝脏和脾脏中。在一些实施方案中,本公开文本的组合物分布到肝脏的细胞中以促进肝脏的细胞(例如,肝细胞)对其中包含的circRNA的递送和后续表达。靶向细胞可以作为能够产生并且全身地排泄功能性蛋白或酶的生物“储器(reservoir)”或“储库(depot)”起作用。因此,在本公开文本的一个实施方案中,所述转移媒介物在递送时可以靶向肝细胞和/或优先分布到肝脏的细胞中。在实施方案中,在转染目标肝细胞之后,翻译媒介物中装载的circRNA,并且产生、排泄和全身地分布功能性蛋白产物。在其他实施方案中,除肝细胞之外的细胞(例如,肺、脾脏、心脏、眼部或中枢神经系统的细胞)可以用作蛋白质生产的储库位置。
在一个实施方案中,本公开文本的组合物促进受试者内源地产生一种或多种功能性蛋白和/或酶。在本公开文本的实施方案中,所述转移媒介物包含编码缺陷型蛋白或酶的circRNA。在将这样的组合物分布到目标组织中并且随后转染这样的目标细胞后,可以在体内翻译装载到转移媒介物(例如,脂质纳米颗粒)中的外源circRNA以产生由外源施用的circRNA编码的功能性蛋白或酶(例如,受试者缺乏的蛋白质或酶)。因此,本公开文本的组合物利用受试者翻译外源或重组制备的circRNA以产生内源翻译的蛋白质或酶的能力,从而产生(并且在适用的情况下排泄)功能性蛋白或酶。所表达或翻译的蛋白质或酶的特征还可以在于体内包含天然的翻译后修饰,在重组制备的蛋白质或酶中通常可能不存在所述修饰,从而进一步降低所翻译的蛋白质或酶的免疫原性。
编码缺陷型蛋白或酶的circRNA的施用避免了将核酸递送到目标细胞内的特定细胞器的需要。而是,在转染目标细胞并且将核酸递送到目标细胞的细胞质后,可以翻译转移媒介物的circRNA内容物并且表达功能性蛋白或酶。
在一些实施方案中,环形RNA包含一个或多个miRNA结合位点。在一些实施方案中,环形RNA包含由存在于一种或多种非目标细胞或非目标细胞类型(例如,库普弗细胞)中而不存在于一种或多种目标细胞或目标细胞类型(例如,肝细胞)中的miRNA识别的一个或多个miRNA结合位点。在一些实施方案中,环形RNA包含由与一种或多种目标细胞或目标细胞类型(例如,肝细胞)相比在一种或多种非目标细胞或非目标细胞类型(例如,库普弗细胞)中以增加的浓度存在的miRNA识别的一个或多个miRNA结合位点。miRNA被认为通过与RNA分子内的互补序列配对而起作用,从而导致基因沉默。
6.6.3.药物组合物/施用
在一些实施方案中,本文提供了包含本文提供的治疗剂的组合物(例如,药物组合物)。在一些实施方案中,所述治疗剂是本文提供的环形RNA多核苷酸。在一些实施方案中,所述治疗剂是本文提供的载体。在一些实施方案中,所述治疗剂是包含本文提供的环形RNA或载体的细胞。在一些实施方案中,所述组合物进一步包含药学上可接受的载体。在一些实施方案中,本文提供的组合物包含本文提供的治疗剂与其他药物活性剂或药物的组合。在优选的实施方案中,所述药物组合物包含本文提供的细胞或其群体。
关于药物组合物,药学上可接受的载体可以是常规使用的载体中的任一种,并且仅受化学物理考虑(如溶解性和缺乏与一种或多种活性剂的反应性)和施用途径的限制。本文所述的药学上可接受的载体(例如,媒介物、佐剂、赋形剂和稀释剂)是本领域技术人员熟知的,并且是公众易于获得的。优选的是,药学上可接受的载体是对一种或多种治疗剂具有化学惰性的载体,以及在使用条件下没有有害副作用或毒性的载体。
载体的选择将部分地取决于特定的治疗剂以及用于施用治疗剂的特定方法。因此,存在多种本文提供的药物组合物的合适的配制品。
在一些实施方案中,所述药物组合物包含防腐剂。在一些实施方案中,合适的防腐剂可以包括例如对羟基苯甲酸甲酯、对羟基苯甲酸丙酯、苯甲酸钠和苯扎氯铵。任选地,可以使用两种或更多种防腐剂的混合物。防腐剂或其混合物通常以按总组合物的重量计约0.0001%至约2%的量存在。
在一些实施方案中,所述药物组合物包含缓冲剂。在一些实施方案中,合适的缓冲剂可以包括例如柠檬酸、柠檬酸钠、磷酸、磷酸钾以及多种其他酸和盐。任选地,可以使用两种或更多种缓冲剂的混合物。缓冲剂或其混合物通常以按总组合物的重量计约0.001%至约4%的量存在。
在一些实施方案中,所述药物组合物中治疗剂的浓度可以变化,例如按重量计小于约1%,或至少约1%、2%、3%、4%、5%、6%、7%、8%、9%、10%、15%、20%、25%、30%、35%、40%、45%或约50%或更多,并且可以根据所选择的特定施用方式主要依据流体体积和粘度来选择。
以下用于口服、气雾剂、肠胃外(例如,皮下、静脉内、动脉内、肌内、皮内、腹膜内和鞘内)和外用施用的配制品仅仅是示例性的,而绝不是限制性的。可以使用多于一种途径来施用本文提供的治疗剂,并且在一些情况下,特定途径可以提供比另一种途径更直接且更有效的反应。
适合于口服施用的配制品可以包括以下或由以下组成:(a)液体溶液,如溶解于稀释剂(如水、盐水或橙汁)中的有效量的治疗剂;(b)胶囊剂、小袋、片剂、锭剂和糖锭剂,其各自含有预定量的呈固体或颗粒的活性成分;(c)散剂;(d)适当液体中的混悬剂;和(e)合适的乳剂。液体配制品可以包括稀释剂,如水和醇(例如,乙醇、苄醇和聚乙烯醇),添加或不添加药学上可接受的表面活性剂。胶囊剂形式可以属于含有例如表面活性剂、润滑剂和惰性填充剂(如乳糖、蔗糖、磷酸钙和玉米淀粉)的普通的硬壳或软壳明胶类型。片剂形式可以包括以下中的一种或多种:乳糖、蔗糖、甘露醇、玉米淀粉、马铃薯淀粉、海藻酸、微晶纤维素、阿拉伯胶、明胶、瓜尔胶、胶体二氧化硅、交联羧甲纤维素钠、滑石、硬脂酸镁、硬脂酸钙、硬脂酸锌、硬脂酸和其他赋形剂、着色剂、稀释剂、缓冲剂、崩解剂、润湿剂、防腐剂、调味剂和其他药理学上相容的赋形剂。锭剂形式可以包含治疗剂与调味剂(通常是蔗糖、阿拉伯胶或黄芪胶)。糖果锭剂可以包含治疗剂与惰性基质(如明胶和甘油,或蔗糖和阿拉伯胶、乳液、凝胶等),还含有如本领域已知的这样的赋形剂。
适合于肠胃外施用的配制品包括水性和非水性的等渗无菌注射溶液,其可以含有抗氧化剂、缓冲剂、抑菌剂和使配制品与预期接受者的血液等渗的溶质;以及水性和非水性的无菌混悬剂,其可以包括助悬剂、增溶剂、增稠剂、稳定剂和防腐剂。在一些实施方案中,本文提供的治疗剂可以在药物载体中的生理学上可接受的稀释剂中施用,所述稀释剂如包括以下的无菌液体或液体混合物:水、盐水、水性右旋糖和相关糖溶液、醇如乙醇或十六醇、二醇如丙二醇或聚乙二醇、二甲亚砜、甘油、缩酮如2,2-二甲基-1,3-二氧戊环-4-甲醇、醚、聚(乙二醇)400、油、脂肪酸、脂肪酸酯或甘油酯或乙酰化的脂肪酸甘油酯,添加或不添加药学上可接受的表面活性剂(如肥皂或洗涤剂)、助悬剂(如果胶、卡波姆、甲基纤维素、羟丙基甲基纤维素或羧甲基纤维素)或乳化剂和其他药物佐剂。
在一些实施方案中,可用于肠胃外配制品中的油包括石油、动物油、植物油或合成油。油的具体例子包括花生油、大豆油、芝麻油、棉籽油、玉米油、橄榄油、矿脂和矿物油。用于在肠胃外配制品中使用的合适的脂肪酸包括油酸、硬脂酸和异硬脂酸。油酸乙酯和肉豆蔻酸异丙酯是合适的脂肪酸酯的例子。
用于在肠胃外配制品的一些实施方案中使用的合适的肥皂包括脂肪碱金属、铵和三乙醇胺盐,并且合适的洗涤剂包括(a)阳离子洗涤剂,例如像二甲基二烷基铵卤化物和烷基吡啶鎓卤化物;(b)阴离子洗涤剂,例如像烷基、芳基和烯烃的磺酸盐,烷基、烯烃、醚和甘油单酯的硫酸盐和磺基琥珀酸盐;(c)非离子洗涤剂,例如像脂肪胺氧化物、脂肪酸烷醇酰胺和聚氧乙烯聚丙烯共聚物;(d)两性洗涤剂,例如像烷基-b-氨基丙酸盐和2-烷基-咪唑啉季铵盐;以及(e)其混合物。
在一些实施方案中,所述肠胃外配制品在溶液中将含有按重量计例如从约0.5%至约25%的治疗剂。可以使用防腐剂和缓冲剂。为了最小化或消除注射部位处的刺激,这样的组合物可以含有一种或多种具有例如从约12至约17的亲水亲油平衡值(HLB)的非离子表面活性剂。这样的配制品中表面活性剂的量的范围通常将为按重量计例如从约5%至约15%。合适的表面活性剂包括聚乙二醇、脱水山梨醇脂肪酸酯(如脱水山梨醇单油酸酯)和环氧乙烷与疏水性基质的高分子量加合物,所述疏水性基质是由环氧丙烷与丙二醇缩合形成的。所述肠胃外配制品可以提供在单位剂量或多剂量密封容器(如安瓿或小瓶)中,并且可以储存在冷冻干燥(冻干)条件下,仅需要在即将使用前添加用于注射的无菌液体赋形剂(例如,水)。临时注射溶液和混悬剂可以由前述种类的无菌散剂、颗粒剂和片剂制备。
在一些实施方案中,本文提供了可注射配制品。对于可注射组合物的有效药物载体的要求是本领域普通技术人员熟知的(参见例如,Pharmaceutics and PharmacyPractice,J.B.Lippincott Company,宾夕法尼亚州费城,Banker和Chalmers编辑,第238-250页(1982),和ASHP Handbook on Injectable Drugs,Toissel,第4版,第622-630页(1986))。
在一些实施方案中,本文提供了外用配制品。外用配制品(包括可用于经皮药物释放的外用配制品)在本文提供的某些实施方案的上下文中适合于应用至皮肤。在一些实施方案中,所述治疗剂单独或与其他合适的组分组合可以制成待经由吸入施用的气雾剂配制品。可以将这些气雾剂配制品置入加压的可接受的推进剂中,所述推进剂如二氯二氟甲烷、丙烷、氮气等。还可以将它们配制为用于非加压制剂的药物,如在喷雾器或雾化器中。这样的喷雾配制品还可用于喷雾粘膜。
在一些实施方案中,可以将本文提供的治疗剂配制为包含物复合物,如环糊精包合物复合物,或配制为脂质体。脂质体可用于将治疗剂靶向特定组织。脂质体还可用于增加治疗剂的半衰期。许多方法可用于制备脂质体,如例如Szoka等人,Ann.Rev.Biophys.Bioeng.,9,467(1980)以及美国专利4,235,871、4,501,728、4,837,028和5,019,369中所描述的。
在一些实施方案中,将本文提供的治疗剂配制在定时释放、延迟释放或持续释放递送系统中,使得所述组合物的递送发生在待治疗部位的致敏之前并且有足够的时间引起致敏。这样的系统可以避免治疗剂的重复施用,从而增加对受试者和医师的便利性,并且可以特别适合于本文提供的某些组合物实施方案。在一个实施方案中,这样配制本公开文本的组合物,使得它们适合于其中含有的circRNA的延长释放。这样的延长释放组合物可以以延长的给药间隔便利地施用至受试者。例如,在一个实施方案中,将本公开文本的组合物每天两次、每天一次或每隔一天一次施用至受试者。在实施方案中,将本公开文本的组合物每周两次、每周一次、每十天一次、每两周一次、每三周一次、每四周一次、每月一次、每六周一次、每八周一次、每三个月一次、每四个月一次、每六个月一次、每八个月一次、每九个月一次或每年一次施用至受试者。
在一些实施方案中,目标细胞产生由本文所述的多核苷酸编码的蛋白质持续持续量的时间。例如,施用后所述蛋白质可以产生超过一小时、超过四小时、超过六小时、超过12小时、超过24小时、超过48小时或超过72小时。在一些实施方案中,在施用后约六小时所述治疗产物以峰值水平表达。在一些实施方案中,所述治疗产物的表达至少以治疗水平持续。在一些实施方案中,施用后所述治疗产物至少以治疗水平表达持续超过一小时、超过四小时、超过六小时、超过12小时、超过24小时、超过48小时或超过72小时。在一些实施方案中,在患者血清或组织(例如,肝脏或肺)中以治疗水平可检测到所述治疗产物。在一些实施方案中,一定水平的可检测治疗产物来自所述circRNA组合物在施用后超过一小时、超过四小时、超过六小时、超过12小时、超过24小时、超过48小时或超过72小时的时间段内的连续表达。
在一些实施方案中,由本文所述的多核苷酸编码的蛋白质以高于正常生理水平的水平产生。与对照相比,蛋白质水平可以增加。在一些实施方案中,所述对照是在正常个体中或在正常个体群体中所述治疗产物的基线生理水平。在其他实施方案中,所述对照是在具有相关蛋白或多肽缺陷的个体中或在具有相关蛋白或多肽缺陷的个体群体中所述治疗产物的基线生理水平。在一些实施方案中,所述对照可以是在被施用所述组合物的个体中相关蛋白或多肽的正常水平。在其他实施方案中,所述对照是在其他治疗干预后(例如,在直接注射相应治疗产物后)在一个或多个可比较的时间点所述治疗产物的表达水平。
在一些实施方案中,在施用后3天、4天、5天或1周或更长时间,可检测到一定水平的由本文所述的多核苷酸编码的蛋白质。可以在血清和/或组织(例如,肝脏或肺)中观察到增加水平的分泌的蛋白质。
在一些实施方案中,所述方法导致由本文所述的多核苷酸编码的蛋白质的持久的循环半衰期。例如,可以比经由皮下注射所述蛋白质或编码所述蛋白质的mRNA观察到的半衰期长数小时或数天检测到所述蛋白质。在一些实施方案中,所述蛋白质的半衰期为1天、2天、3天、4天、5天或1周或更长时间。
许多类型的释放递送系统是可用的并且是本领域普通技术人员已知的。它们包括基于聚合物的系统,如聚(丙交酯-乙交酯)、共聚草酸酯、聚己内酯、聚酯酰胺、聚原酸酯、聚羟基丁酸和聚酸酐。前述含有药物的聚合物的微胶囊描述于例如美国专利5,075,109中。递送系统还包括非聚合物系统,其为脂质,包括固醇,如胆固醇、胆固醇酯和脂肪酸,或中性脂肪,如甘油单酯、甘油二酯和甘油三酯;水凝胶释放系统;硅橡胶系统;基于肽的系统;蜡涂层;使用常规粘合剂和赋形剂的压缩片剂;部分融合的植入物;等。具体的例子包括但不限于:(a)活性组合物以在基质内的形式被包含的侵蚀系统,如美国专利4,452,775、4,667,014、4,748,034和5,239,660中所描述的那些;以及(b)活性组分以受控的速率从聚合物中渗透的扩散系统,如美国专利3,832,253和3,854,480中所描述的那些。此外,可以使用基于泵的硬件递送系统,其中的一些适用于植入。
在一些实施方案中,所述治疗剂可以通过连接部分与靶向部分直接或间接缀合。用于将治疗剂与靶向部分缀合的方法是本领域已知的。参见例如,Wadwa等人,J,DrugTargeting 3:111(1995)和美国专利5,087,616。
在一些实施方案中,将本文提供的治疗剂配制成储库形式,使得治疗剂被释放到它所施用的机体中的方式关于在机体内的时间和位置而受控(参见例如,美国专利4,450,150)。治疗剂的储库形式可以是例如可植入的组合物,其包含治疗剂和多孔或无孔材料,如聚合物,其中所述治疗剂被所述材料包封或扩散到整个材料中和/或通过无孔材料的降解而扩散。然后将储库植入体内的所需位置,并且以预定的速率从植入物中释放治疗剂。
6.6.4.用途
取决于目标序列的种类,通过本发明的构建体或方法生成的环形RNA可用于实现多种用途。例如,在目标序列包含蛋白编码序列或由蛋白编码序列组成时,形成的环形RNA可以用于蛋白质的表达。本发明的环形RNA还可以用于调节miRNA活性、中和RNA结合蛋白结合、表达适配体等多种功能。
7.测定
可以测试各种IRES样序列变体、内源性IRES序列变体或其组合吸引真核核糖体翻译起始复合物和/或促进翻译起始的能力。以下测定是针对IRES样序列描述的,但是对于内源性IRES序列、IRES样序列和内源性IRES序列的组合、包含一个或多个IRES样序列或内源性IRES序列的序列,可以类似地进行。
7.1.确定II型内含子的二级结构和劈分位点
茎环结构是一种类型的RNA二级结构,其可以通过任何合适的多核苷酸折叠算法来确定。一些程序是基于最小吉布斯自由能的计算。一种这样的算法的一个例子是mFold,并且由Zuker和Stiegler(Nu cleic Acids Res.9(1981),133-148)描述。另一种示例性折叠算法是由维也纳大学理论化学研究所使用质心结构预测算法(例如,AR Gruber等人,2008,Cell 106).(1):23-24;以及PACarr和GM Church,2009,Nature Biotechnology 27(12):1151-62)开发的在线网络服务器RNAfol d。另外的算法可以在美国临时专利申请号61/836,080(代理人案卷号44790.11.2022;泛参考编号BI-2013/004A)中找到,将其通过引用并入本文。II型内含子主要包括6个茎环结构,称为结构域1-6(D1-D6),6个结构域顺序排列,其中含有多个外显子结合序列(EBS),如EBS1、EBS2、EBS3。这些EBS序列与外显子区域中的内含子结合序列(IBS)进行相互作用如互补配对,依靠EBS核酸序列内部自身的羟基触发剪接。II型内含子的二级结构的示例性结构示于图9中。在一个实施方案中,使用在线预测工具或预测软件来鉴定II型内含子。这种在线预测工具的一个例子是卡尔加里大学Zimmerly实验室创建的在线网络服务器“http://webapps2.ucalgary.ca/”。
在一个实施方案中,自催化自剪接II型内含子从D1结构域被劈分成两个片段,并在劈分的内含子片段之间插入目标序列。在一个实施方案中,自催化自剪接II型内含子从D2结构域被劈分成两个片段,并在劈分的内含子片段之间插入目标序列。在一个实施方案中,自催化自剪接II型内含子从D3结构域被劈分成两个片段,并在劈分的内含子片段之间插入目标序列。
在优选的实施方案中,自催化自剪接II型内含子从D4结构域被劈分成两个片段,并在劈分的内含子片段之间插入目标序列,如图12所示。
在一个实施方案中,自催化自剪接II型内含子从D5结构域被劈分成两个片段,并在劈分的内含子片段之间插入目标序列。在一个实施方案中,自催化自剪接II型内含子从D6结构域被劈分成两个片段,并在劈分的内含子片段之间插入目标序列。
a.体外circRNA产生
通过体外转录产生前体RNA,然后将其通过cRNAzyme系统环化。
可以使用分子生物学的标准技术制造本文提供的载体。例如,本文提供的载体的各种元件可以使用重组方法获得,如通过筛选来自细胞的cDNA和基因组文库或者通过从已知包括多核苷酸的载体获得所述多核苷酸而获得。
还可以基于已知序列合成地产生(而不是克隆)本文提供的载体的各种元件。完整序列可以由通过标准方法制备的重叠寡核苷酸组装并且组装成完整序列。参见例如,Edge,Nature(1981)292:756;Nambair等人,Science(1984)223:1299;和Jay等人,J.Biol.Chem.(1984)259:631 1。
因此,特定的核苷酸序列可以从含有所需序列的载体获得,或者完全或部分地使用本领域已知的各种寡核苷酸合成技术(如适当情况下的定点诱变和聚合酶链式反应(PCR)技术)合成。获得编码所需载体元件的核苷酸序列的一种方法是通过对在常规的自动化多核苷酸合成器中产生的重叠合成寡核苷酸的互补集进行退火,然后用适当的DNA连接酶连接并且经由PCR扩增连接的核苷酸序列。参见例如,Jayaraman等人,Proc.Natl.Acad.Sci.USA(1991)88:4084-4088。此外,可以使用寡核苷酸指导的合成(Jones等人,Nature(1986)54:75-82)、先前存在的核苷酸区域的寡核苷酸指导的诱变(Riechmann等人,Nature(1988)332:323-327和Verhoeyen等人,Science(1988)239:1534-1536)以及使用T4 DNA聚合酶酶促填充带缺口的寡核苷酸(Queen等人,Proc.Natl.Acad.Sci.USA(1989)86:10029-10033)。
可以通过在允许由载体编码的前体RNA转录的条件下孵育本文提供的载体来生成本文提供的前体RNA。例如,在一些实施方案中,通过将本文提供的包含其5'双链体形成区和/或表达序列上游的RNA聚合酶启动子的载体与相容的RNA聚合酶在允许体外转录的条件下一起孵育来合成前体RNA。在一些实施方案中,将所述载体通过噬菌体RNA聚合酶在细胞内孵育,或通过宿主RNA聚合酶P在细胞的细胞核中孵育。
在一些实施方案中,本文提供了通过使用本文提供的载体作为模板(例如,本文提供的具有位于5'同源区上游的RNA聚合酶启动子的载体)进行体外转录来生成前体RNA的方法。
在一些实施方案中,所得前体RNA可用于生成环形RNA(例如,本文提供的环形RNA多核苷酸)。
因此,在一些实施方案中,本文提供了制造环形RNA的方法。在一些实施方案中,所述方法包括通过使用本文提供的载体作为模板进行转录(例如,失控转录)合成前体RNA,以及将所得前体RNA在适合于环化的条件下孵育,以形成环形RNA。
在一些实施方案中,已经对包含环形RNA的组合物进行纯化。环形RNA可以通过本领域常用的任何已知方法(如柱色谱、凝胶过滤色谱和尺寸排阻色谱)纯化。在一些实施方案中,纯化包括以下步骤中的一个或多个:磷酸酶处理、HPLC尺寸排阻纯化和RNase R消化。在一些实施方案中,纯化按顺序包括以下步骤:RNase R消化、磷酸酶处理和HPLC尺寸排阻纯化。在一些实施方案中,纯化包括反相HPLC。在一些实施方案中,与未纯化的RNA相比,纯化的组合物含有更少的双链RNA、DNA夹板、三磷酸化的RNA、磷酸酶蛋白、蛋白质连接酶、加帽酶和/或带切口的RNA。
7.2.评估治疗产物的表达水平和/或活性
可以通过本领域已知或本文所述的任何方法来确定治疗产物(如多肽、蛋白质、抗体或酶)的水平。例如,可以通过使用例如Northern印迹、PCR分析、实时PCR分析或者本领域已知或本文所述的任何其他技术评估(例如,量化)样品中蛋白质的转录RNA来确定组织样品中治疗产物(如多肽、蛋白质、抗体或酶)的水平。在一个实施方案中,可以通过评估(例如,量化)样品中蛋白质的mRNA来确定组织样品中治疗产物(如多肽、蛋白质、抗体或酶)的水平。还可以通过使用例如免疫组织化学分析、Western印迹、ELISA、免疫沉淀、流式细胞术分析或者本领域已知或本文所述的任何其他技术评估(例如,量化)样品中治疗产物的多肽或蛋白质表达水平来确定组织样品中治疗产物(如多肽、蛋白质、抗体或酶)的水平。在特殊的实施方案中,通过能够量化存在于患者的组织样品中(例如,人血清中)的治疗产物的量的方法,和/或能够检测用circRNA或包含circRNA的配制品处理后蛋白质水平的校正的方法来确定蛋白质的水平。
7.3.筛选测定
可以测试各种IRES样序列变体、内源性IRES序列变体或其组合吸引真核核糖体翻译起始复合物和/或促进翻译起始的能力。以下测定是针对IRES样序列描述的,但是对于内源性IRES序列、IRES样序列和内源性IRES序列的组合、包含一个或多个IRES样序列或内源性IRES序列的序列,可以类似地进行。
7.3.1.表达水平
例如,可以评估本文公开的IRES样序列、内源性IRES序列或其变体或它们的组合对治疗产物表达水平的影响。在一些实施方案中,可以通过包含本文公开的IRES样序列、内源性IRES序列或其变体或它们的组合的RNA多核苷酸或环形RNA在无细胞系统中的体外翻译来评估所述影响。在一些实施方案中,可以通过用包含本文公开的IRES样序列、内源性IRES序列或其变体或它们的组合的RNA多核苷酸或环形RNA转染/转化细胞并且表达所述RNA多核苷酸或环形RNA来评估所述影响。可以根据本领域已知和/或本文所述的任何方法(例如,免疫组织化学分析、Western印迹、ELISA、免疫沉淀和流式细胞术分析)来评估治疗产物的表达水平。
在一些实施方案中,可以进一步评估基于它们对治疗产物表达水平的影响(例如,表达标记物如荧光蛋白或萤光素酶)而鉴定的IRES样序列、内源性IRES序列或其变体或它们的组合在体外促进、促成或调节(例如,增加或减少)治疗产物表达的能力。在一些实施方案中,可以在动物模型中进一步评估IRES样序列、内源性IRES序列或其变体或它们的组合在体内促进、促成或调节(例如,增加或减少)治疗产物表达的能力。在一些实施方案中,可以在动物模型中进一步评估IRES样序列、内源性IRES序列或其变体或它们的组合促进、促成或调节(例如,增加或减少)治疗产物在特定组织或器官中的表达的能力。
下文提供了各种测定或方法的非限制性说明性例子。
体外转染和翻译
可以使用转染试剂(如Lipofectamine 3000(Invitrogen))将希望量的环形RNA转染到细胞(例如,原核细胞或真核细胞)中。可以在无细胞裂解液中进行希望量的环形RNA的体外翻译。孵育后可以收集细胞裂解物以分析蛋白质表达。
Western印迹
可以裂解根据本文所述的方法或技术用本公开文本的包含IRES样序列、内源性IRES序列或其变体或它们的组合的RNA多核苷酸或环形RNA转染的细胞。例如通过如用4%-20%ExpressPlus
萤光素酶测定
可以裂解根据本文所述的方法或技术用本公开文本的包含IRES样序列、内源性IRES序列或其变体或它们的组合的RNA多核苷酸或环形RNA转染的细胞。将萤光素酶报告物测定(如来自Promega
流式细胞术
收集根据本文所述的方法或技术用本公开文本的包含IRES样序列、内源性IRES序列或其变体或它们的组合的RNA多核苷酸或环形RNA转染的细胞进行流式细胞术,例如通过使用BD FACSAria II进行。为了选择单细胞(singlet),SSC-A与FSC-A可用于选择293T细胞。单细胞的两轮选择可以使用SSC-W与FSC-H和FSC-W与FSC-H。FITC-A与FSC-A可用于选择GFP阳性细胞,并且可以通过荧光水平确定表达水平。
ELISA
可以根据Bull World Health Organ.54(2):129-39(1976)(PMID:798633)进行ELISA。
任选地,所述治疗产物可以用其他化合物衍生化并且具有促进所述化合物分离的衍生化基团。衍生化基团的非限制性例子包括生物素、荧光素、地高辛(digoxygenin)、绿色荧光蛋白、同位素、多组氨酸、磁珠、谷胱甘肽S转移酶(GST)、可光活化的交联剂或其任何组合。任选地,可以通过测量衍生化基团的水平来评估所述治疗产物的表达水平。
7.3.2.生物活性
可以根据本领域已知和/或本文所述的任何方法(例如,体内成像和PET)评估本文公开的IRES样序列、内源性IRES序列或其变体或它们的组合的生物活性或功能。例如,生物活性可以包括抑制肿瘤生长,并且可以在动物模型(如细胞系衍生的异种移植物(CDX)和患者衍生的异种移植物(PDX)模型)中评估所述生物活性。
下文提供了各种测定或方法的非限制性说明性例子。
体内成像
为了检测体内表达,可以使用6-8周龄的雌性BALB/c小鼠来施用本公开文本的包含IRES样序列、内源性IRES序列或其变体或它们的组合的RNA多核苷酸或环形RNA(例如,编码萤光素酶的环形RNA)。施用可以经由肌内(i.m.)、皮下(s.c.)或鼻内(i.n.)途径。在施用后的某些时间,向动物腹膜内(i.p.)注射萤光素酶底物。例如通过IVIS光谱仪器(PerkinElmer)收集荧光信号。对于体外成像,立即收集来自动物的组织(包括脑、心脏、肝脏、脾脏、肺、肾脏和肌肉),并且例如通过IVIS成像仪测量每种组织的荧光信号。例如通过使用Living Image3.0来量化目标区域(ROI)中的荧光信号。
PET
用肿瘤细胞系形成小鼠肿瘤异种移植物。在向动物施用本公开文本的包含IRES样序列、内源性IRES序列或其变体或它们的组合的RNA多核苷酸或环形RNA之前和之后进行PET扫描。例如,可以在3.7MBq至7.4MBq施用后1h进行PET扫描。可以在进一步施用后的合适时间点进行第二次PET扫描。
例如,所述测定可用于获得所表达的治疗产物在生物系统中的竞争性结合能力。
例如,所述测定可用于获得所表达的治疗产物在生物系统中的酶促能力。
例如,所述测定可用于获得所表达的治疗产物在生物系统中的抑制活性。
在无细胞系统(如可以用纯化或半纯化的蛋白质衍生)中进行的测定通常优选作为“主要”筛选,因为它们可以经生成以允许快速发展并且相对容易地检测由测试化合物介导的分子目标中的改变。此外,在体外系统中,测试化合物的细胞毒性或生物利用度的影响通常可以忽略,而是所述测定主要集中于所述治疗产物对分子目标的作用。
序列
表1.:序列表第1部分
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
*关于SEQ ID NO:2(II型内含子Cte_原始)的序列,来自外显子的核苷酸由大写字母表示。来自外显子并且与EBS区相互作用的核苷酸由
表2.:II型内含子序列
/>
/>
/>
/>
表3.:3’内含子片段
/>
/>
/>
表4.:E2
表5.:E1
表6.:5’内含子片段
/>
/>
/>
/>
/>
/>
表7.:目标序列的5’臂
/>
表8.:目标序列的3’臂
/>
表9.:同源臂序列
表10.:目标序列
/>
/>
/>
/>
/>
/>
/>
表11.:氨基酸序列
/>
/>
表12.:EBS1
表13.:IBS1
表14.:δ及其上游序列
表15.:IBS1及其下游序列
表16.:真细菌内含子
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
表17.:古生菌内含子
/>
表18.:无ORF的内含子
表19.:连生内含子(Twintron)(外内含子)
表20.:连生内含子(内内含子)
表21.:线粒体内含子
/>
/>
表22.:叶绿体内含子
表23.:细菌内含子片段
/>
/>
/>
表24.:古细菌内含子片段
8.实施例
为了更全面地理解和应用本发明,下文将参考实施例和附图详细描述本发明,所述实施例仅是意图举例说明本发明,而不是意图限制本发明的范围。本发明的范围由后附的权利要求具体限定。
8.1.实施例1.II型内含子的筛选
本实施例涉及确认天然II型内含子的体外自剪接能力的方法。
首先,根据II型内含子的天然序列直接合成DNA序列(Genewiz,苏州),合成的DNA序列除了II型内含子序列本身之外,还包括天然存在于侧翼的外显子E1、E2序列,特别是与II型内含子紧邻的外显子中的内含子结合区的全部或部分。将所述DNA序列通过分子生物学方法克隆到含有Rluc(海肾萤光素酶)编码序列、T7启动子和T7终止子的经修饰的表达载体psiCHECK-2(Promega,C8021)中。具体来说,将psiCHECK-2用内切酶XhoI(New EnglandBiolabs(NEB))进行单酶切,然后将所述合成的DNA序列使用DNA无缝组装方法(ABclonalTechnology,武汉)克隆进入经酶消化的载体中,位于Rluc的3’下游,以获得相应的构建体。该表达载体的骨架是包含T7启动子和T7终止子的psiCHECK-2。
使用针对T7启动子和T7终止子的通用引物,从上述载体进行PCR扩增,以获得转录用模板DNA。该PCR反应的条件为:95℃ 30s,60℃ 20s,72℃ 60s,23-25个循环。将PCR扩增得到的模板DNA用酚氯仿1:1体积抽提,再用2.5倍体积无水乙醇沉淀,由此进行纯化。
将纯化后的模板DNA通过T7 RNA聚合酶(NEB或Promega)进行体外转录,转录反应按照制造商说明书推荐的条件进行。将转录产物用DNase I在37℃消化30分钟以降解PCR模板。再通过柱纯化对转录产物进行纯化,得到高纯度RNA。
将经过柱纯化的转录产物RNA加入自剪接缓冲液(10、20、50或100mM MgCl2,50mMNaCl,40mM Tris-HCl,pH=7.5)进行自剪接反应。反应条件为95℃ 1min,75℃至45℃(-0.5℃,15sec/循环,共60个循环),45℃保持并加入缓冲液,45℃ 5min,53℃ 15-30min(参见图2B)。在体外发生自剪接反应后,取200ng产物使用浓度为1.5%的琼脂糖凝胶进行电泳分析,以检测II型内含子的自剪接效率。
如果成功发生自剪接,则会产生大小不同的两条RNA,未剪接的RNA尺寸较大,在凝胶电泳图中位于上方;剪接后的RNA较小,在凝胶电泳图中位于下方。例如,图3中显示了两种经上述方法鉴定可以发生自剪接的II型内含子的电泳结果,即来自苏云金芽孢杆菌的II型内含子Bth和来自破伤风梭菌的II型内含子Cte(图3)。图3中的箭头分别示出了通过电泳分开的未剪接和剪接后的RNA。将通过本实施例的方法确认的II型内含子及其侧翼外显子序列(如SEQ ID NO:1或SEQ ID NO:2,其分别包含Bth和Cte的II型内含子及其侧翼6个核苷酸的E1和侧翼6个核苷酸的E2)作为制备自剪接核酶构建体cRNAzyme的前体,或称为cRNAzyme前体。
8.2.实施例2.制备包含II型内含子的表达构建体
在通过筛选得到的具有自剪接性质的II型内含子cRNAzyme前体基础上,进一步制备自剪接核酶cRNAzyme构建体。如上所述,设计cRNAzyme构建体的总原则是内含子序列和E1、E2序列的总长度尽可能小,环化率尽可能高。
本实施例详细阐述使用实施例1中筛选出的Cte cRNAzyme前体来设计和制备cRNAzyme构建体的过程。
在Cte cRNAzyme前体序列(SEQ ID NO:2,共1,028个核苷酸长,包含Cte内含子本身以及两端各6个核苷酸的外显子序列,即E1和E2均为6个核苷酸长)的基础上,删除了结构域4中的310个核苷酸的内含子编码蛋白(IEP)序列(SEQ ID NO:2的核苷酸位置625-934),保留了可以进行正确折叠而保持自剪接活性的序列,包括两端少量外显子(各6nt,即IBS1和IBS3),获得E1-Cte△IEP-E2序列。然后将E1-Cte△IEP-E2序列从内含子内部的位置处劈分。将分割后的两个片段互换位置,将由E1和5’内含子片段组成的第一片段构建到插入片段Rluc的3’端,将由3’内含子片段和E2组成的第二片段构建到插入片段Rluc的5’端。在此基础上,在新形成的片段的5’端插入AATACCTTA CTTAATAGTAACAATAGAAAATC(SEQ ID NO:14),3’端插入AAG CTAGATCATATTACTATTAAGTAAGGTATT(SEQ ID NO:15),由此获得cRNAzyme_Cte构建体。SEQ ID NO:14和SEQ ID NO:15这两段插入序列作为“同源臂”可使5’和3’剪接位点彼此接近,提高剪接效率。在分割内含子时,尝试了三种不同的分割位置,分别位于结构域1(第369与370位之间)、结构域3(第560与561位之间)和结构域4(第825与826位之间)中的环状区域中,因此形成了三种cRNAzyme,分别称为cRNAzyme_Cte V1、cRNAzyme_Cte V2、cRNAzyme_Cte V3。在阳离子的存在下,在体外进行自剪接环化反应,以测试获得的cRNAz yme的环化活性。从图4b可见,不同位置分割后剪接效果不同,其中第三种分割方式(V3)剪接效果最好,以其进行后续实验,该构建体的序列如SEQ ID NO:16所示。
根据片段大小,认为图4b的凝胶电泳图中以圆形示出的条带代表了环形RNA。通过RT-PCR和Sanger测序对其即序列进行验证,确定了该条带是包含跨E1E2接头的序列,证明E1、E2已经连接到了一起。但据此尚无法确认其为环形RNA,因为该条带也可能是来自于反向剪接的转录产物,或其他序列。为了确认环形RNA的成功形成,基于环形RNA头尾共价闭合的特征,使用了以下三种方法来验证它的结构(图4c)。
方法I
对Cte中的剪接位点进行了突变,使其丧失环化能力,作为长度相同但不能环化的线性RNA对照。具体来说,将SEQ ID NO:2中的剪接位点(第1-26位核苷酸)进行突变,将第3位的C变为A,第5位的T变为G,第17位的G突变为T,第18位的C突变为T,第21位的A突变为C,第26位的T突变为G。本领域技术人员能够理解此突变旨在破坏环化的能力,也可以进行数量不同、位置不同、类型不同的其他突变来实现类似的目的。将突变后的Cte称为Cte-mut(SEQ ID NO:3)。然后使用多聚腺苷酸酶为此突变的线性RNA添加polyA尾巴。由于环形RNA首尾闭合且没有3’末端,因此不能加尾,而线性RNA可以通过该反应加上数百个腺苷酸,通过琼脂糖凝胶电泳分辨加尾前后RNA大小变化。
具体步骤包括:
(1)将纯化的环形RNA或对照线性RNA通过poly(A)加尾酶(NEB)进行加尾,反应条件是37℃,30min;
(2)柱纯化RNA。
如图4c上部小图所示,泳道3、4是添加了polyA尾巴的产物,对比泳道1、2是未添加polyA尾巴的产物。在未环化的情况下,基于Cte和Cte-mut的cRNAzyme前体RNA条带在进行加polyA反应后均出现上移,表明分子变大,polyA添加成功。与之不同的是,推定未环形RNA的条带在进行和不进行加polyA反应的条件下基本上处于相同的位置,具有相同的大小。
方法II
使用RNase R进行消化。RNase R是一种3’-5’核糖核酸外切酶,可以降解掉线性的RNA分子,闭环结构的环形RNA则不能被降解。通过琼脂糖凝胶电泳可以分辨RNA是否被消化。
具体步骤包括:
(1)将纯化的环形RNA或对照线性RNA通过poly(A)加尾酶(NEB)进行加尾,反应条件是37℃,30min;
(2)加入RNase R(Lucigen)消化线性RNA,反应条件是37℃,30min;
(3)柱纯化RNA。
如图4C上部小图所示,泳道5、6为RNase R处理后的结果,泳道1-4为未进行RNaseR处理的结果。可以看到较大的线性RNA条带在经过RNase R处理之后消失(泳道5和6),而泳道5中推定为环形RNA的条带仍然存在。
方法III
使用RNase H进行消化。RNase H是一种核糖核酸内切酶,可以特异性地水解DNA-RNA杂合链中的RNA。因为线性RNA和环形RNA结构不同,与相同DNA探针结合后,可以被RNaseH切割成长度不一的片段。通过琼脂糖凝胶电泳可以分辨被切割产生的RNA片段的长度,从而反推RNA本来的结构。具体来说,对于环形RNA而言,用两个DNA探针与RNA进行结合,然后进行切割,会得到两条条带。与之不同,如果没有环化,仍然保持线性,采用同样的方法应该会得到三条条带。
(1)通过退火反应,将DNA探针结合到RNA上,反应条件是95℃反应2min,然后缓慢逐级降温至25℃。探针序列见SEQ ID NO:7和8。
(2)用RNase H(NEB)消化DNA/RNA双链,反应条件是37℃,30min;
(3)柱纯化RNA。如图4C下部小图所示,本申请的产物获得了两条条带。
通过以上三种方法获得的结果,均证实了环形RNA的成功形成,验证了构建的cRNAzyme_Cte的环化活性。
8.3.实施例3.通过优化反应体系和改造构建体提高自剪接效率
为了提高最终环形RNA产率,首先需要提高环化效率(图5a)。发明人从两方面对表达构建体的环化效率进行了优化。
反应条件优化
发明人尝试了反应体系中多种不同的离子浓度(50mM和100mM的NaCl;2mM、5mM、10mM、20mM的Mg2+)和多种不同的反应时间(5分钟、15分钟、30分钟)的组合,来确定最优反应体系。
发现在20mM Mg2+、50mM NaCl的反应体系中,进行15和30分钟的条件下,可将环化效率从现有的30%提高至60%以上(图5B)。
序列优化
根据RNA二级结构进一步改造了序列。具体来说,在插入不同目标序列之后,有些序列会因为结构原因而不能有效地进行剪接。在这种情况下,通过在目标序列中纳入一些间隔序列增加结构的柔性来提高剪接效率,例如该间隔序列可以是富含AT的序列。
在实施例2的基础上,通过分子克隆手段在cRNAzyme_Cte中Rluc前端插入了三种不同的间隔序列,即SEQ ID No:4的间隔序列1,SEQ ID NO:5的间隔序列2,和SEQ ID NO:6的间隔序列3,得到三种进一步优化的构建体。在实施例2中确定的最优自剪接反应体系(10mM Mg,50mM NaCl,30分钟反应时长)中使这三种带有间隔序列的前体RNA发生了体外环化。从图5C的结果可知,加入3种间隔序列后的环化效率分别约为60%、80%、98%(图5c)。
8.4.实施例4.制备不含疤痕序列的“无痕”环形RNA
使用前述方法获得的环形RNA仍然会含有少量的非目标序列,即来自外显子E1、E2的序列。为了去除这些序列,可以对构建体进行进一步的改造。
在制备cRNAzyme构建体时,目标序列的两端分别为长度较短的E2和E1,它们分别来源于II型内含子侧翼外显子区的内含子结合序列(IBS),一般而言长度在0-20个核苷酸之间。在形成环形RNA时,不希望包括非目标序列之外的序列,如外显子序列E1和E2。若直接去掉E1和E2,则会因为缺乏与内含子中的EBS或δ序列相互作用的IBS序列而影响自剪接环化过程。本发明的发明人创新性地想到,可以将目标序列的一部分直接视为“IBS”序列,通过修改内含子中的EBS或δ,使其能够与目标序列中被视为“IBS”的区域发生相互作用。这样的方法在保证cRNAzyme构建体具有自剪接功能的同时,摆脱了对外显子序列E1和E2的依赖,将其从构建体和最终环化产物中去除。
仍然以Cte核酶为例,配合不同的目标序列(GFP、Gluc和2A肽)来说明本实施例的cRNAzyme构建体设计思路。
根据各个目标序列两端各6个核苷酸的序列,将II型内含子中的EBS1和EBS1的上游序列(包括δ)分别替换为这两段6核苷酸的序列至少部分互补配对的序列。具体来说,使EBS1的上游序列(包括δ)与目标序列处于线性状态时(如在cRNAzyme构建体自剪接环化之前的状态)5’端的6个核苷酸互补配对,EBS1与目标序列处于线性状态时3’端的6个核苷酸互补配对。具体修饰序列示于更新的图6B右侧小图中,显示了针对三种不同的目标序列设计的用于修饰的EBS1和EBS1的上游序列(包括δ)。值得注意的是,改造过的EBS1和EBS1的上游序列(包括δ)不必与充当“IBS1”和“IBS3”的目标序列片段完美互补配对。可以允许其中有一定比例的错配,或是A和G、G和U这种牢固程度稍低的配对方式。一般来说,用于对II型内含子中的相应序列进行取代的EBS1和EBS1的上游序列(包括δ)与目标序列相应区域在至少60%的核苷酸位置上互补配对,或与目标序列相应区域的互补配对序列至少60%相同。
从电泳结果可以看到,在使用不同目标序列(GFP、Gluc和2A肽)的情况下,该改造方法均有效生成了环形RNA(图6B,左)。通过Sanger测序结果可以确认这种改造可将外源疤痕序列完全消除(图6B,右)。如图6B所示,具有如上修饰的EBS和EBS1的上游序列(包括δ)的cRNAzyme构建体在环化之后,目标序列的两端(带有阴影的两段6核苷酸序列,分别充当“IBS1”和“IBS3”)直接首尾相接,中间不再以E1和E2分隔,这更有利于随后应用生成的环形RNA。将以这种方式改造的构建体称为“无痕”构建体,环化后的环形RNA称为“无痕”RNA。
8.5.实施例5.不同长度目标序列的环化结果
基于实施例2和3中的方法,发明人还尝试了不同长度的目标片段。这些目标片段分别为555个核苷酸的Gluc,其核苷酸序列如SEQ ID NO:9所示;936个核苷酸的Rluc1,其核苷酸序列如SEQ ID NO:10所示;以及1,160个核苷酸的Rluc2,其核苷酸序列如SEQ ID NO:11所示。Gluc和Rluc1的构建体中未加入间隔序列,Rluc2构建体由Cat1IRES序列和Rluc组成。
发现测试的片段均可以高效地进行自剪接,得到环形RNA产物(图7)。
8.6.实施例6.使用本发明的构建体表达目的基因
在此基础上,进一步测试了不同目标序列的环形RNA产物在转染细胞之后的表达情况。
为了尽量减少线性RNA引起的免疫降解,在转染前,对RNA产物进行以下三步处理。
(1)RNase R处理,以消化线性RNA。反应条件是37℃,30min。
(2)HPLC纯化,以去除小的线性RNA。HPLC条件为:凝胶排阻色谱柱:Waters
将目标RNA用lipo RNAmax(Invitrogen)进行转染,转染条件参考供应商的说明书,转染时间为24小时。
采用实施例2和3中的构建方法(采用间隔序列2),并进行了实施例4中描述的可实现无痕环化的修饰,测试了不同目标序列。为了便于检测蛋白的表达,构建了含有荧光蛋白编码序列的构建体包括IRES-GFP(SEQ ID NO:12)和IRES-Gluc(SEQ ID NO:13)。IRES的加入可启动不依赖帽子结构的非经典翻译,使环形RNA中的编码序列能够翻译成蛋白。
针对不同的目标序列,使用了不同的方法来检测蛋白的表达。
对于翻译产物是GFP的情况,通过在显微镜下观察荧光,用RIPA裂解液(Beyotime)裂解细胞,然后通过Western印迹检测蛋白表达。确定通过本发明的方法获得了GFP的表达(图8A)。
对于翻译产物是萤光素酶的情况,先用Passive裂解液(Promega)裂解细胞,然后用萤光素酶检测试剂盒(Promega)通过酶标仪检测蛋白表达。确定通过本发明的方法获得了Gluc蛋白的表达(图8B)。
8.7.实施例7.环形RNA的体外设计与产生
为了有效地产生环形RNA,我们利用II型内含子的自催化剪接反应,II型内含子是主要在细菌和细胞器基因组中发现的移动遗传元件(Lambowitz,A.M.和Zimmerly,S.(2011).Group II introns:mobile ribozymes that invade DNA.Cold Spring HarbPerspect Biol 3,a003616)。所有II型内含子均具有六个结构域(D1至D6),其中结构域1(D1)是最大的结构域并且含有几个短外显子结合位点(EBS)来决定剪接特异性(图12A)。结构域2和3在组装活性内含子结构和刺激剪接反应中起关键作用,而D4是茎环结构,其中长环含有成熟酶的ORF。高度保守的D5是自剪接活性位点的关键(Rybak-Wolf等人(2015).Circular RNAs in the Mammalian Brain Are Highly Abundant,Conserved,andDynamically Expressed.Mol Cell 58,870-885.),而D6含有凸起的腺苷作为分支位点(图12A)。II型内含子的体外自剪接仅需要内含子RNA结构的正确折叠和Mg2+(Peebles,C.L.等人(1986).A self-splicing RNA excises an intron lariat.Cell 44,213-223),然而体内剪接需要成熟酶的辅助(Pyle,A.M.(2016).Group II Intron Self-Splicing.Annu RevBiophys 45,183-205)。
基于该结构域配置,我们从D4处劈分了来自破伤风梭菌的表层蛋白的II型自剪接内含子(McNeil,B.A.,Simon,D.M.和Zimmerly,S.(2014).Alternative splicing of agroup II intron in a surface layer protein gene in Clostridium tetani.NucleicAcids Res 42,1959-1969),生成含有由内含子的上游D5-D6和下游D1-D2-D3侧翼的定制外显子的劈分内含子系统。将D4茎的一部分分离并且放入所得RNA的每个末端,从而形成互补结构以帮助活性内含子的折叠(图12A,绿线)。在内含子和定制外显子的每个接头处均包括两个短的6nt的序列IBS3(内含子结合位点3)和IBS1,以提供与内含子的EBS3和EBS1的远程相互作用。在体外失控转录后,所得RNA前体可以被II型内含子自剪接以产生定制外显子的环形RNA和分支的内含子RNA。通过在目标基因(GOI)的上游包括IRES序列,所得环形RNA可以作为mRNA起作用以通过帽非依赖性翻译来指导蛋白质合成(图12A)。我们将该系统命名为环形编码RNA(CirCode),它可以用作产生任何给定的circRNA进行蛋白质翻译的通用平台。
为了验证该设计,我们将海肾萤光素酶基因的IRES和ORF包括在定制外显子中,并且翻译该D5-D5-外显子-D1。我们发现所得RNA前体确实可以在体外自剪接以产生对应于circRNA的额外条带,而剪接接头的突变(在IBS1处)未能产生circRNA(图1B)。我们进一步使用了两种不同的方法来确认前体RNA下面的另外的条带确实是circRNA,因为它不能通过poly A聚合酶的加尾反应而延伸,并且对RNse R处理的消化具有抗性(图13)。此外,我们从凝胶中纯化了circRNA,并且使用接头的RNase H消化(图14)和直接测序(图15)确认了环形身份。最后,我们通过在IVT步骤之后在环化缓冲液中使用不同浓度的MgCl2和NaCl优化了反应条件(参见方法),并且发现使用10-20mM Mg2+与50-100mM NaCl是RNA环化的最佳反应条件(图5B)。我们还观察到即使当环化缓冲液不含任何Mg2+时,也有一定程度的RNA环化(约30%),这可能是因为在含有24mM MgCl2的IVT缓冲液中共转录地发生了RNA环化。
circRNA产生与翻译的平台优化
先前的使用I型内含子产生circRNA的自剪接系统也将来自T4噬菌体或Anabaena的无关序列引入到最终产物中。该“疤痕”序列通常为约80-180nt长,这限制了目标circRNA的设计灵活性,并且可能在药物开发期间引入一些不想要的效果。我们最初的设计使用了两个短序列(IBS1和IBS3)进行内含子-外显子识别,这留下了较短的12nt的疤痕。为了减少疤痕序列的潜在干扰,我们通过改变D1结构域中的外显子结合位点进一步修改了设计,使EBS1和EBS3分别与环形外显子的3'和5'端形成碱基对(图16,左)。这种新设计使得能够生成没有无关序列的“无痕circRNA”。唯一的序列要求是环形外显子5'端的“AG”,这使得环形RNA的设计完全可编程。我们通过生成编码EGFP和Rluc-P2A的两种不同的circRNA来验证该设计(图16,右),并且观察在不同离子条件下无痕circRNA的有效环化。通过最终circRNA产物的测序进一步证实了circRNA的无痕自剪接(图16,右下)。总之,我们设计出了CirCode系统,其可以有效地产生任何序列的无痕circRNA,为使用circRNA作为不同的治疗工具提供了通用平台。
先前使用PIE方法进行的研究表明,在IRES前添加短间隔区可能有助于IRES的正确折叠和/或内含子的活性结构(Wesselhoeft,R.A.等人,2018,同上),因此我们在环形外显子的每个末端引入了几种形式的间隔序列,以优化它们的环化效率(图17)。我们发现不同的间隔子确实影响RNA环化效率,其范围从47%(对于SP5)到83%(对于SP4)。此外,我们以不同剂量将两种具有不同间隔子的circRNA转染到三个细胞系中,并且观察到如通过萤光素酶活性测定判断的蛋白质产生的剂量依赖性增加(图18)。有趣的是,间隔序列对翻译效率的影响似乎很小,并且取决于circRNA剂量和细胞系。我们进一步检查了circRNA转染后蛋白质产生的时间过程,并且发现可以在转染后六小时内检测到活性蛋白,并且在24小时达到表达峰值(图19)。
8.8.实施例8.circRNA介导延长的蛋白质产生
环形mRNA的主要优点是由于缺乏自由端而具有极佳的稳定性,因此与线性对应物相比,对于蛋白质表达,circRNA应该具有良好的保质期。为了直接测试这一点,我们合成了编码Gaussia萤光素酶(Gluc)的线性和环形mRNA,并且在将它们转染到293T细胞中前,将它们平行地在室温下在纯水中储存不同天数。我们发现circRNA指导蛋白质翻译的活性在两周内基本上没有变化,而线性mRNA到储存的第三天失去了大约一半的活性(图3A),表明circRNA可以在室温下稳定储存。此外,我们发现在转染到细胞中后,来自线性mRNA的蛋白质产生迅速下降,在第2天下降>80%,而来自circRNA的翻译持续5-7天(图3B)。延长的蛋白质翻译也与我们先前使用反向剪接的circRNA报告物的结果(Wang,Y.和Wang,Z.(2015).Efficient backsplicing produces translatable circular mRNAs.RNA 21,172-179)一致。还值得注意的是,在该实验中,来自circRNA的蛋白质产生也可能在结束时受到抑制,因为我们在整个实验的一周内都没有改变培养基(图16)。
我们将来自线性mRNA和使用PIE方案或新的CirCode系统产生的circRNA的蛋白质产生进行了进一步比较。使用IVT生成了具有相同Gluc编码序列的加帽的和未经修饰的线性mRNA,并且与含有CVB3 IRES和Gluc ORF的两种类型的circRNA平行地转染到293T细胞中。我们发现与线性mRNA相比,来自两种circRNA的蛋白质表达更稳健(图23,上),支持了先前使用PIE circRNA的报道(Wesselhoeft,R.A.等人,2018,同上)。当我们在6天的时间跨度内测量积累的萤光素酶活性时,在circRNA中观察到蛋白质产生的甚至更大的增加(图23,下),这可能是因为circRNA的优异稳定性。此外,从两种不同的方案生成的circRNA在指导蛋白质翻译方面显示出相似的能力(图24)。
8.9.实施例9.纯化的circRNA可以指导目标蛋白的稳健翻译
发现mRNA纯度是蛋白质产生和诱导先天免疫的关键因素,因为通过HPLC去除dsRNA可以消除免疫激活并且改善线性核苷修饰的mRNA的翻译(Kariko,K.等人,(2011).Generating the optimal mRNA for therapy:HPLC purification eliminates immuneactivation and improves translation of nucleoside-modified,protein-encodingmRNA.Nucleic Acids Res 39,e142)。然而,关于circRNA的免疫原性存在一些争论。虽然早期的报道表明,体外合成的circRNA比线性RNA更容易诱导细胞免疫反应(Chen,Y.G.等人,(2017).Sensing Self and Foreign Circular RNAs by Intron Identity.Mol Cell 67,228-238e225),但后来报道了从IVT和环化反应的副产物(包括dsRNA、线性RNA片段和三磷酸RNA)中纯化circRNA可以消除circRNA的细胞毒性和免疫原性(Wesselhoeft,R.A.等人,(2019).RNACircularization Diminishes Immunogenicity and Can ExtendTranslation Duration In Vivo.Mol Cell 74,508-520e504;Breuer,J.等人,(2022).What goes around comes around:artificial circular RNAs bypass cellularantiviral responses.Mol Ther Nucleic Acids)。最近的研究还表明,序列身份和结构是circRNA细胞免疫的主要决定因素,因为通过不同方法产生的circRNA显示出不同的免疫原性(Liu,C.X.等人,(2022).RNA circles with minimized immunogenicity as potentPKR inhibitors.Mol Cell82,420-434e426.)。为了检查使用CirCode平台产生的circRNA是否可以诱导先天免疫反应和细胞毒性,我们用凝胶纯化或HPLC纯化了circRNA(图3D),并且测量了circRNA是否可以在circRNA转染后诱导细胞免疫反应。我们发现,与模拟转染相比,未纯化的circRNA的转染导致大量细胞死亡,而纯化的circRNA没有显示出可检测的细胞毒性(图25)。此外,与通过诱导RIG-I和IFN-B1(干扰素-β1)刺激先天免疫反应的未纯化的circRNA相比,纯化的circRNA显著降低了免疫原性(图26),支持了先前的观察结果,即circRNA的免疫原性主要是由IVT反应的副产物引起的(Wesselhoeft,R.A.等人,2019,同上)。
8.10.实施例10.LNP包封的circRNA在小鼠中指导稳健的蛋白质产生
circRNA的治疗应用的一个重要问题是,circRNA的产生是否可以可靠地放大,以及不同生产批次之间的再现性如何。由于CirCode平台使用自剪接内含子进行RNA环化,而没有RNA连接酶或其他共激活剂的参与,因此放大程序相对简单。为了测试该系统的可扩展性,我们将IVT和环化反应系统扩大了50倍(从20μl到1ml),并且发现高环化效率(约70%)基本上保持不变,而RNA产物的总量在单次反应中达到7.5mg(图27)。此外,我们发现circRNA的IVT/环化和HPLC纯化在不同批次之间是高度可再现的(图28),为circRNA的体内应用奠定了基础。
我们进一步生成了用脂质纳米颗粒(LNP)包封的circRNA用于它们的体内递送(图29)。水溶液中的circRNA被可电离的阳离子脂质填充,其与其他脂质组分(如DMG-PEG2000和胆固醇)形成纳米颗粒(参见方法),实现了约95%的包封效率,有效直径为约80nm。我们在我们的配制品中测试了三种不同的可电离阳离子脂质以包封编码Gluc的circRNA,并且通过肌内(IM)或腹膜内(IP)注射将所得LNP注射到BALC/c小鼠中(每个实验组n=3)。在该实验中测试了使用不同的可电离阳离子脂质(MC3、SM-102和ALC-0315)的三种配制品。使用血清萤光素酶发光测定(图30)或动物的生物发光成像(图31)测定了萤光素酶的表达。我们从两种不同的LNP-circRNA配制品中发现了萤光素酶的稳健表达,表明通过我们的程序产生的circRNA可以可靠地诱导体内蛋白质表达。
8.11.实施例11.SARS-coV-2的潜在circRNA疫苗的生成
我们通过改造编码来自SARS-cov-2的S蛋白(其可以潜在地用于生产mRNA疫苗)的受体结合结构域(RBD)的circRNA进一步测试了circRNA在mRNA疗法中的应用。根据先前的报道,将两种不同的抗原设计构建到circRNA中(图32,上)(Dai,L.和Gao,G.F.(2021).Viral targets for vaccines against COVID-19.Nat Rev Immunol 21,73-82)。第一种使用了与来自T4次要纤维蛋白(fibritin)的折叠子(其能够实现RBD的三聚化)融合的单个RBD(Meier,S.等人,(2004).Foldon,the natural trimerization domain of T4fibritin,dissociates into a monomeric A-state formcontaining a stable beta-hairpin:atomic details of trimer dissociation and local beta-hairpinstability from residual dipolar couplings.J Mol Biol 344,1051-1069)。另一种使用在试验研究中显示诱导强抗体产生的RBD二聚体(Dai,L.等人,(2020).AUniversalDesign of Betacoronavirus Vaccines against COVID-19,MERS,and SARS.Cell 182,722-733e711;Dai,L.等人,2021,同上)。我们用经修饰的IRES设计了两种蛋白质的编码序列,并且构建了circRNA载体。验证了所得circRNA的产生和纯化(图32,左下),并且通过转染到293细胞中进一步验证了并通过western印迹抗原产生检测了circRNA编码的蛋白质的翻译(图32,右下)。
我们使用了两种不同的配制品来产生LNP-circRNA颗粒,并且实现了高包封效率(>90%),典型的纳米颗粒尺寸为90-100nm(图33显示了使用SM-102配制品的代表性结果)。将LNP-circRNA用两次分开的IM注射接种到BALB/c小鼠中,并且在每次接种后2周收集血液样品用于进一步分析(图34)。我们测试了几种配制品和circRNA设计,然而为了更好地与所公开的结果进行比较,显示了使用SM-102配制品的LNP-circRNA-RBP的结果。使用流式细胞术,我们发现大部分记忆B细胞(即,CD19+CD27+B淋巴细胞)在第二次接种后两周具有强烈的RBD抗体表达(图35、36),指示抗原诱导的持久免疫反应。具体来说,>70%的CD19+CD27+B细胞表达了RBD特异性抗体(即,RBD+),并且约50%的CD19+CD27+B细胞也呈IgD+RBD+(图36),表明强烈的抗体反应。作为对照,单独注射LNP不会诱导RBD抗体的激活,表明接种后会产生特异性反应。
为了测试RBD抗体在小鼠血清中的活性,我们接下来进行了抗体阻断测定以检查小鼠血清是否可以阻断荧光标记的RDB与稳定表达ACE2的293T细胞的结合(图37)。我们发现,血清可以以适度的稀释度(1:20)有效地阻断所有RBD变体与细胞表面的结合。鉴于我们使用了相当高浓度的RBD(0.5μg/ml),这种保护非常令人印象深刻。即使以非常高的稀释比率(1:200),血清仍然完全阻断了野生型RBD的结合并且显著降低了德尔塔变体的RBD结合。然而,血清没有阻断奥米克戎RBD的结合,这与目前基于野生型S蛋白的mRNA疫苗针对奥米克戎变体的保护较弱的发现是一致的。
我们进一步测量了每次接种后针对RDB的中和抗体的滴度,并且发现了针对RBD的IgG产生的稳健产生(图38-39)。抗体滴度在第一次注射后两周接近105,并且在第二次注射后达到接近108,这是使用具有类似配制品的LNP包封的线性mRNA或使用另一种circRNA设计所报道的小鼠结果的至少10倍。此外,IgG2/IgG1的比率约为0.75,这类似于先前使用线性mRNA疫苗报道的。最后,我们使用SARS-CoV-2假病毒的保护测定测量了抗体滴度,并且发现具有接近105的滴度的强保护(图40)。总的来说,我们在小鼠模型中的初步数据表明,CirCode平台在设计和生成针对SARS-CoV-2的circRNA疫苗方面具有强大的性能,可以将其潜在地扩展到其他病毒病原体的疫苗开发。
质粒构建
从GENEWIZ化学合成了破伤风梭菌(CTE)中的II型内含子和IRES序列的片段,并且通过PCR扩增了不同的蛋白编码片段。通过Gibson组装将这些片段克隆到含有T7 RNA聚合酶启动子和终止子的NheI和XbaI消化的骨架中。
RNA合成和环化
在未经修饰的NTP的存在下,使用T7 RiboMAX
实施例14.circRNA鉴定
对于poly A加尾和RNase R处理,将来自IVT的总RNA用RNA净化柱纯化,然后按照制造说明书用大肠杆菌(E.coli)Poly A聚合酶(NEB,M0276S)处理。该步骤将在未剪接的RNA前体的自由端添加poly A尾。Poly A加尾后,将纯化的RNA按照制造商的说明书用RNaseR核糖核酸外切酶(Lucigen,RNR07520)消化,并且通过柱纯化富集的circRNA。
对于RNase H产切口测定,我们将24nt ssDNA探针以1:20比率进行了孵育,将富含RNase R的circRNA在65℃下加热5min。将RNase H缓冲液立即添加到DNA-RNA混合物中。然后,将混合物缓慢冷却到室温。退火后,在37℃下将RNase H(Thermo Scientific,EN0201)添加到混合物中持续20min。ssDNA探针的序列为5’-TGGTGCTCGTAGGAGTAGTGAAAG-3’。
RNA凝胶电泳与纯化
使用冰冷的DEPC处理的MOPS缓冲液使RNA在120V下在低熔点琼脂糖凝胶(sigmaAldrich,A4018)上跑胶。电泳后,将circRNA泳道切下并且用Zymoclean凝胶RNA回收试剂盒(ZYMO research,R1011)纯化。转染前,将柱纯化的circRNA用磷酸酶(NEB,M0525S)处理,以去除可能产生免疫原性的潜在的5'磷酸。
circRNA的翻译产物的测量
在转染前一天将细胞接种到24孔板中。根据制造商的手册,使用LipofectamineMessenger Max(Invitrogen,LMRNA001)将纯化的circRNA转染到细胞中。转染后,将细胞在37℃下培养24h。收集细胞裂解物和上清液用于使用
环形RNA的RT-PCR
用琼脂糖凝胶对IVT后柱纯化的RNA进行电泳。将对应于circRNA的条带切离并且使用Zymoclean凝胶RNA提取试剂盒(Zymogen,R1011)提取。使用具有随机引物的PrimeScript RT试剂盒(TAKARA,RR037B)将纯化的RNA逆转录为cDNA,然后采用可以扩增跨越剪接接头的转录产物的引物进行PCR。对PCR产物进行Sanger测序以确认环形RNA的反向剪接。
环形RNA的HPLC纯化
为了获得高质量的circRNA,用高效液相色谱分辨来自IVT的旋转柱纯化的DNaseI处理的RNA。为了用SHIMADZ LC-20A(日本京都)进行半制备,将40μg RNA装载到4.6×300mm的尺寸排阻柱(Waters XBridge,BEH450A,
用毛细管电泳分析环形RNA
在RNA模式下,用Agilent 2100生物分析仪用毛细管电泳进一步分析从HPLC纯化的环形RNA。将样品稀释至适当的浓度,并且根据制造说明书进行分析。
LNP产生方法
如先前所述(Corbett,K.S.等人,(2020).SARS-CoV-2mRNA vaccine designenabled by prototype pathogen preparedness.Nature 586,567-571;Polack,F.P.等人,(2020).Safety and Efficacy of the BNT162b2mRNACovid-19 Vaccine.N Engl JMed 383,2603-2615),经由NanoAssemblr Ignite系统将环形RNA包封在脂质纳米颗粒中。简言之,将pH 4.0的circRNA水溶液与溶解于乙醇中的脂质混合物快速混合,所述脂质混合物含有不同的可电离阳离子脂质、二硬脂酰磷脂酰胆碱(DSPC)、DMG-PEG2000和胆固醇。脂质混合物的比率为:对于配制品1,MC3:DSPC:胆固醇:PEG-2000=50:10:38.5:1.5;对于配制品2,SM-102:DSPC:胆固醇:PEG-2000=50:10:38.5:1.5;对于配制品3,ALC-0315:DSPC:胆固醇:ALC-0159=46.3:9.4:42.7:1.6。然后将所得LNP混合物用PBS透析,并且在-80℃下以0.5μg/μl的浓度储存用于进一步应用。
将LNP-circRNA施用至小鼠
8周龄的雌性BALB/C小鼠购自Shanghai Model Organisms Center。将在PBS中的20μg的CircRNA-LNP用3/10胰岛素注射器(BD biosciences)肌内施用至小鼠。施用LNP后24小时收集血清,并且将50μl血清用于体外萤光素酶活性测定。用IVIS光谱仪(RoperScientific)进行生物发光成像。Gluc-LNP注射后24小时,将2mg/kg的腔肠素(Coelenterazine)(MedChemExpress,MCE)腹膜内施用至小鼠。然后将小鼠在接受底物后在室中用2.5%异氟醚(RWD Life Science Co.)麻醉,并且放置在成像平台上,同时经由鼻锥维持接受2%异氟醚。在底物注射后5分钟以30秒的曝光时间对小鼠进行成像,以确保有效且充分地获得信号。
对于免疫原性研究,使用了8周龄的雌性BALB/c小鼠(Shanghai Model OrganismsCenter,lnc)。对于初免和加强注射,将10μg的CircRNA-RBD-LNP稀释于50μl 1XPBS中,并且肌内施用至小鼠相同的后腿。对照组的小鼠接受了PBS和空LNP。在初免和加强注射后2周,收集血液样品。还在终点(加强后2周)收获小鼠脾脏进行免疫染色和流式细胞术。
序列表
<110>上海环码生物医药有限公司
<120>制备环形RNA的构建体、方法及其用途
<130>TPG03550A-CS12901CCD33CN
<150>CN202110594352.4
<151>2021-05-28
<160>134
<170>PatentIn version 3.5
<210>1
<211>989
<212>DNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>1
caaaggctta cctatcacta gcgcgacacg ttcctaagtg aaaagcttag gcactgtcga60
actcaacagt tcagcagtga actgtcattc taagaagtca aatgaaggag taacgtctgg 120
aagggcttcc cttaatcctc cgacatgcag gaaagtaggc aagtactgaa ctgtgtgaag 180
ctcggtgaag tcggttgaag gttaccgtaa attagtatct ctaatacgaa agctatccag 240
cggtggatgg tgtaactgat agaccggagg tctataaaac actcaaggtt aggatgcgcg 300
atgaactaga ggcgatcgct agtaagcgca gacgaatccc tgatggtacg ggtctatatc 360
gggagggaat cgaaaggttc tctgacacaa ataagtgtcg ctactgtggg tgagtaaaac 420
tctcctttat gaaagcccat atatcgttac aggcgttatt aaggtagcag gctcataggg 480
gaaacctaaa agtgtatgta cagataagaa tgacggaacg tggtaagctg ccgacatgga 540
gggcttgttc tctttgaagt gttgccaagg aaagtcacaa tgagattagt tgtcgatata 600
acttggttta acggcagtga aagtggtggc acagtaccga tgaaacgtgt aatgaacgtg 660
gagggatagc cactagtcga ttgaagattg aaggttacta ttggttaaca tggtttcgag 720
taagactaag agatgtaatg ctccaaagta ataaggaggt tacagcccat gttaaagaaa 780
accaagctaa gacataacga atattatgat acacaaaaaa agtgtatgac aatttatact 840
cgaacagtct taacggtaac aatttctttc aattggaaac gatggaacgc cgtatgcccg 900
gaaacgggcg cgtacggtgt ggagtggggg aaaagctgga gataatctca aaggcttacc 960
tatcactatc gcgacacgtt cctaagtga 989
<210>2
<211>1028
<212>DNA
<213>破伤风梭菌
<400>2
gccatacaat aaaagtgcga aacgttatcc tataagtaag aaagttttaa aattttctta60
cgaaaaggat agaacttaaa agttctaact gttctactaa agtaataagt gaaaatctta 120
tttaaagcaa acaaccaagt agctttaagt ctaagtcccc tacacaagtt ttatactact 180
atgcaaaact tgtgaagcta ggtaaggtcg taatccgtga aagtcggatg cggggctcct 240
taaaagatta ctatggtaaa cataagctaa tccattaaga tgcgatttat atgtatttta 300
tactgttaaa tatttttgtg cttgtggctt ggtataaaac agttaagatg aagtacttaa 360
ctggttttgg aataattggt tgttaaacta aaacattata aatcgttagt ggatacctaa 420
ggtaatcaaa aatagggata ggtagaatgg aacgtttgat gctgtatatg aagaggttta 480
gtagaaccta ggacacatat acgggctcag caggttcata gtagctatga tactcagccg 540
gaagtcaatt aattttgaaa tacttctatg gtaacatagg agaaggataa aactgagtga 600
gccaaggaac ctagtcggta atagaaaagt ggaagttaaa acaaatataa gattttagaa 660
ttaatttaat taatgaacgg aattaattta atgatattta aagttagacg gttataaatt 720
aaacatttca aaattaaacc atatccaaat tcataaatat agctagatca tatcactagt 780
ttaaaaataa ataaatcatt tcaaattact attaagtaag gtattaatac cttacttaat 840
agtaatctca ttacataaga gaattactag attagcagac agattcataa aaactatatc 900
aactaggaca atagaaaata tatttataca cttcctatta tcgagcgaac gccttatgcg 960
atgaaagtcg cacgtagggt gtagaccaag cgaaatccta tgcatttagg atagtgaggt1020
atagcaaa 1028
<210>3
<211>1028
<212>DNA
<213>人工序列
<220>
<223>II型内含子Cte_mut
<400>3
gcaagacaat aaaagtttga cacgtgatcc tataagtaag aaagttttaa aattttctta60
cgaaaaggat agaacttaaa agttctaact gttctactaa agtaataagt gaaaatctta 120
tttaaagcaa acaaccaagt agctttaagt ctaagtcccc tacacaagtt ttatactact 180
atgcaaaact tgtgaagcta ggtaaggtcg taatccgtga aagtcggatg cggggctcct 240
taaaagatta ctatggtaaa cataagctaa tccattaaga tgcgatttat atgtatttta 300
tactgttaaa tatttttgtg cttgtggctt ggtataaaac agttaagatg aagtacttaa 360
ctggttttgg aataattggt tgttaaacta aaacattata aatcgttagt ggatacctaa 420
ggtaatcaaa aatagggata ggtagaatgg aacgtttgat gctgtatatg aagaggttta 480
gtagaaccta ggacacatat acgggctcag caggttcata gtagctatga tactcagccg 540
gaagtcaatt aattttgaaa tacttctatg gtaacatagg agaaggataa aactgagtga 600
gccaaggaac ctagtcggta atagaaaagt ggaagttaaa acaaatataa gattttagaa 660
ttaatttaat taatgaacgg aattaattta atgatattta aagttagacg gttataaatt 720
aaacatttca aaattaaacc atatccaaat tcataaatat agctagatca tatcactagt 780
ttaaaaataa ataaatcatt tcaaattact attaagtaag gtattaatac cttacttaat 840
agtaatctca ttacataaga gaattactag attagcagac agattcataa aaactatatc 900
aactaggaca atagaaaata tatttataca cttcctatta tcgagcgaac gccttatgcg 960
atgaaagtcg cacgtagggt gtagaccaag cgaaatccta tgcatttagg atagtgaggt1020
atagcaaa 1028
<210>4
<211>60
<212>DNA
<213>人工序列
<220>
<223>间隔序列1
<400>4
gcaatagccg aaaaacaaaa aacaaaaaaa acaaaaaaaa aaccaaaaaa acaaaacaca60
<210>5
<211>20
<212>DNA
<213>人工序列
<220>
<223>间隔序列2
<400>5
aaattataat aattataata20
<210>6
<211>224
<212>DNA
<213>人工序列
<220>
<223>间隔序列3
<400>6
atgaaaccgg ctcggattcc gcccgcgtgc gccatcccct cagctagcag gtgtgagcgg60
ctttctgccc gcagtctcta cacagctcag catcctgacg cctcctcccc ttgcaggggc 120
gtgaagctac ttcagactct gctgtgacga cttggccgcc aggcaccgat cctccccggt 180
gagaaggtcc acgaatctta ctgcagacag atttgctcag cgcg224
<210>7
<211>24
<212>DNA
<213>人工序列
<220>
<223>探针1
<400>7
ctttcactac tcctacgagc acca 24
<210>8
<211>25
<212>DNA
<213>人工序列
<220>
<223>探针2
<400>8
gaccatgctc ccaagcaaga tcatg25
<210>9
<211>555
<212>DNA
<213>人工序列
<220>
<223>Gluc
<400>9
atgggagtca aagttctgtt tgccctgatc tgcatcgctg tggccgaggc caagcccacc60
gagaacaacg aagacttcaa catcgtggcc gtggccagca acttcgcgac cacggatctc 120
gatgctgacc gcgggaagtt gcccggcaag aagctgccgc tggaggtgct caaagagatg 180
gaagccaatg cccggaaagc tggctgcacc aggggctgtc tgatctgcct gtcccacatc 240
aagtgcacgc ccaagatgaa gaagttcatc ccaggacgct gccacaccta cgaaggcgac 300
aaagagtccg cacagggcgg cataggcgag gcgatcgtcg acattcctga gattcctggg 360
ttcaaggact tggagcccat ggagcagttc atcgcacagg tcgatctgtg tgtggactgc 420
acaactggct gcctcaaagg gcttgccaac gtgcagtgtt ctgacctgct caagaagtgg 480
ctgccgcaac gctgtgcgac ctttgccagc aagatccagg gccaggtgga caagatcaag 540
ggggccggtg gtgac555
<210>10
<211>936
<212>DNA
<213>人工序列
<220>
<223>Rluc1
<400>10
atggcttcca aggtgtacga ccccgagcaa cgcaaacgca tgatcactgg gcctcagtgg60
tgggctcgct gcaagcaaat gaacgtgctg gactccttca tcaactacta tgattccgag 120
aagcacgccg agaacgccgt gatttttctg catggtaacg ctgcctccag ctacctgtgg 180
aggcacgtcg tgcctcacat cgagcccgtg gctagatgca tcatccctga tctgatcgga 240
atgggtaagt ccggcaagag cgggaatggc tcatatcgcc tcctggatca ctacaagtac 300
ctcaccgctt ggttcgagct gctgaacctt ccaaagaaaa tcatctttgt gggccacgac 360
tggggggctt gtctggcctt tcactactcc tacgagcacc aagacaagat caaggccatc 420
gtccatgctg agagtgtcgt ggacgtgatc gagtcctggg acgagtggcc tgacatcgag 480
gaggatatcg ccctgatcaa gagcgaagag ggcgagaaaa tggtgcttga gaataacttc 540
ttcgtcgaga ccatgctccc aagcaagatc atgcggaaac tggagcctga ggagttcgct 600
gcctacctgg agccattcaa ggagaagggc gaggttagac ggcctaccct ctcctggcct 660
cgcgagatcc ctctcgttaa gggaggcaag cccgacgtcg tccagattgt ccgcaactac 720
aacgcctacc ttcgggccag cgacgatctg cctaagatgt tcatcgagtc cgaccctggg 780
ttcttttcca acgctattgt cgagggagct aagaagttcc ctaacaccga gttcgtgaag 840
gtgaagggcc tccacttcag ccaggaggac gctccagatg aaatgggtaa gtacatcaag 900
agcttcgtgg agcgcgtgct gaagaacgag cagtaa 936
<210>11
<211>1160
<212>DNA
<213>人工序列
<220>
<223>Rluc2
<400>11
atgaaaccgg ctcggattcc gcccgcgtgc gccatcccct cagctagcag gtgtgagcgg60
ctttctgccc gcagtctcta cacagctcag catcctgacg cctcctcccc ttgcaggggc 120
gtgaagctac ttcagactct gctgtgacga cttggccgcc aggcaccgat cctccccggt 180
gagaaggtcc acgaatctta ctgcagacag atttgctcag cgcgatggct tccaaggtgt 240
acgaccccga gcaacgcaaa cgcatgatca ctgggcctca gtggtgggct cgctgcaagc 300
aaatgaacgt gctggactcc ttcatcaact actatgattc cgagaagcac gccgagaacg 360
ccgtgatttt tctgcatggt aacgctgcct ccagctacct gtggaggcac gtcgtgcctc 420
acatcgagcc cgtggctaga tgcatcatcc ctgatctgat cggaatgggt aagtccggca 480
agagcgggaa tggctcatat cgcctcctgg atcactacaa gtacctcacc gcttggttcg 540
agctgctgaa ccttccaaag aaaatcatct ttgtgggcca cgactggggg gcttgtctgg 600
cctttcacta ctcctacgag caccaagaca agatcaaggc catcgtccat gctgagagtg 660
tcgtggacgt gatcgagtcc tgggacgagt ggcctgacat cgaggaggat atcgccctga 720
tcaagagcga agagggcgag aaaatggtgc ttgagaataa cttcttcgtc gagaccatgc 780
tcccaagcaa gatcatgcgg aaactggagc ctgaggagtt cgctgcctac ctggagccat 840
tcaaggagaa gggcgaggtt agacggccta ccctctcctg gcctcgcgag atccctctcg 900
ttaagggagg caagcccgac gtcgtccaga ttgtccgcaa ctacaacgcc taccttcggg 960
ccagcgacga tctgcctaag atgttcatcg agtccgaccc tgggttcttt tccaacgcta1020
ttgtcgaggg agctaagaag ttccctaaca ccgagttcgt gaaggtgaag ggcctccact1080
tcagccagga ggacgctcca gatgaaatgg gtaagtacat caagagcttc gtggagcgcg1140
tgctgaagaa cgagcagtaa1160
<210>12
<211>1061
<212>DNA
<213>人工序列
<220>
<223>IRES-EgFP
<400>12
agcaaagacc ccaacgagaa gcgcgatcac atggtcctgc tggagttcgt gaccgccgcc60
gggatcactc tcggcatgga cgagctgtac aagtaacaaa caaacaaaac aaaaacactc 120
ccctgtgagg aactactgtc ttcacgcaga aagcgtctag ccatggcgtt agtatgagtg 180
tcgtgcagcc tccaggaccc cccctcccgg gagagccata gtggtctgcg gaaccggtga 240
gtacaccgga attgccagga cgaccgggtc ctttcttgga taaacccgct caatgcctgg 300
agatttgggc gtgcccccgc aagactgcta gccgagtagt gttgggtcgc gaaaggcctt 360
gtggtactgc ctgatagggt gcttgcgagt gccccgggag gtctcgtaga ccgtgcacca 420
tgagcacgaa tcctaaaatg gtgagcaagg gcgaggagct gttcaccggg gtggtgccca 480
tcctggtcga gctggacggc gacgtaaacg gccacaagtt cagcgtgtcc ggcgagggcg 540
agggcgatgc cacctacggc aagctgaccc tgaagttcat ctgcaccacc ggcaagctgc 600
ccgtgccctg gcccaccctc gtgaccaccc tgacctacgg cgtgcagtgc ttcagccgct 660
accccgacca catgaagcag cacgacttct tcaagtccgc catgcccgaa ggctacgtcc 720
aggagcgcac catcttcttc aaggacgacg gcaactacaa gacccgcgcc gaggtgaagt 780
tcgagggcga caccctggtg aaccgcatcg agctgaaggg catcgacttc aaggaggacg 840
gcaacatcct ggggcacaag ctggagtaca actacaacag ccacaacgtc tatatcatgg 900
ccgacaagca gaagaacggc atcaaggtga acttcaagat ccgccacaac atcgaggacg 960
gcagcgtgca gctcgccgac cactaccagc agaacacccc catcggcgac ggccccgtgc1020
tgctgcccga caaccactac ctgagcaccc agtccgccct g1061
<210>13
<211>899
<212>DNA
<213>人工序列
<220>
<223>IRES-gluc
<400>13
agcagttcat cgcacaggtc gatctgtgtg tggactgcac aactggctgc ctcaaagggc60
ttgccaacgt gcagtgttct gacctgctca agaagtggct gccgcaacgc tgtgcgacct 120
ttgccagcaa gatccagggc caggtggaca agatcaaggg ggccggtggt gactaacaaa 180
caaacaaaac aaaaacactc ccctgtgagg aactactgtc ttcacgcaga aagcgtctag 240
ccatggcgtt agtatgagtg tcgtgcagcc tccaggaccc cccctcccgg gagagccata 300
gtggtctgcg gaaccggtga gtacaccgga attgccagga cgaccgggtc ctttcttgga 360
taaacccgct caatgcctgg agatttgggc gtgcccccgc aagactgcta gccgagtagt 420
gttgggtcgc gaaaggcctt gtggtactgc ctgatagggt gcttgcgagt gccccgggag 480
gtctcgtaga ccgtgcacca tgagcacgaa tcctaaaatg ggagtcaaag ttctgtttgc 540
cctgatctgc atcgctgtgg ccgaggccaa gcccaccgag aacaacgaag acttcaacat 600
cgtggccgtg gccagcaact tcgcgaccac ggatctcgat gctgaccgcg ggaagttgcc 660
cggcaagaag ctgccgctgg aggtgctcaa agagatggaa gccaatgccc ggaaagctgg 720
ctgcaccagg ggctgtctga tctgcctgtc ccacatcaag tgcacgccca agatgaagaa 780
gttcatccca ggacgctgcc acacctacga aggcgacaaa gagtccgcac agggcggcat 840
aggcgaggcg atcgtcgaca ttcctgagat tcctgggttc aaggacttgg agcccatgg899
<210>14
<211>32
<212>DNA
<213>人工序列
<220>
<223>臂1
<400>14
aataccttac ttaatagtaa caatagaaaa tc32
<210>15
<211>33
<212>DNA
<213>人工序列
<220>
<223>臂2
<400>15
aagctagatc atattactat taagtaaggt att 33
<210>16
<211>1719
<212>DNA
<213>人工序列
<220>
<223>cRNAzyme (Cte-Rluc)
<400>16
aataccttac ttaatagtaa caatagaaaa tcctattatc gagcgaacgc cttatgcgat60
gaaagtcgca cgtagggtgt agaccaagcg aaatcctatg catttaggat agtgaggtat 120
agcaaaatgg cttccaaggt gtacgacccc gagcaacgca aacgcatgat cactgggcct 180
cagtggtggg ctcgctgcaa gcaaatgaac gtgctggact ccttcatcaa ctactatgat 240
tccgagaagc acgccgagaa cgccgtgatt tttctgcatg gtaacgctgc ctccagctac 300
ctgtggaggc acgtcgtgcc tcacatcgag cccgtggcta gatgcatcat ccctgatctg 360
atcggaatgg gtaagtccgg caagagcggg aatggctcat atcgcctcct ggatcactac 420
aagtacctca ccgcttggtt cgagctgctg aaccttccaa agaaaatcat ctttgtgggc 480
cacgactggg gggcttgtct ggcctttcac tactcctacg agcaccaaga caagatcaag 540
gccatcgtcc atgctgagag tgtcgtggac gtgatcgagt cctgggacga gtggcctgac 600
atcgaggagg atatcgccct gatcaagagc gaagagggcg agaaaatggt gcttgagaat 660
aacttcttcg tcgagaccat gctcccaagc aagatcatgc ggaaactgga gcctgaggag 720
ttcgctgcct acctggagcc attcaaggag aagggcgagg ttagacggcc taccctctcc 780
tggcctcgcg agatccctct cgttaaggga ggcaagcccg acgtcgtcca gattgtccgc 840
aactacaacg cctaccttcg ggccagcgac gatctgccta agatgttcat cgagtccgac 900
cctgggttct tttccaacgc tattgtcgag ggagctaaga agttccctaa caccgagttc 960
gtgaaggtga agggcctcca cttcagccag gaggacgctc cagatgaaat gggtaagtac1020
atcaagagct tcgtggagcg cgtgctgaag aacgagcagt aagccataca ataaaagtgc1080
gaaacgttat cctataagta agaaagtttt aaaattttct tacgaaaagg atagaactta1140
aaagttctaa ctgttctact aaagtaataa gtgaaaatct tatttaaagc aaacaaccaa1200
gtagctttaa gtctaagtcc cctacacaag ttttatacta ctatgcaaaa cttgtgaagc1260
taggtaaggt cgtaatccgt gaaagtcgga tgcggggctc cttaaaagat tactatggta1320
aacataagct aatccattaa gatgcgattt atatgtattt tatactgtta aatatttttg1380
tgcttgtggc ttggtataaa acagttaaga tgaagtactt aactggtttt ggaataattg1440
gttgttaaac taaaacatta taaatcgtta gtggatacct aaggtaatca aaaataggga1500
taggtagaat ggaacgtttg atgctgtata tgaagaggtt tagtagaacc taggacacat1560
atacgggctc agcaggttca tagtagctat gatactcagc cggaagtcaa ttaattttga1620
aatacttcta tggtaacata ggagaaggat aaaactgagt gagccaagga acctagtcgg1680
taatagaagc tagatcatat tactattaag taaggtatt 1719
<210>17
<211>989
<212>RNA
<213>苏云金芽孢杆菌
<400>17
caaaggcuua ccuaucacua gcgcgacacg uuccuaagug aaaagcuuag gcacugucga60
acucaacagu ucagcaguga acugucauuc uaagaaguca aaugaaggag uaacgucugg 120
aagggcuucc cuuaauccuc cgacaugcag gaaaguaggc aaguacugaa cugugugaag 180
cucggugaag ucgguugaag guuaccguaa auuaguaucu cuaauacgaa agcuauccag 240
cgguggaugg uguaacugau agaccggagg ucuauaaaac acucaagguu aggaugcgcg 300
augaacuaga ggcgaucgcu aguaagcgca gacgaauccc ugaugguacg ggucuauauc 360
gggagggaau cgaaagguuc ucugacacaa auaagugucg cuacuguggg ugaguaaaac 420
ucuccuuuau gaaagcccau auaucguuac aggcguuauu aagguagcag gcucauaggg 480
gaaaccuaaa aguguaugua cagauaagaa ugacggaacg ugguaagcug ccgacaugga 540
gggcuuguuc ucuuugaagu guugccaagg aaagucacaa ugagauuagu ugucgauaua 600
acuugguuua acggcaguga aagugguggc acaguaccga ugaaacgugu aaugaacgug 660
gagggauagc cacuagucga uugaagauug aagguuacua uugguuaaca ugguuucgag 720
uaagacuaag agauguaaug cuccaaagua auaaggaggu uacagcccau guuaaagaaa 780
accaagcuaa gacauaacga auauuaugau acacaaaaaa aguguaugac aauuuauacu 840
cgaacagucu uaacgguaac aauuucuuuc aauuggaaac gauggaacgc cguaugcccg 900
gaaacgggcg cguacggugu ggaguggggg aaaagcugga gauaaucuca aaggcuuacc 960
uaucacuauc gcgacacguu ccuaaguga 989
<210>18
<211>1028
<212>RNA
<213>破伤风梭菌
<400>18
gccauacaau aaaagugcga aacguuaucc uauaaguaag aaaguuuuaa aauuuucuua60
cgaaaaggau agaacuuaaa aguucuaacu guucuacuaa aguaauaagu gaaaaucuua 120
uuuaaagcaa acaaccaagu agcuuuaagu cuaagucccc uacacaaguu uuauacuacu 180
augcaaaacu ugugaagcua gguaaggucg uaauccguga aagucggaug cggggcuccu 240
uaaaagauua cuaugguaaa cauaagcuaa uccauuaaga ugcgauuuau auguauuuua 300
uacuguuaaa uauuuuugug cuuguggcuu gguauaaaac aguuaagaug aaguacuuaa 360
cugguuuugg aauaauuggu uguuaaacua aaacauuaua aaucguuagu ggauaccuaa 420
gguaaucaaa aauagggaua gguagaaugg aacguuugau gcuguauaug aagagguuua 480
guagaaccua ggacacauau acgggcucag cagguucaua guagcuauga uacucagccg 540
gaagucaauu aauuuugaaa uacuucuaug guaacauagg agaaggauaa aacugaguga 600
gccaaggaac cuagucggua auagaaaagu ggaaguuaaa acaaauauaa gauuuuagaa 660
uuaauuuaau uaaugaacgg aauuaauuua augauauuua aaguuagacg guuauaaauu 720
aaacauuuca aaauuaaacc auauccaaau ucauaaauau agcuagauca uaucacuagu 780
uuaaaaauaa auaaaucauu ucaaauuacu auuaaguaag guauuaauac cuuacuuaau 840
aguaaucuca uuacauaaga gaauuacuag auuagcagac agauucauaa aaacuauauc 900
aacuaggaca auagaaaaua uauuuauaca cuuccuauua ucgagcgaac gccuuaugcg 960
augaaagucg cacguagggu guagaccaag cgaaauccua ugcauuuagg auagugaggu1020
auagcaaa 1028
<210>19
<211>1028
<212>RNA
<213>人工序列
<220>
<223>II型内含子Cte_mut
<400>19
gcaagacaau aaaaguuuga cacgugaucc uauaaguaag aaaguuuuaa aauuuucuua60
cgaaaaggau agaacuuaaa aguucuaacu guucuacuaa aguaauaagu gaaaaucuua 120
uuuaaagcaa acaaccaagu agcuuuaagu cuaagucccc uacacaaguu uuauacuacu 180
augcaaaacu ugugaagcua gguaaggucg uaauccguga aagucggaug cggggcuccu 240
uaaaagauua cuaugguaaa cauaagcuaa uccauuaaga ugcgauuuau auguauuuua 300
uacuguuaaa uauuuuugug cuuguggcuu gguauaaaac aguuaagaug aaguacuuaa 360
cugguuuugg aauaauuggu uguuaaacua aaacauuaua aaucguuagu ggauaccuaa 420
gguaaucaaa aauagggaua gguagaaugg aacguuugau gcuguauaug aagagguuua 480
guagaaccua ggacacauau acgggcucag cagguucaua guagcuauga uacucagccg 540
gaagucaauu aauuuugaaa uacuucuaug guaacauagg agaaggauaa aacugaguga 600
gccaaggaac cuagucggua auagaaaagu ggaaguuaaa acaaauauaa gauuuuagaa 660
uuaauuuaau uaaugaacgg aauuaauuua augauauuua aaguuagacg guuauaaauu 720
aaacauuuca aaauuaaacc auauccaaau ucauaaauau agcuagauca uaucacuagu 780
uuaaaaauaa auaaaucauu ucaaauuacu auuaaguaag guauuaauac cuuacuuaau 840
aguaaucuca uuacauaaga gaauuacuag auuagcagac agauucauaa aaacuauauc 900
aacuaggaca auagaaaaua uauuuauaca cuuccuauua ucgagcgaac gccuuaugcg 960
augaaagucg cacguagggu guagaccaag cgaaauccua ugcauuuagg auagugaggu1020
auagcaaa 1028
<210>20
<211>60
<212>RNA
<213>人工序列
<220>
<223>间隔序列 1
<400>20
gcaauagccg aaaaacaaaa aacaaaaaaa acaaaaaaaa aaccaaaaaa acaaaacaca60
<210>21
<211>20
<212>RNA
<213>人工序列
<220>
<223>间隔序列2
<400>21
aaauuauaau aauuauaaua20
<210>22
<211>224
<212>RNA
<213>人工序列
<220>
<223>间隔序列 3
<400>22
augaaaccgg cucggauucc gcccgcgugc gccauccccu cagcuagcag gugugagcgg60
cuuucugccc gcagucucua cacagcucag cauccugacg ccuccucccc uugcaggggc 120
gugaagcuac uucagacucu gcugugacga cuuggccgcc aggcaccgau ccuccccggu 180
gagaaggucc acgaaucuua cugcagacag auuugcucag cgcg224
<210>23
<211>24
<212>RNA
<213>人工序列
<220>
<223>探针1
<400>23
cuuucacuac uccuacgagc acca 24
<210>24
<211>25
<212>RNA
<213>人工序列
<220>
<223>探针2
<400>24
gaccaugcuc ccaagcaaga ucaug25
<210>25
<211>555
<212>RNA
<213>人工序列
<220>
<223>Gluc
<400>25
augggaguca aaguucuguu ugcccugauc ugcaucgcug uggccgaggc caagcccacc60
gagaacaacg aagacuucaa caucguggcc guggccagca acuucgcgac cacggaucuc 120
gaugcugacc gcgggaaguu gcccggcaag aagcugccgc uggaggugcu caaagagaug 180
gaagccaaug cccggaaagc uggcugcacc aggggcuguc ugaucugccu gucccacauc 240
aagugcacgc ccaagaugaa gaaguucauc ccaggacgcu gccacaccua cgaaggcgac 300
aaagaguccg cacagggcgg cauaggcgag gcgaucgucg acauuccuga gauuccuggg 360
uucaaggacu uggagcccau ggagcaguuc aucgcacagg ucgaucugug uguggacugc 420
acaacuggcu gccucaaagg gcuugccaac gugcaguguu cugaccugcu caagaagugg 480
cugccgcaac gcugugcgac cuuugccagc aagauccagg gccaggugga caagaucaag 540
ggggccggug gugac555
<210>26
<211>936
<212>RNA
<213>人工序列
<220>
<223>Rluc1
<400>26
auggcuucca agguguacga ccccgagcaa cgcaaacgca ugaucacugg gccucagugg60
ugggcucgcu gcaagcaaau gaacgugcug gacuccuuca ucaacuacua ugauuccgag 120
aagcacgccg agaacgccgu gauuuuucug caugguaacg cugccuccag cuaccugugg 180
aggcacgucg ugccucacau cgagcccgug gcuagaugca ucaucccuga ucugaucgga 240
auggguaagu ccggcaagag cgggaauggc ucauaucgcc uccuggauca cuacaaguac 300
cucaccgcuu gguucgagcu gcugaaccuu ccaaagaaaa ucaucuuugu gggccacgac 360
uggggggcuu gucuggccuu ucacuacucc uacgagcacc aagacaagau caaggccauc 420
guccaugcug agagugucgu ggacgugauc gaguccuggg acgaguggcc ugacaucgag 480
gaggauaucg cccugaucaa gagcgaagag ggcgagaaaa uggugcuuga gaauaacuuc 540
uucgucgaga ccaugcuccc aagcaagauc augcggaaac uggagccuga ggaguucgcu 600
gccuaccugg agccauucaa ggagaagggc gagguuagac ggccuacccu cuccuggccu 660
cgcgagaucc cucucguuaa gggaggcaag cccgacgucg uccagauugu ccgcaacuac 720
aacgccuacc uucgggccag cgacgaucug ccuaagaugu ucaucgaguc cgacccuggg 780
uucuuuucca acgcuauugu cgagggagcu aagaaguucc cuaacaccga guucgugaag 840
gugaagggcc uccacuucag ccaggaggac gcuccagaug aaauggguaa guacaucaag 900
agcuucgugg agcgcgugcu gaagaacgag caguaa 936
<210>27
<211>1160
<212>RNA
<213>人工序列
<220>
<223>Rluc2
<400>27
augaaaccgg cucggauucc gcccgcgugc gccauccccu cagcuagcag gugugagcgg60
cuuucugccc gcagucucua cacagcucag cauccugacg ccuccucccc uugcaggggc 120
gugaagcuac uucagacucu gcugugacga cuuggccgcc aggcaccgau ccuccccggu 180
gagaaggucc acgaaucuua cugcagacag auuugcucag cgcgauggcu uccaaggugu 240
acgaccccga gcaacgcaaa cgcaugauca cugggccuca guggugggcu cgcugcaagc 300
aaaugaacgu gcuggacucc uucaucaacu acuaugauuc cgagaagcac gccgagaacg 360
ccgugauuuu ucugcauggu aacgcugccu ccagcuaccu guggaggcac gucgugccuc 420
acaucgagcc cguggcuaga ugcaucaucc cugaucugau cggaaugggu aaguccggca 480
agagcgggaa uggcucauau cgccuccugg aucacuacaa guaccucacc gcuugguucg 540
agcugcugaa ccuuccaaag aaaaucaucu uugugggcca cgacuggggg gcuugucugg 600
ccuuucacua cuccuacgag caccaagaca agaucaaggc caucguccau gcugagagug 660
ucguggacgu gaucgagucc ugggacgagu ggccugacau cgaggaggau aucgcccuga 720
ucaagagcga agagggcgag aaaauggugc uugagaauaa cuucuucguc gagaccaugc 780
ucccaagcaa gaucaugcgg aaacuggagc cugaggaguu cgcugccuac cuggagccau 840
ucaaggagaa gggcgagguu agacggccua cccucuccug gccucgcgag aucccucucg 900
uuaagggagg caagcccgac gucguccaga uuguccgcaa cuacaacgcc uaccuucggg 960
ccagcgacga ucugccuaag auguucaucg aguccgaccc uggguucuuu uccaacgcua1020
uugucgaggg agcuaagaag uucccuaaca ccgaguucgu gaaggugaag ggccuccacu1080
ucagccagga ggacgcucca gaugaaaugg guaaguacau caagagcuuc guggagcgcg1140
ugcugaagaa cgagcaguaa1160
<210>28
<211>1061
<212>RNA
<213>人工序列
<220>
<223>IRES-EgFP
<400>28
agcaaagacc ccaacgagaa gcgcgaucac augguccugc uggaguucgu gaccgccgcc60
gggaucacuc ucggcaugga cgagcuguac aaguaacaaa caaacaaaac aaaaacacuc 120
cccugugagg aacuacuguc uucacgcaga aagcgucuag ccauggcguu aguaugagug 180
ucgugcagcc uccaggaccc ccccucccgg gagagccaua guggucugcg gaaccgguga 240
guacaccgga auugccagga cgaccggguc cuuucuugga uaaacccgcu caaugccugg 300
agauuugggc gugcccccgc aagacugcua gccgaguagu guugggucgc gaaaggccuu 360
gugguacugc cugauagggu gcuugcgagu gccccgggag gucucguaga ccgugcacca 420
ugagcacgaa uccuaaaaug gugagcaagg gcgaggagcu guucaccggg guggugccca 480
uccuggucga gcuggacggc gacguaaacg gccacaaguu cagcgugucc ggcgagggcg 540
agggcgaugc caccuacggc aagcugaccc ugaaguucau cugcaccacc ggcaagcugc 600
ccgugcccug gcccacccuc gugaccaccc ugaccuacgg cgugcagugc uucagccgcu 660
accccgacca caugaagcag cacgacuucu ucaaguccgc caugcccgaa ggcuacgucc 720
aggagcgcac caucuucuuc aaggacgacg gcaacuacaa gacccgcgcc gaggugaagu 780
ucgagggcga cacccuggug aaccgcaucg agcugaaggg caucgacuuc aaggaggacg 840
gcaacauccu ggggcacaag cuggaguaca acuacaacag ccacaacguc uauaucaugg 900
ccgacaagca gaagaacggc aucaagguga acuucaagau ccgccacaac aucgaggacg 960
gcagcgugca gcucgccgac cacuaccagc agaacacccc caucggcgac ggccccgugc1020
ugcugcccga caaccacuac cugagcaccc aguccgcccu g1061
<210>29
<211>899
<212>RNA
<213>人工序列
<220>
<223>IRES-gluc
<400>29
agcaguucau cgcacagguc gaucugugug uggacugcac aacuggcugc cucaaagggc60
uugccaacgu gcaguguucu gaccugcuca agaaguggcu gccgcaacgc ugugcgaccu 120
uugccagcaa gauccagggc cagguggaca agaucaaggg ggccgguggu gacuaacaaa 180
caaacaaaac aaaaacacuc cccugugagg aacuacuguc uucacgcaga aagcgucuag 240
ccauggcguu aguaugagug ucgugcagcc uccaggaccc ccccucccgg gagagccaua 300
guggucugcg gaaccgguga guacaccgga auugccagga cgaccggguc cuuucuugga 360
uaaacccgcu caaugccugg agauuugggc gugcccccgc aagacugcua gccgaguagu 420
guugggucgc gaaaggccuu gugguacugc cugauagggu gcuugcgagu gccccgggag 480
gucucguaga ccgugcacca ugagcacgaa uccuaaaaug ggagucaaag uucuguuugc 540
ccugaucugc aucgcugugg ccgaggccaa gcccaccgag aacaacgaag acuucaacau 600
cguggccgug gccagcaacu ucgcgaccac ggaucucgau gcugaccgcg ggaaguugcc 660
cggcaagaag cugccgcugg aggugcucaa agagauggaa gccaaugccc ggaaagcugg 720
cugcaccagg ggcugucuga ucugccuguc ccacaucaag ugcacgccca agaugaagaa 780
guucauccca ggacgcugcc acaccuacga aggcgacaaa gaguccgcac agggcggcau 840
aggcgaggcg aucgucgaca uuccugagau uccuggguuc aaggacuugg agcccaugg899
<210>30
<211>32
<212>RNA
<213>人工序列
<220>
<223>臂1
<400>30
aauaccuuac uuaauaguaa caauagaaaa uc32
<210>31
<211>33
<212>RNA
<213>人工序列
<220>
<223>臂2
<400>31
aagcuagauc auauuacuau uaaguaaggu auu 33
<210>32
<211>1719
<212>RNA
<213>人工序列
<220>
<223>cRNAzyme (Cte-Rluc)
<400>32
aauaccuuac uuaauaguaa caauagaaaa uccuauuauc gagcgaacgc cuuaugcgau60
gaaagucgca cguagggugu agaccaagcg aaauccuaug cauuuaggau agugagguau 120
agcaaaaugg cuuccaaggu guacgacccc gagcaacgca aacgcaugau cacugggccu 180
cagugguggg cucgcugcaa gcaaaugaac gugcuggacu ccuucaucaa cuacuaugau 240
uccgagaagc acgccgagaa cgccgugauu uuucugcaug guaacgcugc cuccagcuac 300
cuguggaggc acgucgugcc ucacaucgag cccguggcua gaugcaucau cccugaucug 360
aucggaaugg guaaguccgg caagagcggg aauggcucau aucgccuccu ggaucacuac 420
aaguaccuca ccgcuugguu cgagcugcug aaccuuccaa agaaaaucau cuuugugggc 480
cacgacuggg gggcuugucu ggccuuucac uacuccuacg agcaccaaga caagaucaag 540
gccaucgucc augcugagag ugucguggac gugaucgagu ccugggacga guggccugac 600
aucgaggagg auaucgcccu gaucaagagc gaagagggcg agaaaauggu gcuugagaau 660
aacuucuucg ucgagaccau gcucccaagc aagaucaugc ggaaacugga gccugaggag 720
uucgcugccu accuggagcc auucaaggag aagggcgagg uuagacggcc uacccucucc 780
uggccucgcg agaucccucu cguuaaggga ggcaagcccg acgucgucca gauuguccgc 840
aacuacaacg ccuaccuucg ggccagcgac gaucugccua agauguucau cgaguccgac 900
ccuggguucu uuuccaacgc uauugucgag ggagcuaaga aguucccuaa caccgaguuc 960
gugaagguga agggccucca cuucagccag gaggacgcuc cagaugaaau ggguaaguac1020
aucaagagcu ucguggagcg cgugcugaag aacgagcagu aagccauaca auaaaagugc1080
gaaacguuau ccuauaagua agaaaguuuu aaaauuuucu uacgaaaagg auagaacuua1140
aaaguucuaa cuguucuacu aaaguaauaa gugaaaaucu uauuuaaagc aaacaaccaa1200
guagcuuuaa gucuaagucc ccuacacaag uuuuauacua cuaugcaaaa cuugugaagc1260
uagguaaggu cguaauccgu gaaagucgga ugcggggcuc cuuaaaagau uacuauggua1320
aacauaagcu aauccauuaa gaugcgauuu auauguauuu uauacuguua aauauuuuug1380
ugcuuguggc uugguauaaa acaguuaaga ugaaguacuu aacugguuuu ggaauaauug1440
guuguuaaac uaaaacauua uaaaucguua guggauaccu aagguaauca aaaauaggga1500
uagguagaau ggaacguuug augcuguaua ugaagagguu uaguagaacc uaggacacau1560
auacgggcuc agcagguuca uaguagcuau gauacucagc cggaagucaa uuaauuuuga1620
aauacuucua ugguaacaua ggagaaggau aaaacugagu gagccaagga accuagucgg1680
uaauagaagc uagaucauau uacuauuaag uaagguauu 1719
<210>33
<211>426
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>33
cucucuaaau agcaauauuu accuuuggag ggaaaaguua ucaggcaugc accugguagc60
uagucuuuaa accaauagau ugcaucgguu uaaaaggcaa gaccgucaaa uugcgggaaa 120
ggggucaaca gccguucagu accaagucuc aggggaaacu uugagauggc cuugcaaagg 180
guaugguaau aagcugacgg acaugguccu aaccacgcag ccaaguccua agucaacaga 240
ucuucuguug auauggaugc aguucacaga cuaaaugucg gucggggaag auguauucuu 300
cucauaagau auagucggac cucuccuuaa ugggagcuag cggaugaagu gaugcaacac 360
uggagccgcu gggaacuaau uuguaugcga aaguauauug auuaguuuug gaguacucgu 420
aaggua426
<210>34
<211>787
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>34
ugcaugugau gcaggucgug gugcgaaucg uuccuaagug aaaagcuuag gcaucuuaga60
caagggaagg ccccauuaag auagaacuaa ugauucgaga gacagaauga uagaguaacg 120
ucuugaaaug uuuccccuaa gucuccgaca ugcauugaaa gguaggaugu uuaaaugugu 180
gaagcucggu gaagacggcu gacagauacc guagugaaaa auguguuuua accgaaaguc 240
cuuuuaguaa gaggauguau cccauaggcc ggggguccgu aaaguaucua uggugagaau 300
guuauaugaa auaacugacg aacuuucgaa uuaacgaguc uauaaccaua caaucagaaa 360
ugauaguaaa ccauauuuug uguugugugu gagguuaagu aaauucgcgg cuaugaacaa 420
ccuuacagua uuauagguac ugucuagcac gcaggcucau agaaggcacc uaaggguacc 480
auauagugga uagaaucauu ggaacgauga aagcucagga cgcagagaac cuacauucug 540
ugaagcggug guaaggaaaa ggaggaaucc uauaacuuau cuuuauuuga gugauagcga 600
ugucauagua gcguggaauu uauggaaaca uaaaggagcg aagggcauua gucaauauug 660
ugaggaaaaa gcaauauuga uggcaagccg uaugaaggga aacuuucaug uacgguuuag 720
ugugggggaa aaagcagaga uuauaucaaa guuuuaccua ucacaauaag gagagaauau 780
uuuuaau 787
<210>35
<211>989
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>35
caaaggcuua ccuaucacua gcgcgacacg uuccuaagug aaaagcuuag gcacugucga60
acucaacagu ucagcaguga acugucauuc uaagaaguca aaugaaggag uaacgucugg 120
aagggcuucc cuuaauccuc cgacaugcag gaaaguaggc aaguacugaa cugugugaag 180
cucggugaag ucgguugaag guuaccguaa auuaguaucu cuaauacgaa agcuauccag 240
cgguggaugg uguaacugau agaccggagg ucuauaaaac acucaagguu aggaugcgcg 300
augaacuaga ggcgaucgcu aguaagcgca gacgaauccc ugaugguacg ggucuauauc 360
gggagggaau cgaaagguuc ucugacacaa auaagugucg cuacuguggg ugaguaaaac 420
ucuccuuuau gaaagcccau auaucguuac aggcguuauu aagguagcag gcucauaggg 480
gaaaccuaaa aguguaugua cagauaagaa ugacggaacg ugguaagcug ccgacaugga 540
gggcuuguuc ucuuugaagu guugccaagg aaagucacaa ugagauuagu ugucgauaua 600
acuugguuua acggcaguga aagugguggc acaguaccga ugaaacgugu aaugaacgug 660
gagggauagc cacuagucga uugaagauug aagguuacua uugguuaaca ugguuucgag 720
uaagacuaag agauguaaug cuccaaagua auaaggaggu uacagcccau guuaaagaaa 780
accaagcuaa gacauaacga auauuaugau acacaaaaaa aguguaugac aauuuauacu 840
cgaacagucu uaacgguaac aauuucuuuc aauuggaaac gauggaacgc cguaugcccg 900
gaaacgggcg cguacggugu ggaguggggg aaaagcugga gauaaucuca aaggcuuacc 960
uaucacuauc gcgacacguu ccuaaguga 989
<210>36
<211>867
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>36
uuauaacaaa guaguuauuu gugcgacacg uuucuuuaua agugugcaaa cacgaaguag60
gaggguuauc aauuugauga caacaacgga uugacugcaa guaggaauga aagccgucag 120
guugagcuga aacuacuuau cugauacucc uauaugcaag gcgugauagu agucacaaau 180
uguaugaagc uaggugaagu cggcugaaca aaaccuaagu gagaaaucau augguaaugg 240
auaggucggg augcuacaaa acaucuaugg ugagaauguc cuaacggacu ggcgaaugua 300
cagguuuaaa ggauuaauuc auuagaaaug uguauauugu caacgacgac gcuaucuacc 360
gaaaaguaag aguaaauaau augaaauucg gaagaucuaa cgaugaggau guaaagauaa 420
caggcuuaca gcaagcaccu aaagauauau guauagcuaa gucauucaga acgugguaag 480
caagagacug ucacaaaugc cuacuaacag acaaggugca uauaagguuc uaacgaacca 540
aaauugcuuu auucuuguga agguggggac acaguaccga cgaagcaugu aacaaaugug 600
gagggauagu cccuagucuu guucguugaa aacuaaauca acuggauaua acucacagga 660
ucgaguaaga ugaugugacu uuucguaaga aaagggaaug aauacguugg ugaguacaac 720
augguuugua aucuaguugu uuuaguauaa aaggauaaga uggggcgcug uaugcgauga 780
aagucgcacg uacaguguca agcgggggaa aagauggaga uaacuuuaaa gucuuaccua 840
ucgcaacgga uaaaaagaau cccugcu 867
<210>37
<211>1046
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>37
gaauauacua uagccauaca auaaaagugc gaaacguuau ccuauaagua agaaaguuuu60
aaaauuuucu uacgaaaagg auagaacuua aaaguucuaa cuguucuacu aaaguaauaa 120
gugaaaaucu uauuuaaagc aaacaaccaa guagcuuuaa gucuaagucc ccuacacaag 180
uuuuauacua cuaugcaaaa cuugugaagc uagguaaggu cguaauccgu gaaagucgga 240
ugcggggcuc cuuaaaagau uacuauggua aacauaagcu aauccauuaa gaugcgauuu 300
auauguauuu uauacuguua aauauuuuug ugcuuguggc uugguauaaa acaguuaaga 360
ugaaguacuu aacugguuuu ggaauaauug guuguuaaac uaaaacauua uaaaucguua 420
guggauaccu aagguaauca aaaauaggga uagguagaau ggaacguuug augcuguaua 480
ugaagagguu uaguagaacc uaggacacau auacgggcuc agcagguuca uaguagcuau 540
gauacucagc cggaagucaa uuaauuuuga aauacuucua ugguaacaua ggagaaggau 600
aaaacugagu gagccaagga accuagucgg uaauagaaaa guggaaguua aaacaaauau 660
aagauuuuag aauuaauuua auuaaugaac ggaauuaauu uaaugauauu uaaaguuaga 720
cgguuauaaa uuaaacauuu caaaauuaaa ccauauccaa auucauaaau auagcuagau 780
cauaucacua guuuaaaaau aaauaaauca uuucaaauua cuauuaagua agguauuaau 840
accuuacuua auaguaaucu cauuacauaa gagaauuacu agauuagcag acagauucau 900
aaaaacuaua ucaacuagga caauagaaaa uauauuuaua cacuuccuau uaucgagcga 960
acgccuuaug cgaugaaagu cgcacguagg guguagacca agcgaaaucc uaugcauuua1020
ggauagugag guauagcaaa ggagaa 1046
<210>38
<211>626
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>38
acucuaggua gacgagagug cgacuaguaa agugcuuaau aacaaugugg ugaaagccca60
ccagauaacc cauuaucuga gcucagacgg uaugcauuag aaguaauucu uuuguguaaa 120
gcccguuuaa uuugguaaac agaccaacca accuauauau aaggauggug agcuauauua 180
cuauggauaa aauuuuuaug aauucacguu cgaagcguau uagagugagu uuuauuuaga 240
aggaaaaaag uaaauaaaau ucuaauuaau uguauaaaca auuuuucguu uguuuauuaa 300
ugggucuuau auuaauguac guauagugaa auccuaaggu aguaaauaag guguuauuaa 360
guaaacuagg uaagcccaau aauaucuuca uaugauagua ugaagaaguu caaguguaaa 420
uuugaauaua uauuaguggg uaaaggauau uuuaaaaagc gaaugucuca uauuaauagu 480
gagaauaggu uuaugacuaa uucgaaagaa ugcugacuua aaauuaauau uaauauuucg 540
auauuaauau uugagccgua ugcgaugaaa gucgcacgua cgguucuuag agggggaaag 600
uccaagaggg ccuaccuauc ucaaca626
<210>39
<211>639
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>39
cucuagauag acgagagugc gacuagauaa guacuuaaua gcaauguggu gaaaguccac60
cugauaaccc auagucugag cucagacggu augcauuaga aguaauucuu uuguguaaag 120
cccguuuaau cuggucuaaa ggaccuucca cccuaaauau auagggggag agcuauaaua 180
cuaaggauaa aauuuuuuug aauucauguu uaaagcguau uagaggaaau uuauuucuau 240
gaaaaagaag aaauaaauuu uuaauuaacu guauaaacaa guuuucguuu guuuauuagu 300
gggucuuaug uuaauguacg uauaguggaa uccuaaggua uuaauuaagg uguuauuaag 360
uaaacuaggu aagcccaaua gugucuucau auuauaauau gaagaaguuc aauugugaaa 420
uugaauaugc auuagugggu aaaggauauu uuaaaaagcg aaugucucau auuaauagug 480
agaauagguc uaugacuaau ucgaaagaau gcugacuuaa aauugguauu aauauaugcg 540
uuucgaugua uauauuaaua uuugagccgu augcgaugaa agucgcacgu acgguucuua 600
gagggggaaa ucuuaguaau aagacgaccu aucucgaca639
<210>40
<211>573
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>40
ggguagcaau aaggaugugc gacuuguuaa guuuuaacaa aaauuguaua acguuuauua60
augauuauac auuguauuuc aucuuacaau agccuaauua gauaugcauu uaggguaacu 120
uuuuuguaua aagcucuaau uauaagugua aauacacuuu uaggcuucuu cuaugguuag 180
agaaaucgaa ccaauguaau uaaaacuuug auguauuagg cauuuaacgu guccuugguu 240
aaaugaagau gaacauaagu auacaaagua aaauuggaac cuaaggaaga auuguuuuug 300
uuaagaaaca agguaauacc uauaacuggc uaauauaaau uugcaagguu uauuguaaaa 360
uaaacuauag guuagaggua aaaggauaau guaaaaagcg aaugcaauuc uguaauggaa 420
uugauagggu auauaccuaa cuugaaagag ugcugacuua cauauagaug uuauuuacgu 480
uucgacguaa auaauguuug agccguaugc uaugaaagua gcacguacgg uucuaagagg 540
gggaaagucc gagaggaccu accuaucuca acu573
<210>41
<211>659
<212>RNA
<213>人工序列
<220>
<223>II型内含子
<400>41
acucuaggua gacgagagug cgacuaguaa aguguuuaau aauaaugugg ugaaaaccca60
ccagauaacc cauuaucuga gcucauacgg ugugcauuaa aaguaauuuu uuuguauaaa 120
gcccguuuaa ucagguaaau aauccuucca uccuaauuau aucuauagau auaaaaggau 180
ggugagcuau auuacuaugg auaagauuuu uuugaauuca cguuugaagc guauuagagu 240
aaguuuuauu uaaaaggaaa aaaaaaaauu aaauaaaauu aaauuaauug uauaaauaag 300
uuuucguuua uuuauuaaug ggucuuauau uaauguacgu auagugaaau ccuaauguag 360
uaauuaaggu guuauuaaau aaacuaggua agcccaauaa ugucuucaua auaugaagaa 420
guucaagugu aaauuugaau auacauuagu ggguaaagga uauuuuaaaa agcgaauguc 480
ucauauuaau agugagaaua ggucuaugac uaauucgaaa gaaugcugac uuaaaauuaa 540
uauuaauauu aauauauaua uauuaauauu ugagccguau gcgaugaaaa ucgcacguac 600
gguucuuaga gggggaaagc ucgagagggc cuaccuaucu ccacagaccc uaugcagcu659
<210>42
<211>261
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>42
cuuugagaug gccuugcaaa ggguauggua auaagcugac ggacaugguc cuaaccacgc60
agccaagucc uaagucaaca gaucuucugu ugauauggau gcaguucaca gacuaaaugu 120
cggucgggga agauguauuc uucucauaag auauagucgg accucuccuu aaugggagcu 180
agcggaugaa gugaugcaac acuggagccg cugggaacua auuuguaugc gaaaguauau 240
ugauuaguuu uggaguacuc g 261
<210>43
<211>97
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>43
gcaauauuga uggcaagccg uaugaaggga aacuuucaug uacgguuuag ugugggggaa60
aaagcagaga uuauaucaaa guuuuaccua ucacaau 97
<210>44
<211>98
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>44
auuggaaacg auggaacgcc guaugcccgg aaacgggcgc guacggugug gaguggggga60
aaagcuggag auaaucucaa aggcuuaccu aucacuau98
<210>45
<211>95
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>45
ggauaagaug gggcgcugua ugcgaugaaa gucgcacgua cagugucaag cgggggaaaa60
gauggagaua acuuuaaagu cuuaccuauc gcaac 95
<210>46
<211>88
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>46
cuauuaucga gcgaacgccu uaugcgauga aagucgcacg uaggguguag accaagcgaa60
auccuaugca uuuaggauag ugagguau 88
<210>47
<211>85
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>47
auauuaauau uugagccgua ugcgaugaaa gucgcacgua cgguucuuag agggggaaag60
uccaagaggg ccuaccuauc ucaac85
<210>48
<211>93
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>48
auguauauau uaauauuuga gccguaugcg augaaagucg cacguacggu ucuuagaggg60
ggaaaucuua guaauaagac gaccuaucuc gac 93
<210>49
<211>88
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>49
acguaaauaa uguuugagcc guaugcuaug aaaguagcac guacgguucu aagaggggga60
aaguccgaga ggaccuaccu aucucaac 88
<210>50
<211>85
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>50
auauuaauau uugagccgua ugcgaugaaa aucgcacgua cgguucuuag agggggaaag60
cucgagaggg ccuaccuauc uccac85
<210>51
<211>408
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>51
gaauaauugg uuguuaaacu aaaacauuau aaaucguuag uggauaccua agguaaucaa60
aaauagggau agguagaaug gaacguuuga ugcuguauau gaagagguuu aguagaaccu 120
aggacacaua uacgggcuca gcagguucau aguagcuaug auacucagcc ggaagucaau 180
uaauuuugaa auacuucuau gguaacauag gagaaggaua aaacugagug agccaaggaa 240
ccuagucggu aauagaagcu agaucauauu acuauuaagu aagguauuaa uaccuuacuu 300
aauaguaaca auagaaaauc cuauuaucga gcgaacgccu uaugcgauga aagucgcacg 360
uaggguguag accaagcgaa auccuaugca uuuaggauag ugagguau408
<210>52
<211>217
<212>RNA
<213>人工序列
<220>
<223>3'内含子片段
<400>52
uacuucuaug guaacauagg agaaggauaa aacugaguga gccaaggaac cuagucggua60
auagaagcua gaucauauua cuauuaagua agguauuaau accuuacuua auaguaacaa 120
uagaaaaucc uauuaucgag cgaacgccuu augcgaugaa agucgcacgu aggguguaga 180
ccaagcgaaa uccuaugcau uuaggauagu gagguau217
<210>53
<211>7
<212>RNA
<213>人工序列
<220>
<223>E2
<400>53
uaaggua 7
<210>54
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>54
aaggag6
<210>55
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>55
aaguga6
<210>56
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>56
ccugcu6
<210>57
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>57
agcagu6
<210>58
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>58
agcaaa6
<210>59
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>59
agagaa6
<210>60
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>60
agcaaa6
<210>61
<211>1
<212>RNA
<213>人工序列
<220>
<223>E2
<400>61
a 1
<210>62
<211>1
<212>RNA
<213>人工序列
<220>
<223>E2
<400>62
u 1
<210>63
<211>6
<212>RNA
<213>人工序列
<220>
<223>E2
<400>63
agaccc6
<210>64
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>64
cucucu6
<210>65
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>65
gucgug6
<210>66
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>66
caaagg6
<210>67
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>67
uuauuu6
<210>68
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>68
ccaugg6
<210>69
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>69
gcccug6
<210>70
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>70
cguuga6
<210>71
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>71
gccaua6
<210>72
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>72
acgaga6
<210>73
<211>6
<212>RNA
<213>人工序列
<220>
<223>E1
<400>73
aaggau6
<210>74
<211>8
<212>RNA
<213>人工序列
<220>
<223>E1
<400>74
agacgaga8
<210>75
<211>148
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>75
aaauagcaau auuuaccuuu ggagggaaaa guuaucaggc augcaccugg uagcuagucu60
uuaaaccaau agauugcauc gguuuaaaag gcaagaccgu caaauugcgg gaaagggguc 120
aacagccguu caguaccaag ucucaggg148
<210>76
<211>641
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>76
gugcgaaucg uuccuaagug aaaagcuuag gcaucuuaga caagggaagg ccccauuaag60
auagaacuaa ugauucgaga gacagaauga uagaguaacg ucuugaaaug uuuccccuaa 120
gucuccgaca ugcauugaaa gguaggaugu uuaaaugugu gaagcucggu gaagacggcu 180
gacagauacc guagugaaaa auguguuuua accgaaaguc cuuuuaguaa gaggauguau 240
cccauaggcc ggggguccgu aaaguaucua uggugagaau guuauaugaa auaacugacg 300
aacuuucgaa uuaacgaguc uauaaccaua caaucagaaa ugauaguaaa ccauauuuug 360
uguugugugu gagguuaagu aaauucgcgg cuaugaacaa ccuuacagua uuauagguac 420
ugucuagcac gcaggcucau agaaggcacc uaaggguacc auauagugga uagaaucauu 480
ggaacgauga aagcucagga cgcagagaac cuacauucug ugaagcggug guaaggaaaa 540
ggaggaaucc uauaacuuau cuuuauuuga gugauagcga ugucauagua gcguggaauu 600
uauggaaaca uaaaggagcg aagggcauua gucaauauug u 641
<210>77
<211>666
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>77
gcgcgacacg uuccuaagug aaaagcuuag gcacugucga acucaacagu ucagcaguga60
acugucauuc uaagaaguca aaugaaggag uaacgucugg aagggcuucc cuuaauccuc 120
cgacaugcag gaaaguaggc aaguacugaa cugugugaag cucggugaag ucgguugaag 180
guuaccguaa auuaguaucu cuaauacgaa agcuauccag cgguggaugg uguaacugau 240
agaccggagg ucuauaaaac acucaagguu aggaugcgcg augaacuaga ggcgaucgcu 300
aguaagcgca gacgaauccc ugaugguacg ggucuauauc gggagggaau cgaaagguuc 360
ucugacacaa auaagugucg cuacuguggg ugaguaaaac ucuccuuuau gaaagcccau 420
auaucguuac aggcguuauu aagguagcag gcucauaggg gaaaccuaaa aguguaugua 480
cagauaagaa ugacggaacg ugguaagcug ccgacaugga gggcuuguuc ucuuugaagu 540
guugccaagg aaagucacaa ugagauuagu ugucgauaua acuugguuua acggcaguga 600
aagugguggc acaguaccga ugaaacgugu aaugaacgug gagggauagc cacuagucga 660
uugaag666
<210>78
<211>604
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>78
gugcgacacg uuucuuuaua agugugcaaa cacgaaguag gaggguuauc aauuugauga60
caacaacgga uugacugcaa guaggaauga aagccgucag guugagcuga aacuacuuau 120
cugauacucc uauaugcaag gcgugauagu agucacaaau uguaugaagc uaggugaagu 180
cggcugaaca aaaccuaagu gagaaaucau augguaaugg auaggucggg augcuacaaa 240
acaucuaugg ugagaauguc cuaacggacu ggcgaaugua cagguuuaaa ggauuaauuc 300
auuagaaaug uguauauugu caacgacgac gcuaucuacc gaaaaguaag aguaaauaau 360
augaaauucg gaagaucuaa cgaugaggau guaaagauaa caggcuuaca gcaagcaccu 420
aaagauauau guauagcuaa gucauucaga acgugguaag caagagacug ucacaaaugc 480
cuacuaacag acaaggugca uauaagguuc uaacgaacca aaauugcuuu auucuuguga 540
agguggggac acaguaccga cgaagcaugu aacaaaugug gagggauagu cccuagucuu 600
guuc604
<210>79
<211>618
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>79
caauaaaagu gcgaaacguu auccuauaag uaagaaaguu uuaaaauuuu cuuacgaaaa60
ggauagaacu uaaaaguucu aacuguucua cuaaaguaau aagugaaaau cuuauuuaaa 120
gcaaacaacc aaguagcuuu aagucuaagu ccccuacaca aguuuuauac uacuaugcaa 180
aacuugugaa gcuagguaag gucguaaucc gugaaagucg gaugcggggc uccuuaaaag 240
auuacuaugg uaaacauaag cuaauccauu aagaugcgau uuauauguau uuuauacugu 300
uaaauauuuu aaugcuuuau gguugguaua aaacaguuaa gaugaaguac uuaacugguu 360
uuggaauaau ugguuguuaa acuaaaacau uauaaaucgu uaguggauac cuaagguaau 420
caaaaauagg gauagguaga auggaacguu ugaugcugua uaugaagagg uuuaguagaa 480
ccuaggacac auauacgggc ucagcagguu cauaguagcu augauacuca gccggaaguc 540
aauuaauuuu gaaauacuuc uaugguaaca uaggagaagg auaaaacuga gugagccaag 600
gaaccuaguc gguaauag 618
<210>80
<211>618
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>80
caauaaaagu gcgaaacguu auccuauaag uaagaaaguu uuaaaauuuu cuuacgaaaa60
ggauagaacu uaaaaguucu aacuguucua cuaaaguaau aagugaaaau cuuauuuaaa 120
gcaaacaacc aaguagcuuu aagucuaagu ccccuacaca aguuuuauac uacuaugcaa 180
aacuugugaa gcuagguaag gucguaaucc gugaaagucg gaugcggggc uccuuaaaag 240
auuacuaugg uaaacauaag cuaauccauu aagaugcgau uuauauguau uuuauacugu 300
uaaauauuuu ugugcuuagg gcuugguaua aaacaguuaa gaugaaguac uuaacugguu 360
uuggaauaau ugguuguuaa acuaaaacau uauaaaucgu uaguggauac cuaagguaau 420
caaaaauagg gauagguaga auggaacguu ugaugcugua uaugaagagg uuuaguagaa 480
ccuaggacac auauacgggc ucagcagguu cauaguagcu augauacuca gccggaaguc 540
aauuaauuuu gaaauacuuc uaugguaaca uaggagaagg auaaaacuga gugagccaag 600
gaaccuaguc gguaauag 618
<210>81
<211>618
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>81
caauaaaagu gcgaaacguu auccuauaag uaagaaaguu uuaaaauuuu cuuacgaaaa60
ggauagaacu uaaaaguucu aacuguucua cuaaaguaau aagugaaaau cuuauuuaaa 120
gcaaacaacc aaguagcuuu aagucuaagu ccccuacaca aguuuuauac uacuaugcaa 180
aacuugugaa gcuagguaag gucguaaucc gugaaagucg gaugcggggc uccuuaaaag 240
auuacuaugg uaaacauaag cuaauccauu aagaugcgau uuauauguau uuuauacugu 300
uaaauauuuu ugcucuucaa cguugguaua aaacaguuaa gaugaaguac uuaacugguu 360
uuggaauaau ugguuguuaa acuaaaacau uauaaaucgu uaguggauac cuaagguaau 420
caaaaauagg gauagguaga auggaacguu ugaugcugua uaugaagagg uuuaguagaa 480
ccuaggacac auauacgggc ucagcagguu cauaguagcu augauacuca gccggaaguc 540
aauuaauuuu gaaauacuuc uaugguaaca uaggagaagg auaaaacuga gugagccaag 600
gaaccuaguc gguaauag 618
<210>82
<211>618
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>82
caauaaaagu gcgaaacguu auccuauaag uaagaaaguu uuaaaauuuu cuuacgaaaa60
ggauagaacu uaaaaguucu aacuguucua cuaaaguaau aagugaaaau cuuauuuaaa 120
gcaaacaacc aaguagcuuu aagucuaagu ccccuacaca aguuuuauac uacuaugcaa 180
aacuugugaa gcuagguaag gucguaaucc gugaaagucg gaugcggggc uccuuaaaag 240
auuacuaugg uaaacauaag cuaauccauu aagaugcgau uuauauguau uuuauacugu 300
uaaauauuuu ugugcuugug gcuugguaua aaacaguuaa gaugaaguac uuaacugguu 360
uuggaauaau ugguuguuaa acuaaaacau uauaaaucgu uaguggauac cuaagguaau 420
caaaaauagg gauagguaga auggaacguu ugaugcugua uaugaagagg uuuaguagaa 480
ccuaggacac auauacgggc ucagcagguu cauaguagcu augauacuca gccggaaguc 540
aauuaauuuu gaaauacuuc uaugguaaca uaggagaagg auaaaacuga gugagccaag 600
gaaccuaguc gguaauag 618
<210>83
<211>519
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>83
gugcgacuag uaaagugcuu aauaacaaug uggugaaagc ccaccagaua acccauuauc60
ugagcucaga cgguaugcau uagaaguaau ucuuuugugu aaagcccguu uaauuuggua 120
aacagaccaa ccaaccuaua uauaaggaug gugagcuaua uuacuaugga uaaaauuuuu 180
augaauucac guucgaagcg uauuagagug aguuuuauuu agaaggaaaa aaguaaauaa 240
aauucuaauu aauuguauaa acaauuuuuc guuuguuuau uaaugggucu uauauuaaug 300
uacguauagu gaaauccuaa gguaguaaau aagguguuau uaaguaaacu agguaagccc 360
aauaauaucu ucauaugaua guaugaagaa guucaagugu aaauuugaau auauauuagu 420
ggguaaagga uauuuuaaaa agcgaauguc ucauauuaau agugagaaua gguuuaugac 480
uaauucgaaa gaaugcugac uuaaaauuaa uauuaauau519
<210>84
<211>525
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>84
gugcgacuag auaaguacuu aauagcaaug uggugaaagu ccaccugaua acccauaguc60
ugagcucaga cgguaugcau uagaaguaau ucuuuugugu aaagcccguu uaaucugguc 120
uaaaggaccu uccacccuaa auauauaggg ggagagcuau aauacuaagg auaaaauuuu 180
uuugaauuca uguuuaaagc guauuagagg aaauuuauuu cuaugaaaaa gaagaaauaa 240
auuuuuaauu aacuguauaa acaaguuuuc guuuguuuau uagugggucu uauguuaaug 300
uacguauagu ggaauccuaa gguauuaauu aagguguuau uaaguaaacu agguaagccc 360
aauagugucu ucauauuaua auaugaagaa guucaauugu gaaauugaau augcauuagu 420
ggguaaagga uauuuuaaaa agcgaauguc ucauauuaau agugagaaua ggucuaugac 480
uaauucgaaa gaaugcugac uuaaaauugg uauuaauaua ugcgu 525
<210>85
<211>464
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>85
gugcgacuug uuaaguuuua acaaaaauug uauaacguuu auuaaugauu auacauugua60
uuucaucuua caauagccua auuagauaug cauuuagggu aacuuuuuug uauaaagcuc 120
uaauuauaag uguaaauaca cuuuuaggcu ucuucuaugg uuagagaaau cgaaccaaug 180
uaauuaaaac uuugauguau uaggcauuua acguguccuu gguuaaauga agaugaacau 240
aaguauacaa aguaaaauug gaaccuaagg aagaauuguu uuuguuaaga aacaagguaa 300
uaccuauaac uggcuaauau aaauuugcaa gguuuauugu aaaauaaacu auagguuaga 360
gguaaaagga uaauguaaaa agcgaaugca auucuguaau ggaauugaua ggguauauac 420
cuaacuugaa agagugcuga cuuacauaua gauguuauuu acgu464
<210>86
<211>538
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>86
gugcgacuag uaaaguguuu aauaauaaug uggugaaaac ccaccagaua acccauuauc60
ugagcucaua cggugugcau uaaaaguaau uuuuuuguau aaagcccguu uaaucaggua 120
aauaauccuu ccauccuaau uauaucuaua gauauaaaag gauggugagc uauauuacua 180
uggauaagau uuuuuugaau ucacguuuga agcguauuag aguaaguuuu auuuaaaagg 240
aaaaaaaaaa auuaaauaaa auuaaauuaa uuguauaaau aaguuuucgu uuauuuauua 300
augggucuua uauuaaugua cguauaguga aauccuaaug uaguaauuaa gguguuauua 360
aauaaacuag guaagcccaa uaaugucuuc auaauaugaa gaaguucaag uguaaauuug 420
aauauacauu aguggguaaa ggauauuuua aaaagcgaau gucucauauu aauagugaga 480
auaggucuau gacuaauucg aaagaaugcu gacuuaaaau uaauauuaau auuaauau 538
<210>87
<211>363
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>87
caauaaaagu gcgaaacguu auccuauaag uaagaaaguu uuaaaauuuu cuuacgaaaa60
ggauagaacu uaaaaguucu aacuguucua cuaaaguaau aagugaaaau cuuauuuaaa 120
gcaaacaacc aaguagcuuu aagucuaagu ccccuacaca aguuuuauac uacuaugcaa 180
aacuugugaa gcuagguaag gucguaaucc gugaaagucg gaugcggggc uccuuaaaag 240
auuacuaugg uaaacauaag cuaauccauu aagaugcgau uuauauguau uuuauacugu 300
uaaauauuuu ugugcuugug gcuugguaua aaacaguuaa gaugaaguac uuaacugguu 360
uug 363
<210>88
<211>441
<212>RNA
<213>人工序列
<220>
<223>5'内含子片段
<400>88
caauaaaagu gcgaaacguu auccuauaag uaagaaaguu uuaaaauuuu cuuacgaaaa60
ggauagaacu uaaaaguucu aacuguucua cuaaaguaau aagugaaaau cuuauuuaaa 120
gcaaacaacc aaguagcuuu aagucuaagu ccccuacaca aguuuuauac uacuaugcaa 180
aacuugugaa gcuagguaag gucguaaucc gugaaagucg gaugcggggc uccuuaaaag 240
auuacuaugg uaaacauaag cuaauccauu aagaugcgau uuauauguau uuuauacugu 300
uaaauauuuu ugugcuugug gcuugguaua aaacaguuaa gaugaaguac uuaacugguu 360
uuggaauaau ugguuguuaa acuaaaacau uauaaaucgu uaguggauac cuaagguaau 420
caaaaauagg gauagguaga a 441
<210>89
<211>60
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>89
gcaauagccg aaaaacaaaa aacaaaaaaa acaaaaaaaa aaccaaaaaa acaaaacaca60
<210>90
<211>58
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>90
cgaaaggagg aggaggagga aaaaaaggag gaggaggaaa ccaaaaaaac aaaacaca58
<210>91
<211>93
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>91
gcaauggagg aggaggagga aaaaaaggag gaggaggaag ccgaaaaaca aaaaacaaaa60
aaaacaaaaa aaaaaccaaa aaaacaaaac aca 93
<210>92
<211>93
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>92
gcaauccucc uccuccuaaa aaaccuccuc cuccuccuag ccgaaaaaca aaaaacaaaa60
aaaacaaaaa aaaaaccaaa aaaacaaaac aca 93
<210>93
<211>39
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>93
cgaaaggagg aggaggagga accaaaaaaa caaaacaca 39
<210>94
<211>40
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>94
cgaaaccucc uccuccuccu aaccaaaaaa acaaaacaca40
<210>95
<211>58
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>95
cgaaaccucc uccuccuaaa aaaccuccuc cuccuccuaa ccaaaaaaac aaaacaca58
<210>96
<211>58
<212>RNA
<213>人工序列
<220>
<223>目标序列的5'臂
<400>96
cgaaaccucc uccuccuaaa aaaccuccuc cuccuccuaa ccaaaaaaac aaaacaca58
<210>97
<211>19
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>97
aaaaaacaaa aaacaaaac 19
<210>98
<211>52
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>98
aaaaaacaaa aaacaccucc uccuccuaaa aaaccuccuc cuccuccuaa aa52
<210>99
<211>52
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>99
aaaaaacaaa aaacaccucc uccuccuaaa aaaccuccuc cuccuccuaa ac52
<210>100
<211>52
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>100
aaaaaacaaa aaacaggagg aggaggagga aaaaaaggag gaggaggaaa ac52
<210>101
<211>34
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>101
aaaaaacaaa aaacaccucc uccuccuccu aaaa34
<210>102
<211>33
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>102
aaaaaacaaa aaacaggagg aggaggagga aaa 33
<210>103
<211>51
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>103
aaaaaacaaa aaacaggagg aggaggagga aaaaaaggag gaggaggaaa a 51
<210>104
<211>100
<212>RNA
<213>人工序列
<220>
<223>目标序列的3'臂
<400>104
auuagagaca auuugaaaua auuuagauug gcuuaacccu acugugcuaa ccgaaccaga60
uaacgguaca guagggguaa auucuccgca uucggugcgg 100
<210>105
<211>20
<212>RNA
<213>人工序列
<220>
<223>5'同源臂序列
<400>105
aauaccuuac uuaauaguaa20
<210>106
<211>20
<212>RNA
<213>人工序列
<220>
<223>3'同源臂序列
<400>106
uuacuauuaa guaagguauu20
<210>107
<211>558
<212>RNA
<213>人工序列
<220>
<223>目标序列
<400>107
augggaguca aaguucuguu ugcccugauc ugcaucgcug uggccgaggc caagcccacc60
gagaacaacg aagacuucaa caucguggcc guggccagca acuucgcgac cacggaucuc 120
gaugcugacc gcgggaaguu gcccggcaag aagcugccgc uggaggugcu caaagagaug 180
gaagccaaug cccggaaagc uggcugcacc aggggcuguc ugaucugccu gucccacauc 240
aagugcacgc ccaagaugaa gaaguucauc ccaggacgcu gccacaccua cgaaggcgac 300
aaagaguccg cacagggcgg cauaggcgag gcgaucgucg acauuccuga gauuccuggg 360
uucaaggacu uggagcccau ggagcaguuc aucgcacagg ucgaucugug uguggacugc 420
acaacuggcu gccucaaagg gcuugccaac gugcaguguu cugaccugcu caagaagugg 480
cugccgcaac gcugugcgac cuuugccagc aagauccagg gccaggugga caagaucaag 540
ggggccggug gugacuag 558
<210>108
<211>720
<212>RNA
<213>人工序列
<220>
<223>目标序列
<400>108
auggugagca agggcgagga gcuguucacc gggguggugc ccauccuggu cgagcuggac60
ggcgacguaa acggccacaa guucagcgug uccggcgagg gcgagggcga ugccaccuac 120
ggcaagcuga cccugaaguu caucugcacc accggcaagc ugcccgugcc cuggcccacc 180
cucgugacca cccugaccua cggcgugcag ugcuucagcc gcuaccccga ccacaugaag 240
cagcacgacu ucuucaaguc cgccaugccc gaaggcuacg uccaggagcg caccaucuuc 300
uucaaggacg acggcaacua caagacccgc gccgagguga aguucgaggg cgacacccug 360
gugaaccgca ucgagcugaa gggcaucgac uucaaggagg acggcaacau ccuggggcac 420
aagcuggagu acaacuacaa cagccacaac gucuauauca uggccgacaa gcagaagaac 480
ggcaucaagg ugaacuucaa gauccgccac aacaucgagg acggcagcgu gcagcucgcc 540
gaccacuacc agcagaacac ccccaucggc gacggccccg ugcugcugcc cgacaaccac 600
uaccugagca cccaguccgc ccugagcaaa gaccccaacg agaagcgcga ucacaugguc 660
cugcuggagu ucgugaccgc cgccgggauc acucucggca uggacgagcu guacaaguag 720
<210>109
<211>936
<212>RNA
<213>人工序列
<220>
<223>目标序列
<400>109
auggcuucca agguguacga ccccgagcaa cgcaaacgca ugaucacugg gccucagugg60
ugggcucgcu gcaagcaaau gaacgugcug gacuccuuca ucaacuacua ugauuccgag 120
aagcacgccg agaacgccgu gauuuuucug caugguaacg cugccuccag cuaccugugg 180
aggcacgucg ugccucacau cgagcccgug gcuagaugca ucaucccuga ucugaucgga 240
auggguaagu ccggcaagag cgggaauggc ucauaucgcc uccuggauca cuacaaguac 300
cucaccgcuu gguucgagcu gcugaaccuu ccaaagaaaa ucaucuuugu gggccacgac 360
uggggggcuu gucuggccuu ucacuacucc uacgagcacc aagacaagau caaggccauc 420
guccaugcug agagugucgu ggacgugauc gaguccuggg acgaguggcc ugacaucgag 480
gaggauaucg cccugaucaa gagcgaagag ggcgagaaaa uggugcuuga gaauaacuuc 540
uucgucgaga ccaugcuccc aagcaagauc augcggaaac uggagccuga ggaguucgcu 600
gccuaccugg agccauucaa ggagaagggc gagguuagac ggccuacccu cuccuggccu 660
cgcgagaucc cucucguuaa gggaggcaag cccgacgucg uccagauugu ccgcaacuac 720
aacgccuacc uucgggccag cgacgaucug ccuaagaugu ucaucgaguc cgacccuggg 780
uucuuuucca acgcuauugu cgagggagcu aagaaguucc cuaacaccga guucgugaag 840
gugaagggcc uccacuucag ccaggaggac gcuccagaug aaauggguaa guacaucaag 900
agcuucgugg agcgcgugcu gaagaacgag caguaa 936
<210>110
<211>1653
<212>RNA
<213>人工序列
<220>
<223>目标序列
<400>110
auggccgaug cuaagaacau uaagaagggc ccugcucccu ucuacccucu ggaggauggc60
accgcuggcg agcagcugca caaggccaug aagagguaug cccuggugcc uggcaccauu 120
gccuucaccg augcccacau ugagguggac aucaccuaug ccgaguacuu cgagaugucu 180
gugcgccugg ccgaggccau gaagagguac ggccugaaca ccaaccaccg caucguggug 240
ugcucugaga acucucugca guucuucaug ccagugcugg gcgcccuguu caucggagug 300
gccguggccc cugcuaacga cauuuacaac gagcgcgagc ugcugaacag caugggcauu 360
ucucagccua ccgugguguu cgugucuaag aagggccugc agaagauccu gaacgugcag 420
aagaagcugc cuaucaucca gaagaucauc aucauggacu cuaagaccga cuaccagggc 480
uuccagagca uguacacauu cgugacaucu caucugccuc cuggcuucaa cgaguacgac 540
uucgugccag agucuuucga cagggacaaa accauugccc ugaucaugaa cagcucuggg 600
ucuaccggcc ugccuaaggg cguggcccug ccucaucgca ccgccugugu gcgcuucucu 660
cacgcccgcg acccuauuuu cggcaaccag aucauccccg acaccgcuau ucugagcgug 720
gugccauucc accacggcuu cggcauguuc accacccugg gcuaccugau uugcggcuuu 780
cggguggugc ugauguaccg cuucgaggag gagcuguucc ugcgcagccu gcaagacuac 840
aaaauucagu cugcccugcu ggugccaacc cuguucagcu ucuucgcuaa gagcacccug 900
aucgacaagu acgaccuguc uaaccugcac gagauugccu cuggcggcgc cccacugucu 960
aaggaggugg gcgaagccgu ggccaagcgc uuucaucugc caggcauccg ccagggcuac1020
ggccugaccg agacaaccag cgccauucug auuaccccag agggcgacga caagccuggc1080
gccgugggca agguggugcc auucuucgag gccaaggugg uggaccugga caccggcaag1140
acccugggag ugaaccagcg cggcgagcug ugugugcgcg gcccuaugau uauguccggc1200
uacgugaaua acccugaggc cacaaacgcc cugaucgaca aggacggcug gcugcacucu1260
ggcgacauug ccuacuggga cgaggacgag cacuucuuca ucguggaccg ccugaagucu1320
cugaucaagu acaagggcua ccagguggcc ccagccgagc uggagucuau ccugcugcag1380
cacccuaaca uuuucgacgc cggaguggcc ggccugcccg acgacgaugc cggcgagcug1440
ccugccgccg ucgucgugcu ggaacacggc aagaccauga ccgagaagga gaucguggac1500
uauguggcca gccaggugac aaccgccaag aagcugcgcg gcggaguggu guucguggac1560
gaggugccca agggccugac cggcaagcug gacgcccgca agauccgcga gauccugauc1620
aaggcuaaga aaggcggcaa gaucgccgug uaa 1653
<210>111
<211>816
<212>RNA
<213>人工序列
<220>
<223>目标序列
<400>111
auggacgcca ugaaacgggg acugugcugc gugcugcugc uguguggcgc cguguucgug60
ucaccuagcc gggugcagcc uaccgagagc aucgugcggu ucccuaacau cacaaaccug 120
uguccauucg gcgagguguu caacgccacc agauucgcca gcguguacgc uuggaauaga 180
aaaagaaucu cuaauugcgu ggccgauuac agcgugcugu acaacagcgc cuccuucagc 240
accuucaagu gcuacggcgu gucccccacc aagcugaacg accugugcuu cacaaauguc 300
uacgccgaua gcuucgugau uagaggcgac gaggugaggc agaucgcucc aggccagacc 360
ggcaagaucg cugauuacaa cuacaagcug ccugaugacu ucacaggaug ugugaucgcc 420
uggaacagca acaaccucga cagcaaggug ggaggcaacu acaauuaccu guauagacug 480
uucagaaagu ccaaccugaa gcccuucgag agagacauca gcaccgaaau cuaccaggcc 540
ggcuccaccc cuugcaacgg aguggaaggc uucaacugcu acuucccccu gcagagcuac 600
gguuuucagc cuaccaacgg cgugggcuac cagcccuacc gcgugguugu gcugagcuuc 660
gagcugcugc acgccccagc uacagugugc ggcccuaaga aaucuaccaa ccuggugaag 720
aacaagggcu auauccccga ggccccuaga gacggccaag ccuacgugcg gaaggacggc 780
gaaugggucc ugcucagcac auuccugggc agcuga 816
<210>112
<211>3345
<212>RNA
<213>人工序列
<220>
<223>目标序列
<400>112
auggccccaa agaagaagcg gaaggucggu auccacggag ucccagcagc caagcggaac60
uacauccugg gccuggacau cggcaucacc agcgugggcu acggcaucau cgacuacgag 120
acacgggacg ugaucgaugc cggcgugcgg cuguucaaag aggccaacgu ggaaaacaac 180
gagggcaggc ggagcaagag aggcgccaga aggcugaagc ggcggaggcg gcauagaauc 240
cagagaguga agaagcugcu guucgacuac aaccugcuga ccgaccacag cgagcugagc 300
ggcaucaacc ccuacgaggc cagagugaag ggccugagcc agaagcugag cgaggaagag 360
uucucugccg cccugcugca ccuggccaag agaagaggcg ugcacaacgu gaacgaggug 420
gaagaggaca ccggcaacga gcuguccacc aaagagcaga ucagccggaa cagcaaggcc 480
cuggaagaga aauacguggc cgaacugcag cuggaacggc ugaagaaaga cggcgaagug 540
cggggcagca ucaacagauu caagaccagc gacuacguga aagaagccaa acagcugcug 600
aaggugcaga aggccuacca ccagcuggac cagagcuuca ucgacaccua caucgaccug 660
cuggaaaccc ggcggaccua cuaugaggga ccuggcgagg gcagccccuu cggcuggaag 720
gacaucaaag aaugguacga gaugcugaug ggccacugca ccuacuuccc cgaggaacug 780
cggagcguga aguacgccua caacgccgac cuguacaacg cccugaacga ccugaacaau 840
cucgugauca ccagggacga gaacgagaag cuggaauauu acgagaaguu ccagaucauc 900
gagaacgugu ucaagcagaa gaagaagccc acccugaagc agaucgccaa agaaauccuc 960
gugaacgaag aggauauuaa gggcuacaga gugaccagca ccggcaagcc cgaguucacc1020
aaccugaagg uguaccacga caucaaggac auuaccgccc ggaaagagau uauugagaac1080
gccgagcugc uggaucagau ugccaagauc cugaccaucu accagagcag cgaggacauc1140
caggaagaac ugaccaaucu gaacuccgag cugacccagg aagagaucga gcagaucucu1200
aaucugaagg gcuauaccgg cacccacaac cugagccuga aggccaucaa ccugauccug1260
gacgagcugu ggcacaccaa cgacaaccag aucgcuaucu ucaaccggcu gaagcuggug1320
cccaagaagg uggaccuguc ccagcagaaa gagaucccca ccacccuggu ggacgacuuc1380
auccugagcc ccgucgugaa gagaagcuuc auccagagca ucaaagugau caacgccauc1440
aucaagaagu acggccugcc caacgacauc auuaucgagc uggcccgcga gaagaacucc1500
aaggacgccc agaaaaugau caacgagaug cagaagcgga accggcagac caacgagcgg1560
aucgaggaaa ucauccggac caccggcaaa gagaacgcca aguaccugau cgagaagauc1620
aagcugcacg acaugcagga aggcaagugc cuguacagcc uggaagccau cccucuggaa1680
gaucugcuga acaaccccuu caacuaugag guggaccaca ucauccccag aagcgugucc1740
uucgacaaca gcuucaacaa caaggugcuc gugaagcagg aagaaaacag caagaagggc1800
aaccggaccc cauuccagua ccugagcagc agcgacagca agaucagcua cgaaaccuuc1860
aagaagcaca uccugaaucu ggccaagggc aagggcagaa ucagcaagac caagaaagag1920
uaucugcugg aagaacggga caucaacagg uucuccgugc agaaagacuu caucaaccgg1980
aaccuggugg auaccagaua cgccaccaga ggccugauga accugcugcg gagcuacuuc2040
agagugaaca accuggacgu gaaagugaag uccaucaaug gcggcuucac cagcuuucug2100
cggcggaagu ggaaguuuaa gaaagagcgg aacaaggggu acaagcacca cgccgaggac2160
gcccugauca uugccaacgc cgauuucauc uucaaagagu ggaagaaacu ggacaaggcc2220
aaaaaaguga uggaaaacca gauguucgag gaaaagcagg ccgagagcau gcccgagauc2280
gaaaccgagc aggaguacaa agagaucuuc aucacccccc accagaucaa gcacauuaag2340
gacuucaagg acuacaagua cagccaccgg guggacaaga agccuaauag agagcugauu2400
aacgacaccc uguacuccac ccggaaggac gacaagggca acacccugau cgugaacaau2460
cugaacggcc uguacgacaa ggacaaugac aagcugaaaa agcugaucaa caagagcccc2520
gaaaagcugc ugauguacca ccacgacccc cagaccuacc agaaacugaa gcugauuaug2580
gaacaguacg gcgacgagaa gaauccccug uacaaguacu acgaggaaac cgggaacuac2640
cugaccaagu acuccaaaaa ggacaacggc cccgugauca agaagauuaa guauuacggc2700
aacaaacuga acgcccaucu ggacaucacc gacgacuacc ccaacagcag aaacaagguc2760
gugaagcugu cccugaagcc cuacagauuc gacguguacc uggacaaugg cguguacaag2820
uucgugaccg ugaagaaucu ggaugugauc aaaaaagaaa acuacuacga agugaauagc2880
aagugcuaug aggaagcuaa gaagcugaag aagaucagca accaggccga guuuaucgcc2940
uccuucuaca acaacgaucu gaucaagauc aacggcgagc uguauagagu gaucggcgug3000
aacaacgacc ugcugaaccg gaucgaagug aacaugaucg acaucaccua ccgcgaguac3060
cuggaaaaca ugaacgacaa gaggcccccc aggaucauua agacaaucgc cuccaagacc3120
cagagcauua agaaguacag cacagacauu cugggcaacc uguaugaagu gaaaucuaag3180
aagcacccuc agaucaucaa aaagggcaaa aggccggcgg ccacgaaaaa ggccggccag3240
gcaaaaaaga aaaagggauc cuacccauac gauguuccag auuacgcuua cccauacgau3300
guuccagauu acgcuuaccc auacgauguu ccagauuacg cuuaa3345
<210>113
<211>185
<212>PRT
<213>人工序列
<220>
<223>目标序列
<400>113
Met Gly Val Lys Val Leu Phe Ala Leu Ile Cys Ile Ala Val Ala Glu
1 5 1015
Ala Lys Pro Thr Glu Asn Asn Glu Asp Phe Asn Ile Val Ala Val Ala
202530
Ser Asn Phe Ala Thr Thr Asp Leu Asp Ala Asp Arg Gly Lys Leu Pro
354045
Gly Lys Lys Leu Pro Leu Glu Val Leu Lys Glu Met Glu Ala Asn Ala
505560
Arg Lys Ala Gly Cys Thr Arg Gly Cys Leu Ile Cys Leu Ser His Ile
65707580
Lys Cys Thr Pro Lys Met Lys Lys Phe Ile Pro Gly Arg Cys His Thr
859095
Tyr Glu Gly Asp Lys Glu Ser Ala Gln Gly Gly Ile Gly Glu Ala Ile
100 105 110
Val Asp Ile Pro Glu Ile Pro Gly Phe Lys Asp Leu Glu Pro Met Glu
115 120 125
Gln Phe Ile Ala Gln Val Asp Leu Cys Val Asp Cys Thr Thr Gly Cys
130 135 140
Leu Lys Gly Leu Ala Asn Val Gln Cys Ser Asp Leu Leu Lys Lys Trp
145 150 155 160
Leu Pro Gln Arg Cys Ala Thr Phe Ala Ser Lys Ile Gln Gly Gln Val
165 170 175
Asp Lys Ile Lys Gly Ala Gly Gly Asp
180 185
<210>114
<211>239
<212>PRT
<213>人工序列
<220>
<223>目标序列
<400>114
Met Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu
1 5 1015
Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly
202530
Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile
354045
Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
505560
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met Lys
65707580
Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu
859095
Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu
100 105 110
Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly
115 120 125
Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr
130 135 140
Asn Tyr Asn Ser His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn
145 150 155 160
Gly Ile Lys Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser
165 170 175
Val Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly
180 185 190
Pro Val Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Ala Leu
195 200 205
Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe
210 215 220
Val Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys
225 230 235
<210>115
<211>311
<212>PRT
<213>人工序列
<220>
<223>目标序列
<400>115
Met Ala Ser Lys Val Tyr Asp Pro Glu Gln Arg Lys Arg Met Ile Thr
1 5 1015
Gly Pro Gln Trp Trp Ala Arg Cys Lys Gln Met Asn Val Leu Asp Ser
202530
Phe Ile Asn Tyr Tyr Asp Ser Glu Lys His Ala Glu Asn Ala Val Ile
354045
Phe Leu His Gly Asn Ala Ala Ser Ser Tyr Leu Trp Arg His Val Val
505560
Pro His Ile Glu Pro Val Ala Arg Cys Ile Ile Pro Asp Leu Ile Gly
65707580
Met Gly Lys Ser Gly Lys Ser Gly Asn Gly Ser Tyr Arg Leu Leu Asp
859095
His Tyr Lys Tyr Leu Thr Ala Trp Phe Glu Leu Leu Asn Leu Pro Lys
100 105 110
Lys Ile Ile Phe Val Gly His Asp Trp Gly Ala Cys Leu Ala Phe His
115 120 125
Tyr Ser Tyr Glu His Gln Asp Lys Ile Lys Ala Ile Val His Ala Glu
130 135 140
Ser Val Val Asp Val Ile Glu Ser Trp Asp Glu Trp Pro Asp Ile Glu
145 150 155 160
Glu Asp Ile Ala Leu Ile Lys Ser Glu Glu Gly Glu Lys Met Val Leu
165 170 175
Glu Asn Asn Phe Phe Val Glu Thr Met Leu Pro Ser Lys Ile Met Arg
180 185 190
Lys Leu Glu Pro Glu Glu Phe Ala Ala Tyr Leu Glu Pro Phe Lys Glu
195 200 205
Lys Gly Glu Val Arg Arg Pro Thr Leu Ser Trp Pro Arg Glu Ile Pro
210 215 220
Leu Val Lys Gly Gly Lys Pro Asp Val Val Gln Ile Val Arg Asn Tyr
225 230 235 240
Asn Ala Tyr Leu Arg Ala Ser Asp Asp Leu Pro Lys Met Phe Ile Glu
245 250 255
Ser Asp Pro Gly Phe Phe Ser Asn Ala Ile Val Glu Gly Ala Lys Lys
260 265 270
Phe Pro Asn Thr Glu Phe Val Lys Val Lys Gly Leu His Phe Ser Gln
275 280 285
Glu Asp Ala Pro Asp Glu Met Gly Lys Tyr Ile Lys Ser Phe Val Glu
290 295 300
Arg Val Leu Lys Asn Glu Gln
305 310
<210>116
<211>550
<212>PRT
<213>人工序列
<220>
<223>目标序列
<400>116
Met Ala Asp Ala Lys Asn Ile Lys Lys Gly Pro Ala Pro Phe Tyr Pro
1 5 1015
Leu Glu Asp Gly Thr Ala Gly Glu Gln Leu His Lys Ala Met Lys Arg
202530
Tyr Ala Leu Val Pro Gly Thr Ile Ala Phe Thr Asp Ala His Ile Glu
354045
Val Asp Ile Thr Tyr Ala Glu Tyr Phe Glu Met Ser Val Arg Leu Ala
505560
Glu Ala Met Lys Arg Tyr Gly Leu Asn Thr Asn His Arg Ile Val Val
65707580
Cys Ser Glu Asn Ser Leu Gln Phe Phe Met Pro Val Leu Gly Ala Leu
859095
Phe Ile Gly Val Ala Val Ala Pro Ala Asn Asp Ile Tyr Asn Glu Arg
100 105 110
Glu Leu Leu Asn Ser Met Gly Ile Ser Gln Pro Thr Val Val Phe Val
115 120 125
Ser Lys Lys Gly Leu Gln Lys Ile Leu Asn Val Gln Lys Lys Leu Pro
130 135 140
Ile Ile Gln Lys Ile Ile Ile Met Asp Ser Lys Thr Asp Tyr Gln Gly
145 150 155 160
Phe Gln Ser Met Tyr Thr Phe Val Thr Ser His Leu Pro Pro Gly Phe
165 170 175
Asn Glu Tyr Asp Phe Val Pro Glu Ser Phe Asp Arg Asp Lys Thr Ile
180 185 190
Ala Leu Ile Met Asn Ser Ser Gly Ser Thr Gly Leu Pro Lys Gly Val
195 200 205
Ala Leu Pro His Arg Thr Ala Cys Val Arg Phe Ser His Ala Arg Asp
210 215 220
Pro Ile Phe Gly Asn Gln Ile Ile Pro Asp Thr Ala Ile Leu Ser Val
225 230 235 240
Val Pro Phe His His Gly Phe Gly Met Phe Thr Thr Leu Gly Tyr Leu
245 250 255
Ile Cys Gly Phe Arg Val Val Leu Met Tyr Arg Phe Glu Glu Glu Leu
260 265 270
Phe Leu Arg Ser Leu Gln Asp Tyr Lys Ile Gln Ser Ala Leu Leu Val
275 280 285
Pro Thr Leu Phe Ser Phe Phe Ala Lys Ser Thr Leu Ile Asp Lys Tyr
290 295 300
Asp Leu Ser Asn Leu His Glu Ile Ala Ser Gly Gly Ala Pro Leu Ser
305 310 315 320
Lys Glu Val Gly Glu Ala Val Ala Lys Arg Phe His Leu Pro Gly Ile
325 330 335
Arg Gln Gly Tyr Gly Leu Thr Glu Thr Thr Ser Ala Ile Leu Ile Thr
340 345 350
Pro Glu Gly Asp Asp Lys Pro Gly Ala Val Gly Lys Val Val Pro Phe
355 360 365
Phe Glu Ala Lys Val Val Asp Leu Asp Thr Gly Lys Thr Leu Gly Val
370 375 380
Asn Gln Arg Gly Glu Leu Cys Val Arg Gly Pro Met Ile Met Ser Gly
385 390 395 400
Tyr Val Asn Asn Pro Glu Ala Thr Asn Ala Leu Ile Asp Lys Asp Gly
405 410 415
Trp Leu His Ser Gly Asp Ile Ala Tyr Trp Asp Glu Asp Glu His Phe
420 425 430
Phe Ile Val Asp Arg Leu Lys Ser Leu Ile Lys Tyr Lys Gly Tyr Gln
435 440 445
Val Ala Pro Ala Glu Leu Glu Ser Ile Leu Leu Gln His Pro Asn Ile
450 455 460
Phe Asp Ala Gly Val Ala Gly Leu Pro Asp Asp Asp Ala Gly Glu Leu
465 470 475 480
Pro Ala Ala Val Val Val Leu Glu His Gly Lys Thr Met Thr Glu Lys
485 490 495
Glu Ile Val Asp Tyr Val Ala Ser Gln Val Thr Thr Ala Lys Lys Leu
500 505 510
Arg Gly Gly Val Val Phe Val Asp Glu Val Pro Lys Gly Leu Thr Gly
515 520 525
Lys Leu Asp Ala Arg Lys Ile Arg Glu Ile Leu Ile Lys Ala Lys Lys
530 535 540
Gly Gly Lys Ile Ala Val
545 550
<210>117
<211>271
<212>PRT
<213>人工序列
<220>
<223>目标序列
<400>117
Met Asp Ala Met Lys Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 1015
Ala Val Phe Val Ser Pro Ser Arg Val Gln Pro Thr Glu Ser Ile Val
202530
Arg Phe Pro Asn Ile Thr Asn Leu Cys Pro Phe Gly Glu Val Phe Asn
354045
Ala Thr Arg Phe Ala Ser Val Tyr Ala Trp Asn Arg Lys Arg Ile Ser
505560
Asn Cys Val Ala Asp Tyr Ser Val Leu Tyr Asn Ser Ala Ser Phe Ser
65707580
Thr Phe Lys Cys Tyr Gly Val Ser Pro Thr Lys Leu Asn Asp Leu Cys
859095
Phe Thr Asn Val Tyr Ala Asp Ser Phe Val Ile Arg Gly Asp Glu Val
100 105 110
Arg Gln Ile Ala Pro Gly Gln Thr Gly Lys Ile Ala Asp Tyr Asn Tyr
115 120 125
Lys Leu Pro Asp Asp Phe Thr Gly Cys Val Ile Ala Trp Asn Ser Asn
130 135 140
Asn Leu Asp Ser Lys Val Gly Gly Asn Tyr Asn Tyr Leu Tyr Arg Leu
145 150 155 160
Phe Arg Lys Ser Asn Leu Lys Pro Phe Glu Arg Asp Ile Ser Thr Glu
165 170 175
Ile Tyr Gln Ala Gly Ser Thr Pro Cys Asn Gly Val Glu Gly Phe Asn
180 185 190
Cys Tyr Phe Pro Leu Gln Ser Tyr Gly Phe Gln Pro Thr Asn Gly Val
195 200 205
Gly Tyr Gln Pro Tyr Arg Val Val Val Leu Ser Phe Glu Leu Leu His
210 215 220
Ala Pro Ala Thr Val Cys Gly Pro Lys Lys Ser Thr Asn Leu Val Lys
225 230 235 240
Asn Lys Gly Tyr Ile Pro Glu Ala Pro Arg Asp Gly Gln Ala Tyr Val
245 250 255
Arg Lys Asp Gly Glu Trp Val Leu Leu Ser Thr Phe Leu Gly Ser
260 265 270
<210>118
<211>1114
<212>PRT
<213>人工序列
<220>
<223>目标序列
<400>118
Met Ala Pro Lys Lys Lys Arg Lys Val Gly Ile His Gly Val Pro Ala
1 5 1015
Ala Lys Arg Asn Tyr Ile Leu Gly Leu Asp Ile Gly Ile Thr Ser Val
202530
Gly Tyr Gly Ile Ile Asp Tyr Glu Thr Arg Asp Val Ile Asp Ala Gly
354045
Val Arg Leu Phe Lys Glu Ala Asn Val Glu Asn Asn Glu Gly Arg Arg
505560
Ser Lys Arg Gly Ala Arg Arg Leu Lys Arg Arg Arg Arg His Arg Ile
65707580
Gln Arg Val Lys Lys Leu Leu Phe Asp Tyr Asn Leu Leu Thr Asp His
859095
Ser Glu Leu Ser Gly Ile Asn Pro Tyr Glu Ala Arg Val Lys Gly Leu
100 105 110
Ser Gln Lys Leu Ser Glu Glu Glu Phe Ser Ala Ala Leu Leu His Leu
115 120 125
Ala Lys Arg Arg Gly Val His Asn Val Asn Glu Val Glu Glu Asp Thr
130 135 140
Gly Asn Glu Leu Ser Thr Lys Glu Gln Ile Ser Arg Asn Ser Lys Ala
145 150 155 160
Leu Glu Glu Lys Tyr Val Ala Glu Leu Gln Leu Glu Arg Leu Lys Lys
165 170 175
Asp Gly Glu Val Arg Gly Ser Ile Asn Arg Phe Lys Thr Ser Asp Tyr
180 185 190
Val Lys Glu Ala Lys Gln Leu Leu Lys Val Gln Lys Ala Tyr His Gln
195 200 205
Leu Asp Gln Ser Phe Ile Asp Thr Tyr Ile Asp Leu Leu Glu Thr Arg
210 215 220
Arg Thr Tyr Tyr Glu Gly Pro Gly Glu Gly Ser Pro Phe Gly Trp Lys
225 230 235 240
Asp Ile Lys Glu Trp Tyr Glu Met Leu Met Gly His Cys Thr Tyr Phe
245 250 255
Pro Glu Glu Leu Arg Ser Val Lys Tyr Ala Tyr Asn Ala Asp Leu Tyr
260 265 270
Asn Ala Leu Asn Asp Leu Asn Asn Leu Val Ile Thr Arg Asp Glu Asn
275 280 285
Glu Lys Leu Glu Tyr Tyr Glu Lys Phe Gln Ile Ile Glu Asn Val Phe
290 295 300
Lys Gln Lys Lys Lys Pro Thr Leu Lys Gln Ile Ala Lys Glu Ile Leu
305 310 315 320
Val Asn Glu Glu Asp Ile Lys Gly Tyr Arg Val Thr Ser Thr Gly Lys
325 330 335
Pro Glu Phe Thr Asn Leu Lys Val Tyr His Asp Ile Lys Asp Ile Thr
340 345 350
Ala Arg Lys Glu Ile Ile Glu Asn Ala Glu Leu Leu Asp Gln Ile Ala
355 360 365
Lys Ile Leu Thr Ile Tyr Gln Ser Ser Glu Asp Ile Gln Glu Glu Leu
370 375 380
Thr Asn Leu Asn Ser Glu Leu Thr Gln Glu Glu Ile Glu Gln Ile Ser
385 390 395 400
Asn Leu Lys Gly Tyr Thr Gly Thr His Asn Leu Ser Leu Lys Ala Ile
405 410 415
Asn Leu Ile Leu Asp Glu Leu Trp His Thr Asn Asp Asn Gln Ile Ala
420 425 430
Ile Phe Asn Arg Leu Lys Leu Val Pro Lys Lys Val Asp Leu Ser Gln
435 440 445
Gln Lys Glu Ile Pro Thr Thr Leu Val Asp Asp Phe Ile Leu Ser Pro
450 455 460
Val Val Lys Arg Ser Phe Ile Gln Ser Ile Lys Val Ile Asn Ala Ile
465 470 475 480
Ile Lys Lys Tyr Gly Leu Pro Asn Asp Ile Ile Ile Glu Leu Ala Arg
485 490 495
Glu Lys Asn Ser Lys Asp Ala Gln Lys Met Ile Asn Glu Met Gln Lys
500 505 510
Arg Asn Arg Gln Thr Asn Glu Arg Ile Glu Glu Ile Ile Arg Thr Thr
515 520 525
Gly Lys Glu Asn Ala Lys Tyr Leu Ile Glu Lys Ile Lys Leu His Asp
530 535 540
Met Gln Glu Gly Lys Cys Leu Tyr Ser Leu Glu Ala Ile Pro Leu Glu
545 550 555 560
Asp Leu Leu Asn Asn Pro Phe Asn Tyr Glu Val Asp His Ile Ile Pro
565 570 575
Arg Ser Val Ser Phe Asp Asn Ser Phe Asn Asn Lys Val Leu Val Lys
580 585 590
Gln Glu Glu Asn Ser Lys Lys Gly Asn Arg Thr Pro Phe Gln Tyr Leu
595 600 605
Ser Ser Ser Asp Ser Lys Ile Ser Tyr Glu Thr Phe Lys Lys His Ile
610 615 620
Leu Asn Leu Ala Lys Gly Lys Gly Arg Ile Ser Lys Thr Lys Lys Glu
625 630 635 640
Tyr Leu Leu Glu Glu Arg Asp Ile Asn Arg Phe Ser Val Gln Lys Asp
645 650 655
Phe Ile Asn Arg Asn Leu Val Asp Thr Arg Tyr Ala Thr Arg Gly Leu
660 665 670
Met Asn Leu Leu Arg Ser Tyr Phe Arg Val Asn Asn Leu Asp Val Lys
675 680 685
Val Lys Ser Ile Asn Gly Gly Phe Thr Ser Phe Leu Arg Arg Lys Trp
690 695 700
Lys Phe Lys Lys Glu Arg Asn Lys Gly Tyr Lys His His Ala Glu Asp
705 710 715 720
Ala Leu Ile Ile Ala Asn Ala Asp Phe Ile Phe Lys Glu Trp Lys Lys
725 730 735
Leu Asp Lys Ala Lys Lys Val Met Glu Asn Gln Met Phe Glu Glu Lys
740 745 750
Gln Ala Glu Ser Met Pro Glu Ile Glu Thr Glu Gln Glu Tyr Lys Glu
755 760 765
Ile Phe Ile Thr Pro His Gln Ile Lys His Ile Lys Asp Phe Lys Asp
770 775 780
Tyr Lys Tyr Ser His Arg Val Asp Lys Lys Pro Asn Arg Glu Leu Ile
785 790 795 800
Asn Asp Thr Leu Tyr Ser Thr Arg Lys Asp Asp Lys Gly Asn Thr Leu
805 810 815
Ile Val Asn Asn Leu Asn Gly Leu Tyr Asp Lys Asp Asn Asp Lys Leu
820 825 830
Lys Lys Leu Ile Asn Lys Ser Pro Glu Lys Leu Leu Met Tyr His His
835 840 845
Asp Pro Gln Thr Tyr Gln Lys Leu Lys Leu Ile Met Glu Gln Tyr Gly
850 855 860
Asp Glu Lys Asn Pro Leu Tyr Lys Tyr Tyr Glu Glu Thr Gly Asn Tyr
865 870 875 880
Leu Thr Lys Tyr Ser Lys Lys Asp Asn Gly Pro Val Ile Lys Lys Ile
885 890 895
Lys Tyr Tyr Gly Asn Lys Leu Asn Ala His Leu Asp Ile Thr Asp Asp
900 905 910
Tyr Pro Asn Ser Arg Asn Lys Val Val Lys Leu Ser Leu Lys Pro Tyr
915 920 925
Arg Phe Asp Val Tyr Leu Asp Asn Gly Val Tyr Lys Phe Val Thr Val
930 935 940
Lys Asn Leu Asp Val Ile Lys Lys Glu Asn Tyr Tyr Glu Val Asn Ser
945 950 955 960
Lys Cys Tyr Glu Glu Ala Lys Lys Leu Lys Lys Ile Ser Asn Gln Ala
965 970 975
Glu Phe Ile Ala Ser Phe Tyr Asn Asn Asp Leu Ile Lys Ile Asn Gly
980 985 990
Glu Leu Tyr Arg Val Ile Gly ValAsn Asn Asp Leu LeuAsn Arg Ile
995 1000 1005
Glu ValAsn Met Ile Asp IleThr Tyr Arg Glu TyrLeu Glu Asn
1010 1015 1020
Met AsnAsp Lys Arg Pro ProArg Ile Ile Lys ThrIle Ala Ser
1025 1030 1035
Lys ThrGln Ser Ile Lys LysTyr Ser Thr Asp IleLeu Gly Asn
1040 1045 1050
Leu TyrGlu Val Lys Ser LysLys His Pro Gln IleIle Lys Lys
1055 1060 1065
Gly LysArg Pro Ala Ala ThrLys Lys Ala Gly GlnAla Lys Lys
1070 1075 1080
Lys LysGly Ser Tyr Pro TyrAsp Val Pro Asp TyrAla Tyr Pro
1085 1090 1095
Tyr AspVal Pro Asp Tyr AlaTyr Pro Tyr Asp ValPro Asp Tyr
1100 1105 1110
Ala
<210>119
<211>6
<212>RNA
<213>人工序列
<220>
<223>EBS1
<400>119
uagggc6
<210>120
<211>6
<212>RNA
<213>人工序列
<220>
<223>EBS1
<400>120
uuaugg6
<210>121
<211>6
<212>RNA
<213>人工序列
<220>
<223>EBS1
<400>121
ucaacg6
<210>122
<211>6
<212>RNA
<213>人工序列
<220>
<223>EBS1
<400>122
uguggc6
<210>123
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1
<400>123
gcccug6
<210>124
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1
<400>124
ccaugg6
<210>125
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1
<400>125
cguuga6
<210>126
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1
<400>126
gccaua6
<210>127
<211>6
<212>RNA
<213>人工序列
<220>
<223>δ及其上游序列
<400>127
ugugcu6
<210>128
<211>6
<212>RNA
<213>人工序列
<220>
<223>δ及其上游序列
<400>128
aaugcu6
<210>129
<211>6
<212>RNA
<213>人工序列
<220>
<223>δ及其上游序列
<400>129
ugcucu6
<210>130
<211>6
<212>RNA
<213>人工序列
<220>
<223>δ及其上游序列
<400>130
ugugcu6
<210>131
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1及其下游序列
<400>131
agcaaa6
<210>132
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1及其下游序列
<400>132
agcagu6
<210>133
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1及其下游序列
<400>133
agagaa6
<210>134
<211>6
<212>RNA
<213>人工序列
<220>
<223>IBS1及其下游序列
<400>134
agcaaa6
