一种基于恒力模式下单分子力谱测量的核酸检测方法
文献发布时间:2024-04-18 20:01:30
技术领域
本发明涉及核酸检测技术领域,尤其是涉及一种基于恒力模式下单分子力谱测量的核酸检测方法。
背景技术
核酸检测是一种重要的生物技术手段,除了在生命科学以及医学研究领域有着广泛的应用,在公共卫生安全、食品安全、生物安全等多个方面也发挥着极为重要的作用。目前常规的核酸检测包括基因测序和荧光定量PCR(qPCR)。由于这两种方法需要聚合酶扩增,因而不适合用于短片段的核酸检测。
单分子技术是近年发展的一种可以检测和操纵单个分子的技术,单分子技术的出现为高灵敏度、高特异性的短片段核酸检测提供了一类新的方法,单分子技术包括单分子荧光显微镜和单分子力谱等。目前,已有研究采用单分子荧光显微镜检测miRNA,其方法是将miRNA固定在玻片表面,随后加入能与miRNA结合的荧光探针,利用单分子荧光显微镜检测荧光探针结合的动力学过程,可以判断待测miRNA的检出,虽然该方法灵敏度高,特异性好,但是该方法需要单分子荧光显微镜等昂贵的仪器,以及荧光信号受光漂白的影响,未被广泛使用;对于单分子力谱,其主要是通过施加力来实时操纵感兴趣的分子,被广泛应用于研究核酸、蛋白等生物大分子,常用的单分子力谱技术有光镊、磁镊、原子力显微镜等,其中磁镊相比于其他单分子力谱技术,有高通量追踪多个分子,且能长时间观测稳定等优势,已被研究者用于核酸分析。相关技术表明,使用力诱导的链侵入可以形成发夹探针,而该探针可用于核酸分析,其具体过程是待测核酸结合探针后,将力降至特定力,计算该力下待测核酸解离前和解离后探针的伸长量的变化,从而确定待测核酸是否结合。然而,仅根据伸长量的变化较难识别核苷酸否含有错配,即可能出现假阳性检测结果。因此,需要寻求一种核酸检测方法,其能够识别碱基错配,且能达到单碱基的特异性。
发明内容
本发明旨在至少解决现有技术中存在的技术问题之一。为此,本发明提出一种基于恒力模式下单分子力谱测量的核酸检测方法,该方法可以识别碱基错配,并且能达到单碱基的特异性,较好地解决了核酸检测过程中相似序列的核酸带来的假阳性的问题。
本发明的一种基于恒力模式下单分子力谱测量的核酸检测方法,包括:
将T型发夹探针与待测核苷酸混合,并对T型发夹探针施加恒定力,通过测量所述T型发夹探针结合所述待测核苷酸后的伸长量变化值计算结合碱基数,和/或通过比较无错配核苷酸和所述待测核苷酸分别与所述T型发夹探针的解离速率常数判断是否存在碱基错配。
根据本发明实施例的方法,至少具有如下有益效果:本发明检测核酸是通过直接在一恒定力下观测目标核酸结合导致的探针伸长量变化这一过程,该方法可以识别对碱基错配进行识别,并且该方法能达到单碱基的特异性识别,较好地解决了核酸检测过程中相似序列的核酸带来的假阳性的问题。
此外,本发明提供的基于恒力模式下单分子力谱测量的核酸检测方法,可以实现快速、无需扩增,无需蛋白参与、无需对待测物进行标记、适用于短序列的核酸检测,能够估计待测核酸与探针结合的碱基数,并且该方法可以实现高特异性的单碱基错配核酸检测。
在本发明的一些实施方式中,所述T型发夹探针包含地高辛标记的手柄、生物素标记的手柄、目标核苷酸结合部分和帽子结构。
在本发明的一些实施方式中,所述探针的目标核苷酸结合部分与所述待测核苷酸部分或完全互补配对;
优选地,所述目标核苷酸结合部分与所述无错配核苷酸完全互补配对。所述无错配核苷酸为用于对照的标准品,其与所述T型发夹探针的目标核苷酸结合部分能够完全匹配。
在本发明的一些实施方式中,所述T型发夹探针的制备方法包括:
步骤S1、将所述地高辛标记的手柄和生物素标记的手柄按1:1摩尔浓度进行连接,得到T型通用手柄;
步骤S2、在T4 DNA连接酶条件下,将所述T型通用手柄、目标核苷酸结合部分和帽子结构按照摩尔浓度1:5:25的比例连接,即得。
在本发明的一些实施方式中,所述待测核苷酸包括单链DNA和RNA中的至少一种;
优选地,所述待测核苷酸的长度大于18nt。
在本发明的一些实施方式中,所述恒定力范围为18pN~60pN;
优选地,所述恒定力范围为35pN~55pN。
在本发明的一些实施方式中所述施加恒定力的工具为单分子力谱工具,其中包括光镊、磁镊、原子力显微镜等。
在本发明的一些实施方式中,所述特征结合时间为待测核苷酸与T型发夹探针结合后在结合态驻留的时间。
在本发明的一些实施方式中,所述解离速率常数的计算公式为:
N=N
其中,N
在本发明的一些实施方式中,计算所述待测核苷酸与所述T型发夹探针结合的碱基数的方法为:
①当所述待测核苷酸为单链DNA时,计算公式为:
②当所述待测核苷酸为RNA时,计算公式为:
其中,ΔN
所述x
其中,f为一固定力,L
在本发明的一些实施方式中,所述x
其中,f为一固定力,h=0.34nm,a
其中,f为一固定力,L
在本发明的一些实施方式中,所述x
其中,f为一固定力,L
本发明的其它特征和优点将在随后的说明书中阐述,并且,部分地从说明书中变得显而易见,或者通过实施本发明而了解。
附图说明
下面结合附图和实施例对本发明做进一步的说明,其中:
图1为本发明实施例T型发夹探针的合成过程图。
图2为本发明实施例恒力模式下单分子力谱核酸检测原理图。
图3为本发明实施例不同力下单链DNA、双链DNA和杂交链的伸长量对比图。
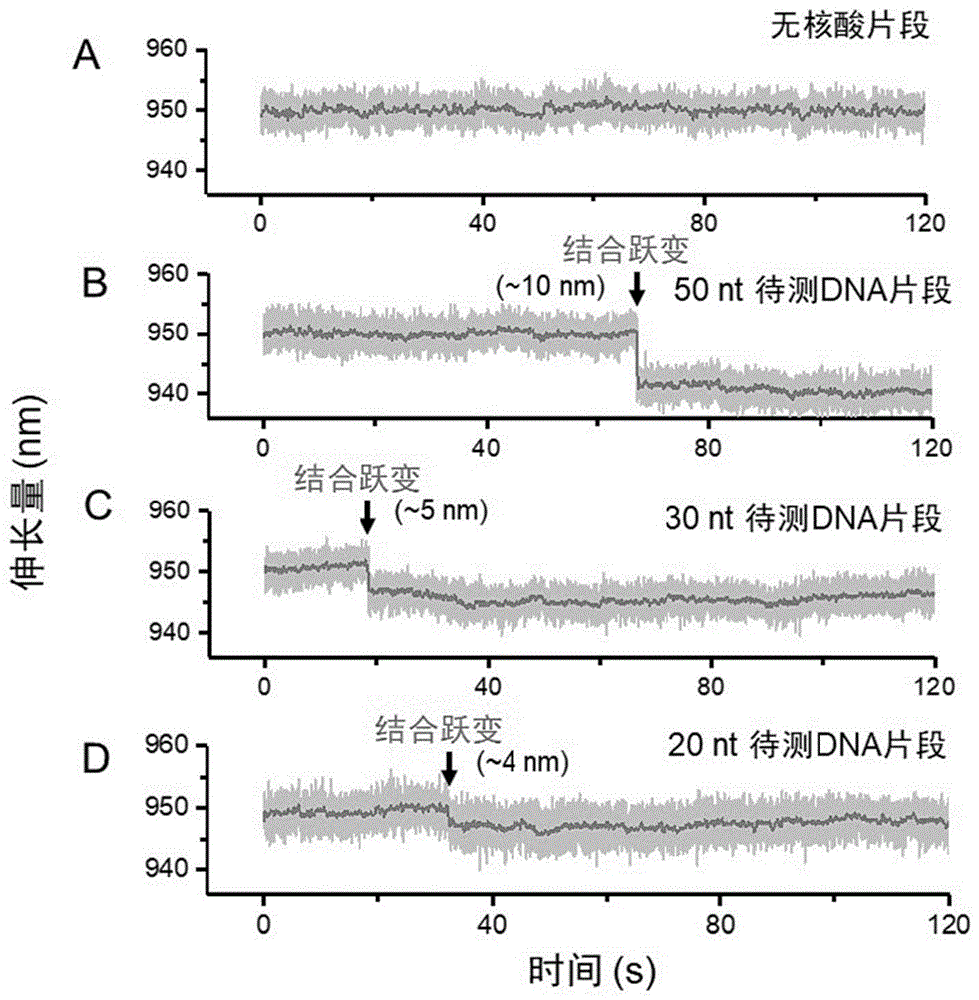
图4为本发明实施例特定恒定力下检测不同的待测核酸片段的结果图,其中A为无核苷酸片段时探针伸长量随时间变化图,B为含50nt待测DNA片段时探针伸长量随时间变化图,C为含30nt待测DNA片段时探针伸长量随时间变化图,D为含20nt待测DNA片段时探针伸长量随时间变化图。
图5为本发明实施例特定恒定力下随机核酸片段的检测结果图。
图6为本发明实施例利用单分子动力学方法比较同一力下20nt无错配核酸片段和含有单碱基错配的核酸片段的解离速率图,其中A为完全互补的无错配核酸片段解离动态变化,B为完全互补的无错配核酸片段与DNA探针的结合态随时间的衰减及由单指数拟合得到解离速率,C为含单碱基错配的核酸片段解离动态变化,D为含单碱基错配的核酸片段与DNA探针的结合态随时间的衰减及由单指数拟合得到解离速率。
图7为本发明实施例不同恒定力下无错配核酸片段和含有单碱基错配的核酸片段的解离速率变化图。
图8为本发明实施例在42.0pN恒力下完全互补的let-7a和含有单碱基错配的let-7c的解离速率图,其中A为测量无错配let-7a miRNA结合-解离的两态动力学过程,B为测量含有单碱基错配let-7c miRNA结合-解离的两态动力学过程,C为无错配let-7a miRNA结合态的衰减曲线,D为单碱基错配let-7a miRNA结合态的衰减曲线。
图9为本发明实施例不同恒定力下无错配的let-7a和含有单碱基错配的let-7c的解离速率变化图。
具体实施方式
以下将结合实施例对本发明的构思及产生的技术效果进行清楚、完整地描述,以充分地理解本发明的目的、特征和效果。显然,所描述的实施例只是本发明的一部分实施例,而不是全部实施例,基于本发明的实施例,本领域的技术人员在不付出创造性劳动的前提下所获得的其他实施例,均属于本发明保护的范围。
本发明的描述中,参考术语“一个实施例”、“一些实施例”、“示意性实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不一定指的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任何的一个或多个实施例或示例中以合适的方式结合。
本发明的描述中,参考术语“解离速率”、“解离速率常数”和“K
实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
T型发夹结构探针的制备
本发明实施例中的T型发夹探针(DNA探针)的设计过程如图1所示,首先本发明以含有地高辛和生物素标记的引物通过PCR得到一段含有地高辛标记(H1)与一段含有生物素标记(H2)的双链DNA手柄;然后对这两段手柄分别进行酶切产生含有4个碱基的黏性末端,并与特定设计的DNA接头片段结合,生成含有35nt黏性末端的地高辛手柄和39nt黏性末端的生物素手柄,其中,35nt的黏性末端与39nt的黏性末端可完全配对,这两段手柄经过退火生成T型通用手柄;再将含有新型冠状病毒N蛋白基因序列的双链DNA、帽子结构和T型通用手柄连接得到最终的DNA探针。
该探针的发夹结构包含了125bp双链茎和5nt的单链环,发夹结构两端分别通过~1.2kb的地高辛手柄连接玻片表面和~1.3kb的生物素标记的双链DNA(dsDNA)手柄连接2.8μm的磁球,从而形成“系链体”,用于核酸检测。
本发明实施例中的T型发夹探针具体制备步骤如下:
1、地高辛标记手柄(H1)制备
(1)以pJM-1质粒为模板进行PCR扩增:将0.4μM HAF引物,0.4μM 1.1kb-BsaI引物,0.4μM dNTPs(TOYOBO,NTP-201),0.02ng/μL pJM-1质粒(实验室制备),1×KOD buffer,0.02U/μL KOD DNA聚合酶(TOYOBO,Code No.KFX-101),混合后,置于PCR仪,设置PCR程序:第一步,起始变性阶段,94℃2min;第二步,变性阶段,98℃10s;第三步,退火阶段,63℃30s;第四步,延伸阶段,68℃,1.2min;第二步至第四步循环36次。
(2)PCR产物纯化:将PCR产物用PCR纯化试剂盒(qiagen,28104)纯化去除多余的引物和酶,将5倍PCR体积的PB buffer与PCR产物混合,混合后加入Qiaquick纯化柱,16000×g离心1min。去除洗脱液后,再加入750μL PE buffer后,16000×g离心1min后,去除洗脱液。将Qiaquick纯化柱继续16000×g离心2min后,去除洗脱液。加入30μL EB buffer,室温静置孵育1min后16000×g离心2min,回收纯化产物。
(3)酶切与纯化:取400nM上述纯化后的PCR产物,在1×cutsmart buffer中与2.8U/μLBsaI内切酶(NEB,R3733S)混匀后,37℃孵育4h;然后用PCR纯化试剂盒纯化酶切产物,得到372nM H1-BsaI cut酶切产物。
(4)H1 adaptor退火:将2μM H1 upper引物与Y-DNA H1 lower在1×annealingbuffer中按照95℃孵育5min,按照-0.1℃/s进行程序降温,直至4℃。其中1×annealingbuffer含有10mM Tris-HCl,pH 8.0,50mM NaCl,1mM EDTA,pH 8.0。
(5)H1 adaptor与H1-BsaI cut酶切产物连接:H1-BsaI cut酶切产物按照1:10的摩尔浓度用T4 DNA连接酶(NEB,M0202S)进行连接,16℃孵育16h。
2、生物素标记手柄(H2)制备
(1)以pJM-1质粒为模板进行PCR扩增:将0.4μM 1-kb-BbsI引物,0.4μM HBR引物,0.4μM dNTPs(TOYOBO,NTP-201),0.02ng/μL pJM-1质粒,1×KOD buffer,0.02U/μL KODDNA聚合酶(TOYOBO,Code No.KFX-101),混合后,置于PCR仪,设置PCR程序:第一步,起始变性阶段,94℃2min;第二步,变性阶段,98℃10s;第三步,退火阶段,63℃30s;第四步,延伸阶段,68℃,1.2min;第二步至第四步循环36次。
(2)PCR产物纯化:将PCR产物用PCR纯化试剂盒(qiagen,28104)纯化去除多余的引物和酶,将5倍PCR体积的PB buffer与PCR产物混合,混合后加入Qiaquick纯化柱,16000×g离心1min。去除洗脱液后,再加入750μL PE buffer后,16000×g离心1min后,去除洗脱液。将Qiaquick纯化柱继续16000×g离心2min后,去除洗脱液。加入30μL EB buffer,室温静置孵育1min后16000×g离心2min,回收纯化产物。
(3)酶切与纯化:取600nM纯化后的PCR产物,在1×cutsmart buffer中与0.76U/μLBbsI内切酶(NEB,R3539S)混匀后,37℃孵育4h;然后用PCR纯化试剂盒纯化酶切产物,纯化后得到566nM H2-BbsI cut酶切产物
(4)H2 adaptor退火:将2μM Y-DNA H2 upper引物与Y-DNA H2 lower在1×annealing buffer中按照95℃孵育5min,按照-0.1℃/s进行程序降温,直至4℃。
(5)H2 adaptor与H2-BbsI cut酶切产物连接:H2-BbsI cut酶切产物按照1:10的摩尔浓度用T4 DNA连接酶进行连接,16℃孵育16h。
3、T型通用手柄的获得
将H1连接产物与H2连接产物按1:1摩尔浓度在1×annealing buffer中,80℃孵育5min,按照-0.1℃/s进行程序降温,直至4℃,即得T型通用手柄。
4、发夹结构的合成
(1)待测核酸结合部分退火:将2μM Trunk forward引物和2μM Trunk reverse引物在1×annealing buffer中按照95℃孵育5min,按照-0.1℃/s进行程序降温,直至4℃。
(2)帽子结构淬火:取5μM Y-DNA cap引物,在95℃加热5min,冰上放置2min,即可。
5、DNA探针合成
将上述T型通用手柄、双链待测核酸结合结构和帽子结构按照摩尔浓度1:5:25的比例用T4 DNA连接酶进行连接,16℃孵育16h,即得DNA探针。
本发明实施例中,所用的引物序列如表1所示。
表1:引物序列
其中,“*”代表地高辛修饰,“#”代表生物素修饰,“/5Phos/”代表5’端磷酸化修饰,红色碱基代表错配碱基。
实施例1
本实施例提供了一种恒力模式下利用单分子力谱技术检测目标核酸与DNA探针结合碱基数方法,具体包括以下步骤:
(1)确定待测物是否含有目的核酸序列。
通过单分子力谱对T型发夹探针施加恒定力,该恒定力的范围在18~60pN之间,在外力的作用下,T型发夹结构探针的发夹结构被打开,使单链DNA暴露出来,如图2所示。
由于同一力下,双链DNA和杂交链比单链DNA的伸长量更短,因此根据DNA探针的伸长量缩短就可判断目标核酸是否与DNA探针结合,确认目标核酸的检出,伸长量关系具体如图3所示。
(2)计算目标核酸与DNA探针结合的碱基数。
根据DNA探针的伸长量变化值可以计算目标核酸与DNA探针的结合的碱基数。
①、当目标核酸为单链DNA时,计算目标核酸与DNA探针的结合的碱基数方法如下:
其中,ΔN
x
型(式3)给出:
其中,f为一固定力,h=0.34nm,a
f
其中,f为一固定力,L
x
其中,f为一固定力,L
②、当目标核酸为RNA时,计算目标核酸与DNA探针结合的碱基数方法如下:
其中,ΔN
其中,f为一固定力,L
(3)通过比较待测核酸与已知序列核酸的解离速率,进行特异性分析。
由于含有一个或几个碱基错配的核酸片段也有可能与探针结合,从而有可能降低核酸检测的准确度,导致出现假阳性。受检测系统探测精度的限制,很难通过单纯测量DNA探针伸长量变化,来区分目标核酸片段与错配核酸片段。因此有必要在上述单纯测量伸长量的单分子实验方法基础上加以改进,来进一步提高检测系统的特异性。考虑到碱基间的错配,即使只有一个错配,由于其导致的“边界效应”,也会带来比较大的自由能“惩罚”,使得错配核酸序列与探针的结合变得不稳定而加速发生解离。但是在传统实验中,例如印迹实验较难应用这一原理来辨别单碱基错配。因为这些实验中就算存在单碱基错配,其解离速率也较低,所以在传统方法中较难测定单碱基错配核酸的解离速率。而当拉力存在时,核酸的自由能发生改变,从而导致结合速率和解离速率出现明显的力学依赖关系,这时可以通过控制力来增大探针与目标核酸序列的解离速率,使得其在实验上更容易被测量。
考虑到探针与单碱基错配片段和无错配片段的解离速率有着不同的力学依赖关系,根据这一原理,本发明设计了单分子动力学检测法来识别单碱基错配,本发明通过在特定张力下,利用单分子力谱记录无错配核酸片段与待测核酸片段在DNA探针上的结合-解离的两态动力学过程,通过测量核酸片段与DNA探针的特征结合时间(在结合态驻留的时间)计算解离速率,并与无错配核酸片段的解离速率相比较,从而实现对无错配核酸片段与单碱基错配核酸片段的区分识别。
实施例2
本实施例提供了一种恒力模式下利用单分子力谱技术检测不同长度核酸序列的方法,其中待测核酸片段序列均来源于新型冠状病毒的N蛋白基因对应的DNA序列,结果表明采用该方法能够检测到长度低至20nt的单链DNA,说明该方法适用于短序列核酸检测,例如检测miRNA。同时,根据目标核酸结合后探针伸长量的变化,可推测目标核酸结合的碱基数。
具体包括以下步骤:
(1)首先将10pM DNA探针利用其一端标记的地高辛与盖玻片表面的地高辛抗体偶联,将探针DNA锚定在盖玻片上,然后将1mg/mL链霉亲和素修饰的磁球M270(Invitrogen,65305)流入并进行孵育10min,利用链霉亲和素与DNA另一端的生物素标记特异性结合,将磁球与DNA探针的另一端连接形成“系链体”;
(2)将10nM不同长度待测核酸片段流入,之后利用磁镊对DNA探针施加35.2pN拉力,使DNA探针的发夹结构打开,暴露单链DNA,如果待测物包含有目标核酸,则能通过碱基互补配对作用与探针结合;
(3)在35.2pN恒力作用下,通过测量探针伸长量的变化值,推测待测核酸片段结合的碱基数。
采用上述方法分别对不同长度待测核酸片段(50nt、30nt和20nt)进行检测,其中对照组未添加核酸片段,检测结果如图4所示,其中灰色线是原始数据,采样率为198Hz,黑色线为平滑后数据,采样率为2Hz。黑色箭头代表在该时刻待测核酸结合至DNA探针上。
结果表明:不同长度待测核酸片段对应的DNA探针伸长量有明显的跃变过程,该过程对应ssDNA与探针的结合。经多次重复测量统计后发现,50nt、30nt和20nt目标核酸的结合导致DNA探针伸长量减少分别是9.48±0.22nm,5.03±0.60nm和3.49±0.85nm,而根据这些值推测结合的碱基数分别是47±1nt,25±3nt和18±4nt。可见,采用本发明恒力模式的核酸检测方法可以用于微小核酸(~20nt)的检测,并且根据伸长变化值可以推测目标核酸结合的碱基数,该方法的长度检测下限能够满足大部分生物标志物的检测需求,例如miRNA,mRNA检测等。
进一步地,由于在实际检测的生物样品中,核酸成分往往较为复杂,除了检测的目标核酸片段外,还可能存在不同序列的核酸片段。为了验证非目标序列的核酸片段是否会结合探针,造成检测假阳性结果,本实施例还采用了上述方法对20nt的随机核酸片段进行了检测,结果如图5所示。
结合图5可以看出,检测随机核酸片段时,其DNA探针的伸长量未发生明显改变,说明该方法受其他非目标核酸的干扰小,可用于复杂样品检测。
实施例3
本实施例提供了一种恒力模式下利用单分子力谱技术识别单链DNA是否存在单碱基错配的方法,具体如下:
(1)首先将10pM DNA探针利用其一端标记的地高辛与盖玻片表面的地高辛抗体偶联,将探针DNA锚定在盖玻片上,然后将1mg/mL链霉亲和素修饰的磁球M270流入并进行孵育10min,利用链霉亲和素与DNA另一端的生物素标记特异性结合,将磁球与DNA探针的另一端连接形成“系链体”。
(2)分别加入100nM 20nt完全互补目标单链DNA和单碱基错配的目标单链DNA,在45.0pN恒力下观测两种核酸片段的结合和解离动态过程,并分别统计完全互补目标单链DNA和单碱基错配的目标单链DNA与DNA探针的结合时间(在结合态驻留的时间),进行单指数拟合得到解离速率(k
N=N
其中,N
(3)通过比较完全互补目标单链DNA或单碱基错配目标单链DNA解离速率,判断是否存在错配。
采用上述方法在45.0pN力下观测上述两种核酸片段的结合和解离动态过程,结果如图6所示,其中图6中的A为完全互补的无错配核酸片段解离动态变化;B为完全互补的无错配核酸片段与DNA探针的结合态随时间的衰减及由单指数拟合得到解离速率;C为含单碱基错配的核酸片段解离动态变化;D为含单碱基错配的核酸片段与DNA探针的结合态随时间的衰减及由单指数拟合得到解离速率。
从图6中可以发现,相同力下无错配核酸片段更倾向于与探针结合,而错配核酸片段则更易解离,且在该恒力下单碱基错配核酸片段的解离速率是无错配核酸片段的20倍。由此可见,依据二者解离速率的显著差异,该方法可以成功识别微小核酸片段的单碱基错配,从而极大程度的降低检测假阳性率。
进一步地,本实施例还采用了上述方法比较了其他力下(38.3~52.3pN)两种核酸片段的解离速率,具体结果如图7所示。
从图7中可以得出,两种核酸片段的解离速率的力学依赖关系显著不同,相比于无错配的核酸片段,单碱基错配的核酸片段与探针结合更不稳定,在更低力下即达到较高的解离速率,再次验证了根据解离速率的差异可以识别错配核酸片段。
实施例4
miRNA同一家族成员间具有很高的相似性,部分同一家族的miRNA仅仅只相差1~2个碱基,例如let-7c miRNA与let-7a miRNA相比只存在单个碱基错配,二者的鉴定也是核酸检测的一个难点。
本实施例提供了一种恒力模式下利用单分子力谱技术识别RNA是否错配的方法,采用该方法分别对无错配let-7a miRNA和单碱基错配let-7c miRNA进行单碱基错配检测,具体步骤如下:
(1)首先将10pM DNA探针利用其一端标记的地高辛与盖玻片表面的地高辛抗体偶联,将探针DNA锚定在盖玻片上,然后将1mg/mL链霉亲和素修饰的磁球M270流入并进行孵育10min,利用链霉亲和素与DNA另一端的生物素标记特异性结合,将磁球与DNA探针的另一端连接形成“系链体”;
(2)分别加入100nM对无错配let-7a miRNA和单碱基错配let-7c miRNA,在42.0pN力下观测两种核酸片段的结合和解离动态过程,并分别统计let-7a miRNA与let-7c miRNA与DNA探针的结合时间(在结合态驻留的时间),进行单指数拟合得到解离速率(k
(3)通过比较无错配let-7a miRNA和单碱基错配let-7c miRNA解离速率,判断是否存在错配。
采用上述方法在42.0pN力下观测两种miRNA的结合和解离动态过程如图8所示,其中图8中A和B为在42.0pN恒定力下测量无错配let-7a miRNA和含有单碱基错配let-7cmiRNA结合-解离的两态动力学过程;C和D为在42.0pN的恒定力下,无错配let-7a miRNA和含有单碱基错配let-7c miRNA结合态的衰减曲线,通过单指数拟合可得k
从图8中可以看出,在42.0pN恒力下正配(完全互补)与错配的miRNA的两态分布明显不同,正配let-7a miRNA更倾向于与探针结合,而错配let-7c miRNA则更易与探针解离;通过测量let-7a miRNA和let-7c miRNA与DNA探针在结合态的驻留时间拟合得到解离速率(如图8中的C和D所示),发现在同一恒力(即42.0pN)下let-7c miRNA的解离速率是let-7amiRNA的13倍,这可能是由于含有单碱基错配的let-7c miRNA结合探针更不稳定所致。因此,通过比较同一恒力下的解离速率,可以辨别两种miRNA,从而实现单碱基特异性检测miRNA。
进一步地,本实施例还采用了上述方法比较了其他力下(37.9~48.3pN)上述两种miRNA核酸片段的解离速率,具体结果如图9所示。
图9是通过统计两种miRNA不同力下的特征结合时间,从而得到解离速率与力的依赖关系,从图9中可以看出两种miRNA在不同力下的解离速率均不相同,并且let-7c miRNA在更低的作用力下就可达到较高的解离速率。基于这些结果,可以认为恒力模式的单分子动力学方法不仅可以识别ssDNA单碱基错配,也可以识别miRNA单碱基错配,这也说明该方法有希望成为一种高效快捷和高特异性miRNA检测手段。
综上所述,本发明提出了一种基于恒力模式下单分子力谱测量的核酸检测方法及其应用,其可以实现快速、无需扩增,无需蛋白参与、无需对待测物进行标记、适用于短序列的核酸检测,能够估计待测核酸与探针结合的碱基数、并且该方法可以实现高特异性的单碱基错配核酸检测。
上面对本发明实施例作了详细说明,但是本发明不限于上述实施例,在所属技术领域普通技术人员所具备的知识范围内,还可以在不脱离本发明宗旨的前提下作出各种变化。此外,在不冲突的情况下,本发明的实施例及实施例中的特征可以相互组合。
