与肝癌相关的生物标识物及治疗靶点的应用及筛选方法
文献发布时间:2024-04-18 20:01:55
技术领域
本发明涉及肝癌早期诊断治疗领域,尤其是涉及与肝癌相关的微生物作为肝癌生物标识物及治疗靶点的应用及筛选方法。
背景技术
肝癌作为一种发病率很高的恶性肿瘤,是导致全球癌症相关性死亡的第三大病因,然而大多数患者在接受治疗时基本都处于晚期而导致疾病治疗效果很差。因此,我们急需提高肝癌早期诊治水平以改善肝癌患者的生存结局。
同时,肝癌作为一种多因素复杂性恶性疾病,与机体免疫、代谢、遗传息息相关,近几十年来关于其发生发展机制的研究层出不穷,但可用的有效治疗方案选择仍然十分有限。
随着高通量测序技术的发展及其在病原微生物学方面的不断进步,实现对大量分离菌更准确、更高通量、更快速及低成本的分析,使得肿瘤微生态领域出现了跳跃性的发展。多项研究显示瘤内微生物群与肿瘤的发生发展密切相关,组织中的微生物参与调控癌症易感性和肿瘤进展,肿瘤组织中的细菌还通过影响代谢和免疫系统信号来影响肿瘤的发生和进展,这可能会为肿瘤防治提供新的视角和思路,但其在肝癌中的分布特征及微生态对肝癌调控作用网络尚未得到全面阐释。
发明内容
本发明目的在于提供一种与肝癌相关的生物标识物及治疗靶点的应用及筛选方法。
本发明通过对比接受手术治疗的HBV相关肝癌患者肝癌组织和癌旁组织的微生物多样性分析发现了与肝癌发生正相关的微生物菌属,包括Acinetobacter、Bacteriaunclassified、Brevundimonas、Corynebacterium、Curvibacter、Delftia、Glycocaulis、Paraburkholderia、Pelagibacterium、Phyllobacteriaceae、Pseudomonas、Ruminococcaceae、Sediminibacterium、Sphingomonas、Staphylococcus、Stenotrophomonas;以及与肝癌发生负相关的微生物菌属,包括Clostridiales、Halomonas、Microcella、Nesterenkonia;
上述微生物在制备肝癌诊断试剂、治疗或预防肝癌药物、抑制肝癌增殖药物中应用,提高肝癌早期诊治水平以改善肝癌患者的生存结局。
本发明所述的与肝癌相关的生物标识物及治疗靶点的筛选方法,包括以下步骤:
S1,收集接受手术治疗的HBV相关肝癌患者癌组织及对应的癌旁组织样本;
S2,提取所述样本的DNA,进行PCR扩增、产物纯化,再进行定量分析和均一化处理,构建测序文库,并使用Illumina Miseq platform对所述测序文库进行质检;
S3,以步骤S2中所述的DNA为模板,使用特异性引物对其中的微生物菌16S rDNA基因V3-V4可变区进行PCR扩增、测序拼接、质控、过滤得到有效数据;
S4,通过OUT聚类分析、群落组成分析、Alpha多样性分析、β多样性分析、微生物群落差异分析、微生物菌群结构和功能分析,得到肝癌患者癌组织及正常组织的差异微生物;
S5,采用PICRUSt2进行微生物群功能的预测分析,挑选丰度前10的通路;采用富集分析肝癌组织和癌旁正常组织中富集信号通路的平均相对丰度,确定肝癌组织和癌旁正常组织中的差异性通路;结合肝癌患者癌组织及正常组织的所述差异微生物,构建微生物菌落共发生网络图,得到所述与肝癌发生正或负相关的微生物菌属。
进一步地,S4步中所述微生物群落差异分析、微生物菌群结构和功能分析包括以下内容:采用Metastats进行所述差异微生物群落属水平分析,得到了丰度排名前20的微生物属;采用Linear discriminant analysis Effect Size方法分析肝癌组织和癌旁正常组织中从门到属水平的微生物群落,找到造成肝癌和癌旁微生物差异的的核心贡献菌;进一步取metagenomeSeq程序包, 设置筛选阈值p-value <0.05;Metastats程序包,设置筛选阈值p-value <0.05;Lefse分析,设置筛选阈值LDA score> 2,p- value<0.05,筛选的差异结果的交集得到20个所述差异微生物。
本发明优点在于阐释了与HBV相关的肝癌组织及癌旁组织中存在着独特的细菌,且在肿瘤调控中发挥了关键作用。这些菌群可以帮助诊断HBV相关肝癌的发生,或利用肿瘤组织中菌群信息帮助判断肿瘤进展和预后。针对特种细菌或者细菌相关代谢产物,从而开发工程菌,或者特异性抑制肿瘤生长和促进肿瘤细胞凋亡的微生物代谢产物,为探索肿瘤菌群与肿瘤相互作用的深层机制提供思路。也即本发明一定程度上揭示了肝癌组织与正常组织中微生物的差异化。探究肿瘤中菌群生理调节功能与肿瘤发生的相关性。该结论可作为制备肝癌诊断试剂、治疗或预防肝癌药物、抑制肝癌细胞增殖药物的新型生物标志物及治疗靶点,提高肝癌早期诊治水平以改善肝癌患者的生存结局的效果。
附图说明
图1是本发明所述方法中OUT聚类分析示意图。
图2是本发明所述方法中微生物菌属群落结构分布差异图。
图3是本发明所述方法中微生物的Alpha多样性分析图。
图4是本发明所述方法中微生物的β多样性分析图。
图5是本发明所述方法中LDA分析法展示的差异微生物图。
图6是本发明所述方法中取metagenomeSeq程序包、Metastats程序包、Lefse分析差异结果交集,筛选出20个差异微生物的示意图。
图7是本发明所述方法中的了PICRUSt2进行菌群功能的预测分析,挑选丰度前10的通路示意图。
具体实施方式
下面将对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明的一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
本发明采用多组学的方法探究肝癌肿瘤微环境,从而为肝脏肿瘤的诊断和治疗策略提供更全面的理解。本发明收集来自浙江大学附属第一医院的47例与HBV相关的肝癌患者的癌组织及癌旁组织的肝脏样本进行微生物16s测序。
实施例1:筛选与肝癌发生具有相关性的一组微生物
首先,本发明筛选了与肝癌发生具有相关性的一组微生物,具体筛选方法如下:
S1为了探究肝癌患者癌组织和癌旁正常组织的微生物菌落结构及差异、物种间的进化关系以及群落的多样性,本发明收集来自浙江大学附属第一医院的接收手术治疗的47例与HBV相关的对肝癌患者癌组织及癌旁组织的肝脏样本进行微生物多样性检测。
S2,提取以上样本的DNA, 进行PCR扩增及产物纯化,经过定量分析和均一化处理后构建测序文库,并使用Illumina Miseq platform (Illumina, San Diego, CA)进行文库质检。
S3,以每个患者癌组织及癌旁组织的肝脏样本DNA为模板,使用特异性引物对其中的微生物菌进行16S rDNA基因V3-V4可变区PCR扩增,测序得到原始数据,并进行拼接、质控和过滤得到有效数据。
S4,接着进行OUT聚类分析、群落组成分析、Alpha多样性分析(α多样性),β多样性分析,微生物群落差异分析,微生物菌群结构和功能分析,分析肝癌患者癌组织及正常组织的差异微生物,得到与肝癌严重程度具有相关性的20个微生物;
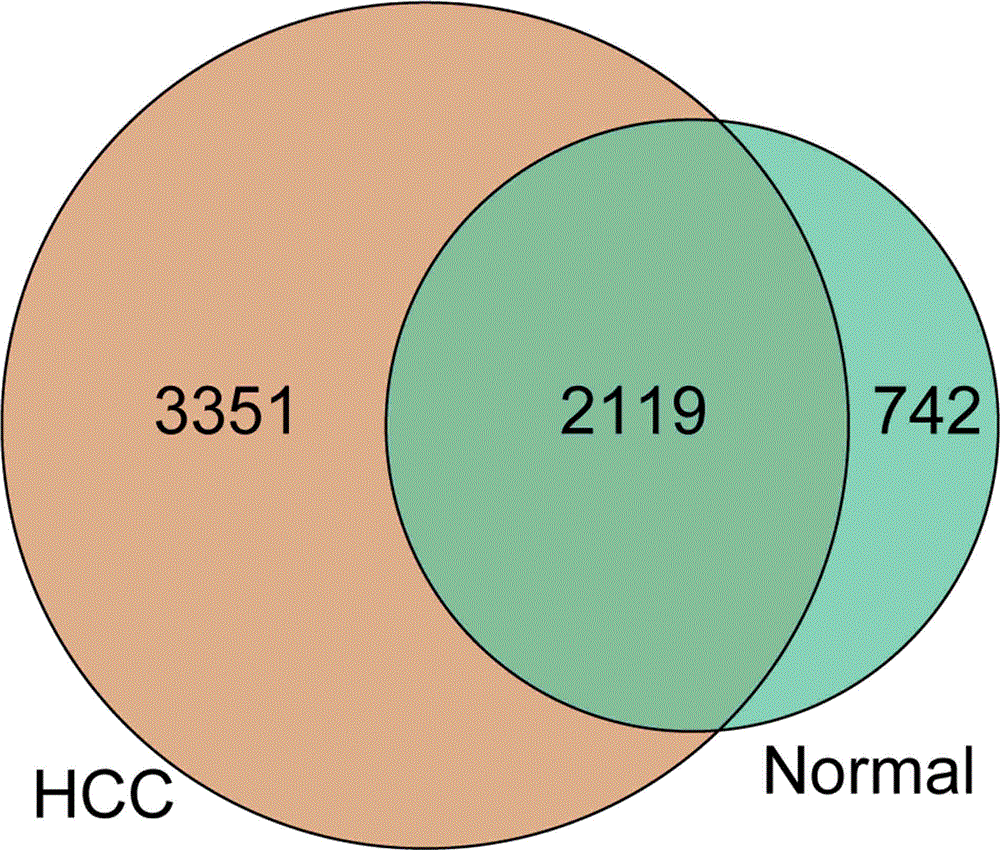
发明人对47对肝癌患者癌组织和癌旁正常组织样本进行微生物检测,其中OUT聚类分析显示,其中2119个OTUs(中文释义:可执行的分类操作单元,英文全称:Operationaltaxonomic units,主要是按照16sDNA测序结果计算遗传距离,人为规定遗传距离,然后依此距离区别微生物类群的分类单)在肝癌组织和癌旁组织中共同存在,742个OTUs只在癌旁组织中存在,3351个OTUs只在癌组织中存在(如图1所示,其中HCC代表癌组织,Normal代表癌旁组织),发明人发现肝癌组织与癌旁组织在门、科、属水平上的微生物种群结构不同。进一步对OTUs水平的菌落结构进行分析,发现肝癌患者癌组织和癌旁组织样本中的菌属群落结构分布差异显著,如图2所示。
微生物的Alpha多样性分析发现,Ace指数,Chao指数,Invsimpson指数,Shannon指数,Simpson指数和Sobs指数在肝癌组织的微生物中显著高于癌旁正常组织,具有显著差异(p<0.0001),如图3所示为Alpha多样性分析中p<0.0001时,癌组织HCC和癌旁组织Normal的Shannon指数分析图。PcoA(中文释义:主坐标分析;英文全拼:principal co-ordinatesanalysis)和PCA (中文释义:主成分分析;英文全拼:Principal Components Analysis)分析也发现肝癌组织和癌旁组织中微生物具有较好的分区聚类。
进一步,微生物的β多样性分析同样表明肝癌组织和癌旁组织中微生物群落结构具有显著的差异,如图4所示,显示了PC1:79.64%, PC2:10.27%时,肝癌组织和癌旁组织中微生物群落的分区聚类情况。
为了探究肝癌组织和癌旁正常组织中微生物群落结构的具体差异,采用Metastats进行差异属水平分析,得到了丰度排名前20的微生物属;采用Lineardiscriminant analysis Effect Size方法(英文缩写为:Lefse,是一种用于发现和解释高维度数据的基因、通路和分类单元相关等生物标识的分析工具,通常可以进行两个或多个分组的比较,能够找出组与组之间有差异的生物标识)分析肝癌组织和癌旁正常组织中从门到属水平的微生物群落,以Cladograms(中文:物种树/进化分支树)图进行展示,找到可能造成肝癌组织和癌旁正常组织中微生物差异的的核心贡献菌,如图5所示,为肝癌组织和癌旁正常组织中LDA>3. 6的微生物;进一步采用metagenomeSeq程序包, 设置筛选阈值P-value <0.05,Metastats程序包,设置筛选阈值p-value <0.05,和Lefse分析设置筛选阈值LDA score> 2,p- value<0.05,筛选核心贡献菌,并取这三个方法筛选的差异结果的交集,得到20个微生物种,如图6所示,使用Cladogram进化树展示差异菌之间的进化关系。
接着,发明人确定了这20个微生物种与肝癌发生的具体相关性
S5,为进一步了解这20个微生物种对肝癌组织和癌旁组织所产生的功能,发明人采用了PICRUSt2进行菌群功能的预测分析,挑选丰度前10的通路,用Circos(是一个基因组数据可视化软件,能够将基因组数据映射到环形的基因组坐标上,用相互嵌套的环道来展示基因组数据,还可以通过连接线来呈现基因组区块之间的关系)图来展示,如图7所示。接着再采用富集分析肝癌组织和癌旁组织中富集信号通路的平均相对丰度,确定肝癌组织和癌旁正常组织中的差异性通路。
以20个微生物种、丰度前10的通路以及确定的肝癌组织和癌旁组织中的差异性通路为基础,构建微生物菌落共发生网络图,确定了与HBV相关的肝癌发生呈正或负相关的微生物菌落分布;
其中,正相关的微生物菌落包括:Acinetobacter(厌氧菌)、Bacteriaunclassified(未定义细菌属,具体见)、Brevundimonas(短波单胞菌属)、Corynebacterium(棒状杆菌属)、Curvibacter(弯曲杆菌属)、Delftia(代尔夫特菌属)、Glycocaulis(甘草菌属)、Paraburkholderia(副伯克霍尔德氏菌属)、Pelagibacterium(浮游杆菌属)、Phyllobacteriaceae(叶杆菌属)、Pseudomonas(假单胞菌属)、Ruminococcaceae(疣微菌属)、Sediminibacterium(细小杆菌属)、Sphingomonas(鞘氨醇单胞菌)、Staphylococcus(葡萄球菌属)、Stenotrophomonas(寡养单胞菌属);
负相关的微生物菌落包括:Clostridiales(梭菌属)、Halomonas(盐单胞菌属)、Microcella(微细胞)、Nesterenkonia(涅斯捷连科氏菌)。
实施例2 验证与肝癌发生正或负相关的微生物的可靠性
本发明通过对15对肝癌患者癌组织和癌旁正常组织样本的代谢组学分析,使用LogFC值为1.5,VIP≥1,p-value ≤ 0.05 且 Fold change ≥ 1.5或≤0.667为筛选阈值,获得差异代谢物;进一步根据肝癌组织和癌旁正常组织中差异代谢物水平,使用FDR<0.05,|log2(FC T/N)|>1筛选,得到24个差异代谢物。通过R(v3.2.1) 分析软件计算这24个差异代谢物的ROC曲线,发现Acetaminophen(对乙酰氨基酚)、L-Arginine(L-精氨酸)、O-Phosphoethanolamine(邻磷酸乙醇胺)、Rosmarinic acid(迷迭香酸)在肝癌组织中水平显著升高,其他20个差异代谢物如牛磺酸(Bovinocidin),在肝癌组织中水平显著降低。此时观察与肝癌严重程度正或负相关的微生物后发现,当与肝癌严重程度负相关的微生物在肝癌中丰度显著升高时,代谢物Acetaminophen(对乙酰氨基酚)、L-Arginine(L-精氨酸)、O-Phosphoethanolamine(邻磷酸乙醇胺)、Rosmarinic acid(迷迭香酸)在肝癌组织中水平显著降低,其他20个差异代谢物如牛磺酸(Bovinocidin),在肝癌组织中水平显著升高。也就是说,本发明所说的与HBV相关的肝癌发生正或负相关的微生物菌落能够有效的指示肝癌发生,可以作为肝癌的治疗或预防肝癌药物、抑制肝癌细胞增殖药物的新型治疗靶点,为肝癌的治疗提供了新的思路。
