与豇豆千粒重主效QTL紧密连锁的分子标记及其应用
文献发布时间:2024-04-18 20:01:55
技术领域
本发明属于生物分子技术领域,涉及豇豆千粒重基因紧密连锁的分子标记的引物及其选育方法。
背景技术
豇豆(Vigna unguiculata(L.)Walp.)(2n=2x=22)隶属豆科(Fabaceae)菜豆族(Trib.PhasoleaeDC.)豇豆属(Vigna Savi),是热带和亚热带地区重要的豆类作物之一。豇豆为自花授粉二倍体,基因组大小约620Mb。豇豆起源于非洲,野生豇豆在西非或东非地区首先被驯化成粮用豇豆(普通豇豆),随后粮用豇豆向东传播,在东南亚或南亚地区进一步驯化出菜用豇豆(长豇豆)。豇豆是一种多用途作物,其幼嫩豆荚可以做蔬菜用,干籽粒可以做粮食食用,成熟后植株整体可做饲料使用。豇豆籽粒富含蛋白质,是非洲地区发展中国家人民重要的蛋白来源,也是主要的粮食作物。在我国,豇豆干籽粒主要做杂粮消费,是我国六大食用豆类作物之一,此外,长豇豆也是我国重要的豆类蔬菜作物。
籽粒重量是豇豆重要的产量性状之一,选育千粒重较大的品种是豇豆育种家长期关注的重要目标。和其它产量因素一样,粒重是一个复杂的数量性状,受环境影响较大,育种上基于表型选择的精确性和效率较低。挖掘控制粒重的关键基因/QTL,开发与之连锁的分子标记,是开展豇豆产量性状分子育种的前提和关键。国外研究者曾利用SSR标记,从一个双亲群体中定位出7个控制粒重的QTL(Andargie et al.2014),并利用关联分析的方法定位出13个与粒重显著相关的SNP位点(Lo et al.2019),但在国内尚未有豇豆粒重相关基因/QTL定位的报道。
随着豇豆高质量基因组的释放和测序技术的快速发展,通过重测序技术可以快速获得不同种质高质量的SNP基因型数据,结合其表型数据进行全基因组范围内的关联分析,可以挖掘出控制目标性状遗传变异,为豇豆分子育种提供目标QTL/基因。单核苷酸多态性标记(single nucleotide polymorphism,SNP)由于其数量多、多态性丰富逐步成为豇豆遗传研究的主流标记,近年来,结合竞争性等位变异特异性PCR(Kompetitive AlleleSpecific PCR,KASP)技术,可以针对海量的SNP标记进行灵活、个性化的筛选和应用,成为作物分子育种研究中有力的遗传工具。因此,利用GWAS快速挖掘控制豇豆粒重的QTL/基因,并开发基于PCR技术的KASP标记,将有助于促进豇豆产量性状分子育种。
发明内容
有鉴于此,本发明的目的在于提供与豇豆千粒重主效QTL紧密连锁的分子标记及其引物,并提供利用该标记进行对千粒重辅助育种的方法。该方法首先利用全基因组关联分析的方法,挖掘与千粒重显著相关的QTL或基因组区段,再挑选与基因紧密连锁的SNP转化为KASP标记进行验证,并利用该标记扩增需要鉴定的材料,根据其基因型判断样本千粒重的有利等位变异,从而为豇豆千粒重性状的遗传改良提供新的技术支撑。
为达到上述目的,本发明提供如下技术方案:
1、本发明首先构建了一个含344份种质的豇豆核心种质群体,随后对其进行千粒重表型和基因型鉴定,千粒重表型于2021年在杭州和广州两地分别进行,采用随机区组设计,每份材料种植4穴,每穴保留2-3株材料,参照《豇豆种质资源描述规范和数据标准》,每份材料收获后取200-300粒种子,利用自动考种仪计算种子个数和重量,最终获得千粒重的表型数据。利用重测序技术对344份种质进行基因型鉴定,鉴定出1,262,497个高质量SNP用于群体结构分析和关联分析,结合该群体的千粒重表型,利用GEMMA软件在全基因组范围内鉴定出2个与千粒重显著相关的区段,其中TSW-9.1位于9号染色体1074288-1288721bp之间,其峰值位置为1189905,LOD值达到6.51,该峰值标记位于一个未知蛋白基因VuG9809G001610下游1899bp处,推测该基因可能为TSW-9.1的候选基因(VuTSW1)。本发明根据VuG9809G001610的基因组序列在344份种质中的基因型,挑选出位于1186708bp处的SNPG/A转化为一个KASP标记2_13994,发现该标记可以指示千粒重基因VuTSW1的筛选,利用该标记扩增需要鉴定的材料,根据其基因型即可判断样本是否携带控制嫩千粒重的有利等位变异。
2、VuTSW1基因在1186708bp处的SNP为G/A,其在群体中序列变异为:TCGCCATTGCCTTGCACGCCAAAAGGGTTGTGTCGAAATCGCGGTGGGACGCGAAAGTT G[G/A]TGCCTTTAAGATGTGGGGTTGGTCTGAGGAAATGGTTATTGATTCTTTCAGGAAGC ATCC
利用该序列设计2_13994的两条特异性引物和一条通用引物,所述引物序列如下:
2_13994Primer1:
5’-GAAGGTGACCAAGTTCATGCTAATCGCGGTGGGACGCGAAAGTTGG-3’;
2_13994Primer2:
5’-GAAGGTCGGAGTCAACGGATTAATCGCGGTGGGACGCGAAAGTTGA-3’;
2_13994Primer1和2_13994Primer2为2条特异性引物,分别连接不同的荧光基团;
2_13994Primer_Common:5’-CCATTTCCTCAGACCAACCCCA-3’。
进一步,其中2_13994Primer1连接FAM基团,2_13994Primer2连接HEX基团。
3、以上技术方案中所述KASP引物、含有KASP引物的试剂或试剂盒在检测豇豆千粒重基因中的应用。
4、利用豇豆千粒重基因紧密连锁的分子标记的KASP引物进行选育不同千粒重豇豆品种的方法,所述方法具体包括以下步骤:
S1、提取豇豆植株样本基因组DNA;
S2、以豇豆植株样本基因组DNA为模板,利用2_13994的KASP引物进行KASP反应检测;
S3、读取KASP反应检测的荧光信号,若豇豆植株样本基因组DNA显示2_13994Primer1所带荧光基团信号,则即可判断该样本携带小粒基因型;若豇豆植株样本基因组DNA显示2_13994Primer2所带荧光基团信号,则即可判断该样本携带大粒基因型。
进一步,选育不同千粒重豇豆品种的方法中,其中Primer1连接FAM基团,Primer2连接HEX基团。
进一步,选育不同千粒重豇豆品种的方法中,KASP反应检测还包括2X KASPMaster mix试剂。
进一步,选育不同千粒重豇豆品种的方法中,KASP反应检测的PCR体系为:DNA0.8μl,2x KASP Master mix 0.75μl,Primer mix 0.05μl。
进一步,选育不同千粒重豇豆品种的方法中,KASP反应检测的PCR反应程序为94℃预变性15分钟,94℃变性20秒,61~55℃梯度复性60秒,每个循环复性温度降低0.6℃,55℃延伸60秒,循环10次;再94℃变性20秒,55℃复性60秒,延伸60秒,循环26次。
本发明的有益效果在于:
(1)本发明提供了一个与豇豆千粒重基因紧密连锁的SNP位点2_13994,将SNP转化为KASP标记进行验证,还提供了相关的KASP引物,并利用该标记扩增需要鉴定的材料,根据其基因型判断样本千粒重的有利等位变异,从而为豇豆千粒重性状的遗传改良提供新的技术支撑。
(2)利用本发明提供的分子标记及其引物进行豇豆不同千粒重性状的筛选,可以在短时间内进行高通量检测,大大节省人力物力,为豇豆遗传多样性分析、基因定位及未来的分子育种提供充足的标记资源。与传统基于表型选择相比,选择结果更准确,不受测量工具、人的影响,便于在早期育苗期就可快速筛选出具有大粒性状的豇豆品种或品系,提高豇豆产量。结合KASP的选育方法也大大提高育种效率,从而加快豇豆育种进程。
附图说明
为了使本发明的目的、技术方案和有益效果更加清楚,提供如下附图进行说明:
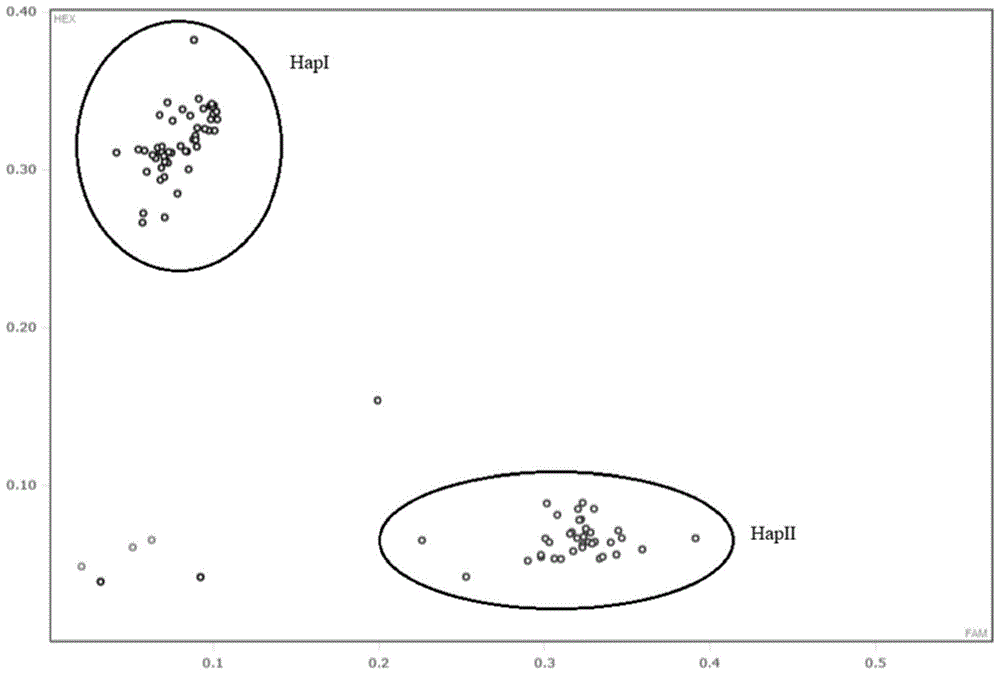
图1为2_13994标记位点相关基因KASP标记分型图。
图2为2_13994标记位点千粒重等位变异差异图。
具体实施方式
下面将结合附图,对本发明的优选实施例进行详细的描述。需要理解的是以下实施例的给出仅是为了起到说明的目的,并不是用于对本发明的范围进行限制。本领域的技术人员在不背离本发明的宗旨和精神的情况下,可以对本发明进行各种修改和替换。
下述实施例中所使用的实验方法如无特殊说明,通常按照常规条件或按照制造厂商所建议的条件。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1
本发明首先构建了一个含344份种质的豇豆核心种质群体,随后对其进行千粒重表型、荚长表型和基因型鉴定,千粒重表型于2021年在杭州和广州两地分别进行,采用随机区组设计,每份材料种植4穴,每穴保留2-3株材料,参照《豇豆种质资源描述规范和数据标准》,每份材料收获后取200-300粒种子,利用自动考种仪计算种子个数和重量,最终获得千粒重的表型数据。利用重测序技术对344份种质进行基因型鉴定,鉴定出1,262,497个高质量SNP用于群体结构分析和关联分析,结合该群体的千粒重表型,利用GEMMA软件在全基因组范围内鉴定出2个与千粒重显著相关的区段,其中TSW-9.1位于9号染色体1074288-1288721bp之间,其峰值位置为1189905,LOD值达到6.51,该峰值标记位于一个未知蛋白基因VuG9809G001610下游1899bp处,推测该基因可能为TSW-9.1的候选基因(VuTSW1)。本发明根据VuG9809G001610的基因组序列在344份种质中的基因型,最终挑选出位于1186708bp处的SNP G/A转化为一个KASP标记2_13994,发现该标记可以指示千粒重基因VuTSW1的筛选,利用该标记扩增需要鉴定的材料,根据其基因型即可判断样本是否携带控制嫩千粒重的有利等位变异。
VuTSW1基因在1186708bp处的SNP为G/A,其在群体中序列变异为:TCGCCATTGCCTTGCACGCCAAAAGGGTTGTGTCGAAATCGCGGTGGGACGCGAAAGTT G[G/A]TGCCTTTAAGATGTGGGGTTGGTCTGAGGAAATGGTTATTGATTCTTTCAGGAAGC ATCC SEQ ID No:1;
利用该序列设计2_13994的两条特异性引物和一条通用引物,所述引物序列如下:
2_13994Primer1:
5’-GAAGGTGACCAAGTTCATGCTAATCGCGGTGGGACGCGAAAGTTGG-3’;SEQ IDNo:2;
2_13994Primer2:
5’-GAAGGTCGGAGTCAACGGATTAATCGCGGTGGGACGCGAAAGTTGA-3’;SEQ IDNo:3;
2_13994Primer1和2_13994Primer2为2条特异性引物,分别连接不同的荧光基团;
本实施例中2_13994Primer1连接FAM基团,2_13994Primer 2连接HEX基团;
2_13994Primer_Common:5’-CCATTTCCTCAGACCAACCCCA-3’;SEQ ID No:4。
实施例2验证
本实施例中“ZN016”可以从浙江省农业科学院蔬菜研究所获得,“特早30”可以从市场商业购买,比如浙江之豇种业有限责任公司。
利用短荚地方品种“ZN016”和长荚育成品种“特早30”构建了一个含120个株系的重组自交系群体,各株系经6代自交,均为高代纯系材料。
于2013年和2019年分别在浙江海宁对该重组自交系群体进行表型鉴定,田间试验采用完全随机区组设计,2次重复,畦宽连沟1.5m,株距40cm,行距75cm,每份材料每个重复种植4穴,田间肥水管理和生产上保持一致。参照《豇豆种质资源描述规范和数据标准》,每个株系随机200-300粒成熟的干种子,利用自动考种仪计算籽粒个数和重量,最终得到千粒重的表型数据(表1)。
表1“ZN016”和“特早30”及其重组自交系千粒重(g)变异情况
取各株系生长两周的幼苗叶片,采用CTAB方法提取基因组DNA,使用IntelliQube基因分型平台,利用2_13994标记(SEQ ID No:2-SEQ ID No:4)扩增上述群体的基因组DNA。PCR反应体系,包含模板DNA 0.8μl,2x KASP Master mix 0.75μl,Primer mix 0.05μl(Primer1、Primer2、Primer_Common引物先分别稀释至100μM,然后按照12%、12%和30%的比例混合在一起,其中ddH
利用IntelliQube机器进行荧光数据读取和分析,结果如图1所示,若豇豆样本基因组DNA显示HEX信号(蓝色),则即可判断该样本携带大粒性状等位变异AA(HapI);若豇豆样本基因组DNA显示FAM信号(红色),则即可判断该样本携带小粒性状等位变异GG(HapII)。结合其千粒重表型发现(表2),携带GG(HapII)基因型的株系在2013和2019年平均千粒重分别为135.35g和140.78g,而携带AA(HapI)基因型的株系在2013和2019年平均千粒重分别为145.37g和148.98g,AA基因型可以分别增加千粒重10.02g和8.20g,两种基因型株系的千粒重之间达到显著水平(p=0.01,p=0.03),说明该标记可以指示千粒重基因VuTSW1,并根据所检测的基因型区别判断千粒重表型,最终达到了分子标记筛选的目的。图1为2_13994标记分型图,图2为携带2_13994不同等位变异株系的千粒重比较图。
表2重组自交系群体部分株系的基因型和表型
最后说明的是,虽然上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
