超氧化物歧化酶1变体和使用其生产谷胱甘肽或其衍生物的方法
文献发布时间:2024-04-18 20:02:40
技术领域
本申请涉及超氧化物歧化酶1变体和使用其生产谷胱甘肽或其衍生物的方法。
背景技术
谷胱甘肽(GSH)是一种在细胞中最常见的有机硫化合物,并且它以三肽的形式存在,其中三个氨基酸(即甘氨酸、谷氨酸和半胱氨酸)缀合。
在体内,谷胱甘肽以两种形式存在:还原型谷胱甘肽(GSH)和氧化型谷胱甘肽(GSSG)。还原型谷胱甘肽通常以相对高的百分比存在。其主要分散在人体的肝脏及皮肤细胞中,并且具有分解和清除氧自由基等抗氧化功能、清除外来化合物(即有毒物质)等解毒功能以及抑制黑色素产生的美白功能等重要作用。
由于这些多种功能,谷胱甘肽作为药物、保健功能食品、化妆品等各领域的材料而备受关注,并且其还可用于制备调味品(flavoring ingredients)、食品和饲料添加剂。已知谷胱甘肽具有显著的增加原料风味和保持浓郁风味的作用,并且可单独用作浓厚风味增强剂或与其它物质组合使用。一般来说,浓厚味(kokumi)材料比鲜味(umami)材料如核酸和MSG的风味更浓郁,并且已知是通过蛋白质的分解和熟化产生的。此外,谷胱甘肽还可被转化为其它γ-谷氨酰肽(Sofyanovich,O.A.et al.,(2019)Multiple pathways for theformation of theγ-glutamyl peptidesγ-glutamyl-valine andγ-glutamyl-valylglycine in Saccharomyces cerevisiae.PLoS ONE 14(5):e0216622)。γ-谷氨酰肽是在分子的N-末端具有谷氨酸的γ-羧基的低分子量化合物,并且已知被广泛用作浓厚风味增强剂。
然而,尽管对如上所述可用于各种领域的谷胱甘肽及其衍生物的需求增大,但由于高生产成本,酶合成法尚未商业化,因此,由于工业生产谷胱甘肽及其衍生物所需的相当大的成本,市场并不十分活跃。
发明内容
【技术问题】
为解决这些问题,本申请提供了超氧化物歧化酶1变体和使用其生产谷胱甘肽或其衍生物的方法。
【技术方案】
本申请的一个目的是提供超氧化物歧化酶1变体,其中对应于自SEQ ID NO:1的氨基酸序列的N-末端起第37位的氨基酸被不同的氨基酸取代。
本申请的另一个目的是提供编码所述变体的多核苷酸和包含所述多核苷酸的载体。
本申请的又一个目的是提供微生物,其包括任一种或多种所述变体和编码所述变体的多核苷酸。
本申请的又另一个目的是提供生产谷胱甘肽或其衍生物的方法,包括培养所述微生物。
【有利效果】
本申请的新型超氧化物歧化酶1变体能够有效用于高产谷胱甘肽或其衍生物。
附图说明
图1和图2显示了通过将超氧化物歧化酶1变体导入酿酒酵母(Saccharomycescerevisiae)菌株中来确认谷胱甘肽生产水平的结果。
具体实施方式
下面将详细描述本公开。与此同时,本文公开的各项描述和实施方式均可分别适用于其它描述和实施方式。也就是说,本文公开的各种要素的所有组合都落入本公开的范围内。此外,本申请的范围不受下文描述的具体描述限制。
此外,本领域普通技术人员仅使用常规实验就能识别或确认本文描述的本发明具体方面的多种等同物。此外,意图使这些等同物也包括在本申请中。
此外,在整个说明书中引用了多篇文章和专利文件。引用的文章和专利文件的内容通过引用以其整体并入本文,并且本发明所属技术领域的水平和本发明的内容将得以更清楚地描述。
本申请的一个方面提供了超氧化物歧化酶1变体,其包含在具有超氧化物歧化酶1(SOD1)活性的蛋白质中的氨基酸取代,其中所述取代包括用不同的氨基酸取代对应于自SEQ ID NO:1的N-末端起第37位的氨基酸。
所述变体可以是蛋白质变体,其中对应于自SEQ ID NO:1的氨基酸序列的N-末端起第37位的氨基酸被不同的氨基酸取代。
本申请的“超氧化物歧化酶1”是也被称为“SOD1”或“超氧化物歧化酶[Cu-Zn]”的酶。已知超氧化物歧化酶催化以下反应方案:
2O
如本文所用,超氧化物歧化酶1的氨基酸序列是sod1基因编码的氨基酸序列,并且可称为“SOD1蛋白”。构成本申请的超氧化物歧化酶1的氨基酸可从已知数据库NCBI的Genbank获得。超氧化物歧化酶1可以是包含SEQ ID NO:1的氨基酸序列的蛋白质,但不限于此。在另一个实例中,超氧化物歧化酶1可衍生自酿酒酵母。在又一个实例中,其可以是对应于SEQ ID NO:1的氨基酸序列中第37位的氨基酸是甘氨酸的蛋白质,但不限于此,并且可无限制地包括任何具有与上述氨基酸序列相同的超氧化物歧化酶1活性的序列。
在具体实例中,本申请的超氧化物歧化酶1可以是包含SEQ ID NO:1的氨基酸序列的蛋白质或与其具有80%、85%、90%、95%、96%、97%、98%、或99%或更高的同源性或同一性的氨基酸序列。此外,显而易见的是,任何具有氨基酸序列的蛋白质,其中氨基酸序列的一部分被缺失、修饰、取代或添加,都可落入本申请的修饰所针对的蛋白质的范围内,只要其是具有这样的同源性或同一性并且表现出与上述蛋白质的效果相应的效果即可。
此外,尽管在本申请中,超氧化物歧化酶1在一个实例中被定义为包含SEQ ID NO:1的氨基酸序列的蛋白质,但其不排除可能通过在SEQ ID NO:1的氨基酸序列的上游或下游添加无意义序列而发生的或者可能天然发生的突变,或其沉默突变,并且对本领域技术人员显而易见的是,任何与由SEQ ID NO:1的氨基酸序列组成的蛋白质具有相同或相应活性的蛋白质都可落入本申请的超氧化物歧化酶1的范围内。
也就是说,在本申请中,尽管将其表达为“具有特定SEQ ID NO的氨基酸序列的蛋白质或多肽”或“包含特定SEQ ID NO的氨基酸序列的蛋白质或多肽”,但显而易见的是,任何具有氨基酸序列的蛋白质,其中氨基酸序列的一部分被缺失、修饰、取代或添加,也都可用于本申请中,只要所述蛋白质具有与由相应SEQ ID NO的氨基酸序列组成的多肽相同或相应的活性即可。
如本文所用,术语“变体”或“修饰多肽”是指通过保守取代和/或修饰而具有与所叙述序列不同的一个或多个氨基酸使得所述蛋白质的功能或特性得以保留的蛋白质。
这些变体与通过几个氨基酸的取代、缺失或添加而鉴定的序列不同。这种变体通常可通过修饰所述蛋白质的上述氨基酸序列中的一个或多个并评估修饰蛋白质的特性来鉴定。也就是说,变体的能力相对于天然蛋白质可以是增强的、不变的或降低的。此外,一些变体可包括修饰多肽,其中一个或多个区域如N-末端前导序列或跨膜区已被去除。其它变体可包括那些某个区域已从成熟蛋白质的N-末端和/或C-末端去除的变体。术语“变体”或“修饰多肽”可与诸如修饰、修饰蛋白质、突变体、突变蛋白、差异的(divergent)、变体等术语互换使用,只要这些术语用于表示变异即可,但这些术语不限于此。
如本文所用,术语“保守取代”是指一种氨基酸被具有相似的结构特性和/或化学特性的另一种氨基酸取代。所述变体可具有例如一个或多个保守取代,同时仍保留至少一种生物活性。这种氨基酸取代通常可基于残基的极性、电荷、可溶性、疏水性、亲水性和/或两亲性的相似性发生。
例如,在具有带电荷侧链的氨基酸当中,带正电的(碱性)氨基酸包括精氨酸、赖氨酸和组氨酸;带负电的(酸性)氨基酸包括谷氨酸和天冬氨酸;而具有不带电侧链的氨基酸包括甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、甲硫氨酸、苯丙氨酸、色氨酸、脯氨酸、丝氨酸、苏氨酸、半胱氨酸、酪氨酸、天冬酰胺和谷氨酰胺。
此外,所述变体还可包括对多肽的特性和二级结构影响最小的氨基酸的缺失或添加。例如,多肽可与参与共翻译地或翻译后地蛋白质转移的N-末端信号(或前导)序列缀合。此外,多肽还可与其它序列或连接体缀合以鉴定、纯化或合成多肽。
在一个实施方式中,本申请的变体可以是超氧化物歧化酶1变体,其中对应于自SEQ ID NO:1的氨基酸序列的N-末端起第37位的氨基酸被不同的氨基酸取代,或者是上述超氧化物歧化酶1当中具有超氧化物歧化酶1活性的修饰多肽。本申请的变体,即“超氧化物歧化酶1变体”可表达为“具有超氧化物歧化酶1活性的(修饰)多肽”或“SOD1变体”,或者可以是与天然野生型多肽或未修饰多肽相比能够增加谷胱甘肽产量的变体,但不限于此。
在上述实施方式中的任一项中,所述变体可以是超氧化物歧化酶1变体,其中对应于自SEQ ID NO:1的氨基酸序列的N-末端起第37位的氨基酸被不同的氨基酸取代。
在本申请中,“用不同的氨基酸取代”不受限制,只要其是与取代前的氨基酸不同的氨基酸即可。与此同时,在本申请中,当表达“特定氨基酸已被取代”时,显而易见的是,所述氨基酸被与取代前的氨基酸不同的氨基酸取代,即使没有具体说明所述氨基酸已被不同的氨基酸取代。
在上述实施方式中的任一项中,“不同的氨基酸”可以是除甘氨酸外的氨基酸。具体地,不同的氨基酸可以是选自丙氨酸、缬氨酸、亮氨酸、异亮氨酸、甲硫氨酸、苯丙氨酸、色氨酸、脯氨酸、丝氨酸、苏氨酸、半胱氨酸、酪氨酸、天冬酰胺、谷氨酰胺、天冬氨酸、谷氨酸、赖氨酸、精氨酸和组氨酸的氨基酸,但不限于此。
如本文所用,“相应位置”是指蛋白质或肽中所叙述位置的氨基酸残基,或与蛋白质或肽中所叙述残基相似、相同或同源的氨基酸残基。如本文所用,术语“相应区域”通常是指相关蛋白质或参考蛋白质中类似或相应的位置。
本申请中,可对本文使用的蛋白质中的氨基酸残基位置使用具体的编号。例如,可通过将本申请的蛋白质的肽序列与待比较的目标蛋白进行比对,将本申请的蛋白质的氨基酸残基位置重新编号为相应位置。
超氧化物歧化酶1变体,其中对应于自SEQ ID NO:1的氨基酸序列的N-末端起第37位的氨基酸被不同的氨基酸取代,可以是在SEQ ID NO:1的氨基酸序列当中,对应于SEQ IDNO:1的第37位的氨基酸被不同的氨基酸取代的蛋白质,或者是与其具有80%、85%、90%、95%、96%、97%、98%或99%或更高的同源性或同一性的氨基酸序列。
这样的变体可以是与SEQ ID NO:1具有80%、85%、90%、95%、96%、97%、98%或99%或更高的同源性或同一性,并且与SEQ ID NO:1具有小于100%的同源性或同一性的变体,但不限于此。
超氧化物歧化酶1变体,其中对应于自SEQ ID NO:1的氨基酸序列的N-末端起第37位的氨基酸被不同的氨基酸取代,可包含SEQ ID NO:12至30的氨基酸序列中的任一个。具体地,所述变体可主要由SEQ ID NO:12至30的氨基酸序列中的任一个组成,以及更具体地,由SEQ ID NO:12至30的氨基酸序列中的任一个组成。
此外,所述变体可包含SEQ ID NO:12至30的氨基酸序列中的任一个,也可包含所述氨基酸序列中第37位的氨基酸固定(即,在所述变体的氨基酸序列中,对应于SEQ ID NO:12至30的氨基酸序列中第37位的氨基酸与SEQ ID NO:12至30的氨基酸序列中第37位的氨基酸相同)且与其具有80%或更高的同源性或同一性的氨基酸序列,但不限于此。
具体地,本申请的变体可具有SEQ ID NO:12至30的氨基酸序列中的任一个,并且可与SEQ ID NO:12至30的氨基酸序列中的任一个的氨基酸序列具有至少80%、90%、95%、96%、97%、98%或99%的同源性或同一性,其中对应于SEQ ID NO:12至30中的任一个的氨基酸序列中第37位的氨基酸可包括氨基酸除甘氨酸外的多肽。此外,显而易见的是,任何具有氨基酸序列的蛋白质,其中氨基酸序列的一部分被缺失、修饰、取代或添加,都可落入本申请的范围内,只要所述氨基酸序列具有这样的同源性或同一性并且显示出与所述变体的功效相应的功效即可。
如本文所用,术语“同源性”或“同一性”是指两个给定氨基酸序列或核苷酸序列之间的相关度,并且可表示成百分比。术语同源性和同一性常常可彼此互换使用。
可通过标准比对算法确定保守多核苷酸或多肽的序列同源性或同一性,并且所用程序建立的默认空位罚分可与其联用。基本上,同源或同一的序列可在中度或高度严格条件下杂交,使得序列的全长或全长的至少约50%、60%、70%、80%、或90%或更多可以杂交。显而易见的是,与含通用密码子或简并密码子的多核苷酸的杂交也包括在内。
任意两个多核苷酸或多肽序列是否具有同源性、相似性或同一性都可通过已知的计算机算法如“FASTA”程序(Pearson et al.,(1988)Proc.Natl.Acad.Sci.USA 85:2444)使用默认参数来确定。可选地,其可通过Needleman–Wunsch算法(Needleman和Wunsch,1970,J.Mol.Biol.48:443–453)来确定,该算法是使用EMBOSS包(EMBOSS:The EuropeanMolecular Biology Open Software Suite,Rice et al.,2000,Trends Genet.16:276–277)(优选5.0.0版本或更高版本)的Needleman程序来执行的(GCG程序包(Devereux,J.,etal.,Nucleic Acids Research 12:387(1984))、BLASTP、BLASTN、FASTA(Atschul,S.F.,etal.,J MOLEC BIOL 215:403(1990);Guide to Huge Computers,Martin J.Bishop,ed.,Academic Press,San Diego,1994和CARILLO et al.(1988)SIAM J Applied Math 48:1073)。例如,可利用National Center for Biotechnology Information(NCBI)的BLAST或ClustalW来确定同源性、相似性或同一性。
多核苷酸或多肽的同源性、相似性或同一性可通过使用例如,如Smith和Waterman,Adv.Appl.Math(1981)2:482中公开的GAP计算机程序(如Needleman et al.(1970),J Mol Biol.48:443)来确定。总之,GAP程序将同源性、相似性或同一性定义为通过将相似对准的符号(即核苷酸或氨基酸)的数量除以两个序列中较短者中的符号总数。GAP程序的默认参数可包括(1)二元比较矩阵(包含同一性值为1和非同一性值为0)和Schwartz与Dayhoff编著的Atlas Of Protein Sequence And Structure,National BiomedicalResearch Foundation,pp.353–358(1979)中公开的Gribskov et al.(1986)Nucl.AcidsRes.14:6745的加权比较矩阵(或EDNAFULL替换矩阵(NCBI NUC4.4的EMBOSS版));(2)每个空位3.0的罚分和每个空位中每个符号另外0.10的罚分(或10的空位开放罚分和0.5的空位扩展罚分);和(3)末端空位无罚分。
此外,任意两个多核苷酸或多肽序列彼此是否具有同源性、相似性或同一性,可通过在如所定义的严格条件下的DNA杂交实验中进行序列比较来鉴定,并且所定义的适当的杂交条件在本领域技术人员的能力范围内,并且可通过本领域技术人员公知的方法来确定(例如,J.Sambrook et al.,Molecular Cloning,A Laboratory Manual,第2版,ColdSpring Harbor Laboratory press,Cold Spring Harbor,New York,1989;F.M.Ausubelet al.,Current Protocols in Molecular Biology,John Wiley&Sons,Inc.,New York,9.50–9.51,11.7–11.8)。
本申请的另一方面可提供编码所述变体的多核苷酸。
如本文所用,术语“多核苷酸”,其由通过共价键连接在长链中的核苷酸单体组成的核苷酸的聚合物,是具有至少一定长度的DNA或RNA链。
编码本申请的超氧化物歧化酶1的基因可以是sod1基因。
所述基因可衍生自酵母。具体地,其可衍生自酿酒酵母属(Saccharomyces),更具体地,衍生自酿酒酵母。具体地,可非限制地包括任何基因,只要其编码具有衍生自酿酒酵母的超氧化物歧化酶1的活性的多肽即可。在一个实施方式中,其可以是编码SEQ ID NO:1的氨基酸序列的基因,而在另一个实施方式中,其可包含SEQ ID NO:2或SEQ ID NO:3的核苷酸序列,但不限于此。
本申请的编码超氧化物歧化酶1变体的多核苷酸可非限制地包括编码超氧化物歧化酶1变体的任何多核苷酸或具有与其相应的活性的多肽。
由于密码子简并性或考虑到待表达多肽的生物体中优选的密码子,本申请的超氧化物歧化酶1或编码其变体的多核苷酸可在不改变多肽的氨基酸序列的范围内于编码区中经历各种修饰。
具体地,本申请的编码超氧化物歧化酶1变体的多核苷酸可非限制地包括任何多核苷酸序列,只要其编码蛋白质变体——其中对应于SEQ ID NO:1的氨基酸序列中第37位的氨基酸被不同的氨基酸取代——即可。
例如,本申请的编码蛋白质变体的多核苷酸可以是本申请的蛋白质变体,具体是编码SEQ ID NO:12至30的氨基酸序列中的任一个或与其具有同源性或同一性的多肽的多核苷酸序列,以及编码对应于SEQ ID NO:12至30的任一个氨基酸序列中第37位的氨基酸的序列可以是编码除甘氨酸外的氨基酸的多核苷酸序列,但不限于此。同源性和同一性如上所述。
此外,本申请的编码蛋白质变体的多核苷酸可包括可由已知基因序列——例如,任何编码对应于SEQ ID NO:1的氨基酸序列中第37位的氨基酸被不同的氨基酸取代的蛋白质变体的序列——制备的探针,其可在严格条件下无限制地与核苷酸序列的全部或部分互补的序列杂交。
“严格条件”是指允许多核苷酸之间特异性杂交的条件。这种条件在文献(例如,J.Sambrook et al.)中有具体描述。例如,严格条件可包括具有40%或更高,具体是90%或更高,更具体是95%或更高、96%或更高、97%或更高、98%或更高,甚至再更具体是99%或更高的高同源性或同一性的基因彼此杂交,而具有比上述同源性或同一性低的同源性或同一性的基因彼此不杂交的条件,或DNA杂交的洗涤条件,即在对应于60℃,1×SSC,和0.1%SDS,具体是60℃,0.1×SSC,0.1% SDS,以及更具体是68℃,0.1×SSC,0.1% SDS的盐浓度和温度下洗涤1次,具体是2次或3次。
杂交要求两个核酸含有互补序列,但是根据杂交的严格性,碱基间可能会错配。术语“互补”用于描述能够彼此杂交的核苷酸碱基之间的关系。例如,关于DNA,腺嘌呤与胸腺嘧啶互补,而胞嘧啶与鸟嘌呤互补。因此,本公开的多核苷酸可包含与整个序列互补的分离核酸片段以及基本上与其相似的核酸序列。
具体地,可利用杂交条件——包括在上述条件下,T
使多核苷酸杂交的适当的严格性取决于多核苷酸的长度和互补程度,并且这些变量是本领域公知的(例如,Sambrook et al.,上文,9.50–9.51,11.7–11.8)。
本申请的又一方面可提供包含编码所述蛋白变体的多核苷酸的载体。
如本文所用,术语“载体”是指含有编码目标蛋白的多核苷酸的核苷酸序列的DNA构建体,所述核苷酸序列可操作地连接到合适的表达调控区(表达调控序列),从而能够在合适的宿主细胞中表达目标多肽。表达调控序列可包括能够起始转录的启动子、用于调控转录的任何操纵基因序列、编码合适的mRNA核糖体结合位点的序列以及用于调控转录和翻译终止的序列。一旦转化到合适的宿主细胞中,载体就可独立于宿主基因组复制或发挥作用,也可整合到其基因组中。
在一个实例中,可通过用于细胞内染色体插入的载体,用修饰多核苷酸置换染色体中编码目标蛋白的多核苷酸。多核苷酸向染色体中的插入可通过本领域已知的任意方法(例如,通过同源重组)执行,但不限于此。载体可进一步包括选择标记来确认插入到染色体中。选择标记用于选择转化了载体的细胞,也就是说,用于确认是否插入了目标核酸分子,并且可使用提供可选择表型如抗药性、营养缺陷、细胞毒剂抗性或表面蛋白表达的标记。在用选择剂处理的环境下,只有表达选择标记的细胞才能存活或显示出不同表型,因此可选出转化细胞。
本申请中使用的载体没有特别限制,并且可使用本领域已知的任何载体。酵母表达载体可包括酵母整合型质粒(YIp)和染色体外质粒载体。染色体外质粒载体可包括酵母附加体质粒(Yep)、酵母复制型质粒(YRp)和酵母着丝粒质粒(YCp)。此外,酵母人工染色体(YAC)也可用作本申请的载体。可使用的载体的具体实例包括pESCHIS、pESC-LEU、pESC-TRP、pESC-URA、Gateway pYES-DEST52、pAO815、pGAPZ A、pGAPZ B、pGAPZ C、pGAPαA、pGAPαB、pGAPαC、pPIC3.5K、pPIC6 A、pPIC6 B、pPIC6 C、pPIC6αA、pPIC6αB、pPIC6αC、pPIC9K、pYC2/CT、pYD1酵母展示载体、pYES2、pYES2/CT、pYES2/NT A、pYES2/NT B、pYES2/NT C、pYES2/CT、pYES2.1、pYES-DEST52、pTEF1/Zeo、pFLD1、PichiaPinkTM、p427-TEF、p417-CYC、pGAL-MF、p427-TEF、p417-CYC、PTEF-MF、pBY011、pSGP47、pSGP46、pSGP36、pSGP40、ZM552、pAG303GAL-ccdB、pAG414GAL-ccdB、pAS404、pBridge、pGAD-GH、pGAD T7、pGBK T7、pHIS-2、pOBD2、pRS408、pRS410、pRS418、pRS420、pRS428、酵母微米A型(yeast micron A form)、pRS403、pRS404、pRS405、pRS406、pYJ403、pYJ404、pYJ405和pYJ406,但不限于此。
如本文所用,术语“转化”是指将包含编码目标蛋白的多核苷酸的载体导入宿主细胞中,使得由多核苷酸编码的蛋白可在宿主细胞中表达。只要转化的多核苷酸可在宿主细胞中表达,无论转化的多核苷酸是插入宿主细胞的染色体中并定位在其中,还是定位在染色体之外都无妨,并且两种情况都可以包括。此外,多核苷酸可包括编码目标蛋白的DNA和RNA。多核苷酸可以任意形式导入,只要其能够导入宿主细胞并在其中表达即可。例如,可以表达盒的形式将多核苷酸导入宿主细胞,表达盒是包括其自主表达所需的所有元件的基因构建体。表达盒通常可包括与多核苷酸可操作地连接的启动子、转录终止子、核糖体结合位点或翻译终止子。表达盒可以是能够自我复制的表达载体的形式。此外,多核苷酸可以其自身的形式导入宿主细胞中,并且可操作地连接到宿主细胞中表达所必需的序列,但不限于此。
此外,如本文所用,术语“可操作地连接”意为基因序列与起始和介导本申请的编码目标多肽的多核苷酸的转录的启动子序列功能连接。
用于转化本申请的载体的方法可包括能够将核酸导入细胞的任何方法,并且可通过根据宿主细胞选择本领域已知的合适的标准技术来进行转化。例如,方法可包括电穿孔、磷酸钙(CaPO
本申请可提供包括超氧化物歧化酶1和编码所述变体的多核苷酸中的任一种或多种的微生物。
如本文所用,术语“微生物”包括所有野生型微生物,或通过天然或人工手段进行遗传修饰的微生物,并且是包括由于插入外来基因,或增强或弱化内源基因的活性而特定机制得到弱化或增强的所有微生物的综合概念。
在一个实施方式中,微生物可以是生产谷胱甘肽的微生物。如本文所用,术语“生产谷胱甘肽的微生物”可与诸如“产谷胱甘肽微生物”、“具有谷胱甘肽生产能力的微生物”、“产谷胱甘肽菌株”、“具有谷胱甘肽生产能力的菌株”等术语互换使用。
在上述任一项实施方式中,微生物可以是生产谷胱甘肽衍生物的微生物。
在上述任一项实施方式中,本申请的微生物可包括载体,所述载体包括编码超氧化物歧化酶1的多核苷酸。
本申请的“谷胱甘肽”可与“谷胱甘肽”或“GSH”互换使用,并且是指由三个氨基酸(即谷氨酸、半胱氨酸、甘氨酸)组成的三肽。谷胱甘肽可用作药物、保健功能食品、调味材料、食品、饲料添加剂、化妆品等的原料,但不限于此。
本申请的“谷胱甘肽衍生物”可非限制地包括可用谷胱甘肽作为起始材料进行转化的任何物质。
在本申请中,谷胱甘肽或其衍生物可统称为“γ-谷氨酰肽”。γ-谷氨酰肽是指在分子的N-末端具有谷氨酸的γ-羧基的低分子量化合物。
具体地,γ-谷氨酰肽可以是由以下通式1表示的化合物:
[通式1]
γ-Glu–X–Y
在上述通式中,X是半胱氨酸、谷氨酸、酪氨酸、甲硫氨酸、缬氨酸、色氨酸、天冬氨酸、异亮氨酸、亮氨酸、天冬酰胺、谷氨酰胺、组氨酸、赖氨酸、鸟氨酸、精氨酸、S-甲基半胱氨酸或S-乙基半胱氨酸;
Y是甘氨酸、丙氨酸、亮氨酸、异亮氨酸、丝氨酸、苏氨酸、甲硫氨酸、半胱氨酸、天冬氨酸、天冬酰胺、谷氨酰胺、赖氨酸、鸟氨酸、精氨酸、苯丙氨酸、酪氨酸、脯氨酸、羟脯氨酸、α-氨基丁酸或不存在。
在一个实施方式中,γ-谷氨酰肽可选自γ-Glu-Cys-Gly(谷胱甘肽)、γ-Glu-Glu(γ-EF)、γ-Glu-Tyr(γ-EY)、γ-Glu-Met(γ-EM)、γ-Glu-Val(γ-EV)、γ-Glu-Trp(γ-EW)、γ-Glu-Asp(γ-ED)、γ-Glu-Ile(γ-EI)、γ-Glu-Leu(γ-EL)、γ-Glu-Cys-Gly(γ-ECG)、γ-Glu-Val-Gly(γ-EVG)、γ-谷氨酰-S-甲基-半胱氨酸(γ-E-S-甲基-C)、γ-谷氨酰-S-乙烯基-半胱氨酸(γ-E-S-乙烯基-C)和γ-谷氨酰-半胱氨酰-β-丙氨酸(γ-E-C-β-A)。
在任一项上述实施方式中,γ-谷氨酰肽可以是谷胱甘肽,但不限于此。
本申请的微生物可指与野生型或未修饰微生物相比能够生产过量的所需谷胱甘肽(包括超氧化物歧化酶1)的微生物。只要微生物能够生产谷胱甘肽,其就不受其类型的特别限制,但可以是酿酒酵母属微生物,具体是酿酒酵母。然而,微生物不限于此。
包含所述变体的产谷胱甘肽微生物的亲本菌株没有特别限制,只要其能够生产谷胱甘肽即可。微生物还可包括突变,如加强生物合成途径以提高谷胱甘肽生产能力、释放反馈抑制或弱化降解途径或生物合成途径的基因失活,并且这些突变不排除那些天然存在的突变。
如本文所用,术语多肽活性的“增强”意为多肽的活性与其内源活性相比是增加的。增强可与诸如激活、上调、过表达、增加等术语互换使用。具体地,激活、增强、上调、过表达和增加可包括两种情况——表现出最初所不具有的活性,或者与内源活性或修饰前的活性相比活性被增强。“内源活性”是指当性状通过因由天然或人工因素引起的遗传修饰而改变时,转化前的亲本菌株或未修饰微生物最初具有的特定多肽的活性,并且可与“修饰前的活性”互换使用。多肽的活性与其内源活性相比“增强”、“上调”、“过表达”或“增加”意为多肽的活性和/或浓度(表达水平)与转化前的亲本菌株或未修饰微生物最初具有的特定多肽的活性和/或浓度(表达水平)相比被增强。
增强可通过导入外来多肽,或通过增强内源多肽的活性和/或浓度(表达水平)来实现。多肽的活性的增强可通过多肽的活性的水平、表达水平或由多肽分泌的产物的量的增加来确认。
多肽的活性的增强可通过本领域公知的各种方法来施加,并且不受限制,只要其与修饰前的微生物相比能够增强目标多肽的活性即可。具体地,可使用本领域技术人员公知的基因工程和/或蛋白质工程,这是分子生物学的常用方法,但方法不限于此(例如,Sitnicka et al.Functional Analysis of Genes.Advances in Cell Biology.2010,Vol.2.1–16,Sambrook etal.Molecular Cloning 2012,etc.)。
具体地,本申请的多肽的增强可通过以下方式实现:
1)增加编码所述多肽的多核苷酸的细胞内拷贝数;
2)用具有强活性的序列置换染色体上编码所述多肽的基因的表达调控区;
3)修饰核苷酸序列,所述核苷酸序列编码:编码所述多肽的基因转录物的起始密码子或5′-UTR;
4)修饰所述多肽的氨基酸序列使得多肽的活性被增强;
5)修饰编码所述多肽的多核苷酸序列使得所述多肽活性被增强(例如,修饰所述多肽基因的多核苷酸序列以编码经修饰以增强所述多肽的活性的多肽);
6)导入表现出所述多肽活性的外来多肽或编码其的外来多核苷酸;
7)对编码所述多肽的多核苷酸密码子优化;
8)分析所述多肽的三级结构,从而选择和修饰暴露位点,或化学修饰暴露位点;或
9)选自上述1)至8)中的两种或更多种的组合,但不特别限于此。
更具体地,
增加编码所述多肽的多核苷酸的细胞内拷贝数的方法1)可通过将载体导入宿主细胞来实现,所述载体与编码所述多肽的多核苷酸可操作地连接并且能够复制和发挥功能而与宿主细胞无关。可选地,所述方法可通过将编码所述多肽的多核苷酸的一个拷贝或两个拷贝导入宿主细胞的染色体来实现。可通过将能够将多核苷酸插入宿主细胞的染色体的载体导入宿主细胞来进行染色体导入,但不限于此。载体如上所述。
用具有强活性的序列置换染色体上编码所述多肽的基因的表达调控区(或表达调控序列)的方法2)可通过经缺失、插入、非保守或保守取代,或其组合诱导对序列的修饰以进一步增强表达调控区的活性,或通过用活性更强的序列置换序列来实现。表达调控区可包括但不特别限于启动子、操纵基因序列、编码核糖体结合位点的序列和调控转录与翻译终止的序列。在一个实例中,所述方法可包括用强启动子置换原始启动子,但不限于此。
真核生物已知启动子的实例包括翻译延伸因子1(TEF1)、甘油-3-磷酸脱氢酶1(GPD1)、3-磷酸甘油酸激酶或其它糖酵解酶的启动子,例如烯醇化酶、甘油醛-3-磷酸脱氢酶、己糖激酶、丙酮酸脱羧酶、磷酸果糖激酶、葡萄糖-6-磷酸异构酶、3-磷酸甘油酸变位酶、丙酮酸激酶、磷酸丙糖异构酶、磷酸葡萄糖异构酶和葡糖激酶;其它酵母启动子——即另外具有通过生长条件控制转录的优点的诱导型启动子——的实例包括醇脱氢酶2、isocytochrome C、酸性磷酸酶、与氮代谢相关的降解酶、金属硫蛋白、甘油醛-3-磷酸脱氢酶以及负责麦芽糖和半乳糖利用的酶的启动子;宿主细胞是酵母时可使用的启动子可包括TEF1启动子、TEF2启动子、GAL10启动子、GAL1启动子、ADH1启动子、ADH2启动子、PHO5启动子、GAL1-10启动子、TDH3启动子(GPD启动子)、TDH2启动子、TDH1启动子、PGK1启动子、PYK2启动子、ENO1启动子、ENO2启动子和TPI1启动子;以及在欧洲专利号073657中进一步描述了用于酵母表达的合适的载体和启动子,但启动子不限于此。此外,酵母增强剂也可有利地与酵母启动子组合使用,但不限于此。
修饰核苷酸序列,所述核苷酸序列编码:编码所述多肽的基因转录物的起始密码子或5′-UTR的方法3)可例如通过用编码与内源起始密码子相比具有更高的多肽表达率的另一个起始密码子的核苷酸序列取代核苷酸序列来实现,但不限于此。
修饰氨基酸序列或多核苷酸序列的方法4)和5)可通过以下方式实现:通过缺失、插入、非保守或保守取代所述多肽的氨基酸序列或编码所述多肽的多核苷酸序列,或其组合来诱导对序列的修饰以增强所述多肽的活性,或通过用经修饰以具有更强活性的氨基酸序列或多核苷酸序列,或经修饰以增强活性的氨基酸序列或多核苷酸序列来置换序列,但不限于此。置换可通过同源重组将多核苷酸插入染色体而特异性地执行,但不限于此。本文使用的载体可进一步包含选择标记以确认插入到染色体中。选择标记如上所述。
导入表现出所述多肽的活性的外来多核苷酸的方法6)可通过将编码与所述多肽表现出相同/相似活性的多肽的外来多核苷酸导入宿主细胞来实现。外来多核苷酸可以不受限制地使用,无论其来源或序列如何,只要其表现出与所述多肽相同/相似的活性即可。导入可由本领域普通技术人员通过适当选择本领域已知的转化方法来执行,并且导入的多核苷酸在宿主细胞中的表达能够产生所述多肽,从而增加其活性。
对编码所述多肽的多核苷酸密码子优化的方法7)可通过对内源多核苷酸密码子优化以增加宿主细胞内的转录或翻译,或通过优化其密码子使得可在宿主细胞内实现外来多核苷酸的优化的转录和翻译来实现。
分析所述多肽的三级结构,从而选择和修饰暴露位点,或化学修饰其的方法8)可例如通过下列来实现:将待分析多肽的序列信息与存储已知蛋白质的序列信息的数据库进行比较,以根据序列相似度确定模板蛋白候选物,并因此基于所述信息确认结构,从而选出和转化或修饰待修饰或化学修饰的暴露位点。
所述多肽活性的这种增强可意为相应多肽的活性或浓度相对于野生型或修饰前微生物中表达的所述多肽的活性或浓度是增加的,或者从所述多肽产生的产物的量是增加的,但不限于此。
对本申请的微生物中多核苷酸的部分或全部的修饰可通过以下方式实现:(a)利用用于微生物中染色体插入的载体进行同源重组或使用工程化核酸酶(例如,CRISPR-Cas9)进行基因组编辑和/或(b)可通过光如紫外线和辐射等和/或化学处理来诱导,但不限于此。修饰基因的部分或全部的方法可包括利用DNA重组技术的方法。例如,可通过将核苷酸序列或含有与目标基因同源的核苷酸序列的载体注射到微生物中以诱导同源重组来缺失基因的部分或全部。注射的核苷酸序列或载体可包含显性选择标记,但不限于此。
如本文所用,术语多肽的“弱化”是一个与其内源活性相比活性降低或没有活性的综合概念。弱化可与失活、缺乏、下调、减少、降低和衰减等术语互换使用。
弱化还可包括:由于编码多肽的多核苷酸的突变,与微生物最初具有的多肽的活性相比,多肽活性自身降低或消除的情况;由于对编码多肽的多核苷酸的基因的表达的抑制或对其向多肽的翻译等的抑制,细胞内多肽活性和/或浓度(表达水平)的总体水平与天然菌株相比降低的情况;多核苷酸完全不表达的情况;和/或即使多核苷酸被表达,也观察不到多肽活性的情况。如本文所用,术语“内源活性”是指当性状因由天然或人工因素引起的遗传修饰而改变时,转化前的亲本菌株、野生型或未修饰微生物最初具有的特定多肽的活性,并且可与“修饰前的活性”互换使用。“与其内源活性相比,多肽活性‘失活、缺乏、减少、下调、降低或衰减’”这一表述意为,与转化前的亲本菌株或未修饰微生物最初具有的特定多肽的活性相比多肽活性减少。
多肽活性的弱化可通过本领域已知的任何方法进行,但是方法不限于此,以及可通过应用本领域公知的各种方法来实现(例如,Nakashima N.et al.,Bacterial cellularengineering by genome editing and gene silencing.Int J Mol Sci.2014;15(2):2773–2793,Sambrook et al.Molecular Cloning 2012,etc.)。
具体地,本申请的多肽活性的弱化可通过以下方式实现:
1)使编码所述多肽的基因的部分或全部缺失;
2)修饰表达调控区(表达调控序列)使得编码所述多肽的基因的表达减少;
3)修饰构成所述多肽的氨基酸序列使得所述多肽活性被消除或弱化(例如,对氨基酸序列进行一个或多个氨基酸的缺失/取代/添加);
4)修饰编码所述多肽的基因序列使得所述多肽活性被消除或弱化(例如,对所述多肽的核苷酸序列进行一个或多个核苷酸的缺失/取代/添加以编码经修饰以消除或弱化所述多肽的活性的多肽);
5)修饰核苷酸序列,所述核苷酸序列编码:编码所述多肽的基因转录物的起始密码子或5′-UTR;
6)导入与编码所述多肽的基因转录物互补结合的反义寡核苷酸(例如,反义RNA);
7)在编码所述多肽的基因的Shine-Dalgarno(SD)序列的前端添加与SD序列互补的序列以形成二级结构,从而抑制核糖体附着;
8)逆转录工程(RTE),其对编码所述多肽的基因序列的开放阅读框(ORF)的3′末端添加待逆转录的启动子;或
9)选自以上方法1)至8)中的两种或更多种的组合,但不特别限于此。
例如,
使编码所述多肽的基因的部分或全部缺失的方法1)可通过使染色体内编码内源目标多肽的所有多核苷酸缺失,或通过用具有部分缺失的核苷酸的多核苷酸或标记基因置换多核苷酸来实现。
修饰表达调控区(表达调控序列)的方法2)可通过对表达调控区(表达调控序列)通过缺失、插入、非保守或保守取代,或其任意组合诱导修饰;或通过用活性较弱的序列置换序列来实现。表达调控区包括启动子、操纵基因序列、编码核糖体结合位点的序列和用于调控转录和翻译的序列,但不限于此。
修饰核苷酸序列,所述核苷酸序列编码:编码所述多肽的基因转录物的起始密码子或5′-UTR的方法5)可通过例如用编码另一个具有比内源起始密码子低的多肽表达率的起始密码子的核苷酸序列取代核苷酸序列来实现,但不限于此。
修饰氨基酸序列或多核苷酸序列的方法3)和4)可通过以下方式实现:通过缺失、插入、非保守或保守取代所述多肽的氨基酸序列或编码所述多肽的多核苷酸序列,或其组合来诱导对序列的修饰以弱化所述多肽的活性,或通过用经修饰以具有较弱活性的氨基酸序列或多核苷酸序列,或经修饰以不具有活性的氨基酸序列或多核苷酸序列来取代序列,但不限于此。例如,可通过将突变导入多核苷酸序列以形成终止密码子来抑制或弱化基因的表达,但不限于此。
导入与编码所述多肽的基因转录物互补结合的反义寡核苷酸(例如,反义RNA)的方法6)可在文献(Weintraub,H.et al.,Antisense-RNA as a molecular tool forgenetic analysis,Reviews-Trends in Genetics,Vol.1(1)1986)中找到。
在编码所述多肽的基因的Shine-Dalgarno(SD)序列的前端添加与SD序列互补的序列以形成二级结构,从而抑制核糖体附着的方法7)可通过抑制mRNA翻译或降低其速度来实现。
逆转录工程(RTE),其对编码所述多肽的基因序列的开放阅读框(ORF)的3′末端添加待逆转录的启动子的方法8)可通过形成与编码所述多肽的基因转录物互补的反义核苷酸以弱化活性来实现。
在上述任一项实施方式中,本申请的微生物可包括谷氨酸–半胱氨酸连接酶(GSH1)的表达调控区中增加谷胱甘肽生产能力的突变。突变可以是GSH1 ORF上游选自-250(C→T)、-252(G→A)、-398(A→T)、-399(A→C)、-407(T→C)和-409(T→C)的任一种或多种突变。
在上述任一项实施方式中,微生物可包括突变,其中谷氨酸–半胱氨酸连接酶(GSH1)的第86个氨基酸和/或第653个氨基酸被不同的氨基酸取代。具体地,微生物可包括第86个氨基酸被精氨酸取代并且第653个氨基酸被甲硫氨酸取代的突变,但突变不限于此。
在上述任一项实施方式中,微生物可包括谷氨酸–半胱氨酸连接酶(GSH1)的活性被增强的突变,但不限于此。
包括本申请的超氧化物歧化酶1变体中的任一种或多种和编码所述变体的多核苷酸的微生物可以是“表达超氧化物歧化酶1的微生物,其中对应于SEQ ID NO:1的氨基酸序列中第37位的氨基酸被不同的氨基酸取代”,但不限于此。
超氧化物歧化酶1及其变体如上所述。
如本文所用,关于蛋白质的术语“待表达/表达”是指目标蛋白被导入微生物或目标蛋白被修饰以在微生物中表达的状态。
本申请的表达蛋白质变体的微生物可以是经修饰以表达本申请的蛋白质变体的微生物,因此,本申请的又一方面提供了生产表达本申请的蛋白质变体的微生物的方法。
如本文所用,术语“修饰前的菌株”或“修饰前的微生物”不排除含有可在微生物中天然发生的突变的菌株,并且可指野生型菌株或天然菌株本身,或由于天然或人工因素引起的遗传修饰而改变性状前的菌株。“修饰前的菌株”或“修饰前的微生物”可与“未突变菌株”、“未修饰菌株”、“未突变微生物”、“未修饰微生物”或“参考微生物”互换使用。
在本申请中,包括本申请的超氧化物歧化酶1变体的微生物、编码所述变体的多核苷酸或包括所述多核苷酸的载体可以是重组微生物,并且重组可通过遗传修饰如转化来实现。
例如,微生物可以是通过用包含多核苷酸的载体转化所制备的重组微生物,但不限于此。重组微生物可以是酵母,例如酵母属微生物,具体是酿酒酵母,但不限于此。
本申请的再又一方面提供了生产谷胱甘肽或其衍生物的方法,包括:培养微生物。微生物、谷胱甘肽或其衍生物如上所述。
用于培养本申请的菌株的培养基和其它培养条件可以是用于常规培养酵母属微生物的任何培养基,而无任何特别限制。然而,本申请的菌株可在有氧或需氧条件下于含有适当的碳源、氮源、磷源、无机化合物、氨基酸和/或维生素的常规培养基中,同时调节温度、pH等进行培养。
在本申请中,碳源可包括碳水化合物,如葡萄糖、果糖、乳糖、蔗糖、麦芽糖等;糖醇,如甘露醇、山梨糖醇等;有机酸,如丙酮酸、乳酸、柠檬酸等;氨基酸,如谷氨酸、甲硫氨酸、赖氨酸等,但不限于此。此外,碳源还可包括天然有机营养物,如淀粉水解物、糖蜜、赤糖糊、米糠、木薯、甘蔗糖蜜和玉米浆等,以及可使用碳水化合物如葡萄糖和灭菌的预处理糖蜜(即转化为还原糖的糖蜜)。此外,还可以不受限制地以适当的量使用各种其它碳源。这些碳源可单独使用或以其两种或更多种的组合使用,但不限于此。
氮源可包括无机氮源,如氨、硫酸铵、氯化铵、醋酸铵、磷酸铵、碳酸铵、硝酸铵等;和有机氮源,如氨基酸、蛋白胨、NZ-胺、肉提取物、酵母提取物、麦芽提取物、玉米浆、酪蛋白水解物、鱼或其分解产物、脱脂豆饼或其分解产物等。这些氮源可单独使用或以两种或更多种的组合使用,但不限于此。
磷源可包括磷酸二氢钾、磷酸氢钾或对应的含钠盐等。无机化合物的实例可包括氯化钠、氯化钙、氯化铁、硫酸镁、硫酸铁、硫酸锰、碳酸钙等。
此外,培养基中可包含氨基酸、维生素和/或适当的前体。具体地,L-氨基酸等可添加到菌株的培养基中。具体地,可添加甘氨酸、谷氨酸和/或半胱氨酸,并且当需要时可进一步添加L-氨基酸,如赖氨酸,但是本申请不限于此。
培养基或前体可以分批或连续的方式添加到培养产物中,但不限于此。
在本申请中,培养产物的pH可在菌株培养过程中通过以适当的方式向培养产物中添加化合物如氢氧化铵、氢氧化钾、氨、磷酸、硫酸等来调节。此外,在培养过程中,可添加消泡剂如脂肪酸聚乙二醇酯防止泡沫产生。此外,可将氧气或含氧气体注入培养产物中以维持培养产物的需氧状态;也可注入氮气、氢气或二氧化碳气体,或者不注入气体,以维持培养基的厌氧或微需氧状态。
培养产物的温度可在25℃至40℃的范围内,具体是28℃至37℃,但不限于此。可继续培养,直到获得有所需产量的有用物质,具体培养1至100小时,但不限于此。
生产方法可进一步包括培养步骤之后另外的过程。可根据谷胱甘肽或其衍生物的用途适当选择另外的过程。
具体地,生产谷胱甘肽或其衍生物的方法可包括回收通过培养步骤在细胞中累积的谷胱甘肽或其衍生物的步骤,例如,所述方法可包括在培养步骤后从选自菌株、其干燥产物、其提取物、其培养产物和其裂解物的至少一种材料中回收谷胱甘肽或其衍生物的步骤。在另一个实例中,生产谷胱甘肽衍生物的方法可包括将谷胱甘肽转化为衍生物的步骤。
所述方法可进一步包括在回收步骤前或同时裂解菌株的步骤。菌株的裂解可通过本申请所属技术领域常用的方法进行,例如通过裂解缓冲溶液、超声仪、热处理和法压机(French press)等。此外,裂解步骤可包括但不限于酶促反应,如细胞壁裂解酶、核酸裂解酶、核酸转移酶和蛋白酶等。
可通过本申请的生产方法制备含有高含量谷胱甘肽或其衍生物的干酵母、酵母提取物、酵母提取物混合粉末、纯谷胱甘肽或其衍生物,但不限于此,并且可根据所需产品进行适当制备。
在本申请中,干酵母可与术语如“菌株的干燥产物”互换使用。干酵母可通过干燥累积了谷胱甘肽的酵母细胞来制备,并且可具体地包含在饲料用组合物、食品用组合物等中,但不限于此。
如本文所用,酵母提取物可与术语如“菌株的提取物”互换使用。菌株的提取物可意为从菌株细胞分离细胞壁后剩余的物质。具体地,其可意为从通过细胞裂解获得的组分中除去细胞壁的剩余组分。菌株的提取物包含谷胱甘肽或其衍生物,而其它组分可包含蛋白质、碳水化合物、核酸和纤维中的一种或多种组分,但不限于此。
在回收步骤中,可通过本领域已知的合适的方法回收目标材料即谷胱甘肽或其衍生物。
回收步骤可包括纯化过程。纯化过程可以是通过仅从菌株中分离谷胱甘肽或其衍生物的纯粹纯化。纯谷胱甘肽可由纯化过程制备。
如有必要,谷胱甘肽的生产方法则还可包括将选自培养步骤后获得的菌株、其干燥物、其提取物、其培养产物、其裂解物和从其中回收的谷胱甘肽的材料与赋形剂混合的步骤。通过混合步骤,可制备酵母提取物混合粉末。同样适用于谷胱甘肽的衍生物。
赋形剂可根据预期用途或形式适当地选择和使用,例如,其可选自淀粉、葡萄糖、纤维素、乳糖、糖原、D-甘露醇、山梨糖醇、乳糖醇、麦芽糊精、碳酸钙、合成硅酸铝、磷酸氢钙、硫酸钙、氯化钠、碳酸氢钠、纯化羊毛脂、糊精、海藻酸钠、甲基纤维素、胶态硅胶、羟丙基淀粉、羟丙基甲基纤维素、丙二醇、酪蛋白、乳酸钙、
赋形剂可包括例如防腐剂、润湿剂、分散剂、悬浮剂、缓冲剂、稳定剂或等渗剂,但不限于此。
实施例
将通过实施例和实验实施例来详细描述本申请。然而,提供这些实施例和实验实施例仅仅是为了说明的目的,而本申请的范围不旨在被这些实施例和实验实施例所限制。
实施例1:产谷胱甘肽菌株的改良
为进一步提高保藏号为KCCM12568P的产谷胱甘肽菌株CJ-5菌株(KR 10-2222210B1)的谷胱甘肽生产能力,通过以下方式诱导突变。
在固体培养基上培养CJ-5菌株,然后将其接种到肉汤中以获得培养液,并且使用UV灯对细胞进行UV照射。此后,只分离得到通过将UV照射的培养液涂在平板培养基上形成菌落的突变菌株,并分离谷胱甘肽生产能力提高最大的菌株。
作为确认分离菌株的基因组的结果,GSH1 ORF上游发生-250(C→T)、-252(G→A)、-398(A→T)、-399(A→C)、-407(T→C)和-409(T→C)突变,并确认gsh1基因编码的GSH1蛋白第86位的氨基酸半胱氨酸被精氨酸取代,以及第653位氨基酸(甘氨酸)被精氨酸取代。
上述菌株被命名为CC02-2816,并根据布达佩斯条约于2020年12月8日保藏在韩国微生物保藏中心(Korean Culture Center of Microorganisms,KCCM),保藏号为KCCM12891P。
实施例2:菌株的附加改良实验
为进一步提高实施例1的CC02-2816菌株的谷胱甘肽生产能力,以下列方式诱导突变。
在固体培养基上培养CC02-2816菌株,然后将其接种到肉汤中以获得培养液,并且使用UV灯用UV光对细胞进行照射。此后,只分离得到通过将UV照射的培养液涂在平板培养基上形成菌落的突变菌株,并分析核苷酸序列。
作为实验结果,确认了由编码谷胱甘肽含量提高26%的菌株的超氧化物歧化酶1的sod1编码的蛋白质即SOD1(SEQ ID NO:1)的第37位氨基酸(甘氨酸)被精氨酸取代。
实施例3:SOD1蛋白第37个残基突变实验
根据实施例2的结果,确定了SOD1蛋白的第37位将对于谷胱甘肽生产很重要,并构建了野生型酿酒酵母CEN.PK2-1D和实施例1的CC02-2816菌株以表达突变蛋白,其中SOD1蛋白的第37个氨基酸被不同的氨基酸取代,从而确定谷胱甘肽产量是否增加。
为制备来自酿酒酵母的SOD1蛋白的第37个氨基酸被精氨酸取代的菌株,参照文献Lee T.H.et al.(J.Microbiol.Biotechnol.(2006),16(6),979–982)中公开的内容,使用pWAL100和pWBR100质粒。
具体地,使用CJ-5菌株的基因组DNA作为模板,如下进行PCR。使用SEQ ID NO:4和SEQ ID NO:6的引物进行PCR获得包括N-末端BamHI侧接序列、SOD1 ORF的起始密码子和G37R突变编码序列的SOD1 N-末端的部分序列,并使用SEQ ID NO:5和SEQ ID NO:7的引物进行PCR获得包括C-末端XhoI侧接序列、SOD1 ORF终止密码子和G37R突变编码序列的SOD1C-末端的部分序列。此后,作为以这两个序列作为模板、使用SEQ ID NO:4和SEQ ID NO:7进行重叠PCR的结果,获得了SOD1 ORF片段,其包含第37个氨基酸被精氨酸取代的SOD1突变蛋白编码序列、N-末端BamHI和C-末端XhoI限制酶。在用BamHI和XhoI处理后,将ORF片段克隆到用相同的酶处理过的pWAL100载体中,并制备pWAL100-SOD1(G37R)载体。
此外,以CJ-5菌株的基因组DNA为模板,使用SEQ ID NO:8和SEQ ID NO:9进行PCR获得SOD1 ORF终止密码子下游500bp片段,其包含N-末端SpeI和C-末端NcoI限制酶序列,并用SpeI和NcoI限制酶处理这一片段。在用相同的限制酶处理过的pWBR100中克隆这一片段后,制备pWBR100-SOD1载体。
为了制备最终导入酵母的DNA片段,以先前制备的pWAL100-SOD1(G37R)载体为模板,使用SEQ ID NO:4和SEQ ID NO:10的引物获得含有精氨酸突变编码序列和部分KlURA3的PCR产物,并以pWBR100-SOD1载体为模板,使用SEQ ID NO:11和SEQ IDNO:9的引物获得含有部分KlURA3和SOD1终止密码子下游500bp片段的PCR产物。以相同摩尔比,将各PCR产物转化至酿酒酵母CEN.PK2-1D和酿酒酵母CC02-2816中。在以下条件下进行PCR:95℃变性5分钟,53℃退火1分钟和72℃聚合1分钟/1kb。对于酵母转化,使用根据Geitzs所著文献(Nucleic Acid Research,20(6),1425)修改的醋酸锂法。具体地,在用醋酸锂/TE缓冲液洗涤O.D.为0.7至1.2的酵母细胞两次后,将PCR产物和单链DNA(Sigma D-7656)混合在一起,并在醋酸锂/TE/40% PEG缓冲液中于30℃下静置培养30分钟和在42℃下静置培养15分钟。此后,在不含尿嘧啶的SC(2%葡萄糖)琼脂平板上培养细胞,直至观察到菌落,获得导入SOD1 G37R突变编码序列和KlURA3基因的菌株。随后,为除去KlURA3,将各菌株在2mL YPD中培养过夜,稀释100倍,并铺板在含有0.1%5-FOA的SC(2%葡萄糖)琼脂平板上以构建酿酒酵母CEN.PK2-1D SOD1G37R突变菌株和已去除尿嘧啶标记的酿酒酵母CC02-2816 SOD G37R突变菌株。还以除了使用SEQ ID NO:5和SEQ ID NO:6的引物序列上第37个精氨酸编码序列被编码不同的氨基酸的序列取代的引物对以外相同的方式,制备能够表达被除精氨酸外的氨基酸取代的SOD1突变蛋白的菌株。
[表1]
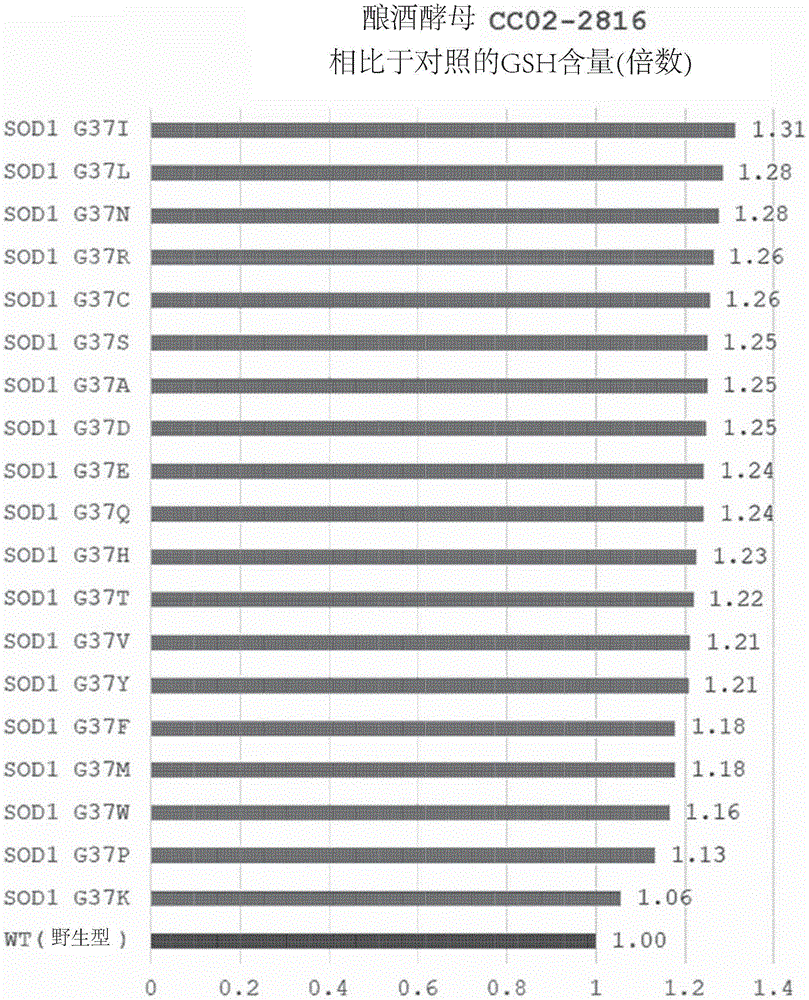
通过培养上述制备的各菌株26小时所生产的谷胱甘肽(GSH)的浓度和含量的测量结果示于下表2和3以及图1和2中。
[表2]
[表3]
/>
基于这些结果,确认了通过用不同的氨基酸取代第37位的氨基酸能够提高谷胱甘肽生产能力,因此,可推测本申请中提供的SOD1蛋白变体能够有效用于γ-谷氨酰肽的生产。
根据前述内容,本申请所属领域技术人员将能够理解,在不修改本申请的技术构思或必要特征的情况下,本申请可以其它具体形式体现。在这一点上,本文公开的示例性实施例仅用于说明的目的,而不应当被解释为限制本申请的范围。因此,本申请的范围由所附权利要求而非由前面的描述来指示。落入权利要求的等同含义和范围内的所有变化都包含在本申请的范围内。
/>
<110>CJ第一制糖株式会社
<120>超氧化物歧化酶1变体和使用其生产谷胱甘肽或其衍生物的方法
<130>OPA22037
<150>KR 10-2021-0075689
<151>2021-06-10
<160>30
<170>KoPatentIn 3.0
<210>1
<211>154
<212>PRT
<213>酿酒酵母
<400>1
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Gly Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>2
<211>465
<212>DNA
<213>酿酒酵母
<400>2
atggttcaag cagtcgcagt gttaaagggt gatgccggtg tctctggtgt tgtcaagttc60
gaacaggctt ccgaatccga gccaaccact gtctcttacg agatcgctgg taacagtcct 120
aacgcagaac gtgggttcca cattcatgag tttggagatg ccaccaatgg ttgtgtctct 180
gctggtcctc acttcaatcc tttcaagaag acacatggtg ctccaactga cgaagtcaga 240
catgtcggtg acatgggtaa cgtaaagacg gacgaaaatg gtgtggccaa gggctccttc 300
aaggactctt tgatcaagct tatcggtcct acctccgttg taggcagaag cgtcgttatc 360
cacgccggcc aagatgactt aggtaagggt gacactgaag aatctttgaa gactggtaat 420
gccggtccaa gaccagcctg tggtgtcatt ggtctaacca actaa 465
<210>3
<211>465
<212>DNA
<213>未知
<220>
<223>酿酒酵母_CC02-2816_sod1
<400>3
atggttcaag cagtcgcagt gttaaagggt gatgccggtg tctctggtgt tgtcaagttc60
gaacaggctt ccgaatccga gccaaccact gtctcttacg agatcgctgg taacagtcct 120
aacgcagaac gtgggttcca tattcatgag tttggagatg ccaccaatgg ttgtgtctct 180
gctggtcctc acttcaatcc tttcaagaag acacacggtg ctccaactga cgaagtcaga 240
catgtcggtg acatgggtaa cgtaaagacg gacgaaaatg gtgtggccaa gggctccttc 300
aaggactctt tgatcaagct tatcggtcct acctccgttg taggcagaag cgtcgttatc 360
cacgccggcc aagatgactt aggtaagggt gacactgaag aatctttgaa gactggtaat 420
gccggtccaa gaccagcctg tggtgtcatt ggtctaacca actaa 465
<210>4
<211>35
<212>DNA
<213>人工序列
<220>
<223>F_BamHI_SOD1
<400>4
ggtaggatcc atggttcaag cagtcgcagt gttaa 35
<210>5
<211>25
<212>DNA
<213>人工序列
<220>
<223>F_SOD1_G37R
<400>5
ttacgagatc gctcgcaaca gtcct25
<210>6
<211>25
<212>DNA
<213>人工序列
<220>
<223>R_SOD1_G37R
<400>6
aggactgttg cgagcgatct cgtaa25
<210>7
<211>35
<212>DNA
<213>人工序列
<220>
<223>R_XhoI_SOD1
<400>7
atgactcgag ttagttggtt agaccaatga cacca 35
<210>8
<211>34
<212>DNA
<213>人工序列
<220>
<223>F_SpeI_SOD1_DW
<400>8
tagaactagt tgttaatgat aatatacttg aata34
<210>9
<211>35
<212>DNA
<213>人工序列
<220>
<223>R_NcoI_SOD1_DW
<400>9
gctgccatgg acactactag ctcctgaaga ccaag 35
<210>10
<211>27
<212>DNA
<213>人工序列
<220>
<223>R_AL杀手
<400>10
gagcaatgaa cccaataacg aaatctt27
<210>11
<211>24
<212>DNA
<213>人工序列
<220>
<223>F_BR杀手
<400>11
cttgacgttc gttcgactga tgag 24
<210>12
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37A
<400>12
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Ala Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>13
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37C
<400>13
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Cys Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>14
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37D
<400>14
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Asp Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>15
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37E
<400>15
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Glu Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>16
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37F
<400>16
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Phe Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>17
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37H
<400>17
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala His Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>18
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37I
<400>18
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Ile Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>19
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37K
<400>19
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Lys Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>20
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37L
<400>20
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Leu Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>21
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37M
<400>21
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Met Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>22
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37N
<400>22
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Asn Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>23
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37P
<400>23
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Pro Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>24
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37Q
<400>24
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Gln Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>25
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37R
<400>25
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Arg Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>26
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37S
<400>26
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Ser Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>27
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37T
<400>27
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Thr Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>28
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37V
<400>28
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Val Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>29
<211>154
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37W
<400>29
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Trp Asn Ser Pro Asn Ala Glu Arg Gly Phe His Ile
354045
His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro His
505560
Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val Arg
65707580
His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val Ala
859095
Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr Ser
100 105 110
Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu Gly
115 120 125
Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro Arg
130 135 140
Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150
<210>30
<211>155
<212>PRT
<213>人工序列
<220>
<223>SOD1 G37Y
<400>30
Met Val Gln Ala Val Ala Val Leu Lys Gly Asp Ala Gly Val Ser Gly
1 5 1015
Val Val Lys Phe Glu Gln Ala Ser Glu Ser Glu Pro Thr Thr Val Ser
202530
Tyr Glu Ile Ala Ala Tyr Asn Ser Pro Asn Ala Glu Arg Gly Phe His
354045
Ile His Glu Phe Gly Asp Ala Thr Asn Gly Cys Val Ser Ala Gly Pro
505560
His Phe Asn Pro Phe Lys Lys Thr His Gly Ala Pro Thr Asp Glu Val
65707580
Arg His Val Gly Asp Met Gly Asn Val Lys Thr Asp Glu Asn Gly Val
859095
Ala Lys Gly Ser Phe Lys Asp Ser Leu Ile Lys Leu Ile Gly Pro Thr
100 105 110
Ser Val Val Gly Arg Ser Val Val Ile His Ala Gly Gln Asp Asp Leu
115 120 125
Gly Lys Gly Asp Thr Glu Glu Ser Leu Lys Thr Gly Asn Ala Gly Pro
130 135 140
Arg Pro Ala Cys Gly Val Ile Gly Leu Thr Asn
145 150 155
PCT/RO/134表
