用于酵母的前导序列
文献发布时间:2023-06-19 11:32:36
本申请要求于2018年11月19日提交的美国申请第62/769,242号的优先权权益,所述美国申请的内容通过引用整体并入本文。
本申请包含根据“按照专利合作条约(PCT)在国际专利申请中呈现核苷酸和氨基酸序列表的标准(Standard for the Presentation of Nucleotide and Amino AcidSequence Listings in International Patent Applications Under the PatentCooperation Treaty(PCT))”ST.25以ASC II文本(.txt)文件呈计算机可读形式(CRF)的核苷酸和氨基酸序列表。序列表在以下鉴别出,并且以全文引用的方式并且出于所有目的并入本申请的本说明书中。
技术领域
本发明涉及促进从宿主细胞分泌重组蛋白的前导肽和编码所述前导肽的核酸序列以及包括这种前导序列的表达盒、载体和宿主细胞。还公开了一种用于使用所述前导肽产生蛋白质的方法。
背景技术
法夫驹形氏酵母(Komagataella phaffii)(以前被称为巴斯德毕赤酵母(Pichiapastoris))是一种易于操作和培养的单细胞微生物。法夫驹形氏酵母是一种真核生物,其能够进行由高级真核细胞进行的许多翻译后修饰,如蛋白水解加工、折叠、二硫键形成和糖基化。因此,相比于无法像真核细胞那样进行翻译后修饰的细菌系统,优选法夫驹形氏酵母系统。进一步地,在细菌系统中,如果可能的话,蛋白质可能以不溶形式产生,这需要昂贵的工艺来重新折叠和回收蛋白质。另外,已经证明,与许多细菌系统相比,法夫驹形氏酵母系统可提供更多的可溶性和相对纯净的分泌蛋白。因此,可以将需要翻译后修饰的外来蛋白质产生为法夫驹形氏酵母中的生物活性分子,并且法夫驹形氏酵母已被用于产生多种重组蛋白。
由于大多数酵母不会分泌大量内源蛋白,而且到目前为止,它们的细胞外蛋白质组尚未得到广泛表征,因此可用于酵母的分泌序列的数量有限。因此,通常将靶蛋白与来自酿酒酵母(S.cerevisiae)的交配因子α(MFa)的前导肽融合,以驱动许多酵母物种中的分泌表达(Kurjan和Herskowitz(1982)《细胞学》(Cell)30(3):933-943)。然而,Kex2蛋白酶对MFa的蛋白水解加工通常会在产物中产生异质的N端氨基酸残基。
EP 0 324 274 B1描述了使用截短的酿酒酵母α-因子前导序列改善酵母中异源蛋白的表达和分泌。
巴斯德毕赤酵母的基因组测序导致鉴定出54个推定的信号肽(De Schutter等人(2009)《自然生物技术(Nature Biotechnol.)》27(6):561-566和补充信息)。
WO 2014/067926 A1公开了使用突变的Epx1前导肽的蛋白质表达和分泌。
然而,仍然需要能够从酵母细胞中影响重组蛋白的高水平分泌的前导肽。
发明内容
本发明人已经分离了前导肽,所述前导肽提供与其相关的蛋白质的强表达和分泌,并因此可以用于产生重组蛋白。
因此,本发明涉及一种分离的前导肽,其选自由以下组成的组:
(a)包括根据SEQ ID No:1所述的氨基酸序列或其功能变体的肽;
(b)包括选自SEQ ID No:2、SEQ ID No:3、SEQ ID No:4和SEQ ID No:5的组的氨基酸序列或其功能变体的肽;以及
(c)包括与根据SEQ ID No:1、SEQ ID No:2、SEQ ID No:3、SEQ ID No:4和SEQ IDNo:5中任一个所述的氨基酸序列具有至少80%同一性的氨基酸序列的肽。
本发明进一步涉及一种分离的核酸分子,其包括编码根据权利要求1所述的前导肽的核酸序列。
在一个实施例中,所述核酸序列选自由以下组成的组:
(a)编码包括根据SEQ ID No:1、SEQ ID No:2、SEQ ID No:3、SEQ ID No:4和SEQID No:5中任一个所述的氨基酸序列的肽的核酸序列;
(b)包括根据SEQ ID No:6、SEQ ID No:7、SEQ ID No:8、SEQ ID No:9和SEQ IDNo:10中任一个所述的序列的核酸序列;
(c)与根据SEQ ID No:6、SEQ ID No:7、SEQ ID No:8、SEQ ID No:9和SEQ ID No:10中任一个所述的核酸序列具有至少80%同一性的核酸序列;以及
(d)在严格条件下与根据SEQ ID No:6、SEQ ID No:7、SEQ ID No:8、SEQ ID No:9和SEQ ID No:10中任一个所述的核酸序列杂交的核酸序列。
本发明进一步涉及一种表达盒,其包括与编码蛋白质的核酸序列可操作地连接的本发明的核酸分子。
所述蛋白质可以是酶、肽、抗体或其抗原结合片段、蛋白质抗生素、融合蛋白、疫苗或疫苗样蛋白质或颗粒、生长因子、激素或细胞因子。
所述酶可以选自由以下组成的组:脂肪酶、淀粉酶、葡糖淀粉酶、蛋白酶、木聚糖酶、葡聚糖酶、纤维素酶、甘露聚糖酶和植酸酶。
所述表达盒可以进一步包括可操作地连接至所述核酸分子的启动子。
本发明进一步涉及包括本发明的表达盒的载体以及包括本发明的表达盒或本发明的载体的宿主细胞。
所述宿主细胞可以是酵母细胞,所述酵母细胞选自由以下组成的组:驹形氏酵母(Komagataella)、假丝酵母(Candida)、球拟酵母(Torulopsis)、阿克氏酵母(Arxula)、汉逊酵母(Hansenula)、汉逊酵母(Ogatea)、耶氏酵母(Yarrowia)、克鲁维酵母(Kluyveromyces)、阿舒氏酵母(Ashbya)和酿酒酵母属(Saccharomyces)。
本发明进一步涉及一种用于在宿主细胞中产生蛋白质的方法,所述方法包括以下步骤:
(a)提供本发明的宿主细胞;
(b)在合适的条件下培养所述宿主细胞;以及
(c)获得所述蛋白质。
本发明进一步涉及本发明的核酸序列或本发明的前导肽用于从宿主细胞分泌蛋白质和/或用于增加从宿主细胞分泌蛋白质的用途。
附图说明
图1A-1B:与α因子前导肽或根据SEQ ID No:2所述的前导肽(被称为AmyTZ)(a)或根据SEQ ID No:3所述的前导肽(被称为Nectria)(b)融合的脂肪酶A的表达。
图2:与α因子前导肽、根据SEQ ID No:2所述的前导肽(被称为AmyTZ)或天然信号肽融合的木聚糖酶的表达。数字1-4代表不同的菌落。
图3:与α因子前导肽(右图)或根据SEQ ID No:2所述的前导肽(被称为AmyTZ)(左图)融合的淀粉酶的表达。每个通道代表单独的转化体。
图4A-4B:与α因子前导肽或根据SEQ ID No:2所述的前导肽(被称为AmyTZ)融合的脂肪酶B的表达(a)和活性(b)。
具体实施方式
尽管将针对特定实施例描述本发明,但是该描述不应被理解为限制性的。
在详细描述本发明的示例性实施例之前,给出对于理解本发明而言重要的定义。除非另有说明或从定义的性质显而易见,否则这些定义适用于本文所述的所有方法和用途。
如在本说明书和所附权利要求书中所使用的,单数形式的“一个/种(a/an)”也包含各自的复数,除非上下文另外明确指出。在本发明的上下文中,术语“约(about)”和“大约(approximately)”表示本领域技术人员将理解以仍然确保所讨论特征的技术效果的精度范围。该术语通常表示与所指示的数值的偏差为±20%,优选地为±15%,更优选地为±10%,并且甚至更优选地为±5%。
应当理解,术语“包括”不是限制性的。出于本发明的目的,术语“由...组成”被认为是术语“包括”的优选实施例。如果在下文中组被定义为包括至少一定数量的实施例,则这意味着还涵盖优选地仅由这些实施例组成的组。
此外,说明书和权利要求书中的术语“第一”、“第二”、“第三”或“(a)”“(b)”“(c)”“(d)”等用于区分相似要素,而不一定用于描述顺序或时间次序。应该理解的是,如此使用的术语在适当情况下是可互换的,并且本文描述的本发明的实施例能够以不同于本文描述或说明的其它顺序操作。在术语“第一”、“第二”、“第三”或“(a)”“(b)”“(c)”“(d)”、“i”、“ii”等涉及方法或用途或测定的步骤的情况下,步骤之间不存在时间或时间间隔的连贯性,即,这些步骤可以同时执行,或者在这些步骤之间可能存在几秒钟、几分钟、几小时、几天、几周、几个月甚至几年的时间间隔,除非在上文或下文中提出的申请中另有说明。
应当理解,本发明不限于本文描述的特定方法、方案、试剂等,因为这些可以变化。还应理解,本文所使用的术语仅出于描述特定实施例的目的,并不旨在限制本发明的范围,本发明的范围仅由所附权利要求书限制。除非另外定义,否则本文所使用的所有技术术语和科学术语具有与本领域普通技术人员通常所理解的含义相同的含义。
术语“分离的核酸分子”是指已经与其天然缔合的环境(如基因组)分离的核酸分子。在本文公开的前导肽的上下文中,该术语特别是指编码前导肽的分离的核酸分子已经与编码前导肽天然连接的蛋白质的核酸分子分离。
术语“核酸”、“核酸序列”或“核酸分子”具有其通常的含义,并且可以包含但不限于例如多核苷酸(如脱氧核糖核酸(DNA)或核糖核酸(RNA))、寡核苷酸、通过聚合酶链反应(PCR)生成的片段以及通过连接、切割、核酸内切酶作用和核酸外切酶作用中的任何一种生成的片段。糖修饰包含例如用卤素、烷基、胺和叠氮基代替一个或多个羟基,或者糖可以被官能化为醚或酯。此外,整个糖部分可以被空间上和电子上相似的结构代替,如氮杂糖和碳环糖类似物。碱基部分的修饰的实例包含烷基化的嘌呤和嘧啶、酰化的嘌呤或嘧啶或其它众所周知的杂环取代基。核酸单体可以通过磷酸二酯键或此类连接的类似物连接。磷酸二酯键的类似物包含硫代磷酸酯、二硫代磷酸酯、硒代磷酸酯、二硒代磷酸酯、硫代苯胺磷酸酯、苯胺磷酸酯、氨基磷酸酯等。核酸可以是单链或双链的。在一些实施例中,提供了编码融合蛋白或重组蛋白的核酸序列,其中所述蛋白质与本发明的前导肽连接。
本发明的核酸序列进一步涵盖密码子优化的序列,其编码本发明的前导肽。通过系统地改变重组DNA中的密码子来优化核酸,以使其在不同于分离核酸的细胞的宿主细胞中表达,使得密码子与用于表达的生物体中的密码子使用模式相匹配,从而增强被表达的蛋白质的产率。然而,密码子优化的序列编码具有与天然蛋白相同的氨基酸序列的蛋白。
如本文所使用的,术语“编码(coding for)或(encoding)”具有其通常的含义,并且可以包含但不限于例如多核苷酸(如基因、cDNA或mRNA)中的核苷酸特定序列的性质,以用作合成其它大分子(如确定的氨基酸序列)的模板。因此,如果对应于基因的mRNA的转录和翻译在细胞或其它生物系统中产生蛋白质,则该基因编码该蛋白质。在本发明的一些实施例中,使用了编码蛋白质的核酸序列,其中编码蛋白质的核酸序列与编码本发明的前导肽的核酸序列可操作地连接。
如本文所使用的,术语“前导肽”是指指导蛋白质分泌的肽。从细胞分泌的蛋白质的前导肽位于蛋白质的N端,一旦新生蛋白质链跨过粗糙的内质网开始输出,该前导肽就会从成熟的蛋白质上被切割下来。前导肽使被表达的蛋白质能够运输到质膜或穿过质膜,从而易于分离和纯化被表达的蛋白质。通常,在蛋白质被转运到质膜上或穿过质膜后,前导肽通过专门的细胞肽酶从蛋白质上被切割下来。
如本文所使用的,关于本发明的前导肽的术语“功能变体”是指在氨基酸序列中具有一个或两个点突变的那些变体,与未修饰的序列相比,这些变体具有基本上相同的前导活性。因此,与SEQ ID No:1相比,根据SEQ ID No:1所述的肽的功能变体具有一个或两个氨基酸交换,并且具有与根据SEQ ID No:1所述的未修饰的肽基本上相同的前导活性。与SEQID No:2到5中任一个的对应序列相比,根据SEQ ID No:2到5中任一个所述的肽的功能变体具有一个或两个氨基酸交换,并且具有与根据SEQ ID No:2到5中任一个所述的对应的未修饰的肽基本上相同的前导活性。
如果变体前导肽与蛋白质的融合导致重组宿主细胞将所述蛋白质分泌到上清液中(与未修饰的前导序列与所述蛋白质的融合基本上相同),则本发明的前导肽的功能变体与未修饰的序列具有基本上相同的前导活性。基本上相同的分泌是指表达前导肽的功能变体的宿主细胞的上清液中蛋白质的量是表达未修饰的前导肽的宿主细胞的上清液中蛋白质的量的至少50%或60%,优选地,至少70%或75%,更优选地,至少80%或85%,并且最优选地,至少90%、92%、95%或98%。
“序列同一性”、“%序列同一性”、“%同一性”、“%一致性”或“序列比对”是指第一氨基酸序列与第二氨基酸序列的比较或第一核酸序列与第二核酸序列的比较,并基于该比较计算为百分比。这一计算结果可以描述为“一致百分比”或“ID百分比”。
通常,序列比对可用于通过两种不同方法之一来计算序列同一性。在第一种方法中,在最终序列同一性计算中,将单个位置的不匹配项和单个位置的空位均计为不一致位置。在第二种方法中,在最终序列同一性计算中,将单个位置的不匹配项计为不一致位置;然而,在最终序列同一性计算中,单个位置的空位不计为(忽略)不一致位置。换句话说,在第二种方法中,在最终序列同一性计算中忽略空位。这两种方法之间的差异(即,将空位计为不一致位置而不是忽略空位)可能会导致两个序列之间的序列同一性值发生变化。
序列同一性由程序确定,所述程序产生比对,并且在最终序列同一性计算中将单个位置的不匹配项和单个位置的空位均计为不一致位置,从而计算同一性。例如程序Needle(EMBOS),所述程序已实现Needleman和Wunsch的算法(Needleman和Wunsch,1970,《分子生物学杂志(J.Mol.Biol.)》48:443-453),并且所述程序如下根据默认设置计算序列同一性:首先在第一个序列和第二个序列之间产生比对,然后计算比对长度上相同位置的数目,然后将相同残基的数目除以比对长度,然后将该数目乘以100来生成序列同一性百分比[序列同一性百分比=(相同残基数/比对长度)x100)]。
可以根据成对的比对计算出序列同一性,所述成对的比对显示全长上的两个序列,因此显示出其全长上的第一序列和第二序列(“全局序列同一性”)。举例来说,程序Needle(EMBOSS)产生这样的比对;序列同一性百分比=(相同残基数/比对长度)x100)]。
可以根据仅显示第一序列或第二序列的局部区域的成对的比对计算序列同一性(“局部同一性”)。举例来说,程序Blast(NCBI)产生这样的比对;序列同一性百分比=(相同残基数/比对长度)x100)]。
优选地通过使用Needleman和Wunsch的算法(《分子生物学杂志》(1979)48,第443-453页)产生序列比对。优选地,将程序“NEEDLE”(欧洲分子生物学开放软件套件(EMBOSS))与程序的默认参数一起使用(空位开放=10.0,空位延伸=0.5,对于蛋白质,矩阵=EBLOSUM62,而对于核苷酸,矩阵=EDNAFULL)。然后,可以根据比对计算出序列同一性,所述比对显示全长上的两个序列,因此显示出其全长上的第一序列和第二序列(“全局序列同一性”)。举例来说:序列同一性百分比=(相同残基数/比对长度)x100)]。
通过参考与各个亲本肽的核酸序列具有至少n%同一性的核酸序列来描述变体核酸序列,其中“n”是介于80和100之间的整数。变体核酸序列包含与根据SEQ ID No:6-10中任一个所述的亲本核酸的全长序列相比具有至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的序列,其中所述变体核酸编码具有与亲本肽基本上相同的前导活性的肽。
通过参考与各个亲本肽的氨基酸序列具有至少n%同一性的氨基酸序列来描述变体肽,其中“n”是介于80和100之间的整数。变体肽包含与根据SEQ ID No:1-5中任一个所述的亲本肽的全长序列相比具有至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的序列,其中所述变体肽具有与亲本肽基本上相同的前导活性。
在严格条件下与选自由SEQ ID No:1、SEQ ID No:2、SEQ ID No:3、SEQ ID No:4和SEQ ID No:5组成的组的核酸序列的互补序列杂交的核酸序列编码具有与根据SEQ ID No:1-5中任一个所述的亲本肽基本上相同的前导活性的肽。
术语“在严格条件下杂交”在本发明的上下文中表示杂交是在足够严格以确保特异性杂交的条件下在体外实施的。严格的体外杂交条件是本领域技术人员已知的,并且可以从文献中获得(例如,Sambrook和Russell(2001)《分子克隆:实验室手册(MolecularCloning:A Laboratory Manual)》,第3版,冷泉港实验室出版社,冷泉港,纽约)。术语“特异性杂交”是指这样的情况:分子在严格条件下优选地结合某个核酸序列,即靶序列,如果该序列是例如DNA或RNA分子的复杂混合物的一部分,但不与其它序列结合或至少很少与其它序列结合。
严格条件视情况而定。较长的序列在较高温度下特异性杂交。通常,选择严格的条件,使得杂交温度在限定的离子强度和限定的pH值下比特定序列的熔点(T
如果在上述缓冲液中存在有机溶剂(例如50%甲酰胺),则标准条件下温度为约42℃。优选地,DNA:DNA杂合物的杂交条件为例如0.1 x SSC和20℃到45℃,优选地,30℃到45℃。优选地,DNA:RNA杂合物的杂交条件为例如0.1 x SSC和30℃到55℃,优选地,介于45℃和55℃之间。例如对于长度为100个碱基对并且在不存在甲酰胺的情况下具有50%的G/C含量的核酸,确定上述杂交温度。本领域技术人员知道如何使用教科书(如上文提到的教科书或以下教科书)来确定所需的杂交条件:《分子生物学的当前方案(Current Protocols inMolecular Biology)》,约翰威利父子出版社(John Wiley&Sons),纽约(1989),Hames和Higgins(出版)1985,《核酸杂交:一种实用方法(Nucleic Acids Hybridization:APractical Approach)》,牛津大学出版社的IRL出版社,牛津;Brown(出版)1991,《基本分子生物学:一种实用方法(Essential Molecular Biology:A Practical Approach)》,牛津大学出版社的IRL出版社,牛津。
典型的杂交和洗涤缓冲液例如具有以下组成:
预杂交溶液:0.5%SDS
5x SSC
50mM磷酸钠,pH 6.8
0.1%焦磷酸钠
5x邓哈特溶液(Denhardt's solution)
100μg/mL鲑鱼精子DNA
杂交溶液:预杂交溶液
1x10
20x SSC:3M NaCl
0.3M柠檬酸钠
ad pH 7,具有HCl
50x邓哈特试剂:5g聚蔗糖(Ficoll)
5 g聚乙烯吡咯烷酮
5 g牛血清白蛋白
ad 500mL蒸馏水
杂交的典型程序如下:
任选的:于65℃下,在1x SSC/0.1%SDS中将印迹洗涤30分钟
预杂交:在50-55℃下,至少2小时
杂交:在55-60℃下过夜
洗涤:
本领域技术人员知道,取决于应用,可以修改或必须修改给定的溶液和所给出的方案。
如上所述,“基本上相同的前导活性”意指前导肽(其与根据SEQ ID No:1-5中任一个所述的未修饰的前导肽具有上述序列同一性)与蛋白质的融合导致重组宿主细胞将所述蛋白质分泌到上清液中(与未修饰的前导序列与所述蛋白质的融合基本上相同)。基本上相同的分泌是指表达前导肽(其与根据SEQ ID No:1-5中任一个所述的未修饰的前导肽具有上述序列同一性)的功能变体的宿主细胞的上清液中蛋白质的量是表达未修饰的前导肽的宿主细胞的上清液中蛋白质的量的至少50%或60%,优选地,至少70%或75%,更优选地,至少80%或85%,并且最优选地,至少90%、92%、95%或98%。
术语“表达盒”是指含有蛋白质的编码序列和控制序列(如例如可操作连接的启动子)的核酸分子,使得用这些序列转化或转染的宿主细胞能够产生被编码的蛋白质。表达盒可以是载体的一部分或可以整合到宿主细胞染色体中。在本发明的表达盒中,编码本发明前导肽的核酸序列可操作地连接至编码蛋白质的核酸序列,使得在核酸序列转录和翻译后,前导肽通过肽键与蛋白质连接在一起。
可以使用本发明的前导肽表达和分泌的蛋白质可以是任何蛋白质,如任何真核、原核和合成蛋白质。该蛋白质可以与宿主细胞同源,即其可以由宿主细胞天然表达,或者可以与宿主细胞异源,即其可以不由宿主细胞天然表达。该蛋白质可以包含但不限于酶、肽、抗体及其抗原结合片段和重组蛋白。市场上已存在的通过在法夫驹形氏酵母中异源表达而获得的蛋白质包含植酸酶、胰蛋白酶、硝酸还原酶、磷脂酶C、胶原蛋白、蛋白酶K、艾卡拉肽(ecallantide)、奥克纤溶酶(ocriplasmin)、人胰岛素、菌丝霉素肽衍生物NZ2114、弹性蛋白酶抑制剂、重组细胞因子和生长因子、人胱抑素C、HB-EGF、干扰素-α2b、人血清白蛋白和人血管抑素。
在一个实施例中,所述蛋白质是酶。所述酶可以选自由以下组成的组:脂肪酶、淀粉酶、葡糖淀粉酶、蛋白酶、木聚糖酶、葡聚糖酶、纤维素酶、甘露聚糖酶和植酸酶。
在一个实施例中,所述蛋白质是脂肪酶。脂肪酶可以具有与SEQ ID No:23的氨基酸序列具有至少80%序列同一性的氨基酸序列。在一个实施例中,脂肪酶具有根据SEQ IDNo:23所述的氨基酸序列。脂肪酶由与SEQ ID No:22的核酸序列具有至少80%序列同一性的核酸序列编码。在一个实施例中,脂肪酶由根据SEQ ID No:22所述的核酸序列编码。具有与SEQ ID No:23的氨基酸序列具有至少80%同一性的氨基酸序列或由与SEQ ID No:22的核酸序列具有至少80%同一性的核酸序列编码的蛋白质具有脂肪酶活性。术语“脂肪酶活性”是指蛋白质可以切割脂质中的酯键。通过将蛋白质与合适的脂肪酶底物(如PNP-辛酸酯、1-油精、半乳糖脂、磷脂酰胆碱和三酰基甘油)一起孵育并确定相对于对照脂肪酶的脂肪酶活性,可以确定蛋白质的脂肪酶活性。
在一个实施例中,与SEQ ID No:23的氨基酸序列相比,脂肪酶包括一个或多个氨基酸插入、缺失或取代。在一个实施例中,与SEQ ID No:23的氨基酸序列相比,氨基酸插入、缺失或取代位于选自氨基酸残基23、33、82、83、84、85、160、199、254、255、256、258、263、264、265、268、308和311的氨基酸残基处。在一个实施例中,与SEQ ID No:23的氨基酸序列相比,氨基酸取代选自由以下组成的组:Y23A、K33N、S82T、S83D、S83H、S83I、S83N、S83R、S83T、S83Y、S84S、S84N、I85A、I85C、I85F、I85H、I85L、I85M、I85P、I85S、I85T、I85V、I85Y、K160N、P199I、P199V、I254A、I254C、I254E、I254F、I254G、I254L、I254M、I254N、I254R、I254S、I2454V、I254W、I254Y、I255A、I255L、A256D、L258A、L258D;L258E、L258G、L258H、L258N、L258Q、L258R、L258S、L258T、L258V、D263G、D263K、D263P、D263R、D263S;T264A、T264D、T264G、T264I、T264L、T264N、T264S、D265A、D265G、D265K、D265L、D265N、D265S、D265T、T268A、T268G、T268K、T268L、T268N、T268S、D308A和Y311E。
下表1中示出了与根据SEQ ID No:23所述的序列相比具有一个或多个氨基酸取代或插入的另外的合适的脂肪酶,其中LIP062是指根据SEQ ID No:23所述的脂肪酶。
表:1
表:1
表:1
表:1
表:1
在一个实施例中,表达盒进一步包括与编码前导肽的核酸分子可操作地连接的启动子。
如本文所使用的,术语“启动子”是指指导结构基因转录的核苷酸序列。在一些实施例中,启动子位于基因的5'非编码区中,最接近结构基因的转录起始位点。在启动转录中起作用的启动子内的序列元件也可以通过共有核苷酸序列来表征。这些启动子元件包含RNA聚合酶结合位点、TATA序列、CAAT序列、分化特异性元件(DSE;McGehee等人,《分子内分泌学(Mol.Endocrinol.)》7:551(1993))、环状AMP反应元件(CRE)、血清反应元件(SRE;Treisman,《癌症生物学研讨会(Seminars in Cancer Biol.)》1:47(1990))、糖皮质激素应答元件(GRE)和其它转录因子的结合位点,如CRE/ATF(O'Reilly等人,《生物化学杂志(J.Biol.Chem.》267:19938(1992))、AP2(Ye等人,《生物化学杂志》269:25728(1994))、SP1、cAMP反应元件结合蛋白(CREB;Loeken,《基因表达(Gene Expr.)》3:253(1993))和八聚体因子(一般参见,编者:Watson等人,《基因的分子生物学(Molecular Biology of theGene)》,第4版(本杰明/卡明斯出版公司(The Benjamin/Cummings Publishing Company,Inc.)1987;)以及Lemaigre和Rousseau,《生物化学杂志(Biochem.J.)》303:1(1994))。
启动子可以是组成型活性的、抑制型或诱导型。如果启动子是诱导型启动子,则转录起始速率响应于诱导剂而增加,或者该启动子在存在诱导剂的情况下提供基因表达,但在不存在诱导剂的情况下不提供基因表达。相反,如果启动子是组成型启动子,则转录起始速率不受诱导剂的调节。因此,组成型启动子通常在细胞的大多数条件下具有活性。抑制型启动子也是已知的。
用于酵母细胞中(并且具体地说,法夫驹形氏酵母中)的蛋白质表达的组成型启动子包含但不限于:GAP(甘油醛-3-磷酸脱氢酶;Waterham等人(1997)《基因(Gene)》186:37-44)、TEF1(翻译伸长因子1(Ahn等人(2007)《应用微生物学与生物技术(Appl.Microbiol.Biotechnol.)》74:601-608)、PGK1(3-磷酸甘油酸酯激酶;de Almeida等人(2005)《酵母(Yeast)》22:725-737)、GCW14(Liang等人(2013)《生物技术通讯(Biotechnol.Lett.)》35:1865-1871)、G1(高亲和力葡萄糖转运蛋白;Prielhofer(2013)《微生物细胞工厂(Microb.Cell Factories)》12:5)和G6启动子(Prielhofer(2013)《微生物细胞工厂)》12:5)。
用于酵母细胞中(并且具体地说,法夫驹形氏酵母中)的蛋白质表达的组成型启动子可以是甲醇诱导型启动子。当将甲醇添加到培养基中时,甲醇诱导型启动子驱动基因表达。甲醇诱导型启动子包含但不限于AOX1(酒精氧化酶1;Tschopp等人(1987)《核酸研究(Nucleic Acids Res.)》15:3859-3876)、DAS(二羟基丙酮合酶;Ellis等人(1985)《分子细胞生物学(Mol.Cell.Biol.)》5:1111-1121)和FLD1(甲醛脱氢酶1;Shen等人(1998)《基因》216:93-102)。在一个实施例中,使用了AOX1启动子。
例如,启动子可以对细菌、哺乳动物或酵母的表达具有特异性。优选地,启动子在酵母细胞中有功能。在一些实施例中,该启动子对酵母中的表达具有特异性,即该启动子在酵母细胞中而不是在其它细胞中起始转录。
在一些实施方案中,启动子是可用于独立于甲醇来驱动蛋白质表达的启动子,其中该启动子驱动无甲醇的培养基中的蛋白质表达。这意味着该启动子在不存在甲醇的情况下是有活性的。表述“在不存在甲醇的情况下是有活性的启动子”在本文中可以与“启动子独立于甲醇来驱动蛋白质表达”和“在不存在甲醇的情况下允许蛋白质表达增加的启动子”互换使用。此类启动子在美国临时申请第62/682,053号中公开,并在本文中作为SEQ IDNo:11-17。
启动子还可以被甲醇以外的物质诱导。在不存在葡萄糖的情况下和/或通过添加乙醇来诱导异柠檬酸裂合酶ICL1启动子(Menendez等人(2003)《酵母》20:1097-1108)。通过磷酸饥饿来诱导PHO89启动子(Ahn等人(2009)《应用与环境微生物学(Appl.Environ.Microbiol.)》75:3528-3534)。通过硫胺素抑制THI11启动子(Stadlmayr等人(2010)《生物技术杂志(J.Biotechnol.)》150:519-529)。醇脱氢酶ADH1启动子在葡萄糖和甲醇上被抑制,并由甘油和乙醇诱导(US 8,222,386)。烯醇酶ENO1启动子在葡萄糖、乙醇和甲醇上被抑制,并在甘油上被诱导(US 8,222,386)。甘油激酶GUT1启动子在甲醇上被抑制,并在葡萄糖、甘油和乙醇上被诱导(US 8,222,386)。
启动子可操作地连接至编码前导肽的核酸分子,这意味着该启动子能够影响该前导肽的表达。如果编码前导肽的核酸分子与编码蛋白质的核酸序列可操作地连接,则启动子能够影响前导肽和蛋白质的表达。在一个实施例中,彼此可操作地连接的核酸序列被立即连接,即在启动子与编码前导肽的核酸序列之间和/或在编码前导肽的核酸序列与编码蛋白质的核酸序列之间没有另外的元件或核酸序列。
表达盒可以进一步含有与编码蛋白质的核酸序列可操作地连接的合适的终止子序列。合适的终止子序列包含但不限于AOX1(醇氧化酶)终止子、CYC1(细胞色素c)终止子和TEF(翻译延伸因子)终止子。
术语“载体”是指在合适的宿主生物体中克隆的重组核苷酸序列(即重组基因)的转录及其mRNA翻译所需的DNA序列。表达载体包括表达盒,并且通常另外地包括宿主细胞或基因组整合位点中自主复制的起点、一个或多个可选择标志物(例如氨基酸合成基因或赋予对抗生素(如博莱霉素、卡那霉素、G418或潮霉素)的抗性的基因)、多个限制酶切割位点、合适的启动子序列和转录终止子,这些组分可操作地连接在一起。
如本文所使用的,术语“载体”包含自主复制性核苷酸序列以及基因组整合性核苷酸序列。载体包含但不限于质粒、小环、酵母、酵母整合质粒、附加型质粒、着丝粒质粒、人工染色体和病毒基因组。可用的商业载体是本领域技术人员已知的。商业载体可从例如欧洲分子生物学实验室和Atum获得。
在优选的实施例中,根据本发明的表达载体是适合以每细胞单拷贝或多拷贝整合到宿主细胞基因组中的质粒。编码前导肽的核酸序列(任选地与蛋白质可操作地连接)也可以以每个细胞单拷贝或多拷贝提供在自主复制质粒上。优选的质粒是真核表达载体,优选地,酵母表达载体。表达载体可以是能够在宿主生物体的基因组中复制或整合到宿主生物体的基因组中的任何载体。优选地,载体在酵母细胞(如法夫驹形氏酵母细胞中)起作用。
载体可以通过本领域已知的任何方法产生。例如,连接编码前导肽和蛋白质的核酸序列并将连接的序列插入合适的载体中的程序是已知的并描述于以下文献中:例如Green和Sambrook(2012)《分子克隆(Molecular Cloning)》,第4版,冷泉港实验室出版社(Cold Spring Harbor Laboratory Press)。
术语“宿主细胞”具有其典型含义,并且可以包含但不限于例如其中已引入了含有编码本发明的前导肽的核酸序列的核酸分子或载体的细胞,优选地,编码前导肽的核酸序列与编码蛋白质的核酸序列可操作地连接。因此,宿主细胞通常是重组宿主细胞,其不同于天然存在的细胞,因为其含有天然存在的细胞中不存在的一个或多个核酸序列。在一些实施例中,宿主细胞是分离的细胞。通过根据本领域已知的方法用本发明的表达盒或载体转化细胞,可以产生重组宿主细胞。用于转化和培养法夫驹形氏酵母细胞的方法描述于以下文献中:例如,《毕赤酵母方案(Pichia Protocols)》,第2版,2007,编者:James M.Cregg,ISBN:978-1-58829-429-6。
在一个实施例中,宿主细胞是酵母细胞。合适的酵母细胞可以选自由以下组成的属组:毕赤酵母属(Pichia)、假丝酵母属(Candida)、球拟酵母属(Torulopsis)、阿克氏酵母属(Arxula)、汉逊酵母属(Hansenula)、汉逊酵母属(Ogatea)、耶氏酵母属(Yarrowia)、克鲁维酵母属(Kluyveromyces)、酿酒酵母属(Saccharomyces)、阿舒氏酵母属(Ashbya)和驹形氏酵母属(Komagataella)。
在一个实施例中,宿主细胞是甲基营养型酵母细胞。如本文所使用的,术语“甲基营养型酵母”包含但不限于例如可以使用还原的一碳化合物(如甲醇或甲烷)以及不具有碳键的多碳化合物(如二甲基醚和二甲胺)的酵母物种。例如,这些物种可以使用甲醇作为细胞生长的唯一碳源和能量源。不受限制地,甲基营养型酵母物种可以包含例如甲烷八叠球菌属(Methanoscacina)、荚膜甲基球菌(Methylococcus capsulatus)、多形汉逊酵母(Hansenula polymorpha)、博伊丁假丝酵母(Candida boidinii)、法夫驹形氏酵母(Komagataella phaffii)和法夫驹形氏酵母(Komagataella phaffii)。优选地,宿主细胞是法夫驹形氏酵母细胞。在一个实施例中,法夫驹形氏酵母菌株是营养缺陷型菌株GS115,其在his4基因中具有突变,并因此无法合成组氨酸。
在用于产生蛋白质的方法中,在获得蛋白质之前,在合适的条件下培养包括本发明的表达盒或载体的宿主细胞。合适的条件是允许蛋白质表达和分泌的条件。合适的条件是本领域技术人员众所周知的,并且包含以分批模式、分批进料模式和连续模式进行培养。
可以以工业规模培养宿主细胞,所述工业规模可以采用至少10升(优选地,至少50升,最优选地,至少100升)的培养基体积。
可以在生长条件下培养宿主细胞以获得至少1g/L细胞干重(更优选地,至少10g/L细胞干重,优选地,至少20g/L细胞干重)的细胞密度。
宿主细胞产生的蛋白质可以通过任何已知的用于分离和纯化蛋白质的工艺获得。这些工艺包含但不限于盐析和溶剂沉淀、超滤、凝胶电泳、离子交换色谱、亲和色谱、反相高效液相色谱、疏水相互作用色谱、混合模式色谱、羟基磷灰石色谱和等电聚焦。
本发明的前导肽影响与前导肽可操作地连接的蛋白质的分泌。如本文所使用的,术语“分泌”是指蛋白质跨质膜和细胞壁两者的转运。优选地,由于分泌,蛋白质存在于宿主细胞的上清液中。
优选地,使用本发明的前导肽可增加蛋白质从宿主细胞的分泌。该蛋白质与本发明的前导肽可操作地连接。与可操作地连接至酿酒酵母的交配因子α(MFa)的前导肽的蛋白质的分泌相比,分泌增加。与可操作地连接至酿酒酵母的交配因子α(MFa)的前导肽的蛋白质的分泌相比,分泌增加了至少2%,优选地,至少5%,更优选地,至少8%,并且最优选地,至少10%。与可操作地连接至酿酒酵母的交配因子α(MFa)的前导肽的蛋白质的分泌相比,分泌增加了2%到15%或5%到12%或8%到10%。增加
通过测定本发明宿主细胞的上清液和对照细胞(例如,其中所述蛋白质与酿酒酵母的交配因子α(MFa)的前导肽可操作地连接的细胞)的上清液中蛋白质的量并比较这些量,可以确定所述蛋白质的分泌的增加。
提供以下实例用于说明性目的。因此,应当理解,这些实例不应解释为限制性的。技术人员将显然能够设想对本文提出的原理的进一步修改。
实例
1.用于法夫驹形氏酵母(毕赤酵母)表达的通用方法
将前导序列克隆到pPICz骨架(Thermo Fischer)中的所关注基因(例如脂肪酶、淀粉酶或木聚糖酶)的上游。所关注基因的表达受pPICz骨架中存在的甲醇诱导型AOX1启动子或克隆到pPICz骨架中以取代AOX1启动子的根据SEQ ID No:11所述的无甲醇组成型启动子调控。将表达载体转化到法夫驹形氏酵母菌株X-33中,并通过博莱霉素选择筛选以进行转化,如以下文献所述:《pPICZ A、B和C的用户手册(User Manual for pPICZ A,B and C)》,修订日期:2010年7月7日,手册部件号25-0148。在YPD培养基(1%酵母提取物、2%蛋白胨、2%葡萄糖,于无菌水中)中的微量滴定板中培养用包括根据SEQ ID No:11所述的无甲醇组成型启动子的质粒转化的菌株的单个菌落。在BMMY培养基(2%蛋白胨、1%酵母提取物、1.34%磷酸钾,pH 6.0,100mM酵母氮碱(不含氨基酸)、0.4μg/mL生物素、0.5%甲醇)中的微量滴定板中生长用包括AOX1启动子的质粒转化的菌株的单个菌落。通过活性和蛋白质凝胶分析在24小时或48小时测定上清液中是否存在分泌的酶。
2.脂肪酶A的表达
测试了三个前导序列(α因子、AmyTZ、Nectria)在法夫驹形氏酵母中辅助脂肪酶A分泌的能力。脂肪酶的表达受甲醇诱导型AOX1启动子驱动。使单个转化体在微量滴定板中生长,并使用甲醇诱导表达48小时。通过在30℃的温度和7.5的pH下将上清液与作为底物的对辛酸酯一起孵育10分钟来测试上清液的相对脂肪酶活性。图1a)和b)显示,根据SEQ IDNo:2所述的AmyTZ前导序列(a)或根据SEQ ID No:3所述的Nectria前导序列(b)与脂肪酶的融合导致更多的转化体具有比α因子前导序列更高的活性分泌脂肪酶水平。单独的培养基或仅带有空pPICz载体的法夫驹形氏酵母菌株(Neg)用作对照。
3.木聚糖酶的表达
测试了三个前导序列(α因子、AmyTZ和天然木聚糖酶前导序列)在法夫驹形氏酵母中辅助根据SEQ ID No:21所述的木聚糖酶分泌的能力。木聚糖酶的表达受根据SEQ ID No:11所述的组成型启动子驱动。使单个转化体在微量滴定板中生长24小时。通过蛋白质染色凝胶分析测试了四个单个转化体的上清液中木聚糖酶的存在。
图2显示,与α因子或天然前导序列相比,AmyTZ前导序列的融合导致更高的分泌木聚糖酶水平。仅带有空pPICz载体的法夫驹形氏酵母菌株(Neg)和纯化的木聚糖酶(金标准;GS)用作对照。
4.淀粉酶的表达
测试了两个前导序列(AmyTZ和α因子)在法夫驹形氏酵母中辅助根据SEQ ID No:19所述的淀粉酶分泌的能力。淀粉酶的表达受根据SEQ ID No:11所述的组成型启动子驱动。使单个转化体在微量滴定板中生长48小时。通过蛋白质染色凝胶分析测试了六个单个转化体的上清液中淀粉酶的存在。
图3显示,与α因子前导序列(右)相比,AmyTZ前导序列(左)导致更高的分泌淀粉酶水平。
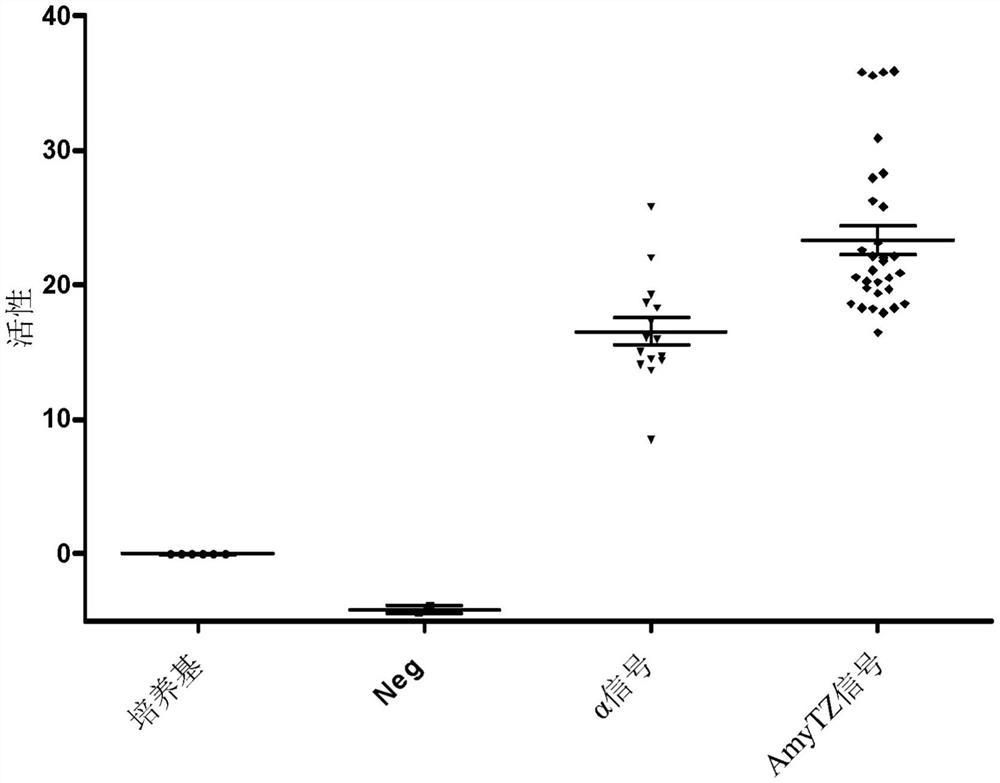
5.脂肪酶B的表达
测试了两个前导序列(α因子和AmyTZ)在法夫驹形氏酵母中辅助脂肪酶B分泌的能力。脂肪酶的表达受甲醇诱导型AOX1启动子驱动。使单个转化体在微量滴定板中生长,并使用甲醇诱导表达48小时。在30℃的温度和7.5的pH下,使用对辛酸作为底物通过蛋白染色凝胶或通过相对脂肪酶活性来测试上清液中脂肪酶的存在,持续10分钟。
图4显示,与α因子前导序列相比,AmyTZ信号的融合导致更多的转化体具有更高的活性分泌脂肪酶水平。仅带有空pPICz载体的法夫驹形氏酵母菌株(Neg)或已知表达脂肪酶C的法夫驹形氏酵母菌株(pos)用作对照。
序列表
<110> 巴斯夫股份公司(BASF SE)
<120> 用于酵母的前导序列
<130> 180263US01
<160> 23
<170> PatentIn 3.5版
<210> 1
<211> 31
<212> PRT
<213> 人工
<220>
<223> 前导肽
<220>
<221> misc_feature
<222> (13)..(13)
<223> Xaa可以是任何天然存在的氨基酸
<220>
<221> misc_feature
<222> (30)..(30)
<223> Xaa可以是任何天然存在的氨基酸
<400> 1
Met Arg Leu Leu Pro Leu Leu Ser Val Val Thr Leu Xaa Ala Ala Ser
1 5 10 15
Pro Ile Ala Ser Val Gln Glu Tyr Thr Asp Ala Leu Glu Xaa Arg
20 25 30
<210> 2
<211> 31
<212> PRT
<213> 腐皮镰刀菌(Fusarium solani)
<400> 2
Met Arg Leu Leu Pro Leu Leu Ser Val Val Thr Leu Thr Ala Ala Ser
1 5 10 15
Pro Ile Ala Ser Val Gln Glu Tyr Thr Asp Ala Leu Glu Lys Arg
20 25 30
<210> 3
<211> 31
<212> PRT
<213> 腐皮镰刀菌(Fusarium solani)
<400> 3
Met Arg Leu Leu Pro Leu Leu Ser Val Val Thr Leu Ala Ala Ala Ser
1 5 10 15
Pro Ile Ala Ser Val Gln Glu Tyr Thr Asp Ala Leu Glu Thr Arg
20 25 30
<210> 4
<211> 31
<212> PRT
<213> 人工
<220>
<223> 前导肽的变体
<400> 4
Met Arg Leu Leu Pro Leu Leu Ser Val Val Thr Leu Ala Ala Ala Ser
1 5 10 15
Pro Ile Ala Ser Val Gln Glu Tyr Thr Asp Ala Leu Glu Lys Arg
20 25 30
<210> 5
<211> 31
<212> PRT
<213> 人工
<220>
<223> 前导肽的变体
<400> 5
Met Arg Leu Leu Pro Leu Leu Ser Val Val Thr Leu Thr Ala Ala Ser
1 5 10 15
Pro Ile Ala Ser Val Gln Glu Tyr Thr Asp Ala Leu Glu Thr Arg
20 25 30
<210> 6
<211> 93
<212> DNA
<213> 人工
<220>
<223> 编码前导肽的变体的核酸序列
<220>
<221> misc_feature
<222> (37)..(39)
<223> n为a、 c、g或 t
<220>
<221> misc_feature
<222> (88)..(90)
<223> n为a、 c、g或 t
<400> 6
atgaggctgc ttccactgtt gtccgtcgtt acattgnnng ccgcttcccc aatcgcctct 60
gtccaggaat acaccgacgc tttggaannn aga 93
<210> 7
<211> 93
<212> DNA
<213> 腐皮镰刀菌(Fusarium solani)
<400> 7
atgaggctgc ttccactgtt gtccgtcgtt acattgactg ccgcttcccc aatcgcctct 60
gtccaggaat acaccgacgc tttggaaaaa aga 93
<210> 8
<211> 93
<212> DNA
<213> 腐皮镰刀菌(Fusarium solani)
<400> 8
atgaggctgc ttccactgtt gtccgtcgtt acattggctg ccgcttcccc aatcgcctct 60
gtccaggaat acaccgacgc tttggaaaca aga 93
<210> 9
<211> 93
<212> DNA
<213> 人工
<220>
<223> 编码前导肽变体的核酸
<400> 9
atgaggctgc ttccactgtt gtccgtcgtt acattggctg ccgcttcccc aatcgcctct 60
gtccaggaat acaccgacgc tttggaaaaa aga 93
<210> 10
<211> 93
<212> DNA
<213> 人工
<220>
<223> 编码前导肽变体的核酸序列
<400> 10
atgaggctgc ttccactgtt gtccgtcgtt acattgactg ccgcttcccc aatcgcctct 60
gtccaggaat acaccgacgc tttggaaaca aga 93
<210> 11
<211> 1501
<212> DNA
<213> 人工
<220>
<223> 启动子pSD001
<400> 11
tccagtgtag cactaaaatc taatatcttc ggctttatac ttttttgttc atccgaaagc 60
ttacgaacaa ttctttctcc tgttttattg tggatataga caatttcgtc agtttcttgg 120
agagaagagt tatttccggt tttggctggc cctataaacg ggttcttgga tttggatcta 180
gtaataaaaa tgtcactgtc attctcggag ctgaactttg tgttgtacga agatgggttg 240
ttccactgtt ttgccagctc ttcattgatg attttcttag tgggtgttct tggaggttca 300
cgttgcctat aatcttgacg ttcttcttca tcactatcga tgccatcaaa attaagcgtc 360
cttattgcag gcttttgtga tttcaactgc aatccttcta tctcttcatc agagctttcg 420
aactgaatac tatcactcaa aactggcgac attgcacatt tccgcaaacc atttcgggaa 480
tctatgctag ctcttctaga cgataaagaa cgaccggaac caatacgggg ttgtgcaggt 540
gggaataaat atgttggttt ggattcttga cgtgaagaag gtattctagt cgatgaagtg 600
gttgataagg atatggcgtc actgagttgt tttcttttcc tatgttgcgg tgttgggtca 660
ggagttaatt gattcacctc cataactctg gaatttcttg aatgtggggt tttcagatgg 720
gcatctttct tgacggggtt gtgagtaacg gaggaacctg gtgtcttggg tgtgaacggt 780
gtttgagcct gtacgcggtt acttctgggc ggagtactcg gagtcatgag agccattgat 840
tagaaggtga atgagggagt caccactcta agcaaacaaa atgaggtcga agcaaaaaat 900
aaagtaaagt agcacttctg gcaggttaga tcaaagagtg acgggagatt tgaagatggc 960
tggtttttcc ttagtcttgg aagaggtttg tgtgggtatc agcgaatatt ccccgattag 1020
gcaaattagt tgcattgaaa ttaacacgac atggtgattt gtggtaacaa atatctattg 1080
gtggttggtg tgtgggtgta atagtggtcg tgtcatgatg atggtgttca ggtgttgtca 1140
tagatcggtc ttcagtaaga gaaggaagct tggtgacgat cacagctatg atgtaataga 1200
aattgctaag caattgtgag gtgtgatgta ttttgcagag caattgtgcg gtacaacggg 1260
gtgttattgt cttcacaagg catttattgc gaatttcgta gttgaaagaa tattttagca 1320
cagggtgctt gacccctatt gttgctcgct aaaccatgat tgctaaatga tgacatagca 1380
atcactttac taagattgct ataaggacac ctttcttagt ataaatggac actcttttcc 1440
cctgctaaac ttcttttatt tttcacactt aaacagttac aaaacacaaa cacaactaga 1500
a 1501
<210> 12
<211> 1501
<212> DNA
<213> 人工
<220>
<223> 启动子pSD002
<400> 12
gtgctaaaat ctgaggttta caagctgtga tgttccccta agatctcaca atcgaacaat 60
cgcgaagcca atgcaagttg tttaagggga aacgactcac tattcctgaa attagtattc 120
aaaacttggt ccggaagaac aatgaggcgg ccgttaaaat actcacgtaa acggtgtcta 180
caagcgcatt aaaatccgtt tgaattcaag caaaagccac cagaggctta tgcttggtta 240
tacccagcat tgacctttgg tatgagcatc tgaaaaacaa ccaggtgttg caaagttaaa 300
catccttctt tgttcatata gaacccacta ttcatggtac tccccaatcg aatttcacat 360
tctggttttg aaattacaca ccacgttagc ttataagatt tcatataact tattgatata 420
cggtttccat tgttcgaata gttgaggttg tatgtaattc gattgaaggg gccatttttg 480
tttcctactt ttcctgggag cttatccgat gcgcttcaaa gctggaattg taaatataga 540
gaaaaagaag gatgttgttt tattcttgaa agagtataat tttacttcta gcaactctcc 600
cacttcgctt gacttcattt atttcttggg cacataggcg tagtaatcta gaccaacaga 660
taatttgccg gaatgatata gcgattggaa aatgaactga aattttttgc tgtctttcaa 720
tttgacgggc agttcatcag tgaccgacca tataaatacg ttgagaatgt tattcttcct 780
cgtagttgaa gtggcttcat aatttcagaa ctcaatagat aaactaggat gttttaaagc 840
aattaatgct cacaagtaag gagcgactct cttgcttttc gaatactaaa agtatcgtcc 900
caacccagaa aaaaagacct cttaactgca aaataaactc tatatatttc ttctaaaaca 960
gtttcaggtt ggatagtatc gcattctcat cacttctaac tagtaggcca tgagatatat 1020
taacgtttac ttgagttcta agttctccga attagatgca cagcacaaac aagattaggt 1080
ttcacttggt acaaaatacg aacagagttt aaggtcgtaa tttcatttcg ttattgatcc 1140
ccacaatcta ttcttatcac agtcatcaga tagtcgcgaa aaagcatgca gaaaaggggg 1200
tcgtccctat ctaagttgta gcattacaac aaatatgact acactcagtg tcgcaatcgg 1260
tatagccaac gctgcaaaat ggattctact gagaatggta tgatgatccc aggatcaatt 1320
tcccaaaaat taaaaaaagt aaaataaaaa gcatcagata ttagggaggt ggtaagattg 1380
ctctgcaagc gatcacgaga ttttaggttt tcctttatgt actatataaa gcgcagattg 1440
gatgccgctt ttccctcctg ggctatgata atatagcgaa cgaaatacac gccaaaataa 1500
a 1501
<210> 13
<211> 1500
<212> DNA
<213> 人工
<220>
<223> 启动子pSD003
<400> 13
tcacattcat agcatctctc gcctgcaata gcttccacga taggaatatc tgtgaaagtg 60
aacatgctat ttcgatgata taagacttta agatctggca tgtttgtgtt ggaggttacc 120
ctggggtcaa taaccctaat tatctccttc actaaaaatg atgaagattc ttcggattcg 180
tttttgaaca gagttaatgc catttcttcg tcaatagaaa aatcaatatc tggtatctca 240
tcttttacat attgaggatt tagttttctt ccctttggat agtacattat gatcaatgta 300
ttcctgtctt tattgataaa gtattggcat tctgcttctt gtacaccttt gaattgtttg 360
tctggaagtg actgacattt ttccacattg ctaacggttt ggcacgaatt acatctaaat 420
aaaatgtctt ctccggattc gtgtattaag tgatactcca atgataaatc cccacctatc 480
gaaccagaat cggcattggc cacagtcaca ggtaacttta ggtcttgaaa aatccttcta 540
taggcttcat tgacattgtc ataagactta agaccatctt ctttggtcaa gtcaaaagaa 600
taggcatctt tcatgagaaa ctctcgtcct ctcaacaaac ctcccctagg tctcaactca 660
tctctatatt tgcgggaaat ttggtacacg agaaggggta aatctttata tgacgaacat 720
aagtcaccaa ctaagtttgt gatttcctct tcacaagttg gcactaaaca gtagtctcta 780
tccttggagt ctttgaactt gaacaattca ttgttgtccc atctcttagt tctctcccat 840
aaatgcttgg aagacaggct acttaattcc atttccagcc caccagcctg atccattctt 900
ttcctaatta cattttgaag ctttttatag gtacggagtc ctaatggaag ccagtgaact 960
attcctgctg caggctggta aataaacctt gattgaagga gcatatcatg agtagtaagg 1020
tcctttacag aaaatagttt acttccttga agagaagtag aataaaacct catgttgggt 1080
ctccatgaaa ggttcaaagg cattgatcct ttaggtactt caggatgttt aagtcatcaa 1140
actgtccatc aaaggtagta tagtatttac catctagata gtgatgtatg ggtgtaacac 1200
aacatttaaa tgttgtaaat taacattagg actgagtccg gagatgctat tgtcacctaa 1260
atctattaga aagcacttca gttatatcat cgatagaggt ttgaagataa acctattgtt 1320
gataaataac cccattaccc gtttacgtag caaggttcaa aaatttgctt agatcggagc 1380
taaaaattcg actgacttct ttcgaaaatg tggattatgc aagcaacgtt gctatcggaa 1440
tagtatataa ggtcgatctg ccccattaca aattgtaaag caacaaacat cctacgcaaa 1500
<210> 14
<211> 1500
<212> DNA
<213> 人工
<220>
<223> 启动子pSD004
<400> 14
tcagtttcac ggttatgtga gctgtctccg cgtgaggcag taacctctgt gtcatggata 60
caggctggta cacatttggc agtaggaaca caatctggtt tagttgaaat atgggacgcc 120
acgacgtcca aatgtacaag atcaatgact gggcattcgg cccgaacctc agcgctgagt 180
tggaaccgtc atgttttgag ttctggttca agagatcgca gtatcttaca tcgggatgta 240
cgtgcagcag ctcactatac aagtcgcatt gttgaacacc gccaagaggt ttgtggctta 300
cgttggaacg tggatgaaaa caagctggcc agtggttcca atgataaccg tatgatggta 360
tgggatgcac tgcgtgtaga acagcccctt atgaaagttg aagagcatac tgcggctgtt 420
aaggcgttgg catggtcacc tcatcaacgt ggaatactgg cttcgggtgg aggtactgct 480
gacagacgta tcaaggtgtg gaatacttta acaggatcca agctgcacga tgttgatact 540
ggatctcaag tttgtaatct cttgtggtct cgcaattcta atgaattggt aagtactcat 600
ggatattctc gaaaccaagt cgttatttgg aaatatccgc aaatgaagca actagcatct 660
ttgactggtc atacttatcg agtcctttac ctttccatgt cacctgatgg aactacagtc 720
gtaacggggg ctggagacga aactttaaga ttttggaact gtttcgagaa gtcacgacaa 780
agcggaggag gatcaatatt actagacgct tttagtcagc ttcgttaaat taccaccaaa 840
tttggtgcaa aagggcccat atggtgctac aaccaaagga actttctaat tttgataatg 900
atgtcatttc tctcatcggg atgaaaatag aagtcgaaag gatttttgtc actatttcaa 960
gccccacctg cagctggcag catttctatt gtttatgcat tgtcatttat gggaaaacta 1020
agaaagttcc tctccacccg gactccactg gtaaatatgc gatatcggaa tcatgaccaa 1080
cccatatttt gatcctaatc atttcggttc tagtctccga tcggactccg taaaactgcg 1140
gagtgaactc caacggagaa tactgcagcc aatctcatat ttcatttgtt atttgtccct 1200
caactgtctc gataaggtca tctgtgtttg actagatgtt cgtcattggc atgtcaaaca 1260
aggctagacc ttacaatcat ctcttacgaa tgtaagtgaa tgtaactata ttttccttgc 1320
tactttaacg aggttaacca acccccgcac atccccacac caccgctctt gataagcatc 1380
tccgaaaatg catgacgcga caacttcaag catgttgtat ttactgagtt ttcagcctca 1440
ctatcgatac ctctataaat agaggcactt tcgtctcttc tccctcccca caagaaacca 1500
<210> 15
<211> 1500
<212> DNA
<213> 人工
<220>
<223> 启动子pSD005
<400> 15
agaagtactg ttatgaatcg atcgacgtga catgttgttg atggttctga cttcttgatg 60
tccgcgtttt ctgtctctca atagtggtgt tcgggggaag tatggttcta atacttaaca 120
ggtaagatgg ttgcaatgag cacctggtaa agcaacttga atttcctgcc ctgtctccgt 180
taagttatat tcgactcaag gtccttgctt cctgtctgtt ctgtaaaact tccctttggt 240
gtcttctata tcaactttaa aaacaaggta gtgtgtcgag cgatagtact gtgtcttttt 300
ccctatgaaa aaaatcgcac catccaagac ttctcacctt caacagcttc aacatcatgt 360
tcggtccttt tagagctacg ctggtcgatc taggaggtct gctatggaaa cgtccttgga 420
gaatgtccaa accacagaaa tatagactcc gcaaaagaat gcaacttgta gactccaata 480
tcgacattat ttaccaggga ctgactgagg agggtctgtc ttgcaaagtg atagataact 540
tgaaacaaaa cttcccaaag gagcatgaag tgctccccaa aaacaagtat accgtgttta 600
acaagacagc caaaaactat agaaagggtg ttcatttggt tccaaaatgg accaagaagt 660
ctttgagaga gaaccccgag ttcttctaat tgcacatttc ttcctgttca tagattatcc 720
cacacatagt tgctcacaaa aaaatcacta taattttcct ccaccggcag tatatcacta 780
acacctttat ctttattgta gattataatc tgatctttat ccttagatgt atctatcatc 840
aaccccatgc tcttgaaaag cttgagtctt aacactgtcg aatcgtagtt ttcttgtaga 900
tcattcgata tcactgcttt ttcttgctct tctaattcgt tgagattctg ggtcaaacta 960
gagattgaat tctgaaggtg attcatgttc atctccagat ctgttattga ttttgctaat 1020
ttaaattttt cgtgttcaag ctcttcgata ctctttaggg tctgttgacg gtcttctgtt 1080
tccaataatt gcttgttgaa ctctttaagt tcgtctctct gtttactgat acgtgacaac 1140
aaatctagct ggtgatcgag tttaagtttc cgtttggagc tcaacagaga aagattttca 1200
ttaatttggt tgatagtttg cacgtccggt tcgatctgaa aattctctat agtcgacctg 1260
attaaggaca cagtctcttg aagatcggac attggattta tggagaaggg agatcaaagc 1320
ggaaccagtt gcactgttta cctttccagt cgagatactt atcccacagg gccctcactt 1380
tccaggcaga agtcacctag gaggcgcatc cctccgtttg cttccctcgc gacaaactcc 1440
cctgtaaaag aaaacttcac tgaatcgtac acctaatcat acgacactaa cacagatata 1500
<210> 16
<211> 1500
<212> DNA
<213> 人工
<220>
<223> 启动子pSD007
<400> 16
gtcctttcca aatttttggt tgaaggcatc gcttaaatta tgagcaggat cggtggaaat 60
aagcaggtat ttcttgttag gattgtgaag ggcaagctgg atagatatag aagaagatgt 120
cgtggtttta ccgacacccc ccttacctcc aacaaagatc cacttcagcg attcgtggtt 180
cacaattgat cgcaaacttg gctctgcctc aatatccatg gttgatgtct agttgagtgg 240
cgtttgtggt ctcttgatga gttcaaggcg aaagaatatg ataggaaagc atggtttgaa 300
cttttcgcga aagaaggaat actgttccgc gagaaactcc ccggtgccag aaccttccat 360
tgaggttaat cggtgggagg tgttcgaatg acaatgtcag acaaggcgaa cacgtcttgt 420
gacaccagct ggactaagaa gattcggtat gcaccgaaga agaaggccgt gtctcaattg 480
gcaactttgc aacaaactac ggaggaaaag tctcacaagc ttttaaccaa gttgaatcac 540
gacgacaacg ataaagaaat cctcaaccat ctaacacatg aagtacaaag tagaaatgtg 600
atcttattgg acaaactaga ggagctcaac aaggaactgg gctggattaa agaccgaaaa 660
tgaggaacca tgagcactgg gcgtttccag aaaaactgca accaacgatg ggaaaatgat 720
accacactac tatggtcacc ccacattgtg aaatttcaaa ccaaaaaaga tcaaccccat 780
aattccccag agggttttcc caacaatttt ccaacggact tgataatgag tcagatcatt 840
tgagcatatt catcttaccc cttattccgt gacaatttac ctattccatt caaagcatac 900
ggtatcccgt gaccttctca tggagatcat tctccaccga tacagcatat acacagatat 960
acccaactaa tatcaattgg accttgatat ggtcgacctt gatggtcccg tccaacctta 1020
aaacttagtt taatgctata ctttcgcctt gaaccaaatc tgtctccccc tcaatcatct 1080
ctatgcaaga aggtcaacac tgattacgtg agcaacagcc agcaatcgtt cgagtccccg 1140
ccaaaaaagg cggagttact gctccttgtg accacacccc ctgagaccac gtccctaaac 1200
gatccttgtc ggttccttcg tccaattggc aattgccacg catacgtgaa tcgttattgt 1260
ttcgcctacc ttgcgtcatt cgttccagaa tgttcgacat actcctctag aacataccgt 1320
cacaccacca tcttaagtta tcttcacgtg accatgacgt acattgtagt tgactacccc 1380
attctcatca ttccgatgcg gccaaaaatc tctatataaa gaccgtatcc cctaatattc 1440
tcttcttgtt aagacattaa cttagttaat tcaccaatta ctcacttata aacaaacaaa 1500
<210> 17
<211> 1500
<212> DNA
<213> 人工
<220>
<223> 启动子pSD008
<400> 17
gtttctcttg gggagatact tttttcgcgt gctcctccgt gcggaacttc cttctgagct 60
tctacctctc agattagtct aatcgcatca ggaataagac tgagaatgct tttaaggaga 120
ggcttgagat tggctaattg cgttccgaag tactctttca aaaggagtta tacccctctc 180
aactacgatt ctctaaagaa ttatcgtagg catgctcagg cgcctcaacc ccatcagttt 240
gacgccacta gatgggacca acaaccagtt actaatgagc aaggagtaat actcccatcc 300
gactcaattg caaacattct gagacaacca actctggtca tagaacggca aatggaaatg 360
atgaatatat ttttaggatt tgagcaggcg aaccgatatg ttatcatgga tcctacagga 420
agtattttgg gttacatgct agaaagggat ctgggcatca ccaaagctat attgagacag 480
atctaccgtt tgcatcgacc ttttacagtg gatgtaatgg atactgcagg aaatgtatta 540
atgacaatca agaggccgtt tagtttcatc aattcgcaca tcaaagctat attaccccct 600
ttcaggaaca gcgacccaga cgaacatgta attggagaat ccgttcaaag ctggcatcct 660
tggagacgaa gatacaatct atttacagca caaattggcg aaaaggacac tgtctacgat 720
cagttcgggt acattgacgc accgtttctt tcctttgagt ttcctgtact ttcagaatct 780
aggcaaacgc taggtgctgt ctctagaaac ttcgtgggct ttgcaagaga gcttttcaca 840
gatacaggag tttacatcat ccgtatgggg cctgaatctt ttgtagggct agaagggaac 900
tacgggaaca atgtggccca acatgccctt acgctggacc aaagggctgt attattagcc 960
aatgccgttt caattgactt tgattacttt tctaggcact cgtcacacag tggtggcttc 1020
attgggtttg aggaatagac agggtctcgt caactcagct cctgccacca aaccaatcat 1080
tgatcaacga gcacactttt gtccacgtga gatcgctttc gcttgcagaa agagcaatgc 1140
atgaaaacgg caaacgcaaa acgagcaaaa aaacgagtaa ataactacaa tttcaccacc 1200
aacagggtca aagagctttt gagacactat aaaaggggcc ctttcccccc aggttccttg 1260
aaatcctcat tcaattatgt tttttactca taatttgact caattggcat cttcttcttt 1320
gttcatatac agtaattgat atgacgctta gtcattatta gtgttctcga ctagcagtgg 1380
cgaaaaaagg gggagttatt ttctagaacc gaccgcaaac tataaaagaa agctgcccct 1440
catatacctt tcgaattctt tattttctgt gtttcttccc tatttaacat ctacacaaaa 1500
<210> 18
<211> 1302
<212> DNA
<213> 人工
<220>
<223> 淀粉酶
<400> 18
ggtgtcatgg ttcacttgtt ccaatggaaa tacaatgaca ttgccaacga gtgtgagaag 60
gttcttggtc caaaaggtta tgaagctgtg cagattactc cacctgctga gcacttgcaa 120
ggatcttcct ggtgggttgt ctaccaacct gtttcctaca agaacttcac ttctctggga 180
ggtaacgagg ctgaattaaa atctatgatc gctagatgta aggctgccgg tgtcaagatt 240
tacgctgacg ctgtattcaa ccaattggct ggtggatcag gtgtcggtac aggtggatct 300
agctacaacg ccggttcctt ctcataccca caatttggct acaacgattt ccatcacgct 360
ggtccattga ccaactatac tgacagaaac aatgtgcaaa acggtgcctt gcacggtttg 420
ccagacttgg ataccggatc tgcctatgtt caagaccagc ttgctaccta catgaagacc 480
ttgagtggct ggggagttgc tggttttcgt cttgacgcag ctaagcacat gtctgttgcc 540
gatttatcgg ccattgtctc aaaggctggt aacccttttg tctactccga ggttattggt 600
gccactggtg agccaatcca accaggtgaa tacacaggaa ttggtgcagt tactgaattc 660
aaatacggta ctgacctagc ttccaacttc aagggacaga ttaagaactt aaagtctatg 720
ggcgagtcat ggggtttgct tgctagtaac aaggctgaag tctttgttgt caaccacgac 780
cgtgagagag gtcatggagg tggaggtatg ttgacttaca aggatggtgc tttgtacaat 840
ctggccaaca tcttcatgct ggcttggcca tatggtgctt atcctcaggt tatgtccggt 900
tacgacttcg gtaccaacac tgatattggt ggtccatctg ctaccccttg ttcttccggt 960
tcttcctgga actgcgaaca cagatggtct aacatcgcta acatggtctc tttccacaat 1020
gctgcccaag gaacttccat gaccaactgg tgggacaatg gtaataacca gattgctttc 1080
ggtagaggtg ccaaagcttt tgttgtcatc aacaatgagt cttccacttt gagaaagaag 1140
ttgcaaactg gtctgccagc tggtgagtac tgtaacattt tggccggtga tgctttgtgt 1200
tctggttcca ccatcaaggt tgatgcttct ggtatggcta ccttcaacgt tgcaggtatg 1260
aaggctgcag ctatccatat caatgccaag ccagattcct aa 1302
<210> 19
<211> 433
<212> PRT
<213> 人工
<220>
<223> 淀粉酶
<400> 19
Gly Val Met Val His Leu Phe Gln Trp Lys Tyr Asn Asp Ile Ala Asn
1 5 10 15
Glu Cys Glu Lys Val Leu Gly Pro Lys Gly Tyr Glu Ala Val Gln Ile
20 25 30
Thr Pro Pro Ala Glu His Leu Gln Gly Ser Ser Trp Trp Val Val Tyr
35 40 45
Gln Pro Val Ser Tyr Lys Asn Phe Thr Ser Leu Gly Gly Asn Glu Ala
50 55 60
Glu Leu Lys Ser Met Ile Ala Arg Cys Lys Ala Ala Gly Val Lys Ile
65 70 75 80
Tyr Ala Asp Ala Val Phe Asn Gln Leu Ala Gly Gly Ser Gly Val Gly
85 90 95
Thr Gly Gly Ser Ser Tyr Asn Ala Gly Ser Phe Ser Tyr Pro Gln Phe
100 105 110
Gly Tyr Asn Asp Phe His His Ala Gly Pro Leu Thr Asn Tyr Thr Asp
115 120 125
Arg Asn Asn Val Gln Asn Gly Ala Leu His Gly Leu Pro Asp Leu Asp
130 135 140
Thr Gly Ser Ala Tyr Val Gln Asp Gln Leu Ala Thr Tyr Met Lys Thr
145 150 155 160
Leu Ser Gly Trp Gly Val Ala Gly Phe Arg Leu Asp Ala Ala Lys His
165 170 175
Met Ser Val Ala Asp Leu Ser Ala Ile Val Ser Lys Ala Gly Asn Pro
180 185 190
Phe Val Tyr Ser Glu Val Ile Gly Ala Thr Gly Glu Pro Ile Gln Pro
195 200 205
Gly Glu Tyr Thr Gly Ile Gly Ala Val Thr Glu Phe Lys Tyr Gly Thr
210 215 220
Asp Leu Ala Ser Asn Phe Lys Gly Gln Ile Lys Asn Leu Lys Ser Met
225 230 235 240
Gly Glu Ser Trp Gly Leu Leu Ala Ser Asn Lys Ala Glu Val Phe Val
245 250 255
Val Asn His Asp Arg Glu Arg Gly His Gly Gly Gly Gly Met Leu Thr
260 265 270
Tyr Lys Asp Gly Ala Leu Tyr Asn Leu Ala Asn Ile Phe Met Leu Ala
275 280 285
Trp Pro Tyr Gly Ala Tyr Pro Gln Val Met Ser Gly Tyr Asp Phe Gly
290 295 300
Thr Asn Thr Asp Ile Gly Gly Pro Ser Ala Thr Pro Cys Ser Ser Gly
305 310 315 320
Ser Ser Trp Asn Cys Glu His Arg Trp Ser Asn Ile Ala Asn Met Val
325 330 335
Ser Phe His Asn Ala Ala Gln Gly Thr Ser Met Thr Asn Trp Trp Asp
340 345 350
Asn Gly Asn Asn Gln Ile Ala Phe Gly Arg Gly Ala Lys Ala Phe Val
355 360 365
Val Ile Asn Asn Glu Ser Ser Thr Leu Arg Lys Lys Leu Gln Thr Gly
370 375 380
Leu Pro Ala Gly Glu Tyr Cys Asn Ile Leu Ala Gly Asp Ala Leu Cys
385 390 395 400
Ser Gly Ser Thr Ile Lys Val Asp Ala Ser Gly Met Ala Thr Phe Asn
405 410 415
Val Ala Gly Met Lys Ala Ala Ala Ile His Ile Asn Ala Lys Pro Asp
420 425 430
Ser
<210> 20
<211> 1161
<212> DNA
<213> 青霉素(Penicillium sp.)
<400> 20
gccggcttga acaccgccgc taaggctatt ggtttgaagt acttcggtac tgctactgac 60
aacccagagt tatctgatac tgcttatgaa acccagctaa acaatactca agatttcggc 120
cagttgactc cagcaaactc tatgaagtgg gacgccactg agcccgagca aaatgtcttc 180
actttctctg ctggtgacca aatcgctaat ttggcaaaag ctaacggtca gatgcttaga 240
tgtcacaatt tggtttggta caaccagctg ccatcctggg tcacctcggg atcatggacc 300
aatgagacac tcctggcagc catgaagaac cacattacca acgtcgttac ccattacaag 360
ggtcagtgct atgcttggga tgtggtcaac gaagccttaa acgacgatgg tacttaccgt 420
tccaacgtct tctaccaata cattggtgaa gcttatatcc ctatcgcttt cgccactgct 480
gccgccgccg accctaacgc caaactttac tataacgatt acaacatcga ataccctggt 540
gctaaggcta ctgctgccca aaacctggtt aagttggtgc aatcctacgg agctagaatt 600
gatggtgtcg gtttgcagtc acactttatt gttggtgaga ctccttctac ttcttcccaa 660
cagcagaata tggctgcctt tacagcattg ggcgttgagg ttgctatcac cgaattggat 720
attagaatgc aattgccaga gaccgaggcc ttgctgactc aacaggccac tgactaccaa 780
tcaactgttc aagcttgtgc caacaccaaa ggttgtgtcg gaattaccgt ttgggactgg 840
accgataagt atagttgggt tccatctact ttttccggtt acggggacgc ttgcccttgg 900
gacgctaact atcagaagaa accagcttac gagggtatct tgaccggtct aggtcaaacg 960
gtgacctcca ctacatacat cattagccca accacttctg ttggtaccgg taccacaact 1020
tcttccggag gttccggtgg aaccactgga gttgctcaac attgggagca gtgtggtgga 1080
ttgggttgga ccggtccaac tgtttgtgcc tctggttaca cttgtactgt cattaatgaa 1140
tactattctc aatgtttgta a 1161
<210> 21
<211> 386
<212> PRT
<213> 青霉素(Penicillium sp.)
<400> 21
Ala Gly Leu Asn Thr Ala Ala Lys Ala Ile Gly Leu Lys Tyr Phe Gly
1 5 10 15
Thr Ala Thr Asp Asn Pro Glu Leu Ser Asp Thr Ala Tyr Glu Thr Gln
20 25 30
Leu Asn Asn Thr Gln Asp Phe Gly Gln Leu Thr Pro Ala Asn Ser Met
35 40 45
Lys Trp Asp Ala Thr Glu Pro Glu Gln Asn Val Phe Thr Phe Ser Ala
50 55 60
Gly Asp Gln Ile Ala Asn Leu Ala Lys Ala Asn Gly Gln Met Leu Arg
65 70 75 80
Cys His Asn Leu Val Trp Tyr Asn Gln Leu Pro Ser Trp Val Thr Ser
85 90 95
Gly Ser Trp Thr Asn Glu Thr Leu Leu Ala Ala Met Lys Asn His Ile
100 105 110
Thr Asn Val Val Thr His Tyr Lys Gly Gln Cys Tyr Ala Trp Asp Val
115 120 125
Val Asn Glu Ala Leu Asn Asp Asp Gly Thr Tyr Arg Ser Asn Val Phe
130 135 140
Tyr Gln Tyr Ile Gly Glu Ala Tyr Ile Pro Ile Ala Phe Ala Thr Ala
145 150 155 160
Ala Ala Ala Asp Pro Asn Ala Lys Leu Tyr Tyr Asn Asp Tyr Asn Ile
165 170 175
Glu Tyr Pro Gly Ala Lys Ala Thr Ala Ala Gln Asn Leu Val Lys Leu
180 185 190
Val Gln Ser Tyr Gly Ala Arg Ile Asp Gly Val Gly Leu Gln Ser His
195 200 205
Phe Ile Val Gly Glu Thr Pro Ser Thr Ser Ser Gln Gln Gln Asn Met
210 215 220
Ala Ala Phe Thr Ala Leu Gly Val Glu Val Ala Ile Thr Glu Leu Asp
225 230 235 240
Ile Arg Met Gln Leu Pro Glu Thr Glu Ala Leu Leu Thr Gln Gln Ala
245 250 255
Thr Asp Tyr Gln Ser Thr Val Gln Ala Cys Ala Asn Thr Lys Gly Cys
260 265 270
Val Gly Ile Thr Val Trp Asp Trp Thr Asp Lys Tyr Ser Trp Val Pro
275 280 285
Ser Thr Phe Ser Gly Tyr Gly Asp Ala Cys Pro Trp Asp Ala Asn Tyr
290 295 300
Gln Lys Lys Pro Ala Tyr Glu Gly Ile Leu Thr Gly Leu Gly Gln Thr
305 310 315 320
Val Thr Ser Thr Thr Tyr Ile Ile Ser Pro Thr Thr Ser Val Gly Thr
325 330 335
Gly Thr Thr Thr Ser Ser Gly Gly Ser Gly Gly Thr Thr Gly Val Ala
340 345 350
Gln His Trp Glu Gln Cys Gly Gly Leu Gly Trp Thr Gly Pro Thr Val
355 360 365
Cys Ala Ser Gly Tyr Thr Cys Thr Val Ile Asn Glu Tyr Tyr Ser Gln
370 375 380
Cys Leu
385
<210> 22
<211> 954
<212> DNA
<213> 腐皮镰刀菌(Fusarium solani)
<400> 22
gccattactg cttctcaatt ggactacgaa aacttcaagt tttacatcca gcacggtgcc 60
gctgcttact gtaactccga aactgcctct ggtcaaaaga tcacttgttc cgacaacggt 120
tgcaaaggtg tcgaagctaa caacgctatt attgtcgcct ctttcgttgg aaaaggtact 180
ggtattggtg gttacgtttc tactgataac gttagaaagg agatcgtttt gtctattaga 240
ggttcttcca acattcgtaa ctggttgact aacgtcgact tcggacaatc ctcttgttct 300
tacgttagag attgtggagt tcacactggt ttcagaaatg cttgggacga gattgcccaa 360
agagctagag acgctgtcgc taaagctaga actatgaacc catcttacaa ggttatcgct 420
actggtcact ctttgggtgg tgctgttgcc actttgggtg ctgctgattt gagatccaag 480
ggtactgccg tcgatatctt tacttttggt gccccaagag ttggtaacgc tgagttgtcc 540
gctttcatca ctgctcaggc tggtggtgag ttcagagtta ctcacggacg tgatccagtt 600
ccacgtttgc cacctatcgt cttcggttac agacacacct ctccagagta ctggttggct 660
ggtggtgctt ccaccaagac tgattatact gttaacgata tcaaggtttg tgaaggtgcc 720
gctaacttgg cctgtaatgg tggtactttg ggattggata tcattgctca tttgagatac 780
ttccaagaca ctgacgcctg tactgctggt ggtatctcct ggaagagagg tgacaaagct 840
aagagagatg agattccaaa aagacaagaa ggaatgactg atgaggagtt ggaacaaaaa 900
ctgaacgact atgtcgccat ggataaggag tacgttgagt ccaacaagat gtaa 954
<210> 23
<211> 317
<212> PRT
<213> 腐皮镰刀菌(Fusarium solani)
<400> 23
Ala Ile Thr Ala Ser Gln Leu Asp Tyr Glu Asn Phe Lys Phe Tyr Ile
1 5 10 15
Gln His Gly Ala Ala Ala Tyr Cys Asn Ser Glu Thr Ala Ser Gly Gln
20 25 30
Lys Ile Thr Cys Ser Asp Asn Gly Cys Lys Gly Val Glu Ala Asn Asn
35 40 45
Ala Ile Ile Val Ala Ser Phe Val Gly Lys Gly Thr Gly Ile Gly Gly
50 55 60
Tyr Val Ser Thr Asp Asn Val Arg Lys Glu Ile Val Leu Ser Ile Arg
65 70 75 80
Gly Ser Ser Asn Ile Arg Asn Trp Leu Thr Asn Val Asp Phe Gly Gln
85 90 95
Ser Ser Cys Ser Tyr Val Arg Asp Cys Gly Val His Thr Gly Phe Arg
100 105 110
Asn Ala Trp Asp Glu Ile Ala Gln Arg Ala Arg Asp Ala Val Ala Lys
115 120 125
Ala Arg Thr Met Asn Pro Ser Tyr Lys Val Ile Ala Thr Gly His Ser
130 135 140
Leu Gly Gly Ala Val Ala Thr Leu Gly Ala Ala Asp Leu Arg Ser Lys
145 150 155 160
Gly Thr Ala Val Asp Ile Phe Thr Phe Gly Ala Pro Arg Val Gly Asn
165 170 175
Ala Glu Leu Ser Ala Phe Ile Thr Ala Gln Ala Gly Gly Glu Phe Arg
180 185 190
Val Thr His Gly Arg Asp Pro Val Pro Arg Leu Pro Pro Ile Val Phe
195 200 205
Gly Tyr Arg His Thr Ser Pro Glu Tyr Trp Leu Ala Gly Gly Ala Ser
210 215 220
Thr Lys Thr Asp Tyr Thr Val Asn Asp Ile Lys Val Cys Glu Gly Ala
225 230 235 240
Ala Asn Leu Ala Cys Asn Gly Gly Thr Leu Gly Leu Asp Ile Ile Ala
245 250 255
His Leu Arg Tyr Phe Gln Asp Thr Asp Ala Cys Thr Ala Gly Gly Ile
260 265 270
Ser Trp Lys Arg Gly Asp Lys Ala Lys Arg Asp Glu Ile Pro Lys Arg
275 280 285
Gln Glu Gly Met Thr Asp Glu Glu Leu Glu Gln Lys Leu Asn Asp Tyr
290 295 300
Val Ala Met Asp Lys Glu Tyr Val Glu Ser Asn Lys Met
305 310 315
- 用于酵母的前导序列
- 用于识别前导序列以及用于估计整数载频偏移的方法和装置
