一种核酸文库的制备方法及测序方法
文献发布时间:2023-06-19 09:51:02
技术领域
本发明涉及分子生物学领域。具体而言,本发明涉及核酸文库的制备方法及测序方法。
背景技术
序列捕获技术是针对已知的特定基因组区域设计探针进行有选择性的分离或者富集基因组的特定片段。常用的序列捕获的方法有探针捕获、PCR捕获。利用探针捕获的方法进行序列捕获通常是利用探针与目标序列互补的特性,捕获目标序列,所捕获的目标序列也是单链序列。
二代测序和单分子测序在文库构建方面,常常涉及DNA片段与特定接头的接头连接形成测序文库。接头的种类直接影响连接效率,同时影响测序文库中有效文库的比率。不同的测序平台需要不同的测序接头,各平台都需要优化建库方法和所用的接头,如Illumina建库中,Y接头和Bubble接头具有不同的优势。测序领域中,上机前的样本处理包括核酸处理、文库构建等还待进一步优化,以提高建库效率、简化操作流程。
发明内容
本发明提供一种核酸文库的制备方法,利用此方法制备的核酸文库可提高探针捕获核酸文库的效率,进一步地,对该文库进行测序,能够获得高比例的有效数据。
本发明第一方面公开了一种捕获核酸分子的方法,所述核酸分子包括互补的第一核酸链和第二核酸链,所述核酸分子具有至少一个单链末端,至少一个所述单链末端位于所述第一核酸链上,该方法包括利用探针捕获所述核酸分子,使所述探针和所述第一核酸链上的至少一部分互补结合,以获得杂交复合体,所述第一核酸链上的至少一部分包含所述单链末端。
可以理解的是,所述的核酸分子的第一核酸链和第二核酸链为不同的两条链,也可以为一条链,当核酸分子为一条链时第一核酸链和第二核酸链在双链区的一端连接在一起,形成的一端为单链区的核酸分子。
单链末端可以位于第一核酸链的3’端和5’端,单链区的主要作用是与探针互补配对。任选地,上述单链末端位于第一核酸链的3’端。基于核酸互补配对的原理可知,当探针与第一核酸链配对时,单链末端位于第一核酸链的3’端时探针可以作为引物,第一核酸链可以作为模板进行PCR扩增。
同时,可以理解的是,与第一核酸链配对的序列探针含有寡核苷酸链,当寡核苷酸链位于探针的一端时,探针的另一端可以为核苷酸序列也可为其它序列,如氨基酸序列,另一端的序列同时可以有其它修饰,如氨基修饰,荧光修饰等。
探针中的寡核苷酸链的选择会综合考虑退火温度、核苷酸组成、长度及寡核苷酸链的内部二级结构等因素,在本申请中一个实施例中,寡核苷酸链的长度为20~80nt。
进一步地,上述的寡核苷酸链3’末端的核苷酸含有羟基(-OH),这样寡核苷酸链的3’端才能与其它核苷酸通过磷酸二酯键进行连接。
在一个实施例中,上述的探针固定在固体介质上,所述固体介质选自玻璃、塑料和磁珠中的至少一种。将探针固定在固体介质上,一方面,方便探针捕获核酸序列后的分离纯化核酸序列;另一方面,捕获核酸序列后可以进行其它检测或操作,如PCR扩增、测序反应等。
在一个实施例中,上述核酸分子通过连接第一核酸片段和第二核酸片段而获得,所述第一核酸片段和所述第二核酸片段均为双链DNA,所述第一核酸片段为已知序列,所述第一核酸片段包括互补的第一链和第二链,所述单链末端位于所述第一链上。
核酸片段的连接方式可以选择平末端连接和/或粘性末端连接。对于两端时平末端的核酸片段,可以通过末端加T/A后再进行连接,这样可以提高连接效率及保证连接的方向性。
可以理解,核酸片段在通过化学键进行连接时,可以采用磷酸二酯键但不局限于磷酸二酯键,当采用磷酸二酯键将两者连接时,所述第一链的5'端具有磷酸基团。天然核苷酸含有磷酸基团及羟基基团,采用磷酸二酯键进行化学连接,连接产物不会破坏核苷酸链的自然结构特征。
在一个实施例中,上述第二链的3’端无羟基,从而第二链的3’不能通过磷酸二酯键与第二核酸片段连接。采用此方案时,连接产物会有两种形式,一种形式是第二链通过非磷酸二酯键与第二核酸片段连接;另一种形式是连接产物中不含有第二链,这两种形式的连接产物在连接产物的两条链作为PCR底物时,第二链都不能被有效扩增,若连接产物作为测序底物,可减少无效数据的检测,提高有效检测数据量。
使用的第一核酸片段中两条互补的链,经过筛选,两者的温度差至少要为10℃,不高于80℃,即80℃≥Tm1-Tm2≥10℃并且90℃≥Tm1≥50℃,Tm1为所述第一链的溶解温度,Tm2为所述第二链的溶解温度。选择合适的溶解温度差,从而保证在变性退火后,第一链优先与探针结合。探针可与第一链的所有序列或者部分序列互补配对,优选的一个方案为第一链的所有序列与探针互补配对。可以理解,第一链与探针互补配对是为了保证在选择的退火温度下,第一链优先和探针杂交,因此,在满足上述要求下,第一链的所有序列与探针的配对序列中允许存在错配序列。
本申请的一个实施例中,上述Tm1为71℃,Tm2=45.6℃。在两条链的溶解温度确定后,选择与探针杂交的退火温度T,Tm2-5℃≤T≤Tm1-5℃,本申请的一个实施例中,选择的退火温度为55℃。
在本申请的一种实现方式中,本申请的捕获核酸分子的方法具体包括以下步骤:连接第一核酸片段和第二核酸片段获得核酸分子;变性处理,使核酸分子解链为单链;快速降温,将含有解链后的核酸分子溶液快速置于冰上;杂交捕获;其中第一核酸片段和所述第二核酸片段均为双链DNA,所述第一核酸片段为已知序列,所述第一核酸片段包括互补的第一链和第二链,所述单链末端位于所述第一链上,第一核酸链和第二核酸链,所述核酸分子具有至少一个单链末端,至少一个所述单链末端位于所述第一核酸链上,其中第一链的5'端具有磷酸基团,第二链的3’端无羟基,且第一链的所有序列与捕获所用的探针互补配对。通过该方法捕获核酸分子可以大大提高捕获效率,排除第二链对捕获的影响。
当核酸分子与探针互补结合时,需要直观的判断两者的结合情况,尤其是结合效率、结合位置,从而判断该方法是否成功。当探针固定时,判断固定的核酸分子的数量即可判断结合效率,为此,优选的一个方案是在上述第一链的3’端带有光学可检测标记,优先选择荧光分子如Cy3,Cy5等,从而可简单直观的检测杂交效率。
为了捕获单链核酸分子,在一个示例中,将双链核酸分子解链为单链核酸分子,例如通过变性,变性后探针与单链核酸分子杂交形成杂交复合体。变性后的核酸分子在杂交溶液中,与探针互补结合。
通过上述捕获方法,一方面可以捕获双链核酸分子,另一方面可以捕获单链核酸分子。通过此方法捕获双链核酸分子,一方面可以将目标双链分子进行纯化分离、检测,另一发面捕获双链核酸分子后形成的杂交复合体在链置换反应聚合酶存在的条件下,合成目的序列,如合成核酸片段的探针序列。通过此方法捕获单链核酸分子后形成的杂交复合体可用于序列测序,从而确定核酸片段的具体序列信息。
上述捕获核酸分子的方法,巧妙了利用了双链核酸分子具有单链区的特点,可以有效捕获双链核酸序列,同时利用此方法也可以捕获单链核苷酸。利用单链区和双链区的Tm的关系,设计合适的杂交捕获温度,减少非特异性杂交的同时,同时降低第二链与第一链杂交的几率,提高探针与第一链的杂交效率。
本发明第二方面提供了一种杂交复合体,所述的杂交复合体通过上述的捕获方法获得。杂交复合体有两种类型,第一种类型是通过探针与未变性的核酸分子即双链核酸分子杂交形成,探针与单链末端配对形成杂交复合体,此时杂交复合体含有三条序列即核酸分子的两条序列及探针序列;第二种类型是通过探针与变性成单链的核酸分子杂交形成,与探针配对杂交的序列至少含有单链末端序列,此时杂交复合体含有两条序列即一条单链核酸分子序列及探针序列。
本发明第三方面提供了一种试剂盒,包括上面所述的核酸分子。进一步的,试剂盒还包含探针和/或杂交复合体。试剂盒中的核酸分子包括互补的第一核酸链和第二核酸链,且具有至少一个单链末端位于所述第一核酸链上,试剂盒中的探针和第一核酸链单链末端序列互补。此试剂盒可用序列杂交捕获实验、探针序列杂交捕获实验的对照实验以及测序中。
本发明第四方面提供了上述试剂盒在在序列杂交捕获和/或测序中的用途。利用此试剂盒进行序列杂交捕获和/或测序中时,可以作为对照组实验对序列杂交捕获进行质控。
本发明第五方面提供了一种测序方法,包括对杂交复合体进行序列测定,其中杂交复合体利用上述方法获得。进行测序时,当杂交复合体为三条序列的复合体时,测序采用链置换DNA聚合酶进行测序,如Phi29DNA聚合酶;当杂交复合体为两条序列的复合体时,测序采用测序常用DNA聚合酶即可,如taq DNA聚合酶。
本发明第六方面提供了一种核酸文库的制备方法,包含连接第一核酸片段和第二核酸片段,以获得所述核酸文库,所述第一核酸片段和所述第二核酸片段均为双链DNA,所述第一核酸片段为已知序列,所述第一核酸片段包括互补的第一链和第二链,所述第一核酸片段具有单链末端,所述单链末端位于所述第一链的3'端,并且,80℃≥(Tm1-Tm2)≥10℃,90℃≥Tm1≥50℃,Tm1为所述第一链的溶解温度,Tm2为所述第二链的溶解温度为Tm2。
本申请的一个实施例中,上述Tm1为71℃,Tm2为45.6℃。
可以理解,上述核酸片段在通过化学键进行连接时,可以采用磷酸二酯键但不局限于磷酸二酯键,当采用磷酸二酯键将两者连接时,所述第一链的5'端具有磷酸基团。天然核苷酸含有磷酸基团及羟基基团,采用磷酸二酯键进行化学连接,连接产物不会破坏核苷酸链的自然结构特征。
在一个实施例中,上述第二链的3’端无羟基,利用具有此特征的第二链构建核酸文库,核酸文库中的连接产物有两种形式,一种形式为:第二链通过非磷酸二酯键与第二核酸片段连接;另外一种形式为:连接产物中不含有第二链,这两种形式的连接产物在连接产物的两条链作为PCR底物时,第二链都不能被有效扩增,若连接产物作为测序底物,可减少无效数据的检测,提高有效检测数据量。
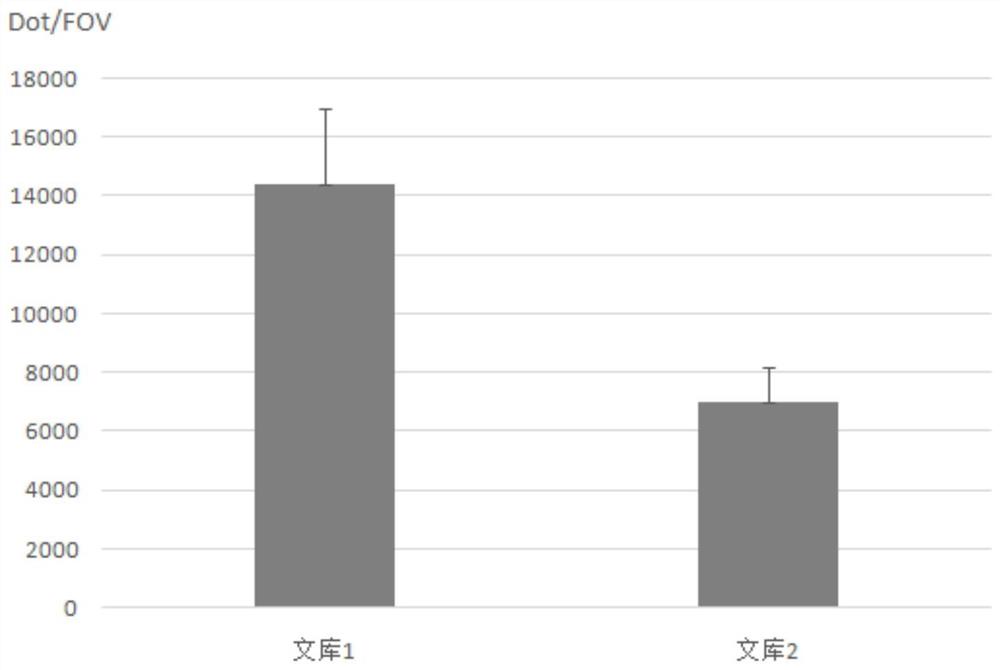
上述核酸文库的制备方法制备的核酸文库用于测序时,尤其用于单分子测序时,可提高与探针的杂交效率,从而增加有效测序的目标片段。在本发明的实施例中,利用本发明的第一核酸片段与平末端接头分别构建核酸文库用于探针捕获杂交,对捕获杂交效率进行了对比,用探针捕获核酸文库片段后检测荧光信号判断捕获的核酸文库的多少,从检测的结果可看出,利用本发明文库构建方法获得的核酸文库与探针的捕获效率是平末端接头构建文库捕获效率的2倍多。
构建好的文库有多种用途,如芯片序列杂交、测序等。当核酸文库与探针互补结合时,需要直观的判断两者的结合情况,尤其是结合效率、结合位置,从而判断操作是否成功。当探针固定时,判断固定的核酸分子的数量即可判断结合效率、结合位置,为此,优选的一个方案为:在上述第一链的3’端带有光学可检测标记,优先选择荧光分子如Cy3,Cy5等,从而可简单直观的检测杂交效率。
核酸文库制备方法中核酸片段的连接方式可以选择平末端连接和/或粘性末端连接。对于两端时平末端的核酸片段,可以通过末端加T/A后再进行连接,这样可以提高连接效率及保证连接的方向性。
两种核酸片段进行连接时,已知序列的核酸片段即第一核酸片段的摩尔量要多于第二核酸片段,从而提高第二核酸片段与第一核酸片段进行连接的概率,基于多次实验中两种核酸片段摩尔浓度的比列优化结果可知,当第一核酸片段和所述第二核酸片段的混合比例为摩尔比50~500:1范围时,第一核酸片段和所述第二核酸片段能进行有效的连接,且当混合比例为摩尔比200:1时,第二核酸片段90%以上都连接上了第一核酸片段,且第二核酸片段自连率(第二核酸片段之间进行连接)大大降低。
在本申请的一种实现方式中,本申请的制备方法具体包括以下步骤:包含连接第一核酸片段和第二核酸片段,以获得所述核酸文库,所述第一核酸片段和所述第二核酸片段均为双链DNA,所述第一核酸片段为已知序列,所述第一核酸片段包括互补的第一链和第二链,所述第一核酸片段具有单链末端,所述单链末端位于所述第一链的3'端,并且,80℃≥(Tm1-Tm2)≥10℃,90℃≥Tm1≥50℃,Tm1为所述第一链的溶解温度,Tm2为所述第二链的溶解温度为Tm2。其中,第二链的3’端无羟基,第一链的5’端含有磷酸基团,第一链可与第二核酸片段通过磷酸二酯键连接。利用该方法构建核酸文库,连接产物中不含有第二链,或者第二链通过非磷酸二酯键与第二核酸片段连接。这两种形式的连接产物在连接产物的两条链作为PCR底物时,第二链都不能被有效扩增,若连接产物作为测序底物,可减少无效数据的检测,提高有效检测数据量。
本发明第七方面提供了一种核酸文库,核酸文库为利用上述的制备方法获得的连接产物,其中连接产物是本发明第三方面提供的核酸片段与第二核酸片段直接连接的产物或是直接连接的产物经过n轮(1≤n≤6,且n为整数)PCR扩增后的产物。
本发明第八方面提供了一种捕获核酸分子的方法,包含如下步骤:
1)将上述的核酸文库变性形成单链;
2)将步骤1)的单链产物与探针杂交,形成杂交复合体。
其中,探针含有寡核苷酸链。探针一端的寡核苷酸链的作用为与核酸分子中的序列互补杂交,可以理解的是,探针的另一端可以为核苷酸序列也可为其它序列,如氨基酸序列,另一端的序列同时可以有其它修饰,如氨基修饰,荧光修饰等。在本发明中的一个实施例中,探针的序相同,且3’末端的核苷酸含有羟基(-OH)。基于核酸互补配对的原理可知,当探针与第一核酸链配对时,单链末端位于第一核酸链的3’端时探针可以作为引物,第一核酸链可以作为模板进行PCR扩增。任选地,寡核苷酸链为DNA链。
探针可与第一链的所有序列或者部分序列互补配对,优选的一个方案为第一链的所有序列与探针互补配对。可以理解,第一链与探针互补配对是为了保证在选择的退火温度下,第一链优先和探针杂交,因此,在满足上述要求下,第一链所有序列与探针的配对序列中允许存在错配序列。
为了有效的捕获含有第一链的核酸序列,选择的杂交温度为T,Tm2-5℃ 同时,可以理解的是,与第一核酸链配对的序列探针含有寡核苷酸链,当寡核苷酸链位于探针的一端时,探针的另一端可以为核苷酸序列也可为其它序列,如氨基酸序列,另一端的序列同时可以有其它修饰,如氨基修饰,荧光修饰等。 探针中的寡核苷酸链的选择会综合考虑退火温度、核苷酸组成、长度及寡核苷酸链的内部二级结构等因素,在本申请中一个实施例中,寡核苷酸链的长度为20~80nt。 进一步地,上述的寡核苷酸链3’末端的核苷酸含有羟基(-OH),这样寡核苷酸链的3’端才能与其它核苷酸通过磷酸二酯键进行连接。 在一个实施例中,上述的探针固定在固体介质上,所述固体介质选自玻璃、塑料磁珠中的至少一种。将探针固定在固体介质上,一方面,方便探针捕获核酸序列后的分离纯化核酸序列;另一方面,捕获核酸序列后可以进行其它检测或操作,如PCR扩增、测序反应等。 本发明第九方面提供了一种测序方法,包括对杂交复合体进行序列测定,其中杂交复合体利用上述方法获得。进行测序时,当杂交复合体为三条序列的复合体时,测序采用链置换DNA聚合酶进行测序,如Phi29DNA聚合酶;当杂交复合体为两条序列的复合体时,测序采用测序常用DNA聚合酶即可,如taq DNA聚合酶。 本发明第十方面提供了一种用于制备核酸文库、捕获和/或测序的试剂盒,该试剂盒包含第一核酸片段,在一个示例中,第一核酸片段为接头。进一步,该试剂盒还包含探针,探针能够捕获上述第一核酸片段,所述探针的3’末端的核苷酸含有-OH。基于核酸互补配对的原理可知,当探针与第一核酸链配对时,单链末端位于第一核酸链的3’端时探针可以作为引物,第一核酸链可以作为模板进行PCR扩增。 可以理解的是,探针一端的寡核苷酸链的作用为与核酸分子中的序列互补杂交,探针的另一端可以为核苷酸序列也可为其它序列,如氨基酸序列,另一端的序列同时可以有其它修饰,如氨基修饰,荧光修饰等。在一个示例中,寡核苷酸链为DNA链。 探针中的寡核苷酸链的选择会综合考虑退火温度、核苷酸组成、长度及寡核苷酸链的内部二级结构等因素,在本申请中一个实施例中,寡核苷酸链的长度为20~80nt。 在一个实施例中,所述探针固定在固体介质上,所述固体介质选自玻璃、塑料和磁珠中的至少一种。 附图说明 图1示意本发明一个实施例中的文库1和文库2与探针的杂交结果。横坐标为文库1和文库2,纵坐标为特定视野范围(FOV,例如110×110μm)内的亮点(dot)数目,表示为Dot/FOV。术语解释: 本文中所称的“核酸”、“核酸分子”和“核酸片段”,包括DNA、RNA、RNA-DNA和/或者cDNA,双链或者单链,是由核苷酸和/或核苷酸类似物(衍生物)组成的序列。 本文中所称的“接头”为序列已知的核酸片段。 实施例 下面参考具体实施例和附图,对本发明进行说明。需要说明的是,这些实施例仅仅是说明性的,而不能理解为对本发明的限制。实施例中未注明具体技术或条件的,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市场购买获得的常规产品。 1.接头制备 设计并合成表1所示的核苷酸序列。 表1
注:5'-PHO:5’端磷酸化修饰;3'Cy3:3’端Cy3修饰;5'-NH 利用在线软件(http://biotools.nubic.northwestern.edu/OligoCalc.html)计算每条链的溶解温度Tm,SEQ ID NO:1:溶解温度为45.6℃;SEQ ID NO:2:溶解温度为71℃。 接头制备流程:将合成的表1所示的核苷酸序列分别用1×Annealing Buffer(10mMTris pH7.5-8.0,50mMNaCl,1mM EDTA)溶解至100pmol/μl,然后使用Nanodrop 2000对其定量。根据定量结果,用1×Annealing Buffer分别将SEQ ID NO:1-4所示的核苷酸序列稀释到100pmol/μl。 分别取10μl,100pmol/μl的oligo1.1、oligo1.2于洁净的200μl PCR管中,吸打混匀,用于制备接头1。同样,分别取10μl,100pmol/μl的10μl oligo2.1、10μl oligo2.2于另一PCR管中混匀,用于制备接头2。将混合后的序列至于PCR仪中进行缓慢退火制备接头1和接头2,接头制备设置条件如表2所示: 表2
将制备好的接头1和2用Qubit 2.0定量仪、Qubit 2.文库构建 使用诺唯赞的试剂盒(VAHTS Universal DNA library prep kit for IlluminaVazyme cat,货号ND606)进行DNA片段修复并加接头,其中DNA片段和接头的摩尔量比值为1:200,即DNA:接头=1:200。利用接头1构建文库1,利用接头2构建文库2。文库1和文库2的建库条件只有使用的接头不同,其它建库条件都相同。具体操作步骤如下: 步骤一:末端修复 将长度为100-500bp(主带为200bp)片段化的DNA末端补平,并在5’端进行磷酸化和3’端加dA尾。 在灭菌PCR管中配置末端补平反应体系,具体组成如表3所示: 表3
将末端补平反应体系混匀后,并短暂离心将反应液收集至管底。 末端修复反应条件如表4所示: 表4
反应结束后获得末端修复产物。 步骤二:接头连接 将步骤一中末端修复后的产物与接头连接。 (1)在步骤一中末端修复的PCR管中配置接头连接反应体系,接头连接反应体系如表5所示: 表5
使用移液器轻轻吹打混匀,并短暂离心将反应液收集至PCR管底。 (2)将(1)中配置好的接头连接反应体系PCR管置于PCR仪中进行反应,接头连接反应条件按照表6所示条件进行设置: 表6
反应结束后获得接头连接产物。 步骤三:纯化 使用VAHTS DNA Clean Beads对步骤2)中接头连接产物进行纯化,纯化步骤如下: a)磁珠平衡至室温后,涡旋振荡混匀VAHTS DNA Clean Beads。 b)吸取60μl VAHTS DNA Clean Beads至100μl接头连接产物中,涡旋振荡或使用移液器轻轻吹打10次充分混匀。 c)室温孵育5min。 d)将PCR管短暂离心并置于磁力架中分离磁珠和液体,待溶液澄清后(约5min),小心移除上清。 e)保持PCR管始终置于磁力架中,加入200μl新鲜配制的80%乙醇漂洗磁珠,室温孵育30sec,小心移除上清。 f)重复步骤5,总计漂洗两次。 g)保持PCR管始终置于磁力架中,开盖空气干燥磁珠5-10min至无乙醇残留。 h)将PCR管从磁力架中取出,进行洗脱: 加入22.5μl洗脱液(10mMTris-HCl,pH 8.0-pH 8.5)洗脱,涡旋振荡或使用移液器轻轻吹打充分混匀,于室温放置2min,将PCR管短暂离心并置于磁力架中静置,待溶液澄清后(约5min),小心移取20μl上清至新EP管中,切勿触碰磁珠。 使用Qubit 2.0定量仪、QubitTM dsDNA BR Assay Kit对纯化后的文库定量,文库1连接的接头为接头1,文库2连接的接头为接头2。 3.杂交捕获 利用公开专利申请CN201510501968.7说明书中公开的方法在芯片上固定探针(SEQ IDNO:4),制备的文库1和文库2用3*SSC杂交液稀释,然后与芯片上固定的探针进行杂交。然后根据Cy3的信号来判断接头序列与探针杂交的数目。 文库芯片杂交的流程如下: (1)芯片选择:所用的芯片的基地玻璃为SCHOTT公司的环氧基修饰的玻璃芯片,通过探针上的氨基和芯片表面的环氧基团反应的方法,例如,可参看公开专利申请号CN201811191589.2披露的方法来固定探针(序列:TTTTTTTTTTTCCTTGATACCTGCGACCATCCAGTTCCACTCAGATGTGTATAAGAGACAG),在110×110μM区域内固定的探针密度约为18000Dot/FOV即在110×110μM视野内有18000个亮点。 (2)杂交液配置:杂交液配置体系如表7所示,所用缓冲液为20×SSC缓冲液(西格玛,#S6639-1L),终浓度为成3×SSC,文库的终浓度为1nM,总体积为40μL。将配置好的杂交液95℃变性2min,迅速至冰上冷却。 表7
(3)将上述变性后的杂交液迅速加载至芯片上,再将芯片放置55℃30min,使文库与芯片表面的探针进行杂交。 (4)依次用3×SSC、1×SSC、0.1×SSC冲洗芯片。 (5)然后将芯片放置测序仪上,在532波长下拍照,统计每个视野下的点数。 4.实验结果 实验结果见图1,横坐标为文库1和文库2,纵坐标为Dot/FOV(观测区域为110×110μm范围的亮点数)。文库1和文库2杂交结果相比,二者在同等杂交浓度和同等的视野(在110×110μM区域内)下,文库1的杂交密度(平均14400亮点数)远高于文库2(平均7000亮点数),从文库与探针杂交密度的差异可知,文库1与探针杂交的效率比文库2探针杂交的效率高。 在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。 尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。 序列表 <110> 深圳市真迈生物科技有限公司 <120> 一种核酸文库的制备方法及测序方法 <130> PI2019003 <160> 4 <170> SIPOSequenceListing 1.0 <210> 1 <211> 20 <212> DNA <213> Artificial Sequence <220> <221> misc_feature <222> (1)..(20) <223> 人工序列 <400> 1 agatgtgtat aagagacagt 20 <210> 2 <211> 51 <212> DNA <213> Artificial Sequence <220> <221> misc_feature <222> (1)..(51) <223> 人工序列 <400> 2 ctgtctctta tacacatctg agtggaactg gatggtcgca ggtatcaagg a 51 <210> 3 <211> 52 <212> DNA <213> Artificial Sequence <220> <221> misc_feature <222> (1)..(52) <223> 人工序列 <400> 3 tccttgatac ctgcgaccat ccagttccac tcagatgtgt ataagagaca gt 52 <210> 4 <211> 61 <212> DNA <213> Artificial Sequence <220> <221> misc_feature <222> (1)..(61) <223> 人工序列 <400> 4 tttttttttt tccttgatac ctgcgaccat ccagttccac tcagatgtgt ataagagaca 60 g 61
