鉴定靶蛋白染色质结合图谱的快速建库方法
文献发布时间:2023-06-19 10:25:58
技术领域
本发明涉及一种鉴定靶蛋白染色质结合图谱的快速建库方法,属于生物技术领域。
背景技术
染色质免疫共沉淀技术(chromatin immunoprecipitation assay, ChIP)是研究基因表达调控、DNA损伤修复和表观遗传修饰的关键手段,也是人们探索生命进程和细胞病变原因的重要工具。随着高通量测序技术的兴起和发展,染色质免疫共沉淀测序技术(ChIP-seq)成为人们研究DNA结合蛋白在染色质上的结合图谱的高效工具。截至到目前位置,人类DNA元件百科全书计划(encyclopedia of dna elements,ENCODE)和表观基因组学路线图计划(Roadmap Epigenomics Project)已经收纳了超过200种DNA结合蛋白的染色质结合图谱,包括表观遗传修饰及其调控蛋白、转录调控因子、染色质结构调控蛋白等。近年来,越来越多的疾病如白血病、实体瘤和神经退行性疾病等被报道出与DNA结合蛋白的染色质结合图谱改变息息相关,众多靶向药也进入了临床试验阶段。
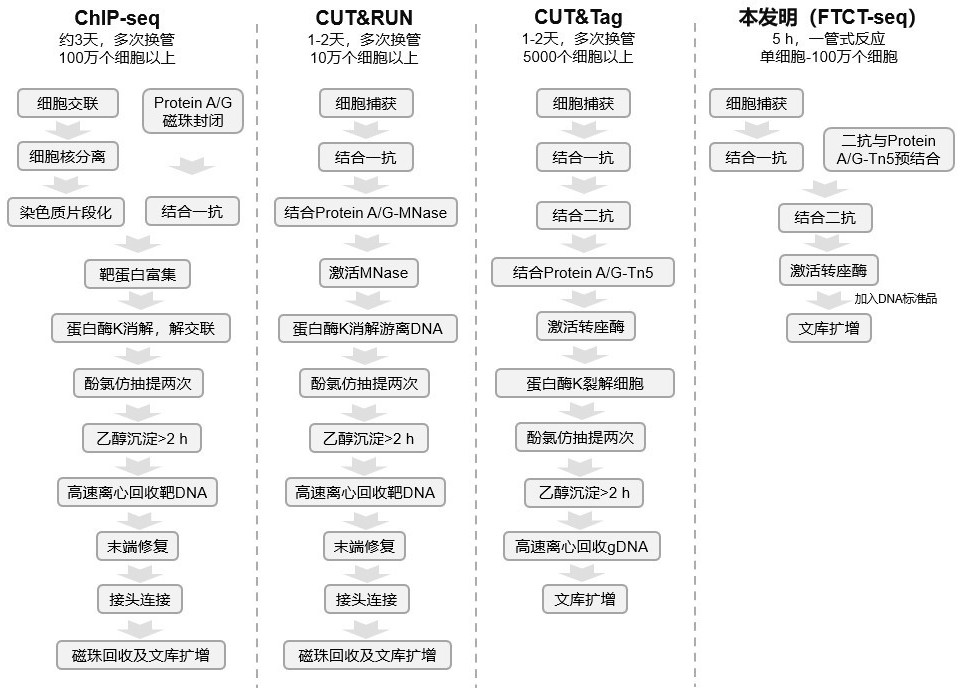
现有的染色质免疫共沉淀测序技术主要分为三种。一是传统的ChIP-seq,由抗体的靶向富集DNA进行建库测序,该方法具有耗时长(需要3天)、对起始材料要求量大(500-2000万细胞)、成本高、操作复杂、对抗体要求严格等缺陷,一直难以大范围推广。第二种是CUT&RUN技术,该技术利用Protein A/G-MNase和抗体识别靶蛋白,将靶蛋白邻近区域的染色质切断游离出来进行回收建库。这种方法背景较重,且灵敏度较低、操作繁琐。另一种方法是中国专利文献CN109400714A-抗体靶向的重组融合蛋白以及其在表观遗传学中的应用公开的CUT&Tag技术,该技术利用Protein A/G-Tn5转座酶复合物,实现在细胞内原位对靶蛋白结合DNA序列的快速标记和片段化,进而利用基因组DNA进行靶向文库扩增。该方法具有耗时短(约1-2天)、操作简单、对细胞起始量要求较低(100-100万个细胞)、成本低等优势,成为代替传统ChIP-seq的有效手段。但这种技术仍然存在操作时间过长、操作过程损失大等缺陷,限制了该技术在珍贵样本和单细胞上的发展和应用。
发明内容
本发明的目的是提供一种适用于低细胞起始量的鉴定靶蛋白染色质结合图谱的可定量快速建库方法,显著简化了传统方法的流程,降低了此类技术对样本的要求。同时,通过将三条不同浓度的DNA标准品掺入到建库过程中,可以达到对文库中DNA拷贝数精确定量的效果。
本发明采用的技术方案为:
一种适用于低细胞起始量的鉴定靶蛋白染色质结合图谱的可定量快速建库方法,其特征在于其步骤包括:
(1)用离心的方式收集样本细胞,将细胞重悬于缓冲液中;
(2)将活化的磁珠加入细胞中孵育,分离收集磁珠;
(3)步骤(2)获得的磁珠与含细胞穿孔剂的一抗进行孵育,分离收集磁珠;
(4)二抗与重组融合转座酶预结合,步骤(3)获得的磁珠与预结合的二抗和重组融合转座酶进行孵育,分离收集磁珠;
(5)步骤(4)获得的磁珠中加入转座酶激活剂,孵育一段时间;
(6)终止转座酶反应;
(7)文库扩增。
优选的,所述磁珠为伴刀豆蛋白A磁珠或抗体偶联免疫磁珠。伴刀豆蛋白A磁珠可以和细胞表面的糖蛋白结合,抗体偶联免疫磁珠可以和细胞表面的特异性蛋白抗原结合,从而快速从溶液中高效分离细胞,同时保证细胞处于正常生理状态,避免离心等应力操作对细胞生理的影响,保证细胞环境的真实性。
优选的,所述细胞穿孔剂为非离子型表面活性剂。非离子型表面活性剂的作用是对细胞膜进行穿孔,同时保证细胞核的完整性以及抗体和转座酶复合物可以进入细胞。穿孔的细胞也可以释放细胞基质降低背景。
优选的,所述细胞穿孔剂为聚乙二醇辛基苯基醚、乙基苯基聚乙二醇、毛地黄皂苷或吐温,处理细胞的浓度为0.005-0.5%。
优选的,所述重组融合转座酶为Protein A-Tn5、Protein G-Tn5或Protein AG-Tn5。
优选的,所述转座酶激活剂为二价金属离子。
优选的,所述转座酶激活剂为镁离子、锰离子、锌离子、钙离子或钴离子,或者上述离子的混合物。
优选的,在步骤(6)中同时掺入DNA标准品。标准品的加入可以提高定量的准确性,传统的CUT&Tag没有DNA标准品,检测不出升高和降低的趋势和程度,会出现很多假阴性和假阳性的情况。本专利中加入标准品后就可以对每个样品进行定量分析,可以计算出测序数据变化的真实值。
优选的,DNA标准品可以为lambda DNA、酵母基因组DNA或者果蝇基因组DNA中的一个或者多个片段。
优选的,终止转座酶反应的方法为SDS热失活、蛋白酶K降解或EDTA螯合金属离子。
优选的,步骤(1)中的缓冲液为磷酸盐缓冲液、Tris缓冲液、HEPES缓冲液或PIPES缓冲液,缓冲液pH为7.5-8.5,NaCl浓度为150 mM,缓冲液的条件接近细胞内真实的条件,以减少缓冲液对细胞状态的影响。
优选的,步骤(3)中一抗先采用抗体稀释液来稀释,抗体稀释液包括pH 7.5的10-100 mM HEPES-KOH、100-500 mM NaCl、1-10 mM 亚精胺、1-10 mM EDTA或0.1-1% mM BSA。
本发明的有益效果:
本发明方法命名为FTCT-seq(Fast Targeted Chromatin Tagmentation andSequencing),无需提取回收基因组DNA即可直接进行文库扩增,显著简化了传统方法CUT&Tag(CN109400714A)的流程,缩短了建库时间(从1-2天缩短至5 h),提高了建库效率(可以兼容1-1,000,000细胞投入量),损失小(利用一管式反应简化操作,不需要换管),对建库材料要求更低。同时,通过将三条不同浓度的DNA标准品掺入到建库过程中,可以达到对文库中DNA拷贝数精确定量的效果。相比于传统CUT&Tag,采用二抗和重组融合转座酶预结合的方式,不仅可以使得二抗与重组融合转座酶结合更加充分,而且可以减少转座酶与染色质直接孵育的时间和机会,从而减少转座酶自身随机结合产生的非特异性,提高建库质量。本方法非常适合用于对少量、珍贵样本或者不同处理状态样本的研究,可以广泛应用在表观遗传学、基因表达调控和三维染色质结构等各种生理病理学领域,尤其是在稀有珍贵样本或者需要快速检测样本上的研究。
附图说明
图1本发明FTCT-seq和现有技术的比较。
图2 ENCODE H3K4me3和H3K27me3 ChIP-seq信号在qPCR区域的分布。
图3 Qsep验证Protein A-Tn5 CUT&Tag文库大小分布。
图4 Qsep验证Protein AG-Tn5 CUT&Tag文库大小分布。
图5 qPCR验证Protein A-Tn5 CUT&Tag文库特异性。
图6 qPCR验证Protein AG-Tn5 CUT&Tag文库特异性。
图7 二抗与重组融合转座酶预结合方式构建文库流程图。
图8 Protein A-Tn5在二抗与重组融合转座酶预结合方式与传统CUT&Tag在文库产量上的比较。
图9 qPCR验证Protein A-Tn5在二抗与重组融合转座酶预结合方式构建文库的特异性(Protein A-Tn5)。
图10 qPCR验证Protein A-Tn5在二抗与重组融合转座酶预结合方式构建文库的特异性(Protein AG-Tn5)。
图11 二抗与转座酶预结合与传统CUT&Tag流程特异性对比。
图12 三种不同基因组DNA回收方式建库示意图。
图13 三种基因组DNA回收方法的建库产量。
图14 qPCR验证三种基因组DNA回收方法的建库特异性。
图15 FTCT-seq与CUT&Tag在建库产量上的比较。
图16 qPCR验证Protein A-Tn5使用FTCT-seq构建文库的特异性。
图17 qPCR验证Protein AG-Tn5使用FTCT-seq构建文库的特异性。
图18 FTCT-seq和CUT&Tag流程特异性对比。
图19 Qsep验证Protein A-Tn5 在FTCT-seq文库大小分布。
图20 Qsep验证Protein AG-Tn5 FTCT-seq文库大小分布。
图21 FTCT-seq与CUT&Tag数据在基因组比对率和重复率上的比较。
图22 FTCT-seq与CUT&Tag、ChIP-seq在测序上的相关性(H3K4me3)。
图23 FTCT-seq与CUT&Tag、ChIP-seq在测序上的相关性(H3K27me3)。
图24 FTCT-seq与CUT&Tag在基因组上测序数据展示案例 (H3K4me3)。
图25 FTCT-seq与CUT&Tag在基因组上测序数据展示案例 (H3K27me3)。
图26 qPCR验证不同细胞投入量条件下FTCT-seq建库效果。
图27 不同细胞投入量条件下FTCT-seq文库大小分布。
图28 不同细胞投入量条件下FTCT-seq测序质量。
图29 不同细胞投入量条件下FTCT-seq测序数据相关性。
图30 不同细胞投入量条件下FTCT-seq测序数据展示。
图31 Qsep验证三种DNA标准品的纯度和长度。
图32 Qsep验证DNA标准品混合物的纯度和浓度比例。
图33 H3K27me3促进剂和抑制剂处理后文库产量。
图34 Qsep检测H3K27me3促进剂和抑制剂处理后文库大小分布。
图35不同处理条件下H3K27me3 FTCT-seq富集效果。
图36测量值和理论值之间的拟合定量函数。
图37 三组数据的相关性。
图38 两种不同标准化方式对FTCT-seq信号的定量的准确性(qPCR验证)。
图39 两种不同标准化方式对FTCT-seq信号的定量与qPCR验证之间的相关性。
图40两种不同标准化方式对FTCT-seq信号定量的案例展示。
下面结合附图对本发明的具体实施方式做进一步说明。
具体实施方式
通过以下详细说明结合附图可以进一步理解本发明的特点和优点。所提供的实施例仅是对本发明方法的说明,而不以任何方式限制本发明揭示的其余内容。本实施例所使用的重组融合转座酶分别购买于南京诺唯赞生物科技股份有限公司(Protein A-Tn5,TD901)和对岸蛋白质科技有限公司(Protein AG-Tn5,N259-YH01)。使用的引物序列如表1所示。SEQ NO: 1-16对用于实施例中qPCR检测利用FTCT-seq和CUT&Tag两种方法构建的文库的特异性,其中qGADPH、qRPS18、qRPL15和qKDM2A这四对引物的PCR靶位点是组蛋白H3K4me3的富集区域(根据ENCODE计划中人胚胎干细胞H9的H3K4me3 ChIP-seq数据鉴定,https://www.encodeproject.org/),其中qEVX1、qALOX126、qHOXC13和qHOXD13这四对引物的PCR靶位点是组蛋白H3K27me3的富集区域(根据ENCODE计划中人胚胎干细胞H9的H3K4me3ChIP-seq数据鉴定),这八对引物所在的基因位点和ChIP-seq信号强度见图2。SEQ NO: 17-22用于标准品DNA的制备。
表1:引物序列
对比例1:Protein A-Tn5和Protein AG-Tn5重组融合转座酶结合抗体的能力和特异性测定。
利用CN109400714A公开的CUT&Tag技术流程验证结合抗体的能力和特异性,具体流程如下:
1. 收集细胞。取约100,000个293FT细胞,600x g室温离心5 min收集细胞。用清洗缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM 亚精胺,1x Protease inhibitorscocktail)清洗2次。将细胞重悬于90 μL 清洗缓冲液中。
2. 活化磁珠及捕获细胞。取10 μL ConA磁珠,加入结合缓冲液(20 mM HEPES(pH7.5),10 mM KCl,1 mM CaCl2,1 mM MnCl2)清洗磁珠1次。将磁珠重悬于10μL 结合缓冲液中。将磁珠加入到细胞中,室温旋转孵育5-10 min。
3. 一抗结合。用一抗稀释缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM亚精胺,0.05% Digitonin,2 mM EDTA,0.1% BSA,1x Protease inhibitors cocktail)按照1:100分别稀释三种抗体。分别是Histone H3K4me3 antibody(Active Motif, AB_2687512)、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb (Cell SignalingTechnology, #9733)和Normal Rabbit IgG(Cell Signaling Technology, #2729)。将结合好细胞的磁珠置于磁分离架上,去上清。用加入一抗的缓冲液重悬磁珠,室温旋转孵育2h。
4. 二抗结合。用二抗稀释缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM亚精胺,0.05% Digitonin,1x Protease inhibitors cocktail)按照1:100稀释二抗(Guinea pig anti-rabbit secondary antibody ABIN101961)。将结合好一抗的磁珠置于磁分离架上,去上清。用加入二抗的缓冲液重悬磁珠,室温旋转孵育0.5 h。
5. 转座酶结合。用转座酶结合缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,0.5mM 亚精胺,0.01% Digitonin,1x Protease inhibitors cocktail)按照1:100分别稀释重组融合转座酶Protein A-Tn5和Protein AG-Tn5。将结合好二抗的磁珠置于磁分离架上,去上清。用二抗稀释缓冲液清洗磁珠2次。用加入重组融合转座酶的缓冲液重悬磁珠,室温旋转孵育1.5 h。
6. 转座酶激活。用转座酶结合缓冲液清洗磁珠3次。用300 µL转座酶激活缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,10 mM MgCl2,0.5 mM 亚精胺,0.01% Digitonin,1xProtease inhibitors cocktail)重悬磁珠,37 ºC旋转孵育1 h。
7. 提取基因组DNA。加入10 µL 0.5 M EDTA,3 µL 10%SDS和2.5 µL 20 mg / mL蛋白酶K,终止片段化反应。50 ºC反应1 h。加入等体积DNA提取液(Solarbio,P1012),充分震荡混匀。12000x g离心5 min,取上清。加入等体积DNA提取液,充分震荡混匀。12000x g离心5 min,取上清。加入1/10体积3 M 醋酸钠(pH 5.2),等体积异丙醇,混匀后放置于-80 ºC沉淀,不少于1 h。4 ºC 12000 x g离心15 min,去上清。预冷无水乙醇洗涤沉淀,4 ºC12000 x g离心15 min,去上清。室温晾干15 min。加入22 µL 超纯属溶解gDNA。
8. 文库扩增。使用翊圣生物的Hieff NGS® Fast Tagment DNA Library PrepKit for Illumina试剂盒(Cat#12207)按表2的体系和表3的程序进行文库扩增。
表2 扩增体系
吹打混匀后瞬离。按照以下反应程序进行文库扩增。
表3 扩增程序
加入65 μL Hieff NGS® DNA Selection Beads (Yeasen, 12601),充分吹打混匀,室温孵育5 min。将PCR管置于磁力架中分离磁珠和液体,待溶液澄清后(约3 min),小心移除上清。保持PCR管始终置于磁力架中,加入 200 μL Nuclease free H2O新鲜配制的80%乙醇漂洗磁珠,室温孵育 30 sec 后,小心移除上清。重复漂洗一次。用 10 μL移液器吸干净残留液体。保持 PCR 管始终置于磁力架中,室温下开盖干燥磁珠(5 min)。加入22 μLddH2O,吹打至充分混匀,室温静置5 min。将PCR管短暂离心并置于磁力架中静置,待溶液澄清后(约5 min),小心移取20 μL上清至新PCR管中。用Qubit测定文库浓度, Qsep检测文库大小分布。结果见表4、图3和图4。将文库1:100000稀释,用表1 中的引物进行qPCR定量。定量结果见图5和图6。
表4 利用CUT&TAG方法检测重组融合蛋白的建库产量
由图3和图4可知,重组融合转座酶Protein A-Tn5和Protein AG-Tn5能够有效切割核小体之间的DNA,产生由一个或多个核小体保护的DNA插入片段。相比于IgG阴性对照,阳性抗体的文库产量有显著升高(表4)。由图5和图6的qPCR结果可知,文库显著富集在该一抗靶蛋白结合的染色质区域,这说明重组融合转座酶能够特异性结合抗体。
实施例1:二抗与重组融合转座酶Protein A-Tn5和Protein AG-Tn5预结合对文库产量的影响。
流程示意图见图7,具体流程如下:
1. 收集细胞。取约100,000个293FT细胞,600x g室温离心5 min收集细胞。用清洗缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM 亚精胺,1x Protease inhibitorscocktail)清洗2次。将细胞重悬于90 μL 清洗缓冲液中。
2. 活化磁珠及捕获细胞。取10 μL ConA磁珠,加入结合缓冲液(20 mM HEPES(pH7.5),10 mM KCl,1 mM CaCl2,1 mM MnCl2)清洗磁珠2次。将磁珠重悬于10μL 结合缓冲液中。将磁珠加入到细胞中,室温旋转孵育5-10 min。
3. 一抗结合。用一抗稀释缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM亚精胺,0.05% Digitonin,2 mM EDTA,0.1% BSA,1x Protease inhibitors cocktail)按照1:100分别稀释三种抗体。分别是Histone H3K4me3 antibody(Active Motif, AB_2687512)、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb (Cell SignalingTechnology, #9733)和Normal Rabbit IgG(Cell Signaling Technology, #2729)。将结合好细胞的磁珠置于磁分离架上,去上清。用加入一抗的缓冲液重悬磁珠,室温旋转孵育2h。
4. 二抗与转座酶复合物预结合。在细胞孵育一抗的同时,用转座酶结合缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,0.5 mM 亚精胺,0.01% Digitonin,1x Proteaseinhibitors cocktail)按照1:50分别稀释重组融合蛋白Protein A-Tn5和Protein AG-Tn5(每个样品约1 U),往其中按照1:100加入二抗(Guinea pig anti-rabbit secondaryantibody ABIN101961)。室温旋转孵育1-2 h。
5. 转座酶结合。将结合好一抗的磁珠置于磁分离架上,去上清。用转座酶和二抗预结合的缓冲液重悬磁珠,室温旋转孵育0.5 h。
6. 转座酶激活。用转座酶结合缓冲液清洗磁珠3次。用300 µL转座酶激活缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,10 mM MgCl2,0.5 mM 亚精胺,0.01% Digitonin,1xProtease inhibitors cocktail)重悬磁珠,37 ºC旋转孵育1 h。
7. 提取基因组DNA。加入10 µL 0.5 M EDTA,3 µL 10%SDS和2.5 µL 20 mg / mL蛋白酶K,终止片段化反应。50 ºC反应1 h。加入等体积DNA提取液(Solarbio,P1012),充分震荡混匀。12000x g离心5 min,取上清。加入等体积DNA提取液,充分震荡混匀。12000x g离心5 min,取上清。加入1/10体积3 M 醋酸钠(pH 5.2),等体积异丙醇,混匀后放置于-80 ºC沉淀,不少于1 h。4 ºC 12000 x g离心15 min,去上清。预冷无水乙醇洗涤沉淀,4 ºC12000 x g离心15 min,去上清。室温晾干15 min。加入22 µL 超纯属溶解gDNA。
8. 文库扩增。使用翊圣生物的Hieff NGS® Fast Tagment DNA Library PrepKit for Illumina试剂盒(Cat#12207)按表5的扩增体系和表6的程序进行文库扩增。
表5 扩增反应体系
混匀后加入到含细胞和磁珠的PCR管中,混匀。
表6 扩增反应程序
反应结束后将PCR管短暂离心置于磁分离架上,取95 µL上清,加入120 μL HieffNGS® DNA Selection Beads (Yeasen, 12601),重复上述磁珠回收步骤。用Qubit测定文库浓度,结果见表7和图8。将文库1:100000稀释,用表1 中的引物进行qPCR定量。定量结果见图9和图10。
表7 二抗与转座酶Protein A-Tn5和Protein AG-Tn5预结合方式构建文库的产量
由表7可知,相较于传统的CUT&Tag流程,二抗与重组融合转座酶预结合能够缩短文库构建的时间(约缩短一小时),且在文库产量上有明显的升高(见表7和图8),文库特异性有明显升高(见图9、图10和图11)。
实施例2:不同基因组DNA提取方式对文库产量的影响。
利用重组融合转座酶Protein AG-Tn5和H3K27me3抗体验证不同基因组DNA提取方式对文库产量的影响,流程示意图见图12,具体流程如下:
1. 收集细胞。取约100,000个293FT细胞,600x g室温离心5 min收集细胞。用清洗缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM 亚精胺,1x Protease inhibitorscocktail)清洗2次。将细胞重悬于90 μL 清洗缓冲液中。
2. 活化磁珠及捕获细胞。取10 μL ConA磁珠,加入结合缓冲液(20 mM HEPES(pH7.5),10 mM KCl,1 mM CaCl2,1 mM MnCl2)清洗磁珠2次。将磁珠重悬于10μL 结合缓冲液中。将磁珠加入到细胞中,室温旋转孵育5-10 min。
3. 一抗结合。用一抗稀释缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM亚精胺,0.05% Digitonin,2 mM EDTA,0.1% BSA,1x Protease inhibitors cocktail)按照1:100分别稀释两种抗体。分别是Tri-Methyl-Histone H3 (Lys27) (C36B11) RabbitmAb (Cell Signaling Technology, #9733)和Normal Rabbit IgG(Cell SignalingTechnology, #2729)。将结合好细胞的磁珠置于磁分离架上,去上清。用加入一抗的缓冲液重悬磁珠,室温旋转孵育2 h。
4. 二抗结合。用二抗稀释缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM亚精胺,0.05% Digitonin,1x Protease inhibitors cocktail)按照1:100稀释二抗(Guinea pig anti-rabbit secondary antibody ABIN101961)。将结合好一抗的磁珠置于磁分离架上,去上清。用加入二抗的缓冲液重悬磁珠,室温旋转孵育0.5 h。
5. 转座酶结合。用转座酶结合缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,0.5mM 亚精胺,0.01% Digitonin,1x Protease inhibitors cocktail)按照1:100稀释重组融合转座酶Protein AG-Tn5。将结合好二抗的磁珠置于磁分离架上,去上清。用二抗稀释缓冲液清洗磁珠2次。用加入重组融合转座酶的缓冲液重悬磁珠,室温旋转孵育1.5 h。
6.1-6.3和7.1-7.3是三种不同的回收gDNA的方式及其配套的转座酶激活方式。
6.1抽提法转座酶激活。用转座酶结合缓冲液清洗磁珠3次。用300 μL转座酶激活缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,10 mM MgCl2,0.5 mM 亚精胺,0.01%Digitonin,1x Protease inhibitors cocktail)重悬磁珠,37 ºC旋转孵育1 h。
7.1抽提法回收基因组DNA。加入10 µL 0.5 M EDTA,3 µL 10%SDS和2.5 µL 20mg / mL蛋白酶K,终止片段化反应。50 ºC反应1 h。加入等体积DNA提取液(Solarbio,P1012),充分震荡混匀。12000x g离心5 min,取上清。加入等体积DNA提取液,充分震荡混匀。12000x g离心5 min,取上清。加入1/10体积3 M 醋酸钠(pH 5.2),等体积异丙醇,混匀后放置于-80 ºC沉淀,不少于1 h。4 ºC 12000 x g离心15 min,去上清。预冷无水乙醇洗涤沉淀,4 ºC 12000 x g离心15 min,去上清。室温晾干15 min。加入22 µL 超纯属溶解gDNA。
6.2 磁珠法转座酶激活。用转座酶结合缓冲液清洗磁珠3次。用30 µL转座酶激活缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,10 mM MgCl2,0.5 mM 亚精胺,0.01%Digitonin,1x Protease inhibitors cocktail)重悬磁珠,37 ºC旋转孵育1 h。
7.2 磁珠法回收基因组DNA。加入1 µL 0.5 M EDTA,0.3 µL 10%SDS和0.25 µL20 mg / mL蛋白酶K,终止片段化反应。50 ºC反应1 h。离心后取30 µL上清,加入40 µLHieff NGS® DNA Selection Beads (Yeasen, 12601),重复上述磁珠回收步骤,22 µLddH2O洗脱。
6.3 直接PCR法转座酶激活。用转座酶结合缓冲液清洗磁珠3次。用10 μL转座酶激活缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,10 mM MgCl2,0.5 mM 亚精胺,0.01%Digitonin,1x Protease inhibitors cocktail)重悬磁珠,37 ºC反应1 h。
7.3 直接PCR法回收基因组DNA。加入0.5 μL 0.5% SDS(终浓度0.05%),65 ºC孵育10 min。加入1 μL 10% Triton-X 100(终浓度1%)。
8. 文库扩增。使用翊圣生物的Hieff NGS® Fast Tagment DNA Library PrepKit for Illumina试剂盒(Cat#12207)按表5的扩增体系和表6的程序进行文库扩增。
表8 扩增反应体系
混匀后加入到含细胞和磁珠的PCR管中,混匀。
表9 扩增反应程序
反应结束后将PCR管短暂离心置于磁分离架上,取95 µL上清,加入120 μL HieffNGS® DNA Selection Beads (Yeasen, 12601),重复上述磁珠回收步骤。用Qubit测定文库浓度,结果见表10、图13。将文库1:100000稀释,用表1 中的引物进行qPCR定量。定量结果见图14。
表10 三种不同基因组DNA回收方法构建建库的产量
从表10、图13和图14可以看出,磁珠法和直接PCR法的文库产量要显著高于抽提法(传统CUT&Tag),产量是抽提法的12倍以上。且磁珠法和直接PCR法能够节省大量的时间,提高了建库效率和可操作性,其中磁珠法只需要6 h(一次换管),直接PCR法只需要5 h(无需换管),而抽提法需要1-2天(多次换管)。
实施例3:Protein A-Tn5和Protein AG-Tn5在FTCT-seq中的建库效果。
根据实施例1和实施例2的改进,我们发明了一种鉴定靶蛋白DNA结合图谱的快速建库方法,并命名为FTCT-seq。并利用重组融合转座酶Protein A-Tn5和Protein AG-Tn5验证本发明方法流程的建库效果,具体流程如下:
1. 收集细胞。取约100,000个293FT细胞,600x g室温离心5 min收集细胞。用清洗缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM 亚精胺,1x Protease inhibitorscocktail)清洗2次。将细胞重悬于90 μL 清洗缓冲液中。
2. 活化磁珠及捕获细胞。取10 μL ConA磁珠,加入结合缓冲液(20 mM HEPES(pH7.5),10 mM KCl,1 mM CaCl2,1 mM MnCl2)清洗磁珠2次。将磁珠重悬于10μL 结合缓冲液中。将磁珠加入到细胞中,室温旋转孵育5-10 min。
3. 一抗结合。用一抗稀释缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM亚精胺,0.05% Digitonin,2 mM EDTA,0.1% BSA,1x Protease inhibitors cocktail)按照1:100分别稀释三种抗体。分别是Histone H3K4me3 antibody(Active Motif, AB_2687512)、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb (Cell SignalingTechnology, #9733)和Normal Rabbit IgG(Cell Signaling Technology, #2729)。将结合好细胞的磁珠置于磁分离架上,去上清。用加入一抗的缓冲液重悬磁珠,室温旋转孵育2h。
4. 二抗与转座酶复合物预结合。在细胞孵育一抗的同时,用转座酶结合缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,0.5 mM 亚精胺,0.01% Digitonin,1x Proteaseinhibitors cocktail)按照1:50分别稀释重组融合蛋白Protein A-Tn5和Protein G-Tn5(每个样品约1 U),往其中按照1:100加入二抗(Guinea pig anti-rabbit secondaryantibody ABIN101961)。室温旋转孵育1-2 h。
5. 转座酶结合。将结合好一抗的磁珠置于磁分离架上,去上清。用转座酶和二抗预结合的缓冲液重悬磁珠,室温旋转孵育0.5 h。
6. 转座酶激活。用转座酶结合缓冲液清洗磁珠3次。用10 μL转座酶激活缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,10 mM MgCl2,0.5 mM 亚精胺,0.01% Digitonin,1xProtease inhibitors cocktail)重悬磁珠,37 ºC反应1 h。加入0.5 μL 0.5% SDS(终浓度0.05%),65 ºC孵育10 min。加入1 μL 10% Triton-X 100(终浓度1%)。
7. 文库扩增。使用翊圣生物的Hieff NGS® Fast Tagment DNA Library PrepKit for Illumina试剂盒(Cat#12207)按表11的扩增体系和表12的程序进行文库扩增。
表11 扩增反应体系
混匀后加入到含细胞和磁珠的PCR管中,混匀。
表12 扩增反应程序
反应结束后将PCR管短暂离心置于磁分离架上,取95 µL上清,加入120 μL HieffNGS® DNA Selection Beads (Yeasen, 12601),重复上述磁珠回收步骤。用Qubit测定文库浓度, Qsep检测文库大小分布。结果见表13、图15、图16和图17。将文库1:100000稀释,用表1 中的引物进行qPCR定量。定量结果见图18、19、20和图21。
表13 FTCT-seq构建建库的产量
由表13可知,相较于IgG,H3K4me3抗体和H3K27me3抗体在FTCT-seq流程中显著提高了建库的产量,这于CUT&Tag流程(对比例1)的结果趋势是相符的。而且H3K4me3抗体和H3K27me3抗体在FTCT-seq流程中的文库产量要显著高于CUT&Tag(10倍以上),但是IgG抗体经过两种方法构建的文库产量并没有变化(见图15),这说明这种文库产量升高是抗体靶位点特异性的升高。从qPCR结果也能看出,相较于CUT&Tag,FTCT-seq构建的文库在所使用的抗体靶蛋白DNA结合位点具有更特异性的富集(见图5,图6,图16,图17和图18)。FTCT-seq的文库大小和CUT&Tag呈现相似的分布(不同数量核小体结合DNA的梯度分布,见图3、图4、图19和图20)。这些结果表明FTCT-seq比CUT&Tag具有更高的建库产量,对靶蛋白结合DNA具有更特异性的富集,FTCT-seq能够容纳更低细胞投入量的样本或者细胞丰度更低的DNA结合蛋白。
实施例4:Protein AG-Tn5 FTCT-seq与CUT&Tag测序数据的比较。
将对比例1和实施例3中构建好的文库在Illumina NovaSeq 6000平台上进行测序分析,分析后的结果见表14、图21-图25。
表14:FTCT-seq与CUT&Tag测序数据质量的比较。
由表14和图21可知,相较于CUT&Tag,FTCT-seq构建的文库与人类基因组具有更高的比对率和更低的重复率,这说明经过FTCT-seq流程构建的文库质量更高。FTCT-seq测序数据与CUT&Tag和ChIP-seq具有较好的相关性,说明FTCT-seq构建的文库具有与CUT&Tag和ChIP-seq相似水平的特异性(见图22、图23、图24和图25)。
实施例5:Protein AG-Tn5 FTCT-seq在不同细胞投入量中的建库效果。
按照实施例3的方法,对1-100万细胞投入量的293FT细胞样本进行H3K27me3FTCT-seq建库,文库扩增前取1 μL产物稀释100倍,利用翊圣生物的Hieff UNICON® qPCRSYBR Green Master Mix (Cat#11199)进行定量分析,引物用翊圣生物的Hieff NGS®Fast Tagment DNA Library Prep Kit for Illumina试剂盒(Cat#12207)中的文库扩增引物。qPCR结果见图26。余下的产物进行文库扩增,PCR循环数和文库产量见表15。Qsep鉴定文库大小分布,结果见图27。文库使用NovaSeq 6000进行测序,测序质量和结果见表16,图28-30。
表15 不同细胞投入量条件下FTCT-seq的PCR循环数和文库产量
表16 不同细胞投入量条件下FTCT-seq的测序质量
从图26和表16可知,FTCT-seq在单细胞-100万细胞投入量条件下都有较好的文库产出。在10个细胞以上的投入量条件下文库大小分布仍可以维持基本的不同数量核小体结合DNA梯度分布(见图27)。从表16和图28可知,当细胞投入量大于1000时,测序数据在人类基因组上的比对率高于98%,重复率低于15%。当细胞投入量大于100时,测序数据在人类基因组上的比对率高于92%,重复率低于20%。当细胞投入量大于10时,测序数据在人类基因组上的比对率高于80%,重复率低于55%。当细胞投入量为单细胞时,测序数据在人类基因组上的比对率达到62.15%,重复率为72.57%。不同细胞投入量的FTCT-seq数据呈现出较好的相关性(图29,图30)这些结果表明FTCT-seq能够容纳从单细胞到100万细胞投入量的样本。
实施例6:标准品DNA的制备。
利用表1 的引物,以Lambda DNA(New England Biolabs,N3011S)为模板,进行DNA标准品扩增。按照表17的体系进行配制:
表17
混匀后加入到含细胞和磁珠的PCR管中,混匀。
表18 扩增反应程序
反应结束后,切胶回收,Qsep验证片段纯度,Sanger测序验证序列确定性。结果见图31。将3个Spikein DNA标准品按照摩尔浓度比1:3:10混合成Spikein Mix,稀释成10 pg/µl。Qsep验证混合之后的浓比是否正确,见图32。
实施例7: FTCT-seq(含Spikein mix) 在测序数据定量中的效果。
分别利用H3K27me3抑制剂EPZ005687(甲基转移酶复合物EZH2抑制剂,Selleck,Cat#S7004, 10 µM终浓度)和H3K27me3促进剂GSK-J4(去甲基转移酶复合物JMJD3/KDM6B和 UTX/KDM6A抑制剂,Selleck, Cat#S7070,30 µM终浓度)提前处理细胞一天,对照用DMSO。
1. 收集细胞。取约100,000个293FT细胞,600x g室温离心5 min收集细胞。用清洗缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM 亚精胺,1x Protease inhibitorscocktail)清洗2次。将细胞重悬于90 μL 清洗缓冲液中。
2. 活化磁珠及捕获细胞。取10 μL ConA磁珠,加入结合缓冲液(20 mM HEPES(pH7.5),10 mM KCl,1 mM CaCl2,1 mM MnCl2)清洗磁珠2次。将磁珠重悬于10μL 结合缓冲液中。将磁珠加入到细胞中,室温旋转孵育5-10 min。
3. 一抗结合。用一抗稀释缓冲液(20 mM HEPES(pH 7.5),150 mM NaCl,0.5 mM亚精胺,0.05% Digitonin,2 mM EDTA,0.1% BSA,1x Protease inhibitors cocktail)按照1:100稀释一抗(Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb (CellSignaling Technology, #9733))将结合好细胞的磁珠置于磁分离架上,去上清。用加入一抗的缓冲液重悬磁珠,室温旋转孵育2 h。
4. 二抗与转座酶复合物预结合。在细胞孵育一抗的同时,用转座酶结合缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,0.5 mM 亚精胺,0.01% Digitonin,1x Proteaseinhibitors cocktail)按照1:50稀释重组融合蛋白Protein AG-Tn5(每个样品约1 U),往其中按照1:100加入二抗(Guinea pig anti-rabbit secondary antibody ABIN101961)。室温旋转孵育1-2 h。
5. 转座酶结合。将结合好一抗的磁珠置于磁分离架上,去上清。用转座酶和二抗预结合的缓冲液重悬磁珠,室温旋转孵育0.5 h。
6. 转座酶激活。用转座酶结合缓冲液清洗磁珠3次。用10 μL转座酶激活缓冲液(20 mM HEPES(pH 7.5),300 mM NaCl,10 mM MgCl2,0.5 mM 亚精胺,0.01% Digitonin,1xProtease inhibitors cocktail)重悬磁珠,37 ºC反应1 h。加入0.5 μL 0.5% SDS(终浓度0.05%),65 ºC孵育10 min。加入1 μL Spikein mix(10 pg/µl,每100,000个细胞加入10pg标准品DNA),加入1 μL 10% Triton-X 100(终浓度1%)。
7. 文库扩增。使用翊圣生物的Hieff NGS® Fast Tagment DNA Library PrepKit for Illumina试剂盒(Cat#12207)按表19的扩增体系和表20的程序进行文库扩增。
表19扩增反应体系
混匀后加入到含细胞和磁珠的PCR管中,混匀。
表20扩增反应程序
反应结束后将PCR管短暂离心置于磁分离架上,取95 µL上清,加入120 μL HieffNGS® DNA Selection Beads (Yeasen, 12601),重复上述磁珠回收步骤。用Qubit测定文库浓度,Qsep检测文库大小分布。结果见表21、图33和图34。将文库1:100000稀释,用表1 中的引物进行qPCR定量。定量结果见图35。
表21 FTCT-seq(含Spikein)构建建库的产量
由表21、图33和图34可知,H3K27me3促进剂GSK-J4和抑制剂EPZ005687不会显著影响文库的大小分布,但是会影响到文库的产量和特异性富集。促进剂能够提高文库的产量(约2.5倍),提高文库的特异性;而抑制剂会降低文库的产量(降低至9%),也显著降低了文库的特异性。
将构建好的文库在Illumina NovaSeq 6000平台上进行测序分析,分析后的结果见表22、图36-图40。
表22 不同处理状态H3K27me3 FTCT-seq数据质量
根据表22、图36和图37可知,加入DNA标准品Spikein mix测序结果中实际测量读长数和理论比例呈现显著的相关性(R
给本领域技术人员提供上述实施例,以完全公开和描述如何实施和使用所主张的实施方案,而不是用于限制本文公开的范围。对于本领域技术人员而言显而易见的修饰将在所附权利要求的范围内。
