一种应用于NGS平台的超微量核酸样本建库方法
文献发布时间:2023-06-19 10:41:48
技术领域
本申请涉及高通量测序技术领域,特别是涉及一种应用于NGS平台的超微量核酸样本建库方法。
背景技术
二代测序(NGS)系统因其可同时进行大量平行测序反应而广为人知。虽然不同的二代测序平台在技术细节上有许多不同,但在测序前都需要建立文库。而低质量的核酸样本,例如福尔马林固定石蜡包埋(Formalin-Fixed and Paraffin-Embedded,缩写FFPE)的样本,即FFPE样本,在建立文库时存在诸多问题。
FFPE样本,作为生物和医学领域的重要样本来源,广受研究者的青睐。但由于多种因素影响,一般从FFPE尤其是穿刺组织制成的FFPE样本中难以获得高质量且足量的DNA。所以如何通过穿刺切片样本中获得的DNA建立合格的文库已成为当前测序研究领域亟待解决的问题。
常规NGS平台建库包括,首先提取样本中的DNA,然后对DNA进行片段化并纯化,再依序进行末端修复、加A和纯化、接头连接和纯化双筛、pre-PCR和纯化、杂交捕获和洗脱、post-PCR和纯化等步骤。可高通量检测多种基因突变。但是每一步反应进行纯化损失大量DNA,样本分子多样性受影响;因此要求高建库起始量。而针对大量穿刺切片样本或显微切割的石蜡样本无法满足高建库起始量,因此亟需研发一种应用于NGS平台的超微量核酸样本建库方法。
发明内容
本申请的目的是提供一种改进的应用于NGS平台的超微量核酸样本建库方法。
为了实现上述目的,本申请采用了以下技术方案:
本申请公开了一种应用于NGS平台的超微量核酸样本建库方法,包括在对超微量核酸样本进行DNA提取之前,向超微量核酸样本中加入核酸修复酶,对微量核酸样本进行核酸修复;然后对含有核酸修复酶的超微量核酸样本进行DNA提取;对提取的DNA进行文库构建,获得超微量核酸样本的测序文库。
需要说明的是,本申请的超微量核酸样本建库方法,在进行DNA提取之前,对样本DNA进行核酸酶修复;并且,在DNA提取的过程中,都有核酸修复酶存在;能够有效的修复超微量核酸样本的DNA损伤,提高超微量核酸样本提取的DNA的质量,解决了提取的DNA质量差总量低,无法有效建库的问题。可以理解,本申请的关键之一是采用核酸修复酶对超微量核酸样本的DNA进行损伤修复;至于DNA提取的具体过程可以参考现有的超微量核酸样本DNA提取试剂盒,后续的测序文库建库也可以参考现有的文库构建流程;在此不作具体限定。但是,为了进一步提高超微量核酸样本的建库质量,本申请优选的方案中,对后续的建库流程也进行了改进,详见以下技术方案。
优选的,本申请的超微量核酸样本建库方法中,核酸修复酶为UDG酶,采用UDG酶进行核酸修复的反应条件为,50℃恒温孵育至少1小时。
需要说明的是,UDG酶只是本申请的一种实现方式中具体采用的核酸修复酶,不排除还可以采用其它类似功能的核酸修复酶,在此不作具体限定。其中,50℃恒温孵育至少1小时,也是本申请的一种实现方式中具体采用的反应条件;因为,UDG酶的最佳反应温度是50℃;可以理解,基于其它目的,也可以在此基础上进行温度和孵育时间的调整,只要能够起到核酸修复作用即可。
优选的,本申请的超微量核酸样本建库方法中,对提取的DNA进行文库构建,具体包括,对提取的DNA进行酶切、末端修复和加A组合在一起的一步法反应;然后,对反应产物依序进行加接头和纯化、Pre-PCR扩增和纯化、杂交捕获、post-PCR和纯化,即获得所述超微量核酸样本的测序文库。
需要说明的是,本申请的酶切、末端修复和加A组合在一起的一步法反应是指,在本申请的一种实现方式中,在反应体系中添加酶切、末端修复和加A的所有反应试剂,在一个反应中同步进行酶切、末端修复和加A;这样不仅节省了程序,而且减少了酶切后纯化的步骤;步骤精简,不仅提高了DNA利用效率、降低DNA损失,而且能够更好的保存分子的多样性。
优选的,本申请的超微量核酸样本建库方法还包括,在进行Pre-PCR扩增之前,对加接头纯化产物进行定量检测,根据定量检测结果确定Pre-PCR扩增循环数,然后再进行Pre-PCR扩增。
需要说明的是,本申请创造性的根据加接头纯化产物的定量结果确定Pre-PCR扩增的循环数,而不是根据DNA的原始投入量;这样更加准确和有针对性,采用最佳的扩增循环数,能够有效的提高建库质量。
优选的,本申请的超微量核酸样本建库方法中,超微量核酸样本为FFPE样本。
需要说明的是,FFPE样本是生物和医学领域比较常见的超微量核酸样本,也是本申请的一种实现方式中具体采用的样本。可以理解,本申请的建库方法不仅仅适用于FFPE样本,也可以用于其它超微量核酸样本,例如法医或其它领域获得的高度腐烂或损伤的样本、考古获得的古生物样本或其它生物样本等,在此不作具体限定。
优选的,本申请的超微量核酸样本建库方法中,在向FFPE样本中加入核酸修复酶进行核酸修复之前,对FFPE样本进行预处理;该预处理包括,刮取FFPE样本,加入脱蜡液,充分混合后56℃孵育;孵育完成后加入RNase-free water和蛋白酶K,56℃消化4小时以上,然后90℃灭活;灭活完成后,在温度降至石蜡凝固之前,提取下层的澄清液,即获得预处理的FFPE样本,向提取的下层澄清液中加入核酸修复酶进行核酸修复。
需要说明的是,本申请的FFPE样本预处理就是在有利的环境或条件下去除石蜡,本申请优选的加入RNase-free water和蛋白酶K进行消化处理,为后续的核酸酶修复和DNA提取奠定基础。
基于本申请的超微量核酸样本建库方法,本申请的另一面公开了一种特征针对FFPE样本的建库方法,具体包括以下步骤,
(1)刮取FFPE样本,对FFPE样本进行预处理,去除石蜡;
(2)向预处理的FFPE样本中加入核酸修复酶进行核酸修复;
(3)对含有核酸修复酶的FFPE样本进行DNA提取;
(4)对提取的DNA进行酶切、末端修复和加A组合在一起的一步法反应;
(5)对步骤(4)的反应产物进行加接头,并对加接头产物进行纯化;
(6)对步骤(5)的纯化产物进行定量检测;
(7)根据步骤(6)定量检测的结果确定Pre-PCR扩增循环数,进行Pre-PCR扩增,并对Pre-PCR扩增产物进行纯化;
(8)对步骤(7)的纯化产物进行杂交捕获、洗脱;
(9)对步骤(8)的产物进行post-PCR扩增,并对post-PCR扩增产物进行纯化,即获得本申请的FFPE样本的测序文库。
需要说明的是,本申请的超微量核酸样本建库方法中,其中一个关键改进就是在DNA提取前和提取时采用了核酸修复酶;该方法也可以单独用于超微量核酸样本的DNA提取。
因此,本申请的再一面公开了一种超微量核酸样本的DNA提取方法,包括在对超微量核酸样本进行DNA提取之前,向超微量核酸样本中加入核酸修复酶,对微量核酸样本进行核酸修复;然后对含有核酸修复酶的超微量核酸样本进行DNA提取。其中,核酸修复酶可以参考本申请的超量核酸样本建库方法,可以是UDG酶,在采用UDG酶进行核酸修复时,其反应条件为,50℃恒温孵育至少1小时。
由于采用以上技术方案,本申请的有益效果在于:
本申请应用于NGS平台的超微量核酸样本建库方法,在进行DNA提取之前,对样本DNA进行核酸酶修复;并且,在DNA提取的过程中添加核酸修复酶,能够有效的修复超微量核酸样本的DNA损伤,降低反应假阳性,提高超微量核酸样本提取的DNA的质量,使得构建的文库能够更好的适用于NGS平台测序。本申请的建库方法解决了FFPE样本提取的DNA质量差总量低,无法有效建库的问题,为生物或医学领域的FFPE样本的核酸研究奠定了基础。
附图说明
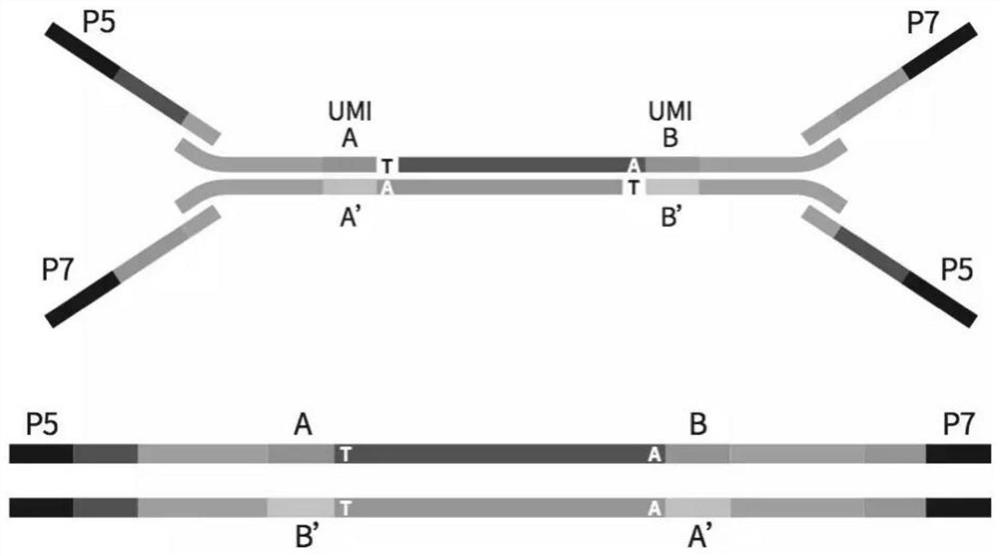
图1是本申请实施例中双端UMI接头的结构示意图。
具体实施方式
FFPE样本由于DNA损伤、DNA含量低等问题,难以提取获得满足NGS测序平台文库构建所需要的足够量的高质量DNA;因此,如何有效的进行FFPE样本DNA提取,并对其进行合格的文库构建和核酸测序,是本领域的研究重点和难点。
本申请创造性的采用核酸修复酶对FFPE样本的DNA损伤进行修复,从而提高FFPE样本的DNA质量。因此,本申请提出了一种改进的应用于NGS平台的超微量核酸样本建库方法,包括在对超微量核酸样本进行DNA提取之前,向超微量核酸样本中加入核酸修复酶,对微量核酸样本进行核酸修复;然后对含有核酸修复酶的超微量核酸样本进行DNA提取;对提取的DNA进行文库构建,获得超微量核酸样本的测序文库。
在进一步的改进方案中,本申请在建库过程中采用酶切、末端修复和加A组合在一起的一步法反应;并且,在进行Pre-PCR扩增之前,对加接头纯化产物进行定量检测,根据定量检测结果确定Pre-PCR扩增循环数;prePCR后只需进行单筛,即0.8×纯化,去除引物、接头序列等小片段。
本申请的应用于NGS平台的超微量核酸样本建库方法,只需1ng-50ng的超微量起始量DNA,即可有效的构建应用于NGS平台的测序文库,并满足测序和基因突变检测的使用需求。本申请的建库方法,通过修复DNA损伤,降低反应假阳性,提高DNA提取质量;优化建库方案,精简步骤,不仅能够降低DNA损失,而且能够更好的保存分子的多样性。本申请的建库方法,有效解决了超微量核酸样本或稀有样本,提取DNA质量差总量低,无法有效建库,难以利用NGS平台测序的难题。本申请的建库方法应用于临床医学FFPE样本的文库构建时,为药效评估以及靶向治疗动态监测提供了重要的参考依据;并且,为靶向用药提供关键的靶标突变位点信息,可根据患者的个体差异性,辅助医生选择合适的治疗药物、制定个体化的治疗方案,提高患者生活质量,延长患者的生存时间。
本申请中涉及的名词解释如下:
NGS,高通量测序技术(High-throughput sequencing)又称“下一代”测序技术(“Next-generation”sequencing technology),以能一次并行对几十万到几百万条DNA分子进行序列测定和一般读长较短等为标志。
UDG酶,选择性水解断裂含有dU的双链或单链DNA中的尿嘧啶糖苷键,形成的有缺失碱基的DNA链,在碱性介质以及高温下会进一步水解断裂,从而被消除;UDG酶的最佳活性温度为50℃,95℃灭活。
FFPE样本,福尔马林固定石蜡包埋处理的样本(Formalin-Fixed and Parrffin-Embedded,FFPE)可以在常温保留很久,所以对肿瘤组织进行石蜡包埋保存就成了医院里最常用的样本储存手段。
下面通过具体实施例对本申请作进一步详细说明。以下实施例仅对本申请进行进一步说明,不应理解为对本申请的限制。
实施例
本例应用于NGS平台的超微量核酸样本建库方法主要包括,从FFPE样本中提取DNA,在提取DNA之前先进行核酸酶修复处理;并且,在DNA提取过程中添加UDG;后续在建库过程中,去除多余纯化步骤,优化各步骤反应体系,使得构建的测序文库能够更好的适用于NGS平台测序;进而使得基于NGS测序的FFPE样本DNA检测的灵敏度幅度提升。本例的FFPE样本选用深圳海普洛斯医学检验实验室临床样本,样本编号为20191012001-b、20191013001-a、20191022001-b。本例的建库方法详细如下:
一、DNA提取优化
FFPE样本中的DNA提取质量较差,部分损伤修复方案容易造成检测结果假阳性,且影响建库效率,因此保证DNA提取质量至关重要。
本例的FFPE样本DNA提取方案详细步骤如下:
首先,采用手术刀片刮取FFPE样本。
石蜡肿瘤组织切片/石蜡穿刺组织样本:每个样本刮取后置于对应编号的2mL离心管内;若是组织蜡块,则需先用刀片去除表面氧化蜡层,再使刀片刀锋垂直于蜡面,横向轻柔刮取样本。
原则上,一个刀片仅处理一个样本,避免交叉污染。
每个2mL离心管加入160μL脱蜡液,充分涡旋10s,掌式离心机离心5s;
将2mL离心管置于金属浴内,56℃孵育3min,涡旋混匀,掌式离心机离心5s。
向2mL离心管中加入50μL RNase-free water,30μL UB和20μL蛋白酶K(Proteinase K),充分涡旋混匀,掌式离心机离心5s。
将2mL离心管置于金属浴中,56℃消化4小时以上或过夜消化。
将2mL离心管置于金属浴中,90℃,灭活60min。
准备新的1.5mL离心管,标写对应的编号。将2.0mL离心管从金属浴中取出,快速离心3s,趁降温之前,将下层的澄清液转移至对应编号的1.5mL离心管中,补充115μL RNase-free water。待液体降为室温时,加入35μL的UDG酶混匀,50℃孵育1小时。
离心,富集管壁微滴。然后,采用QIAamp DNA FFPE Tissue Kit进行DNA提取,具体如下:
每个离心管内加入2μL RNase A,充分涡旋混匀,室温放置2min。
每个离心管内再加入200μL Buffer AL,再加入200μL无水乙醇,充分涡旋混匀,离心3s,4℃孵育10min。
准备好4℃保存的黄色吸附柱,即QIAamp MinElute column,以及2mL收集管,一个样本准备一个吸附柱,写好对应的编号。取600μL上述步骤中的全部溶液置于对应编号的吸附柱内,12000rpm离心1min,丢弃收集管内的废液。
向吸附柱中加入500μL Buffer AW1,12000rpm离心1min,弃废液,将吸附柱放回收集管中。
向吸附柱中加入500μL Buffer AW2,12000rpm离心1min,弃废液。将吸附柱放回收集管中。
向吸附柱内加入250μL无水乙醇,12000rpm离心1min,弃废液,将吸附柱放回收集管中。
将空的吸附柱以12000rpm转速离心2min。开盖,室温放置5min挥发酒精。
将吸附柱转入一个干净的1.5mL离心管中,离心管盖上标记相应的编号,向吸附膜中间位置悬空滴加40μL的56℃预热的Nuclease-free Water,室温放置5min,12000rpm离心2min,将溶液收集到离心管中,即完成FFPE样本DNA提取,提取的DNA可以直接使用或者-20℃保存备用。
二、提取的DNA超微起始量建库
本例采用优化的文库构建方法进行建库,具体操作如下:
2.1计算提取的三个样本的DNA浓度,每个样本针对1ng和10ng两种超微量投入量,分别计算取样体积,具体如表1所示。
表1建库起始用量
2.2酶切末端修复加A反应:
本例采用5×WGS Fragmentation mix(天根,Y9410),10×Fragmentation buffer(天根,B0330)进行酶切、末端修复和加A组合在一起的一步法反应。具体的,按照表2配制反应体系,表2为各组份单次反应用量,本例在200μL的PCR管中准备反应体系。
表2酶切、末端修复和加A反应体系
表2中,“X”为提取的DNA的添加体积,按照表1的建库起始用量分别添加各样本的1ng和10ng的投入量体积;“33-X”是指无核酸水(Nuclease-free H
用移液器轻柔吸打6-8次混匀,将离心管瞬时离心后,立即放入预冷至4℃的PCR仪中,进入反应程序。
使用PCR仪,将热盖温度设置至70℃,进行如下反应程序:4℃1min、32℃反应8min、65℃恒温30min,然后4℃待机。
2.3接头连接
本例采用
表3接头信息及添加量
根据上表,向上一步得到的反应产物中加入对应体积接头,去相应量的体积补水至5μL后加入上一步的反应产物中。本例具体取0.5μL浓度15μmol/L的接头,加Nuclease-free H
本例的连接反应采用T4 DNA Ligase(600000u/mL)(天根,L6030-600000)和5×rapld ligation buffer(天根,B9020L),具体的,按照表4配制连接反应体系,表4为各组份单次反应用量。
表4连接反应体系
分别向样本管加入45μL接头连接反应体系,以及5μL接头,进行连接反应的混合液中包括45μL接头连接反应体系+5μL接头+50μL“2.2酶切末端修复加A反应”步骤产物,总计100μL。用移液器吸打混匀后瞬时离心。置PCR仪中20℃反应15min。
2.4接头纯化
磁珠HyperCap Bead Kit,96rxn(Roche,08286400001)
1)加入80μL充分混匀的磁珠至连接产物中,混匀离心。
2)室温孵育10min后上架吸附,弃去上清。
3)用500μL 80%乙醇洗涤磁珠,弃去上清。重复本洗涤步骤一次。
4)将离心管取下磁力架,开盖干燥至磁珠表面不反光。
5)每个样本管内加入22μL Nuclease-Free water,重悬浮磁珠,充分混匀后室温静置5min。
6)准备一批新的0.2mLPCR管,管盖管壁标注对应的样本编号。将样本管置于磁力架,进行磁珠吸附,直至溶液澄清后,将上清液转移至对应编号的PCR管中,作为PCR实验的模板。
7)配置Qubit Buffer 199μL,加入1μL DNA样本,涡旋混匀,避光静置2min,测试浓度。
表5三个样本的起始浓度
根据检测的浓度设置后续Pre-PCR扩增反应的循环数,本例具体的,选择Pre-PCR扩增反应的循环数为12个循环。
2.5Pre-PCR
按照表6所示,配制Pre-PCR扩增反应体系,配置好反应体系后移至加样槽内。2×高保真酶反应预混液:KAPA HiFi Hotstart readyrMix(2×)(Roche,7958935001);UDIPrimer Mix(5μmol/L):KAPA IDT
表6 Pre-PCR反应体系
每个0.2mL样本管内加入30μL的Pre-PCR扩增反应体系,涡旋混匀。
按照表7所示设置PCR扩增过程温度和时间参数。
表7 Pre-PCR反应条件
其余投入量定量,循环数选择如表8所示。
表8 Pre-PCR扩增循环数参考表
2.6Pre-PCR后纯化
具体步骤如下:
A.将反应加入40μL混合均匀的磁珠,充分涡旋混匀后,室温静置10min。
B.置于磁力架上,进行磁珠吸附,直至溶液澄清。
C.小心移除上清液,再加入200μL 80%乙醇,180度旋转八连管使磁珠穿过溶液吸至另一侧管壁,旋转2-3次,静置15s后弃上清液。
D.重复上述步骤C一次。
E.将管自磁力架上取下,快速离心,然后置于磁力架上再次分离、移除残留的酒精溶液。将样本管自磁力架上取下,打开管盖,常温下干燥磁珠,挥发乙醇,以免过多乙醇影响后续反应体系中酶的效果。
F.每管样本管内加入22μL Nuclease-Free water,充分混匀后室温静置5min。
G.准备一批新的离心管,管盖上标注所属项目,收样日期,样本名称,g-PrePCR;管壁上标注接头信息,建库日期。
H.将样本管置于磁力架上,进行磁珠吸附,直至溶液澄清后,将上清液转移至对应的新的写有样本信息的1.5mL离心管。
对获得的Pre-PCR扩增产物的纯化产物,按照常规的建库流程进行基因组DNA杂交捕获洗脱、post-PCR和纯化,以及polling上机测序。根据KAPA HyperCap workflow和Illumina Novaseq 6000使用说明进行上机测序。即本例采用双端测序方案对构建的测序文库进行测序Illumina Novaseq 6000。
三、文库构建结果数值分析
本例的测序深度为2200×,本例分别对三个样本分别在1ng和10ng的建库起始量的情况下,采用本例的文库构建方法进行建库,测试本例的文库构建方法在超底起始量下的建库效果。本例的测序数据统计分析和结果如表9所示。
表9测序结果分析
表9的结果显示,本例的超微量建库方法测序结果能够满足分析,建库成功。即本例的应用于NGS平台的超微量核酸样本建库方法,针对FFPE样本这样的超微量核酸样本,也能够构建出满足NGS测序需求的测序文库。
本例的应用于NGS平台的超微量核酸样本建库方法,以稀有FFPE样本中获得的低质量超微量DNA为对象,优化DNA的提取,修复DNA损伤,降低反应假阳性,优化酶切末端修复加A反应体系,去除之后纯化步骤,优化prePCR反应循环数,精简步骤,保存分子的多样性。本例的超微量核酸样本建库方法,有效的解决了稀有样本,提取DNA质量差总量低,无法有效建库,难以利用NGS平台测序的难题;采用本例的建库方法,能够获得满足NGS平台测序和后续测序结果分析的测序文库,能够对稀有FFPE样本进行点突变、插入缺失、基因融合,基因拷贝数变化等基因变异信息检测,从而给出更精准的用药指导信息,为个体化精准用药指导、药效评估以及靶向治疗动态监测提供了重要的参考依据。
以上内容是结合具体的实施方式对本申请所作的进一步详细说明,不能认定本申请的具体实施只局限于这些说明。对于本申请所属技术领域的普通技术人员来说,在不脱离本申请构思的前提下,还可以做出若干简单推演或替换。
