基于图卷积网络和自注意力机制的DNA存储编码方法
文献发布时间:2024-04-18 19:52:40
技术领域
本发明属于DNA存储中编码技术领域,涉及机器学习算法和DNA存储编码,具体涉及一种基于图卷积神经网络及自注意力机制的DNA存储编码方法。本发明可以根据建模后的DNA存储编码图的结构特征和节点属性,通过图神经网络和自注意力机制对图结构和节点进行特征提取,构建DNA存储编码预测模型。
背景技术
DNA作为一种高密度、高耐久性,并且在自然界中广泛存在的存储介质,在应对海量数据方面成为了一种可能的方案。尤其在当前大环境下,DNA在存储冷数据时有得天独厚的优势。相比传统存储介质,DNA存储是一种维护能耗基本为零的存储方式,可以在室温和干燥的环境中保存数千年。基于DNA的存储的基本原理是数字数据(以二进制信息表示)和DNA分子(以硅基DNA序列表示)之间的转换。因此,编码是DNA存储中最基础也是最重要的步骤之一。DNA存储中最基本的生化技术是DNA合成(“书写”信息)、聚合酶链反应(PCR)扩增(“复制”信息)和DNA测序(“阅读”信息)。利用DNA存储非生物信息可以追溯到1940年,Davis把DNA克隆到质粒中用于保存数据。但当时合成和测序技术限制了DNA存储的发展,而近年来合成和测序技术持续进步,DNA存储再次成为全球研究的热点。然而,DNA存储中的编码效率仍然不够高,但是随着各种高效编码算法的提出,DNA存储在将来会是磁介质存储的有力替代方案。
发明内容
本发明提出了一种基于图卷积网络和自注意力机制的DNA存储编码方法,该方法首先对已满足约束的编码集进行筛选,构造训练数据集;其次,利用基于图卷积网络和自注意力机制的神经网络在数据集上训练编码预测模型;接着,将编码处理为图数据,输入预测模型,进行满足约束的编码预测;最后,比对当前节点概率值是否大于阈值,将符合条件的加入DNA存储编码集合;该方法可以预测出数量较优的DNA编码序列。
为实现上述目的,本发明的技术方案为:
基于图卷积网络和自注意力机制的DNA存储编码方法,其具体为:预测满足组合约束条件的DNA编码序列。首先对已有DNA编码进行筛选和数据清洗,构造DNA存储编码训练集;其次,训练基于图卷积神经网络和自注意力机制的预测模型,利用自注意力机制捕捉局部DNA编码的关系;然后,将处理为图的DNA编码输入预测模型,进行满足组合约束的编码预测;最后,输出符合满足约束条件的DNA存储编码集合。具体步骤如下:
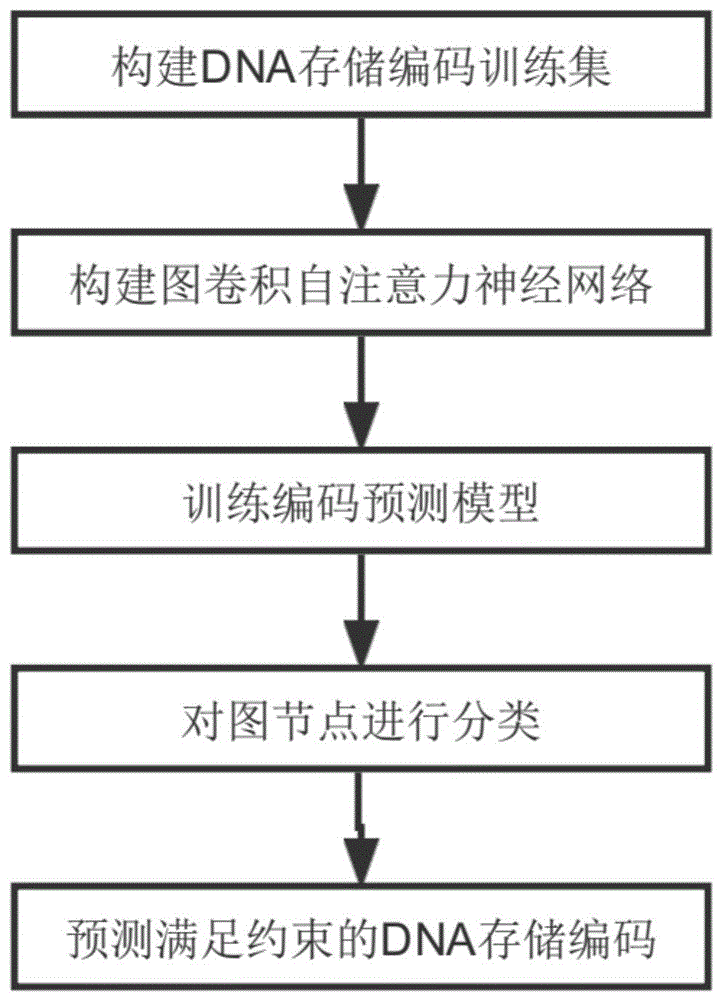
步骤1:构建满足组合约束的DNA存储编码训练集。
DNA存储编码一般需要满足给定的组合约束,除距离约束外还需要满足GC含量(GC-Content)、全不连续约束(No-runlength constraint)、非相邻子序列约束(Non-adjacent subsequence constraint)、末端约束(End-constraint)和自补约束(Self-complementary constraint)等。
构建DNA存储编码训练集需要从前人工作中,收集不同约束下DNA存储编码结果。并对DNA存储编码数据进行预处理,即将DNA存储编码间的关系和编码中的信息映射为图中节点和边,最终完成DNA存储编码训练集的构建。
步骤2:构建图卷积自注意力神经网络。
搭建由多层卷积层和自注意力机制层组成的图卷积自注意力神经网络,结构依次为输入层、第一卷积层、第一池化层、第二卷积层、第二池化层、自注意力层、第三卷积层、激活层和输出层。并将图卷积自注意力神经网络池化层设置平均池化方式,激活层使用非线性激活函数ReLU,在最后一层使用sigmoid函数。
步骤3:训练图卷积自注意力神经网络。
将步骤1中构建的带有类别标签的满足组合约束的DNA存储编码训练集输入步骤2中构建的图卷积自注意力神经网络迭代更新网络参数,直至初步预训练的损失函数预测得到的类别标签与实际类别标签之间的差异值收敛为止,得到初步训练的图卷积自注意力神经网络模型。
所述初步训练的损失函数如下:
其中l是节点分类标签,l
步骤4:对不包含类别标签的DNA存储编码图进行节点分类。
将一个不含类别标签的DNA编码图输入到步骤3中训练好的图卷积自注意力神经网络中,网络输出一个预测概率的特征向量,通过特征向量与阈值的比较,确定每一个节点是否属于DNA存储编码集合。
本发明的有益效果:
1)本发明的编码结果克服了编码长度增加带来的编码障碍,提高DNA存储系统的存储密度;
2)利用图卷积网络提取特征和自注意力机制捕捉局部DNA码字的关系,并对DNA编码向量进行加权聚合,提高DNA存储编码效率;
3)本发明能够构建出更多满足约束的DNA存储编码。
附图说明
图1为本发明的实现流程图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施中的技术方案进行清楚、完整的描述,可以理解的是,所描述的实例仅仅是本发明的一部分实例,而不是全部的实施例。基于本发明的实施例,本领域的技术人员在没有做出创造性劳动的前提下所获得的所有其他实施例,都属于本发明的保护范围。
详细步骤如下所示:
步骤1:构建满足组合约束的DNA存储编码训练集。
DNA存储编码一般需要满足给定的组合约束,除距离约束外还需要满足GC含量(GC-Content)、全不连续约束(No-runlength constraint)、非相邻子序列约束(Non-adjacent subsequence constraint)、末端约束(End-constraint)和自补约束(Self-complementary constraint)等。
构建DNA存储编码训练集主要分为如下步骤:
步骤1.1:从前人工作中进行不同约束下的所有情况DNA存储编码结果的收集,本实施例统计了5种不同组合约束下的编码数据集,包括589651个阴性样本,70734个阳性样本。
步骤1.2:对数据进行分类和筛选,将长度小于5的DNA编码筛选掉,因为长度小于5的序列在四元码(ATGC)情况下只有4^4=256种排列方式,候选解集太小。
步骤1.3:将五种不同组合约束的编码数据集分别命名为GNH_db、GNHN_db、GNHE_db、GNHS_db、GNE_db。
步骤1.4:并对DNA存储编码数据进行预处理,即通过将DNA存储编码间的关系和编码中的信息映射为图中节点和边,构建DNA存储编码图,构建DNA存储编码训练集。
步骤2:构建图卷积自注意力神经网络。
搭建由多层卷积层和自注意力机制层组成的图卷积自注意力神经网络,结构依次为输入层、第一卷积层、第一池化层、第二卷积层、第二池化层、自注意力层、第三卷积层、激活层和输出层。图卷积自注意力神经网络由多层卷积层{H
其中,
将图卷积自注意力神经网络池化层设置平均池化方式,激活层使用非线性激活函数ReLU,在最后一层使用sigmoid函数。并且利用自注意力机制捕捉数据的内部相关性,对于输入向量L,使用单头注意力模块的输出向量L*是所有输入的特征向量的加权和,通过公式(2)计算:
其中,d
步骤3:训练图卷积自注意力神经网络。
将步骤1中构建的带有类别标签的满足组合约束的DNA存储编码训练集输入步骤2中构建的图卷积自注意力神经网络迭代更新网络参数,直至初步预训练的损失函数预测得到的类别标签与实际类别标签之间的差异值收敛为止,得到初步训练的图卷积自注意力神经网络模型。
所述初步训练的损失函数如下:
其中l是节点分类标签,l
步骤4:对不包含类别标签的DNA存储编码图进行节点分类。
将一个不含类别标签的DNA编码图输入到步骤3中训练的图卷积自注意力神经网络中,网络输出一个预测概率的特征向量,通过特征向量与阈值的比较,确定每一个节点是否属于DNA存储编码集合。
具体来说,
下面结合仿真实验对本发明的效果做进一步的说明。
仿真实施例1
1.仿真实验条件:
本发明的仿真实验的硬件平台为:处理器为intel i9-9900K,主频为3.6GHz,显卡RTX 3090,内存256GB。
本发明的仿真软件平台为:Ubuntu 18.04操作系统和Python3.6.
2.仿真内容及其结果分析:
本发明仿真实验是采用本发明和现有技术的编码方法(末端约束编码(EP),生物约束编码(BC),哈萨克鹰编码(NOL-HHO),K聚类多元宇宙编码(KMVO),布朗多元宇宙编码(BMVO),分别在相同组合约束的条件下对DNA存储编码进行设计,最终得到满足组合约束的DNA存储编码集合。
现有技术EP存储编码方法是指,Wu等人在“Enhancing Physical andThermodynamic Properties of DNA Storage Sets with End-constraint,in IEEEtransactions on nanobioscience,21(2):184-193,2021.”中提出的基于End-constrain的编码算法,简称EP。
现有技术BC存储编码方法是指,Rasool等人在“Bio-Constrained Codes withNeural Network for Density-Based DNA Data Storage,in Mathematics,10(5):845,2022.”中提出的基于生物约束码的编码算法,简称BC。
现有技术NOL-HHO存储编码方法是指,Yin等人在“An Intelligent OptimizationAlgorithm for Constructing a DNA Storage Code:NOL-HHO,in Internationaljournal of molecular sciences,21:6,2020”中提出的基于NOL-HHO的编码算法,简称NOL-HHO。
现有技术KMVO存储编码方法是指,Cao等人在“K-means multi-verse optimizer(KMVO)algorithm to construct DNA storage codes,in IEEE Access,8:29547-29556,2020.”中提出的K-means多元宇宙DNA存储编码算法,简称KMVO。
现有技术BMVO存储编码方法是指,Zhang等人在“Minimum free energy codingfor DNA storage,in IEEE Transactions on NanoBioscience,2:212-222,2021.”中提出的布朗多元宇宙DNA存储编码算法,简称BMVO。
本仿真实验中使用的编码数据集选取在GNH_db中,5 编码正确率=预测正确的编码数/数据集中编码总数。 表1中n表示编码长度,d表示汉明距离,结合表1可以看出,本发明在编码长度n=7的所有情况下达到了现有技术的平均水平,而在其他情况下均超越了现有技术,证明了本发明可以得到更多的满足组合约束的DNA存储编码。 表1六种DNA存储编码方法的编码性能比较 以上所述仅是本发明的优选实施方式,并不用于限制本发明,应当指出,对于本技术领域的普通技术人员,在不脱离本发明技术原理的前提下,还可以做出若干改进和变型,这些改进和变型也应视为本发明的保护范围。
