一种利用L/RPA快速检测SARS-CoV-2的探针及引物组、试剂盒、检测方法
文献发布时间:2023-06-19 12:08:44
技术领域
本发明涉及生物检测领域,特别涉及一种利用L/RPA快速检测SARS-CoV-2的探针及引物组、试剂盒、检测方法。
背景技术
新型冠状病毒(SARS-CoV-2)已经对全世界人民的健康安全产生严重的安全威胁。有效的疫苗,准确的诊断对于控制传染病至关重要。在现阶段基因组学研究深入的前提下,关于新型冠状病毒的分子诊断技术也获得了飞速的发展。目前成为临床分子诊断最主要的检测方法是逆转录荧光定量PCR方法(RT-qPCR),该方法将冠状病毒RNA转录成为cDNA,并且通过聚合酶链式反应对冠状病毒的生物标志物进行特异性的扩增,最终实现荧光信号的实时检出。然而,RT-qPCR检测方法对于实验室和专业人员的依赖程度非常高,这并不适合在资源匮乏的地区甚至国家进行有效的病毒检测。为了满足资源匮乏地区以及早发现早治疗的现实需求,利用LAMP、RPA等技术开发的等温扩增检测方法也相继被报道,这些方法在一定程度上减少了检测时对于设备的依赖性。除此之外,为了使得检测更灵敏,结果更直观,侧向流试纸条和便携式荧光检测仪同样被应用到了病毒检测中,这些检测方法无疑为SARS-CoV-2的分子检测提供了更多的选择。
然而,基于PCR、LAMP、RPA技术开发出的分子诊断方法只能够扩增DNA模板,无法直接扩增RNA模板,因此必须将病毒的RNA逆转录成cDNA才能够进行后续的扩增。众所周知,RNA很容易受到环境中RNase的污染,而逆转录实验本身对于RNA的浓度和纯度要求都很高,这也就直接要求了操作人员不仅需要足够专业,同样还需要足够小心以避免环境对待检样本的污染,因此基于逆转录步骤的分子检测方法的应用受到了很大的限制。
为了避免逆转录步骤,减少临床样本中病毒RNA的损失以及提高检测的准确度,本项目发明了一种利用连接酶、重组酶聚合酶扩增(L/RPA)快速检测SARS-CoV-2的试剂盒。本发明针对中国疾控中心报告出的SARS-CoV-2生物标志物N基因和ORF1ab基因分别设计连接探针,这些探针中有一部分与目标基因互补配对,而另一部分能够促进RPA扩增。当待测样本中存在病毒RNA时,探针能够与RNA互补配对,接着在连接酶的作用下两条探针能够形成一条完整的DNA链,一旦形成完整的DNA链,就可以成为RPA扩增的模板,另一组引物组合可以与形成的DNA链通过RPA体系进行扩增,如此一来即可避免逆转录步骤,并且在RPA扩增体系内加入SYBR Green I染料即可实现荧光信号的实时读取。
发明内容
本发明旨在提供一种不依赖仪器设备,反应快速灵敏,操作简单,且可以避免样本污染的L/RPA检测方法,且提供了该方法所用的探针组及引物组,并进一步探索了最佳检测体系。
为了实现上述目的,本发明具体采用如下技术方案:一种利用L/RPA快速检测SARS-CoV-2的探针及引物组,所述的检测探针及引物组分别为针对N基因和针对ORF1ab基因的探针及引物组;
所述针对N基因的探针及引物组包括:探针组:N-Probe 2A-3、N-Probe 2B-3、引物组N-Primer 3F、N-Primer 3R;
所述针对ORF1ab基因的探针及引物组包括:探针组:O-Probe 1A-2、O-Probe 1B、引物组O-Primer 2F、O-Primer 2R;
所述N-Probe 2A-3序列为SEQ ID NO.5所述的序列;
所述N-Probe 2B-3序列为SEQ ID NO.8所述的序列;
所述O-Probe 1A-2序列为SEQ ID NO.18所述的序列;
所述O-Probe 1B序列为SEQ ID NO.19所述的序列;
所述N-Primer 3F序列为SEQ ID NO.14所述的序列;
所述N-Primer 3R序列为SEQ ID NO.16所述的序列;
所述O-Primer 2F序列为SEQ ID NO.26所述的序列;
所述O-Primer 2R序列为SEQ ID NO.27所述的序列。
优选的,所述探针N-Probe 2A-3的5’端带有磷酸基团;所述探针O-Probe 1A-2的5’端带有磷酸基团。
一种试剂盒,所述试剂盒含有前述所述的利用L/RPA快速检测SARS-CoV-2的探针及引物组。
一种利用L/RPA快速检测SARS-CoV-2的方法,所述的利用L/RPA快速检测的方法包括如下步骤:
(1)将RNA模板与前述所述的针对N基因的探针组或针对ORF1ab基因的探针组加热至85℃~95℃并保持40s~80s得到混合物;
(2)将混合物冷却,使探针组与RNA模板退火形成杂合链;
(3)加入T4 DNA连接酶,T4 DNA连接酶Buffer,并且在37℃下连接8-10min后升温至95℃,2min后失活T4 DNA连接酶得到连接产物;
(4)向RAA反应管中加入连接产物、前述所述的针对N基因的引物组或针对ORF1ab基因的引物组、ABuffer、B Buffer、核酸荧光染料,将RAA反应管置于实时荧光定量PCR仪器中,检测温度37℃,检测间隔20s,检测时间15min。
优选的,所述核酸荧光染料选自SYBR Green I荧光染料。
优选的,所述RNA模板与探针组的体积比为:3:1。
优选的,所述T4 DNA连接酶的添加量为500U。
进一步优选的,所述利用L/RPA快速检测的体系包括连接体系和RPA扩增体系;
所述连接体系,以5μL计,包括:
RNA模板:3μL
针对N基因或针对ORF1ab基因的探针组:各探针浓度0.1~10μM,各探针体积0.5μLT4 DNA连接酶:0.5μL
T4 DNA连接酶Buffer:0.5μL
最后获得5μL的连接产物;
所述RPA扩增体系,以50μL计,包括:
针对N基因的引物组或针对ORF1ab基因的引物组:各引物浓度10μM、正反引物各2μL
ABuffer:36μL
B Buffer:2.5μL
核酸荧光染料:2.5μL
连接体系所得连接产物:5μL。
有益效果:
本发明探针及引物组、检测方法不依赖仪器设备,反应快速灵敏,操作简单,更有望应用于病毒突变株的检测中;
该发明还提供了一种较为新颖的检测体系,巧妙利用连接酶结合重组酶聚合酶实现快速检测,并且该发明囊括了RPA耗时短,连接酶避免逆转录步骤的众多优势。该发明的设计思路对于未来新冠病毒突变体甚至其他RNA病毒的检测都具有重大意义。该发明中所涉及的反应体系(探针与模板之间的配比,连接酶使用的比例)同样是非常有价值的。除此之外,本发明能够利用手持荧光仪在37℃下实现实时、实地、即时的快速现场检测。目前尚无这一系列的方法应用到RNA病毒的检测中,而本发明不仅仅提供了一种L/RPA实验技术,还体现了一种实验思路,每一步优化都为整个实验体系的可靠性提供了保障。
附图说明
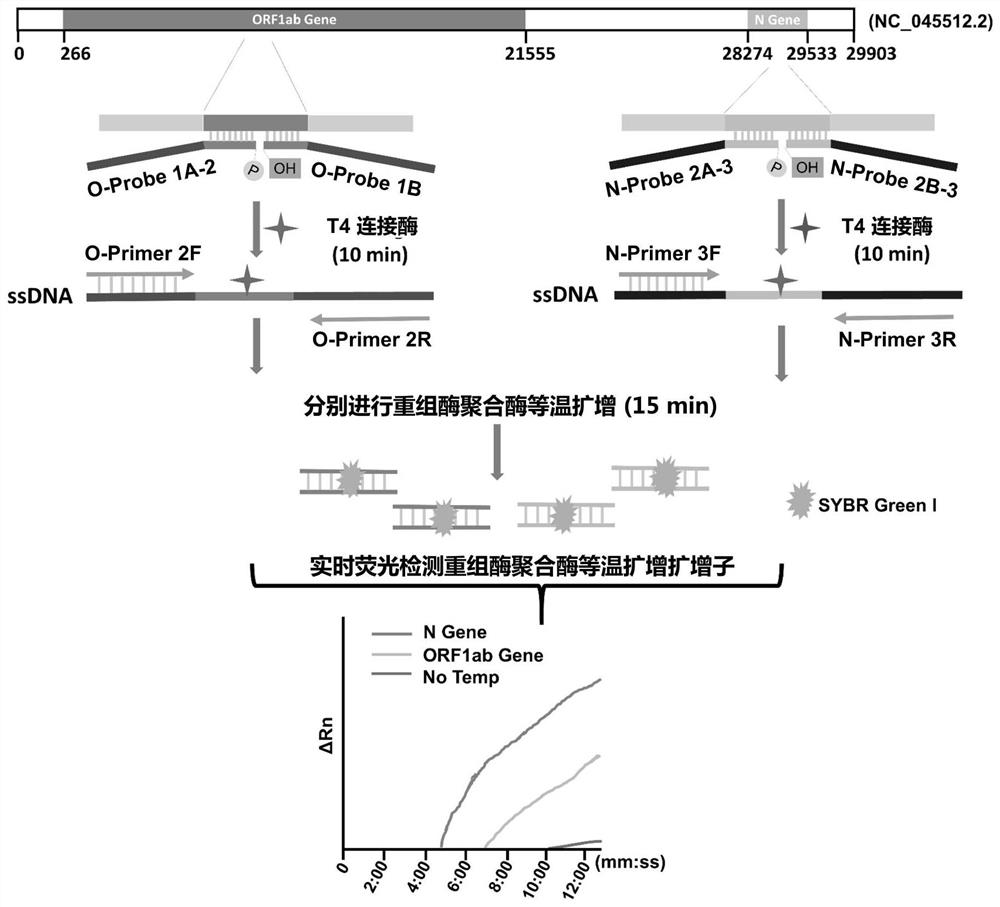
图1本发明连接酶/聚合酶重组酶扩增(L/RPA)原理示意图。
图2L/RPA筛选N基因最佳探针组合结果示意图。
图3L/RPA筛选ORF1ab基因最佳探针组合结果示意图。
图4L/RPA筛选N基因最佳RPA引物组合结果示意图。
图5L/RPA筛选ORF1ab基因最佳RPA引物组合结果示意图。
图6L/RPA与RT-qPCR方法检测假病毒RNA中N基因灵敏度的荧光定量结果图。
图7L/RPA与RT-qPCR方法检测假病毒RNA中ORF1ab基因灵敏度的荧光定量结果图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。以下对至少一个示例性实施例的描述实际上仅仅是说明性的,决不作为对本发明及其应用或使用的任何限制。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
需要注意的是,这里所使用的术语仅是为了描述具体实施方式,而非意图限制根据本申请的示例性实施方式。如在这里所使用的,除非上下文另外明确指出,否则单数形式也意图包括复数形式,此外,还应当理解的是,当在本说明书中使用术语“包含”和/或“包括”时,其指明存在特征、步骤、操作、器件、组件和/或它们的组合。
下面结合附图对本发明进行详细说明,以方便本领域技术人员理解本发明。
1、实验材料
含有ORF1ab、E、N基因的假病毒购自上海复百澳生物科技有限公司;RNA提取试剂盒购自北京天根生物技术有限公司;T4连接酶购自赛默飞科技有限公司;RAA试剂盒购自杭州众测生物科技有限公司;SYBR Green I荧光染料购自索莱宝生物科技有限公司;探针由安徽通用生物系统有限公司合成。
2、样本准备
利用购自北京天根生物技术有限公司的RNA提取试剂盒提取出上述各假病毒中的RNA,利用含有N基因与ORF1ab基因的质粒进行体外转录。
3、引物和探针设计
针对SARS-CoV-2基因组(GenBank:NC_045512.2)的N基因和ORF1ab基因分别设计含有用于T4连接酶的连接段和重组酶聚合酶的扩增段的探针。其中靶标区域的选择参考中国疾控中心列出的RT-qPCR检测区域,以此为参考依据可以在一定程度上确保特异性。针对探针再设计相对应的RPA引物。体系中用于T4连接酶的连接段能够与靶标序列的两端完全匹配,而用于RPA实验的扩增段不仅需要排除与任何微生物或人类核酸序列的相似,还要遵循RPA引物设计原则的要求,并且最终需要将设计好的序列进行引物二聚体分析。因此在整个发明过程中,首先针对连接酶所使用的探针进行了筛选,在引物设计过程中为了找到连接效果最好的探针组合,因此针对RT-qPCR检测区域中3个区域设计了探针,接着对于用于RPA扩增的引物进行筛选,以及为了实现最佳的扩增效率,最后进一步对于引物探针进行了修改。本发明实施例中所有序列信息见表1,表中探针带有下划线的序列即为连接酶所需的与模板匹配的区域,剩余序列即为RPA扩增所需的匹配序列,且与表中命名为引物的序列互补。
表1本发明所用序列信息表
3.1探针和引物初步设计
如上表所示,针对N基因,本发明首先设计了三组探针组,分别为:
N-Probe1组:N-Probe 1A N-Probe 1B
N-Probe2组:N-Probe 2A N-Probe 2B
N-Probe3组:N-Probe 3A N-Probe 3B
并针对ORF1ab基因也设计了三组探针:
O-Probe1组:O-Probe 1A O-Probe 1B
O-Probe2组:O-Probe 2A O-Probe 2B
O-Probe3组:O-Probe 3A O-Probe 3B
针对上述各个基因的三组探针进行筛选,筛选过程如下:
针对N基因筛选:
针对N基因的三组探针,设计对应的RPA引物:上表中的N-Primer 1F和N-Primer1R。将设计好的三组探针(N-Probe1组、N-Probe2组、N-Probe3组)分别与引物组合(N-Primer1F/N-Primer 1R)结合进行L/RPA检测,这样筛选目的是为了能够确保无模板(NoTemplate)检测时间与有模板的检测时间相差足够大,才能够确保该体系在后续检测中具有较高的灵敏度。检测结果如图2和表2所示,图2中,横坐标表明检测时间,纵坐标表明检测产生的荧光信号值,No Temp代表无模板对照,10
针对ORF1ab基因筛选:
针对ORF1ab基因的三组探针,设计对应的RPA引物:上表中的O-Primer 1F和O-Primer1R。
将设计好的三组探针(O-Probe1组,O-Probe2组,O-Probe3组)分别与引物组合(O-Primer1F/O-Primer 1R)结合进行L/RPA检测,这样能够确保无模板(No Template)检测时间与有模板的检测时间相差足够大,才能够确保该体系在后续检测中具有较高的灵敏度。检测结果如图3和表2所示,图3中横坐标表明检测时间,纵坐标表明检测产生的荧光信号值,No Temp代表无模板对照,10
表2 L/RPA筛选N基因与ORF1ab基因最佳探针实验结果
上述所述L/RPA检测体系如下:
如图1所示,将3μL RNA模板与针对N基因或针对ORF1ab基因探针组中的两种探针各0.5μL,各浓度均为0.1~10μM,加热至85℃~95℃并保持2min,随后将混合物冰上冷却,使得探针与RNA模板退火形成杂合链。接着加入0.5μLT4 DNA连接酶,0.5μL 10X T4 DNA连接酶Buffer并且在37℃下连接8-10min后95℃,2min失活T4 DNA连接酶。向RAA反应管中分别加入5μL连接产物、2μL RPA正反向引物(10μM)、36μLA Buffer、2.5μL BBuffer、2.5μLSYBR Green I荧光染料(15X),将反应管置于ABI 7900实时荧光定量PCR仪器中,检测温度37℃,检测间隔20s,检测时间15min。
3.2引物和探针的进一步筛选和修改
通过上述步骤筛选出了较好的N基因与ORF1ab基因的探针,但这并不意味着体系没有继续进一步的空间,因此该发明又通过RPA原理,及引物二聚体分析等方法巧妙地设计了多组RPA引物,并且以最佳的连接酶探针来筛选最佳的RPA扩增引物。并且由于RPA引物在发生改变,相对应的连接酶的探针序列也发生了改变。
N基因:针对N基因RPA引物的筛选共有3组测试组合:
N-Primer-set 1:引物组合+探针组合为:N-Primer 1F/N-Primer 1R+N-Probe2A/N-Probe2B
N-Primer-set 2:引物组合+探针组合为:N-Primer 2F/N-Primer 2R+N-Probe2A-2/N-Probe2B-2
N-Primer-set 3:引物组合+探针组合为:N-Primer 3F/N-Primer 3R+N-Probe2A-3/N-Probe2B-3
使用3.1节中的L/RPA检测体系分别对上述三组组合进行筛选检测。
结果如图4,表3所示,图4中横坐标表明检测时间,纵坐标表明检测产生的荧光信号值,No Temp代表无模板对照,10
ORF1ab基因:针对ORF1ab基因RPA引物的筛选共有2组测试组合
O-Primer-set 1:O-Primer 1F/O-Primer 1R+O-Probe 1AO-Probe 1B
O-Primer-set 2:O-Primer 2F/O-Primer 2R+O-Probe 1A-2/O-Probe 1B
使用3.1节中的L/RPA检测体系分别对上述两组组合进行筛选检测。
结果如图5,表3所示,图5中横坐标表明检测时间,纵坐标表明检测产生的荧光信号值,No Temp代表无模板对照,Blank代表空白反应,10
表3 L/RPA筛选N基因与ORF1ab基因最佳RPA引物组合
L/RPA筛选N基因与ORF1ab基因最佳RPA引物及探针组合,上表体现出了在不同模板量的情况下,探针与引物的组合对于靶标的扩增效率,并且通过与无模板对照的检出时间进行对比,确定了针对N基因与ORF1ab基因的最佳探针引物组合。
4、连接酶酶量的筛选
不同浓度的酶量对于反应速率以及反应效果都有着一定的影响,因此本发明探究了T4连接酶在每个反应中用量不同对于反应的影响。如表4所示,当酶量为40U/reaction或200U/reaction时,低丰度模板的检出时间与无模板对照的检出时间差值很小,这样会存在着将阳性判定为阴性的情况,不利于实际检测。但是我们发现在将酶量加大至500U/reaction时,低丰度模板检出时间与无模板对照检出时间的差值增大了,这样就非常有利于结果的判定,避免了将低丰度模板误判为阴性的风险。并且在针对N基因与PRF1ab基因的数据进行统计时发现,酶量对于反应体系至关重要,而这一方面的反应优化常常是不被人重视的。
表4不同量的T4连接酶对于L/RPA体系的检测能力
上表中检测能力以样本检出时间与无模板对照检出时间的差值为体现,其中差值越大,表明扩增效率越好,假阳性风险越低。
5、灵敏度检测与RT-qPCR对标
为了进一步对比L/RPA方法与RT-qPCR方法的检测能力,将含有N基因与ORF1ab基因的SARS-CoV-2假病毒提取RNA,并且以提取出的RNA作为1,在此基础上再10倍稀释5个梯度,以L/PCR和RT-qPCR方法同时检测,并且比较二者之间的准确度与灵敏度。
L/RPA检测体系:
将3μLRNA模板与两个探针(1μM)各0.5μL加热至85℃并保持2min,随后将混合物冰上冷却,使得探针与RNA模板退火形成杂合链。接着加入0.5μLT4 DNA连接酶,0.5μL 10XT4DNA连接酶Buffer并且在37℃下连接8-10min后95℃,2min失活T4 DNA连接酶。向RAA反应管中分别加入5μL连接产物、2μLRPA正反向引物(10μM)、36μLABuffer、2.5μLB Buffer、2.5μLSYBR Green I荧光染料(15X),将反应管置于ABI 7900实时荧光定量PCR仪器中,检测温度37℃,检测间隔20s,检测时间15min。
本实施例针对N基因和ORF1ab基因所用探针和引物组合分别为N-Primer-set 3、O-Primer-set 2组合进行检测,T4 DNA连接酶酶量为500U。
RT-qPCR检测体系:
RT-qPCR反应是根据新型冠状病毒
所用仪器为ABI 7900HT快速荧光定量仪,其中针对ORF1ab基因的检测通道为VIC,针对N基因的检测通道为FAM。
在此体系中,针对N基因和ORF1ab基因所用探针和引物组合分别如表1所示,
上述检测体系的检测结果如图6和图7,表5所示,图6中左图横坐标表明检测时间,纵坐标表明检测产生的荧光信号值,No Temp代表无模板对照,Blank代表空白反应;右图横坐标表明RT-qPCR检测的循环数,纵坐标表明检测产生的荧光信号值。此外,图中曲线1代表直接提取的RNA的量,曲线10-10
表5 L/RPA与RT-qPCR方法检测假病毒RNA中N基因与ORF1ab基因灵敏度的检测时间(Tt)及循环数(Ct)
本发明所涉及核酸序列表如下:
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。
SEQUENCE LISTING
<110> 江苏海洋大学
<120> 一种利用L/RPA快速检测 SARS-CoV-2的探针及引物组、试剂盒、检测方法
<130> 20210510
<160> 33
<170> PatentIn version 3.3
<210> 1
<211> 37
<212> DNA
<213> 人工合成
<400> 1
agaagttccc cgacgaggga aagagttgta cctaaat 37
<210> 2
<211> 37
<212> DNA
<213> 人工合成
<400> 2
tgtataggaa tcccactgaa tttttcattc tagcagg 37
<210> 3
<211> 36
<212> DNA
<213> 人工合成
<400> 3
gcagcagcaa gacgagggaa agagttgtac ctaaat 36
<210> 4
<211> 34
<212> DNA
<213> 人工合成
<400> 4
gcagcagcaa gacgagggaa agagcagtac ctaa 34
<210> 5
<211> 34
<212> DNA
<213> 人工合成
<400> 5
gcagcagcaa gacgagggaa agagcagtac ctaa 34
<210> 6
<211> 36
<212> DNA
<213> 人工合成
<400> 6
tgtataggaa tcccactgaa tttttcaatc tgtcaa 36
<210> 7
<211> 33
<212> DNA
<213> 人工合成
<400> 7
tgtgtacgaa tcccacaaga atcaatctgt caa 33
<210> 8
<211> 35
<212> DNA
<213> 人工合成
<400> 8
tgtgtacgaa tcccactaat tcgccaatct gtcaa 35
<210> 9
<211> 36
<212> DNA
<213> 人工合成
<400> 9
tctcaagctg gacgagggaa agagttgtac ctaaat 36
<210> 10
<211> 38
<212> DNA
<213> 人工合成
<400> 10
tgtataggaa tcccactgaa tttttccaga cattttgc 38
<210> 11
<211> 26
<212> DNA
<213> 人工合成
<400> 11
atttaggtac aactctttcc ctcgtc 26
<210> 12
<211> 26
<212> DNA
<213> 人工合成
<400> 12
tgtataggaa tcccactgaa tttttc 26
<210> 13
<211> 24
<212> DNA
<213> 人工合成
<400> 13
ttaggtactg ctctttccct cgtc 24
<210> 14
<211> 24
<212> DNA
<213> 人工合成
<400> 14
ttaggtactg ctctttccct cgtc 24
<210> 15
<211> 23
<212> DNA
<213> 人工合成
<400> 15
tgtgtacgaa tcccacaaga atc 23
<210> 16
<211> 25
<212> DNA
<213> 人工合成
<400> 16
tgtgtacgaa tcccactaat tcgcc 25
<210> 17
<211> 37
<212> DNA
<213> 人工合成
<400> 17
aacccacagg ggataaggga gataataaga gttgggt 37
<210> 18
<211> 37
<212> DNA
<213> 人工合成
<400> 18
aacccacagg gcaataggga gatcatagga gttggct 37
<210> 19
<211> 36
<212> DNA
<213> 人工合成
<400> 19
taactcatat tgtagaagag tagaagttaa gtgtaa 36
<210> 20
<211> 41
<212> DNA
<213> 人工合成
<400> 20
acataccgca gacgggataa gggagataat aagagttggg t 41
<210> 21
<211> 39
<212> DNA
<213> 人工合成
<400> 21
taactcatat tgtagaagag tagaagccat aacctttcc 39
<210> 22
<211> 36
<212> DNA
<213> 人工合成
<400> 22
catcagctga gataagggag ataataagag ttgggt 36
<210> 23
<211> 35
<212> DNA
<213> 人工合成
<400> 23
taactcatat tgtagaagag tagaagacga ttgtg 35
<210> 24
<211> 26
<212> DNA
<213> 人工合成
<400> 24
acccaactct tattatctcc cttatc 26
<210> 25
<211> 26
<212> DNA
<213> 人工合成
<400> 25
taactcatat tgtagaagag tagaag 26
<210> 26
<211> 26
<212> DNA
<213> 人工合成
<400> 26
taactcatat tgtagaagag tagaag 26
<210> 27
<211> 26
<212> DNA
<213> 人工合成
<400> 27
agccaactcc tatgatctcc ctattg 26
<210> 28
<211> 22
<212> DNA
<213> 人工合成
<400> 28
ggggaacttc tcctgctaga at 22
<210> 29
<211> 20
<212> DNA
<213> 人工合成
<400> 29
ttgctgctgc ttgacagatt 20
<210> 30
<211> 22
<212> DNA
<213> 人工合成
<400> 30
cagacatttt gctctcaagc tg 22
<210> 31
<211> 21
<212> DNA
<213> 人工合成
<400> 31
ccctgtgggt tttacactta a 21
<210> 32
<211> 28
<212> DNA
<213> 人工合成
<400> 32
ccgtctgcgg tatgtggaaa ggttatgg 28
<210> 33
<211> 19
<212> DNA
<213> 人工合成
<400> 33
acgattgtgc atcagctga 19
- 一种利用L/RPA快速检测SARS-CoV-2的探针及引物组、试剂盒、检测方法
- 一种基于RPA方法快速检测非洲猪瘟病毒的引物组和探针、试剂盒以及检测方法
