使用逆转录病毒整合酶-Cas9融合蛋白通过定向非同源DNA插入进行的基因组编辑
文献发布时间:2023-06-19 12:19:35
相关申请的交叉引用
本申请要求2018年10月22日提交的美国临时申请序列号62/748,703的优先权,所述美国临时申请以引用的方式整体并入本文。
发明背景
CRISPR-Cas9大大提高了为基础研究和临床应用快速改变哺乳动物基因组的能力。CRISPR-Cas9使用向导RNA来将Cas9引导至特异性DNA靶序列,在其中它诱导双链DNA裂解并触发细胞修复途径,以引入移码突变或通过同源定向修复(HDR)插入供体序列。尽管取得了这些重大进展,但靶向递送大DNA序列以使用CRISPR-Cas9介导的HDR进行基因组编辑的效率仍然低下,需要含有显著侧翼同源性区域的供体模板并诱导p53 DNA损伤途径(Byrne等人,2015,NAR 43:e21;Happaniemi等人,2018,Nat Med 24:927-30;Ihry等人,2018,Nat Med 24:939-46)。总之,这些大大限制了CRISPR-Cas9基因组编辑的效率。因此,需要改进的整合基因组编辑。
相比之下,通过不需要靶序列同源性的方法,慢病毒酶整合酶(IN)对于催化大的慢病毒基因组插入宿主细胞DNA中而言是必要且足够的。IN介导的慢病毒DNA插入在几乎没有DNA靶序列特异性情况下发生,部分是由于其C末端结构域与DNA非特异性结合(Lutzke和Plasterk 1998,J Virol 72:4841-48)。
基因治疗技术的当前局限性阻止了大多数人类单基因疾病的治疗。CRISPR-Cas9基因编辑一直是开发治疗方法以校正哺乳动物基因组中的有害突变的新焦点。由于人类基因组内可能引起疾病和病症的众多患者特异性突变,这仍然是重大挑战。设计用于靶向外显子-内含子边界的CRISPR向导RNA可允许外显子跳跃策略以靶向这些突变组,然而,这些策略的功效仍有待测试并且不适用于所有患者。
许多基因的转基因表达可预防和逆转动物模型中的疾病结果,然而一些基因的大的尺寸大大超出了传统基因编辑方法(如CRISPR-Cas9)或传统病毒基因治疗方法如AAV(约4.9kb限制)的尺寸限制,从而阻止其用于人类基因疗法。使用通关AAV递送的较小工程化基因的方法目前正在临床试验中,然而这些策略是否提供长期恢复并且仅适用于具有特定突变的患者尚待确定。
相比之下,慢病毒载体能够递送大基因并通过整合到宿主基因组中允许永久性校正。然而,目前慢病毒整合的随机性质有可能导致脱靶突变和疾病,这阻止了其用于临床应用(Milone等人,2018,Leukemia 23:1529-41)。慢病毒序列通过病毒编码的酶整合酶(IN)插入宿主基因组中,所述酶利用基因组整合所需的非特异性DNA结合结构域(Andrake等人,2015,Annu Rev Virol 2:241-64)。
因此,需要改进的编辑基因组物质。本发明满足了这一需求。
发明内容
在一个方面,本发明提供了一种融合蛋白。在一个实施方案中,所述融合蛋白包含具有第一氨基酸序列的逆转录病毒整合酶(IN)或其片段;具有第二氨基酸序列的CRISPR相关(Cas)蛋白;以及具有第三氨基酸序列的核定位信号(NLS)。
在一个实施方案中,所述逆转录病毒IN选自由以下组成的组:人免疫缺陷病毒(HIV)IN、劳斯肉瘤病毒(RSV)IN、小鼠乳腺肿瘤病毒(MMTV)IN、莫洛尼鼠白血病病毒(MoLV)IN、牛白血病病毒(BLV)IN、人嗜T淋巴细胞病毒(HTLV)IN、禽肉瘤白血病病毒(ASLV)IN、猫白血病病毒(FLV)IN、异嗜性鼠白血病病毒相关病毒(XMLV)IN、猿猴免疫缺陷病毒(SIV)IN、猫免疫缺陷病毒(FIV)IN、马传染性贫血病毒(EIAV)IN、原型泡沫病毒(PFV)IN、猿猴泡沫病毒(SFV)IN、人泡沫病毒(HFV)IN、大眼鲈皮肤肉瘤病毒(WDSV)IN和牛免疫缺陷病毒(BIV)IN。
在一个实施方案中,所述逆转录病毒IN片段包含IN N末端结构域(NTD)和IN催化核心结构域(CCD)。在一个实施方案中,所述逆转录病毒IN包含与SEQ ID NO:1-40中的一者至少70%同一的序列。在一个实施方案中,所述逆转录病毒IN包含SEQ ID NO:1-40中的一者的序列。
在一个实施方案中,所述Cas蛋白选自由以下组成的组:Cas9、Cas13和Cpf1。在一个实施方案中,所述Cas蛋白是催化缺陷型的(dCas)。在一个实施方案中,所述Cas蛋白包含与SEQ ID NO:41-46中的一者至少95%同一的序列。在一个实施方案中,所述Cas蛋白包含SEQ ID NO:41-46中的一者的序列。
在一个实施方案中,所述NLS是逆转录转座子NLS。在一个实施方案中,所述逆转录转座子NLS是Ty1或Ty2 NLS。在一个实施方案中,所述NLS是Ty1样NLS。在一个实施方案中,所述NLS包含与SEQ ID NO:47-56、254-257和275-887中的一者至少70%同一的序列。在一个实施方案中,所述NLS包含SEQ ID NO:47-56、254-257和275-887中的一者的序列。
在一个实施方案中,所述融合蛋白包含与SEQ ID NO:57-98中的一者至少70%同一的序列。在一个实施方案中,所述融合蛋白包含SEQ ID NO:57-98中的一者的序列。
在一个方面,本发明提供了一种编码本发明的融合蛋白的核酸。在一个实施方案中,所述核酸包含与SEQ ID NO:155-196中的一者至少70%同一的序列。在一个实施方案中,所述核酸包含选自SEQ ID NO:155-196的序列。
在一个方面,本发明提供了一种编辑遗传物质的方法。在一个实施方案中,所述方法包括向所述遗传物质施用:(a)本发明的融合蛋白或编码本发明的融合蛋白的核酸分子,(b)包含与所述遗传物质中的靶区域互补的靶向核苷酸序列的向导核酸,以及(c)包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,所述编辑遗传物质的方法是体外方法。在一个实施方案中,所述编辑遗传物质的方法是体内方法。
在一个方面,本发明提供了一种用于编辑遗传物质的系统。在一个实施方案中,所述系统在一种或多种载体中包含:(a)编码本发明的融合蛋白的核酸序列,(b)编码CRISPR-Cas系统向导RNA的核酸序列,和(c)编码供体模板核酸的核酸序列,其中所述供体模板核酸包含U3序列、U5序列和供体模板序列。在一个实施方案中,所述融合蛋白包含逆转录病毒整合酶(IN)或其片段;CRISPR相关(Cas)蛋白和核定位信号(NLS)。在一个实施方案中,所述核酸位于同一载体上。在一个实施方案中,所述核酸位于不同的载体上。
在一个实施方案中,所述CRISPR-Cas系统向导RNA基本上与所述基因中的靶DNA序列杂交。在一个实施方案中,所述U3序列和U5序列对所述逆转录病毒IN具有特异性。
在方面,本发明提供了一种用于递送基因组编辑组分的系统。在一个实施方案中,所述系统包含:(a)包装质粒,所述包装质粒包含编码gag-pol多蛋白的序列,所述gag-pol多蛋白包含与催化性死亡Cas(dCas)蛋白融合的整合酶;(b)转移质粒,所述转移质粒包含编码供体序列的序列、5’LTR和3’LTR;以及(c)包膜质粒,所述包膜质粒包含编码包膜蛋白的核酸序列。在一个实施方案中,所述包装质粒还包含编码向导RNA序列的序列。
在一个实施方案中,所述系统包含:(a)包装质粒,所述包装质粒包含编码gag-pol多蛋白的序列;(b)转移质粒,所述转移质粒包含编码供体序列的序列、5’LTR和3’LTR;(c)包膜质粒,所述包膜质粒包含编码包膜蛋白的核酸序列;以及(d)VPR-IN-dCas质粒,所述VPR-IN-dCas质粒包含编码融合蛋白的核酸序列,所述融合蛋白包含VPR、整合酶和催化性死亡Cas(dCas)。在一个实施方案中,所述VPR-IN-dCas质粒还包含编码向导RNA序列的序列。
在一个实施方案中,所述系统包含(a)包装质粒,所述包装质粒包含编码gag-pol多蛋白的核酸序列;(b)转移质粒,所述转移质粒包含编码向导RNA的核酸序列、包含整合酶和催化性死亡Cas的融合蛋白、5’LTR和3’LTR;以及(c)包膜质粒,所述包膜质粒包含编码包膜蛋白的核酸序列。
附图说明
在结合附图阅读时,将更好地理解本发明的实施方案的以下详细描述。然而,应理解,本发明不限于附图中所示的实施方案的精确安排和手段。
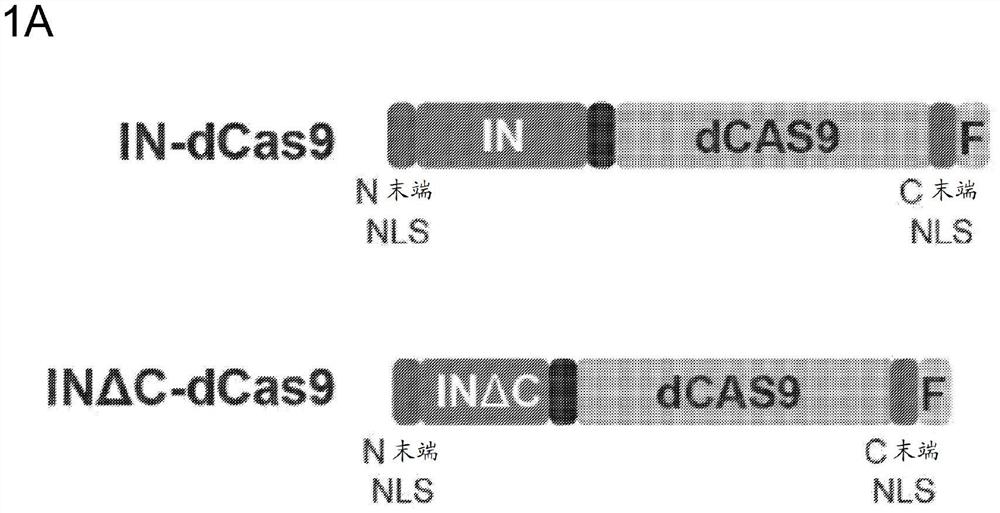
图1,包括图1A至图1C,描绘实验结果,所述实验结果证明用于编辑哺乳动物基因组DNA的逆转录病毒整合酶-dCas9融合蛋白的核定位增强。图1A描绘IN-dCas9融合蛋白的示意图。图1B描绘IN-dCas9融合蛋白的核定位。图1C描绘实验结果,所述实验结果证明INΔC-dCas9融合蛋白整合靶向HEK293细胞中EF1-α的3’UTRE的IRES-mCherry模板的酶活性。
图2描绘核酸编辑技术的示意图,其示出病毒整合酶(IN)与CRISPR-dCas9的融合物允许以靶标特异性方式整合大DNA序列。这种方法允许通常超过非整合AAV载体限制的大基因序列的安全且永久性递送。
图3描绘用于定量和表征哺乳动物细胞中的单独整合事件的保真度的GFP报告基因细胞系的实验设计和实验结果。
图4描绘CRISPER-Cas9介导的同源定向修复和逆转录病毒整合酶介导的随机DNA整合的示意图。
图5描绘整合酶-Cas基因组编辑的示意图。
图6描绘供体载体、产生平末端模板和产生3'-加工模板的示意图。
图7描绘INsrt模板的共转染的实验设计,将靶向amilCP序列的IN-dCas9载体共转染到Cos7细胞中。
图8描绘对人EF1-α基因座的3'UTR具有特异性的成对向导RNA将IGR-mCherry-2A-嘌呤霉素-pA盒敲入人HEK293细胞系中的实验设计,以及转染后48小时mCherry-阳性细胞的图像。
图9描绘证明定向编辑的示意图。
图10描绘示意图,所述示意图展示用于产生floxed等位基因的多重基因组编辑。
图11,包括图11A至图11C,描绘实验结果,所述实验结果证明Ty1 NLS样序列对INΔC-Cas9融合蛋白的核定位的效率。图11A描绘使用抗FLAG抗体检测含有在Cos-7细胞中表达的C末端经典SV40、Ty1或Ty2 NLS的INΔC-dCas9融合蛋白。图11B描绘从酵母蛋白分离的Ty1 NLS样序列可提供稳健核定位(MAK11)或无明显定位活性(INO4和STH1)。图11C描绘Ty1、Ty2和Ty1 NLS样序列的序列。Ty1和Ty2在长度和残基组成上均高度保守。比例尺=10μm。
图12,包括图12A至图12C,描绘实验结果,所述实验结果证明Ty1 NLS增强哺乳动物细胞中的Cas9 DNA编辑。图12A描绘px330 CRISPR-Cas9表达质粒的图,所述质粒编码hU6驱动的单向导RNA(sgRNA)和含有N末端3x FLAG标签、SV40 NLS和C末端NPM NLS的CAG驱动的Cas9蛋白。克隆Ty1 NLS代替px330中的NPM NLS(px330-Ty1)。图12B描绘产生移码激活的荧光素酶报告基因,其中上游20nt靶序列(ts)中断从下游荧光素酶开放阅读框的开放阅读。通过非同源末端连接(NHEJ)诱导的移码重构下游报告基因,并且允许荧光素酶表达。图12C描绘移码响应性荧光素酶报告基因和px330的共表达产生相对于非靶向sgRNA,荧光素酶活性的约20倍激活,所述px330含有对靶序列具有特异性的单向导RNA。px330-Ty1的共表达产生相对于px330的约44%增强。
图13,包括图13A至图13E,描绘用于编辑的基因组靶向策略。可取决于所需的应用使DNA供体序列的整合靶向不同的基因组位置。图13A描绘可使携带基因盒的DNA供体序列的递送靶向基因间‘安全港(safe harbor)’基因座,以防止破坏相邻或必需基因表达。图13B描绘可使携带基因盒的DNA供体序列的递送靶向非必需‘安全港(safe harbor)’基因座,以防止破坏相邻或必需基因表达。图13C描绘可将编码剪接受体序列(SA)的DNA序列的整合递送至基因(例如,疾病基因基因座)的内含子区域,这将允许整合序列的表达并阻止下游序列的表达。图13D描绘可将编码剪接受体序列(SA)的DNA序列的整合递送至基因(例如,疾病基因基因座)的内含子区域,这将允许整合序列的表达并阻止下游序列的表达。图13E描绘将含有内部核糖体进入序列(IRES)的DNA供体序列整合到3’UTR中可允许表达而不破坏来自内源基因座的表达。
图14描绘慢病毒生命周期的图。慢病毒(逆转录病毒的一个亚类)是单链RNA病毒,其将其前病毒基因组的永久双链DNA(dsDNA)拷贝整合到宿主细胞DNA中。在病毒转导后,慢病毒RNA基因组通过病毒编码的逆转录酶(RT)复制为平末端dsDNA,并通过整合酶I(IN)插入宿主基因组中。慢病毒基因组在其U3和U5末端侧接短(约20个碱基对)的序列基序,所述序列基序是IN进行前病毒基因组整合所必需的。IN介导的逆转录病毒DNA插入在几乎没有DNA靶序列特异性的情况下发生,并且可整合到活性基因基因座中,其可破坏正常基因功能并有可能导致人类疾病。
图15,包括图15A至图15E,描绘哺乳动物细胞中的基因组编辑。慢病毒整合酶与dCas9的融合允许含有短病毒末端的供体DNA序列的靶向非同源插入。图15A描绘编码人U6驱动的单向导RNA(sgRNA)和整合酶-dCas9融合蛋白的哺乳动物表达载体的图。图15B描绘显示含有侧接U3/U5病毒基序的IGR IRES-mCherry-2A-嘌呤霉素(puro)盒的dsDNA供体模板的图。图15C描绘这种供体模板的整合酶-Cas9介导的整合到在COS-7细胞中稳定表达的CMV-eGFP报告基因中的示意图。图15D描绘示意图,所述示意图展示这种供体模板的整合酶-Cas9介导的整合到在COS-7中稳定表达的CMV-eGFP报告基因转基因种可导致eGFP表达的破坏,同时允许mCherry表达。图15E描绘实验结果,所述实验结果证明在编辑的COS-7细胞中eGFP表达的损失和mCherry表达的获得。
图16,包括图16A至图16C,描绘传统慢病毒基因递送系统。图16A描绘慢病毒基因组的图,所述慢病毒基因组在侧翼长末端重复序列(LTR)之间编码病毒蛋白。图16B和图16C描绘示意图,所述示意图证明慢病毒基因组已被用作稳健基因传递工具。慢病毒颗粒可用于包装、递送和稳定表达供体转基因序列。对于慢病毒载体基因表达系统,将病毒多蛋白从病毒基因组中除去,并使用单独的哺乳动物表达质粒表达。然后可克隆供体DNA序列以代替侧翼LTR序列之间的病毒多蛋白。这些载体在哺乳动物包装细胞中的共转染允许形成能够递送和整合所编码的供体序列的慢病毒颗粒,然而不需要整合酶和随后病毒繁殖所必需的其它病毒蛋白的编码信息。慢病毒颗粒是用于递送病毒蛋白(例如整合酶和逆转录酶)和dsDNA供体序列两者的天然载体,其含有整合酶介导的插入哺乳动物细胞中所需的必要病毒末端序列。图16B描绘慢病毒载体的产生。图16C描绘递送并稳定表达供体转基因序列的慢病毒颗粒的转导。
图17,包括图17A至图17C,描绘靶向慢病毒整合。为了用于哺乳动物细胞中的基因组编辑的靶向慢病毒供体模板整合的目的,可修改现有慢病毒递送系统以并入编辑组分。图17A描绘一种方法,其中将dCas9直接融合至编码gag-pol多蛋白的慢病毒包装质粒(例如psPax2)内的整合酶(或缺乏其C末端非特异性DNA结合结构域的整合酶)。图17B描绘将修饰的gag-pol多蛋白与其它病毒组分一起翻译为多蛋白,负载向导RNA并包装到慢病毒颗粒中。对于这种方法,IN-dCas9融合蛋白保留蛋白酶裂解(PR)所需的序列,并且因此在颗粒成熟过程中通常从gag-pol多蛋白裂解。哺乳动物细胞的转导产生病毒蛋白,包括IN-dCas9融合蛋白、sgRNA和慢病毒供体序列的递送。图17C描绘在慢病毒转导后,ssRNA基因组通过逆转录酶的逆转录产生含有正确病毒末端序列(U3和U5)的dsDNA序列,所述dsDNA序列通过IN-dCas9融合蛋白被整合到哺乳动物基因组中。
图18,包括图18A至图18C,描绘通过与病毒蛋白融合的靶向慢病毒整合。图18A描绘IN-dCas9表达和包装为与病毒蛋白(例如,病毒蛋白R,VPR)的N末端和C末端融合物,作为实现靶向慢病毒基因整合的一种方法。在VPR与IN-dCas9融合蛋白之间包含病毒蛋白酶裂解序列,因此成熟后,IN-dCas9将从VPR释放。图18B描绘包装细胞与慢病毒组分的共转染产生含有VPR-IN-dCas9蛋白和sgRNA的病毒颗粒。病毒颗粒形成所需的包装质粒(例如psPax2)在整合酶中含有突变,以抑制其在包装质粒背景下的催化活性,从而防止非整合酶-Cas9介导的整合。图18C描绘在病毒转导后,IN-dCas9蛋白被递送为蛋白质并介导慢病毒供体序列的整合。递送IN-dCas9融合物和作为核糖蛋白的sgRNA的益处是它仅在靶细胞中瞬时表达。
图19,包括图19A至图19C,描绘经由并入到转移质粒中的靶向慢病毒整合。图19A描绘从病毒转移质粒(或其它病毒载体,如AAV)中表达IN-dCas9融合蛋白和/或向导RNA是实现靶向慢病毒基因整合的一种方法。图19B描绘在这种方法中,将含有IN-dCas9融合蛋白和sgRNA的转移质粒与产生慢病毒颗粒所需的包装质粒和包膜质粒共转染。如果使用慢病毒,则包装质粒在整合酶中含有催化突变,以抑制非特异性整合。图19C描绘在转导哺乳动物细胞后,IN-dCas9融合蛋白和sgRNA的表达产生能够靶向其自身的病毒供体载体以进行靶向整合(自我整合)的组分。这种方法用于靶向基因破坏或用作基因驱动。
图20,包括图20A至图20D,描绘慢病毒供体序列的共同递送。图20A描绘与编码供体DNA序列的慢病毒颗粒共转导可充当整合的供体模板。图20B和图20C描绘,通过使用来自不同逆转录病毒家族成员的整合酶及其相应的转移质粒,可实现以这种方法阻止其自身病毒编码序列的自我整合。图20B描绘编码IN(FIV)-dCas9融合蛋白的HIV慢病毒颗粒的产生。图20C描绘包含FIV转移质粒的FIV慢病毒颗粒的产生。图20D描绘利用编码IN(FIV)-dCas9融合蛋白的HIV慢病毒颗粒来整合在FIV慢病毒颗粒中编码的FIV供体模板。
图21描绘原代哺乳动物细胞中的靶向慢病毒整合。此数据证明将编码IRES-tdTO盒的慢病毒供体模板慢病毒包装、递送和靶向整合到小鼠胚胎成纤维细胞的ROSA26
图22描绘哺乳动物稳定细胞系中的靶向慢病毒整合。此数据证明将编码IRES-tdTO盒的慢病毒供体模板慢病毒包装、递送和靶向整合到COS-7细胞中的稳定表达的CMV-eGFP中。
图23,包括图23A至图23C,描绘用于慢病毒颗粒的靶向整合的DNA结合结构域。用dCas9的可编程DNA结合结构域代替整合酶的非特异性DNA结合结构域允许经由慢病毒颗粒中的递送进行dsDNA供体模板的靶向整合。替代DNA结合结构域(如TALEN)可作为与病毒整合酶的融合物用于靶向整合。使用类似的慢病毒产生方法,用靶向特定序列的TALEN代替先前包装策略中的dCas9。图23A描绘在gag-pol多蛋白的背景下作为与整合酶的融合物包装和递送的TALEN。图23B描绘作为与整合酶(作为与病毒蛋白的融合物)的融合物包装和递送的TALEN。图23C描绘作为与在转移质粒内编码的整合酶的融合物包装和递送的TALEN。
图24,包括图24A至图24C,描绘实验结果,所述实验结果证明Ty1 NLS增强哺乳动物细胞中的Cas9 DNA编辑。图24A描绘px330 CRISPR-Cas9表达质粒的图,所述质粒编码hU6驱动的单向导RNA(sgRNA)和含有N末端3x FLAG标签、SV40 NLS和C末端NPM NLS的CAG驱动的Cas9蛋白。克隆Ty1 NLS代替px330中的NPM NLS(px330-Ty1)。图24B描绘结果,所述结果证明产生移码激活的荧光素酶报告基因,其中上游20nt靶序列(ts)中断从下游荧光素酶开放阅读框的开放阅读。通过非同源末端连接(NHEJ)诱导的移码重构下游报告基因,并且允许荧光素酶表达。图24C描绘结果,所述结果证明移码响应性荧光素酶报告基因和px330的共表达产生相对于非靶向sgRNA,荧光素酶活性的约20倍激活,所述px330含有对靶序列具有特异性的单向导RNA。px330-Ty1的共表达产生相对于px330的约44%增强。
图25描绘示意图,所述示意图证明TALEN可用于指导逆转录病毒整合酶介导的供体DNA模板整合。
图26描绘质粒DNA整合测定的示意图。
图27描绘实验数据,所述实验数据证明相隔16bp的TALEN对产生多约6倍的氯霉素抗性菌落,而相隔28bp的TALEN对与非靶向整合酶相似。
图29,包括图29A至图29C,描绘实验结果。图29A描绘在大肠杆菌中表达amilCP色蛋白产生紫色大肠杆菌(白色箭头)。整合酶-Cas介导的含有病毒末端的供体序列整合破坏amilCP表达(橙色箭头)(在卡那霉素板上生长)。图29B描绘具有平末端(ScaI裂解的)或3'加工模拟物(FauI裂解的)末端的Insrt IGR-CAT供体模板整合到哺乳动物细胞中的pCRII-amilCP报告基因中。令人感兴趣地,作为与dCas9的融合物,C末端非特异性DNA结合结构域的缺失不会抑制整合酶-Cas介导的整合。使用模拟3'加工的末端显示出CAT抗性克隆增加约2倍。图29C描绘对质粒DNA中整合酶-Cas介导的整合的整合酶突变的评估。使用IGR-CAT供体模板的双向导RNA靶向整合到amilCP中,抑制二聚化的突变(E85G和E85F)不会破坏整合酶-Cas介导的整合。然而,IN E87G突变不能通过配对的靶向sgRNA挽救。令人感兴趣地,与dCas9的串联INΔC融合物(tdINΔC-dCas9)显示增强约2倍的整合。
具体实施方式
本发明涉及融合蛋白、编码融合蛋白的核酸、用于编辑遗传物质的系统和方法。在一个实施方案中,本发明涉及逆转录病毒整合酶(IN)-CRISPR相关(Cas)融合蛋白和编码逆转录病毒IN-Cas融合蛋白的核酸分子。在一个实施方案中,IN-Cas融合蛋白还包含核定位信号(NLS)。
本发明的融合蛋白、核酸分子、系统和方法具有将供体DNA序列递送至靶基因组位置的能力。此外,本发明消除了对同源臂的需要,并且依靠由向导RNA进行的靶向,从而大大简化了编辑遗传物质。
在一个方面,本发明提供了一种IN-Cas融合蛋白。在一个实施方案中,所述融合蛋白包含具有第一氨基酸序列的逆转录病毒IN或其片段;具有第二氨基酸序列的Cas蛋白;以及具有第三氨基酸序列的NLS。
在一个方面,本发明提供了一种编码IN-Cas融合蛋白的核酸分子。在一个实施方案中,所述核酸分子包含编码逆转录病毒IN或其片段的第一核酸序列;编码Cas蛋白的第二核酸序列;以及编码NLS的第三核酸序列。
在一个实施方案中,逆转录病毒IN可以是人免疫缺陷病毒(HIV)IN、劳斯肉瘤病毒(RSV)IN、小鼠乳腺肿瘤病毒(MMTV)IN、莫洛尼鼠白血病病毒(MoLV)IN、牛白血病病毒(BLV)IN、人嗜T淋巴细胞病毒(HTLV)IN、禽肉瘤白血病病毒(ASLV)IN、猫白血病病毒(FLV)IN、异嗜性鼠白血病病毒相关病毒(XMLV)IN、猿猴免疫缺陷病毒(SIV)IN、猫免疫缺陷病毒(FIV)IN、马传染性贫血病毒(EIAV)IN、原型泡沫病毒(PFV)IN、猿猴泡沫病毒(SFV)IN、人泡沫病毒(HFV)IN、大眼鲈皮肤肉瘤病毒(WDSV)IN或牛免疫缺陷病毒(BIV)IN。在一个实施方案中,Cas蛋白是Cas9或Cpf1。在一个实施方案中,NLS是逆转录转座子NLS,如Ty1 NLS。在一个实施方案中,逆转录转座子NLS增加核定位。
在一个方面,本发明提供了一种用于编辑遗传物质的系统。在一个实施方案中,所述系统在一种或多种载体中包含:编码融合蛋白的核酸序列,其中所述融合蛋白包含逆转录病毒IN或其片段、Cas蛋白和NLS;编码CRISPR-Cas系统向导RNA的核酸序列;以及编码供体模板核酸的核酸序列,其中所述供体模板核酸包含U3序列、U5序列和供体模板序列。
在一个方面,本发明提供了一种用于编辑遗传物质的方法。在一个实施方案中,所述方法包括施用本发明的核酸分子;包含与基因中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。
除非另外定义,否则本文所用的所有技术和科学术语都具有与本发明所属领域中的普通技术人员通常所理解的相同含义。
一般来说,本文所用的命名法和细胞培养、分子遗传学、有机化学和核酸化学以及杂交中的实验室程序是本领域中众所周知并普遍采用的那些。
标准技术用于核酸和肽合成。通常根据本领域的常规方法和各种一般参考文献(例如,Sambrook和Russell,2012,Molecular Cloning,A Laboratory Approach,ColdSpring Harbor Press,Cold Spring Harbor,NY;和Ausubel等人,2012,CurrentProtocols in Molecular Biology,John Wiley&Sons,NY)来进行所述技术和程序,所述文献在本文档中提供。
本文所用的命名法以及下文所述的分析化学和有机合成中所用的实验室程序是本领域众所周知并普遍采用的那些。标准技术或其修改用于化学合成和化学分析。
除非本文另外指示或者与上下文含义明显相悖,否则在本发明的上下文中(尤其在权利要求书的上下文中)使用的术语“一个/种(a/an)”、“所述”以及类似的术语应解释为涵盖单数和复数两者。
当涉及可测量的值(如量、持续时间等)时,如本文所用的“约”是指涵盖与指定值的±20%、或±10%、或±5%、或±1%或或±0.1%的偏差,因为此类偏差对于执行所公开的方法是适当的。
“反义”特别是指编码蛋白质的双链DNA分子的非编码链的核酸序列,或与所述非编码链基本上同源的序列。如本文所定义,反义序列与编码蛋白质的双链DNA分子的序列互补。反义序列不必仅与DNA分子的编码链的编码部分互补。反义序列可与编码蛋白质的DNA分子的编码链上指定的调控序列互补,所述调控序列控制编码序列的表达。
“疾病”是动物的一种健康状态,其中所述动物不能维持体内平衡,并且其中如果所述疾病得不到改善,则动物的健康状况将继续恶化。
相比之下,动物的“病症”是所述动物能够维持体内平衡但动物的健康状态比没有病症的情况下不利的一种健康状态。如果不进行治疗,病症不一定会导致动物的健康状态进一步下降。
如果疾病或病症的体征或症状的严重程度、患者经历这种体征或症状的频率或两者均降低,则疾病或病症得以“减轻”。
“编码”是指多核苷酸(如基因、cDNA或mRNA)中的核苷酸的特定序列充当在生物学过程中合成具有限定的核苷酸序列(即,rRNA、tRNA和mRNA)或限定的氨基酸序列的其它聚合物和大分子的模板的固有性质和由此产生的生物学性质。因此,基因编码蛋白质,如果对应于所述基因的mRNA的转录和翻译在细胞或其它生物系统中产生所述蛋白质的话。编码链(其核苷酸序列与mRNA序列相同并且通常提供在序列表中)和非编码链(用作基因或cDNA的转录模板)两者均可称为编码所述基因或cDNA的蛋白质或其它产物。
术语“患者”、“受试者”、“个体”等在本文中可互换使用,并且是指无论在体外或是在体内可顺从本文所述方法的任何动物或其细胞。在某些非限制性实施方案中,患者、受试者或个体是人。
如本文关于抗体使用的术语“特异性地结合”是指识别特定抗原但基本上不识别或结合样品中的其它分子的抗体。例如,特异性地结合至来自一种物种的抗原的抗体也可结合至来自一种或多种物种的所述抗原。但是,这种跨物种反应性本身并不改变抗体分类为特异性的。在另一实例中,特异性地结合至抗原的抗体也可结合至所述抗原的不同等位基因形式。然而,此交叉反应性本身并不改变抗体分类为特异性的。
在一些情况下,术语“特异性结合”或“特异性地结合”可关于抗体、蛋白质或具有第二化学物质的肽的相互作用被用来指所述相互作用依赖于所述化学物质上特定结构(例如,抗原决定簇或表位)的存在;例如,抗体识别并结合至特定的蛋白质结构,而不是广泛地结合至蛋白质。如果抗体对表位“A”具有特异性,则在含有标记的“A”和抗体的反应中,含有表位A(或游离的未标记A)的分子的存在将降低结合至所述抗体的标记的A的量。
基因的“编码区”由分别与mRNA分子的编码区同源或互补的所述基因的编码链的核苷酸残基和所述基因的非编码链的核苷酸组成,mRNA分子的编码区通过基因转录产生。
mRNA分子的“编码区”还由mRNA分子的核苷酸残基组成,所述核苷酸残基在mRNA分子的翻译过程中与转移RNA分子的反密码子区匹配或编码终止密码子。因此,编码区可包含核苷酸残基,所述核苷酸残基包含不存在于由mRNA分子编码的成熟蛋白质中的氨基酸残基(例如,蛋白质输出信号序列中的氨基酸残基)的密码子。
如本文关于核酸使用的“互补”是指两个核酸链的区域之间或同一核酸链的两个区域之间的序列互补性的广义概念。已知第一核酸区域的腺嘌呤残基能够与第二核酸区域的残基形成特异性氢键(“碱基配对”),如果第二核酸区域的残基是胸腺嘧啶或尿嘧啶,则所述第二核酸区域与所述第一区域反向平行。类似地,已知第一核酸链的胞嘧啶残基能够与第二核酸链的残基碱基配对,如果所述第二核酸链的残基是鸟嘌呤,则所述第二核酸链与所述第一链反向平行。如果当两个区域以反向平行方式排列时,第一区域的至少一个核苷酸残基能够与第二区域的残基碱基配对,则核酸的第一区域与相同或不同核酸的第二区域互补。在一个实施方案中,所述第一区域包含第一部分并且所述第二区域包括第二部分,由此,当所述第一部分和第二部分以反向平行方式排列时,所述第一部分的至少约50%、至少约75%、至少约90%或至少约95%的核苷酸残基能够与所述第二部分中的核苷酸残基碱基配对。在一个实施方案中,所述第一部分的所有核苷酸残基能够与所述第二部分中的核苷酸残基碱基配对。
如本文所用的术语“DNA”被定义为脱氧核糖核酸。
如本文所用的术语“表达”被定义为特定核苷酸序列由其启动子驱动的转录和/或翻译。
如本文所用的术语“表达载体”是指含有编码能够被转录的基因产物的至少一部分的核酸序列的载体。在一些情况下,RNA分子然后被翻译成蛋白质、多肽或肽。在其它情况下,这些序列在例如反义分子、siRNA、核酶等的产生中不翻译。表达载体可含有多种控制序列,所述控制序列是指在特定宿主生物体中可操作连接的编码序列的转录和可能地翻译所必需的核酸序列。除了控制转录和翻译的控制序列外,载体和表达载体还可含有具有其它功能的核酸序列。
如本文所用的术语“野生型”是本领域技术人员所理解的术语,并且表示生物体、菌株、基因的典型形式或者当它在自然界存在时区别于突变体或变体形式的特征。
术语“同源性”是指互补程度。可存在部分同源性或完全同源性(即,同一性)。同源性通常使用序列分析软件(例如,遗传学计算机组的序列分析软件包.威斯康星大学生物技术中心.1710University Avenue.Madison,Wis.53705)来测量。这种软件通过对不同的取代、缺失、插入以及其它修饰的同源性的程度进行赋值而将相似的序列进行匹配。保守取代通常包括以下组内的取代:甘氨酸,丙氨酸;缬氨酸,异亮氨酸,亮氨酸;天冬氨酸,谷氨酸,天冬酰胺,谷氨酰胺;丝氨酸,苏氨酸;赖氨酸,精氨酸;以及苯丙氨酸,酪氨酸。
“分离的”是指从天然状态发生改变或去除。例如,在活动物体中在其正常环境中天然存在的核酸或肽不是“分离的”,但部分地或完全地从其天然环境的共存物质分离出来的相同核酸或肽是“分离的”。分离的核酸或蛋白质可以大致上纯化的形式存在,或者可存在于非天然环境(例如,宿主细胞)中。
当关于核酸使用时,如“分离的寡核苷酸”或“分离的多核苷酸”中的术语“分离的”是指从在其来源中通常与其缔合的至少一种污染物鉴定并分离的核酸序列。因此,分离的核酸以与自然界中发现的不同的形式或环境存在。相比之下,非分离的核酸(例如DNA和RNA)以它们在自然界中存在的状态被发现。例如,给定DNA序列(例如基因)在靠近相邻基因的宿主细胞染色体上发现;RNA序列(例如,编码特定蛋白质的特定mRNA序列)在细胞中以与编码多种蛋白质的许多其它mRNA的混合物形式发现。然而,例如,分离的核酸在通常表达所述核酸的细胞中包含这种核酸,其中所述核酸处于与天然细胞的染色体位置不同的染色体位置中,或者另外侧接与自然界中发现的不同的核酸序列。分离的核酸或寡核苷酸可以单链或双链形式存在。当使用分离的核酸或寡核苷酸来表达蛋白质时,所述寡核苷酸至少含有有义链或编码链(即,寡核苷酸可以是单链的),但可含有有义链和反义链两者(即,寡核苷酸可以是双链的)。
当关于多肽使用时,如“分离的蛋白质”或“分离的多肽”中的术语“分离的”是指从在其来源中通常与其缔合的至少一种污染物鉴定并分离的多肽。因此,分离的多肽以与自然界中发现的不同的形式或环境存在。相比之下,非分离的多肽(例如蛋白质和酶)以它们在自然界中存在的状态被发现。
“核酸”是指任何核酸,无论是由脱氧核糖核苷还是核糖核苷组成,以及无论是由磷酸二酯键联还是经修饰的键联组成,如磷酸三酯、氨基磷酸酯、硅氧烷、碳酸酯、羧甲基酯、乙酰胺酯、氨基甲酸酯、硫醚、桥联氨基磷酸酯、桥联亚甲基膦酸酯、硫代磷酸酯、甲基膦酸酯、二硫代磷酸酯、桥联硫代磷酸酯或砜键联以及此类键联的组合。术语核酸还具体地包括由不同于五种生物学上存在的碱基(腺嘌呤、鸟嘌呤、胸腺嘧啶、胞嘧啶和尿嘧啶)的碱基组成的核酸。术语“核酸”通常是指大的多核苷酸。
本文中使用常规符号来描述多核苷酸序列:单链多核苷酸序列的左端是5'-端;将双链多核苷酸序列的左侧方向称为5'-方向。
将核苷酸的5'至3'添加至新生RNA转录物的方向称为转录方向。与mRNA具有相同序列的DNA链被称为“编码链”;DNA链上位于DNA上的参考点5'的序列被称为“上游序列”;DNA链上为DNA上的参考点3'的序列被称为“下游序列”。
“表达盒”是指核酸分子,所述核酸分子包含可操作地连接至编码序列的转录和任选地翻译所必需的启动子/调控序列的编码序列。
如本文所用的术语“可操作地连接”是指核酸序列的连接处于这种方式,所述方式使得产生能够指导给定基因的转录和/或所续蛋白质分子的合成的核酸分子。所述术语还指编码氨基酸的序列的连接处于这种方式,所述方式使得产生功能性(例如酶促活性、能够与结合配偶体结合、能够抑制等)蛋白质或多肽。
如本文所用,术语“启动子/调控序列”意指表达可操作地连接至启动子/调控因子序列的基因产物所需的核酸序列。在一些情况下,此序列可以是核心启动子序列,并且在其它情况下,此序列还可包含增强子序列和表达基因产物所需的其它调控元件。启动子/调控序列可以是例如以诱导型方式表达基因产物的启动子/调控序列。
如本文所用,用于杂交的“严格条件”是指与靶序列具有互补性的核酸主要与所述靶序列杂交并且基本上不与非靶序列杂交的条件。严格条件通常是序列依赖性的,并且取决于许多因素而变化。一般来说,序列越长,序列与其靶序列特异性杂交的温度越高。严格条件的非限制性实例在Tijssen(1993),Laboratory Techniques In Biochemistry AndMolecular Biology-Hybridization With Nucleic Acid Probes第1部分,第二章“Overview of principles of hybridization and the strategy of nucleic acidprobe assay”,Elsevier,N.Y.中详细描述。
“杂交”是指一种或多种多核苷酸反应形成经由核苷酸残基的碱基之间的氢键合而稳定的复合物的反应。氢键合可通过沃森/克里克碱基配对、Hoogstein结合或以任何其它序列特异性方式发生。所述复合物可包含形成双链体结构的两条链,形成多链复合物的三条或更多条链,单一自我杂交链,或这些的任何组合。杂交反应可构成更广泛过程中的步骤,如PCR的起始或酶对多核苷酸的裂解。能够与给定序列杂交的序列被称为给定序列的“互补序列”。
“诱导型”启动子是当与编码或指定基因产物的多核苷酸可操作地连接时致使基因产物基本上仅在对应于启动子的诱导物存在时产生的核苷酸序列。
“组成型”启动子是当与编码或指定基因产物的多核苷酸可操作地连接时致使基因产物在细胞的大多数或全部生理条件下在细胞中产生的核苷酸序列。
如本文所用的术语“多肽”被定义为核苷酸的链。此外,核酸是核苷酸的聚合物。因此,如本文所用的核酸和多核苷酸是可互换的。本领域的技术人员具备核酸是可水解为单体“核苷酸”的多核苷酸的一般知识。单体核苷酸可水解为核苷。如本文所用,多核苷酸包含但不限于通过本领域中可用的任何手段获得的所有核酸序列,所述手段包括但不限于重组手段(即,克隆来自重组文库或细胞基因组的核酸序列、使用普通的克隆技术和PCR等)和通过合成手段。
在本发明的上下文中,使用普遍存在的核酸碱基的以下缩写。“A”是指腺苷,“C”是指胞嘧啶,“G”是指鸟苷,“T”是指胸苷,并且“U”是指尿苷。
如本文所用,术语“肽”、“多肽”和“蛋白质”可互换使用,并且是指由通过肽键共价连接的氨基酸残基构成的化合物。蛋白质或肽必须含有至少两个氨基酸并且对于可构成蛋白质的或肽的序列的氨基酸的最大数目没有设限。多肽包括含有通过肽键彼此连接的两个或更多个氨基酸的任何肽或蛋白质。如本文所用,所述术语是指短链和长链,所述短链在本领域中还通常称为例如肽、寡肽和低聚物,所述长链在本领域中通常称为蛋白质,其中存在许多种类型。“多肽”包括例如生物活性片段、基本上同源的多肽、寡肽、同源二聚体、异源二聚体、多肽的变体、修饰的多肽、衍生物、类似物、融合蛋白等。多肽包括天然肽、重组肽、合成肽或其组合。
如本文所用的术语“RNA”被定义为核糖核酸。
“重组多核苷酸”是指具有不天然连接在一起的序列的多核苷酸。扩增或组装的重组多核苷酸可包含在合适的载体中,并且所述载体可用于转化合适的宿主细胞。
重组多核苷酸也可起到非编码功能(例如启动子、复制起点、核糖体结合位点等)的作用。
如本文所用的术语“重组多肽”被定义为通过使用重组DNA方法产生的多肽。
如本文所用,“转录激活因子样效应物核酸酶(TALEN)”是通过将TAL效应子DNA结合结构域融合至DNA裂解结构域而产生的人工限制性酶。这些试剂可实现高效、可编程且特异性的DNA裂解,并且代表用于原位编辑遗传物质的强大工具。转录激活因子样效应物(TALE)可快速工程化以结合几乎任何DNA序列。如本文所用,术语TALEN是广泛的并且包括可在没有另一TALEN帮助的情况下裂解双链DNA的单体TALEN。术语TALEN也可用来指一对TALEN中的一者或两个成员,所述TALEN被工程化为共同作用以在同一位点裂解DNA。共同作用的TALEN可被称为左-TALEN和右-TALEN,它们引用了DNA的偏手性。参见美国序列号12/965,590;美国序列号13/426,991(美国专利号8,450,471);美国序列号13/427,040(美国专利号8,440,431);美国序列号13/427,137(美国专利号8,440,432);以及美国序列号13/738,381,其全部以引用的方式整体并入本文)。
如所述术语在本文所用,“变体”是序列分别不同于参考核酸序列或肽序列、但保留参考分子的基本生物学性质的核酸序列或肽序列。核酸变体的序列中的变化可能不会改变参考核酸所编码的肽的氨基酸序列,或者可能产生氨基酸取代、添加、缺失、融合和截短。肽变体的序列中的变化通常是有限的或保守的,使得参考多肽和变体的序列总体上是紧密类似的并且在许多区域中是相同的。变体和参考肽可通过任何组合的一种或多种取代、添加、缺失而在氨基酸序列上有所不同。核酸或肽的变体可以是天然存在的,诸如等位基因变体,或者可以是未知为天然存在的变体。核酸和肽的非天然存在的变体可通过诱变技术或通过直接合成来制备。
“载体”是包含分离的核酸并且可用于向细胞的内部递送分离的核酸的物质的组合物。多种载体在本领域中是已知的,包括但不限于线性多核苷酸、与离子化合物或两亲性化合物缔合的多核苷酸、质粒以及病毒。因此,术语“载体”包括自主复制的质粒或病毒。所述术语还应解释为包括有助于将核酸转移到细胞中的非质粒和非病毒化合物,例如像聚赖氨酸化合物、脂质体等。病毒载体的实例包括但不限于腺病毒载体、腺相关病毒载体、逆转录病毒载体等。
范围:贯穿本公开,本发明的各个方面可以范围形式呈现。应理解,呈范围形式的描述仅仅是为了方便和简洁,并且不应解释为对本发明范围的硬性限制。因此,范围的描述应被认为是具有确切公开的所有可能的子范围以及所述范围内的单独数值。例如,诸如1至6的范围的描述应被认为具有确切公开的子范围,如1至3、1至4、1至5、2至4、2至6、3至6等,以及所述范围内的单独数字,例如1、2、2.7、3、4、5、5.3和6。这在任何宽度范围的条件下均适用。
在一个方面,本发明是基于有效地递送至细胞核的编辑蛋白的新型融合物的开发。在一个方面,本发明提供了融合蛋白,所述融合蛋白包含编辑蛋白和具有第二氨基酸序列的核定位信号(NLS)。
在一个实施方案中,编辑蛋白包括但不限于CRISPR相关(Cas)蛋白、基于转录激活因子样效应物的核酸酶(TALEN)蛋白、锌指核酸酶(ZFN)蛋白和具有DNA结合结构域的蛋白质。
Cas蛋白的非限制性实例包括Cas1、Cas1B、Cas2、Cas3、Cas4、Cas5、Cas6、Cas7、Cas8、Cas9、Cas10、Csy1、Csy2、Csy3、Cse1、Cse2、Csc1、Csc2、Csa5、Csn2.Csm2、Csm3、Csm4、Csm5、Csm6、Cmr1、Cmr3、Cmr4、Cmr5、Cmr6、Csb1、Csb2、Csb3、Csx17、Csx14、Csx10、Csx16、CsaX、Csx3、Csxl、Csx15、Csf1、Csf2、Csf3、Csf4、SpCas9、StCas9、NmCas9、SaCas9、CjCas9、CjCas9、AsCpf1、LbCpf1、FnCpf1、VRER SpCas9、VQR SpCas9、xCas9 3.7,其同源物、其直向同源物或其修饰型式。在一些实施方案中,Cas蛋白具有DNA或RNA裂解活性。在一些实施方案中,Cas蛋白指导核酸分子的一条或两条链在靶序列位置处(如在靶序列内和/或在靶序列的互补序列内)的裂解。在一些实施方案中,Cas蛋白指导一条或两条链在距靶序列的第一个或最后一个核苷酸约1、2、3、4、5、6、7、8、9、10、15、20、25、50、100、200、500个或更多个碱基对内裂解。在一个实施方案中,Cas蛋白是Cas9、Cas13或Cpf1。在一个实施方案中,Cas蛋白是Cas9。在一个实施方案中,Cas蛋白是催化缺陷型的(dCas)。
在一个实施方案中,Cas蛋白包含与SEQ ID NO:41-46中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,Cas蛋白包含SEQ ID NO:41-46中的一者的序列。
在一个实施方案中,NLS是逆转录转座子NLS。在一个实施方案中,NLS源自Ty1、酵母GAL4、SKI3、L29或组蛋白H2B蛋白、多瘤病毒大T蛋白、VP1或VP2衣壳蛋白、SV40 VP1或VP2衣壳蛋白、腺病毒El a或DBP蛋白、流感病毒NS1蛋白、肝炎病毒核心抗原或哺乳动物核纤层蛋白、c-myc、max、c-myb、p53、c-erbA、jun、Tax、类固醇受体或Mx蛋白、核质蛋白(NPM2)、核仁磷酸蛋白(NPM1)或猿猴vims 40(“SV40”)T抗原。在一个实施方案中,NLS是Ty1或Ty1来源的NLS、Ty2或Ty2来源的NLS或MAK11或MAK11来源的NLS。在一个实施方案中,Ty1 NLS包含SEQ ID NO:51的氨基酸序列。在一个实施方案中,Ty2 NLS包含SEQ ID NO:254的氨基酸序列。在一个实施方案中,MAK11 NLS包含SEQ ID NO:256的氨基酸序列。在一个实施方案中,NLS包含与SEQ ID NO:47-56和254-257中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,NLS蛋白包含SEQ ID NO:47-56和254-257中的一者的序列。
在一个实施方案中,NLS是Ty1样NLS。例如,在一个实施方案中,Ty样NLS包含KKRX基序。在一个实施方案中,Ty1样NLS在N末端包含KKRX基序。在一个实施方案中,Ty1样NLS包含KKR基序。在一个实施方案中,Ty1样NLS在C末端包含KKR基序。在一个实施方案中,Ty1样NLS包含KKRX和KKR基序。在一个实施方案中,Ty1样NLS在N末端包含KKRX基序,并且在C末端包含KKR基序。在一个实施方案中,Ty1样NLS包含至少20个氨基酸。在一个实施方案中,Ty1样NLS包含介于20与40个之间的氨基酸。在一个实施方案中,Ty1样NLS包含与SEQ ID NO:275-887中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,Ty1样NLS蛋白包含SEQ ID NO:275-887中的一者的序列。
在一个实施方案中,融合蛋白包含与SEQ ID NO:249-250中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,融合蛋白包含SEQ ID NO:249-250中的一者的序列。
在一个方面,本发明是基于有效地递送至细胞核的编辑蛋白和逆转录病毒整合酶蛋白的新型融合物的开发。这些融合蛋白组合病毒整合酶的DNA整合活性和催化性死亡Cas的可编程DNA靶向能力。因此,由于这种融合蛋白不依赖于细胞途径进行DNA插入或需要细胞能量源(如ATP),因此这种酶可在许多环境下起作用,如从体外至原核细胞、至分裂或非分裂真核细胞。此外,因为整合酶不需要同源性区域用于插入,所以仅对每个整合酶家族具有特异性的小的末端基序序列,这些融合蛋白编辑可利用单个DNA供体模板进行多重基因组整合(如果由多个向导RNA引导)。
因此,在一个方面,本发明提供了融合蛋白,所述融合蛋白包含具有第一氨基酸序列的CRISPR相关(Cas)蛋白、具有第二氨基酸序列的核定位信号(NLS)以及具有第三氨基酸序列的逆转录病毒整合酶(IN)或其片段或变体。
在一个实施方案中,逆转录病毒IN是人免疫缺陷病毒(HIV)IN、劳斯肉瘤病毒(RSV)IN、小鼠乳腺肿瘤病毒(MMTV)IN、莫洛尼鼠白血病病毒(MoLV)IN、牛白血病病毒(BLV)IN、人嗜T淋巴细胞病毒(HTLV)IN、禽肉瘤白血病病毒(ASLV)IN、猫白血病病毒(FLV)IN、异嗜性鼠白血病病毒相关病毒(XMLV)IN、猿猴免疫缺陷病毒(SIV)IN、猫免疫缺陷病毒(FIV)IN、马传染性贫血病毒(EIAV)IN、原型泡沫病毒(PFV)IN、猿猴泡沫病毒(SFV)IN、人泡沫病毒(HFV)IN、大眼鲈皮肤肉瘤病毒(WDSV)IN或牛免疫缺陷病毒(BIV)IN。
在一个实施方案中,整合酶是逆转录转座子整合酶。在一个实施方案中,逆转录转座子整合酶是Ty1或Ty2。在一个实施方案中,整合酶是细菌整合酶。在一实施方案中,细菌整合酶是insF。
在一个实施方案中,逆转录病毒IN是HIV IN。在一个实施方案中,HIV IN包含一个或多个氨基酸取代,其中所述取代提高催化活性,提高溶解度或增加与一种或多种宿主细胞辅因子的相互作用。在一个实施方案中,HIV IN包含选自由以下组成的组的一个或多个、两个或更多个、三个或更多个、四个或更多个、五个或更多个、六个或更多个、七个或更多个、八个或更多个或九个氨基酸取代:E85G、E85F、D116N、F185K、C280S、T97A、Y134R、G140S和Q148H。在一个实施方案中,HIV IN包含氨基酸取代F185K和C280S。在一个实施方案中,HIV IN包含氨基酸取代T97A和Y134R。在一个实施方案中,HIV IN包含氨基酸取代G140S和Q148H。
在一个实施方案中,逆转录病毒IN片段包含IN N末端结构域(NTD)和IN催化核心结构域(CCD)。在一个实施方案中,逆转录病毒IN片段包含IN CCD和IN C-末端结构域(CTD)。在一个实施方案中,逆转录病毒IN片段包含IN NTD。在一个实施方案中,逆转录病毒IN片段包含IN CCD。在一个实施方案中,逆转录病毒IN片段包含IN CTD。在一个实施方案中,整合酶的片段保留全长整合酶的至少一种活性。逆转录病毒整合酶功能和片段是本领域已知的,并且可在例如Li,等人,2011,Virology 411:194-205和Maertens等人,2010,Nature 468:326-29中找到,所述文献以引用的方式并入本文。
在一个实施方案中,逆转录病毒IN包含与SEQ ID NO:1-40中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,逆转录病毒IN包含SEQ ID NO:1-40中的一者的序列。
在一些实施方案中,CRISPR-Cas结构域包含Cas蛋白。Cas蛋白的非限制性实例包括Cas1、Cas1B、Cas2、Cas3、Cas4、Cas5、Cas6、Cas7、Cas8、Cas9、Cas10、Csy1、Csy2、Csy3、Cse1、Cse2、Csc1、Csc2、Csa5、Csn2.Csm2、Csm3、Csm4、Csm5、Csm6、Cmr1、Cmr3、Cmr4、Cmr5、Cmr6、Csb1、Csb2、Csb3、Csx17、Csx14、Csx10、Csx16、CsaX、Csx3、Csxl、Csx15、Csf1、Csf2、Csf3、Csf4、SpCas9、StCas9、NmCas9、SaCas9、CjCas9、CjCas9、AsCpf1、LbCpf1、FnCpf1、VRERSpCas9、VQR SpCas9、xCas9 3.7,其同源物、其直向同源物或其修饰型式。在一些实施方案中,Cas蛋白具有DNA或RNA裂解活性。在一些实施方案中,Cas蛋白指导核酸分子的一条或两条链在靶序列位置处(如在靶序列内和/或在靶序列的互补序列内)的裂解。在一些实施方案中,Cas蛋白指导一条或两条链在距靶序列的第一个或最后一个核苷酸约1、2、3、4、5、6、7、8、9、10、15、20、25、50、100、200、500个或更多个碱基对内裂解。在一个实施方案中,Cas蛋白是Cas9、Cas13或Cpf1。在一个实施方案中,Cas蛋白是催化缺陷型的(dCas)。
在一个实施方案中,Cas蛋白包含与SEQ ID NO:41-46中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,Cas蛋白包含SEQ ID NO:41-46中的一者的序列。
在一个实施方案中,NLS是逆转录转座子NLS。在一个实施方案中,NLS源自Ty1、酵母GAL4、SKI3、L29或组蛋白H2B蛋白、多瘤病毒大T蛋白、VP1或VP2衣壳蛋白、SV40 VP1或VP2衣壳蛋白、腺病毒El a或DBP蛋白、流感病毒NS1蛋白、肝炎病毒核心抗原或哺乳动物核纤层蛋白、c-myc、max、c-myb、p53、c-erbA、jun、Tax、类固醇受体或Mx蛋白、核质蛋白(NPM2)、核仁磷酸蛋白(NPM1)或猿猴vims 40(“SV40”)T抗原。在一个实施方案中,NLS是Ty1或Ty1来源的NLS、Ty2或Ty2来源的NLS或MAK11或MAK11来源的NLS。在一个实施方案中,Ty1 NLS包含SEQ ID NO:51的氨基酸序列。在一个实施方案中,Ty2 NLS包含SEQ ID NO:254的氨基酸序列。在一个实施方案中,MAK11 NLS包含SEQ ID NO:256的氨基酸序列。在一个实施方案中,NLS包含与SEQ ID NO:47-56和254-257中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,NLS蛋白包含SEQ ID NO:47-56和254-257中的一者的序列。
在一个实施方案中,NLS是Ty1样NLS。例如,在一个实施方案中,Ty样NLS包含KKRX基序。在一个实施方案中,Ty1样NLS在N末端包含KKRX基序。在一个实施方案中,Ty1样NLS包含KKR基序。在一个实施方案中,Ty1样NLS在C末端包含KKR基序。在一个实施方案中,Ty1样NLS包含KKRX和KKR基序。在一个实施方案中,Ty1样NLS在N末端包含KKRX基序,并且在C末端包含KKR基序。在一个实施方案中,Ty1样NLS包含至少20个氨基酸。在一个实施方案中,Ty1样NLS包含介于20与40个之间的氨基酸。在一个实施方案中,Ty1样NLS包含与SEQ ID NO:275-887中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,Ty1样NLS蛋白包含SEQ ID NO:275-887中的一者的序列。
在一个实施方案中,融合蛋白包含与SEQ ID NO:249-250中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,融合蛋白包含SEQ ID NO:249-250中的一者的序列。
在一个实施方案中,NLS包含两个不同NLS的组合。例如,在一个实施方案中,NLS包括Ty1来源的NLS和SV40来源的NLS。在一个实施方案中,NLS是Ty1或Ty1来源的NLS、Ty2或Ty2来源的NLS或MAK11或MAK11来源的NLS。在一个实施方案中,Ty1 NLS包含SEQ ID NO:51的氨基酸序列。在一个实施方案中,Ty2NLS包含SEQ ID NO:254的氨基酸序列。在一个实施方案中,MAK11NLS包含SEQ ID NO:256的氨基酸序列。
在一个实施方案中,NLS包含同一NLS的两个拷贝。例如,在一个实施方案中,NLS包含第一Ty1来源的NLS和第二Ty1来源的NLS的多聚体。
在一个实施方案中,NLS包含与SEQ ID NO:47-56、254-257和275-887中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的第一序列,以及与SEQ ID NO:47-56、254-257和275-887中的一者至少至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的第二序列。在一个实施方案中,第一序列和第二序列是相同的。在一个实施方案中,第一序列和第二序列是不同的。
在一个实施方案中,融合蛋白包含与SEQ ID NO:57-98中的一者70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,融合蛋白包含SEQ ID NO:57-98中的一者的序列。
本发明的肽可使用化学方法来制备。例如,肽可通过固相技术(Roberge J Y等人(1995)Science 269:202-204)来合成、从树脂裂解、并且通过制备型高效液相色谱法来纯化。自动合成可例如根据制造商提供的说明书使用ABI 431A肽合成仪(Perkin Elmer)来实现。
本发明还应解释为包括与本文所公开的融合蛋白具有实质同源性的肽的任何形式。在一个实施方案中,“基本上同源”的肽与本文公开的融合蛋白的氨基酸序列约50%同源性、约70%同源、约80%同源、约90%同源、约95%同源或约99%同源。
所述肽可替代性地通过重组手段或通过从较长的多肽裂解来制备。肽的组成可通过氨基酸分析或测序来确认。
根据本发明的肽的变体可以是:(i)其中氨基酸残基中的一者或多个用保守的或非保守的氨基酸残基取代并且这种取代的氨基酸残基可以是或可以不是通过遗传密码编码的残基的肽,(ii)其中存在一个或多个修饰的氨基酸残基(例如,通过连接取代基进行修饰的残基)的肽,(iii)其中肽是本发明的肽的替代剪接变体的肽,(iv)肽的片段和/或(v)其中肽与另一种肽(诸如前导序列或分泌序列或采用来用于纯化(例如,His-标签)或用于检测(例如,Sv5表位标签)的序列)融合的肽。所述片段包括通过原始序列的蛋白水解裂解(包括多位点蛋白水解)生成的肽。变体可以是翻译后修饰的或化学修饰的。根据本文的教义,此类变体应在本领域的技术人员的范围内。
如本领域中已知的,两种肽之间的“相似性”通过将一种多肽的氨基酸序列及其保守氨基酸取代物与第二多肽的序列进行比较来确定。变体被定义为包括不同于原始序列的肽序列。在一个实施方案中,变体在每个目标区段的少于40%的残基中不同于原始序列、在每个目标区段的少于25%的残基中不同于原始序列、在每个目标区段的少于10%的残基中不同于原始序列或在每个目标区段的仅一些残基中不同于原始序列,并且同时与原始序列足够同源,以保留原始序列的功能性和/或刺激干细胞分化为成骨细胞谱系的能力。本发明包括与原始氨基酸序列至少60%、65%、70%、72%、74%、76%、78%、80%、90%或95%相似或相同的氨基酸序列。使用本领域的技术人员广泛已知的计算机算法和方法来确定两种肽之间的同一性程度。可通过使用BLASTP算法[BLAST Manual,Altschul,S.,等人,NCBINLM NIH Bethesda,Md.20894,Altschul,S.,等人,J.Mol.Biol.215:403-410(1990)]来确定两个氨基酸序列之间的同一性。
本发明的肽可进行翻译后修饰。例如,落入本发明范围内的翻译后修饰包括信号肽裂解、糖基化、乙酰化、异戊二烯化、蛋白水解、豆蔻酰化、蛋白质折叠以及蛋白水解加工等。一些修饰或加工事件需要引入额外的生物学机器。例如,加工事件(诸如信号肽裂解和核心糖基化)通过向标准翻译反应添加犬微粒体膜或非洲爪蟾卵提取物(美国专利号6,103,489)来检验。
本发明的肽可包含通过翻译后修饰或通过在翻译期间引入非天然氨基酸形成的非天然氨基酸。各种方法对于在蛋白质翻译期间引入非天然氨基酸是可用的。
本发明的肽或蛋白质可使用常规方法,诸如在Reedijk等人(The EMBO Journal11(4):1365,1992)中所描述的方法进行磷酰化。
本发明的肽的环状衍生物也是本发明的一部分。环化可使肽呈现对于与其它分子的缔合更有利的构象。环化可使用本领域中已知的技术来实现。例如,二硫键可在具有游离巯基的两种适当间隔的组分之间形成,或者酰胺键可在一种组分的氨基与另一种组分的羧基之间形成。环化还可使用含偶氮苯的氨基酸来实现,如由Ulysse,L.,等人,J.Am.Chem.Soc.1995,117,8466-8467所描述的。形成所述键的组分可以是氨基酸的侧链、非氨基酸组分或两者的组合。在本发明的一个实施方案中,环状肽可包含在右位置中的β-转角。β-转角可通过在右位置处添加氨基酸Pro-Gly来引入到本发明的肽中。
可能希望产生比如上所述的含有肽键连接的环状肽更具柔性的环状肽。更具柔性的肽可通过在肽的右位置和左位置处引入半胱氨酸并且在两个半胱氨酸之间形成二硫桥来制备。布置两个半胱氨酸以便不使β-折叠和β-转角变形。由于β-折叠部分中的二硫键的长度和氢键的较小数量,所述肽是更具柔性的。环状肽的相对柔性可通过分子动力学模拟来测定。
本发明还涉及融合至靶蛋白和/或能够将嵌合蛋白导向至所需的细胞组分或细胞类型或组织的靶向结构域或整合到所述靶蛋白和/或靶向结构域中的肽,所述肽包含IN-Cas9肽。嵌合蛋白还可含有额外的氨基酸序列或结构域。嵌合蛋白是重组的,因为各种组分来自于不同的来源,并且因为此类组分并不一起存在于自然界中(即,是异源的)。
在一个实施方案中,靶向结构域可以是跨膜结构域、膜结合结构域或指导蛋白质例如与囊泡或与核缔合的序列。在一个实施方案中,靶向结构域可使肽靶向特定的细胞类型或组织。例如,靶向结构域可以是针对靶组织的细胞表面抗原的细胞表面配体或抗体。靶向结构域可使本发明的肽靶向细胞组分。
本发明的肽可通过常规技术合成。例如,所述肽或嵌合蛋白可通过使用固相肽合成的化学合成来合成。这些方法采用固相或液相合成方法(参见例如,关于固相合成技术J.M.Stewart,和J.D.Young,Solid Phase Peptide Synthesis,第2版,Pierce ChemicalCo.,Rockford Ill.(1984)以及G.Barany和R.B.Merrifield,The Peptides:AnalysisSynthesis,Biology编辑E.Gross和J.Meienhofer第2卷Academic Press,New York,1980,第3-254页;并且关于经典的溶液合成M Bodansky,Principles of Peptide Synthesis,Springer-Verlag,Berlin 1984,以及E.Gross和J.Meienhofer,编辑,The Peptides:Analysis,Synthesis,Biology,suprs,第1卷)。通过举例的方式,本发明的肽可使用9-芴基甲氧羰基(Fmoc)固相化学来合成,其中直接并入磷酸苏氨酸作为N-芴基甲氧基-羰基-O-苄基-L-磷酸苏氨酸衍生物。
包含与其它分子缀合的本发明的肽或嵌合蛋白的N末端或C末端融合蛋白可通过重组技术将所述肽或嵌合蛋白的N末端或C末端和具有所需生物功能的所选蛋白质或选择性标记物的序列融合来制备。所得的融合蛋白含有融合至如本文所述的所选蛋白质或标记物蛋白的IN-Cas9肽。可用于制备融合蛋白的蛋白质的实例包括免疫球蛋白、谷胱甘肽-S-转移酶(GST)、血凝素(HA)以及截短的myc。
本发明的肽可使用生物学表达系统来开发。使用这些系统允许产生随机肽序列的大文库并且针对结合至特定蛋白质的肽序列筛选这些文库。可通过将编码随机肽序列的合成DNA克隆到适当的表达载体中来产生文库(参见,Christian的人1992,J.Mol.Biol.227:711;Devlin的人,1990Science 249:404;Cwirla的人1990,Proc.Natl.Acad,Sci.USA,87:6378)。还可通过重叠肽的并行合成来构建文库(参见美国专利号4,708,871)。
本发明的肽和嵌合蛋白可通过与无机酸或有机酸反应来转化为药物盐,所述无机酸诸如盐酸、硫酸、氢溴酸、磷酸等,所述有机酸诸如甲酸、乙酸、丙酸、乙醇酸、乳酸、丙酮酸、草酸、琥珀酸、苹果酸、酒石酸、柠檬酸、苯甲酸、水杨酸、苯磺酸和甲苯磺酸。
在一个实施方案中,本发明是一种编码融合蛋白的核酸分子。在一个实施方案中,核酸分子包含编码编辑蛋白的第一核酸序列;和编码核定位信号(NLS)的第二核酸序列。
在一个实施方案中,编辑蛋白包括但不限于CRISPR相关(Cas)蛋白、基于转录激活因子样效应物的核酸酶(TALEN)蛋白、锌指核酸酶(ZFN)蛋白和具有DNA结合结构域的蛋白质。在一个实施方案中,编辑蛋白是Cas蛋白。
Cas蛋白的非限制性实例包括Cas1、Cas1B、Cas2、Cas3、Cas4、Cas5、Cas6、Cas7、Cas8、Cas9、Cas10、Csy1、Csy2、Csy3、Cse1、Cse2、Csc1、Csc2、Csa5、Csn2.Csm2、Csm3、Csm4、Csm5、Csm6、Cmr1、Cmr3、Cmr4、Cmr5、Cmr6、Csb1、Csb2、Csb3、Csx17、Csx14、Csx10、Csx16、CsaX、Csx3、Csxl、Csx15、Csf1、Csf2、Csf3、Csf4、SpCas9、StCas9、NmCas9、SaCas9、CjCas9、CjCas9、AsCpf1、LbCpf1、FnCpf1、VRER SpCas9、VQR SpCas9、xCas9 3.7,其同源物、其直向同源物或其修饰型式。在一些实施方案中,Cas蛋白具有DNA或RNA裂解活性。在一些实施方案中,Cas蛋白指导核酸分子的一条或两条链在靶序列位置处(如在靶序列内和/或在靶序列的互补序列内)的裂解。在一些实施方案中,Cas蛋白指导一条或两条链在距靶序列的第一个或最后一个核苷酸约1、2、3、4、5、6、7、8、9、10、15、20、25、50、100、200、500个或更多个碱基对内裂解。在一个实施方案中,Cas蛋白是Cas9、Cas13或Cpf1。在一个实施方案中,Cas蛋白是Cas9。在一个实施方案中,Cas蛋白是催化缺陷型的(dCas)。
在一个实施方案中,编码Cas蛋白的第一核酸序列包含编码与SEQ ID NO:41-46中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的氨基酸序列的核酸序列。在一个实施方案中,编码Cas蛋白的第一核酸序列包含编码SEQ ID NO:41-46中的一者的核酸序列。
在一个实施方案中,编码Cas蛋白的第一核酸序列包含与SEQ ID NO:139-144中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码Cas蛋白的第一核酸序列包含SEQ ID NO:139-144中的一者的核酸序列。
在一个实施方案中,第二核酸序列编码核定位信号(NLS)。在一个实施方案中,NLS是逆转录转座子NLS。在一个实施方案中,NLS源自酵母GAL4、SKI3、L29或组蛋白H2B蛋白、多瘤病毒大T蛋白、VP1或VP2衣壳蛋白、SV40 VP1或VP2衣壳蛋白、腺病毒El a或DBP蛋白、流感病毒NS1蛋白、肝炎病毒核心抗原或哺乳动物核纤层蛋白、c-myc、max、c-myb、p53、c-erbA、jun、Tax、类固醇受体或Mx蛋白、核质蛋白(NPM2)、核仁磷酸蛋白(NPM1)或猿猴vims40(“SV40”)T抗原。在一个实施方案中,NLS是Ty1或Ty1来源的NLS、Ty2或Ty2来源的NLS或MAK11或MAK11来源的NLS。在一个实施方案中,Ty1 NLS包含SEQ ID NO:51的氨基酸序列。在一个实施方案中,Ty2 NLS包含SEQ ID NO:254的氨基酸序列。在一个实施方案中,MAK11NLS包含SEQ ID NO:256的氨基酸序列。
在一个实施方案中,NLS是Ty1样NLS。例如,在一个实施方案中,Ty样NLS包含KKRX基序。在一个实施方案中,Ty1样NLS在N末端包含KKRX基序。在一个实施方案中,Ty1样NLS包含KKR基序。在一个实施方案中,Ty1样NLS在C末端包含KKR基序。在一个实施方案中,Ty1样NLS包含KKRX和KKR基序。在一个实施方案中,Ty1样NLS在N末端包含KKRX基序,并且在C末端包含KKR基序。在一个实施方案中,Ty1样NLS包含至少20个氨基酸。在一个实施方案中,Ty1样NLS包含介于20与40个之间的氨基酸。
在一个实施方案中,逆转录转座子NLS增加核定位。在一个实施方案中,与非逆转录转座子NLS相比,逆转录转座子NLS显著更多地增加核定位。
在一个实施方案中,编码NLS的第二核酸序列包含编码与SEQ ID NO:47-56、254-257和275-887中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的氨基酸序列的核酸序列。在一个实施方案中,编码NLS的第二核酸序列包含编码SEQ ID NO:47-56、254-257和275-887中的一者的核酸序列。
在一个实施方案中,编码NLS蛋白的第二核酸序列包含与SEQ ID NO:145-154中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码NLS的第二核酸序列包含SEQ ID NO:145-154中的一者的核酸序列。
在一个实施方案中,核酸分子编码融合蛋白,所述融合蛋白包含与SEQ ID NO:249-250中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,核酸分子编码包含SEQ ID NO:249-250中的一者的序列的融合蛋白。
在一个实施方案中,核酸分子包含:编码编辑蛋白的第一核酸序列;编码核定位信号(NLS)的第二核酸序列;以及编码逆转录病毒整合酶(IN)或其片段的第三核酸序列。
在一个实施方案中,逆转录病毒IN是人免疫缺陷病毒(HIV)IN、劳斯肉瘤病毒(RSV)IN、小鼠乳腺肿瘤病毒(MMTV)IN、莫洛尼鼠白血病病毒(MoLV)IN、牛白血病病毒(BLV)IN、人嗜T淋巴细胞病毒(HTLV)IN、禽肉瘤白血病病毒(ASLV)IN、猫白血病病毒(FLV)IN、异嗜性鼠白血病病毒相关病毒(XMLV)IN、猿猴免疫缺陷病毒(SIV)IN、猫免疫缺陷病毒(FIV)IN、马传染性贫血病毒(EIAV)IN、原型泡沫病毒(PFV)IN、猿猴泡沫病毒(SFV)IN、人泡沫病毒(HFV)IN、大眼鲈皮肤肉瘤病毒(WDSV)IN或牛免疫缺陷病毒(BIV)IN。
在一个实施方案中,逆转录病毒IN是HIV IN。在一个实施方案中,HIV IN包含一个或多个氨基酸取代,其中所述取代提高催化活性,提高溶解度或增加与一种或多种宿主细胞辅因子的相互作用。在一个实施方案中,HIV IN包含选自由以下组成的组的一个或多个、两个或更多个、三个或更多个、四个或更多个、五个或更多个、六个或更多个、七个或更多个、八个或更多个或九个氨基酸取代:E85G、E85F、D116N、F185K、C280S、T97A、Y134R、G140S和Q148H。在一个实施方案中,HIV IN包含氨基酸取代F185K和C280S。在一个实施方案中,HIV IN包含氨基酸取代T97A和Y134R。在一个实施方案中,HIV IN包含氨基酸取代G140S和Q148H。
在一个实施方案中,逆转录病毒IN片段包含IN N末端结构域(NTD)和IN催化核心结构域(CCD)。在一个实施方案中,逆转录病毒IN片段包含IN CCD和IN C-末端结构域(CTD)。在一个实施方案中,逆转录病毒IN片段包含IN NTD。在一个实施方案中,逆转录病毒IN片段包含IN CCD。在一个实施方案中,逆转录病毒IN片段包含IN CTD。在一个实施方案中,整合酶的片段保留全长整合酶的至少一种活性。逆转录病毒整合酶功能和片段是本领域已知的,并且可在例如Li,等人,2011,Virology 411:194-205和Maertens等人,2010,Nature 468:326-29中找到,所述文献以引用的方式并入本文。
在一个实施方案中,编码逆转录病毒IN的第三核酸序列包含编码与SEQ ID NO:1-40中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的氨基酸序列的核酸序列。在一个实施方案中,编码逆转录病毒IN的第三核酸序列包含编码SEQ ID NO:1-40中的一者的核酸序列。
在一个实施方案中,编码逆转录病毒IN的第三核酸序列包含与SEQ ID NO:99-138中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码逆转录病毒IN的第三核酸序列包含SEQ ID NO:99-138中的一者的核酸序列。
在一个实施方案中,编辑蛋白包括但不限于CRISPR相关(Cas)蛋白、基于转录激活因子样效应物的核酸酶(TALEN)蛋白、锌指核酸酶(ZFN)蛋白和DNA-结合蛋白。在一个实施方案中,编辑蛋白是Cas蛋白。在一个实施方案中,Cas蛋白是Cas9、Cas13或Cpf1。在一个实施方案中,Cas蛋白是催化缺陷型的(dCas)。
在一个实施方案中,第一核酸序列编码Cas蛋白。在一个实施方案中,编码Cas蛋白的第一核酸序列包含编码与SEQ ID NO:41-46中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的氨基酸序列的核酸序列。在一个实施方案中,编码Cas蛋白的第一核酸序列包含编码SEQ ID NO:41-46中的一者的核酸序列。
在一个实施方案中,编码Cas蛋白的第一核酸序列包含与SEQ ID NO:139-144中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码Cas蛋白的第一核酸序列包含SEQ ID NO:139-144中的一者的核酸序列。
在一个实施方案中,第二核酸序列编码核定位信号(NLS)。在一个实施方案中,NLS是逆转录转座子NLS。在一个实施方案中,NLS源自酵母GAL4、SKI3、L29或组蛋白H2B蛋白、多瘤病毒大T蛋白、VP1或VP2衣壳蛋白、SV40 VP1或VP2衣壳蛋白、腺病毒El a或DBP蛋白、流感病毒NS1蛋白、肝炎病毒核心抗原或哺乳动物核纤层蛋白、c-myc、max、c-myb、p53、c-erbA、jun、Tax、类固醇受体或Mx蛋白、核质蛋白(NPM2)、核仁磷酸蛋白(NPM1)或猿猴vims40(“SV40”)T抗原。在一个实施方案中,NLS是Ty1或Ty1来源的NLS、Ty2或Ty2来源的NLS或MAK11或MAK11来源的NLS。在一个实施方案中,Ty1 NLS包含SEQ ID NO:51的氨基酸序列。在一个实施方案中,Ty2 NLS包含SEQ ID NO:254的氨基酸序列。在一个实施方案中,MAK11NLS包含SEQ ID NO:256的氨基酸序列。
在一个实施方案中,逆转录转座子NLS增加核定位。在一个实施方案中,与非逆转录转座子NLS相比,逆转录转座子NLS显著更多地增加核定位。
在一个实施方案中,编码NLS的第二核酸序列包含编码与SEQ ID NO:47-56、254-257和275-87中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的氨基酸序列的核酸序列。在一个实施方案中,编码NLS的第二核酸序列包含编码SEQ ID NO:47-56、254-257和275-887中的一者的核酸序列。
在一个实施方案中,编码NLS蛋白的第二核酸序列包含与SEQ ID NO:145-154中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码NLS的第二核酸序列包含SEQ ID NO:145-154中的一者的核酸序列。
在一个实施方案中,核酸分子编码融合蛋白,所述融合蛋白包含与SEQ ID NO:57-98中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,核酸分子编码包含SEQ ID NO:57-98中的一者的序列的融合蛋白。
在一个实施方案中,核酸分子包含与SEQ ID NO:155-196中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,核酸分子包含SEQ ID NO:155-196中的一者的核酸序列。
编码融合蛋白的分离的核酸序列可使用本领域中已知的许多重组方法中的任一种来获得,例如像通过筛选来自表达所述基因的细胞的文库、通过从已知含有所述基因的载体得到所述基因、或通过使用标准技术直接从含有所述基因的细胞和组织分离来获得。或者,目标基因可合成产生而并非克隆。
分离的核酸可包括任何类型的核酸,包括但不限于DNA和RNA。例如,在一个实施方案中,组合物包含分离的DNA分子,包括例如编码本发明的融合蛋白的分离的cDNA分子。在一个实施方案中,组合物包含编码本发明的融合蛋白的分离的RNA分子或其功能片段。
本发明的核酸分子可进行修饰以提高在血清或在用于细胞培养的生长培养基中的稳定性。可添加修饰以增强本发明的核酸分子的稳定性、功能性和/或特异性并且使免疫刺激性质最小化。例如,为了增强稳定性,3’-残基可针对降解进行稳定化,例如,它们可被选择使得其由嘌呤核苷酸、具体地是腺苷或鸟苷核苷酸组成。或者,用修饰的类似物取代嘧啶核苷酸,例如用2'-脱氧胸苷取代尿苷是可容忍的并且不影响分子的功能。
在本发明的一个实施方案中,核酸分子可含有至少一种修饰的核苷酸类似物。例如,端部可通过并入修饰的核苷酸类似物来稳定化。
核苷酸类似物的非限制性实例包括糖修饰的和/或主链修饰的核糖核苷酸(即,包括对磷酸糖主链的修饰)。例如,天然RNA的磷酸二酯键联可进行修饰以包含氮或硫杂原子中的至少一个。在示例性主链修饰的核糖核苷酸中,连接至相邻核糖核苷酸的磷酸酯基团被修饰的基团(例如,硫代磷酸酯基团的修饰的基团)代替。在示例性糖修饰的核糖核苷酸中,2’OH-基团被选自H、OR、R、卤基、SH、SR、NH
修饰的其它实例是核苷酸修饰的核糖核苷酸,即含有至少一个非天然存在的核碱基而不是天然存在的核碱基的核糖核苷酸。碱基可进行修饰以阻断腺苷脱氨酶的活性。示例性修饰的核碱基包括但不限于在5-位置处修饰的尿苷和/或胞苷,例如5-(2-氨基)丙基尿苷、5-溴尿苷;在8位置处修饰的腺苷和/或鸟苷,例如8-溴鸟苷;脱氮核苷酸,例如7-脱氮-腺苷;O-和N-烷基化核苷酸,例如N6-甲基腺苷是合适的。应注意,以上修饰可进行组合。
在一些情况下,核酸分子包含以下化学修饰中的至少一种:一个或多个核苷酸的2’-H、2’-O-甲基或2’-OH修饰。在某些实施方案中,本发明的核酸分子可具有对于核酸酶的增强的抗性。针对增加的核酸酶抗性,核酸分子可包含例如2’-修饰的核糖单元和/或硫代磷酸酯键联。例如,2’羟基(OH)可使用多种不同的“氧基”或“脱氧”取代基修饰或代替。针对增强的核碱基抗性,本发明的核酸分子可包含2’-O-甲基、2'-氟、2’-O-甲氧基乙基、2’-O-氨基丙基、2’-氨基和/或硫代磷酸酯键联。包含锁核酸(LNA)、亚乙基核酸(ENA)(例如,2’-4’-亚乙基-桥联核酸)和某些核碱基修饰(诸如2-氨基-A、2-硫代(例如,2-硫代-U)、G-夹环修饰)也可增加对靶标的结合亲和力。
在一个实施方案中,核酸分子包含2’-修饰的核苷酸,例如2’-脱氧、2’-脱氧-2’-氟、2’-O-甲基、2’-O-甲氧基乙基(2’-O-MOE)、2’-O-氨基丙基(2’-O-AP)、2’-O-二甲基氨基乙基(2’-O-DMAOE)、2’-O-二甲基氨基丙基(2’-O-DMAP)、2’-O-二甲基氨基乙基氧基乙基(2’-O-DMAEOE)或2’-O-N-甲基乙酰胺基(2’-O-NMA)。在一个实施方案中,核酸分子包含至少一个2’-O-甲基修饰的核苷酸,并且在一些实施方案中,核酸分子的所有核苷酸包含2’-O-甲基修饰。
在某些实施方案中,本发明的核酸分子具有以下性质中的一种或多种:
本文所讨论的核酸剂包括以其它方式未修饰的RNA和DNA以及进行修饰例如以提高功效的RNA和DNA、以及核苷替代物的聚合物。未修饰的RNA是指其中核酸的组分(即,糖、碱基和磷酸部分)与自然界中存在的组分、或与人体中天然存在的组分相同或基本上相同的分子。本领域将罕见的或不寻常的但是天然存在的RNA称为修饰的RNA,参见例如Limbach等人(Nucleic Acids Res.,1994,22:2183-2196)。此类罕见的或不寻常的RNA(常常称作修饰的RNA)通常是转录后修饰的结果并且在如本文所使用的术语未修饰的RNA内。如本文所使用的修饰的RNA是指其中核酸的组分(即,糖、碱基和磷酸部分)中的一种或多种不同于自然界中存在的组分,或不同于人体中存在的组分。虽然它们被称为“修饰的RNA”,但是因为修饰,它们当然包括严格地讲不是RNA的分子。核苷替代物是其中核糖磷酸主链使用允许碱基以正确的空间关系呈现、使得杂交与使用核糖磷酸主链所看到的情况大体上相似的非核糖磷酸构建体(例如,核糖磷酸主链的不带电荷的模拟物)代替的分子。
本发明的核酸的修饰可存在于磷酸基团、糖基团、主链、N-末端、C-末端、或核碱基中的一者或多个处。
本发明还包括一种本发明的分离的核酸插入其中的载体。本领域有很多可用于本发明中的合适的载体。
简言之,编码本发明的融合蛋白的天然的或合成核酸的表达通常通过将编码本发明的融合蛋白的核酸或其部分可操作地连接至启动子并且将构建体并入表达载体中来实现。待使用的载体适于在真核细胞中复制并且任选地整合在真核细胞中。典型的载体含有可用于调控所需核酸序列的表达的转录和翻译终止子、起始序列和启动子。
本发明的载体还可使用标准基因递送方案用于核酸免疫和基因疗法。用于基因递送的方法是本领域已知的。参见例如,美国专利号5,399,346、5,580,859、5,589,466,所述专利以引用的方式整体并入本文。在另一个实施方案中,本发明提供一种基因治疗载体。
本发明的分离的核酸可被克隆到多种类型的载体中。例如,所述核酸可被克隆到载体中,所述载体包括但不限于质粒、噬菌粒、噬菌体衍生物、动物病毒和粘粒。特别感兴趣的载体包括表达载体、复制载体、探针生成载体和测序载体。
此外,所述载体可以病毒载体的形式提供给细胞。病毒载体技术在本领域中是熟知,并且描述于例如Sambrook等人(2012,Molecular Cloning:A Laboratory Manual,ColdSpring Harbor Laboratory,New York)和其它病毒学和分子生物学手册中。可用作载体的病毒包括但不限于逆转录病毒、腺病毒、腺相关病毒、疱疹病毒以及慢病毒。一般来说,合适的载体含有在至少一种生物体中发挥功能的复制起点、启动子序列、便利的限制核酸内切酶位点以及一种或多种选择性标记物(例如WO 01/96584;WO 01/29058;和美国专利号6,326,193)。
在一个方面,本发明涉及新型慢病毒包装和递送系统的开发。慢病毒颗粒将病毒酶作为蛋白质递送。以这种方式,慢病毒酶的寿命很短,从而限制由于贯穿细胞的整个生命周期的长期表达所致的脱靶编辑的可能性。考虑到它们所需的活性仅在短时间段内需要,将编辑成分或传统CRISPR-Cas编辑组分作为蛋白质并入慢病毒颗粒中是有利的。因此,在一个实施方案中,本发明提供了慢病毒递送系统,以及递送本发明的组合物、编辑遗传物质和使用慢病毒递送系统进行核酸递送的方法。
例如,在一个方面,递送系统包含(1)包装质粒,(2)转移质粒和(3)包膜质粒。在一个实施方案中,包装质粒包含编码经修饰的gag-pol多蛋白的核酸序列。在一个实施方案中,经修饰的gag-pol多蛋白包含与编辑蛋白融合的整合酶。在一个实施方案中,经修饰的gag-pol多蛋白包含与Cas蛋白融合的整合酶。在一个实施方案中,经修饰的gag-pol多蛋白包含与催化性死亡Cas蛋白(dCas)融合的整合酶。在一个实施方案中,包装质粒还包含编码sgRNA序列的序列。
在一个实施方案中,转移质粒包含供体序列。供体序列可以是待递送至基因组的任何核酸序列。在一个实施方案中,转移质粒包含5'长末端重复(LTR)序列和3’LTR序列。在一个实施方案中,3’LTR是自灭活(SIN)LTR。因此,在一个实施方案中,5’LTR包含U3序列、R序列和U5序列,并且3’LTR包含R序列和U5序列、但不包含U3序列。在一个实施方案中,5’LTR和3’LTR对Insctriptr包装质粒中的整合酶具有特异性。
在一个实施方案中,包膜质粒包含编码包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码HIV包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码水疱性口炎病毒g蛋白包膜蛋白的核酸序列。在一个实施方案中,可基于所需的细胞类型选择包膜蛋白。
在一个实施方案中,将包装质粒、转移质粒和包膜质粒引入细胞中。在一个实施方案中,细胞转录并翻译编码经修饰的gag-pol蛋白的核酸序列,以产生经修饰的gag-pol蛋白。在一个实施方案中,细胞转录编码sgRNA的核酸序列。在一个实施方案中,sgRNA结合至整合酶-Cas融合蛋白。在一个实施方案中,细胞转录并翻译编码包膜蛋白的核酸序列以产生包膜蛋白。在一个实施方案中,细胞转录供体序列以提供供体序列RNA分子。在一个实施方案中,将与sgRNA、包膜多蛋白和供体序列RNA结合的经修饰的gag-pol蛋白包装到病毒颗粒中。在一个实施方案中,从细胞培养基收集病毒颗粒。在一个实施方案中,病毒颗粒转导靶细胞,其中sgRNA结合细胞DNA的靶区域,从而靶向IN-Cas9融合蛋白,并且整合酶催化供体序列整合到细胞DNA中。
在一个方面,递送系统包含(1)包装质粒,(2)转移质粒,(3)包膜质粒,以及(4)VPR-IN-dCas质粒。在一个实施方案中,包装质粒包含编码gag-pol多蛋白的核酸序列。在一个实施方案中,gag-pol多蛋白包含催化性死亡整合酶。在一个实施方案中,gag-pol多蛋白包含D116N整合酶突变。
在一个实施方案中,转移质粒包含供体序列。供体序列可以是待递送至基因组的任何核酸序列。在一个实施方案中,转移质粒包含5'长末端重复(LTR)序列和3’LTR序列。在一个实施方案中,3’LTR是自灭活(SIN)LTR。因此,在一个实施方案中,5’LTR包含U3序列、R序列和U5序列,并且3’LTR包含R序列和U5序列、但不包含U3序列。在一个实施方案中,5’LTR和3’LTR对VPR-IN-dCas包装质粒中的整合酶具有特异性。
在一个实施方案中,包膜质粒包含编码包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码HIV包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码水疱性口炎病毒g蛋白(VSV-g)包膜蛋白的核酸序列。在一个实施方案中,可基于所需的细胞类型选择包膜蛋白。
在一个实施方案中,VPR-IN-dCas质粒包含编码融合蛋白的核酸序列,所述融合蛋白包含VPR、整合酶和编辑蛋白。在一个实施方案中,VPR-IN-dCas质粒包含编码融合蛋白的核酸序列,所述融合蛋白包含VPR、整合酶和Cas蛋白。在一个实施方案中,VPR-IN-dCas质粒包含编码融合蛋白的核酸序列,所述融合蛋白包含VPR、整合酶和dCas蛋白。在一个实施方案中,融合蛋白包含在VPR与整合酶之间的蛋白酶裂解位点。在一个实施方案中,VPR-IN-dCas质粒包装质粒还包含编码sgRNA序列的序列。
在一个实施方案中,将包装质粒、转移质粒、包膜质粒和VPR-IN-dCas质粒引入细胞中。在一个实施方案中,细胞转录并翻译编码gag-pol蛋白的核酸序列以产生gag-pol多蛋白。在一个实施方案中,细胞转录并翻译编码包膜蛋白的核酸序列以产生包膜蛋白。在一个实施方案中,细胞转录供体序列以提供供体序列RNA分子。在一个实施方案中,细胞转录并翻译融合蛋白以产生VPR-整合酶编辑蛋白融合蛋白。在一个实施方案中,细胞转录并翻译融合蛋白以产生VPR-整合酶-dCas融合蛋白。在一个实施方案中,细胞转录编码sgRNA的核酸序列。在一个实施方案中,sgRNA结合至VPR-整合酶-dCas融合蛋白。
在一个实施方案中,将gag-pol蛋白、包膜多蛋白、供体序列RNA和与sgRNA结合的VPR-整合酶-dCas9蛋白包装到病毒颗粒中。在一个实施方案中,从细胞培养基收集病毒颗粒。在一个实施方案中,经由蛋白酶位点从病毒颗粒中的融合蛋白裂解VPR以提供IN-dCas融合蛋白。在一个实施方案中,病毒颗粒转导靶细胞,其中sgRNA结合细胞DNA的靶区域,从而靶向IN-dCas融合蛋白,并且整合酶催化供体序列整合到细胞DNA中。
在一个方面,递送系统包含(1)转移质粒,(2)包装质粒和(3)包膜质粒。在一个实施方案中,包装质粒包含编码gag-pol多蛋白的核酸序列。在一个实施方案中,gag-pol多蛋白包含催化性死亡整合酶。在一个实施方案中,gag-pol多蛋白包含D116N整合酶突变。
在一个实施方案中,转移质粒包含编码sgRNA的核酸和编码融合蛋白的核酸序列,所述融合蛋白包含整合酶和编辑蛋白。在一个实施方案中,转移质粒包含5'长末端重复(LTR)序列和3’LTR序列。在一个实施方案中,3’LTR是自灭活(SIN)LTR。因此,在一个实施方案中,5’LTR包含U3序列、R序列和U5序列,并且3’LTR包含R序列和U5序列、但不包含U3序列。在一个实施方案中,5’LTR和3’LTR对融合蛋白的整合酶具有特异性。在一个实施方案中,融合蛋白包含整合酶和Cas蛋白。在一个实施方案中,融合蛋白包含整合酶和dCas蛋白。在一个实施方案中,5’LTR和3’LTR位于编码融合蛋白的序列和编码sgRNA的序列的侧翼。
在一个实施方案中,包膜质粒包含编码包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码HIV包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码水疱性口炎病毒g蛋白(VSV-g)包膜蛋白的核酸序列。在一个实施方案中,可基于所需的细胞类型选择包膜蛋白。
在一个实施方案中,将包装质粒、转移质粒和包膜质粒引入细胞中。在一个实施方案中,细胞转录并翻译编码gag-pol蛋白的核酸序列以产生gag-pol多蛋白。在一个实施方案中,细胞转录并翻译编码包膜蛋白的核酸序列以产生包膜蛋白。在一个实施方案中,细胞转录编码sgRNA的核酸序列。在一个实施方案中,细胞转录编码融合蛋白的核酸序列。
在一个实施方案中,将gag-pol蛋白、包膜多蛋白、供体序列RNA和与sgRNA结合的VPR-整合酶-dCas9蛋白包装到病毒颗粒中。在一个实施方案中,从细胞培养基收集病毒颗粒。在一个实施方案中,病毒颗粒转导靶细胞,其中病毒反向翻译,并且细胞表达融合蛋白和sgRNA。在一个实施方案中,sgRNA结合至融合蛋白的Cas蛋白和另一种病毒DNA转录物,其中整合酶催化自身整合。在一个实施方案中,sgRNA结合至融合蛋白的Cas蛋白和细胞DNA的靶区域,从而破坏靶基因。
在一个方面,递送系统包含(1)转移质粒、(2)第一包装质粒、(3)第一包膜质粒、(4)第二包装质粒、(5)第二包膜质粒和(6)转移质粒。在一个实施方案中,第一包装质粒包含编码gag-pol多蛋白的核酸序列。在一个实施方案中,第二包装质粒包含编码gag-pol多蛋白的核酸序列。在一个实施方案中,gag-pol多蛋白包含催化性死亡整合酶。在一个实施方案中,gag-pol多蛋白包含D116N或D64V整合酶突变。
在一个实施方案中,第一包膜质粒包含编码包膜蛋白的核酸序列。在一个实施方案中,第二包膜质粒包含编码包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码HIV包膜蛋白的核酸序列。在一个实施方案中,包膜质粒包含编码水疱性口炎病毒g蛋白(VSV-g)包膜蛋白的核酸序列。在一个实施方案中,可基于所需的细胞类型选择包膜蛋白。
在一个实施方案中,转移质粒包含编码sgRNA的核酸和编码融合蛋白的核酸序列,所述融合蛋白包含整合酶和编辑蛋白。在一个实施方案中,融合蛋白包含整合酶和Cas蛋白。在一个实施方案中,融合蛋白包含整合酶和dCas蛋白。在一个实施方案中,与第一和第二包装质粒的gag-pol多蛋白相比,融合蛋白的整合酶来自不同种类的慢病毒。例如,在一个实施方案中,转移质粒包含编码融合蛋白的核酸,所述融合蛋白包含FIV整合酶和Cas;并且第一和第二包装质粒包含编码HIV gag-pol多蛋白的核酸序列。在一个实施方案中,使用不同的慢病毒种类阻止自我整合。
在一个实施方案中,转移质粒包含5'长末端重复(LTR)序列和3’LTR序列。在一个实施方案中,3’LTR是自灭活(SIN)LTR。因此,在一个实施方案中,5’LTR包含U3序列、R序列和U5序列,并且3’LTR包含R序列和U5序列、但不包含U3序列。在一个实施方案中,5’LTR和3’LTR对gag-pol多蛋白的整合酶具有特异性。在一个实施方案中,5’LTR和3’LTR位于编码融合蛋白的序列和编码sgRNA的序列的侧翼。
在一个实施方案中,转移质粒包含供体序列。供体序列可以是待递送至基因组的任何核酸序列。在一个实施方案中,转移质粒包含5'长末端重复(LTR)序列和3’LTR序列。在一个实施方案中,3’LTR是自灭活(SIN)LTR。因此,在一个实施方案中,5’LTR包含U3序列、R序列和U5序列,并且3’LTR包含R序列和U5序列、但不包含U3序列。在一个实施方案中,5’LTR和3’LTR对Inscrtipter转移质粒中的整合酶具有特异性。
在一个实施方案中,将第一包装质粒、转移质粒和第一包膜质粒引入细胞中。在一个实施方案中,细胞转录并翻译编码gag-pol蛋白的核酸序列以产生gag-pol多蛋白。在一个实施方案中,细胞转录并翻译编码包膜蛋白的核酸序列以产生包膜蛋白。在一个实施方案中,细胞转录编码sgRNA的核酸序列。在一个实施方案中,细胞转录编码融合蛋白的核酸序列。在一个实施方案中,将gag-pol蛋白、包膜多蛋白、gRNA和融合蛋白RNA包装到第一病毒颗粒中。在一个实施方案中,从细胞培养基收集第一病毒颗粒。
在一个实施方案中,将第二包装质粒、转移质粒和第二包膜质粒引入细胞中。在一个实施方案中,细胞转录并翻译编码gag-pol多蛋白的核酸序列以产生gag-pol多蛋白。在一个实施方案中,细胞转录并翻译编码包膜蛋白的核酸序列以产生包膜蛋白。在一个实施方案中,细胞转录供体序列以提供供体序列RNA分子。在一个实施方案中,将gag-pol多蛋白、包膜多蛋白和供体序列RNA包装到第二病毒颗粒中。在一个实施方案中,从细胞培养基中收集第二病毒颗粒。
在一个实施方案中,将第一包装质粒、转移质粒、第一包膜质粒、第二包装质粒、转移质粒和第二包膜质粒引入同一细胞中。在一个实施方案中,将第一包装质粒、转移质粒、第一包膜质粒与第二包装质粒、转移质粒和第二包膜质粒引入不同的细胞中。
在一个实施方案中,第一病毒颗粒和第二病毒颗粒转导靶细胞。在一个实施方案中,病毒反向翻译,并且细胞表达融合蛋白和sgRNA,其中sgRNA结合至所述融合蛋白的dCas。在一个实施方案中,病毒将供体序列RNA反向翻译成供体DNA序列,所述供体DNA序列结合至融合蛋白的整合酶。在一个实施方案中,sgRNA结合细胞DNA的靶区域,从而靶向IN-dCas融合蛋白,并且整合酶催化供体DNA序列整合到细胞DNA中。
此外,多种基于另外的病毒的系统已被开发用于将基因转移到哺乳动物细胞中。例如,逆转录病毒为基因递送系统提供便利的平台。可使用本领域已知的技术将选择的基因插入载体中并包装在逆转录病毒颗粒中。然后可分离重组病毒并体内或离体递送至受试者的细胞。许多逆转录病毒系统是本领域已知的。在一些实施方案中,使用腺病毒载体。许多腺病毒载体是本领域已知的。在一个实施方案中,使用慢病毒载体。
例如,来源于逆转录病毒(诸如慢病毒)的载体是适于实现长期基因转移的工具,因为它们允许转基因及其子代长期稳定地整合在子细胞中。慢病毒具有优于来源于致癌性逆转录病毒(诸如鼠白血病病毒)的附加优点,因为慢病毒可转导非增殖性细胞,诸如肝细胞。它们还具有低免疫原性的附加优点。
在一个实施方案中,所述组合物包含来源于腺相关病毒(AAV)的载体。术语“AAV载体”是指源自腺相关病毒血清型的载体,所述腺相关病毒血清型包括但不限于AAV-1、AAV-2、AAV-3、AAV-4、AAV-5、AAV-6、AAV-7、AAV-8和AAV-9。AAV载体已成为用于治疗各种病症的强力的基因递送工具。AAV载体具有使得它们理想地适于基因治疗的多种特征,所述特征包括缺少致病力、最低的免疫原性、以及以稳定且高效的方式转导分裂期后细胞的能力。AAV载体内包含的特定基因的表达可通过选择AAV血清型、启动子和递送方法的适当组合来特异性地靶向到一种或多种类型的细胞。
AAV载体可具有全部或部分缺失的AAV野生型基因中的一者或多个,优选为rep和/或cap基因,但保留功能性侧接ITR序列。尽管具有高度同源性,但不同的血清型对于不同的组织具有嗜性。AAV1的受体未知;然而,已知AAV1比AAV2更有效地转导骨骼肌和心肌。由于大多数研究是用假型化载体完成的,其中将侧接有AAV2 ITR的载体DNA包装到替代血清型的衣壳中,因此很明显,生物学差异与衣壳有关,而不是与基因组有关。最近的证据表明,与包装在AAV2衣壳中的DNA表达盒相比,包装在AAV 1衣壳中的DNA表达盒在转导心肌细胞方面的效率高至少1log 10。在一个实施方案中,病毒递送系统是腺相关病毒递送系统。腺相关病毒可具有血清型1(AAV1)、血清型2(AAV2)、血清型3(AAV3)、血清型4(AAV4)、血清型5(AAV5)、血清型6(AAV6)、血清型7(AAV7)、血清型8(AAV8)或血清型9(AAV9)。
用于组装成载体的期望的AAV片段包含cap蛋白,包括vp1、vp2、vp3和高变区;rep蛋白,包括rep 78、rep 68、rep 52和rep 40;以及编码这些蛋白的序列。这些片段可容易地用于多种载体系统和宿主细胞中。此类片段可单独使用、与其它AAV血清型序列或片段组合使用或与来自其它AAV或非AAV病毒序列的元件组合使用。如本文所用,人工AAV血清型包括但不限于具有非天然存在的衣壳蛋白的AAV。可通过任何合适的技术,使用选择的AAV序列(例如,vp1衣壳蛋白的片段)与异源序列的组合来产生这样的人工衣壳,所述异源序列可获自不同的选择的AAV血清型、相同AAV血清型的非连续部分、非AAV病毒来源或非病毒来源。人工AAV血清型可以是但不限于嵌合AAV衣壳、重组AAV衣壳或“人源化”AAV衣壳。因此,适于表达一种或多种蛋白质的示例性AAV或人工AAV包括AAV2/8(参见美国专利号7,282,199)、AAV2/5(可从美国国立卫生研究院获得)、AAV2/9(国际专利公布号WO2005/033321)、AAV2/6(美国专利号6,156,303)和AAVrh8(国际专利公布号WO2003/042397)等。
在某些实施方案中,载体还包含常规控制元件,所述控制元件以允许其在用质粒载体转染或用由本发明产生的病毒感染的细胞中转录、翻译和/或表达的方式可操作地连接至转基因。如本文所用,“可操作地连接的”序列包括与目标基因邻接的表达控制序列和以反式或隔开一定距离起作用以控制目标基因的表达控制序列两者。表达控制序列包括:适当转录起始序列、终止序列、启动子序列和增强子序列;高效RNA加工信号,诸如剪接信号和聚腺苷酸化(polyA)信号;使细胞质mRNA稳定的序列;增强翻译效率的序列(即,Kozak共有序列);增强蛋白质稳定性的序列;以及当需要时,增强所编码的产物的分泌的序列。多种表达控制序列(包括天然的、组成型的、诱导型的和/或组织特异性的启动子)是本领域中已知的并且可被利用。
额外的启动子元件(例如,增强子)调控转录起始的频率。通常,这些启动子位于起始位点上游30-110bp区域中,不过许多启动子最近已被证明也含有起始位点下游的功能元件。启动子元件之间的间隔经常是灵活的,使得当元件相对于彼此反转或移动时保留启动子功能。在胸苷激酶(tk)启动子中,在活性开始下降之前,启动子元件之间的间隔可增加至相隔50bp。取决于启动子,似乎单独元件可协同或独立地作用来激活转录。
合适的启动子的一个实例是即刻早期巨细胞病毒(CMV)启动子序列。这种启动子序列是强大的组成型启动子序列,其能够驱动任何与其可操作地连接的多核苷酸序列的高水平表达。合适的启动子的另一个实例是伸长生长因子-1α(EF-1α)。然而,也可使用其它组成型启动子序列,包括但不限于猿猴病毒40(SV40)早期启动子、小鼠乳腺肿瘤病毒(MMTV)、人类免疫缺陷病毒(HIV)长末端重复序列(LTR)启动子、MoMuLV启动子、禽白血病病毒启动子、爱泼斯坦-巴尔(Epstein-Barr)病毒立即早期启动子、劳氏(Rous)肉瘤病毒启动子以及人类基因启动子,诸如但不限于肌动蛋白启动子、肌球蛋白启动子、血红蛋白启动子以及肌酸激酶启动子。此外,本发明不应该被局限于使用组成型启动子。诱导型启动子也可考虑作为本发明的一部分。使用诱导型启动子提供了一种分子开关,所述分子开关能够在需要此表达时开启与它操作性地连接的多核苷酸序列的表达,或者在不需要表达时关闭所述表达。诱导型启动子的实例包括但不限于金属硫蛋白启动子、糖皮质激素启动子、黄体酮启动子和四环素启动子。
载体上存在的增强子序列还调控包含在其中的基因的表达。通常,增强子与蛋白质因子结合以增强基因的转录。增强子可位于其所调控的基因的上游或下游。增强子还可以是组织特异性的,以增强特定细胞或组织类型中的转录。在一个实施方案中,本发明的载体含有一种或多种增强子以加强存在于载体内的基因的转录。
为了评估本发明的融合蛋白的表达,待引入到细胞中的表达载体也可含有选择性标记物基因或报告基因或两者,以便有助于从试图通过病毒载体转染或感染的细胞群体中鉴定和选择表达细胞。在其它方面,选择性标记物可携带在一段单独的DNA上,并且在共转染程序中使用。选择性标记物和报告基因均可侧接有适当的调空序列,使得在宿主细胞中能够表达。有用的选择性标记物包括例如抗生素抗性基因,诸如neo等。
报告基因用于鉴定潜在被转染的细胞并用于评价调控序列的功能性。一般来说,报告基因是不存在于接受体生物体或组织中或不由所述接受体生物体或组织表达并且编码通过一些易于检测的性质(例如,酶活性)显现表达的多肽的基因。在DNA已被引入到接受体细胞中后的合适时间,测定报告基因的表达。合适的报告基因可包括编码荧光素酶、β-半乳糖苷酶、氯霉素乙酰转移酶、分泌型碱性磷酸酶的基因或绿色荧光蛋白基因(例如,Ui-Tei等人,2000FEBS Letters 479:79-82)。合适的表达系统是熟知的,并且可使用已知的技术来制备或商购获得。一般来说,具有显示报告基因的最高水平表达的最小5'侧翼区的构建体被鉴定为启动子。这样的启动子区域可连接至报告基因,并用于评估剂调节启动子驱动的转录的能力。
将基因引入到细胞中并表达基因的方法是本领域中已知的。在表达载体的情况下,所述载体可易于通过本领域的任何方法引入到宿主细胞(例如,哺乳动物、细菌、酵母或昆虫细胞)中。例如,可通过物理、化学或生物学手段将表达载体转移到宿主细胞中。
用于将多核苷酸引入到宿主细胞中的物理方法包括磷酸钙沉淀、脂质转染、粒子轰击、显微注射、电穿孔等。用于产生包含载体和/或外源核酸的细胞的方法是本领域众所周知的。参见例如,Sambrook等人(2012,Molecular Cloning:A Laboratory Manual,ColdSpring Harbor Laboratory,New York)。用于将多核苷酸引入到宿主细胞中的示例性方法是磷酸钙转染。
用于将目标多核苷酸引入到宿主细胞中的生物学方法包括使用DNA和RNA载体。病毒载体,并且尤其是逆转录病毒载体,已成为用于将基因插入到哺乳动物(例如,人类细胞)中的最广泛使用的方法。其它病毒载体可来源于慢病毒、痘病毒、单纯疱疹病毒I、腺病毒和腺相关病毒等。参见例如,美国专利号5,350,674和5,585,362。
用于将多核苷酸引入到宿主细胞中的化学手段包括胶体分散体系,如大分子复合物、纳米囊、微球体、珠粒;以及基于脂质的系统,包括水包油型乳剂、胶束、混合型胶束和脂质体。在体外和体内用作递送媒介物的示例性胶体系统是脂质体(例如,人工膜囊泡)。
在利用非病毒递送系统的情况下,一种示例性递送媒介物是脂质体。关于将核酸引入宿主细胞中(体外、离体或体内)可考虑使用脂质制剂。在另一个方面,核酸可与脂质缔合。与脂质缔合的核酸可包封在脂质体的含水内部、散布在脂质体的脂质双层内、通过与脂质体和寡核苷酸两者缔合的连接分子连接到脂质体、包埋在脂质体中、与脂质体复合、分散在含有脂质的溶液中、与脂质混合、与脂质组合、以悬浮液形式含于脂质中、含于胶束中或与胶束复合、或以其它方式与脂质缔合。脂质、脂质/DNA或脂质/表达载体缔合的组合物不限于在溶液中的任何具体结构。例如,它们可存在于双层结构中、以胶束形式存在、或具有“塌陷的”结构。它们也可仅散布在溶液中,可能形成大小或形状不均一的聚集体。脂质是脂肪物质,它们可以是天然存在的或合成的脂质。例如,脂质包括天然存在于细胞质中的脂肪滴,以及含有长链脂肪族烃和其衍生物的一类化合物,如脂肪酸、醇、胺、氨基醇和醛。
适于使用的脂质可从商业来源获得。例如,二肉豆蔻磷脂酰胆碱(“DMPC”)可从Sigma,St.Louis,MO获得;磷酸双十六烷基酯(“DCP”)可从K&K Laboratories(Plainview,NY)获得;胆固醇(“Choi”)可从Calbiochem-Behring获得;二肉豆蔻磷脂酰甘油(“DMPG”)和其它脂质可从Avanti Polar Lipids,Inc.(Birmingham,AL)获得。脂质在氯仿或者氯仿/甲醇中的储备溶液可以储存在约-20℃下。因为氯仿比甲醇更易于蒸发,所以氯仿作为唯一的溶剂使用。“脂质体”是通用术语,涵盖各种单层和多层脂质媒介物,所述脂质媒介物通过生成封闭的脂质双层或聚集体而形成。脂质体可被表征为具有囊泡结构,所述囊泡结构具有磷脂双层膜和内部水介质。多层脂质体具有通过水介质分隔的多个脂质层。它们在磷脂悬浮于过量水溶液中时自发形成。脂质组分在形成封闭结构之前进行自我重排,并将水和溶解的溶质包埋在脂质双层之间(Ghosh等人,1991Glycobiology 5:505-10)。然而,也涵盖与正常囊泡结构相比在溶液中具有不同结构的组合物。例如,脂质可呈现胶束结构或仅仅以非均匀脂质分子聚集体形式存在。lipofectamine-核酸复合物也考虑在内。
不管使用何种方法将外源核酸引入到宿主细胞中,为了确认重组DNA序列存在于宿主细胞中,可进行各种测定。此类测定包括例如本领域的技术人员熟知的“分子生物学”测定,诸如DNA印迹和RNA印迹、RT-PCR和PCR;“生物化学”测定,诸如检测液滴多肽存在或不存在,所述测定例如通过免疫学手段(ELISA和蛋白质印迹法)或通过本文所述的鉴定落在本发明范围内的剂的测定来进行。
在一个方面,本发明提供了一种用于编辑遗传物质如核酸分子、基因组或基因的系统。在一个实施方案中,所述系统在一种或多种载体中包含:编码融合蛋白的核酸序列,其中所述融合蛋白包含逆转录病毒整合酶(IN)或其片段、CRISPR相关(Cas)蛋白和核定位信号(NLS);编码CRISPR-Cas系统向导RNA的核酸序列;以及编码供体模板核酸的核酸序列,其中所述供体模板核酸包含U3序列、U5序列和供体模板序列。在一个实施方案中,CRISPR-Cas系统向导RNA基本上与所述基因中的靶DNA序列杂交。
在一个实施方案中,所述系统在一种或多种载体中包含:编码融合蛋白的核酸序列,其中所述融合蛋白包含逆转录病毒整合酶(IN)或其片段、CRISPR相关(Cas)蛋白和核定位信号(NLS);编码第一CRISPR-Cas系统向导RNA的核酸序列;编码第二CRISPR-Cas系统向导RNA的核酸序列;以及编码供体模板核酸的核酸序列,其中所述供体模板核酸包含U3序列、U5序列和供体模板序列。在一个实施方案中,第一CRISPR-Cas系统向导RNA基本上与第一DNA序列杂交,并且第二CRISPR-Cas系统向导RNA基本上与第二DNA序列杂交。在一个实施方案中,第一DNA序列和第二DNA序列位于靶标插入区域的侧翼。在一个实施方案中,所述系统催化供体模板核酸插入到靶标插入区域中。
在一个实施方案中,所述系统在一种或多种载体中包含:编码第一融合蛋白的核酸序列,其中所述第一融合蛋白包含逆转录病毒整合酶(IN)或其片段、CRISPR相关(Cas)蛋白和核定位信号(NLS);编码第一CRISPR-Cas系统向导RNA的核酸序列;编码第二融合蛋白的核酸序列,其中所述第二融合蛋白包含逆转录病毒整合酶(IN)或其片段、CRISPR相关(Cas)蛋白和核定位信号(NLS);编码第一CRISPR-Cas系统向导RNA的核酸序列;编码第二CRISPR-Cas系统向导RNA的核酸序列;以及编码供体模板核酸的核酸序列,其中所述供体模板核酸包含U3序列、U5序列和供体模板序列。
在一个实施方案中,第一融合蛋白和第二融合蛋白是相同的或不同的。例如,在一个实施方案中,第一融合蛋白包含HIV IN或其片段、dCas9蛋白和NLS;并且第二融合蛋白包含BIV IN或其片段、Cpf1 Cas蛋白和NLS。
在一个实施方案中,U3对第一融合蛋白的逆转录病毒IN具有特异性,并且U5对第二融合蛋白的逆转录病毒IN具有特异性。例如,在一个实施方案中,第一融合蛋白包含HIVIN或其片段、dCas9蛋白和NLS;第二融合蛋白包含BIV IN或其片段、Cpf1 Cas蛋白和NLS;U3序列对HIV IN具有特异性,并且U5序列对BIV IN具有特异性。
在一个实施方案中,第一CRISPR-Cas系统向导RNA基本上与第一DNA序列杂交,并且第二CRISPR-Cas系统向导RNA基本上与第二DNA序列杂交。在一个实施方案中,第一DNA序列和第二DNA序列位于靶标插入区域的侧翼。在一个实施方案中,所述系统催化供体模板核酸插入到靶标插入区域中。
在一个实施方案中,所述系统包含编码融合蛋白的核酸序列,其中所述融合蛋白包含逆转录病毒整合酶(IN)或其片段、CRISPR相关(Cas)蛋白和核定位信号(NLS);CRISPR-Cas系统向导RNA;供体模板核酸,其中所述供体模板核酸包含U3序列、U5序列和供体模板序列。
在一个实施方案中,编码融合蛋白的核酸序列、编码CRISPR-Cas系统向导RNA的核酸序列和编码供体模板核酸的核酸序列在相同或不同的载体上。
在一个实施方案中,编码融合蛋白的核酸序列编码编码融合蛋白,所述融合蛋白包含与SEQ ID NO:57-98中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,编码融合蛋白的核酸序列编码包含SEQ ID NO:57-98中的一者的序列的融合蛋白。
在一个实施方案中,编码融合蛋白的核酸序列包含与SEQ ID NO:155-196中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码融合蛋白的核酸序列包含SEQ ID NO:155-196中的一者的核酸序列。
在一个实施方案中,U3序列和U5序列对逆转录病毒IN具有特异性。例如,在一个实施方案中,逆转录病毒IN是HIV IN,并且U3序列包含与SEQ ID NO:197至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:198至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是RSV IN,并且U3序列包含与SEQ ID NO:199至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:20095%同一的序列。
在一个实施方案中,逆转录病毒IN是HFV IN,并且U3序列包含与SEQ ID NO:201至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:202至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是EIAV IN,并且U3序列包含与SEQ ID NO:203至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:204至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是MoLV IN,并且U3序列包含与SEQ ID NO:205至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:206至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是MMTV IN,并且U3序列包含与SEQ ID NO:207至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:208至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是WDSV IN,并且U3序列包含与SEQ ID NO:209至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:210至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是BLV IN,并且U3序列包含与SEQ ID NO:211至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:212至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是SIV IN,并且U3序列包含与SEQ ID NO:213至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:214至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是FIV IN,并且U3序列包含与SEQ ID NO:215至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:216至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,逆转录病毒IN是BIV IN,并且U3序列包含与SEQ ID NO:217至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:218至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,IN是TY1,并且U3序列包含与SEQ ID NO:219至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:220至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
在一个实施方案中,IN是InsF IN,并且U3序列是IS3 IRL序列,并且U5序列是IS3IRR序列。在一个实施方案中,IN是InsF IN,并且U3序列包含与SEQ ID NO:221至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列,并且U5序列包含与SEQ ID NO:222至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。
可设计系统和载体以在原核或真核细胞中表达CRISPR转录物(例如核酸转录物、蛋白质或酶)。例如,CRISPR转录物可表达于诸如大肠杆菌的细菌细胞、昆虫细胞(使用杆状病毒表达载体)、酵母细胞或哺乳动物细胞中。适合的宿主细胞进一步讨论于Goeddel,GENEEXPRESSION TECHNOLOGY:METHODS IN ENZYMOLOGY 185,Academic Press,San Diego,Calif.(1990)中。或者,可例如使用T7启动子调控序列和T7聚合酶在体外转录和翻译重组表达载体。
载体可被引入原核生物中并且在其中增殖。在一些实施方案中,原核生物用于扩增待引入真核细胞中的载体或作为产生待引入真核细胞中的载体中的中间载体的拷贝(例如,扩增质粒作为病毒载体包装系统的一部分)。在一些实施方案中,原核生物用于扩增载体的多个拷贝并且表达一种或多种核酸,以便提供用于递送至宿主细胞或宿主生物体的一种或多种蛋白质的来源。蛋白质在原核生物中的表达最经常在大肠杆菌中用含有指导融合蛋白或非融合蛋白的表达的组成型或诱导型启动子的载体来进行。融合载体将许多氨基酸添加至其中所编码的蛋白质上,如添加至重组蛋白的氨基末端。此类融合载体可用于一个或多个目的,如:(i)增加重组蛋白的表达;(ii)增加重组蛋白的溶解性;以及(iii)通过在亲和纯化中充当配体来帮助纯化重组蛋白。经常,在融合表达载体中,将蛋白裂解位点引入在融合部分与重组蛋白的接点处以使得能够在纯化融合蛋白之后将重组蛋白与融合部分分离。此类酶以及其同源识别序列包括因子Xa、凝血酶和肠激酶。融合表达载体的实例包括pGEX(Pharmacia Biotech Inc;Smith和Johnson,1988.Gene 67:31-40)、pMAL(NewEngland Biolabs,Beverly,Mass.)和pRIT5(Pharmacia,Piscataway,N.J.),它们分别将谷胱甘肽S-转移酶(GST)、麦芽糖E结合蛋白或蛋白A融合至靶重组蛋白。
合适的诱导型非融合大肠杆菌表达载体的实例包括pTrc(Amrann等人,(1988)Gene 69:301-315)和pET 11d(Studier等人,GENE EXPRESSION TECHNOLOGY:METHODS INENZYMOLOGY 185,Academic Press,San Diego,Calif.(1990)60-89)。
在一些实施方案中,载体是酵母表达载体。用于在酵母酿酒酵母中表达的载体的实例包括pYepSec1(Baldari,等人,1987.EMBO J.6:229-234)、pMFa(Kuijan和Herskowitz,1982.Cell 30:933-943)、pJRY88(Schultz等人,1987.Gene 54:113-123)、pYES2(Invitrogen Corporation,San Diego,Calif.)和picZ(InVitrogen Corp,San Diego,Calif.)。
在一些实施方案中,载体使用杆状病毒表达载体驱动昆虫细胞中的蛋白质表达。可用于在培养的昆虫细胞(例如SF9细胞)中表达蛋白质的杆状病毒载体包括pAc系列(Smith,等人,1983.Mol.Cell.Biol.3:2156-2165)和pVL系列(Lucklow和Summers,1989.Virology 170:31-39)。
在一些实施方案中,使用哺乳动物表达载体,载体能够驱动一种或多种序列在哺乳动物细胞中表达。哺乳动物表达载体的实例包括pCDM8(Seed,1987.Nature 329:840)和pMT2PC(Kaufman,等人,1987.EMBO J.6:187-195)。当用于哺乳动物细胞中时,表达载体的控制功能通常由一种或多种调控元件提供。例如,通常使用的启动子来源于多瘤病毒、腺病毒2、巨细胞病毒、猿猴病毒40以及本文公开和本领域已知的其它启动子。对于用于原核和真核细胞的其它合适的表达系统,参见例如,Sambrook,等人,MOLECULAR CLONING:ALABORATORY MANUAL.第2版,Cold Spring Harbor Laboratory,Cold Spring HarborLaboratory Press,Cold Spring Harbor,N.Y.,1989的第16和17章。
在一些实施方案中,重组哺乳动物表达载体能够指导核酸优先在特定细胞类型中表达(例如,使用组织特异型调控元件来表达核酸)。组织特异性调控元件是本领域已知的。合适的组织特异性启动子的非限制性实例包括白蛋白启动子(肝特异性;Pinkert,等人,1987.Genes Dev.1:268-277)、淋巴特异性启动子(Calame和Eaton,1988.Adv.Immunol.43:235-275),特别是T细胞受体(Winoto和Baltimore,1989.EMBO J.8:729-733)和免疫球蛋白(Baneiji,等人,1983.Cell 33:729-740;Queen和Baltimore,1983.Cell 33:741-748)的启动子、神经元特异性启动子(例如,神经丝启动子;Byrne和Ruddle,1989.Proc.Natl.Acad.Sci.USA 86:5473-5477)、胰腺特异性启动子(Edlund,等人,1985.Science 230:912-916)和乳腺特异性启动子(例如,乳清启动子;美国专利号4,873,316和欧洲申请公布号264,166)。还涵盖发育调控启动子,例如鼠hox启动子(Kessel和Gruss,1990.Science 249:374-379)和α-胎蛋白启动子(Campes和Tilghman,1989.GenesDev.3:537-546)。
在一些实施方案中,调控元件可操作地连接至CRISPR系统的一个或多个元件,以驱动CRISPR系统的一个或多个元件的表达。一般而言,CRISPR(成簇规律间隔的短回文重复序列),也称为SPIDR(SPacer间隔的正向重复序列),构成通常对于特定细菌物种而言特异性的DNA基因座的家族。CRISPR基因座包含在大肠杆菌中识别的一类独特的散布短序列重复序列(SSR)(Ishino等人,J.Bacteriol.,169:5429-5433[1987];和Nakata等人,J.Bacteriol.,171:3553-3556[1989])以及相关的基因。在地中海嗜盐菌、酿脓链球菌、鱼腥藻和结核分枝杆菌中也鉴定了类似的散布SSR(参见Groenen等人,Mol.Microbiol.,10:1057-1065[1993];Hoe等人,Emerg.Infect.Dis.,5:254-263[1999];Masepohl等人,Biochim.Biophys.Acta 1307:26-30[1996];以及Mojica等人,Mol.Microbiol.,17:85-93[1995])。CRISPR基因座通常与其它SSR的不同之处在于重复序列的结构,所述重复序列被称为短规律间隔的重复序列(SRSR)(Janssen等人,OMICS J.Integ.Biol.,6:23-33[2002];和Mojica等人,Mol.Microbiol.,36:244-246[2000])。一般而言,所述重复序列是以簇存在的短元件,其被具有基本上恒定长度的独特插入序列规律地间隔开(Mojica等人,[2000],同上)。尽管重复序列在菌株之间高度保守,但是散布的重复序列的数量和间隔区的序列通常因菌株而异(van Embden等人,J.Bacteriol.,182:2393-2401[2000])。已在超过40种原核生物中鉴定了CRISPR基因座(参见例如,Jansen等人,Mol.Microbiol.,43:1565-1575[2002];和Mojica等人,[2005]),包括但不限于气火菌属、火棒菌属、硫化叶菌属、古生球菌属、嗜盐小盒菌属(Halocarcula)、甲烷杆菌属、产甲烷球菌属、甲烷八叠球菌属、甲烷火菌属、火球菌属、嗜酸菌属、Thernioplasnia、棒状杆菌属、分枝杆菌属、链霉菌属、产液菌属(Aquifrx)、卟啉单胞菌属(Porphvromonas)、绿菌属、栖热菌属、芽孢杆菌属、李斯特菌属、葡萄球菌属、梭菌属、高温厌氧杆菌属、支原体属、梭形杆菌属、Azarcus、色杆菌属、奈瑟氏菌属、亚硝化单胞菌属、脱硫弧菌属、土杆菌属、Myrococcus、弯曲杆菌属、沃林氏菌属、不动杆菌属、欧文氏菌属、埃希氏菌属、军团菌属、甲基球菌属、巴斯德氏菌属、发光杆菌属、沙门氏菌属、黄单胞菌属、耶尔森氏菌属、密螺旋体属和栖热袍菌属。
如本文所用,“靶序列”是指向导序列被设计成与其具有互补性的序列,其中靶序列与向导序列之间的杂交促进CRISPR复合物的形成。不一定需要完全互补性,条件是存在足够的互补性以引起杂交并促进CRISPR复合物的形成。靶序列可包含任何多核苷酸,如DNA或RNA多核苷酸。在一些实施方案中,靶序列位于细胞的细胞核或细胞质中。在一些实施方案中,靶序列可在真核细胞的细胞器内,例如线粒体或叶绿体内。可用于重组到包含靶序列的靶向基因座中的序列或模板被称为“编辑模板”或“编辑多核苷酸”或“编辑序列”。在本发明的方面,外源性模板多核苷酸可被称为编辑模板。在本发明的一个方面,重组是同源重组。
向导序列可被选择为靶向任何靶序列。在一些实施方案中,靶序列是细胞基因组内的序列。示例性靶序列包括靶基因组中独特的那些。例如,对于酿脓链球菌Cas9,基因组中的独特靶序列可包含MMMMMMMMNNNNNNNNNNNNXGG形式的Cas9靶位点,其中NNNNNNNNNNNNXGG(N是A、G、T或C;并且X可以是任何物质)在基因组中只出现一次。基因组中的独特靶序列可包含MMMMMMMMMNNNNNNNNNNNXGG形式的酿脓链球菌Cas9靶位点,其中NNNNNNNNNNNXGG(N是A、G、T或C;并且X可以是任何物质)在基因组中只出现一次。对于嗜热链球菌CRISPR1Cas9,基因组中的独特靶序列可包含MMMMMMMMNNNNNNNNNNNNXXAGAAW(SEQID NO:1)形式的Cas9靶位点,其中NNNNNNNNNNNNXXAGAAW(SEQ ID NO:2)(N是A、G、T或C;X可以是任何物质;并且W是A或T)在基因组中只出现一次。C基因组中的独特靶序列可包含MMMMMMMMMNNNNNNNNNNNXXAGAAW(SEQ ID NO:3)形式的嗜热链球菌CRISPR1 Cas9靶位点,其中NNNNNNNNNNNXXAGAAW(SEQ ID NO:4)(N是A、G、T或C;X可以是任何物质;并且W是A或T)在基因组中只出现一次。对于酿脓链球菌Cas9,基因组中的独特靶序列可包含MMMMMMMMNNNNNNNNNNNNXGGXG形式的Cas9靶位点,其中NNNNNNNNNNNNXGGXG(N是A、G、T或C;并且X可以是任何物质)在基因组中只出现一次。基因组中的独特靶序列可包含MMMMMMMMMNNNNNNNNNNNXGGXG形式的酿脓链球菌Cas9靶位点,其中NNNNNNNNNNNXGGXG(N是A、G、T或C;并且X可以是任何物质)在基因组中只出现一次。在这些序列的每一个中,“M”可以是A、G、T或C,并且在将序列鉴定为独特时无需考虑。
在一些实施方案中,选择向导序列以降低向导序列内的二级结构的程度。可通过任何合适的多核苷酸折叠算法来确定二级结构。一些程序是基于计算最小吉布斯(Gibbs)自由能。一种这样的算法的实例是mFold,如Zuker和Stiegler(Nucleic Acids Res.9(1981),133-148)所描述。折叠算法的另一个实例是在线网络服务器RNAfold,它是由维也纳大学理论化学研究所开发的,使用质心结构预测算法(参见例如A.R.Gruber等人,2008,Cell 106(1):23-24;以及PA Carr和GM Church,2009,Nature Biotechnology 27(12):1151-62)。
一般来说,tracr配对序列包括与tracr序列具有足够互补性以促进以下中的一种或多种的任何序列:(1)在含有相应tracr序列的细胞中,侧接tracr配对序列的向导序列的切除;和(2)在靶序列处形成CRISPR复合物,其中所述CRISPR复合物包含与tracr序列杂交的tracr配对序列。一般而言,互补性程度是关于沿着两个序列中较短者的长度,tracr配对序列和tracr序列的最佳比对。最佳比对可通过任何合适的比对算法来确定,并且可进一步考虑二级结构,如tracr序列或tracr配对序列内的自身互补性。在一些实施方案中,当最佳比对时,沿着两者中较短者的长度,tracr序列与tracr配对序列之间的互补性程度是约或大于约25%、30%、40%、50%、60%、70%、80%、90%、95%、97.5%、99%或更高。在一些实施方案中,tracr序列的长度是约或多于约5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、25、30、40、50个或更多个核苷酸。在一些实施方案中,tracr序列和tracr配对序列包含在单个转录物中,使得两者之间的杂交产生具有二级结构(如发夹)的转录物。在一个实施方案中,用于发夹结构的环形成序列的长度是四个核苷酸。在一个实施方案中,用于发夹结构的环形成序列具有序列GAAA。然而,可使用更长或更短的环序列,也可使用替代序列。序列可包含核苷酸三联体(例如AAA)和另外的核苷酸(例如C或G)。环形成序列的实例包括CAAA和AAAG。在本发明的一个实施方案中,转录物或转录的多核苷酸序列具有至少两个或更多个发夹。在一些实施方案中,转录物具有两个、三个、四个或五个发夹。在本发明的另一实施方案中,转录物具有至多五个发夹。在一些实施方案中,单个转录物还包含转录终止序列;在一些实施方案中,这是polyT序列,例如六个T核苷酸。
在一个实施方案中,本发明提供了编辑遗传物质如核酸分子、基因组或基因的方法。例如,在一个实施方案中,编辑是整合。在一个实施方案中,编辑是CIRSPR介导的编辑。
在一个实施方案中,所述方法包括向所述遗传物质施用:编码融合蛋白的核酸分子;包含与所述遗传物质中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,所述方法包括向所述遗传物质施用:融合蛋白;包含与所述遗传物质中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,所述方法是体外方法或体内方法。
在一个实施方案中,本发明提供了将核酸序列递送至遗传物质的方法。在一个实施方案中,所述方法包括向所述基因施用:编码融合蛋白的核酸分子;包含与所述基因中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,所述方法包括向所述遗传物质施用:融合蛋白;包含与所述遗传物质中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,所述方法是体外方法或体内方法。
在一个实施方案中,所述方法包括向细胞施用:编码融合蛋白的核酸分子;包含与所述基因中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,所述方法包括向细胞施用:融合蛋白;包含与所述基因中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。
在一个实施方案中,编辑遗传物质的方法是编辑基因的方法。在一个实施方案中,所述基因位于细胞的基因组中。在一个实施方案中,编辑遗传物质的方法是编辑核酸的方法。
在一个实施方案中,本发明提供了将供体模板序列插入靶序列中的方法。在一个实施方案中,所述方法将供体模板序列插入细胞中的靶序列中。在一个实施方案中,所述方法包括向所述细胞施用:编码融合蛋白的核酸分子;包含与所述靶序列中的区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,所述方法包括向所述细胞施用:融合蛋白;包含与所述靶序列中的区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。
使用CRISPR-Cas9介导的HDR用于基因组编辑的大DNA序列的靶向递送仍然效率低下。然而,本发明提供了用于将大供体模板序列插入细胞中的靶序列制剂的方法。在一个实施方案中,所述方法插入至少1kb或更大、至少2kb或更大、至少3kb或更大、至少4kb或更大、至少5kb或更大、至少6kb或更大、至少7kb或更大、至少8kb或更大、至少9kb或更大、至少10kb或更大、至少11kb或更大、至少12kb或更大、至少13kb或更大、至少14kb或更大、至少15kb或更大、至少16kb或更大、至少17kb或更大、或至少18kb或更大的供体模板序列。在一个实施方案中,所述方法包括向所述细胞施用:融合蛋白或编码融合蛋白的核酸分子;包含与所述靶序列中的区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。
在一个实施方案中,靶序列位于基因内。在一个实施方案中,供体模板序列破坏基因的序列,从而抑制或减少基因的表达。在一个实施方案中,靶序列具有突变,并且供体模板序列将校正的序列插入靶序列中,从而校正所述基因突变。在一个实施方案中,供体模板序列是基因序列,并且将所述供体模板序列插入细胞中的靶序列中允许表达所述基因。
在一个实施方案中,将供体模板序列插入安全港位点中。因此,在一个实施方案中,向导核酸包含与基因中的安全港区域互补的核苷酸序列。安全港区域允许在不影响邻近基因表达的情况下表达治疗性基因。安全港区域可包括远离邻近基因例如H11的基因间区域,或‘非必需’基因例如CCR5、hROSA26或AAVS1内的基因间区域。示例性的安全港区域和与这些序列互补的向导核酸序列可例如在Pellenz等人,New Human Chromosomal Siteswith“Safe Harbor”Potential for Targeted Transgene Insertion,2019,Hum GeneTher 30(7):814-28中找到,所述文献以引用的方式并入本文。
在一个实施方案中,将供体模板序列插入3'非翻译区(UTR)中,从而允许供体模板序列的表达受其它基因的启动子控制。
在一个实施方案中,核酸分子包含编码逆转录病毒整合酶(IN)或其片段的第一核酸序列;编码CRISPR相关(Cas)蛋白的第二核酸序列;以及编码核定位信号(NLS)的第三核酸序列。
在一个实施方案中,逆转录病毒IN是人免疫缺陷病毒(HIV)IN、劳斯肉瘤病毒(RSV)IN、小鼠乳腺肿瘤病毒(MMTV)IN、莫洛尼鼠白血病病毒(MoLV)IN、牛白血病病毒(BLV)IN、人嗜T淋巴细胞病毒(HTLV)IN、禽肉瘤白血病病毒(ASLV)IN、猫白血病病毒(FLV)IN、异嗜性鼠白血病病毒相关病毒(XMLV)IN、猿猴免疫缺陷病毒(SIV)IN、猫免疫缺陷病毒(FIV)IN、马传染性贫血病毒(EIAV)IN、原型泡沫病毒(PFV)IN、猿猴泡沫病毒(SFV)IN、人泡沫病毒(HFV)IN、大眼鲈皮肤肉瘤病毒(WDSV)IN或牛免疫缺陷病毒(BIV)IN。
在一个实施方案中,逆转录病毒IN是HIV IN。在一个实施方案中,HIV IN包含一个或多个氨基酸取代,其中所述取代提高催化活性,提高溶解度或增加与一种或多种宿主细胞辅因子的相互作用。在一个实施方案中,HIV IN包含选自由以下组成的组的一个或多个氨基酸取代:E85G、E85F、D116N、F185K、C280S、T97A、Y134R、G140S和Q148H。在一个实施方案中,HIV IN包含氨基酸取代F185K和C280S。在一个实施方案中,HIV IN包含氨基酸取代T97A和Y134R。在一个实施方案中,HIV IN包含氨基酸取代G140S和Q148H。
在一个实施方案中,逆转录病毒IN片段包含IN N末端结构域(NTD)和IN催化核心结构域(CCD)。在一个实施方案中,逆转录病毒IN片段包含IN CCD和IN C-末端结构域(CTD)。在一个实施方案中,逆转录病毒IN片段包含IN NTD。在一个实施方案中,逆转录病毒IN片段包含IN CCD。在一个实施方案中,逆转录病毒IN片段包含IN CTD。
在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含编码与SEQ ID NO:1-40中的一者至少95%同一的序列的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含编码与SEQ ID NO:1-40中的一者至少96%同一的序列的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含编码与SEQ ID NO:1-40中的一者至少97%同一的序列的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含编码与SEQ ID NO:1-40中的一者至少98%同一的序列的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含编码与SEQ ID NO:1-40中的一者至少99%同一的序列的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含编码SEQ IDNO:1-40中的一者的核酸序列。
在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含与SEQ ID NO:99-138中的一者至少95%同一的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含与SEQ ID NO:99-138中的一者至少96%同一的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含与SEQ ID NO:99-138中的一者至少97%同一的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含与SEQ ID NO:99-138中的一者至少98%同一的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含与SEQ ID NO:99-138中的一者至少99%同一的核酸序列。在一个实施方案中,编码逆转录病毒IN的第一核酸序列包含SEQ ID NO:99-138中的一者的核酸序列。
在一个实施方案中,Cas蛋白是Cas9、Cas13或Cpf1。在一个实施方案中,Cas蛋白是催化缺陷型的(dCas)。
在一个实施方案中,编码Cas蛋白的第二核酸序列包含编码与SEQ ID NO:41-46中的一者至少95%同一的序列的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含编码与SEQ ID NO:41-46中的一者至少96%同一的序列的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含编码与SEQ ID NO:41-46中的一者至少97%同一的序列的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含编码与SEQ ID NO:41-46中的一者至少98%同一的序列的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含编码与SEQ ID NO:41-46中的一者至少99%同一的序列的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含编码SEQ ID NO:41-46中的一者的核酸序列。
在一个实施方案中,编码Cas蛋白的第二核酸序列包含与SEQ ID NO:139-144中的一者至少95%同一的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含与SEQ ID NO:139-144中的一者至少96%同一的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含与SEQ ID NO:139-144中的一者至少97%同一的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含与SEQ ID NO:139-144中的一者至少98%同一的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含与SEQ ID NO:139-144中的一者至少99%同一的核酸序列。在一个实施方案中,编码Cas蛋白的第二核酸序列包含SEQ ID NO:139-144中的一者的核酸序列。
在一个实施方案中,NLS是逆转录转座子NLS。在一个实施方案中,NLS源自酵母GAL4、SKI3、L29或组蛋白H2B蛋白、多瘤病毒大T蛋白、VP1或VP2衣壳蛋白、SV40 VP1或VP2衣壳蛋白、腺病毒El a或DBP蛋白、流感病毒NS1蛋白、肝炎病毒核心抗原或哺乳动物核纤层蛋白、c-myc、max、c-myb、p53、c-erbA、jun、Tax、类固醇受体或Mx蛋白或猿猴vims 40(“SV40”)T抗原。在一个实施方案中,NLS是Ty1或Ty1来源的NLS、Ty2或Ty2来源的NLS或MAK11或MAK11来源的NLS。在一个实施方案中,Ty1 NLS包含SEQ ID NO:51的氨基酸序列。在一个实施方案中,Ty2 NLS包含SEQ ID NO:254的氨基酸序列。在一个实施方案中,MAK11 NLS包含SEQ IDNO:256的氨基酸序列。
在一个实施方案中,编码NLS的第三核酸序列包含编码与SEQ ID NO:47-56中的一者至少95%同一的序列的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含编码与SEQ ID NO:47-56中的一者至少96%同一的序列的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含编码与SEQ ID NO:47-56中的一者至少97%同一的序列的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含编码与SEQ ID NO:47-56中的一者至少98%同一的序列的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含编码与SEQ ID NO:47-56中的一者至少99%同一的序列的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含编码SEQ ID NO:47-56中的一者的核酸序列。
在一个实施方案中,编码NLS的第三核酸序列包含与SEQ ID NO:145-154中的一者至少95%同一的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含与SEQ ID NO:145-154中的一者至少96%同一的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含与SEQ ID NO:145-154中的一者至少97%同一的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含与SEQ ID NO:145-154中的一者至少98%同一的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含与SEQ ID NO:145-154中的一者至少99%同一的核酸序列。在一个实施方案中,编码NLS的第三核酸序列包含SEQ ID NO:145-154中的一者的核酸序列。
在一个实施方案中,核酸分子编码融合蛋白,所述融合蛋白包含与SEQ ID NO:57-98中的一者至少95%同一的序列。在一个实施方案中,核酸分子编码融合蛋白,所述融合蛋白包含与SEQ ID NO:57-98中的一者至少96%同一的序列。在一个实施方案中,核酸分子编码融合蛋白,所述融合蛋白包含与SEQ ID NO:57-98中的一者至少97%同一的序列。在一个实施方案中,核酸分子编码融合蛋白,所述融合蛋白包含与SEQ ID NO:57-98中的一者至少98%同一的序列。在一个实施方案中,核酸分子编码融合蛋白,所述融合蛋白包含与SEQ IDNO:57-98中的一者至少99%同一的序列。在一个实施方案中,核酸分子编码包含SEQ IDNO:57-98中的一者的序列的融合蛋白。
在一个实施方案中,核酸分子包含与SEQ ID NO:155-196中的一者至少95%同一的核酸序列。在一个实施方案中,核酸分子包含与SEQ ID NO:155-196中的一者至少96%同一的核酸序列。在一个实施方案中,核酸分子包含与SEQ ID NO:155-196中的一者至少97%同一的核酸序列。在一个实施方案中,核酸分子包含与SEQ ID NO:155-196中的一者至少98%同一的核酸序列。在一个实施方案中,核酸分子包含与SEQ ID NO:155-196中的一者至少99%同一的核酸序列。在一个实施方案中,核酸分子包含SEQ ID NO:155-196中的一者的核酸序列。
在一个实施方案中,U3序列和U5序列对逆转录病毒IN具有特异性。
在一些实施方案中,基因是任何目标靶基因。例如,在一个实施方案中,基因是与患病或发展疾病的风险增加相关的任何基因。在一些实施方案中,所述方法包括引入编码融合蛋白的核酸分子;包含与所述基因中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,IN-Cas9融合蛋白结合至靶多核苷酸以实现所述基因内靶多核苷酸的裂解。在一个实施方案中,IN-Cas9融合蛋白与向导核酸复合,所述向导核酸与靶多核苷酸内的靶序列杂交。在一个实施方案中,IN-Cas9融合蛋白与编码供体模板核酸的核酸序列复合。在一个实施方案中,IN-Cas9融合蛋白与编码向导核酸的核酸序列复合。在一个实施方案中,IN-Cas9融合蛋白与编码向导核酸的核酸序列和编码供体模板核酸的核酸序列复合。在一个实施方案中,IN-Cas9融合蛋白与向导核酸和供体模板核酸复合,所述向导核酸与靶多核苷酸内的靶序列杂交。在一个实施方案中,IN-Cas9融合蛋白与供体模板核酸复合。在一个实施方案中,IN-Cas9融合蛋白与向导核酸复合。在一个实施方案中,IN-Cas9融合蛋白与向导核酸和供体模板核酸复合。
在一些实施方案中,IN-Cas9催化供体模板整合到基因中。在一个实施方案中,所述整合将一种或多种突变引入到基因中。在一些实施方案中,所述突变导致来自包含靶序列的基因的蛋白质表达中的一者或多个氨基酸变化。
在一个实施方案中,IN介导的DNA序列整合可在靶DNA序列的任一方向上发生。在一个实施方案中,Cas和IN逆转录病毒类别蛋白的不同组合用于促进定向编辑。例如,在一个实施方案中,来自逆转录病毒类别的IN的融合物结合至第一催化性死亡Cas,从而允许利用Cas特异性向导RNA结合至特定靶序列。在一个实施方案中,供体序列包含HIV和BIV LTR序列。因此,在一个实施方案中,所述序列与靶DNA以单一取向整合。
在一个实施方案中,在目标基因周围并入侧接LoxP(Floxed)序列。包含floxed序列允许CRE介导的重组和条件诱变。使用CRISPR-Cas9产生Floxed等位基因的当前方法效率低下。最广泛使用的方法是使用两种向导RNA来诱导侧接靶序列处的DNA裂解,并使用同源定向修复来插入含有LoxP序列的ssDNA模板。然而,当使用双sgRNA来诱导裂解时,最有利的反应是插入序列的缺失,从而导致全基因缺失。因此,在一个实施方案中,使用整合酶-Cas介导的基因插入增加了DNA序列的串联插入的效率。在一个实施方案中,含有反向LoxP序列的序列的整合允许侧接LoxP序列的重组,因为IN介导的整合可在任一方向上发生。
本发明提供了治疗疾病或病症、减轻所述疾病或病症的症状和/或降低发展所述疾病或病症的风险和/或遗传修饰以产生所需表型结果的方法。例如,在一个实施方案中,本发明的方法治疗哺乳动物的疾病或病症、减轻所述疾病或病症的症状和/或降低发展所述疾病或病症的风险。在一个实施方案中,本发明的方法治疗植物中的疾病或病症、减轻所述疾病或病症的症状和/或降低发展所述疾病或病症的风险。在一个实施方案中,本发明的方法治疗酵母生物体中的疾病或病症、减轻所述疾病或病症的症状和/或降低发展所述疾病或病症的风险。
在一个实施方案中,所述疾病或病症由基因组基因座中的一种或多种突变引起。因此,在一个实施方案中,通过引入与具有所述一种或多种突变的区域的野生型序列相对应的核酸序列和/或引入阻止或减少具有所述一种或多种突变的基因组序列的表达的元件,可治疗、减轻所述疾病或病症或者可降低所述风险。因此,在一个实施方案中,所述方法包括在靶序列中的基因组基因座的编码、非编码或调控元件内操纵靶序列。
例如,在一个实施方案中,疾病是单基因疾病。在一个实施方案中,所述疾病包括但不限于:杜氏肌营养不良(肌营养不良蛋白中存在突变)、肢带型肌营养不良2B型(LGMD2B)和Miyoshi肌病(Dysferlin中存在突变)、囊性纤维化(CFTR中存在突变)、威尔逊氏病(ATP7B中存在突变)和斯塔加特(Stargardt)黄斑变性(ABCA4中存在突变)。
本发明还提供了调节基因或遗传物质的表达的方法。例如,在一个实施方案中,本发明的方法提供了递送遗传物质以在细胞或生物体中赋予表型。例如,在一个实施方案中,所述方法提供对病原体的抗性。在一个实施方案中,所述方法提供代谢途径的调节。在一个实施方案中,所述方法提供生物体中的物质的产生和使用。例如,在一个实施方案中,所述方法在诸如真核生物、酵母、细菌或植物的生物体中产生诸如生物制剂、药物和生物燃料的物质。
在一个实施方案中,所述方法包括施用融合蛋白或编码融合蛋白的核酸分子;包含与所述基因中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列的供体模板核酸。在一个实施方案中,所述方法还包括施用供体模板序列。
在一个实施方案中,靶序列位于基因内。在一个实施方案中,供体模板序列破坏基因的序列,从而抑制或减少基因的表达。在一个实施方案中,靶序列具有突变,并且供体模板序列将校正的序列插入靶序列中,从而校正所述基因突变。在一个实施方案中,供体模板序列是基因序列,并且将所述供体模板序列插入细胞中的靶序列中允许表达所述基因。
在一个实施方案中,融合蛋白包含CRISPR相关(Cas)蛋白和核定位信号(NLS)。在一个实施方案中,融合蛋白包含Cas蛋白、NLS和逆转录病毒整合酶(IN)或其片段。
在一个实施方案中,逆转录病毒IN是人免疫缺陷病毒(HIV)IN、劳斯肉瘤病毒(RSV)IN、小鼠乳腺肿瘤病毒(MMTV)IN、莫洛尼鼠白血病病毒(MoLV)IN、牛白血病病毒(BLV)IN、人嗜T淋巴细胞病毒(HTLV)IN、禽肉瘤白血病病毒(ASLV)IN、猫白血病病毒(FLV)IN、异嗜性鼠白血病病毒相关病毒(XMLV)IN、猿猴免疫缺陷病毒(SIV)IN、猫免疫缺陷病毒(FIV)IN、马传染性贫血病毒(EIAV)IN、原型泡沫病毒(PFV)IN、猿猴泡沫病毒(SFV)IN、人泡沫病毒(HFV)IN、大眼鲈皮肤肉瘤病毒(WDSV)IN或牛免疫缺陷病毒(BIV)IN。
在一个实施方案中,逆转录病毒IN是HIV IN。在一个实施方案中,HIV IN包含一个或多个氨基酸取代,其中所述取代提高催化活性,提高溶解度或增加与一种或多种宿主细胞辅因子的相互作用。在一个实施方案中,HIV IN包含选自由以下组成的组的一个或多个氨基酸取代:E85G、E85F、D116N、F185K、C280S、T97A、Y134R、G140S和Q148H。在一个实施方案中,HIV IN包含氨基酸取代F185K和C280S。在一个实施方案中,HIV IN包含氨基酸取代T97A和Y134R。在一个实施方案中,HIV IN包含氨基酸取代G140S和Q148H。
在一个实施方案中,逆转录病毒IN片段包含IN N末端结构域(NTD)和IN催化核心结构域(CCD)。在一个实施方案中,逆转录病毒IN片段包含IN CCD和IN C-末端结构域(CTD)。在一个实施方案中,逆转录病毒IN片段包含IN NTD。在一个实施方案中,逆转录病毒IN片段包含IN CCD。在一个实施方案中,逆转录病毒IN片段包含IN CTD。
在一个实施方案中,逆转录病毒IN包含与SEQ ID NO:1-40中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,逆转录病毒IN包含SEQ ID NO:1-40中的一者的序列。
在一个实施方案中,编码逆转录病毒IN的核酸包含与SEQ ID NO:99-138至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码逆转录病毒IN的核酸包含SEQ ID NO:99-138中的一者的核酸序列。
在一个实施方案中,Cas蛋白是Cas9、Cas13或Cpf1。在一个实施方案中,Cas蛋白是催化缺陷型的(dCas)。
在一个实施方案中,Cas蛋白包含与SEQ ID NO:41-46中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,Cas蛋白包含SEQ ID NO:41-46中的一者的序列。
在一个实施方案中,编码Cas蛋白的核酸序列包含与SEQ ID NO:139-144中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码Cas蛋白的核酸序列包含SEQ ID NO:139-144中的一者的核酸序列。
在一个实施方案中,NLS是逆转录转座子NLS。在一个实施方案中,NLS源自酵母GAL4、SKI3、L29或组蛋白H2B蛋白、多瘤病毒大T蛋白、VP1或VP2衣壳蛋白、SV40 VP1或VP2衣壳蛋白、腺病毒El a或DBP蛋白、流感病毒NS1蛋白、肝炎病毒核心抗原或哺乳动物核纤层蛋白、c-myc、max、c-myb、p53、c-erbA、jun、Tax、类固醇受体或Mx蛋白或猿猴vims 40(“SV40”)T抗原。在一个实施方案中,NLS是Ty1或Ty1来源的NLS、Ty2或Ty2来源的NLS或MAK11或MAK11来源的NLS。在一个实施方案中,Ty1 NLS包含SEQ ID NO:51的氨基酸序列。在一个实施方案中,Ty2 NLS包含SEQ ID NO:254的氨基酸序列。在一个实施方案中,MAK11 NLS包含SEQ IDNO:256的氨基酸序列。
在一个实施方案中,NLS包含核酸序列,所述核酸序列编码与SEQ ID NO:47-56、254-256和275-887中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,NLS包含编码SEQ ID NO:47-56、254-256和275-887中的一者的核酸序列。
在一个实施方案中,编码NLS的核酸序列包含与SEQ ID NO:145-154中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的核酸序列。在一个实施方案中,编码NLS的核酸序列包含SEQ ID NO:145-154中的一者的核酸序列。
在一个实施方案中,融合蛋白包含与SEQ ID NO:57-98中的一者至少70%、至少71%、至少72%、至少73%、至少74%、至少75%、至少76%、至少77%、至少78%、至少79%、80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%同一的序列。在一个实施方案中,融合蛋白包含SEQ ID NO:57-98中的一者的序列。
在一个实施方案中,U3序列和U5序列对逆转录病毒IN具有特异性。
在一些实施方案中,基因是任何目标靶基因。例如,在一个实施方案中,基因是与患病或发展疾病的风险增加相关的任何基因。在一些实施方案中,所述方法包括引入编码融合蛋白的核酸分子;包含与所述基因中的靶区域互补的靶向核苷酸序列的向导核酸;以及包含U3序列、U5序列和供体模板序列的供体模板核酸。在一个实施方案中,IN-Cas9融合蛋白结合至靶多核苷酸以实现所述基因内靶多核苷酸的裂解。在一个实施方案中,IN-Cas9融合蛋白与向导核酸复合,所述向导核酸与靶多核苷酸内的靶序列杂交。在一个实施方案中,IN-Cas9融合蛋白与编码供体模板核酸的核酸序列复合。在一个实施方案中,IN-Cas9融合蛋白与编码向导核酸的核酸序列复合。在一个实施方案中,IN-Cas9融合蛋白与编码向导核酸的核酸序列和编码供体模板核酸的核酸序列复合。在一个实施方案中,IN-Cas9融合蛋白与向导核酸和供体模板核酸复合,所述向导核酸与靶多核苷酸内的靶序列杂交。在一个实施方案中,IN-Cas9融合蛋白与供体模板核酸复合。在一个实施方案中,IN-Cas9融合蛋白与向导核酸复合。在一个实施方案中,IN-Cas9融合蛋白与向导核酸和供体模板核酸复合。
在一些实施方案中,IN-Cas9催化供体模板整合到基因中。在一个实施方案中,所述整合将一种或多种突变引入到基因中。在一些实施方案中,所述突变导致来自包含靶序列的基因的蛋白质表达中的一者或多个氨基酸变化。
实验实施例
可参考以下实验实施例对本发明进一步详述。这些实施例仅出于说明目的而提供且并非意图进行限制,除非另外指明。因此,本发明不应以任何方式被解释为局限于以下实施例,而是应被解释为涵盖因本文提供的教义而变得显而易见的任何和所有变化。
无需进一步描述,据信本领域的普通技术人员可使用前述的描述和以下说明性实施例,制备和利用本发明并实践所要求保护的方法。因此,以下工作实施例特别指出本发明的某些实施方案,并且不应被解释为以任何方式限制本公开的其余部分。
哺乳动物基因组DNA的有效CRISPR-Cas9编辑需要Cas9的核定位,Cas9是通常在缺乏核膜的原核细胞中起作用的一种大的细菌RNA引导的核酸内切酶。研究表明,Cas9在哺乳动物细胞中的有效核定位需要添加至少两种哺乳动物核定位信号,一种位于N末端,并且一种位于C末端(Cong等人,2013,Science 339:819-23)。
为了促进逆转录病毒整合酶-dCas9融合蛋白的核定位以进行编辑,除了dCas9上的C末端SV40 NLS之外,整合酶上还包含N末端SV40 NLS(图1A)。出人意料地,当在哺乳动物细胞中表达时,只有一小部分的IN-dCas9融合蛋白被核定位,如使用识别dCas9上的C末端3xFLAG表位的FLAG抗体检测的(图1B)。令人感兴趣地,虽然全长IN-dCas9融合蛋白产生了细胞质聚集体,但整合酶的C末端结构域的缺失(INΔC)产生更大的溶解度和增加的核定位(图1B)。
逆转录病毒整合酶与dCas9的融合似乎大大降低了其定位于细胞核的能力。为了进一步增强整合酶Cas9融合蛋白的核定位,测试了许多不同的哺乳动物核定位序列指导IN-dCas9核输入的能力(图1B)。对SV40 NLS的3个拷贝的多聚化(3xSV40)对IN-dCas9或INΔC-dCas9的核定位程度没有明显影响。然而,添加来自核质蛋白(NPM)的二分NLS提供增加的INΔC-dCas9融合蛋白核定位,但不提供全长IN融合蛋白的核定位。3xSV40和NPM NLS的组合看起来类似于单独的NPM。
令人感兴趣地,酵母LTR-逆转录转座子(例如Ty1)是逆转录病毒的进化祖先,并且通过逆转录细胞质中的RNA中间体来复制其基因组(Curcio等人,2015,Microbiol Spectr3:MDNA3-0053-2014)。LTR-逆转录转座子含有整合酶,所述整合酶是逆转录转座子基因组的插入所必需的。与在细胞分裂过程中经历开放有丝分裂的高级真核生物相反,酵母经历封闭有丝分裂,由此其核被膜保持完整。因此,对于Ty1生物发生,整合酶/逆转录转座子基因组复合物的核输入需要主动核输入。因此,与哺乳动物整合酶对比,Ty1整合酶含有逆转录转座所需的大的C末端二分体NLS(Moore等人,1998,Mol Cell Biol18:1105-14)。令人感兴趣地,本文呈现的结果证明,Ty1 NLS与两种IN-dCas9融合蛋白的C末端的融合可在哺乳动物细胞中提供稳健的核定位(图1B)。
INΔC-dCas9融合蛋白的增加的核定位显著增强了培养中分裂哺乳动物细胞的编辑。Ty1 NLS的添加增强了INΔC-dCas9融合蛋白的活性,以整合靶向HEK293细胞中的EF1-α的3’UTRE的IRES-mCherry模板(图1C)。利用稳健的Ty1 NLS可进一步允许在始终保持核被膜的非分裂细胞中进行编辑(例如,体内治疗应用)。
如本文其它地方所证明的,慢病毒整合酶与CRISPR-Cas9的融合允许大的DNA序列序列特异性整合到基因组DNA中。这种方法可用于将治疗有益的基因递送至非病原性基因组位置(安全港),以永久校正人类遗传疾病(图2)。这项技术允许含有短病毒末端基序的大的DNA供体序列的序列特异性整合。
本发明的基因治疗方法的主要优点是能够将供体DNA序列递送至靶向基因组位置。此外,这种方法消除了对同源臂的需求,并且依靠由向导RNA进行的靶向,从而大大简化了基因组编辑。因此,一旦产生了特定报告基因供体序列,就可将其引导至任何位置(或多个位置)以用于多种应用。
慢病毒整合酶与dCas9的融合足以在哺乳动物细胞中使用CRISPR向导RNA将含有短病毒末端的供体DNA序列插入靶序列中(图3)。为了监测哺乳动物细胞中整合酶-Cas介导的整合,产生了含有IGR IRES序列、随后mCherry-2a-嘌呤霉素基因和SV40聚腺苷酸化序列的供体载体(图3)。接下来,设计了靶向COS-7细胞中的稳定人CMV-eGFP稳定细胞系的sgRNA。hCMV-eGFP稳定转基因提供了异源靶序列,所述异源靶序列可用于确定在稳健表达、但非必需表达基因座处的编辑。纯化供体mCherry-2a-puro模板,并与sgRNA和IN-dCas9一起共转染到GFP稳定细胞中,并培养48小时。48小时后,mCherry阳性细胞在培养物中可见,并替代GFP阳性信号(图3)。
整合酶-Cas介导的基因递送指导大的DNA序列序列特异性整合到哺乳动物基因组DNA中。整合酶-Cas用于在培养的细胞中使用对人AAVS1和小鼠ROSA26基因组DNA具有特异性的CRISPR向导RNA,在人α-骨骼肌动蛋白(HSA)启动子的控制下将人肌营养不良蛋白基因递送至安全港位置。使用基于PCR的基因分型评估肌营养不良蛋白的正确靶向。
在肌营养不良最常用的小鼠模型MDX小鼠品系中确定Inscritpr介导的人肌营养不良蛋白递送的功效。全身递送后,在2、4和6个月的时间过程中,使用抗肌营养不良蛋白抗体定量并测量肢体骨骼肌、心脏和膈肌中肌营养不良蛋白表达的水平。通过定量血清肌酸激酶(CK)(骨骼肌损伤的标志物和DMD患者的诊断标志物)、握力和肢体骨骼肌、心脏和膈肌的组织学分析来评估DMD疾病发病机制的缓解。
基因表达的组织学分析。
在8周龄时,收获左后肢股四头肌、心脏和膈肌,称重并固定在PBS中的4%甲醛中,并使用石蜡组织学的常规方法进行加工。在DMD
整合酶-Cas介导的递送减轻杜氏肌营养不良的小鼠模型中的疾病发病机制。
对横向组织学切片进行的苏木精和曙红(H&E)、冯库萨(von Kossa)和马森氏(Masson’s)三色染色分别用于鉴定含有中央核、矿化和肌内膜纤维化的肌纤维。定量比较和统计分析用于比较肌纤维与中央核的比率,或比较股四头肌肢体肌肉中染色的矿化或纤维化区域。对于每种肌肉比较来自每种基因型的3只不同小鼠的至少三个不同截面平面。经整合酶-Cas处理的Dmd
血清肌酸激酶(CK)测量。
血清CK是骨骼肌损伤的相关标志物,并且是DMD患者的诊断标志物。在第2周、第4周、第6周和第8周使用非致死程序对上述动物群组进行CK测量。简言之,从眶周血管丛收获的血液直接进入微量血细胞比容管,在室温下凝结30分钟,然后以1,700×g离心10分钟。与Dmd
本文提供的数据证明优化的整合酶-Cas能够实现哺乳动物基因组的有效编辑。
优化的编辑
为了优化IN介导的整合,确定增强整合酶催化活性、溶解度或与宿主细胞辅因子的相互作用的氨基酸突变是否增强编辑。此外,评估了从七种独特类别的逆转录病毒分离的IN蛋白的效率和保真度。
为了定量和表征哺乳动物细胞中IN-dCas9介导的整合,使用了基于质粒的报告基因系统,所述系统利用来自多孔鹿角珊瑚的蓝色色蛋白(amilCP),当在大肠杆菌中表达时所述色蛋白产生深蓝色菌落。对amilCP开放阅读框的破坏消除蓝色蛋白表达,所述蓝色蛋白可用作靶向保真度的直接读数。此外,产生了编码氯霉素抗生素抗性基因的供体模板,所述供体模板侧接来自HIV的U3和U5逆转录病毒末端序列。这种供体模板的整合赋予对氯霉素的抗性,其可用于监测整合酶-Cas介导的DNA整合。在这种报告基因测定中,将含有IN-dCas9融合蛋白、靶向amilCP的sgRNA和供体模板的表达质粒与细菌amilCP报告基因一起共转染到哺乳动物COS-7细胞中。48小时后,使用柱纯化回收总质粒DNA,并转化到大肠杆菌中。IN-dCas9足以将氯霉素编码模板DNA整合到amilCP报告基因质粒中,从而破坏amilCP表达并赋予对氯霉素的抗性。这种快速测定(其允许对单独整合事件的定量和克隆序列分析)可用于优化编辑。
编辑哺乳动物基因组DNA的效率
使用稳定的CMV驱动的GFP报告基因细胞系确定编辑哺乳动物基因组DNA的效率和保真度,并产生含有RFP和嘌呤霉素选择盒的供体模板。对整合事件进行定量和克隆表征,以确定所述方法作为新颖的基因组编辑技术的功效和保真度。
功能性IN-dCas9融合蛋白的产生。
为了产生用于哺乳动物细胞中的功能性IN-dCas9融合蛋白,将全长逆转录病毒IN从HIV-1(gag-pol多蛋白的氨基酸1148-1435)克隆至人密码子优化的dCas9的N末端,通过15个氨基酸的柔性接头[(GGGGS)3)]分隔(图6)。SV40核定位信号(NLS)包含在IN的N末端,所述SV40核定位信号与dCas9上的C末端SV40 NLS一起提供IN-dCas9融合蛋白的核定位。为了产生缺少C末端非特异性DNA结合结构域的IN-dCas9融合物,产生了仅含有IN的N末端和催化核心结构域(氨基酸1148-1369)的另外构建体,作为与dCas9的N末端融合物(图6)。
用于监测质粒DNA的编辑的报告基因的产生。
为了定量和表征哺乳动物细胞中IN-dCas9介导的整合,设计了基于质粒的报告基因测定,所述测定利用来自多孔鹿角珊瑚的蓝色色蛋白(amilCP),当在大肠杆菌中表达时所述色蛋白产生深蓝色菌落(图6)。对amilCP开放阅读框的破坏消除蓝色蛋白表达,所述蓝色蛋白可用作靶向保真度的直接读数,以及用作整合酶-Cas介导的整合的靶DNA。设计了具有‘PAM输出’取向的通过16bp间隔区序列隔开的单向导RNA(sgRNA)靶序列,以促进靶DNA处的N末端dCas9融合蛋白的有效二聚化(图4)。
用于整合酶-Cas介导的整合的病毒末端供体序列的产生。
为了构建可用于产生用于整合酶-Cas介导的整合的供体序列的靶向载体,将包含U3和U5 HIV末端的30个碱基对亚克隆到pCRII中(图6)。为了促进供体序列的亚克隆,在U3与U5之间包含含有9种独特限制酶的多克隆位点。由于U3和U5在其末端共享相同的3个核苷酸(分别ACT和AGT),因此包含了另外的半位点序列,以在每个末端产生可用于从质粒主链产生平末端供体序列的ScaI限制性位点(图6)。此外,对于FauI包含侧翼IIS型限制性酶位点,所述FauI切割并留下两个5'核苷酸突出端,从而模拟具有暴露的CA二核苷酸的3'预加工的病毒末端(图6)。为了有助于FauI消化的模板从质粒主链的凝胶纯化和分离,使用多位点定向诱变除去pCR II质粒主链中存在的六个FauI位点。
方案:制备用于转染的INsrt供体模板
1)建立INsrt质粒DNA的限制性消化
2)限制性消化反应
3)凝胶纯化来自主链DNA的供体模板
4)洗脱用于转染的供体DNA。
整合酶-Cas介导的供体序列整合到哺乳动物细胞中的质粒DNA中。
为了允许协同IN-dCas9介导的整合的阳性选择,设计了携带氯霉素抗性基因(CAT)的INsrt供体载体,所述基因在表达质粒的报告基因中不存在(图7)。CAT基因前面包含来自斯氏珀蝽(Plautia stali)肠病毒(PSIV)的IGR IRES,所述IGR IRES可在原核细胞和真核细胞中起始翻译,以有助于多个整合位点的翻译。使用ScaI(平末端)或FauI(加工的末端)消化含有氯霉素抗性基因和病毒末端的模板,并从质粒主链DNA凝胶纯化。INsrt模板的共转染,将靶向amilCP序列的IN-dCas9载体共转染到Cos7细胞中(图7)。48小时后,使用柱纯化回收总质粒DNA,并转化到大肠杆菌中。对于全长IN和INDC-dCas9融合蛋白两者均观察到氯霉素抗性克隆。对质粒的测序表明,IG3-CAT质粒序列已整合到amilCP报告基因中。令人感兴趣地,使用FauI消化的供体序列(其模拟病毒DNA末端的预3'加工)产生与ScaI消化的平末端模板相比,两倍的氯霉素抗性克隆。整合酶-Cas介导的整合含有HIV IN慢病毒整合的标志,包括在整合位点侧翼的宿主DNA的5个碱基对重复序列。令人感兴趣地,整合位点没有出现在两个sgRNA靶位点之间,而是出现在amilCP靶序列的任一侧。
将具有平末端(ScaI裂解的)或3'加工模拟物(FauI裂解的)末端的Insrt IGR-CAT供体模板整合到哺乳动物细胞中的pCRII-amilCP报告基因中。令人感兴趣地,作为与dCas9的融合物,C末端非特异性DNA结合结构域的缺失不会抑制整合酶-Cas介导的整合。使用模拟3'加工的末端显示出CAT抗性克隆增加约2倍。(图29B)使用IGR-CAT供体模板的双向导RNA靶向整合到amilCP中,抑制二聚化的突变(E85G和E85F)不会破坏整合酶-Cas介导的整合。然而,IN E87G突变不能通过配对的靶向sgRNA挽救。令人感兴趣地,与dCas9的串联INΔC融合物(tdINΔC-dCas9)显示增强约2倍的整合(图29C)。
方案:整合酶Cas介导的供体序列整合到哺乳动物细胞中的质粒DNA中
1)将多顺反子sgRNA和IN-dCas9质粒、细菌amilCP报告基因质粒和INsrt供体模板共转染到哺乳动物(例如Cos7)细胞中。
a.在涂铺细胞之前立即建立转染反应。
b.收获和涂铺和转染细胞
2)从转染的细胞中回收质粒DNA:
3)将回收的质粒DNA转化到化学感受态大肠杆菌中。
用于整合酶-Cas介导的整合到基因组DNA的CMV-GFP稳定哺乳动物细胞系的产生。
产生了可用于定量和表征哺乳动物细胞中的单独整合事件的保真度的稳定GFP报告基因细胞系(图3)。将在人CMV启动子控制下编码GFP的质粒(pcDNA3.1-GFP)线性化并转染到Cos7细胞中,并使用G418和连续稀释选择稳定的克隆。这种人工基因座允许稳健的基因表达,可靶向所述人工基因座以进行破坏而不损害正常细胞活力,否则当靶向必需宿主基因时可能发生损害正常细胞活力。
整合酶Cas介导的供体序列整合到哺乳动物基因组DNA中。
为了定量CMV-GFP基因座处的整合事件,构建了供体模板,所述供体模板含有IGR-mCherry-2A-嘌呤霉素-pA盒和靶向GFP编码序列的成对向导RNA(图3)。供体盒整合到CMV-GFP基因座中将驱动mCherry表达并破坏GFP表达,并且提供对抗生素嘌呤霉素的抗性。转染和培养48小时后,观察到mCherry阳性细胞,其中一些仍含有微弱但可检测的GFP表达水平(图3)。
在内源性基因座处整合酶-Cas介导的供体序列整合。
设计了靶向策略,并且选择对人EF1-α基因座的3'UTR具有特异性的向导RNA,以将IGR-mCherry-2A-嘌呤霉素-pA盒敲入人HEK293细胞系中(图8)。靶向3’UTR以允许表达IGR-mCherry盒,而不会破坏EF1-α表达的开放阅读框。转染和培养48小时后,在培养物中观察到mCherry阳性细胞(图8)。
方案:整合酶Cas介导的供体序列整合到哺乳动物基因组DNA中。
1)使用Fugene6或Lipofectamine2000将编码sgRNA、IN-dCas9和INsrt供体模板的共转染质粒1:1:1共转染到哺乳动物细胞(COS7,HEK293等)中。
a.收获、涂铺和转染细胞。
2)整合序列的抗生素选择
a.用10ml含抗生素选择的培养基洗涤细胞并涂铺
b.培养细胞,然后产生克隆。
定向编辑。
IN介导的DNA序列整合可在靶DNA序列的任一方向上发生。利用Cas和IN逆转录病毒类别蛋白的不同组合提供促进定向编辑的能力。例如,与催化性死亡LbCpf1(LbCpf1)融合的来自BIV(牛免疫缺陷病毒或其它HIV相关病毒)的IN的融合物允许利用Cpf1特异性向导RNA结合至特定靶序列。利用含有HIV和BIV末端序列两者的供体序列将与靶DNA的结合锁定至单一取向。(图9)。
用于产生Floxed等位基因的多重基因组编辑。
目标基因周围的侧翼LoxP(Floxed)序列的并入允许CRE介导的重组和条件诱变。使用CRISPR-Cas9产生Floxed等位基因的当前方法效率低下。最广泛使用的方法是使用两种向导RNA来诱导侧接靶序列处的DNA裂解,并使用同源定向修复来插入含有LoxP序列的ssDNA模板。然而,当使用双sgRNA来诱导裂解时,最有利的反应是插入序列的缺失,从而导致全基因缺失。如果用宿主DNA进行IN介导的链转移不允许插入序列的有效缺失,那么使用整合酶-Cas介导的基因插入可为DNA序列的串联插入提供替代且更有效的方法。由于IN介导的整合可在任一方向上发生,因此含有反向LoxP序列的序列整合允许侧翼LoxP序列的重组(图10)。
来自酵母Ty1逆转录转座子的整合酶含有非经典二分核定位信号,所述核定位信号由通过较大接头序列分隔的串联KKR基序组成。酵母中的先前研究证明了这些基本基序对于核定位和Ty1转位的必要性(Kenna等人,1998,Mol Cell Biol 18,1115-1124;Moore等人,1998,Mol Cell Biol 18,1105-1114)。Ty1转位绝对取决于Ty1 NLS的存在,并且令人感兴趣地,经典NLS不足以概括转位所需的Ty1 NLS活性。令人感兴趣地,另外的酵母蛋白共享这一串联KKR基序,鉴于这些蛋白中的许多是核定位的,所述基序可起到NLS的作用(Kenna等人,1998,Mol Cell Biol 18,1115-1124)。
如实施例1所示,酵母Ty1 NLS在哺乳动物细胞中提供Cas蛋白和Cas-融合蛋白的稳健核定位。为了确定这种活性是否是Ty1 NLS的独特特征,测试了来自Ty2整合酶的密切相关的NLS和其它酵母Ty1 NLS样基序是否足以将整合酶-dCas9融合蛋白(INΔC-Cas9)定位于哺乳动物细胞中的细胞核。令人感兴趣地,与Ty1 NLS高度保守的Ty2 NLS在核定位方面与Ty1 NLS同等有效(图11)。在酵母中鉴定的与Ty1/Ty2 NLS序列不同的三种不同Ty1NLS样序列的融合物(Kenna等人,1998)显示出稳健的NLS活性(MAK11)或无明显NLS活性(INO4和STH1)。MAK11序列源自酵母核蛋白,其也出现在进一步筛选的蛋白的C末端,从而表明此序列确实起着NLS的作用。使用基序KKR
CRISPR-Cas DNA裂解系统源自细菌,并且Cas蛋白既大又缺乏内在哺乳动物核定位信号(NLS),从而阻止了它们在哺乳动物细胞中的有效核定位。先前已经表明需要添加两种经典核定位信号(N末端SV40和C末端核质蛋白(NPM)二分NLS)来在哺乳动物细胞中通过CRISPR-Cas9进行DNA的有效核定位和编辑(Cong等人,2013,Science 339,819-823)。由于非经典酵母逆转录转座子Ty1 NLS用于在哺乳动物细胞中定位Cas融合蛋白的稳健性(实施例1),因此测试了Ty1 NLS是否还能够增强哺乳动物细胞中传统CRISPR-Cas9的编辑效率。
为了确定Ty1是否增强CRISPR-Cas9编辑,通过用非经典Ty1NLS(px330-Ty1)代替C末端NPM NLS来对现有CRISPR-Cas9表达质粒(px330)进行了修饰(图12A)。接下来,产生了移码响应性荧光素酶报告基因,其编码靶序列(ts)下游的框外荧光素酶编码序列(图12B)。对于这种报告基因测定,靶序列附近的裂解和通过细胞非同源末端连接(NHEJ)途径进行的不完全修复可诱导核苷酸插入或缺失,所述核苷酸插入或缺失可能重构荧光素酶编码序列并产生荧光素酶表达。
荧光素酶报告基因与编码Cas9的载体的共表达相对于非靶向向导RNA产生荧光素酶活性相对于背景的约20倍增加,所述Cas9含有NPM NLS和对20个核苷酸的靶序列具有特异性的单向导RNA(图12C)。值得注意的是,与含有NPM NLS的Cas9相比,含有Ty1 NLS的Cas9的表达在COS-7细胞中产生报告基因活性的显著增强(约44%)(图12C)。
可根据所需的结果使得使用整合酶-DNA结合融合蛋白的DNA供体序列的靶向整合靶向基因组内的不同位置。例如,可使由基因表达盒(例如启动子、基因序列和转录终止子)组成的治疗性DNA供体序列靶向‘安全港’位置(有关人基因组中的安全港位点的综述和列表,参见Pellenz等人,2019,Hum Gene Ther 30,814-828),这将允许治疗性基因的表达而不影响邻近基因表达。这些可包括远离邻近基因例如H11的基因间区域,或‘非必需’基因例如CCR5、hROSA26或AAVS1内的基因间区域(图13A和13b)。
为了恢复引起疾病的基因突变的表达,将治疗性基因序列靶向整合到内源性疾病基因基因座中可能是有利的,因为此基因座已经是缺陷型的并且此基因座的时间和空间表达处于内源性调控控制之下。在一次迭代中,可将编码含有剪接受体的治疗性基因的DNA供体序列整合到内源性基因基因座的第一内含子中,以使得剪接将1)允许所引入基因序列的表达,以及2)阻止突变的序列的下游表达(由于来自整合的poly(A)序列或LTR序列的终止)(图13C)。如果靶向下游内含子,则可递送或表达较小的DNA供体序列(图13D)。
将含有IRES序列的DNA供体序列靶向插入基因的3'非翻译区(3'UTR)中可能是有益的,因为这种方法将允许以与靶向基因座相同的时间和空间表达进行表达,并且将不太可能破坏靶向基因基因座(图13E)。
本文提供的数据证明将慢病毒供体序列递送和靶向整合到哺乳动物基因组中的三种不同方法。
慢病毒生命周期
慢病毒是单链RNA病毒,其将其前病毒基因组的永久双链DNA(dsDNA)拷贝整合到宿主细胞DNA中(图14)。慢病毒基因组侧接长末端重复序列(LTR),所述长末端重复序列控制病毒基因转录,并且在其U3和U5末端含有前病毒基因组整合所需的短(约20个碱基对)序列基序。在病毒感染后,慢病毒RNA基因组通过病毒编码的逆转录酶(RT)复制为平末端dsDNA,并通过整合酶(IN)插入宿主基因组中。IN由三个对IN活性至关重要的功能结构域组成,包括非特异性地结合至DNA的C末端结构域(CTD)。IN介导的逆转录病毒DNA插入在几乎没有DNA靶序列特异性的情况下发生,并且可整合到活性基因基因座中,其可破坏正常基因功能并有可能导致人类疾病。尽管具有大的序列携带能力的益处,但是这限制慢病毒载体用于基因疗法的用途。
基因组编辑
CRISPR-Cas9通过利用短的单向导RNA来识别并结合DNA而允许可编程的DNA靶向。催化失活的Cas9(dCas9)保留靶向DNA的能力,并且最近已被重新用作可编程的DNA结合平台,用于基因组询问和调控的各种应用。如实施例1所示,慢病毒整合酶与dCas9的融合物足以在哺乳动物细胞中使用CRISPR向导RNA将含有短病毒末端的供体DNA序列插入靶序列(图15)。为了监测整合酶-Cas介导的在哺乳动物细胞中的整合,产生了含有IGR IRES序列、随后是mCherry-2a-嘌呤霉素基因和SV40聚腺苷酸化序列的供体载体(图15B)。设计了靶向COS-7细胞中的稳定人CMV-eGFP稳定细胞系的sgRNA(图15C和15D)。hCMV-eGFP稳定转基因提供了异源靶序列,所述异源靶序列可用于确定在稳健表达、但非必需表达基因座处的编辑。纯化供体mCherry-2a-puro模板,并与sgRNA和IN-dCas9一起共转染到GFP稳定细胞中,并培养48小时。48小时后,mCherry阳性细胞在培养物中可见,并替代GFP阳性信号(图15E)。将编辑组分(整合酶-CRISPR-Cas9融合物)并入慢病毒颗粒中允许靶向且可容易编程的慢病毒基因组整合到宿主DNA中,从而消除慢病毒基因疗法的主要局限性(即非特异性慢病毒整合)。这种方法对于基础研究和治疗应用两者均是有用的。
慢病毒基因递送系统
慢病毒载体已被用作研究应用的稳健基因递送工具(图16)。慢病毒的结构蛋白和酶蛋白被转录并翻译为大的多蛋白(gag-pol和包膜)(图16A)。一旦并入出芽病毒颗粒中,多蛋白就被病毒蛋白酶加工成单独蛋白质。对于慢病毒载体基因表达系统,将这些多蛋白从病毒基因组中除去,并使用单独的哺乳动物表达质粒表达(图16B)。然后可克隆供体DNA序列以代替侧翼LTR序列之间的病毒多蛋白。这些载体在哺乳动物细胞中的共转染允许形成能够递送和整合所编码的供体序列的慢病毒颗粒,然而不需要整合酶和随后病毒繁殖所必需的其它病毒蛋白的编码信息(图16B)。慢病毒颗粒是用于递送病毒蛋白(例如整合酶和逆转录酶)和dsDNA供体序列两者的天然载体,其含有整合酶介导的插入哺乳动物细胞中所需的必要病毒末端序列(图16C)。
将整合酶-dCas9融合蛋白包装到慢病毒颗粒中。
为了用于哺乳动物细胞中的基因组编辑的靶向慢病毒供体模板整合的目的,可修改现有慢病毒递送系统以并入编辑组分(图17-20)。本文描述了将慢病毒供体序列递送和靶向整合到哺乳动物基因组中的三种不同方法。
第一种方法是直接并入dCas9作为与编码gag-pol多蛋白的慢病毒包装质粒(例如psPax2)内的整合酶(或缺乏其C末端非特异性DNA结合结构域的整合酶,INΔC)的融合物(图17A)。在这种方法中,将修饰的gag-pol多蛋白与其它病毒组分一起翻译为多蛋白,负载向导RNA并包装到慢病毒颗粒中(图4B)。整合酶-dCas9融合蛋白保留蛋白酶裂解(PR)所需的序列,并且因此在颗粒成熟过程中通常从gag-pol多蛋白裂解。哺乳动物细胞的转导产生病毒蛋白,包括IN-dCas9融合蛋白、sgRNA和慢病毒供体序列的递送。ssRNA基因组通过逆转录酶的逆转录产生含有正确病毒末端序列(U3和U5)的dsDNA序列,所述dsDNA序列然后通过IN-dCas9融合蛋白被整合到哺乳动物基因组中。
第二种方法是产生整合酶-dCas9与HIV病毒蛋白R(VPR)的N末端和C末端融合物(图18A)。VPR作为辅助蛋白被有效地包装到慢病毒颗粒中,并且已被用于将异源蛋白(例如GFP)包装到慢病毒颗粒中。在VPR与IN-dCas9融合蛋白之间包含病毒蛋白酶裂解序列,因此成熟后,IN-dCas9从VPR释放(图18A)。包装细胞与慢病毒组分的共转染产生含有VPR-IN-dCas9蛋白和sgRNA的病毒颗粒。病毒颗粒形成所需的包装质粒(例如psPax2)在整合酶中含有突变,以抑制其催化活性,从而防止非介导的整合(图18B)。在病毒转导后,整合酶-dCas9蛋白被递送并介导慢病毒供体序列的整合(图18C)。递送IN-dCas9融合物和作为核糖蛋白的sgRNA的益处是它仅在靶细胞中瞬时表达。
第三种方法是将整合酶-dCas9融合蛋白和sgRNA表达盒直接并入慢病毒转移质粒或其它病毒载体(如AAV)中(图19A)。将含有IN-dCas9融合蛋白和sgRNA的转移质粒与产生慢病毒颗粒所需的包装质粒和包膜质粒共转染。如果使用慢病毒,则包装质粒在整合酶中含有催化突变,以抑制非特异性整合(图19B)。在转导哺乳动物细胞后,IN-dCas9融合蛋白和sgRNA的表达产生能够靶向其自身的病毒供体载体以进行靶向整合(自我整合)的组分(图19C)。这种方法用于靶向基因破坏或用作基因驱动。或者,与编码供体序列的另外慢病毒颗粒共转导充当整合的供体模板(图19)。通过使用来自不同逆转录病毒家族成员的整合酶及其相应的转移质粒,实现以这种方法阻止其自身病毒编码序列的自我整合。例如,利用编码FIV IN-dCas9融合蛋白的HIV慢病毒颗粒来整合在FIV慢病毒颗粒中编码的FIV供体模板(图20)。
产生单基因座、组成型活性的遍在ROSA26
ROSA26 mT/mG报告基因小鼠品系(Jackson Labs,Stock#007576)含有floxed膜定位的tdTO(mT)荧光报告基因盒,其在与CRE重组酶重组时可除去mT报告基因并允许表达膜定位的eGFP(mG)报告基因。为了产生单基因座体内GFP报告基因系,将ROSA26 mT/mG小鼠与通用CAG-CRE重组酶小鼠杂交,以产生组成型且遍在表达的ROSA26 mG报告基因小鼠。从杂合ROSA26
将组分包装到慢病毒颗粒中,以靶向整合到ROSA-mGFP基因座中。
为了将IRES-tdTO序列靶向整合到ROSA26
哺乳动物细胞中的靶向慢病毒整合
使用Incriptr修饰的慢病毒颗粒来转导培养物中的ROSA26
用于靶向整合慢病毒颗粒的替代DNA结合结构域。
此数据证明用dCas9的可编程DNA结合结构域代替整合酶的非特异性DNA结合结构域允许dsDNA供体模板的靶向整合,或经由慢病毒颗粒中的递送,以用于递送慢病毒编码的供体序列。CRISPR-Cas系统是双组分的,依赖于Cas蛋白和小向导RNA两者进行靶向。在一些情况下,利用单组分DNA靶向蛋白(如TALEN)用于经由慢病毒颗粒递送可能是有益的,因为这些蛋白仅被所编码的蛋白靶向。使用类似的慢病毒产生方法,用靶向给定序列(例如,eGFP或安全港基因座)的TALEN代替先前包装策略中的dCas9允许慢病毒包装和靶向,而无需递送向导RNA(图23)。例如,TALEN在gag-pol多蛋白的背景下作为与整合酶的融合物包装并递送(图23A),IN-TALEN作为与病毒并入蛋白如VPR的融合物包装并递送(图23B),或者IN-TALEN在转移质粒内递送(图23C)。
CRISPR-Cas DNA裂解系统源自细菌,并且Cas蛋白既大又缺乏内在哺乳动物核定位信号(NLS),从而阻止了它们在哺乳动物细胞中的有效核定位。
为了确定Ty1是否增强CRISPR-Cas9编辑,通过用非经典Ty1NLS(px330-Ty1)代替C末端NPM NLS来对现有CRISPR-Cas9表达质粒(px330)进行了修饰(图24A)。接下来,产生了移码响应性荧光素酶报告基因,其编码靶序列(ts)下游的框外荧光素酶编码序列(图24B)。对于这种报告基因测定,靶序列附近的裂解和通过细胞非同源末端连接(NHEJ)途径进行的不完全修复可诱导核苷酸插入或缺失,所述核苷酸插入或缺失可能重构荧光素酶编码序列并产生荧光素酶表达。
荧光素酶报告基因与编码Cas9的载体的共表达相对于非靶向向导RNA产生荧光素酶活性相对于背景的约20倍增加,所述Cas9含有NPM NLS和对20个核苷酸的靶序列具有特异性的单向导RNA(图24C)。值得注意的是,与含有NPM NLS的Cas9相比,含有Ty1 NLS的Cas9的表达在COS-7细胞中产生报告基因活性的显著增强(约44%)(图24C)。
转录激活因子样效应物核酸酶(TALEN)是经过充分研究的可编程DNA结合蛋白,其由单独核苷酸靶向结构域的串联组装构建(Reyon等人,2012)。在针对Inscriptr展示的类似方法中,TALEN可用于指导逆转录病毒整合酶介导的供体DNA模板整合(图25)。为了产生TALEN-整合酶融合蛋白,构建哺乳动物表达载体以接受来自先前描述的TALEN表达载体的TALEN靶向重复序列,以产生IN-TALEN或TALEN-IN融合物。每种融合蛋白均并入3xFLAG表位、Ty1 NLS和TALEN重复序列,在缺少C末端非特异性DNA结合结构域的HIV整合酶(INΔC)之间通过接头序列隔开。在一些情况下,可并入IN突变以改变IN活性、二聚化、与细胞蛋白的相互作用、对二聚化抑制剂的抗性或INΔC的串联拷贝(tdINΔC)。例如,可并入E85G突变以抑制专性二聚体形成。
先前已经描述了靶向eGFP的TALEN对,并验证了靶向效率(Reyon等人,2012;可从Addgene获得)。将TALEN对(ClaI/BamHI片段)亚克隆以产生针对eGFP的具有长度为16bp或28bp的间隔区的TALEN-IN融合蛋白。
使用质粒DNA整合测定(图26),靶向eGFP的TALEN-IN对的共转染,将编码IGR-CAT抗性基因的线性双链DNA供体序列和amilCP细菌表达报告基因共转染到哺乳动物COS-7细胞中。转染后两天,从哺乳动物细胞中回收编辑的质粒,并转化到大肠杆菌中,并且在氯霉素板上选择。令人感兴趣地,相隔16bp的TALEN对产生多约6倍的氯霉素抗性菌落,而相隔28bp的TALEN对与非靶向整合酶相似(图27)。这些数据表明TALEN对的接近性对于靶向和整合是重要的,这是先前已经针对TALEN-FokI介导的dsDNA裂解报告的一种特征。
本文中引用的每一和各个专利、专利申请和公布的公开内容特此以引用的方式整体并入本文。
虽然已参考具体实施方案公开了本发明,但是显而易见的是,本发明的其它实施方案和变化可在不脱离本发明的真实精神和范围的情况下由本领域其它技术人员设计。所附权利要求书意图被解释为包括所有此类实施方案和等效变化。
- 使用逆转录病毒整合酶-Cas9融合蛋白通过定向非同源DNA插入进行的基因组编辑
- 一种融合蛋白及使用其进行同源重组的方法
