一种基于图注意力机制的circRNA与疾病关联关系预测方法
文献发布时间:2023-06-19 13:29:16
技术领域
本发明属于生物信息领域,涉及一种基于图注意力机制的circRNA与疾病关联关系预测方法。
背景技术
环状核糖核酸(Circular RNAs,以下简称为circRNA)是一种新的内源性非编码RNA,缺乏5’帽和3’聚腺苷化尾。自从20世纪70年代首次被发现以来,它们一直被认为是剪接错误。在过去的十年中,随着高通量测序技术的发展,在哺乳动物细胞中发现了大量的circRNA。研究人员发现,circRNA在人体组织中广泛表达,具有稳定的结构和组织特异性。目前,circRNA表达的机制仍然不清楚。研究表明,许多circRNA通过充当微小核糖核酸(miRNA)或核糖核酸结合蛋白的海绵、调节蛋白功能来实现其生物学功能。
许多circRNA参与了人类疾病,特别是癌症。例如,circHIPK3在结直肠癌(CRC) 组织中被发现显著上调,这是通过海绵化miR-7来抑制miR-7的活性。 Hsa_circ_0000190在胃癌组织和胃癌患者血浆中表达下调,可作为胃癌诊断的一种新型生物标志物。CircANRIL通过与pescadillo homolog 1(PES1)结合而与动脉粥样硬化疾病相关,进而损害pre-rRAN的加工和核糖体的生物发生,从而诱导细胞凋亡并抑制增殖。此外,研究人员还建立了相关数据库,如circRNADisease、 CircR2Disease、Circ2Disease和circAtlas。
实验验证方法识别疾病相关circRNA既昂贵又耗时。近年来,研究人员逐渐引入计算方法推断circRNA与疾病的潜在关联。Lei等人首先提出了一种路径加权方法来预测与疾病相关的circRNA。他们计算了疾病语义相似度、疾病功能相似度和高斯相互作用普相似。然后,构建了一个异构网络,并采用深度优先搜索 (DFS)遍历网络中的节点,计算预测得分。Yan等人开发了基于Kronecker积核正则化最小二乘的DWNN-RLS方法预测circRNA与疾病相关性,Xiao等人开发了一种具有双流形规则的加权低秩近似优化方法来推断潜在的circRNA-疾病关联。深度学习算法也被引入该领域。Deepthi等人提出了一种集成方法AE-RF,通过深度自编码器提取特征,利用随机森林进行预测。Li等人使用DeepWalk提取circRNA-疾病网络中的节点特征,并使用网络一致性投影算法预测。Wang等人使用FastGCN设计了GCNCDA来提取circRNA和疾病特征,使用Forest PA 分类器进行预测。
上述计算方法取得了不错的预测性能,但还存在一些问题和缺陷,还有进一步提升的空间。上述文献在提取circRNA和疾病特征时,通常直接使用相似性矩阵作为特征,或者使用相似性矩阵低维表示作为特征,导致circRNA和疾病特征的表示不准确。此外,特征的表示直接决定了模型的预测性能。
发明内容
为了解决现有技术的问题,本发明提供了一种基于图注意力机制的circRNA 与疾病关联关系预测方法,解决现有技术中circRNA和疾病特征的表示不准确进而影响模型的预测性能的问题。
本发明的技术方案如下:
一种基于图注意力机制的circRNA与疾病关联关系预测方法,包括步骤如下:
1)构建已知circRNA-疾病关联网络
从已知的circRNA-疾病关系,定义邻接矩阵
2)计算疾病语义相似性
根据Disease Ontology(DO)数据库中的疾病本体之间定义的关系,每个疾病构建一个有向无环图,根据代表两个疾病的有向无环图之间的重合度,计算疾病之间的相似性,得到疾病语义相似性;
3)计算circRNA和疾病高斯核相似性,circRNA功能相似性
根据已知circRNA-疾病邻接矩阵Y,分别计算出circRNA和疾病高斯核相似性;根据疾病语义相似性核circRNA-疾病邻接矩阵Y,计算circRNA功能相似性;
4)构建融合的circRNA和疾病相似性
针对circRNA功能相似性矩阵和疾病语义相似性矩阵稀疏性问题,结合疾病语义相似性矩阵、circRNA功能相似性矩阵,以及相应的高斯核相似性矩阵;若疾病语义相似性矩阵中元素非0,则保持不变,否则替换为疾病高斯核相似性矩阵中对应位置的值;类似地,若circRNA功能相似性中元素非0,则保持不变,否则替换为circRNA高斯核相似性矩阵中对应的值;分别获得融合circRNA和疾病相似性矩阵SC和SD;
5)基于图注意力机制的circRNA和疾病特征提取
针对circRNA和疾病特征缺失的问题,首先定义两个投影参数矩阵MC和MD,使得SC和SD映射到同一空间,矩阵中的一行表示每个circRNA和疾病的初始特征;然后,基于图注意力机制,在已知circRNA-疾病网络Y中进行进一步提取 circRNA和疾病的特征表示;
6)构建多层神经网络的模型进行circRNA-疾病关系的预测
根据已知circRNA-疾病关系,随机选取同样数量的未知关联作为负样本,构建多层神经网络的模型,计算所有circRNA和某疾病的关联概率,分值越大,则表示潜在的未知关联关系可能性越大。
与现有技术相比,本发明的有益效果是:
本发明融合circRNA和疾病相似性矩阵,以及已知circRNA-疾病关联关系,基于图注意力机制提取circRNA和疾病的低维表示,基于多层神经网络的模型准确预测未知circRNA-疾病之间的关联关系。本发明能够基于已知circRNA-疾病的关联,推断潜在的、未知的circRNA-疾病关联关系。本发明通过计算方法可以高效、可靠地预测与疾病相关的circRNA,本发明预测结果可以为进一步生物实验验证提供了可靠的依据,节省实验时间和人力财力花销。
附图说明
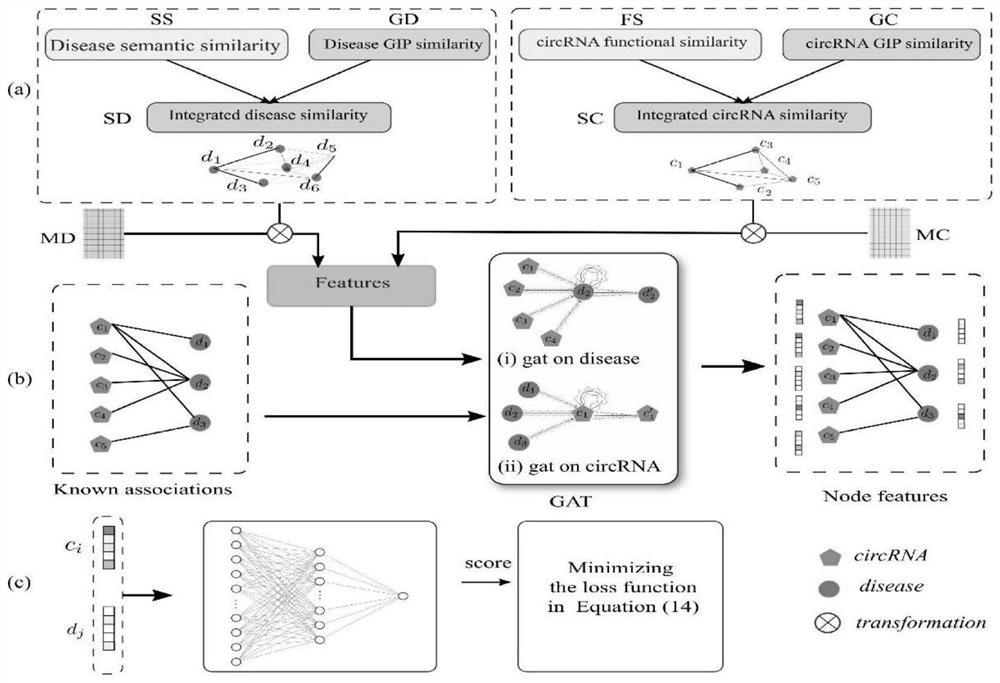
图1是本发明实施的流程图;(a)为circRNA和疾病相似性融合,(b)为基于图注意力机制的特征提取,(c)为基于多层神经网络的circRNA-疾病关联预测;
图2是本发明在五折交叉验证下AUC和PR指标示意图;(a)为基于五折交叉验证的AUC性能,(b)为基于五折交叉验证的AUPR性能;
图3是本发明在不同结构选择下获得AUC指标示意图;
图4是本发明在不同参数选择下获得AUC指标示意图。(a)为不同特征维度下对性能的影响,(b)为不同图注意力机制头数下对性能的影响,(c)为正则化因子的不同取值对性能的影响;
具体实施方式
下面通过结合本发明实施例及附图,说明本发明提出的基于图注意力机制的circRNA与疾病关联关系预测方法的实施过程。
1:构建已知circRNA-疾病关联,计算相似性。
从CircR2Disease数据库下载已知circRNA-疾病关联,表示为
从Disease Ontology(DO)数据库下载疾病本体数据,每个疾病构建一个有向无环图,疾病d表示为DAG
D
D
其中,Δ表示衰减因子。其次,定义疾病d的语义值如下:
然后,计算两个疾病d
其中,
与两个circRNA相关的疾病越相似,它们的功能就越相似。本发明用FS(c
其中,
根据已知circRNA-疾病邻接矩阵Y,分别计算circRNA和疾病高斯核相似性:
GC(c
GD(d
其中,GC和GD分别表示circRNA和疾病高斯核相似性矩阵。Y
其中,N
其中,D
表示疾病d和组D之间的相似性,|D
2:进一步融合疾病和circRNA的相似性,如图1(a)所示。
由于疾病语义相似性矩阵SS和circRNA功能相似性矩阵FS的稀疏性,本发明融合高斯核相似性,构建疾病和circRNA相似性矩阵,分别记为SC和SD。
其中,SC和SD为融合后的circRNA和疾病相似性矩阵。由于SC和SD的维度不一致,为了获得circRNA和疾病特征表示,引入参数矩阵
X=concat(SC×MC,SD×MD)
其中,
3:提出基于图注意力机制的circRNA与疾病特征提取方法,如图1(b)所示。
根据已知circRNA-疾病关联矩阵Y构建异构注意力图模型,进一步进行特征提取。对于节点v
其中,K表示注意力机制的头数,σ是非线性激活函数,
其中,softmax表示归一化所有邻居节点对该节点的贡献系数,每个邻居节点j 对该节点i的贡献,计算公式如下:
其中,a为单层神经网络,W
4:提出基于多层神经网络的circRNA与疾病关联关系预测方法,如图1(c) 所示。
首先构造样本,本发明随机选取同样数量的未知关联作为负样本,全部已知circRNA-疾病关联作为正样本,设计多层神经网络预测模型,计算所有circRNA 和某疾病的关联概率,第k层的输出表示为:
h
其中,σ1是非线性激活函数LeakyReLU,W
f(c,d)=h
其中,σ2表示激活函数sigmoid(·),某circRNA和特定疾病之间的关联得分 f(c,d)∈(0,1),作为判断潜在的未知关联关系大小的依据。
最终,预测模型的损失函数定义为:
其中,其中N为训练样本总数。λ表示正则化的控制因子,Θ是模型的参数。
5:模型结构和参数对预测性能的影响,如图3和图4所示。
本发明定量评估了不同结构和参数对预测性能的影响,使用基于 CircR2Disease数据集的5折交叉验证进行性能评价。首先,本发明选择是否使用相似性集成、图注意力机制和多层神经网络的分类器,将模型的变种定义为:
变种1(不使用相似性特征):使用随机初始化的SD和SC作为疾病和circRNA 的特征。
变种2(不使用图注意力机制):从模型中去除图注意力机制,使用集成的相似性作为特征,并使用一个两层神经网络作为预测器。
变种3(不使用多层神经网络分类器):使用点积计算得分,而不是用多层神经网络作为预测器。
结果如图3所示。本发明使用随机初始化特征获得的AUC和AUPR值最低,表明将集成相似性作为初始节点特征可以大大提高预测性能。本发明不带图注意力机制和不带多层神经网络的性能下降约10%。因此,本发明提出的方法结合了这些组件的优点,以获得最佳性能。
同时,本发明评估几个重要的超参数对预测性能的影响,如图4所示。首先,选择了不同的特征维度大小{8,16,32,64,128,256},测试其对性能的影响。如图4(a) 所示,当维度设置为8时,本发明的AUC和AUPR最低,在32时性能最好。当维度超过32时,性能略有下降。结果表明,过小的维度可能导致疾病和circRNA 的表达能力降低,而过大的维度可能导致高噪声。其次,本发明对图注意力机制的不同头数进行了实验。由图4(b)可知,本发明在4个图注意力机制头时的AUC 最好,在1个图注意力机制头时的AUPR最好。考虑到大多数方法使用AUC作为性能比较的标准,我们最终选择4作为图注意力机制的默认头数。此外,还计算正则化因子λ的影响。如图4(c)所示,本发明在λ=1e-2处获得最佳AUC和 AUPR。
6:验证本发明的预测性能,如图2,表1和表2所示。
表1.基于CircR2Disease数据库的五折交叉验证结果.
表2.基于CircR2Disease数据库五折交叉验证下AUC值比较.
本发明使用5折交叉验证评估预测性能。首先,所有样本被随机分成5份,每份轮流作为测试集,其他4份作为训练集。结果如图2所示,在CircR2Disease 数据集上,平均AUC和AUPR分别为0.9740,0.9673。如表1所示,本发明的平均准确率为0.9315,精度为0.9714,召回率为0.9615,F1值为0.9336。
此外,基于5折交叉验证的评价指标,进行了50次实验,选择本发明的最佳性能和平均性能,与其他9种方法进行比较。结果见表2所示,可以看出,本发明优于其他9种方法。值得注意的是,后两种方法都是基于图神经网络的。本发明的AUC值大大优于这些方法,这表明本发明可以有效、准确地预测潜在的疾病相关circRNA。
7:典型疾病案例分析,如表3和表4所示。
以乳腺癌和肝细胞癌为例,进一步验证本发明的预测性能。案例分析在CircR2Disease数据集上训练,然后在circRNADisease和circAtlas v2.0数据集上验证预测的circRNA。第一个案例研究是乳腺癌,这是女性最常见的癌症之一。特别地,实验使用CircR2Disease中所有已知关联作为正样本。同时,从未知的关联中随机抽取相同数量的负样本。基于这些训练样本训练模型,然后计算了乳腺癌与每个circRNA之间的得分。最后,我们选择排名前20的相关circRNA进行分析。如表3所示,前20个预测中有18个得到了验证。其他2个预测circRNA 已在最近发表的文献中得到验证。
第二个疾病案例是关于肝细胞癌。它是最常见的一种肝癌,在长期肝病患者中发病率较高。我们利用本发明计算其与circRNA的相关得分,然后按降序排序。前20个与肝细胞癌相关的circRNA列在表4中。可以看到,前20个候选circRNA 中有10个在验证数据集中得到了验证,其他8个候选circRNA在最近的文献中也得到了验证。
表3.与乳腺癌相关的前20个circRNA.
I,II表示circRNADisease,circAtlas v2.0数据库.
表4.与肝细胞癌相关的前20个circRNA.
I,II表示circRNADisease,circAtlas v2.0数据库。
- 一种基于图注意力机制的circRNA与疾病关联关系预测方法
- 一种circRNA和疾病关联关系的高效预测方法
