基于自适应基因交互正则化弹性网络模型的基因选择方法及系统
文献发布时间:2023-06-19 13:49:36
技术领域
本发明属于生物信息技术领域,尤其涉及一种基于自适应基因交互正则化弹性网络模型的基因选择方法及系统。
背景技术
肿瘤现在已经成为威胁人类生命健康的主要疾病之一,根据2018全球癌症统计数据报告显示,2018年全球新增癌症病例将达到1810万人,死亡人数将达到960万人,并且确诊癌症的人数每年还在快速上涨,然而对于癌症的治疗手段和预防手段等方面的研究还不是很全面。随着大量基因芯片技术的应用和发展完善,人类已经能够陆续获得各类组织正常的基因表达信息,而从基因芯片上所测量得到的海量基因中找出不同疾病类别上具有差异性表达的少量基因,是进行准确的疾病判断和给出可靠诊断依据的关键所在。同时这也将为进一步抗病药物的研制提供便利。
基于机器学习和统计分析的学习方法从大量基因表达数据中筛选出的特征基因有助于为肿瘤诊断、癌症分类、临床结果预测等提供重要的参考,因此引起了大量学者的广泛关注。在生物医学中,微阵列数据广泛应用在癌症分类和预测的研究中。通常使用矩阵的形式来表示DNA微阵列数据,矩阵内元素的数值表示基因表达水平信息。具体形式如表1所示,其中行表示样本,列表示一种基因,最右边的一列表示该样本的类别标签。
表1.DNA微阵列数据的矩阵形式
基因选择是对基因表达谱进行研究和分析的重要步骤,基因选择需要从高维的基因表达谱中选择出少量含有关键基因信息子集,在进行基因选择的过程中不改变原来样本中的基因值,而是通过去除冗余基因并保留与分类相关的基因。
基因选择的过程如下:(1)获取微阵列数据,(2)对所获取的数据进行预处理,(3)提取特征基因,(4)进行分类建模,(5)对分类的结果进行分析。具体分类过程如图1所示。
目前,有许多研究利用微阵列数据并根据基因的表达水平对疾病进行分类。大量研究表明,基因的表达水平是发现特征基因和分类的重要工具。逻辑回归是一种常用的特征分类方法,但是对于微阵列数据(即预测变量的数量p远远大于样本数n),它可能产生不稳定的估计结果。此外,最大似然法在预测变量之间存在的多重共线性时也会产生不稳定的结果。因此,现有的逻辑回归方法不适用于基于基因表达水平的疾病分类。基于L
随着大规模的癌症基因组研究和个性化医学的兴起,利用多组学数据进行临床结果综合预测成为了新兴研究课题。由于在许多癌症中,DNA甲基化和基因表达在预测癌症分期和患者生存方面的相对优势不太明显,通过将基因表达谱数据、甲基化谱数据和其他基因测量方法结合起来预测临床结果,可以提高预测性能。然而,这需要从大量的患者中收集和整合基因组数据,任务量比较大。
发明内容
本发明针对现有基因选择方法存在的估计效率低下、未充分考虑基因相互作用、任务量比较大的问题,提出一种基于自适应基因交互正则化弹性网络模型的基因选择方法及系统,克服了上述缺陷,可以自适应地选择与肿瘤的产生高度相关的重要基因,并去除冗余、不相关的基因和噪声基因。
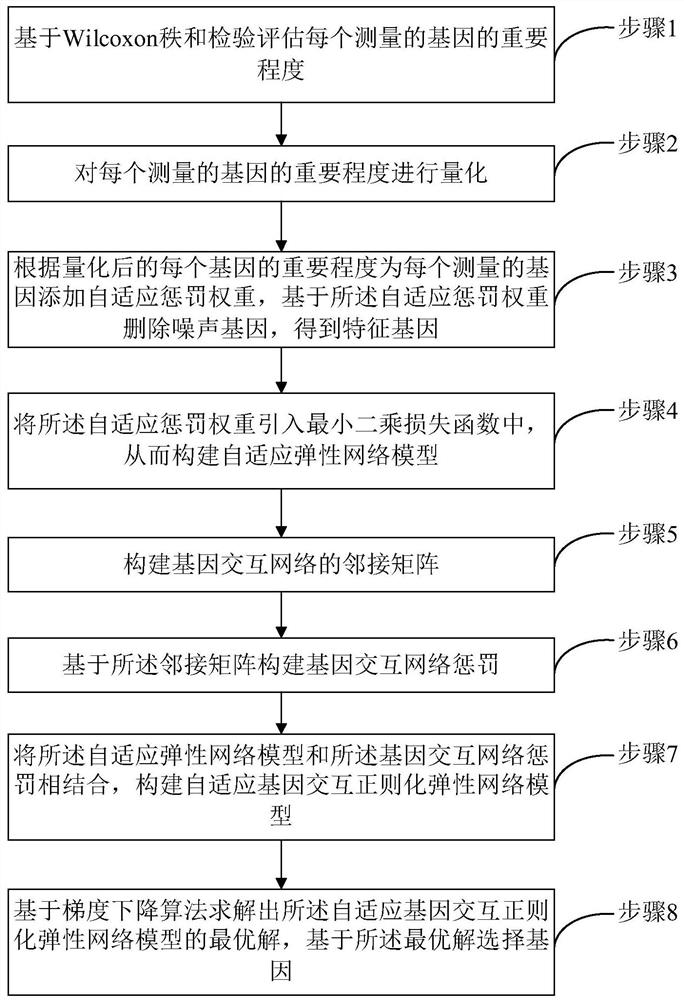
为了实现上述目的,本发明的一种基于自适应基因交互正则化弹性网络模型的基因选择方法,基于Wilcoxon秩和检验(Wilcoxon Rank Sum Test,WRST)评估每个基因的重要性,然后根据测量的基因的重要性程度对每个基因施加自适应型惩罚,从而将噪声基因从模型中删除,识别出特征基因;将基因测量和基因与基因之间的交互信息整合到自适应弹性网络模型中,增强结构的稀疏性,并利用分组效应来选取特征基因降低冗余;利用迭代梯度下降算法求解正则化弹性网络模型。该方法具体包括如下步骤:
步骤1:基于Wilcoxon秩和检验评估每个测量的基因的重要程度;
步骤2:对每个测量的基因的重要程度进行量化;
步骤3:根据量化后的每个基因的重要程度为每个测量的基因添加自适应惩罚权重,基于所述自适应惩罚权重删除噪声基因,得到特征基因;
步骤4:将所述自适应惩罚权重引入最小二乘损失函数中,从而构建自适应弹性网络模型;
步骤5:构建基因交互网络的邻接矩阵;
步骤6:基于所述邻接矩阵构建基因交互网络惩罚;
步骤7:将所述自适应弹性网络模型和所述基因交互网络惩罚相结合,构建自适应基因交互正则化弹性网络模型;
步骤8:基于梯度下降算法求解出所述自适应基因交互正则化弹性网络模型的最优解,基于所述最优解选择基因。
进一步地,所述步骤1包括:
基于Wilcoxon秩和检验,按照下式评估每个测量的基因的重要程度:
其中I(.)为指示函数;
进一步地,所述步骤2包括:
按照下式将基因进行排序:
R(g
当s(g
进一步地,所述步骤3中,自适应惩罚权重的表达式为:
其中n为样本个数。
进一步地,所述自适应弹性网络模型的表达式为:
其中O
进一步地,所述步骤5中,按照下式构建基因相互作用网络的邻接矩阵:
A=[a
其中R代表实数集;A表示基因相互作用网络的邻接矩阵;a
进一步地,所述步骤6中,按照下式构建基因交互网络惩罚:
其中O
进一步地,所述自适应基因交互正则化弹性网络模型的表达式为:
其中F(X,A,β)表示自适应基因交互正则化弹性网络模型,X为输入矩阵,
本发明另一方面还提出一种基于自适应基因交互正则化弹性网络模型的基因选择系统,包括:
基因重要程度评估模块,用于基于Wilcoxon秩和检验评估每个测量的基因的重要程度;
基因重要程度量化模块,用于对每个测量的基因的重要程度进行量化;
加权模块,用于根据量化后的每个基因的重要程度为每个测量的基因添加自适应惩罚权重,基于所述自适应惩罚权重删除噪声基因,得到特征基因;
第一构建模块,用于将所述基因权重引入最小二乘损失函数中,从而构建自适应弹性网络模型;
第二构建模块,用于构建基因交互网络的邻接矩阵;
第三构建模块,用于基于所述邻接矩阵构建基因交互网络惩罚;
第四构建模块,用于将所述自适应弹性网络模型和所述基因交互网络惩罚相结合,构建自适应基因交互正则化弹性网络模型;
基因得出模块,用于基于梯度下降算法求解出所述自适应基因交互正则化弹性网络模型的最优解,基于所述最优解选择基因。
与现有技术相比,本发明具有的有益效果:
本发明的自适应基因交互正则化弹性网络模型扩展并整合了基因交互网络信息和自适应弹性网络模型,以达到更好的分类目的。普通的弹性网络模型不考虑基因之间交互的信息,而提出的自适应弹性网络模型包含基因交互的信息。将自适应弹性网络模型与基因交互网络融合在一起,采用梯度下降算法求解模型的最优解,本发明方便地整合基因重要性和基因交互信息来识别特征基因,降低冗余;还可以自适应地选择与肿瘤的产生高度相关的重要基因,并去除冗余、不相关的基因和噪声基因。
附图说明
图1为基因选择过程示意图;
图2为本发明实施例一种基于自适应基因交互正则化弹性网络模型的基因选择方法的基本流程图;
图3为本发明实施例一种基于自适应基因交互正则化弹性网络模型的基因选择系统的架构示意图。
具体实施方式
下面结合附图和具体的实施例对本发明做进一步的解释说明:
如图2所示,一种基于自适应基因交互正则化弹性网络模型的基因选择方法,包括:
步骤1:基于Wilcoxon秩和检验评估每个测量的基因的重要程度;
步骤2:对每个测量的基因的重要程度进行量化;
步骤3:根据量化后的每个基因的重要程度为每个测量的基因添加自适应惩罚权重,基于所述自适应惩罚权重删除噪声基因,得到特征基因;
步骤4:将所述自适应惩罚权重引入最小二乘损失函数中,从而构建自适应弹性网络模型;
步骤5:构建基因交互网络的邻接矩阵;
步骤6:基于所述邻接矩阵构建基因交互网络惩罚;
步骤7:将所述自适应弹性网络模型和所述基因交互网络惩罚相结合,构建自适应基因交互正则化弹性网络模型(AGIREN);
步骤8:基于梯度下降算法求解出所述自适应基因交互正则化弹性网络模型的最优解,基于所述最优解选择基因。
具体地,为了能够有效地挑选出重要的基因进行分类,通过施加自适应L
进一步地,所述步骤1包括:
基于Wilcoxon秩和检验,按照下式评估每个测量的基因的重要程度:
其中I(.)为指示函数;
进一步地,所述步骤2包括:
虽然通过Wilcoxon秩和检验可以对每个基因的重要程度进行度量,但是由于该统计量不能直接用于自适应惩罚权重,为了对每个基因的重要性进行量化,按照下式将基因进行排序:
R(g
其中s(g
进一步地,为了在分类时根据每个基因的重要程度进行区别惩罚,所述步骤3中,自适应惩罚权重的表达式为:
其中n为样本个数。噪声基因会获得相对较大的惩罚权重,而关键的特征基因获得较小的惩罚权重。
进一步地,将基因对分类的重要性,即基因权重w
其中O
进一步地,所述步骤5中,按照下式构建基因相互作用网络的邻接矩阵:
A=[a
其中R代表实数集,A表示基因相互作用网络的邻接矩阵;a
进一步地,为了保证已知的相互作用的基因具有相似的系数,从而更有可能分在同一组,需要最大化基因相互作用网络中的总分组效应,按照下式构建基因交互网络惩罚:
其中O
进一步地,所述步骤7中,所述自适应基因交互正则化弹性网络模型的表达式为:
其中F(X,A,β)表示自适应基因交互正则化弹性网络模型,X为输入矩阵,
进一步地,所述步骤8中,基于梯度下降算法求解出的自适应基因交互正则化弹性网络模型的表达式为:
其中
进一步地,在所述步骤8之后,还包括:
基于选择的基因进行分类,并对分类结果进行分析。
具体地,在进行疾病分类时存在的一些高度相关的基因应该作为一个基因群体,同时被选择或消除。作为一种新的正则化方法,弹性网络模型及其各种推广能够在创建分类器的过程中产生群体效应。为了能够有效地挑选出重要的基因进行分类,通过施加自适应L
基因与基因之间的相互作用是理解复杂疾病的根本要素,而表现型被认为是多个关键基因之间相互交互的结果。当进行癌症分类时,需要考虑基因的相互作用,当多个基因相互作用时,不用将所有的基因都看作为特征基因,因为由于基因交互的作用,它们所携带的信息会不可避免地具有相关性。为了避免冗余的产生,可以定义一个基于基因相互作用的网络约束,这样在网络中的任何变量都有可能被放置到相同的集合中。为了保证已知的相互作用的基因具有相似的系数,从而更有可能分在同一组,需要最大化基因相互作用网络中的总分组效应,即基因交互正则化模型
根据自适应弹性网络模型和基因交互正则化模型构建自适应基因交互正则化弹性网络模型(AGIREN):
在上述实施例的基础上,如图3所示,本发明还提出一种基于自适应基因交互正则化弹性网络模型的基因选择系统,包括:
基因重要程度评估模块,用于基于Wilcoxon秩和检验评估每个测量的基因的重要程度;
基因重要程度量化模块,用于对每个测量的基因的重要程度进行量化;
加权模块,用于根据量化后的每个基因的重要程度为每个测量的基因添加自适应惩罚权重,基于所述自适应惩罚权重删除噪声基因,得到特征基因;
第一构建模块,用于将所述基因权重引入最小二乘损失函数中,从而构建自适应弹性网络模型;
第二构建模块,用于构建基因交互网络的邻接矩阵;
第三构建模块,用于基于所述邻接矩阵构建基因交互网络惩罚;
第四构建模块,用于将所述自适应弹性网络模型和所述基因交互网络惩罚相结合,构建自适应基因交互正则化弹性网络模型;
基因得出模块,用于基于梯度下降算法求解出所述自适应基因交互正则化弹性网络模型的最优解,基于所述最优解选择基因。
综上,
(1)本发明通过Wilcoxon秩和检验在基于基因排序的分类方法中引入基因的重要性以更好的选择出对分类有重要贡献的特征基因;
(2)本发明对每个基因施加自适应惩罚权重,因此,噪声基因会有较大的惩罚从而被模型剔除,而特征基因的惩罚较小而被保留下来;
(3)由于基因与基因之间会存在大量的冗余信息,为了有效地剔除冗余基因,本发明构造基因与基因交互网络惩罚;
(4)结合上述三点。本发明提出的自适应基因交互正则化弹性网络模型,所构建的网络模型具有以下两个明显的特点。首先,自适应基因交互正则化弹性网络模型建立在自适应弹性网络模型的基础上,所以具有稀疏性,并根据回归系数选择相对较少的特征基因,选出的特征基因在癌症分类、临床结果预测等过程中发挥关键作用。其次,构建基因交互网络模型可以减少基因之间的冗余信息,并适用于各种各样的数据类型,例如DNA甲基化数据、基因表达谱数据、蛋白质相互作用等等。
以上所示仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
- 基于自适应基因交互正则化弹性网络模型的基因选择方法及系统
- 基于知识分类获取的交互式AI人工智能基因分析系统
