柠檬酸合酶活性减弱的修饰多肽及利用其产生L-氨基酸的方法
文献发布时间:2023-06-19 09:24:30
本申请是分案申请,原申请的申请日为2019年2月12日,申请号为201980001012.7,发明名称为“柠檬酸合酶活性减弱的修饰多肽及利用其产生L-氨基酸的方法”。
[技术领域]
本公开涉及柠檬酸合酶活性减弱的修饰多肽及利用该修饰多肽生产L-氨基酸的方法。
[背景技术]
棒杆菌(Corynebacterium)属微生物,特别是谷氨酸棒杆菌(Corynebacteriumglutamicum),是广泛用于L-氨基酸和其他有用物质的生产的革兰氏阳性微生物。对于L-氨基酸和其他有用物质的生产,正在进行各种研究以开发生产高效的微生物和发酵过程技术。例如,主要使用目标物质特异性方法(例如,增加编码L-赖氨酸生物合成涉及的酶或去除对生物合成不必要基因的基因的表达)(韩国专利号10-0838038)。
同时,在L-氨基酸中,L-赖氨酸、L-苏氨酸、L-甲硫氨酸、L-异亮氨酸、和L-甘氨酸是天冬氨酸(aspartate)衍生的氨基酸,并且草酰乙酸(即,天冬氨酸的前体)的生物合成水平可影响这些L-氨基酸的生物合成水平。
柠檬酸合酶(CS)是通过催化乙酰辅酶A和微生物糖酵解期间生成的草酰乙酸的缩合而生成柠檬酸的酶,并且其也是用于决定进入TCA途径的碳流(carbon-flow)的重要酶。
此前文献(Ooyen等,Biotechnol.Bioeng.,109(8):2070-2081,2012)中报道了由于编码柠檬酸合酶的gltA基因的缺失导致L-赖氨酸生产菌株中的表型改变。然而,gltA基因缺失的这些菌株的缺点在于,不仅其生长受到抑制而且其糖消耗速率显著降低,因此导致每单位时间的赖氨酸产量低。因此,仍需要可同时考虑有效增加L-氨基酸生产力和菌株生长两者的研究。
[发明内容]
[技术问题]
本发明人已经确认,当使用其中柠檬酸合酶活性减弱到一定水平的新型修饰多肽时,可在不延迟菌株的生长速率的情况下增加L-氨基酸生产量,从而完成本公开。
[技术方案]
本公开的一个目的是提供具有柠檬酸合酶活性的修饰多肽,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(天冬酰胺)被另一种氨基酸取代。
本公开的另一个目的是提供编码该修饰多肽的多核苷酸。
本公开的又另一个目的是提供包含该修饰多肽的生产天冬氨酸衍生的L-氨基酸的棒杆菌属微生物。
本公开的又另一个目的是提供用于生产L-氨基酸的方法,其包括在培养基中培养该棒杆菌属微生物;以及从培养的微生物或培养基中回收L-氨基酸。
[有益效果]
当使用具有减弱的柠檬酸合酶活性的本公开的新型修饰多肽时,可在不延迟生长速率的情况下进一步提高天冬氨酸衍生的L-氨基酸的生产量。
[附图说明]
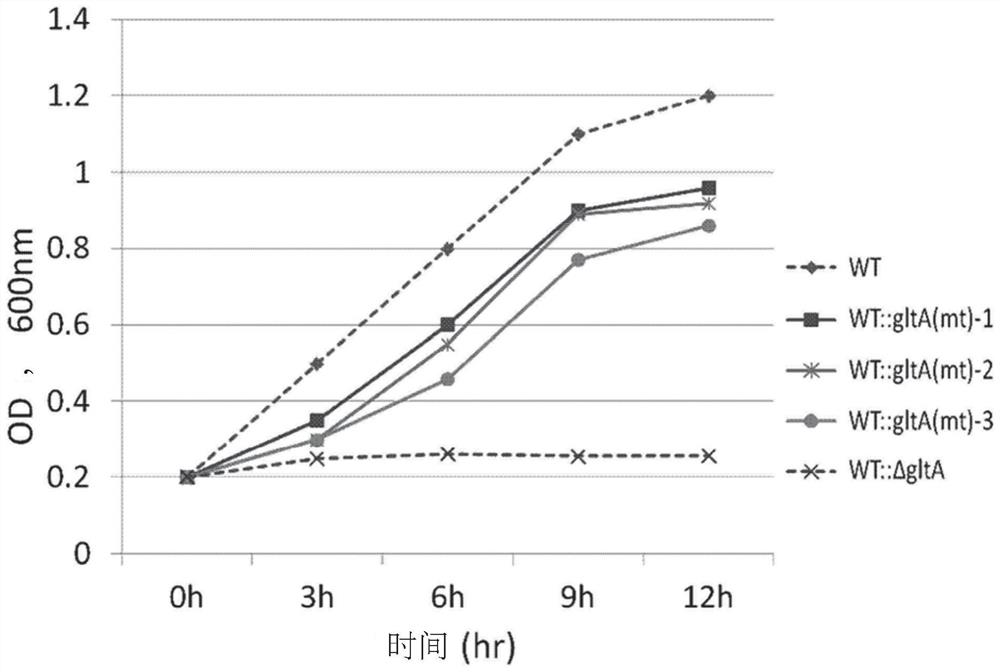
图1示出了菌株的生长曲线,其中引入了gltA基因的缺失和修饰。
[具体实施方式]
本公开详述如下。同时,本公开中公开的各个描述和实施方式也可应用于其他描述和实施方式。也就是说,本公开中公开的各种要素的所有组合落入本公开的范围内。此外,本公开的范围不受以下具体描述的限制。
为了实现上述目的,本公开的一个方面提供具有柠檬酸合酶活性的修饰多肽,其中修饰多肽包括SEQ ID NO:1的氨基酸的至少一个修饰并且该至少一个修饰包括SEQ IDNO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被另一种氨基酸取代。
具体地,修饰多肽可被描述为具有柠檬酸合酶活性的修饰多肽,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被另一种氨基酸取代。
在本公开中,SEQ ID NO:1是指具有柠檬酸合酶活性的氨基酸序列,并且具体地,具有由gltA基因编码的柠檬酸合酶的活性的蛋白质序列。SEQ ID NO:1的氨基酸序列可从公共数据库NCBI GenBank获得。例如,SEQ ID NO:1的氨基酸序列可源自谷氨酸棒杆菌,但该氨基酸序列不限于此,并且可非限制地包括与上述氨基酸序列具有相同活性的任何序列。此外,该氨基酸序列可包括SEQ ID NO:1的氨基酸序列或与SEQ ID NO:1的氨基酸序列具有80%以上同源性或同一性的任何氨基酸序列,但该氨基酸序列不限于此。具体地,该氨基酸序列可包括SEQ ID NO:1的氨基酸序列和与SEQ ID NO:1的氨基酸序列具有至少80%、90%、95%、96%、97%、98%、或99%以上同源性的任何氨基酸序列。此外,显然,具有如下氨基酸序列的任何蛋白质也可用于本公开:其中部分氨基酸序列被缺失(被删除,deleted)、修饰、取代、或添加,只要该蛋白质与上述蛋白质具有这样的氨基酸序列同源性或同一性并且呈现相应于上述蛋白质的效果。
也就是说,在本公开中,尽管描述为“具有具体SEQ ID NO的氨基酸序列的蛋白质或多肽”或“由具体SEQ ID NO的氨基酸序列组成的蛋白质或多肽”,但显然,与由相应SEQID NO的氨基酸序列组成的多肽具有基本上相同或等同的生物活性的任何蛋白质可用于本公开中,即使氨基酸序列的部分序列可具有缺失、修饰、取代、或添加。例如,显然,“由SEQID NO:1的氨基酸序列组成的多肽”可属于“由SEQ ID NO:1的氨基酸序列组成的多肽”。此外,在多肽具有与本公开的修饰多肽相同或等同的活性的情况下,除了在第241位氨基酸上的修饰或与其相应的修饰之外,不排除还有其中可因相应SEQ ID NO的氨基酸序列的上游或下游的无意义序列添加而导致的突变、天然存在的突变、或沉默突变,并且显然,在多肽具有这种序列添加或突变的情况下,所得肽也可属于本公开的范围。
如本文所用,术语“同源性”或“同一性”代表两个给定氨基酸序列或核苷酸序列之间的相关性并且可表示为百分比。这两个术语“同源性”和“同一性”通常彼此可互换地使用。
保守多核苷酸或多肽序列的序列同源性或同一性可通过标准比对算法确定并且可与通过在用程序建立的默认空位罚分(default gap penalty)一起使用。通常预期基本上同源或同一的序列在中等或高度严格条件下,沿着目标多核苷酸或多肽的全长或全长的至少约50%、约60%、约70%、约80%、或约90%杂交。关于杂交,还考虑含有简并密码子——代替杂交多肽的密码子——的多核苷酸。
任何两个多核苷酸或多肽序列是否具有同源性或同一性可利用已知的计算机算法确定,如Pearson等(Proc.Natl.Acad.Sci.USA 85:2444,(1988))描述的利用默认参数的“FASTA”程序。或者,同源性或同一性可利用以下确定:Needleman-Wunsch算法(Needlemanand Wunsch,1970,J.Mol.Biol.48:443-453)——其在EMBOSS包的Needleman程序(EMBOSS:The European Molecular Biology Open Software Suite,Rice等,2000,TrendsGenet.16:276-277)(优选地,版本5.0.0或其之后的版本)(GCG程序包(Devereux,J.,等,Nucleic Acids Research 12:387(1984))中进行、BLASTP、BLASTN、FASTA(Atschul,[S.][F.,]等人,J Molec Bio 215]:403(1990);Guide to Huge Computers,Martin J.Bishop,[ED.],Academic Press,San Diego,1994,和[CARILLO ETA/.](1988)SIAM J AppliedMath 48:1073)。例如,同源性、相似性、或同一性可利用National Center forBiotechnology Information的BLAST或ClustalW来确定。
多核苷酸或多肽之间的同源性、相似性、或同一性可通过比较序列信息来确定——利用例如GAP计算机程序(例如,Needleman等(1970),J Mol Biol.48:443),如文献(Smith和Waterman,Adv.Appl.Math(1981)2:482)中公开。总之,GAP程序将同源性、相似性、或同一性限定为通过比对相似的符号(symbol)(即,核苷酸或氨基酸)的数量除以两个序列中较短者中的符号总数而获得的值。GAP程序的默认参数可包括(1)一元比较矩阵(含有同一性值1和非同一性值0)和Gribskov等(1986),Nucl.Acids Res.14:6745的加权比较矩阵,如文献(Schwartz和Dayhoff,eds.,Atlas of Protein Sequence and Structure,National Biomedical Research Foundation,pp.353-358,1979)中公开;(2)每个空位3.0的罚分和每个空位中每个符号额外0.10罚分(或空位开口罚分(gap opening penalty)10和空位延伸罚分(gap extension penalty)0.5);以及(3)对末端空位(end gaps)无罚分。
此外,任何两个多核苷酸或多肽序列是否具有同源性、相似性、或同一性可通过经由Southern杂交实验比较这些序列来确定,并且将要限定的适当杂交条件可通过本领域技术人员已知的方法来确定(例如,J.Sambrook等,Molecular Cloning,A LaboratoryManual,2nd Edition,Cold Spring Harbor Laboratory press,Cold Spring Harbor,NewYork,1989;F.M.Ausubel等,Current Protocols in Molecular Biology,John Wiley&Sons,Inc.,New York)。
如本文所用,术语“修饰多肽”是指这样的多肽:其中一个或多个氨基酸区别于保守取代和/或修饰的所述序列,但维持了多肽的功能或性质。该修饰多肽区别于通过几个氨基酸的取代、缺失、或添加而鉴定的那些序列。这种修饰多肽可总体上通过修饰多肽序列中的一个和评价修饰多肽的性质来鉴定。也就是说,修饰多肽的能力可相对于天然蛋白质提高、不变、或降低。这种修饰多肽可总体上通过修饰多肽序列中的一个和评价修饰多肽的反应性来鉴定。此外,部分修饰多肽可包括其中其中的一个或多个部分(例如,N-端前导序列、跨膜结构域等)被去除的修饰多肽。其他修饰多肽可包括其中从各成熟蛋白质的N-和/或C-端去除其中一部分的那些多肽。在本公开中,术语“修饰”可与诸如下列的术语互换使用:如修饰、修饰蛋白质、修饰多肽、突变体、突变蛋白质、趋异体(divergent)、变体等,并且该术语不受限制,只要其用作修饰的含义。
如本文所用,术语“保守取代”是指氨基酸被具有相似结构和/或化学性质的另一种氨基酸取代。变体(修饰多肽)可具有例如一个或多个保守取代,同时维持一种或多种生物活性。这种氨基酸取代可总体上基于氨基酸残基的极性、电荷、可溶性、疏水性、亲水性、和/或两亲性的相似性来进行。例如,在带电荷的氨基酸中,带正电荷的(碱性)氨基酸包括精氨酸、赖氨酸、和组氨酸;以及带负电荷的(酸性)氨基酸包括谷氨酸和天冬氨酸;以及在不带电荷的氨基酸中,非极性氨基酸包括甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、甲硫氨酸、苯丙氨酸、色氨酸、和脯氨酸;极性或亲水性氨基酸包括丝氨酸、苏氨酸、半胱氨酸、酪氨酸、天冬酰胺、和谷氨酰胺;以及在极性氨基酸中,芳族氨基酸包括苯丙氨酸、色氨酸、和酪氨酸。
此外,变体(修饰多肽)可包括对多肽的特征和二级结构具有最小影响的氨基酸缺失或添加。例如,多肽可与蛋白质N-端的信号(或前导)序列缀合,该信号(或前导)序列涉及共翻译地或翻译后地蛋白质转移。此外,多肽还可与另一个将被鉴定、纯化、或合成的序列或连接体缀合。
本公开的修饰多肽可以是与SEQ ID NO:1的氨基酸序列相比具有减弱的柠檬酸合酶活性的修饰多肽,其中修饰多肽包括SEQ ID NO:1的氨基酸的至少一个修饰,并且所述至少一个修饰包括SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被另一种氨基酸取代。修饰多肽可被描述为相比于SEQ ID NO:1的氨基酸序列具有减弱的柠檬酸合酶活性的修饰多肽,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸被另一种氨基酸取代。
“被另一种氨基酸取代”不受限制,只要取代后的氨基酸不同于取代前的氨基酸。也就是说,当SEQ ID NO:1的氨基酸序列中的第241位氨基酸被另一种氨基酸取代时,所述另一种氨基酸不受限制,只要所述另一种氨基酸是天冬酰胺以外的氨基酸。
本公开的修饰多肽可以是相比于修饰前多肽、天然野生型多肽、或未修饰多肽具有降低的或减弱的柠檬酸合酶活性的多肽,但修饰多肽不限于此。
具体地,本公开的修饰多肽可以是其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被甘氨酸、丙氨酸、精氨酸、天冬氨酸、半胱氨酸、谷氨酸(glutamate)、谷氨酰胺、组氨酸、脯氨酸、丝氨酸、酪氨酸、异亮氨酸、亮氨酸、赖氨酸、色氨酸、缬氨酸、甲硫氨酸、苯丙氨酸、或苏氨酸取代的多肽。更具体地,修饰多肽可以是其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被赖氨酸以外的氨基酸取代的多肽,但取代不限于此。或者,修饰多肽可以是其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被酸性或碱性氨基酸以外的氨基酸取代,或被具有不带电荷的氨基酸的氨基酸取代的多肽,但取代不限于此。或者,修饰多肽可以是其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被非极性氨基酸或亲水性氨基酸,具体地被芳族氨基酸(例如,苯丙氨酸、色氨酸、和酪氨酸)或亲水性氨基酸(例如,丝氨酸、苏氨酸、酪氨酸、半胱氨酸、天冬酰胺、和谷氨酰胺)取代的多肽,但修饰多肽不限于此。更具体地,修饰多肽可以是具有减弱的柠檬酸合酶活性的多肽,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被苏氨酸、丝氨酸、或酪氨酸取代,但修饰多肽不限于此。甚至更具体地,修饰多肽可以是具有减弱的柠檬酸合酶活性的多肽,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被苏氨酸取代,但修饰多肽不限于此。
这种修饰多肽具有与具有SEQ ID NO:1的氨基酸序列的多肽相比减弱的柠檬酸合酶活性。显然,修饰多肽——其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸被另一种氨基酸取代——包括其中相应于第241位氨基酸的任何氨基酸被另一种氨基酸取代的修饰多肽。
具体地,在修饰多肽中,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被另一种氨基酸取代的修饰多肽可以是由SEQ ID NO:3、59、和61组成的多肽,并且更具体地,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)被苏氨酸、丝氨酸、或酪氨酸取代的修饰多肽可以是由SEQ ID NO:3、59、和61中的每一种组成的多肽,但修饰多肽不限于此。此外,修饰多肽可包括SEQ ID NO:3、59、和61的氨基酸序列、或与SEQID NO:3、59、和61的氨基酸序列的每一种具有至少80%的同源性的氨基酸序列,但修饰多肽不限于此。具体地,本公开的修饰多肽可包括SEQ ID NO:3、59、和61的氨基酸序列、或与SEQ ID NO:3、59、和61的氨基酸序列的每一种具有至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、或至少99%的同源性的氨基酸序列。此外,显然,具有除了氨基酸序列的第241位氨基酸之外还在部分氨基酸序列中具有缺失、修饰、取代、或添加的氨基酸序列的任何蛋白质也可被包括在本公开的范围内,只要该氨基酸序列具有上述同源性且具有相应于该蛋白质的效果。
如本文所用,术语“柠檬酸合酶(CS)”是指通过催化乙酰辅酶A和微生物糖酵解期间生成的草酰乙酸的缩合而生成柠檬酸的酶,并且其是决定进入TCA途径的碳流的重要酶。具体地,柠檬酸合酶作为用于合成柠檬酸的酶在TCA循环的第一步中充当调节速率的因子。此外,柠檬酸合酶催化来自乙酰辅酶A的二碳乙酸残基与4-碳草酰乙酸酯分子的缩合反应以形成6-碳乙酸酯。在本公开中,柠檬酸合酶可与“用于合成柠檬酸的酶”或“CS”互换使用。
本公开的另一方面提供编码修饰多肽的多核苷酸。
如本文所用,术语“多核苷酸”——其是由在长链中通过共价键连接的核苷酸单体构成的核苷酸聚合物——是具有至少一定长度的DNA或RNA链,且更具体地,编码修饰多肽的多核苷酸片段。
编码本公开的修饰多肽的多核苷酸可不受限制地包括编码本公开的具有减弱的柠檬酸合酶活性的修饰多肽的任何多核苷酸序列。在本公开中,编码柠檬酸合酶多肽的氨基酸序列的基因可以是gltA基因,具体地源自谷氨酸棒杆菌的基因,但基因不限于此。
由于密码子简并性或考虑到表达多核苷酸的生物中优选的密码子,本公开的多核苷酸可在编码区经历各种修饰而不改变多核苷酸的氨基酸序列。具体地,可不受限制地包括编码修饰多肽的任何多核苷酸序列,其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸被另一种氨基酸取代。例如,本公开的多核苷酸可以是分别由SEQ ID NO:3、59、和61的氨基酸序列组成的多核苷酸、或编码与这些多肽具有序列同源性的多肽的多核苷酸序列,但多核苷酸不限于此。更具体地,本公开的多核苷酸可以是由SEQ ID NO:4、60、和62的各多核苷酸序列组成的多核苷酸,但本公开的多核苷酸不限于此。
此外,可非限制地包括探针,该探针可由已知基因序列制备——例如,可在严格条件下全部或部分所述多核苷酸序列的互补序列杂交以编码蛋白质的任何序列,该蛋白质具有其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸被另一种氨基酸取代的修饰多肽的活性。
“严格条件”是指这样的条件:在该条件下允许在多核苷酸之间的特定杂交。这种条件被具体描述于文献(例如,J.Sambrook等,同上)中。严格条件可包括这样的条件:在该条件下具有高同源性,例如,40%以上的同源性,具体地90%以上的同源性,更具体地95%以上的同源性,更具体地97%以上的同源性,还更具体地99%以上的同源性的基因彼此杂交,并且具有低于上述同源性的同源性的基因不彼此杂交;或Southern杂交的普通洗涤条件(即,在相应于60℃,1×SSC,0.1%SDS,具体地60℃,0.1×SSC,0.1%SDS,更具体地68℃,0.1×SSC,0.1%SDS的盐浓度和温度下洗涤一次,具体地两次或三次),但严格条件不限于此,并且可由本领域技术人员适当地调整。
杂交需要两个多核苷酸含有互补序列,尽管根据杂交的严格性,碱基之间的错配是可能的。术语“互补”用于描述可彼此杂交的核苷酸碱基之间的关系。例如,关于DNA,腺苷与胸腺嘧啶互补且胞嘧啶与鸟嘌呤互补。因此,本公开可包括与整个序列互补的分离的核苷酸片段以及与其基本上相似的多核苷酸序列。
具体地,可利用包括在上述条件下中55℃的T
适于杂交多核苷酸的严格性取决于多核苷酸的长度和互补的程度,并且这些变量是本领域公知的(参见Sambrook等,同上,9.50-9.51,11.7-11.8)。
本公开的又另一方面提供包含修饰多肽的微生物。具体地,本公开提供包含修饰多肽的生产L-氨基酸的棒杆菌属微生物。更具体地,本公开提供包含修饰多肽的生产天冬氨酸衍生的L-氨基酸的棒杆菌属微生物。例如,提供的微生物可以是用含有编码修饰多肽的多核苷酸的载体转化的微生物,但微生物不限于此。
包含本公开的修饰多肽的微生物相比于包含野生型多肽的微生物具有提高的生产L-氨基酸的能力,而不抑制微生物的生长或糖消耗速率。因此,可从这些微生物以高产率获得L-氨基酸。具体地,可解释为包含修饰多肽的微生物可通过控制柠檬酸合酶的活性在进入TCA途径的碳流与草酰乙酸(用作L-氨基酸生物合成的前体)的供应量之间建立适当的平衡,并且因此,包含修饰多肽的微生物可增加L-氨基酸的生产量,但解释不限于此。
如本文所用,术语“L-氨基酸”是指含有胺和羧基官能团的有机化合物,并且具体地,α-氨基酸形式或L立体异构体形式(L-形式)的氨基酸。L-氨基酸可以是天冬酰胺、甘氨酸、丙氨酸、精氨酸、天冬氨酸、半胱氨酸、谷氨酸、谷氨酰胺、组氨酸、脯氨酸、丝氨酸、酪氨酸、异亮氨酸、亮氨酸、赖氨酸、色氨酸、缬氨酸、甲硫氨酸、苯丙氨酸、或苏氨酸。此外,L-氨基酸可以是L-高丝氨酸(α-氨基酸)或作为L-氨基酸前体的其衍生物,但L-氨基酸不限于此。L-高丝氨酸衍生物可以是,例如,选自O-乙酰高丝氨酸、O-琥珀酰高丝氨酸、和O-磷酸高丝氨酸中的一种,但L-高丝氨酸衍生物不限于此。
如本文所用,术语“天冬氨酸”是指用于蛋白质的生物合成的α-氨基酸并且可以与天门冬氨酸互换地使用。总体上,天冬氨酸由作为天冬氨酸前体的草酰乙酸生成,并且可在体内转化为L-赖氨酸、L-甲硫氨酸、L-高丝氨酸或其衍生物、L-苏氨酸、L-异亮氨酸等。
如本文所用,术语“天冬氨酸衍生的L-氨基酸(天冬氨酸源L-氨基酸)”是指可利用天冬氨酸作为前体生物合成的物质,并且天冬氨酸衍生的L-氨基酸不限于此,只要L-氨基酸可利用天冬氨酸作为前体经由生物合成生成。天冬氨酸衍生的L-氨基酸可不仅包括天冬氨酸衍生的L-氨基酸而且包括其衍生物。例如,L-氨基酸和其衍生物可以是L-赖氨酸、L-苏氨酸、L-甲硫氨酸、L-甘氨酸、高丝氨酸或其衍生物(O-乙酰高丝氨酸、O-琥珀酰高丝氨酸、和O-磷酸高丝氨酸)、L-异亮氨酸、和/或尸胺,但L-氨基酸和其衍生物不限于此。具体地,L-氨基酸和其衍生物可以是L-赖氨酸、L-苏氨酸、L-甲硫氨酸、高丝氨酸或其生物、和/或L-异亮氨酸、且更具体地,L-氨基酸和其衍生物可以是L-赖氨酸、L-苏氨酸和/或L-异亮氨酸,但L-氨基酸和其衍生物不限于此。
如本文所用,术语“载体”是指这样的DNA产物:包含编码目标蛋白质的多核苷酸的核苷酸序列,该核苷酸序列与适当的控制序列可操作地连接以在适当的宿主中表达目标蛋白质。控制序列包括能够起始转录的启动子、用于控制转录的任意操纵子序列、编码适当的mRNA核糖体结合位点的序列、和用于控制转录和翻译的终止的序列。在转化到适当的宿主细胞后,载体可独立于宿主基因组复制或作用,或者可整合到基因组自身中。
本公开中所使用的载体不受具体限制,只要其可在宿主细胞中复制,并且可使用本领域已知的任何载体。常规使用的载体实例可包括天然或重组质粒、粘粒、病毒、和噬菌体。例如,作为噬菌体载体或粘粒载体,可使用pWE15、M13、MBL3、MBL4、IXII、ASHII、APII、t10、t11、Charon4A、Charon21A等;且作为质粒载体,可使用基于pBR、pUC、pBluescriptII、pGEM、pTZ、pCL、pET等的载体。具体地,可使用pDZ、pACYC177、pACYC184、pCL、pECCG117、pUC19、pBR322、pMW118、pCC1BAC载体等。
可在本公开中所使用的载体不受具体限制,且可使用任何已知的表达载体。此外,可利用用于细胞内染色体插入的载体将编码目标蛋白质的多核苷酸插入到染色体中。多核苷酸在染色体中的插入可通过本领域已知的任何方法(例如,同源重组)进行,但方法不限于此。载体可进一步包含选择标记以确认基因成功插入染色体中。选择标记用于筛选被载体转化的细胞,也就是说,用于确定目标多核苷酸分子是否已被插入。可使用提供可选择表型(例如,抗药性、营养缺陷(auxotrophy)、细胞毒剂抗性、或表面蛋白表达)的标记。在用选择剂处理的情况下,只有表达选择标记的细胞可存活或表达其他表型性状,因此可选择转化的细胞。
如本文所用,术语“转化”是指将包含编码目标蛋白质的多核苷酸的载体引入到宿主细胞中,使得由多核苷酸编码的蛋白质可在宿主细胞中表达。只要转化的多核苷酸可在宿主细胞中表达,转化的多核苷酸是否整合到宿主细胞的染色体中以及是位于其中还是位于染色体外是无关紧要的。此外,多核苷酸包含编码目标蛋白质的DNA和RNA。多核苷酸可以任何形式引入,只要其可被引入到宿主细胞中并在其中表达。例如,多核苷酸可以表达盒的形式被引入到宿主细胞中,表达盒是包含其自主表达所需的所有元件的基因构建体。表达盒可包含可操作地连接到多核苷酸的启动子、转录终止子、核糖体结合位点、或翻译终止子。表达盒可以是可自我复制表达载体的形式。此外,多核苷酸可以被原样引入到宿主细胞中并且可操作地连接到在宿主细胞中表达所需的序列,但多核苷酸向细胞中的引入不限于此。转化方法包括将多核苷酸引入细胞的任何方法,并且可通过根据宿主细胞选择本领域已知的适当标准技术来进行。例如,方法可包括电穿孔、磷酸钙(Ca(H
此外,如本文所用,术语“可操作的连接”意指多核苷酸序列与启动子序列功能性连接,启动子序列该启动并介导编码本公开的目标蛋白质的多核苷酸的转录。可操作的连接可利用本领域已知的基因重组技术准备,并且位点特异性DNA切割和连接可利用本领域已知的用于切割和连接的酶等准备,但可操作的连接的准备不限于此。
如本文所用,术语“包含修饰多肽的微生物”是指包含编码修饰多肽的多核苷酸的宿主细胞或微生物、或用包含编码修饰多肽的多核苷酸的载体转化并且因此能够在其中表达修饰多肽的宿主细胞或微生物。宿主细胞或微生物可以是天然野生型的或其中已发生天然或人工基因修饰的宿主细胞或微生物。具体地,本公开的微生物可以是具有柠檬酸合酶活性的其中第241位氨基酸(天冬酰胺)被另一种氨基酸取代从而表达修饰多肽的微生物,但微生物不限于此。此外,包含修饰多肽的微生物可以是生产L-氨基酸的微生物。具体地,包含修饰多肽的微生物可以是与其天然型或未修饰的母体菌株相比具有提高的L-氨基酸生产能力的微生物,但微生物不限于此。此外,包含修饰多肽的微生物可以是生产天冬氨酸衍生的L-氨基酸的微生物。具体地,包含修饰多肽的微生物可以是与其天然型或未修饰的母体菌株相比具有提高的天冬氨酸衍生的L-氨基酸的生产能力的微生物,但微生物不限于此。
微生物的实例包括埃希氏菌(Escherichia)、沙雷氏菌(Serratia)、欧文氏菌(Erwinia)、肠杆菌(Enterobacteria)、沙门氏菌(Salmonella)、链霉菌(Streptomyces)、假单胞菌(Pseudomonas)、短杆菌(Brevibacterium)、棒杆菌属等微生物菌株,且特别是棒杆菌属微生物。
例如,棒杆菌属微生物可以是谷氨酸棒杆菌、产氨棒杆菌(Corynebacteriumammoniagenes)、乳酸发酵短杆菌(Brevibacterium lactofermentum)、黄色短杆菌(Brevibacterium flavum)、热产氨棒杆菌(Corynebacterium thermoaminogenes)、有效棒杆菌(Corynebacterium efficiens)等,但棒杆菌属微生物不必限制于此。更具体地,棒杆菌属微生物可以是谷氨酸棒杆菌,但微生物不限于此。
在具体实施方式中,微生物可以是生产L-赖氨酸的微生物,其中gltA的修饰被引入棒杆菌属微生物中,其中由三种修饰的pyc、hom、和lysC基因编码的蛋白质的活性增加并且因此生产L-赖氨酸的能力增加。
此外,微生物可以是生产L-苏氨酸和L-异亮氨酸的微生物,其中在编码生成高丝氨酸(即,L-苏氨酸和L-异亮氨酸的生物合成途径中的常见中间体)的高丝氨酸脱氢酶的基因中引入修饰,从而增强该基因的活性,但微生物不限于此。具体地,微生物可以是生产L-异亮氨酸的微生物,其中在编码L-苏氨酸脱水酶的基因中引入进一步修饰,从而增强该基因的活性,但微生物不限于此。因此,基于本公开的目的,生产L-氨基酸的微生物可以是其中进一步添加修饰多肽并且因此增加产生目标L-氨基酸的能力的微生物。
本公开的另一方面提供用于生产L-氨基酸的方法,包括在培养基中培养微生物;以及从培养的微生物或培养基回收L-氨基酸。具体地,L-氨基酸可以是天冬氨酸衍生的L-氨基酸。
该方法可容易由本领域技术人员在优化培养条件和酶活性条件下确定。具体地,微生物可通过已知的分批培养、连续培养、补料分批培养等培养,但培养方法不具体限制于此。具体地,培养条件不受具体限制,但pH(例如,pH 5至pH 9,具体地pH 6至pH 8,且更具体地pH 6.8)可用碱性化合物(例如,氢氧化钠、氢氧化钾、或氨)或酸性化合物(例如,磷酸或硫酸)适当地调整。可通过向培养物添加氧气或含氧气体混合物来维持有氧条件。培养温度可维持在20℃至45℃,且具体地在25℃至40℃,并且培养可进行约10至160小时,但培养条件不限制于此。通过培养产生的L-氨基酸可被分泌到培养基中或可保留在细胞内。
此外,在培养基中,可单独使用或组合使用碳源,如糖和碳水化合物(例如,葡萄糖、蔗糖、乳糖、果糖、麦芽糖、糖蜜、淀粉、和纤维素)、油和脂肪(例如,大豆油、葵花籽油、花生油、和椰子油)、脂肪酸(例如,棕榈酸、硬脂酸、和亚油酸)、醇(例如,甘油和乙醇)、和有机酸(例如,乙酸),但碳源不限于此;可单独使用或组合使用氮源,如含氮有机化合物(例如,蛋白胨、酵母提取物、肉汁、麦芽提取物、玉米浆、大豆粉、和尿素)、或无机化合物(例如,硫酸铵、氯化铵、磷酸铵、碳酸铵、和硝酸铵),但氮源不限于此;以及可单独使用或组合使用钾源,如磷酸二氢钾、磷酸氢二钾,或其相应的含钠盐,但钾源不限于此。此外,培养基中可含有其他重要的生长刺激物质,包括金属盐(例如,硫酸镁或硫酸铁)、氨基酸、和维生素。
在回收在本公开的培养步骤中产生的L-氨基酸的方法中,可以根据培养方法利用本领域已知的适当方法从培养物收集目标氨基酸。例如,可利用离心、过滤、阴离子交换色谱、结晶、HPLC等,并且可利用本领域已知的适当方法从培养基或微生物回收期望的L-氨基酸。
此外,回收步骤可包括纯化过程。纯化过程可利用本领域已知的适当方法进行。因此,被回收的L-氨基酸可以是纯化形式或含有L-氨基酸的微生物发酵液。
[发明详细描述]
在下文中,将通过示例性实施方式详细地描述本公开。然而,这些示例性实施方式仅是用于说明性目的,并不旨在限制本公开的范围。
为了发现其中谷氨酸棒杆菌的gltA基因的表达水平或活性减弱的修饰菌株,通过以下方法制备文库。
首先,利用GenemorphII Random Mutagenesis试剂盒(Stratagene),在每1kb包含gltA基因(1,314bp)的DNA片段(1,814bp)中引入0至4.5个修饰。利用谷氨酸棒杆菌ATCC13032(WT)的染色体作为模板和引物(SEQ ID NO:5和6)进行易错PCR(表1)。具体地,含有WT菌株(500ng)的染色体、引物5和6(各125ng)、Mutazyme II反应缓冲液(1Х)、dNTP混合物(40mM)、和Mutazyme II DNA聚合酶(2.5U)的反应溶液进行94℃变性2分钟,然后进行25个循环的94℃变性1分钟、56℃退火1分钟、以及72℃聚合3分钟,然后72℃聚合10分钟。
利用TOPO TA Cloning试剂盒(Invitrogen)将扩增的基因片段连接到pCRII载体,转化到大肠杆菌(E.coli)DH5α中,并且将转化的大肠杆菌DH5α平皿接种在含有卡那霉素(25mg/L)的固体LB培养基上。选择20个转化的菌落,并且对从中获得的质粒进行序列分析。结果是,确认修饰以0.5突变/kb的频率被引入彼此不同的位点。最后,收集约10,000个大肠杆菌转化菌落,并且从中提取质粒并命名为pTOPO-gltA(mt)文库。
[表1]
为了制备其中gltA基因被删除的野生型谷氨酸棒杆菌ATCC13032菌株,如下制备了其中gltA基因被删除的pDZ-ΔgltA载体。具体地,制备了pDZ-ΔgltA载体(韩国专利号10-0924065),使得位于gltA基因的5′和3′的DNA片段(各600bp)连接到pDZ-ΔgltA载体。合成了引物(SEQ ID NO:7和8),其中基于报道的gltA基因(SEQ ID NO:2)的核苷酸序列,限制酶(XbaI)的识别位点分别被插入5'片段和3'片段;和引物(SEQ ID NO:9和10),各自均位于距引物(SEQ ID NO:7和8)600bp(表2)处。利用谷氨酸棒杆菌ATCC13032的染色体作为模板和引物(SEQ ID NO:7和9),通过PCR制备5'-端片段。同样地,利用引物(SEQ ID NO:8和10),通过PCR制备位于gltA基因的3'端的基因片段。PCR如下进行:在94℃下变性2分钟;在94℃下变性1分钟,在56℃下退火1分钟,和在72℃下聚合40秒,30个循环;以及在72℃下聚合10分钟。
同时,将如上所述通过PCR扩增的DNA片段连接到pDZ载体,将其用限制酶(XbaI)切割,然后在65℃下热处理20分钟,转化到大肠杆菌DH5α中,并且将转化的大肠杆菌DH5α平皿接种在含有卡那霉素(25mg/L)的固体LB培养基上。选择被载体转化的菌落——其中利用引物(SEQ ID NO:7和8)通过PCR将目标基因插入到其中,并且利用常规质粒提取方法从菌落获得质粒,并且命名为pDZ-ΔgltA。
[表2]
通过电脉冲法(Van der Rest等,Appl.Microbiol.Biotecnol.52:541-545,1999)将制备的pDZ-ΔgltA载体转化到谷氨酸棒杆菌ATCC13032中,并且通过同源重组制备其中缺失gltA基因的菌株。gltA基因缺失的菌株命名为谷氨酸棒杆菌WT::ΔgltA。
此外,利用WT::ΔgltA菌株通过电脉冲法转化pTOPO-gltA(mt)文库。将转化的菌株平皿接种在含有卡那霉素(25mg/L)的复合平板培养基上,并且从中获得了约500个菌落。将所获得的菌落接种到其中含有种子培养基(200μL/孔)的96孔板中,并且将菌株在32℃下以1,000rpm的速率培养约9小时。
<复合平板培养基(pH 7.0)>
葡萄糖(10g)、蛋白胨(10g)、牛肉提取物(5g)、酵母提取物(5g)、脑心浸液(18.5g)、NaCl(2.5g)、尿素(2g)、山梨糖醇(91g)、琼脂(20g)(基于1L蒸馏水)
<种子培养基(pH 7.0)>
葡萄糖(20g)、蛋白胨(10g)、酵母提取物(5g)、尿素(1.5g)、KH
利用UV分光光度计微型读数器(Shimazu)监测培养期间的细胞生长(图1)。WT和WT::ΔgltA菌株用作对照组。选择三种菌株——其中相比于野生型(WT)菌株,细胞团(cellmass)较小但细胞生长速率维持在较高速率,并命名为WT::gltA(mt)-1至3。其余497个菌株显示,与WT和WT::ΔgltA菌株(对照组)相比相似或增加的细胞团或降低的生长速率。
为了确认三种所选菌株(即,WT::gltA(mt)-1至3)的gltA基因的核苷酸序列,使用实施例1中指定的引物(SEQ ID NO:5和6)扩增染色体中包含gltA基因的DNA片段。PCR如下进行:在94℃下变性2分钟;在94℃下变性1分钟,在56℃下退火1分钟,和在72℃下聚合40秒,30个循环;以及在72℃下聚合10分钟。
分析扩增基因的核苷酸序列,并且结果是,确认这些核苷酸序列共同显示1至2个修饰被引入位于gltA基因ORF起始密码子下游721bp至723bp的核苷酸序列。也就是说,确认WT::gltA(mt)-1至3菌株是柠檬酸合酶(CS)的修饰菌株,其中第721至723位核苷酸序列从原来的‘AAC’变成‘ACC’或‘ACT’(即,从gltA基因的自N端第241位氨基酸由天冬酰胺变成苏氨酸)。
尝试用天冬酰胺以外的氨基酸取代SEQ ID NO:1的氨基酸序列中的第241位氨基酸(即,天冬酰胺)(野生型菌株所具有)。
为引入包括N241T(其是实施例3中确认的修饰)在内的19种异质核苷酸取代修饰,如下制备了各重组载体。
首先,使用从WT菌株提取的基因组DNA作为模板,合成引物(SEQ ID NO:11和12)——其中限制酶(XbaI)的识别位点被分别插入5'片段和3'片段,于下游或上游距gltA基因的第721至723位核苷酸序列位置约600bp。为了引入19种异质核苷酸取代修饰,合成引物(SEQ ID NO:13至48),用于取代gltA基因的第721至723位核苷酸序列(表3)。
具体地,以将位于gltA基因的5′和3′端的DNA片段(各600bp)连接到pDZ载体(韩国专利号2009-0094433)的形式,制备pDZ-gltA(N241A)质粒。利用WT菌株的染色体作为模板和引物(SEQ ID NO:11和13),通过PCR制备gltA基因的5′端基因片段。PCR如下进行:在94℃下变性2分钟;在94℃下变性1分钟,在56℃下退火1分钟,和在72℃下聚合40秒,30个循环;以及在72℃下聚合10分钟。同样,利用引物(SEQ ID NO:12和14),通过PCR制备gltA基因的3′端基因片段。使用PCR纯化试剂盒(Quiagen)纯化扩增的DNA片段,并且用作插入DNA片段(insertion DMA fragments)以制备载体。
同时,通过PCR扩增插入DNA片段,并且将pDZ载体(其用限制酶(XbaI)切割,然后在65℃下热处理20分钟)用Infusion Cloning试剂盒连接,然后转化到大肠杆菌DH5α中。将菌株平皿接种在含有卡那霉素(25mg/L)的固体LB培养基上。选择转化的菌落——其中目标基因通过PCR用引物(SEQ ID NO:11和12)插入到载体中,并且利用公知的质粒提取方法获得质粒,并命名为pDZ-gltA(N241A)。
同样地,制备质粒如下:pDZ-gltA(N241V),使用引物(SEQ ID NO:11和15与SEQ IDNO:12和16);pDZ-gltA(N241Q),使用引物(SEQ ID NO:11和17与SEQ ID NO:12和18);pDZ-gltA(N241H),使用引物(SEQ ID NO:11和19与SEQ ID NO:12和20);pDZ-gltA(N241R),使用引物(SEQ ID NO:11和21与SEQ ID NO:12和22);pDZ-gltA(N241P),使用引物(SEQ ID NO:11和23与SEQ ID NO:12和24);pDZ-gltA(N241L),使用引物(SEQ ID NO:11和25与SEQ IDNO:12和26);pDZ-gltA(N241Y),使用引物(SEQ ID NO:11和27与SEQ ID NO:12和28);pDZ-gltA(N241S),使用引物(SEQ ID NO:11和29与SEQ ID NO:12和30);pDZ-gltA(N241K),使用引物(SEQ ID NO:11和31与SEQ ID NO:12和32);pDZ-gltA(N241M),使用引物(SEQ ID NO:11和33与SEQ ID NO:12和34);pDZ-gltA(N241I),使用引物(SEQ ID NO:11和35与SEQ IDNO:12和36);pDZ-gltA(N241E),使用引物(SEQ ID NO:11和37与SEQ ID NO:12和38);pDZ-gltA(N241D),使用引物(SEQ ID NO:11和39与SEQ ID NO:12和40);pDZ-gltA(N241G),使用引物(SEQ ID NO:11和41与SEQ ID NO:12和42);pDZ-gltA(N241W),使用引物(SEQ ID NO:11和43与SEQ ID NO:12和44);pDZ-gltA(N241C),使用引物(SEQ ID NO:11和45与SEQ IDNO:12和46);pDZ-gltA(N241F),使用引物(SEQ ID NO:11和47与SEQ ID NO:12和48);pDZ-gltA(N241T),使用引物(SEQ ID NO:11和49与SEQ ID NO:12和50)。
[表3]
通过电脉冲法将各制备的载体转化到生产赖氨酸的谷氨酸棒杆菌KCCM11016P菌株(韩国专利号10-0159812)中。19种菌株——其中异质核苷酸取代修饰被引入各菌株的gltA基因——命名如下:KCCM11016P::gltA(N241A)、KCCM11016P::gltA(N241V)、KCCM11016P::gltA(N241Q)、KCCM11016P::gltA(N241H)、KCCM11016P::gltA(N241R)、KCCM11016P::gltA(N241P)、KCCM11016P::gltA(N241L)、KCCM11016P::gltA(N241Y)、KCCM11016P::gltA(N241S)、KCCM11016P::gltA(N241K)、KCCM11016P::gltA(N241M)、KCCM11016P::gltA(N241I)、KCCM11016P::gltA(N241E)、KCCM11016P::gltA(N241D)、KCCM11016P::gltA(N241G)、KCCM11016P::gltA(N241W)、KCCM11016P::gltA(N241C)、KCCM11016P::gltA(N241F)、和KCCM11016P::gltA(N241T)。
通过此前报道的方法(Ooyen等,Biotechnol.Bioeng.,109(8):2070-2081,2012)测量实施例4中制备的菌株的柠檬酸合酶(CS)活性。通过实施例1中使用的方法删除KCCM11016P菌株的gltA基因,并且将所得菌株命名为KCCM11016P::ΔgltA。当使用KCCM11016P和KCCM11016P::ΔgltA菌株作为对照组时,将选择的19种菌株如下所述培养,并且测量糖消耗速率、赖氨酸的产率、谷氨酸(GA)(即,培养基中的代表性副产物)的浓度、和CS酶活性。
首先,将各菌株接种到含有25mL种子培养基的250mL角-挡板烧瓶(corner-baffleflask)中,并且在振荡培养箱(200rpm)中在30℃下培养20小时。然后,对含有24mL L-赖氨酸生产培养基的各250mL角-挡板烧瓶接种1mL种子培养液(seed culture broth)并且在振荡培养箱(200rpm)中在32℃下培养72小时。种子培养基和生产培养基的组成如下所示。培养完成后,通过HPLC(Waters 2478)测量各培养物中L-赖氨酸和谷氨酸的浓度。
<种子培养基(pH 7.0)>
葡萄糖(20g)、蛋白胨(10g)、酵母提取物(5g)、尿素(1.5g)、KH
<生产培养基(pH 7.0)>
葡萄糖(100g)、(NH
为了测量CS酶活性,将细胞通过离心回收,用100mM Tris-HCl缓冲液(pH 7.2,3mML-半胱氨酸,10mM MgCl
[表4]赖氨酸生产能力、培养液的组成、和CS酶活性(%)的测量
在缺失gltA基因的菌株的情况下,菌株的赖氨酸产率显示与其母体菌株相比增加约5.5%p,但该菌株直到培养后期才能消耗糖。也就是说,在gltA基因缺失并因此菌株几乎没有CS活性的情况下,菌株的生长受到抑制,因此使该菌株的工业应用困难。确认在菌株包含修饰多肽(其中SEQ ID NO:1的氨基酸序列中的第241位氨基酸被不同氨基酸取代)的所有情况下,CS活性减弱,同时菌株的生长维持在工业适用水平。此外,随着菌株的CS活性减弱,菌株的赖氨酸产率相比于其母体菌株倾向于增加约3%p至5%p。具体地,在其中CS活性减弱到30%至60%水平的修饰菌株中的三种修饰菌株(即,N241S、N241Y、和N241T)的情况下,相比于其母体菌株,这些菌株显示赖氨酸产率增加约3%p至5%p,同时显示糖消耗速率水平相似。此外,确认赖氨酸产率相比于其母体菌株增加的菌株显示培养液中谷氨酸(GA)量减少。也就是说,解释了本公开的修饰在这些菌株中的引入具有提高这些菌株的赖氨酸产率同时减少这些菌株的副产物的作用。
这些结果显示赖氨酸生产量可经由进入TCA途径的碳流和草酰乙酸(即,赖氨酸生物合成的前体)的供应量之间的适当平衡而增加。具体地,考虑到谷氨酸(在赖氨酸培养期间通常作为副产物大量产生)的量减少,确认gltA基因活性的减弱抑制进入TCA途径的碳流并且从而将碳流导向赖氨酸生物合成的方向,因此显著有效提高赖氨酸生产力。
在上述制备的菌株中,KCCM11016P::gltA(N241T)菌株于2017年11月20日被保藏于韩国微生物保藏中心(KCCM)(韩国,首尔)的韩国保藏(Korean Collection),根据布达佩斯条约的国际保藏机构,并且被分配登录号KCCM12154P(谷氨酸棒杆菌(Corynebacteriumglutamicum)CA01-7513)。
将实施例5中选择的三种gltA修饰菌株的修饰引入生产L-赖氨酸的谷氨酸棒杆菌菌株(即,KCCM10770P(韩国专利号10-0924065)和KCCM11347P(韩国专利号10-0073610))中。这三种菌株是基于其具有降低的CS活性、具有与其母体菌株相似的糖消耗速率、和与其母体菌株相比增加的赖氨酸产率的标准来选择的。将实施例4的三种载体(即,pDZ-gltA(N241S)、pDZ-gltA(N241Y)、和pDZ-gltA(N241T))通过电脉冲法引入到两种谷氨酸棒杆菌菌株(即,KCCM10770P和KCCM11347P)中,以制备六种菌株(即,KCCM10770P::gltA(N241S)、KCCM10770P::gltA(N241Y)、KCCM10770P::gltA(N241T)、KCCM11347P::gltA(N241S)、KCCM11347P::gltA(N241Y)、和KCCM11347P::gltA(N241T))。将两种对照组菌株(即,KCCM10770P和KCCM11347P)和具有gltA基因的核苷酸取代修饰的六种菌株以与实施例5中相同的方式培养,并分析这些菌株的赖氨酸生产能力、糖消耗速率、和培养液组成。
在培养这些菌株一定时间段后,分析赖氨酸生产能力、糖消耗速率和培养液组成。结果显示在下表5中。
[表5]gltA修饰菌株的赖氨酸生产能力、糖消耗速率和培养液组成的分析
如在上表5的结果中所示,在其中gltA序列第241位氨基酸被另一种氨基酸取代修饰的两种赖氨酸生产菌株(即,KCCM10770P和KCCM11347P)的情况下,所有菌株均显示与其母体菌株相比赖氨酸产率增加、副产物地产率减少、和糖消耗速率相似。确认在这三种修饰中,具有修饰(N241T)(即,其中第241位氨基酸(天冬酰胺)被苏氨酸取代的修饰)的菌株显示出赖氨酸产率的增加最高,同时显示与其母体菌株相比相似水平或显著增加水平的糖消耗速率。此外,确认N241T修饰显示谷氨酸产率的降低水平最高。由这些结果确认,gltA基因活性的减弱导致进入TCA途径的减少,从而引起培养液中谷氨酸量减少,如在实施例6的结果确认。
为了确认其他生产L-赖氨酸的谷氨酸棒杆菌菌株是否也具有上述相同效果,使用谷氨酸棒杆菌CJ3P(Binder等,Genome Biology,2012,13:R40)制备其中引入gltA(N241T)修饰的菌株,CJ3P被提供有L-赖氨酸生产能力——通过以与实施例6中相同的方式向野生型菌株引入三种修饰[即,pyc(P458S)、hom(V59A)、和lysC(T311I)]。由此制备的菌株被命名为CJ3::gltA(N241T)。以与实施例5中相同的方式培养对照组(即,CJ3P和CJ3::gltA(N241T)菌株),并且分析赖氨酸生产能力、糖消耗速率和培养液组成,结果显示在下表6中。
[表6]CJ3P衍生的gltA修饰菌株的赖氨酸产生能力、糖消耗速率和培养液组成的分析
赖氨酸生产能力、糖消耗速率和培养液组成的分析结果是,确认引入gltA(N241T))修饰的菌株显示赖氨酸产率增加和谷氨酸浓度降低,同时维持相似水平的糖消耗速率。
为了明确确认引入gltA(N241T)修饰导致L-苏氨酸生产能力变化,通过修饰使编码产生高丝氨酸(即,L-苏氨酸和L-异亮氨酸的生物合成途径中的相同中间体)的高丝氨酸脱氢酶的基因过表达。具体地,制备菌株,其中已知的hom(G378E)修饰(R.Winkels,S.等,Appl.Microbiol.Biotechnol.45,612-620,1996)被引入实施例7中使用的CJ3P::gltA(N241T)菌株。此外,制备其中仅hom(G378E)修饰被引入CJ3P的菌株作为对照组。通过下述方法制备用于引入修饰的重组载体。
为了制备用于引入hom(G378E)的载体,首先,利用从WT菌株提取的基因组DNA作为模板,合成引物(SEQ ID NO:51和52),其中限制酶(XbaI)的识别位点被分别插入5'-端片段和3'-端片段,于下游或上游距hom基因的第1,131至1,134位核苷酸序列位置约600bp。用于取代hom基因的核苷酸序列的引物(SEQ ID NO:53和54)(表7)。制备pDZ-hom(G378E)质粒——以位于5'和3'端每一端的DNA片段(各600bp)连接到pDZ载体(韩国专利号2009-0094433)的形式。使用WT菌株的染色体作为模板和引物(SEQ ID NO:51和53),通过PCR制备hom基因的5'-端片段。PCR如下进行:在94℃下变性2分钟;在94℃下变性1分钟,在56℃下退火1分钟,和在72℃下聚合40秒,30个循环;以及在72℃下聚合10分钟。同样地,使用引物(SEQ ID NO:52和54),通过PCR制备hom基因的3'-端片段。将扩增的DNA片段利用PCR纯化试剂盒(Quiagen)纯化,并作为插入DNA片段用于载体制备。同时,使用Infusion Cloning试剂盒,将用限制酶XbaI处理并在65℃下热处理20分钟的pDZ载体连接到通过PCR扩增的插入DNA片段,并且将连接的产物转化到大肠杆菌DH5α中并平皿接种在含有卡那霉素(25mg/L)的固体LB培养基上。选择被其中插入了利用引物(SEQ ID NO:51和52)通过PCR获得的目标基因的载体转化的菌落,并通过公知的质粒提取方法获得质粒,从而制备用于在染色体上引入hom(G378E)核苷酸取代修饰的载体(即,pDZ-hom(G378E))。
[表7]
获得了菌株(即,CJ3P::hom(G378E)和CJ3P::gltA(N241T)-hom(G378E)),其中利用与实施例6中相同的方法通过pDZ-hom(G378E)载体在CJ3P和CJ3P::gltA(N241T)菌株的hom基因中引入了核苷酸修饰。利用与实施例5中相同的方法培养所得两种菌株,并且分析苏氨酸浓度、糖消耗速率和培养液组成。结果显示在下表8中。
[表8]苏氨酸浓度、糖消耗速率、和培养液组成。
苏氨酸浓度、糖消耗速率和培养液组成的分析结果是,确认在引入了gltA(N241T)修饰的菌株中,苏氨酸浓度以与糖消耗速率相似的水平下增加,同时谷氨酸浓度降低。
为了确认gltA(N241T)修饰的引入对L-异亮氨酸生产能力的影响,通过此前报道的修饰,还使编码L-苏氨酸脱水酶的基因过表达。具体地,制备了如下菌株:其中将已知的ilvA(V323A)修饰(S.Morbach等,Appl.Enviro.Microbiol.,62(12):4345-4351,1996)被引入实施例7中使用的CJ3P::gltA(N241T)-hom(G378E)菌株中。此外,制备其中仅ilvA(V323A)修饰被引入CJ3P::hom(G378E)的菌株作为对照组。通过下述方法制备用于引入修饰的重组载体。
为了制备用于引入ilvA(V323A)的载体,首先,利用从WT菌株提取的基因组DNA作为模板,合成引物(SEQ ID NO:55和56),其中限制酶(XbaI)的识别位点被分别插入5'-端片段和3'-端片段,于下游或上游距ilvA基因的第966至969位核苷酸序列位置约600bp中。此外,用于取代ilvA基因的核苷酸序列的引物(SEQ ID NO:57和58)(表9)。制备pDZ-ilvA(V323A)质粒,其形式为位于5'和3'端每一端的DNA片段(各600bp)连接到pDZ载体(韩国专利号2009-0094433)。使用WT菌株的染色体作为模板和引物(SEQ ID NO:55和57),通过PCR制备ilvA基因的5'-端片段。PCR如下进行:在94℃下变性2分钟;在94℃下变性1分钟,在56℃下退火1分钟,和在72℃下聚合40秒,30个循环;以及在72℃下聚合10分钟。
同样地,使用引物(SEQ ID NO:56和58),通过PCR制备ilvA基因的3'-端片段。将扩增的DNA片段利用PCR纯化试剂盒(Quiagen)纯化,并作为插入DNA片段用于载体制备。同时,使用Infusion Cloning试剂盒,将用限制酶XbaI处理并在65℃下热处理20分钟的pDZ载体连接到通过PCR扩增的插入DNA片段,并且将连接的产物转化到大肠杆菌DH5α中并平皿接种在含有卡那霉素(25mg/L)的固体LB培养基上。选择被其中插入了使用引物(SEQ ID NO:55和56)通过PCR获得的目标基因的载体转化的菌落,并且通过公知的质粒提取方法获得质粒,从而制备用于在染色体上引入ilvA(V323A)核苷酸取代修饰的载体(即,pDZ-ilvA(V323A))。
[表9]
获得了菌株(即,CJ3P::hom(G378E)-ilvA(V323A)和CJ3P::gltA(N241T)-hom(G378E)-ilvA(V323A)),其中利用与实施例6中相同的方法通过pDZ-ilvA(G378E)载体在CJ3P::hom(G378E)和CJ3P::gltA(N241T)-hom(G378E)菌株的ilvA基因中引入了核苷酸修饰。利用与实施例5中相同的方法培养所获得的两种菌株,并分析培养液的异亮氨酸和GA浓度和糖消耗速率。结果显示在下表10中。
[表10]异亮氨酸浓度、糖消耗速率和培养液组成
苏氨酸浓度、糖消耗速率和培养液组成的分析结果是,确认在引入gltA(N241T)修饰的菌株中,异亮氨酸浓度以与糖消耗速率相似的水平增加,同时谷氨酸浓度降低。
由上可知,本公开所属领域技术人员将能够理解,在不改变本公开的技术思路或本质特征的情况下,本公开可以其他具体形式实施。在这方面,本文公开的示例性实施方式仅用于说明性目的而不应被解释为限制本公开的范围。相反,本公开意图不仅覆盖示例性实施方式,而且覆盖可包含在由所附权利要求所限定的本公开的精神和范围内的各种替代形式、修改、等同形式、和其他实施方式。
<110> CJ第一制糖株式会社
<120> 柠檬酸合酶活性减弱的修饰多肽及利用其产生L-氨基酸的方法
<130> OPA18433
<150> KR 10-2018-0017400
<151> 2018-02-13
<160> 62
<170> KoPatentIn 3.0
<210> 1
<211> 437
<212> PRT
<213> 谷氨酸棒杆菌
<220>
<221> 肽
<222> (1)..(437)
<223> gltA
<400> 1
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Asn Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Ser Asn His Gly Gly Asp Ala Thr Glu Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 2
<211> 1314
<212> DNA
<213> 谷氨酸棒杆菌
<220>
<221> 基因
<222> (1)..(1314)
<223> gltA
<400> 2
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggacca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aatgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
aactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagagcaac cacggtggcg acgcaaccga gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 3
<211> 437
<212> PRT
<213> 未知
<220>
<223> N241T
<400> 3
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Thr Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Ser Asn His Gly Gly Asp Ala Thr Glu Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 4
<211> 1314
<212> DNA
<213> 未知
<220>
<223> N241T
<400> 4
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggacca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aatgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
acctgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagagcaac cacggtggcg acgcaaccga gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 5
atgtttgaaa gggatatcgt g 21
<210> 6
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 6
ttagcgctcc tcgcgaggaa c 21
<210> 7
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 7
cggggatcct ctagacgatg aaaaacgccc 30
<210> 8
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 8
caggtcgact ctagactgca cgtggatcgt 30
<210> 9
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 9
actgggacta tttgttcgga aaaa 24
<210> 10
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 10
cgaacaaata gtcccagttc aacg 24
<210> 11
<211> 35
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 11
cggggatcct ctagaagatg ctgtctgaga ctgga 35
<210> 12
<211> 35
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 12
caggtcgact ctagacgcta aatttagcgc tcctc 35
<210> 13
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 13
ggaggtggag catgcctgct cgtggtcagc 30
<210> 14
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 14
gaccacgagc aggcatgctc cacctccacc 30
<210> 15
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 15
ggaggtggag cagacctgct cgtggtcagc 30
<210> 16
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 16
gaccacgagc aggtctgctc cacctccacc 30
<210> 17
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 17
ggaggtggag cactgctgct cgtggtcagc 30
<210> 18
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 18
gaccacgagc agcagtgctc cacctccacc 30
<210> 19
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 19
ggaggtggag cagtgctgct cgtggtcagc 30
<210> 20
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 20
gaccacgagc agcactgctc cacctccacc 30
<210> 21
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 21
ggaggtggag cagcgctgct cgtggtcagc 30
<210> 22
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 22
gaccacgagc agcgctgctc cacctccacc 30
<210> 23
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 23
ggaggtggag catggctgct cgtggtcagc 30
<210> 24
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 24
gaccacgagc agccatgctc cacctccacc 30
<210> 25
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 25
ggaggtggag cacagctgct cgtggtcagc 30
<210> 26
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 26
gaccacgagc agctgtgctc cacctccacc 30
<210> 27
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 27
ggaggtggag cagtactgct cgtggtcagc 30
<210> 28
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 28
gaccacgagc agtactgctc cacctccacc 30
<210> 29
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 29
ggaggtggag caggactgct cgtggtcagc 30
<210> 30
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 30
gaccacgagc agtcctgctc cacctccacc 30
<210> 31
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 31
ggaggtggag cacttctgct cgtggtcagc 30
<210> 32
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 32
gaccacgagc agaagtgctc cacctccacc 30
<210> 33
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 33
ggaggtggag cacatctgct cgtggtcagc 30
<210> 34
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 34
gaccacgagc agatgtgctc cacctccacc 30
<210> 35
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 35
ggaggtggag cagatctgct cgtggtcagc 30
<210> 36
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 36
gaccacgagc agatctgctc cacctccacc 30
<210> 37
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 37
ggaggtggag cattcctgct cgtggtcagc 30
<210> 38
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 38
gaccacgagc aggaatgctc cacctccacc 30
<210> 39
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 39
ggaggtggag cagtcctgct cgtggtcagc 30
<210> 40
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 40
gaccacgagc aggactgctc cacctccacc 30
<210> 41
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 41
ggaggtggag cagccctgct cgtggtcagc 30
<210> 42
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 42
gaccacgagc agggctgctc cacctccacc 30
<210> 43
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 43
ggaggtggag cagcactgct cgtggtcagc 30
<210> 44
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 44
gaccacgagc agtggtgctc cacctccacc 30
<210> 45
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 45
ggaggtggag cagcactgct cgtggtcagc 30
<210> 46
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 46
gaccacgagc agtgctgctc cacctccacc 30
<210> 47
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 47
ggaggtggag cagaactgct cgtggtcagc 30
<210> 48
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 48
gaccacgagc agttctgctc cacctccacc 30
<210> 49
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 49
ggaggtggag caggtctgct cgtggtcagc 30
<210> 50
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 50
gaccacgagc agacctgctc cacctccacc 30
<210> 51
<211> 29
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 51
tcctctagac tggtcgcctg atgttctac 29
<210> 52
<211> 29
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 52
gactctagat tagtcccttt cgaggcgga 29
<210> 53
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 53
gccaaaacct ccacgcgatc 20
<210> 54
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 54
atcgcgtgga ggttttggct 20
<210> 55
<211> 28
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 55
acggatccca gactccaaag caaaagcg 28
<210> 56
<211> 28
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 56
acggatccaa ccaaacttgc tcacactc 28
<210> 57
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 57
acaccacggc agaaccaggt gcaaaggaca 30
<210> 58
<211> 30
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 58
ctggttctgc cgtggtgtgc atcatctctg 30
<210> 59
<211> 437
<212> PRT
<213> 未知
<220>
<223> N241S
<400> 59
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Ser Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Ser Asn His Gly Gly Asp Ala Thr Glu Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 60
<211> 1314
<212> DNA
<213> 未知
<220>
<223> N241S
<400> 60
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggacca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aatgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
tcctgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagagcaac cacggtggcg acgcaaccga gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
<210> 61
<211> 437
<212> PRT
<213> 未知
<220>
<223> N241Y
<400> 61
Met Phe Glu Arg Asp Ile Val Ala Thr Asp Asn Asn Lys Ala Val Leu
1 5 10 15
His Tyr Pro Gly Gly Glu Phe Glu Met Asp Ile Ile Glu Ala Ser Glu
20 25 30
Gly Asn Asn Gly Val Val Leu Gly Lys Met Leu Ser Glu Thr Gly Leu
35 40 45
Ile Thr Phe Asp Pro Gly Tyr Val Ser Thr Gly Ser Thr Glu Ser Lys
50 55 60
Ile Thr Tyr Ile Asp Gly Asp Ala Gly Ile Leu Arg Tyr Arg Gly Tyr
65 70 75 80
Asp Ile Ala Asp Leu Ala Glu Asn Ala Thr Phe Asn Glu Val Ser Tyr
85 90 95
Leu Leu Ile Asn Gly Glu Leu Pro Thr Pro Asp Glu Leu His Lys Phe
100 105 110
Asn Asp Glu Ile Arg His His Thr Leu Leu Asp Glu Asp Phe Lys Ser
115 120 125
Gln Phe Asn Val Phe Pro Arg Asp Ala His Pro Met Ala Thr Leu Ala
130 135 140
Ser Ser Val Asn Ile Leu Ser Thr Tyr Tyr Gln Asp Gln Leu Asn Pro
145 150 155 160
Leu Asp Glu Ala Gln Leu Asp Lys Ala Thr Val Arg Leu Met Ala Lys
165 170 175
Val Pro Met Leu Ala Ala Tyr Ala His Arg Ala Arg Lys Gly Ala Pro
180 185 190
Tyr Met Tyr Pro Asp Asn Ser Leu Asn Ala Arg Glu Asn Phe Leu Arg
195 200 205
Met Met Phe Gly Tyr Pro Thr Glu Pro Tyr Glu Ile Asp Pro Ile Met
210 215 220
Val Lys Ala Leu Asp Lys Leu Leu Ile Leu His Ala Asp His Glu Gln
225 230 235 240
Tyr Cys Ser Thr Ser Thr Val Arg Met Ile Gly Ser Ala Gln Ala Asn
245 250 255
Met Phe Val Ser Ile Ala Gly Gly Ile Asn Ala Leu Ser Gly Pro Leu
260 265 270
His Gly Gly Ala Asn Gln Ala Val Leu Glu Met Leu Glu Asp Ile Lys
275 280 285
Ser Asn His Gly Gly Asp Ala Thr Glu Phe Met Asn Lys Val Lys Asn
290 295 300
Lys Glu Asp Gly Val Arg Leu Met Gly Phe Gly His Arg Val Tyr Lys
305 310 315 320
Asn Tyr Asp Pro Arg Ala Ala Ile Val Lys Glu Thr Ala His Glu Ile
325 330 335
Leu Glu His Leu Gly Gly Asp Asp Leu Leu Asp Leu Ala Ile Lys Leu
340 345 350
Glu Glu Ile Ala Leu Ala Asp Asp Tyr Phe Ile Ser Arg Lys Leu Tyr
355 360 365
Pro Asn Val Asp Phe Tyr Thr Gly Leu Ile Tyr Arg Ala Met Gly Phe
370 375 380
Pro Thr Asp Phe Phe Thr Val Leu Phe Ala Ile Gly Arg Leu Pro Gly
385 390 395 400
Trp Ile Ala His Tyr Arg Glu Gln Leu Gly Ala Ala Gly Asn Lys Ile
405 410 415
Asn Arg Pro Arg Gln Val Tyr Thr Gly Asn Glu Ser Arg Lys Leu Val
420 425 430
Pro Arg Glu Glu Arg
435
<210> 62
<211> 1314
<212> DNA
<213> 未知
<220>
<223> N241Y
<400> 62
atgtttgaaa gggatatcgt ggctactgat aacaacaagg ctgtcctgca ctaccccggt 60
ggcgagttcg aaatggacat catcgaggct tctgagggta acaacggtgt tgtcctgggc 120
aagatgctgt ctgagactgg actgatcact tttgacccag gttatgtgag cactggctcc 180
accgagtcga agatcaccta catcgatggc gatgcgggaa tcctgcgtta ccgcggctat 240
gacatcgctg atctggctga gaatgccacc ttcaacgagg tttcttacct acttatcaac 300
ggtgagctac caaccccaga tgagcttcac aagtttaacg acgagattcg ccaccacacc 360
cttctggacg aggacttcaa gtcccagttc aacgtgttcc cacgcgacgc tcacccaatg 420
gcaaccttgg cttcctcggt taacattttg tctacctact accaggacca gctgaaccca 480
ctcgatgagg cacagcttga taaggcaacc gttcgcctca tggcaaaggt tccaatgctg 540
gctgcgtacg cacaccgcgc acgcaagggt gctccttaca tgtacccaga caactccctc 600
aatgcgcgtg agaacttcct gcgcatgatg ttcggttacc caaccgagcc atacgagatc 660
gacccaatca tggtcaaggc tctggacaag ctgctcatcc tgcacgctga ccacgagcag 720
tactgctcca cctccaccgt tcgtatgatc ggttccgcac aggccaacat gtttgtctcc 780
atcgctggtg gcatcaacgc tctgtccggc ccactgcacg gtggcgcaaa ccaggctgtt 840
ctggagatgc tcgaagacat caagagcaac cacggtggcg acgcaaccga gttcatgaac 900
aaggtcaaga acaaggaaga cggcgtccgc ctcatgggct tcggacaccg cgtttacaag 960
aactacgatc cacgtgcagc aatcgtcaag gagaccgcac acgagatcct cgagcacctc 1020
ggtggcgacg atcttctgga tctggcaatc aagctggaag aaattgcact ggctgatgat 1080
tacttcatct cccgcaagct ctacccgaac gtagacttct acaccggcct gatctaccgc 1140
gcaatgggct tcccaactga cttcttcacc gtattgttcg caatcggtcg tctgccagga 1200
tggatcgctc actaccgcga gcagctcggt gcagcaggca acaagatcaa ccgcccacgc 1260
caggtctaca ccggcaacga atcccgcaag ttggttcctc gcgaggagcg ctaa 1314
