一种基于结构特征的DNA绑定残基预测方法
文献发布时间:2023-06-19 11:05:16
技术领域
本发明涉及生物信息学、模式识别与计算机应用领域,具体而言涉及一种基于结构特征的DNA绑定残基预测方法。
背景技术
蛋白质与配体相互作用在生命过程中是普遍存在且不可或缺的,这种相互作用在生物分子的识别和信号传递过程中起着非常重要的作用。DNA分子是重要的一类配体分子,准确识别蛋白质序列中DNA分子的绑定残基,有助于理解蛋白质功能、分析蛋白质与DNA分子之间的相互作用机制及设计药物靶蛋白,具有重要的生物学意义。
调研文献发现,许多用于预测蛋白质序列中DNA绑定残基的方法已被提出,如:DNAPred(Zhu Y,Hu J,Song X,et al.DNAPred:Accurate Identification of DNA-Binding Sites from Protein Sequence by Ensembled Hyperplane-Distance-BasedSupport Vector Machines[J].Journal of Chemical Information and Modeling,2019,59(6):3057-3071.即:Zhu Y等.集成基于超平面距离的支持向量机来准确识别蛋白质序列中的DNA绑定位点[J].化学信息和建模期刊,2019,59(6):3057-3071)、EL_PSSM-RT(ZhouJ,Lu Q,Xu R,et al.EL_PSSM-RT:DNA-binding residue prediction by integratingensemble learning with PSSM Relation Transformation[J].BMC Bioinformatics,2017,18(1):1-16.即:Zhou J等.通过结合集成学习与PSSM关系转化预测DNA绑定残基[J].生物信息学,2017,18(1):1-16)、CNNsite(Zhou J,Lu Q,Xu R,et al.CNNsite:predictionof DNA-binding residues in proteins using convolutional neural network withsequence features[J],2016:78-85.即:Zhou J等.基于序列特征的卷积神经网络预测蛋白质DNA绑定残基[J],2016:78-85)、DP-Bind(Hwang S,Gou Z,Kuznetsov A I B.DP-Bind:a web server for sequence-based prediction of DNA-binding residues in DNA-binding proteins.[J].Bioinformatics,2007,23(5):634-636.即:Hwang S等.一个基于序列的蛋白质DNA绑定残基预测服务器[J].生物信息学,2017,23(5):634-636)等。尽管已有方法可以用于预测蛋白质序列中的DNA绑定残基,但是蛋白质的三维结构信息没有得到足够的关注,预测精度并不能保证是最优的,有待进一步提升。
综上所述,已有的DNA绑定残基的预测方法在计算代价、预测精度两个方面距离实际应用的要求还有很大差距,迫切地需要改进。
发明内容
为了克服已有的DNA绑定残基预测方法在计算代价、预测精度两个方面的不足,本发明提出一种计算代价小、预测精度高的基于结构特征的DNA绑定残基预测方法。
本发明解决其技术问题所采用的技术方案是:
一种基于结构特征的DNA绑定残基预测方法,所述方法包括以下步骤:
1)输入一个残基数为L的待进行DNA绑定残基预测的蛋白质三维结构信息S;
2)对蛋白质三维结构信息S中的任一残基P
2.1)分别统计该球内20种天然氨基酸类型的残基出现的频率,组成一个含有20个元素的向量,记作
2.2)分别统计该球内三类二级结构类别(折叠、螺旋、卷曲)的残基出现的频率,组成一个含有3个元素的向量,记作
2.3)分别统计该球内三类溶剂可及性类别(埋藏、中间状态、暴露)的残基出现的频率,组成一个含有3个元素的向量,记作
2.4)将步骤2.1)至2.3)中的
3)从蛋白质结构数据库PDB(https://www.rcsb.org/)中获取已知DNA绑定残基状态的蛋白质三维结构信息作为训练蛋白质集合,使用步骤2)生成每个残基的特征向量;结合每个残基绑定的DNA状态信息,构建训练样本集;
4)搭建一维卷积神经网络预测蛋白质三维结构信息S的DNA绑定残基,该网络共有三个模块,第一个模块和第二个模块为卷积层模块,第三个模块为全连接层模块,每一个卷积层模块分别包含一个卷积核大小为3的一维卷积层,一个池化层和一个ReLU层,每一个卷积层输入输出通道分别为1和16,16和32,,全连接层模块包含三个全连接层,三个全连接层的输出神经数量分别是512、256、2,每一模块的输出作为下一模块的输入,使用sigmoid激活函数使网络的输出值在(0,1)范围内,该网络的输出记为:
g(I)=sigmoid(mod3(mod2(mod1(I))))
其中,I表示网络的输入,mod1,mod2,mod3分别表示第一个模块、第二个模块、第三个模块的运算;
5)使用步骤3)中构建的训练样本集训练4)中搭建的一维卷积神经网络,训练阶段都采用二分类交叉熵损失函数调整网络中的参数,二分类交叉熵损失函数记作:
其中,u表示蛋白质三维结构信息中残基的真实标签,
6)将蛋白质三维结构信息S中每个残基的特征向量输入到步骤5)训练的模型中,根据模型的输出概率是否大于判定阈值threshold,来判断对应残基是否为DNA绑定残基。
本发明的技术构思为:首先,根据输入的待进行DNA绑定残基预测的蛋白质结构信息,以结构信息中任一残基为球心,R为半径,通过以下四步获取该残基的特征向量:1)分别统计该球内20种天然氨基酸类型的残基出现的频率,记作
本发明的有益效果表现在:一方面,从蛋白质结构信息中残基的特征向量出发,生成残基样本,为模型的训练做好了准备;另一方面,搭建一维卷积网络预测模型,进一步提高了DNA绑定残基的预测效率与精确性。
附图说明
图1为一种基于结构特征的DNA绑定残基预测方法的示意图。
图2为使用一种基于结构特征的预测方法对蛋白质2M2W三维结构信息进行DNA绑定残基预测的结果。
具体实施方式
下面结合附图对本发明作进一步描述。
参照图1和图2,一种基于结构特征的DNA绑定残基预测方法,包括以下步骤:
1)输入一个残基数为L的待进行DNA绑定残基预测的蛋白质三维结构信息S;
2)对蛋白质三维结构信息S中的任一残基P
2.1)分别统计该球内20种天然氨基酸类型的残基出现的频率,组成一个含有20个元素的向量,记作
2.2)分别统计该球内三类二级结构类别(折叠、螺旋、卷曲)的残基出现的频率,组成一个含有3个元素的向量,记作
2.3)分别统计该球内三类溶剂可及性类别(埋藏、中间状态、暴露)的残基出现的频率,组成一个含有3个元素的向量,记作
2.4)将步骤2.1)至2.3)中的
3)从蛋白质结构数据库PDB(https://www.rcsb.org/)中获取已知DNA绑定残基状态的蛋白质三维结构信息作为训练蛋白质集合,使用步骤2)生成每个残基的特征向量;结合每个残基绑定的DNA状态信息,构建训练样本集;
4)搭建一维卷积神经网络预测蛋白质三维结构信息S的DNA绑定残基,该网络共有三个模块,第一个模块和第二个模块为卷积层模块,第三个模块为全连接层模块,每一个卷积层模块分别包含一个卷积核大小为3的一维卷积层,一个池化层和一个ReLU层,每一个卷积层输入输出通道分别为1和16,16和32,全连接层模块包含三个全连接层,三个全连接层的输出神经数量分别是512、256、2,每一模块的输出作为下一模块的输入,使用sigmoid激活函数使网络的输出值在(0,1)范围内,该网络的输出记为:
g(I)=sigmoid(mod3(mod2(mod1(I))))
其中,I表示网络的输入,mod1,mod2,mod3分别表示第一个模块、第二个模块、第三个模块的运算;
5)使用步骤3)中构建的训练样本集训练4)中搭建的一维卷积神经网络,训练阶段都采用二分类交叉熵损失函数调整网络中的参数,二分类交叉熵损失函数记作:
其中,u表示蛋白质三维结构信息中残基的真实标签,
6)将蛋白质三维结构信息S中每个残基的特征向量输入到步骤5)训练的模型中,根据模型的输出概率是否大于判定阈值threshold,来判断对应残基是否为DNA绑定残基。
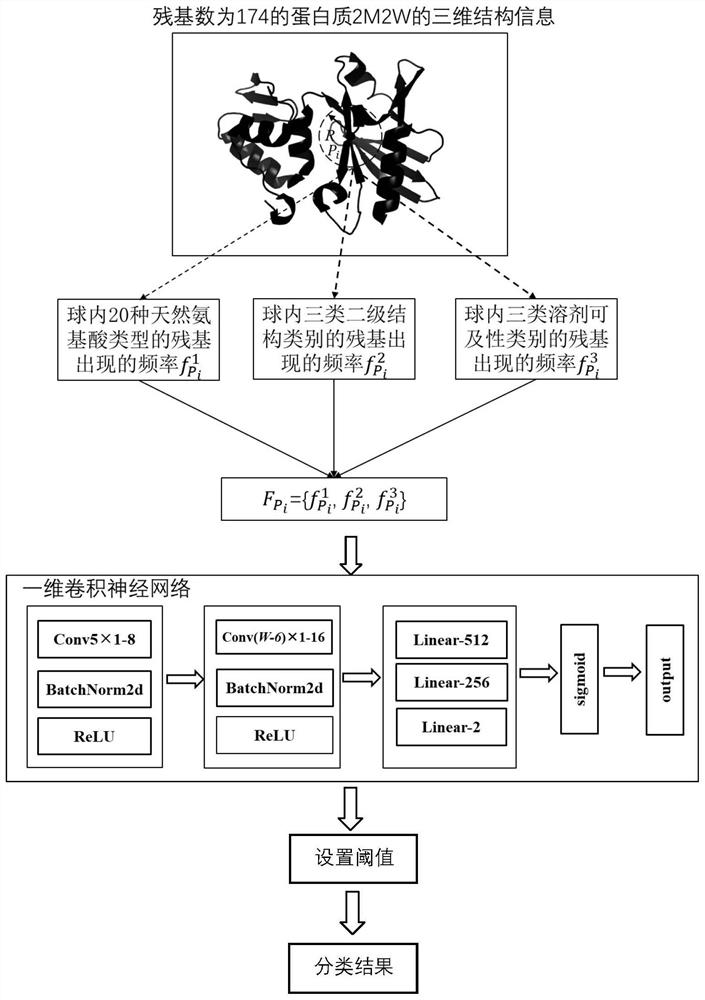
本实施例以蛋白质2M2W三维结构信息的DNA绑定残基预测为实施例,一种基于结构特征的DNA绑定残基预测方法,包括以下步骤:
1)输入一个残基数为174的待进行DNA绑定残基预测的蛋白质2M2W的三维结构信息,记作S;
2)对蛋白质三维结构信息S的任一残基P
2.1)分分别统计该球内20种天然氨基酸类型的残基出现的频率,组成一个含有20个元素的向量,记作
2.2)分别统计该球内三类二级结构类别(折叠、螺旋、卷曲)的残基出现的频率,组成一个含有3个元素的向量,记作
2.3)分别统计该球内三类溶剂可及性类别(埋藏、中间状态、暴露)的残基出现的频率,组成一个含有3个元素的向量,记作
2.4)将步骤2.1)至2.3)中的
3)从蛋白质结构数据库PDB(https://www.rcsb.org/)中获取已知DNA绑定残基状态的蛋白质三维结构信息作为训练蛋白质集合,使用步骤2)生成每个残基的特征向量;结合每个残基绑定的DNA状态信息,构建训练样本集;
4)搭建一维卷积神经网络预测蛋白质三维结构信息S的DNA绑定残基,该网络共有三个模块,第一个模块和第二个模块为卷积层模块,第三个模块为全连接层模块,每一个卷积层模块分别包含一个卷积核大小为3的一维卷积层,一个池化层和一个ReLU层,每一个卷积层输入输出通道分别为1和16,16和32,全连接层模块包含三个全连接层,三个全连接层的输出神经数量分别是512、256、2,每一模块的输出作为下一模块的输入,使用sigmoid激活函数使网络的输出值在(0,1)范围内,该网络的输出记为:
g(I)=sigmoid(mod3(mod2(mod1(I))))
其中,I表示网络的输入,mod1,mod2,mod3分别表示第一个模块、第二个模块、第三个模块的运算;
5)使用步骤3)中构建的训练样本集训练4)中搭建的一维卷积神经网络,训练阶段都采用二分类交叉熵损失函数调整网络中的参数,二分类交叉熵损失函数记作:
其中,u表示蛋白质三维结构信息中残基的真实标签,
6)将蛋白质2M2W的三维结构信息S中每个残基的特征向量输入到步骤5)训练的模型中,根据模型的输出概率是否大于判定阈值0.5,来判断对应残基是否为DNA绑定残基。
以蛋白质2M2W三维结构信息的DNA绑定残基预测为实施例,运用以上方法划分得到的预测结果如图2所示。
以上说明是本发明以蛋白质2M2W三维结构信息的DNA绑定残基为实例所得出的预测结果,并非限定本发明的实施范围,在不偏离本发明基本内容所涉及范围的前提下对其做各种变形和改进,不应排除在本发明的保护范围之外。
- 一种基于结构特征的DNA绑定残基预测方法
- 一种基于深度卷积神经网络的DNA绑定残基预测方法
