CAPS分子标记在鉴定谷子株高性状的用途及引物和检测试剂盒
文献发布时间:2023-06-19 11:59:12
技术领域
本发明属于植物基因工程与分子育种技术领域,具体涉及一种CAPS分子标记在鉴定谷子株高性状的用途及引物和检测试剂盒。
背景技术
20世纪60年代以来小麦和水稻等农作物先后实现了矮化育种,作物产量有了大幅度的提升,为解决全球饥饿问题做出了巨大贡献。小麦矮化育种的实现主要依靠农林10号的Rht-B1b和Rht-D1b两个矮秆基因,这两个基因为拟南芥GAI的同源基因,编码赤霉素信号转导的DELLA蛋白(Peng J,Richards DE,Hartley NM,Murphy GP,Devos KM,Flintham JE,Beales J,Fish LJ,Worland AJ,Pelica F,Sudhakar D,Christou P,Snape JW,Gale MD,Harberd NP.'Green revolution'genes encode mutant gibberellin responsemodulators.Nature,1999,400(6741):256-261)。类似地,水稻矮化育种是通过赤霉素合成关键基因Semi-dwarf 1(SD1)来实现的,该基因编码赤霉素生物合成的关键酶GA20ox2,功能缺失后导致水稻株高显著降低,绝大部分矮秆籼稻品种中都含有SD1基因(Sasaki A,Ashikari M,Ueguchi-Tanaka M,Itoh H,Nishimura A,Swapan D,Ishiyama K,Saito T,Kobayashi M,Khush GS,Kitano H,Matsuoka M.Green revolution:a mutantgibberellin-synthesis gene in rice.Nature,2002,416(6882):701-702)。目前,SD1的突变等位基因仍然是培育水稻半矮秆性状的主要基因(Liu C,Zheng S,Gui J,Fu C,Yu H,Song D,Shen J,Qin P,Liu X,Han B,Yang Y,Li L.Shortened Basal Internodesencodes a gibberellin 2-oxidase and contributes to lodging resistance inrice.Molecular Plant,2018,11(2):288-299)。目前,已经定位的水稻矮秆基因至少有18个,这些基因主要与赤霉素、油菜素内酯和独脚金内酯等激素的生物合成和信号转导相关(http://www.ricedata.cn/gene/gene_sd.htm)。
谷子(Setaria italica)起源于中国,是我国几千年来的主栽作物和中华民族的哺育作物。近年来,随着人们生活的不断提高及食品的多样化和营养化,小米倍受青睐。生产上推广的谷子品种株高较高,株高较高的谷子产量较低,严重限制谷子产业的发展,因此有必要对谷子进行矮化育种。早在1998年,陈金桂等就研究了3种矮秆谷子突变体对赤霉素的反应,为谷子矮秆基因的克隆奠定了基础(陈金桂,张玉宗,周燮.赤霉素反应敏感型和不敏感型谷子矮秆突变体的鉴定.华北农学报,1998,12(1):46-52)。2013年,中国农业科学院作物科学研究所的赵美丞博士从矮秆突变体84133中克隆到一个半显性矮秆基因SiDw1,该基因属于GRAS家族的DELLA蛋白基因,与拟南芥的GAI/RGA、水稻的SLRl、玉米的D8、小麦的Rht具有很高的同源性。尽管如此,谷子的矮化基因的克隆及矮化分子育种仍远远落后于水稻和小麦,克隆谷子矮秆基因并将其应用于谷子矮化育种对于谷子产业的健康发展具有极其重要的意义。
酶切扩增多态性序列(Cleaved Amplified Polymorphic Sequences,CAPS)是一种简单、经济、可靠的共显性分子标记,其原理是先利用PCR技术扩增该位点上的某一DNA片段;接着用专一性的限制性内切酶切割得到扩增带并进行RFLP分析(Konieczny A,AusubelFM.A procedure for mapping Arabidopsis mutations using co-dominant ecotype-specific PCR-based markers.Plant Journal,1993,4(2):403-410)。与RFLP技术一样,CAPS技术检测的多态性其实是酶切片段大小的差异。作为特异引物PCR和限制性内切酶酶切的一种结合技术,CAPS标记有以下优点:第一,引物与限制性内切酶的组合非常多,这就使多态性较高;第二,CAPS标记是一种共显性标记,即可以区分纯合基因型和杂合基因型;第三,所需DNA的量较少;第四,由于是特异引物扩增,使用常规PCR的退火温度,所以结果稳定可靠,重复性高,便于不同实验室间的数据交流;第五,CAPS无需同位素标记,可以用琼脂糖凝胶电泳分析,操作简便,自动化程度高,成本较低,便于在发展中国家推广使用。由于以上优点,CAPS标记在理论研究与分子标记辅助选择育种中得到了广泛应用。
因此,开发谷子株型改良相关的基因标记非常有必要,然而目前并未见谷子株高鉴定相关的分子标记技术及相应的引物或检测试剂盒。
发明内容
为了解决上述技术问题,本发明提供了一种CAPS分子标记在鉴定谷子株高性状的用途及引物和检测试剂盒。
本发明的第一个目的是提供一种CAPS分子标记在鉴定谷子株高性状中的用途,所述CAPS分子标记包括SiSD1NciI-1或SiSD1NciI-2,SiSD1NciI-1的核苷酸序列为SEQ IDNo_7,SiSD1NciI-2的核苷酸序列为SEQ ID No_8。
优选的,上述CAPS分子标记在鉴定谷子株高性状中的用途,所述SiSD1NciI-1用于鉴定、选育高秆性状植物,所述SiSD1NciI-2用于鉴定、选育矮秆性状植物。
优选的,上述CAPS分子标记在鉴定谷子株高性状中的用途,所述植物包括但不限于拟南芥、水稻和谷子。
优选的,上述CAPS分子标记在鉴定谷子株高性状中的用途,利用基因编辑的方法对SiSD1基因中的此位点进行编辑,选育矮秆谷子种质;
其中,所述CAPS分子标记对应的PCR扩增片段为SEQ ID No_7和SEQ ID No_8所示的序列,或者是与SEQ ID No_7和SEQ ID No_8具有90%以上同源性的序列。
优选的,上述CAPS分子标记在鉴定谷子株高性状中的用途,根据所述CAPS分子标记设计PCR扩增引物,据此开发用于鉴定谷子株高的特异性引物,并用于谷子株型改良。
本发明的第二个目的是提供一种上述CAPS分子标记的引物,包括SiSD1NciIF和SiSD1NciIB,所述SiSD1NciIF核苷酸序列参见SEQ ID No_5,所述SiSD1NciIB核苷酸序列参见SEQ ID No_6。
本发明的第三个目的是提供一种上述CAPS分子标记的引物在植物种质资源改良中的应用。
本发明的第四个目的是提供一种所述CAPS分子标记的检测试剂盒,包括引物SiSD1NciIF和SiSD1NciIB、限制性内切核酸酶NciI。
本发明的第五个目的是提供一种所述的试剂盒在植物种质资源改良中的应用。
优选的,上述应用中,植物种质资源改良的技术手段包括但不限于分子标记辅助育种。
与现有技术相比,本发明具有以下有益效果:
本发明用基于高通量测序的Super-BSA方法定位了谷子半矮秆基因SiSD1,并获得了其等位突变基因sisd1。上述基因可以控制谷子株高性状,用于选育高秆或者矮杆性状植物,基因SiSD1及其等位基因sisd1和CAPS分子标记在谷子矮化育种及遗传改良方面具有重要的实际意义,在农业领域具有广阔的应用和市场前景。
附图说明
图1为本发明实施例1的矮秆谷子品种S2和高秆谷子品种酒谷表型及其株高统计数据;
A:大田种植的S2(左)和酒谷(右);B:S2和酒谷株高统计数据,每个品种统计40株,取平均值;
图2为本发明实施例2的F
图3为本发明实施例4的高秆极端混池和矮秆极端混池以及亲本间的SNP统计Venn图;
图4为本发明实施例5的高秆极端混池和矮秆极端混池以及亲本间的InDel统计Venn图;
图5为本发明实施例6SNP的ED关联值在染色体上的分布;
图6为本发明实施例7InDel的ED关联值在染色体上的分布;
图7为本发明实施例8的矮秆谷子品种S2的SiSD1基因突变情况;
A:SiSD1基因重测序Reads与参考基因组的比对情况,中间碱基G为S2缺失的碱基;B:S2中SiSD1等位基因sisd1基因结构;
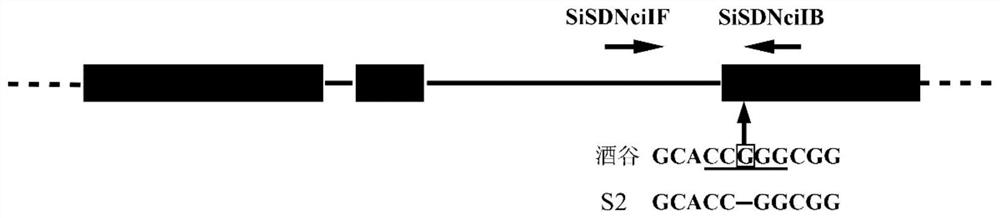
图8为本发明实施例9的CAPS分子标记SiSD1NciI扩增位点示意图。
具体实施方式
为了使本领域技术人员更好地理解本发明的技术方案能予以实施,下面结合具体实施例和附图对本发明作进一步说明。
下述实施例中所用方法如无特别说明均为常规方法,所用引物由生工生物工程(上海)股份有限公司合成;高效植物基因组DNA提取试剂盒购买自天根生化科技(北京)有限公司,货号DP350。
实施例1谷子矮秆种质S2(现存地址:山西省太谷区山西农业大学,获取时间:2015年)和高秆种质酒谷株高分析
将矮秆谷子种质S2和高秆种质酒谷正季种植于山西农业大学杂粮团队试验田内(山西省太谷区),正常水肥管理。如图1A所示,矮秆谷子S2株高较矮,而酒谷株高显著高于S2。测量了S2和酒谷株高,每个品种测定40株。S2平均株高为98.0cm,株高最低为58.2cm,最高为119.8cm;酒谷平均株高为176.80cm,株高最低为147.8cm,最高为200.1cm(图1B)。S2和酒谷平均株高相差78.8cm。
实施例2谷子矮秆基因定位群体S2×酒谷的构建及株高分析
将矮秆谷子S2作为母本,酒谷作为父本,进行杂交获得F
实施例3两个极端混池和亲本的高通量测序及分析
从S2×酒谷的F
高通量测序得到的原始图像数据文件,经碱基识别(Base Calling)分析转化为原始测序序列(Sequenced Reads),即Raw Reads。Raw Reads经过过滤去杂,高秆极端混池、矮秆极端混池、S2和酒谷分别获得16.43G、17.00G、18.32G和18.60G的高质量碱基,测序深度分别为33×、33×、39×和40×,结果参见表1。
表1极端混池和亲本高通量测序数据统计
实施例4极端混池和亲本的SNP检测
首先,使用BWA软件(Li H,Durbin R.Fast and accurate short read alignmentwith Burrows Wheeler Transform.Bioinformatics,2009,25:1754-1760)将矮秆极端混池、高秆极端混池及亲本S2和酒谷的重测序数据与豫谷1号参考基因组(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Sitalica)进行比对,从而将重测序获得的测序reads重新定位到参考基因组上;然后,根据Clean Reads在参考基因组的定位结果,使用Picard(https://sourceforge.net/projects/picard/)进行去重复,屏蔽PCR duplication的影响;最后,利用GATK软件(McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A,Garimella K,Altshuler D,Gabriel S,DalyM,DePristo MA.The Genome Analysis Toolkit:a MapReduce framework for analyzingnext generation DNA sequencing data.Genome Research,2010,20:1297-1303)进行单核苷酸多态性(Single Nucleotide Polymorphism,SNP)检测,共计获得1595214个SNP位点。进一步对上述SNP进行过滤,过滤掉2390个有多个基因型的SNP位点,33196个read支持度小于4的SNP位点,385618个混池之间基因型一致的SNP位点以及283445个隐性混池基因不是来自于隐性亲本的SNP位点。最终得到高质量的可信SNP位点890565个,这些SNP位点将用于下一步谷子S2×酒谷F
实施例5极端混池和亲本的InDel检测
根据样品的Clean Reads在参考基因组上的定位结果,检测样品与参考基因组之间是否存在InDel。样品的InDel检测使用GATK软件进行。样品间共检测到280610个InDel位点,在使用InDel进行关联分析前,首先对InDel进行过滤,过滤标准与SNP分析相同,最终得到高质量的可信InDel位点157598个(图4)。InDel变异一般比SNP变异少,但同样反映了样品与参考基因组之间的差异,并且编码区的InDel会引起移码突变,导致基因功能上的变化。
实施例6S2×酒谷F
采用欧式距离(Euclidean Distance,ED)算法对S2×酒谷杂交群体株高基因进行关联进行。ED算法是利用测序数据寻找混池间存在显著差异标记,并以此评估与性状关联区域的方法(Hill JT,Demarest BL,Bisgrove BW,Gorsi B,Su YC,Yost HJ.MMAPPR:mutation mapping analysis pipeline for pooled RNA-seq.Genome Research,2013,23(4):687-97)。理论上,构建的高秆极端混池和矮秆极端混池间除了目标性状相关位点存在差异,其他位点均趋向于一致,因此非目标位点的ED值应趋向于0。ED值越大表明该标记在两混池间的差异越大。为消除背景噪音,对原始ED值进行乘方处理,取原始ED的5次方作为关联值以达到消除背景噪音的功能,然后采用DISTANCE方法对ED值进行拟合,关联值分布如图5所示。图5的横坐标为染色体名称,灰色的点代表每个SNP位点的ED值,非坐标轴的实线为拟合后的ED值,虚线代表显著性关联阈值,ED值越高,代表该点关联效果越好。
取所有位点拟合值的median+3SD作为分析的关联阈值,计算得0.36。根据关联阈值判定,将株高相关基因定位在Scaffold 5 39110000-44720000bp的一个区间。该区间总长度为5.61Mb,包括1022个基因。
实施例7 S2×酒谷F
InDel关联分析使用与SNP关联分析相同的分析方法(ED方法),关联值分布如图6所示。图6的横坐标为染色体名称,灰色的点代表每个InDel位点的ED值,非坐标轴的实线为拟合后的ED值,虚线代表显著性关联阈值,ED值越高,代表该点关联效果越好。
取所有位点拟合值的median+3SD作为分析的关联阈值,计算得0.42。将株高相关基因定位在Scaffold 5 37980000-44630000bp的一个区间。该区间总长度为6.65Mb,包括1194个基因。
实施例8S2×酒谷F2群体株高相关基因分析
对实施例6和实施例7中SNP和InDel对应的关联区域取交集,最终将S2×酒谷F2群体株高相关基因定位在第5染色体40360000-44630000bp之间4.27Mb的区域,该区域共有767个基因。深入分析发现,上述767个基因中共有40个基因在亲本间存在非同义突变,其中发生SNP变异的为37个,InDel变异的为6个。矮秆品种S2基因组Scaffold 5 43166915bp的碱基G发生了缺失,此处共有32条测序Reads覆盖,具有非常高的可信度(图7A)。该碱基的缺失导致谷子Seita.5G404900基因发生移码突变和提前终止,缺失了83个氨基酸(图7B)。Seita.5G404900基因编码赤霉素合成的关键酶GA20ox2,功能缺失将导致赤霉素生物合成受阻,从而使谷子株高降低。序列分析表明,Seita.5G404900基因与水稻半矮秆基因OsSD1(LOC_Os01g66100)为同源基因。因此,将其命名为SiSD1,将S2中的突变基因(等位基因)命名为sisd1。
SiSD1基因的序列全长为2900bp,包括155个碱基的5′UTR区,开放阅读框架为5′端第156到第2525位碱基,3′UTR区为第2526到第2900位碱基。自5′端第156-808位碱基为该基因的第一个外显子,自5′端第809-903位碱基为第一个内含子,自5′端第904-1225位碱基为第二个外显子,自5′端第1226-2228位碱基为第二个内含子,自5′端第2229-2525位碱基为第三个外显子,自5′端第2526-2900位碱基为3′非编码区。SiSD1基因核苷酸序列如SEQ IDNo_1所示,SiSD1基因编码的蛋白质序列如SEQ ID No_2所示。
SiSD1基因的等位基因sisd1缺失了第三外显子第2266位的G碱基,导致移码突变和提前终止(对应SEQ ID No_1所示核苷酸序列的线框部分),sisd1基因核苷酸序列如SEQID No_3所示,sisd1基因编码的蛋白质序列如SEQ ID No_4所示。
实施例9SiSD1和sisd1基因CAPS标记开发及应用
本发明利用CAPS技术来快速鉴定谷子半矮秆基因SiSD1的等位基因,尤其是sisd1基因。等位基因sisd1中碱基G的缺失导致该位置NciI酶切位点的缺失,从而可以利用NciI酶切来检测这一突变。首先,设计PCR引物SiSD1NciIF(SEQ ID No_5)和SiSD1NciIB(SEQ IDNo_6),两条引物分别位于第二内含子和第三外显子上(图8)。
分别以S2、酒谷和S2×酒谷F
理论上,酒谷PCR扩增片段长度为482bp(SEQ ID No_7),该分子标记记作SiSD1NciI-1,用于鉴定高秆谷子品种;而S2中由于单碱基的缺失,PCR扩增片段长度为481bp(SEQ ID No_8),该分子标记记作SiSD1NciI-2,用于鉴定矮秆谷子品种;而S2×酒谷F
需要说明的是,本发明中涉及数值范围时,应理解为每个数值范围的两个端点以及两个端点之间任何一个数值均可选用,由于采用的步骤方法与实施例相同,为了防止赘述,本发明描述了优选的实施例。尽管已描述了本发明的优选实施例,但本领域内的技术人员一旦得知了基本创造性概念,则可对这些实施例做出另外的变更和修改。所以,所附权利要求意欲解释为包括优选实施例以及落入本发明范围的所有变更和修改。
显然,本领域的技术人员可以对本发明进行各种改动和变型而不脱离本发明的精神和范围。这样,倘若本发明的这些修改和变型属于本发明权利要求及其等同技术的范围之内,则本发明也意图包含这些改动和变型在内。
序列表
SEQ ID No_1,SiSD1基因,其第2266位碱基(方框标注)为其等位基因sisd1缺失的碱基:
ATGGCCTCCCCTGTGGGGCGGGTCCCACATGAGGTAGCAAATGTTTCCTCCTTCCCTTGTTTCTGTCGTTGCTCGCGAACTCCCCCTCCTCCCCTGCTACAAATACCCCCACCGGCCCGGACAGGTCTCCTGCACACTCGCAGCTCGCACATCTCATGGTGTCCCAAGCACAGCAAGAGCCAGCTCTGCCTCACAGCAGCAGCACCGCCAAGCGCGCAGCCGCGTCACTCATGGACGCCCGCCCGGCCCAGCCTCTCCTCCTCCGCGCCCCGACTCCCAGCATTGACCTCCCCGCGTCCAAGCCGGACAGGGCCGCCGCGGCGGCCGGCAAGGCCGCCGCCGCCTCCGTGTTCGACCTGCGGCGGGAGCCCAAGATCCCGGCGCCATTCGTGTGGCCGCACGACGACGCGCGGCCGGCGTCGGCGGCGGAGCTGGACGTGCCGTTGGTGGACGTGGGCGTGCTGCGCAATGGCGACCGCGCGGGGCTGCGGCGCGCTGCGGCGCAGGTGGCCGCGGCGTGCGCGACGCACGGGTTCTTCCAGGTGTGCGGGCACGGCGTGGGCGCGGACCTGGCGCGCGCGGCGCTGGACGGCGCCAGTGACTTCTTCCGGCTGCCGCTGGCGGAGAAGCAGCGCGCCCGGCGCGTCCCGGGGACCGTGTCCGGGTACACGAGCGCGCACGCCGACCGGTTCGCGTCCAAGCTCCCCTGGAAGGAGACCCTCTCCTTCGGGTTCCACGACGGCGCCGCGTCGCCCGTCGTCGTCGACTACTTCGCCGGCACCCTCGGGCAGGACTTCGAGGCAGTGGGGTAAGTATGTAGGAATGAACTTGGCACGCATTGCATCCACATGGCGTGCTGATCGAACGAGCTGAGCCAACCGGCATGCACACATGGCGTGGCAGGCGGGTGTACCAGAGGTACTGCGAGGAGATGAAGGCTCTGTCGCTGACGATCATGGAGCTCCTGGAGCTGAGCCTGGGCGTGGAGCGCGGCTACTACCGCGACTTCTTCGAGGACAGCCGCTCCATCATGCGGTGCAACTACTACCCGCCGTGCCCGGAGCCGGAGCGCACGCTGGGCACGGGCCCGCACTGCGACCCCACCGCGCTGACCATCCTCCTCCAGGACGACGTCGGCGGGCTCGAGGTCCTCGTCGACGGCGACTGGCGCCCCGTCCGCCCCGTCCCCGGCGCCATGGTCATCAACATCGGCGACACCTTCATGGTACGGCCGCCGCTAATCCATCCTTTTGTTGCTCTTATCTCCTCTGGCGAGTGCGAGTAACGAAAGCGCTAGCTCCCCTGCTCCTTGTCCTGCTCTGTTTCCCAAGTCCTAATGGAGCTAACCGGGCAGACTGCAACACGCACGCGTAGGCATGTCACGTAGCCACCACTTGCACTGTGCTGCGCAGCGACGACGCAACGCGGACGTGCGTTCGAGTCGGTTCCATCTCGGCGCCGCTACACGCGGCCGCGGCTCCTAGCCTCCTAGGGCTCCCTGATCCCTATCCCCGAGCCCTTCCGCGGGAAAAGTTCGTTGGCGACGGCAGAGGAGAGCCGACGGGTCCGTGCCGTTGGAGCGTGGCGGCAGGAGAGGCCGGGAGGGTGTTTTGTTGCGTTGCGCGGCGGCGCGGAGGATGCGATGGCGCGGGCGGGCGGCGCTTTCGGCGGTGGCCCCCGCGACCCACGTGCGCGCGCGGTCTCGTCGCCTTCCCTGTTTTGGTGCCACCTCTCTGTGTCCGGGAATGGGTTGGCTTAGCGGCGACCGAGACCGGGCGGTGGTCTGGCCTGCTCCCGGCGCCCATCCCGCCTGGTCTCTCATCCTGCTCCTCCTATGCGCGAGGGGGCCTGTAGCGGCTGGAGTACAAGCAGATTGGTTGGGTTGGGTTGCTGCTGCTTGGCTGTTGCCCGCCCGCTTTCTAGCCGTTTCCGCTCGCCATCCGGCACGCGGCGCCCACGCCGGGGCTCCAGCTCGGCCCCTTTGGCCGTGTGGGTGGCAGGCACCCCTGCATCGTCTCGTGCGTCCGGTTTCCGCGCCTGGCCCCCCGCCTTGAGGTTTCCCTGTGCTTTTGACAAGACTTTCGTAGATATATGTGTGTGTATGTGTGTGTGTGCGTGCGCGCGTGTGTGTATATATATATATAAATAAATAACATCTGTGAATGATGGATTACACGTGTAGCTGACCGGCTGATTGTGTTCGCGTGTGTGTCTTCGATGCATTGCAGGCTCTGTCCAACGGGCGGTACAAGAGCTGCCTGCACC
SEQ ID No_2,SiSD1蛋白质序列:
MVSQAQQEPALPHSSSTAKRAAASLMDARPAQPLLLRAPTPSIDLPASKPDRAAAAAGKAAAASVFDLRREPKIPAPFVWPHDDARPASAAELDVPLVDVGVLRNGDRAGLRRAAAQVAAACATHGFFQVCGHGVGADLARAALDGASDFFRLPLAEKQRARRVPGTVSGYTSAHADRFASKLPWKETLSFGFHDGAASPVVVDYFAGTLGQDFEAVGRVYQRYCEEMKALSLTIMELLELSLGVERGYYRDFFEDSRSIMRCNYYPPCPEPERTLGTGPHCDPTALTILLQDDVGGLEVLVDGDWRPVRPVPGAMVINIGDTFMALSNGRYKSCLHRAVVNQRQERRSLAFFLCPREDRVVRPPASGAVGEAPRRYPDFTWADLMRFTQRHYRADTRTLDAFTRWLSHGPAQDAPVAAAAST
SEQ ID No_3,谷子SiSD1基因的等位基因sisd1核酸序列:
ATGGCCTCCCCTGTGGGGCGGGTCCCACATGAGGTAGCAAATGTTTCCTCCTTCCCTTGTTTCTGTCGTTGCTCGCGAACTCCCCCTCCTCCCCTGCTACAAATACCCCCACCGGCCCGGACAGGTCTCCTGCACACTCGCAGCTCGCACATCTCATGGTGTCCCAAGCACAGCAAGAGCCAGCTCTGCCTCACAGCAGCAGCACCGCCAAGCGCGCAGCCGCGTCACTCATGGACGCCCGCCCGGCCCAGCCTCTCCTCCTCCGCGCCCCGACTCCCAGCATTGACCTCCCCGCGTCCAAGCCGGACAGGGCCGCCGCGGCGGCCGGCAAGGCCGCCGCCGCCTCCGTGTTCGACCTGCGGCGGGAGCCCAAGATCCCGGCGCCATTCGTGTGGCCGCACGACGACGCGCGGCCGGCGTCGGCGGCGGAGCTGGACGTGCCGTTGGTGGACGTGGGCGTGCTGCGCAATGGCGACCGCGCGGGGCTGCGGCGCGCTGCGGCGCAGGTGGCCGCGGCGTGCGCGACGCACGGGTTCTTCCAGGTGTGCGGGCACGGCGTGGGCGCGGACCTGGCGCGCGCGGCGCTGGACGGCGCCAGTGACTTCTTCCGGCTGCCGCTGGCGGAGAAGCAGCGCGCCCGGCGCGTCCCGGGGACCGTGTCCGGGTACACGAGCGCGCACGCCGACCGGTTCGCGTCCAAGCTCCCCTGGAAGGAGACCCTCTCCTTCGGGTTCCACGACGGCGCCGCGTCGCCCGTCGTCGTCGACTACTTCGCCGGCACCCTCGGGCAGGACTTCGAGGCAGTGGGGTAAGTATGTAGGAATGAACTTGGCACGCATTGCATCCACATGGCGTGCTGATCGAACGAGCTGAGCCAACCGGCATGCACACATGGCGTGGCAGGCGGGTGTACCAGAGGTACTGCGAGGAGATGAAGGCTCTGTCGCTGACGATCATGGAGCTCCTGGAGCTGAGCCTGGGCGTGGAGCGCGGCTACTACCGCGACTTCTTCGAGGACAGCCGCTCCATCATGCGGTGCAACTACTACCCGCCGTGCCCGGAGCCGGAGCGCACGCTGGGCACGGGCCCGCACTGCGACCCCACCGCGCTGACCATCCTCCTCCAGGACGACGTCGGCGGGCTCGAGGTCCTCGTCGACGGCGACTGGCGCCCCGTCCGCCCCGTCCCCGGCGCCATGGTCATCAACATCGGCGACACCTTCATGGTACGGCCGCCGCTAATCCATCCTTTTGTTGCTCTTATCTCCTCTGGCGAGTGCGAGTAACGAAAGCGCTAGCTCCCCTGCTCCTTGTCCTGCTCTGTTTCCCAAGTCCTAATGGAGCTAACCGGGCAGACTGCAACACGCACGCGTAGGCATGTCACGTAGCCACCACTTGCACTGTGCTGCGCAGCGACGACGCAACGCGGACGTGCGTTCGAGTCGGTTCCATCTCGGCGCCGCTACACGCGGCCGCGGCTCCTAGCCTCCTAGGGCTCCCTGATCCCTATCCCCGAGCCCTTCCGCGGGAAAAGTTCGTTGGCGACGGCAGAGGAGAGCCGACGGGTCCGTGCCGTTGGAGCGTGGCGGCAGGAGAGGCCGGGAGGGTGTTTTGTTGCGTTGCGCGGCGGCGCGGAGGATGCGATGGCGCGGGCGGGCGGCGCTTTCGGCGGTGGCCCCCGCGACCCACGTGCGCGCGCGGTCTCGTCGCCTTCCCTGTTTTGGTGCCACCTCTCTGTGTCCGGGAATGGGTTGGCTTAGCGGCGACCGAGACCGGGCGGTGGTCTGGCCTGCTCCCGGCGCCCATCCCGCCTGGTCTCTCATCCTGCTCCTCCTATGCGCGAGGGGGCCTGTAGCGGCTGGAGTACAAGCAGATTGGTTGGGTTGGGTTGCTGCTGCTTGGCTGTTGCCCGCCCGCTTTCTAGCCGTTTCCGCTCGCCATCCGGCACGCGGCGCCCACGCCGGGGCTCCAGCTCGGCCCCTTTGGCCGTGTGGGTGGCAGGCACCCCTGCATCGTCTCGTGCGTCCGGTTTCCGCGCCTGGCCCCCCGCCTTGAGGTTTCCCTGTGCTTTTGACAAGACTTTCGTAGATATATGTGTGTGTATGTGTGTGTGTGCGTGCGCGCGTGTGTGTATATATATATATAAATAAATAACATCTGTGAATGATGGATTACACGTGTAGCTGACCGGCTGATTGTGTTCGCGTGTGTGTCTTCGATGCATTGCAGGCTCTGTCCAACGGGCGGTACAAGAGCTGCCTGCACCGGCGGTGGTGAACCAGCGGCAGGAGCGGCGGTCGCTGGCCTTCTTCCTGTGCCCGCGCGAGGACCGGGTGGTGCGCCCGCCGGCCAGCGGCGCCGTCGGCGAGGCGCCCCGCCGCTACCCGGACTTCACCTGGGCCGACCTCATGCGCTTCACGCAGCGCCACTACCGCGCCGACACCCGCACGCTGGACGCCTTCACACGCTGGCTCTCCCACGGCCCGGCCCAGGACGCGCCAGTGGCGGCGGCGGCTTCCACCTAGCTAGCGGCGCGGATCCGACCGAGCCCATTGACGACGCCGTCCCTTTCCGCCGCCGCCGGGGCCCGCGCGGGGGTTCACCCCACGTGCGCGCCCAGGTGGGCGAGGTGGCGGCCTCGTGGCCCGCGGGCCCCGCGCCGCCTTCCCATTTTTGGGCGCTGCCGCCCCGCGCGCATGCCGGATGCGTGCGTCCACGGCCTACTGCTGCTACTAGTGTACATATACAAACATACATATATACGTAGTATAAATATATAAGCAAGCGGCCCGGTGCCCCTTTTCGTTTTCTTGTTTTGTCGATCACAATCTCTGGATTCGATGGATGGATAAATGTTTGTACGCATGCATGTAGATGGGCTCATGAAATTTCAGAATCTG
SEQ ID No_4,sisd1蛋白质序列:
MVSQAQQEPALPHSSSTAKRAAASLMDARPAQPLLLRAPTPSIDLPASKPDRAAAAAGKAAAASVFDLRREPKIPAPFVWPHDDARPASAAELDVPLVDVGVLRNGDRAGLRRAAAQVAAACATHGFFQVCGHGVGADLARAALDGASDFFRLPLAEKQRARRVPGTVSGYTSAHADRFASKLPWKETLSFGFHDGAASPVVVDYFAGTLGQDFEAVGRVYQRYCEEMKALSLTIMELLELSLGVERGYYRDFFEDSRSIMRCNYYPPCPEPERTLGTGPHCDPTALTILLQDDVGGLEVLVDGDWRPVRPVPGAMVINIGDTFMALSNGRYKSCLHRRW
SEQ ID No_5,CAPS分子标记SiSD1NciI引物序列:
SiSD1NciIF:5′GGGCCTGTAGCGGCTGGAGTACAA3′
SEQ ID No_6,CAPS分子标记SiSD1NciI引物序列:
SiSD1NciIB:5′TCGCGCGGGCACAGGAAGAAGG3′
SEQ ID No_7,CAPS分子标记SiSD1NciI酒谷PCR产物序列:
GGGCCTGTAGCGGCTGGAGTACAAGCAGATTGGTTGGGTTGGGTTGCTGCTGCTTGGCTGTTGCCCGCCCGCTTTCTAGCCGTTTCCGCTCGCCATCCGGCACGCGGCGCCCACGCCGGGGCTCCAGCTCGGCCCCTTTGGCCGTGTGGGTGGCAGGCACCCCTGCATCGTCTCGTGCGTCCGGTTTCCGCGCCTGGCCCCCCGCCTTGAGGTTTCCCTGTGCTTTTGACAAGACTTTCGTAGATATATGTGTGTGTATGTGTGTGTGTGCGTGCGCGCGTGTGTGTATATATATATATAAATAAATAACATCTGTGAATGATGGATTACACGTGTAGCTGACCGGCTGATTGTGTTCGCGTGTGTGTCTTCGATGCATTGCAGGCTCTGTCCAACGGGCGGTACAAGAGCTGCCTGCACCGGGCGGTGGTGAACCAGCGGCAGGAGCGGCGGTCGCTGGCCTTCTTCCTGTGCCCGCGCGA
SEQ ID No_8,CAPS分子标记SiSD1NciI S2 PCR产物序列:
GGGCCTGTAGCGGCTGGAGTACAAGCAGATTGGTTGGGTTGGGTTGCTGCTGCTTGGCTGTTGCCCGCCCGCTTTCTAGCCGTTTCCGCTCGCCATCCGGCACGCGGCGCCCACGCCGGGGCTCCAGCTCGGCCCCTTTGGCCGTGTGGGTGGCAGGCACCCCTGCATCGTCTCGTGCGTCCGGTTTCCGCGCCTGGCCCCCCGCCTTGAGGTTTCCCTGTGCTTTTGACAAGACTTTCGTAGATATATGTGTGTGTATGTGTGTGTGTGCGTGCGCGCGTGTGTGTATATATATATATAAATAAATAACATCTGTGAATGATGGATTACACGTGTAGCTGACCGGCTGATTGTGTTCGCGTGTGTGTCTTCGATGCATTGCAGGCTCTGTCCAACGGGCGGTACAAGAGCTGCCTGCACCGGCGGTGGTGAACCAGCGGCAGGAGCGGCGGTCGCTGGCCTTCTTCCTGTGCCCGCGCGA
序列表
<120> CAPS分子标记在鉴定谷子株高性状的用途及引物和检测试剂盒
<160> 8
<170> PatentIn version 3.3
<210> 1
<211> 2900
<212> DNA
<213> 谷子
<400> 1
atggcctccc ctgtggggcg ggtcccacat gaggtagcaa atgtttcctc cttcccttgt 60
ttctgtcgtt gctcgcgaac tccccctcct cccctgctac aaataccccc accggcccgg 120
acaggtctcc tgcacactcg cagctcgcac atctcatggt gtcccaagca cagcaagagc 180
cagctctgcc tcacagcagc agcaccgcca agcgcgcagc cgcgtcactc atggacgccc 240
gcccggccca gcctctcctc ctccgcgccc cgactcccag cattgacctc cccgcgtcca 300
agccggacag ggccgccgcg gcggccggca aggccgccgc cgcctccgtg ttcgacctgc 360
ggcgggagcc caagatcccg gcgccattcg tgtggccgca cgacgacgcg cggccggcgt 420
cggcggcgga gctggacgtg ccgttggtgg acgtgggcgt gctgcgcaat ggcgaccgcg 480
cggggctgcg gcgcgctgcg gcgcaggtgg ccgcggcgtg cgcgacgcac gggttcttcc 540
aggtgtgcgg gcacggcgtg ggcgcggacc tggcgcgcgc ggcgctggac ggcgccagtg 600
acttcttccg gctgccgctg gcggagaagc agcgcgcccg gcgcgtcccg gggaccgtgt 660
ccgggtacac gagcgcgcac gccgaccggt tcgcgtccaa gctcccctgg aaggagaccc 720
tctccttcgg gttccacgac ggcgccgcgt cgcccgtcgt cgtcgactac ttcgccggca 780
ccctcgggca ggacttcgag gcagtggggt aagtatgtag gaatgaactt ggcacgcatt 840
gcatccacat ggcgtgctga tcgaacgagc tgagccaacc ggcatgcaca catggcgtgg 900
caggcgggtg taccagaggt actgcgagga gatgaaggct ctgtcgctga cgatcatgga 960
gctcctggag ctgagcctgg gcgtggagcg cggctactac cgcgacttct tcgaggacag 1020
ccgctccatc atgcggtgca actactaccc gccgtgcccg gagccggagc gcacgctggg 1080
cacgggcccg cactgcgacc ccaccgcgct gaccatcctc ctccaggacg acgtcggcgg 1140
gctcgaggtc ctcgtcgacg gcgactggcg ccccgtccgc cccgtccccg gcgccatggt 1200
catcaacatc ggcgacacct tcatggtacg gccgccgcta atccatcctt ttgttgctct 1260
tatctcctct ggcgagtgcg agtaacgaaa gcgctagctc ccctgctcct tgtcctgctc 1320
tgtttcccaa gtcctaatgg agctaaccgg gcagactgca acacgcacgc gtaggcatgt 1380
cacgtagcca ccacttgcac tgtgctgcgc agcgacgacg caacgcggac gtgcgttcga 1440
gtcggttcca tctcggcgcc gctacacgcg gccgcggctc ctagcctcct agggctccct 1500
gatccctatc cccgagccct tccgcgggaa aagttcgttg gcgacggcag aggagagccg 1560
acgggtccgt gccgttggag cgtggcggca ggagaggccg ggagggtgtt ttgttgcgtt 1620
gcgcggcggc gcggaggatg cgatggcgcg ggcgggcggc gctttcggcg gtggcccccg 1680
cgacccacgt gcgcgcgcgg tctcgtcgcc ttccctgttt tggtgccacc tctctgtgtc 1740
cgggaatggg ttggcttagc ggcgaccgag accgggcggt ggtctggcct gctcccggcg 1800
cccatcccgc ctggtctctc atcctgctcc tcctatgcgc gagggggcct gtagcggctg 1860
gagtacaagc agattggttg ggttgggttg ctgctgcttg gctgttgccc gcccgctttc 1920
tagccgtttc cgctcgccat ccggcacgcg gcgcccacgc cggggctcca gctcggcccc 1980
tttggccgtg tgggtggcag gcacccctgc atcgtctcgt gcgtccggtt tccgcgcctg 2040
gccccccgcc ttgaggtttc cctgtgcttt tgacaagact ttcgtagata tatgtgtgtg 2100
tatgtgtgtg tgtgcgtgcg cgcgtgtgtg tatatatata tataaataaa taacatctgt 2160
gaatgatgga ttacacgtgt agctgaccgg ctgattgtgt tcgcgtgtgt gtcttcgatg 2220
cattgcaggc tctgtccaac gggcggtaca agagctgcct gcaccgggcg gtggtgaacc 2280
agcggcagga gcggcggtcg ctggccttct tcctgtgccc gcgcgaggac cgggtggtgc 2340
gcccgccggc cagcggcgcc gtcggcgagg cgccccgccg ctacccggac ttcacctggg 2400
ccgacctcat gcgcttcacg cagcgccact accgcgccga cacccgcacg ctggacgcct 2460
tcacacgctg gctctcccac ggcccggccc aggacgcgcc agtggcggcg gcggcttcca 2520
cctagctagc ggcgcggatc cgaccgagcc cattgacgac gccgtccctt tccgccgccg 2580
ccggggcccg cgcgggggtt caccccacgt gcgcgcccag gtgggcgagg tggcggcctc 2640
gtggcccgcg ggccccgcgc cgccttccca tttttgggcg ctgccgcccc gcgcgcatgc 2700
cggatgcgtg cgtccacggc ctactgctgc tactagtgta catatacaaa catacatata 2760
tacgtagtat aaatatataa gcaagcggcc cggtgcccct tttcgttttc ttgttttgtc 2820
gatcacaatc tctggattcg atggatggat aaatgtttgt acgcatgcat gtagatgggc 2880
tcatgaaatt tcagaatctg 2900
<210> 2
<211> 423
<212> PRT
<213> 谷子
<400> 2
Met Val Ser Gln Ala Gln Gln Glu Pro Ala Leu Pro His Ser Ser Ser
1 5 10 15
Thr Ala Lys Arg Ala Ala Ala Ser Leu Met Asp Ala Arg Pro Ala Gln
20 25 30
Pro Leu Leu Leu Arg Ala Pro Thr Pro Ser Ile Asp Leu Pro Ala Ser
35 40 45
Lys Pro Asp Arg Ala Ala Ala Ala Ala Gly Lys Ala Ala Ala Ala Ser
50 55 60
Val Phe Asp Leu Arg Arg Glu Pro Lys Ile Pro Ala Pro Phe Val Trp
65 70 75 80
Pro His Asp Asp Ala Arg Pro Ala Ser Ala Ala Glu Leu Asp Val Pro
85 90 95
Leu Val Asp Val Gly Val Leu Arg Asn Gly Asp Arg Ala Gly Leu Arg
100 105 110
Arg Ala Ala Ala Gln Val Ala Ala Ala Cys Ala Thr His Gly Phe Phe
115 120 125
Gln Val Cys Gly His Gly Val Gly Ala Asp Leu Ala Arg Ala Ala Leu
130 135 140
Asp Gly Ala Ser Asp Phe Phe Arg Leu Pro Leu Ala Glu Lys Gln Arg
145 150 155 160
Ala Arg Arg Val Pro Gly Thr Val Ser Gly Tyr Thr Ser Ala His Ala
165 170 175
Asp Arg Phe Ala Ser Lys Leu Pro Trp Lys Glu Thr Leu Ser Phe Gly
180 185 190
Phe His Asp Gly Ala Ala Ser Pro Val Val Val Asp Tyr Phe Ala Gly
195 200 205
Thr Leu Gly Gln Asp Phe Glu Ala Val Gly Arg Val Tyr Gln Arg Tyr
210 215 220
Cys Glu Glu Met Lys Ala Leu Ser Leu Thr Ile Met Glu Leu Leu Glu
225 230 235 240
Leu Ser Leu Gly Val Glu Arg Gly Tyr Tyr Arg Asp Phe Phe Glu Asp
245 250 255
Ser Arg Ser Ile Met Arg Cys Asn Tyr Tyr Pro Pro Cys Pro Glu Pro
260 265 270
Glu Arg Thr Leu Gly Thr Gly Pro His Cys Asp Pro Thr Ala Leu Thr
275 280 285
Ile Leu Leu Gln Asp Asp Val Gly Gly Leu Glu Val Leu Val Asp Gly
290 295 300
Asp Trp Arg Pro Val Arg Pro Val Pro Gly Ala Met Val Ile Asn Ile
305 310 315 320
Gly Asp Thr Phe Met Ala Leu Ser Asn Gly Arg Tyr Lys Ser Cys Leu
325 330 335
His Arg Ala Val Val Asn Gln Arg Gln Glu Arg Arg Ser Leu Ala Phe
340 345 350
Phe Leu Cys Pro Arg Glu Asp Arg Val Val Arg Pro Pro Ala Ser Gly
355 360 365
Ala Val Gly Glu Ala Pro Arg Arg Tyr Pro Asp Phe Thr Trp Ala Asp
370 375 380
Leu Met Arg Phe Thr Gln Arg His Tyr Arg Ala Asp Thr Arg Thr Leu
385 390 395 400
Asp Ala Phe Thr Arg Trp Leu Ser His Gly Pro Ala Gln Asp Ala Pro
405 410 415
Val Ala Ala Ala Ala Ser Thr
420
<210> 3
<211> 2899
<212> DNA
<213> 谷子
<400> 3
atggcctccc ctgtggggcg ggtcccacat gaggtagcaa atgtttcctc cttcccttgt 60
ttctgtcgtt gctcgcgaac tccccctcct cccctgctac aaataccccc accggcccgg 120
acaggtctcc tgcacactcg cagctcgcac atctcatggt gtcccaagca cagcaagagc 180
cagctctgcc tcacagcagc agcaccgcca agcgcgcagc cgcgtcactc atggacgccc 240
gcccggccca gcctctcctc ctccgcgccc cgactcccag cattgacctc cccgcgtcca 300
agccggacag ggccgccgcg gcggccggca aggccgccgc cgcctccgtg ttcgacctgc 360
ggcgggagcc caagatcccg gcgccattcg tgtggccgca cgacgacgcg cggccggcgt 420
cggcggcgga gctggacgtg ccgttggtgg acgtgggcgt gctgcgcaat ggcgaccgcg 480
cggggctgcg gcgcgctgcg gcgcaggtgg ccgcggcgtg cgcgacgcac gggttcttcc 540
aggtgtgcgg gcacggcgtg ggcgcggacc tggcgcgcgc ggcgctggac ggcgccagtg 600
acttcttccg gctgccgctg gcggagaagc agcgcgcccg gcgcgtcccg gggaccgtgt 660
ccgggtacac gagcgcgcac gccgaccggt tcgcgtccaa gctcccctgg aaggagaccc 720
tctccttcgg gttccacgac ggcgccgcgt cgcccgtcgt cgtcgactac ttcgccggca 780
ccctcgggca ggacttcgag gcagtggggt aagtatgtag gaatgaactt ggcacgcatt 840
gcatccacat ggcgtgctga tcgaacgagc tgagccaacc ggcatgcaca catggcgtgg 900
caggcgggtg taccagaggt actgcgagga gatgaaggct ctgtcgctga cgatcatgga 960
gctcctggag ctgagcctgg gcgtggagcg cggctactac cgcgacttct tcgaggacag 1020
ccgctccatc atgcggtgca actactaccc gccgtgcccg gagccggagc gcacgctggg 1080
cacgggcccg cactgcgacc ccaccgcgct gaccatcctc ctccaggacg acgtcggcgg 1140
gctcgaggtc ctcgtcgacg gcgactggcg ccccgtccgc cccgtccccg gcgccatggt 1200
catcaacatc ggcgacacct tcatggtacg gccgccgcta atccatcctt ttgttgctct 1260
tatctcctct ggcgagtgcg agtaacgaaa gcgctagctc ccctgctcct tgtcctgctc 1320
tgtttcccaa gtcctaatgg agctaaccgg gcagactgca acacgcacgc gtaggcatgt 1380
cacgtagcca ccacttgcac tgtgctgcgc agcgacgacg caacgcggac gtgcgttcga 1440
gtcggttcca tctcggcgcc gctacacgcg gccgcggctc ctagcctcct agggctccct 1500
gatccctatc cccgagccct tccgcgggaa aagttcgttg gcgacggcag aggagagccg 1560
acgggtccgt gccgttggag cgtggcggca ggagaggccg ggagggtgtt ttgttgcgtt 1620
gcgcggcggc gcggaggatg cgatggcgcg ggcgggcggc gctttcggcg gtggcccccg 1680
cgacccacgt gcgcgcgcgg tctcgtcgcc ttccctgttt tggtgccacc tctctgtgtc 1740
cgggaatggg ttggcttagc ggcgaccgag accgggcggt ggtctggcct gctcccggcg 1800
cccatcccgc ctggtctctc atcctgctcc tcctatgcgc gagggggcct gtagcggctg 1860
gagtacaagc agattggttg ggttgggttg ctgctgcttg gctgttgccc gcccgctttc 1920
tagccgtttc cgctcgccat ccggcacgcg gcgcccacgc cggggctcca gctcggcccc 1980
tttggccgtg tgggtggcag gcacccctgc atcgtctcgt gcgtccggtt tccgcgcctg 2040
gccccccgcc ttgaggtttc cctgtgcttt tgacaagact ttcgtagata tatgtgtgtg 2100
tatgtgtgtg tgtgcgtgcg cgcgtgtgtg tatatatata tataaataaa taacatctgt 2160
gaatgatgga ttacacgtgt agctgaccgg ctgattgtgt tcgcgtgtgt gtcttcgatg 2220
cattgcaggc tctgtccaac gggcggtaca agagctgcct gcaccggcgg tggtgaacca 2280
gcggcaggag cggcggtcgc tggccttctt cctgtgcccg cgcgaggacc gggtggtgcg 2340
cccgccggcc agcggcgccg tcggcgaggc gccccgccgc tacccggact tcacctgggc 2400
cgacctcatg cgcttcacgc agcgccacta ccgcgccgac acccgcacgc tggacgcctt 2460
cacacgctgg ctctcccacg gcccggccca ggacgcgcca gtggcggcgg cggcttccac 2520
ctagctagcg gcgcggatcc gaccgagccc attgacgacg ccgtcccttt ccgccgccgc 2580
cggggcccgc gcgggggttc accccacgtg cgcgcccagg tgggcgaggt ggcggcctcg 2640
tggcccgcgg gccccgcgcc gccttcccat ttttgggcgc tgccgccccg cgcgcatgcc 2700
ggatgcgtgc gtccacggcc tactgctgct actagtgtac atatacaaac atacatatat 2760
acgtagtata aatatataag caagcggccc ggtgcccctt ttcgttttct tgttttgtcg 2820
atcacaatct ctggattcga tggatggata aatgtttgta cgcatgcatg tagatgggct 2880
catgaaattt cagaatctg 2899
<210> 4
<211> 340
<212> PRT
<213> 谷子
<400> 4
Met Val Ser Gln Ala Gln Gln Glu Pro Ala Leu Pro His Ser Ser Ser
1 5 10 15
Thr Ala Lys Arg Ala Ala Ala Ser Leu Met Asp Ala Arg Pro Ala Gln
20 25 30
Pro Leu Leu Leu Arg Ala Pro Thr Pro Ser Ile Asp Leu Pro Ala Ser
35 40 45
Lys Pro Asp Arg Ala Ala Ala Ala Ala Gly Lys Ala Ala Ala Ala Ser
50 55 60
Val Phe Asp Leu Arg Arg Glu Pro Lys Ile Pro Ala Pro Phe Val Trp
65 70 75 80
Pro His Asp Asp Ala Arg Pro Ala Ser Ala Ala Glu Leu Asp Val Pro
85 90 95
Leu Val Asp Val Gly Val Leu Arg Asn Gly Asp Arg Ala Gly Leu Arg
100 105 110
Arg Ala Ala Ala Gln Val Ala Ala Ala Cys Ala Thr His Gly Phe Phe
115 120 125
Gln Val Cys Gly His Gly Val Gly Ala Asp Leu Ala Arg Ala Ala Leu
130 135 140
Asp Gly Ala Ser Asp Phe Phe Arg Leu Pro Leu Ala Glu Lys Gln Arg
145 150 155 160
Ala Arg Arg Val Pro Gly Thr Val Ser Gly Tyr Thr Ser Ala His Ala
165 170 175
Asp Arg Phe Ala Ser Lys Leu Pro Trp Lys Glu Thr Leu Ser Phe Gly
180 185 190
Phe His Asp Gly Ala Ala Ser Pro Val Val Val Asp Tyr Phe Ala Gly
195 200 205
Thr Leu Gly Gln Asp Phe Glu Ala Val Gly Arg Val Tyr Gln Arg Tyr
210 215 220
Cys Glu Glu Met Lys Ala Leu Ser Leu Thr Ile Met Glu Leu Leu Glu
225 230 235 240
Leu Ser Leu Gly Val Glu Arg Gly Tyr Tyr Arg Asp Phe Phe Glu Asp
245 250 255
Ser Arg Ser Ile Met Arg Cys Asn Tyr Tyr Pro Pro Cys Pro Glu Pro
260 265 270
Glu Arg Thr Leu Gly Thr Gly Pro His Cys Asp Pro Thr Ala Leu Thr
275 280 285
Ile Leu Leu Gln Asp Asp Val Gly Gly Leu Glu Val Leu Val Asp Gly
290 295 300
Asp Trp Arg Pro Val Arg Pro Val Pro Gly Ala Met Val Ile Asn Ile
305 310 315 320
Gly Asp Thr Phe Met Ala Leu Ser Asn Gly Arg Tyr Lys Ser Cys Leu
325 330 335
His Arg Arg Trp
340
<210> 5
<211> 24
<212> DNA
<213> 人工序列
<400> 5
gggcctgtag cggctggagt acaa 24
<210> 6
<211> 22
<212> DNA
<213> 人工序列
<400> 6
tcgcgcgggc acaggaagaa gg 22
<210> 7
<211> 482
<212> DNA
<213> 谷子
<400> 7
gggcctgtag cggctggagt acaagcagat tggttgggtt gggttgctgc tgcttggctg 60
ttgcccgccc gctttctagc cgtttccgct cgccatccgg cacgcggcgc ccacgccggg 120
gctccagctc ggcccctttg gccgtgtggg tggcaggcac ccctgcatcg tctcgtgcgt 180
ccggtttccg cgcctggccc cccgccttga ggtttccctg tgcttttgac aagactttcg 240
tagatatatg tgtgtgtatg tgtgtgtgtg cgtgcgcgcg tgtgtgtata tatatatata 300
aataaataac atctgtgaat gatggattac acgtgtagct gaccggctga ttgtgttcgc 360
gtgtgtgtct tcgatgcatt gcaggctctg tccaacgggc ggtacaagag ctgcctgcac 420
cgggcggtgg tgaaccagcg gcaggagcgg cggtcgctgg ccttcttcct gtgcccgcgc 480
ga 482
<210> 8
<211> 481
<212> DNA
<213> 谷子
<400> 8
gggcctgtag cggctggagt acaagcagat tggttgggtt gggttgctgc tgcttggctg 60
ttgcccgccc gctttctagc cgtttccgct cgccatccgg cacgcggcgc ccacgccggg 120
gctccagctc ggcccctttg gccgtgtggg tggcaggcac ccctgcatcg tctcgtgcgt 180
ccggtttccg cgcctggccc cccgccttga ggtttccctg tgcttttgac aagactttcg 240
tagatatatg tgtgtgtatg tgtgtgtgtg cgtgcgcgcg tgtgtgtata tatatatata 300
aataaataac atctgtgaat gatggattac acgtgtagct gaccggctga ttgtgttcgc 360
gtgtgtgtct tcgatgcatt gcaggctctg tccaacgggc ggtacaagag ctgcctgcac 420
cggcggtggt gaaccagcgg caggagcggc ggtcgctggc cttcttcctg tgcccgcgcg 480
a 481
- CAPS分子标记在鉴定谷子株高性状的用途及引物和检测试剂盒
- 鉴定万寿菊瓣型的CAPS分子标记、检测引物及检测试剂盒
