一种基于RPA-CRISPR/Cas12a检测病原微生物的引物及其应用
文献发布时间:2024-04-18 19:58:53
技术领域
本发明属于分子生物学检测技术领域,涉及一种基于RPA-CRISPR/Cas12a检测病原微生物(淋病奈瑟菌及FC428头孢曲松耐药克隆)的引物及其应用和检测方法。
背景技术
淋病是由淋病奈瑟菌(Neisseria gonorrhoeae,NG)感染引起的一种常见的性传播疾病。世界卫生组织2019年预估淋病发病率位列细菌性性传播疾病的第2位,15至49岁成年人中每年淋病的新发病例数达8690万。目前大多数国家推荐的淋病奈瑟菌一线治疗药物是头孢曲松。然而,全球范围内淋病奈瑟菌对头孢曲松的耐药水平正在上升。其中,最初于2015年在日本发现对头孢曲松耐药的FC428克隆,在国际上逐渐流行,造成了严重的公共卫生威胁。FC428克隆对多种抗生素耐药,面临无药可治的风险。因此,开发淋病奈瑟菌FC428头孢曲松耐药克隆的及时诊断方法,为临床医生提供及时的用药指导,对耐药淋病奈瑟菌的的临床治疗、现场防治和监测预警至关重要。
目前,检测淋病奈瑟菌FC428头孢曲松耐药克隆的常用方法主要包括抗生素敏感性测试(Antimicrobial susceptibility testing,AST)、聚合酶链式反应(polymerasechain reaction,PCR)、实时荧光PCR法(quantitative real-time PCR,qPCR)等,然而,这些方法受限于花费大、检测周期长、需要专业技术人员和庞大设备支持等缺点。作为发展迅速分子诊断技术,基于规律成簇的间隔短回文重复序列(clustered regularlyinterspaced short palindromic repeats,CRISPR)/CRISPR相关(CRISPR-associated,Cas)系统具有强特异性和可编程性,易与现场即时检测设备相结合,已在核酸检测领域展现出巨大潜力。为提高基于CRISPR检测方法的灵敏度,其与等温扩增方法如重组酶聚合酶扩增技术(recombinase polymerase amplification,RPA)结合后,可实现单分子检测。因此,应用RPA-CRISPR系统检测淋病奈瑟菌FC428头孢曲松耐药克隆提供了一种快速(检测时间1小时)、方便(38℃等温反应)、简便(易与现场即使检测设备结合)的方法,具有重要意义。
微流控芯片技术可集成不同检测操作单元,由微管道联系整个系统。由于其在生物、化学、医学等领域展现处的潜力,微流控芯片技术已发展成多学科交叉的崭新研究领域。搭载多个反应腔室,实现高通量检测的微流控芯片简化了实验操作,有利于现场防治,大大提升现场即时筛查的能力。
发明内容
本发明的目的在于提供一种特异性强、灵敏度高的鉴定一种基于RPA-CRISPR/Cas12a检测病原微生物(淋病奈瑟菌及FC428头孢曲松耐药克隆)的引物及其应用和检测方法。
为实现上述目的,本发明采用的技术方案为:
一种鉴定病原微生物的引物,病原微生物为淋病奈瑟菌或淋病奈瑟菌FC428头孢曲松;其中,
检测淋病奈瑟菌专用等温扩增引物为一组RPA扩增引物,碱基序列依次为SEQ IDNO.1-2所示;
检测淋病奈瑟菌FC428头孢曲松耐药克隆专用等温扩增引物为一组RPA扩增引物,碱基序列依次为SEQ ID NO.3-4所示。
鉴定病原体的引物的应用,所述检测淋病奈瑟菌专用等温扩增引物在淋病奈瑟菌种属鉴定中的应用;
检测淋病奈瑟菌FC428头孢曲松耐药克隆专用等温扩增引物在检测淋病奈瑟菌FC428头孢曲松耐药克隆中的应用。
一种淋病奈瑟菌的crRNA,用于CRISPR反应鉴别淋病奈瑟菌的特异性crRNA,碱基序列依次为SEQ ID NO.5所示。
一种淋病奈瑟菌FC428头孢曲松耐药克隆的crRNA,用于CRISPR反应鉴别淋病奈瑟菌FC428头孢曲松耐药克隆的特异性crRNA,碱基序列依次为SEQ ID NO.6所示。
一种所述的crRNA的应用,所述crRNA在鉴别淋病奈瑟菌中的应用;
所述crRNA在鉴别淋病奈瑟菌FC428头孢曲松耐药克隆中的应用。
上述淋病奈瑟菌鉴定,是指能准确将淋病奈瑟菌鉴定出。选取淋病奈瑟菌种间特异、种内保守的基因保守区设计RPA扩增引物;同时根据CRISPR原则设计一条特异性的crRNA;
上述用于淋病奈瑟菌FC428头孢曲松耐药克隆鉴别的专用引物,是选取penA种间变异区的基因或区域作为RPA扩增靶标及CRISPR/Cas12a检测靶标,并筛选包含CRISPR/Cas12a检测所需原型间区序列邻近基序(protospacer adjacent motif,PAM)位点的靶标序列设计crRNA。
上述淋病奈瑟菌鉴别专用RPA引物和crRNA,所述专用RPA引物共一组,为SEQ IDNO.1-2两条引物组成,一条为正向引物,一条为反向引物,用于RPA扩增反应;所述特异性crRNA共一条,为SEQ ID NO.5。上述淋病奈瑟菌FC428头孢曲松耐药克隆鉴别专用RPA引物和crRNA,所述专用RPA引物共一组,为SEQ ID NO.3-4两条引物组成,一条为正向引物,一条为反向引物,用于RPA扩增反应;所述特异性crRNA共一条,为SEQ ID NO.6。引物序列信息见表1。
表1引物及crRNA序列信息
一种用于检测淋病奈瑟菌FC428头孢曲松耐药克隆的微流控芯片装配体检测平台,含SEQ ID NO.1-4所示用于检测淋病奈瑟菌及淋病奈瑟菌FC428头孢曲松耐药克隆的RPA引物,以及SEQ ID NO.5-6所示检测淋病奈瑟菌及淋病奈瑟菌FC428头孢曲松耐药克隆crRNA。
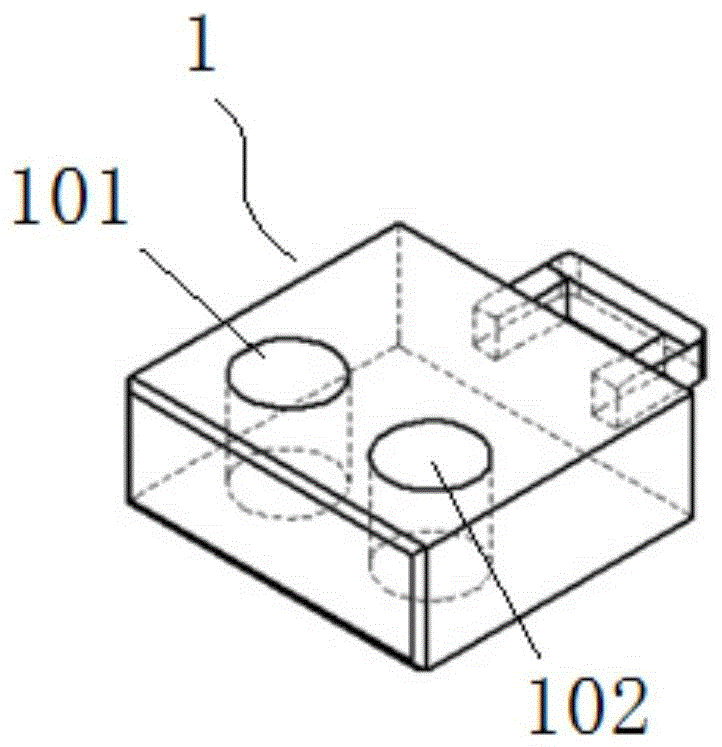
所述检测平台包括第一零件1和第二零件2,其中第一零件1上设有第一下腔体101和第二下腔体102,第二零件2下部设有插槽105,第二零件2上部设有第一上腔体201、第二上腔体202和进样口203,并且所述进样口203设于第一上腔体201和第二上腔体202之间,所述进样口203两侧设有连接通道204分别与对应侧的上腔体连通,第一零件1插装于第二零件2下部的插槽105中,并且第一零件1插入后,第一下腔体101与第一上腔体201对应衔接形成淋病奈瑟菌反应腔,第二下腔体102与第二上腔体202对应衔接形成淋病奈瑟菌FC428头孢曲松耐药克隆反应腔。
所述第一下腔体101承载淋病奈瑟菌的CRISPR体系,第二下腔体201承载淋病奈瑟菌FC428头孢曲松耐药克隆的CRISPR体系;
第一上腔体201承载淋病奈瑟菌的RPA反应体系,第二上腔体202承载淋病奈瑟菌FC428头孢曲松耐药克隆的RPA反应体系。
所述第一下腔体101与第一上腔体201之间;以及第二下腔体102与第二上腔体202之间使用3M
零件2中的进样口设于中心,中心进样口通过毛细管通道204连接两侧的两个RPA反应腔。
其中,微流控芯片装配体的构建为:微流控芯片经Solidworks2021软件设计,芯片包含两个零件。CRISPR反应腔所在零件长12.4mm,宽11.2mm,高4.5mm;CRISPR反应腔体为直径3.6mm,高4mm的圆柱。RPA反应腔室所在零件长15mm,宽11.2mm,高11.1mm。RPA反应腔室为直径为3mm,高6mm的圆柱体,中心进样口直径1.4mm。将设计稿保存为.stl格式后,用3D打印机(Formlabs
一种利用所述检测平台检测淋病奈瑟菌及淋病奈瑟菌FC428头孢曲松耐药克隆的方法:
(1)使用商品化DNA提取试剂盒完成测试样本核酸的提取;
(2)以待测样本的核酸为模板,在利用权利要求1所述引物的引导下配制RPA扩增反应体系,进行各检测反应孔特异性扩增;
其中,RPA体系的构建为:RPA试剂盒购自英国TwistDx公司。往RPA干粉里加入25μL缓冲液、2μL正向引物RPA-porA-F(10μM)、2μL反向引物RPA-porA-R(10μM)、2μL正向引物RPA-penA-F(10μM)、2μL反向引物RPA-penA-R(10μM)、8μL H
其中,反应条件为:金属浴模块中38℃恒温孵育20min。
(3)将2μL扩增产物加入分别包含特异性crRNA的CRISPR/Cas12a体系中;根据Cas12a蛋白对荧光报告探针的切割,实验组反应前后荧光从无到有的变化,检测样本中淋病奈瑟菌及淋病奈瑟菌FC428头孢曲松耐药克隆有无。
所述每个CRISPR/Cas12a反应体系的构建为:由终浓度为100nM Cas12a、100nMcrRNA、600nM荧光报告探针、1x NEB r2.1 Buffer以及无核酶水共同配制合成终体积为13.5μL的反应体系;所述每个CRISPR/Cas12a反应体系中荧光报告探针为FAM-TTATT-BHQ1。
本发明方法,种属鉴定的目的是对阳性淋病奈瑟菌样品进行分类,使其不与非淋病奈瑟菌菌株发生交叉反应。
所述通过荧光变化判定,若样本为淋病奈瑟菌阳性,则在包含淋病奈瑟菌种属鉴定的反应腔室可观察到荧光信号,若样本为淋病奈瑟菌FC428头孢曲松耐药克隆crRNA阳性,则在种属鉴定和FC428头孢曲松耐药克隆特异性的反应腔室均可观察到荧光信号。
进一步的说,在CRISPR/Cas12a反应中,由于目标序列的序列特异性,仅能与其完全碱基互补配对的crRNA结合,激活相应Cas12a蛋白,根据Cas12a蛋白对荧光报告探针的切割,实验组反应前后荧光从无到有的变化,在QuantStudio 7 Flex Real-Time PCR System进行CRISPR/Cas12a反应的条件为:38℃孵育30min完成反应,每30秒收集一次荧光信号。反应结束后使用QuantStudio 6 and 7 Flex Real-Time PCR software v1.0软件提取各组荧光强度数值,运用GraphPad Prism 8、PowerPoint进行结果图的绘制。若荧光强度显著高于阴性对照组,则表明样本中存在检测靶标。
在微流控芯片装配体上进行CRISPR/Cas12a反应的条件为:38℃孵育20min完成反应。当样本不是淋病奈瑟菌时,两个腔室不产生荧光信号;当样本时淋病奈瑟菌而非FC428克隆时,荧光信号仅在包含淋病奈瑟菌特异性crRNA的腔室中产生;当样本为淋病奈瑟菌FC428头孢曲松耐药克隆,含有淋病奈瑟菌特异性crRNA以及FC428克隆特异性crRNA的两个腔室中均产生荧光信号。为了使系统的性能最大化,我们优化了RPA和CRISPR反应的条件。
与现有技术相比,本发明的积极效果是:
首先,本发明方法利用RPA-CRISPR方法鉴定淋病奈瑟菌、鉴别淋病奈瑟菌FC428头孢曲松耐药克隆,反应在恒温38℃条件下进行,没有复杂的温控需求;本方法反应时间方面,基于PCR管的分步反应时间为RPA 20min、CRISPR反应30min,共50min,而基于微流控芯片装配体的分步法总反应时间为40min,即RPA反应20min、CRISPR反应20min,实现快速检测;本发明方法设计的RPA专用引物,可在同一体系内分别扩增出包含淋病耐瑟菌保守区和淋病奈瑟菌FC428头孢曲松耐药克隆变异区的靶标序列,后偶联CRISPR/Cas12a反应体系,实现方便、特异检测;进一步,本方法将RPA-CRISPR反应集成至微流控芯片,根据芯片上各反应腔室的荧光有无即可判断淋病奈瑟菌FC428头孢曲松耐药克隆。因此本方法为开发淋病奈瑟菌FC428头孢曲松耐药克隆的检测方法及检测平台提供新的技术手段。
附图说明
图1为本发明实施例提供的鉴定淋病奈瑟菌和淋病奈瑟菌FC428头孢曲松耐药克隆的微流控芯片装配体检测平台。
图2为本发明实施例提供的鉴定淋病奈瑟菌和淋病奈瑟菌FC428头孢曲松耐药克隆的微流控芯片装配体检测平台。
图3为本发明实施例提供的基于PCR管荧光监测的鉴定淋病奈瑟菌和淋病奈瑟菌FC428头孢曲松耐药克隆的结果示意图。
图4为本发明实施例提供的检测方法原理图。
图5为本发明实施例提供的基于微流控芯片平台的鉴定淋病奈瑟菌和淋病奈瑟菌FC428头孢曲松耐药克隆的结果示意图。
具体应用方式
以下结合实例对本发明的具体实施方式做进一步说明,应当指出的是,此处所描述的具体实施方式只是为了说明和解释本发明,并不局限于本发明。
本发明为实现淋病奈瑟菌和淋病奈瑟菌FC428头孢曲松耐药克隆鉴定设计了RPA扩增引物,扩增淋病奈瑟菌保守区及淋病奈瑟菌FC428头孢曲松耐药克隆变异区的核酸片段,后偶联基于规律成簇的间隔短回文重复序列(clustered regularly interspacedshort palindromic repeats,CRISPR)/CRIPSR相关(CRISPR-associated,Cas)12a检测系统,通过设计两种crRNA序列,分别用于检测淋病奈瑟菌特异性,以及淋病奈瑟菌FC428头孢曲松耐药克隆。上述检测方法被进一步集成到微流控芯片上,通过判读各个反应腔室的荧光信号有无,即可实现淋病奈瑟菌的种属鉴定和FC428头孢曲松耐药克隆的鉴定。
实施例1:微流控芯片装配体检测平台设计
如图1-2所述,检测平台包括第一零件1(图1)和第二零件2(图2),其中第一零件1上设有第一下腔体101和第二下腔体102,第二零件2下部设有插槽105,第二零件2上部设有第一上腔体201、第二上腔体202和进样口203,并且所述进样口203设于第一上腔体201和第二上腔体202之间,所述进样口203两侧设有连接通道204分别与对应侧的上腔体连通,第一零件1插装于第二零件2下部的插槽105中,并且第一零件1插入后,第一下腔体101与第一上腔体201对应衔接形成淋病奈瑟菌反应腔,第二下腔体102与第二上腔体202对应衔接形成淋病奈瑟菌FC428头孢曲松耐药克隆反应腔。
所述第一下腔体101承载淋病奈瑟菌的CRISPR体系,第二下腔体201承载淋病奈瑟菌FC428头孢曲松耐药克隆的CRISPR体系;
第一上腔体201承载淋病奈瑟菌的RPA反应体系,第二上腔体202承载淋病奈瑟菌FC428头孢曲松耐药克隆的RPA反应体系。
所述第一下腔体101与第一上腔体201之间以及第二下腔体102与第二上腔体202之间使用3M
零件2中的进样口设于中心,中心进样口通过毛细管通道204连接两侧的两个RPA反应腔。
其中,微流控芯片装配体的构建为:微流控芯片经Solidworks 2021软件设计,芯片包含两个零件。CRISPR反应腔所在零件长12.4mm,宽11.2mm,高4.5mm;CRISPR反应腔体为直径3.6mm,高4mm的圆柱。RPA反应腔室所在零件长15mm,宽11.2mm,高11.1mm。RPA反应腔室为直径为3mm,高6mm的圆柱体,中心进样口直径1.4mm。将设计稿保存为.stl格式后,用3D打印机(Formlabs
首先在零件1的两个反应孔中分别预先储存针对淋病奈瑟菌和FC428头孢曲松耐药克隆的CRISPR体系,将零件1完全插入零件2中,使RPA反应腔体下端接近密封状态。再将RPA体系通过进样口注射入RPA反应腔。将装置置于38℃下孵育20min待RPA完全反应,在轻拉零件1,使得RPA与CRISPR反应孔贯通,从而混合体系。继续在38℃下反应20min,即可判读实验结果。
实施例2:对干样本实施基于PCR管的淋病奈瑟菌种属鉴定和淋病奈瑟菌FC428头孢曲松耐药克隆鉴别。
(1)样本采集:采集患者拭子于装有样品保存液的采样管中,保存于-80℃超低温冰箱。
(2)样本核酸提取:根据QIAGEN QIAamp DNA Mini Kit试剂盒操作说明提取样本中的DNA。最终所得DNA样品溶于50μL无核酶水备用。
(3)以待测样本的核酸为模板,在上述专用引物组(SEQ ID NO.1-4所示引物)的引导下配制RPA扩增反应体系;
其中,RPA体系的构建为:RPA试剂盒购自英国TwistDx公司。往RPA干粉里加入25μL缓冲液、2μL正向引物RPA-porA-F(10μM)、2μL反向引物RPA-porA-R(10μM)、2μL正向引物RPA-penA-F(10μM)、2μL反向引物RPA-penA-R(10μM)、8μL H
其中,反应条件为:金属浴模块中38℃恒温孵育20min。
(4)将1.5μL扩增产物加入分别包含特异性crRNA的CRISPR/Cas12a体系中;根据Cas12a蛋白对荧光报告探针的切割,实验组反应前后荧光从无到有的变化,检测样本中淋病奈瑟菌有无,并鉴别FC428头孢曲松耐药克隆。
所述每个CRISPR/Cas12a反应体系的构建为:由终浓度为100nM Cas12a、100nMcrRNA、600nM荧光报告探针、1x NEB r2.1 Buffer以及无核酶水共同配制合成终体积为13.5μL的反应体系;所述每个CRISPR/Cas12a反应体系中荧光报告探针为FAM-TTATT-BHQ1。
其中,反应条件为:在QuantStudio 7 Flex Real-Time PCR System38℃孵育30min完成反应,每30秒收集一次荧光信号。
(5)结果判读:反应结束后使用QuantStudio 6 and 7 Flex Real-Time PCRsoftware v1.0软件提取各组荧光强度数值,运用GraphPad Prism 8、PowerPoint进行结果图的绘制。若荧光强度显著高于阴性对照组,则表明样本中存在检测靶标。若样本种存在淋病奈瑟菌FC428头孢曲松耐药克隆,应引起两个CRISPR/Cas12a反应均输出荧光信号。
(6)最终该方法的测试结果为:porA-crRNA、penA-crRNA产生明显荧光信号,则测得靶标为淋病奈瑟菌FC428头孢曲松耐药克隆(参见图3)。实施例3:利用上述实施例1获得微流控芯片对拭子样本实施微流控芯片检测,原理参见图4。
(1)样本采集:采集患者拭子于装有样品保存液的采样管中,保存于-80℃超低温冰箱。
(2)样本核酸提取:根据QIAGEN QIAamp DNA Mini Kit试剂盒操作说明提取样本中的DNA。最终所得DNA样品溶于50μL无核酶水备用。
(3)以待测样本的核酸为模板,在上述专用引物组(SEQ ID NO.1-4所示引物)的引导下配制RPA扩增反应体系;
其中,RPA体系的构建为:RPA试剂盒购自英国TwistDx公司。往RPA干粉里加入25μL缓冲液、2μL正向引物RPA-porA-F(10μM)、2μL反向引物RPA-porA-R(10μM)、2μL正向引物RPA-penA-F(10μM)、2μL反向引物RPA-penA-R(10μM)、10μL H
所述样品为携带淋病奈瑟菌FC428头孢曲松耐药克隆患者拭子经过核酸提取后获取的DNA。
(4)将配好的25μLRPA反应体系自微流控芯片中心进样口加入,分别流入包含两种特异性crRNA的CRISPR/Cas12a体系中。在CRISPR/Cas12a反应中,由于目标序列的序列特异性,仅能与其完全碱基互补配对的crRNA结合,激活相应Cas12a蛋白,根据Cas12a蛋白对荧光报告探针的切割,实验组反应前后荧光从无到有的变化,检测样本中淋病奈瑟菌有无,并鉴别淋病奈瑟菌FC428克隆。
其中,CRISPR/Cas12a反应体系的构建为:由终浓度为100nM Cas12a、100nMcrRNA、600nM荧光报告探针、1x NEB r2.1 Buffer以及无核酶水共同配制合成终体积为13.5μL的反应体系。分别包含两种用于鉴定淋病奈瑟菌和FC428头孢曲松耐药克隆的crRNA的CRISPR/Cas12a体系被预先置于两侧反应腔室中。
(4)金属浴模块中38℃恒温孵育20min后,拉动下侧零件1,使得RPA与CRISPR反应孔贯通,从而混合体系。继续在38℃下反应20min,即可判读实验结果。
(5)结果判读:检测结果的判定,若样本为淋病奈瑟菌阳性,则在左侧包含淋病奈瑟菌种属鉴定porA-crRNA的反应腔室可观察到荧光信号;根据淋病奈瑟菌FC428头孢曲松耐药克隆特异性penA-crRNA的反应腔室荧光信号有无,判断样本是否感染淋病奈瑟菌FC428头孢曲松耐药克隆(参见图5)。
(6)最终该方法的测试结果为:根据各反应腔室荧光信号有无,判别该样本是否感染淋病奈瑟菌种属鉴定和淋病奈瑟菌FC428头孢曲松耐药克隆。
- 一种基于BTNAA体系检测犬瘟热病毒的引物对、引物和探针组合物、试剂及试剂盒
- 一种基于BTNAA体系检测犬细小病毒的引物对、引物和探针组合物、试剂及试剂盒
- 一种基于高通量测序技术检测DMD基因变异的引物及其应用
- 一种基于RPA-CRISPR/Cas12a检测大丽轮枝菌的引物和试剂盒及检测方法
- 一种基于RPA-CRISPR/Cas12a的人型支原体检测系统及其应用
