通过功能结构域嫁接提高聚合酶大片段活性的方法及应用
文献发布时间:2023-06-19 09:26:02
技术领域
本发明涉及一种通过功能结构域嫁接的方法提高嗜热脂肪土芽孢杆菌属(Geobacillus stearothermophilus)DNA聚合酶大片段即Bst DNA聚合酶大片段对底物亲和力的方法及应用,属于分子生物学、蛋白质工程、酶工程领域。
背景技术
DNA聚合酶,又称DNA依赖的DNA聚合酶(DNA dependent DNA polymerase),它是以亲代DNA为模板,催化底物dNTP分子聚合形成子代DNA的一类酶,广泛存在于原核生物和真核生物中,是基因复制的基础,是维持机体基因组稳定和完整遗传的关键。DNA聚合酶有三种功能:①聚合作用:在引物3'-OH末端,以dNTP为底物,按模板DNA上的指令,即A与T,C与G的配对原则,逐步逐个、连续地将dNTP加到延伸中的DNA分子3'-OH末端,逐步合成延长中的子链DNA。这是DNA聚合酶的主要作用;②3’→5’外切酶活性(校对作用):从3’→5’方向识别和切除不配对的子链DNA末端的核苷酸,主要功能是校对作用,保证复制过程的保真性和准确性;③5’→3’外切酶活性(切除修复作用):从5’→3’方向水解DNA延长链前方的DNA链(即只对DNA上双链处的磷酸二酯键有切割作用),产生5'—脱氧核苷酸。这种酶活性在DNA损伤的修复中可能起着重要作用。
嗜热脂肪土芽孢杆菌属(Geobacillus stearothermophilus)DNA聚合酶,即BstDNA聚合酶,基因全长2631bp,氨基酸序列全长876aa,Bst DNA聚合酶大片段是Bst DNA聚合酶第291位氨基酸至876位氨基酸序列酶,其不具有Bst DNA聚合酶5’→3’外切酶活性(切除修复作用),无水解母链DNA分子5’端的单核苷酸,因其继承了5’端的单核苷酸的链置换反应、高保真、耐高温等特点,而成为一种重要的商用DNA多重置换扩增酶。
Sso7d是一种来源于嗜热硫化裂片菌(Sulfolobus solfataricus)的双链DNA结合蛋白,分子量为7kd,可非特异性的结合双链DNA片段,结合后会产生不可预料的效果(或降低活性,或提高活性,或灭活,或无变化)。本发明尝试将Sso7d、Sso7a、Sso7c3、Sso7c4系列双链DNA结合蛋白的DNA结合结构域与Bst DNA聚合酶大片段融合,旨在提高Bst DNA聚合酶大片段的反应速率。
单链结合蛋白(Single Strand Binding protein,SSB)又称DNA结合蛋白,其功能在于非特异性的和单链DNA结合,稳定解开的DNA单链,阻止复性和保护单链部分不被核酸酶水解,主要参与DNA复制、修复和重组。单链结合蛋白是177个氨基酸组成的蛋白,发挥功能是以四聚体的形式与单链DNA结合,并和其他单链结合蛋白四聚体、相邻单链DNA结合,被结合的单链DNA是呈伸展状态的,这种功能有利于以单链DNA作为模板进行的核酸扩增。因此,将单链结合蛋白与Bst DNA聚合酶大片段共价连接或许可以提高DNA聚合酶对底物的亲和力,以提高反应速率。
发明内容
针对上述现有技术,本发明提供了一种通过功能结构域嫁接提高聚合酶大片段活性的方法,本发明分别将Sso7d DNA结合蛋白、SSB单链结合蛋白与嗜热脂肪土芽孢杆菌(Geobacillus stearothermophilus)Bst DNA聚合酶大片段通过共价键连接,得到了两种重组的Bst DNA聚合酶大片段:Sso7d-Bst DNA聚合酶,SSB-Bst DNA聚合酶;通过反应速率检测结果显示:Sso7d-Bst DNA聚合酶大片段较野生型的Bst DNA聚合酶大片段反应速率提高,而SSB-Bst DNA聚合酶大片段较野生型的Bst DNA聚合酶大片段反应速率降低。
本发明是通过以下技术方案实现的:
一种通过功能结构域嫁接提高聚合酶大片段活性的方法:将Sso7d DNA结合蛋白的基因与野生型Bst DNA聚合酶大片段的基因连接,并转入原核表达载体进行表达、纯化;
所述Sso7d DNA结合蛋白的基因序列如下所示,如SEQ ID NO.1所示;
所述野生型Bst DNA聚合酶大片段的基因序列如下所示,如SEQ ID NO.2所示。
Sso7d DNA结合蛋白的基因序列(5’→3’)(SEQ ID NO.1):
CATATGATGGCAACCGTAAAGTTCAAGTACAAAGGCGAAGAAAAAGAGGTAGACATCTCCAAGATCAAGAAAGTATGGCGTGTGGGCAAGATGATCTCCTTCACCTACGACGAGGGCGGTGGCAAGACCGGCCGTGGTGCGGTAAGCGAAAAGGACGCGCCGAAGGAGCTGCTGCAGATGCTGGAGAAGCAGAAAAAG。
野生型Bst DNA聚合酶大片段的基因序列:(5’→3’)(SEQ ID NO.2):
CATATGGAAGGGGAGAAACCGCTTGAGGAGATGGAGTTTGCCATCGTTGACGTCATTACCGAAGAGATGCTTGCCGACAAGGCAGCGCTTGTCGTTGAGGTGATGGAAGAAAACTACCACGATGCCCCGATTGTCGGAATCGCACTAGTGAACGAGCATGGGCGATTTTTTATGCGCCCGGAGACCGCGCTGGCTGATTCGCAATTTTTAGCATGGCTTGCCGATGAAACGAAGAAAAAAAGCATGTTTGACGCCAAGCGGGCAGTCGTTGCCTTAAAGTGGAAAGGAATTGAGCTTCGCGGCGTCGCCTTTGATTTATTGCTCGCTGCCTATTTGCTCAATCCGGCTCAAGATGCCGGCGATATCGCTGCGGTGGCGAAAATGAAACAATATGAAGCGGTGCGGTCGGATGAAGCGGTCTATGGCAAAGGCGTCAAGCGGTCGCTGCCGGACGAACAGACGCTTGCTGAGCATCTCGTTCGCAAAGCGGCAGCCATTTGGGCGCTTGAGCAGCCGTTTATGGACGATTTGCGGAACAACGAACAAGATCAATTATTAACGAAGCTTGAGCAGCCGCTGGCGGCGATTTTGGCTGAAATGGAATTCACTGGGGTGAACGTGGATACAAAGCGGCTTGAACAGATGGGTTCGGAGCTCGCCGAACAACTGCGTGCCATCGAGCAGCGCATTTACGAGCTAGCCGGCCAAGAGTTCAA CATTAACTCACCAAAACAGCTCGGAGTCATTTTATTTGAAAAGCTGCAGCTACCGGTGCTGAAGAAGACGAAAACAGGCTATTCGACTTCGGCTGATGTGCTTGAGAAGCTTGCGCCGCATCATGAAATCGTCGAAAACATTTTGCATTACCGCCAGCTTGGCAAACTGCAATCAACGTATATTGAAGGATTGTTGAAAGTTGTGCGCCCTGATACCGGCAAAGTGCATACGATGTTCAACCAAGCGCTGACGCAAACTGGGCGGCTCAGCTCGGCCGAGCCGAACTTGCAAAACATTCCGATTCGGCTCGAAGAGGGGCGGAAAATCCGCCAAGCGTTCGTCCCGTCAGAGCCGGACTGGCTCATTTTCGCCGCCGATTACTCACAAATTGAATTGCGCGTCCTCGCCCATATCGCCGATGACGACAATCTAATTGAAGCGTTCCAACGCGATTTGGATATTCACACAAAAACGGCGATGGACATTTTCCATGTGAGCGAAGAGGAAGTCACGGCCAACATGCGCCGCCAGGCAAAGGCCGTTAACTTCGGTATCGTTTACGGAATTAGCGATTACGGATTGGCGCAAAACTTGAACATTACGCGCAAAGAAGCTGCCGAATTTATCGAACGTTACTTCGCCAGCTTTCCGGGCGTAAAGCAGTATATGGAAAACATTGTGCAAGAAGCGAAACAGAAAGGATATGTGACAACGCTGTTGCATCGGCGCCGCTATTTGCCTGATATTACAAGCCGCAATTTCAACGTCCGCAGTTTTGCAGAGCGGACGGCCATGAACACGCCAATTCAAGGAAGCGCCGCTGACATTATTAAAAAAGCGATGATTGATTTAGCGGCACGGCTGAAAGAAGAGCAGCTTCAGGCTCGTCTTTTGCTGCAAGTGCATGACGAGCTCATTTTGGAAGCGCCAAAAGAGGAAATTGAGCGATTATGTGAGCTTGTTCCGGAAGTGATGGAGCAGGCCGTTACGCTCCGCGTGCCGCTGAAAGTCGACTACCATTACGGCCCAACATGGTATGATGCCAAATAAGGATCC。
进一步地,通过Linker将Sso7d DNA结合蛋白的基因与野生型Bst DNA聚合酶大片段的基因连接;所述Linker序列如下所示,如SEQ ID NO.3所示。
Linker序列(5’→3’)(SEQ ID NO.3):
GGAGGCGGAGGCTCAGGAGGCGGAGGCTCAGGAGGCGGAGGCTCA。
通过Linker将Sso7d DNA结合蛋白的基因与野生型Bst DNA聚合酶大片段的基因连接后基因序列如下所示,如SEQ ID NO.5所示。
Sso7d-Bst DNA聚合酶大片段的基因序列(5’→3’)(SEQ ID NO.5)(其中,双下划线部分为Sso7d DNA双链结合蛋白的基因序列,单下划线部分为Linker的基因序列,其余为Bst DNA聚合酶大片段的基因序列):
进一步地,具体方法如下:通过Linker将Sso7d DNA结合蛋白的基因与野生型BstDNA聚合酶大片段的基因连接,克隆至大肠杆菌表达载体pET-16b载体上,酶切位点选择NdeI和XhoI,并在XhoI酶切位点后添加终止密码子,得到重组质粒;将重组质粒转入Rosetta(DE3)表达感受态细胞,诱导表达目的蛋白,通过亲和层析、离子层析、分子筛等纯化方法得到纯度较高的Sso7d-Bst DNA聚合酶。通过BSA蛋白浓度测定法测定蛋白浓度,用SDS-PAGE法鉴定蛋白纯度,通过单碱基延伸法对Sso7d-Bst DNA聚合酶进行反应速率的测定,以野生型Bst DNA聚合酶大片段做对照。
所述通过功能结构域嫁接改变Bst DNA聚合酶大片段活性的方法在提高Bst DNA聚合酶大片段活性中的应用。
一种Sso7d-Bst DNA聚合酶,其氨基酸序列如下所示,如SEQ ID NO.6所示,共计687个氨基酸,预测分子量为77.4kda,等电点为5.97。
Sso7d-Bst DNA聚合酶的氨基酸序列如下所示(SEQ ID NO.6)(5’→3’)(注:波浪下划线部分为pET-16b载体本身表达带有的氨基酸序列,双下划线部分为Sso7d双链DNA结合蛋白的氨基酸序列,单下划线为Linker的氨基酸序列,其余为野生型Bst DNA聚合酶大片段氨基酸序列):
本发明的通过功能结构域嫁接提高聚合酶大片段活性的方法,将Sso7d DNA结合蛋白、SSB单链结合蛋白与嗜热脂肪土芽孢杆菌(Geobacillus stearothermophilus)BstDNA聚合酶大片段通过共价键连接,得到了两种重组的Bst DNA聚合酶大片段:Sso7d-BstDNA聚合酶,SSB-Bst DNA聚合酶。通过反应速率检测结果显示:Sso7d-Bst DNA聚合酶大片段较野生型的Bst DNA聚合酶大片段反应速率提高,而SSB-Bst DNA聚合酶大片段较野生型的Bst DNA聚合酶大片段反应速率降低。本发明的方法以及融合DNA聚合酶,有着广阔的应用前景。
本发明使用的各种术语和短语具有本领域技术人员公知的一般含义。提及的术语和短语如有与公知含义不一致的,以本发明所表述的含义为准。
附图说明
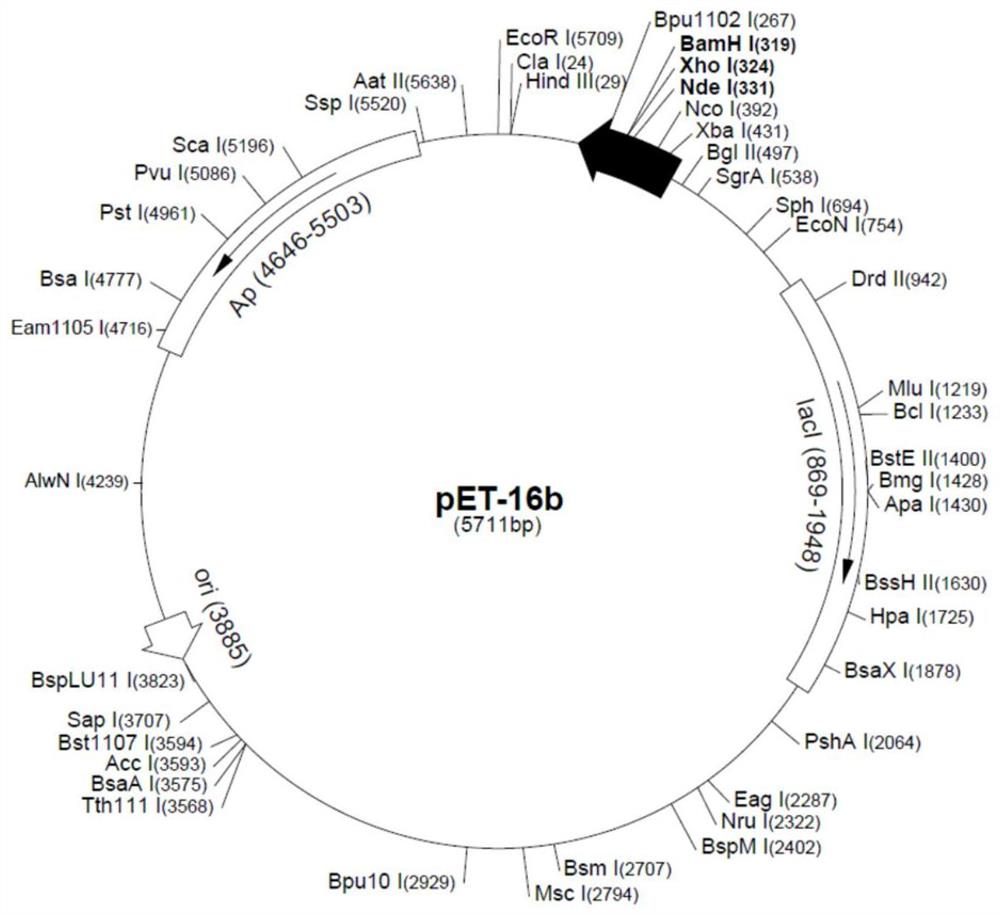
图1:pET16B质粒的结构示意图。
图2:pET16B质粒的克隆表达区域示意图。
图3:酶表达验证结果示意图。
图4:酶纯度鉴定结果示意图。
图5:BSA标准曲线示意图。
图6:底物分子的结构式。
图7:单碱基延伸实验图。
具体实施方式
下面结合实施例对本发明作进一步的说明。然而,本发明的范围并不限于下述实施例。本领域的专业人员能够理解,在不背离本发明的精神和范围的前提下,可以对本发明进行各种变化和修饰。
本发明对试验中所使用到的材料以及试验方法进行一般性和/或具体的描述。虽然为实现本发明目的所使用的许多材料和操作方法是本领域公知的,但是本发明仍然在此作尽可能详细描述。
下述实施例中所涉及的仪器、试剂、材料等,若无特别说明,均为现有技术中已有的常规仪器、试剂、材料等,可通过正规商业途径获得。下述实施例中所涉及的实验方法,检测方法等,若无特别说明,均为现有技术中已有的常规实验方法,检测方法等。
实施例1通过功能结构域嫁接改变Bst DNA聚合酶大片段活性的实验
(一)确定野生型Bst DNA聚合酶大片段(如SEQ ID NO.2所示)、Sso7d DNA结合蛋白(如SEQ ID NO.1所示)、SSB单链结合蛋白的基因序列(如下所示),设计共价连接Linker序列(如SEQ ID NO.3所示),将两段基因通过linker序列连接起来(即Sso7d-LLinker-Bst、SSB-LLinker-Bst),同野生型Bst DNA聚合酶大片段基因序列,分别克隆大肠杆菌表达载体pET-16b载体(如图1、图2所示)上,酶切位点选择NdeI和XhoI,并在XhoI酶切位点后添加终止密码子。
基因合成和克隆设计委托给通用生物系统(安徽)有限公司(通用生物)。
Sso7d DNA结合蛋白的基因序列如下所示(5’→3’)(SEQ ID NO.1):
CATATGATGGCAACCGTAAAGTTCAAGTACAAAGGCGAAGAAAAAGAGGTAGACATCTCCAAGATCAAGAAAGTATGGCGTGTGGGCAAGATGATCTCCTTCACCTACGACGAGGGCGGTGGCAAGACCGGCCGTGGTGCGGTAAGCGAAAAGGACGCGCCGAAGGAGCTGCTGCAGATGCTGGAGAAGCAGAAAAAG。
SSB单链结合蛋白的基因序列如下所示(5’→3’):
CATATGATCGAGGGAAGGATTTCAGAAAATCTTTATTTTCAAGGTATGGAAGAAAAAGTAGGTAATCTGAAACCAAATATGGAAAGCGTAAATGTAACCGTACGAGTTTTGGAAGCAAGCGAAGCACGTCAAATCCAGACAAAGAACGGTGTTCGGACAATCAGTGAGGCTATTGTTGGAGATGAAACGGGACGAGTAAAGTTAACATTATGGGGAAAACATGCAGGTAGTATCAAAGAAGGTCAAGTGGTAAAGATTGAAAACGCGTGGACCACCGCTTTTAAGGGTCAAGTACAGTTAAATGCTGGAAGCAAAACTAAGATCGCTGAAGCTTCAGAAGATGGATTTCCAGAATCATCTCAAATTCCAGAAAATACACCAACAGCTCCTCAGCAAATGCGTGGAGGAGGACGCGGATTCCGCGGTGGGGGACGTCGGTATGGACGACGTGGTGGTCGCCGGCAAGAAAACGAAGAAGGTGAAGAGGAGGGAAGCGGAGGGGTCGAC。
野生型Bst DNA聚合酶大片段的基因序列如下所示(5’→3’)(SEQ ID NO.2):
CATATGGAAGGGGAGAAACCGCTTGAGGAGATGGAGTTTGCCATCGTTGACGTCATTACCGAAGAGATGCTTGCCGACAAGGCAGCGCTTGTCGTTGAGGTGATGGAAGAAAACTACCACGATGCCCCGATTGTCGGAATCGCACTAGTGAACGAGCATGGGCGATTTTTTATGCGCCCGGAGACCGCGCTGGCTGATTCGCAATTTTTAGCATGGCTTGCCGATGAAACGAAGAAAAAAAGCATGTTTGACGCCAAGCGGGCAGTCGTTGCCTTAAAGTGGAAAGGAATTGAGCTTCGCGGCGTCGCCTTTGATTTATTGCTCGCTGCCTATTTGCTCAATCCGGCTCAAGATGCCGGCGATATCGCTGCGGTGGCGAAAATGAAACAATATGAAGCGGTGCGGTCGGATGAAGCGGTCTATGGCAAAGGCGTCAAGCGGTCGCTGCCGGACGAACAGACGCTTGCTGAGCATCTCGTTCGCAAAGCGGCAGCCATTTGGGCGCTTGAGCAGCCGTTTATGGACGATTTGCGGAACAACGAACAAGATCAATTATTAACGAAGCTTGAGCAGCCGCTGGCGGCGATTTTGGCTGAAATGGAATTCACTGGGGTGAACGTGGATACAAAGCGGCTTGAACAGATGGGTTCGGAGCTCGCCGAACAACTGCGTGCCATCGAGCAGCGCATTTACGAGCTAGCCGGCCAAGAGTTCAACATTAACTCACCAAAACAGCTCGGAGTCATTTTATTTGAAAAGCTGCAGCTACCGGTGCTGAAGAAGACGAAAACAGGCTATTCGACTTCGGCTGATGTGCTTGAGAAGCTTGCGCCGCATCATGAAATCGTCGAAAACATTTTGCATTACCGCCAGCTTGGCAAACTGCAATCAACGTATATTGAAGGATTGTTGAAAGTTGTGCGCCCTGATACCGGCAAAGTGCATACGATGTTCAACCAAGCGCTGACGCAAACTGGGCGGCTCAGCTCGGCCGAGCCGAACTTGCAAAACATTCCGATTCGGCTCGAAGAGGGGCGGAAAATCCGCCAAGCGTTCGTCCCGTCAGAGCCGGACTGGCTCATTTTCGCCGCCGATTACTCACAAATTGAATTGCGCGTCCTCGCCCATATCGCCGATGACGACAATCTAATTGAAGCGTTCCAACGCGATTTGGATATTCACACAAAAACGGCGATGGACATTTTCCATGTGAGCGAAGAGGAAGTCACGGCCAACATGCGCCGCCAGGCAAAGGCCGTTAACTTCGGTATCGTTTACGGAATTAGCGATTACGGATTGGCGCAAAACTTGAACATTACGCGCAAAGAAGCTGCCGAATTTATCGAACGTTACTTCGCCAGCTTTCCGGGCGTAAAGCAGTATATGGAAAACATTGTGCAAGAAGCGAAACAGAAAGGATATGTGACAACGCTGTTGCATCGGCGCCGCTATTTGCCTGATATTACAAGCCGCAATTTCAACGTCCGCAGTTTTGCAGAGCGGACGGCCATGAACACGCCAATTCAAGGAAGCGCCGCTGACATTATTAAAAAAGCGATGATTGATTTAGCGGCACGGCTGAAAGAAGAGCAGCTTCAGGCTCGTCTTTTGCTGCAAGTGCATGACGAGCTCATTTTGGAAGCGCCAAAAGAGGAAATTGAGCGATTATGTGAGCTTGTTCCGGAAGTGATGGAGCAGGCCGTTACGCTCCGCGTGCCGCTGAAAGTCGACTACCATTACGGCCCAACATGGTATGATGCCAAATAAGGATCC。
Linker序列如下所示(5’→3’)(SEQ ID NO.3):
GGAGGCGGAGGCTCAGGAGGCGGAGGCTCAGGAGGCGGAGGCTCA。
野生型Bst DNA聚合酶大片段理论表达氨基酸序列如下所示(5’→3’)(SEQ IDNO.4)(注:波浪下划线部分是pET-16b载体本身表达带有的氨基酸序列,其余部分为BstDNA聚合酶氨基酸序列):
共计608个氨基酸,预测分子量为69.2kda,等电点为5.67。
Sso7d-Bst DNA聚合酶大片段的基因序列如下所示(5’→3’)(SEQ ID NO.5)(其中,双下划线部分为Sso7d DNA双链结合蛋白的基因序列,单下划线部分为Linker的基因序列,其余为Bst DNA聚合酶大片段的基因序列):
Sso7d-Bst DNA聚合酶大片段基因理论表达氨基酸序列如下所示(SEQ ID NO.6)(5’→3’)(注:波浪下划线部分为pET-16b载体本身表达带有的氨基酸序列,双下划线部分为Sso7d双链DNA结合蛋白的氨基酸序列,单下划线为Linker的氨基酸序列,其余为野生型Bst DNA聚合酶大片段氨基酸序列):
共计687个氨基酸,预测分子量为77.4kda,等电点为5.97。
SSB-Bst DNA聚合酶大片段的基因序列如下所示(5’→3’)(其中,双下划线部分为SSB DNA单链结合蛋白的基因序列,单下划线部分为Linker的基因序列,其余为Bst DNA聚合酶大片段的基因序列):
SSB-Bst DNA聚合酶大片段基因理论表达氨基酸序列如下所示(5’→3’)(注:波浪下划线为pET-16b载体本身表达带有的氨基酸序列,双下划线部分为SSB单链DNA结合蛋白的氨基酸序列,单下划线为Linker的氨基酸序列,其余为野生型Bst DNA聚合酶大片段氨基酸序列):
共计790个氨基酸,预测分子量为88.3kda,等电点为5.74。
(二)分别将野生型、Sso7d-Bst和SSB-Bst的质粒转入Rosetta(DE3)表达感受态细胞,并诱导表达目的蛋白。
所需培养基、试剂:LB液体培养基,LB固体培养基,100mg/ml氨苄霉素溶液,氨苄抗性平板(含100mg/ml氨苄霉素的LB固体培养基),1M IPTG溶液。均按常规方法配制。
具体操作步骤如下:
1.质粒转化:3种质粒均按照以下步骤分别进行转化。
(1)取50ul灭菌过滤的纯化水加入装有质粒粉末的离心管中,混匀。
(2)取Rosetta(DE3)感受态细胞冰浴融化。
(3)取1ul质粒加入融化的Rosetta(DE3)感受态细胞,轻弹混匀,放在冰浴中30min。
(4)设置水浴锅温度42℃。
(5)将冰浴后含有质粒的感受态细胞放入水浴锅中热激30s。
(6)取800ul LB液体培养基入热激后的细胞中。
(7)将细胞转移至恒温振荡培养箱中37℃、150rpm孵育1h。
(8)取200ul培养后的菌液涂板在氨苄抗性平板中。
(9)倒置放入恒温培养箱,37℃静置培养过夜。
2.菌体活化:3种单克隆菌株均按照以下步骤分别进行活化。
(1)挑取1个单克隆点加入到含有100ug/ml的氨苄霉素的150ml LB液体培养基中。
(2)放入恒温振荡培养箱中37℃、200rpm,培养过夜。
3.扩大培养:3种单克隆菌液均按照以下步骤分别进行培养。
(1)取10ml过夜菌加入含有100ug/ml的1L/瓶的LB液体培养基中。
(2)放入恒温振荡培养箱中37℃、200rpm培养至OD
4.诱导表达:3种菌液均按照以下步骤分别进行诱导表达。
(1)取1ml 1M IPTG加入1L/瓶的菌液中。
(2)放入恒温振荡培养箱中16℃、200rpm培养16~20h。
5.离心收菌:3种菌液均按照以下步骤分别进行收菌。
(1)将诱导表达完毕的菌液,分装入离心瓶中,并用天平两两配平。
(2)离心机收菌7000rpm、15min、16℃。
6.验证目的蛋白的表达:3种蛋白均按照以下步骤分别进行验证表达。
(1)做胶:Bst DNA聚合酶大片段分子量约66Kda,采用10%分离胶加5%浓缩胶配制。按照康为世纪说明书制备SDS-PAGE蛋白胶。
(2)样品处理:
A、650ul菌液12000rpm离心1min,弃上清,用30ul Lysis buffer悬起混匀。
B、取80ul,5*loading buffer加入其中,混匀,100℃,10min加热。
C、12000rpm离心2min。
(3)10ul上样,设置140V恒压,至loading至胶底部。
(4)用康为世纪染色液加热染色,用纯化水加热脱色。结果如图3所示,Sso7d-BstDNA聚合酶、SSB-Bst DNA聚合酶在Marker 75.6kda以上有目的蛋白条带,Bst DNA聚合酶在Marker66.2kda左右有目的蛋白条带。
(三)通过亲和层析、离子层析、分子筛等纯化方法,分别得到纯度较高的野生型、Sso7d-Bst和SSB-Bst DNA聚合酶。
1.所需缓冲液如下:
A、Lysis buffer:150mM NaCl,30mM Tris,pH8.0。
B、Ni Elution buffer:150mM NaCl,250mM咪唑,30mM Tris,pH8.0。
C、Q-Elution buffer:500mM NaCl,30mM Tris,pH8.0。
D、2*storagse buffer:100mM KCl,20mM Tris pH7.4。
具体操作步骤如下:
2.样品处理:
(1)用200ml Lysis buffer充分溶解悬起20g菌体。
(2)用高压均质机破碎菌体。
(3)60℃水浴锅中热激破碎液。
(4)将破碎液转移至离心瓶中,两两配平后,用高速冷冻离心机4℃、12000rpm离心30min。
(5)弃沉淀,收集上清液,并用0.2um滤膜过滤上清液。
3.Ni柱纯化:
(1)将5ml Ni预装柱连接AKTA系统,A、B泵分别放入过滤抽气后的Lysis buffer和Ni elution buffer,并泵洗AKTA系统。
(2)用Lysis buffer平衡Ni柱。
(3)将过滤后上清液上样Ni柱。
(4)Ni Elution buffer梯度洗脱目的蛋白(2ml/min,30min,100%)。
(5)收集洗脱峰。
4.Q柱纯化:
(1)将5ml Q预装柱连接AKTA系统,A、B泵分别放入过滤抽气后的Lysis buffer和Qelution buffer,并泵洗AKTA系统。
(2)用Lysis buffer平衡Q预装柱。
(3)将Ni柱洗脱后的目的蛋白,上样Q预装柱。
(4)Q Elution buffer梯度洗脱目的蛋白(2ml/min,30min,100%)。
(5)收集洗脱峰。
5.脱盐处理:
(1)将Superdex 200预装柱连接AKTA系统,A泵放入过滤抽气后的2*storagsebuffer,并泵洗AKTA系统。
(2)用2*storagse buffer平衡Superdex 200预装柱。
(3)将Q柱洗脱后的目的蛋白,上样Superdex 200预装柱。
(4)收集洗脱峰。
6.目的蛋白处理:将目的蛋白用膜包分别浓缩到3.2mg/ml,然后加入等体积的甘油,形成理论浓度为1.6mg/ml的成品酶。
(四)检验:通过BSA蛋白浓度测定法测定蛋白浓度,用SDS-PAGE法鉴定蛋白纯度,通过单碱基延伸法对野生型Sso7d-Bst和SSB-Bst DNA聚合酶进行反应速率的测定。
1.SDS-PAGE法鉴定酶纯度:
(1)按照康为世纪SDS-PAGE试剂盒说明书制作分离胶是12%、浓缩胶是5%的蛋白胶。
(2)样品处理:取20ul样品和5ul 5*loading buffer于1.5ml离心管中,100℃,加热10min后混合备用。
(3)取3ul样品上样,设置恒压140V进行电泳,待loading buffer颜色至蛋白胶底部后停止电泳。
(4)用康为世纪染色液加热染色,用纯化水加热脱色。
结果如图4所示,Bst DNA聚合酶、Sso7d-Bst DNA聚合酶、SSB-Bst DNA聚合酶经纯化后的纯度均能达到98%以上。
2.BCA法鉴定酶浓度:
(1)按照康为世纪BCA蛋白浓度测定试剂盒说明书,配制工作液。按200ul/酶标孔将工作液分入其中。
(2)样品处理:取50ul纯化水和50ul样品混合待测。
(3)取10ul BSA标准品和样品加入含有工作液的酶标孔中充分混匀,放入恒温培养箱中,静置30min,每点重复测试3次。
(4)酶标仪在562nm处进行紫外吸收检测。
(5)数据处理:制作BCA对照品标准曲线,得出Y=kX±b,依照公式,样品检测值为X,计算浓度Y。结果如表1、表2、图5所示。
表1 BSA标准曲线数据
表2酶浓度数据
结论:SSB-Bst DNA聚合酶浓度为3.44mg/ml;Sso7d-Bst DNA聚合酶浓度为3.30mg/ml;Bst DNA聚合酶浓度为3.25mg/ml。
3.单碱基延伸法检测酶活性和反应速率:
(1)模板制备:引物1和引物2结合后,暴露碱基为T,延伸A底物。
引物序列如下:
引物1(5’→3’):AGAAGCTAGTACTAATTGATGGCAACAGTGTGGCA。
引物2(5’→3’):TGCCACACTGTTGCCATCAATTAGT。
A.将引物1和引物2分别用seq buffer稀释至100uM,按照引物1:引物2(v/v)=4:6,混合均匀。
B.将引物混合物放置在90℃金属浴中,10min,然后2~8℃静置30min。标记浓度为40uM,待用。
(2)底物:5’末端荧光标记的核苷酸,此分子(结构如图6所示)在单独存在时无荧光信号,被DNA聚合酶识别并正确结合到模板上时,可在碱性磷酸酶作用下快速释放荧光信号(涉及和参考的专利CN 104844674 A、CN 105315698 B,且此底物由北京大学北京未来基因诊断高精尖创新中心黄岩谊实验室提供)。
(3)缓冲液:Seq buffer:20mM Tris-HCl,20mM KCl,20mM NH
(4)反应体系:如表3所示。
表3单碱基延伸实验体系
注:g=g/mol*mol,Kd=10
例如:3.25mg/ml*2.1*10
结果及分析:结果如图7、表4所示。
表4单碱基延伸实验数据
从反应起始10s内,单碱基A延伸所释放信号的速率对应酶的反应速率。
在Sso7系列双链DNA结合蛋白系列中,本发明尝试过Sso7d、Sso7a、Sso7c3、Sso7c4的DNA结合结构域与Bst DNA聚合酶大片段的融合,Sso7a、Sso7c3、Sso7c4与Bst DNA聚合酶的融合蛋白均无活性,仅Sso7d对Bst DNA聚合酶的反应速率有提高。
从表4可看出,与野生型Bst DNA聚合酶大片段的反应速率相比(234.8593),SSB-Bst DNA聚合酶的反应速率(71.8672)较低,说明SSB单链结合蛋白通过Linker与Bst DNA聚合酶大片段以收尾相连的形式融合时,会抑制Bst DNA聚合酶大片段的酶活性。Sso7d-BstDNA聚合酶的反应速率(252.3505)较高,较野生型的Bst DNA聚合酶活性提高7.4%,说明Sso7d双链结合蛋白通过Linker与Bst DNA聚合酶大片段以收尾相连的形式融合时,会提高Bst DNA聚合酶大片段的酶活性。
结论:Sso7d双链结合蛋白与Bst DNA聚合酶大片段,可提高了其反应速率。
给本领域技术人员提供上述实施例,以完全公开和描述如何实施和使用所主张的实施方案,而不是用于限制本文公开的范围。对于本领域技术人员而言显而易见的修饰将在所附权利要求的范围内。
序列表
<110> 赛纳生物科技(北京)有限公司
<120> 通过功能结构域嫁接提高聚合酶大片段活性的方法及应用
<141> 2019-07-05
<160> 6
<170> SIPOSequenceListing 1.0
<210> 1
<211> 198
<212> DNA
<213> Sulfolobus solfataricus
<400> 1
catatgatgg caaccgtaaa gttcaagtac aaaggcgaag aaaaagaggt agacatctcc 60
aagatcaaga aagtatggcg tgtgggcaag atgatctcct tcacctacga cgagggcggt 120
ggcaagaccg gccgtggtgc ggtaagcgaa aaggacgcgc cgaaggagct gctgcagatg 180
ctggagaagc agaaaaag 198
<210> 3
<211> 1773
<212> DNA
<213> Geobacillus stearothermophilus
<400> 3
catatggaag gggagaaacc gcttgaggag atggagtttg ccatcgttga cgtcattacc 60
gaagagatgc ttgccgacaa ggcagcgctt gtcgttgagg tgatggaaga aaactaccac 120
gatgccccga ttgtcggaat cgcactagtg aacgagcatg ggcgattttt tatgcgcccg 180
gagaccgcgc tggctgattc gcaattttta gcatggcttg ccgatgaaac gaagaaaaaa 240
agcatgtttg acgccaagcg ggcagtcgtt gccttaaagt ggaaaggaat tgagcttcgc 300
ggcgtcgcct ttgatttatt gctcgctgcc tatttgctca atccggctca agatgccggc 360
gatatcgctg cggtggcgaa aatgaaacaa tatgaagcgg tgcggtcgga tgaagcggtc 420
tatggcaaag gcgtcaagcg gtcgctgccg gacgaacaga cgcttgctga gcatctcgtt 480
cgcaaagcgg cagccatttg ggcgcttgag cagccgttta tggacgattt gcggaacaac 540
gaacaagatc aattattaac gaagcttgag cagccgctgg cggcgatttt ggctgaaatg 600
gaattcactg gggtgaacgt ggatacaaag cggcttgaac agatgggttc ggagctcgcc 660
gaacaactgc gtgccatcga gcagcgcatt tacgagctag ccggccaaga gttcaacatt 720
aactcaccaa aacagctcgg agtcatttta tttgaaaagc tgcagctacc ggtgctgaag 780
aagacgaaaa caggctattc gacttcggct gatgtgcttg agaagcttgc gccgcatcat 840
gaaatcgtcg aaaacatttt gcattaccgc cagcttggca aactgcaatc aacgtatatt 900
gaaggattgt tgaaagttgt gcgccctgat accggcaaag tgcatacgat gttcaaccaa 960
gcgctgacgc aaactgggcg gctcagctcg gccgagccga acttgcaaaa cattccgatt 1020
cggctcgaag aggggcggaa aatccgccaa gcgttcgtcc cgtcagagcc ggactggctc 1080
attttcgccg ccgattactc acaaattgaa ttgcgcgtcc tcgcccatat cgccgatgac 1140
gacaatctaa ttgaagcgtt ccaacgcgat ttggatattc acacaaaaac ggcgatggac 1200
attttccatg tgagcgaaga ggaagtcacg gccaacatgc gccgccaggc aaaggccgtt 1260
aacttcggta tcgtttacgg aattagcgat tacggattgg cgcaaaactt gaacattacg 1320
cgcaaagaag ctgccgaatt tatcgaacgt tacttcgcca gctttccggg cgtaaagcag 1380
tatatggaaa acattgtgca agaagcgaaa cagaaaggat atgtgacaac gctgttgcat 1440
cggcgccgct atttgcctga tattacaagc cgcaatttca acgtccgcag ttttgcagag 1500
cggacggcca tgaacacgcc aattcaagga agcgccgctg acattattaa aaaagcgatg 1560
attgatttag cggcacggct gaaagaagag cagcttcagg ctcgtctttt gctgcaagtg 1620
catgacgagc tcattttgga agcgccaaaa gaggaaattg agcgattatg tgagcttgtt 1680
ccggaagtga tggagcaggc cgttacgctc cgcgtgccgc tgaaagtcga ctaccattac 1740
ggcccaacat ggtatgatgc caaataagga tcc 1773
<210> 4
<211> 45
<212> DNA
<213> Artificial Sequence
<400> 4
ggaggcggag gctcaggagg cggaggctca ggaggcggag gctca 45
<210> 7
<211> 608
<212> PRT
<213> Artificial Sequence
<400> 7
Met Gly His His His His His His His His His His Ser Ser Gly His
1 5 10 15
Ile Glu Gly Arg His Met Glu Gly Glu Lys Pro Leu Glu Glu Met Glu
20 25 30
Phe Ala Ile Val Asp Val Ile Thr Glu Glu Met Leu Ala Asp Lys Ala
35 40 45
Ala Leu Val Val Glu Val Met Glu Glu Asn Tyr His Asp Ala Pro Ile
50 55 60
Val Gly Ile Ala Leu Val Asn Glu His Gly Arg Phe Phe Met Arg Pro
65 70 75 80
Glu Thr Ala Leu Ala Asp Ser Gln Phe Leu Ala Trp Leu Ala Asp Glu
85 90 95
Thr Lys Lys Lys Ser Met Phe Asp Ala Lys Arg Ala Val Val Ala Leu
100 105 110
Lys Trp Lys Gly Ile Glu Leu Arg Gly Val Ala Phe Asp Leu Leu Leu
115 120 125
Ala Ala Tyr Leu Leu Asn Pro Ala Gln Asp Ala Gly Asp Ile Ala Ala
130 135 140
Val Ala Lys Met Lys Gln Tyr Glu Ala Val Arg Ser Asp Glu Ala Val
145 150 155 160
Tyr Gly Lys Gly Val Lys Arg Ser Leu Pro Asp Glu Gln Thr Leu Ala
165 170 175
Glu His Leu Val Arg Lys Ala Ala Ala Ile Trp Ala Leu Glu Gln Pro
180 185 190
Phe Met Asp Asp Leu Arg Asn Asn Glu Gln Asp Gln Leu Leu Thr Lys
195 200 205
Leu Glu Gln Pro Leu Ala Ala Ile Leu Ala Glu Met Glu Phe Thr Gly
210 215 220
Val Asn Val Asp Thr Lys Arg Leu Glu Gln Met Gly Ser Glu Leu Ala
225 230 235 240
Glu Gln Leu Arg Ala Ile Glu Gln Arg Ile Tyr Glu Leu Ala Gly Gln
245 250 255
Glu Phe Asn Ile Asn Ser Pro Lys Gln Leu Gly Val Ile Leu Phe Glu
260 265 270
Lys Leu Gln Leu Pro Val Leu Lys Lys Thr Lys Thr Gly Tyr Ser Thr
275 280 285
Ser Ala Asp Val Leu Glu Lys Leu Ala Pro His His Glu Ile Val Glu
290 295 300
Asn Ile Leu His Tyr Arg Gln Leu Gly Lys Leu Gln Ser Thr Tyr Ile
305 310 315 320
Glu Gly Leu Leu Lys Val Val Arg Pro Asp Thr Gly Lys Val His Thr
325 330 335
Met Phe Asn Gln Ala Leu Thr Gln Thr Gly Arg Leu Ser Ser Ala Glu
340 345 350
Pro Asn Leu Gln Asn Ile Pro Ile Arg Leu Glu Glu Gly Arg Lys Ile
355 360 365
Arg Gln Ala Phe Val Pro Ser Glu Pro Asp Trp Leu Ile Phe Ala Ala
370 375 380
Asp Tyr Ser Gln Ile Glu Leu Arg Val Leu Ala His Ile Ala Asp Asp
385 390 395 400
Asp Asn Leu Ile Glu Ala Phe Gln Arg Asp Leu Asp Ile His Thr Lys
405 410 415
Thr Ala Met Asp Ile Phe His Val Ser Glu Glu Glu Val Thr Ala Asn
420 425 430
Met Arg Arg Gln Ala Lys Ala Val Asn Phe Gly Ile Val Tyr Gly Ile
435 440 445
Ser Asp Tyr Gly Leu Ala Gln Asn Leu Asn Ile Thr Arg Lys Glu Ala
450 455 460
Ala Glu Phe Ile Glu Arg Tyr Phe Ala Ser Phe Pro Gly Val Lys Gln
465 470 475 480
Tyr Met Glu Asn Ile Val Gln Glu Ala Lys Gln Lys Gly Tyr Val Thr
485 490 495
Thr Leu Leu His Arg Arg Arg Tyr Leu Pro Asp Ile Thr Ser Arg Asn
500 505 510
Phe Asn Val Arg Ser Phe Ala Glu Arg Thr Ala Met Asn Thr Pro Ile
515 520 525
Gln Gly Ser Ala Ala Asp Ile Ile Lys Lys Ala Met Ile Asp Leu Ala
530 535 540
Ala Arg Leu Lys Glu Glu Gln Leu Gln Ala Arg Leu Leu Leu Gln Val
545 550 555 560
His Asp Glu Leu Ile Leu Glu Ala Pro Lys Glu Glu Ile Glu Arg Leu
565 570 575
Cys Glu Leu Val Pro Glu Val Met Glu Gln Ala Val Thr Leu Arg Val
580 585 590
Pro Leu Lys Val Asp Tyr His Tyr Gly Pro Thr Trp Tyr Asp Ala Lys
595 600 605
<210> 5
<211> 2010
<212> DNA
<213> Artificial Sequence
<400> 5
catatgatgg caaccgtaaa gttcaagtac aaaggcgaag aaaaagaggt agacatctcc 60
aagatcaaga aagtatggcg tgtgggcaag atgatctcct tcacctacga cgagggcggt 120
ggcaagaccg gccgtggtgc ggtaagcgaa aaggacgcgc cgaaggagct gctgcagatg 180
ctggagaagc agaaaaaggg aggcggaggc tcaggaggcg gaggctcagg aggcggaggc 240
tcagaagggg agaaaccgct tgaggagatg gagtttgcca tcgttgacgt cattaccgaa 300
gagatgcttg ccgacaaggc agcgcttgtc gttgaggtga tggaagaaaa ctaccacgat 360
gccccgattg tcggaatcgc actagtgaac gagcatgggc gattttttat gcgcccggag 420
accgcgctgg ctgattcgca atttttagca tggcttgccg atgaaacgaa gaaaaaaagc 480
atgtttgacg ccaagcgggc agtcgttgcc ttaaagtgga aaggaattga gcttcgcggc 540
gtcgcctttg atttattgct cgctgcctat ttgctcaatc cggctcaaga tgccggcgat 600
atcgctgcgg tggcgaaaat gaaacaatat gaagcggtgc ggtcggatga agcggtctat 660
ggcaaaggcg tcaagcggtc gctgccggac gaacagacgc ttgctgagca tctcgttcgc 720
aaagcggcag ccatttgggc gcttgagcag ccgtttatgg acgatttgcg gaacaacgaa 780
caagatcaat tattaacgaa gcttgagcag ccgctggcgg cgattttggc tgaaatggaa 840
ttcactgggg tgaacgtgga tacaaagcgg cttgaacaga tgggttcgga gctcgccgaa 900
caactgcgtg ccatcgagca gcgcatttac gagctagccg gccaagagtt caacattaac 960
tcaccaaaac agctcggagt cattttattt gaaaagctgc agctaccggt gctgaagaag 1020
acgaaaacag gctattcgac ttcggctgat gtgcttgaga agcttgcgcc gcatcatgaa 1080
atcgtcgaaa acattttgca ttaccgccag cttggcaaac tgcaatcaac gtatattgaa 1140
ggattgttga aagttgtgcg ccctgatacc ggcaaagtgc atacgatgtt caaccaagcg 1200
ctgacgcaaa ctgggcggct cagctcggcc gagccgaact tgcaaaacat tccgattcgg 1260
ctcgaagagg ggcggaaaat ccgccaagcg ttcgtcccgt cagagccgga ctggctcatt 1320
ttcgccgccg attactcaca aattgaattg cgcgtcctcg cccatatcgc cgatgacgac 1380
aatctaattg aagcgttcca acgcgatttg gatattcaca caaaaacggc gatggacatt 1440
ttccatgtga gcgaagagga agtcacggcc aacatgcgcc gccaggcaaa ggccgttaac 1500
ttcggtatcg tttacggaat tagcgattac ggattggcgc aaaacttgaa cattacgcgc 1560
aaagaagctg ccgaatttat cgaacgttac ttcgccagct ttccgggcgt aaagcagtat 1620
atggaaaaca ttgtgcaaga agcgaaacag aaaggatatg tgacaacgct gttgcatcgg 1680
cgccgctatt tgcctgatat tacaagccgc aatttcaacg tccgcagttt tgcagagcgg 1740
acggccatga acacgccaat tcaaggaagc gccgctgaca ttattaaaaa agcgatgatt 1800
gatttagcgg cacggctgaa agaagagcag cttcaggctc gtcttttgct gcaagtgcat 1860
gacgagctca ttttggaagc gccaaaagag gaaattgagc gattatgtga gcttgttccg 1920
gaagtgatgg agcaggccgt tacgctccgc gtgccgctga aagtcgacta ccattacggc 1980
ccaacatggt atgatgccaa ataaggatcc 2010
<210> 8
<211> 687
<212> PRT
<213> Artificial Sequence
<400> 8
Met Gly His His His His His His His His His His Ser Ser Gly His
1 5 10 15
Ile Glu Gly Arg His Met Met Ala Thr Val Lys Phe Lys Tyr Lys Gly
20 25 30
Glu Glu Lys Glu Val Asp Ile Ser Lys Ile Lys Lys Val Trp Arg Val
35 40 45
Gly Lys Met Ile Ser Phe Thr Tyr Asp Glu Gly Gly Gly Lys Thr Gly
50 55 60
Arg Gly Ala Val Ser Glu Lys Asp Ala Pro Lys Glu Leu Leu Gln Met
65 70 75 80
Leu Glu Lys Gln Lys Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
85 90 95
Gly Gly Gly Gly Ser Glu Gly Glu Lys Pro Leu Glu Glu Met Glu Phe
100 105 110
Ala Ile Val Asp Val Ile Thr Glu Glu Met Leu Ala Asp Lys Ala Ala
115 120 125
Leu Val Val Glu Val Met Glu Glu Asn Tyr His Asp Ala Pro Ile Val
130 135 140
Gly Ile Ala Leu Val Asn Glu His Gly Arg Phe Phe Met Arg Pro Glu
145 150 155 160
Thr Ala Leu Ala Asp Ser Gln Phe Leu Ala Trp Leu Ala Asp Glu Thr
165 170 175
Lys Lys Lys Ser Met Phe Asp Ala Lys Arg Ala Val Val Ala Leu Lys
180 185 190
Trp Lys Gly Ile Glu Leu Arg Gly Val Ala Phe Asp Leu Leu Leu Ala
195 200 205
Ala Tyr Leu Leu Asn Pro Ala Gln Asp Ala Gly Asp Ile Ala Ala Val
210 215 220
Ala Lys Met Lys Gln Tyr Glu Ala Val Arg Ser Asp Glu Ala Val Tyr
225 230 235 240
Gly Lys Gly Val Lys Arg Ser Leu Pro Asp Glu Gln Thr Leu Ala Glu
245 250 255
His Leu Val Arg Lys Ala Ala Ala Ile Trp Ala Leu Glu Gln Pro Phe
260 265 270
Met Asp Asp Leu Arg Asn Asn Glu Gln Asp Gln Leu Leu Thr Lys Leu
275 280 285
Glu Gln Pro Leu Ala Ala Ile Leu Ala Glu Met Glu Phe Thr Gly Val
290 295 300
Asn Val Asp Thr Lys Arg Leu Glu Gln Met Gly Ser Glu Leu Ala Glu
305 310 315 320
Gln Leu Arg Ala Ile Glu Gln Arg Ile Tyr Glu Leu Ala Gly Gln Glu
325 330 335
Phe Asn Ile Asn Ser Pro Lys Gln Leu Gly Val Ile Leu Phe Glu Lys
340 345 350
Leu Gln Leu Pro Val Leu Lys Lys Thr Lys Thr Gly Tyr Ser Thr Ser
355 360 365
Ala Asp Val Leu Glu Lys Leu Ala Pro His His Glu Ile Val Glu Asn
370 375 380
Ile Leu His Tyr Arg Gln Leu Gly Lys Leu Gln Ser Thr Tyr Ile Glu
385 390 395 400
Gly Leu Leu Lys Val Val Arg Pro Asp Thr Gly Lys Val His Thr Met
405 410 415
Phe Asn Gln Ala Leu Thr Gln Thr Gly Arg Leu Ser Ser Ala Glu Pro
420 425 430
Asn Leu Gln Asn Ile Pro Ile Arg Leu Glu Glu Gly Arg Lys Ile Arg
435 440 445
Gln Ala Phe Val Pro Ser Glu Pro Asp Trp Leu Ile Phe Ala Ala Asp
450 455 460
Tyr Ser Gln Ile Glu Leu Arg Val Leu Ala His Ile Ala Asp Asp Asp
465 470 475 480
Asn Leu Ile Glu Ala Phe Gln Arg Asp Leu Asp Ile His Thr Lys Thr
485 490 495
Ala Met Asp Ile Phe His Val Ser Glu Glu Glu Val Thr Ala Asn Met
500 505 510
Arg Arg Gln Ala Lys Ala Val Asn Phe Gly Ile Val Tyr Gly Ile Ser
515 520 525
Asp Tyr Gly Leu Ala Gln Asn Leu Asn Ile Thr Arg Lys Glu Ala Ala
530 535 540
Glu Phe Ile Glu Arg Tyr Phe Ala Ser Phe Pro Gly Val Lys Gln Tyr
545 550 555 560
Met Glu Asn Ile Val Gln Glu Ala Lys Gln Lys Gly Tyr Val Thr Thr
565 570 575
Leu Leu His Arg Arg Arg Tyr Leu Pro Asp Ile Thr Ser Arg Asn Phe
580 585 590
Asn Val Arg Ser Phe Ala Glu Arg Thr Ala Met Asn Thr Pro Ile Gln
595 600 605
Gly Ser Ala Ala Asp Ile Ile Lys Lys Ala Met Ile Asp Leu Ala Ala
610 615 620
Arg Leu Lys Glu Glu Gln Leu Gln Ala Arg Leu Leu Leu Gln Val His
625 630 635 640
Asp Glu Leu Ile Leu Glu Ala Pro Lys Glu Glu Ile Glu Arg Leu Cys
645 650 655
Glu Leu Val Pro Glu Val Met Glu Gln Ala Val Thr Leu Arg Val Pro
660 665 670
Leu Lys Val Asp Tyr His Tyr Gly Pro Thr Trp Tyr Asp Ala Lys
675 680 685
- 通过功能结构域嫁接提高聚合酶大片段活性的方法及应用
- 一种通过点突变提高大片段嗜热脂肪地芽孢杆菌DNA聚合酶活性的方法及应用
