Exendin-4融合基因修饰的MSC及其应用
文献发布时间:2023-06-19 13:45:04
技术领域
本发明涉及Exendin-4融合基因修饰的MSC及其应用,属于基因工程技术领域。
背景技术
2型糖尿病(T2D)是一种慢性进行性疾病,其特征是由胰岛素抵抗、受损的细胞功能逐年下降、高血凝素血症和不适当的肝半乳糖生成等因素共同导致的血糖调节缺陷。进食会刺激多种胃肠道激素的分泌,包括胰岛素、胰高血糖素、GIP、GLP-1等,这些激素参与调节肠道运动、胃酸和胰酶的分泌、胆囊收缩和营养吸收,肠道激素也通过刺激胰腺内分泌分泌胰岛素来促进消化吸收葡萄糖。
GLP-1是通过与结构上不同的G蛋白偶联受体(GPCRs)结合来发挥作用的。GLP-1受体(GLP-1R)在胰岛α和β细胞以及周围组织中表达,包括中枢和周围神经系统、心脏、肾脏、肺和胃肠道。GLP-1还可抑制胰高血糖素分泌、胃排空和食物摄入,并通过神经机制促进葡萄糖的增强处理。目前,针对GLP1开发的降糖药物主要是抑制GLP1降解的DPP4抑制剂和GLP1类似物,但都面临半衰期短的问题,患者用药频繁。虽然输注天然GLP-1在降低2型糖尿病患者血糖方面非常有效,但单次皮下注射这种天然肽会在几分钟内迅速降解并从血液循环中消失。因此,大多数开发GLP-1模拟剂的制药方法都集中在长效降解耐药肽的开发上。
近年来,间充质干细胞(mesenchymal stem cells, MSCs)的移植治疗糖尿病受到越来越多的关注,这种细胞容易获得,且易增殖,移植后可以存活较长时间。鉴于这样的移植已在各种应用中被证明是安全的,且MSCs具有有效的免疫调节和趋化现象的属性,利用这些细胞作为载体,为治疗目的运送或生产有益的蛋白质,一直是许多研究人员关注的焦点。
CN109929806A提供了一种双基因修饰的干细胞,采用的基因为FGF21和GLP-1或其变体;CN104805058A同样使用的是GLP-1基因修饰的间充质干细胞;WO2021/042321A1专利单独采用的Exendin-4基因修饰的间充质干细胞,没有与Fc结构域相连形成融合蛋白以延长其半衰期。
开发出一种半衰期长的GLP-1蛋白相关药物,减少糖尿病患者的用药,是亟待解决的技术问题。
发明内容
本发明针对现有技术存在的不足,本发明提供Exendin-4融合基因修饰的MSC及其应用,实现以下发明目的:Exendin-4融合基因修饰的MSC的Exendin-4的表达量高,且半衰期长,减少糖尿病患者的用药,延长用药时间。
为解决上述技术问题,本发明采取以下技术方案:
Exendin-4融合基因修饰的MSC,所述Exendin-4融合基因为Exendin-4与IgG2、Albumin-1、Albumin、Ig kappa chain之一串联得到。
以下是对上述技术方案的进一步改进:
所述Exendin-4的核酸人工序列为SEQ ID NO.3所示;所述IgG2核酸人工序列为SEQ ID NO.4所示,所述Albumin-1的核酸人工序列为SEQ ID NO.5所示,所述Albumin的核酸人工序列为SEQ ID NO.6所示,所述Ig kappa chain的核酸人工序列为SEQ ID NO.7所示。
所述Exendin-4融合基因为Exendin-4-IgG2、Exendin-4-Albumin-1、Exendin-4-Albumin、Exendin-4-Ig kappa chain中的一种;所述Exendin-4-IgG2的核酸序列为SEQ IDNO.1所示;所述Exendin-4-Albumin-1的核酸序列为SEQ ID NO.8所示;所述Exendin-4-Albumin的核酸序列为SEQ ID NO.9所示;所述Exendin-4-Ig kappa chain的核酸序列为SEQ ID NO.10所示。
所述Exendin-4融合基因修饰的MSC的制备方法包括合成融合基因、制备重组质粒、重组质粒转染间充质干细胞、筛选。
所述制备重组质粒,采用载体pIRES2-eGFP负载Exendin-4融合基因,转化到E.coli,再提取得到重组质粒,浓度为855-956 ng/µL。
所述重组质粒转染间充质干细胞,每5×10
所述筛选,电转完成后,每孔加入2mL含有400µg/mL G418的筛选培养基,每隔3天更换一次筛选培养基,直至细胞融合度达到60%,对细胞进行传代到小瓶子中,更换10mL含有200µg/mL G418的维持培养基,对细胞进行扩增培养。
所述筛选培养基为添加400µg/mL G418的含有血小板裂解物的Hyclone间充质培养基;所述维持培养基为添加200µg/mL G418的含有血小板裂解物的Hyclone间充质培养基。
Exendin-4融合基因修饰的MSC的应用,所述Exendin-4融合基因修饰的MSC在制备GLP-1蛋白相关药物的应用。
与现有技术相比,本发明取得以下有益效果:
(1)本发明对Exendin-4基因进行氨基酸和密码子优化,可以提高Exendin-4融合基因修饰的MSC的Exendin-4表达量。
(2)本发明采用Exendin-4和IgG2、Albumin-1、Albumin、Ig kappa chain之一得到融合基因,可以延长Exendin-4蛋白的半衰期,用药后第7天仍然可以保持体内Exendin-4蛋白的高浓度。
附图说明
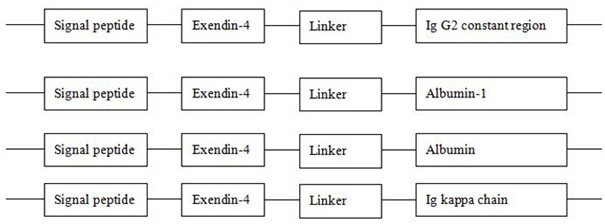
图1为Exendin-4融合基因的结构图;
图2为实施例2中双酶切验证重组表达载体pIRES2-eGFP-Exendin-4-IgG2的电泳图;
图3为P3代脐带间充质干细胞显微镜图;
图4为流式细胞术检测间充质干细胞的CD105标志物的流式图;
图5为流式细胞术检测间充质干细胞的CD90标志物的流式图;
图6为流式细胞术检测间充质干细胞的CD73标志物的流式图;
图7为流式细胞术检测间充质干细胞的CD34标志物的流式图;
图8为流式细胞术检测间充质干细胞的HLA-DR标志物的流式图;
图9为流式细胞仪检测Exendin-4-IgG2-MSC中GFP的表达率的流式图;
图10为流式细胞仪检测Exendin-4-Albumin-1-MSC中GFP的表达率的流式图;
图11为流式细胞仪检测Exendin-4-Albumin-MSC中GFP的表达率的流式图;
图12为流式细胞仪检测Exendin-4-Ig kappa chain-MSC中GFP的表达率的流式图;
图13为流式细胞仪检测Exendin-4-MSC中GFP的表达率的流式图;
图14为Exendin-4的RNA相对表达量的柱状图;
图15为葡萄糖刺激后MIN6细胞中胰岛素的分泌量的折线图。
具体实施方式
实施例1 融合基因的结构
修饰间充质干细胞的融合基因(Exendin-4-IgG2),包括信号肽核酸人工序列、Exendin-4核酸人工序列、Linker核酸人工序列和IgG2核酸人工序列,其中IgG2核酸人工序列可以由Albumin-1、Albumin、Ig kappa chain代替,得到Exendin-4-Albumin-1融合基因、Exendin-4-Albumin融合基因、Exendin-4-Ig kappa chain融合基因,如图1所示。Exendin-4-IgG2融合基因由信号肽核酸人工序列、Exendin-4核酸人工序列、Linker核酸人工序列和IgG2核酸人工序列依次串联而成,其核酸序列为SEQ ID NO.1。其中,所述信号肽核酸人工序列为SEQ ID NO.2;所述Exendin-4核酸人工序列为SEQ ID NO.3;所述IgG2核酸人工序列为SEQ ID NO.4,所述Albumin-1的核酸人工序列为SEQ ID NO.5,Albumin的核酸人工序列为SEQ ID NO.6,Ig kappa chain的核酸人工序列为SEQ ID NO.7;所述Exendin-4-Albumin-1融合基因的核酸序列为SEQ ID NO.8;所述Exendin-4-Albumin融合基因的核酸序列为SEQ ID NO.9;所述Exendin-4-Ig kappa chain融合基因的核酸序列为SEQ IDNO.10。
实施例2 重组质粒的制备
本实施例是以IgG2为例进行说明,当采用Albumin-1、Albumin、Ig kappa chain中其他的任一个时,其制备方法基本相同,在此不做过多赘述。
将融合基因(Exendin-4-IgG2)委托上海捷瑞生物工程有限公司合成,将合成的融合基因序列用EcoRI 、BamHI进行双酶切,获得具有粘性末端的目的片段。同时将载体pIRES2-eGFP(购自上海泽叶生物科技有限公司)进行EcoRI 、BamHI双酶切,获得线性化载体片段,用T4连接酶连接目的片段和线性化载体片段,转化到E.coli(Top10),进行酶切验证(见图2),经测序正确后,使用OMEGA公司的质粒提取试剂盒提取质粒,获得重组表达载体pIRES2-eGFP-Exendin-4-IgG2、pIRES2-eGFP-Exendin-4-Albumin-1、pIRES2-eGFP-Exendin-4-Albumin、pIRES2-eGFP-Exendin-4-Ig kappa chain、pIRES2-eGFP-Exendin-4。本发明中提取到的重组表达载体pIRES2-eGFP-Exendin-4-IgG2的浓度为950ng/µL,pIRES2-eGFP-Exendin-4-Albumin-1的浓度为870ng/µL,pIRES2-eGFP-Exendin-4-Albumin的浓度为924ng/µL,pIRES2-eGFP-Exendin-4-Ig kappa chain的浓度为855ng/µL,pIRES2-eGFP-Exendin-4的浓度为956ng/µL。
实施例3 脐带间充质干细胞的制备
取医院捐献的新生儿脐带,在超净工作台中先用75wt%酒精消毒两次,放在培养皿中,用镊子剥离出其中的华氏胶组织,用剪刀剪碎至0.5mm
实施例4 重组质粒转染间充质干细胞并筛选
电转前准备工作:将T75瓶中的处于对数期(实施例3得到的P3代的脐带间充质干细胞)的脐带间充质干细胞用TrypLE™ Select 酶 (购自gibco,货号12563029)消化后,收集到50mL离心管中,400g离心5min,去掉上清,备用。将电转液(购自LONZA的 P1 PrimaryCell 4D X Kit L 核转试剂盒,V4XP-1012)从4℃冰箱中拿出,取1.5mL电转液置于离心管中恢复至室温,剩余的电转液放回4℃冰箱;取7.5×10
将电转杯放入LONZA 4D电转仪中,选择间充质干细胞电转程序进行电转,电转完成后将细胞立即转移至预热的间充质干细胞培养基中,每孔中加入5×10
所述预热的间充质培养基预先加入六孔板中,每孔中加入2mL培养基,并预热到37℃;
所述间充质干细胞培养基为实施例3中的含有血小板裂解物的Hyclone间充质培养基(购自Hyclone)。
换液后继续培养24h后,弃掉六孔板中的培养基,加入含有400µg/mL G418的筛选培养基(每孔加入2mL筛选培养基,即添加G418的含有血小板裂解物的Hyclone间充质培养基),每隔3天更换一次筛选培养基。直至细胞融合度达到60%,对细胞进行传代到小瓶子中,更换10mL含有200µg/mL G418的维持培养基(添加200µg/mL G418的含有血小板裂解物的Hyclone间充质培养基),对细胞进行扩增培养。同时利用流式细胞仪检测筛选后的间充质干细胞中GFP的表达率,其中Exendin-4-IgG2-MSC的GFP表达率为93.1%,Exendin-4-Albumin-1-MSC的GFP表达率为94.2%,Exendin-4-Albumin-MSC的GFP表达率为94.5%,Exendin-4-Ig kappa chain-MSC的GFP表达率为93.6%,Exendin-4-MSC的GFP表达率为95.6%(见图9-13)。
实施例5 融合基因修饰的MSC中Exendin-4的基因表达量
取相同数量的不同细胞分别接种到T75瓶中,每瓶加入10mL间充质干细胞培养基,培养48h,收集细胞。利用QIAGEN的RNeasy Mini Kit(货号:74104)试剂盒提取细胞的总RNA,根据反转录试剂盒(购自艾科瑞,货号:AG11728)说明书进行反转录,利用SYBR green试剂盒(购自索莱宝,货号:SR2110-50)进行实时荧光定量PCR反应,检测5种基因修饰的间充质干细胞与正常间充质干细胞中Exendin-4的基因表达量。
实验结果如图14所示,经过基因修饰的间充质干细胞与正常间充质干细胞相比,其Exendin-4的基因表达量均明显上升,其中Exendin-4-IgG2-MSC中Exendin-4的表达量为正常间充质干细胞的2958倍,Exendin-4-Albumin-1-MSC中Exendin-4的表达量为正常间充质干细胞的2832倍,Exendin-4-Albumin-MSC中Exendin-4的表达量为正常间充质干细胞的2896倍,Exendin-4-Ig kappa chain-MSC中Exendin-4的表达量为正常间充质干细胞的2780倍,Exendin-4-MSC中Exendin-4的表达量为正常间充质干细胞的3050倍。
实施例6 融合基因修饰的MSC中Exendin-4的蛋白表达量
将不同融合基因修饰的MSC以1×10
(1)从室温平衡 60min 后的铝箔袋中取出所需板条,剩余板条用自封袋密封放回4℃。
(2)设置标准品孔,空白孔和样本孔,标准品孔各加不同浓度的标准品50μL。
(3)样本孔中加待测样本 50μL;空白孔加样本稀释液 50μL。
(4)空白孔,标准品孔和样本孔中每孔加入辣根过氧化物酶 (HRP)标记的检测抗体100μL,用封板膜封住反应孔,37℃水浴锅或恒温箱温育60min。
(5)弃去液体,吸水纸上拍干,每孔加满洗涤液350μL, 静置 1min,甩去洗涤液,吸水纸上拍干,如此重复洗板5次。
(6)每孔加入底物 A、B 各 50μL,37℃避光孵育15min。
(7)每孔加入终止液 50μL,15min 内,在 450nm波长处 测定各孔的OD值。
表1不同融合基因修饰的MSC分泌Exendin-4的量
结果如表1所示,随着时间的延长,不同融合基因修饰的间充质干细胞其分泌的Exendin-4蛋白的量是不断增加的,在96h时,Exendin-4-IgG2-MSC分泌Exendin-4蛋白的量达到356.54ng/mL。
实施例7 融合基因修饰的MSC对胰岛素分泌的影响
将MIN6细胞(购自上海通派生物科技有限公司)铺板在六孔板中,共铺板36孔,分为六组,每组6孔,每孔接种密度为1×10
24h后全部孔中加入终浓度为10mM的葡萄糖刺激细胞分泌,分别在30min、60min、12h、1d、3d、5d时各取一孔进行检测,用小鼠胰岛素ELISA试剂盒(购自北京索莱宝生物科技有限公司,货号:SEKM0141)测定细胞培养上清中胰岛素的分泌量。
如图15所示,胰岛素的分泌量在0.5h后开始升高,在24h时达到最高,随着时间的延长,胰岛素的分泌量缓慢下降。与普通MSC细胞处理的MIN6对照组相比,融合基因修饰的间充质干细胞促进胰岛素的分泌。在24h时,Exendin-4-IgG2-MSC实验组中胰岛素的分泌量为446ng,Exendin-4-Albumin-1-MSC实验组中胰岛素的分泌量为432ng,Exendin-4-Albumin-MSC实验组中胰岛素的分泌量为421ng,Exendin-4-Ig kappa chain-MSC实验组中胰岛素的分泌量为438ng,Exendin-4-MSC实验组中胰岛素的分泌量为426ng。
实施例8 融合基因修饰的MSC对小鼠胰岛瘤细胞凋亡的影响
将MSCs和MIN6细胞分别铺板在六孔板上,每种细胞铺板18孔,每孔接种密度为1×10
细胞培养过夜后,用终浓度为25mM的葡萄糖处理5天。然后用终浓度为0.125 mM的H
(1)用去离子水将10×Binding Buffer稀释成1×Binding Buffer;
(2)细胞收集。用TrypLE™ Select 酶消化收集后,于室温2000rpm离心5分钟,收集细胞;
(3)细胞洗涤,用预冷1×PBS(4℃)重悬细胞一次,2000rpm离心5分钟,洗涤细胞;
(4)加入300μL 的1×Binding Buffer 悬浮细胞;
(5)Annexin V-FITC标记:加入5μL的Annexin V-FITC混匀后,避光,室温孵育30分钟;
(6)PI标记:上机前5分钟再加入5μL的PI染色。
(7)上机前,补加200μL的1×Binding Buffer。
表2 MIN6和MSC细胞的凋亡率
结果如表2所示,融合基因修饰的MSC细胞与MIN6细胞共培养后,抑制MIN6的凋亡,提高MIN6细胞的存活率,但对普通的MSC细胞没有作用,说明Exendin-4基因抑制胰岛细胞的凋亡,提高存活率。
实施例9 融合基因修饰的MSC在小鼠体内Exendin-4的表达量
18-22g雄性BLAB/C小鼠(购自沈阳蓝谱达斯实验用品科技有限公司)于动物房饲养(室温23±2℃,湿度50%±10%),利用高脂饲料进行喂养,分别收集1×10
表3小鼠血清中Exendin-4的表达量
结果如表3所示,融合基因修饰的MSC注射的小鼠体内Exendin-4的表达量在第6天时达到高峰,Exendin-4-MSC注射的小鼠体内Exendin-4的表达量在第4天时达到高峰,随后逐渐下降。但Exendin-4-IgG2-MSC组Exendin-4的表达量下降的趋势明显要比Exendin-4-MSC组Exendin-4的表达量下降趋势慢。说明添加了IgG2等融合基因的Exendin-4的降解速度明显比Exendin-4单独基因的降解速度慢,延长Exendin-4蛋白的存在时间。
序列表
<110> 山东兴瑞生物科技有限公司
<120> Exendin-4融合基因修饰的MSC及其应用
<130> 2021
<160> 10
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1209
<212> DNA
<213> Homo sapiens
<400> 1
atgaaaatca tcctgtggct gtgtgttttt gggctgttcc ttgcaacttt attccctatc 60
agctggcaac atggtgaagg cacatttacc tctgacttgt caaagcagat ggaggaggaa 120
gcagtgcggt tatttattga gtggcttaag aacggaggac caagtagcgg ggcacctccg 180
tctaagaaaa agaaaaagaa aggaggagga ggaagcggag gaggaggaag cgcttcaact 240
aaaggcccgt ctgtgttccc tctggctcct tgttcaagat ctacatcaga atcaaccgcc 300
gcactcggct gtctggtgaa ggactatttt cctgaacccg ttaccgtttc ctggaacagc 360
ggtgcgctca ccagcggcgt gcacaccttc ccagctgtcc tacagtcctc aggactctac 420
tccctcagca gcgtggtgac cgtgccctcc agcaacttcg gcacccagac ctacacctgc 480
aacgtagatc acaagcccag caacaccaag gtggacaaga cagttgagcg caaatgttgt 540
gtcgagtgcc caccgtgccc agcaccacct gtggcaggac cgtcagtctt cctcttcccc 600
ccaaaaccca aggacaccct catgatctcc cggacccctg aggtcacgtg cgtggtggtg 660
gacgtgagcc acgaagaccc cgaggtccag ttcaactggt acgtggacgg cgtggaggtg 720
cataatgcca agacaaagcc acgggaggag cagttcaaca gcacgttccg tgtggtcagc 780
gtcctcaccg ttgtgcacca ggactggctg aacggcaagg agtacaagtg caaggtctcc 840
aacaaaggcc tcccagcccc catcgagaaa accatctcca aaaccaaagg gcagccccga 900
gaaccacagg tgtacaccct gcccccatcc cgggaggaga tgaccaagaa ccaggtcagc 960
ctgacctgcc tggtcaaagg cttctacccc agcgacatcg ccgtggagtg ggagagcaat 1020
gggcagccgg agaacaacta caagaccaca cctcccatgc tggactccga cggctccttc 1080
ttcctctaca gcaagctcac cgtggacaag agcaggtggc agcaggggaa cgtcttctca 1140
tgctccgtga tgcatgaggc tctgcacaac cactacacgc agaagagcct ctccctgtct 1200
ccgggtaaa 1209
<210> 2
<211> 69
<212> DNA
<213> Homo sapiens
<400> 2
atgaaaatca tcctgtggct gtgtgttttt gggctgttcc ttgcaacttt attccctatc 60
agctggcaa 69
<210> 3
<211> 132
<212> DNA
<213> Homo sapiens
<400> 3
catggtgaag gcacatttac ctctgacttg tcaaagcaga tggaggagga agcagtgcgg 60
ttatttattg agtggcttaa gaacggagga ccaagtagcg gggcacctcc gtctaagaaa 120
aagaaaaaga aa 132
<210> 4
<211> 978
<212> DNA
<213> Homo sapiens
<400> 4
gcttcaacta aaggcccgtc tgtgttccct ctggctcctt gttcaagatc tacatcagaa 60
tcaaccgccg cactcggctg tctggtgaag gactattttc ctgaacccgt taccgtttcc 120
tggaacagcg gtgcgctcac cagcggcgtg cacaccttcc cagctgtcct acagtcctca 180
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcaacttcgg cacccagacc 240
tacacctgca acgtagatca caagcccagc aacaccaagg tggacaagac agttgagcgc 300
aaatgttgtg tcgagtgccc accgtgccca gcaccacctg tggcaggacc gtcagtcttc 360
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacgtgc 420
gtggtggtgg acgtgagcca cgaagacccc gaggtccagt tcaactggta cgtggacggc 480
gtggaggtgc ataatgccaa gacaaagcca cgggaggagc agttcaacag cacgttccgt 540
gtggtcagcg tcctcaccgt tgtgcaccag gactggctga acggcaagga gtacaagtgc 600
aaggtctcca acaaaggcct cccagccccc atcgagaaaa ccatctccaa aaccaaaggg 660
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 720
caggtcagcc tgacctgcct ggtcaaaggc ttctacccca gcgacatcgc cgtggagtgg 780
gagagcaatg ggcagccgga gaacaactac aagaccacac ctcccatgct ggactccgac 840
ggctccttct tcctctacag caagctcacc gtggacaaga gcaggtggca gcaggggaac 900
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 960
tccctgtctc cgggtaaa 978
<210> 5
<211> 576
<212> DNA
<213> Homo sapiens
<400> 5
cgtggtgtgt tccgtcgtga tgctcacaaa tctgaagtag cacaccgttt caaagatctg 60
ggtgaggaaa actttaaagc gctggtcctg atcgccttcg cccagtatct gcagcagtgc 120
ccattcgagg accacgttaa gttggttaac gaggttactg agttcgctaa gacttgtgtt 180
gctgacgaat ccgctgagaa ctgtgataag tccttgcaca ctttgttcgg tgacaagttg 240
tgtactgttg ctactttgag agaaacttac ggtgagatgg ctgactgttg tgctaagcaa 300
gagcctgaga gaaacgagtg tttcttgcaa cacaaggacg acaacccaaa cttgccaaga 360
ttggttagac cagaggttga cgttatgtgt actgctttcc acgacaacga agagactttc 420
ttgaagaagt acttgtacga gatcgctaga agacacccat acttctacgc tccagagttg 480
ttgttcttcg ctaagagata caaggctgct ttcactgagt gttgtcaggc tgctgataag 540
gctgcttgtt tgttgccaaa gttggacgag ttgaga 576
<210> 6
<211> 1773
<212> DNA
<213> Homo sapiens
<400> 6
cgtggtgtgt tccgtcgtga tgctcacaaa tctgaagttg cgcaccgttt taaagacctg 60
ggcgaagaga actttaaagc cctggtactg atcgctttcg ctcaatacct gcaacagtgc 120
ccgttcgaag accacgtgaa actggttaac gaagtaactg aatttgcaaa aacctgcgtc 180
gctgacgaat ctgctgaaaa ctgtgacaag tctttgcaca ctttgttcgg tgacaagttg 240
tgtactgttg ctactttgag agaaacttac ggtgaaatgg ctgactgttg tgctaagcaa 300
gaaccagaaa gaaacgaatg tttcttgcaa cacaaggacg acaacccaaa cttgccaaga 360
ttggttagac cagaagtcga cgttatgtgt actgctttcc acgacaacga agaaactttc 420
ttgaagaagt acttgtacga aattgctaga agacacccat acttctacgc tccagaattg 480
ttgttcttcg ctaagagata caaggctgct ttcactgaat gttgtcaagc tgctgacaag 540
gctgcttgtt tgttgccaaa gttggacgaa ttgagagacg aaggtaaggc ttcttctgct 600
aagcaaagat tgaagtgtgc ttctttgcaa aagttcggtg aaagagcttt caaagcttgg 660
gctgttgcta gattgtctca aagattccca aaggctgaat ttgctgaagt ttctaagttg 720
gttactgact tgactaaggt tcacactgaa tgttgtcacg gtgacttgtt ggaatgtgct 780
gacgacagag ctgacttggc taagtacatt tgtgaaaacc aagactctat ttcttctaag 840
ttgaaggaat gttgtgaaaa gccattgttg gaaaagtctc actgtattgc tgaagttgaa 900
aacgacgaaa tgccagctga cttgccatct ttggctgctg acttcgttga atctaaggac 960
gtttgtaaga actacgctga agctaaggac gttttcttgg gtatgttctt gtacgaatac 1020
gctagaagac acccagacta ctctgttgtt ttgttgttga gattggctaa gacttacgaa 1080
actactttgg aaaagtgttg tgcggccgct gacccacacg aatgttacgc taaggttttc 1140
gacgaattta agccattggt tgaagaacca caaaacttga ttaagcaaaa ctgtgaattg 1200
ttcgaacaat tgggtgaata caagttccaa aacgctttgt tggttagata cactaagaag 1260
gttccacaag tttctactcc aactttggtt gaagtttcta gaaacttggg taaggttggt 1320
tctaagtgtt gtaagcaccc agaagctaag agaatgccat gtgctgaaga ctacttgtct 1380
gttgttttga accaattgtg tgttttgcac gaaaagactc cagtttctga cagagttact 1440
aagtgttgta ctgaatcttt ggttaacaga agaccatgtt tctctgcttt ggaagttgac 1500
gaaacttacg ttccaaagga atttaacgct gaaactttca ctttccacgc tgacatttgt 1560
actttgtctg aaaaggaaag acaaattaag aagcaaactg ctttggttga attggttaag 1620
cacaagccaa aggctactaa ggaacaattg aaggctgtta tggacgactt cgctgctttc 1680
gttgagaaat gctgcaaagc ggatgacaaa gaaacgtgct ttgcggaaga aggtaaaaaa 1740
ctggtagcgg cgtcccaagc agctctgggt ctg 1773
<210> 7
<211> 300
<212> DNA
<213> Homo sapiens
<400> 7
gctccgtctg ttttcatctt cccgccgtcc gatgaacagc tgaaatccgg tactgcgagc 60
gttgtttgtc tgctgaataa cttctatccc agagaggcca aagtacagtg gaaggtggat 120
aacgccctcc aatcgggtaa ctcccaggag agtgtcacag agcaggacag caaggacagc 180
acctacagcc tcagcagcac cctgacgctg agcaaagcag actacgagaa acacaaagtc 240
tacgcctgcg aagtcaccca tcagggcctg agctcgcccg tcacaaagag cttcaacagg 300
<210> 8
<211> 807
<212> DNA
<213> Homo sapiens
<400> 8
atgaaaatca tcctgtggct gtgtgttttt gggctgttcc ttgcaacttt attccctatc 60
agctggcaac atggtgaagg cacatttacc tctgacttgt caaagcagat ggaggaggaa 120
gcagtgcggt tatttattga gtggcttaag aacggaggac caagtagcgg ggcacctccg 180
tctaagaaaa agaaaaagaa aggaggagga ggaagcggag gaggaggaag ccgtggtgtg 240
ttccgtcgtg atgctcacaa atctgaagta gcacaccgtt tcaaagatct gggtgaggaa 300
aactttaaag cgctggtcct gatcgccttc gcccagtatc tgcagcagtg cccattcgag 360
gaccacgtta agttggttaa cgaggttact gagttcgcta agacttgtgt tgctgacgaa 420
tccgctgaga actgtgataa gtccttgcac actttgttcg gtgacaagtt gtgtactgtt 480
gctactttga gagaaactta cggtgagatg gctgactgtt gtgctaagca agagcctgag 540
agaaacgagt gtttcttgca acacaaggac gacaacccaa acttgccaag attggttaga 600
ccagaggttg acgttatgtg tactgctttc cacgacaacg aagagacttt cttgaagaag 660
tacttgtacg agatcgctag aagacaccca tacttctacg ctccagagtt gttgttcttc 720
gctaagagat acaaggctgc tttcactgag tgttgtcagg ctgctgataa ggctgcttgt 780
ttgttgccaa agttggacga gttgaga 807
<210> 9
<211> 2004
<212> DNA
<213> Homo sapiens
<400> 9
atgaaaatca tcctgtggct gtgtgttttt gggctgttcc ttgcaacttt attccctatc 60
agctggcaac atggtgaagg cacatttacc tctgacttgt caaagcagat ggaggaggaa 120
gcagtgcggt tatttattga gtggcttaag aacggaggac caagtagcgg ggcacctccg 180
tctaagaaaa agaaaaagaa aggaggagga ggaagcggag gaggaggaag ccgtggtgtg 240
ttccgtcgtg atgctcacaa atctgaagtt gcgcaccgtt ttaaagacct gggcgaagag 300
aactttaaag ccctggtact gatcgctttc gctcaatacc tgcaacagtg cccgttcgaa 360
gaccacgtga aactggttaa cgaagtaact gaatttgcaa aaacctgcgt cgctgacgaa 420
tctgctgaaa actgtgacaa gtctttgcac actttgttcg gtgacaagtt gtgtactgtt 480
gctactttga gagaaactta cggtgaaatg gctgactgtt gtgctaagca agaaccagaa 540
agaaacgaat gtttcttgca acacaaggac gacaacccaa acttgccaag attggttaga 600
ccagaagtcg acgttatgtg tactgctttc cacgacaacg aagaaacttt cttgaagaag 660
tacttgtacg aaattgctag aagacaccca tacttctacg ctccagaatt gttgttcttc 720
gctaagagat acaaggctgc tttcactgaa tgttgtcaag ctgctgacaa ggctgcttgt 780
ttgttgccaa agttggacga attgagagac gaaggtaagg cttcttctgc taagcaaaga 840
ttgaagtgtg cttctttgca aaagttcggt gaaagagctt tcaaagcttg ggctgttgct 900
agattgtctc aaagattccc aaaggctgaa tttgctgaag tttctaagtt ggttactgac 960
ttgactaagg ttcacactga atgttgtcac ggtgacttgt tggaatgtgc tgacgacaga 1020
gctgacttgg ctaagtacat ttgtgaaaac caagactcta tttcttctaa gttgaaggaa 1080
tgttgtgaaa agccattgtt ggaaaagtct cactgtattg ctgaagttga aaacgacgaa 1140
atgccagctg acttgccatc tttggctgct gacttcgttg aatctaagga cgtttgtaag 1200
aactacgctg aagctaagga cgttttcttg ggtatgttct tgtacgaata cgctagaaga 1260
cacccagact actctgttgt tttgttgttg agattggcta agacttacga aactactttg 1320
gaaaagtgtt gtgcggccgc tgacccacac gaatgttacg ctaaggtttt cgacgaattt 1380
aagccattgg ttgaagaacc acaaaacttg attaagcaaa actgtgaatt gttcgaacaa 1440
ttgggtgaat acaagttcca aaacgctttg ttggttagat acactaagaa ggttccacaa 1500
gtttctactc caactttggt tgaagtttct agaaacttgg gtaaggttgg ttctaagtgt 1560
tgtaagcacc cagaagctaa gagaatgcca tgtgctgaag actacttgtc tgttgttttg 1620
aaccaattgt gtgttttgca cgaaaagact ccagtttctg acagagttac taagtgttgt 1680
actgaatctt tggttaacag aagaccatgt ttctctgctt tggaagttga cgaaacttac 1740
gttccaaagg aatttaacgc tgaaactttc actttccacg ctgacatttg tactttgtct 1800
gaaaaggaaa gacaaattaa gaagcaaact gctttggttg aattggttaa gcacaagcca 1860
aaggctacta aggaacaatt gaaggctgtt atggacgact tcgctgcttt cgttgagaaa 1920
tgctgcaaag cggatgacaa agaaacgtgc tttgcggaag aaggtaaaaa actggtagcg 1980
gcgtcccaag cagctctggg tctg 2004
<210> 10
<211> 531
<212> DNA
<213> Homo sapiens
<400> 10
atgaaaatca tcctgtggct gtgtgttttt gggctgttcc ttgcaacttt attccctatc 60
agctggcaac atggtgaagg cacatttacc tctgacttgt caaagcagat ggaggaggaa 120
gcagtgcggt tatttattga gtggcttaag aacggaggac caagtagcgg ggcacctccg 180
tctaagaaaa agaaaaagaa aggaggagga ggaagcggag gaggaggaag cgctccgtct 240
gttttcatct tcccgccgtc cgatgaacag ctgaaatccg gtactgcgag cgttgtttgt 300
ctgctgaata acttctatcc cagagaggcc aaagtacagt ggaaggtgga taacgccctc 360
caatcgggta actcccagga gagtgtcaca gagcaggaca gcaaggacag cacctacagc 420
ctcagcagca ccctgacgct gagcaaagca gactacgaga aacacaaagt ctacgcctgc 480
gaagtcaccc atcagggcct gagctcgccc gtcacaaaga gcttcaacag g 531
