一种RNA中肌苷单碱基分辨率定量检测的方法
文献发布时间:2023-06-19 19:16:40
技术领域
本发明属于生物技术领域,具体涉及一种RNA中肌苷单碱基分辨率定量检测的方法。
背景技术
肌苷是最丰富的RNA修饰类型之一,也是核酸中发现的最早的碱基修饰之一。肌苷是由次黄嘌呤形成的嘌呤核苷,由作用于RNA的腺苷脱氨酶(adenosine deaminasesacting on RNA,ADARs)催化RNA双链区域中的腺苷水解脱氨基生成。这个过程也称为A-to-I RNA编辑。无脊椎动物和脊椎动物的正常行为和发育都需要ADARs酶的参与。
在RNA中,肌苷有两个主要功能。tRNA反密码子上的肌苷扩大了tRNA可以识别的三联体的范围。在mRNA中,肌苷可以改变双链区域的三维结构,从而影响与RNA结合蛋白的相互作用。肌苷由于可以和胞苷(cytidine,C)配对,因此被认为是鸟苷(guanosine,G),从而可以影响转录和翻译的准确性。因此肌苷是翻译效率和准确性的主要调节因子,有助于跨物种蛋白质组的多样性。A-to-I RNA编辑对于许多生物学过程至关重要,如免疫系统激活和神经功能。因此它的失调与自身免疫性疾病、神经系统疾病和几种类型的癌症直接相关。
鉴定肌苷的方法很多。液相色谱-电喷雾串联质谱(LC-ESI-MS/MS)是一种高效的定量方法,但它需要事先纯化大量RNA,并且依赖昂贵的仪器设备。而且LC-ESI-MS/MS法也不能提供肌苷修饰具体的位置信息。特异性RNA酶(如RNA酶T1和核酸内切酶V)可用于切割含肌苷的RNA,并且后续可通过凝胶电泳分离酶切后的RNA。尽管酶切法操作简单且花费少,但这种方法的通量很低并且不是完全定量的。鉴定A-to-I RNA编辑位点的最常用方法是将测序得到的cDNA序列与相应的基因组序列进行比较。由于肌苷可以与C配对,如果基因组序列中的腺苷(adenosine,A)在cDNA序列中的相应位点被G取代,则该位点可能是A-to-I RNA编辑位点。然而,这种方法难以排除由PCR错误、测序错误或背景干扰引起的A至G突变。研究者们开发了一种基于生物化学的方法,肌苷化学擦除测序法(inosine chemical erasuresequence,ICE-seq)。当总RNA中的肌苷与丙烯腈反应时,产物N1氰乙基肌苷(N1-cyanoethylinosine,ce
腺苷的脱氨作用改变了碱基的分子结构和氢键模式,导致肌苷与C配对,从而将这些位点重新编码为G。RNA中的肌苷通过迈克尔加成反应与丙烯腈进行氰乙基化,形成ce
发明内容
本发明的目的在于解决现有技术存在的问题,提供一种RNA中肌苷单碱基分辨率定量检测的分析方法,即一种能应用于RNA修饰单碱基分辨率定量领域中,具有高灵敏度、高选择性且操作简便的特异性位点的肌苷定量方法。
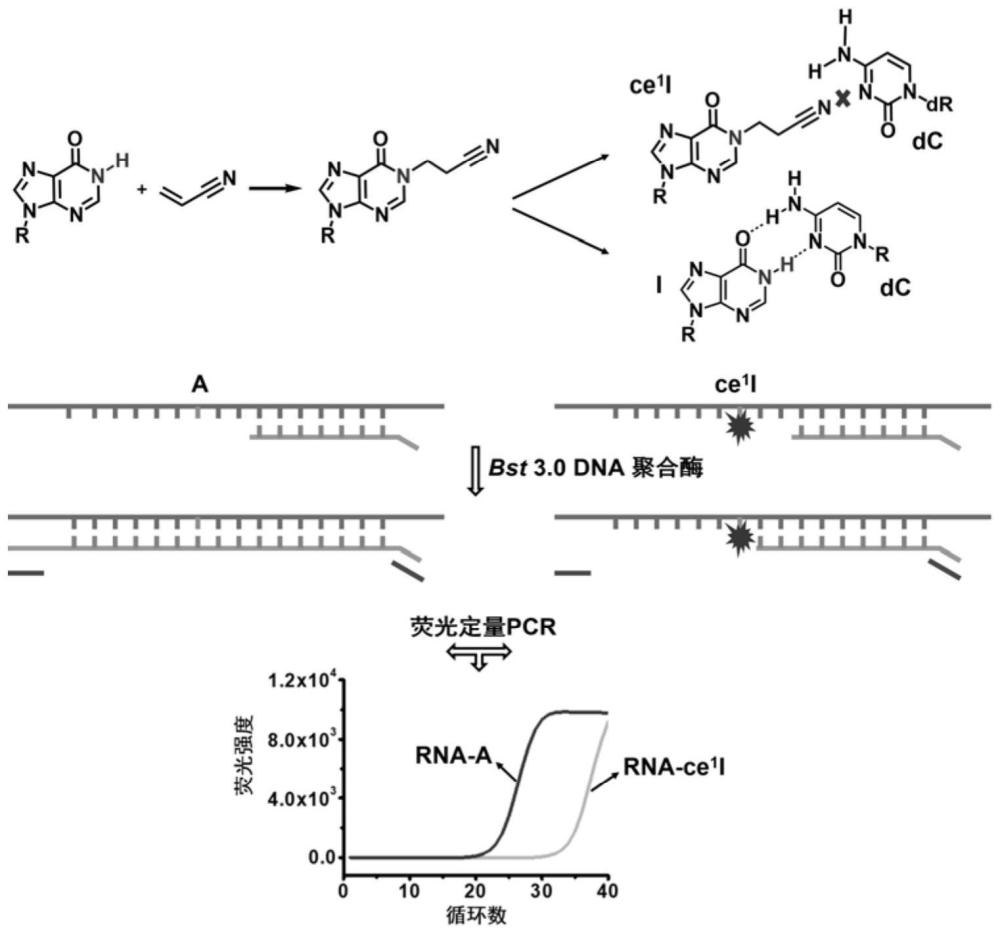
本发明原理见图1,本发明提出了一种通过丙烯腈标记介导的延伸停止法(ALES)对RNA中肌苷进行单碱基分辨率的位点特异性定量。在ALES法中,RNA中的肌苷(I)首先与丙烯腈氰乙基化形成ce
利用qPCR实现单碱基分辨率定量分析肌苷的原理:某位点处的肌苷修饰水平的计算公式为I%=(某RNA的总量-含有A的某RNA的量)/某RNA的总量。当用Bst 3.0DNA聚合酶扩增未经氰乙基化处理的RNA样品时,可通过qPCR测定某RNA的总量。当RNA样品用丙烯腈处理时,通过qPCR测定的就是含有A的某RNA的量。因此,只需要将样本一分为二,一份加丙烯腈处理,另一份不加丙烯腈处理,然后进行逆转录及qPCR,根据Ct值及绘制的定量方程就可以实现肌苷的单碱基分辨率定量分析。
本发明的目的通过下述技术方案实现:
一种RNA中肌苷单碱基分辨率定量检测的方法,包含如下步骤:
(1)将待检RNA均分为两份,一份用丙烯腈进行氰乙基化处理,一份不进行氰乙基化处理(只加对应的缓冲液,不加丙烯腈)。
(2)向两份样品中分别加入DNA探针,该探针一部分与目标RNA位点下游互补配对,另一部分用于后续的PCR扩增。用Bst 3.0DNA聚合酶对两份RNA样品分别进行逆转录。
(3)根据DNA探针的一部分及生成的DNA的下游序列设计qPCR引物,然后将逆转录的产物进行qPCR,计算RNA的量。未进行氰乙基化处理的RNA,通过qPCR测定的是RNA的总量;经氰乙基化处理的RNA,通过qPCR测定的是含有A的RNA的量。待检RNA某位点处的肌苷修饰水平通过下述公式计算:
肌苷修饰水平I%=(RNA的总量-含有A的RNA的量)/RNA的总量。
步骤(1)中,待检RNA用丙烯腈进行氰乙基化处理,包括如下步骤:
1)配制反应体系:30μL CE溶液(50%乙醇,1.1M三乙基乙酸铵缓冲液,pH 8.6),4μL丙烯腈,1-10μg待检RNA,总体积为38μL。反应体系置于70℃,反应45分钟。
2)将反应体系置于冰上结束反应,然后将体系置于氮吹仪下除去有机溶剂。
3)向上述体系中加入无酶水然后置于离心浓缩仪除去所有溶液,再次彻底去除反应体系中残留的有机试剂后可进行下一步操作。
步骤(3)中,RNA的量根据Ct值和RNA浓度的线性方程计算,所述的Ct值和RNA浓度的线性方程优选通过包括如下步骤的方法得到:用合成的含目标位点上下游序列的RNA链配制一系列不同浓度的标品进行Bst 3.0DNA聚合酶介导的逆转录及qPCR,然后绘制Ct值和RNA浓度的线性方程。
本发明方法简单、高效、灵敏度高、操作简单,可成功用于低丰度生物样品中肌苷的单碱基分辨率准确测定,为今后肌苷的功能研究和动态控制奠定了基础。本发明的优点及有益效果如下:
1)本发明中,方法操作简便,无需昂贵的仪器,无需繁琐的处理步骤,整个实验流程一天内即可完成,有利于推广使用。
2)本发明可以十分精确的对给定位点的肌苷水平进行定量分析。
3)本发明可快速估算不同位点的肌苷修饰水平,无需复杂的优化及定量过程。
4)本发明无需采用复杂且昂贵的第二代及第三代测序技术,只需采用简单的qPCR即可完成对肌苷的单碱基分辨率定量分析。
5)本发明可直接用于临床样本中重要肌苷位点修饰水平的分析,为深入研究肌苷在不同疾病中发挥的作用提供了有力的工具。
6)本发明涉及的方法能够广泛应用于不同生物来源肌苷修饰位点的定量研究,并有助于对肌苷的生物学功能进行更加深入的研究。
附图说明
图1为本发明中ALES法对RNA中肌苷单碱基分辨率定量检测的示意图。
图2为本发明中通过LC-ESI-MS/MS检测RNA-I链中肌苷的丙烯腈标记效率及琼脂糖凝胶电泳分析ce
图3为本发明中睡眠剥夺和对照小鼠不同组织的Ino80dos RNA在Chr1:63117284位点的肌苷定量分析结果。(A)通过ALES方法获得的RNA-A和链的标准曲线。(B)睡眠剥夺小鼠和对照小鼠肺、大脑和肾组织Ino80dos RNA中Chr1:63117284处肌苷的定量和统计分析。
图4为本发明中ALES法检测RNA中chr18:60954690和Chr5:34477808位点肌苷水平的结果。
具体实施方式
下面结合附图和具体实施例子对本发明进行进一步说明,但不应理解为对本发明的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。若未特别指明,实施例中所用的技术手段包括组织样本RNA的提取、RNA的酶解、qPCR以及琼脂糖凝胶电泳为本领域技术人员所熟知的常规手段。
实施例1:丙烯腈标记RNA的反应条件优化
配制总体积为38μL的反应体系:30μL CE溶液(50%乙醇,1.1M三乙基乙酸铵缓冲液,pH 8.6),4μL丙烯腈及0.76pmol的RNA-I链(5’-UUCACAAAGAUCCCCUUGCUUCUICCUCCUGGGUGCUGGAAUUAA-3’)。用LC-ESI-MS/MS评估不同反应温度和反应时间下合成的RNA-I链中肌苷的氰乙基化效率。
将上述反应体系分别在37℃、50℃、60℃及70℃四个不同温度下进行标记反应,结果显示在70℃时反应效率最高,因此选择70℃作为肌苷氰乙基化的反应温度。
将上述反应体系在70℃下反应15-60分钟,结果显示45分钟时丙烯腈标记效率达到平台期,因此选择45分钟作为肌苷氰乙基化的反应时间。
因此,最终优化的反应条件为上述反应体系在70℃下反应45分钟。
实施例2:通过丙烯腈标记介导的延伸停止法(ALES)
(1)分别向合成的RNA-A(5’-UUCACAAAGAUCCCCUUGCUUCUACCUCCUGGGUGCUGGAAUUAA-3’)或RNA-I链中加入30μL CE溶液(50%乙醇,1.1M三乙基乙酸铵缓冲液,pH8.6)和4μL丙烯腈及一定量的水至总体积为38μL,其中RNA-A和或RNA-I在反应体系中的浓度为20nM。将上述反应体系在70℃下孵育45分钟进行肌苷的氰乙基化。
(2)氰乙基化反应结束后,在氮吹仪下除去反应体系中的有机溶剂对RNA进行纯化。然后将两组纯化的RNA分别与10pmol DNA寡核苷酸探针RI(CCCATTCCGTGACTACGTGTCTTCCAGCACCCAGGAGG)混合。将混合物在85℃变性3分钟,然后缓慢冷却至25℃,使RNA模板和DNA寡核苷酸探针RI进行退火。然后,加入200nmol Mg
(3)最后,通过qPCR扩增上一步产物。qPCR体系为10μL
通过LC-ESI-MS/MS检测RNA-I链中肌苷的丙烯腈标记效率及琼脂糖凝胶电泳分析ce
实施例3:定量小鼠Ino80dos RNA Chr1:63117284位肌苷修饰水平
具体步骤如下:
(1)首先配制一系列不同浓度的RNA-A链进行Bst 3.0DNA聚合酶介导的逆转录及qPCR,绘制用于定量计算的线性方程。
(2)将8只C57BL/6J小鼠随机平分为两组,一组为正常组(control)另一组为急性睡眠剥夺组,control组正常饲养,急性睡眠剥夺组小鼠采用改良多平台水环境法进行连续睡眠剥夺24小时。改良多平台水环境法,其原理是利用小鼠进入快速动眼睡眠(REM)时,全身肌紧张性下降,使得面部接触或浸到水面而突然惊醒,此行为可反复发生,这样就选择性破坏了REM。其装置是由长宽高为40*28*19cm的睡眠剥夺箱和高于水面1cm、直径2.5cm、高度为5cm的20个圆柱形平台组成。
收集睡眠剥夺组和正常组小鼠的肺、肾和大脑组织,然后提取小鼠不同组织的大RNA(>200nt)进行ALES实验,分别得到加丙烯腈处理和不加丙烯腈处理组的Ct值。
其中,DNA探针:oligo-1:CCCATTCCGTGACTACGTGTCTTCCAGCACCCAGGAGG;
qPCR的引物:FP-7284:CGCTATTCACAAAGATCCCC、RP-7284:CCCATTCCGTGACTACGTGT。
(3)根据第一步的定量曲线及第二步得到的Ct值计算肌苷修饰的水平。
结果见图3,绘制的定量方程(图3A)为Ct=-3.435lgC
实施例4:判断其他位点是否含有肌苷修饰
采用ALES方法检测了文献中报道的另外两个肌苷位点(chr18:60954690和chr5:34477808)及一个不含肌苷修饰的对照位点(chr1:63117082)。实验方法同上,只是需要把实验中所用的DNA探针及引物换成对应位点的探针及引物。
检测chr18:60954690所用到的DNA探针和qPCR引物的序列分别为:
DNA探针oligo 690:CTGCCTCTAAAGAGCCAGGAAGTTTGAGCCCTGGGGCCTGGGGCT;
qPCR引物:FP-690:GGTCCCACATCCAAGTCTCATC,RP-690:TCCAAAGCCCTGCTCTTCAC。
检测chr5:34477808所用到的DNA探针和qPCR引物的序列分别为:
DNA探针oligo 808:CCAGTTTTCTGTTTATGTCAGAATCCCAAAGATGTGGGCTCTAAT;
qPCR引物:FP-808:CCTTCTGGTGGCAGCCTACA,RP-808:AGAGTGAGGGCAAACAGGTAAAGA。
检测chr1:63117082所用到的DNA探针和qPCR引物的序列分别为:
DNA探针oligo A:TAACACTCTTCACTTGCCAGGACCTGGAAGGCAGAG;
qPCR引物:RP-082:CGTAACACTCTTCACTTGCCAC,FP-082:ATGCCCTCGAACTCAGAGC。
结果见图4,丙烯腈处理组的Ct值显著增加,表明chr18:60954690和chr5:34477808位点存在肌苷修饰。相反,对照位点(Chr1:63117082)的Ct值在使用或不使用丙烯腈处理的RNA之间没有变化,表明Chr1:63117082处不存在肌苷修饰。通过方程1-2
- 一种简单定性/定量检测单碱基基因编辑技术工作效率的报告系统
- 一种基于全基因组测序检测腺嘌呤单碱基编辑系统脱靶效应的方法及其在基因编辑中的应用
- 一种基于单光镊介质微球高分辨率光学检测方法
- 一种内切酶辅助的RNA中肌苷修饰的单碱基分辨率定位分析方法
- 一种单碱基分辨率检测RNA中N6-异戊烯基腺嘌呤修饰的方法
