一种基于食蟹猴改造后的APOBEC3A的胞嘧啶碱基编辑器
文献发布时间:2023-06-19 19:30:30
技术领域
本发明涉及一种基于食蟹猴改造后的APOBEC3A的胞嘧啶碱基编辑器,属于基因工程技术领域。
背景技术
当前基因编辑技术不断快速发展,通过改造特定的基因,可以研究基因功能、遗传病的发病机理、开发新药以及用于基因治疗和改良作物等。近年来,基于CRISPR-Cas系统的各种衍生技术更是广泛应用在生命科学和医学领域,其中的单碱基编辑器(Base editor,BE)已成为基因编辑技术的重要组成部分。BE是在CRISPR-Cas9系统的基础上设计的,将野生型的Cas9蛋白进行改造后与胞嘧啶脱氨酶或腺嘌呤脱氨酶连接,然后通过sgRNA(smallguide RNA)的引导,在不产生DNA双链断裂(Double stranded break,DSB)的情况下直接进行单个碱基的编辑。BE主要分为两大类:胞嘧啶碱基编辑器(Cytosine Base Editor,CBE)和腺嘌呤碱基编辑器(Adenine Base Editor,ABE)。2016年Dvid R Liu.团队开发的CBE系统中采用的胞嘧啶脱氨酶可将非互补链中相应的胞嘧啶(Cytosine,C)经过脱氨基作用变为尿嘧啶(Uracil,U),在DNA复制或修复的过程中U被识别为胸腺嘧啶(Thymine,T),而互补链上相对应的鸟嘌呤(Guanine,G)将会变成腺嘌呤(Adenine,A),最终实现非互补链上C>T和互补链上G>A的转换。2017年,Dvid R Liu.团队又开发ABE系统,原理和CBE系统相似,只是将胞嘧啶脱氨酶换成了腺嘌呤脱氨酶,可以完成非互补链上A>G和互补链上T>C的编辑,进一步补充了单碱基编辑器的类型。
BE系统中的Cas9蛋白可进一步优化,一种是可结合靶基因但不切割靶基因的无核酸内切酶活性的dCas9(Catalytically dead Cas9),另一种是具有单链DNA切口酶活性的nCas9(Cas9 nickase),两种Cas9蛋白均不会产生DBS,从而避免了非同源末端连接(Non-homologous end-joining,NHEJ)的错配,也不用考虑同源重组修复(Homology-directedrepair,HDR)的低效率问题。随后又在BE中加入了尿嘧啶糖基化酶抑制剂(uracilglycosylase inhibitor,UGI)可抑制中间产物U的切除,提高了DNA链上C>T的编辑效率。目前,BE系统发展迅速,CBE系统已优化到第四代BE4,ABE系统也优化到ABE7.10版本,甚至开发出一些将CBE和ABE系统功能融合的双碱基编辑器,可在相同的靶位点同时实现C>T和A>G的转换。
传统的CBE系统中采用的胞嘧啶脱氨酶是大鼠的Apobec1(rA1),Jason M Gehrke等人2018年时尝试采用人的改造后的APOBEC3A(eA3A)去替换第三代CBE系统(BE3)中的rA1,随后他们将携带hA3A-N57G突变的BE3用于β-地中海贫血的治疗研究,将其编辑的精确度与BE3相比提高了将近40倍,但是在HBB
发明内容
现有的CBE系统中采用的胞嘧啶脱氨酶大多是大鼠的Apobec1(rA1),少数是人的APOBEC3A(hA3A)和APOBEC3G(hA3G),暂无非人灵长类动物食蟹猴的胞嘧啶脱氨酶在CBE系统中的应用。
为了解决上述问题,本发明的目的在于提供一种编辑效率高,产物纯度高,编辑窗口特异性强的基因编辑工具。
本发明的第一个目的是提供一种来源于食蟹猴的胞嘧啶脱氨酶突变体,所述突变体是在氨基酸序列如SEQ ID NO:1所示的亲本酶的基础上,删除第27~29连续的三位氨基酸。即,删除第27位丝氨酸,第28位缬氨酸和第29位精氨酸。
在一种实施方式中,所述突变体的氨基酸序列如SEQ ID NO:2所示。
本发明的第二个目的是提供编码上述胞嘧啶脱氨酶突变体的基因。
在一种实施方式中,所述基因的核苷酸序列如SEQ ID NO:3所示。
本发明的第三个目的是提供一种表达盒,所述表达盒包含上述编码上述胞嘧啶脱氨酶突变体的基因。
在一种实施方式中,所述表达盒还含有启动子、nCas9(D10A)、尿嘧啶DNA糖基化酶抑制剂(UGI)、核定位序列NLS、终止序列。
在一种实施方式中,所述启动子启动编码上述胞嘧啶脱氨酶突变体的基因表达,按连接顺序依次为启动子、所述胞嘧啶脱氨酶突变体、nCas9(D10A)、尿嘧啶DNA糖基化酶抑制剂(UGI)、核定位序列NLS和终止序列。
在一种实施方式中,所述启动子包括但不限于CMV启动子。
在一种实施方式中,所述nCas9(D10A)的核苷酸序列如SEQ ID NO:4所示,UGI的核苷酸序列如SEQ ID NO:5所示,所述NLS的核苷酸序列如SEQ ID NO:6所示,所述终止序列的核苷酸序列如SEQ ID NO:7所示。
本发明的第四个目的是提供一种基于食蟹猴改造后的胞嘧啶脱氨酶(APOBEC3A)的CBE单碱基编辑器BE4-mA3A-delSVR,所述CBE单碱基编辑系统包括四个部分,第一部分为转染效率指示部分,包含红色荧光蛋白(dTomato)和启动子;第二部分为sgRNA转录单元,包含插入sgRNA序列的携带框架及其启动子;第三部分包含上述表达盒,第四部分包含来自大肠杆菌素因子(colE1)的高拷贝复制起点ori以及氨苄青霉素抗性筛选基因AmpR。
在一种实施方式中,所述启动子包括CMV启动子和U6启动子。
在一种实施方式中,红色荧光蛋白的核苷酸序列如SEQ ID NO:8所示。
在一种实施方式中,所述sgRNA转录单元的核苷酸序列如SEQ ID NO:9所示。
本发明的第五个目的是提供上述CBE单碱基编辑器在基因编辑领域中的应用。
在本发明的一种实施方式中,根据靶基因设计sgRNA序列并插入至上述CBE单碱基编辑系统的sgRNA转录单元,获得具有特异性靶向基因的CBE单碱基编辑系统,将CBE单碱基编辑系统导入受体细胞中,实现靶标碱基C突变为T。
在本发明的一种实施方式中,所述细胞为真核生物细胞。
在本发明的一种实施方式中,所述真核生物细胞为哺乳动物细胞。
在本发明的一种实施方式中,所述哺乳动物细胞包括HEK293T细胞。
本发明的第六个目的是提供所述CBE单碱基编辑系统BE4-mA3A-delSVR的构建方法,具体的构建步骤如下:
(1)以pSpCas9(BB)-2A-dTomato(PX458)质粒为模板扩增得到红色荧光蛋白dTomato基因及其CMV增强子和启动子,获得载体骨架Part1;
(2)以BE3-rA1质粒为模板,扩增获得插入sgRNA序列的携带框架及其U6启动子,获得载体骨架Part2;
(3)以食蟹猴的cDNA为模板扩增获得APOBEC3A(mA3A-B5)的核苷酸序列,获得载体骨架Part3;
(4)以质粒BE4-rA1为模板扩增获得包括nCas9(D10A)、UGI、NLS、终止序列及CMV增强子和启动子基因序列的载体骨架Part4;
(5)以质粒BE4-rA1为模板扩增获得colE1高拷贝复制起点和氨苄青霉素抗性筛选基因及其氨苄青霉素启动子序列的载体骨架Part5;
(6)按照Part1、Part2、Part3、Part4、Part5的顺序连接五个片段,获得载体质粒BE4-mA3A-B5。
(7)以质粒pcDNA3.1-mA3A-GFP为模板,通过特定的引物进行PCR扩增获得删除SVR序列的mA3A序列(即mA3A-delSVR,见图5);
(8)将步骤(7)的删除SVR序列的mA3A序列与BamHI和SmaI双酶切去除食蟹猴的APOBEC3A(即mA3A)的BE4-mA3A-B5载体进行连接,获得载体质粒BE4-mA3A-delSVR。
(9)设计结合靶基因的sgRNA序列并插入到步骤(2)中的携带框架,获得可以针对不同靶基因相应位点的单碱基编辑器。
在一种实施方式中,pSpCas9(BB)-2A-dTomato(PX458)质粒的构建方式为在质粒pSpCas9(BB)-2A-GFP(PX458)的基础上将GFP替换为dTomato,所述dTomato的核苷酸序列如SEQ ID NO:8所示。
在一种实施方式中,质粒pcDNA3.1-mA3A-GFP的构建方式为以pcDNA3.1-2xFLAG-SREBP-2质粒模板,将mA3A片段和GFP片段与EcoRⅠ和XbaⅠ双酶切后的载体连接而成。
在一种实施方式中,mA3A片段(NCBI的参考序列)的核苷酸序列如SEQ ID NO:12所示。
在一种实施方式中,mA3A-delSVR片段的核苷酸序列如SEQ ID NO:3所示。
在一种实施方式中,GFP片段的核苷酸序列如SEQ IDNO:10所示。
本发明还提供上述突变体在基因编辑领域的应用。
本发明还提供上述表达盒在基因编辑领域的应用。
本发明的有益效果:
1、本发明构建的单碱基编辑器BE4-mA3A-delSVR上含有sgRNA转录单元,在利用其进行基因编辑时,只需将BE4-mA3A-delSVR质粒转染细胞,质粒上携带的靶向目的基因的sgRNA通过碱基互补配对原则引导融合蛋白结合到靶基因DNA区域,然后删除SVR序列的mA3A结合到由nCas9蛋白、sgRNA及基因组DNA形成的R-loop区的单链DNA(single-strandedDNA,ssDNA)处,将该ssDNA一定范围内(20bp原间隔子序列)的胞嘧啶(C)脱氨基变成尿嘧啶(U),进而通过DNA复制或修复将U转变为胸腺嘧啶(T),最终实现CG碱基对至TA碱基对的直接替换。此外,由于nCas9只具有单链DNA切口酶活性,不会在PAM(Protospacer AdjacentMotif)序列上游2-3碱基间将靶基因的DNA双链切断,从而不会形成DSB,进而大大降低脱靶效率。
2、本发明敲除了食蟹猴来源的胞嘧啶脱氨酶mA3A氨基酸序列的第27~29位氨基酸,成功构建了一种在编辑效率,产物纯度和编辑特异性都优于原始BE4-rA1的胞嘧啶碱基编辑器BE4-mA3A-delSVR,并将其成功用于HEK293T细胞中的site3、RNF2、EMX1、HEK2和HEK4基因中靶位点C>T的编辑。本发明构建的单碱基编辑器特异性的在第C5和C6位进行C>T转换,编辑效率和产物纯度较高。充分证明了改造后的mA3A-delSVR是优于rA1甚至原始mA3A的用于CBE基因编辑系统优化的胞嘧啶脱氨酶,从而为进一步优化和改造CBE系统提供了一个更加灵活可控的原型以及新的思路和方向,丰富了基因编辑的工具包,也为遗传病的基因治疗提供一个潜在的工具。(备注:site3、HEK2和HEK4为已发表文章中的命名。site3的基因名为LINC01509、HEK2的基因名为AC114971.1和HEK4的基因名为DNMT3B。)
附图说明
图1:胞嘧啶碱基编辑器BE4-mA3A-B5的质粒图谱;
图2:过表达质粒pcDNA3.1-mA3A-GFP的质粒图谱。
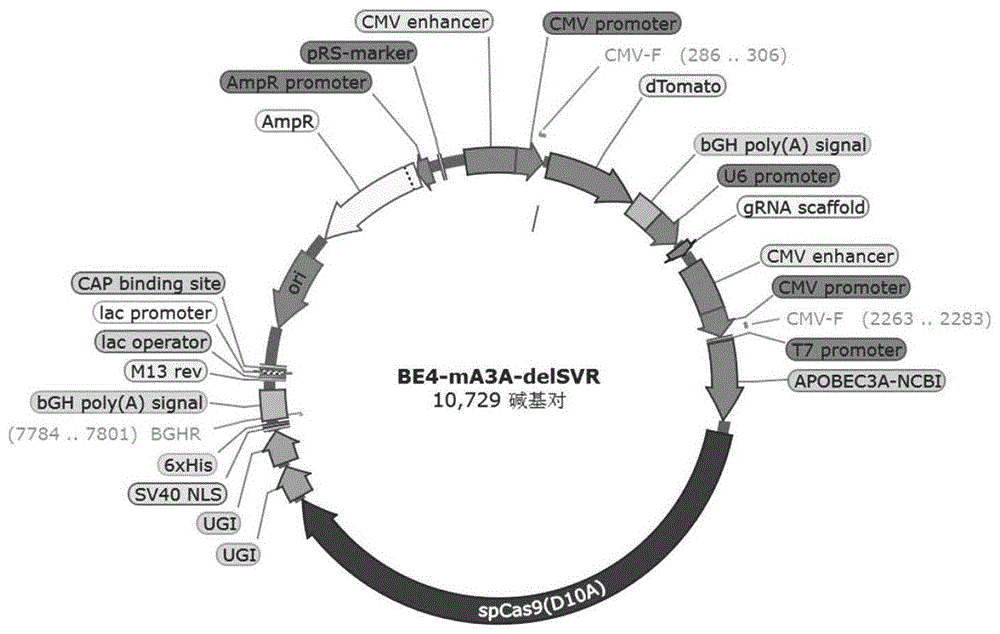
图3:胞嘧啶碱基编辑器BE4-mA3A-delSVR的质粒图谱;
图4:BamHI和SmaI双酶切BE4-mA3A-B5(~11kb)质粒的电泳图;
图5:NCBI中食蟹猴APOBEC3A的参考序列(mA3A)及其删除SVR序列的突变体(mA3A-delSVR)的电泳图;
图6:Sanger测序验证不同胞嘧啶单碱基编辑器(BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR)在site3基因靶向位点的编辑效率(n=3);
图7:Sanger测序验证不同胞嘧啶单碱基编辑器(BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR)在RNF2基因靶向位点的编辑效率(n=3);
图8:Sanger测序验证不同胞嘧啶单碱基编辑器(BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR)在EMX1基因靶向位点的编辑效率(n=3);
图9:Sanger测序验证不同胞嘧啶单碱基编辑器(BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR)在HEK2基因靶向位点的编辑效率(n=3);
图10:Sanger测序验证不同胞嘧啶单碱基编辑器(BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR)在HEK4基因靶向位点的编辑效率(n=3);
图11:BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR编辑器在site3、RNF2、EMX1、HEK2和HEK4基因靶向位点的C>T编辑效率对比图(n=3);
图12:BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR编辑器在site3-C5、RNF2-C6、EMX1-C5、EMX1-C6、HEK2-C6和HEK4-C5位点产物(T、G、A)分布比较图(n=3);
图13:根据胞嘧啶C在原间隔子(protospacer)中的位置(PAM位于21-23位)对5个基因位点中Cs的平均编辑效率进行了整理,不同形状和灰度代表不同的编辑器(n=3,6,9);
图14:BE4-mA3A和BE4-mA3A-delSVR编辑器在5个基因位点中Cs的平均编辑效率对比图(n=3,6,9)。
具体实施方式
下述实施例中涉及到的质粒:
BE4质粒:Addgene Plasmid#100802。
pSpCas9(BB)-2A-dTomato(PX458)质粒:本实验室在pSpCas9(BB)-2A-GFP(PX458)(Addgene Plasmid#48138)质粒的基础上,化学合成dTomato(SEQ ID NO:8)替换GFP构建的。
BE3-rA1质粒:杨辉教授赠送,已在此Off-target RNA mutation induced by DNAbase editing and its elimination by mutagenesis文章中报道,文章中的BE3在本发明中即为BE3-rA1。
BE4-rA1质粒:本实验室构建的,在BE4质粒(Addgene Plasmid#100802)的基础上,在NotⅠ酶切位点依次插入化学合成的dTomato基因序列(SEQ ID NO:8)以及BE3-rA1质粒的U6promoter+sgRNA scaffold+CMV enhancer+CMVpromoter片段。
pcDNA3.1-mA3A-GFP质粒:本实验室构建的,以pcDNA3.1-2xFLAG-SREBP-2(Addgene Plasmid#26807)质粒为骨架,EcoRⅠ和XbaⅠ双酶切载体后,将化学合成的mA3A片段(NCBI上参考序列NP_001306288.1,SEQ ID NO:12)和GFP片段(SEQ ID NO:10)与酶切后的载体连接而成。质粒图谱见图2。
BE4-mA3A-B5质粒:本实验室构建的,具体构建方法见实施例1。质粒图谱见图1。
本发明所涉及的食蟹猴购买于广州相观生物科技有限公司(生产许可证号:SCXK(粤)2018-0043),实验前经记录和兽医检查证实健康状况良好,动物设施符合国家实验动物标准(GB14925-2010)。随后挑选了经过19个月高糖高脂饮食处理产生高胆固醇血症并且可自行恢复到正常血脂的食蟹猴进行本发明胞嘧啶单碱基编辑器(BE4-mA3A-B5)的构建。
实施例1:胞嘧啶单碱基编辑器(BE4-mA3A-B5)的构建
(1)目的基因的获取(mA3A-B5)
本发明采用Paxgene管取得了食蟹猴的血液样品,并通过TIANGEN的血液RNA提取试剂盒提取该食蟹猴的RNA,通过Takara的反转试剂盒获得该食蟹猴的cDNA序列,然后以其cDNA序列为模板,通过引物对5’-TATAGGGAGAGCCGCCACCATGGAAGCCAGCCCAG-3’和5’-ACCAGAAGAACCACCAGAGTTTCCCTGATTCTGG-3’进行PCR扩增得到mA3A-B5基因片段(即Part3,SEQ IDNO:11),再用TIANGEN的琼脂糖凝胶DNA提取试剂盒纯化PCR产物。PCR反应体系如表1。
表1 PCR反应体系
反应程序如下:95℃预变性3min;95℃ 15s,60℃ 15s,72℃ 25s,进行35个循环;72℃延伸5min,降温至4℃,最终得到mA3A-B5(Part3)。
(2)线性化质粒载体的制备(BE4)
以BE4-rA1质粒为模板,通过引物对5’-TCTGGTGGTTCTTCTGGTGGTTCTAGCGGC-3’和5’-GGTGGCGGCTCTCCCTATAGTGAGTCGTAT-3’进行PCR扩增,获得PCR产物Part4,包括nCas9(D10A)、UGI、NLS、终止序列及CMV增强子和启动子基因序列;
以BE4-rA1质粒为模板,通过引物对5’-CGGTGGCTTCGATAGCCCTACAGTTGCCT-3’和5’-CTACTAGGACAGAATAGGCAACTGTAGGGC-3’进行PCR扩增,获得PCR产物Part5,包括colE1高拷贝复制起点和氨苄青霉素抗性筛选基因及其氨苄青霉素启动子序列。
通过TIANGEN的琼脂糖凝胶DNA提取试剂盒纯化PCR产物Part4和Part5。
(3)红色荧光蛋白(dTomato)的基因获取
以pSpCas9(BB)-2A-dTomato(PX458)质粒为模板,通过引物5’-TAGAGATCCGCGCCACCATGGTGAGC-3’和5’-GAAGGCACAGTTACTTGTACAGCTCG-3’进行PCR扩增,获得红色荧光蛋白dTomato基因及其CMV增强子和启动子,即载体骨架Part1。再用TIANGEN的琼脂糖凝胶DNA提取试剂盒纯化PCR产物Part1。
(4)sgRNA的携带框架及U6启动子的基因获取
以BE3-rA1质粒为模板,利用以下引物5’-GCTCACATGTGAGGGCCTATTTCCC-3’和5’-ATAGGCCCTCACATGTGAGCAAAAG-3’进行PCR扩增,获得用于插入sgRNA序列的携带框架及其U6启动子,即载体骨架Part2。再用TIANGEN的琼脂糖凝胶DNA提取试剂盒纯化PCR产物Part2。
(5)构建载体质粒BE4-mA3A-B5(同源重组法)
采用诺唯赞公司的MultiS One Step Cloning Kit试剂盒将步骤(1)~(4)中纯化后的五个PCR片段Part1、Part2、Part3、Part4和Part5进行连接,获得连接产物,转化大肠杆菌DH5α并涂布在含有0.05%Amp(浓度为100μg/mL的氨苄青霉素)抗性的LB平板上,37℃倒置过夜培养。
在每个克隆平板上挑选3个菌落接种至含有0.1%Amp(浓度为100μg/mL的氨苄青霉素)抗性的液体LB培养基中培养8h以上,然后将菌液送到金唯智公司进行Sanger测序验证。将片段成功插入的克隆载体进行扩大培养,然后采用康为世纪公司的无内毒素的质粒提取试剂盒获得载体质粒并命名为BE4-mA3A-B5,-20℃存放备用。BE4-mA3A-B5质粒图谱如图1所示。
实施例2:基于食蟹猴APOBEC3A(mA3A)及其突变体的胞嘧啶单碱基编辑器的构建
(1)线性化质粒载体的制备(BE4-mA3A-B5)
通过Takara公司的快切酶BamHI和SmaI双酶切实施例1中获得的BE4-mA3A-B5载体,去除载体中食蟹猴的APOBEC3A(即核苷酸序列如SEQ ID NO:11所示的mA3A-B5基因片段),以便接下来胞嘧啶脱氨酶的替换成核苷酸序列如SEQ ID NO:12所示的mA3A基因片段,酶切体系及反应条件如表2。然后使用TIANGEN的普通DNA产物纯化试剂盒纯化酶切产物,获得线性化载体(见图4)。
表2 BamHI和SmaI双酶切体系
(2)目的基因的获取(mA3A和mA3A-delSVR)
以过表达mA3A的质粒pcDNA3.1-mA3A-GFP为模板,通过引物对5’-CCCAGACACTTGATGGATCCAAACACGTTCACTTTC-3’和5’-GGACTCTGAGGTCCCGGGAGTCTCGCTGCC-3’进行PCR扩增得到mA3A的基因序列;以过表达mA3A的质粒pcDNA3.1-mA3A-GFP为模板通过引物对5’-CCCAGACACTTGATGGATCCAAACACGTTCACTTTCAACTTTAACAATGACCTTGGACGGCACCAGACC-3’和5’-GGACTCTGAGGTCCCGGGAGTCTCGCTGCC-3’进行PCR扩增得到mA3A-delSVR基因片段(见图5)。再用TIANGEN的琼脂糖凝胶DNA提取试剂盒纯化PCR产物,在进行PCR的时候引物两端均加上与载体切口互补配对的同源臂以备克隆到载体中,PCR反应体系如表3。
表3 PCR反应体系
反应程序如下:95℃预变性3min;95℃ 15s,60℃ 15s,72℃ 25s,进行35个循环;72℃延伸5min,降温至4℃,最终得到mA3A和mA3A-delSVR基因片段。
(3)构建载体质粒BE4-mA3A-delSVR(同源重组法)
采用诺唯赞公司的ClonExpress II One Step Cloning Kit试剂盒将步骤(2)纯化后的mA3A或mA3A-delSVR基因片段分别与步骤(1)纯化后的线性化载体BE4-mA3A-B5进行连接,获得连接产物,转化大肠杆菌DH5α并涂布在含有0.05%Amp(浓度为100μg/mL的氨苄青霉素)抗性的LB平板上,37℃倒置过夜培养。
在每个克隆平板上挑选3个菌落接种至含有0.1%Amp(浓度为100μg/mL的氨苄青霉素)抗性的液体LB培养基中培养8h以上,然后将菌液送到金唯智公司进行Sanger测序验证。将片段成功插入的克隆载体进行扩大培养,然后采用康为世纪公司的无内毒素的质粒提取试剂盒获得载体质粒并命名为BE4-mA3A和BE4-mA3A-delSVR,-20℃存放备用。BE4-mA3A-delSVR质粒图谱如图3所示。
实施例3:胞嘧啶碱基编辑器BE4-mA3A-delSVR的应用方法
(1)插入特异性靶向基因的sgRNA
由于单碱基编辑器是基于CRISPR/Cas9系统的,它的靶向特异性是由两部分组成的,一部分是sgRNA和靶DNA序列之间的碱基互补配对,另一部分是Cas9蛋白和位于靶DNA序列3’末端的短DNA序列决定的。其中靶DNA序列被称为原间隔子(细菌天然免疫防御时将外来DNA片段称为原间隔子,即protospacer),位于靶DNA序列3’末端的短DNA序列被称为PAM(protospacer adjacent motif)。
为了更好的和已有研究做对比,采用的sgRNA是已经有文献报道的序列,具体见表4。
表4:sgRNA及其PAM序列表
1)利用BbsI分别酶切原始的BE3-rA1质粒、BE4-rA1质粒以及实施例2中构建的胞嘧啶碱基编辑器(BE4-mA3A和BE4-mA3A-delSVR)获得线性化载体,酶切体系如表5;
2)分别化学合成表6中靶点site3、RNF2、EMX1、HEK2和HEK4的sgRNA的克隆引物,并通过热激退火使克隆引物自连成双链寡核苷酸片段(sgRNA自连体系见表7),将双链寡核苷酸片段与步骤1)中线性化载体连接(连接体系见表8),将连接好的载体转化至感受态大肠杆菌细胞(DH5α),质粒提取并测序鉴定,最后获得一系列靶向site3、RNF2、EMX1、HEK2和HEK4基因的胞嘧啶单碱基编辑器。
表5 BbsI酶切体系
表6 sgRNA克隆引物表
/>
表7 sgRNA自连体系
表8连接体系
(2)细胞转染
高糖DMEM完全培养基的配制:高糖DMEM培养基中含10%胎牛血清(FBS)和1%三抗(青霉素-链霉素-庆大霉素)。
1)细胞培养:从液氮罐中取出冻存的HEK293T细胞,置于37℃水浴中迅速融化,将融化的细胞悬液加入到10mL高糖DMEM完全培养基中,离心收集细胞沉淀,使用高糖DMEM完全培养基重悬细胞并加入到含有10mL高糖DMEM完全培养基的细胞培养皿中,并放置于含有5%CO
2)细胞铺板:待HEK293T细胞长满培养皿,用0.25%胰酶消化细胞1min,高糖DMEM完全培养基终止消化,1500rpm平衡离心2min,弃去上清,用1mL高糖DMEM完全培养基重悬细胞,并按照1×10
3)细胞转染:在一支1.5mL的EP管中,用125μL Opti-MEM培养基稀释3.75μL的转染试剂Lipofectamine 3000,充分混匀,获得混合液A;在另一支1.5mL的EP管中,使用125μLOpti-MEM培养基稀释2.5μg步骤(1)中构建的靶向site3、RNF2、EMX1、HEK2或HEK4基因的胞嘧啶单碱基编辑器,并添加5μLP3000辅助转染试剂,充分混匀,获得混合液B;将混合液A和B以1:1的比例混合,室温孵育15min,获得混合物;将混合物添加至步骤2)中刚刚铺好细胞的6孔板中,轻柔混匀,避免损伤细胞;然后将6孔板放回37℃,5%CO
(3)流式分选
先用0.25%胰酶消化转染72h后的细胞,高糖DMEM完全培养基终止消化,1500rpm平衡离心2min收集细胞沉淀,然后加入2mL含有2%胎牛血清(FBS)的PBS重悬清洗细胞,1500rpm平衡离心2min,重复清洗两次后用1mL含有2%FBS的PBS重悬细胞,然后将细胞悬液通过带有滤膜的流式分选小管进行收集,去除较大的细胞碎片聚集物,避免分选时堵塞仪器。
将制备好的细胞悬液和对应的含有2mL高糖DMEM完全培养基的收集管均放置于冰上,然后进行流式上机分选操作,因为本发明构建的胞嘧啶碱基编辑器上携带有红色荧光蛋白(dTomato),根据激发波长(554nm)和发射波长(581nm)直接进行分选收集获得dTomato阳性细胞。BE3-rA1质粒携带的是绿色荧光蛋白(GFP),根据其激发波长(488nm)和发射波长(507nm)直接进行分选收集获得GFP阳性细胞。每个样品至少分得10万细胞。
(4)Sanger测序验证
将步骤(3)中收集的dTomato阳性细胞或GFP阳性细胞,1500rpm平衡离心2min后收集沉淀,然后通过诺唯赞的细胞/组织DNA提取试剂盒提取细胞的DNA。以DNA为模板,采用高保真酶进行PCR扩增靶基因的特定序列(~400bp),即包含sgRNA靶向结合的20bp片段的扩增子,获得PCR产物,测序引物见表9。取5μLPCR产物进行琼脂糖凝胶电泳,将条带大小符合预期的PCR产物送到金唯智公司进行Sanger测序。通过采用Chromas软件对Sanger测序的峰图进行查看,通过SnapGene Viewer软件对Sanger测序的结果进行分析各个碱基的占比。
表9 sgRNA的Sanger测序引物表
实施例4:胞嘧啶碱基编辑器的编辑效率对比
将靶向site3、RNF2、EMX1、HEK2和HEK4基因的一系列编辑器(如BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR)分别转染HEK293T细胞,分选出转染成功的细胞,分析各个编辑器在同一基因位点上的编辑情况,具体实施方式同实施例3的步骤(2)~(4)。
不同编辑器在site3、RNF2、EMX1、HEK2和HEK4基因靶向位点的编辑效率对比结果分别见图6、图7、图8、图9和图10。结果显示,相比原始的BE3-rA1和BE4-rA1质粒,携带mA3A或mA3A-delSVR的胞嘧啶碱基编辑器在C>T的编辑效率上都具有一定的优势,整体提高了至少1倍,尤其是在EMX1基因靶向位点提高了近5倍。此外,删除SVR序列的BE4-mA3A-delSVR与未进行改造的BE4-mA3A相比,编辑的特异性显著增强,其中在site3基因上的编辑位点从C4、C5和C14,变成主要编辑C5位点,在RNF2和HEK2基因上则主要编辑C6位点,且在C5和C6位点的编辑效率基本保持相对稳定,除了在HEK4基因靶向位点的编辑效率降低了,其余基因靶向位点均保持与BE4-mA3A相当的编辑效率甚至更高(在site3-C5位点),见图11。
实施例5:胞嘧啶碱基编辑器的产物纯度对比
将靶向site3、RNF2、EMX1、HEK2和HEK4基因的一系列编辑器(如BE3-rA1、BE4-rA1、BE4-mA3A和BE4-mA3A-delSVR)分别转染HEK293T细胞,分选出转染成功的细胞,分析各个编辑器在同一基因位点上的编辑情况,具体实施方式同实施例3的步骤(2)~(4)。
不同编辑器在site3、RNF2、EMX1、HEK2和HEK4基因靶向位点的产物纯度对比结果分别见图12。已知删除SVR序列的BE4-mA3A-delSVR与BE4-mA3A相比,主要编辑C5和C6位点,所以本发明分析了不同编辑器在site3-C5、RNF2-C6、EMX1-C5、EMX1-C6、HEK2-C6和HEK4-C5位点的产物纯度。结果显示,相比与原始的BE3-rA1和BE4-rA1质粒,携带mA3A或mA3A-delSVR的胞嘧啶碱基编辑器在C5和C6位点的产物纯度都有所提高,C被编辑成T的占比均大于70%,都保持远高于BE3-rA1和BE4-rA1的水平(C被编辑成T的占比越高,说明产物纯度越高,即产生的非T产物占比大大减少),尤其在EMX1-C6位点,纯度比原始的编辑器提高了近10倍(见图12)。此外,删除SVR序列的BE4-mA3A-delSVR与BE4-mA3A相比,在C5和C6位点的产物纯度也是保持相当的水平,在EMX1-C6位点,产物纯度可达到约96%。
实施例6:胞嘧啶碱基编辑器的编辑窗口对比
整合所有测试过的基因(site3、RNF2、EMX1、HEK2和HEK4)中C碱基的编辑情况,根据C碱基在原间隔子中的位置(PAM位于21-23位)对这5个基因位点中C的平均编辑效率进行了整理。其中BE3和BE4的编辑窗口较小,只有C4-C6之间才能有效编辑,且编辑效率较低(最高不超过30%)。与BE3-rA1和BE4-rA1相比,可以看出BE4-mA3A的编辑窗口是扩大的(C3-C14位点)而且整体编辑效率更高,BE4-mA3A-delSVR的编辑窗口是最小的,只编辑C5和C6位点,特异性更强,且最高编辑效率和产物纯度保持稳定(图13)。
单独比较BE4-mA3A和BE4-mA3A-delSVR,可以明显看出BE4-mA3A-delSVR的编辑窗口更特异,对C5和C6位点有明显的偏好性,且在这两个位点的C>T编辑效率和BE4-mA3A相似(图14)。因此食蟹猴特有的SVR序列对RNA编辑酶本身的活性和选择性都有很大的影响,进一步推测SVR序列可能是通过影响编辑口袋的大小,进而导致更精准的编辑。所以本发明为今后编辑器的优化提供了一个很好的思路和方向,本发明构建的BE4-mA3A-delSVR编辑器丰富了基因编辑工具包,在单基因遗传病的基因治疗方面具有很大的应用潜力。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
SEQUENCE LISTING
<110> 江南大学
<120> 一种基于食蟹猴改造后的APOBEC3A的胞嘧啶碱基编辑器
<130> BAA211146A
<160> 12
<170> PatentIn version 3.3
<210> 1
<211> 202
<212> PRT
<213> 人工序列
<400> 1
Met Glu Ala Ser Pro Ala Ser Arg Pro Arg His Leu Met Asp Pro Asn
1 5 1015
Thr Phe Thr Phe Asn Phe Asn Asn Asp Leu Ser Val Arg Gly Arg His
202530
Gln Thr Tyr Leu Cys Tyr Glu Val Glu Arg Leu Asp Asn Gly Thr Trp
354045
Val Pro Met Asp Glu Arg Arg Gly Phe Leu Cys Asn Lys Ala Lys Asn
505560
Val Pro Cys Gly Asp Tyr Gly Cys His Ala Glu Leu Cys Phe Leu Gly
65707580
Glu Val Pro Ser Trp Gln Leu Asp Pro Ala Gln Thr Tyr Arg Val Thr
859095
Trp Phe Ile Ser Trp Ser Pro Cys Phe Arg Arg Gly Cys Ala Glu Gln
100 105 110
Val Arg Ala Phe Leu Gln Glu Asn Thr His Met Arg Leu Arg Ile Phe
115 120 125
Ala Ala Arg Ile Tyr Asp Tyr Asp Leu Leu Tyr Gln Glu Ala Leu Arg
130 135 140
Thr Leu Arg Asp Ala Gly Ala Gln Val Ser Ile Met Thr Tyr Glu Glu
145 150 155 160
Phe Lys His Cys Trp Asp Thr Phe Val Asp Arg Gln Gly Arg Pro Phe
165 170 175
Gln Pro Trp Asp Gly Leu Asp Glu His Ser Gln Ala Leu Ser Gly Arg
180 185 190
Leu Arg Asp Ile Leu Gln Asn Gln Gly Asn
195 200
<210> 2
<211> 199
<212> PRT
<213> 人工序列
<400> 2
Met Glu Ala Ser Pro Ala Ser Arg Pro Arg His Leu Met Asp Pro Asn
1 5 1015
Thr Phe Thr Phe Asn Phe Asn Asn Asp Leu Gly Arg His Gln Thr Tyr
202530
Leu Cys Tyr Glu Val Glu Arg Leu Asp Asn Gly Thr Trp Val Pro Met
354045
Asp Glu Arg Arg Gly Phe Leu Cys Asn Lys Ala Lys Asn Val Pro Cys
505560
Gly Asp Tyr Gly Cys His Ala Glu Leu Cys Phe Leu Gly Glu Val Pro
65707580
Ser Trp Gln Leu Asp Pro Ala Gln Thr Tyr Arg Val Thr Trp Phe Ile
859095
Ser Trp Ser Pro Cys Phe Arg Arg Gly Cys Ala Glu Gln Val Arg Ala
100 105 110
Phe Leu Gln Glu Asn Thr His Met Arg Leu Arg Ile Phe Ala Ala Arg
115 120 125
Ile Tyr Asp Tyr Asp Leu Leu Tyr Gln Glu Ala Leu Arg Thr Leu Arg
130 135 140
Asp Ala Gly Ala Gln Val Ser Ile Met Thr Tyr Glu Glu Phe Lys His
145 150 155 160
Cys Trp Asp Thr Phe Val Asp Arg Gln Gly Arg Pro Phe Gln Pro Trp
165 170 175
Asp Gly Leu Asp Glu His Ser Gln Ala Leu Ser Gly Arg Leu Arg Asp
180 185 190
Ile Leu Gln Asn Gln Gly Asn
195
<210> 3
<211> 600
<212> DNA
<213> 人工序列
<400> 3
atggaagcca gcccagcatc caggcccaga cacttgatgg atccaaacac gttcactttc 60
aactttaaca atgaccttgg acggcaccag acctacttgt gctacgaggt ggagcgcctg 120
gacaatggca cctgggtccc gatggacgag cgcaggggct ttctatgcaa caaggctaag 180
aatgttccct gtggtgatta tggctgccac gcggagctgt gcttcctggg cgaggttcct 240
tcttggcagt tggacccggc ccagacgtac agggtcactt ggttcatctc ctggagcccc 300
tgcttcagga ggggctgtgc cgagcaagtg cgtgcgttcc ttcaggagaa cacacacatg 360
agactgcgca tctttgctgc ccgcatctat gattacgatc tcctgtatca ggaggcactg 420
cgaacgctgc gggatgctgg ggcccaagtc tccatcatga cctacgagga atttaagcac 480
tgctgggaca cctttgtgga ccgccaggga cgtcccttcc agccctggga tggactagat 540
gagcacagcc aagccctgag tgggaggctt cgggacattc tccagaatca gggaaactga 600
<210> 4
<211> 4101
<212> DNA
<213> 人工序列
<400> 4
gataaaaagt attctattgg tttagccatc ggcactaatt ccgttggatg ggctgtcata 60
accgatgaat acaaagtacc ttcaaagaaa tttaaggtgt tggggaacac agaccgtcat 120
tcgattaaaa agaatcttat cggtgccctc ctattcgata gtggcgaaac ggcagaggcg 180
actcgcctga aacgaaccgc tcggagaagg tatacacgtc gcaagaaccg aatatgttac 240
ttacaagaaa tttttagcaa tgagatggcc aaagttgacg attctttctt tcaccgtttg 300
gaagagtcct tccttgtcga agaggacaag aaacatgaac ggcaccccat ctttggaaac 360
atagtagatg aggtggcata tcatgaaaag tacccaacga tttatcacct cagaaaaaag 420
ctagttgact caactgataa agcggacctg aggttaatct acttggctct tgcccatatg 480
ataaagttcc gtgggcactt tctcattgag ggtgatctaa atccggacaa ctcggatgtc 540
gacaaactgt tcatccagtt agtacaaacc tataatcagt tgtttgaaga gaaccctata 600
aatgcaagtg gcgtggatgc gaaggctatt cttagcgccc gcctctctaa atcccgacgg 660
ctagaaaacc tgatcgcaca attacccgga gagaagaaaa atgggttgtt cggtaacctt 720
atagcgctct cactaggcct gacaccaaat tttaagtcga acttcgactt agctgaagat 780
gccaaattgc agcttagtaa ggacacgtac gatgacgatc tcgacaatct actggcacaa 840
attggagatc agtatgcgga cttatttttg gctgccaaaa accttagcga tgcaatcctc 900
ctatctgaca tactgagagt taatactgag attaccaagg cgccgttatc cgcttcaatg 960
atcaaaaggt acgatgaaca tcaccaagac ttgacacttc tcaaggccct agtccgtcag 1020
caactgcctg agaaatataa ggaaatattc tttgatcagt cgaaaaacgg gtacgcaggt 1080
tatattgacg gcggagcgag tcaagaggaa ttctacaagt ttatcaaacc catattagag 1140
aagatggatg ggacggaaga gttgcttgta aaactcaatc gcgaagatct actgcgaaag 1200
cagcggactt tcgacaacgg tagcattcca catcaaatcc acttaggcga attgcatgct 1260
atacttagaa ggcaggagga tttttatccg ttcctcaaag acaatcgtga aaagattgag 1320
aaaatcctaa cctttcgcat accttactat gtgggacccc tggcccgagg gaactctcgg 1380
ttcgcatgga tgacaagaaa gtccgaagaa acgattactc catggaattt tgaggaagtt 1440
gtcgataaag gtgcgtcagc tcaatcgttc atcgagagga tgaccaactt tgacaagaat 1500
ttaccgaacg aaaaagtatt gcctaagcac agtttacttt acgagtattt cacagtgtac 1560
aatgaactca cgaaagttaa gtatgtcact gagggcatgc gtaaacccgc ctttctaagc 1620
ggagaacaga agaaagcaat agtagatctg ttattcaaga ccaaccgcaa agtgacagtt 1680
aagcaattga aagaggacta ctttaagaaa attgaatgct tcgattctgt cgagatctcc 1740
ggggtagaag atcgatttaa tgcgtcactt ggtacgtatc atgacctcct aaagataatt 1800
aaagataagg acttcctgga taacgaagag aatgaagata tcttagaaga tatagtgttg 1860
actcttaccc tctttgaaga tcgggaaatg attgaggaaa gactaaaaac atacgctcac 1920
ctgttcgacg ataaggttat gaaacagtta aagaggcgtc gctatacggg ctggggacga 1980
ttgtcgcgga aacttatcaa cgggataaga gacaagcaaa gtggtaaaac tattctcgat 2040
tttctaaaga gcgacggctt cgccaatagg aactttatgc agctgatcca tgatgactct 2100
ttaaccttca aagaggatat acaaaaggca caggtttccg gacaagggga ctcattgcac 2160
gaacatattg cgaatcttgc tggttcgcca gccatcaaaa agggcatact ccagacagtc 2220
aaagtagtgg atgagctagt taaggtcatg ggacgtcaca aaccggaaaa cattgtaatc 2280
gagatggcac gcgaaaatca aacgactcag aaggggcaaa aaaacagtcg agagcggatg 2340
aagagaatag aagagggtat taaagaactg ggcagccaga tcttaaagga gcatcctgtg 2400
gaaaataccc aattgcagaa cgagaaactt tacctctatt acctacaaaa tggaagggac 2460
atgtatgttg atcaggaact ggacataaac cgtttatctg attacgacgt cgatcacatt 2520
gtaccccaat cctttttgaa ggacgattca atcgacaata aagtgcttac acgctcggat 2580
aagaaccgag ggaaaagtga caatgttcca agcgaggaag tcgtaaagaa aatgaagaac 2640
tattggcggc agctcctaaa tgcgaaactg ataacgcaaa gaaagttcga taacttaact 2700
aaagctgaga ggggtggctt gtctgaactt gacaaggccg gatttattaa acgtcagctc 2760
gtggaaaccc gccaaatcac aaagcatgtt gcacagatac tagattcccg aatgaatacg 2820
aaatacgacg agaacgataa gctgattcgg gaagtcaaag taatcacttt aaagtcaaaa 2880
ttggtgtcgg acttcagaaa ggattttcaa ttctataaag ttagggagat aaataactac 2940
caccatgcgc acgacgctta tcttaatgcc gtcgtaggga ccgcactcat taagaaatac 3000
ccgaagctag aaagtgagtt tgtgtatggt gattacaaag tttatgacgt ccgtaagatg 3060
atcgcgaaaa gcgaacagga gataggcaag gctacagcca aatacttctt ttattctaac 3120
attatgaatt tctttaagac ggaaatcact ctggcaaacg gagagatacg caaacgacct 3180
ttaattgaaa ccaatgggga gacaggtgaa atcgtatggg ataagggccg ggacttcgcg 3240
acggtgagaa aagttttgtc catgccccaa gtcaacatag taaagaaaac tgaggtgcag 3300
accggagggt tttcaaagga atcgattctt ccaaaaagga atagtgataa gctcatcgct 3360
cgtaaaaagg actgggaccc gaaaaagtac ggtggcttcg atagccctac agttgcctat 3420
tctgtcctag tagtggcaaa agttgagaag ggaaaatcca agaaactgaa gtcagtcaaa 3480
gaattattgg ggataacgat tatggagcgc tcgtcttttg aaaagaaccc catcgacttc 3540
cttgaggcga aaggttacaa ggaagtaaaa aaggatctca taattaaact accaaagtat 3600
agtctgtttg agttagaaaa tggccgaaaa cggatgttgg ctagcgccgg agagcttcaa 3660
aaggggaacg aactcgcact accgtctaaa tacgtgaatt tcctgtattt agcgtcccat 3720
tacgagaagt tgaaaggttc acctgaagat aacgaacaga agcaactttt tgttgagcag 3780
cacaaacatt atctcgacga aatcatagag caaatttcgg aattcagtaa gagagtcatc 3840
ctagctgatg ccaatctgga caaagtatta agcgcataca acaagcacag ggataaaccc 3900
atacgtgagc aggcggaaaa tattatccat ttgtttactc ttaccaacct cggcgctcca 3960
gccgcattca agtattttga cacaacgata gatcgcaaac gatacacttc taccaaggag 4020
gtgctagacg cgacactgat tcaccaatcc atcacgggat tatatgaaac tcggatagat 4080
ttgtcacagc ttgggggtga c 4101
<210> 5
<211> 249
<212> DNA
<213> 人工序列
<400> 5
actaatctgt cagatattat tgaaaaggag accggtaagc aactggttat ccaggaatcc 60
atcctcatgc tcccagagga ggtggaagaa gtcattggga acaagccgga aagcgatata 120
ctcgtgcaca ccgcctacga cgagagcacc gacgagaatg tcatgcttct gactagcgac 180
gcccctgaat acaagccttg ggctctggtc atacaggata gcaacggtga gaacaagatt 240
aagatgctc 249
<210> 6
<211> 21
<212> DNA
<213> 人工序列
<400> 6
cccaagaaga agaggaaagt c 21
<210> 7
<211> 225
<212> DNA
<213> 人工序列
<400> 7
ctgtgccttc tagttgccag ccatctgttg tttgcccctc ccccgtgcct tccttgaccc 60
tggaaggtgc cactcccact gtcctttcct aataaaatga ggaaattgca tcgcattgtc 120
tgagtaggtg tcattctatt ctggggggtg gggtggggca ggacagcaag ggggaggatt 180
gggttgacaa tagcaggcat gctggggatg cggtgggctc tatgg 225
<210> 8
<211> 705
<212> DNA
<213> 人工序列
<400> 8
atggtgagca agggcgagga ggtcatcaaa gagttcatgc gcttcaaggt gcgcatggag 60
ggctccatga acggccacga gttcgagatc gagggcgagg gcgagggccg cccctacgag 120
ggcacccaga ccgccaagct gaaggtgacc aagggcggcc ccctgccctt cgcctgggac 180
atcctgtccc cccagttcat gtacggctcc aaggcgtacg tgaagcaccc cgccgacatc 240
cccgattaca agaagctgtc cttccccgag ggcttcaagt gggagcgcgt gatgaacttc 300
gaggacggcg gtctggtgac cgtgacccag gactcctccc tgcaggacgg cacgctgatc 360
tacaaggtga agatgcgcgg caccaacttc ccccccgacg gccccgtaat gcagaagaaa 420
accatgggct gggaggcctc caccgagcgc ctgtaccccc gcgacggcgt gctgaagggc 480
gagatccacc aggccctgaa gctgaaggac ggcggccact acctggtgga gttcaagacc 540
atctacatgg ccaagaagcc cgtgcaactg cccggctact actacgtgga caccaagctg 600
gacatcacct cccacaacga ggactacacc atcgtggaac agtacgagcg ctccgagggc 660
cgccaccacc tgttcctgta cggcatggac gagctgtaca agtaa 705
<210> 9
<211> 343
<212> DNA
<213> 人工序列
<400> 9
gagggcctat ttcccatgat tccttcatat ttgcatatac gatacaaggc tgttagagag 60
ataattggaa ttaatttgac tgtaaacaca aagatattag tacaaaatac gtgacgtaga 120
aagtaataat ttcttgggta gtttgcagtt ttaaaattat gttttaaaat ggactatcat 180
atgcttaccg taacttgaaa gtatttcgat ttcttggctt tatatatctt gtggaaagga 240
cgaaacaccg ggtcttcgag aagacctgtt ttagagctag aaatagcaag ttaaaataag 300
gctagtccgt tatcaacttg aaaaagtggc accgagtcgg tgc 343
<210> 10
<211> 720
<212> DNA
<213> 人工序列
<400> 10
atggtgagca agggcgagga gctgttcacc ggggtggtgc ccatcctggt cgagctggac 60
ggcgacgtaa acggccacaa gttcagcgtg tccggcgagg gcgagggcga tgccacctac 120
ggcaagctga ccctgaagtt catctgcacc accggcaagc tgcccgtgcc ctggcccacc 180
ctcgtgacca ccctgaccta cggcgtgcag tgcttcagcc gctaccccga ccacatgaag 240
cagcacgact tcttcaagtc cgccatgccc gaaggctacg tccaggagcg caccatcttc 300
ttcaaggacg acggcaacta caagacccgc gccgaggtga agttcgaggg cgacaccctg 360
gtgaaccgca tcgagctgaa gggcatcgac ttcaaggagg acggcaacat cctggggcac 420
aagctggagt acaactacaa cagccacaac gtctatatca tggccgacaa gcagaagaac 480
ggcatcaagg tgaacttcaa gatccgccac aacatcgagg acggcagcgt gcagctcgcc 540
gaccactacc agcagaacac ccccatcggc gacggccccg tgctgctgcc cgacaaccac 600
tacctgagca cccagtccgc cctgagcaaa gaccccaacg agaagcgcga tcacatggtc 660
ctgctggagt tcgtgaccgc cgccgggatc actctcggca tggacgagct gtacaagtaa 720
<210> 11
<211> 609
<212> DNA
<213> 人工序列
<400> 11
atggaagcca gcccagcatc caggcccaga cacttgatgg atccaaacac gttcactttc 60
aactttaaca atgacctttc ggtccgtgga cggcaccaga cctacttgtg ctacgaggtg 120
gagcgcctgg acaatggcac ctgggtcccg atggacgagc gcaggggctt tctatgcaac 180
aaggctaaga atgttccctg tggtgattat ggctgccacg cggagctgtg cttcctgggc 240
gaggttcctt cttggcagtt ggacccggcc cagacgtaca gggtcacttg gttcatctcc 300
tggagcccct gcttcaggag gggctgtgcc gagcaagtgc gtgcgttcct tcaggagaac 360
acacacatga gactgcgcat ctttgctgcc cgcatctatg attacgattt cctgtatcag 420
gaggcactgc gaacgctgcg ggatgctggg gcccaagtct ccatcatgac ctacgaggaa 480
tttaagcact gctgggacac ctttgtggac cgccagggac gtcccttcca gccctgggat 540
ggactagatg agcacagcca agccctgagt gggaggcttc gggacattct ccagaatcag 600
ggaaactga 609
<210> 12
<211> 609
<212> DNA
<213> 人工序列
<400> 12
atggaagcca gcccagcatc caggcccaga cacttgatgg atccaaacac gttcactttc 60
aactttaaca atgacctttc ggtccgtgga cggcaccaga cctacttgtg ctacgaggtg 120
gagcgcctgg acaatggcac ctgggtcccg atggacgagc gcaggggctt tctatgcaac 180
aaggctaaga atgttccctg tggtgattat ggctgccacg cggagctgtg cttcctgggc 240
gaggttcctt cttggcagtt ggacccggcc cagacgtaca gggtcacttg gttcatctcc 300
tggagcccct gcttcaggag gggctgtgcc gagcaagtgc gtgcgttcct tcaggagaac 360
acacacatga gactgcgcat ctttgctgcc cgcatctatg attacgatct cctgtatcag 420
gaggcactgc gaacgctgcg ggatgctggg gcccaagtct ccatcatgac ctacgaggaa 480
tttaagcact gctgggacac ctttgtggac cgccagggac gtcccttcca gccctgggat 540
ggactagatg agcacagcca agccctgagt gggaggcttc gggacattct ccagaatcag 600
ggaaactga 609
- 一种基于食蟹猴APOBEC3A及其突变体的胞嘧啶碱基编辑器
- 一种基于人的改造后的APOBEC3A的胞嘧啶碱基编辑器
