一种多重核酸原位检测方法及探针和试剂盒
文献发布时间:2024-04-18 19:58:53
技术领域
本发明涉及基因检测技术领域,尤其涉及一种多重核酸原位检测方法及探针和试剂盒。
背景技术
空间转录组是一项快速发展的技术,因为它有望帮助人们了解各种组织的内在异质性。其中,基于成像的方法具有最高的空间分辨率,可以解析RNA在细胞甚至亚细胞中的分布。由于光学通道的限制,通常一次只能用少于5个荧光通道来标记RNA(如,RNA Scope,Stellaris smFISH,一次染色最多可以检测4种不同的RNA)。虽然利用超分辨(单轮32种)或荧光寿命显微镜(例如MOSAICA,文章中做到了10种)等策略可以突破传统光学通道的限制,但它依赖于专门的仪器。因此,通常采用多轮标记和条带循环来编码更多的RNA种类,并通过荧光序列进行解码。
然而,循环荧光标记也带来了诸多不便和问题。它需要复杂的仪器设备,包括流体、成像和温度控制,因此很难在大多数生物实验室推广。此外,一个信号点在不同周期的荧光信号需要精确定位,微小的组织变形或位移也会造成巨大影响。一些技术为了解决这个问题,利用每一轮的单轮单通道信息进行RNA编码,无需通过轮间信息来解码RNA,即可支持十几条甚至更多的RNA图谱,例如:RNAscope HiPlex v2可以通过3轮反应完成12种RNA的检测,pai-FISH在2轮反应内完成24种RNA的检测,但这些技术在成本、时间和RNA多重性可扩展性方面仍存在不便。此外,由于液体在厚组织中的扩散和交换速度较慢,三维原位RNA分析较为困难,通常需要更长的多轮反应和成像时间。
因此,有必要提出一种通过单轮染色实现多重核酸原位检测的方法。
发明内容
为了解决上述技术问题,本发明提供了一种多重核酸原位检测方法及探针和试剂盒,可以通过单轮染色就可以实现多重核酸的原位检测。
本发明提供了一种多重核酸原位检测方法,至少包括以下步骤:
S1、设计挂锁探针集,挂锁探针集中的挂锁探针的两个末端序列为和靶标核酸序列毗邻互补的结合序列,中间的连接序列为编码序列,编码序列用于对结合序列进行编码;
每条编码序列包括m个区段序列,每个区段序列用于结合一种颜色荧光探针,编码序列结合的m种荧光探针的颜色不同;2≤m≤8;每种颜色的区段序列包括分别至少两个子区段序列,子区段序列用于与同一颜色、不同强度值的荧光探针结合;
S2、取待测样品,用挂锁探针集进行原位杂交,进行扩增反应得到扩增产物,对编码序列的信息进行信号放大;
S3、采用荧光探针对扩增产物进行染色,对染色后的扩增产物成像,得到多个信号点,待测样品中的一个靶标核酸序列对应一个信号点;
S4、对每个信号点采用m个通道对颜色和强度值进行解码,根据解码结果获得待测样品中靶标核酸的原位分布信息;解码原则为:全部编码序列中,m-p个通道中的m-p个颜色的总强度值相同,0≤p≤3。
可选的,在S1中,在m种颜色的区段序列中,选定p种颜色的区段序列包括分别与强度值为0、100%的荧光探针结合的两个子区段序列;其余(m-p)种颜色的区段序列包括分别与不同的强度值的荧光探针结合的多个子区段序列,(m-p)种颜色的强度值总和为0或100%,2≤子区段序列的个数≤10,0≤p≤3。
可选的,其余(m-p)种颜色的区段序列包括分别与强度值为0、1/n±a
可选的,其余(m-p)种颜色的区段序列包括分别与强度值为0、1/n、2/n、3/n、……、(n-1)/n、1的荧光探针结合的n+1个子区段序列,1≤n≤9,0≤p≤3。
可选的,通过荧光标记探针与非荧光标记探针混合,获得同一颜色、不同强度值的荧光探针。
可选的,步骤S4中解码包括:提取信号点的m个通道的颜色,并识别每个通道的颜色的强度值,根据每个通道的颜色的强度值,得到解码结果,解码结果为m位的数值,每一位数值对应一种颜色的强度值,所选定颜色识别强度值0和强度值100%,其余(m-p)种颜色的强度值之和为0或100%。
可选的,得到解码结果后,根据挂锁探针集设计时所确立的编码序列和靶标核酸对应关系,根据解码结果获得靶标RNA在待测样品中的种类和分布信息。
可选的,在S2中,扩增反应采用滚环复制技术。
可选的,待测样品选自:冷冻组织切片、石蜡包埋样品、厚度不大于100微米的组织。
可选的,当待测样本的厚度大于10微米时,S2完成后进行除脂,并在成像前进行折射率匹配。
本发明提出一种挂锁探针集,挂锁探针集包括多条挂锁探针;挂锁探针集中的挂锁探针的两个末端序列为和靶标核酸序列毗邻互补的结合序列,中间的连接序列为编码序列,编码序列用于对结合序列进行编码;每条编码序列包括m个区段序列,每个区段序列用于结合一种颜色荧光探针,编码序列结合的m种荧光探针的颜色不同;2≤m≤8;每种颜色的区段序列包括分别至少两个子区段序列,子区段序列用于与同一颜色、不同强度值的荧光探针结合。
本发明提出一种多重核酸原位检测试剂盒,包括上述挂锁探针集;试剂盒还包括荧光探针集,包括m种颜色的荧光探针;(m–p)种颜色的荧光探针包括强度值分别为0、1/n±a
<……<(n-1)/n±a
可选的,通过荧光标记探针与非荧光标记探针混合,获得同一颜色、不同强度值的荧光探针。
本发明提出上述方法或上述试剂盒在基因敲除、药物测试、肿瘤分布检测、细胞分布分析或病原体分布分析中的应用;应用以单张切片、连续切片或厚组织的形式进行检测。
本发明实施例提供的技术方案与现有技术相比具有如下优点:
本发明提出了PRISM(Profiling of RNA In-situ through Single-roundiMaging):利用padlock挂锁探针扩增产物的总和恒定及比例荧光编码原则,通过一次性染色和成像,可在大尺度/三维组织中以光学极限分辨率对至少31个不同基因的核酸进行空间分辨。实验工作流程只需要常规的设备和传统荧光显微镜或共聚焦显微镜,不需要额外的仪器和流体系统。
本发明实施例提出的PRISM具有快速、易用、经济高效的技术优势。整个实验从样本到数据可在一天内完成。
本发明实施例的方法应用范围广泛,可以用于多种不同的组织,除冷冻组织切片外,还能用于石蜡包埋样品,或者厚度不大于100微米的组织块。
附图说明
图1为本发明实施例1-4的实验过程流程图;
图2为当m为不同取值时的编码策略;
图3为当n为不同取值时的编码策略;
图4为本发明实施例Padlock探针设计的示意图;
图5为本发明实施例中是否采用总和恒定编码策略的效果对比图,证明该编码策略可以消除扩增带来的信号差异;
图6为本发明实施例中信号解码的流程示意图,通过四种颜色相对间的比例在颜色空间中区分不同条形码;
图7为本发明实施例1中实际的信号点结果,根据信号点在颜色空间中的位置将30种不同的条形码区分开,并根据挂锁探针集设计时所确立的所述编码序列和所述靶标核酸对应关系,根据所述解码结果获得所述靶标RNA在所述待测样品中的种类和分布信息;
图8为本发明实施例1中小鼠大脑冠状切片中靶标RNA的原位分布图;
图9为本发明实施例1中小鼠大脑冠状切片中靶标RNA的原位分布图与Allen脑数据库中每种RNA的原位分布图对比;
图10为本发明实施例1中PRISM解码准确性实验结果;
图11为实施例1中小鼠大脑冠状切片中30个靶标RNA的原位分布图;
图12为本发明实施例2中小鼠E13.5胚胎的矢状切片中靶标RNA的原位分布图;
图13~图15为本发明实施例2中细胞的空间分布与其各自位置的投影关系;
图16为实施例2中根据30种基因分布划出的不同细胞类型,右侧为相邻切片的HE染色对比图;
图17为本发明实施例2中神经系统亚型区分图;
图18为本发明实施例3中不同系统的各种早期发育结构的原位分布图;
图19为本发明实施例4中乙型肝炎病毒阳性肝细胞癌样本中31个靶标RNA的原位分布图;
图20为本发明实施例5中厚组织脑片的实验流程示意图;
图21为本发明实施例5中在小鼠大脑切片上使用30个基因的大尺度ROI原位分布图;
图22为本发明实施例5中试剂对厚组织的穿透的效果图,如右侧的曲线图可知,100微米范围内可以检测到的信号点个数在z轴上较为均一。
具体实施方式
为了能够更清楚地理解本发明的上述目的、特征和优点,下面将对本发明的方案进行进一步描述。需要说明的是,在不冲突的情况下,本发明的实施例及实施例中的特征可以相互组合。
在下面的描述中阐述了很多具体细节以便于充分理解本发明,但本发明还可以采用其他不同于在此描述的方式来实施;显然,说明书中的实施例只是本发明的一部分实施例,而不是全部的实施例。
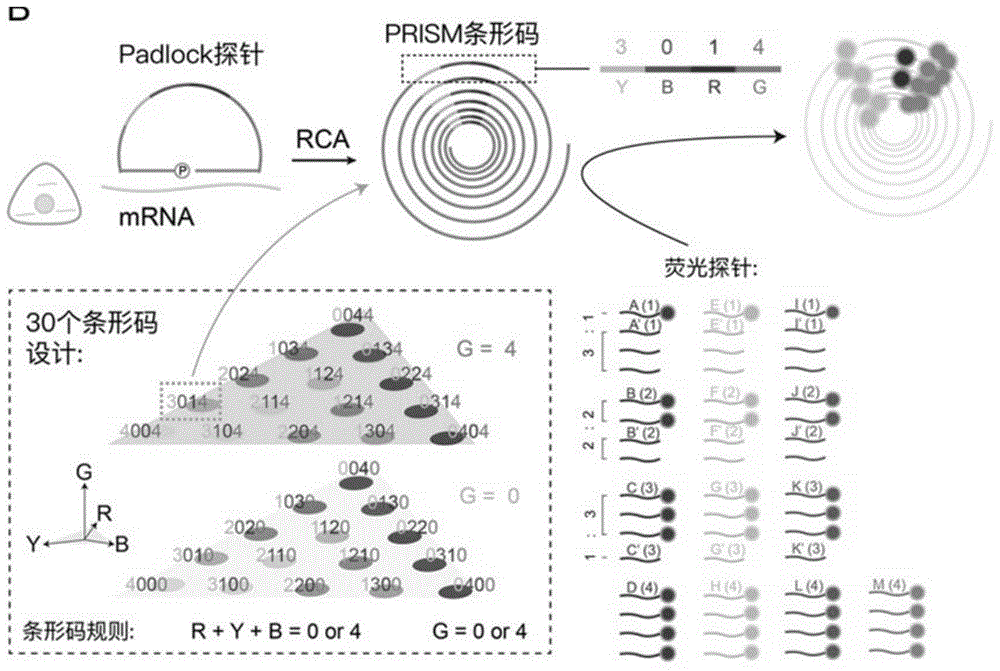
本发明实施例提出的一种多重核酸原位检测方法,简写为PRISM(Profiling ofRNA In-situ through Single-round iMaging),包括挂锁探针(Padlock探针)结合、信号放大(Padlock探针连接和RCA扩增)、荧光探针染色和成像的步骤。本发明实施例利用Padlock探针上的编码序列(barcode)为每个Padlock探针上的结合序列编码,通过Padlock探针在待测样本上的原位结合,并根据编码序列结合不同颜色、强度的荧光探针,从而实现了单次染色和成像过程中同时检测样本上数十种核酸的分布。通过连续切片可以构建待测样本的时空图谱,克服了现有技术中所检测靶标核酸种类少、多轮分析所需时间长、成本高及需要特殊的仪器等缺陷。其中,荧光探针上连接后荧光基团,从而实现了核酸在样本上的原位显示。荧光基团可选自荧光染料或量子点。
本发明实施例创新性的采用了针对Padlock探针上与荧光探针结合的编码序列,通过颜色组合和多强度的多级编码显著提高了单轮成像的信息密度。
为了获得更大的物理空间进行信息编码,采用了扩增技术,将单个Padlock探针上的编码序列扩增转化为挂锁探针上的数百个编码序列,增加了荧光探针的结合位点,扩增了荧光信号的强度,在更容易被低倍物镜捕获的同时,也使得我们利用荧光探针和非荧光探针的竞争结合从而实现单通道的多强度分级成为可能。
作为本发明实施例的一种改进,本发明实施例针对Padlock探针所选用的扩增技术为滚环复制技术(RCA,rolling circle replication)。但是,由于RCA中所使用的酶的扩增活性不尽相同,不同扩增产物之间的拷贝数可能会有差异。这将导致一些编码序列无法区分,例如,来自1000份拷贝扩增子的编码序列"2222"和来自500份拷贝扩增子的编码序列"4444",其表现出的荧光信息是完全一样的。为了克服这一问题,本发明实施例采用了"总和恒定编码"原则,即最终文库中每个编码序列(又称为PRISM编码序列barcode)的不同通道比例水平总和为一个固定的总水平。通过计算一个信号点内不同通道的相对水平比,控制每个信号点的通道绝对亮度值,可以消除来自RCA的偏差。进一步的,通过实验发现,在不同组织的成像过程中,可能会出现某些通道的高自发荧光背景,会影响该通道的多级分割。因此,本发明实施例在解码时,选择至少一个通道作为妥协通道,不对其荧光强度进行检测,只做“有”和“无”的区分。
具体的,本发明实施例中的多重核酸原位检测方法至少包括以下步骤:
S1、设计挂锁探针(Padlock探针)集,挂锁探针集中的挂锁探针的两个末端序列为和靶标核酸序列毗邻互补的结合序列,中间的连接序列为编码序列(又称为PRISM编码序列barcode),编码序列用于对结合序列进行编码;
每条编码序列包括m个区段序列,每个区段序列用于结合一种颜色荧光探针,编码序列结合的m种荧光探针的颜色不同;2≤m≤8;每种颜色的区段序列包括分别至少两个子区段序列,子区段序列用于与同一颜色、不同强度值的荧光探针结合;
S2、取待测样品,用挂锁探针集进行原位杂交,进行扩增反应得到扩增产物(又称为DNA纳米球),对编码序列的信息进行信号放大;
S3、采用荧光探针对扩增产物进行染色,对染色后的扩增产物成像,得到多个信号点,待测样品中的一个靶标核酸序列对应一个信号点;
S4、对每个信号点采用m个通道对颜色和强度值进行解码,根据解码结果获得待测样品中靶标核酸的原位分布信息;
编码解码原则为:全部编码序列中,m-p个通道中的m-p个颜色的总强度值相同,0≤p≤3。
以组织切片为例,本发明实施例的多重核酸原位检测方法的过程示意图如图1所示。
其中,靶标核酸的选取原则为每种基因的核酸序列上一段独有的20~60bp序列,优选为40bp序列,该序列拆分成10~30bp+10~30bp两段序列后,两段的Tm值应尽可能相近,且总Tm值应在60℃以上。
本发明实施例的靶标核酸包括RNA序列和DNA序列。当靶标核酸序列为DNA时,仅在步骤“挂锁探针连接”时,采用DNA-DNA双链所使用的连接酶如T4连接酶和其缓冲液,当靶标核酸序列为DNA时,仅在步骤“挂锁探针连接”时,采用DNA-RNA双链连接所使用的SplintR连接酶及其缓冲液。
作为本发明实施例的一种改进,结合现阶段一般所采用显微镜的分辨程度,m的范围的最大值可以为8、7、6、5、4、3。具体的,每种颜色的区段序列包括分别至少两个子区段序列,子区段序列的最大值可以为10、9、8、7、6、5、4、3、2。其中,m的范围的最大值以及子区段序列的最大值可以根据显微镜的通道数,扩增产物的大小以及光源的功率的改进而继续提高,本发明实施例并不进行特别的限制。m的数量取决于显微镜的光学通道,光学通道数量主要取决于显微镜的光源,滤光片等硬件。一般常见的荧光显微镜/共聚焦显微镜的通道数为2~8。因此m取值为2~8。
在具体实施例1-5中m=4即代表可适用于拥有4个通道的荧光显微镜。增加m数可大幅度增加可使用的编码序列以编码更多种类的基因。图2所示为固定n,p=1,将m从4提升至5后的编码情况,其中当m=4时,编码颜色依次为黄色、蓝色、红色和绿色;当m=5时,编码颜色依次为黄色、蓝色、红色、紫色和绿色。
作为本发明实施例的一种改进,步骤S1中,挂锁探针集设计包括:m种颜色的区段序列中,选定p种颜色的区段序列包括分别与强度值为0、100%的荧光探针结合的两个子区段序列;其余(m-p)种颜色的区段序列包括分别与不同的强度值的荧光探针结合的多个子区段序列,(m-p)种颜色的强度值总和为0或100%,2≤子区段序列的个数≤10,0≤p≤3。可选的,其余(m-p)种颜色的区段序列包括分别与强度值为0、1/n±a
可选的,其余(m-p)种颜色的区段序列包括分别与强度值为0、1/n、2/n、3/n、……、(n-1)/n、1的荧光探针结合的n+1个子区段序列,1≤n≤9,0≤p≤3。当n为不同取值时,编码策略如图3所示。
其中,所选定的颜色即为在该待测样本中高自发荧光的荧光颜色,高自发荧光的荧光颜色优选为0种、1种或2种,但不超过3种。
作为本发明实施例的一种改进,步骤S2中染色包括:所选定颜色的荧光探针包括强度值分别为0、100%的荧光探针的集合;(m-p)种颜色的荧光探针包括强度值分别为0、1/n、2/n、3/n、……、(n-1)/n、1的荧光探针的集合,1≤n≤9,0≤p≤3。
作为本发明实施例的一种改进,步骤S4中解码包括:提取信号点的m个通道的颜色,并识别每个通道的颜色的强度值,根据每个通道的颜色的强度值,得到解码结果,解码结果为m位的数值,每一位数值对应一种颜色的强度值,所选定颜色识别强度值0和强度值100%。因为采用"总和恒定编码"原则,(m-p)个通道中的(m-p)个颜色的总强度值相同。为了将荧光强度归一化,将(m-p)个通道的总强度值设定为100%。
具体的,当m=4、p=1、n=4时,每条编码序列包括4个区段序列;选定一种颜色的区段序列包括分别与强度值为0、100%的荧光探针结合的两个子区段序列;其余3种颜色的区段序列包括分别与强度值为0、25%、50%、75%、100%的荧光探针结合的5个子区段序列,这三种颜色的强度值在任一barcode中的总和为100%。步骤S2中染色包括,所选定颜色的荧光探针包括强度值分别为0、100%的荧光探针的集合;其余3种颜色的荧光探针包括强度值分别为0、25%、50%、75%、100%的荧光探针的集合,荧光探针的强度值通过荧光探针与非荧光标记的探针的比例实现;例如,25%的红色对应的荧光探针,即该序列的荧光探针与非荧光标记的探针的比例为1:3。步骤S4中解码包括:提取信号点的4个通道的颜色,并识别每个通道的颜色的强度值,根据每个通道的颜色的强度值,得到解码结果,解码结果为4位的数值,每一位数值对应一种颜色的强度值,所选定颜色识别强度值0和强度值100%,其余3种颜色的强度值之和为0或100%,所有颜色不能同时为0。解码结果示例如表1所示:
表1
由表1的第一行可知,第一颜色强度值、第二颜色强度值、第三颜色强度值之和:0+25%+75%=100%,所选定颜色识别强度值100%;由第二行可知,第一颜色强度值、第二颜色强度值、第三颜色强度值之和:25%+25%+50%=100%,所选定颜色识别强度值0;由第三行可知,第一颜色强度值、第二颜色强度值、第三颜色强度值之和为0,所选定颜色识别强度值100%;
上述解码结果排列组合,可以得到31个编码序列库,编码如表2所示:为了表达的简洁性,将1代表25%、2代表50%、3代表75%、4代表100%。
表2
在具体的实施方式中,当待测样本高自发荧光为绿色,选择通道(532nm)为妥协通道,其余三个通道(488nm、594nm、647nm)进行荧光强度的识别,并采用"总和恒定编码"原则,3个通道中的3个颜色的总强度值相同。为了荧光强度的最大化,将总强度值设定为100%。Padlock探针设计示意图如图4所示,在编码序列中第一个颜色为黄色、第二个颜色为蓝色、第三个颜色为红色,第四个颜色为绿色。
由于RCA中所使用的酶的扩增活性不尽相同,不同扩增产物之间的拷贝数可能会有差异。这将导致一些编码序列无法区分。当扩增产物的扩增效率不同时,单独通道中的荧光强度值具有非常显著的差异,会给编码结果准确性带来严重偏差。当采用总和恒定编码策略时,如图5所示,通过计算一个点内不同通道的相对水平比,可以消除来自RCA的偏差,从而获得相同的解码结果“0314”。
本发明实施例利用总和恒定编码策略使本发明的方法能够实现对滚动圆积的大小无偏编码。本发明实施例的PRISM可通过一次性染色和成像在原位解析31种不同的核酸。多级编码有可能在不增加探针长度的情况下提高编码复用性。此外,本发明实施例将一个通道折衷为2级识别,并容忍组织本底荧光,这对高荧光背景组织(如人体肿瘤样本)很有帮助。
作为本发明实施例的一种改进,获得解码结果后,根据挂锁探针集设计时所确立的编码序列和靶标核酸对应关系,根据解码结果获得靶标核酸在待测样品中的种类和分布信息。
当m=4的示例中,本发明实施例在挂锁探针(Padlock探针)集设计节点,选择30个靶标核酸,按上述策略进行编码,实现了靶标核酸与编码序列的唯一对应关系。为了从原始图像中解码四色维度的信号点,将三种颜色(总和为4的三个)降维到xy平面,并将第四种颜色(妥协通道)设为z轴,从而将所有信号点置于三维颜色空间中,如图6所示。根据z值(0或4级)可将斑点分为两层,然后将每层投影到xy平面。由于缩小了维度,所有信号点都出现在一个三角形内,三个顶点分别是三个纯色斑点(400X、040X和004X)。由两种颜色组成的斑点出现在三角形的边上,可通过每条边上的频率分布加以区分。最后,由三种颜色组成的信号点会在三角形内部形成独立的三群。
根据所有信号点在颜色空间中的位置为其调用的编码信息(编码信息对应靶标核酸),示意图如图7所示。
为了验证解码的准确性,在拍照后清洗掉荧光探针,然后重新杂交特定基因的荧光探针并成像,编码序列的准确率平均为94.6%以上。
本发明实施例可针对各种不同的组织样本进行检测和分析,例如,冷冻组织切片(厚度一般为10微米)、石蜡包埋样品(厚度一般为10微米)。除了组织mRNA外,本发明实施例的检测方法还能检测病毒感染后组织中的短病毒片段(20~40bp)。此外,本发明实施例的检测方法还能对三维组织进行原位染色,例如厚度不大于100微米的组织,以获得三维原位的转录本信息。
为了保留精确的三维结构,直接对厚组织进行处理和成像至关重要。目前的三维多重RNA成像方法在多轮杂交和条带化过程中的信号点配准方面存在挑战(如EASI-FISH,STARmap等)。尤其是考虑到厚组织中试剂扩散时间较长,会使得这一问题更为严重。在本发明实施例的检测方法中,单轮染色和成像可以完美地避免这一问题。此外,本发明实施例的检测方法还能最大限度地减少光学衰减的影响,因为解码依靠的是荧光的相对比率而不是绝对强度。
当待测样本为厚度为大于10微米,且不大于100微米的组织时,S2完成后还包括有使用Cubic-L试剂除脂的步骤,并使用0.9M的碘海醇进行折射率匹配,以提高在厚组织中的光学透射率。
本发明实施例提出的PRISM具有快速、易用、经济高效的技术优势。标准化试剂和台上设置使其在普通生物实验室和医疗机构中得到普及。首先,采用本发明实施例的检测方法进行快速病理诊断,可提供丰富的分子模式信息,包括亚微米分辨率的病原体分布。其次,可以快速分析基因敲除等扰动带来的组织细胞层面的变化。为了全面捕捉这种变化,本发明实施例的检测方法可以在短时间内以低成本分析连续切片。此外,本发明实施例的检测方法还能帮助人们实现低成本、快速的药物发现。空间分辨基因表达谱分析对于全面了解组织对药物治疗的反应非常重要。然而,目前的多重方法(如RNAscope或空间转录组)对于高通量药物测试来说非常昂贵。本发明实施例的检测方法可以大大降低这种成本,从而加速药物发现过程。
本发明实施例还提出一种挂锁探针集,挂锁探针集包括多条挂锁探针;挂锁探针集中的挂锁探针的两个末端序列为和靶标RNA序列毗邻互补的结合序列,中间的连接序列为编码序列,编码序列用于对结合序列进行编码;
每条编码序列包括m个区段序列,每个区段序列用于结合一种颜色荧光探针,编码序列结合的m种荧光探针的颜色不同;2≤m≤8;每种颜色的区段序列包括分别至少两个子区段序列,子区段序列用于与同一颜色、不同强度值的荧光探针结合。
作为本发明实施的一种改进,m种颜色的区段序列中,选定p种颜色的区段序列包括分别与强度值为0、100%的荧光探针结合的两个子区段序列;其余(m-p)种颜色的区段序列包括分别与不同的强度值的荧光探针结合的多个子区段序列,(m-p)种颜色的强度值总和为0或100%,2≤子区段序列的个数≤10,0≤p≤3。
作为本发明实施的一种改进,其余(m-p)种颜色的区段序列包括分别与强度值为0、1/n±a
作为本发明实施的一种改进,其余(m-p)种颜色的区段序列包括分别与强度值为0、1/n、2/n、3/n、……、(n-1)/n、1的荧光探针结合的n+1个子区段序列,1≤n≤9,0≤p≤3。
作为本发明实施的一种改进,每条编码序列包括4个区段序列;选定一种颜色的区段序列包括分别与强度值为0、100%的荧光探针结合的两个子区段序列;其余3种颜色的区段序列包括分别与强度值为0、25%、50%、75%、100%的荧光探针结合的5个子区段序列。此时设计得到挂锁探针集包括31条挂锁探针。
作为本发明实施的一种改进,选定的一种颜色为绿色,其余三种颜色分别为红色、黄色和蓝色。
本发明实施例还提出一种多重核酸原位检测试剂盒,包括如上述挂锁探针集;试剂盒还包括荧光探针集,包括m种颜色的荧光探针;所选定p种颜色的荧光探针包括强度值分别为0、1的荧光探针的集合;(m–p)种颜色的荧光探针包括强度值分别为0、1/n±a
作为本发明实施的一种改进,(m-p)种颜色的荧光探针包括强度值分别为:0、1/n、2/n、3/n、……、(n-1)/n、1的荧光探针的集合,1≤n≤9,0≤p≤3。
作为本发明实施的一种改进,通过荧光标记探针与非荧光标记探针混合,获得同一颜色、不同强度值的荧光探针。
具体的,当m=4、p=1、n=4时,所选定颜色的荧光探针包括强度值分别为0、100%的荧光探针的集合;其余3种颜色的荧光探针包括强度值分别为0、25%、50%、75%、100%的荧光探针的集合,荧光探针的强度值通过荧光探针与非荧光标记的探针的比例实现。例如,25%的红色对应的荧光探针,即该序列的荧光探针与非荧光标记的探针的比例为1:3。
本发明实施例还提出上述方法或如上述试剂盒在基因敲除、药物测试、肿瘤分布检测、细胞分布分析或病原体分布分析中的应用;应用以单张切片、连续切片或厚组织的形式进行检测。
本发明实施例所用的实验装置、试剂等均为市售。
实施例1
在小鼠大脑冠状切片上测试本发明实施例的检测方法。实验的流程图如图1所示。
1、挂锁探针集的设计:挂锁探针集中的挂锁探针的两个末端序列为和靶标RNA序列毗邻互补的结合序列,中间的连接序列为编码序列,编码序列用于对结合序列进行编码;靶标RNA序列选择30个用于进行细胞分类的基因
每条编码序列包括4个区段序列,每个区段序列分别用于结合黄色、蓝色、红色、绿色的荧光探针;绿色的区段序列包括两个子区段序列,分别与强度值为0、100%的荧光探针结合;黄色、蓝色、红色的区段序列包括分别与强度值为0、25%、50%、75%、100%的荧光探针结合的5个子区段序列;31种编码序列如表4所示。
表4
/>
30种挂锁探针序列(5’端磷酸修饰)如表5所示:
表5
/>
/>
2、取小鼠大脑冠状冷冻切片,用4%多聚甲醛固定30分钟后,用PBST(PBS,0.05%Tween,下同)清洗一次,依次对组织进行消化(0.01%的胃蛋白酶和0.1M盐酸在37℃处理2分钟,然后用PBST清洗),脱水(80%乙醇处理10分钟,采用100%乙醇处理2分钟,然后用PBST处理3遍2分钟)和封闭(在Ampligase缓冲液中加入100nM的oligo dT、20%甲酰胺、50mM KCl、20μg/mL牛血清蛋白、20μg/mL酵母tRNA以及1U/μL的RNA酶抑制剂,室温处理30分钟)。
用设计的挂锁探针集合进行原位杂交,在Ampligase缓冲液[Lucigen]中加入挂锁探针使得每种挂锁探针终浓度为200nM/μL,20%甲酰胺,50mM KCl,20μg/mL牛血清蛋白,20μg/mL酵母tRNA[AM7119,Invitrogen]以及1U/μL的RNA酶抑制剂[Thermo Scientific]。在55℃下处理15分钟,然后在45℃下处理2小时。用10%甲酰胺和2x SSC溶液进行10分钟的清洗,清洗重复三次,随后用PBST再清洗三次。随后进行连接反应,反应体系为:SplintR缓冲液,20μg/mL牛血清蛋白,20μg/mL牛血清蛋白,1U/μL的RNA酶抑制剂以及2.5U/μL的SplintR连接酶[New England Biolabs]。连接反应在37℃下进行2小时,然后用PBST进行清洗。
然后进行RCA扩增;反应体系为:Phi29聚合酶缓冲液,0.25U/μL Phi29聚合酶(Thermo scientific),250μM dNTP,50μM氨基dUTP(Thermo scientific),10%甘油,20μg/mL牛血清蛋白以及600nM的扩增引物。扩增条件为30℃过夜。采用PBST清洗两遍,然后用10μg/μL的BS(PEG)9(Thermo scientific)固定,并用65%的甲酰胺清洗三遍。
3、采用4种颜色荧光探针对扩增产物进行染色和成像,待测样品中的每个靶标mRNA序列形成一个信号点;荧光探针序列如表6所示:
表6
/>
荧光探针按一定的比例加入,例如,同样是红色强度值100%对应的第一种子序列,荧光标记的探针与非荧光标记的探针的比例为4:0;同样是红色强度值75%对应的第二种子序列,荧光标记的探针与非荧光标记的探针的比例为3:1,以此类推。每一种子序列的荧光与非荧光探针的终浓度和为120nM,体系中除了17对子序列的探针外,还加入2×SSC溶液和20%的甲酰胺溶液。荧光探针加入组织后在50℃度处理9分钟,并在37℃度处理21分钟。随后用10%甲酰胺和2×SSC溶液进行10分钟的清洗,清洗重复三次,随后用PBST再清洗三次,然后即可用于成像。
绿色荧光探针包括强度值分别为0、100%的荧光探针的集合;
黄色、蓝色、红色的荧光探针包括强度值分别为0、25%、50%、75%、100%的荧光探针的集合。
4、对每个信号点采用4个通道的颜色和强度值进行解码,根据解码结果获得小鼠大脑冠状切片中靶标RNA的原位分布信息,得到的结果图如图8所示。
在实验后用65%甲酰胺洗脱荧光探针,然后重新杂交特定基因的荧光探针并成像,实验结果如图9所示,通过计算,各种编码序列的准确率平均为94.6%,结果如图10所示。图9为本发明实施例1中小鼠大脑冠状切片中靶标RNA的原位分布图与Allen脑数据库中每种RNA的原位分布图对比。
实施例2
采用本发明实施例方法分析E13.5期小鼠胚胎的矢状切片。
1、挂锁探针集的设计:挂锁探针集中的挂锁探针的两个末端序列为和靶标RNA序列毗邻互补的结合序列,中间的连接序列为编码序列,编码序列用于对结合序列进行编码;靶标RNA序列选择30个用于不同类型器官和组织的标记基因;
每条编码序列包括4个区段序列,每个区段序列分别用于结合黄色、蓝色、红色、绿色的荧光探针;绿色的区段序列包括两个子区段序列,分别与强度值为0、100%的荧光探针结合;黄色、蓝色、红色的区段序列包括分别与强度值为0、25%、50%、75%、100%的荧光探针结合的5个子区段序列;
30种挂锁探针序列(5’端磷酸修饰)如表7所示:
表7
/>
/>
2、取小鼠胚胎的矢状切片,固定条件同实施例1。消化时间延长至10分钟。脱水,封闭等条件同实施例1。
探针杂交和连接条件同实施例1。
RCA扩增反应时长增加至24小时,其余条件等同于实施例1。
3、采用4种颜色荧光探针对扩增产物进行染色和成像,待测样品中的每个靶标mRNA序列形成一个信号点;
荧光探针的设计同实施例1一致。
绿色荧光探针包括强度值分别为0、100%的荧光探针的集合;
黄色、蓝色、红色的荧光探针包括强度值分别为0、25%、50%、75%、100%的荧光探针的集合。
4、对每个信号点采用4个通道的颜色和强度值进行解码,根据解码结果获得小鼠胚胎的矢状切片中靶标RNA的原位分布信息,得到的结果如图11所示。如图12所示,该切片包含30个代表不同类型器官和组织的标记基因。根据每个细胞中表达丰度最高的基因进行细胞种类的标注,共有209,631个细胞被注释为12种细胞类型,包括表皮、骨骼和胃肠道等。然后将这些细胞的空间分布投影到它们各自的位置上,如图13、图14、图15所示。图16为实施例2中根据30种基因分布划出的不同细胞类型,右侧为相邻切片的HE染色对比图;此外,在一般细胞类型中,还可以进一步区分亚型,如神经系统,如图17所示。
实施例3
采用实施例2的方法,对单个E12.5小鼠胚胎的4个组织切片,单个E13.5小鼠胚胎的11个组织切片以及单个E14.5小鼠胚胎的11个组织切片(平均间隔280微米)进行了检测,绘制出具有单细胞分辨率的E12.5到E14.5胚胎空间图谱,不同平面上共有4,257,418个注释细胞。通过检查不同的切片,能够观察和分析不同系统的各种早期发育结构,实验结果如图18所示。
实施例4
PRISM描绘肝脏肿瘤微环境的复杂组织学图谱。
1、挂锁探针集的设计:挂锁探针集中的挂锁探针的两个末端序列为和靶标RNA序列毗邻互补的结合序列,中间的连接序列为编码序列,编码序列用于对结合序列进行编码;靶标RNA序列选择31个与细胞增殖、肝肿瘤、癌相关成纤维细胞(CAF)、内皮细胞和免疫细胞相关的标记基因,还包括针对28bp HBV核心蛋白编码RNA序列的特异性探针;
每条编码序列包括4个区段序列,每个区段序列分别用于结合黄色、蓝色、红色、绿色的荧光探针;绿色的区段序列包括两个子区段序列,分别与强度值为0、100%的荧光探针结合;黄色、蓝色、红色的区段序列包括分别与强度值为0、25%、50%、75%、100%的荧光探针结合的5个子区段序列;
挂锁探针序列(5’端磷酸修饰)如表8所示:
表8
/>
/>
/>
/>
/>
/>
2、取乙型肝炎病毒(HBV)阳性肝细胞癌(HCC)样本,后续所有步骤同实施例1。
3、采用4种颜色荧光探针对扩增产物进行染色和成像,待测样品中的每个靶标mRNA序列形成一个信号点;
荧光探针的设计同实施例1一致。
绿色荧光探针包括强度值分别为0、100%的荧光探针的集合;
黄色、蓝色、红色的荧光探针包括强度值分别为0、25%、50%、75%、100%的荧光探针的集合。
4、对每个信号点采用4个通道的颜色和强度值进行解码,根据解码结果获得乙型肝炎病毒(HBV)阳性肝细胞癌(HCC)样本中靶标RNA的原位分布信息,得到的结果图如图19所示。
结果表明,与传统的HE染色法相比,PRISM能够提供类似的组织学洞察力,能在分子水平上提供更有价值的信息。
实施例5
厚组织中的三维RNA原位染色
1、挂锁探针集的设计:挂锁探针集中的挂锁探针的两个末端序列为和靶标RNA序列毗邻互补的结合序列,中间的连接序列为编码序列,编码序列用于对结合序列进行编码;靶标RNA序列选择31个与细胞增殖、肝肿瘤、癌相关成纤维细胞(CAF)、内皮细胞和免疫细胞相关的标记基因,还包括针对28bp HBV核心蛋白编码RNA序列的特异性探针;
每条编码序列包括4个区段序列,每个区段序列分别用于结合黄色、蓝色、红色、绿色的荧光探针;绿色的区段序列包括两个子区段序列,分别与强度值为0、100%的荧光探针结合;黄色、蓝色、红色的区段序列包括分别与强度值为0、25%、50%、75%、100%的荧光探针结合的5个子区段序列;
挂锁探针序列同实施例1一致。
2、取100微米厚的小鼠脑组织样本,用PBST处理20分钟,重复三次,随后进行封闭(在Ampligase缓冲液中加入100nM的oligo dT,20%甲酰胺,50mM KCl,20μg/mL牛血清蛋白,20μg/mL酵母tRNA以及1U/μL的RNA酶抑制剂,室温处理30分钟)。
用设计的挂锁探针集合进行原位杂交,在Ampligase缓冲液中加入挂锁探针使得每种挂锁探针终浓度为200nM/μL,20%甲酰胺,50mM KCl,20μg/mL牛血清蛋白,20μg/mL酵母tRNA以及1U/μL的RNA酶抑制剂。在45℃下处理24小时。用10%甲酰胺和2×SSC溶液进行20分钟的清洗,清洗重复三次,PBST清洗三次。随后进行连接反应,反应体系为:SplintR缓冲液,20μg/mL牛血清蛋白,20μg/mL牛血清蛋白,1U/μL的RNA酶抑制剂以及2.5U/μL的SplintR连接酶。连接反应在37℃下过夜完成,完成后用PBST清洗两遍。
然后进行RCA扩增;反应体系为:Phi29聚合酶缓冲液,0.25U/μL Phi29聚合酶,250μM dNTP,50μM氨基dUTP,10%甘油,20μg/mL牛血清蛋白以及600nM的扩增引物。扩增条件为30℃24小时。扩增后采用PBST清洗两遍,用10μg/μL的BS(PEG)9固定,并用65%的甲酰胺清洗三遍。
3、采用4种颜色荧光探针对扩增产物进行染色和共聚焦成像,待测样品中的每个靶标mRNA序列形成一个信号点;
荧光探针的设计同实施例1一致。
绿色荧光探针包括强度值分别为0、100%的荧光探针的集合;
黄色、蓝色、红色的荧光探针包括强度值分别为0、25%、50%、75%、100%的荧光探针的集合。
4、首先在整个脑部的小区域取样,流程图如图20所示。检测沿Z方向的信号密度分布,这反映了试剂的穿透性和光透射率。结果表明,z方向的检测效率是一致的(图22)。在同一小鼠大脑切片(0.1毫米厚)上使用30个基因面板绘制了大尺度ROI(Region ofInterest),包括皮层、海马、丘脑和下丘脑,如图21所示。
以上所述仅是本发明的具体实施方式,使本领域技术人员能够理解或实现本发明。对这些实施例的多种修改对本领域的技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本发明的精神或范围的情况下,在其它实施例中实现。因此,本发明将不会被限制于本文所述的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
- 一种用于检测副猪嗜血杆菌的RPA引物、RPA探针、试剂盒及核酸检测方法
- 检测CHO核酸残留的引物、探针、试剂盒及检测方法
- 一种自组装核酸适配体/蛋白质复合纳米探针、制备方法、试剂盒及其应用
- 基于高分辨率熔解的多重环介导等温核酸扩增检测方法及试剂盒
- 应用多重探针熔解曲线法检测脑炎病毒核酸试剂盒及方法
- 基于探针熔解曲线分析的多重核酸检测方法和试剂盒
