嵌合抗原受体T细胞
文献发布时间:2024-04-18 20:01:23
本发明涉及嵌合抗原受体(CAR)T细胞,并且具体地但不排他地涉及抗CD4 CAR,以及它们在免疫疗法中的用途,以及用于治疗、预防或改善癌症(例如T细胞淋巴瘤)、各种微生物感染(例如HIV和TB)以及自身免疫性疾病的用途。本发明特别涉及CAR工程化粘膜相关恒定T(MAIT)细胞的用途,以及用于刺激、分离和扩增高度纯化的MAIT细胞的新方法,然后可以将其工程化改造为此类CAR-MAIT细胞。本发明延伸至遗传构建体本身,以及它们在产生CAR-MAIT细胞中的用途,以及转导的CAR-MAIT细胞本身。本发明还延伸至构建体和转导的CAR-MAIT细胞的各种医学用途,以及包含这些构建体和CAR-MAIT细胞的药物组合物。
T细胞淋巴瘤是一组异质性临床侵袭性疾病,包括外周T细胞淋巴瘤(PTCL),例如由人类T淋巴细胞病毒I型(HTLV-1)引起的成人T细胞白血病/淋巴瘤(ATL),皮肤T细胞淋巴瘤(CTCL),例如塞扎里综合征(SS)。T细胞恶性肿瘤比B细胞恶性肿瘤更难治疗。ATL和SS代表一种罕见且通常具有侵袭性的T细胞淋巴瘤类型,目前还没有足够的患者参加随机试验来建立治疗标准。因此,常用的一线疗法与治疗其他类型T细胞淋巴瘤的疗法相同。例如,目前获得许可的治疗T细胞恶性肿瘤的药物包括化疗药物、生物反应调节剂(例如干扰素、贝沙罗汀和HDAC抑制剂)、单克隆抗体(阿仑单抗、莫加穆利珠单抗、本妥昔单抗)、造血干细胞移植(HSCT)和体外光分离疗法(ECP)等。然而,没有一种治疗方案在其总体反应率或反应持续时间方面优于其他治疗方案。尽管同种异体(HSCT)是唯一潜在的治疗方案,但由于高龄和合并症以及HSCT相关死亡率,大量患者可能不适合HSCT。因此,需要更有效的治疗,因为目前ATL治疗的中位生存期仅为8个月(Katsuya等,2015)。
最近FDA批准的基于嵌合抗原受体(CAR)的T细胞疗法(CAR-T)被认为是治疗B细胞恶性肿瘤最重大的突破之一,通过靶向CD19抗原实现了近100%的缓解(Park等,2018)。尽管CAR-T治疗B细胞恶性肿瘤有效,但针对T细胞衍生恶性肿瘤的类似方法尚未建立。与大多数含有相同免疫球蛋白基因重排(即克隆)并表达泛B细胞标记物(例如CD19)的B细胞恶性肿瘤一样,大多数(>95%)ATL和CTCL源自表达确定的T细胞受体(TCR)基因(即克隆TCR-Vbeta链)和泛T辅助细胞标记物CD4的优势T细胞克隆。因此,通过单克隆抗体(mAb)靶向泛T细胞标记物(例如CD4或TCR-Vb链)来治疗T淋巴瘤已经过测试,但在小型临床试验中仅取得了部分疗效。
目前的CAR-T疗法主要基于传统的αβT细胞。然而,αβT细胞的抗原识别受到MHC的严重限制,这使得其适合自体治疗,但异体过继转移非常困难。此外,由于αβT细胞的肿瘤浸润不足,传统的CAR-T疗法在实体瘤(如CTCL)中显示出较低的疗效,但在液态肿瘤治疗中具有广阔的前景。然而,传统的CAR-T细胞疗法有一些主要缺点,限制了其进一步应用。例如,目前的CAR-T疗法:(i)受到移植物抗宿主病(GVHD)导致的自体输血的限制,(ii)受到细胞因子释放综合征的靶向/脱瘤毒性的限制;(iii)自体CAR-T的缺点,例如患者T细胞功能的变异性、产品标准化和成本。
因此,需要为T细胞恶性肿瘤(例如T细胞淋巴瘤,包括PTCL和CTCL)以及治疗微生物感染(例如HIV和TB)提供改进的免疫疗法。
为了治疗T细胞恶性肿瘤,发明人将注意力集中在粘膜相关恒定T细胞(MAIT细胞)上,它们是免疫系统中T细胞的一个亚群,表现出先天的类效应性。MAIT细胞由T细胞受体链Vα7.2的恒定使用定义,受主要组织相容性复合体(MHC)-Ib相关蛋白MR1的限制,并表现出C型凝集素CD161和IL18受体的高表达。在人类中,MAIT细胞存在于血液、肝脏、肺和粘膜中,可防御微生物活动和感染。MHC I类蛋白MR1负责将细菌产生的维生素B代谢物(例如5-OP-RU)呈递给MAIT细胞。MR1呈递外源抗原后,MAIT细胞会分泌促炎细胞因子,并能够裂解细菌感染的细胞。
目前的MAIT细胞扩增方法需要使用人同种异体饲养细胞来支持MAIT细胞在体外培养的生长,这对于人类适应性免疫治疗的大规模生产和质量控制来说是困难的。此外,使用这些已知方法产生的MAIT细胞含有一定比例的其他细胞亚群,例如常规CD4+T细胞和CD8+T细胞,因此这使得所得MAIT分离物完全不适合用于同种异体过继转移,因为这些细胞亚群会导致移植物抗宿主病(GvHD)。
因此,还需要一种改进的方法来刺激和分离纯MAIT细胞培养物,而不需要同种异体饲养细胞,以扩大MAIT细胞的生产,从而用于同种异体过继转移。
如示例中所述,发明人开发了一种用于从人PBMC中刺激和分离高纯度MAIT细胞培养物的新方法。发明人还开发了一些新颖的遗传构建体和载体(在本文中称为“CART4”和“CARTVb7.1”),它们编码嵌合抗原受体(CAR),然后转导至纯MAIT细胞中,从而产生CAR-MAIT细胞,这种细胞会特异性地靶向T细胞上的CD4分子(使用“CART4”)或TCR-Vbeta 7.1链(使用“CARTVb7.1”)。这是通过创建包含以下任一者的scFv的新型遗传构建体来实现的:(i)抗CD4 mAb(例如Hu5A8)或(ii)抗TCR-Vb 7.1mAb(例如3G5),与CD28/4-1BB/CD3zeta链信号传导部分一起形成第三代CAR。然后将这些CAR转导至从外周血单核细胞(PBMC)纯化的MAIT细胞中,以产生靶向T细胞上的CD4或T细胞上的TCR-Vbeta 7.1链的最终CAR-MAIT细胞。发明人已经证实,这些CAR-MAIT细胞令人惊讶地表现出与常规CAR-T细胞相当甚至更优异的抗T淋巴瘤活性。
因此,在本发明的第一方面,本文提供了表达嵌合抗原受体(CAR)的粘膜相关恒定T(MAIT)细胞。
如本文所述,MAIT细胞是表达恒定T细胞受体(TCR)的非常规和先天性T细胞,在哺乳动物进化过程中高度保守,识别MR1蛋白呈递的微生物抗原,并存在于人类血液中并维持组织稳态以获得广泛的抗菌反应性。此外,有利的是,MAIT细胞的抗原识别机制不依赖于MHC,这使得MAIT细胞成为同种异体T细胞杀伤疗法的有力的候选者,使得其不限于自体疗法,如当前依赖于MHC的T细胞疗法。换句话说,MAIT细胞具有较低的同种异体反应性,并且不易诱发人类移植物抗宿主病(GVHD),因此代表了同种异体CAR-T疗法的理想T细胞亚群。相比之下,本发明的CART-MAIT细胞和由此产生的细胞疗法可以容易地进行同种异体转移,并且MAIT细胞的内源性特征使其成为浸润到外周组织用于实体瘤治疗的有希望的候选者。因此,CAR-MAIT细胞能够有效地渗透到实体瘤中,这使得基于MAIT细胞的细胞疗法成为实体瘤和液态肿瘤的有前景的疗法。
有利的是,本发明的CAR-MAIT细胞可用于治疗T细胞恶性肿瘤,例如由人类T淋巴细胞病毒I型(HTLV-1)引起的成人T细胞白血病/淋巴瘤(ATL),皮肤T细胞淋巴瘤(CTCL),例如塞扎里综合征(SS)。应当理解,T细胞恶性肿瘤是一组异质性临床侵袭性疾病,并且比B细胞恶性肿瘤更难治疗。有利的是,CAR-MAIT疗法预计对实体瘤表现出增强的功效并且是同种异体的,从而不需要自体移植,因此将其定位为即用型疗法。
优选地,在一个实施方案中,CAR-MAIT细胞表达靶向T细胞上的CD4抗原的CAR。因此,优选地,CAR对T细胞上的CD4抗原具有特异性。应当理解,CD4抗原是在免疫细胞例如T辅助细胞、单核细胞、巨噬细胞和树突细胞表面上发现的糖蛋白(T细胞表面糖蛋白CD4[智人]UniProt No.P01730.1;NCBI参考序列NP_000607.1)。CD4抗原的多肽序列的一个实施方案在本文中表示为SEQ ID No:1,如下:
[SEQ ID No:1]
因此,优选地,CAR对包含基本上如SEQ ID No:1所示的氨基酸序列或其变体或片段的CD4抗原具有特异性。
优选地,在另一个实施方案中,CAR-MAIT细胞表达靶向T细胞上的T细胞受体(TCR)β链可变区(Vbeta)的CAR。应当理解,T细胞受体(TCR)是在T细胞或T淋巴细胞表面上发现的蛋白质复合物,其负责将抗原片段识别为与主要组织相容性复合物(MHC)分子结合的肽。TCR由两条不同的蛋白质链组成。在人类中,95%的T细胞中的TCR由TRA编码的alpha(α)链和TRB编码的beta(β)链组成。
下面的表1列出了T细胞上的Vbeta区以及相关的编码基因,并且它们中的任何一个或多个可以被本发明的CAR-MAIT细胞上的CAR靶向。
表1-T细胞上的β链可变区(Vbeta)
CAR-MAIT细胞可以表达靶向T细胞上的多个T细胞受体(TCR)β链可变区(Vbeta)的CAR。优选地,多个Vbeta区可以选自表1中所示的一组Vbeta区。例如,CAR-MAIT细胞可以表达靶向T细胞上的至少两个、至少三个或至少四个T细胞受体(TCR)β链可变区(Vbeta)的CAR,优选地如表1所示。CAR-MAIT细胞可以表达靶向T细胞上的至少五个、至少六个或至少七个T细胞受体(TCR)β链可变区(Vbeta)的CAR,优选地如表1所示。多个TCR Vbeta区可以是相同或不同的Vbeta区。
以下Vb家族被认为经常与T细胞淋巴瘤相关,即Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20。因此,在优选的实施方案中,CAR-MAIT细胞表达靶向T细胞上的一个或多个TCR Vbeta区的CAR,这些Vbeta区选自以下:Vb 1、Vb 2、Vb3、Vb 5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20。优选地,CAR-MAIT细胞表达靶向T细胞上的至少两个或三个TCR Vbeta区的CAR,这些Vbeta区选自以下:Vb 1、Vb 2、Vb 3、Vb 5.1、Vb7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20。
因此,优选地,CAR对T细胞上的至少一个或多个TCR Vbeta区具有特异性,最优选TCR-Vbeta 7.1链。TCR Vbeta 7.1区的多肽序列(智人重排的TCR Vbeta 7.1UniProtKB/Swiss-Prot:A0A577.1)的一个实施方案在本文中表示为SEQ ID No:2,如下:
MGCRLLCCAVLCLLGAVPIDTEVTQTPKHLVMGMTNKKSLKCEQHMGHRAMYWYKQKAKKPPELMFVYSYEKLSINESVPSRFSPECPNSSLLNLHLHALQPEDSALYLCASSQ
[SEQ ID No:2]
因此,优选地,CAR对包含基本上如SEQ ID No:2所示的氨基酸序列或其变体或片段的至少一个或多个TCR Vbeta区(更优选TCR-Vbeta 7.1链)具有特异性。
优选地,CAR-MAIT细胞被配置为通过诱导细胞凋亡来直接杀死靶向T细胞。
优选地,CAR-MAIT细胞包含一种或多种编码序列,其允许可控地或诱导地消除CAR-MAIT细胞,例如在患者不良反应的情况下。技术人员可以将该一种或多种编码序列称为所谓的“自杀基因”。
因此,在一个实施方案中,一种或多种编码序列可以编码表皮生长因子受体(EGFR)或截短的表皮生长因子受体(tEGFR)(参考)(UniProt No.P00533;NCBI参考序列NP_001333826.1)。tEGFR的表达可以通过抗EGFR mAb(西妥昔单抗)来控制,以监测或消除患者体内的CAR-T细胞。应当理解,EGFR在人类中被称为HER1,并且是跨膜蛋白,其是细胞外蛋白配体的表皮生长因子(EGF)成员的受体(UniProt No.P01133;NCBI参考序列NP_001171601.1)。
在另一个实施方案中,一种或多种编码序列可以编码诱导型半胱天冬酶9(iC9)(Mol.Therapy,Diaconu et al.,580,25,3,March 2017)。iC9是一种修饰的人半胱天冬酶9(UniProt No.P55211;NCBI参考序列NP_001220.2),与人FK506结合蛋白(UniProtNo.P62942;NCBI参考序列NP_000792.1)融合,允许使用化学诱导剂进行条件二聚化(半胱天冬酶诱导药物(CID),Rimiducid),触发表达融合蛋白的CAR-T细胞凋亡。
优选地,CAR-MAIT细胞包含编码截短的表皮生长因子受体(tEGFR)和/或诱导型半胱天冬酶9(iC9)的编码序列。更优选地,CAR-MAIT细胞包含编码截短的表皮生长因子受体(tEGFR)和诱导型半胱天冬酶9(iC9)的编码序列。
应当理解,CAR-MAIT细胞是通过用编码CAR的核酸或遗传构建体转导MAIT细胞来产生的。将高度纯化的MAIT细胞的培养物用于CAR转导步骤以产生T细胞特异性和活性CAR-MAIT细胞是重要的。因此,本发明人开发了一种从人外周血单核细胞(PBMC)中分离纯化的MAIT细胞的有效方法,通过结合磁激活细胞分选(MACS)和荧光激活细胞分选(FACS),使得所得方法得到大量具有高扩增倍数的MAIT细胞。
因此,优选地,MAIT细胞分离自人外周血单核细胞(PBMC)。优选地,通过磁激活细胞分选(MACS)和/或荧光激活细胞分选(FACS),更优选MACS和FACS两者,从PBMC分离MAIT细胞。发明人相信他们的MAIT分离方法本身是新颖的。
因此,在第二方面,本文提供了分离MAIT细胞的方法,该方法包括:
(i)提供外周血单核细胞(PBMC);和
(ii)对PBMC进行磁激活细胞分选(MACS)和/或荧光激活细胞分选(FACS)以从中分离MAIT细胞。
优选地,本发明的方法导致纯的离体MAIT细胞的分离。在一个实施方案中,该方法包括对PBMC进行MACS或FACS以从中分离MAIT细胞。然而,优选地,该方法包括对PBMC进行MACS和FACS两者以从中分离MAIT细胞。优选地,首先对PBMC进行MACS,然后进行FACS。优选地,该方法包括通过MACS从PBMC分离TCR Vα7.2+细胞,然后使用MR1-5-OP-RU四聚体染色对这些细胞进行FACS,以分离MAIT细胞。
MACS程序(在步骤(ii)中)可以包括收集PBMC,然后用结合缓冲液洗涤细胞。可弃去上清液,并将所得细胞沉淀重悬于MACS缓冲液中(例如浓度为1x 10
FACS程序(在步骤(ii)中)可以包括收集磁分离的细胞,然后离心(例如以300x g离心5分钟)。可以将细胞重悬(例如用FACS缓冲液以10
在用编码CAR的核酸转导分离的MAIT细胞之前,优选地在第二方面的方法中在步骤(ii)之后的后续步骤中激活MAIT细胞。因此,该方法还包括用抗CD3抗体激活分离的MAIT细胞,优选在体外激活。该方法包括用抗CD28抗体激活分离的MAIT细胞,优选在体外激活。优选地,用抗CD3和抗CD28抗体基本上同时地或依次地激活分离的MAIT细胞。依次激活可以包括使MAIT细胞首先与抗CD3接触,然后与抗CD28抗体接触,或者首先与抗CD28抗体接触,然后与抗CD3抗体接触。与抗体接触可以持续至少一天、两天或三天。
MAIT细胞激活程序可以包括通过离心(例如以300x g离心5分钟)收集分选的MAIT细胞。可以弃去上清液,并且可以将沉淀重悬(例如在R10培养基中至10
然后,该方法可以包括用编码CAR的核酸转导分离的且现已激活的MAIT细胞。
MAIT细胞是先天性T细胞的一个亚群,定义为CD3
因此,在优选的实施方案中,该方法可以包括在对PBMC进行MACS和/或FACS步骤(即步骤ii)之前刺激PBMC。优选地,该初始的刺激步骤包括使PBMC接触:(a)包含MR1/5-OP-RU或5-OP-RU的抗原;和/或(b)细胞因子。优选地,刺激步骤包括使PBMC接触:(a)包含MR1/5-OP-RU或5-OP-RU的抗原;和(b)细胞因子。刺激步骤可以包括使PBMC接触抗原和/或细胞因子至少1天、2天或3天。刺激步骤可以持续至少4天、5天或6天。优选地,刺激步骤包括使PBMC接触体外培养物中的抗原和/或细胞因子。
WO 2015/149130描述了MR1/5-OP-RU和5-OP-RU,其全部内容通过引用并入本文。因此,优选地,抗原包含MR1/5-OP-RU或5-OP-RU,如WO 2015/149130(PCT/AU2015/050148)中所述。
细胞因子可以是任何白细胞介素。然而,优选地,细胞因子可以是选自IL-2、IL-7、IL-12、IL-15、IL-18和IL-23或其任意组合中的一种或多种白细胞介素。白细胞介素的浓度可以是至少5ng/ml、10ng/ml或20ng/ml,优选至少30ng/ml、40ng/ml或50ng/ml。
例如,一种或多种白细胞介素可包含:(i)单独的IL-2(图15中的条件1);(ii)IL-12和IL-18(图15中的条件11);(iii)IL-2、IL-12和IL-18(图15中的条件3);(iv)IL-12、IL-18和IL-23(图15中的条件12);(v)IL-2、IL-12、IL-18和IL-23(图15中的条件13),或(vi)IL-7、IL-15、IL-12和IL-18(图15中的条件8)。
最优选地,一种或多种白细胞介素可以包含IL-12、IL-18和IL-23的组合。因此,优选地,刺激步骤包括使PBMC接触:(a)包含MR1/5-OP-RU或5-OP-RU的抗原;和(b)IL-12、IL-18和IL-23的组合。
发明人相信他们已经设计出一种在PBMC培养物中刺激MAIT细胞的新方法。
因此,在另一个方面,本文提供了在PBMC培养物中刺激MAIT细胞的方法,该方法包括使PMBC培养物接触:(a)包含MR1/5-OP-RU或5-OP-RU的抗原;和/或(b)选自IL-2、IL-7、IL-12、IL-15、IL-18和IL-23或其任意组合中的一种或多种白细胞介素。
优选地,一种或多种白细胞介素是IL-12、IL-18和/或IL-23。更优选地,一种或多种白细胞介素是IL-12、IL-18和IL-23。
发明人相信他们的产生CAR-MAIT细胞的方法本身也是新颖的。
因此,在第三方面,本文提供了一种产生CAR-MAIT细胞的方法,该方法包括:
(i)提供外周血单核细胞(PBMC);
(ii)对PBMC进行MACS和/或FACS以从中分离MAIT细胞;
(iii)激活分离的MAIT细胞,任选地通过使它们与抗CD3和/或抗CD28抗体接触;和
(iv)用编码CAR的核酸转导激活的MAIT细胞,从而产生CAR-MAIT细胞。
第三方面的方法的步骤(i)、(ii)和/或(iii)可以与本文中关于第二方面的方法描述的步骤相同,因此这些方法步骤是可互换的。
另外,优选地,该方法包括在对PBMC进行MACS和/或FACS步骤(即步骤ii)之前刺激PBMC。优选地,该初始的刺激步骤包括使PBMC接触:(a)包含MR1/5-OP-RU或5-OP-RU的抗原;和/或(b)细胞因子。优选地,刺激步骤包括使PBMC接触:(a)包含MR1/5-OP-RU或5-OP-RU的抗原;和(b)细胞因子。细胞因子可以是关于第二方面描述的白细胞介素,优选选自IL-2、IL-7、IL-12、IL-15、IL-18和IL-23或其任意组合中的一种或多种白细胞介素,如上所述。
最优选地,一种或多种白细胞介素可以包含IL-12、IL-18和IL-23的组合。因此,优选地,刺激步骤包括使PBMC接触:(a)包含MR1/5-OP-RU或5-OP-RU的抗原;和(b)IL-12、IL-18和IL-23的组合。
优选地,在步骤(iv)用编码CAR的核酸转导分离的MAIT细胞之前,在步骤(iii)中激活MAIT细胞。分离的MAIT细胞可用抗CD3抗体激活,优选在体外激活。分离的MAIT细胞可以用抗CD28抗体激活,优选在体外激活。优选地,用抗CD3和抗CD28抗体基本上同时地或依次地激活分离的MAIT细胞,如关于第二方面的方法所述。
MAIT细胞转导程序(即步骤(iv))可以包括用编码CAR的核酸转导MAIT细胞。转导步骤可以包括病毒转导。优选地,MAIT细胞转导程序包括用编码CAR的核酸通过逆转录病毒转导MAIT细胞。MAIT细胞可以用如本文所述的编码CAR的任何核酸转导,例如根据第五方面或根据第六方面的载体。核酸可编码靶向T细胞上的CD4抗原或至少一个或多个TCR Vbeta区的CAR。优选地,核酸可以编码靶向T细胞上表1中所示的一个或多个TCR Vbeta区(并且更优选TCR-Vbeta 7.1链)的CAR。该核酸可以编码靶向T细胞上的一个或多个TCR Vbeta区的CAR,该TCR Vbeta区选自以下:Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb17和Vb 20。
优选地,转导在MAIT细胞激活后至少34小时、36小时或48小时进行。在转导之前(例如大约1天前),可以制备RetroNectin包被的板。可以将RetroNectin(例如约15μg)与PBS(例如约1ml)接触以形成溶液。该方法可以包括将溶液引入到未经组织培养物处理的24孔板的一个孔中。可以将板包裹起来(例如用保鲜膜)并在约4℃下保存(例如在冰箱中过夜)。在基因转移当天,将未结合的RetroNectin从孔中除去。可以清洗孔(例如用2ml PBS清洗一次或两次)。优选地,不允许孔干燥。逆转录病毒上清液可以解冻(例如在37℃水浴中)。优选将病毒上清液(例如约1ml)转移至RetroNectin包被的板的每个孔中。板可以用保鲜膜包裹。可将板离心(例如在32℃下以1000x g离心2小时)。离心时,可以收集激活的MAIT细胞。收集的细胞可以重悬(例如在含有100IU/ml IL-2的新鲜R10培养基中,至浓度1x10
在第三方面的方法中,本发明的方法优选地包括在步骤(iv)之后的后续步骤中扩增CAR-MAIT细胞。CAR-MAIT细胞扩增步骤可以包括在转导后一天或两天收获转导的CAR-MAIT细胞,优选使用逆转录病毒或病毒。可以对收获的细胞进行计数(例如通过血细胞计数器)。然后可以将收获的细胞转移到板的孔中(例如,将1x 10
有利的是,如实施例中所述,培养11天产生100倍扩增水平的CAR-MAIT细胞,且转导效率高于50%。发明人观察到,CAR-MAIT细胞令人惊讶地表现出至少与表达常规CAR的CD8+T细胞相当的细胞毒性效力。
在第四方面,本文提供了通过第三方面的方法获得的或可获得的CAR-MAIT细胞。
如本文所述,使用第二方面或第三方面的方法获得的分离的MAIT细胞可以被激活,并且最终用编码CAR的核酸构建体转导以产生第一方面或第四方面的CAR-MAIT细胞。如本文所述,发明人开发了编码CAR的新型遗传构建体和重组载体,其特异性靶向CD4分子(在这种情况下构建体和载体在本文中称为“CART4”)或一个或多个TCR-Vbeta区,例如T细胞上的TCR-Vbeta 7.1链(在这种情况下构建体和载体在本文中称为“CARTVb7.1”)。这些构建体和载体中的任一种都可以用于在第三方面或第四方面的方法中转导MAIT细胞。
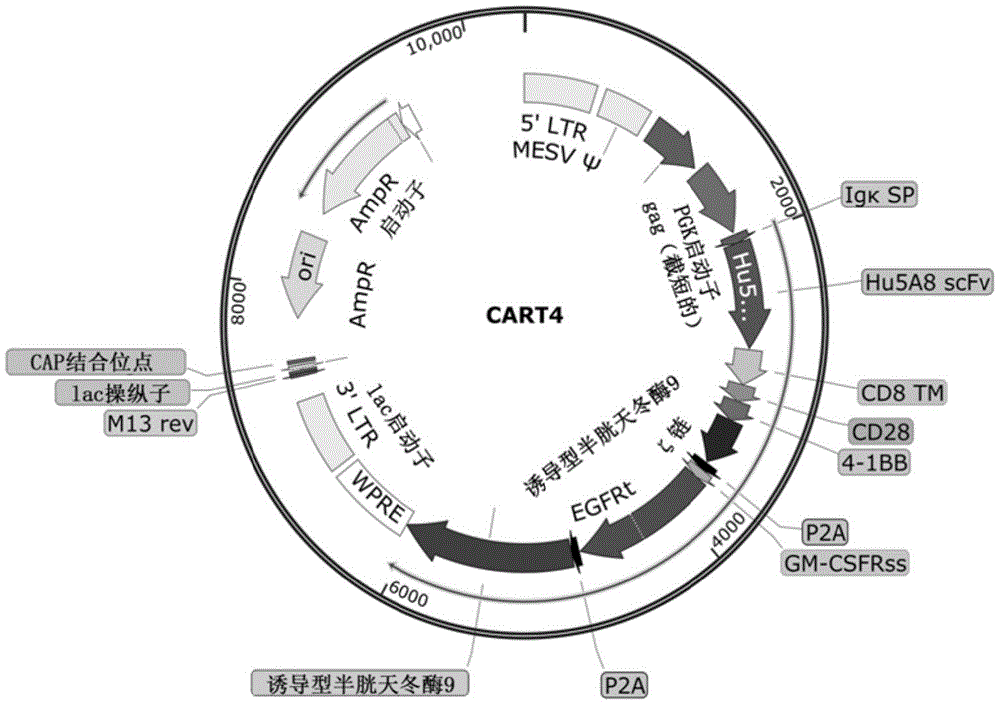
遗传构建体包含以下任一者的scFv:(i)抗CD4 mAb(例如Hu5A8)或(ii)抗TCR-VbmAb,例如抗TCR-Vb 7.1mAb(例如3G5),与CD28/4-1BB/CD3zeta链信号传导部分一起形成第三代CAR。编码CAR的构建体还包含至少一个由所谓的自杀基因编码的安全开关,例如截短的表皮生长因子受体(tEGFR)和/或诱导型半胱天冬酶9(iC9),并且其能够清除所得的CAR-T细胞,因此在治疗中使用CAR-T细胞时提供了一个精妙的监控系统或安全机制。tEGFR的表达可以被抗EGFR mAb识别(例如西妥昔单抗)用于监测或消除CAR-T细胞,而iC9是一种与人FK506结合蛋白融合的修饰人半胱天冬酶9,可使用二聚化化学诱导剂(例如Rimiducid)进行条件二聚化,从而触发表达融合蛋白的CAR-T细胞凋亡。发明人相信,他们开发的CAR编码核酸构建体本身是新颖的。参考图1A(1和2),该示意图说明了包括在根据本发明的CAR编码构建体的两个实施例中的功能元件,即,图1A(1)示出“CART4”,图1A(2)示出“CARTVb7.1”。然而,应当理解,具有靶向Vbeta 7.1家族的抗TCR-Vb 7.1的“CARTVb7.1”纯粹是说明性的,任何Vbeta区都可以被CAR靶向,例如表1中所示的任何Vbeta,尤其是Vb 1、Vb 2、Vb 3、Vb5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20中的任一个。
因此,在第五方面,本文提供了包含可操作地连接至第一编码序列的启动子,所述第一编码序列编码抗CD4嵌合抗原受体(CAR)或抗T细胞受体(TCR)Vbeta CAR。
启动子可以是任何合适的启动子,包括组成型启动子、激活型启动子、诱导型启动子或组织特异型启动子。组成型启动子允许异源基因(也称为转基因)在宿主细胞中组成型表达。本文考虑的示例性组成型启动子包括但不限于巨细胞病毒(CMV)启动子、人延伸因子-1α(hEFla)、泛素C启动子(UbiC)、磷酸甘油激酶启动子(PGK)、猿猴病毒40早期启动子(SV40),以及与CMV早期增强子(CAGG)偶联的鸡β-肌动蛋白启动子。诱导型启动子属于调控型启动子的范畴。诱导型启动子可以由一种或多种条件诱导,例如物理条件、工程化免疫效应细胞的微环境或工程化免疫效应细胞的生理状态、诱导物(即诱导剂)或其组合。在一些实施方案中,诱导条件不诱导工程化哺乳动物细胞中和/或接受药物组合物的受试者中内源基因的表达。在一些实施方案中,诱导条件选自以下:诱导剂、辐射(例如电离辐射、光)、温度(例如热)、氧化还原状态、肿瘤环境和工程化哺乳动物细胞的激活状态。
在一个实施方案中,启动子可以是PGK启动子(EMBL NO:A19297.1)。在一个实施方案中,PGK启动子在本文中被称为SEQ ID No:3,如下:
GGGTAGGGGAGGCGCTTTTCCCAAGGCAGTCTGGAGCATGCGCTTTAGCAGCCCCGCTGGGCACTTGGCGCTACACAAGTGGCCTCTGGCCTCGCACACATTCCACATCCACCGGTAGGCGCCAACCGGCTCCGTTCTTTGGTGGCCCCTTCGCGCCACCTTCTACTCCTCCCCTAGTCAGGAAGTTCCCCCCCGCCCCGCAGCTCGCGTCGTGCAGGACGTGACAAATGGAAGTAGCACGTCTCACTAGTCTCGTGCAGATGGACAGCACCGCTGAGCAATGGAAGCGGGTAGGCCTTTGGGGCAGCGGCCAATAGCAGCTTTGCTCCTTCGCTTTCTGGGCTCAGAGGCTGGGAAGGGGTGGGTCCGGGGGCGGGCTCAGGGGCGGGCTCAGGGGCGGGGCGGGCGCCCGAAGGTCCTCCGGAGGCCCGGCATTCTGCACGCTTCAAAAGCGCACGTCTGCCGCGCTGTTCTCCTCTTCCTCATTCTCCGGGCCTTTCG
[SEQ ID No:3]
因此,优选地,启动子可以包含基本上如SEQ ID No:3所示的核苷酸序列或其片段或变体。
核酸构建体可以包含编码信号肽的核苷酸序列。有利的是,信号肽被配置为将CAR(即,其是融合蛋白)引导至T细胞外膜。优选地,编码信号肽的序列位于启动子的3’。优选地,信号肽是Igκ信号肽。在一个实施方案中,信号肽可具有在本文中称为SEQ ID No:4的氨基酸序列,如下:
METDTLLLWVLLLWVPGSTGD
[SEQ ID No:4]
因此,优选地,构建体包含编码具有基本上如SEQ ID No:4所示的氨基酸序列的信号肽的核苷酸序列或其片段或变体。
在一个实施方案中,编码信号肽的核苷酸序列在本文中称为SEQ ID No:5,如下:
ATGGAGACAGACACACTCCTGCTATGGGTGCTGCTGCTCTGGGTTCCAGGTTCCACAGGTGAC
[SEQ ID No:5]
因此,优选地,构建体包含基本上如SEQ ID No:5所示的核苷酸序列或其片段或变体。
优选地,第一编码序列位于编码信号肽的序列的3’。在核酸构建体的第一实施方案中,第一编码序列编码抗CD4嵌合抗原受体(CAR)。优选地,CAR对包含基本上如SEQ IDNo:1所示的氨基酸序列或其变体或片段的CD4抗原具有特异性。
如图1A(1)(“CART4”)所示,第一编码序列可编码scFv区,其可包含VL(可变轻链)序列和VH(可变重链)序列。优选地,VL序列位于VH序列的上游(即5’)。然而,在一些实施方案中,VH序列可以位于VL序列的上游。
在一个实施方案中,VL和VH序列可以是用于结合T细胞上的CD4抗原的Hu5A8(即抗CD4单克隆抗体的杂交瘤克隆名称)轻链可变区和重链可变区。
因此,在一个实施方案中,第一编码序列(其可编码用于结合CD4的VL序列)编码在本文中称为SEQ ID No:6的氨基酸序列,如下:
DIVMTQSPDSLAVSLGERVTMNCKSSQSLLYSTNQKNYLAWYQQKPGQSPKLLIYWASTRESGVPDRFSGSGSGTDFTLTISSVQAEDVAVYYCQQYYSYRTFGGGTKLEIK
[SEQ ID No:6]
因此,优选地,第一编码序列包含编码基本上如SEQ ID No:6所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,第一编码序列(其可编码用于结合CD4的VL序列)包含在本文中称为SEQ ID No:7的核苷酸序列,如下:
GACATTGTGATGACTCAGAGCCCCGACAGCCTGGCCGTCTCACTGGGCGAAAGGGTGACCATGAATTGTAAATCTTCTCAGAGCCTGCTGTACAGTACAAACCAGAAAAATTACCTGGCCTGGTATCAGCAGAAACCCGGCCAGAGCCCTAAGCTGCTGATCTATTGGGCAAGTACCCGAGAGTCAGGAGTGCCAGACAGATTCTCCGGGTCTGGAAGTGGCACAGACTTCACCCTGACAATTAGCTCCGTGCAGGCCGAGGACGTGGCTGTCTACTATTGCCAGCAGTACTATAGCTACCGAACTTTCGGCGGGGGAACCAAACTGGAAATCAAG
[SEQ ID No:7]
因此,优选地,第一编码序列包含基本上如SEQ ID No:7所示的核苷酸序列或其片段或变体。
在一个实施方案中,第一编码序列(其可编码用于结合CD4的VH序列)编码在本文中称为SEQ ID No:8的氨基酸序列,如下:
QVQLQQSGPEVVKPGASVKMSCKASGYTFTSYVIHWVRQKPGQGLDWIGYINPYNDGTDYDEKFKGKATLTSDTSTSTAYMELSSLRSEDTAVYYCAREKDNYATGAWFAYWGQGTLVTVSS
[SEQ ID No:8]
因此,优选地,第一编码序列包含编码基本上如SEQ ID No:8所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,第一编码序列(其可编码用于结合CD4的VH序列)包含在本文中称为SEQ ID No:9的核苷酸序列,如下:
CAGGTGCAGCTGCAGCAGTCCGGACCAGAGGTGGTCAAACCCGGCGCTAGCGTCAAAATGTCCTGTAAGGCATCTGGCTACACTTTCACCTCTTATGTGATTCACTGGGTCAGACAGAAGCCTGGGCAGGGACTGGACTGGATCGGGTACATTAACCCATATAATGATGGAACTGACTACGATGAAAAGTTTAAAGGCAAGGCCACACTGACTTCCGACACCTCAACAAGCACTGCTTATATGGAGCTGTCTAGTCTGAGGTCTGAAGACACAGCAGTGTACTATTGCGCCCGCGAGAAGGATAACTACGCCACTGGCGCTTGGTTTGCATATTGGGGCCAGGGGACCCTGGTGACAGTCTCATCC
[SEQ ID No:9]
因此,优选地,第一编码序列包含基本上如SEQ ID No:9所示的核苷酸序列或其片段或变体。
优选地,VH(例如SEQ ID No:9)和VL(例如SEQ ID No:7)序列,当处于任一方向时,均由接头序列分隔开。在一个实施方案中,接头序列可以是G4S接头序列,其在本文中称为SEQ ID No:10,如下:
GGGGSGGGGSGGGGS
[SEQ ID No:10]
因此,优选地,第一编码序列包含编码基本上如SEQ ID No:10所示的氨基酸序列或其片段或变体。
在一个实施方案中,接头序列可以由在本文中称为SEQ ID No:11的核苷酸序列编码,如下:
GAGGAGGAGGCAGTGGCGGAGGAGGGTCAGGAGGAGGAGGAAGC
[SEQ ID No:11]
因此,优选地,接头序列包含基本上如SEQ ID No:11所示的核苷酸序列或其片段或变体。
然而,在核酸构建体(“CARTVb7.1”)的第二实施方案中,第一编码序列编码抗T细胞受体(TCR)Vbeta区CAR。应当理解,任何Vbeta区都可以被CAR靶向。例如,表1中列出的任何Vbeta区可以被第一编码序列编码的CAR靶向。
第一编码序列可以编码多个T细胞受体(TCR)β链可变区(Vbeta)CAR。优选地,多个Vbeta区可以选自表1中所示的一组Vbeta区。还可以在同一构建体上组合两个或多个靶向Vbeta区的CAR。例如,构建体可以包含编码至少两个、至少三个或至少四个T细胞受体(TCR)β链可变区(Vbeta)靶向CAR的编码序列,优选地如表1中所示。构建体可以包含编码至少五个、至少六个或至少七个T细胞受体(TCR)β链可变区(Vbeta)靶向CAR的编码序列,优选地如表1中所列。多个TCR Vbeta区可以是相同或不同的Vbeta区。
以下Vb家族被认为经常与T细胞淋巴瘤相关,即Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20。因此,在一个优选的实施方案中,构建体包含编码至少一种CAR的编码序列,该CAR靶向T细胞上的一个或多个TCR Vbeta区,其选自以下Vbeta区:Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb 8、Vb 12,Vb 13.1、Vb 17和Vb 20。优选地,构建体包含编码至少一种CAR的编码序列,该CAR靶向T细胞上的至少两个或三个TCR Vbeta区,其选自以下Vbeta区:Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20。
因此,优选地,本文提供了包含可操作地连接至第一编码序列的启动子的核酸构建体,其中第一编码序列编码多个抗T细胞受体(TCR)Vbeta CAR,其中T细胞受体(TCR)Vbeta CAR上的不同Vbeta区细胞被靶向。
优选地,CAR对TCR Vbeta区(优选TCR-Vbeta 7.1链)具有特异性,其包含基本上如SEQ ID No:2所示的氨基酸序列或其变体或片段。
如图1A(2)所示,第一编码序列可编码scFv区,其可包含VL(可变轻链)序列和VH(可变重链)序列。优选地,VL序列位于VH序列的上游(即5’)。然而,在一些实施方案中,VH序列可以位于VL序列的上游。优选地,VH和VL编码序列当处于任一方向时均被接头序列(例如G4S接头序列)分隔开。
在一个实施方案中,VL和VH序列可以是用于结合TCR Vbeta 7.1抗原的3G5(即抗TCR Vbeta 7.1单克隆抗体的杂交瘤克隆名称)轻链可变区和重链可变区。
在一个实施方案中,第一编码序列(其可编码用于结合TCR Vbeta、优选TCR-Vbeta7.1链的VL序列)编码在本文中称为SEQ ID No:12的氨基酸序列,如下:
QVQLQQPGAELVKPGASVKMSCKASGYTFTRYWITWVKQRPGQGLEWIGDIYPGSGFTKYNEKFKSKATLTVDTSSSTAYMQLSSLTSEDSAVYYCAREGGNYWYFDVWGTGTTVTVSS
[SEQ ID No:12]
因此,优选地,第一编码序列包含编码基本上如SEQ ID No:12所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,第一编码序列(其可编码用于结合TCR Vbeta、优选TCR-Vbeta7.1链的VL序列)包含在本文中称为SEQ ID No:13的核苷酸序列,如下:
CAAGTTCAGCTGCAACAGCCTGGCGCCGAGCTTGTGAAACCTGGCGCCTCTGTGAAGATGAGCTGCAAGGCCTCCGGCTACACCTTCACCAGATACTGGATCACCTGGGTCAAGCAGAGGCCTGGACAGGGACTCGAGTGGATCGGCGATATCTATCCTGGCTCCGGCTTCACCAAGTACAACGAGAAGTTCAAGAGCAAGGCCACACTGACCGTGGACACCAGCAGCAGCACAGCCTACATGCAGCTGTCTAGCCTGACCAGCGAGGACAGCGCCGTGTACTACTGTGCTAGAGAAGGCGGCAACTACTGGTACTTCGACGTGTGGGGCACCGGCACCACAGTGACAGTTAGTTCT
[SEQ ID No:13]
因此,优选地,第一编码序列包含基本上如SEQ ID No:13所示的核苷酸序列或其片段或变体。
在一个实施方案中,第一编码序列(其可编码用于结合TCR Vbeta、优选TCR-Vbeta7.1链的VH序列)编码在本文中称为SEQ ID No:34的氨基酸序列,如下:
DIQMTQSPSSLSASLGGKVTLTCKASQDINKYIAWYQHKPGKGPRLLIHYTSTLQPGIPSRFSGSGSGRDYSFSISNLEPEDVATYYCLQYDNLRTFGGGTKLEIKRTD
[SEQ ID No:34]
因此,优选地,第一编码序列包含编码基本上如SEQ ID No:34所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,第一编码序列(其可编码用于结合TCR Vbeta、优选TCR-Vbeta7.1链的VH序列)包含在本文中称为SEQ ID No:35的核苷酸序列,如下:
GACATCCAGATGACACAGAGCCCTAGCAGCCTGTCTGCCTCTCTCGGCGGAAAAGTGACCCTGACATGCAAGGCCAGCCAGGACATCAACAAGTATATCGCCTGGTATCAGCACAAGCCCGGCAAGGGACCTAGACTGCTGATCCACTACACCAGCACACTGCAGCCTGGCATCCCCAGCAGATTTTCTGGCAGCGGCTCCGGCAGAGACTACAGCTTCAGCATCAGCAACCTGGAACCTGAGGACGTGGCCACCTACTACTGCCTGCAGTACGACAACCTGCGGACCTTTGGCGGCGGAACAAAGCTGGAAATCAAGCGGACAGAT
[SEQ ID No:35]
因此,优选地,第一编码序列包含基本上如SEQ ID No:35所示的核苷酸序列或其片段或变体。
优选地,VH(例如SEQ ID No:35)和VL(例如SEQ ID No:13)序列当处于任一方向时均被接头序列分隔开。在一个实施方案中,接头序列可以是G4S接头序列,其可包含基本上如SEQ ID No:10所示的氨基酸序列或其片段或变体或由其组成。因此,优选地,接头序列包含基本上如SEQ ID No:11所示的核苷酸序列或其片段或变体。
核酸构建体可以包含编码CD8a铰链和跨膜(TM)结构域的核苷酸序列。有利的是,铰链和TM结构域被配置用于CAR展示并锚定在CAR-T细胞上。优选地,编码铰链和TM结构域的序列位于第一编码序列的3’。
在一个实施方案中,CD8a铰链和跨膜结构域的氨基酸序列在本文中称为SEQ IDNo:14,如下:
FVPVFLPAKPTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCNHRN
[SEQ ID No:14]
因此,优选地,构建体包含编码基本上如SEQ ID No:14所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,编码CD8a铰链和跨膜结构域的核苷酸序列在本文中称为SEQID No:15,如下:
TTCGTGCCGGTCTTCCTGCCAGCGAAGCCCACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCCTGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAACCACAGGAAC
[SEQ ID No:15]
因此,优选地,构建体包含基本上如SEQ ID No:15所示的核苷酸序列或其片段或变体。
核酸构建体可包含编码胞内结构域的核苷酸序列,所述胞内结构域可包含CD28的信号传导结构域、4-1BB的信号传导结构域和/或CD3ζ链,其中更优选CD28的信号传导结构域、4-1BB的信号传导结构域和CD3ζ链。应当理解,这些组分形成了第三代CAR的基础并且是触发细胞内信号传导途径所必需的。优选地,胞内结构域位于编码铰链和跨膜结构域的序列的3'处。CD28的信号传导结构域可以是4-1BB的信号传导结构域的5'。4-1BB的信号传导结构域可能是CD3ζ链的5'。
CD28信号传导结构域的一个实施方案可以具有在本文中称为SEQ ID No:16的氨基酸序列,如下:
RSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRS
[SEQ ID No:16]
因此,优选地,构建体包含编码基本上如SEQ ID No:16所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,CD28信号传导结构域可以由在本文中称为SEQ ID No:17的核酸序列编码,如下:
AGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCC
[SEQ ID No:17]
因此,优选地,构建体包含基本上如SEQ ID No:17所示的核苷酸序列或其片段或变体。
4-1BB信号传导结构域的一个实施方案可以具有在本文中称为SEQ ID No:18的氨基酸序列,如下:
RFSVVKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL
[SEQ ID No:18]
因此,优选地,构建体包含编码基本上如SEQ ID No:18所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,4-1BB信号传导结构域可以由在本文中称为SEQ ID No:19的核酸序列编码,如下:
CGTTTCTCTGTTGTTAAACGGGGCAGAAAGAAGCTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTG
[SEQ ID No:19]
因此,优选地,构建体包含基本上如SEQ ID No:19所示的核苷酸序列或其片段或变体。CD3ζ链的一个实施方案可以具有在本文中称为SEQ ID No:20的氨基酸序列,如下:
RVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPR
[SEQ ID No:20]
因此,优选地,构建体包含编码基本上如SEQ ID No:20所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,CD3ζ链可以由在本文中称为SEQ ID No:21的核酸序列编码,如下:
AGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGC
[SEQ ID No:21]
因此,优选地,构建体包含基本上如SEQ ID No:21所示的核苷酸序列或其片段或变体。
优选地,核酸构建体包含第二编码序列,其编码至少一种自杀蛋白,更优选至少两种自杀蛋白。本发明的构建体的优点在于存在编码至少一种自杀蛋白的第二编码序列,这意味着用该构建体转导的所得CAR-T细胞可以被可控地或诱导地检测或消除,例如在患者出现不良反应的情况下。
因此,优选地,在一个实施方案中,本文提供了一种核酸构建体,其包含可操作地连接至第一编码序列和第二编码序列的启动子,其中该第一编码序列编码抗CD4嵌合抗原受体(CAR),该第二编码序列编码至少一种自杀蛋白,更优选至少两种自杀蛋白。
优选地,在另一个实施方案中,本文提供了一种核酸构建体,其包含可操作地连接至第一编码序列和第二编码序列的启动子,其中该第一编码序列编码抗T细胞受体(TCR)Vbeta CAR,该第二编码序列编码至少一种自杀蛋白,更优选至少两种自杀蛋白。
在又一个实施方案中,本文优选地提供了一种核酸构建体,其包含可操作地连接至第一编码序列和第二编码序列的启动子,其中该第一编码序列编码多个抗T细胞受体(TCR)Vbeta CAR,其中T细胞上不同的Vbeta区被靶向,该第二编码序列编码至少一种自杀蛋白,更优选至少两种自杀蛋白。
在一个实施方案中,第二编码序列可以编码表皮生长因子受体(EGFR)或截短的表皮生长因子受体(tEGFR)(UniProt No.P00533;NCBI参考序列NP_001333826.1)。tEGFR的表达可以通过抗EGFR mAb(西妥昔单抗)来控制,以监测或消除患者体内的CAR-T细胞。在一个实施方案中,tEGFR的氨基酸序列在本文中可以称为SEQ ID No:22,如下:
MLLLVTSLLLCELPHPAFLLIPRKVCNGIGIGEFKDSLSINATNIKHFKNCTSISGDLHILPVAFRGDSFTHTPPLDPQELDILKTVKEITGFLLIQAWPENRTDLHAFENLEIIRGRTKQHGQFSLAVVSLNITSLGLRSLKEISDGDVIISGNKNLCYANTINWKKLFGTSGQKTKIISNRGENSCKATGQVCHALCSPEGCWGPEPRDCVSCRNVSRGRECVDKCNLLEGEPREFVENSECIQCHPECLPQAMNITCTGRGPDNCIQCAHYIDGPHCVKTCPAGVMGENNTLVWKYADAGHVCHLCHPNCTYGCTGPGLEGCPTNGPKIPSIATGMVGALLLLLVVALGIGLFM
[SEQ ID No:22]
因此,优选地,构建体包含编码基本上如SEQ ID No:22所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,tEGFR可以由在本文中称为SEQ ID No:23的核酸序列编码,如下:
ATGCTTCTCCTGGTGACAAGCCTTCTGCTCTGTGAGTTACCACACCCAGCATTCCTCCTGATCCCACGCAAAGTGTGTAACGGAATAGGTATTGGTGAATTTAAAGACTCACTCTCCATAAATGCTACGAATATTAAACACTTCAAAAACTGCACCTCCATCAGTGGCGATCTCCACATCCTGCCGGTGGCATTTAGGGGTGACTCCTTCACACATACTCCTCCTCTGGATCCACAGGAACTGGATATTCTGAAAACCGTAAAGGAAATCACAGGGTTTTTGCTGATTCAGGCTTGGCCTGAAAACAGGACGGACCTCCATGCCTTTGAGAACCTAGAAATCATACGCGGCAGGACCAAGCAACATGGTCAGTTTTCTCTTGCAGTCGTCAGCCTGAACATAACATCCTTGGGATTACGCTCCCTCAAGGAGATAAGTGATGGAGATGTGATAATTTCAGGAAACAAAAATTTGTGCTATGCAAATACAATAAACTGGAAAAAACTGTTTGGGACCTCCGGTCAGAAAACCAAAATTATAAGCAACAGAGGTGAAAACAGCTGCAAGGCCACAGGCCAGGTCTGCCATGCCTTGTGCTCCCCCGAGGGCTGCTGGGGCCCGGAACCCAGGGACTGCGTCTCTTGCCGGAATGTCAGCCGAGGCAGGGAATGCGTGGACAAGTGCAACCTTCTGGAGGGTGAGCCAAGGGAGTTTGTGGAGAACTCTGAGTGCATACAGTGCCACCCAGAGTGCCTGCCTCAGGCCATGAACATCACCTGCACAGGACGGGGACCAGACAACTGTATCCAGTGTGCCCACTACATTGACGGCCCCCACTGCGTCAAGACCTGCCCGGCAGGAGTCATGGGAGAAAACAACACCCTGGTCTGGAAGTACGCAGACGCCGGCCATGTGTGCCACCTGTGCCATCCAAACTGCACCTACGGATGCACTGGGCCAGGTCTTGAAGGCTGTCCAACGAATGGGCCTAAGATCCCGTCCATCGCCACTGGGATGGTGGGGGCCCTCCTCTTGCTGCTGGTGGTGGCCCTGGGGATCGGCCTCTTCATG
[SEQ ID No:23]
因此,优选地,构建体包含基本上如SEQ ID No:23所示的核苷酸序列或其片段或变体。
在另一个实施方案中,第二编码序列可以编码诱导型半胱天冬酶9(iC9)。iC9是一种修饰的人半胱天冬酶9(UniProt No.P55211;NCBI参考序列NP_001220.2),与人FK506结合蛋白(UniProt No.P62942;NCBI参考序列NP_000792.1)融合,允许使用二聚化化学诱导剂进行条件二聚化(半胱天冬酶诱导药物(CID),Rimiducid),触发表达融合蛋白的CAR-T细胞凋亡。在一个实施方案中,iC9的氨基酸序列在本文中可以称为SEQ ID No:24,如下:
MLEGVQVETISPGDGRTFPKRGQTCVVHYTGMLEDGKKVDSSRDRNKPFKFMLGKQEVIRGWEEGVAQMSVGQRAKLTISPDYAYGATGHPGIIPPHATLVFDVELLKLESGGGSGVDGFGDVGALESLRGNADLAYILSMEPCGHCLIINNVNFCRESGLRTRTGSNIDCEKLRRRFSSLHFMVEVKGDLTAKKMVLALLELAQQDHGALDCCVVVILSHGCQASHLQFPGAVYGTDGCPVSVEKIVNIFNGTSCPSLGGKPKLFFIQACGGEQKDHGFEVASTSPEDESPGSNPEPDATPFQEGLRTFDQLDAISSLPTPSDIFVSYSTFPGFVSWRDPKSGSWYVETLDDIFEQWAHSEDLQSLLLRVANAVSVKGIYKQMPGCFNFLRKKLFFKTSVDYPYDVPDYALD*
[SEQ ID No:24]
因此,优选地,构建体包含编码基本上如SEQ ID No:24所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,iC9可以由在本文中称为SEQ ID No:25的核酸序列编码,如下:
ATGCTCGAGGGAGTGCAGGTGGAAACCATCTCCCCAGGAGACGGGCGCACCTTCCCCAAGCGCGGCCAGACCTGCGTGGTGCACTACACCGGGATGCTTGAAGATGGAAAGAAAGTTGATTCCTCCCGGGACAGAAACAAGCCCTTTAAGTTTATGCTAGGCAAGCAGGAGGTGATCCGAGGCTGGGAAGAAGGGGTTGCCCAGATGAGTGTGGGTCAGAGAGCCAAACTGACTATATCTCCAGATTATGCCTATGGTGCCACTGGGCACCCAGGCATCATCCCACCACATGCCACTCTCGTCTTCGATGTGGAGCTTCTAAAACTGGAATCTGGCGGTGGATCCGGAGTCGACGGATTTGGTGATGTCGGTGCTCTTGAGAGTTTGAGGGGAAATGCAGATTTGGCTTACATCCTGAGCATGGAGCCCTGTGGCCACTGCCTCATTATCAACAATGTGAACTTCTGCCGTGAGTCCGGGCTCCGCACCCGCACTGGCTCCAACATCGACTGTGAGAAGTTGCGGCGTCGCTTCTCCTCGCTGCATTTCATGGTGGAGGTGAAGGGCGACCTGACTGCCAAGAAAATGGTGCTGGCTTTGCTGGAGCTGGCGCAGCAGGACCACGGTGCTCTGGACTGCTGCGTGGTGGTCATTCTCTCTCACGGCTGTCAGGCCAGCCACCTGCAGTTCCCAGGGGCTGTCTACGGCACAGATGGATGCCCTGTGTCGGTCGAGAAGATTGTGAACATCTTCAATGGGACCAGCTGCCCCAGCCTGGGAGGGAAGCCCAAGCTCTTTTTCATCCAGGCCTGTGGTGGGGAGCAGAAAGACCATGGGTTTGAGGTGGCCTCCACTTCCCCTGAAGACGAGTCCCCTGGCAGTAACCCCGAGCCAGATGCCACCCCGTTCCAGGAAGGTTTGAGGACCTTCGACCAGCTGGACGCCATATCTAGTTTGCCCACACCCAGTGACATCTTTGTGTCCTACTCTACTTTCCCAGGTTTTGTTTCCTGGAGGGACCCCAAGAGTGGCTCCTGGTACGTTGAGACCCTGGACGACATCTTTGAGCAGTGGGCTCACTCTGAAGACCTGCAGTCCCTCCTGCTTAGGGTCGCTAATGCTGTTTCGGTGAAAGGGATTTATAAACAGATGCCTGGTTGCTTTAATTTCCTCCGGAAAAAACTTTTCTTTAAAACATCAGTCGACTATCCGTACGACGTACCAGACTACGCACTCGACTAA
[SEQ ID No:25]
因此,优选地,构建体包含基本上如SEQ ID No:25所示的核苷酸序列或其片段或变体。
优选地,本发明的核酸构建体包含编码EGFR或截短的表皮生长因子受体(tEGFR)和/或诱导型半胱天冬酶9(iC9)的编码序列。当用于治疗时,具有一个自杀基因可以对CAR和CAR-MAIT细胞的表达提供强有力的控制(检测和/或消除)。
然而,更优选地,核酸构建体包含编码截短的表皮生长因子受体(tEGFR)和诱导型半胱天冬酶9(iC9)的编码序列。有利的是,当用于治疗时,具有两个自杀基因可以对CAR和CAR-MAIT细胞的表达提供更严格的控制(检测和/或消除)。
优选地,核酸构建体包含编码肽间隔区的核苷酸序列,其中肽间隔区可被消化(或自切割)从而产生间隔区两侧作为单独分子的编码多肽,例如胞内结构域和自杀基因编码的蛋白质,可以是tEGFR和/或iC9。因此,肽间隔区可被称为自切割肽。
优选地,间隔序列包含并编码病毒肽间隔序列,更优选病毒2A肽间隔序列(FurlerS,Paterna J-C,Weibel M and Bueler H Recombinant AAV vectors containing thefoot and mouth disease virus 2A sequence confer efficient bicistronic geneexpression in cultured cells and rat substantia nigra neurons Gene Ther.2001,vol.8,PP:864-873)。
优选地,编码2A肽序列的间隔序列将编码胞内结构域的序列与编码自杀蛋白的序列连接。在构建体编码两种自杀蛋白(例如,EGFR/tEGFR和iC9)的实施方式中,核酸构建体包含布置在编码胞内结构域的序列与编码第一自杀蛋白的序列之间的第一间隔区序列,以及布置在编码第一自杀蛋白的序列和编码第二自杀蛋白的序列之间的第二间隔区序列。第一自杀蛋白可以是EGFR/tEGFR,第二自杀蛋白可以是iC9。在另一个实施方案中,第一自杀蛋白可以是iC9并且第二自杀蛋白可以是EGFR/tEGFR。
2A间隔序列可以是任何已知的变体,其包括称为E2A、F2A、P2A和T2A的那些序列,如Wang Y等公开的报道所述(Wang Y et al.Scientific Reports 2015,5)。优选地,自切割肽是P2A。在一个实施方案中,P2A间隔区具有在本文中称为SEQ ID No:26的氨基酸序列,如下:
GSGATNFSLLKQAGDVEENPGP
[SEQ ID No:26]
因此,优选地,构建体包含编码基本上如SEQ ID No:26所示的氨基酸序列的核苷酸序列或其片段或变体。
在一个实施方案中,2A间隔区可由在本文中称为SEQ ID No:27的核酸序列编码,如下:
GGATCCGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCT
因此,优选地,构建体包含基本上如SEQ ID No:27所示的核苷酸序列或其片段或变体。
在进一步的实施方案中,构建体还可包含编码土拨鼠肝炎病毒转录后调节元件(WPRE)的核苷酸序列,其增强转基因的表达。优选地,WPRE编码序列位于编码自杀蛋白的序列的3’。具体地,WPRE序列优选地是iC9编码序列的3'。
WPRE的一个实施方案是592bp长,包括γ-α-β元件,并且在本文中称为SEQ ID No:28,如下:
AATCAACCTCTGGATTACAAAATTTGTGAAAGATTGACTGGTATTCTTAACTATGTTGCTCCTTTTACGCTATGTGGATACGCTGCTTTAATGCCTTTGTATCATGCTATTGCTTCCCGTATGGCTTTCATTTTCTCCTCCTTGTATAAATCCTGGTTGCTGTCTCTTTATGAGGAGTTGTGGCCCGTTGTCAGGCAACGTGGCGTGGTGTGCACTGTGTTTGCTGACGCAACCCCCACTGGTTGGGGCATTGCCACCACCTGTCAGCTCCTTTCCGGGACTTTCGCTTTCCCCCTCCCTATTGCCACGGCGGAACTCATCGCCGCCTGCCTTGCCCGCTGCTGGACAGGGGCTCGGCTGTTGGGCACTGACAATTCCGTGGTGTTGTCGGGGAAGCTGACGTCCTTTCCATGGCTGCTCGCCTGTGTTGCCACCTGGATTCTGCGCGGGACGTCCTTCTGCTACGTCCCTTCGGCCCTCAATCCAGCGGACCTTCCTTCCCGCGGCCTGCTGCCGGCTCTGCGGCCTCTTCCGCGTCTTCGCCTTCGCCCTCAGACGAGTCGGATCTCCCTTTGGGCCGCCTCCCCGCCTG
[SEQ ID NO:28]
优选地,核酸包含基本上如SEQ ID No:28所示的核酸序列或其片段或变体。
优选地,构建体包含左(即5’)和/或右(即3’)长末端重复序列(LTR)。优选地,每个LTR位于构建体的5’和/或3’端。
在一个优选的实施方案中,核酸构建体以该特定顺序包含5’启动子;编码对CD4或TCR Vbeta区特异性的scFv的序列;和编码胞内结构域的3’序列。5’和3’的使用指示该特征是上游或下游,并且并不旨在指示该特征必然是终端特征。
在一个优选的实施方案中,核酸构建体以该特定顺序包含5’启动子;编码对CD4或TCR Vbeeta区特异性的scFv的序列;编码胞内结构域的序列;和编码至少一种自杀蛋白的3’序列。
在更优选的实施方案中,核酸构建体以该特定顺序包含5’启动子(优选PGK启动子);编码对CD4或一个或多个TCR Vbeta区(优选VL和/或VH结构域)特异性的scFv的序列;编码胞内结构域(优选CD28、4-1BB和/或CD3ζ链)的序列;编码至少一种自杀蛋白(优选EGFR/EGFRt和/或iC9)的3’序列。
在又一个更优选的实施方案中,核酸构建体以该特定顺序包含5’启动子(优选PGK启动子);编码信号肽(SP)的序列;编码对CD4或一个或多个TCR Vbeta区(优选由G4S接头分隔的VL和VH结构域)特异性的scFv的序列;编码CD8a铰链和跨膜结构域的序列;编码胞内结构域(优选CD28、4-1BB和CD3ζ链)的序列;编码至少一种自杀蛋白(优选EGFR/EGFRt和iC9)的3’序列,任选地在编码胞内结构域的序列与自杀蛋白编码序列之间具有自切割肽间隔区。
在第一最优选的实施方案(称为“CART4”)中,核酸构建体以该特定顺序包含5’PGK启动子;编码信号肽(SP)的序列;编码对CD4特异性的scFv的VL和VH结构域的序列(优选由G4S接头分隔);编码CD8a铰链和跨膜结构域的序列;编码胞内结构域的CD28、4-1BB和CD3ζ链的序列;编码第一自切割肽间隔区的序列;编码EGFR/EGFRt的序列;编码第二自切割肽间隔区的序列;编码iC9的3’序列。
在第二最优选的实施方案(称为“CARTVb7.1”)中,核酸构建体以该特定顺序包含5’PGK启动子;编码信号肽(SP)的序列;编码对一个或多个TCR Vbeta区、优选TCR-Vbeta7.1链(优选由G4S接头分隔)特异性的scFv的VL和VH结构域的序列;编码CD8a铰链和跨膜结构域的序列;编码胞内结构域的CD28、4-1BB和CD3ζ链的序列;编码第一自切割肽间隔区的序列;编码EGFR/EGFRt的序列;编码第二自切割肽间隔区的序列;编码iC9的3’序列。
因此,核酸构建体(称为“CART4”)的一个优选实施方案具有在本文中称为SEQ IDNo:29的氨基酸序列,如下:
METDTLLLWVLLLWVPGSTGDDIVMTQSPDSLAVSLGERVTMNCKSSQSLLYSTNQKNYLAWYQQKPGQSPKLLIYWASTRESGVPDRFSGSGSGTDFTLTISSVQAEDVAVYYCQQYYSYRTFGGGTKLEIKGGGGSGGGGSGGGGSQVQLQQSGPEVVKPGASVKMSCKASGYTFTSYVIHWVRQKPGQGLDWIGYINPYNDGTDYDEKFKGKATLTSDTSTSTAYMELSSLRSEDTAVYYCAREKDNYATGAWFAYWGQGTLVTVSSAAAFVPVFLPAKPTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCNHRNRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRFSVVKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRGSGATNFSLLKQAGDVEENPGPMLLLVTSLLLCELPHPAFLLIPRKVCNGIGIGEFKDSLSINATNIKHFKNCTSISGDLHILPVAFRGDSFTHTPPLDPQELDILKTVKEITGFLLIQAWPENRTDLHAFENLEIIRGRTKQHGQFSLAVVSLNITSLGLRSLKEISDGDVIISGNKNLCYANTINWKKLFGTSGQKTKIISNRGENSCKATGQVCHALCSPEGCWGPEPRDCVSCRNVSRGRECVDKCNLLEGEPREFVENSECIQCHPECLPQAMNITCTGRGPDNCIQCAHYIDGPHCVKTCPAGVMGENNTLVWKYADAGHVCHLCHPNCTYGCTGPGLEGCPTNGPKIPSIATGMVGALLLLLVVALGIGLFMGSGATNFSLLKQAGDVEENPGPMLEGVQVETISPGDGRTFPKRGQTCVVHYTGMLEDGKKVDSSRDRNKPFKFMLGKQEVIRGWEEGVAQMSVGQRAKLTISPDYAYGATGHPGIIPPHATLVFDVELLKLESGGGSGVDGFGDVGALESLRGNADLAYILSMEPCGHCLIINNVNFCRESGLRTRTGSNIDCEKLRRRFSSLHFMVEVKGDLTAKKMVLALLELAQQDHGALDCCVVVILSHGCQASHLQFPGAVYGTDGCPVSVEKIVNIFNGTSCPSLGGKPKLFFIQACGGEQKDHGFEVASTSPEDESPGSNPEPDATPFQEGLRTFDQLDAISSLPTPSDIFVSYSTFPGFVSWRDPKSGSWYVETLDDIFEQWAHSEDLQSLLLRVANAVSVKGIYKQMPGCFNFLRKKLFFKTSVDYPYDVPDYALD*
[SEQ ID No:29]
因此,优选地,构建体包含编码基本上如SEQ ID No:29所示的氨基酸序列的核苷酸序列或其片段或变体。
优选地,核酸构建体(称为“CART4”)的实施方案具有在本文中称为SEQ ID No:30核苷酸序列,如下:
ATGGAGACAGACACACTCCTGCTATGGGTGCTGCTGCTCTGGGTTCCAGGTTCCACAGGTGACGACATTGTGATGACTCAGAGCCCCGACAGCCTGGCCGTCTCACTGGGCGAAAGGGTGACCATGAATTGTAAATCTTCTCAGAGCCTGCTGTACAGTACAAACCAGAAAAATTACCTGGCCTGGTATCAGCAGAAACCCGGCCAGAGCCCTAAGCTGCTGATCTATTGGGCAAGTACCCGAGAGTCAGGAGTGCCAGACAGATTCTCCGGGTCTGGAAGTGGCACAGACTTCACCCTGACAATTAGCTCCGTGCAGGCCGAGGACGTGGCTGTCTACTATTGCCAGCAGTACTATAGCTACCGAACTTTCGGCGGGGGAACCAAACTGGAAATCAAGGGAGGAGGAGGCAGTGGCGGAGGAGGGTCAGGAGGAGGAGGAAGCCAGGTGCAGCTGCAGCAGTCCGGACCAGAGGTGGTCAAACCCGGCGCTAGCGTCAAAATGTCCTGTAAGGCATCTGGCTACACTTTCACCTCTTATGTGATTCACTGGGTCAGACAGAAGCCTGGGCAGGGACTGGACTGGATCGGGTACATTAACCCATATAATGATGGAACTGACTACGATGAAAAGTTTAAAGGCAAGGCCACACTGACTTCCGACACCTCAACAAGCACTGCTTATATGGAGCTGTCTAGTCTGAGGTCTGAAGACACAGCAGTGTACTATTGCGCCCGCGAGAAGGATAACTACGCCACTGGCGCTTGGTTTGCATATTGGGGCCAGGGGACCCTGGTGACAGTCTCATCCGCGGCCGCATTCGTGCCGGTCTTCCTGCCAGCGAAGCCCACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCCTGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAACCACAGGAACAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCCGTTTCTCTGTTGTTAAACGGGGCAGAAAGAAGCTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCGGATCCGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTTCTCCTGGTGACAAGCCTTCTGCTCTGTGAGTTACCACACCCAGCATTCCTCCTGATCCCACGCAAAGTGTGTAACGGAATAGGTATTGGTGAATTTAAAGACTCACTCTCCATAAATGCTACGAATATTAAACACTTCAAAAACTGCACCTCCATCAGTGGCGATCTCCACATCCTGCCGGTGGCATTTAGGGGTGACTCCTTCACACATACTCCTCCTCTGGATCCACAGGAACTGGATATTCTGAAAACCGTAAAGGAAATCACAGGGTTTTTGCTGATTCAGGCTTGGCCTGAAAACAGGACGGACCTCCATGCCTTTGAGAACCTAGAAATCATACGCGGCAGGACCAAGCAACATGGTCAGTTTTCTCTTGCAGTCGTCAGCCTGAACATAACATCCTTGGGATTACGCTCCCTCAAGGAGATAAGTGATGGAGATGTGATAATTTCAGGAAACAAAAATTTGTGCTATGCAAATACAATAAACTGGAAAAAACTGTTTGGGACCTCCGGTCAGAAAACCAAAATTATAAGCAACAGAGGTGAAAACAGCTGCAAGGCCACAGGCCAGGTCTGCCATGCCTTGTGCTCCCCCGAGGGCTGCTGGGGCCCGGAACCCAGGGACTGCGTCTCTTGCCGGAATGTCAGCCGAGGCAGGGAATGCGTGGACAAGTGCAACCTTCTGGAGGGTGAGCCAAGGGAGTTTGTGGAGAACTCTGAGTGCATACAGTGCCACCCAGAGTGCCTGCCTCAGGCCATGAACATCACCTGCACAGGACGGGGACCAGACAACTGTATCCAGTGTGCCCACTACATTGACGGCCCCCACTGCGTCAAGACCTGCCCGGCAGGAGTCATGGGAGAAAACAACACCCTGGTCTGGAAGTACGCAGACGCCGGCCATGTGTGCCACCTGTGCCATCCAAACTGCACCTACGGATGCACTGGGCCAGGTCTTGAAGGCTGTCCAACGAATGGGCCTAAGATCCCGTCCATCGCCACTGGGATGGTGGGGGCCCTCCTCTTGCTGCTGGTGGTGGCCCTGGGGATCGGCCTCTTCATGGGATCTGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTCGAGGGAGTGCAGGTGGAAACCATCTCCCCAGGAGACGGGCGCACCTTCCCCAAGCGCGGCCAGACCTGCGTGGTGCACTACACCGGGATGCTTGAAGATGGAAAGAAAGTTGATTCCTCCCGGGACAGAAACAAGCCCTTTAAGTTTATGCTAGGCAAGCAGGAGGTGATCCGAGGCTGGGAAGAAGGGGTTGCCCAGATGAGTGTGGGTCAGAGAGCCAAACTGACTATATCTCCAGATTATGCCTATGGTGCCACTGGGCACCCAGGCATCATCCCACCACATGCCACTCTCGTCTTCGATGTGGAGCTTCTAAAACTGGAATCTGGCGGTGGATCCGGAGTCGACGGATTTGGTGATGTCGGTGCTCTTGAGAGTTTGAGGGGAAATGCAGATTTGGCTTACATCCTGAGCATGGAGCCCTGTGGCCACTGCCTCATTATCAACAATGTGAACTTCTGCCGTGAGTCCGGGCTCCGCACCCGCACTGGCTCCAACATCGACTGTGAGAAGTTGCGGCGTCGCTTCTCCTCGCTGCATTTCATGGTGGAGGTGAAGGGCGACCTGACTGCCAAGAAAATGGTGCTGGCTTTGCTGGAGCTGGCGCAGCAGGACCACGGTGCTCTGGACTGCTGCGTGGTGGTCATTCTCTCTCACGGCTGTCAGGCCAGCCACCTGCAGTTCCCAGGGGCTGTCTACGGCACAGATGGATGCCCTGTGTCGGTCGAGAAGATTGTGAACATCTTCAATGGGACCAGCTGCCCCAGCCTGGGAGGGAAGCCCAAGCTCTTTTTCATCCAGGCCTGTGGTGGGGAGCAGAAAGACCATGGGTTTGAGGTGGCCTCCACTTCCCCTGAAGACGAGTCCCCTGGCAGTAACCCCGAGCCAGATGCCACCCCGTTCCAGGAAGGTTTGAGGACCTTCGACCAGCTGGACGCCATATCTAGTTTGCCCACACCCAGTGACATCTTTGTGTCCTACTCTACTTTCCCAGGTTTTGTTTCCTGGAGGGACCCCAAGAGTGGCTCCTGGTACGTTGAGACCCTGGACGACATCTTTGAGCAGTGGGCTCACTCTGAAGACCTGCAGTCCCTCCTGCTTAGGGTCGCTAATGCTGTTTCGGTGAAAGGGATTTATAAACAGATGCCTGGTTGCTTTAATTTCCTCCGGAAAAAACTTTTCTTTAAAACATCAGTCGACTATCCGTACGACGTACCAGACTACGCACTCGACTAA
[SEQ ID No:30]
因此,优选地,构建体包含基本上如SEQ ID No:30所示的核苷酸序列或其片段或变体。
因此,核酸构建体(称为“CARTVb7.1”)的第二优选实施方案具有在本文中称为SEQID No:31的氨基酸序列,如下:
MALPVTALLLPLALLLHAARPDIQMTQSPSSLSASLGGKVTLTCKASQDINKYIAWYQHKPGKGPRLLIHYTSTLQPGIPSRFSGSGSGRDYSFSISNLEPEDVATYYCLQYDNLRTFGGGTKLEIKRTDGGGGSGGGGSGGGGSQVQLQQPGAELVKPGASVKMSCKASGYTFTRYWITWVKQRPGQGLEWIGDIYPGSGFTKYNEKFKSKATLTVDTSSSTAYMQLSSLTSEDSAVYYCAREGGNYWYFDVWGTGTTVTVSSAAAAAFVPVFLPAKPTTTPAPRPPTPAPTIASQPLSLRPEACRPAAGGAVHTRGLDFACDIYIWAPLAGTCGVLLLSLVITLYCNHRNRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRSRFSVVKRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCELRVKFSRSADAPAYQQGQNQLYNELNLGRREEYDVLDKRRGRDPEMGGKPRRKNPQEGLYNELQKDKMAEAYSEIGMKGERRRGKGHDGLYQGLSTATKDTYDALHMQALPPRGSGATNFSLLKQAGDVEENPGPMLLLVTSLLLCELPHPAFLLIPRKVCNGIGIGEFKDSLSINATNIKHFKNCTSISGDLHILPVAFRGDSFTHTPPLDPQELDILKTVKEITGFLLIQAWPENRTDLHAFENLEIIRGRTKQHGQFSLAVVSLNITSLGLRSLKEISDGDVIISGNKNLCYANTINWKKLFGTSGQKTKIISNRGENSCKATGQVCHALCSPEGCWGPEPRDCVSCRNVSRGRECVDKCNLLEGEPREFVENSECIQCHPECLPQAMNITCTGRGPDNCIQCAHYIDGPHCVKTCPAGVMGENNTLVWKYADAGHVCHLCHPNCTYGCTGPGLEGCPTNGPKIPSIATGMVGALLLLLVVALGIGLFMGSGATNFSLLKQAGDVEENPGPMLEGVQVETISPGDGRTFPKRGQTCVVHYTGMLEDGKKVDSSRDRNKPFKFMLGKQEVIRGWEEGVAQMSVGQRAKLTISPDYAYGATGHPGIIPPHATLVFDVELLKLESGGGSGVDGFGDVGALESLRGNADLAYILSMEPCGHCLIINNVNFCRESGLRTRTGSNIDCEKLRRRFSSLHFMVEVKGDLTAKKMVLALLELAQQDHGALDCCVVVILSHGCQASHLQFPGAVYGTDGCPVSVEKIVNIFNGTSCPSLGGKPKLFFIQACGGEQKDHGFEVASTSPEDESPGSNPEPDATPFQEGLRTFDQLDAISSLPTPSDIFVSYSTFPGFVSWRDPKSGSWYVETLDDIFEQWAHSEDLQSLLLRVANAVSVKGIYKQMPGCFNFLRKKLFFKTSVDYPYDVPDYALD*
[SEQ ID No:31]
优选地,因此,构建体包含编码基本上如SEQ ID No:31所示的氨基酸序列的核苷酸序列或其片段或变体。
优选地,核酸构建体(称为“CARTVb7.1”)的实施方案具有在本文中称为SEQ IDNo:32的核苷酸序列,如下:ATGGCTCTGCCTGTTACAGCTCTGCTGCTGCCTCTGGCTCTGCTTCTGCATGCCGCCAGACCTGACATCCAGATGACACAGAGCCCTAGCAGCCTGTCTGCCTCTCTCGGCGGAAAAGTGACCCTGACATGCAAGGCCAGCCAGGACATCAACAAGTATATCGCCTGGTATCAGCACAAGCCCGGCAAGGGACCTAGACTGCTGATCCACTACACCAGCACACTGCAGCCTGGCATCCCCAGCAGATTTTCTGGCAGCGGCTCCGGCAGAGACTACAGCTTCAGCATCAGCAACCTGGAACCTGAGGACGTGGCCACCTACTACTGCCTGCAGTACGACAACCTGCGGACCTTTGGCGGCGGAACAAAGCTGGAAATCAAGCGGACAGATGGCGGAGGCGGATCAGGCGGCGGAGGAAGCGGTGGCGGAGGATCTCAAGTTCAGCTGCAACAGCCTGGCGCCGAGCTTGTGAAACCTGGCGCCTCTGTGAAGATGAGCTGCAAGGCCTCCGGCTACACCTTCACCAGATACTGGATCACCTGGGTCAAGCAGAGGCCTGGACAGGGACTCGAGTGGATCGGCGATATCTATCCTGGCTCCGGCTTCACCAAGTACAACGAGAAGTTCAAGAGCAAGGCCACACTGACCGTGGACACCAGCAGCAGCACAGCCTACATGCAGCTGTCTAGCCTGACCAGCGAGGACAGCGCCGTGTACTACTGTGCTAGAGAAGGCGGCAACTACTGGTACTTCGACGTGTGGGGCACCGGCACCACAGTGACAGTTAGTTCTGCGGCCGCGGCCGCATTCGTGCCGGTCTTCCTGCCAGCGAAGCCCACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCCTGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAACCACAGGAACAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCCGTTTCTCTGTTGTTAAACGGGGCAGAAAGAAGCTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCGGATCCGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTTCTCCTGGTGACAAGCCTTCTGCTCTGTGAGTTACCACACCCAGCATTCCTCCTGATCCCACGCAAAGTGTGTAACGGAATAGGTATTGGTGAATTTAAAGACTCACTCTCCATAAATGCTACGAATATTAAACACTTCAAAAACTGCACCTCCATCAGTGGCGATCTCCACATCCTGCCGGTGGCATTTAGGGGTGACTCCTTCACACATACTCCTCCTCTGGATCCACAGGAACTGGATATTCTGAAAACCGTAAAGGAAATCACAGGGTTTTTGCTGATTCAGGCTTGGCCTGAAAACAGGACGGACCTCCATGCCTTTGAGAACCTAGAAATCATACGCGGCAGGACCAAGCAACATGGTCAGTTTTCTCTTGCAGTCGTCAGCCTGAACATAACATCCTTGGGATTACGCTCCCTCAAGGAGATAAGTGATGGAGATGTGATAATTTCAGGAAACAAAAATTTGTGCTATGCAAATACAATAAACTGGAAAAAACTGTTTGGGACCTCCGGTCAGAAAACCAAAATTATAAGCAACAGAGGTGAAAACAGCTGCAAGGCCACAGGCCAGGTCTGCCATGCCTTGTGCTCCCCCGAGGGCTGCTGGGGCCCGGAACCCAGGGACTGCGTCTCTTGCCGGAATGTCAGCCGAGGCAGGGAATGCGTGGACAAGTGCAACCTTCTGGAGGGTGAGCCAAGGGAGTTTGTGGAGAACTCTGAGTGCATACAGTGCCACCCAGAGTGCCTGCCTCAGGCCATGAACATCACCTGCACAGGACGGGGACCAGACAACTGTATCCAGTGTGCCCACTACATTGACGGCCCCCACTGCGTCAAGACCTGCCCGGCAGGAGTCATGGGAGAAAACAACACCCTGGTCTGGAAGTACGCAGACGCCGGCCATGTGTGCCACCTGTGCCATCCAAACTGCACCTACGGATGCACTGGGCCAGGTCTTGAAGGCTGTCCAACGAATGGGCCTAAGATCCCGTCCATCGCCACTGGGATGGTGGGGGCCCTCCTCTTGCTGCTGGTGGTGGCCCTGGGGATCGGCCTCTTCATGGGATCTGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTCGAGGGAGTGCAGGTGGAAACCATCTCCCCAGGAGACGGGCGCACCTTCCCCAAGCGCGGCCAGACCTGCGTGGTGCACTACACCGGGATGCTTGAAGATGGAAAGAAAGTTGATTCCTCCCGGGACAGAAACAAGCCCTTTAAGTTTATGCTAGGCAAGCAGGAGGTGATCCGAGGCTGGGAAGAAGGGGTTGCCCAGATGAGTGTGGGTCAGAGAGCCAAACTGACTATATCTCCAGATTATGCCTATGGTGCCACTGGGCACCCAGGCATCATCCCACCACATGCCACTCTCGTCTTCGATGTGGAGCTTCTAAAACTGGAATCTGGCGGTGGATCCGGAGTCGACGGATTTGGTGATGTCGGTGCTCTTGAGAGTTTGAGGGGAAATGCAGATTTGGCTTACATCCTGAGCATGGAGCCCTGTGGCCACTGCCTCATTATCAACAATGTGAACTTCTGCCGTGAGTCCGGGCTCCGCACCCGCACTGGCTCCAACATCGACTGTGAGAAGTTGCGGCGTCGCTTCTCCTCGCTGCATTTCATGGTGGAGGTGAAGGGCGACCTGACTGCCAAGAAAATGGTGCTGGCTTTGCTGGAGCTGGCGCAGCAGGACCACGGTGCTCTGGACTGCTGCGTGGTGGTCATTCTCTCTCACGGCTGTCAGGCCAGCCACCTGCAGTTCCCAGGGGCTGTCTACGGCACAGATGGATGCCCTGTGTCGGTCGAGAAGATTGTGAACATCTTCAATGGGACCAGCTGCCCCAGCCTGGGAGGGAAGCCCAAGCTCTTTTTCATCCAGGCCTGTGGTGGGGAGCAGAAAGACCATGGGTTTGAGGTGGCCTCCACTTCCCCTGAAGACGAGTCCCCTGGCAGTAACCCCGAGCCAGATGCCACCCCGTTCCAGGAAGGTTTGAGGACCTTCGACCAGCTGGACGCCATATCTAGTTTGCCCACACCCAGTGACATCTTTGTGTCCTACTCTACTTTCCCAGGTTTTGTTTCCTGGAGGGACCCCAAGAGTGGCTCCTGGTACGTTGAGACCCTGGACGACATCTTTGAGCAGTGGGCTCACTCTGAAGACCTGCAGTCCCTCCTGCTTAGGGTCGCTAATGCTGTTTCGGTGAAAGGGATTTATAAACAGATGCCTGGTTGCTTTAATTTCCTCCGGAAAAAACTTTTCTTTAAAACATCAGTCGACTATCCGTACGACGTACCAGACTACGCACTCGACTAA
[SEQ ID No:32]
因此,优选地,构建体包含基本上如SEQ ID No:32所示的核苷酸序列或其片段或变体。
因此,应当理解,使用第二方面或第三方面的方法获得的分离的MAIT细胞可以被激活,并且最终用根据第五方面的编码CAR的核酸构建体转导,从而产生第一方面或第四方面的CAR-MAIT细胞。
在第六方面,本文提供了编码第五方面的核酸构建体的表达载体。
优选地,载体是重组的。优选地,载体是病毒载体,更优选逆转录病毒载体。图9和图10示出本发明载体的两个优选实施方案的特征的图谱。
在一个实施方案中(CART4:CAR4-tEGFR-iC9;参见图9),载体具有在本文中称为SEQ ID No:33的核酸序列,如下:
TGAAAGACCCCACCTGTAGGTTTGGCAAGCTAGCTTAAGTAACGCCATTTTGCAAGGCATGGAAAATACATAACTGAGAATAGAGAAGTTCAGATCAAGGTTAGGAACAGAGAGACAGCAGAATATGGGCCAAACAGGATATCTGTGGTAAGCAGTTCCTGCCCCGGCTCAGGGCCAAGAACAGATGGTCCCCAGATGCGGTCCCGCCCTCAGCAGTTTCTAGAGAACCATCAGATGTTTCCAGGGTGCCCCAAGGACCTGAAATGACCCTGTGCCTTATTTGAACTAACCAATCAGTTCGCTTCTCGCTTCTGTTCGCGCGCTTCTGCTCCCCGAGCTCAATAAAAGAGCCCACAACCCCTCACTCGGCGCGCCAGTCCTCCGATAGACTGCGTCGCCCGGGTACCCGTATTCCCAATAAAGCCTCTTGCTGTTTGCATCCGAATCGTGGACTCGCTGATCCTTGGGAGGGTCTCCTCAGATTGATTGACTGCCCACCTCGGGGGTCTTTCATTTGGAGGTTCCACCGAGATTTGGAGACCCCTGCCCAGGGACCACCGACCCCCCCGCCGGGAGGTAAGCTGGCCAGCGGTCGTTTCGTGTCTGTCTCTGTCTTTGTGCGTGTTTGTGCCGGCATCTAATGTTTGCGCCTGCGTCTGTACTAGTTAGCTAACTAGCTCTGTATCTGGCGGACCCGTGGTGGAACTGACGAGTTCTGAACACCCGGCCGCAACCCTGGGAGACGTCCCAGGGACTTTGGGGGCCGTTTTTGTGGCCCGACCTGAGGAAGGGAGTCGATGTGGAATCCGACCCCGTCAGGATATGTGGTTCTGGTAGGAGACGAGAACCTAAAACAGTTCCCGCCTCCGTCTGAATTTTTGCTTTCGGTTTGGAACCGAAGCCGCGCGTCTTGTCTGCTGCAGCGCTGCAGCATCGTTCTGTGTTGTCTCTGTCTGACTGTGTTTCTGTATTTGTCTGAAAATTAGGGCCAGACTGTTACCACTCCCTTAAGTTTGACCTTAGGTCACTGGAAAGATGTCGAGCGGATCGCTCACAACCAGTCGGTAGATGTCAAGAAGAGACGTTGGGTTACCTTCTGCTCTGCAGAATGGCCAACCTTTAACGTCGGATGGCCGCGAGACGGCACCTTTAACCGAGACCTCATCACCCAGGTTAAGATCAAGGTCTTTTCACCTGGCCCGCATGGACACCCAGACCAGGTCCCCTACATCGTGACCTGGGAAGCCTTGGCTTTTGACCCCCCTCCCTGGGTCAAGCCCTTTGTACACCCTAAGCCTCCGCCTCCTCTTCCTCCATCCGCCCCGTCTCTCCCCCTTGAACCTCCTCGTTCGACCCCGCCTCGATCCTCCCTTTATCCAGCCCTCACTCCTTCTCTAGGCGCCGGAATTAGATCTCTCGAGGTTAACGAATTCTACCGGGTAGGGGAGGCGCTTTTCCCAAGGCAGTCTGGAGCATGCGCTTTAGCAGCCCCGCTGGGCACTTGGCGCTACACAAGTGGCCTCTGGCCTCGCACACATTCCACATCCACCGGTAGGCGCCAACCGGCTCCGTTCTTTGGTGGCCCCTTCGCGCCACCTTCTACTCCTCCCCTAGTCAGGAAGTTCCCCCCCGCCCCGCAGCTCGCGTCGTGCAGGACGTGACAAATGGAAGTAGCACGTCTCACTAGTCTCGTGCAGATGGACAGCACCGCTGAGCAATGGAAGCGGGTAGGCCTTTGGGGCAGCGGCCAATAGCAGCTTTGCTCCTTCGCTTTCTGGGCTCAGAGGCTGGGAAGGGGTGGGTCCGGGGGCGGGCTCAGGGGCGGGCTCAGGGGCGGGGCGGGCGCCCGAAGGTCCTCCGGAGGCCCGGCATTCTGCACGCTTCAAAAGCGCACGTCTGCCGCGCTGTTCTCCTCTTCCTCATTCTCCGGGCCTTTCGACCTGCAGCCCAAGCCACCATGGAGACAGACACACTCCTGCTATGGGTGCTGCTGCTCTGGGTTCCAGGTTCCACAGGTGACGACATTGTGATGACTCAGAGCCCCGACAGCCTGGCCGTCTCACTGGGCGAAAGGGTGACCATGAATTGTAAATCTTCTCAGAGCCTGCTGTACAGTACAAACCAGAAAAATTACCTGGCCTGGTATCAGCAGAAACCCGGCCAGAGCCCTAAGCTGCTGATCTATTGGGCAAGTACCCGAGAGTCAGGAGTGCCAGACAGATTCTCCGGGTCTGGAAGTGGCACAGACTTCACCCTGACAATTAGCTCCGTGCAGGCCGAGGACGTGGCTGTCTACTATTGCCAGCAGTACTATAGCTACCGAACTTTCGGCGGGGGAACCAAACTGGAAATCAAGGGAGGAGGAGGCAGTGGCGGAGGAGGGTCAGGAGGAGGAGGAAGCCAGGTGCAGCTGCAGCAGTCCGGACCAGAGGTGGTCAAACCCGGCGCTAGCGTCAAAATGTCCTGTAAGGCATCTGGCTACACTTTCACCTCTTATGTGATTCACTGGGTCAGACAGAAGCCTGGGCAGGGACTGGACTGGATCGGGTACATTAACCCATATAATGATGGAACTGACTACGATGAAAAGTTTAAAGGCAAGGCCACACTGACTTCCGACACCTCAACAAGCACTGCTTATATGGAGCTGTCTAGTCTGAGGTCTGAAGACACAGCAGTGTACTATTGCGCCCGCGAGAAGGATAACTACGCCACTGGCGCTTGGTTTGCATATTGGGGCCAGGGGACCCTGGTGACAGTCTCATCCGCGGCCGCATTCGTGCCGGTCTTCCTGCCAGCGAAGCCCACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCCTGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAACCACAGGAACAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCCGTTTCTCTGTTGTTAAACGGGGCAGAAAGAAGCTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCGGATCCGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTTCTCCTGGTGACAAGCCTTCTGCTCTGTGAGTTACCACACCCAGCATTCCTCCTGATCCCACGCAAAGTGTGTAACGGAATAGGTATTGGTGAATTTAAAGACTCACTCTCCATAAATGCTACGAATATTAAACACTTCAAAAACTGCACCTCCATCAGTGGCGATCTCCACATCCTGCCGGTGGCATTTAGGGGTGACTCCTTCACACATACTCCTCCTCTGGATCCACAGGAACTGGATATTCTGAAAACCGTAAAGGAAATCACAGGGTTTTTGCTGATTCAGGCTTGGCCTGAAAACAGGACGGACCTCCATGCCTTTGAGAACCTAGAAATCATACGCGGCAGGACCAAGCAACATGGTCAGTTTTCTCTTGCAGTCGTCAGCCTGAACATAACATCCTTGGGATTACGCTCCCTCAAGGAGATAAGTGATGGAGATGTGATAATTTCAGGAAACAAAAATTTGTGCTATGCAAATACAATAAACTGGAAAAAACTGTTTGGGACCTCCGGTCAGAAAACCAAAATTATAAGCAACAGAGGTGAAAACAGCTGCAAGGCCACAGGCCAGGTCTGCCATGCCTTGTGCTCCCCCGAGGGCTGCTGGGGCCCGGAACCCAGGGACTGCGTCTCTTGCCGGAATGTCAGCCGAGGCAGGGAATGCGTGGACAAGTGCAACCTTCTGGAGGGTGAGCCAAGGGAGTTTGTGGAGAACTCTGAGTGCATACAGTGCCACCCAGAGTGCCTGCCTCAGGCCATGAACATCACCTGCACAGGACGGGGACCAGACAACTGTATCCAGTGTGCCCACTACATTGACGGCCCCCACTGCGTCAAGACCTGCCCGGCAGGAGTCATGGGAGAAAACAACACCCTGGTCTGGAAGTACGCAGACGCCGGCCATGTGTGCCACCTGTGCCATCCAAACTGCACCTACGGATGCACTGGGCCAGGTCTTGAAGGCTGTCCAACGAATGGGCCTAAGATCCCGTCCATCGCCACTGGGATGGTGGGGGCCCTCCTCTTGCTGCTGGTGGTGGCCCTGGGGATCGGCCTCTTCATGGGATCTGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTCGAGGGAGTGCAGGTGGAAACCATCTCCCCAGGAGACGGGCGCACCTTCCCCAAGCGCGGCCAGACCTGCGTGGTGCACTACACCGGGATGCTTGAAGATGGAAAGAAAGTTGATTCCTCCCGGGACAGAAACAAGCCCTTTAAGTTTATGCTAGGCAAGCAGGAGGTGATCCGAGGCTGGGAAGAAGGGGTTGCCCAGATGAGTGTGGGTCAGAGAGCCAAACTGACTATATCTCCAGATTATGCCTATGGTGCCACTGGGCACCCAGGCATCATCCCACCACATGCCACTCTCGTCTTCGATGTGGAGCTTCTAAAACTGGAATCTGGCGGTGGATCCGGAGTCGACGGATTTGGTGATGTCGGTGCTCTTGAGAGTTTGAGGGGAAATGCAGATTTGGCTTACATCCTGAGCATGGAGCCCTGTGGCCACTGCCTCATTATCAACAATGTGAACTTCTGCCGTGAGTCCGGGCTCCGCACCCGCACTGGCTCCAACATCGACTGTGAGAAGTTGCGGCGTCGCTTCTCCTCGCTGCATTTCATGGTGGAGGTGAAGGGCGACCTGACTGCCAAGAAAATGGTGCTGGCTTTGCTGGAGCTGGCGCAGCAGGACCACGGTGCTCTGGACTGCTGCGTGGTGGTCATTCTCTCTCACGGCTGTCAGGCCAGCCACCTGCAGTTCCCAGGGGCTGTCTACGGCACAGATGGATGCCCTGTGTCGGTCGAGAAGATTGTGAACATCTTCAATGGGACCAGCTGCCCCAGCCTGGGAGGGAAGCCCAAGCTCTTTTTCATCCAGGCCTGTGGTGGGGAGCAGAAAGACCATGGGTTTGAGGTGGCCTCCACTTCCCCTGAAGACGAGTCCCCTGGCAGTAACCCCGAGCCAGATGCCACCCCGTTCCAGGAAGGTTTGAGGACCTTCGACCAGCTGGACGCCATATCTAGTTTGCCCACACCCAGTGACATCTTTGTGTCCTACTCTACTTTCCCAGGTTTTGTTTCCTGGAGGGACCCCAAGAGTGGCTCCTGGTACGTTGAGACCCTGGACGACATCTTTGAGCAGTGGGCTCACTCTGAAGACCTGCAGTCCCTCCTGCTTAGGGTCGCTAATGCTGTTTCGGTGAAAGGGATTTATAAACAGATGCCTGGTTGCTTTAATTTCCTCCGGAAAAAACTTTTCTTTAAAACATCAGTCGACTATCCGTACGACGTACCAGACTACGCACTCGACTAAACAATCAACCTCTGGATTACAAAATTTGTGAAAGATTGACTGGTATTCTTAACTATGTTGCTCCTTTTACGCTATGTGGATACGCTGCTTTAATGCCTTTGTATCATGCTATTGCTTCCCGTATGGCTTTCATTTTCTCCTCCTTGTATAAATCCTGGTTGCTGTCTCTTTATGAGGAGTTGTGGCCCGTTGTCAGGCAACGTGGCGTGGTGTGCACTGTGTTTGCTGACGCAACCCCCACTGGTTGGGGCATTGCCACCACCTGTCAGCTCCTTTCCGGGACTTTCGCTTTCCCCCTCCCTATTGCCACGGCGGAACTCATCGCCGCCTGCCTTGCCCGCTGCTGGACAGGGGCTCGGCTGTTGGGCACTGACAATTCCGTGGTGTTGTCGGGGAAATCATCGTCCTTTCCTTGGCTGCTCGCCTGTGTTGCCACCTGGATTCTGCGCGGGACGTCCTTCTGCTACGTCCCTTCGGCCCTCAATCCAGCGGACCTTCCTTCCCGCGGCCTGCTGCCGGCTCTGCGGCCTCTTCCGCGTCTTCGCCTTCGCCCTCAGACGAGTCGGATCTCCCTTTGGGCCGCCTCCCCGCCTATCGATAAAATAAAAGATTTTATTTAGTCTCCAGAAAAAGGGGGGAATGAAAGACCCCACCTGTAGGTTTGGCAAGCTAGCTTAAGTAACGCCATTTTGCAAGGCATGGAAAATACATAACTGAGAATAGAGAAGTTCAGATCAAGGTTAGGAACAGAGAGACAGCAGAATATGGGCCAAACAGGATATCTGTGGTAAGCAGTTCCTGCCCCGGCTCAGGGCCAAGAACAGATGGTCCCCAGATGCGGTCCCGCCCTCAGCAGTTTCTAGAGAACCATCAGATGTTTCCAGGGTGCCCCAAGGACCTGAAATGACCCTGTGCCTTATTTGAACTAACCAATCAGTTCGCTTCTCGCTTCTGTTCGCGCGCTTCTGCTCCCCGAGCTCAATAAAAGAGCCCACAACCCCTCACTCGGCGCGCCAGTCCTCCGATAGACTGCGTCGCCCGGGTACCCGTGTATCCAATAAACCCTCTTGCAGTTGCATCCGACTTGTGGTCTCGCTGTTCCTTGGGAGGGTCTCCTCTGAGTGATTGACTACCCGTCAGCGGGGGTCTTTCATGGGTAACAGTTTCTTGAAGTTGGAGAACAACATTCTGAGGGTAGGAGTCGAATATTAAGTAATCCTGACTCAATTAGCCACTGTTTTGAATCCACATACTCCAATACTCCTGAAATAGTTCATTATGGACAGCGCAGAAGAGCTGGGGAGAATTAATTCGTAATCATGGTCATAGCTGTTTCCTGTGTGAAATTGTTATCCGCTCACAATTCCACACAACATACGAGCCGGAAGCATAAAGTGTAAAGCCTGGGGTGCCTAATGAGTGAGCTAACTCACATTAATTGCGTTGCGCTCACTGCCCGCTTTCCAGTCGGGAAACCTGTCGTGCCAGCTGCATTAATGAATCGGCCAACGCGCGGGGAGAGGCGGTTTGCGTATTGGGCGCTCTTCCGCTTCCTCGCTCACTGACTCGCTGCGCTCGGTCGTTCGGCTGCGGCGAGCGGTATCAGCTCACTCAAAGGCGGTAATACGGTTATCCACAGAATCAGGGGATAACGCAGGAAAGAACATGTGAGCAAAAGGCCAGCAAAAGGCCAGGAACCGTAAAAAGGCCGCGTTGCTGGCGTTTTTCCATAGGCTCCGCCCCCCTGACGAGCATCACAAAAATCGACGCTCAAGTCAGAGGTGGCGAAACCCGACAGGACTATAAAGATACCAGGCGTTTCCCCCTGGAAGCTCCCTCGTGCGCTCTCCTGTTCCGACCCTGCCGCTTACCGGATACCTGTCCGCCTTTCTCCCTTCGGGAAGCGTGGCGCTTTCTCATAGCTCACGCTGTAGGTATCTCAGTTCGGTGTAGGTCGTTCGCTCCAAGCTGGGCTGTGTGCACGAACCCCCCGTTCAGCCCGACCGCTGCGCCTTATCCGGTAACTATCGTCTTGAGTCCAACCCGGTAAGACACGACTTATCGCCACTGGCAGCAGCCACTGGTAACAGGATTAGCAGAGCGAGGTATGTAGGCGGTGCTACAGAGTTCTTGAAGTGGTGGCCTAACTACGGCTACACTAGAAGGACAGTATTTGGTATCTGCGCTCTGCTGAAGCCAGTTACCTTCGGAAAAAGAGTTGGTAGCTCTTGATCCGGCAAACAAACCACCGCTGGTAGCGGTGGTTTTTTTGTTTGCAAGCAGCAGATTACGCGCAGAAAAAAAGGATCTCAAGAAGATCCTTTGATCTTTTCTACGGGGTCTGACGCTCAGTGGAACGAAAACTCACGTTAAGGGATTTTGGTCATGAGATTATCAAAAAGGATCTTCACCTAGATCCTTTTAAATTAAAAATGAAGTTTTAAATCAATCTAAAGTATATATGAGTAAACTTGGTCTGACAGTTACCAATGCTTAATCAGTGAGGCACCTATCTCAGCGATCTGTCTATTTCGTTCATCCATAGTTGCCTGACTCCCCGTCGTGTAGATAACTACGATACGGGAGGGCTTACCATCTGGCCCCAGTGCTGCAATGATACCGCGAGACCCACGCTCACCGGCTCCAGATTTATCAGCAATAAACCAGCCAGCCGGAAGGGCCGAGCGCAGAAGTGGTCCTGCAACTTTATCCGCCTCCATCCAGTCTATTAATTGTTGCCGGGAAGCTAGAGTAAGTAGTTCGCCAGTTAATAGTTTGCGCAACGTTGTTGCCATTGCTACAGGCATCGTGGTGTCACGCTCGTCGTTTGGTATGGCTTCATTCAGCTCCGGTTCCCAACGATCAAGGCGAGTTACATGATCCCCCATGTTGTGCAAAAAAGCGGTTAGCTCCTTCGGTCCTCCGATCGTTGTCAGAAGTAAGTTGGCCGCAGTGTTATCACTCATGGTTATGGCAGCACTGCATAATTCTCTTACTGTCATGCCATCCGTAAGATGCTTTTCTGTGACTGGTGAGTACTCAACCAAGTCATTCTGAGAATAGTGTATGCGGCGACCGAGTTGCTCTTGCCCGGCGTCAATACGGGATAATACCGCGCCACATAGCAGAACTTTAAAAGTGCTCATCATTGGAAAACGTTCTTCGGGGCGAAAACTCTCAAGGATCTTACCGCTGTTGAGATCCAGTTCGATGTAACCCACTCGTGCACCCAACTGATCTTCAGCATCTTTTACTTTCACCAGCGTTTCTGGGTGAGCAAAAACAGGAAGGCAAAATGCCGCAAAAAAGGGAATAAGGGCGACACGGAAATGTTGAATACTCATACTCTTCCTTTTTCAATATTATTGAAGCATTTATCAGGGTTATTGTCTCATGAGCGGATACATATTTGAATGTATTTAGAAAAATAAACAAATAGGGGTTCCGCGCACATTTCCCCGAAAAGTGCCACCTGACGTCTAAGAAACCATTATTATCATGACATTAACCTATAAAAATAGGCGTATCACGAGGCCCTTTCGTCTCGCGCGTTTCGGTGATGACGGTGAAAACCTCTGACACATGCAGCTCCCGGAGACGGTCACAGCTTGTCTGTAAGCGGATGCCGGGAGCAGACAAGCCCGTCAGGGCGCGTCAGCGGGTGTTGGCGGGTGTCGGGGCTGGCTTAACTATGCGGCATCAGAGCAGATTGTACTGAGAGTGCACCATATGCGGTGTGAAATACCGCACAGATGCGTAAGGAGAAAATACCGCATCAGGCGCCATTCGCCATTCAGGCTGCGCAACTGTTGGGAAGGGCGATCGGTGCGGGCCTCTTCGCTATTACGCCAGCTGGCGAAAGGGGGATGTGCTGCAAGGCGATTAAGTTGGGTAACGCCAGGGTTTTCCCAGTCACGACGTTGTAAAACGACGGCGCAAGGAATGGTGCATGCAAGGAGATGGCGCCCAACAGTCCCCCGGCCACGGGGCCTGCCACCATACCCACGCCGAAACAAGCGCTCATGAGCCCGAAGTGGCGAGCCCGATCTTCCCCATCGGTGATGTCGGCGATATAGGCGCCAGCAACCGCACCTGTGGCGCCGGTGATGCCGGCCACGATGCGTCCGGCGTAGAGGCGATTAGTCCAATTTGTTAAAGACAGGATATCAGTGGTCCAGGCTCTAGTTTTGACTCAACAATATCACCAGCTGAAGCCTATAGAGTACGAGCCATAGATAAAATAAAAGATTTTATTTAGTCTCCAGAAAAAGGGGGGAA
[SEQ ID NO:33]
优选地,载体包含基本上如SEQ ID NO:33所示的核酸序列或其片段或变体。
在一个实施方案中(CARTVb7.1:CARTVb7.1-tEGFR-iC9;参见图10),载体具有在本文中称为SEQ ID No:36的核酸序列,如下:
TGAAAGACCCCACCTGTAGGTTTGGCAAGCTAGCTTAAGTAACGCCATTTTGCAAGGCATGGAAAATACATAACTGAGAATAGAGAAGTTCAGATCAAGGTTAGGAACAGAGAGACAGCAGAATATGGGCCAAACAGGATATCTGTGGTAAGCAGTTCCTGCCCCGGCTCAGGGCCAAGAACAGATGGTCCCCAGATGCGGTCCCGCCCTCAGCAGTTTCTAGAGAACCATCAGATGTTTCCAGGGTGCCCCAAGGACCTGAAATGACCCTGTGCCTTATTTGAACTAACCAATCAGTTCGCTTCTCGCTTCTGTTCGCGCGCTTCTGCTCCCCGAGCTCAATAAAAGAGCCCACAACCCCTCACTCGGCGCGCCAGTCCTCCGATAGACTGCGTCGCCCGGGTACCCGTATTCCCAATAAAGCCTCTTGCTGTTTGCATCCGAATCGTGGACTCGCTGATCCTTGGGAGGGTCTCCTCAGATTGATTGACTGCCCACCTCGGGGGTCTTTCATTTGGAGGTTCCACCGAGATTTGGAGACCCCTGCCCAGGGACCACCGACCCCCCCGCCGGGAGGTAAGCTGGCCAGCGGTCGTTTCGTGTCTGTCTCTGTCTTTGTGCGTGTTTGTGCCGGCATCTAATGTTTGCGCCTGCGTCTGTACTAGTTAGCTAACTAGCTCTGTATCTGGCGGACCCGTGGTGGAACTGACGAGTTCTGAACACCCGGCCGCAACCCTGGGAGACGTCCCAGGGACTTTGGGGGCCGTTTTTGTGGCCCGACCTGAGGAAGGGAGTCGATGTGGAATCCGACCCCGTCAGGATATGTGGTTCTGGTAGGAGACGAGAACCTAAAACAGTTCCCGCCTCCGTCTGAATTTTTGCTTTCGGTTTGGAACCGAAGCCGCGCGTCTTGTCTGCTGCAGCGCTGCAGCATCGTTCTGTGTTGTCTCTGTCTGACTGTGTTTCTGTATTTGTCTGAAAATTAGGGCCAGACTGTTACCACTCCCTTAAGTTTGACCTTAGGTCACTGGAAAGATGTCGAGCGGATCGCTCACAACCAGTCGGTAGATGTCAAGAAGAGACGTTGGGTTACCTTCTGCTCTGCAGAATGGCCAACCTTTAACGTCGGATGGCCGCGAGACGGCACCTTTAACCGAGACCTCATCACCCAGGTTAAGATCAAGGTCTTTTCACCTGGCCCGCATGGACACCCAGACCAGGTCCCCTACATCGTGACCTGGGAAGCCTTGGCTTTTGACCCCCCTCCCTGGGTCAAGCCCTTTGTACACCCTAAGCCTCCGCCTCCTCTTCCTCCATCCGCCCCGTCTCTCCCCCTTGAACCTCCTCGTTCGACCCCGCCTCGATCCTCCCTTTATCCAGCCCTCACTCCTTCTCTAGGCGCCGGAATTAGATCTCTCGAGGTTAACGAATTCTACCGGGTAGGGGAGGCGCTTTTCCCAAGGCAGTCTGGAGCATGCGCTTTAGCAGCCCCGCTGGGCACTTGGCGCTACACAAGTGGCCTCTGGCCTCGCACACATTCCACATCCACCGGTAGGCGCCAACCGGCTCCGTTCTTTGGTGGCCCCTTCGCGCCACCTTCTACTCCTCCCCTAGTCAGGAAGTTCCCCCCCGCCCCGCAGCTCGCGTCGTGCAGGACGTGACAAATGGAAGTAGCACGTCTCACTAGTCTCGTGCAGATGGACAGCACCGCTGAGCAATGGAAGCGGGTAGGCCTTTGGGGCAGCGGCCAATAGCAGCTTTGCTCCTTCGCTTTCTGGGCTCAGAGGCTGGGAAGGGGTGGGTCCGGGGGCGGGCTCAGGGGCGGGCTCAGGGGCGGGGCGGGCGCCCGAAGGTCCTCCGGAGGCCCGGCATTCTGCACGCTTCAAAAGCGCACGTCTGCCGCGCTGTTCTCCTCTTCCTCATTCTCCGGGCCTTTCGACCTGCAGCCCAAGCCACCATGGCTCTGCCTGTTACAGCTCTGCTGCTGCCTCTGGCTCTGCTTCTGCATGCCGCCAGACCTGACATCCAGATGACACAGAGCCCTAGCAGCCTGTCTGCCTCTCTCGGCGGAAAAGTGACCCTGACATGCAAGGCCAGCCAGGACATCAACAAGTATATCGCCTGGTATCAGCACAAGCCCGGCAAGGGACCTAGACTGCTGATCCACTACACCAGCACACTGCAGCCTGGCATCCCCAGCAGATTTTCTGGCAGCGGCTCCGGCAGAGACTACAGCTTCAGCATCAGCAACCTGGAACCTGAGGACGTGGCCACCTACTACTGCCTGCAGTACGACAACCTGCGGACCTTTGGCGGCGGAACAAAGCTGGAAATCAAGCGGACAGATGGCGGAGGCGGATCAGGCGGCGGAGGAAGCGGTGGCGGAGGATCTCAAGTTCAGCTGCAACAGCCTGGCGCCGAGCTTGTGAAACCTGGCGCCTCTGTGAAGATGAGCTGCAAGGCCTCCGGCTACACCTTCACCAGATACTGGATCACCTGGGTCAAGCAGAGGCCTGGACAGGGACTCGAGTGGATCGGCGATATCTATCCTGGCTCCGGCTTCACCAAGTACAACGAGAAGTTCAAGAGCAAGGCCACACTGACCGTGGACACCAGCAGCAGCACAGCCTACATGCAGCTGTCTAGCCTGACCAGCGAGGACAGCGCCGTGTACTACTGTGCTAGAGAAGGCGGCAACTACTGGTACTTCGACGTGTGGGGCACCGGCACCACAGTGACAGTTAGTTCTGCGGCCGCGGCCGCATTCGTGCCGGTCTTCCTGCCAGCGAAGCCCACCACGACGCCAGCGCCGCGACCACCAACACCGGCGCCCACCATCGCGTCGCAGCCCCTGTCCCTGCGCCCAGAGGCGTGCCGGCCAGCGGCGGGGGGCGCAGTGCACACGAGGGGGCTGGACTTCGCCTGTGATATCTACATCTGGGCGCCCTTGGCCGGGACTTGTGGGGTCCTTCTCCTGTCACTGGTTATCACCCTTTACTGCAACCACAGGAACAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCCGTTTCTCTGTTGTTAAACGGGGCAGAAAGAAGCTCCTGTATATATTCAAACAACCATTTATGAGACCAGTACAAACTACTCAAGAGGAAGATGGCTGTAGCTGCCGATTTCCAGAAGAAGAAGAAGGAGGATGTGAACTGAGAGTGAAGTTCAGCAGGAGCGCAGACGCCCCCGCGTACCAGCAGGGCCAGAACCAGCTCTATAACGAGCTCAATCTAGGACGAAGAGAGGAGTACGATGTTTTGGACAAGAGACGTGGCCGGGACCCTGAGATGGGGGGAAAGCCGAGAAGGAAGAACCCTCAGGAAGGCCTGTACAATGAACTGCAGAAAGATAAGATGGCGGAGGCCTACAGTGAGATTGGGATGAAAGGCGAGCGCCGGAGGGGCAAGGGGCACGATGGCCTTTACCAGGGTCTCAGTACAGCCACCAAGGACACCTACGACGCCCTTCACATGCAGGCCCTGCCCCCTCGCGGATCCGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTTCTCCTGGTGACAAGCCTTCTGCTCTGTGAGTTACCACACCCAGCATTCCTCCTGATCCCACGCAAAGTGTGTAACGGAATAGGTATTGGTGAATTTAAAGACTCACTCTCCATAAATGCTACGAATATTAAACACTTCAAAAACTGCACCTCCATCAGTGGCGATCTCCACATCCTGCCGGTGGCATTTAGGGGTGACTCCTTCACACATACTCCTCCTCTGGATCCACAGGAACTGGATATTCTGAAAACCGTAAAGGAAATCACAGGGTTTTTGCTGATTCAGGCTTGGCCTGAAAACAGGACGGACCTCCATGCCTTTGAGAACCTAGAAATCATACGCGGCAGGACCAAGCAACATGGTCAGTTTTCTCTTGCAGTCGTCAGCCTGAACATAACATCCTTGGGATTACGCTCCCTCAAGGAGATAAGTGATGGAGATGTGATAATTTCAGGAAACAAAAATTTGTGCTATGCAAATACAATAAACTGGAAAAAACTGTTTGGGACCTCCGGTCAGAAAACCAAAATTATAAGCAACAGAGGTGAAAACAGCTGCAAGGCCACAGGCCAGGTCTGCCATGCCTTGTGCTCCCCCGAGGGCTGCTGGGGCCCGGAACCCAGGGACTGCGTCTCTTGCCGGAATGTCAGCCGAGGCAGGGAATGCGTGGACAAGTGCAACCTTCTGGAGGGTGAGCCAAGGGAGTTTGTGGAGAACTCTGAGTGCATACAGTGCCACCCAGAGTGCCTGCCTCAGGCCATGAACATCACCTGCACAGGACGGGGACCAGACAACTGTATCCAGTGTGCCCACTACATTGACGGCCCCCACTGCGTCAAGACCTGCCCGGCAGGAGTCATGGGAGAAAACAACACCCTGGTCTGGAAGTACGCAGACGCCGGCCATGTGTGCCACCTGTGCCATCCAAACTGCACCTACGGATGCACTGGGCCAGGTCTTGAAGGCTGTCCAACGAATGGGCCTAAGATCCCGTCCATCGCCACTGGGATGGTGGGGGCCCTCCTCTTGCTGCTGGTGGTGGCCCTGGGGATCGGCCTCTTCATGGGATCTGGAGCCACGAACTTCTCTCTGTTAAAGCAAGCAGGAGACGTGGAAGAAAACCCCGGTCCTATGCTCGAGGGAGTGCAGGTGGAAACCATCTCCCCAGGAGACGGGCGCACCTTCCCCAAGCGCGGCCAGACCTGCGTGGTGCACTACACCGGGATGCTTGAAGATGGAAAGAAAGTTGATTCCTCCCGGGACAGAAACAAGCCCTTTAAGTTTATGCTAGGCAAGCAGGAGGTGATCCGAGGCTGGGAAGAAGGGGTTGCCCAGATGAGTGTGGGTCAGAGAGCCAAACTGACTATATCTCCAGATTATGCCTATGGTGCCACTGGGCACCCAGGCATCATCCCACCACATGCCACTCTCGTCTTCGATGTGGAGCTTCTAAAACTGGAATCTGGCGGTGGATCCGGAGTCGACGGATTTGGTGATGTCGGTGCTCTTGAGAGTTTGAGGGGAAATGCAGATTTGGCTTACATCCTGAGCATGGAGCCCTGTGGCCACTGCCTCATTATCAACAATGTGAACTTCTGCCGTGAGTCCGGGCTCCGCACCCGCACTGGCTCCAACATCGACTGTGAGAAGTTGCGGCGTCGCTTCTCCTCGCTGCATTTCATGGTGGAGGTGAAGGGCGACCTGACTGCCAAGAAAATGGTGCTGGCTTTGCTGGAGCTGGCGCAGCAGGACCACGGTGCTCTGGACTGCTGCGTGGTGGTCATTCTCTCTCACGGCTGTCAGGCCAGCCACCTGCAGTTCCCAGGGGCTGTCTACGGCACAGATGGATGCCCTGTGTCGGTCGAGAAGATTGTGAACATCTTCAATGGGACCAGCTGCCCCAGCCTGGGAGGGAAGCCCAAGCTCTTTTTCATCCAGGCCTGTGGTGGGGAGCAGAAAGACCATGGGTTTGAGGTGGCCTCCACTTCCCCTGAAGACGAGTCCCCTGGCAGTAACCCCGAGCCAGATGCCACCCCGTTCCAGGAAGGTTTGAGGACCTTCGACCAGCTGGACGCCATATCTAGTTTGCCCACACCCAGTGACATCTTTGTGTCCTACTCTACTTTCCCAGGTTTTGTTTCCTGGAGGGACCCCAAGAGTGGCTCCTGGTACGTTGAGACCCTGGACGACATCTTTGAGCAGTGGGCTCACTCTGAAGACCTGCAGTCCCTCCTGCTTAGGGTCGCTAATGCTGTTTCGGTGAAAGGGATTTATAAACAGATGCCTGGTTGCTTTAATTTCCTCCGGAAAAAACTTTTCTTTAAAACATCAGTCGACTATCCGTACGACGTACCAGACTACGCACTCGACTAAACAATCAACCTCTGGATTACAAAATTTGTGAAAGATTGACTGGTATTCTTAACTATGTTGCTCCTTTTACGCTATGTGGATACGCTGCTTTAATGCCTTTGTATCATGCTATTGCTTCCCGTATGGCTTTCATTTTCTCCTCCTTGTATAAATCCTGGTTGCTGTCTCTTTATGAGGAGTTGTGGCCCGTTGTCAGGCAACGTGGCGTGGTGTGCACTGTGTTTGCTGACGCAACCCCCACTGGTTGGGGCATTGCCACCACCTGTCAGCTCCTTTCCGGGACTTTCGCTTTCCCCCTCCCTATTGCCACGGCGGAACTCATCGCCGCCTGCCTTGCCCGCTGCTGGACAGGGGCTCGGCTGTTGGGCACTGACAATTCCGTGGTGTTGTCGGGGAAATCATCGTCCTTTCCTTGGCTGCTCGCCTGTGTTGCCACCTGGATTCTGCGCGGGACGTCCTTCTGCTACGTCCCTTCGGCCCTCAATCCAGCGGACCTTCCTTCCCGCGGCCTGCTGCCGGCTCTGCGGCCTCTTCCGCGTCTTCGCCTTCGCCCTCAGACGAGTCGGATCTCCCTTTGGGCCGCCTCCCCGCCTATCGATAAAATAAAAGATTTTATTTAGTCTCCAGAAAAAGGGGGGAATGAAAGACCCCACCTGTAGGTTTGGCAAGCTAGCTTAAGTAACGCCATTTTGCAAGGCATGGAAAATACATAACTGAGAATAGAGAAGTTCAGATCAAGGTTAGGAACAGAGAGACAGCAGAATATGGGCCAAACAGGATATCTGTGGTAAGCAGTTCCTGCCCCGGCTCAGGGCCAAGAACAGATGGTCCCCAGATGCGGTCCCGCCCTCAGCAGTTTCTAGAGAACCATCAGATGTTTCCAGGGTGCCCCAAGGACCTGAAATGACCCTGTGCCTTATTTGAACTAACCAATCAGTTCGCTTCTCGCTTCTGTTCGCGCGCTTCTGCTCCCCGAGCTCAATAAAAGAGCCCACAACCCCTCACTCGGCGCGCCAGTCCTCCGATAGACTGCGTCGCCCGGGTACCCGTGTATCCAATAAACCCTCTTGCAGTTGCATCCGACTTGTGGTCTCGCTGTTCCTTGGGAGGGTCTCCTCTGAGTGATTGACTACCCGTCAGCGGGGGTCTTTCATGGGTAACAGTTTCTTGAAGTTGGAGAACAACATTCTGAGGGTAGGAGTCGAATATTAAGTAATCCTGACTCAATTAGCCACTGTTTTGAATCCACATACTCCAATACTCCTGAAATAGTTCATTATGGACAGCGCAGAAGAGCTGGGGAGAATTAATTCGTAATCATGGTCATAGCTGTTTCCTGTGTGAAATTGTTATCCGCTCACAATTCCACACAACATACGAGCCGGAAGCATAAAGTGTAAAGCCTGGGGTGCCTAATGAGTGAGCTAACTCACATTAATTGCGTTGCGCTCACTGCCCGCTTTCCAGTCGGGAAACCTGTCGTGCCAGCTGCATTAATGAATCGGCCAACGCGCGGGGAGAGGCGGTTTGCGTATTGGGCGCTCTTCCGCTTCCTCGCTCACTGACTCGCTGCGCTCGGTCGTTCGGCTGCGGCGAGCGGTATCAGCTCACTCAAAGGCGGTAATACGGTTATCCACAGAATCAGGGGATAACGCAGGAAAGAACATGTGAGCAAAAGGCCAGCAAAAGGCCAGGAACCGTAAAAAGGCCGCGTTGCTGGCGTTTTTCCATAGGCTCCGCCCCCCTGACGAGCATCACAAAAATCGACGCTCAAGTCAGAGGTGGCGAAACCCGACAGGACTATAAAGATACCAGGCGTTTCCCCCTGGAAGCTCCCTCGTGCGCTCTCCTGTTCCGACCCTGCCGCTTACCGGATACCTGTCCGCCTTTCTCCCTTCGGGAAGCGTGGCGCTTTCTCATAGCTCACGCTGTAGGTATCTCAGTTCGGTGTAGGTCGTTCGCTCCAAGCTGGGCTGTGTGCACGAACCCCCCGTTCAGCCCGACCGCTGCGCCTTATCCGGTAACTATCGTCTTGAGTCCAACCCGGTAAGACACGACTTATCGCCACTGGCAGCAGCCACTGGTAACAGGATTAGCAGAGCGAGGTATGTAGGCGGTGCTACAGAGTTCTTGAAGTGGTGGCCTAACTACGGCTACACTAGAAGGACAGTATTTGGTATCTGCGCTCTGCTGAAGCCAGTTACCTTCGGAAAAAGAGTTGGTAGCTCTTGATCCGGCAAACAAACCACCGCTGGTAGCGGTGGTTTTTTTGTTTGCAAGCAGCAGATTACGCGCAGAAAAAAAGGATCTCAAGAAGATCCTTTGATCTTTTCTACGGGGTCTGACGCTCAGTGGAACGAAAACTCACGTTAAGGGATTTTGGTCATGAGATTATCAAAAAGGATCTTCACCTAGATCCTTTTAAATTAAAAATGAAGTTTTAAATCAATCTAAAGTATATATGAGTAAACTTGGTCTGACAGTTACCAATGCTTAATCAGTGAGGCACCTATCTCAGCGATCTGTCTATTTCGTTCATCCATAGTTGCCTGACTCCCCGTCGTGTAGATAACTACGATACGGGAGGGCTTACCATCTGGCCCCAGTGCTGCAATGATACCGCGAGACCCACGCTCACCGGCTCCAGATTTATCAGCAATAAACCAGCCAGCCGGAAGGGCCGAGCGCAGAAGTGGTCCTGCAACTTTATCCGCCTCCATCCAGTCTATTAATTGTTGCCGGGAAGCTAGAGTAAGTAGTTCGCCAGTTAATAGTTTGCGCAACGTTGTTGCCATTGCTACAGGCATCGTGGTGTCACGCTCGTCGTTTGGTATGGCTTCATTCAGCTCCGGTTCCCAACGATCAAGGCGAGTTACATGATCCCCCATGTTGTGCAAAAAAGCGGTTAGCTCCTTCGGTCCTCCGATCGTTGTCAGAAGTAAGTTGGCCGCAGTGTTATCACTCATGGTTATGGCAGCACTGCATAATTCTCTTACTGTCATGCCATCCGTAAGATGCTTTTCTGTGACTGGTGAGTACTCAACCAAGTCATTCTGAGAATAGTGTATGCGGCGACCGAGTTGCTCTTGCCCGGCGTCAATACGGGATAATACCGCGCCACATAGCAGAACTTTAAAAGTGCTCATCATTGGAAAACGTTCTTCGGGGCGAAAACTCTCAAGGATCTTACCGCTGTTGAGATCCAGTTCGATGTAACCCACTCGTGCACCCAACTGATCTTCAGCATCTTTTACTTTCACCAGCGTTTCTGGGTGAGCAAAAACAGGAAGGCAAAATGCCGCAAAAAAGGGAATAAGGGCGACACGGAAATGTTGAATACTCATACTCTTCCTTTTTCAATATTATTGAAGCATTTATCAGGGTTATTGTCTCATGAGCGGATACATATTTGAATGTATTTAGAAAAATAAACAAATAGGGGTTCCGCGCACATTTCCCCGAAAAGTGCCACCTGACGTCTAAGAAACCATTATTATCATGACATTAACCTATAAAAATAGGCGTATCACGAGGCCCTTTCGTCTCGCGCGTTTCGGTGATGACGGTGAAAACCTCTGACACATGCAGCTCCCGGAGACGGTCACAGCTTGTCTGTAAGCGGATGCCGGGAGCAGACAAGCCCGTCAGGGCGCGTCAGCGGGTGTTGGCGGGTGTCGGGGCTGGCTTAACTATGCGGCATCAGAGCAGATTGTACTGAGAGTGCACCATATGCGGTGTGAAATACCGCACAGATGCGTAAGGAGAAAATACCGCATCAGGCGCCATTCGCCATTCAGGCTGCGCAACTGTTGGGAAGGGCGATCGGTGCGGGCCTCTTCGCTATTACGCCAGCTGGCGAAAGGGGGATGTGCTGCAAGGCGATTAAGTTGGGTAACGCCAGGGTTTTCCCAGTCACGACGTTGTAAAACGACGGCGCAAGGAATGGTGCATGCAAGGAGATGGCGCCCAACAGTCCCCCGGCCACGGGGCCTGCCACCATACCCACGCCGAAACAAGCGCTCATGAGCCCGAAGTGGCGAGCCCGATCTTCCCCATCGGTGATGTCGGCGATATAGGCGCCAGCAACCGCACCTGTGGCGCCGGTGATGCCGGCCACGATGCGTCCGGCGTAGAGGCGATTAGTCCAATTTGTTAAAGACAGGATATCAGTGGTCCAGGCTCTAGTTTTGACTCAACAATATCACCAGCTGAAGCCTATAGAGTACGAGCCATAGATAAAATAAAAGATTTTATTTAGTCTCCAGAAAAAGGGGGGAA
[SEQ ID NO:36]
优选地,载体包含基本上如SEQ ID NO:36所示的核酸序列或其片段或变体。
因此,应当理解,使用第二方面或第三方面的方法获得的分离的MAIT细胞可以被激活,并最终用根据第六方面的编码CAR的表达载体转导,从而产生第一方面或第四方面的CAR-MAIT细胞。
在第十一方面,本文提供了包含根据第五构建体的构建体或根据第六方面的载体的T细胞,任选地其中所述T细胞表达抗CD4嵌合抗原受体(CAR)或抗Vbeta CAR。
优选地,T细胞是粘膜相关恒定T(MAIT)细胞。
在第七方面,本文提供了一种药物组合物,其包含根据第十一方面的T细胞,优选根据第一方面或第四方面的MAIT细胞,和药学上可接受的赋形剂。
优选地,药物组合物包含多个本发明的T细胞或MAIT细胞。例如,组合物可包含至少100、1000或10000个T细胞或MAIT细胞。优选地,组合物包含至少100000、至少1000000或至少10000000个T细胞或MAIT细胞。
在第八方面,本文提供了根据第十一方面的T细胞、根据第一方面或第四方面的MAIT细胞或第七方面的药物组合物用于治疗。
在第九方面,本文提供了根据第十一方面的T细胞、根据第一方面或第四方面的MAIT细胞根据第七方面的药物组合物用于(i)免疫疗法;(ii)治疗、预防或改善癌症;(ii)治疗、预防或改善微生物感染;或(iv)治疗、预防或改善自身免疫性疾病。
在第十方面,本发明提供了一种方法:(i)用免疫疗法治疗、预防或改善受试者的疾病;(ii)治疗、预防或改善癌症;(iii)治疗、预防或改善受试者的微生物感染;或(iv)治疗、预防或改善受试者的自身免疫性疾病,该方法包括向需要这种治疗的患者施用或已经施用治疗有效量的根据第十一方面的T细胞、根据第一方面或第四方面的MAIT细胞或第七方面的药物组合物。
优选地,T细胞、MAIT细胞或药物组合物用于治疗、预防或改善T细胞恶性肿瘤,其可以是实体瘤或液态肿瘤。
T细胞恶性肿瘤可以是外周T细胞淋巴瘤(PTCL)或皮肤T细胞淋巴瘤(CTCL)。
外周T细胞淋巴瘤(PTCL)包括多种罕见的侵袭性疾病,该疾病患者的T细胞会发生癌变。PTCL分为三类,即结内的、结外的和白血病的,每一类都包含在本发明内。
PTCL可以是选自下组的PTCL亚型:成人T细胞急性淋巴细胞性淋巴瘤或白血病(ATL)、肠病相关淋巴瘤、肝脾淋巴瘤、皮下脂膜炎样淋巴瘤(SPTCL)、前体T细胞急性淋巴细胞性淋巴瘤或白血病和血管免疫母细胞性T细胞淋巴瘤(AITL)。
成人T细胞急性淋巴细胞性淋巴瘤或白血病(ATL)在日本和加勒比地区比在美国更常见,并且与人T细胞白血病病毒-1(HTLV-1)有关。肠病相关淋巴瘤与乳糜泻有关,乳糜泻是一种慢性肠道疾病,由对小麦、黑麦和大麦中的麸质蛋白过敏引起。症状通常包括胃痛、体重减轻、胃肠道出血或肠穿孔。肠病相关T细胞淋巴瘤患者的治疗包括基于蒽环类药物的化疗方案、营养补充剂以及(如果适用)无麸质饮食。肝脾淋巴瘤是一种极其罕见的侵袭性疾病,始于肝脏或脾脏,通常影响20多岁和30多岁的年轻人。肝脾T细胞淋巴瘤患者的治疗包括基于蒽环类药物的化疗,在某些情况下还包括干细胞移植。
皮下脂膜炎样淋巴瘤(SPTCL)是最罕见且定义最不明确的T细胞淋巴瘤。这种淋巴瘤主要发生在皮下脂肪组织中,导致结节形成。症状包括发烧、发冷、体重减轻和口腔粘膜溃疡。SPTCL可能具有快速攻击性或惰性(生长缓慢)。治疗包括基于蒽环类药物的联合化疗或局部放疗。前体T细胞急性淋巴细胞性淋巴瘤或白血病可能被诊断为白血病或淋巴瘤或两者兼而有之。这种癌症在儿童和成人中都有发现,最常见于青少年和成年男性。新诊断患有前体T细胞急性淋巴细胞性淋巴瘤或白血病的患者的治疗方法是积极的化疗和放疗。奈拉滨
血管免疫母细胞性T细胞淋巴瘤(AITL)是一种以强烈炎症和免疫反应为特征的肿瘤,其临床、病理、细胞和生物学特性证明了这一点。由于肿瘤细胞在表型上类似于滤泡辅助T(Tfh)细胞,因此认为它们的功能在某种程度上与反应性滤泡增生中看到的非肿瘤性Tfh细胞相似。然而,在绝大多数AITL病例中,卵泡并不增生,而是相当耗尽或破坏。据最近报道,AITL占PTCL的36.1%。
皮肤T细胞淋巴瘤(CTCL)约占原发性皮肤淋巴瘤的70-75%。CTCL可以是选自下组的CTCL亚型:蕈样肉芽肿(MF)、塞扎里综合征(SS)和CD4+小中型多形性T细胞淋巴增殖性疾病。
蕈样肉芽肿(MF)是最常见的亚型。塞扎里综合征(SS)是一种更具侵袭性的CTCL类型。SS患者患有红皮病(即影响>80%体表面积[BSA]的皮疹)、淋巴结病以及外周血中存在大量循环肿瘤性CD4+T细胞。
在其他实施方案中,T细胞、MAIT细胞或药物组合物可以用于治疗、预防或改善病毒(例如HIV、HBV、HTLV、EBV、HPV)、细菌(例如TB)或真菌感染。
在其他实施方案中,T细胞、MAIT细胞或药物组合物可用于治疗、预防或改善自身免疫性疾病,例如系统性红斑狼疮、类风湿性关节炎或重症肌无力。
优选地,该方法包括触发编码自杀蛋白的序列。因此,在核酸构建体包含编码EGFR或tEGFR的序列的实施方案中,该方法优选地包括向受试者施用抗EGFR抗体。例如,抗EGFR抗体可以是单克隆抗体西妥昔单抗。施用抗体能够监测或消除受试者体内的CAR-T细胞。
在核酸构建体包含编码iC9的序列的实施方案中,该方法可以包括向受试者施用半胱天冬酶诱导药物(CID)。例如,CID可以包括Rimiducid。施用CID能够使半胱天冬酶有条件地二聚化,从而触发表达融合蛋白的CAR-T细胞凋亡,从而导致受试者体内CAR-T细胞的耗尽。
在最优选的实施方案中,构建体编码两种自杀蛋白,包括iC9和tEGFR,因此抗EGFR抗体和CID的使用能够精确控制CAR-T或CAR-MAIT细胞在接受治疗的受试者中的寿命。
应当理解,根据本发明的CAR-T细胞或CAR-MAIT细胞(在本文中统称为“试剂”)可以用于单一疗法(例如单独使用T细胞或CAR-MAIT细胞),用于治疗(优选在免疫治疗中),其中用于(i)治疗、改善或预防癌症或T细胞恶性肿瘤;或(ii)治疗、预防或改善微生物感染,或(iii)自身免疫性疾病。或者,根据本发明的CAR-T或CAR-MAIT细胞可用作已知免疫疗法的辅助剂或与其组合,或用于治疗微生物感染以及癌症或自身免疫性疾病。
本发明的试剂可以组合在具有多种不同形式的组合物中,具体取决于组合物的使用方式。因此,例如,组合物可以是粉末、片剂、胶囊、液体、软膏、霜剂、凝胶、水凝胶、气雾剂、喷雾剂、胶束溶液、透皮贴剂、脂质体悬浮液的形式或是任何其他适于向需要治疗的人或动物施用的形式。应当理解,根据本发明的药剂载体应该是被施用受试者良好耐受的载体。
包含本发明试剂的药剂可以多种方式使用。例如,可能需要口服施用,在这种情况下,药剂可以包含在组合物内,该组合物可以例如以片剂、胶囊或液体的形式口服摄入。包含本发明试剂和药剂的组合物可以通过吸入(例如鼻内)施用。组合物也可配制用于局部使用。例如,可以将霜剂或软膏施用于皮肤。
根据本发明的试剂和药剂还可以并入缓释或延迟释放装置中。此类装置可以例如插入皮肤上或皮肤下,并且药物可以在数周甚至数月内释放。该装置可以至少位于邻近治疗部位的位置。当需要用根据本发明的药剂进行长期治疗并且通常需要频繁施用(例如至少每天注射)时,此类装置可能特别有利。
在一个优选的实施方案中,根据本发明的试剂和药剂可以通过注射到血流中或直接注射到需要治疗的部位来施用于受试者。注射可以是静脉内(推注或输注)或皮下(推注或输注)或皮内(推注或输注)。
应当理解,所需的遗传构建体或载体(即试剂)的量由其生物活性和生物利用度决定,而生物活性和生物利用度又取决于施用模式、试剂的理化性质以及是否被用作单一疗法或联合疗法。施用频率也将受到所治疗受试者体内药剂的半衰期的影响。待施用的最佳剂量可由本领域技术人员确定,并且将随所使用的具体药剂、药物组合物的强度、施用方式和所治疗的疾病(例如癌症、T细胞恶性肿瘤、微生物感染或自身免疫性疾病)的进展而变化。取决于所治疗的特定受试者的其他因素将导致需要调整剂量,包括受试者年龄、体重、性别、饮食和施用时间。
一般而言,在0.001μg/kg体重至10mg/kg体重之间的本发明试剂的日剂量可用于治疗,特别是用于治疗、改善或预防癌症、T细胞恶性肿瘤、微生物感染或自身免疫性疾病,具体取决于具体试剂。更优选地,试剂的日剂量为0.01μg/kg体重至1mg/kg体重,更优选为0.1μg/kg至100μg/kg体重,最优选为约0.1μg/kg至10μg/kg体重。
或者,施用于受试者的剂量可以在0.5x10
该药剂可以在癌症、T细胞恶性肿瘤、微生物感染或自身免疫性疾病发作之前、期间或之后施用。每日剂量可以作为单次给药(例如每日单次注射)施用。或者,试剂可能会要求一天内给药两次或多次。作为示例,可以以0.07μg至700mg(即假设体重为70kg)之间的每日两次(或更多次,取决于所治疗的疾病的严重程度,例如癌症)剂量来施用药剂。接受治疗的患者可以在醒来时施用第一剂,然后在晚上施用第二剂(如果采用两次剂量方案)或此后每隔3或4小时施用第二剂。或者,该药剂可能需要每周施用一次,甚至每月一次。或者,可以使用缓释装置向患者提供根据本发明的试剂的最佳剂量,而不需要施用重复剂量。已知的程序,例如制药业常规采用的程序(例如体内实验、临床试验等),可用于形成根据本发明的试剂的特定配方和精确的治疗方案(例如试剂的每日剂量和给药频率)。
本发明的药物组合物优选是免疫疗法治疗组合物、自身免疫性疾病治疗组合物、抗感染组合物或抗癌组合物,即用于受试者治疗性改善、预防或治疗癌症的药物制剂。
在第十一方面,本发明还提供了制备根据第七方面的药物组合物的方法,该方法包括将治疗有效量的根据第一方面或第四方面的MAIT细胞与药学上可接受的载体组合。
“受试者”可以是脊椎动物、哺乳动物或家畜。因此,根据本发明的药剂可以用于治疗任何哺乳动物,例如牲畜(例如马)、宠物,或者可以用于其他兽医应用。最优选地,受试者是人类。
遗传构建体或载体的“治疗有效量”是当施用于受试者时,治疗所治疗的疾病(例如癌症)或产生期望效果所需的药剂的任何量。
例如,所使用的遗传构建体或载体的治疗有效量可以是约0.001ng至约1mg,优选约0.01ng至约100ng。优选地,遗传构建体或载体的量为约0.1ng至约10ng,最优选为约0.5ng至约5ng。
本文提及的“药学上可接受的载体”是本领域技术人员已知可用于配制药物组合物的任何已知化合物或已知化合物的组合。
在一实施方案中,药学上可接受的载体可以是固体,组合物可以是粉末或片剂的形式。固体药学上可接受的载体可以包括一种或多种物质,这些物质也可以充当矫味剂、润滑剂、增溶剂、悬浮剂、染料、填充剂、助流剂、压缩助剂、惰性粘合剂、甜味剂、防腐剂、染料、包衣或片剂崩解剂。载体还可以是封装材料。在粉末中,载体是与根据本发明的细分活性剂混合的细分固体。在片剂中,活性剂可以与具有必要的压缩特性的载体以合适的比例混合并压制成所需的形状和尺寸。粉末和片剂优选含有高达99%的活性剂。合适的固体载体包括例如磷酸钙、硬脂酸镁、滑石、糖、乳糖、糊精、淀粉、明胶、纤维素、聚乙烯吡咯烷、低熔点蜡和离子交换树脂。在另一实施方案中,药物载体可以是凝胶并且组合物可以是乳膏等形式。
然而,药物载体可以是液体,药物组合物是溶液的形式。液体载体可用于制备溶液、悬浮液、乳液、糖浆、酏剂和加压组合物。本发明的活性剂可以溶解或悬浮在药学上可接受的液体载体中,例如水、有机溶剂、两者的混合物或药学上可接受的油或脂肪。液体载体可以含有其他合适的药物添加剂,例如增溶剂、乳化剂、缓冲剂、防腐剂、甜味剂、矫味剂、悬浮剂、增稠剂、着色剂、粘度调节剂、稳定剂或渗透压调节剂。用于口服和肠胃外施用的液体载体的合适实例包括水(部分含有如上所述的添加剂,例如纤维素衍生物,优选羧甲基纤维素钠溶液)、醇(包括一元醇和多元醇,例如乙二醇)及其衍生物,以及油(例如分馏椰子油和花生油)。对于肠胃外给药,载体还可以是油酯,例如油酸乙酯和肉豆蔻酸异丙酯。无菌液体载体可用于用于肠胃外施用的无菌液体形式组合物。用于加压组合物的液体载体可以是卤代烃或其他药学上可接受的推进剂。
液体药物组合物为无菌溶液或悬浮液,可通过肌内注射、鞘内注射、硬膜外注射、腹膜内注射、静脉内注射且特别是皮下注射等方式使用。试剂可以配置为无菌固体组合物,其可以在施用时使用无菌水、盐水或其他合适的无菌可注射介质溶解或悬浮。
本发明的试剂和组合物可以以无菌溶液或混悬液的形式口服施用,其中含有其他溶质或混悬剂(例如,足够的盐水或葡萄糖以使溶液具有等渗性)、胆汁盐、阿拉伯胶、明胶、失水山梨糖醇单油酸酯、聚山梨醇酯80(山梨醇及其酸酐与环氧乙烷共聚的油酸酯)等。根据本发明使用的试剂还可以以液体或固体组合物形式口服施用。适合口服施用的组合物包括固体形式,例如丸剂、胶囊、颗粒剂、片剂和粉末,以及液体形式,例如溶液剂、糖浆剂、酏剂和混悬剂。可用于肠胃外施用的形式包括无菌溶液、乳液和悬浮液。
应当理解,本发明延伸至任何核酸或肽或其变体、衍生物或类似物,其基本上包含本文提及的任何序列的氨基酸或核酸序列,包括其变体或片段。术语“基本上氨基酸/核苷酸/肽序列”、“变体”和“片段”可以是与本文中所提及的任一序列的氨基酸/核苷酸/肽序列具有至少40%序列同一性的序列。例如与SEQ ID No:1至SEQ ID No:36等所标识的序列有40%的同一性。
氨基酸/多核苷酸/多肽序列与上述任何序列的序列同一性大于65%,更优选大于70%,甚至更优选大于75%,更优选大于80%,也是可以考虑的。优选地,氨基酸/多核苷酸/多肽序列与本文所提及的任何序列具有至少85%同一性,更优选至少90%同一性,甚至更优选至少92%同一性,甚至更优选至少95%同一性,甚至更优选至少97%同一性,甚至更优选至少98%同一性,最优选至少99%同一性。
技术人员将理解如何计算两个氨基酸/多核苷酸/多肽序列之间的同一性百分比。为了计算两个氨基酸/多核苷酸/多肽序列之间的同一性百分比,必须首先对两个序列进行比对,然后计算序列同一性值。两个序列的百分比同一性可能取不同的值,具体取决于:(i)用于比对序列的方法,例如ClustalW、BLAST、FASTA、Smith-Waterman(在不同程序中实现)或来自3D比较的结构比对;(ii)比对方法使用的参数,例如局部与全局比对、使用的配对分数矩阵(例如BLOSUM62、PAM250、Gonnet等),以及间隙罚分,例如函数形式和常数。
进行比对后,有许多不同的方法来计算两个序列之间的同一性百分比。例如,可以将同一性的数量除以:(i)最短序列的长度;(ii)对齐长度;(iii)序列的平均长度;(iv)无间隙位置的数量;或(v)除悬垂外的等效位置的数量。此外,应当理解,同一性百分比也与长度密切相关。因此,一对序列越短,偶然出现的序列同一性就越高。
因此,应当理解,蛋白质或DNA序列的精确比对是一个复杂的过程。流行的多重比对程序ClustalW(Thompson et al.,1994,Nucleic Acids Research,22,4673-4680;Thompson et al.,1997,Nucleic Acids Research,24,4876-4882)是根据本发明生成蛋白质或DNA的多重比对的首选方法。ClustalW的合适参数如下:对于DNA比对:空位开放罚分=15.0,空位延伸罚分=6.66,矩阵=同一性。对于蛋白质比对:空位开放罚分=10.0,空位延伸罚分=0.2,矩阵=Gonnet。对于DNA和蛋白质比对:ENDGAP=-1,GAPDIST=4。本领域技术人员将意识到,可能有必要改变这些参数和其他参数,以实现最佳序列比对。
优选地,两个氨基酸/多核苷酸/多肽序列之间同一性百分比的计算可以通过这样的比对计算出来,即(N/T)*100,其中N是序列共享相同残基的位置数,T是比对的位置总数,包括空位以及包括或不包括悬垂部分。优选地,计算中包括悬垂部分。因此,用于计算两个序列之间的百分比同一性的最优选方法包括(i)使用ClustalW程序使用一组合适的参数来准备序列比对,例如如上所述的参数;(ii)将N和T的值代入以下公式:-序列同一性=(N/T)*100。
本领域技术人员都知晓用于鉴定相似序列的其他方法。例如,基本相似的核苷酸序列将由在严格条件下与DNA序列或其互补序列杂交的序列编码。所谓严格条件,发明人是指在大约45℃下在3x氯化钠/柠檬酸钠(SSC)中核苷酸与滤膜结合的DNA或RNA杂交,然后在大约20-65℃下在0.2xSSC/0.1%SDS中洗涤至少一次。另外,基本上相似的多肽可以与例如SEQ ID No:1至SEQ ID No:36中所示的氨基酸序列有至少1个、但少于5、10、20、50或100个氨基酸的差异。
由于遗传密码的简并性,显然本文所述的任何核酸序列都可以在不对其编码的蛋白质序列产生实质性影响的情况下进行变化或改变,以提供其功能变体。合适的核苷酸变体是那些具有通过替换序列内编码相同氨基酸的不同密码子而改变的序列的核苷酸变体,从而产生沉默(同义)改变。其他合适的变体是具有同源核苷酸序列但包含全部或部分序列的变体,其通过不同密码子的取代而改变,这些密码子编码具有与其取代的氨基酸具有相似生物物理特性的侧链的氨基酸,以产生保守的改变。例如,小的非极性疏水氨基酸包括甘氨酸、丙氨酸、亮氨酸、异亮氨酸、缬氨酸、脯氨酸和甲硫氨酸。大的非极性疏水氨基酸包括苯丙氨酸、色氨酸和酪氨酸。极性中性氨基酸包括丝氨酸、苏氨酸、半胱氨酸、天冬酰胺和谷氨酰胺。带正电荷的(碱性)氨基酸包括赖氨酸、精氨酸和组氨酸。带负电(酸性)氨基酸包括天冬氨酸和谷氨酸。因此,应当理解哪些氨基酸可以用具有类似生物物理特性的氨基酸取代是显而易见的,熟练的技术人员也知道编码这些氨基酸的核苷酸序列。
本文描述的所有特征(包括任何所附权利要求、摘要和附图)和/或公开的任何方法或过程的所有步骤可以与上述任何方面任意组合,但至少其中某些特征和/或步骤相互排斥的组合除外。
为了更好地理解本发明,并且为了说明如何实施本发明的实施例,现在将通过举例的方式参考附图,其中:
图1示出了根据本发明的实施方案的第三代CD4靶向T细胞的产生。A(1):该图表示包含在根据本发明的CAR构建体(称为“CART4”)的一个实施方案中的功能元件。源自单克隆抗体Hu5A8的scFv与CD8跨膜结构域(TM)、CD28胞内结构域、4-1BB胞内结构域和CD3ζ链融合。tEGFR(截短的表皮生长因子受体)和iC9(诱导型半胱天冬酶9)的基因序列通过自切割2A接头标记在CAR后面。A(2):该图表示包含在根据本发明的CAR构建体(称为“CARTVb7.1”)的另一个实施方案中的功能元件。源自单克隆抗体3G5的scFv与CD8跨膜结构域(TM)、CD28胞内结构域、4-1BB胞内结构域和CD3ζ链融合。tEGFR(截短的表皮生长因子受体)和iC9(诱导型半胱天冬酶9)的基因序列通过自切割2A接头标记在CAR后面。B:转导的T细胞用抗小鼠IgG F(ab)’2抗体和抗EGFR抗体染色。细胞在CD3
图2示出了根据本发明的CART4 T细胞的实施方案的体外功能验证。A:PBMC由Dynabeads人T活化剂和IL7/IL15激活。激活的PBMC包含两个T细胞亚群:CD4+和CD8+(左)。细胞由CART20(中)或CART4(右)逆转录病毒颗粒转导。从转导后第三天开始,用抗CD4和抗CD8抗体对细胞进行染色,并通过流式细胞术进行分析。B:该图中汇总了CD4+比率的统计数据。数据反映了五个健康个体的典型结果。C:原代CD4+T细胞(左)或CD20+B细胞(右)与自体CART4细胞、CART20细胞或非转导CD8+T细胞共培养4小时。使用Countbright珠通过流式细胞术分析对存活靶细胞的绝对数量进行计数。D:用抗CD4抗体对两种T细胞系CEM-ss细胞、Jurkat细胞和一种B细胞系进行染色。通过流式细胞术分析评估CD4表达水平。E:CART4细胞杀伤T肿瘤细胞的代表性结果。每个样本重复三次。数据反映了三个独立实验的典型结果。F:与不同靶细胞共培养的CART4细胞的胞内细胞因子表达。每个样本重复三次。数据反映了三个独立实验的典型结果。
图3示出了CART4细胞特异性杀伤CD4+T肿瘤细胞。在进行流式细胞术分析和共培养实验前,从液氮中复苏ATLL患者的PBMC,并在培养箱中静置过夜。A:PBMC用抗CD4、CD8和特异性TCR Vbeta染色。用PBS洗涤两次后进行流式细胞术分析。在流式细胞术分析之前,将复苏的ATLL(B)或CTCL(C)PBMC与同种异体CART4或CART20细胞共培养4小时。每个条件重复三次。数据以平均值±SEM表示。
图4示出了CART4细胞在体内有效介导抗白血病作用。A:NRG免疫缺陷小鼠注射1x10
图5示出了符合GMP标准的CAR-T细胞制造方法的开发。A:CAR-T细胞制造的时间进程。在逆转录病毒转导CAR之前,先在烧瓶中用CD3/CD28 Dynabeads和IL7/IL15激活人PBMC。。转导两天后,将转导细胞转移至G-Rex板(1x10
图6示出了CARTVb7.1的产生和功能验证。A:转导的T细胞用抗EGFR抗体染色以检测CAR表达。细胞在CD3+单淋巴细胞上增殖,数字表示tEGFR+细胞的百分比。B:CAR转导后五天的内源TCRVb7.1+群体检测。C:TCRVb7.1+原代ATL样本经CFSE染色并与不同数量的效应CAR-T细胞混合。培养6小时后,收集细胞并用DAPI、3G5和CD3抗体染色15分钟。在每个样本中加入固定体积的5uL Countbright珠。将样本载入流式细胞仪中进行绝对定量。D:CARTVb7.1细胞杀伤T肿瘤细胞的代表性结果。每个样本重复三次。
图7示出了MAIT-CART细胞的产生过程。A:MAIT细胞染色的代表性流式细胞术示例。用BV421 MR1-5-OP-RU四聚体和PE抗TCR Vα7.2抗体对PBMC染色20分钟。在通过流式细胞术表征之前,用PBS洗涤细胞两次。B:流式细胞术分选MAIT细胞的门控策略。通过磁分离方法从PBMC中分离TCR Vα7.2+细胞,然后用BV421 MR1-5-OP-RU四聚体和PE抗CD3抗体染色20分钟。细胞经PBS冲洗两次后装入Melody细胞分选仪。对MR1-5-OP-RU四聚体阳性群体进行分选和培养。C:体外培养的MAIT细胞和作为对照的CD8+T细胞的扩增曲线。D:培养12-14天后,超过90%的扩增MAIT细胞具有MR1-5-OP-RU四聚体特异性。E:用抗EGFR流式抗体对CAR转导的CD8+T细胞和MAIT细胞进行染色以检测转导效率。
图8示出了扩增的MAIT和CD8 T细胞与CFSE染色的CD4+细胞系CEMss以E:T 0:1、1:1、3:1和5:1的比例共培养。培养20小时后收获共培养系统。使用Countbright珠通过流式细胞术分析计算存活肿瘤细胞的绝对数量。(A)3:1E:T条件下的流式细胞术数据。(B)细胞毒性统计结果。数据以平均值±SEM表示。
图9示出了用于转导MAIT细胞的表达载体“CART4”的第一实施方案。
图10示出了用于转导MAIT细胞的表达载体“CARTVb7.1”的第二实施方案。
图11示出了从外周血单核细胞(PBMC)中检测人MAIT细胞。对淋巴细胞进行门控,并通过CD3的表达以及与5-OP-RU/MR1四聚体的反应性(A)或CD161和TCRVα7.2的表达(B)来鉴定MAIT细胞。
图12示出了从外周血单核细胞(PBMC)中分离人MAIT细胞。通过磁珠进行Vα7.2表达分离后,Vα7.2阳性细胞群从2.2%(A)富集到>97%。根据与5-OP-RU/MR1四聚体的反应性对MAIT细胞进行分选(B)。
图13示出了CAR-MAIT细胞的产生。培养12-14天后,超过90%的扩增MAIT细胞具有MR1-5-OP-RU四聚体特异性(A)。CAR转导的CD8+T细胞和MAIT细胞用抗EGFR流式抗体染色以检测转导效率(B)。
图14示出了CAR-MAIT4细胞在体内表现出有效的抗白血病功能。A:NSG免疫缺陷小鼠进行静脉注射1x10
图15示出了PBMC中MAIT细胞的富集。在表中所示的不同细胞因子存在的情况下,用MR1/5-OP-RU复合珠(珠与细胞比为1:1)或5-OP-RU抗原(10nM)刺激PBMC 6天。MAIT细胞增加的倍数是通过将第6天的活MAIT(CD3+Va7.2+CD161+)细胞的频率除以第0天的MAIT细胞的原始频率来计算的。前5组用橙色标出(即条件1、3、11、12和13)。IL-12、IL-18和IL-23的组合使PBMC中MAIT细胞的增加倍数最高。
实施例
基于嵌合抗原受体(CAR)的T细胞疗法通过靶向泛B细胞特异性抗原,在治疗B细胞恶性肿瘤方面取得了巨大成功。然而,针对T细胞淋巴瘤的类似策略迄今尚未实现,这主要是由于与B细胞恶性肿瘤相比,T淋巴瘤中整体T细胞耗竭和正常T细胞功能障碍/频率低所带来的潜在严重毒性。为了克服这些限制,发明人设计了两种新型CAR构建体,第一种在本文中被称为“CART4”,其对泛T细胞标记物(CD4)具有特异性,第二种被称为“CARTVb7.1”,其对TCR-Vb同型链具有特异性。这两种CAR构建体均包含一个或两个安全开关,这些开关选自截短的表皮生长因子受体(tEGFR)和诱导型半胱天冬酶9(iC9)。然而,如图1所示,两个安全开关都显示出来了。发明人研究了同种异体反应性低的粘膜相关恒定T(MAIT)细胞在用CAR构建体转导后是否会表现出与传统T细胞类似的抗肿瘤杀伤活性。
此外,众所周知,MAIT细胞是先天性T细胞的一个亚群,定义为CD3+TCRVa7.2+CD161+细胞,可识别MHC I类分子MR1。先前的研究表明,MAIT细胞可以在体外扩增,但需要同种异体饲养细胞的存在。然而,该方法的一个问题是难以大规模生产和质量控制。在本项研究中,发明人现已开发出一种用于体外扩增MAIT细胞的有效方法,其中通过首先用负载抗原(5-OP-RU)的MR1四聚体珠或仅用5-OP-RU刺激PBMC,在多种细胞因子(IL-2、IL-7、IL-15、IL-12、IL-18和IL-23)组合存在下进行长达6天的体外培养。然后通过MACS或FACS分选分离MAIT细胞,随后通过抗CD3/CD28珠进一步扩增以用于基于CAR的治疗,如前面的实施例中所述。
材料与方法
CAR质粒的构建
由Genewiz合成编码Hu5A8和Leu16的scFv以及人Igκ前导序列的DNA片段。NcoI和NotI用于切割这些片段以及MSCV CAR表达逆转录病毒载体。MSCV CAR表达载体由MSCV-IRES-GFP载体(Addgene)改造而成,将IRES-GFP区域替换为人CD8跨膜结构域和第三代CAR胞内信号结构域(CD28和4-1BB的共刺激结构域、CD3ζ信号结构域)。tEGFR的序列从US8802347B2获得,删除了人EGFR蛋白的胞外部分和胞内结构域的结构域I和II。tEGFR由Genewiz合成,具有自切割T2A序列和人粒细胞-巨噬细胞集落刺激因子(GM-CSF)受体的前导肽。iC9的DNA序列由Lishan Su教授(北卡罗来纳大学教堂山分校)友情提供。iC9的DNA片段由截短的半胱天冬酶9组成,包括半胱天冬酶分子的大亚基和小亚基,通过短的Gly-Gly-Gly-Ser(GGGS)柔性接头与一个12-kDa人FK506结合蛋白(FKBP12)连接。
逆转录病毒载体的产生
用MSCV-逆转录病毒质粒和pCMV-VSVG载体(Addgene)通过7ul X-tremeGENE HP转染试剂(Roche)转染Plat-GP细胞(Cellbiolabs)以产生具有VSV包膜的病毒。为了产生高滴度的CAR编码逆转录病毒上清液,随后用PG-13(ATCC)进行了稳定的病毒生产细胞系。PG-13细胞由含有8ug/ml聚凝胺(Sigma)的Plat-GP病毒上清液转导。将板用保鲜膜包裹并在32℃下1000g离心2小时。为了产生高滴度的病毒颗粒,通过胰蛋白酶化将汇合细胞传代。24小时后收集上清液。在300g离心5分钟后,分装培养基并保存在-80℃下。
原代T细胞和肿瘤细胞
通过Ficoll-Paque PLUS密度梯度离心(GE Healthcare)分离健康供体的外周血单核细胞(PBMC),用于CAR-T细胞工程。从ATL或CTCL患者产生的T淋巴瘤细胞系在D10培养基(DMEM,含10%胎牛血清、100IU/mL青霉素、100μg/mL链霉素和2mM L-谷氨酰胺)中培养、T白血病细胞系(Jurket或CEM)在R10培养基(RPMI 1640,含10%胎牛血清、100IU/mL青霉素、100μg/mL链霉素和2mM L-谷氨酰胺)中培养。
MAIT细胞的分离和扩增
使用抗人TCR Vα7.2抗体(Biolegend,Cat#351724)、微珠(Miltenyi,Cat#130-090-485),然后用BV421 MR1-5-OP-RU四聚体(由澳大利亚墨尔本大学的Jim McCluskey教授友情提供),通过两步法从健康PBMC中分离MAIT细胞。简言之,根据制造程序,使用生物素化抗人TCR Vα7.2抗体和抗生物素MicroBeads试剂盒(Miltenyi)从PBMC分离TCR Vα7.2+T细胞。然后通过对BV421 MR1-5-OP-RU四聚体进行染色并使用FACSMelody细胞分选仪(BD)进行FACS分选,将MAIT细胞与TCR Vα7.2+T细胞分离。
通过两步法将MAIT细胞与PBMC分离。对PBMC细胞进行计数,并在15mL试管中的PBS/EDTA缓冲液中稀释至1x 10
MAIT细胞的激活和扩增
对分选的MAIT细胞进行计数,并在R10培养基(90% RPMI+10% FBS+1%青霉素/链霉素+2mM L-谷氨酰胺)中重悬至10
CAR-T或CAR-MAIT细胞的产生
在含有100IU/mL IL-2的R10培养基中用Dynabeads人T活化剂CD3/CD28(LifeTechnologies公司)刺激纯化的PBMC或MAIT细胞48小时。然后用编码CAR构建体的逆转录病毒转染激活的细胞,并在R10中再培养48小时。在收获前,在存在重组IL7和IL15(Miltenyi)的情况下,将CAR-T或CAR-MAIT细胞在G-Rex六孔板(Wilsonwolf)中再维持7天。
CAR-T或CAR-MAIT细胞的产生
在T细胞激活后48小时后进行逆转录病毒转导。如有必要,可以在24小时后重复转导步骤以提高转导效率。将10x10
共培养细胞毒性测定
该非放射性杀伤测定按照之前的报道进行(Rowan等,2014)。简而言之,靶细胞用1uM CFSE(Biolegend)在37℃下染色15分钟。PBS洗涤3次后,将100000个靶细胞与CAR-T或CAR-MAIT细胞按1:1、1:3、1:5的比例混合。将200ul共培养物在37℃培养箱中培养4小时。向样本中加入1ul DAPI和5ul Countbright珠子(BD Biosciences)。通过流式细胞术以恒定速度采集样本。存活的靶细胞的数量计算公式为:试管中的细胞数=(收集的细胞数/收集的珠子数)x加入试管中的珠子总数。
细胞内细胞因子染色
CART4或CART20 T细胞与特定靶细胞在96孔U底板中共培养。所有细胞以2x10
体外自杀测试
用逆转录病毒转导CART4或CART4 w/o iC9构建体生成含有或不含iC9细胞的CART4。CAR-T细胞在转导后保持扩增5至7天。将半胱天冬酶诱导药物(CID)、B/B同二聚体AP20187(Clontech Laboratories)以不同浓度添加到T细胞培养物中。24小时后,使用膜联蛋白-v/7-AAD(BD Biosciences)染色并通过流式细胞术分析评估CID诱导的细胞凋亡。通过计数珠(BD Biosciences)对存活细胞进行定量。存活指数计算方法如下:存活的tEGFR+细胞数/未处理对照样本中存活的tEGFR+细胞数。
体内小鼠异种移植实验
使用6至8周龄的NRG小鼠(Jackson Laboratory)进行T白血病细胞系的体内实验。将共表达Gaussia荧光素酶和EGFP的0.5x10
统计分析
使用GraphPad Prism软件6.0版(GraphPad软件)进行统计分析。使用双尾未配对t检验来比较两组数据。*P<0.05,**P<0.01,***P<0.001。所有带有误差线的数据均以平均值±平均值标准误差(SEM)的形式表示。P值小于0.05为显著。使用GraphPad Prism软件(版本8)分析数据。
1.
1.1.
1.1.1.人外周血单核细胞(PBMC)的制备
1.1.1.1.将5mL志愿者供体的外周血转移至肝素化管中。
1.1.1.2.用等体积的PBS稀释全血。
1.1.1.3.将5mL Histopaque-1077放入15ml离心管中。小心地将10ml稀释的血液溶液沿着管壁轻轻地添加到Histopaque-1077上;不要破坏液体界面。
1.1.1.4.在室温下以400x g离心30分钟。
注意:确保离心机的制动和加速设置处于最低设置,剧烈制动和加速可能会影响层分离。
1.1.1.5.离心后,用巴斯德移液管小心地将上层吸至含有单核细胞的不透明界面0.5cm以内。丢弃上层。
1.1.1.6.通过在250x g下离心10分钟,用10ml PBS清洗收获的PBMC两次。
1.1.1.7.用RPMI1640培养基重新悬浮PBMC沉淀。
1.1.2.BV421标记的5-OP-RU/MR1-四聚体的制备
1.1.2.1.将6.8μl 0.5mg/ml的链霉亲和素-BV421稀释至10.2μl PBS中并充分混合。
1.1.2.2.每10分钟将1/10的链霉亲和素-BV421溶液(1.7μl)添加到18μl MR1-5-OP-RU溶液(5μg)中,并用移液管混合,两步骤之间在室温下暗处培养。
1.1.2.3.将BV421标记的5-OP-RU/MR1溶液保持在4℃。
四聚体应滴定后使用;通常1:500稀释就足够了。
1.1.3.通过流式细胞术检测人MAIT细胞
可以通过流式细胞术检测人MAIT细胞,通过负载5-OP-RU的MR1四聚体或与CD161和TCR Vα7.2链抗体共染色。一般来说,MAIT细胞在外周血中占CD3+T细胞的0.1-10%。
1.1.3.1.以每100μl FACS染色缓冲液1x 10
1.1.3.2.将FACS抗体和/或5-OP-RU/MR1四聚体添加到样本中。
对于人MAIT细胞的检测,可以使用两种方法:
a)四聚体染色:使用BV421标记的人5-OP-RU MR1四聚体(1:500)和APC-H7缀合的抗人CD3(1:200)。
b)取代标记:PE缀合的抗人TCR Va7.2(1:200)、APC-H7缀合的抗人CD3(1:200)和FITC缀合的抗人CD161(1:200)。
1.1.3.3.4℃避光培养30分钟。
1.1.3.4.通过在4℃下以300x g离心5分钟,用FACS染色缓冲液洗涤细胞,并用300μl FACS染色缓冲液重悬。
1.1.3.5.通过流式细胞术分析MAIT细胞(见图11)。
1.2.
1.2.1.Vα7.2+细胞的磁珠分离。
1.2.1.1.收集PBMC,并用结合缓冲液洗涤细胞。弃去上清液,并用浓度为1x 10
1.2.1.2.以300x g离心5分钟,用MACS缓冲液清洗细胞一次。
1.2.1.3.用MACS缓冲液以10
1.2.1.4.用10倍体积的MACS缓冲液清洗细胞一次。以300x g离心5分钟。在1mlMACS缓冲液中重悬。
1.2.1.5.用1ml MACS缓冲液预洗MS柱并组装在磁体上。将细胞加到柱上并洗涤柱3次,每次用1ml MACS缓冲液。
1.2.1.6.从磁体上取下柱子,并在1ml MACS缓冲液中洗脱结合的细胞。
1.2.2.MAIT细胞的流式分选
1.2.2.1.收集磁力分离的细胞并以300x g离心5分钟。
1.2.2.2.用MACS缓冲液以10
1.2.2.3.用10倍体积的MACS缓冲液清洗细胞一次。以300x g离心5分钟。在2mlMACS缓冲液中重悬。
1.2.2.4.打开BD Prodigy分选仪并加载细胞样本。分选CD3+四聚体+细胞群(见图12)。
1.3.
1.3.1.收集分选的MAIT细胞并以300x g离心5分钟。
1.3.2.弃去上清液并在R10培养基中重悬至10
1.3.3.通过涡旋重悬Dynabeads人T活化剂CD3/CD28 30秒。
1.3.4.将所需体积的Dynabeads转移至试管中。
1.3.5.添加等体积的缓冲液并涡旋混合5秒。将试管放在磁体上1分钟,弃去上清液。
1.3.6.从磁体上取下试管,并将洗涤过的Dynabeads重新悬浮在R10介质中。
1.3.7.将所需体积的Dynabeads添加到细胞悬浮液中,在37℃培养箱的24孔板中加入100IU/ml IL-2,使得珠子与细胞之比达到1:1。
1.4.
1.4.1.在MAIT细胞激活48小时后进行逆转录病毒转导。
1.4.2.转导前一天,准备RetroNectin包被板。将15μg RetroNectin添加到1mlPBS中。
充分混合并添加到未经组织培养处理的24孔板的一个孔中。
1.4.3.用保鲜膜包裹板并在4℃冰箱中保存过夜。
1.4.4.在基因转移当天,从孔中除去未结合的RetroNectin。用2ml PBS洗涤两次。避免充分干燥。
1.4.5.在37℃水浴中解冻逆转录病毒上清液。将1ml病毒上清液转移至RetroNectin包被板的每个孔中。
1.4.6.用保鲜膜包裹板并在32℃下以1000x g离心2小时。
1.4.7.在离心过程中,收集激活的MAIT细胞。用含有100IU/ml IL-2的新鲜R10培养基重悬细胞至浓度1x 10
1.4.8.旋转结束后,弃去板上的上清液。每孔中加入1ml细胞悬液。
1.4.9.将板以500x g离心10分钟。
1.4.10.将板放回37℃培养箱中。
1.4.11.如有必要,重复转导步骤以提高转导效率。
注意:转导48小时后可通过流式细胞术检测转导效率。
1.5.
1.5.1.逆转录病毒转导两天后,收获细胞并通过血细胞计数器对细胞进行计数。
1.5.2.将1x 10
1.5.3.每三天将IL-2刷新至终浓度100IU/ml。
1.5.4.培养8-12天后即可收获CAR-MAIT细胞(见图13)。
注意:扩增的CAR-MAIT细胞可用于表型测试、功能测定或液氮冷冻。
结果
实施例1:靶向CD4的T细胞和靶向TCR Vbeta 7.1的T细胞的产生
参考图1A(1)和(2),图中示出了说明根据本发明的CAR构建体的两种不同实施方式中包含的功能元件。
在每个实施方案中,构建体的侧翼是上游和下游长末端重复序列(LTR)。5'启动子位于5'LTR下游,并且其可以是PGK启动子。启动子的3'端有SP,一种Igκ信号肽,用于引导融合蛋白到达T细胞外膜。SP的3'处设置有scFv区,其包括上游VL(可变轻链)序列、中央G4S序列和下游VH(可变重链)序列。在一个实施方案中(如图1A(1)所示),VL和VH序列可以是Hu5A8轻链可变区和重链可变区以结合CD4抗原。在其他实施方案中(如图1A(2)所示),VL和VH序列可以是3G5轻链可变区和重链可变区以结合TCR-Vb7.1。
scFv区的3'处有CD8a铰链和跨膜(TM)结构域,用于CAR展示和锚定。铰链和TM的3’端设有胞内结构域,包括CD28、4-1BB和CD3ζ链的信号结构域,用于触发胞内信号传导通路。P2A自切割肽位于ζ链的3'处和截短型EGFR(EGFRt)的5'处,用于跟踪并充当第一安全开关。第二个P2A自切割肽位于EGFRt的3'处和诱导型半胱天冬酶9(iC9)的5'处,充当第二安全开关。该构建体包括土拨鼠肝炎调节元件(WPRE)(参见图9和图10质粒),可增强表达,最后是末端3'LTR。
对人CD4具有免疫特异性的小鼠IgG抗体人源化5A8(Hu5A8)的DNA序列从CN103282385中找到。Hu5A8也称为TNX-355或艾巴利珠单抗(ibalizumab),在I期和II期临床试验中进行了广泛评估,用于通过阻断CD4分子的HIV结合位点来抑制HIV进入。作为对照,还合成了抗CD20单克隆抗体(Leu16)的VH链和VL链,并在临床前和临床CAR-T研究中评估了其功效。scFv片段被克隆到第三代CAR质粒的骨架中,具有CD8跨膜结构域、CD28胞内结构域、4-1BB胞内结构域和CD3ζ链。经过研究证明,第三代CAR的使用优于第一代和第二代CAR。tEGFR作为一种选择,其作为体内追踪标记物以及消融工程CAR-T细胞的第一安全开关的实用性得到了检验。因此,该残留的tEGFR序列通过T2A-核糖体跳跃序列与CAR序列连接。
CAR-T细胞有时可以在患者体内保留长达数十年,就像抗CD19和抗HIVCAR试验的情况一样。与B细胞再生障碍不同,长期CD4+T细胞再生障碍会危及生命。因此,有必要建立一种安全的方法,在肿瘤或病毒消除后或在CAR-T治疗过程中由于严重副作用而导致的紧急情况下,从患者体内移除本发明的CART4细胞。二聚化药物诱导的半胱天冬酶9(iC9)自杀开关基于人半胱天冬酶9与突变的人FK506结合蛋白(FKBP)的融合,可在小分子化学药物AP20187(称为半胱天冬酶诱导药物,CID)存在的情况下实现条件二聚化。iC9的使用已在半相合HSC移植的临床试验中被证明是安全有效的。因此,随后合成了包含CD4 CAR、tEGFR和iC9的基因片段(图1A.1)并插入逆转录病毒MSCV(鼠干细胞病毒)载体中(如图9所示;10,348bp)。此外,随后合成了包含TCR Vbeta7.1 CAR、tEGFR和iC9的基因片段(图1A.2)并插入逆转录病毒MSCV(鼠干细胞病毒)载体中(如图10所示;10,347bp)。
为了证实CAR和tEGFR的共表达,用Dynabeads人T活化剂激活人PBMC,并通过逆转录病毒转导图9和图10所示的质粒进行基因工程改造以表达CAR/tEGFR/iC9基因构建体。事实上,CAR和tEGFR的表达水平紧密相关,用小鼠scFv特异性抗小鼠IgG F(ab')2抗体结合EGFR特异性抗体进行表面染色后,可检测到双阳性细胞群(图1B)。随着T细胞的扩增,tEGFR+细胞群保持其比例在总细胞数的50%左右(图1C)。
为了评估体外iC9安全开关的效率,克隆了不含iC9基因的CART4变体(CART4 w/oiC9)作为对照。用CART4或不含iC9构建体的CART4转导的T细胞暴露于浓度不断增加的CIDAP20187(0.1nM至100nM)24小时。使用7AAD和膜联蛋白-V进行流式细胞术分析来了解细胞死亡情况。存活细胞群体中的tEGFR阳性百分比随着CID浓度的增加而下降。单次100nM剂量的CID使69.1%的tEGFR高细胞被消除(图1D)。与其他研究的观察结果一致,逃脱杀伤的细胞是表达低水平转基因的细胞,CID后tEGFR的平均荧光强度(MFI)降低了50%(图1E)。因此,无反应的T细胞表达的iC9不足以激活CID的功能。对于临床应用,CAR-T细胞可能需要在给药前进行分选,以获得足够的转基因表达。
实施例2:CART4 T细胞的体外功能验证
在CAR转导后四天内,与未转导的(NTD)和CART20对照相比,CD4+T细胞几乎完全耗尽,其中约45%的细胞仍呈CD4阳性(图2A)。这些数据表明,CART4细胞在T细胞扩增过程中具有强大的抗CD4活性。
针对自体原代健康供体PBMC建立共培养物。CFSE标记的自体PBMC与CD8+CART4细胞或CART20细胞进行共培养。在这两种情况下,CART4/20细胞都能对各自的靶细胞产生高水平的细胞毒性。在4小时共培养期间,在E:T比例3:1的条件下,PBMC中94%的CD4+细胞被CART4细胞裂解。然而,与NTD T细胞相比,CART4对CD20+细胞没有特异性的T细胞毒性(图2C)。
为了进一步评估CART4细胞的功能,发明人使用Jurkat细胞系和CEM-ss细胞系测试了CART4细胞的抗肿瘤功效。Jurkat和CEM-ss细胞系是最初从T细胞白血病或人T4淋巴细胞白血病患者的外周血中建立的T细胞系。这两种细胞系均表达CD4,而CEM-ss细胞系表达更高水平的CD4(图2D)。事实上,CART4细胞根据CD4表达水平靶向T肿瘤细胞系。经过短期培养,CART4细胞以5:1的E:T(效应物:靶标)比例成功消除了CEM-ss细胞。作为对照,还测试了CART4细胞对CD4-淋巴瘤细胞(一种不表达CD4的人类B细胞系,BCL)的活性(图2D)。流式细胞术分析表明,CART4细胞无法靶向BCL(图2E)。此外,通过细胞内细胞因子染色,与CD4+肿瘤细胞一起培养的CART4细胞表现出显着的IFN-γ和TNF-α反应(图2F)。因此,这些数据证明了CART4对CD4表达的强烈的剂量依赖性反应。当CART4细胞与CD4阴性细胞一起培养时,没有观察到杀伤作用。因此,这些结果表明CART4细胞消融对CD4具有特异性。
实施例3:CART4细胞特异性杀死CD4+T肿瘤细胞
为了检测CART4对患者样本的功能,将来自ATLL患者的PBMC解冻并进行表型分析。所有样本的CD4表达率为67.4%至97.7%。大多数CD4+细胞表达一条独特的T细胞受体(TCRVbeta)β链,表明T细胞白血病202-204的克隆发展(图3A)。通过流式细胞术分析量化,ATLL患者样本与CART4细胞共培养4小时后,CD4+恶性肿瘤会快速、明确地消融。所有ATLL共培养物均观察到约80%的消融,与之前显示的母细胞T细胞系的消融一致(图3B)。研究还使用来自六名CTCL患者的样本。同样观察到CART4细胞对新鲜解冻的原代CTCL细胞具有强大的细胞毒性,共培养4小时后,恶性T细胞减少约60%至80%(图3C)。因此,CART4细胞能够有效地消除直接从患者样本中分离出的侵袭性CD4+T恶性肿瘤。这些结果表明,CD4是CD4+T恶性肿瘤很有前景的治疗靶点。
实施例4:CART4细胞有效介导体内抗白血病作用
为了评价体内抗肿瘤活性,发明人使用表达Gaussia荧光素酶的CEM-ss细胞系开发了异种小鼠模型。首先使用单剂量(4x10
尽管复发的肿瘤细胞保留了CD4的表达,但与对照组相比,表达水平下降至约40%MFI(图4E)。然而,这种下调不足以损害CART4细胞消除复发肿瘤的能力(图4F)。这一结果表明,肿瘤复发的主要原因是CAR-T细胞缺乏持久性,而不是抗原逃逸。
实施例5:开发符合GMP标准的CAR-T细胞制造方法
为了评估CAR-T细胞的可扩展性并简化CAR-T细胞制造,使用透气静态细胞培养系统(G-Rex)建立了优化的标准操作程序,用于CAR-T细胞的制造(图5A)。G-Rex系统在板的底部包含一层硅胶膜。通过膜上的气体交换(包括O
有趣的是,G-Rex中产生的CAR-T细胞表现出对中央记忆表型的分化偏好。对记忆标记CD45RO和CD62L的评估显示,与传统培养瓶培养的细胞相比,CD45RO CD62L双阳性细胞的比例更高(77%7.1%vs 41%5.5%)(图5D)。CD45RO CD62L双阳性细胞是中央记忆T细胞,被认为是体内长期持续存在所必需的。因此,这种生物过程优化方法提高了细胞产量和具有中央记忆表型的比例,同时减少了技术人员干预的次数和CAR-T制造的成本。
实施例6:TCRVbeta7.1特异性CAR-T细胞的产生
T细胞恶性肿瘤通常由表达独特TCR的一种单克隆癌细胞发展而来。针对TCRβ(Vβ)链可变(V)区的多种抗体已以直接缀合的多色形式提供,可以评估25个Vβ家族中的22个,覆盖正常循环T细胞谱系的75%。因此,发明人认为TCR Vβ是CAR-T疗法针对T细胞恶性肿瘤的潜在靶点。为了开发靶向CAR-T(CARTVb7.1)细胞的TCR Vβ,发明人将杂交瘤细胞3G5的scFv区克隆到CAR构建体上,该杂交瘤细胞能产生针对人TCR Vβ7.1的单克隆抗体(来自牛津大学Andrew实验室的Margret Callam博士),如图1A(2)所示。CAR转导五天后,内源性TCR Vβ7.1+细胞群几乎完全耗尽,与CART20对照相比,约1.2%的细胞仍保持TCR Vβ7.1阳性(图6B)。这些数据表明,CAR-T细胞在T细胞扩增过程中具有针对TCR Vβ7.1的强大活性。
为了进一步评估CARTVb7.1细胞的功能,发明人使用从被诊断患有TCRVβ7.1阳性肿瘤的ATL患者身上分离的肿瘤细胞测试了抗肿瘤功效。事实上,通过流式细胞术分析量化结果,ATL患者样本与CARTVb7.1细胞共培养6小时后,CD4+恶性肿瘤会快速、明确地消融。所有ATL共培养物均观察到约60%的消融(图6C、D)。这些结果表明,TCR Vβ是T恶性肿瘤有前景的治疗靶点。
实施例7:CAR-MAIT细胞的开发
目前,大多数CAR-T疗法利用自体常规CD3+T细胞。然而,癌症患者的免疫细胞可能功能低下或数量很少。特别是,对T恶性肿瘤患者的PBMC进行扩增和基因改造是有风险的,因为可以用CAR改造肿瘤细胞。因此,需要开发一种可以制造第三方同种异体细胞的免疫疗法。在这里,发明人开发了一种两步法,通过将磁分离和流式细胞术分选相结合,从PBMC中分离出粘膜相关恒定T细胞(MAIT细胞)。第一步根据TCR Vα7.2表达进行分离后,MAIT细胞百分比从0.74%提高到33.3%(图7A、B)。下一步的流式分选可以进一步将MAIT纯度提高到95%。分选的细胞用Dynabeads人T活化剂CD3/CD28激活,并在细胞因子混合物(IL-2、IL-7和IL-15)存在下扩增。该扩增方法在12至14天内实现了约100倍的扩增(图7C)。在收获时,90.9%的扩增细胞保持了对MR1-5-OP-RU四聚体的特异性(图7D)。此外,扩增的MAIT细胞可以通过逆转录病毒转导成功地进行CAR基因改造(图7E)。CAR转导的MAIT(CAR-MAIT)细胞具有与传统CAR-T细胞相当的细胞毒性能力(图8)。
实施例8:CAR-MAIT细胞有效介导体内抗白血病作用
为了评价CAR-MAIT细胞的体内抗肿瘤功能,发明人使用单剂量(4x10
实施例9:人MAIT细胞的检测、分离、扩增和工程化改造
使用本文描述的方法,对人MAIT细胞进行检测、分离、扩增和工程化改造。
如图11所示,通过流式细胞术分析人MAIT细胞。
如图12所示,MAIT细胞经过流式分选。
然后激活MAIT细胞,并用表达CAR的载体转导以产生CAR-MAIT细胞,然后进行扩增,如图13所示。
实施例10:通过刺激PMBC扩增MAIT细胞
MAIT细胞是先天性T细胞的一个亚群,定义为CD3+TCRVa7.2+CD161+细胞,可识别MHC I类分子MR1。先前的研究表明,MAIT细胞可以在体外扩增,但需要同种异体饲养细胞的存在。然而,该方法的一个问题是难以大规模生产和质量控制。在本项研究中,发明人现已开发出一种非常新颖且有效的体外扩增MAIT细胞的方法,通过首先用负载抗原(5-OP-RU)的MR1四聚体珠或仅用5-OP-RU刺激PBMC,在多种细胞因子(IL-2、IL-7、IL-15、IL-12、IL-18和IL-23)组合存在下进行长达6天的体外培养。然后通过MACS或FACS分选分离MAIT细胞,随后通过抗CD3/CD28珠进一步扩增以用于基于CAR的治疗,如前面的实施例中所述。
材料和方法
1.PBMC分离
通过Ficoll-Hypaque密度梯度离心法从健康献血者的血沉棕黄层中分离出PBMC。将分离的PBMC等分试样冷冻并储存在液氮中备用。在开始实验之前,将冷冻的PBMC储存液解冻并在补充有10% FBS的RPMI培养基中于37℃下培养。
2.MR1/5-OP-RU复合珠的制备
通过使用M-280dynabeads和链霉亲和素(来自ThermoFisher)以及生物素化的MR1单体,制成MR1/5-OP-RU四聚体包被珠。负载5-OP-RU的MR1单体由Jim McCluskey博士(澳大利亚墨尔本大学)友情提供。将珠子与负载5-OP-RU的MR1单体(5ug/3x10
3.PBMC中MAIT细胞的富集
PBMC(每孔2x10
4.FACS分析PBMC中MAIT细胞频率
使用抗体在黑暗中对扩增的PBMC进行表面标记染色30分钟。FITC缀合的CD3(克隆BW264/56,Miltenyi)、PE缀合的Va7.2(克隆3C10,Biolegend)、APC缀合的CD161(克隆DX12,BD)以1:100的比例用于标记细胞。MAIT细胞定义为CD3+Va7.2+CD161+细胞。使用LIVE/DEAD
结果
图15显示了在PBMC中富集MAIT细胞的结果。PBMC通过(i)珠子与细胞比例为1:1的MR1/5-OP-RU复合珠,或(ii)10nM的5-OP-RU抗原进行刺激——在不同的细胞因子(IL-2、IL-7、IL-15、IL-12、IL-18和IL-23)存在下进行,持续6天,如表中所示。例如,条件1对应于仅IL-2,条件2对应于IL-7和IL-15,条件3对应于IL-2、IL-12和IL-18,等等。
MAIT细胞增加的倍数是通过将第6天的活MAIT(CD3+Va7.2+CD161+)细胞的频率除以第0天的MAIT细胞的原始频率来计算的。前五组用橙色标出(即条件1、3、11、12和13)。
可以看出,对于MR1/5-OP-RU复合珠(供体1),1、13、12、3和11的细胞因子组合给出了PBMC中MAIT细胞的最高增加倍数。对于MR1/5-OP-RU复合珠(供体2),12、13、1、11和3的细胞因子组合给出了PBMC中MAIT细胞的最高增加倍数。
可以看出,对于5-OP-RU(供体1),3、1、12、13和11的细胞因子组合给出了PBMC中MAIT细胞的最高增加倍数。对于5-OP-RU(供体2),8、13、12、11和3的细胞因子组合给出了PBMC中MAIT细胞的最高增加倍数。
根据这些数据,很明显,不同的细胞因子和细胞因子的组合(IL-2、IL-7、IL-15、IL-12、IL-18和IL-23)会产生不同程度的刺激,从而提高PBMC中MAIT细胞的富集。总体而言,IL-12、IL-18和IL-23的组合使PBMC中MAIT细胞的增加倍数最高。
讨论
目前,与B细胞恶性肿瘤相比,T细胞淋巴瘤尚无成熟的治疗方法,唯一潜在的治疗方案是同种异体造血干细胞移植(HSCT),但是这种方法本身具有显着的治疗相关死亡率。由于大多数(>95%)T细胞淋巴瘤源自表达特定T细胞受体(TCR)基因(即克隆TCR-Vb链)和泛T辅助细胞标记CD4的显性T细胞克隆,因此针对这些标记物的单克隆抗体已被用于治疗T细胞淋巴瘤,在小型临床试验中,其中一些抗体导致了部分淋巴瘤的消退(d’Amore etal.,2010;Hagberg et al.,2005;Kim et al.,2007)。
尽管CAR-T通过靶向泛B细胞标志物CD19是一种非常有效的B细胞恶性肿瘤治疗方法,但这种方法在应用于T细胞淋巴瘤的治疗时遇到了重大障碍。首先,与B细胞耗竭不同,持续性T细胞再生障碍,特别是CD4+T细胞耗竭,会导致严重的毒性,例如慢性HIV感染期间观察到的机会性感染。其次,T细胞淋巴瘤相关的T细胞功能受损和由T细胞肿瘤显性生长引起的正常T细胞计数降低,不能用于产生自体CAR-T细胞,因此需要同种异体CAR-T细胞来治疗T细胞细胞淋巴瘤。最后,大多数T细胞淋巴瘤是与淋巴结和皮肤组织相关的实体瘤,由于组织浸润能力较低以及肿瘤微环境恶劣,传统CAR-T难以治疗。
为了解决这些问题,发明人设计了一种靶向CD4抗原(CART4)的CAR,其含有tEGFR和iC9作为安全开关,以在根除肿瘤细胞后选择性地消除CART4转导的T细胞,从而使自体造血干细胞或同种异体HSCT恢复正常的CD4+T细胞。CD4+T细胞的转运耗竭已被证明在抗CD4抗体治疗自身免疫性疾病中是安全且可耐受的(Hagberg et al.,2005;Kim et al.,2007)。CART4转导的人类T细胞能够在体外杀死从ATLL或CTCL患者分离的CD4+T细胞淋巴瘤细胞系,并在小鼠异种移植模型中抑制体内肿瘤生长。更重要的是,这些CART4+T细胞与tEGFR(通过抗EGFR抗体检测)和iC9(通过CID药物诱导CART4+T细胞体外和体内凋亡而确定)共表达CAR。tEGFR的表达可用于监测CART4+T细胞增殖或体内用抗EGFR抗体消除CART4+T细胞。
一般来说,正常T细胞由高度多样化的TCR库组成,以维持针对病原体感染的细胞免疫。TCR由含有N端可变区和C端恒定区的a和b链的异二聚体组成。TCR-Vb区(链)比TCR-Va更具多态性,通常用于分析免疫反应或T细胞恶性肿瘤的克隆性。目前,TCR-Vb链家族有22种特异性mAbs,覆盖了75%的TCR库。由于大多数T细胞淋巴瘤都来自于表达相同TCR Vb链的单个T细胞克隆,因此针对肿瘤克隆定义的TCR-Vb链的CART,同时保留其余正常T细胞库将是最大限度减少机会性感染的理想方法,并且在输血后无需移除CART细胞。
作为抗TCR-Vb的免疫疗法的概念验证,发明人设计了特异性靶向TCR-Vb 7.1链(CARTVb7.1)的CAR,结果表明CARTVb7.1转导的T细胞能够有效消除从ATL患者中分离出的TCR-Vb7.1阳性肿瘤细胞,表明抗TCR-Vb CAR可以为T细胞淋巴瘤提供一种替代的免疫疗法。
最后,发明人研究了MAIT细胞是否可以作为基于CAR的疗法的效应细胞,因为MAIT细胞相对于传统T细胞具有若干优势,包括:
(1)由于在哺乳动物进化过程中表达高度保守的恒定TCR,因此同种异体反应性低(即诱导移植物抗宿主病、GVHD);
(2)调节功能,包括抑制小鼠模型中的GVHD;
(3)激活诱导GrB、穿孔素和GrA的杀伤活性;和
(4)组织归巢,例如分布于肠道粘膜、皮肤和肺部。
如本文所述,CAR转导的MAIT细胞(CAR-MAIT)在体外和体内表现出至少与常规CAR-T细胞相当的抗肿瘤活性。总之,发明人开发了一种基于CAR-MAIT的新型免疫疗法,可有效治疗T细胞恶性肿瘤,通过用可切换的CAR-T靶向泛T细胞标记物CD4,减少细胞因子释放综合征的靶向/脱瘤毒性或特异性TCR-Vb链(这是恶性T细胞所特有的),从而避免整体免疫抑制。更重要的是,CAR-MAIT细胞可能有潜力开发出治疗T细胞淋巴瘤所需的基于同种异体CAR的疗法。如果持续生产,即大规模体外扩增,它们可用于现成的开发。发明人相信,CAR-MAIT不仅可以为T细胞恶性肿瘤提供有效的治疗新方法,而且还可以为其他非免疫细胞类型的肿瘤提供有效的治疗新方法。
结论
基于嵌合抗原受体(CAR)的T细胞疗法通过靶向泛B细胞特异性抗原,在治疗B细胞恶性肿瘤方面取得了巨大成功。然而,针对T细胞淋巴瘤的类似策略迄今尚未实现,这主要是由于与B细胞恶性肿瘤相比,T淋巴瘤中整体T细胞耗竭和正常T细胞功能障碍/频率低所带来的潜在严重毒性。为了克服这些限制,发明人设计了一种种新型CAR构建体,其对泛T细胞标记物(CD4)或TCR-Vb同型链具有特异性,其中包含两个安全开关:截短的表皮生长因子受体(tEGFR)和诱导型半胱天冬酶9(iC9)。发明人研究了同种异体反应性低的粘膜相关恒定T(MAIT)细胞在用CAR构建体转导后是否会表现出与传统T细胞类似的抗肿瘤杀伤活性。
令人惊讶的是,CAR转导的T细胞不仅对从患者体内分离出来的CD4+T淋巴瘤细胞或TCR-Vb特异性T白血病克隆表现出特异性杀伤作用,而且在体外和体内用诱导剂处理后也会被消除。此外,本发明人还首次证明,CAR-MAIT细胞能够在体外和体内与常规T细胞一样有效地抑制肿瘤生长。该研究为T细胞淋巴瘤的治疗提供了新的策略。
因此,本发明的粘膜相关恒定T(MAIT)细胞是一类免疫细胞,这类免疫细胞参与广泛的感染性和非感染性疾病,并且它们对由组织相容性复合物(MHC)I类蛋白MR1呈递的微生物核黄素衍生物抗原具有不寻常的特异性,它们被开发成一种新型免疫疗法用于治疗癌症患者,通过用嵌合抗原受体(CAR)对MAIT细胞进行基因改造,使它们能够特异性识别和攻击T淋巴瘤。
参考文献
d’Amore,F.,Radford,J.,Relander,T.,Jerkeman,M.,Tilly,H.,Osterborg,A.,Morschhauser,F.,Gramatzki,M.,Dreyling,M.,Bang,B.,&Hagberg,H.(2010).Phase IItrial of zanolimumab(HuMax-CD4)in relapsed or refractory non-cutaneousperipheral T cell lymphoma.British Journal of Haematology,150(5),565–573.https://onlinelibrary.wiley.com/doi/full/10.1111/j.1365-2141.2010.08298.x
Hagberg,H.,Pettersson,M.,Bjerner,T.,&Enblad,G.(2005).Treatment of apatient with a nodal peripheral T-cell lymphoma(Angioimmunoblastic T-celllymphoma)with a human monoclonal antibody against the CD4 antigen(HuMax-CD4).Medical Oncology(Northwood,London,England),22(2),191–194.https://link.springer.com/article/10.1385/MO:22:2:191
Katsuya,H.,Ishitsuka,K.,Utsunomiya,A.,Hanada,S.,Eto,T.,Moriuchi,Y.,Saburi,Y.,Miyahara,M.,Sueoka,E.,Uike,N.,Yoshida,S.,Yamashita,K.,Tsukasaki,K.,Suzushima,H.,Ohno,Y.,Matsuoka,H.,Jo,T.,Amano,M.,Hino,R.,…Project,A.-P.I.(2015).Treatment and survival among 1594patientswith ATL.Blood,126(24),2570–2577.https://ashpublications.org/blood/article/126/24/2570/34701/Treatment-and-survival-among-1594-patients-with
Kim,Y.H.,Duvic,M.,Obitz,E.,Gniadecki,R.,Iversen,L.,Osterborg,A.,Whittaker,S.,Illidge,T.M.,Schwarz,T.,Kaufmann,R.,Cooper,K.,Knudsen,K.M.,Lisby,S.,Baadsgaard,O.,&Knox,S.J.(2007).Clinical efficacy of zanolimumab(HuMax-CD4):two phase 2studies in refractory cutaneous T-cell lymphoma.Blood,109(11),4655–4662.https://ashpublications.org/blood/article/109/11/4655/23083/Clinical-efficacy-of-zanolimumab-HuMaxCD4-two
Park,J.H.,Rivière,I.,Gonen,M.,Wang,X.,Sénéchal,B.,Curran,K.J.,Sauter,C.,Wang,Y.,Santomasso,B.,Mead,E.,Roshal,M.,Maslak,P.,Davila,M.,Brentjens,R.J.,&Sadelain,M.(2018).Long-Term Follow-up of CD19 CAR Therapy in AcuteLymphoblastic Leukemia.New England Journal of Medicine.https://doi.org/10.1056/nejmoa1709919
Rowan,A.G.,Suemori,K.,Fujiwara,H.,Yasukawa,M.,Tanaka,Y.,Taylor,G.P.,&Bangham,C.R.M.(2014).Cytotoxic T lymphocyte lysis of HTLV-1infected cells islimited by weak HBZ protein expression,but non-specifically enhanced oninduction of Tax expression.Retrovirology,11(1),112–116.https://retrovirology.biomedcentral.com/articles/10.1186/s12977-014-0116-6
附文
1.一种表达嵌合抗原受体(CAR)的粘膜相关恒定T(MAIT)细胞。
2.根据第1条所述的MAIT细胞,其中所述CAR-MAIT细胞表达靶向T细胞上的CD4抗原的CAR。
3.根据第2条所述的MAIT细胞,其中所述CAR对包含基本上如SEQ ID No:1所示的氨基酸或其变体或片段的CD4抗原具有特异性。
4.根据前述任一条所述的MAIT细胞,其中所述CAR-MAIT细胞表达靶向T细胞上的T细胞受体(TCR)β链可变区(Vbeta)的CAR,优选如表1所示的任意一个Vbeta区。
5.根据第4条所述的MAIT细胞,其中所述CAR靶向T细胞上的多个T细胞受体(TCR)β链可变区(Vbeta),优选地其中所述多个Vbeta区选自表1所示的一组Vbeta区,任选地其中所述多个TCR Vbeta区是相同或不同的Vbeta区。
6.根据第4条或第5条所述的MAIT细胞,其中所述CAR靶向选自下组的T细胞上的一个或多个TCR Vbeta区:Vb 1、Vb 2、Vb 3、Vb 5.1,Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb17和Vb20。
7.根据第4至6条中任一条所述的MAIT细胞,其中所述CAR对包含基本上如SEQIDNo:2所示的氨基酸或其变体或片段的TCR Vbeta区具有特异性。
8.根据前述任一条所述的MAIT细胞,其中所述CAR-MAIT细胞包含一种或多种编码序列,其允许所述CAR-MAIT细胞被可控地或诱导性地消除。
9.根据第8条所述的MAIT细胞,其中所述一种或多种编码序列编码表皮生长因子受体(EGFR)或截短的表皮生长因子受体(tEGFR)。
10.根据第8条或第9条所述的MAIT细胞,其中所述一种或多种编码序列编码诱导型半胱天冬酶9(iC9)。
11.根据前述任一条所述的MAIT细胞,其中通过磁激活细胞分选(MACS)和/或荧光激活细胞分选(FACS)从人外周血单核细胞(PBMC)分离所述MAIT细胞,更优选MACS和FACS二者。
12.一种核酸构建体,其包含可操作地连接至第一编码序列的启动子,所述第一编码序列编码抗CD4嵌合抗原受体(CAR)或抗T细胞受体(TCR)Vbeta CAR。
13.根据第12条所述的构建体,其中所述启动子是PGK启动子,任选地所述启动子包含基本上如SEQ ID No:3所示的核苷酸序列或其片段或变体。
14.根据第12条或第13条所述的构建体,其中所述第一编码序列编码抗CD4嵌合抗原受体(CAR),任选地其中所述CAR对包含基本上如SEQ ID No:1所示的氨基酸序列或其变体或片段的CD4抗原具有特异性。
15.根据第14条所述的构建体,其中:
(i)所述第一编码序列包含编码基本上如SEQ ID No:6所示的氨基酸序列的核苷酸序列或其片段或变体;
(ii)所述第一编码序列包含基本上如SEQ ID No:7所示的核苷酸序列或其片段或变体;
(iii)所述第一编码序列包含编码基本上如SEQ ID No:8所示的氨基酸序列的核苷酸序列或其片段或变体;和/或
(iv)所述第一编码序列包含基本上如SEQ ID No:9所示的核苷酸序列或其片段或变体。
16.根据第12条或第13条所述的构建体,其中所述第一编码序列编码抗T细胞受体(TCR)Vbeta区CAR,任选地为表1中列出的任何Vbeta区。
17.根据第16条所述的构建体,其中所述第一编码序列编码多个T细胞受体(TCR)β链可变区(Vbeta)CAR,优选地其中所述多个Vbeta区选自表1所示的一组Vbeta区。
18.根据第16条或第17条所述的构建体,其中所述构建体包含编码至少一种CAR的编码序列,所述CAR靶向T细胞上的一个或多个选自以下的TCR Vbeta区:Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20;任选地其中所述构建体包含编码至少一种CAR的编码序列,所述CAR靶向T细胞上的至少两个或三个选自以下的TCR Vbeta区:Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb 8、Vb 12、Vb 13.1、Vb 17和Vb 20。
19.根据第16至18条中任一条所述的构建体,其中所述CAR对包含基本上如SEQ IDNo:2所示的氨基酸序列或其变体或其片段的TCR Vbeta区(优选TCR-Vbeta 7.1链)具有特异性。
20.根据第16至19条中任一条所述的构建体,其中:
(i)所述第一编码序列包含编码基本上如SEQ ID No:12所示的氨基酸序列的核苷酸序列或其片段或变体;
(ii)所述第一编码序列包含基本上如SEQ ID No:13所示的核苷酸序列或其片段或变体;
(iii)所述第一编码序列包含编码基本上如SEQ ID No:34所示的氨基酸序列的核苷酸序列或其片段或变体;和/或
(iv)所述第一编码序列包含基本上如SEQ ID No:35所示的核苷酸序列或其片段或变体。
21.根据第12至20条中任一条所述的构建体,其中所述构建体包含编码CD8a铰链和跨膜(TM)结构域的核苷酸序列,任选地其中所述构建体包含编码基本上如SEQ ID No:14所示的氨基酸序列的核苷酸序列或其片段或变体,和/或其中所述构建体包含基本上如SEQIDNo:15所示的核苷酸序列或其片段或变体。
22.根据第12至21条中任一条所述的构建体,其中所述构建体包含编码胞内结构域的核苷酸序列,所述胞内结构域包含CD28的信号传导结构域、4-1BB的信号传导结构域和/或CD3ζ链,其中更优选CD28的信号结构域、4-1BB的信号结构域和CD3ζ链。
23.根据第22条所述的构建体,其中:
(i)所述构建体包含编码基本上如SEQ ID No:16所示的氨基酸序列的核苷酸序列或其片段或变体;
(ii)所述构建体包含基本上如SEQ ID No:17所示的核苷酸序列或其片段或变体;
(iii)所述构建体包含编码基本上如SEQ ID No:18所示的氨基酸序列的核苷酸序列或其片段或变体;
(iv)所述构建体包含基本上如SEQ ID No:19所示的核苷酸序列或其片段或变体;
(v)所述构建体包含编码基本上如SEQ ID No:20所示的氨基酸序列的核苷酸序列或其片段或变体;和/或
(vi)所述构建体包含基本上如SEQ ID No:21所示的核苷酸序列或其片段或变体。
24.根据第12至23条中任一条所述的构建体,其中所述核酸构建体包含第二编码序列,所述第二编码序列编码至少一种自杀蛋白,更优选至少两种自杀蛋白。
25.根据第24条所述的构建体,其中所述第二编码序列编码:(i)表皮生长因子受体(EGFR)或截短的表皮生长因子受体(tEGFR);和/或(ii)诱导型半胱天冬酶9(iC9)。
26.根据第25条所述的构建体,其中:
(i)所述构建体包含编码基本上如SEQ ID No:22所示的氨基酸序列的核苷酸序列或其片段或变体,任选地其中所述构建体包含基本上如SEQ ID No:23所示的核苷酸序列或其片段或变体;和/或
(ii)所述构建体包含编码基本上如SEQ ID No:24中所示的氨基酸序列的核苷酸序列或其片段或变体,任选地其中所述构建体包含基本上如SEQ ID No:25所示的核苷酸序列或其片段或变体。
27.根据第12至26条中任一条所述的构建体,其中所述构建体包含编码基本上如SEQ ID No:29所示的氨基酸序列的核苷酸序列或其片段或变体,任选地其中所述构建体包含基本上如SEQ ID No:30所示的核苷酸序列或其片段或变体。
28.根据第12至26条中任一条所述的构建体,其中所述构建体包含编码基本上如SEQ ID No:31所示的氨基酸序列的核苷酸序列或其片段或变体,任选地其中所述构建体包含基本上如SEQ ID No:32所示的核苷酸序列或其片段或变体。
29.一种表达载体,其编码根据第12至28条中任一条所述的核酸构建体,任选地其中所述载体包含基本上如SEQ ID No:33或SEQ ID No:36所示的核酸序列或其片段或变体。
30.一种分离MAIT细胞的方法,所述方法包括:
(i)提供外周血单核细胞(PBMC);和
(ii)对PBMC进行磁激活细胞分选(MACS)和/或荧光激活细胞分选(FACS)以从中分离MAIT细胞。
31.一种产生CAR-MAIT细胞的方法,所述方法包括:
(i)提供外周血单核细胞(PBMC);
(ii)对PBMC进行MACS和/或FACS以从中分离MAIT细胞;
(iii)激活分离的MAIT细胞,任选地通过使它们与抗CD3和/或抗CD28抗体接触;和
(iv)用编码CAR的核酸转导激活的MAIT细胞,从而产生CAR-MAIT细胞。
32.根据第30条或第31条所述的方法,其中所述方法包括对PBMC进行MACS和FACS以从中分离MAIT细胞,任选地其中对PBMC进行MACS,然后进行FACS。
33.根据第30至32条中任一条所述的方法,其中所述分离的MAIT细胞用抗CD3抗体和抗CD28抗体激活。
34.根据第31至33条中任一条所述的方法,其中步骤(iv)包括用编码CAR的核酸通过病毒或逆转录病毒转导MAIT细胞,优选地其中所述核酸编码CAR,所述CAR靶向T细胞上的(i)CD4抗原,或(ii)至少一个或多个TCR Vbeta区,优选表1中所示的一个或多个TCR Vbeta区,或选自以下的T细胞上的一个或多个TCR Vbeta区:Vb 1、Vb 2、Vb 3、Vb 5.1、Vb 7.1、Vb8、Vb 12、Vb 13.1、Vb 17和Vb 20。
35.根据第30至43条中任一条所述的方法,其中所述MAIT细胞用根据第12至28条中任一条所述的核酸构建体或根据第29条所述的表达载体来转导。
36.根据第30至35条中任一条所述的方法,其中所述方法包括在步骤(iv)之后的后续步骤中扩增CAR-MAIT细胞。
37.一种通过根据第30至36条中任一条所述的方法获得或可获得的CAR-MAIT细胞。
38.一种药物组合物,其包含根据第1至11条中任一条或第37条所述的MAIT细胞和药学上可接受的赋形剂。
39.根据第1至11条中任一条或第37条所述的MAIT细胞或根据第38条所述的药物组合物用于治疗。
40.根据第1至11条中任一条或第37条所述的MAIT细胞或根据第38条所述的药物组合物用于(i)免疫疗法;(ii)治疗、预防或改善癌症;(ii)治疗、预防或改善微生物感染;或(iv)治疗、预防或改善自身免疫性疾病。
41.根据第1至11条中任一条或第37条所述的MAIT细胞或根据第38条所述的药物组合物用于根据第39条或第40条所述的用途,其中用于治疗、预防或改善T细胞恶性肿瘤,任选实体瘤或液态肿瘤。
42.根据第1至11条中任一条或第37条所述的MAIT细胞或根据第38条所述的药物组合物用于根据第41条所述的用途,其中所述T细胞恶性肿瘤是外周T细胞淋巴瘤(PTCL)或皮肤T细胞淋巴瘤(CTCL)。
43.根据第1至11条中任一条或第37条所述的MAIT细胞或根据第38条所述的药物组合物用于根据第41条所述的用途,其中:
(i)所述PTCL是选自下组的PTCL亚型:成人T细胞急性淋巴细胞性淋巴瘤或白血病(ATL)、肠病相关淋巴瘤、肝脾淋巴瘤、皮下脂膜炎样淋巴瘤(SPTCL)、前体T细胞急性淋巴细胞性淋巴瘤或白血病和血管免疫母细胞性T细胞淋巴瘤(AITL);
(ii)所述CTCL是选自下组的CTCL亚型:蕈样肉芽肿(MF)、塞扎里综合征(SS)和CD4+小中型多形性T细胞淋巴增殖性疾病。
44.根据第1至11条中任一条或第37条所述的MAIT细胞或根据第38条所述的药物组合物用于根据第40至43条中任一条所述的用途,其中用于治疗、预防或改善病毒感染(例如HIV、HBV、HTLV、EBV、HPV)、细菌感染(例如TB)或真菌感染,或者用于治疗、预防或改善自身免疫性疾病,例如系统性红斑狼疮、类风湿性关节炎或重症肌无力。
45.根据第1至11条中任一条或第37条所述的MAIT细胞或根据第38条所述的药物组合物用于根据第40至44条中任一条所述的用途,其中所述用途包括触发编码自杀蛋白的序列,任选地其中所述方法包括向受试者施用抗EGFR抗体和/或半胱天冬酶诱导药物(CID)。
46.一种制备根据第38条所述的药物组合物的方法,所述方法包括将治疗有效量的根据第1至11条中任一条或第37条所述的MAIT细胞与药学上可接受的赋形剂组合。
序列表
<110>帝国理工学院有限公司
<120>嵌合抗原受体T细胞
<130>115551PCT1
<150>2105684.1
<151>2021-04-21
<160>36
<170>PatentIn version 3.5
<210>1
<211>458
<212>PRT
<213>Homo sapiens
<400>1
Met Asn Arg Gly Val Pro Phe Arg His Leu Leu Leu Val Leu Gln Leu
1 5 1015
Ala Leu Leu Pro Ala Ala Thr Gln Gly Lys Lys Val Val Leu Gly Lys
202530
Lys Gly Asp Thr Val Glu Leu Thr Cys Thr Ala Ser Gln Lys Lys Ser
354045
Ile Gln Phe His Trp Lys Asn Ser Asn Gln Ile Lys Ile Leu Gly Asn
505560
Gln Gly Ser Phe Leu Thr Lys Gly Pro Ser Lys Leu Asn Asp Arg Ala
65707580
Asp Ser Arg Arg Ser Leu Trp Asp Gln Gly Asn Phe Pro Leu Ile Ile
859095
Lys Asn Leu Lys Ile Glu Asp Ser Asp Thr Tyr Ile Cys Glu Val Glu
100 105 110
Asp Gln Lys Glu Glu Val Gln Leu Leu Val Phe Gly Leu Thr Ala Asn
115 120 125
Ser Asp Thr His Leu Leu Gln Gly Gln Ser Leu Thr Leu Thr Leu Glu
130 135 140
Ser Pro Pro Gly Ser Ser Pro Ser Val Gln Cys Arg Ser Pro Arg Gly
145 150 155 160
Lys Asn Ile Gln Gly Gly Lys Thr Leu Ser Val Ser Gln Leu Glu Leu
165 170 175
Gln Asp Ser Gly Thr Trp Thr Cys Thr Val Leu Gln Asn Gln Lys Lys
180 185 190
Val Glu Phe Lys Ile Asp Ile Val Val Leu Ala Phe Gln Lys Ala Ser
195 200 205
Ser Ile Val Tyr Lys Lys Glu Gly Glu Gln Val Glu Phe Ser Phe Pro
210 215 220
Leu Ala Phe Thr Val Glu Lys Leu Thr Gly Ser Gly Glu Leu Trp Trp
225 230 235 240
Gln Ala Glu Arg Ala Ser Ser Ser Lys Ser Trp Ile Thr Phe Asp Leu
245 250 255
Lys Asn Lys Glu Val Ser Val Lys Arg Val Thr Gln Asp Pro Lys Leu
260 265 270
Gln Met Gly Lys Lys Leu Pro Leu His Leu Thr Leu Pro Gln Ala Leu
275 280 285
Pro Gln Tyr Ala Gly Ser Gly Asn Leu Thr Leu Ala Leu Glu Ala Lys
290 295 300
Thr Gly Lys Leu His Gln Glu Val Asn Leu Val Val Met Arg Ala Thr
305 310 315 320
Gln Leu Gln Lys Asn Leu Thr Cys Glu Val Trp Gly Pro Thr Ser Pro
325 330 335
Lys Leu Met Leu Ser Leu Lys Leu Glu Asn Lys Glu Ala Lys Val Ser
340 345 350
Lys Arg Glu Lys Ala Val Trp Val Leu Asn Pro Glu Ala Gly Met Trp
355 360 365
Gln Cys Leu Leu Ser Asp Ser Gly Gln Val Leu Leu Glu Ser Asn Ile
370 375 380
Lys Val Leu Pro Thr Trp Ser Thr Pro Val Gln Pro Met Ala Leu Ile
385 390 395 400
Val Leu Gly Gly Val Ala Gly Leu Leu Leu Phe Ile Gly Leu Gly Ile
405 410 415
Phe Phe Cys Val Arg Cys Arg His Arg Arg Arg Gln Ala Glu Arg Met
420 425 430
Ser Gln Ile Lys Arg Leu Leu Ser Glu Lys Lys Thr Cys Gln Cys Pro
435 440 445
His Arg Phe Gln Lys Thr Cys Ser Pro Ile
450 455
<210>2
<211>114
<212>PRT
<213>Homo sapiens
<400>2
Met Gly Cys Arg Leu Leu Cys Cys Ala Val Leu Cys Leu Leu Gly Ala
1 5 1015
Val Pro Ile Asp Thr Glu Val Thr Gln Thr Pro Lys His Leu Val Met
202530
Gly Met Thr Asn Lys Lys Ser Leu Lys Cys Glu Gln His Met Gly His
354045
Arg Ala Met Tyr Trp Tyr Lys Gln Lys Ala Lys Lys Pro Pro Glu Leu
505560
Met Phe Val Tyr Ser Tyr Glu Lys Leu Ser Ile Asn Glu Ser Val Pro
65707580
Ser Arg Phe Ser Pro Glu Cys Pro Asn Ser Ser Leu Leu Asn Leu His
859095
Leu His Ala Leu Gln Pro Glu Asp Ser Ala Leu Tyr Leu Cys Ala Ser
100 105 110
Ser Gln
<210>3
<211>501
<212>DNA
<213>Artificial Sequence
<220>
<223>PGK promoter
<400>3
gggtagggga ggcgcttttc ccaaggcagt ctggagcatg cgctttagca gccccgctgg60
gcacttggcg ctacacaagt ggcctctggc ctcgcacaca ttccacatcc accggtaggc 120
gccaaccggc tccgttcttt ggtggcccct tcgcgccacc ttctactcct cccctagtca 180
ggaagttccc ccccgccccg cagctcgcgt cgtgcaggac gtgacaaatg gaagtagcac 240
gtctcactag tctcgtgcag atggacagca ccgctgagca atggaagcgg gtaggccttt 300
ggggcagcgg ccaatagcag ctttgctcct tcgctttctg ggctcagagg ctgggaaggg 360
gtgggtccgg gggcgggctc aggggcgggc tcaggggcgg ggcgggcgcc cgaaggtcct 420
ccggaggccc ggcattctgc acgcttcaaa agcgcacgtc tgccgcgctg ttctcctctt 480
cctcattctc cgggcctttc g 501
<210>4
<211>21
<212>PRT
<213>Artificial Sequence
<220>
<223>Signalling peptide
<400>4
Met Glu Thr Asp Thr Leu Leu Leu Trp Val Leu Leu Leu Trp Val Pro
1 5 1015
Gly Ser Thr Gly Asp
20
<210>5
<211>63
<212>DNA
<213>Artificial Sequence
<220>
<223>Signalling peptide
<400>5
atggagacag acacactcct gctatgggtg ctgctgctct gggttccagg ttccacaggt60
gac63
<210>6
<211>112
<212>PRT
<213>Artificial Sequence
<220>
<223>First coding sequence (VL for CD4)
<400>6
Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu Ala Val Ser Leu Gly
1 5 1015
Glu Arg Val Thr Met Asn Cys Lys Ser Ser Gln Ser Leu Leu Tyr Ser
202530
Thr Asn Gln Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
354045
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
505560
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65707580
Ile Ser Ser Val Gln Ala Glu Asp Val Ala Val Tyr Tyr Cys Gln Gln
859095
Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210>7
<211>336
<212>DNA
<213>Artificial Sequence
<220>
<223>First coding sequence (VL for CD4)
<400>7
gacattgtga tgactcagag ccccgacagc ctggccgtct cactgggcga aagggtgacc60
atgaattgta aatcttctca gagcctgctg tacagtacaa accagaaaaa ttacctggcc 120
tggtatcagc agaaacccgg ccagagccct aagctgctga tctattgggc aagtacccga 180
gagtcaggag tgccagacag attctccggg tctggaagtg gcacagactt caccctgaca 240
attagctccg tgcaggccga ggacgtggct gtctactatt gccagcagta ctatagctac 300
cgaactttcg gcgggggaac caaactggaa atcaag 336
<210>8
<211>122
<212>PRT
<213>Artificial Sequence
<220>
<223>First coding sequence (VH for CD4)
<400>8
Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Val Val Lys Pro Gly Ala
1 5 1015
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
202530
Val Ile His Trp Val Arg Gln Lys Pro Gly Gln Gly Leu Asp Trp Ile
354045
Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Asp Tyr Asp Glu Lys Phe
505560
Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser Thr Ser Thr Ala Tyr
65707580
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Arg Glu Lys Asp Asn Tyr Ala Thr Gly Ala Trp Phe Ala Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210>9
<211>366
<212>DNA
<213>Artificial Sequence
<220>
<223>First coding sequence (VH for CD4)
<400>9
caggtgcagc tgcagcagtc cggaccagag gtggtcaaac ccggcgctag cgtcaaaatg60
tcctgtaagg catctggcta cactttcacc tcttatgtga ttcactgggt cagacagaag 120
cctgggcagg gactggactg gatcgggtac attaacccat ataatgatgg aactgactac 180
gatgaaaagt ttaaaggcaa ggccacactg acttccgaca cctcaacaag cactgcttat 240
atggagctgt ctagtctgag gtctgaagac acagcagtgt actattgcgc ccgcgagaag 300
gataactacg ccactggcgc ttggtttgca tattggggcc aggggaccct ggtgacagtc 360
tcatcc366
<210>10
<211>15
<212>PRT
<213>Artificial Sequence
<220>
<223>G4S linker sequence
<400>10
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 1015
<210>11
<211>44
<212>DNA
<213>Artificial Sequence
<220>
<223>G4S linker sequence
<400>11
gaggaggagg cagtggcgga ggagggtcag gaggaggagg aagc 44
<210>12
<211>119
<212>PRT
<213>Artificial Sequence
<220>
<223>First coding sequence (VL for TCR V-beta)
<400>12
Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 1015
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Arg Tyr
202530
Trp Ile Thr Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
354045
Gly Asp Ile Tyr Pro Gly Ser Gly Phe Thr Lys Tyr Asn Glu Lys Phe
505560
Lys Ser Lys Ala Thr Leu Thr Val Asp Thr Ser Ser Ser Thr Ala Tyr
65707580
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
859095
Ala Arg Glu Gly Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Thr Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210>13
<211>357
<212>DNA
<213>Artificial Sequence
<220>
<223>First coding sequence (VL for TCR V-beta)
<400>13
caagttcagc tgcaacagcc tggcgccgag cttgtgaaac ctggcgcctc tgtgaagatg60
agctgcaagg cctccggcta caccttcacc agatactgga tcacctgggt caagcagagg 120
cctggacagg gactcgagtg gatcggcgat atctatcctg gctccggctt caccaagtac 180
aacgagaagt tcaagagcaa ggccacactg accgtggaca ccagcagcag cacagcctac 240
atgcagctgt ctagcctgac cagcgaggac agcgccgtgt actactgtgc tagagaaggc 300
ggcaactact ggtacttcga cgtgtggggc accggcacca cagtgacagt tagttct357
<210>14
<211>83
<212>PRT
<213>Artificial Sequence
<220>
<223>CD8a hinge and transmembrane domain
<400>14
Phe Val Pro Val Phe Leu Pro Ala Lys Pro Thr Thr Thr Pro Ala Pro
1 5 1015
Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu
202530
Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg
354045
Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly
505560
Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Asn
65707580
His Arg Asn
<210>15
<211>249
<212>DNA
<213>Artificial Sequence
<220>
<223>CD8a hinge and transmembrane domain
<400>15
ttcgtgccgg tcttcctgcc agcgaagccc accacgacgc cagcgccgcg accaccaaca60
ccggcgccca ccatcgcgtc gcagcccctg tccctgcgcc cagaggcgtg ccggccagcg 120
gcggggggcg cagtgcacac gagggggctg gacttcgcct gtgatatcta catctgggcg 180
cccttggccg ggacttgtgg ggtccttctc ctgtcactgg ttatcaccct ttactgcaac 240
cacaggaac 249
<210>16
<211>41
<212>PRT
<213>Artificial Sequence
<220>
<223>CD28 signalling domain
<400>16
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 1015
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
202530
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
3540
<210>17
<211>123
<212>DNA
<213>Artificial Sequence
<220>
<223>CD28 signalling domain
<400>17
aggagtaaga ggagcaggct cctgcacagt gactacatga acatgactcc ccgccgcccc60
gggcccaccc gcaagcatta ccagccctat gccccaccac gcgacttcgc agcctatcgc 120
tcc 123
<210>18
<211>47
<212>PRT
<213>Artificial Sequence
<220>
<223>4-1BB signalling domain
<400>18
Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
1 5 1015
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
202530
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
354045
<210>19
<211>141
<212>DNA
<213>Artificial Sequence
<220>
<223>4-1BB signalling domain
<400>19
cgtttctctg ttgttaaacg gggcagaaag aagctcctgt atatattcaa acaaccattt60
atgagaccag tacaaactac tcaagaggaa gatggctgta gctgccgatt tccagaagaa 120
gaagaaggag gatgtgaact g 141
<210>20
<211>112
<212>PRT
<213>Artificial Sequence
<220>
<223>CD3zeta chain
<400>20
Arg Val Lys Phe Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly
1 5 1015
Gln Asn Gln Leu Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr
202530
Asp Val Leu Asp Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys
354045
Pro Arg Arg Lys Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys
505560
Asp Lys Met Ala Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg
65707580
Arg Arg Gly Lys Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala
859095
Thr Lys Asp Thr Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg
100 105 110
<210>21
<211>336
<212>DNA
<213>Artificial Sequence
<220>
<223>CD3zeta chain
<400>21
agagtgaagt tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc60
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc 120
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat 180
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc 240
cggaggggca aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc 300
tacgacgccc ttcacatgca ggccctgccc cctcgc 336
<210>22
<211>357
<212>PRT
<213>Artificial Sequence
<220>
<223>Truncated EGFR
<400>22
Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro
1 5 1015
Ala Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly
202530
Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe
354045
Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala
505560
Phe Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu
65707580
Leu Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile
859095
Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu
100 105 110
Glu Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala
115 120 125
Val Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu
130 135 140
Ile Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr
145 150 155 160
Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys
165 170 175
Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly
180 185 190
Gln Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu
195 200 205
Pro Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys
210 215 220
Val Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu
225 230 235 240
Asn Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met
245 250 255
Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala
260 265 270
His Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val
275 280 285
Met Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His
290 295 300
Val Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro
305 310 315 320
Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala
325 330 335
Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly
340 345 350
Ile Gly Leu Phe Met
355
<210>23
<211>1071
<212>DNA
<213>Artificial Sequence
<220>
<223>Truncated EGFR
<400>23
atgcttctcc tggtgacaag ccttctgctc tgtgagttac cacacccagc attcctcctg60
atcccacgca aagtgtgtaa cggaataggt attggtgaat ttaaagactc actctccata 120
aatgctacga atattaaaca cttcaaaaac tgcacctcca tcagtggcga tctccacatc 180
ctgccggtgg catttagggg tgactccttc acacatactc ctcctctgga tccacaggaa 240
ctggatattc tgaaaaccgt aaaggaaatc acagggtttt tgctgattca ggcttggcct 300
gaaaacagga cggacctcca tgcctttgag aacctagaaa tcatacgcgg caggaccaag 360
caacatggtc agttttctct tgcagtcgtc agcctgaaca taacatcctt gggattacgc 420
tccctcaagg agataagtga tggagatgtg ataatttcag gaaacaaaaa tttgtgctat 480
gcaaatacaa taaactggaa aaaactgttt gggacctccg gtcagaaaac caaaattata 540
agcaacagag gtgaaaacag ctgcaaggcc acaggccagg tctgccatgc cttgtgctcc 600
cccgagggct gctggggccc ggaacccagg gactgcgtct cttgccggaa tgtcagccga 660
ggcagggaat gcgtggacaa gtgcaacctt ctggagggtg agccaaggga gtttgtggag 720
aactctgagt gcatacagtg ccacccagag tgcctgcctc aggccatgaa catcacctgc 780
acaggacggg gaccagacaa ctgtatccag tgtgcccact acattgacgg cccccactgc 840
gtcaagacct gcccggcagg agtcatggga gaaaacaaca ccctggtctg gaagtacgca 900
gacgccggcc atgtgtgcca cctgtgccat ccaaactgca cctacggatg cactgggcca 960
ggtcttgaag gctgtccaac gaatgggcct aagatcccgt ccatcgccac tgggatggtg1020
ggggccctcc tcttgctgct ggtggtggcc ctggggatcg gcctcttcat g 1071
<210>24
<211>413
<212>PRT
<213>Artificial Sequence
<220>
<223>Inducible caspase-9 (iC9)
<400>24
Met Leu Glu Gly Val Gln Val Glu Thr Ile Ser Pro Gly Asp Gly Arg
1 5 1015
Thr Phe Pro Lys Arg Gly Gln Thr Cys Val Val His Tyr Thr Gly Met
202530
Leu Glu Asp Gly Lys Lys Val Asp Ser Ser Arg Asp Arg Asn Lys Pro
354045
Phe Lys Phe Met Leu Gly Lys Gln Glu Val Ile Arg Gly Trp Glu Glu
505560
Gly Val Ala Gln Met Ser Val Gly Gln Arg Ala Lys Leu Thr Ile Ser
65707580
Pro Asp Tyr Ala Tyr Gly Ala Thr Gly His Pro Gly Ile Ile Pro Pro
859095
His Ala Thr Leu Val Phe Asp Val Glu Leu Leu Lys Leu Glu Ser Gly
100 105 110
Gly Gly Ser Gly Val Asp Gly Phe Gly Asp Val Gly Ala Leu Glu Ser
115 120 125
Leu Arg Gly Asn Ala Asp Leu Ala Tyr Ile Leu Ser Met Glu Pro Cys
130 135 140
Gly His Cys Leu Ile Ile Asn Asn Val Asn Phe Cys Arg Glu Ser Gly
145 150 155 160
Leu Arg Thr Arg Thr Gly Ser Asn Ile Asp Cys Glu Lys Leu Arg Arg
165 170 175
Arg Phe Ser Ser Leu His Phe Met Val Glu Val Lys Gly Asp Leu Thr
180 185 190
Ala Lys Lys Met Val Leu Ala Leu Leu Glu Leu Ala Gln Gln Asp His
195 200 205
Gly Ala Leu Asp Cys Cys Val Val Val Ile Leu Ser His Gly Cys Gln
210 215 220
Ala Ser His Leu Gln Phe Pro Gly Ala Val Tyr Gly Thr Asp Gly Cys
225 230 235 240
Pro Val Ser Val Glu Lys Ile Val Asn Ile Phe Asn Gly Thr Ser Cys
245 250 255
Pro Ser Leu Gly Gly Lys Pro Lys Leu Phe Phe Ile Gln Ala Cys Gly
260 265 270
Gly Glu Gln Lys Asp His Gly Phe Glu Val Ala Ser Thr Ser Pro Glu
275 280 285
Asp Glu Ser Pro Gly Ser Asn Pro Glu Pro Asp Ala Thr Pro Phe Gln
290 295 300
Glu Gly Leu Arg Thr Phe Asp Gln Leu Asp Ala Ile Ser Ser Leu Pro
305 310 315 320
Thr Pro Ser Asp Ile Phe Val Ser Tyr Ser Thr Phe Pro Gly Phe Val
325 330 335
Ser Trp Arg Asp Pro Lys Ser Gly Ser Trp Tyr Val Glu Thr Leu Asp
340 345 350
Asp Ile Phe Glu Gln Trp Ala His Ser Glu Asp Leu Gln Ser Leu Leu
355 360 365
Leu Arg Val Ala Asn Ala Val Ser Val Lys Gly Ile Tyr Lys Gln Met
370 375 380
Pro Gly Cys Phe Asn Phe Leu Arg Lys Lys Leu Phe Phe Lys Thr Ser
385 390 395 400
Val Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Leu Asp
405 410
<210>25
<211>1242
<212>DNA
<213>Artificial Sequence
<220>
<223>Inducible caspase-9 (iC9)
<400>25
atgctcgagg gagtgcaggt ggaaaccatc tccccaggag acgggcgcac cttccccaag60
cgcggccaga cctgcgtggt gcactacacc gggatgcttg aagatggaaa gaaagttgat 120
tcctcccggg acagaaacaa gccctttaag tttatgctag gcaagcagga ggtgatccga 180
ggctgggaag aaggggttgc ccagatgagt gtgggtcaga gagccaaact gactatatct 240
ccagattatg cctatggtgc cactgggcac ccaggcatca tcccaccaca tgccactctc 300
gtcttcgatg tggagcttct aaaactggaa tctggcggtg gatccggagt cgacggattt 360
ggtgatgtcg gtgctcttga gagtttgagg ggaaatgcag atttggctta catcctgagc 420
atggagccct gtggccactg cctcattatc aacaatgtga acttctgccg tgagtccggg 480
ctccgcaccc gcactggctc caacatcgac tgtgagaagt tgcggcgtcg cttctcctcg 540
ctgcatttca tggtggaggt gaagggcgac ctgactgcca agaaaatggt gctggctttg 600
ctggagctgg cgcagcagga ccacggtgct ctggactgct gcgtggtggt cattctctct 660
cacggctgtc aggccagcca cctgcagttc ccaggggctg tctacggcac agatggatgc 720
cctgtgtcgg tcgagaagat tgtgaacatc ttcaatggga ccagctgccc cagcctggga 780
gggaagccca agctcttttt catccaggcc tgtggtgggg agcagaaaga ccatgggttt 840
gaggtggcct ccacttcccc tgaagacgag tcccctggca gtaaccccga gccagatgcc 900
accccgttcc aggaaggttt gaggaccttc gaccagctgg acgccatatc tagtttgccc 960
acacccagtg acatctttgt gtcctactct actttcccag gttttgtttc ctggagggac1020
cccaagagtg gctcctggta cgttgagacc ctggacgaca tctttgagca gtgggctcac1080
tctgaagacc tgcagtccct cctgcttagg gtcgctaatg ctgtttcggt gaaagggatt1140
tataaacaga tgcctggttg ctttaatttc ctccggaaaa aacttttctt taaaacatca1200
gtcgactatc cgtacgacgt accagactac gcactcgact aa 1242
<210>26
<211>22
<212>PRT
<213>Artificial Sequence
<220>
<223>P2A spacer
<400>26
Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val
1 5 1015
Glu Glu Asn Pro Gly Pro
20
<210>27
<211>66
<212>DNA
<213>Artificial Sequence
<220>
<223>P2A spacer
<400>27
ggatccggag ccacgaactt ctctctgtta aagcaagcag gagacgtgga agaaaacccc60
ggtcct 66
<210>28
<211>592
<212>DNA
<213>Woodchuck hepatitis virus
<400>28
aatcaacctc tggattacaa aatttgtgaa agattgactg gtattcttaa ctatgttgct60
ccttttacgc tatgtggata cgctgcttta atgcctttgt atcatgctat tgcttcccgt 120
atggctttca ttttctcctc cttgtataaa tcctggttgc tgtctcttta tgaggagttg 180
tggcccgttg tcaggcaacg tggcgtggtg tgcactgtgt ttgctgacgc aacccccact 240
ggttggggca ttgccaccac ctgtcagctc ctttccggga ctttcgcttt ccccctccct 300
attgccacgg cggaactcat cgccgcctgc cttgcccgct gctggacagg ggctcggctg 360
ttgggcactg acaattccgt ggtgttgtcg gggaagctga cgtcctttcc atggctgctc 420
gcctgtgttg ccacctggat tctgcgcggg acgtccttct gctacgtccc ttcggccctc 480
aatccagcgg accttccttc ccgcggcctg ctgccggctc tgcggcctct tccgcgtctt 540
cgccttcgcc ctcagacgag tcggatctcc ctttgggccg cctccccgcc tg 592
<210>29
<211>1370
<212>PRT
<213>Artificial Sequence
<220>
<223>Nucleic acid construct (CART4)
<400>29
Met Glu Thr Asp Thr Leu Leu Leu Trp Val Leu Leu Leu Trp Val Pro
1 5 1015
Gly Ser Thr Gly Asp Asp Ile Val Met Thr Gln Ser Pro Asp Ser Leu
202530
Ala Val Ser Leu Gly Glu Arg Val Thr Met Asn Cys Lys Ser Ser Gln
354045
Ser Leu Leu Tyr Ser Thr Asn Gln Lys Asn Tyr Leu Ala Trp Tyr Gln
505560
Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr
65707580
Arg Glu Ser Gly Val Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr
859095
Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala Glu Asp Val Ala Val
100 105 110
Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Arg Thr Phe Gly Gly Gly Thr
115 120 125
Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Val Val
145 150 155 160
Lys Pro Gly Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr
165 170 175
Phe Thr Ser Tyr Val Ile His Trp Val Arg Gln Lys Pro Gly Gln Gly
180 185 190
Leu Asp Trp Ile Gly Tyr Ile Asn Pro Tyr Asn Asp Gly Thr Asp Tyr
195 200 205
Asp Glu Lys Phe Lys Gly Lys Ala Thr Leu Thr Ser Asp Thr Ser Thr
210 215 220
Ser Thr Ala Tyr Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala
225 230 235 240
Val Tyr Tyr Cys Ala Arg Glu Lys Asp Asn Tyr Ala Thr Gly Ala Trp
245 250 255
Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ala
260 265 270
Ala Phe Val Pro Val Phe Leu Pro Ala Lys Pro Thr Thr Thr Pro Ala
275 280 285
Pro Arg Pro Pro Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser
290 295 300
Leu Arg Pro Glu Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr
305 310 315 320
Arg Gly Leu Asp Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala
325 330 335
Gly Thr Cys Gly Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys
340 345 350
Asn His Arg Asn Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr
355 360 365
Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln
370 375 380
Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Phe Ser
385 390 395 400
Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
405 410 415
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
420 425 430
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe
435 440 445
Ser Arg Ser Ala Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu
450 455 460
Tyr Asn Glu Leu Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp
465 470 475 480
Lys Arg Arg Gly Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys
485 490 495
Asn Pro Gln Glu Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala
500 505 510
Glu Ala Tyr Ser Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys
515 520 525
Gly His Asp Gly Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr
530 535 540
Tyr Asp Ala Leu His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala
545 550 555 560
Thr Asn Phe Ser Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro
565 570 575
Gly Pro Met Leu Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro
580 585 590
His Pro Ala Phe Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly
595 600 605
Ile Gly Glu Phe Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys
610 615 620
His Phe Lys Asn Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro
625 630 635 640
Val Ala Phe Arg Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro
645 650 655
Gln Glu Leu Asp Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu
660 665 670
Leu Ile Gln Ala Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu
675 680 685
Asn Leu Glu Ile Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser
690 695 700
Leu Ala Val Val Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu
705 710 715 720
Lys Glu Ile Ser Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu
725 730 735
Cys Tyr Ala Asn Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly
740 745 750
Gln Lys Thr Lys Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala
755 760 765
Thr Gly Gln Val Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly
770 775 780
Pro Glu Pro Arg Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg
785 790 795 800
Glu Cys Val Asp Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe
805 810 815
Val Glu Asn Ser Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln
820 825 830
Ala Met Asn Ile Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln
835 840 845
Cys Ala His Tyr Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala
850 855 860
Gly Val Met Gly Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala
865 870 875 880
Gly His Val Cys His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr
885 890 895
Gly Pro Gly Leu Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser
900 905 910
Ile Ala Thr Gly Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala
915 920 925
Leu Gly Ile Gly Leu Phe Met Gly Ser Gly Ala Thr Asn Phe Ser Leu
930 935 940
Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Leu Glu
945 950 955 960
Gly Val Gln Val Glu Thr Ile Ser Pro Gly Asp Gly Arg Thr Phe Pro
965 970 975
Lys Arg Gly Gln Thr Cys Val Val His Tyr Thr Gly Met Leu Glu Asp
980 985 990
Gly Lys Lys Val Asp Ser Ser Arg Asp Arg Asn Lys Pro Phe Lys Phe
995 10001005
Met Leu Gly Lys Gln Glu Val Ile Arg Gly Trp Glu Glu Gly Val
101010151020
Ala Gln Met Ser Val Gly Gln Arg Ala Lys Leu Thr Ile Ser Pro
102510301035
Asp Tyr Ala Tyr Gly Ala Thr Gly His Pro Gly Ile Ile Pro Pro
104010451050
His Ala Thr Leu Val Phe Asp Val Glu Leu Leu Lys Leu Glu Ser
105510601065
Gly Gly Gly Ser Gly Val Asp Gly Phe Gly Asp Val Gly Ala Leu
107010751080
Glu Ser Leu Arg Gly Asn Ala Asp Leu Ala Tyr Ile Leu Ser Met
108510901095
Glu Pro Cys Gly His Cys Leu Ile Ile Asn Asn Val Asn Phe Cys
110011051110
Arg Glu Ser Gly Leu Arg Thr Arg Thr Gly Ser Asn Ile Asp Cys
111511201125
Glu Lys Leu Arg Arg Arg Phe Ser Ser Leu His Phe Met Val Glu
113011351140
Val Lys Gly Asp Leu Thr Ala Lys Lys Met Val Leu Ala Leu Leu
114511501155
Glu Leu Ala Gln Gln Asp His Gly Ala Leu Asp Cys Cys Val Val
116011651170
Val Ile Leu Ser His Gly Cys Gln Ala Ser His Leu Gln Phe Pro
117511801185
Gly Ala Val Tyr Gly Thr Asp Gly Cys Pro Val Ser Val Glu Lys
119011951200
Ile Val Asn Ile Phe Asn Gly Thr Ser Cys Pro Ser Leu Gly Gly
120512101215
Lys Pro Lys Leu Phe Phe Ile Gln Ala Cys Gly Gly Glu Gln Lys
122012251230
Asp His Gly Phe Glu Val Ala Ser Thr Ser Pro Glu Asp Glu Ser
123512401245
Pro Gly Ser Asn Pro Glu Pro Asp Ala Thr Pro Phe Gln Glu Gly
125012551260
Leu Arg Thr Phe Asp Gln Leu Asp Ala Ile Ser Ser Leu Pro Thr
126512701275
Pro Ser Asp Ile Phe Val Ser Tyr Ser Thr Phe Pro Gly Phe Val
128012851290
Ser Trp Arg Asp Pro Lys Ser Gly Ser Trp Tyr Val Glu Thr Leu
129513001305
Asp Asp Ile Phe Glu Gln Trp Ala His Ser Glu Asp Leu Gln Ser
131013151320
Leu Leu Leu Arg Val Ala Asn Ala Val Ser Val Lys Gly Ile Tyr
132513301335
Lys Gln Met Pro Gly Cys Phe Asn Phe Leu Arg Lys Lys Leu Phe
134013451350
Phe Lys Thr Ser Val Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala
135513601365
Leu Asp
1370
<210>30
<211>4113
<212>DNA
<213>Artificial Sequence
<220>
<223>Nucleic acid construct (CART4)
<400>30
atggagacag acacactcct gctatgggtg ctgctgctct gggttccagg ttccacaggt60
gacgacattg tgatgactca gagccccgac agcctggccg tctcactggg cgaaagggtg 120
accatgaatt gtaaatcttc tcagagcctg ctgtacagta caaaccagaa aaattacctg 180
gcctggtatc agcagaaacc cggccagagc cctaagctgc tgatctattg ggcaagtacc 240
cgagagtcag gagtgccaga cagattctcc gggtctggaa gtggcacaga cttcaccctg 300
acaattagct ccgtgcaggc cgaggacgtg gctgtctact attgccagca gtactatagc 360
taccgaactt tcggcggggg aaccaaactg gaaatcaagg gaggaggagg cagtggcgga 420
ggagggtcag gaggaggagg aagccaggtg cagctgcagc agtccggacc agaggtggtc 480
aaacccggcg ctagcgtcaa aatgtcctgt aaggcatctg gctacacttt cacctcttat 540
gtgattcact gggtcagaca gaagcctggg cagggactgg actggatcgg gtacattaac 600
ccatataatg atggaactga ctacgatgaa aagtttaaag gcaaggccac actgacttcc 660
gacacctcaa caagcactgc ttatatggag ctgtctagtc tgaggtctga agacacagca 720
gtgtactatt gcgcccgcga gaaggataac tacgccactg gcgcttggtt tgcatattgg 780
ggccagggga ccctggtgac agtctcatcc gcggccgcat tcgtgccggt cttcctgcca 840
gcgaagccca ccacgacgcc agcgccgcga ccaccaacac cggcgcccac catcgcgtcg 900
cagcccctgt ccctgcgccc agaggcgtgc cggccagcgg cggggggcgc agtgcacacg 960
agggggctgg acttcgcctg tgatatctac atctgggcgc ccttggccgg gacttgtggg1020
gtccttctcc tgtcactggt tatcaccctt tactgcaacc acaggaacag gagtaagagg1080
agcaggctcc tgcacagtga ctacatgaac atgactcccc gccgccccgg gcccacccgc1140
aagcattacc agccctatgc cccaccacgc gacttcgcag cctatcgctc ccgtttctct1200
gttgttaaac ggggcagaaa gaagctcctg tatatattca aacaaccatt tatgagacca1260
gtacaaacta ctcaagagga agatggctgt agctgccgat ttccagaaga agaagaagga1320
ggatgtgaac tgagagtgaa gttcagcagg agcgcagacg cccccgcgta ccagcagggc1380
cagaaccagc tctataacga gctcaatcta ggacgaagag aggagtacga tgttttggac1440
aagagacgtg gccgggaccc tgagatgggg ggaaagccga gaaggaagaa ccctcaggaa1500
ggcctgtaca atgaactgca gaaagataag atggcggagg cctacagtga gattgggatg1560
aaaggcgagc gccggagggg caaggggcac gatggccttt accagggtct cagtacagcc1620
accaaggaca cctacgacgc ccttcacatg caggccctgc cccctcgcgg atccggagcc1680
acgaacttct ctctgttaaa gcaagcagga gacgtggaag aaaaccccgg tcctatgctt1740
ctcctggtga caagccttct gctctgtgag ttaccacacc cagcattcct cctgatccca1800
cgcaaagtgt gtaacggaat aggtattggt gaatttaaag actcactctc cataaatgct1860
acgaatatta aacacttcaa aaactgcacc tccatcagtg gcgatctcca catcctgccg1920
gtggcattta ggggtgactc cttcacacat actcctcctc tggatccaca ggaactggat1980
attctgaaaa ccgtaaagga aatcacaggg tttttgctga ttcaggcttg gcctgaaaac2040
aggacggacc tccatgcctt tgagaaccta gaaatcatac gcggcaggac caagcaacat2100
ggtcagtttt ctcttgcagt cgtcagcctg aacataacat ccttgggatt acgctccctc2160
aaggagataa gtgatggaga tgtgataatt tcaggaaaca aaaatttgtg ctatgcaaat2220
acaataaact ggaaaaaact gtttgggacc tccggtcaga aaaccaaaat tataagcaac2280
agaggtgaaa acagctgcaa ggccacaggc caggtctgcc atgccttgtg ctcccccgag2340
ggctgctggg gcccggaacc cagggactgc gtctcttgcc ggaatgtcag ccgaggcagg2400
gaatgcgtgg acaagtgcaa ccttctggag ggtgagccaa gggagtttgt ggagaactct2460
gagtgcatac agtgccaccc agagtgcctg cctcaggcca tgaacatcac ctgcacagga2520
cggggaccag acaactgtat ccagtgtgcc cactacattg acggccccca ctgcgtcaag2580
acctgcccgg caggagtcat gggagaaaac aacaccctgg tctggaagta cgcagacgcc2640
ggccatgtgt gccacctgtg ccatccaaac tgcacctacg gatgcactgg gccaggtctt2700
gaaggctgtc caacgaatgg gcctaagatc ccgtccatcg ccactgggat ggtgggggcc2760
ctcctcttgc tgctggtggt ggccctgggg atcggcctct tcatgggatc tggagccacg2820
aacttctctc tgttaaagca agcaggagac gtggaagaaa accccggtcc tatgctcgag2880
ggagtgcagg tggaaaccat ctccccagga gacgggcgca ccttccccaa gcgcggccag2940
acctgcgtgg tgcactacac cgggatgctt gaagatggaa agaaagttga ttcctcccgg3000
gacagaaaca agccctttaa gtttatgcta ggcaagcagg aggtgatccg aggctgggaa3060
gaaggggttg cccagatgag tgtgggtcag agagccaaac tgactatatc tccagattat3120
gcctatggtg ccactgggca cccaggcatc atcccaccac atgccactct cgtcttcgat3180
gtggagcttc taaaactgga atctggcggt ggatccggag tcgacggatt tggtgatgtc3240
ggtgctcttg agagtttgag gggaaatgca gatttggctt acatcctgag catggagccc3300
tgtggccact gcctcattat caacaatgtg aacttctgcc gtgagtccgg gctccgcacc3360
cgcactggct ccaacatcga ctgtgagaag ttgcggcgtc gcttctcctc gctgcatttc3420
atggtggagg tgaagggcga cctgactgcc aagaaaatgg tgctggcttt gctggagctg3480
gcgcagcagg accacggtgc tctggactgc tgcgtggtgg tcattctctc tcacggctgt3540
caggccagcc acctgcagtt cccaggggct gtctacggca cagatggatg ccctgtgtcg3600
gtcgagaaga ttgtgaacat cttcaatggg accagctgcc ccagcctggg agggaagccc3660
aagctctttt tcatccaggc ctgtggtggg gagcagaaag accatgggtt tgaggtggcc3720
tccacttccc ctgaagacga gtcccctggc agtaaccccg agccagatgc caccccgttc3780
caggaaggtt tgaggacctt cgaccagctg gacgccatat ctagtttgcc cacacccagt3840
gacatctttg tgtcctactc tactttccca ggttttgttt cctggaggga ccccaagagt3900
ggctcctggt acgttgagac cctggacgac atctttgagc agtgggctca ctctgaagac3960
ctgcagtccc tcctgcttag ggtcgctaat gctgtttcgg tgaaagggat ttataaacag4020
atgcctggtt gctttaattt cctccggaaa aaacttttct ttaaaacatc agtcgactat4080
ccgtacgacg taccagacta cgcactcgac taa 4113
<210>31
<211>1366
<212>PRT
<213>Artificial Sequence
<220>
<223>Nucleic acid construct (CARTVb7.1)
<400>31
Met Ala Leu Pro Val Thr Ala Leu Leu Leu Pro Leu Ala Leu Leu Leu
1 5 1015
His Ala Ala Arg Pro Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu
202530
Ser Ala Ser Leu Gly Gly Lys Val Thr Leu Thr Cys Lys Ala Ser Gln
354045
Asp Ile Asn Lys Tyr Ile Ala Trp Tyr Gln His Lys Pro Gly Lys Gly
505560
Pro Arg Leu Leu Ile His Tyr Thr Ser Thr Leu Gln Pro Gly Ile Pro
65707580
Ser Arg Phe Ser Gly Ser Gly Ser Gly Arg Asp Tyr Ser Phe Ser Ile
859095
Ser Asn Leu Glu Pro Glu Asp Val Ala Thr Tyr Tyr Cys Leu Gln Tyr
100 105 110
Asp Asn Leu Arg Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
115 120 125
Thr Asp Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
130 135 140
Ser Gln Val Gln Leu Gln Gln Pro Gly Ala Glu Leu Val Lys Pro Gly
145 150 155 160
Ala Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Arg
165 170 175
Tyr Trp Ile Thr Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp
180 185 190
Ile Gly Asp Ile Tyr Pro Gly Ser Gly Phe Thr Lys Tyr Asn Glu Lys
195 200 205
Phe Lys Ser Lys Ala Thr Leu Thr Val Asp Thr Ser Ser Ser Thr Ala
210 215 220
Tyr Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr
225 230 235 240
Cys Ala Arg Glu Gly Gly Asn Tyr Trp Tyr Phe Asp Val Trp Gly Thr
245 250 255
Gly Thr Thr Val Thr Val Ser Ser Ala Ala Ala Ala Ala Phe Val Pro
260 265 270
Val Phe Leu Pro Ala Lys Pro Thr Thr Thr Pro Ala Pro Arg Pro Pro
275 280 285
Thr Pro Ala Pro Thr Ile Ala Ser Gln Pro Leu Ser Leu Arg Pro Glu
290 295 300
Ala Cys Arg Pro Ala Ala Gly Gly Ala Val His Thr Arg Gly Leu Asp
305 310 315 320
Phe Ala Cys Asp Ile Tyr Ile Trp Ala Pro Leu Ala Gly Thr Cys Gly
325 330 335
Val Leu Leu Leu Ser Leu Val Ile Thr Leu Tyr Cys Asn His Arg Asn
340 345 350
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
355 360 365
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
370 375 380
Pro Arg Asp Phe Ala Ala Tyr Arg Ser Arg Phe Ser Val Val Lys Arg
385 390 395 400
Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met Arg Pro
405 410 415
Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe Pro Glu
420 425 430
Glu Glu Glu Gly Gly Cys Glu Leu Arg Val Lys Phe Ser Arg Ser Ala
435 440 445
Asp Ala Pro Ala Tyr Gln Gln Gly Gln Asn Gln Leu Tyr Asn Glu Leu
450 455 460
Asn Leu Gly Arg Arg Glu Glu Tyr Asp Val Leu Asp Lys Arg Arg Gly
465 470 475 480
Arg Asp Pro Glu Met Gly Gly Lys Pro Arg Arg Lys Asn Pro Gln Glu
485 490 495
Gly Leu Tyr Asn Glu Leu Gln Lys Asp Lys Met Ala Glu Ala Tyr Ser
500 505 510
Glu Ile Gly Met Lys Gly Glu Arg Arg Arg Gly Lys Gly His Asp Gly
515 520 525
Leu Tyr Gln Gly Leu Ser Thr Ala Thr Lys Asp Thr Tyr Asp Ala Leu
530 535 540
His Met Gln Ala Leu Pro Pro Arg Gly Ser Gly Ala Thr Asn Phe Ser
545 550 555 560
Leu Leu Lys Gln Ala Gly Asp Val Glu Glu Asn Pro Gly Pro Met Leu
565 570 575
Leu Leu Val Thr Ser Leu Leu Leu Cys Glu Leu Pro His Pro Ala Phe
580 585 590
Leu Leu Ile Pro Arg Lys Val Cys Asn Gly Ile Gly Ile Gly Glu Phe
595 600 605
Lys Asp Ser Leu Ser Ile Asn Ala Thr Asn Ile Lys His Phe Lys Asn
610 615 620
Cys Thr Ser Ile Ser Gly Asp Leu His Ile Leu Pro Val Ala Phe Arg
625 630 635 640
Gly Asp Ser Phe Thr His Thr Pro Pro Leu Asp Pro Gln Glu Leu Asp
645 650 655
Ile Leu Lys Thr Val Lys Glu Ile Thr Gly Phe Leu Leu Ile Gln Ala
660 665 670
Trp Pro Glu Asn Arg Thr Asp Leu His Ala Phe Glu Asn Leu Glu Ile
675 680 685
Ile Arg Gly Arg Thr Lys Gln His Gly Gln Phe Ser Leu Ala Val Val
690 695 700
Ser Leu Asn Ile Thr Ser Leu Gly Leu Arg Ser Leu Lys Glu Ile Ser
705 710 715 720
Asp Gly Asp Val Ile Ile Ser Gly Asn Lys Asn Leu Cys Tyr Ala Asn
725 730 735
Thr Ile Asn Trp Lys Lys Leu Phe Gly Thr Ser Gly Gln Lys Thr Lys
740 745 750
Ile Ile Ser Asn Arg Gly Glu Asn Ser Cys Lys Ala Thr Gly Gln Val
755 760 765
Cys His Ala Leu Cys Ser Pro Glu Gly Cys Trp Gly Pro Glu Pro Arg
770 775 780
Asp Cys Val Ser Cys Arg Asn Val Ser Arg Gly Arg Glu Cys Val Asp
785 790 795 800
Lys Cys Asn Leu Leu Glu Gly Glu Pro Arg Glu Phe Val Glu Asn Ser
805 810 815
Glu Cys Ile Gln Cys His Pro Glu Cys Leu Pro Gln Ala Met Asn Ile
820 825 830
Thr Cys Thr Gly Arg Gly Pro Asp Asn Cys Ile Gln Cys Ala His Tyr
835 840 845
Ile Asp Gly Pro His Cys Val Lys Thr Cys Pro Ala Gly Val Met Gly
850 855 860
Glu Asn Asn Thr Leu Val Trp Lys Tyr Ala Asp Ala Gly His Val Cys
865 870 875 880
His Leu Cys His Pro Asn Cys Thr Tyr Gly Cys Thr Gly Pro Gly Leu
885 890 895
Glu Gly Cys Pro Thr Asn Gly Pro Lys Ile Pro Ser Ile Ala Thr Gly
900 905 910
Met Val Gly Ala Leu Leu Leu Leu Leu Val Val Ala Leu Gly Ile Gly
915 920 925
Leu Phe Met Gly Ser Gly Ala Thr Asn Phe Ser Leu Leu Lys Gln Ala
930 935 940
Gly Asp Val Glu Glu Asn Pro Gly Pro Met Leu Glu Gly Val Gln Val
945 950 955 960
Glu Thr Ile Ser Pro Gly Asp Gly Arg Thr Phe Pro Lys Arg Gly Gln
965 970 975
Thr Cys Val Val His Tyr Thr Gly Met Leu Glu Asp Gly Lys Lys Val
980 985 990
Asp Ser Ser Arg Asp Arg Asn Lys Pro Phe Lys Phe Met Leu Gly Lys
995 10001005
Gln Glu Val Ile Arg Gly Trp Glu Glu Gly Val Ala Gln Met Ser
101010151020
Val Gly Gln Arg Ala Lys Leu Thr Ile Ser Pro Asp Tyr Ala Tyr
102510301035
Gly Ala Thr Gly His Pro Gly Ile Ile Pro Pro His Ala Thr Leu
104010451050
Val Phe Asp Val Glu Leu Leu Lys Leu Glu Ser Gly Gly Gly Ser
105510601065
Gly Val Asp Gly Phe Gly Asp Val Gly Ala Leu Glu Ser Leu Arg
107010751080
Gly Asn Ala Asp Leu Ala Tyr Ile Leu Ser Met Glu Pro Cys Gly
108510901095
His Cys Leu Ile Ile Asn Asn Val Asn Phe Cys Arg Glu Ser Gly
110011051110
Leu Arg Thr Arg Thr Gly Ser Asn Ile Asp Cys Glu Lys Leu Arg
111511201125
Arg Arg Phe Ser Ser Leu His Phe Met Val Glu Val Lys Gly Asp
113011351140
Leu Thr Ala Lys Lys Met Val Leu Ala Leu Leu Glu Leu Ala Gln
114511501155
Gln Asp His Gly Ala Leu Asp Cys Cys Val Val Val Ile Leu Ser
116011651170
His Gly Cys Gln Ala Ser His Leu Gln Phe Pro Gly Ala Val Tyr
117511801185
Gly Thr Asp Gly Cys Pro Val Ser Val Glu Lys Ile Val Asn Ile
119011951200
Phe Asn Gly Thr Ser Cys Pro Ser Leu Gly Gly Lys Pro Lys Leu
120512101215
Phe Phe Ile Gln Ala Cys Gly Gly Glu Gln Lys Asp His Gly Phe
122012251230
Glu Val Ala Ser Thr Ser Pro Glu Asp Glu Ser Pro Gly Ser Asn
123512401245
Pro Glu Pro Asp Ala Thr Pro Phe Gln Glu Gly Leu Arg Thr Phe
125012551260
Asp Gln Leu Asp Ala Ile Ser Ser Leu Pro Thr Pro Ser Asp Ile
126512701275
Phe Val Ser Tyr Ser Thr Phe Pro Gly Phe Val Ser Trp Arg Asp
128012851290
Pro Lys Ser Gly Ser Trp Tyr Val Glu Thr Leu Asp Asp Ile Phe
129513001305
Glu Gln Trp Ala His Ser Glu Asp Leu Gln Ser Leu Leu Leu Arg
131013151320
Val Ala Asn Ala Val Ser Val Lys Gly Ile Tyr Lys Gln Met Pro
132513301335
Gly Cys Phe Asn Phe Leu Arg Lys Lys Leu Phe Phe Lys Thr Ser
134013451350
Val Asp Tyr Pro Tyr Asp Val Pro Asp Tyr Ala Leu Asp
135513601365
<210>32
<211>4101
<212>DNA
<213>Artificial Sequence
<220>
<223>Nucleic acid construct (CARTVb7.1)
<400>32
atggctctgc ctgttacagc tctgctgctg cctctggctc tgcttctgca tgccgccaga60
cctgacatcc agatgacaca gagccctagc agcctgtctg cctctctcgg cggaaaagtg 120
accctgacat gcaaggccag ccaggacatc aacaagtata tcgcctggta tcagcacaag 180
cccggcaagg gacctagact gctgatccac tacaccagca cactgcagcc tggcatcccc 240
agcagatttt ctggcagcgg ctccggcaga gactacagct tcagcatcag caacctggaa 300
cctgaggacg tggccaccta ctactgcctg cagtacgaca acctgcggac ctttggcggc 360
ggaacaaagc tggaaatcaa gcggacagat ggcggaggcg gatcaggcgg cggaggaagc 420
ggtggcggag gatctcaagt tcagctgcaa cagcctggcg ccgagcttgt gaaacctggc 480
gcctctgtga agatgagctg caaggcctcc ggctacacct tcaccagata ctggatcacc 540
tgggtcaagc agaggcctgg acagggactc gagtggatcg gcgatatcta tcctggctcc 600
ggcttcacca agtacaacga gaagttcaag agcaaggcca cactgaccgt ggacaccagc 660
agcagcacag cctacatgca gctgtctagc ctgaccagcg aggacagcgc cgtgtactac 720
tgtgctagag aaggcggcaa ctactggtac ttcgacgtgt ggggcaccgg caccacagtg 780
acagttagtt ctgcggccgc ggccgcattc gtgccggtct tcctgccagc gaagcccacc 840
acgacgccag cgccgcgacc accaacaccg gcgcccacca tcgcgtcgca gcccctgtcc 900
ctgcgcccag aggcgtgccg gccagcggcg gggggcgcag tgcacacgag ggggctggac 960
ttcgcctgtg atatctacat ctgggcgccc ttggccggga cttgtggggt ccttctcctg1020
tcactggtta tcacccttta ctgcaaccac aggaacagga gtaagaggag caggctcctg1080
cacagtgact acatgaacat gactccccgc cgccccgggc ccacccgcaa gcattaccag1140
ccctatgccc caccacgcga cttcgcagcc tatcgctccc gtttctctgt tgttaaacgg1200
ggcagaaaga agctcctgta tatattcaaa caaccattta tgagaccagt acaaactact1260
caagaggaag atggctgtag ctgccgattt ccagaagaag aagaaggagg atgtgaactg1320
agagtgaagt tcagcaggag cgcagacgcc cccgcgtacc agcagggcca gaaccagctc1380
tataacgagc tcaatctagg acgaagagag gagtacgatg ttttggacaa gagacgtggc1440
cgggaccctg agatgggggg aaagccgaga aggaagaacc ctcaggaagg cctgtacaat1500
gaactgcaga aagataagat ggcggaggcc tacagtgaga ttgggatgaa aggcgagcgc1560
cggaggggca aggggcacga tggcctttac cagggtctca gtacagccac caaggacacc1620
tacgacgccc ttcacatgca ggccctgccc cctcgcggat ccggagccac gaacttctct1680
ctgttaaagc aagcaggaga cgtggaagaa aaccccggtc ctatgcttct cctggtgaca1740
agccttctgc tctgtgagtt accacaccca gcattcctcc tgatcccacg caaagtgtgt1800
aacggaatag gtattggtga atttaaagac tcactctcca taaatgctac gaatattaaa1860
cacttcaaaa actgcacctc catcagtggc gatctccaca tcctgccggt ggcatttagg1920
ggtgactcct tcacacatac tcctcctctg gatccacagg aactggatat tctgaaaacc1980
gtaaaggaaa tcacagggtt tttgctgatt caggcttggc ctgaaaacag gacggacctc2040
catgcctttg agaacctaga aatcatacgc ggcaggacca agcaacatgg tcagttttct2100
cttgcagtcg tcagcctgaa cataacatcc ttgggattac gctccctcaa ggagataagt2160
gatggagatg tgataatttc aggaaacaaa aatttgtgct atgcaaatac aataaactgg2220
aaaaaactgt ttgggacctc cggtcagaaa accaaaatta taagcaacag aggtgaaaac2280
agctgcaagg ccacaggcca ggtctgccat gccttgtgct cccccgaggg ctgctggggc2340
ccggaaccca gggactgcgt ctcttgccgg aatgtcagcc gaggcaggga atgcgtggac2400
aagtgcaacc ttctggaggg tgagccaagg gagtttgtgg agaactctga gtgcatacag2460
tgccacccag agtgcctgcc tcaggccatg aacatcacct gcacaggacg gggaccagac2520
aactgtatcc agtgtgccca ctacattgac ggcccccact gcgtcaagac ctgcccggca2580
ggagtcatgg gagaaaacaa caccctggtc tggaagtacg cagacgccgg ccatgtgtgc2640
cacctgtgcc atccaaactg cacctacgga tgcactgggc caggtcttga aggctgtcca2700
acgaatgggc ctaagatccc gtccatcgcc actgggatgg tgggggccct cctcttgctg2760
ctggtggtgg ccctggggat cggcctcttc atgggatctg gagccacgaa cttctctctg2820
ttaaagcaag caggagacgt ggaagaaaac cccggtccta tgctcgaggg agtgcaggtg2880
gaaaccatct ccccaggaga cgggcgcacc ttccccaagc gcggccagac ctgcgtggtg2940
cactacaccg ggatgcttga agatggaaag aaagttgatt cctcccggga cagaaacaag3000
ccctttaagt ttatgctagg caagcaggag gtgatccgag gctgggaaga aggggttgcc3060
cagatgagtg tgggtcagag agccaaactg actatatctc cagattatgc ctatggtgcc3120
actgggcacc caggcatcat cccaccacat gccactctcg tcttcgatgt ggagcttcta3180
aaactggaat ctggcggtgg atccggagtc gacggatttg gtgatgtcgg tgctcttgag3240
agtttgaggg gaaatgcaga tttggcttac atcctgagca tggagccctg tggccactgc3300
ctcattatca acaatgtgaa cttctgccgt gagtccgggc tccgcacccg cactggctcc3360
aacatcgact gtgagaagtt gcggcgtcgc ttctcctcgc tgcatttcat ggtggaggtg3420
aagggcgacc tgactgccaa gaaaatggtg ctggctttgc tggagctggc gcagcaggac3480
cacggtgctc tggactgctg cgtggtggtc attctctctc acggctgtca ggccagccac3540
ctgcagttcc caggggctgt ctacggcaca gatggatgcc ctgtgtcggt cgagaagatt3600
gtgaacatct tcaatgggac cagctgcccc agcctgggag ggaagcccaa gctctttttc3660
atccaggcct gtggtgggga gcagaaagac catgggtttg aggtggcctc cacttcccct3720
gaagacgagt cccctggcag taaccccgag ccagatgcca ccccgttcca ggaaggtttg3780
aggaccttcg accagctgga cgccatatct agtttgccca cacccagtga catctttgtg3840
tcctactcta ctttcccagg ttttgtttcc tggagggacc ccaagagtgg ctcctggtac3900
gttgagaccc tggacgacat ctttgagcag tgggctcact ctgaagacct gcagtccctc3960
ctgcttaggg tcgctaatgc tgtttcggtg aaagggattt ataaacagat gcctggttgc4020
tttaatttcc tccggaaaaa acttttcttt aaaacatcag tcgactatcc gtacgacgta4080
ccagactacg cactcgacta a4101
<210>33
<211>10348
<212>DNA
<213>Artificial Sequence
<220>
<223>Vector (CART4: CAR4-tEGFR-iC9)
<400>33
tgaaagaccc cacctgtagg tttggcaagc tagcttaagt aacgccattt tgcaaggcat60
ggaaaataca taactgagaa tagagaagtt cagatcaagg ttaggaacag agagacagca 120
gaatatgggc caaacaggat atctgtggta agcagttcct gccccggctc agggccaaga 180
acagatggtc cccagatgcg gtcccgccct cagcagtttc tagagaacca tcagatgttt 240
ccagggtgcc ccaaggacct gaaatgaccc tgtgccttat ttgaactaac caatcagttc 300
gcttctcgct tctgttcgcg cgcttctgct ccccgagctc aataaaagag cccacaaccc 360
ctcactcggc gcgccagtcc tccgatagac tgcgtcgccc gggtacccgt attcccaata 420
aagcctcttg ctgtttgcat ccgaatcgtg gactcgctga tccttgggag ggtctcctca 480
gattgattga ctgcccacct cgggggtctt tcatttggag gttccaccga gatttggaga 540
cccctgccca gggaccaccg acccccccgc cgggaggtaa gctggccagc ggtcgtttcg 600
tgtctgtctc tgtctttgtg cgtgtttgtg ccggcatcta atgtttgcgc ctgcgtctgt 660
actagttagc taactagctc tgtatctggc ggacccgtgg tggaactgac gagttctgaa 720
cacccggccg caaccctggg agacgtccca gggactttgg gggccgtttt tgtggcccga 780
cctgaggaag ggagtcgatg tggaatccga ccccgtcagg atatgtggtt ctggtaggag 840
acgagaacct aaaacagttc ccgcctccgt ctgaattttt gctttcggtt tggaaccgaa 900
gccgcgcgtc ttgtctgctg cagcgctgca gcatcgttct gtgttgtctc tgtctgactg 960
tgtttctgta tttgtctgaa aattagggcc agactgttac cactccctta agtttgacct1020
taggtcactg gaaagatgtc gagcggatcg ctcacaacca gtcggtagat gtcaagaaga1080
gacgttgggt taccttctgc tctgcagaat ggccaacctt taacgtcgga tggccgcgag1140
acggcacctt taaccgagac ctcatcaccc aggttaagat caaggtcttt tcacctggcc1200
cgcatggaca cccagaccag gtcccctaca tcgtgacctg ggaagccttg gcttttgacc1260
cccctccctg ggtcaagccc tttgtacacc ctaagcctcc gcctcctctt cctccatccg1320
ccccgtctct cccccttgaa cctcctcgtt cgaccccgcc tcgatcctcc ctttatccag1380
ccctcactcc ttctctaggc gccggaatta gatctctcga ggttaacgaa ttctaccggg1440
taggggaggc gcttttccca aggcagtctg gagcatgcgc tttagcagcc ccgctgggca1500
cttggcgcta cacaagtggc ctctggcctc gcacacattc cacatccacc ggtaggcgcc1560
aaccggctcc gttctttggt ggccccttcg cgccaccttc tactcctccc ctagtcagga1620
agttcccccc cgccccgcag ctcgcgtcgt gcaggacgtg acaaatggaa gtagcacgtc1680
tcactagtct cgtgcagatg gacagcaccg ctgagcaatg gaagcgggta ggcctttggg1740
gcagcggcca atagcagctt tgctccttcg ctttctgggc tcagaggctg ggaaggggtg1800
ggtccggggg cgggctcagg ggcgggctca ggggcggggc gggcgcccga aggtcctccg1860
gaggcccggc attctgcacg cttcaaaagc gcacgtctgc cgcgctgttc tcctcttcct1920
cattctccgg gcctttcgac ctgcagccca agccaccatg gagacagaca cactcctgct1980
atgggtgctg ctgctctggg ttccaggttc cacaggtgac gacattgtga tgactcagag2040
ccccgacagc ctggccgtct cactgggcga aagggtgacc atgaattgta aatcttctca2100
gagcctgctg tacagtacaa accagaaaaa ttacctggcc tggtatcagc agaaacccgg2160
ccagagccct aagctgctga tctattgggc aagtacccga gagtcaggag tgccagacag2220
attctccggg tctggaagtg gcacagactt caccctgaca attagctccg tgcaggccga2280
ggacgtggct gtctactatt gccagcagta ctatagctac cgaactttcg gcgggggaac2340
caaactggaa atcaagggag gaggaggcag tggcggagga gggtcaggag gaggaggaag2400
ccaggtgcag ctgcagcagt ccggaccaga ggtggtcaaa cccggcgcta gcgtcaaaat2460
gtcctgtaag gcatctggct acactttcac ctcttatgtg attcactggg tcagacagaa2520
gcctgggcag ggactggact ggatcgggta cattaaccca tataatgatg gaactgacta2580
cgatgaaaag tttaaaggca aggccacact gacttccgac acctcaacaa gcactgctta2640
tatggagctg tctagtctga ggtctgaaga cacagcagtg tactattgcg cccgcgagaa2700
ggataactac gccactggcg cttggtttgc atattggggc caggggaccc tggtgacagt2760
ctcatccgcg gccgcattcg tgccggtctt cctgccagcg aagcccacca cgacgccagc2820
gccgcgacca ccaacaccgg cgcccaccat cgcgtcgcag cccctgtccc tgcgcccaga2880
ggcgtgccgg ccagcggcgg ggggcgcagt gcacacgagg gggctggact tcgcctgtga2940
tatctacatc tgggcgccct tggccgggac ttgtggggtc cttctcctgt cactggttat3000
caccctttac tgcaaccaca ggaacaggag taagaggagc aggctcctgc acagtgacta3060
catgaacatg actccccgcc gccccgggcc cacccgcaag cattaccagc cctatgcccc3120
accacgcgac ttcgcagcct atcgctcccg tttctctgtt gttaaacggg gcagaaagaa3180
gctcctgtat atattcaaac aaccatttat gagaccagta caaactactc aagaggaaga3240
tggctgtagc tgccgatttc cagaagaaga agaaggagga tgtgaactga gagtgaagtt3300
cagcaggagc gcagacgccc ccgcgtacca gcagggccag aaccagctct ataacgagct3360
caatctagga cgaagagagg agtacgatgt tttggacaag agacgtggcc gggaccctga3420
gatgggggga aagccgagaa ggaagaaccc tcaggaaggc ctgtacaatg aactgcagaa3480
agataagatg gcggaggcct acagtgagat tgggatgaaa ggcgagcgcc ggaggggcaa3540
ggggcacgat ggcctttacc agggtctcag tacagccacc aaggacacct acgacgccct3600
tcacatgcag gccctgcccc ctcgcggatc cggagccacg aacttctctc tgttaaagca3660
agcaggagac gtggaagaaa accccggtcc tatgcttctc ctggtgacaa gccttctgct3720
ctgtgagtta ccacacccag cattcctcct gatcccacgc aaagtgtgta acggaatagg3780
tattggtgaa tttaaagact cactctccat aaatgctacg aatattaaac acttcaaaaa3840
ctgcacctcc atcagtggcg atctccacat cctgccggtg gcatttaggg gtgactcctt3900
cacacatact cctcctctgg atccacagga actggatatt ctgaaaaccg taaaggaaat3960
cacagggttt ttgctgattc aggcttggcc tgaaaacagg acggacctcc atgcctttga4020
gaacctagaa atcatacgcg gcaggaccaa gcaacatggt cagttttctc ttgcagtcgt4080
cagcctgaac ataacatcct tgggattacg ctccctcaag gagataagtg atggagatgt4140
gataatttca ggaaacaaaa atttgtgcta tgcaaataca ataaactgga aaaaactgtt4200
tgggacctcc ggtcagaaaa ccaaaattat aagcaacaga ggtgaaaaca gctgcaaggc4260
cacaggccag gtctgccatg ccttgtgctc ccccgagggc tgctggggcc cggaacccag4320
ggactgcgtc tcttgccgga atgtcagccg aggcagggaa tgcgtggaca agtgcaacct4380
tctggagggt gagccaaggg agtttgtgga gaactctgag tgcatacagt gccacccaga4440
gtgcctgcct caggccatga acatcacctg cacaggacgg ggaccagaca actgtatcca4500
gtgtgcccac tacattgacg gcccccactg cgtcaagacc tgcccggcag gagtcatggg4560
agaaaacaac accctggtct ggaagtacgc agacgccggc catgtgtgcc acctgtgcca4620
tccaaactgc acctacggat gcactgggcc aggtcttgaa ggctgtccaa cgaatgggcc4680
taagatcccg tccatcgcca ctgggatggt gggggccctc ctcttgctgc tggtggtggc4740
cctggggatc ggcctcttca tgggatctgg agccacgaac ttctctctgt taaagcaagc4800
aggagacgtg gaagaaaacc ccggtcctat gctcgaggga gtgcaggtgg aaaccatctc4860
cccaggagac gggcgcacct tccccaagcg cggccagacc tgcgtggtgc actacaccgg4920
gatgcttgaa gatggaaaga aagttgattc ctcccgggac agaaacaagc cctttaagtt4980
tatgctaggc aagcaggagg tgatccgagg ctgggaagaa ggggttgccc agatgagtgt5040
gggtcagaga gccaaactga ctatatctcc agattatgcc tatggtgcca ctgggcaccc5100
aggcatcatc ccaccacatg ccactctcgt cttcgatgtg gagcttctaa aactggaatc5160
tggcggtgga tccggagtcg acggatttgg tgatgtcggt gctcttgaga gtttgagggg5220
aaatgcagat ttggcttaca tcctgagcat ggagccctgt ggccactgcc tcattatcaa5280
caatgtgaac ttctgccgtg agtccgggct ccgcacccgc actggctcca acatcgactg5340
tgagaagttg cggcgtcgct tctcctcgct gcatttcatg gtggaggtga agggcgacct5400
gactgccaag aaaatggtgc tggctttgct ggagctggcg cagcaggacc acggtgctct5460
ggactgctgc gtggtggtca ttctctctca cggctgtcag gccagccacc tgcagttccc5520
aggggctgtc tacggcacag atggatgccc tgtgtcggtc gagaagattg tgaacatctt5580
caatgggacc agctgcccca gcctgggagg gaagcccaag ctctttttca tccaggcctg5640
tggtggggag cagaaagacc atgggtttga ggtggcctcc acttcccctg aagacgagtc5700
ccctggcagt aaccccgagc cagatgccac cccgttccag gaaggtttga ggaccttcga5760
ccagctggac gccatatcta gtttgcccac acccagtgac atctttgtgt cctactctac5820
tttcccaggt tttgtttcct ggagggaccc caagagtggc tcctggtacg ttgagaccct5880
ggacgacatc tttgagcagt gggctcactc tgaagacctg cagtccctcc tgcttagggt5940
cgctaatgct gtttcggtga aagggattta taaacagatg cctggttgct ttaatttcct6000
ccggaaaaaa cttttcttta aaacatcagt cgactatccg tacgacgtac cagactacgc6060
actcgactaa acaatcaacc tctggattac aaaatttgtg aaagattgac tggtattctt6120
aactatgttg ctccttttac gctatgtgga tacgctgctt taatgccttt gtatcatgct6180
attgcttccc gtatggcttt cattttctcc tccttgtata aatcctggtt gctgtctctt6240
tatgaggagt tgtggcccgt tgtcaggcaa cgtggcgtgg tgtgcactgt gtttgctgac6300
gcaaccccca ctggttgggg cattgccacc acctgtcagc tcctttccgg gactttcgct6360
ttccccctcc ctattgccac ggcggaactc atcgccgcct gccttgcccg ctgctggaca6420
ggggctcggc tgttgggcac tgacaattcc gtggtgttgt cggggaaatc atcgtccttt6480
ccttggctgc tcgcctgtgt tgccacctgg attctgcgcg ggacgtcctt ctgctacgtc6540
ccttcggccc tcaatccagc ggaccttcct tcccgcggcc tgctgccggc tctgcggcct6600
cttccgcgtc ttcgccttcg ccctcagacg agtcggatct ccctttgggc cgcctccccg6660
cctatcgata aaataaaaga ttttatttag tctccagaaa aaggggggaa tgaaagaccc6720
cacctgtagg tttggcaagc tagcttaagt aacgccattt tgcaaggcat ggaaaataca6780
taactgagaa tagagaagtt cagatcaagg ttaggaacag agagacagca gaatatgggc6840
caaacaggat atctgtggta agcagttcct gccccggctc agggccaaga acagatggtc6900
cccagatgcg gtcccgccct cagcagtttc tagagaacca tcagatgttt ccagggtgcc6960
ccaaggacct gaaatgaccc tgtgccttat ttgaactaac caatcagttc gcttctcgct7020
tctgttcgcg cgcttctgct ccccgagctc aataaaagag cccacaaccc ctcactcggc7080
gcgccagtcc tccgatagac tgcgtcgccc gggtacccgt gtatccaata aaccctcttg7140
cagttgcatc cgacttgtgg tctcgctgtt ccttgggagg gtctcctctg agtgattgac7200
tacccgtcag cgggggtctt tcatgggtaa cagtttcttg aagttggaga acaacattct7260
gagggtagga gtcgaatatt aagtaatcct gactcaatta gccactgttt tgaatccaca7320
tactccaata ctcctgaaat agttcattat ggacagcgca gaagagctgg ggagaattaa7380
ttcgtaatca tggtcatagc tgtttcctgt gtgaaattgt tatccgctca caattccaca7440
caacatacga gccggaagca taaagtgtaa agcctggggt gcctaatgag tgagctaact7500
cacattaatt gcgttgcgct cactgcccgc tttccagtcg ggaaacctgt cgtgccagct7560
gcattaatga atcggccaac gcgcggggag aggcggtttg cgtattgggc gctcttccgc7620
ttcctcgctc actgactcgc tgcgctcggt cgttcggctg cggcgagcgg tatcagctca7680
ctcaaaggcg gtaatacggt tatccacaga atcaggggat aacgcaggaa agaacatgtg7740
agcaaaaggc cagcaaaagg ccaggaaccg taaaaaggcc gcgttgctgg cgtttttcca7800
taggctccgc ccccctgacg agcatcacaa aaatcgacgc tcaagtcaga ggtggcgaaa7860
cccgacagga ctataaagat accaggcgtt tccccctgga agctccctcg tgcgctctcc7920
tgttccgacc ctgccgctta ccggatacct gtccgccttt ctcccttcgg gaagcgtggc7980
gctttctcat agctcacgct gtaggtatct cagttcggtg taggtcgttc gctccaagct8040
gggctgtgtg cacgaacccc ccgttcagcc cgaccgctgc gccttatccg gtaactatcg8100
tcttgagtcc aacccggtaa gacacgactt atcgccactg gcagcagcca ctggtaacag8160
gattagcaga gcgaggtatg taggcggtgc tacagagttc ttgaagtggt ggcctaacta8220
cggctacact agaaggacag tatttggtat ctgcgctctg ctgaagccag ttaccttcgg8280
aaaaagagtt ggtagctctt gatccggcaa acaaaccacc gctggtagcg gtggtttttt8340
tgtttgcaag cagcagatta cgcgcagaaa aaaaggatct caagaagatc ctttgatctt8400
ttctacgggg tctgacgctc agtggaacga aaactcacgt taagggattt tggtcatgag8460
attatcaaaa aggatcttca cctagatcct tttaaattaa aaatgaagtt ttaaatcaat8520
ctaaagtata tatgagtaaa cttggtctga cagttaccaa tgcttaatca gtgaggcacc8580
tatctcagcg atctgtctat ttcgttcatc catagttgcc tgactccccg tcgtgtagat8640
aactacgata cgggagggct taccatctgg ccccagtgct gcaatgatac cgcgagaccc8700
acgctcaccg gctccagatt tatcagcaat aaaccagcca gccggaaggg ccgagcgcag8760
aagtggtcct gcaactttat ccgcctccat ccagtctatt aattgttgcc gggaagctag8820
agtaagtagt tcgccagtta atagtttgcg caacgttgtt gccattgcta caggcatcgt8880
ggtgtcacgc tcgtcgtttg gtatggcttc attcagctcc ggttcccaac gatcaaggcg8940
agttacatga tcccccatgt tgtgcaaaaa agcggttagc tccttcggtc ctccgatcgt9000
tgtcagaagt aagttggccg cagtgttatc actcatggtt atggcagcac tgcataattc9060
tcttactgtc atgccatccg taagatgctt ttctgtgact ggtgagtact caaccaagtc9120
attctgagaa tagtgtatgc ggcgaccgag ttgctcttgc ccggcgtcaa tacgggataa9180
taccgcgcca catagcagaa ctttaaaagt gctcatcatt ggaaaacgtt cttcggggcg9240
aaaactctca aggatcttac cgctgttgag atccagttcg atgtaaccca ctcgtgcacc9300
caactgatct tcagcatctt ttactttcac cagcgtttct gggtgagcaa aaacaggaag9360
gcaaaatgcc gcaaaaaagg gaataagggc gacacggaaa tgttgaatac tcatactctt9420
cctttttcaa tattattgaa gcatttatca gggttattgt ctcatgagcg gatacatatt9480
tgaatgtatt tagaaaaata aacaaatagg ggttccgcgc acatttcccc gaaaagtgcc9540
acctgacgtc taagaaacca ttattatcat gacattaacc tataaaaata ggcgtatcac9600
gaggcccttt cgtctcgcgc gtttcggtga tgacggtgaa aacctctgac acatgcagct9660
cccggagacg gtcacagctt gtctgtaagc ggatgccggg agcagacaag cccgtcaggg9720
cgcgtcagcg ggtgttggcg ggtgtcgggg ctggcttaac tatgcggcat cagagcagat9780
tgtactgaga gtgcaccata tgcggtgtga aataccgcac agatgcgtaa ggagaaaata9840
ccgcatcagg cgccattcgc cattcaggct gcgcaactgt tgggaagggc gatcggtgcg9900
ggcctcttcg ctattacgcc agctggcgaa agggggatgt gctgcaaggc gattaagttg9960
ggtaacgcca gggttttccc agtcacgacg ttgtaaaacg acggcgcaag gaatggtgca 10020
tgcaaggaga tggcgcccaa cagtcccccg gccacggggc ctgccaccat acccacgccg 10080
aaacaagcgc tcatgagccc gaagtggcga gcccgatctt ccccatcggt gatgtcggcg 10140
atataggcgc cagcaaccgc acctgtggcg ccggtgatgc cggccacgat gcgtccggcg 10200
tagaggcgat tagtccaatt tgttaaagac aggatatcag tggtccaggc tctagttttg 10260
actcaacaat atcaccagct gaagcctata gagtacgagc catagataaa ataaaagatt 10320
ttatttagtc tccagaaaaa ggggggaa10348
<210>34
<211>109
<212>PRT
<213>Artificial Sequence
<220>
<223>First coding sequence (VH for TCR V-beta)
<400>34
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 1015
Gly Lys Val Thr Leu Thr Cys Lys Ala Ser Gln Asp Ile Asn Lys Tyr
202530
Ile Ala Trp Tyr Gln His Lys Pro Gly Lys Gly Pro Arg Leu Leu Ile
354045
His Tyr Thr Ser Thr Leu Gln Pro Gly Ile Pro Ser Arg Phe Ser Gly
505560
Ser Gly Ser Gly Arg Asp Tyr Ser Phe Ser Ile Ser Asn Leu Glu Pro
65707580
Glu Asp Val Ala Thr Tyr Tyr Cys Leu Gln Tyr Asp Asn Leu Arg Thr
859095
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Asp
100 105
<210>35
<211>327
<212>DNA
<213>Artificial Sequence
<220>
<223>First coding sequence (VH for TCR V-beta)
<400>35
gacatccaga tgacacagag ccctagcagc ctgtctgcct ctctcggcgg aaaagtgacc60
ctgacatgca aggccagcca ggacatcaac aagtatatcg cctggtatca gcacaagccc 120
ggcaagggac ctagactgct gatccactac accagcacac tgcagcctgg catccccagc 180
agattttctg gcagcggctc cggcagagac tacagcttca gcatcagcaa cctggaacct 240
gaggacgtgg ccacctacta ctgcctgcag tacgacaacc tgcggacctt tggcggcgga 300
acaaagctgg aaatcaagcg gacagat 327
<210>36
<211>10336
<212>DNA
<213>Artificial Sequence
<220>
<223>Vector (CARTVb7.1: CARTVb7.1-tEGFR-iC9)
<400>36
tgaaagaccc cacctgtagg tttggcaagc tagcttaagt aacgccattt tgcaaggcat60
ggaaaataca taactgagaa tagagaagtt cagatcaagg ttaggaacag agagacagca 120
gaatatgggc caaacaggat atctgtggta agcagttcct gccccggctc agggccaaga 180
acagatggtc cccagatgcg gtcccgccct cagcagtttc tagagaacca tcagatgttt 240
ccagggtgcc ccaaggacct gaaatgaccc tgtgccttat ttgaactaac caatcagttc 300
gcttctcgct tctgttcgcg cgcttctgct ccccgagctc aataaaagag cccacaaccc 360
ctcactcggc gcgccagtcc tccgatagac tgcgtcgccc gggtacccgt attcccaata 420
aagcctcttg ctgtttgcat ccgaatcgtg gactcgctga tccttgggag ggtctcctca 480
gattgattga ctgcccacct cgggggtctt tcatttggag gttccaccga gatttggaga 540
cccctgccca gggaccaccg acccccccgc cgggaggtaa gctggccagc ggtcgtttcg 600
tgtctgtctc tgtctttgtg cgtgtttgtg ccggcatcta atgtttgcgc ctgcgtctgt 660
actagttagc taactagctc tgtatctggc ggacccgtgg tggaactgac gagttctgaa 720
cacccggccg caaccctggg agacgtccca gggactttgg gggccgtttt tgtggcccga 780
cctgaggaag ggagtcgatg tggaatccga ccccgtcagg atatgtggtt ctggtaggag 840
acgagaacct aaaacagttc ccgcctccgt ctgaattttt gctttcggtt tggaaccgaa 900
gccgcgcgtc ttgtctgctg cagcgctgca gcatcgttct gtgttgtctc tgtctgactg 960
tgtttctgta tttgtctgaa aattagggcc agactgttac cactccctta agtttgacct1020
taggtcactg gaaagatgtc gagcggatcg ctcacaacca gtcggtagat gtcaagaaga1080
gacgttgggt taccttctgc tctgcagaat ggccaacctt taacgtcgga tggccgcgag1140
acggcacctt taaccgagac ctcatcaccc aggttaagat caaggtcttt tcacctggcc1200
cgcatggaca cccagaccag gtcccctaca tcgtgacctg ggaagccttg gcttttgacc1260
cccctccctg ggtcaagccc tttgtacacc ctaagcctcc gcctcctctt cctccatccg1320
ccccgtctct cccccttgaa cctcctcgtt cgaccccgcc tcgatcctcc ctttatccag1380
ccctcactcc ttctctaggc gccggaatta gatctctcga ggttaacgaa ttctaccggg1440
taggggaggc gcttttccca aggcagtctg gagcatgcgc tttagcagcc ccgctgggca1500
cttggcgcta cacaagtggc ctctggcctc gcacacattc cacatccacc ggtaggcgcc1560
aaccggctcc gttctttggt ggccccttcg cgccaccttc tactcctccc ctagtcagga1620
agttcccccc cgccccgcag ctcgcgtcgt gcaggacgtg acaaatggaa gtagcacgtc1680
tcactagtct cgtgcagatg gacagcaccg ctgagcaatg gaagcgggta ggcctttggg1740
gcagcggcca atagcagctt tgctccttcg ctttctgggc tcagaggctg ggaaggggtg1800
ggtccggggg cgggctcagg ggcgggctca ggggcggggc gggcgcccga aggtcctccg1860
gaggcccggc attctgcacg cttcaaaagc gcacgtctgc cgcgctgttc tcctcttcct1920
cattctccgg gcctttcgac ctgcagccca agccaccatg gctctgcctg ttacagctct1980
gctgctgcct ctggctctgc ttctgcatgc cgccagacct gacatccaga tgacacagag2040
ccctagcagc ctgtctgcct ctctcggcgg aaaagtgacc ctgacatgca aggccagcca2100
ggacatcaac aagtatatcg cctggtatca gcacaagccc ggcaagggac ctagactgct2160
gatccactac accagcacac tgcagcctgg catccccagc agattttctg gcagcggctc2220
cggcagagac tacagcttca gcatcagcaa cctggaacct gaggacgtgg ccacctacta2280
ctgcctgcag tacgacaacc tgcggacctt tggcggcgga acaaagctgg aaatcaagcg2340
gacagatggc ggaggcggat caggcggcgg aggaagcggt ggcggaggat ctcaagttca2400
gctgcaacag cctggcgccg agcttgtgaa acctggcgcc tctgtgaaga tgagctgcaa2460
ggcctccggc tacaccttca ccagatactg gatcacctgg gtcaagcaga ggcctggaca2520
gggactcgag tggatcggcg atatctatcc tggctccggc ttcaccaagt acaacgagaa2580
gttcaagagc aaggccacac tgaccgtgga caccagcagc agcacagcct acatgcagct2640
gtctagcctg accagcgagg acagcgccgt gtactactgt gctagagaag gcggcaacta2700
ctggtacttc gacgtgtggg gcaccggcac cacagtgaca gttagttctg cggccgcggc2760
cgcattcgtg ccggtcttcc tgccagcgaa gcccaccacg acgccagcgc cgcgaccacc2820
aacaccggcg cccaccatcg cgtcgcagcc cctgtccctg cgcccagagg cgtgccggcc2880
agcggcgggg ggcgcagtgc acacgagggg gctggacttc gcctgtgata tctacatctg2940
ggcgcccttg gccgggactt gtggggtcct tctcctgtca ctggttatca ccctttactg3000
caaccacagg aacaggagta agaggagcag gctcctgcac agtgactaca tgaacatgac3060
tccccgccgc cccgggccca cccgcaagca ttaccagccc tatgccccac cacgcgactt3120
cgcagcctat cgctcccgtt tctctgttgt taaacggggc agaaagaagc tcctgtatat3180
attcaaacaa ccatttatga gaccagtaca aactactcaa gaggaagatg gctgtagctg3240
ccgatttcca gaagaagaag aaggaggatg tgaactgaga gtgaagttca gcaggagcgc3300
agacgccccc gcgtaccagc agggccagaa ccagctctat aacgagctca atctaggacg3360
aagagaggag tacgatgttt tggacaagag acgtggccgg gaccctgaga tggggggaaa3420
gccgagaagg aagaaccctc aggaaggcct gtacaatgaa ctgcagaaag ataagatggc3480
ggaggcctac agtgagattg ggatgaaagg cgagcgccgg aggggcaagg ggcacgatgg3540
cctttaccag ggtctcagta cagccaccaa ggacacctac gacgcccttc acatgcaggc3600
cctgccccct cgcggatccg gagccacgaa cttctctctg ttaaagcaag caggagacgt3660
ggaagaaaac cccggtccta tgcttctcct ggtgacaagc cttctgctct gtgagttacc3720
acacccagca ttcctcctga tcccacgcaa agtgtgtaac ggaataggta ttggtgaatt3780
taaagactca ctctccataa atgctacgaa tattaaacac ttcaaaaact gcacctccat3840
cagtggcgat ctccacatcc tgccggtggc atttaggggt gactccttca cacatactcc3900
tcctctggat ccacaggaac tggatattct gaaaaccgta aaggaaatca cagggttttt3960
gctgattcag gcttggcctg aaaacaggac ggacctccat gcctttgaga acctagaaat4020
catacgcggc aggaccaagc aacatggtca gttttctctt gcagtcgtca gcctgaacat4080
aacatccttg ggattacgct ccctcaagga gataagtgat ggagatgtga taatttcagg4140
aaacaaaaat ttgtgctatg caaatacaat aaactggaaa aaactgtttg ggacctccgg4200
tcagaaaacc aaaattataa gcaacagagg tgaaaacagc tgcaaggcca caggccaggt4260
ctgccatgcc ttgtgctccc ccgagggctg ctggggcccg gaacccaggg actgcgtctc4320
ttgccggaat gtcagccgag gcagggaatg cgtggacaag tgcaaccttc tggagggtga4380
gccaagggag tttgtggaga actctgagtg catacagtgc cacccagagt gcctgcctca4440
ggccatgaac atcacctgca caggacgggg accagacaac tgtatccagt gtgcccacta4500
cattgacggc ccccactgcg tcaagacctg cccggcagga gtcatgggag aaaacaacac4560
cctggtctgg aagtacgcag acgccggcca tgtgtgccac ctgtgccatc caaactgcac4620
ctacggatgc actgggccag gtcttgaagg ctgtccaacg aatgggccta agatcccgtc4680
catcgccact gggatggtgg gggccctcct cttgctgctg gtggtggccc tggggatcgg4740
cctcttcatg ggatctggag ccacgaactt ctctctgtta aagcaagcag gagacgtgga4800
agaaaacccc ggtcctatgc tcgagggagt gcaggtggaa accatctccc caggagacgg4860
gcgcaccttc cccaagcgcg gccagacctg cgtggtgcac tacaccggga tgcttgaaga4920
tggaaagaaa gttgattcct cccgggacag aaacaagccc tttaagttta tgctaggcaa4980
gcaggaggtg atccgaggct gggaagaagg ggttgcccag atgagtgtgg gtcagagagc5040
caaactgact atatctccag attatgccta tggtgccact gggcacccag gcatcatccc5100
accacatgcc actctcgtct tcgatgtgga gcttctaaaa ctggaatctg gcggtggatc5160
cggagtcgac ggatttggtg atgtcggtgc tcttgagagt ttgaggggaa atgcagattt5220
ggcttacatc ctgagcatgg agccctgtgg ccactgcctc attatcaaca atgtgaactt5280
ctgccgtgag tccgggctcc gcacccgcac tggctccaac atcgactgtg agaagttgcg5340
gcgtcgcttc tcctcgctgc atttcatggt ggaggtgaag ggcgacctga ctgccaagaa5400
aatggtgctg gctttgctgg agctggcgca gcaggaccac ggtgctctgg actgctgcgt5460
ggtggtcatt ctctctcacg gctgtcaggc cagccacctg cagttcccag gggctgtcta5520
cggcacagat ggatgccctg tgtcggtcga gaagattgtg aacatcttca atgggaccag5580
ctgccccagc ctgggaggga agcccaagct ctttttcatc caggcctgtg gtggggagca5640
gaaagaccat gggtttgagg tggcctccac ttcccctgaa gacgagtccc ctggcagtaa5700
ccccgagcca gatgccaccc cgttccagga aggtttgagg accttcgacc agctggacgc5760
catatctagt ttgcccacac ccagtgacat ctttgtgtcc tactctactt tcccaggttt5820
tgtttcctgg agggacccca agagtggctc ctggtacgtt gagaccctgg acgacatctt5880
tgagcagtgg gctcactctg aagacctgca gtccctcctg cttagggtcg ctaatgctgt5940
ttcggtgaaa gggatttata aacagatgcc tggttgcttt aatttcctcc ggaaaaaact6000
tttctttaaa acatcagtcg actatccgta cgacgtacca gactacgcac tcgactaaac6060
aatcaacctc tggattacaa aatttgtgaa agattgactg gtattcttaa ctatgttgct6120
ccttttacgc tatgtggata cgctgcttta atgcctttgt atcatgctat tgcttcccgt6180
atggctttca ttttctcctc cttgtataaa tcctggttgc tgtctcttta tgaggagttg6240
tggcccgttg tcaggcaacg tggcgtggtg tgcactgtgt ttgctgacgc aacccccact6300
ggttggggca ttgccaccac ctgtcagctc ctttccggga ctttcgcttt ccccctccct6360
attgccacgg cggaactcat cgccgcctgc cttgcccgct gctggacagg ggctcggctg6420
ttgggcactg acaattccgt ggtgttgtcg gggaaatcat cgtcctttcc ttggctgctc6480
gcctgtgttg ccacctggat tctgcgcggg acgtccttct gctacgtccc ttcggccctc6540
aatccagcgg accttccttc ccgcggcctg ctgccggctc tgcggcctct tccgcgtctt6600
cgccttcgcc ctcagacgag tcggatctcc ctttgggccg cctccccgcc tatcgataaa6660
ataaaagatt ttatttagtc tccagaaaaa ggggggaatg aaagacccca cctgtaggtt6720
tggcaagcta gcttaagtaa cgccattttg caaggcatgg aaaatacata actgagaata6780
gagaagttca gatcaaggtt aggaacagag agacagcaga atatgggcca aacaggatat6840
ctgtggtaag cagttcctgc cccggctcag ggccaagaac agatggtccc cagatgcggt6900
cccgccctca gcagtttcta gagaaccatc agatgtttcc agggtgcccc aaggacctga6960
aatgaccctg tgccttattt gaactaacca atcagttcgc ttctcgcttc tgttcgcgcg7020
cttctgctcc ccgagctcaa taaaagagcc cacaacccct cactcggcgc gccagtcctc7080
cgatagactg cgtcgcccgg gtacccgtgt atccaataaa ccctcttgca gttgcatccg7140
acttgtggtc tcgctgttcc ttgggagggt ctcctctgag tgattgacta cccgtcagcg7200
ggggtctttc atgggtaaca gtttcttgaa gttggagaac aacattctga gggtaggagt7260
cgaatattaa gtaatcctga ctcaattagc cactgttttg aatccacata ctccaatact7320
cctgaaatag ttcattatgg acagcgcaga agagctgggg agaattaatt cgtaatcatg7380
gtcatagctg tttcctgtgt gaaattgtta tccgctcaca attccacaca acatacgagc7440
cggaagcata aagtgtaaag cctggggtgc ctaatgagtg agctaactca cattaattgc7500
gttgcgctca ctgcccgctt tccagtcggg aaacctgtcg tgccagctgc attaatgaat7560
cggccaacgc gcggggagag gcggtttgcg tattgggcgc tcttccgctt cctcgctcac7620
tgactcgctg cgctcggtcg ttcggctgcg gcgagcggta tcagctcact caaaggcggt7680
aatacggtta tccacagaat caggggataa cgcaggaaag aacatgtgag caaaaggcca7740
gcaaaaggcc aggaaccgta aaaaggccgc gttgctggcg tttttccata ggctccgccc7800
ccctgacgag catcacaaaa atcgacgctc aagtcagagg tggcgaaacc cgacaggact7860
ataaagatac caggcgtttc cccctggaag ctccctcgtg cgctctcctg ttccgaccct7920
gccgcttacc ggatacctgt ccgcctttct cccttcggga agcgtggcgc tttctcatag7980
ctcacgctgt aggtatctca gttcggtgta ggtcgttcgc tccaagctgg gctgtgtgca8040
cgaacccccc gttcagcccg accgctgcgc cttatccggt aactatcgtc ttgagtccaa8100
cccggtaaga cacgacttat cgccactggc agcagccact ggtaacagga ttagcagagc8160
gaggtatgta ggcggtgcta cagagttctt gaagtggtgg cctaactacg gctacactag8220
aaggacagta tttggtatct gcgctctgct gaagccagtt accttcggaa aaagagttgg8280
tagctcttga tccggcaaac aaaccaccgc tggtagcggt ggtttttttg tttgcaagca8340
gcagattacg cgcagaaaaa aaggatctca agaagatcct ttgatctttt ctacggggtc8400
tgacgctcag tggaacgaaa actcacgtta agggattttg gtcatgagat tatcaaaaag8460
gatcttcacc tagatccttt taaattaaaa atgaagtttt aaatcaatct aaagtatata8520
tgagtaaact tggtctgaca gttaccaatg cttaatcagt gaggcaccta tctcagcgat8580
ctgtctattt cgttcatcca tagttgcctg actccccgtc gtgtagataa ctacgatacg8640
ggagggctta ccatctggcc ccagtgctgc aatgataccg cgagacccac gctcaccggc8700
tccagattta tcagcaataa accagccagc cggaagggcc gagcgcagaa gtggtcctgc8760
aactttatcc gcctccatcc agtctattaa ttgttgccgg gaagctagag taagtagttc8820
gccagttaat agtttgcgca acgttgttgc cattgctaca ggcatcgtgg tgtcacgctc8880
gtcgtttggt atggcttcat tcagctccgg ttcccaacga tcaaggcgag ttacatgatc8940
ccccatgttg tgcaaaaaag cggttagctc cttcggtcct ccgatcgttg tcagaagtaa9000
gttggccgca gtgttatcac tcatggttat ggcagcactg cataattctc ttactgtcat9060
gccatccgta agatgctttt ctgtgactgg tgagtactca accaagtcat tctgagaata9120
gtgtatgcgg cgaccgagtt gctcttgccc ggcgtcaata cgggataata ccgcgccaca9180
tagcagaact ttaaaagtgc tcatcattgg aaaacgttct tcggggcgaa aactctcaag9240
gatcttaccg ctgttgagat ccagttcgat gtaacccact cgtgcaccca actgatcttc9300
agcatctttt actttcacca gcgtttctgg gtgagcaaaa acaggaaggc aaaatgccgc9360
aaaaaaggga ataagggcga cacggaaatg ttgaatactc atactcttcc tttttcaata9420
ttattgaagc atttatcagg gttattgtct catgagcgga tacatatttg aatgtattta9480
gaaaaataaa caaatagggg ttccgcgcac atttccccga aaagtgccac ctgacgtcta9540
agaaaccatt attatcatga cattaaccta taaaaatagg cgtatcacga ggccctttcg9600
tctcgcgcgt ttcggtgatg acggtgaaaa cctctgacac atgcagctcc cggagacggt9660
cacagcttgt ctgtaagcgg atgccgggag cagacaagcc cgtcagggcg cgtcagcggg9720
tgttggcggg tgtcggggct ggcttaacta tgcggcatca gagcagattg tactgagagt9780
gcaccatatg cggtgtgaaa taccgcacag atgcgtaagg agaaaatacc gcatcaggcg9840
ccattcgcca ttcaggctgc gcaactgttg ggaagggcga tcggtgcggg cctcttcgct9900
attacgccag ctggcgaaag ggggatgtgc tgcaaggcga ttaagttggg taacgccagg9960
gttttcccag tcacgacgtt gtaaaacgac ggcgcaagga atggtgcatg caaggagatg 10020
gcgcccaaca gtcccccggc cacggggcct gccaccatac ccacgccgaa acaagcgctc 10080
atgagcccga agtggcgagc ccgatcttcc ccatcggtga tgtcggcgat ataggcgcca 10140
gcaaccgcac ctgtggcgcc ggtgatgccg gccacgatgc gtccggcgta gaggcgatta 10200
gtccaatttg ttaaagacag gatatcagtg gtccaggctc tagttttgac tcaacaatat 10260
caccagctga agcctataga gtacgagcca tagataaaat aaaagatttt atttagtctc 10320
cagaaaaagg ggggaa 10336
- 基于D-A型酞菁共晶的双极性半导体材料及其制备方法和应用
- 一种酞菁共晶及其制备方法和应用
