个性化基因修饰肿瘤DC疫苗的制备方法
文献发布时间:2023-06-19 11:30:53
技术领域
本发明涉及一种肿瘤DC疫苗及其制备方法,属于基因工程技术领域。
背景技术
癌症是全球第二大死亡原因。在全球范围内,每6例死亡中就有1例死于癌症。对于早期没有扩散到淋巴结和没有转移的癌症,手术切除是一种非常有效的治疗方法。然而,对于更严重的病例,使用标准的、非特异性的癌症治疗,比如化疗和放疗,会对体内的健康细胞产生影响并形成毒性。由于癌症的分子异质性,通常只有不到25%的接受治疗的个体从批准的治疗中获益。
目前以DC为基础的肿瘤疫苗策略,主要有5种。一是通过肿瘤细胞或肿瘤细胞提取物冲击DC,得到肿瘤DC疫苗,如CN102793912A、CN104815323A。二是利用肿瘤抗原肽冲击DC,得到肿瘤DC疫苗。三是利用水溶性蛋白质作为抗原冲击DC,得到肿瘤DC疫苗。四是采用肿瘤抗原DNA/RNA负载到DC细胞中,得到肿瘤DC疫苗,如CN107583042A、CN102153658A。五是利用肿瘤抗原在体内冲击DC,得到肿瘤DC疫苗。由于每个患者肿瘤的特异性抗原尚未明确且易发生抗原位点变异,另外肿瘤细胞株在体外传代过程中表面抗原可能发生改变,DC细胞不能将该肿瘤的所有抗原有效呈递给效应T细胞,从而限制了效应T细胞在体内的抗肿瘤效应。
目前肿瘤抗原DNA/RNA冲击DC,主要利用的是肿瘤中已经公开的相关抗原,而没有针对每个患者的肿瘤特异性。基于患者个性化治疗的个体化药物被认为是解决药物研发创新低疗效、高成本的潜在解决方案。
发明内容
针对现有技术存在的不足,本发明提供一种高效的个体化肿瘤DC疫苗,实现以下发明目的:
制备针对患者肿瘤的个体化特异性疫苗,与免疫细胞共培养后,IFN-γ的释放量、CD8
为解决上述技术问题,本发明采取以下技术方案:
个性化基因修饰肿瘤DC疫苗的制备方法,所述制备方法,包括筛选肿瘤患者的抗原位点;所述筛选肿瘤患者的抗原位点,包括全基因的突变位点的筛选和肿瘤标志物基因抗原表位的筛选;将全基因的突变位点的筛选得到的突变多肽、肿瘤标志物基因抗原表位的筛选得到的肿瘤抗原表位进行连接,得到突变肽,在突变肽的N端连接MHC I类信号分子,在C端连接MHC I类转运信号MITD,MITD通过T2A与增强型细胞因子GM-CSF连接,得到重组抗原肽片段;将全基因的突变位点的筛选得到的突变多肽、肿瘤标志物基因抗原表位的筛选得到的肿瘤抗原表位进行连接时,肿瘤抗原表位在前,突变多肽在后进行连接。
以下是对上述技术方案的进一步改进:
所述全基因的突变位点的筛选,穿刺抽取癌症患者的组织,进行全基因测序,根据全基因测序结果筛选出与该癌症患者所患癌相关联的错义突变的突变位点,查询各突变位点的上下游序列,得到突变多肽;
所述突变多肽的长度为25-35个氨基酸,突变位点位于其对应的突变多肽的氨基酸序列的1/3到2/3处。
所述肿瘤标志物基因抗原表位的筛选,对该癌症患者自身携带的其所患癌的肿瘤标志物基因对应的氨基酸序列,与病人的HLAI、HLAII进行匹配,根据匹配结果,选择%Rank值小于0.5且BindLevel为SW的肽段,筛选得到的肽段,如果有氨基酸重合的,将重合的肽段整合成一个肽段,如有相邻的两个肽段,间隔10个氨基酸以内,则将相邻的这两个肽段和中间间隔的氨基酸一起整合成一个肽段,得到肿瘤抗原表位。
所述肿瘤标志物基因对应的氨基酸序列,与病人的HLAI、HLAII进行匹配,使用的软件为NetMHCpan、SYFPEITHI或免疫表位数据库。
所述DC疫苗的制备方法还包括重组表达载体的构建、腺相关病毒的包装、肿瘤DC疫苗制备;
所述重组表达载体的构建,将表达重组抗原肽片段的目的基因序列与载体连接,得到重组表达载体;
所述腺相关病毒的包装,将重组表达载体转染HEK293细胞,转染后使细胞充分裂解,得到腺相关病毒;
所述肿瘤DC疫苗制备,将腺相关病毒感染imDC,得到肿瘤DC疫苗。
本发明基于对病人特定的癌症突变的识别。具体地说,本发明涉及基于个体化免疫治疗方法的基因组,优选外显子组或转录组测序,旨在免疫治疗地靶向癌症中的多个个体突变。使用下一代测序(NGS)可以快速和经济有效地识别患者特定的癌症突变。
本发明涉及提供在癌症患者中诱导有效和特异性免疫反应的个体化重组癌症疫苗并潜在靶向原发肿瘤和肿瘤转移的有效方法。所述疫苗能够诱导或促进毒性T细胞活性,针对以呈现具有I类和II类MHC的一个或多个癌症表达抗原为特征的癌症疾病。
本发明中的突变肽来自于患者中能够被肿瘤特异性CTL系识别的抗原表位,用于制备肿瘤疫苗。这种疫苗代表了一种免疫原性成分,可用于癌症患者,从而诱导CTL特异性识别肿瘤细胞表达的突变序列,并导致癌细胞死亡。因此,该疫苗组合物包括由本发明所述方法识别的肿瘤特异性新抗原所对应的突变多肽。
本发明中DC疫苗包含至少一个突变肽序列,优选包含多个突变肽序列,如2、3、4、5、6、7、8、9、10或更多,突变肽抗原能够在疫苗制备中被选中,是由于它们能够与接受疫苗的肿瘤患者表达的MHC抗原结合。这些多肽能够被肿瘤特异性的CTL系识别或可以诱导肿瘤特异性的CTL系。
在本发明中,编码各种突变肽序列的载体还可以编码所包含的一种蛋白质以增强免疫原性。例如有益增强免疫反应的蛋白质或多肽,包括细胞因子(如IL2、IL12、GM-CSF),细胞因子诱导分子或共刺激分子。为了提高抗原呈递效率,在N端将MHC I类信号分子与免疫原性抗原连接,并将MHC I类转运信号(MITD,MHC I类跨膜和胞浆结构域)粘附在免疫原性抗原的C端。
上述技术方案取得以下有益效果:
本发明提供了一种高效的个性化肿瘤DC疫苗,与免疫细胞共培养后,IFN-γ的释放量、CD8
附图说明
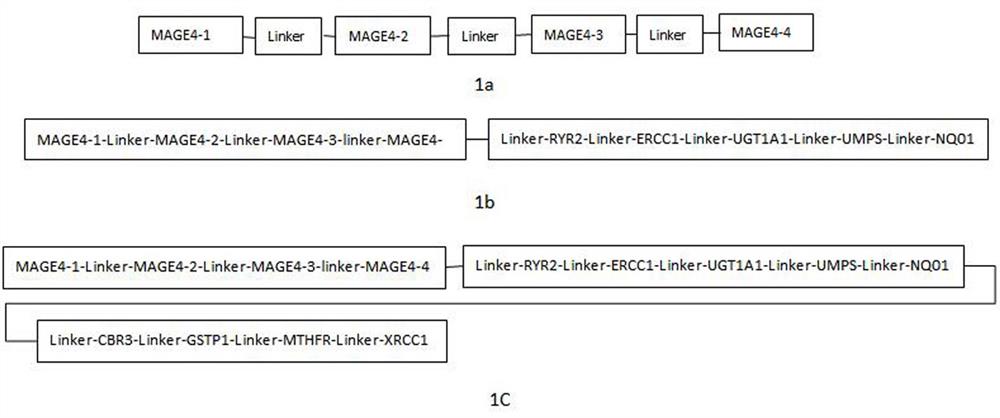
图1为本发明实施例1所述三种突变肽的设计结构;
其中1a为EC-MAGE的设计结构;
1b为EC-LMJ的设计结构;
1c为EC-13的设计结构;
图2为本发明基因序列的设计结构;
图3为双酶切验证质粒的电泳图;
图4为pAAV-EC-LMJ感染DC细胞的免疫荧光图;
图5为流式细胞术检测DC细胞标志物的流式图;
其中5a为流式细胞术检测DC细胞标志物CD80的流式图;
5b为流式细胞术检测DC细胞标志物CD83的流式图;
5c为流式细胞术检测DC细胞标志物CD86的流式图;
图6为pAAV-EC-LMJ感染DC细胞后GFP的表达率的流式图;
图7为免疫细胞中CD8
图8为免疫细胞中CD4
具体实施方式
实施例1 筛选食道癌患者的抗原位点
以食道癌患者为例,穿刺抽取组织,送到华大基因利用二代测序方法进行全基因测序,部分突变位点如下表1所示。碱基的突变不一定会导致氨基酸的突变,根据全基因测序结果筛选出与食道癌相关联的错义突变的5个突变位点,在NCBI上查询上下游序列,选择25-35个氨基酸,使其突变位点位于氨基酸序列的1/3到2/3处。本发明中,选择的突变多肽为RYR2、ERCC1、UGT1A1、UMPS、NQ01。其中RYR2的突变位点位于该基因氨基酸序列的1/2(31个氨基酸第16位)处,ERCC1的突变位点位于该基因氨基酸序列的13/29(29个氨基酸第13位)处,UGT1A1的突变位点位于该基因氨基酸序列的1/2(34个氨基酸第17位)处,UMPS的突变位点位于该基因氨基酸序列的9/17(34个氨基酸第18位)处,NQ01的突变位点位于该基因氨基酸序列的1/2(30个氨基酸第15位)处。
同时对食道癌的肿瘤标志物基因MAGE4对应的氨基酸序列利用NetMHCpan 4.1软件与病人的HLAI、HLAII进行匹配,根据匹配结果,选择%Rank值小于0.5且BindLevel为SW的肽段,这种肽段与MHC I型分子亲和力较强,筛选得到的肽段,如果有氨基酸重合的,将重合的肽段整合成一个肽段,如有相邻的两个肽段,间隔10个氨基酸以内,则将相邻的这两个肽段和中间间隔的氨基酸一起整合成一个肽段,一个肽段作为一个肿瘤新抗原表位。
本发明中,选择了4个不同的肿瘤新抗原表位,分别为MAGE4-1、MAGE4-2、MAGE4-3、MAGE4-4。
将筛选出的9个抗原肽利用常规的Linker连接,Linker的氨基酸序列为GGSGGGGSGG,如图1所示。
在突变肽的N端连接MHC I类信号分子,在C端连接MHC I类转运信号MITD。同时利用T2A与增强型细胞因子GM-CSF连接,如图2所示。本发明中的核苷酸全长序列为SEQ IDNO.1,氨基酸序列为SEQ ID NO.4,其基因序列命名为EC-LMJ,送到南京金斯瑞公司合成。
以肿瘤标志物基因MAGE4筛选出的4个抗原表位连接成的突变肽作为其中一种,命名为EC-MAGE,其核苷酸全长序列为SEQ ID NO.2,氨基酸序列为SEQ ID NO.5。根据检测的突变基因位点,可以选择13个抗原表位连接成的突变肽作为其中一种,是在EC-LMJ的基础上再添加CBR3、GSTP1、MTHFR、XRCC1四个抗原位点,命名为EC-13,其核苷酸全长序列为SEQID NO.3,氨基酸序列为SEQ ID NO.6,结构如图1所示,送到南京金斯瑞公司合成。
表1部分突变位点的检测结果
实施例2 重组表达载体的构建
以EC-LMJ序列为例,构建重组腺相关病毒载体,其余两种序列构建方法一样。将委托合成的EC-LMJ序列进行BamHI、EcoRI双酶切,获得具有粘性末端的目的片段。同时将载体pAAV-IRES(购自Agilent公司)进行BamHI、EcoRI双酶切,获得线性化载体片段,用T4连接酶连接目的片段和线性化载体片段,转化到E.coli(Top10),经酶切验证后(见图3)和测序验证后,使用OMEGA公司的去内毒素质粒提取试剂盒提取质粒,获得重组表达载体pAAV-EC-LMJ、pAAV-EC-MAGE、pAAV-EC-13。本发明中提取到的重组表达载体pAAV-EC-LMJ的浓度为1.02mg/mL、pAAV-EC-MAGE的浓度为0.85mg/mL、pAAV-EC-13的浓度为0.94mg/mL。
实施例3 腺相关病毒的包装及滴度测定
1)包装细胞系的复苏
本发明使用的包装细胞系为HEK293细胞。从液氮罐中取出冻存的HEK293细胞,迅速丢入37℃水浴锅中并快速晃动,尽量在1~2 min内使细胞溶液完全溶解。将细胞溶液转移到50mL离心管中,加入生理盐水清洗DMSO,混匀后离心,1500 rpm 5 min。去掉上清,加入5mL新鲜的高糖DMEM(含10% FBS)培养基重悬细胞,转入T75瓶中,每个瓶中补足到10mL高糖DMEM(含10% FBS)培养基。将培养瓶平稳放入37℃、5 %CO
2)腺相关病毒的包装及滴度测定
以pAAV-EC-LMJ为例,其余两种重组腺相关病毒的包装及滴度测定方法一样,不再重复。
当传代后HEK293细胞铺板达到80%-90%时,用于转染。
转染试剂的准备:在5mL离心管中,分别配制A管与B管试剂(Tube A and Tube B)
配好后,放置5min,然后将A管缓慢加入B管,混合均匀。室温放置20min,形成脂质体-DNA混合物。将混合物加入培养瓶中,轻微混匀。置于37℃,5%CO
实施例4 肿瘤DC疫苗制备
采集该食道癌患者外周静脉血50mL,加入50mL生理盐水进行稀释,在50mL离心管里先加入淋巴细胞分离液(购自天津灏阳生物科技有限公司,LTS10770125)20mL,然后缓慢加入25mL上述稀释后的血液,两者明显分层,900g离心25min。液面分为四层,从上到下依次为血浆、白细胞层、分离液层、红细胞层,吸取中间的白细胞层,加入DC培养基。37℃,5% CO
取出制备好的pAAV-EC-LMJ病毒,以MOI=100感染上述imDC,感染8-12h后,用PBS洗涤细胞2-3次,加入成熟因子TNF-α(终浓度为10ng/mL)继续培养诱导得到成熟的DC细胞(mDC),通过免疫荧光显微镜观察DC的感染情况(如图4)。流式细胞仪分析DC细胞成熟标志CD80、CD83、CD86的表达情况和腺相关病毒感染DC细胞的效率。如图5和6所示,本发明中,DC细胞CD80的表达率为75.1%,CD83的表达率为70.7%,CD86的表达率为70.3%,pAAV-EC-LMJ感染DC细胞后GFP的表达率为57.9%。
以相同方法制备其余两种肿瘤DC疫苗,其中pAAV-EC-MAGE感染DC细胞后GFP的表达率为63.5%,pAAV-EC-13感染DC细胞后GFP的表达率为50.8%。
实施例5 肿瘤DC疫苗对免疫细胞的活化及增殖影响
1)免疫细胞的培养
采集患者30mL外周血,用TBD样本密度分离液(购自天津灏洋华科生物),分离外周血单个核细胞。用免疫细胞培养基(购自CORNING公司,88-551-CM)诱导培养24小时后,加入1500IU/mL的重组白细胞介素2(购自沈阳三生制药)、50ng/mL的OKT-3和5%的患者自体血浆诱导继续培养。
每隔三天倍比加液,培养至第6天,流式细胞术检测T细胞中的CD8
2)肿瘤DC疫苗与免疫细胞联合培养
实验分组为:
A组、免疫细胞
B组、免疫细胞+普通DC细胞
C组、免疫细胞+DC-EC-MAGE
D组、免疫细胞+DC-EC-LMJ
E组、免疫细胞+DC-EC-13
B-E实验组中免疫细胞和DC细胞的数量为10:1,将两种细胞在免疫细胞培养基(购自CORNING的KBM551淋巴细胞无血清培养基)中,37℃、5%CO
序列表
<110> 山东兴瑞生物科技有限公司
<120> 个性化基因修饰肿瘤DC疫苗的制备方法
<130> 2021
<160> 6
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1854
<212> DNA
<213> Homo sapiens
<400> 1
atggcggtca tggcgccccg aaccctcctc ctgctactct cgggggccct ggccctgacc 60
cagacctggg cgcagggagc ctctgcctta cccactacca tcagcttcac ttgctgggga 120
ggatctggag gtggaggttc aggaggattc cgagaagcac tcagtaacaa ggtggatgag 180
ttggctcatt ttctgctccg caagtatcga gccaaggagc tggtcacaaa ggcagaagga 240
ggatctggag gtggaggttc aggaggaatc ttcggcaaag cctccgagtc cctgaagatg 300
atctttggca ttgacgtgaa ggaagtggac cccgccagca acacctacac ccttgtcgga 360
ggatctggag gtggaggttc aggaggaggc agtaatcctg cgcgctatga gttcctgtgg 420
ggtccaaggg ctctggctga aaccagctat gtgaaagtcc tggagcatgt ggtcagggtc 480
aatgcaagag ttcgcattgc ctacccatcc ctgcgtgaag cagctttgtt aggaggatct 540
ggaggtggag gttcaggagg actcaaattt tttgacatgt tcttaaaact aaaggatttg 600
acgtcgtgtg atacttttaa agaatatgac cccgatggca agggagtcat ttccggagga 660
tctggaggtg gaggttcagg aggattcgtc cctccccaga ggggcaatcc cgtactgaag 720
tgggtgcgca atgtgccctg ggaatttggc gacgtaattc ccgactatgt gggaggatct 780
ggaggtggag gttcaggagg acatgaaata gttgtcctag cacctgacgc ctcgttgtac 840
atcagagaca gagcatttta caccttgaag acgtaccctg tgccattcca aagggaggat 900
gtgggaggat ctggaggtgg aggttcagga ggaagagtga agaggtttat tcaggagaat 960
gtctttgtgg cagcgaatca taatgcttct cccctttcta taaaggaagc acccaaagaa 1020
ctcagcttcg gtgcaggagg atctggaggt ggaggttcag gaggacagag tggcattctg 1080
catttctgtg gcttccaagt cttagaatct caactgacat atagcattgg gcacactcca 1140
gcagacgccc gaattggagg atctggaggt ggaggttcag gaggagtggg catcattgct 1200
ggcctggttc tccttggagc tgtgatcact ggagctgtgg tcgctgccgt gatgtggagg 1260
aggaagagct cagatagaaa aggagggagt tacactcagg ctgcaagcag tgacagtgcc 1320
cagggctctg atgtgtccct cacagcttgt aaagtgggct ccggcgaggg caggggaagt 1380
cttctaacat gcggggacgt ggaggaaaat cccggcccaa tgtggctgca gagcctgctg 1440
ctcttgggca ctgtggcctg cagcatctct gcacccgccc gctcgcccag ccccagcaca 1500
cagccctggg agcatgtgaa tgccatccag gaggcccggc gtctcctgaa cctgagtaga 1560
gacactgctg ctgagatgaa tgaaacagta gaagtcatct cagaaatgtt tgacctccag 1620
gagccgacct gcctacagac ccgcctggag ctgtacaagc agggcctgcg gggcagcctc 1680
accaagctca agggcccctt gaccatgatg gccagccact acaaacagca ctgccctcca 1740
accccggaaa cttcctgtgc aacccagatt atcacctttg aaagtttcaa agagaacctg 1800
aaggactttc tgcttgtcat cccctttgac tgctgggagc cagtccagga gtga 1854
<210> 2
<211> 1230
<212> DNA
<213> Homo sapiens
<400> 2
atggcggtca tggcgccccg aaccctcctc ctgctactct cgggggccct ggccctgacc 60
cagacctggg cgcagggagc ctctgcctta cccactacca tcagcttcac ttgctgggga 120
ggatctggag gtggaggttc aggaggattc cgagaagcac tcagtaacaa ggtggatgag 180
ttggctcatt ttctgctccg caagtatcga gccaaggagc tggtcacaaa ggcagaagga 240
ggatctggag gtggaggttc aggaggaatc ttcggcaaag cctccgagtc cctgaagatg 300
atctttggca ttgacgtgaa ggaagtggac cccgccagca acacctacac ccttgtcgga 360
ggatctggag gtggaggttc aggaggaggc agtaatcctg cgcgctatga gttcctgtgg 420
ggtccaaggg ctctggctga aaccagctat gtgaaagtcc tggagcatgt ggtcagggtc 480
aatgcaagag ttcgcattgc ctacccatcc ctgcgtgaag cagctttgtt aggaggatct 540
ggaggtggag gttcaggagg agtgggcatc attgctggcc tggttctcct tggagctgtg 600
atcactggag ctgtggtcgc tgccgtgatg tggaggagga agagctcaga tagaaaagga 660
gggagttaca ctcaggctgc aagcagtgac agtgcccagg gctctgatgt gtccctcaca 720
gcttgtaaag tgggctccgg cgagggcagg ggaagtcttc taacatgcgg ggacgtggag 780
gaaaatcccg gcccaatgtg gctgcagagc ctgctgctct tgggcactgt ggcctgcagc 840
atctctgcac ccgcccgctc gcccagcccc agcacacagc cctgggagca tgtgaatgcc 900
atccaggagg cccggcgtct cctgaacctg agtagagaca ctgctgctga gatgaatgaa 960
acagtagaag tcatctcaga aatgtttgac ctccaggagc cgacctgcct acagacccgc 1020
ctggagctgt acaagcaggg cctgcggggc agcctcacca agctcaaggg ccccttgacc 1080
atgatggcca gccactacaa acagcactgc cctccaaccc cggaaacttc ctgtgcaacc 1140
cagattatca cctttgaaag tttcaaagag aacctgaagg actttctgct tgtcatcccc 1200
tttgactgct gggagccagt ccaggagtga 1230
<210> 3
<211> 2391
<212> DNA
<213> Homo sapiens
<400> 3
atggcggtca tggcgccccg aaccctcctc ctgctactct cgggggccct ggccctgacc 60
cagacctggg cgcagggagc ctctgcctta cccactacca tcagcttcac ttgctgggga 120
ggatctggag gtggaggttc aggaggattc cgagaagcac tcagtaacaa ggtggatgag 180
ttggctcatt ttctgctccg caagtatcga gccaaggagc tggtcacaaa ggcagaagga 240
ggatctggag gtggaggttc aggaggaatc ttcggcaaag cctccgagtc cctgaagatg 300
atctttggca ttgacgtgaa ggaagtggac cccgccagca acacctacac ccttgtcgga 360
ggatctggag gtggaggttc aggaggaggc agtaatcctg cgcgctatga gttcctgtgg 420
ggtccaaggg ctctggctga aaccagctat gtgaaagtcc tggagcatgt ggtcagggtc 480
aatgcaagag ttcgcattgc ctacccatcc ctgcgtgaag cagctttgtt aggaggatct 540
ggaggtggag gttcaggagg actcaaattt tttgacatgt tcttaaaact aaaggatttg 600
acgtcgtgtg atacttttaa agaatatgac cccgatggca agggagtcat ttccggagga 660
tctggaggtg gaggttcagg aggattcgtc cctccccaga ggggcaatcc cgtactgaag 720
tgggtgcgca atgtgccctg ggaatttggc gacgtaattc ccgactatgt gggaggatct 780
ggaggtggag gttcaggagg acatgaaata gttgtcctag cacctgacgc ctcgttgtac 840
atcagagaca gagcatttta caccttgaag acgtaccctg tgccattcca aagggaggat 900
gtgggaggat ctggaggtgg aggttcagga ggaagagtga agaggtttat tcaggagaat 960
gtctttgtgg cagcgaatca taatgcttct cccctttcta taaaggaagc acccaaagaa 1020
ctcagcttcg gtgcaggagg atctggaggt ggaggttcag gaggacagag tggcattctg 1080
catttctgtg gcttccaagt cttagaatct caactgacat atagcattgg gcacactcca 1140
gcagacgccc gaattggagg atctggaggt ggaggttcag gaggagacat ggatgggaaa 1200
gacagcatca ggactatgga ggagggggct gagacccctg tctacttggc cctcttgcct 1260
ccagatgcca ctggaggatc tggaggtgga ggttcaggag gacgtcacct gggccgcacc 1320
cttgggctct atgggaagga ccagcaggag gcagccctgg tggacatggt gaatgacggc 1380
gtggaggacc tccgctgcaa atacatcgga ggatctggag gtggaggttc aggaggaggg 1440
agctttgagg ctgacctgaa gcacttgaag gagaaggtgt ctgcgggagc cgatttcatc 1500
atcacgcagc ttttctttga ggctgacaca ttcttccgct ttggaggatc tggaggtgga 1560
ggttcaggag gaacgcacct catctgtgcc tttgccaaca cccccaagta cagccaggtc 1620
ctaggcctgg gaggccgcat cgtgcgtaag gagtgggtgc tggactgtca ccgcatgcgt 1680
cggcggctgc ccggaggatc tggaggtgga ggttcaggag gagtgggcat cattgctggc 1740
ctggttctcc ttggagctgt gatcactgga gctgtggtcg ctgccgtgat gtggaggagg 1800
aagagctcag atagaaaagg agggagttac actcaggctg caagcagtga cagtgcccag 1860
ggctctgatg tgtccctcac agcttgtaaa gtgggctccg gcgagggcag gggaagtctt 1920
ctaacatgcg gggacgtgga ggaaaatccc ggcccaatgt ggctgcagag cctgctgctc 1980
ttgggcactg tggcctgcag catctctgca cccgcccgct cgcccagccc cagcacacag 2040
ccctgggagc atgtgaatgc catccaggag gcccggcgtc tcctgaacct gagtagagac 2100
actgctgctg agatgaatga aacagtagaa gtcatctcag aaatgtttga cctccaggag 2160
ccgacctgcc tacagacccg cctggagctg tacaagcagg gcctgcgggg cagcctcacc 2220
aagctcaagg gccccttgac catgatggcc agccactaca aacagcactg ccctccaacc 2280
ccggaaactt cctgtgcaac ccagattatc acctttgaaa gtttcaaaga gaacctgaag 2340
gactttctgc ttgtcatccc ctttgactgc tgggagccag tccaggagtg a 2391
<210> 4
<211> 617
<212> PRT
<213> Homo sapiens
<400> 4
Met Ala Val Met Ala Pro Arg Thr Leu Leu Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Ala Leu Thr Gln Thr Trp Ala Gln Gly Ala Ser Ala Leu Pro Thr
20 25 30
Thr Ile Ser Phe Thr Cys Trp Gly Gly Ser Gly Gly Gly Gly Ser Gly
35 40 45
Gly Phe Arg Glu Ala Leu Ser Asn Lys Val Asp Glu Leu Ala His Phe
50 55 60
Leu Leu Arg Lys Tyr Arg Ala Lys Glu Leu Val Thr Lys Ala Glu Gly
65 70 75 80
Gly Ser Gly Gly Gly Gly Ser Gly Gly Ile Phe Gly Lys Ala Ser Glu
85 90 95
Ser Leu Lys Met Ile Phe Gly Ile Asp Val Lys Glu Val Asp Pro Ala
100 105 110
Ser Asn Thr Tyr Thr Leu Val Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Asn Pro Ala Arg Tyr Glu Phe Leu Trp Gly Pro Arg Ala
130 135 140
Leu Ala Glu Thr Ser Tyr Val Lys Val Leu Glu His Val Val Arg Val
145 150 155 160
Asn Ala Arg Val Arg Ile Ala Tyr Pro Ser Leu Arg Glu Ala Ala Leu
165 170 175
Leu Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Leu Lys Phe Phe Asp
180 185 190
Met Phe Leu Lys Leu Lys Asp Leu Thr Ser Cys Asp Thr Phe Lys Glu
195 200 205
Tyr Asp Pro Asp Gly Lys Gly Val Ile Ser Gly Gly Ser Gly Gly Gly
210 215 220
Gly Ser Gly Gly Phe Val Pro Pro Gln Arg Gly Asn Pro Val Leu Lys
225 230 235 240
Trp Val Arg Asn Val Pro Trp Glu Phe Gly Asp Val Ile Pro Asp Tyr
245 250 255
Val Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly His Glu Ile Val Val
260 265 270
Leu Ala Pro Asp Ala Ser Leu Tyr Ile Arg Asp Arg Ala Phe Tyr Thr
275 280 285
Leu Lys Thr Tyr Pro Val Pro Phe Gln Arg Glu Asp Val Gly Gly Ser
290 295 300
Gly Gly Gly Gly Ser Gly Gly Arg Val Lys Arg Phe Ile Gln Glu Asn
305 310 315 320
Val Phe Val Ala Ala Asn His Asn Ala Ser Pro Leu Ser Ile Lys Glu
325 330 335
Ala Pro Lys Glu Leu Ser Phe Gly Ala Gly Gly Ser Gly Gly Gly Gly
340 345 350
Ser Gly Gly Gln Ser Gly Ile Leu His Phe Cys Gly Phe Gln Val Leu
355 360 365
Glu Ser Gln Leu Thr Tyr Ser Ile Gly His Thr Pro Ala Asp Ala Arg
370 375 380
Ile Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Val Gly Ile Ile Ala
385 390 395 400
Gly Leu Val Leu Leu Gly Ala Val Ile Thr Gly Ala Val Val Ala Ala
405 410 415
Val Met Trp Arg Arg Lys Ser Ser Asp Arg Lys Gly Gly Ser Tyr Thr
420 425 430
Gln Ala Ala Ser Ser Asp Ser Ala Gln Gly Ser Asp Val Ser Leu Thr
435 440 445
Ala Cys Lys Val Gly Ser Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys
450 455 460
Gly Asp Val Glu Glu Asn Pro Gly Pro Met Trp Leu Gln Ser Leu Leu
465 470 475 480
Leu Leu Gly Thr Val Ala Cys Ser Ile Ser Ala Pro Ala Arg Ser Pro
485 490 495
Ser Pro Ser Thr Gln Pro Trp Glu His Val Asn Ala Ile Gln Glu Ala
500 505 510
Arg Arg Leu Leu Asn Leu Ser Arg Asp Thr Ala Ala Glu Met Asn Glu
515 520 525
Thr Val Glu Val Ile Ser Glu Met Phe Asp Leu Gln Glu Pro Thr Cys
530 535 540
Leu Gln Thr Arg Leu Glu Leu Tyr Lys Gln Gly Leu Arg Gly Ser Leu
545 550 555 560
Thr Lys Leu Lys Gly Pro Leu Thr Met Met Ala Ser His Tyr Lys Gln
565 570 575
His Cys Pro Pro Thr Pro Glu Thr Ser Cys Ala Thr Gln Ile Ile Thr
580 585 590
Phe Glu Ser Phe Lys Glu Asn Leu Lys Asp Phe Leu Leu Val Ile Pro
595 600 605
Phe Asp Cys Trp Glu Pro Val Gln Glu
610 615
<210> 5
<211> 409
<212> PRT
<213> Homo sapiens
<400> 5
Met Ala Val Met Ala Pro Arg Thr Leu Leu Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Ala Leu Thr Gln Thr Trp Ala Gln Gly Ala Ser Ala Leu Pro Thr
20 25 30
Thr Ile Ser Phe Thr Cys Trp Gly Gly Ser Gly Gly Gly Gly Ser Gly
35 40 45
Gly Phe Arg Glu Ala Leu Ser Asn Lys Val Asp Glu Leu Ala His Phe
50 55 60
Leu Leu Arg Lys Tyr Arg Ala Lys Glu Leu Val Thr Lys Ala Glu Gly
65 70 75 80
Gly Ser Gly Gly Gly Gly Ser Gly Gly Ile Phe Gly Lys Ala Ser Glu
85 90 95
Ser Leu Lys Met Ile Phe Gly Ile Asp Val Lys Glu Val Asp Pro Ala
100 105 110
Ser Asn Thr Tyr Thr Leu Val Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Asn Pro Ala Arg Tyr Glu Phe Leu Trp Gly Pro Arg Ala
130 135 140
Leu Ala Glu Thr Ser Tyr Val Lys Val Leu Glu His Val Val Arg Val
145 150 155 160
Asn Ala Arg Val Arg Ile Ala Tyr Pro Ser Leu Arg Glu Ala Ala Leu
165 170 175
Leu Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Val Gly Ile Ile Ala
180 185 190
Gly Leu Val Leu Leu Gly Ala Val Ile Thr Gly Ala Val Val Ala Ala
195 200 205
Val Met Trp Arg Arg Lys Ser Ser Asp Arg Lys Gly Gly Ser Tyr Thr
210 215 220
Gln Ala Ala Ser Ser Asp Ser Ala Gln Gly Ser Asp Val Ser Leu Thr
225 230 235 240
Ala Cys Lys Val Gly Ser Gly Glu Gly Arg Gly Ser Leu Leu Thr Cys
245 250 255
Gly Asp Val Glu Glu Asn Pro Gly Pro Met Trp Leu Gln Ser Leu Leu
260 265 270
Leu Leu Gly Thr Val Ala Cys Ser Ile Ser Ala Pro Ala Arg Ser Pro
275 280 285
Ser Pro Ser Thr Gln Pro Trp Glu His Val Asn Ala Ile Gln Glu Ala
290 295 300
Arg Arg Leu Leu Asn Leu Ser Arg Asp Thr Ala Ala Glu Met Asn Glu
305 310 315 320
Thr Val Glu Val Ile Ser Glu Met Phe Asp Leu Gln Glu Pro Thr Cys
325 330 335
Leu Gln Thr Arg Leu Glu Leu Tyr Lys Gln Gly Leu Arg Gly Ser Leu
340 345 350
Thr Lys Leu Lys Gly Pro Leu Thr Met Met Ala Ser His Tyr Lys Gln
355 360 365
His Cys Pro Pro Thr Pro Glu Thr Ser Cys Ala Thr Gln Ile Ile Thr
370 375 380
Phe Glu Ser Phe Lys Glu Asn Leu Lys Asp Phe Leu Leu Val Ile Pro
385 390 395 400
Phe Asp Cys Trp Glu Pro Val Gln Glu
405
<210> 6
<211> 796
<212> PRT
<213> Homo sapiens
<400> 6
Met Ala Val Met Ala Pro Arg Thr Leu Leu Leu Leu Leu Ser Gly Ala
1 5 10 15
Leu Ala Leu Thr Gln Thr Trp Ala Gln Gly Ala Ser Ala Leu Pro Thr
20 25 30
Thr Ile Ser Phe Thr Cys Trp Gly Gly Ser Gly Gly Gly Gly Ser Gly
35 40 45
Gly Phe Arg Glu Ala Leu Ser Asn Lys Val Asp Glu Leu Ala His Phe
50 55 60
Leu Leu Arg Lys Tyr Arg Ala Lys Glu Leu Val Thr Lys Ala Glu Gly
65 70 75 80
Gly Ser Gly Gly Gly Gly Ser Gly Gly Ile Phe Gly Lys Ala Ser Glu
85 90 95
Ser Leu Lys Met Ile Phe Gly Ile Asp Val Lys Glu Val Asp Pro Ala
100 105 110
Ser Asn Thr Tyr Thr Leu Val Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Ser Asn Pro Ala Arg Tyr Glu Phe Leu Trp Gly Pro Arg Ala
130 135 140
Leu Ala Glu Thr Ser Tyr Val Lys Val Leu Glu His Val Val Arg Val
145 150 155 160
Asn Ala Arg Val Arg Ile Ala Tyr Pro Ser Leu Arg Glu Ala Ala Leu
165 170 175
Leu Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Leu Lys Phe Phe Asp
180 185 190
Met Phe Leu Lys Leu Lys Asp Leu Thr Ser Cys Asp Thr Phe Lys Glu
195 200 205
Tyr Asp Pro Asp Gly Lys Gly Val Ile Ser Gly Gly Ser Gly Gly Gly
210 215 220
Gly Ser Gly Gly Phe Val Pro Pro Gln Arg Gly Asn Pro Val Leu Lys
225 230 235 240
Trp Val Arg Asn Val Pro Trp Glu Phe Gly Asp Val Ile Pro Asp Tyr
245 250 255
Val Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly His Glu Ile Val Val
260 265 270
Leu Ala Pro Asp Ala Ser Leu Tyr Ile Arg Asp Arg Ala Phe Tyr Thr
275 280 285
Leu Lys Thr Tyr Pro Val Pro Phe Gln Arg Glu Asp Val Gly Gly Ser
290 295 300
Gly Gly Gly Gly Ser Gly Gly Arg Val Lys Arg Phe Ile Gln Glu Asn
305 310 315 320
Val Phe Val Ala Ala Asn His Asn Ala Ser Pro Leu Ser Ile Lys Glu
325 330 335
Ala Pro Lys Glu Leu Ser Phe Gly Ala Gly Gly Ser Gly Gly Gly Gly
340 345 350
Ser Gly Gly Gln Ser Gly Ile Leu His Phe Cys Gly Phe Gln Val Leu
355 360 365
Glu Ser Gln Leu Thr Tyr Ser Ile Gly His Thr Pro Ala Asp Ala Arg
370 375 380
Ile Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Asp Met Asp Gly Lys
385 390 395 400
Asp Ser Ile Arg Thr Met Glu Glu Gly Ala Glu Thr Pro Val Tyr Leu
405 410 415
Ala Leu Leu Pro Pro Asp Ala Thr Gly Gly Ser Gly Gly Gly Gly Ser
420 425 430
Gly Gly Arg His Leu Gly Arg Thr Leu Gly Leu Tyr Gly Lys Asp Gln
435 440 445
Gln Glu Ala Ala Leu Val Asp Met Val Asn Asp Gly Val Glu Asp Leu
450 455 460
Arg Cys Lys Tyr Ile Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
465 470 475 480
Ser Phe Glu Ala Asp Leu Lys His Leu Lys Glu Lys Val Ser Ala Gly
485 490 495
Ala Asp Phe Ile Ile Thr Gln Leu Phe Phe Glu Ala Asp Thr Phe Phe
500 505 510
Arg Phe Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Thr His Leu Ile
515 520 525
Cys Ala Phe Ala Asn Thr Pro Lys Tyr Ser Gln Val Leu Gly Leu Gly
530 535 540
Gly Arg Ile Val Arg Lys Glu Trp Val Leu Asp Cys His Arg Met Arg
545 550 555 560
Arg Arg Leu Pro Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Val Gly
565 570 575
Ile Ile Ala Gly Leu Val Leu Leu Gly Ala Val Ile Thr Gly Ala Val
580 585 590
Val Ala Ala Val Met Trp Arg Arg Lys Ser Ser Asp Arg Lys Gly Gly
595 600 605
Ser Tyr Thr Gln Ala Ala Ser Ser Asp Ser Ala Gln Gly Ser Asp Val
610 615 620
Ser Leu Thr Ala Cys Lys Val Gly Ser Gly Glu Gly Arg Gly Ser Leu
625 630 635 640
Leu Thr Cys Gly Asp Val Glu Glu Asn Pro Gly Pro Met Trp Leu Gln
645 650 655
Ser Leu Leu Leu Leu Gly Thr Val Ala Cys Ser Ile Ser Ala Pro Ala
660 665 670
Arg Ser Pro Ser Pro Ser Thr Gln Pro Trp Glu His Val Asn Ala Ile
675 680 685
Gln Glu Ala Arg Arg Leu Leu Asn Leu Ser Arg Asp Thr Ala Ala Glu
690 695 700
Met Asn Glu Thr Val Glu Val Ile Ser Glu Met Phe Asp Leu Gln Glu
705 710 715 720
Pro Thr Cys Leu Gln Thr Arg Leu Glu Leu Tyr Lys Gln Gly Leu Arg
725 730 735
Gly Ser Leu Thr Lys Leu Lys Gly Pro Leu Thr Met Met Ala Ser His
740 745 750
Tyr Lys Gln His Cys Pro Pro Thr Pro Glu Thr Ser Cys Ala Thr Gln
755 760 765
Ile Ile Thr Phe Glu Ser Phe Lys Glu Asn Leu Lys Asp Phe Leu Leu
770 775 780
Val Ile Pro Phe Asp Cys Trp Glu Pro Val Gln Glu
785 790 795
- 个性化基因修饰肿瘤DC疫苗的制备方法
- 异源融合基因修饰的癌细胞/树突状细胞融合肿瘤疫苗及其制备方法
