用于调节RNA片段的人工RNA
文献发布时间:2024-04-18 20:01:23
技术领域
本发明涉及生物技术领域。特别地,本发明涉及如本发明所定义的人工RNA。更特别地,本发明涉及适合于通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构从而调节一个或多个RNA片段的人工RNA。
背景技术
正在进行扩展一般药物如抗病毒剂范围的大量研究和开发,以治疗目前尚未商业治疗的病原体或治疗癌症。而且,随着病毒和肿瘤细胞突变并产生抗药性的趋势,当务之急是开发和更新具有新疗法的抗肿瘤药和/或抗病毒药的研发管线。根据最近的市场报道(https://www.mordorintelligence.com/industries-reports/global-viral-drugs-market-industries),该市场可利用的机会是(i)对广谱抗病毒药物的需求不断增加和(ii)在新兴医药市场中抗病毒药物的取得。
目前批准和有效的抗病毒治疗包括与病毒生命周期所必需的病毒蛋白特异性相互作用的分子。开发这些治疗分子是昂贵且耗时的。此外,每个分子仅对特定病毒起作用。已进行了多种努力来开发用于靶向病毒RNA基因组的基于RNA的抗病毒治疗[Brice ASullenger and Smita Nair.From the RNA world to the clinic.Science 352(6292),1417-1420]。这些包括设计19个核苷酸长的microRNA(miRNA),这些miRNA与病毒RNA基因组特异性相互作用,导致它们随后被细胞RISC系统降解。尽管在细胞培养中成功,但其临床应用的主要缺点是快速出现并在RNA基因组中选择抑制与miRNA相互作用的突变。miRNA作为抗病毒分子的另一个缺点是它们的稳定性,因为它们通过细胞衰变机制自然降解。
由于开发针对病毒蛋白的新的治疗剂所需的成本和时间,基于RNA的疗法在最近几年中已经引发了许多关注。基于RNA的疗法已经能够处理不能用抗体和小分子方法治疗的靶标[Ling-Ling Chen.The biogenesis and emerging roles of circularRNAs.Nature Reviews Molecular Cell Biology 17,205–211(2016),doi:10.1038/nrm.2015.32]。因此,已创建了多家公司来开发基于RNA的疗法以治疗多种疾病。例如,Moderna公司正在开发针对SARS-CoV-2(Covid-19)引起的感染的基于mRNA的疫苗。这些公司关注反义、siRNA、适体和microRNA模拟物/抗-microRNA的应用。然而,这些分子(i)被快速降解,并且(ii)与基于蛋白质和抗体的疗法一样由病毒的高突变倾向性而出现抗药性。
本发明人特别关注了稳定的人工RNA如环状RNA的方法。环状RNA(circRNA)是天然存在于细胞中的前体mRNA的反向剪接产物(参见综述[Ling-Ling Chen.The biogenesisand emerging roles of circular RNAs.Nature Reviews Molecular Cell Biology 17,205–211(2016),doi:10.1038/nrm.2015.32])。以前被认为是无关的副产物,现在已证明执行几种功能,例如miRNA海绵和RBP(RNA结合蛋白)海绵。环状RNA没有末端。这是非常重要的,因为细胞核酸外切酶在细胞中的大多数mRNA降解途径需要5’或3’端来实现它们的降解功能。因此,circRNA是极其稳定的分子。尚未充分开发利用circRNA作为新的治疗平台的潜力。
WO2017/222911公开了用外源内含子产生的环状RNA刺激免疫应答或用内源内含子产生的环状RNA防止外来RNA的免疫识别的用途。
WO0061595公开了共价闭合的多重反义(CMAS)-寡核苷酸和带状反义(RiAS)-寡核苷酸,其中多重反义(CMAS)-寡核苷酸通过使用互补引物连接而构建形成闭合式,带状反义(RIA)-寡核苷酸由含有多重反义序列的两个环和连接这两个环的茎组成,通过使用两个5引物末端处的互补序列连接而构建。
WO 2013/162350 A2涉及由2个富含嘌呤的结构域组成的环状RNA的用途,所述2个富含嘌呤的结构域可靶向病毒富含嘧啶的区域以形成可阻断病毒复制的三螺旋。
CN 108165549 A涉及环状RNA作为micro-RNA海绵的用途。提供了具有内源circRNA的公知功能的人工制剂,其中多个部分互补区域靶向杂交时激活AGO2途径的成熟micro-RNA。
此外,越来越需要更好地理解构成许多病理学(例如癌症、病毒感染、自身免疫疾病、神经疾病、遗传病症等)的基础的分子机制。在大多数这些病理学中,RNA在潜在的机制中起作用。因此,需要允许研究涉及病理学相关分子机制的RNA的特定功能的工具。
本发明解决了上述问题,并且涉及允许调节RNA片段如病毒基因组的功能的工具,其使得多种应用成为可能。例如,使用本发明的工具(人工RNA),可以研究特定RNA片段的结构-功能关系,或特定RNA片段中的特定区域。此外,利用本发明的RNA,可以预防和/或治疗涉及RNA片段的疾病,例如病毒感染、癌症、免疫疾病、遗传病症等。例如,利用本发明的人工RNA,可以研究结合双链RNA基序的RBP(核糖体结合蛋白)。
发明内容
第一方面,本发明提供了适合于通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构的人工环状RNA,
(a)其中所述人工环状RNA包含150至800个核苷酸,优选200至600个核苷酸;
(b)其中所述人工环状RNA包含两个或更多个杂交区域,所述杂交区域:
(i)与包含于所述一个或多个RNA片段的一个或多个靶破坏结构中的至少一个靶杂交区域完全杂交;和
(ii)具有总共7至100个核苷酸,优选10至50个核苷酸;
(c)其中所述一个或多个靶破坏结构:
(i)包含至少发夹环,所述发夹环在未配对核苷酸的区域之后或之前;和
(ii)包含至少一个靶杂交区域,所述靶杂交区域包含在至少5个核苷酸、优选10个或更多个核苷酸的双链区之后或之前的至少2个核苷酸、优选3个或更多个核苷酸的单链区,其中所述至少一个靶杂交区域与所述人工环状RNA的两个或更多个杂交区域中的每一个完全杂交;和
(d)其中包含于所述人工环状RNA中的两个或更多个杂交区域的进一步特征在于,当与所述靶杂交区域杂交时,如通过RNAcofold所测量的,所述杂交区域和所述至少一个靶杂交区域之间的杂交能量比所述靶破坏区域的能量更负,从而破坏所述靶破坏结构。
优选地,根据第一方面或其任何实施方式的人工环状RNA包含6至20个杂交区域。优选地,所述杂交区域中的至少两个、优选全部能够与相同的靶杂交区域完全杂交。优选地,所述杂交区域中的至少两个、优选全部具有不同的核苷酸序列。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述2个或更多个杂交区域:
a)由大小至多20个核苷酸的非杂交区域分隔;或
b)未由非杂交区域分隔;或
c)是重叠的。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述一个或多个RNA片段选自mRNA、tRNA、rRNA、非编码RNA和病毒基因组RNA。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述一个或多个RNA片段为病毒基因组RNA。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述一个或多个RNA片段为正义单链病毒基因组RNA。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述病毒基因组RNA选自流感病毒、HAV、脊髓灰质炎病毒、柯萨奇病毒B、冠状病毒和鼻病毒(普通感冒)。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述病毒基因组RNA选自丙型肝炎病毒、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒和黄热病毒。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,其中所述病毒基因组RNA为来自严重急性呼吸综合征冠状病毒2(SARS-CoV-2)。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,其中所述至少一个或多个靶破坏结构选自由以下组成的组:
(a)来自丙型肝炎病毒的内部核糖体进入位点(IRES)结构域IV和结构域V、衣壳编码区发夹元件(cHP)或SL427,
(b)来自登革病毒的短茎环(sHP)或衣壳编码区发夹元件(cHP),
(c)来自基孔肯亚病毒的5’非翻译区(5’UTR)、重复序列元件(RSE)或重编码元件,
(d)来自西尼罗病毒的茎环III(SLIII),和/或
(e)来自冠状病毒,优选来自严重急性呼吸综合征冠状病毒2(SARS-CoV-2)的SL-2、复制位点、靶A、靶C、靶D。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述至少一个或多个靶破坏结构包含以下或由以下组成:来自丙型肝炎病毒的选自由SEQ ID NO.:76、77、78和/或79组成的列表中的靶破坏结构,并且其中这些靶破坏结构中的每一个的靶杂交区域分别选自SEQ ID NO.:25、26、27和28。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述至少一个或多个靶破坏结构包含以下或由以下组成:来自登革病毒的选自由SEQ ID NO.:29和/或30组成的列表中的靶破坏结构,并且其中这些靶破坏结构中的每一个的靶杂交区域分别选自SEQ IDNO.:29和30。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述至少一个或多个靶破坏结构包含以下或由以下组成:来自基孔肯亚病毒的选自由SEQ ID NO.:80、81和/或82组成的列表中的靶破坏结构,并且其中这些靶破坏结构中的每一个的靶杂交区域分别选自SEQ ID NO.:33、35和31。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述至少一个或多个靶破坏结构包含以下或由以下组成:来自西尼罗病毒的SEQ ID NO.:83的靶破坏结构,并且其中所述靶杂交区域为SEQ ID NO.:37。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述至少一个或多个靶破坏结构包含以下或由以下组成:来自严重急性呼吸综合征冠状病毒2(SARS-CoV-2)的选自由SEQ ID NO.:84、58、85、86和/或87组成的列表中的靶破坏结构,并且其中这些靶破坏结构中的每一个的靶杂交区域分别选自SEQ ID NO.:62、58、59、60和61。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述至少一个或多个靶破坏结构包含以下或由以下组成:来自登革病毒和丙型肝炎病毒的选自由SEQ ID NO.:30和/或79组成的列表中的靶破坏结构,并且其中这些靶破坏结构中的每一个的靶杂交区域选自SEQ ID NO.:30和28。
优选地,根据第一方面或其任何实施方式的人工环状RNA中,所述至少一个或多个靶破坏结构包含以下或由以下组成:来自登革病毒和西尼罗病毒的选自由SEQ ID NO.:30和/或83组成的列表中的靶破坏结构,并且其中这些靶破坏结构中的每一个的靶杂交区域分别选自SEQ ID NO.:30和37。
优选地,根据第一方面或其任何实施方式的人工环状RNA序列包含以下定义的核苷酸或优选由以下定义的核苷酸组成:SEQ ID NO.:2、3、4、5、6(针对丙型肝炎病毒);8、9、10(针对登革病毒);12、13、14、15、39(针对基孔肯亚病毒);16和17(针对丙型肝炎病毒和登革病毒的广谱活性);24和19(针对西尼罗病毒);21、22和23(针对登革病毒和西尼罗病毒的广谱活性);32(针对丙型肝炎病毒和登革病毒的广谱活性);36、38、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、65、66、67、68、69、70、71、72(针对严重急性呼吸综合征冠状病毒2)。
优选地,与所述两个或更多个杂交区域完全杂交的所述人工环状RNA的一个或多个靶杂交区域包含于SEQ ID NO.:1、SEQ ID NO.:7、SEQ ID NO.:11、SEQ ID NO.:34和/或SEQ ID NO.:20所定义的人工RNA中。
第二方面,本发明涉及一种组合物,其包含本发明的人工环状RNA。
第三方面,本发明涉及一种试剂盒,其包括本发明的人工环状RNA和/或组合物。
第四方面,本发明涉及用作药物,优选应用于预防和/或治疗病毒感染的方法中的本发明的人工RNA和/或组合物。优选地,所述病毒感染由以下病毒引起:丙型肝炎病毒、甲型肝炎病毒、脊髓灰质炎病毒、流感病毒、柯萨奇病毒B、鼻病毒(普通感冒)、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒、黄热病毒或冠状病毒,如SARS和/或MERS,优选SARS-CoV-2。
第六方面,本发明提供了筛选人工环状RNA的方法,所述人工环状RNA包含两个或更多个杂交区域,所述杂交区域能够通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构,其中所述靶破坏结构定义为:
i.第一区域,所述第一区域具有在未配对核苷酸的第二区域之后或之前的至少发夹环;和
ii.包含至少一个靶杂交区域,所述靶杂交区域包含在至少5个核苷酸、优选10个或更多个核苷酸的双链区之后或之前的至少2个核苷酸、优选3个或更多个核苷酸的单链区,
其中所述方法包括以下步骤:
a)鉴别作为总长度为7至100个核苷酸、优选10至50个核苷酸的那些区域的人工环状RNA的两个或更多个杂交区域,当与所述至少一个靶杂交区域杂交时,所述两个或更多个杂交区域与所述至少一个靶杂交区域之间的杂交能量比所述靶破坏结构的能量更负,从而破坏所述一个或多个靶破坏结构;其中包含于所述人工环状RNA中的两个或更多个杂交区域通过RNA反向折叠工具如NUPACK、RNAifold或MoiRNAiFold来鉴别;
b)将步骤a)中鉴别的人工环状RNA设计为包含能够破坏所述一个或多个靶破坏结构的两个或更多个杂交区域,其中所述人工环状RNA的长度为150至800个核苷酸,优选200至600个核苷酸;以及
c)任选地选择步骤b)中所设计的能够通过杂交破坏所述一个或多个靶破坏结构的人工环状RNA,以及任选地将所述人工环状RNA包装成产品。
附图说明
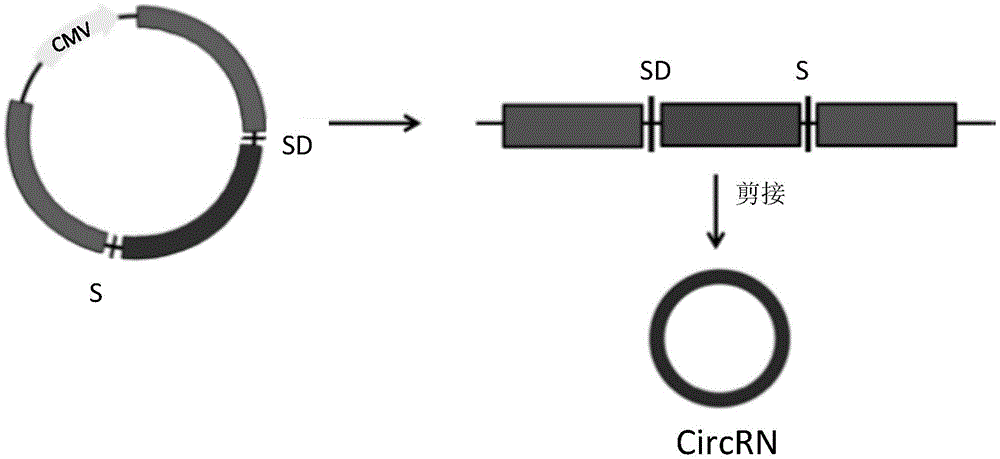
图1图示抗病毒circRNA的细胞内产生。该图描述了环状RNA如何在靶细胞内产生。首先,该图显示含有CMV启动子、围绕候选环状RNA的两个互补重复区域、以及侧翼剪接受体和供体位点的质粒。将该质粒转染到靶细胞中。然后细胞如所示转录线性RNA,并且剪接供体和受体位点之间的区域通过反向剪接而环化。
图2.RNA的合成与环化。工作流程由四个主要步骤组成:(a)通过PCR产生侧翼有T7启动子的模板。(b)通过体外转录制备RNA。(c)产生用于连接的特异性末端。体外转录的RNA在体外环化之前必须去磷酸化。(d)与T4 RNA连接酶1或circRNA连接酶连接。可形成作为副产物的环状或线性低聚物。
图3图示作用方式。该图显示环状RNA阻碍病毒周期的作用方式。病毒在其基因组中包含对其生命周期至关重要的RNA结构。环状RNA通过以发生构象变化的方式与那些结构结合而靶向它们,这阻碍了病毒生命周期的关键步骤。
图4图示相关作用方式的示例。该图显示环状RNA的杂交如何改变病毒周期的示例。首先,RNA结合蛋白(RBP)对于病毒生命周期中的许多步骤是必需的。这些RBP通常结合病毒基因组的特定区域,其中序列和结构(或结构环境)都是必需的。与该图类似,一个示例是PTB在许多小核糖核酸病毒的IRES元件的最后一个结构域中的结合,其与紧接稳定发夹3’的单链嘧啶盒结合。如右图所示,当环状RNA结合IRES的某一结构化区域和该结构化区域前面的单链区时,它以这样的方式改变RNA结构,即嘧啶盒不再在相同的结构环境中,因此RBP不能与其结合。
图5表示靶向病毒基因组的3个不同区域(或3种不同病毒基因组)的circRNA的示例,其中每个靶区域2个杂交序列。具有相同颜色的区域表示它们靶向同一病毒区域。
图6图示HCV基因组开始和结束的RNA二级结构。突出显示的是设计环状RNA所靶向的区域。
图7图示针对HCV基因组设计的4种RNA的结果。circRNA的id对应于前面图中所示的靶区域。可以看出,感染相对于对照降低高达20%。
图8图示DENV基因组的RNA二级结构。突出显示的是设计环状RNA所靶向的区域。
图9图示针对DENV基因组设计的3种RNA的结果。circRNA的id对应于前面图中所示的靶区域。可以看出,感染相对于对照降低高达40%。
图10表示针对CHIKV基因组设计的4种RNA的结果。可以看出,感染相对于对照降低高达50%。
图11表示用于治疗DENV感染时针对DENV和HCV基因组设计的广谱circRNA的结果。HCV_CDS2是实施例1中的circRNA之一,在这里用作阴性对照。DENV1_chp是实施例2中的circRNA之一,在这里用作阳性对照。
图12表示用于治疗HCV感染时针对DENV和HCV基因组设计的广谱circRNA的结果。HCV_CDS2是实施例1中的circRNA之一,在这里用作阳性对照。DENV1_chp是实施例2中的circRNA之一,在这里用作阴性对照。
图13表示用于治疗DENV感染时针对DENV和HCV基因组设计的第二广谱circRNA的结果。HCV_CDS2是实施例1中的circRNA之一,在这里用作阴性对照。DENV1_chp是实施例2中的circRNA之一,在这里用作阳性对照。
图14表示用于治疗HCV感染时针对DENV和HCV基因组设计的第二广谱circRNA的结果。HCV_CDS2是实施例1中的circRNA之一,在这里用作阳性对照。DENV1_chp是实施例2中的circRNA之一,在这里用作阴性对照。
图15表示针对西尼罗病毒(WNV)基因组的设计的两种RNA(circ wnv_sIII 1和circ wnv_sIII 2)的结果。可以看出,感染相对于对照降低。
图16表示用于治疗WNV感染时针对WNV和DENV设计的3种广谱circRNA(dchp_wslI_A、dchp_wslI_B和dchp_wslI_C)的结果。使用来自实施例2和5的阳性对照和阴性对照。可以看出,所有circRNA都显示抑制作用。
图17表示用于治疗DENV感染时针对WNV和DENV设计的3种广谱circRNA(dchp_wslI_A、dchp_wslI_B和dchp_wslI_C)的结果。使用来自实施例2和5的阳性对照和阴性对照。可以看出,所有circRNA都显示抑制作用。
图18表示针对HCV设计的RNA抑制HCV慢性感染细胞的能力。circRNA抑制HCV慢性感染细胞的感染性。Huh7/Scr细胞用HCV感染并在48hpi用circ_hcv_cds2转染。两天后测量荧光素酶值。通过荧光素酶表达水平的变化来确定对感染性的作用。
图19表示针对HCV RNA复制所需的区域设计的RNA及其抑制HCV复制能力的结果。靶向HCV RNA复制所需区域的circ_hcv_cds2抑制HCV RNA复制。在感染后48小时HCV RNA被翻译和复制时,circ_hcv_cds2抑制荧光素酶水平,而在感染后4小时HCV RNA仅被翻译时,circ_hcv_cds2不抑制荧光素酶水平。通过荧光素酶表达水平的变化来确定对感染性的作用。使用T检验计算统计学显著性(*表示p值<0.05)。
图20表示针对DENV RNA复制所需的区域设计的RNA及其抑制DENV复制能力的结果。设计为靶向DENV RNA基因组中指导RNA复制的结构的circ_dv_3utr和circ_dv_cHP_v1抑制DENV RNA复制。circ_dv_3utr和circ_dv_cHP_v1在48小时RNA基因组被翻译和复制时抑制荧光素酶表达水平,而在8小时仅被翻译时不抑制荧光素酶表达水平。所有结果都获自至少三个生物学重复。使用T检验计算统计学显著性(*表示p值<0.05)。
图21.人工circRNA结构和作用方式。1)circRNA包含几个不同的杂交(褐色区域H)和分隔序列(浅灰色区域S)。杂交序列靶向病毒RNA基因组(深灰色区域),并且分隔序列允许杂交序列之间的结构柔性和物理分隔。注意,所有的H和S序列自身是不同的。2)将杂交区域设计为靶向并破坏特定的病毒RNA基因组结构,导致感染性降低。杂交从单链区,外部或发夹环或假结开始,并在螺旋内结束,从而破坏螺旋。
图22.用于circWNV设计的WNV结构。表示来自WNV基因组的5’-UTR(上)和3’-UTR(下)。红色杂交区域用于设计circWNV。UTR:非翻译区[修改自Fernandez-Sanlés et al.,2017,Front.Microbiol.8,1-16]。
图23.用于circCHIKV设计的示意CHIKV结构。表示预测的5’-UTR(A)、RSE(B)和记录元件(C)。在A和B中,所有结构都由C中的circRNA靶向,杂交区域用红色表示。UTR:非翻译区;RSE:重复序列元件。[修改自Adapted from Kendra et al.,2018,Virology 339,200–212;和Khan et al.,2002,J.Gen.Virol.83,3075-3084]。
图24.设计的广谱cirRNA损害DENV和HCV的感染性。用circRNA dv_chp_v1、circRNA hcv_cds2或包含来自circRNA dv_chp_v1和circRNA hcv_cds2的杂交序列的广谱circRNA(circ dchp_hcv_cds2_2)转染细胞。接着,用携带荧光素酶报告基因的DENV或HCV感染细胞,48小时后测量感染性。所有结果都获自至少三个生物学重复。通过荧光素酶表达水平的变化来确定对感染性的作用。使用T检验计算统计学显著性(*表示p值<0.05)。
图25.描述体外获得环状RNA的方案。
图26.显示体外产生的针对DNV和WNV的环状RNA和针对HCV的环状RNA的结果。
图27.显示靶破坏结构(内线)和靶杂交区域(外线)的类型。
图28.例示靶破坏结构、靶杂交区域和杂交区域之间的关系,并显示通过杂交破坏的机制。
图29.不同锤头状核酶切割效率的比较。
图30.具有不同镁条件的凝胶。
图31.显示不同IVT时间的凝胶。
图32.显示RNAse R作用的凝胶,未凝胶纯化。
图33.显示针对SARS-Cov2设计的circRNA的效力结果。
图34.显示体外产生的针对SARS-Cov2的circRNA的效力。
具体实施方式
定义
在描述本公开内容时,将使用以下术语并如下所述定义这些术语。
除非上下文另有说明,术语“一个”、“一种”和“该”包括复数指示物。
术语“约”当提及给定的量或数量时意指包括±5%的偏差。
本文所用的术语“人工环状RNA”或“环状RNA”或“circRNA”是指形成共价闭合的连续环的非编码RNA,其具有连接在一起的3’和5’端,因而它们缺少5’-帽并且因此缺少聚腺苷酸化尾。因此,它们对核酸外切酶介导的降解和脱支酶具有抗性,这赋予它们相对于其他RNA更高的稳定性。
术语“互补”和“互补性”是可互换的,是指多核苷酸彼此形成碱基对的能力。碱基对通常由反向平行多核苷酸链或区域中核苷酸单位之间的氢键形成。互补多核苷酸链或区域可以Watson-Crick方式碱基配对(例如,A与T、A与U、C与G)。100%(或全部)互补是指一条多核苷酸链或区域的每个核苷酸单位可与第二条多核苷酸链或区域的每个核苷酸单位以氢键结合的情况。不完全(或部分)互补是指其中两条链或两个区域的一些但不是全部核苷酸单位可以氢键彼此结合的情况,并且可以以百分比表示。
术语“杂交”用于指由2条独立的RNA链形成的结构,所述RNA链通过由一条链到另一条链的碱基配对形成双链结构。认为这些碱基对是G-C、A-U和G-U(A-腺嘌呤、C-胞嘧啶、G-鸟嘌呤、U-尿嘧啶)。与互补的情况一样,杂交可以是完全或部分的。
术语“转染”用于指细胞对环状RNA的摄取。当环状RNA被引入细胞膜内部时,细胞已被“转染”。
“茎环分子内碱基配对”(“茎环”)是可在单链DNA中或更通常地在RNA中存在的模式。该结构又被称为“发夹”或“发夹环”。当同一条链的以相反的方向读码时通常核苷酸序列互补的两个区域碱基配对从而形成以未配对环终止的双螺旋时,就会发生这种情况。所得结构是许多RNA二级结构的关键构建单元。螺旋的碱基配对不需要是完全的,可以有单链核苷酸段,其被称为“凸起”或“内环”。作为RNA的重要二级结构,它可以指导RNA折叠,保护信使RNA(mRNA)的结构稳定性,提供RNA结合蛋白的识别位点,并用作酶反应的底物。
RNA中的“内环”(又被称为“内部环”)见于双链RNA由于核苷酸之间没有Watson-Crick碱基配对而分隔的地方。可将内环分类为对称的或不对称的,有一些不对称的内环又被称为凸起。内环不同于茎环,因为它们存在于一段双链RNA的中间。
“外环”是发夹环或多环的单链核苷酸段。
“多环”是双链区分支成几个发夹环。
在本发明的上下文中,“靶破坏结构”是茎环(或发夹环),优选在一段未配对的核苷酸(外环,凸起或内环或多环,参见图27)之后或之前。
在本发明的上下文中,“靶杂交区域”是靶破坏结构内的区域,其由双链区之后或之前的单链区组成。因此,靶杂交区域是在另一条RNA链杂交时引起其所属结构破坏的区域。
“通过杂交破坏”:给定靶破坏结构、靶杂交区域和与靶杂交区域完全杂交的人工RNA,当杂交的能量比靶破坏结构的能量更有利(更负)时,则发生通过杂交破坏。这种杂交后,靶破坏结构的结构发生改变,至少对于靶破坏结构和靶杂交区域之间的重叠是完全改变的。这类能量计算可使用完备的软件如RNAcofold(Lorenz,Ronny and Bernhart,Stephan H.and
“病毒保守结构”是指在同一物种、属、科等的成员中保守的病毒基因组RNA结构。那些结构有时已在文献中表征并且已经得到实验验证。在没有这种实验证明的情况下,存在能够在计算机上预测这类结构的软件。例如,同一科的所有病毒基因组都可进行比对,使用例如ClustalW,然后使用RNAz(Improved noncoding RNA detection,Gruber AR,FindeiβS,Washietl S,Hofacker IL,Stadler PF.Pac Symp Biocomput.15:69-79,2010;NucleicAcids Res.1994Nov 11;22(22):4673–4680,doi:10.1093/nar/22.22.4673,PMCID:PMC308517,PMID:7984417;CLUSTAL W:Improving the sensitivity of progressivemultiple sequence alignment through sequence weighting,position-specific gappenalties and weight matrix choice,J D Thompson,D G Higgins,and T J Gibson)来进行。RNAz通过与具有相同长度和序列组成的随机序列进行比较来预测比预期在热力学上更稳定的结构化区域(z-得分),并且另外通过在序列比对中支持补偿性和一致性突变来评估区域。这类保守结构由于其在病毒生命周期中的功能相关性而被认为如此。在实施例14中,我们显示如何发现一些病毒保守结构。
术语5’UTR是指紧靠在起始密码子之前的编码区的上游区域。
术语“IRES”定义为内部核糖体进入位点并且是允许以帽-非依赖性方式进行翻译起始的RNA元件,其作为更大的蛋白质合成过程的一部分。在真核翻译中,起始通常发生在mRNA分子的5’端,因为5’帽识别是装配起始复合物所必需的。IRES元件的位置通常在5’UTR中,但也可存在于mRNA的其他地方。
术语CDS是指信使RNA或病毒基因组的编码区。这是编码相应蛋白质的区域。
术语3’UTR是指紧接在终止密码子之后的CDS下游的病毒基因组区域。
术语“cHP”意指“衣壳编码区发夹元件”,是几种黄病毒基因组的见于CDS内的已知区域。
术语“RSE”意指“保守重复序列元件”,是见于几种甲病毒属基因组的3’UTR的已知结构。
对病毒生命周期至关重要的结构化区域“SRVVLC”,是RNA病毒基因组中在病毒复制、衣壳化和/或翻译上至关重要的结构化区域,即,如果被破坏,病毒不太可能执行其生命周期的基本功能,因而病毒感染性更小。
向细胞“施用”人工RNA包括转导、转染、电穿孔、易位、融合、吞噬、打靶或弹道法等,即核酸可穿过细胞膜转运的任何方法。
“同源性区域”是指在不同病毒株/血清型中具有共同结构和/或功能特征的区域。同源结构不意味着序列一致是必要条件。本领域技术人员能够通过常规方法鉴别其他病毒株/血清型中与如本申请中所定义的同源的区域。
两个序列之间的同一性程度可通过常规方法来确定,例如借助现有技术中已知的标准序列比对算法,例如BLASTn(Altschul S.F.et al.Basic local alignment searchtool.J Mol Biol.1990Oct 5;215(3):403-10)。
在描述权利要求时,词语“包括/包含”及其变体并不旨在排除其他技术特征、添加物、组分或步骤。此外,术语“包括/包含”也可涵盖术语“由……组成”。
发明描述
总体而言,诸如mRNA、tRNA、rRNA、非编码RNA、病毒RNA基因组、病毒mRNA等的RNA片段包含高度结构化区域(具有二级结构的区域),这些区域是其功能所必需的,或者虽然不是必需的但可能在所述RNA片段的功能性中发挥或多或少的重要作用。这些高度结构化区域包括在未配对核苷酸区域之后或之前的发夹环或至少发夹环的部分。如果这些发夹环或发夹环的部分的结构(如二级结构)发生改变,则RNA片段内的构象发生变化。这会导致RNA片段的功能性的变化。
生物RNA是单链的,并且由于其因核糖中额外羟基而形成氢键的能力增加,通常形成错综复杂的碱基对相互作用。RNA的二级结构由同一分子内折叠的单个多核苷酸组成。RNA中的碱基配对发生RNA在互补区域之间折叠时。单链区和双链区通常都存在于RNA分子中。反向平行链形成螺旋形。RNA二级结构中的四个基本构成部分是螺旋、环、凸起和连接。茎环或发夹环是RNA二级结构中最常见的元件。当RNA链自身向后折叠形成被称为茎的双螺旋束,而未配对的核苷酸形成被称为环的单链区时,则形成茎环。
RNA的二级结构可通过计算或实验方法进行预测。计算上,RNA二级结构可使用技术人员可公开获得的工具(例如,如上所述的Vienna程序包)由一个或几个核酸序列预测。
如上所述,RNA结构(如RNA二级结构)在许多生物学过程中都很重要,包括信使RNA的翻译调节、单链RNA病毒的复制以及结构RNA和RNA/蛋白复合物的功能。事实上,对于许多RNA分子来说,二级结构对于RNA的正确功能非常重要——通常比实际序列更重要。
因此,RNA片段二级结构的改变将不可避免地导致RNA片段的功能性的变化。
本发明的人工RNA(优选circRNA)能够改变它们靶向的RNA片段的二级结构。因此,利用本发明的人工RNA,可调节靶RNA片段的功能性。例如,在病毒基因组中,存在对病毒生命周期至关重要的高度结构化区域(SRVVLC)。如果这些区域的构象或二级结构发生改变(如被破坏),则病毒将不太能(优选不能)执行其生命周期的基本功能。因此,改变这些对病毒生命周期至关重要的区域的构象或二级结构将导致病毒感染性降低,理想情况下完全无效。同样的原理也适用于其他RNA片段。例如,tRNA分子构象的变化会导致该特定tRNA的翻译效率降低(或增加)。例如,mRNA分子二级结构的变化会导致该mRNA分子与某种蛋白质甚至核糖体的结合亲和力降低(或增加),从而影响其翻译速率。病毒和细胞的IRES元件的结构变化都会影响翻译起始。mRNA的UTR结构的变化会遮蔽miRNA结合位点,从而影响该mRNA的翻译调节。内含子区域结构的变化会影响剪接等。
特别地,本发明的人工RNA(优选circRNA)能够与靶RNA片段内的特定区域杂交。这样做时,本发明的人工RNA(优选circRNA)能够破坏靶RNA片段内的这些特定靶区域。这些“靶RNA片段内的特定区域”是所谓的“靶破坏结构”。
靶破坏结构包括在未配对核苷酸区域之后或之前的至少发夹环,并且还包括所谓的“靶杂交区域”。靶杂交区域是靶破坏结构内的区域,其包括在双链区之后或之前的至少2个核苷酸的单链区。
本发明的人工RNA(优选circRNA)与靶杂交区域通过所谓的“杂交区域”进行杂交。换句话说,本发明的人工RNA(优选circRNA)包含至少一个杂交区域,所述杂交区域是人工RNA能够与包含于靶RNA片段的靶破坏区域中的一个或多个靶杂交区域完全杂交的区域。
杂交是两个RNA分子通过碱基配对相互作用而相互退火的现象。在本发明的上下文中,仅考虑标准碱基配对(C-G、A-U和G-U)。“完全杂交”应理解为人工RNA杂交区域的所有核苷酸与靶杂交区域的所有核苷酸退火。
当杂交区域与靶杂交区域完全杂交时,杂交区域与靶杂交区域之间的杂交能量比靶破坏区域的能量更负。这种情况发生时,靶破坏结构被破坏,即靶RNA片段的二级结构被改变。因此,靶RNA片段的功能性也被改变:本发明的人工RNA调节了RNA片段的功能性。这可以复现,因此一旦靶杂交区域已知,通过使用RNA设计工具如NUPACK或RNAiFold,就可容易地发现与靶杂交区域完全杂交的杂交区域的候选物,如下所述。
第一方面,本发明涉及适合于通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构的人工RNA,优选circRNA。
“通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构”,应理解为在本发明的上下文中,当本发明的人工RNA的杂交区域与靶破坏结构的相应靶杂交区域完全杂交时,一个或多个RNA片段的一个或多个靶破坏结构的二级结构被改变。
本发明的人工RNA(优选circRNA)可以是任何长度的,只要它包含至少一个如本发明所述的杂交区域,并且适合于通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构。优选地,本发明的RNA(优选circRNA)包含100至1000个核苷酸,更优选150至800个核苷酸,甚至更优选200至600个核苷酸。
如上所述,本发明的人工RNA(优选环状RNA)包含至少一个杂交区域。所述至少一个杂交区域是人工RNA的与包含于所述一个或多个RNA片段的一个或多个靶破坏结构中的至少一个靶杂交区域完全杂交的区域(即,核苷酸序列)。杂交区域的核苷酸数目不受限制,只要其能够与至少一个靶杂交区域完全杂交,但是优选地,其具有与包含于一个或多个靶破坏结构中的靶杂交区域相同的长度,并且在这种情况下破坏一个或多个靶破坏结构(即,改变二级结构)。
在优选实施方式中,至少一个杂交区域具有总共7至100个核苷酸,更优选10至50个核苷酸,甚至更优选15至35个核苷酸,例如15至25个核苷酸。
如上所述,杂交区域与包含于一个或多个RNA片段的一个或多个靶破坏结构中的至少一个靶杂交区域完全杂交。靶破坏结构是RNA片段的包含在未配对核苷酸区域之后或之前的发夹环的至少一部分的区域。靶破坏结构包含至少一个靶杂交区域。靶杂交区域是包含在双链区之后或之前的至少2个核苷酸的单链区的区域(即,核苷酸序列)。靶杂交区域的长度不受限制,只要其包含在双链区之后或之前的至少2个核苷酸的单链区,并且能够与人工RNA的至少一个杂交区域完全杂交。优选地,靶杂交区域的单链区包含3个或更多个核苷酸,如3、4、5、10、15个或更多个核苷酸。优选地,靶杂交区域的双链区包含5个或更多个核苷酸,如5、7、10、15、20、25个或更多个核苷酸。
重要地,本发明的人工RNA(优选是环状RNA)的杂交区域与包含于一个或多个RNA片段的一个或多个靶破坏结构中的至少一个靶杂交区域完全杂交。因此,本发明的人工RNA的至少一个杂交区域的核苷酸数目与与其杂交的至少一个靶杂交区域完全相同。因此,本发明的人工RNA的至少一个杂交区域具有与靶杂交区域的单链区完全杂交的含特定数目的核苷酸的第一区域,和与靶杂交区域的双链区完全杂交的含特定数目的核苷酸的第二区域。
换句话说,第一步中,靶杂交区域的单链区与人工RNA(优选环状RNA)的杂交区域的某些核苷酸退火。第二步中,在单链区之前或之后的靶杂交区域的双链区被破坏,并且在靶杂交区域的双链区的一条链与人工RNA的杂交区域的某些核苷酸之间彼此退火形成新的相互作用。当至少一个杂交区域和至少一个靶杂交区域之间的杂交能量比靶破坏区域的能量更负(更有利)时,发生靶杂交区域的双链区的破坏。杂交能量和破坏一个或多个靶破坏结构的能力可以例如用RNAcofold确定;以至少一个杂交区域和至少一个靶杂交区域之间的杂交能量比靶破坏区域的能量更负(更有利)为特征的本发明的人工RNA的杂交区域的潜在候选物的鉴别,可通过RNA反向折叠工具如NUPACK、RNAifold或MoiRNAiFold容易地鉴别,如实施例4所示。
因此,至少靶杂交区域的双链区被破坏,因而靶破坏结构也被破坏。因此,靶破坏结构的结构发生改变,至少对于靶破坏结构和靶杂交区域之间的重叠是完全改变的。因此,靶RNA片段的二级结构被改变,由此其功能性也发生了变化。
人工RNA的杂交区域和RNA片段的靶破坏结构的靶杂交区域之间的杂交可在体外或体内进行。RNA片段的二级结构的变化也可使用本领域熟知的技术进行检测,例如SHAPE(Poulsen,Line Dahl et al.“SHAPE Selection(SHAPES)enrich for RNA structuresignal in SHAPE sequencing-based probing data.”RNA(New York,N.Y.)vol.21,5(2015):1042-52.doi:10.1261/rna.047068.114)或PARIS(Lu Z,Gong J,Zhang QC.PARIS:Psoralen Analysis of RNA Interactions and Structures with High Throughput andResolution.Methods Mol Biol.2018;1649:59-84.doi:10.1007/978-1-4939-7213-5_4)。
因此,包含于本发明的人工RNA中的至少一个杂交区域的进一步特征在于,当与靶杂交区域杂交时,至少一个杂交区域和至少一个靶杂交区域之间的杂交能量比靶破坏区域的能量更负,从而破坏靶破坏结构。如上所讨论的,本领域技术人员能够预测至少一个杂交区域和至少一个靶杂交区域之间的杂交能量与靶破坏区域的能量。用于该预测的可用工具是,例如已完备的软件如RNAcofold(Lorenz,Ronny and Bernhart,Stephan H.and
优选地,至少一个杂交区域和至少一个靶杂交区域之间的杂交能量与靶破坏区域的能量优选用RNAcofold软件计算,其设置如[Mathews,D.H.,et al.,“Predictingoligonucleotide affinity to nucleic acid targets”,RNA,1999 5:1458-1469]所述。然而,应注意,当使用RNA反向折叠工具如NUPACK、RNAifold或MoiRNAiFold来鉴别以具有至少一个杂交区域和至少一个靶杂交区域之间的杂交能量比靶破坏区域的能量更负(更有利)为特征的本发明的人工RNA的杂交区域的潜在候选物时,这种计算不是必要的。
对于给定的RNA片段,本领域技术人员能够鉴别潜在的靶破坏结构,因为这些是RNA的包含在未配对核苷酸区域之后或之前的发夹环的至少一部分的区域。本领域技术人员可获得许多RNA结构预测软件,并且这些RNA结构预测软件预测给定RNA序列的二级结构。例如,参考Mathews,D.H.,et al.“RNA secondary structure prediction.”Currentprotocols in nucleic acid chemistry vol.Chapter 11(2007):Unit 11.2.doi:10.1002/0471142700.nc1102s28。
此外,本领域技术人员能够鉴别靶破坏结构内的一个或多个靶杂交区域,因为靶杂交区域包含于靶破坏结构中,并且包含在双链区之后或之前的至少2个核苷酸的单链区,如上文所定义。
一旦鉴别了靶杂交区域,本领域技术人员就能够设计与靶杂交区域完全杂交的一个或多个杂交区域。与上述类似,本领域技术人员知道用于设计与给定RNA序列完全杂交的RNA序列的几种软件,例如NUPACK(http://www.nupack.org/design/new,see alsoB.R.Wolfe,N.J.Porubsky,J.N.Zadeh,R.M.Dirks,and N.A.Pierce,“Constrainedmultistate sequence design for nucleic acid reaction pathway engineering”,JAm Chem Soc,139:3134-3144,2017;B.R.Wolfe and N.A.Pierce,“sequence design fora test tube of interacting nucleic acid strands”,ACS Synth Biol,4:1086-1100,2015;J.N.Zadeh,B.R.Wolfe,and N.A.Pierce,“Nucleic acid sequence design viaefficient ensemble defect optimization”J Comput Chem,32:439–452,2011;以及R.M.Dirks,M.Lin,E.Winfree,and N.A.Pierce,“Paradigms for computational nucleicacid design”Nucl Acids Res,32:1392-1403,2004.)、或RNAiFold(https://bioinformatics.bc.edu/clotelab/RNAiFold/,同样参见Juan Antonio Garcia-Martin,Peter Clote,Ivan Dotu,“RNAiFold:A constraint programming algorithm for RNAinverse folding and molecular design,J Bioinform Comput Biol 11(2):1350001,2013;以及Garcia-Martin JA,Dotu I,Clote P.,“RNAiFold 2.0A web server andsoftware to design custom and Rfam-based RNA molecules”,Nucleic AcidsResearch Web Server issue,2015,doi:10.1093/nar/gkv460,“RNAiFold 2.0A webserver and software to design custom and Rfam-based RNA molecules”)。此外,可以确定在给定的特定靶杂交区域的情况下,是否可以通过杂交破坏某个靶破坏结构。这可使用可公开获得的几个RNA反向折叠或RNA设计工具来实现,例如NUPACK(http://www.nupack.org/design/new)、RNAiFold(http://bioinformatics.bc.edu/clotelab/RNAiFold/)或其最新扩展MoiRNAiFold(https://moiraibiodesign.com/design/)。
例如,在RNAiFold(和MoiRNAiFold)中,给定的任何具体的靶破坏结构,比如图28所示的靶破坏结构(AAUAGAGUCCUGCCCAUUGGCGGG)及其靶杂交区域(GAGUCCUGCC),可以使用以下输入文件:
#RNAscdstr
((((((((((&....))))))))))..........
#RNAseqcon
NNNNNNNNNN&AAUAGAGUCCUGCCCAUUGGCGGG
其中输入((((((((((&....))))))))))..........对应于已完备建立的点括号表示法(例如,RNAlib-2.4.18:RNA结构表示法(univie.ac.at),并且其中输入NNNNNNN&AAUAGAGUCCUGCCCAUUGGCGGG表示每个“N”是核苷酸(任何核苷酸A、C、G或U),该核苷酸对应于上面的“(”且必须与以上所示的相应“)”杂交。也就是说,RNAiFold(和MoiRNAiFold)应当返回潜在杂交区域的一个(或多个)解决方案,其在与靶杂交区域GAGUCCUGCC完全杂交时确实可破坏靶破坏结构AAUAGAGUCCUGCCCAUUGGCGGG。
在这种情况下,RNA反向折叠工具返回的10个解决方案的样本集如下:
由此得出,首先,靶破坏结构可通过杂交被破坏,其次,杂交区域(粗体)可用于构建可通过杂交破坏靶破坏结构的circRNA(通过用随机多核苷酸区域将这些区域分隔)。
因此,在这些输入指令的条件下,如果软件工具返回一个(或多个)解决方案,这意味着靶破坏结构通过杂交确实可被破坏。由这组解决方案,结果生成了本发明的circRNA:通过取任意数目的这些解决方案(杂交区域)并用随机核苷酸区域将其分隔。实施例14显示本申请中用作示例的几个靶破坏结构的其他输入文件。
在本发明的上下文中,杂交区域是人工环状RNA内的区域,该区域能够与感兴趣的RNA片段中的靶杂交区域杂交,并通过杂交破坏靶破坏结构(参见图28,描绘所有这些概念如何相互关联)。在优选实施方式中,杂交能量和破坏一个或多个靶破坏结构的能力可用RNA反向折叠工具来计算,例如NUPACK、RNAifold、MoiRNAiFold。
如上所述,在非常优选的实施方式中,本发明的人工RNA是环状RNA。在这个意义上,本发明人已发现,使用本文公开的基于人工RNA(优选环状RNA)的疗法,与其他RNA分子相比具有根本上的优势:
-稳定性。一方面,circRNA极其稳定,因为它们不能被细胞核酸外切酶利用。与其他当前基于RNA的疗法相比,这种稳定性将简化它们在治疗中的使用,当前基于RNA的疗法需要昂贵、非天然的化学修饰,并可能导致毒性问题。
-抵抗逃逸突变体的出现和选择。所设计的人工RNA如circRNA,优选包含(i)较长的序列,这种较长的序列与靶杂交区域的杂交不会受到单个突变的影响,和(ii)能够与病毒RNA基因组中的多个靶杂交区域完全杂交的多个杂交区域,阻碍通过选择单个突变的抗性突变体的出现。
-能够处理多个靶标。目前所有有效的疗法都涉及靶向特定病毒蛋白的分子,因此只能治愈单一感染。通过在设计的人工RNA(如circRNA)中包括能够与包含于不同RNA片段(如不同病毒基因组)的靶破坏结构中的不同靶杂交区域杂交的杂交区域,将能够同时靶向不同的RNA片段,例如不同的病毒。这种广谱疗法对于(i)简化合并感染的治疗和(ii)治疗具有相同地理位置和初始症状的急性感染(如,例如由登革病毒、寨卡病毒和基孔肯亚病毒引起的感染或由SARS-CoV-2和流感引起的感染)具有重要价值。在急性感染中,早期治疗是控制疾病和流行病的关键。广谱疗法甚至可在完成最终诊断之前进行治疗。此外,在流行病期间,使用这类疗法作为预防性治疗是可取的。
-使抗药性风险最小化。此外,为了进一步使抗药性风险最小化,设计的人工RNA(如circRNA)中靶向同一病毒靶破坏结构的杂交区域(例如,参见图21)都不相同,因为本发明的人工RNA(如circRNA)可有利地包含G-U配对。与需要互补的DNA相反,G-U配对在RNA中是有效的杂交配对。
在另一个实施方式中,本发明的人工RNA(优选环状RNA)包含至少一个杂交区,其中至少一个杂交区域能够杂交来自同一RNA片段内或甚至来自不同RNA片段的不同靶破坏结构的靶杂交区域。
在优选实施方式中,本发明的人工RNA(优选环状RNA)包含2个或更多个杂交区域,优选6至20个杂交区域。在另一优选实施方式中,至少两个且优选全部杂交区域能够与同一靶杂交区域完全杂交。因此,根据该优选实施方式的人工RNA将具有更大的概率能破坏RNA片段的靶破坏结构,并且随后将更有效地调节RNA片段的功能性,特别是易于突变的RNA片段(例,病毒和/或肿瘤)。因此,具有多个杂交区域主要影响病毒/肿瘤的突变体逃逸能力。
在另一个更优选实施方式中,本发明的RNA(优选环状RNA)包括至少两个杂交区域,其中至少两个且优选全部杂交区域具有不同的核苷酸序列,即在核苷酸序列方面彼此不同。因此,在该优选实施方式中,人工RNA的至少两个杂交区域(优选全部杂交区域)彼此不同,并且它们都靶向(能够杂交)同一靶杂交区域(“多对一”法)。这样,根据该优选实施方式的人工RNA将具有更大的概率能破坏RNA片段的靶破坏结构,然后将更有效地调节RNA片段的功能性。或者,人工RNA彼此不同的至少两个杂交区域(优选全部杂交区域)靶向(能够杂交)来自同一RNA片段内的或甚至来自不同RNA片段的不同靶破坏结构的不同靶杂交区域。
在另一个优选实施方式中,本发明的人工RNA的两个或更多个杂交区域:
a)由大小至多20个核苷酸的非杂交区域分隔;或
b)未由非杂交区域分隔;或
c)是重叠的。
优选地,所述一个或多个RNA片段选自mRNA、tRNA、rRNA、非编码RNA和病毒基因组RNA。更优选地,RNA片段是病毒基因组RNA。甚至更优选地,一个或多个RNA片段是正义单链(ss)病毒基因组RNA。
病毒RNA基因组和病毒mRNA包含高度结构化区域(“靶破坏结构”),该高度结构化区域包含未配对核苷酸区域之后或之前的发夹环的至少一部分,对其功能至关重要。这些高度结构化区域优选是对病毒生命周期至关重要的结构化区域(SRVVLC)。如果这些区域被破坏,病毒将不太能(优选不能)执行其生命周期的基本功能。因此,破坏这些区域会使病毒感染性降低,理想情况下完全无效。
本发明人设计了人工RNA(优选环状RNA),其包含至少一个(优选多于一个)杂交区域,该杂交区域与病毒基因组RNA中存在的至少一个(如一个、两个或更多个)靶破坏结构内的至少一个(如一个、两个或更多个)靶杂交区域杂交并破坏它,从而减少或甚至抑制病毒感染。如上所述,在优选实施方式中,本发明的人工RNA包含至少两个(优选更多个)杂交区域,它们彼此不同,并且能够与病毒基因组RNA中存在的一个靶破坏结构内的一个靶杂交区域完全杂交并破坏它,从而减少或甚至抑制病毒感染。通过这种方式(“多对一”法)提高了破坏的效率。
在一个实施方式中,一个或多个靶破坏结构包含于病毒基因组的IRES元件中。优选地,包含于病毒基因组IRES元件中的一个或多个靶破坏结构是对病毒生命周期至关重要的结构化区域(SRVVLC)。
在另一个实施方式中,一个或多个靶破坏结构包含于病毒基因组的5’UTR和/或3’UTR中。优选地,包含于病毒基因组的5’UTR和/或3’UTR中的一个或多个靶破坏结构是对病毒生命周期至关重要的结构化区域(SRVVLC)。
在另一个实施方式中,一个或多个靶破坏结构包含于病毒基因组的CDS中。优选地,包含于病毒基因组CDS中的一个或多个靶破坏结构是对病毒生命周期至关重要的结构化区域(SRVVLC)。
在另一个实施方式中,人工RNA的至少两个杂交区域能够与至少一个靶杂交区域完全杂交,所述靶杂交区域包含于至少一个破坏结构中,所述破坏结构存在于以下两个位置的任意组合中:病毒基因组的IRES元件、5’UTR区域、CDS区域和/或3’UTR区域。
在另一个实施方式中,本发明的人工RNA(优选环状RNA)能够通过杂交破坏包含于至少两种病毒基因组RNA中的一个或多个靶破坏结构。优选地,本发明的人工RNA能够通过杂交破坏包含于至少两种病毒基因组RNA中的一个或多个对病毒生命周期至关重要的结构化区域。
在优选实施方式中,一个或多个对病毒生命周期至关重要的结构化区域的破坏使病毒活性降低,更优选地,一个或多个对病毒生命周期至关重要的结构化区域的破坏使病毒完全失活。
作为靶RNA片段的病毒基因组(病毒基因组RNA)不受限制。具有靶破坏结构(即,包含未配对核苷酸区域之后或之前的至少发夹环的区域)的任何病毒基因组可以是适合于本发明的人工RNA的靶RNA片段。根据本发明可作为靶RNA片段的病毒基因组示例如下:丙型肝炎病毒(HCV)、流感病毒、甲型肝炎病毒(HAV)、脊髓灰质炎病毒、柯萨奇病毒B、冠状病毒、鼻病毒(普通感冒)、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒和黄热病毒。
在另一个优选实施方式中,本发明的人工RNA(优选在本发明的人工环状RNA中)能够破坏的靶破坏结构是包含于病毒基因组的5’UTR中的靶破坏结构。在优选实施方式中,DENV(登革病毒)基因组的5’UTR区的靶破坏结构是cHP(衣壳编码发夹区)。
在另一个优选实施方式中,本发明的人工RNA(优选在本发明的人工环状RNA中)能够破坏的靶破坏结构是包含于病毒基因组的IRES元件中的靶破坏结构。在优选实施方式中,HCV(丙型肝炎病毒)病毒基因组的IRES元件的靶破坏结构是IRES1和/或IRES2。
在另一个优选实施方式中,本发明的人工RNA(优选在本发明的人工环状RNA中)能够破坏的靶破坏结构是包含于病毒基因组CDS中的靶破坏结构。在优选实施方式中,病毒基因组CDS的靶破坏结构是CDS1和/或CDS2。优选地,靶破坏结构选自HCV基因组的区域SL388、SL427、SL588和/或SL750。在优选实施方式中,靶破坏结构是HCV基因组的区域SL427和/或区域SL588(SL代表茎环)。
在另一个优选实施方式中,本发明的人工RNA(优选在本发明的人工环状RNA中)能够破坏的靶破坏结构是包含于病毒基因组3’UTR中的靶破坏结构。在一个优选实施方式中,DENV基因组的3’UTR区的靶破坏结构是sHP(短茎环或短发夹区)。在另一个优选实施方式中,WNV(西尼罗病毒)基因组3’UTR区的靶破坏结构是SL_II。
在另一个优选实施方式中,在根据任一前述实施方式的环状RNA中,靶破坏结构见于病毒基因组的IRES元件、5’UTR结构化区域、CDS结构化区域、3’UTR结构化区域中的两个或更多个的组合中。
在另一个优选实施方式中,CHIKV(基孔肯亚病毒)基因组的靶破坏结构为RSE区域和/或重编码元件(RE)。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:1中。具体地,靶杂交区域包含SEQ ID NO.:25至28中的一个或多个所定义的核苷酸序列,或与SEQ IDNO.:25至28中所定义的核苷酸序列具有至少70%同一性、优选至少80%、更优选至少90%、甚至更优选地至少95%同一性的核苷酸序列,或另一HCV株/血清型中的同源区。
在优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含SEQ ID NO.:25至27所定义的核苷酸序列(组合circ_hcv_ires1、circ HCV2和circ_hcv_cds1,参见下表1),或与SEQ ID NO.:25至27所定义的核苷酸序列具有至少70%同一性、优选与SEQ ID NO.:25至27所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列,或另一HCV株/血清型的同源区。
两个序列之间的同一性程度可通过常规方法来确定,例如通过现有技术中已知的标准序列比对算法,例如BLASTn(Altschul S.F.et al.Basic local alignment searchtool.J Mol Biol.1990Oct 5;215(3):403-10)。
“同源性区域”是指在不同病毒株/血清型中具有共同结构和/或功能特征的区域。同源结构不意味着序列一致是必要条件。本领域技术人员能够通过常规方法鉴别其他病毒株/血清型中与如本申请中所定义的同源的区域。
表1
表1.针对HCV设计的circRNA:针对HCV设计的不同circRNA在表中分类,同时列出以下信息:它们通过杂交破坏的靶破坏结构、与人工RNA的杂交区域完全杂交的靶杂交区域的序列、完整的circRNA序列和存在于每个候选物(每个人工环状RNA)中的杂交区域数目。IRES:内部核糖体进入位点;CDS:编码序列;cHP:衣壳发夹。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:7中。具体地,靶杂交区域包含如SEQ ID NO.:29至30中的一个或多个所定义的核苷酸序列,或与SEQ IDNO.:29至30所定义的核苷酸序列具有至少70%同一性、优选与SEQ ID NO.:29至30所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列,或另一DENV株/血清型中的同源区。
表2
表2.针对DENV设计的circRNA:针对DENV设计的不同circRNA在表中分类,同时列出以下信息:它们通过杂交破坏的靶破坏结构、与人工RNA的杂交区域完全杂交的靶杂交区域的序列、完整的circRNA序列和存在于每个候选物(每个人工环状RNA)中的杂交区域数目。UTR:非翻译区;cHP:衣壳区发夹。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:1和SEQ IDNO.:7中的一个或多个。具体地,靶杂交区域包含SEQ ID NO.:30和SEQ ID NO.:28中的一个或多个所定义的核苷酸序列,或与SEQ IDNO.:30和SEQ ID NO.:28所定义的核苷酸序列具有至少70%同一性、优选与SEQ ID NO.:30和SEQ IDNO.:28所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列,或另一DENV/HCV株/血清型中的同源区。
表3
/>
表3.广谱DENV-HCV cirRNA。具有两个靶破坏结构(DENV cHP和HCV CDS)的circ_dv_cHP_v1-circ_hcv_cds2、靶杂交区域、完整的circRNA序列和每个circRNA中杂交区域数目的信息。cHP:衣壳区发夹;CDS:编码序列。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:11中。具体地,靶杂交区域包含SEQ ID NO.:31、33和35中的一个或多个所定义的核苷酸序列,或与SEQ IDNO.:31、33和35所定义的核苷酸序列具有至少70%同一性、优选与SEQ ID NO.:31、33和35所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列,或另一CHIKV株/血清型中的同源区。
表4
表4.针对CHIKV设计的circRNA:针对CHIKV设计的不同circRNA在表中分类,同时列出以下信息:靶破坏结构、靶杂交区域的序列、完整的circRNA序列和存在于每个候选物(每个人工环状RNA)中的杂交区域数目。UTR:非翻译区;CDS:编码序列。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:20中。具体地,靶杂交区域包含SEQ ID NO.:37所定义的核苷酸序列,或与SEQ ID NO.:37所定义的核苷酸序列具有至少70%同一性、优选与SEQ IDNO.:37所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列,或另一WNV株/血清型中的同源区。
表5
表5.针对WNV设计的circRNA:针对WNV设计的不同circRNA在表中分类,同时列出以下信息:靶破坏结构、靶杂交区域的序列、完整的circRNA序列和存在于每个候选物(每个人工环状RNA)中的杂交区域的数目。SLIII:茎环III;UTR:非翻译区。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:20和SEQ IDNO.:7中。具体地,靶杂交区域包含SEQ ID NO.:30和37中的一个或多个所定义的核苷酸序列,或与SEQ ID NO.:30和37所定义的核苷酸序列具有至少70%同一性、优选与SEQ IDNO.:30和37所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列,或另一DENV/WNV株/血清型中的同源区。
表6
表6.针对WNV和DWNV设计的circRNA:针对WNV和DWNV设计的不同circRNA在表中分类,同时列出以下信息:靶破坏结构、靶杂交区域的序列、完整的circRNA序列和存在于每个候选物(每个人工环状RNA)中的杂交区域数目。SLI:茎环I;cHP:衣壳区发夹。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:1、SEQ IDNO.:7、SEQ ID NO.:11和/或SEQ ID NO.:20中。具体地,靶杂交区域包含SEQ ID NO.:25至31、33和35、37中的一个或多个所定义的核苷酸序列,或与SEQ ID NO.:25至31、33和35、37所定义的核苷酸序列具有至少70%同一性的核苷酸序列、优选与SEQ ID NO.:25至31,33和35、37所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列。
在另一个优选实施方式中,HCV病毒基因组中的靶破坏结构见于IRES和/或CDS区和/或它们的组合中。更优选地,HCV基因组的靶破坏结构选自包含以下或由以下组成的靶破坏结构列表:SEQ ID NO.:76、77、78或79,或与SEQ ID NO.:76、77、78或79所定义的核苷酸序列具有至少60%、70%、80%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%同一性的核苷酸序列,或其任何组合。
在另一个优选实施方式中,登革病毒基因组中的靶破坏结构见于3’UTR和/或为cHP和/或它们的组合。更优选地,DENV基因组的靶破坏结构选自包含以下或由以下组成的靶破坏结构列表:SEQ ID NO.:29或30,或与SEQ ID NO.:29或30所定义的核苷酸序列具有至少60%、70%、80%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%同一性的核苷酸序列,或其任何组合。
在另一个优选实施方式中,寨卡病毒基因组中的靶破坏结构见于5’UTR和/或为RSE和/或它们的组合。
在另一个优选实施方式中,基孔肯亚病毒基因组中的靶破坏结构见于5’UTR和/或为RSE和/或它们的组合中。更优选地,CHIKV基因组的靶破坏结构选自包含以下或由以下组成的靶破坏结构列表:SEQ IDNO.:80、81或82,或具有与SEQ ID NO.:80、81或82所定义的核苷酸序列至少60%、70%、80%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%同一性的核苷酸序列,或其任何组合。
在另一个优选实施方式中,西尼罗病毒基因组中的靶破坏结构为茎环III(SLII)。更优选地,WNV基因组的靶破坏结构包含以下或由以下组成的靶破坏结构列表:SEQ IDNO.:83,或与SEQ ID NO.:83所定义的核苷酸序列具有至少60%、70%、80%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%同一性的核苷酸序列。
在另一个优选实施方式中,本发明的人工RNA(优选环状RNA)的杂交区域靶向多于一个存在于一种病毒基因组中的靶破坏结构(即,能够与在存在于一种病毒基因组中的靶破坏结构中的靶杂交区域完全杂交)。
在具体实施方式中,本发明的人工RNA(优选本发明的人工环状RNA)具有针对RNA病毒基因组的广谱活性,优选具有针对HCV、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒和黄热病毒基因组的广谱活性。在本发明中,与本发明的人工RNA(优选环状RNA)相关的“广谱活性”意指所述人工RNA有效针对多种RNA病毒,优选针对HCV、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒和黄热病毒。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:34中。具体地,靶杂交区域包含SEQ ID NO.:58至62中的一个或多个所定义的核苷酸序列,或与SEQ IDNO.:58至62所定义的核苷酸序列具有至少70%同一性、优选与SEQ ID NO.:58至62所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列,或另一SARS-CoV-2株/血清型中的同源区。
在另一个优选实施方式中,SARS-CoV-2病毒基因组中的靶破坏结构见于靶A、B、C、D、3’UTR、5’UTR和/或它们的组合中。更优选地,SARS-CoV-2基因组的靶破坏结构选自包含以下或由以下组成的靶破坏结构列表:SEQ ID NO.:84、58、85、86、87,或与SEQ ID NO.:84、58、85、86、87所定义的核苷酸序列具有至少60%、70%、80%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%同一性的核苷酸序列,或其任何组合。
表7
/>
/>
/>
表7.针对严重急性呼吸综合征冠状病毒2(SARS-CoV-2)分离株Wuhan-Hu-1(完整基因组:SEQ ID NO:34)设计的circRNA:针对SARS-CoV-2设计的不同circRNA在表中分类,同时列出以下信息:靶破坏结构、靶杂交区域的序列、完整的circRNA序列和存在于每个候选物(每个人工环状RNA)中的杂交区域数目。
在另一个优选实施方式中,与根据本发明的人工RNA(优选本发明的人工环状RNA)的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:1、SEQ IDNO.:7、SEQ ID NO.:11和/或SEQ ID NO.:20中的一个或多个。具体地,靶杂交区域包含SEQID NO.:25至31、33、35、37和58至62中的一个或多个所定义的核苷酸序列,或与SEQ IDNO.:25至31、33和35、37所定义的核苷酸序列具有至少70%同一性的核苷酸序列、优选与SEQ ID NO.:25至31、33、35、37和58至62所定义的核苷酸序列具有至少80%、更优选至少90%、甚至更优选至少95%同一性的核苷酸序列。
来自以上公开的相同病毒或不同病毒的靶破坏结构和/或靶杂交区域的所有可能组合都包括在本文中作为设计本发明的circRNA序列的具体实施方式。应注意,对于在本发明中公开的每个靶破坏结构(IRES、CDS等),不同的相应杂交区域可以如本文所述使用和/或组合,以便设计circRNA。
第二方面,本发明提供了组合物,其单独或组合地包含本文公开的任何实施方式中的本发明的人工RNA。
优选地,所述组合物是药物组合物,并且在任何实施方式中优选包含本发明的人工RNA和一种或多种药学上可接受的载体。
考虑临床应用时,药物组合物将以适合于预期应用的形式制备。通常,这需要制备基本上不含热原以及对人或动物有害的其他杂质的组合物。
胶体分散系统,例如大分子复合物、纳米胶囊、微球、珠以及包括水包油乳液、胶束、混合胶束和脂质体的基于脂质的系统,可用作人工RNA(如环状RNA)的递送溶媒。适合于将本公开的核酸递送至组织的市售脂肪乳剂包括Intralipid、Liposyn、Liposyn II、Liposyn III、Nutrilipid和其他类似脂质乳剂。用作体内递送溶媒的胶体系统是脂质体(即,人工膜囊泡)。这类系统的制备和使用在本领域中是公知的。示例性制剂还公开于美国专利号5,981,505;美国专利号6,217,900;美国专利号6,383,512;美国专利号5,783,565;美国专利号7,202,227;美国专利号6,379,965;美国专利号6,127,170;美国专利号5,837,533;美国专利号6,747,014;以及WO03/093449。特别地,可将包含lipofectamine的阳离子脂质体制剂用于递送。lipofectamine可与中性共脂质或辅助脂质一起配制。参见,例如美国专利号7,479,573、Dalby et al.(2004)Science Direct,Methods 33:95-103、Hawley-Nelson et al.(1993)Focus 15:73-79。
通常期望使用适当的盐和缓冲液使递送溶媒稳定并允许靶细胞摄取。将重组细胞(如,用人工RNA(如环状RNA)离体转染)引入患者时,还将使用缓冲液。本公开的水性组合物包含溶解或分散于药学上可接受的载体或水性介质中的有效量的递送溶媒。
短语“药学上可接受的”或“药理学上可接受的”是指当施用于动物或人时不会产生不利反应、过敏反应或其他不良反应的分子实体和组合物。如本文所用,“药学上可接受的载体”包括可接受的用于配制药物(如适合施用于人的药物)的溶剂、缓冲液、溶液、分散介质、包衣、抗菌剂和抗真菌剂、等渗剂和吸收延迟剂等。这类介质和药剂用于药物活性物质的用途是本领域熟知的。
除了与本公开的活性成分不相容的任何常规介质或药剂之外,其在治疗组合物中的用途是预期的。补充的活性成分也可掺入组合物中,只要它们不使组合物的核酸失活即可。
适合于注射使用或导管递送的药物形式包括例如无菌水溶液或分散体和用于临时制备无菌可注射溶液或分散体的无菌粉末。通常,这些制剂是无菌的达到易注射程度的流体。制剂在制造和储存条件下应当是稳定的,并且应当保存以防止微生物(如细菌和真菌)的污染作用。合适的溶剂或分散介质可包含例如水、乙醇、多元醇(如,甘油、丙二醇和液体聚乙二醇等)、它们的合适混合物、以及植物油。例如,通过使用包衣如卵磷脂,通过在分散的情况下维持所需的粒度和通过使用表面活性剂,可以维持适当的流动性。可通过各种抗菌剂和抗真菌剂如对羟苯甲酸酯类、氯丁醇、苯酚、山梨酸、硫柳汞等带来防止微生物的作用。在许多情况下,优选包括等渗剂,例如糖或氯化钠。可通过在组合物中使用延迟吸收的药剂(如单硬脂酸铝和明胶)带来可注射组合物的延长吸收。
无菌可注射溶液可通过将适量的活性化合物与所需的任何其他成分(例如,如上列举的)一起掺入溶剂中,然后过滤灭菌来制备。通常,通过将各种灭菌的活性成分掺入含有基本分散介质和所需的其他成分(例如,如上所列举的)的无菌溶媒中来制备分散体。在用于制备无菌可注射溶液的无菌粉末的情况下,优选的制备方法包括真空干燥和冷冻干燥技术,产生活性成分加上任何额外的所需成分的粉末,所述任何额外的所需成分来自它们的先前无菌过滤的溶液。
本发明的组合物通常可配制成中性或盐形式。药学上可接受的盐包括,例如源自无机酸(如盐酸或磷酸)或源自有机酸(如乙酸、草酸、酒石酸、扁桃酸等)的酸加成盐(其与蛋白质的游离氨基形成)。与蛋白质的游离羧基形成的盐也可源自无机碱(如氢氧化钠、氢氧化钾、氢氧化铵、氢氧化钙或氢氧化铁)或有机碱(如异丙胺、三甲胺、组氨酸、普鲁卡因等)。
配制时,溶液优选以与剂量制剂相容的方式且以治疗有效量施用。制剂可易于以各种剂型施用,例如可注射溶液、药物释放胶囊等。对于以水溶液进行肠胃外施用,例如,溶液通常是适当缓冲的,并且液体稀释剂首先例如用足够的盐水或葡萄糖提供等渗。这类水溶液可用于例如静脉内、肌肉内、皮下和腹膜内施用。优选地,使用无菌水性介质,这是本领域技术人员已知的,特别是鉴于本公开。作为例证,可将单个剂量溶解于1ml等渗NaCl溶液中,或添加到1000ml皮下输注液体,或在建议的输注部位注射(参见,例如,"Remington'sPharmaceutical Sciences"15th Edition,pages 1035-1038and 1570-1580)。用量必然根据待治疗对象的状况发生一些变化。在任何情况下,负责施用的人将决定个体对象的适当剂量。此外,对于人施用,制剂应满足FDA生物制品标准局(FDA Office of Biologiesstandards)所要求的无菌、致热原性和一般安全性和纯度标准。
本发明的药物组合物还可置于注射器、植入装置等中,这取决于预期的递送和使用方式。优选地,如本文所述制备的包含人工RNA(如人工环状RNA)的组合物为单位剂型,意指预先测量或预先包装形式的适合单个剂量的组合物的量。
对于施用,可将至少一个治疗有效周期的人工RNA(如人工环状RNA)治疗施用于对象,以治疗由例如HCV、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒、黄热病毒或冠状病毒(如SARS和/或MERS,优选SARS-CoV-2)引起的病毒感染。
包含人工RNA(如人工环状RNA)的组合物的“治疗有效剂量或量”意指如本文所述进行施用时引起积极治疗反应,如从治疗的病症(如,例如病毒感染、癌症或遗传病症)中改善恢复的量。
将施用多个治疗有效剂量的包含本发明的人工RNA(如本发明的人工环状RNA)和/或一种或多种其他治疗剂的组合物。本发明的组合物通常(但不是必须)经由注射(皮下、静脉内、动脉内或肌肉内)、通过输注或局部施用。还考虑了其他施用方式,例如腹膜内、鞘内、肿瘤内、淋巴内、血管内、病灶内、透皮等。在一些实施方式中,局部施用包含本发明的人工RNA(如人工环状RNA)的药物组合物。根据本领域已知的任何医学上可接受的方法,可使用相同或不同的施用途径来施用包含本发明的人工RNA(如人工环状RNA)和其他药剂的药物组合物。
也可预防性施用包含本发明的人工RNA(如人工环状RNA)的药物组合物,例如,以预防病毒感染和/或癌症和/或遗传病症。
包含本发明的人工RNA(如人工环状RNA)和/或其他药剂的药物组合物在持续释放制剂或使用持续释放装置施用的制剂中。这类装置在本领域中是公知的,并且包括例如可植入微型泵,该可植入微型泵可以以各种剂量以连续稳态的方式随时间提供递送,以实现非持续释放的药物组合物的持续释放效果。
本领域普通技术人员将理解包含本发明人工RNA的组合物可有效治疗和/或预防的状况。待施用的实际剂量将随对象的年龄、体重和一般状况以及所治疗病症的严重程度、健康护理专业人员的判断和所施用的缀合物而变化。
治疗有效量可由本领域技术人员确定,并根据每种具体情况的具体要求进行调整。包含本发明的人工RNA(如人工环状RNA)的组合物可单独施用或与一种或多种其他治疗剂联合施用。具体的给药方案是本领域普通技术人员已知的或可使用常规方法通过实验来确定。示例性给药方案包括但不限于施用每日五次、每日四次、每日三次、每日两次、每日一次、每周三次、每周两次、每周一次、每月两次、每月一次及其任意组合。优选的组合物是需要一天给药不超过一次的那些。
包含本发明的人工RNA(如人工环状RNA)的组合物可在其他药剂之前、同时或之后施用。如果与其他药剂同时提供,本发明的人工RNA可在同一组合物或不同组合物中提供。因此,人工RNA和一种或多种其他药剂可通过同时治疗的方式提供给个体。
“同时治疗”意指施用于对象,使得在接受治疗的对象中引起物质组合的治疗效果。例如,根据特定的给药方案,通过施用一定剂量的包含根据本发明的人工RNA(如人工环状RNA)的药物组合物和一定剂量的包含至少一种其他药剂的药物组合物(其组合包含治疗有效剂量)可实现同时治疗。类似地,可以以至少一个治疗剂量施用本发明的人工RNA和一种或多种其他治疗剂。单独的药物组合物的施用可同时或在不同的时间(即,在同一天或不同天以任一顺序依次)进行,只要这些物质的组合在经历治疗的对象中引起治疗效果即可。
第三方面,本发明提供了试剂盒,其包括在任何实施方式中的本发明的人工RNA或包括本发明的组合物,以及用于使用所述人工RNA或组合物的说明书。
因此,本文所述的任何人工RNA和/或组合物可包括于试剂盒中。例如,本文公开的环状RNA可包括于试剂盒中。试剂盒还可包括一种或多种转染试剂以便于向细胞递送人工RNA。这类试剂盒还可包括保存多核苷酸或防止其降解的组分。这类组分可以是无RNA酶或抗RNA酶的。
这类试剂盒通常将以合适的手段包括用于每个单独试剂或溶液的不同容器。试剂盒可包括一个或多个装有人工RNA和其他药剂的容器。用于组合物的合适容器包括,例如瓶、小瓶、注射器和试管。容器可由包括玻璃或塑料在内的多种材料制成。容器可具有无菌进入孔(例如,容器可以是具有可被皮下注射针刺穿的塞子的小瓶)。
试剂盒还可包括含有药学上可接受的缓冲液如磷酸盐缓冲盐水,林格氏溶液或右旋糖溶液的容器。还可包括对终端用户有用的其他材料,包括其他药学上可接受的配制溶液如缓冲液、稀释剂、过滤器、针头和注射器或其他递送装置。所述递送装置可预先装填所述组合物。
试剂盒还可包括包装插页,所述包装插页包括使用本发明的人工RNA治疗病毒感染的方法的书面说明书。包装插页可以是未经批准的包装插页草案或可以是经食品和药品管理局(FDA)或其他管理机构批准的包装插页。
如上所述,优选地,本发明的组合物或试剂盒中可包含在任何实施方式中的本发明的人工RNA,所述人工RNA包含如以下所定义的核苷酸序列:SEQ ID NO.:2、3、4、5、6(针对HCV);8、9、10(针对登革病毒);12、13、14、15、39(针对基孔肯亚病毒);16和17(针对HCV和登革病毒的广谱活性);24和19(针对西尼罗病毒);21、22和23(针对登革病毒和西尼罗病毒的广谱活性);32(针对HCV和登革病毒的广谱活性),36、38、40、41、42、88、89、65、43、44、45、66、67、46、47、48、49、68、69、70、50、51、52、53、71、72、54、55、56、57(针对SARS-CoV-2病毒)。这些是与包含于下面实施例中描述的人工RNA的杂交区域完全杂交的靶杂交区域。
优选地,本发明的组合物或试剂盒中也可包含在任何实施方式中的本发明的人工RNA,与包含于所述人工RNA中的至少一个杂交区域完全杂交的一个或多个靶杂交区域包含于SEQ ID NO.:1、SEQ ID No.:7、SEQ ID No.:11、SEQ ID No.:20和/或SEQ ID NO.:34中。
第四方面,本发明提供了用作药物的在任何实施方式中的本发明的人工RNA、或本发明的组合物或试剂盒。优选地,本发明提供了用于预防和/或治疗病毒感染的方法中的在任何实施方式中的本发明的人工RNA、或本发明的组合物或试剂盒。优选地,本发明的人工RNA对两种或更多种RNA病毒具有广谱活性。
在另一个实施方式中,本发明提供了用于预防和/或治疗癌症的方法中的在任何实施方式中的本发明的人工RNA、或本发明的组合物或试剂盒。
在另一个实施方式中,本发明提供了用于预防和/或治疗遗传病症的方法中的在任何实施方式中的本发明的人工RNA,或本发明的组合物或试剂盒。
在本发明的上下文中,遗传病症是指由基因组中的一种或多种异常引起的健康问题。可由单个基因(单基因)或多个基因(多基因)中的突变或由染色体异常引起。遗传病症的示例如下:家族性高胆固醇血症、多囊性肾病、I型神经纤维瘤病、遗传性球形红细胞症、马凡(Marfan)综合征、亨廷顿(Huntington's)病、镰状细胞贫血、囊性纤维化、泰-萨克斯(Tay-Sachs)病、苯丙酮尿症、粘多醣贮积症、溶酶体酸性脂肪酶缺乏症、糖原贮积病、半乳糖血症、杜氏(Duchenne)肌营养不良症或血友病。
因此,第四方面,本发明提供了在任何实施方式中的本发明的人工RNA、或本发明的组合物或试剂盒在制备用于预防和/或治疗病毒感染和/或癌症和/或遗传病症的药物中的用途。
在优选实施方式中,病毒感染由HCV、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒、黄热病毒或冠状病毒(如SARS和/或MERS,优选SARS-CoV-2)引起。
如本领域所熟知的(参见例如WO 2017/222911),任何已知的核酸递送方法(如免疫原性或非免疫原性的)可用于将本文公开的人工RNA施用于对象。例如,可通过体内转染施用人工RNA。或者,人工RNA可通过离体转染,随后将用人工RNA转染的细胞转移至对象来施用。
人工RNA(如环状RNA)可与核酸载体(如,阳离子载体)或纳米颗粒(如,脂质纳米颗粒、聚合物纳米颗粒、包含聚合物和肽的组合的纳米颗粒、或静电复合物)一起递送。可使用重组病毒、外来体、脂质体或其他脂囊泡、或工程化以分泌人工RNA(如环状RNA)的细胞将人工RNA递送至对象。
人工RNA(如环状RNA)也可与靶向配体(如小分子、肽或蛋白质)缀合,用于局部递送至对象的特定部位(如,细胞、组织或器官)。人工RNA甚至可与内化序列、蛋白质转导结构域或细胞穿透肽连接以利于进入细胞。
第五方面,本发明提供了用于生产本发明的人工RNA(优选本发明的人工环状RNA)的方法,其中所述方法包括以下步骤:
a)合成本发明的人工RNA;
b)筛选步骤a)的人工RNA,选出能够破坏一个或多个RNA片段的一个或多个靶破坏结构、优选能够破坏SRVVLC和/或使病毒不太能(理想地不能)执行其生命周期的基本功能的人工RNA,即,能够在靶RNA片段中产生构象变化(如,在病毒的RNA中产生阻碍病毒生命周期的基本功能的构象变化)的人工RNA。
circRNA的产生
使用内部软件工具设计人工RNA,如环RNA。该工具允许考虑几个设计约束条件,例如序列的GC含量范围。一旦设计了人工RNA(如circRNA),它们可以在细胞内或通过体外转录产生。
(1)人工RNA(如circRNA)的细胞内产生
DNA形式的相应人工RNA序列(如circRN序列)通过商购获得并使用以下步骤克隆以随后转化到细胞中。
首先,将设计的人工RNA序列在介导剪接和环化的两个序列SA(剪接受体)和SD(剪接供体)之间克隆到质粒(如,pcDNA3-CIRS7质粒)中(克隆由the University of Aarhus(Denmark)的Thomas B.Hansen博士馈赠)(专利申请:WO2014/082644A1)。该质粒含有赋予抗生素氨苄青霉素抗性的基因。然后将衍生的质粒转化到XL1-Blue感受态细胞(Sigma)中。将转化的细胞在含有氨苄青霉素(LB-Amp)的LB培养基中生长进行选择。然后将选择的克隆之一在LB-Amp液体培养基中生长36小时进行扩增。此后,离心培养物以获得细菌细胞,并使用
将这些衍生的质粒导入人细胞时,转录启动子CMV由细胞转录机制识别,并且使用宿主转录机制表达mRNA。为了产生circRNA,通过宿主剪接机制使所得到的RNA环化,从而产生所设计的circRNA(参见图1)。
人工circRNA的体外产生
还可通过化学合成或体外转录获得相应的线性RNA,然后用RNA连接酶使其环化,从而在体外实现circRNA的产生(参见图2)。
更有效的方法是在体外由侧翼有T7启动子的模板产生circRNA。该质粒含有自切割核酶,其在体外转录后被切割,产生允许环化并产生circRNA的末端。对于每个候选物,于37℃下在30分钟期间使用HindIII(FastDigest)将T7启动子质粒线性化。纯化的线性化分子在体外转录并从聚丙烯酰胺凝胶纯化。然后,将使用RtcB(NEB)使RNA环化,并于37℃下在30’期间RNAse R(Lucigen)处理后选择环化的分子(参见图25)。
更具体地,circRNA由含有在两端被核酶包围的感兴趣的circRNA的T7-衍生质粒产生,所述核酶是具有自切割能力的RNA分子。在5’使用茄子潜隐类病毒(ELVd)锤头和在3’使用珊瑚锤头作为一对锤头。在5’锤头中观察到较低的切割效率时,测试了几种天然和人工的核酶,以便决定哪对锤头更有效。如图29所示,除编号3和7外的所有测试的锤头都以不同的切割效率产生所需的切割的RNA分子,锤头5-6-9是最佳的。在从凝胶中提取相应切割的RNA分子的条带后,在三个锤头中观察到类似的量,然而,决定使用人工核酶3(ART-RBZ3),因为该锤头的行为方式与计算机上预测的相同。
然后,测试了在缓冲液中使用不同浓度的镁是否可以增加部分裂解/完全裂解的比率,但是未观察到任何差异(图30)。
一旦建立锤头,继续产生所需的circRNA。按照制造商的说明,将circRNA序列克隆到质粒中,并使用Hind III(Fast Digest)将10微克线性化。对线性化的DNA进行苯酚-氯仿纯化并进一步体外转录。此外,测试了用作模板的线性化质粒的最佳量和反应时间。观察到使用10微克或1微克线性化DNA,使用T7RNA聚合酶(Takara)温育6h(如制造商推荐的)或3h获得相同量的切割的RNA分子(图31)。因此,决定使用1微克模板和3h反应进行IVT。
终止IVT并在尿素凝胶中跑胶4小时,切下所需的条带(完全切割),并进一步从中分离RNA。最后一步产生低产量的回收的RNA,需要大量的时间来产生足够的线性分子以进一步继续该方案。为了改善这一点,通过氯仿/异戊醇萃取代替凝胶跑胶来纯化IVT,这在回收方面具有更好结果。
接着,使用RtcB(New England Biolabs)于37℃下1小时期间进行10微克纯化的线性RNA的环化。为了消除非环状RNA形式,按照制造商说明,将所得到的RNA用RNAse R(Lucigen)处理并纯化。为了验证RNAse R处理,进行尿素凝胶跑胶并选择circRNA条带。如前所述,尿素凝胶纯化引起RNA材料明显损失。因此,决定避免凝胶纯化,增加RNAse R条件(图32)并如先前在方案中进行纯化,获得较高量的circRNA。该方案中的这种改变(参见图25)是以合理的成本和时间获得大量circRNA的关键。最后,cirRNA的存在用TBE-尿素凝胶仅使用1%的材料来验证。
作用方式
设计人工RNA,如circRNA,使得它们与存在于一个或多个RNA片段(如RNA病毒基因组)的靶破坏结构中的选定区域(靶杂交区域)杂交。如上所述,这些靶破坏结构与RNA片段的功能有关。例如,如果RNA片段是RNA病毒基因组,则靶破坏结构可能与病毒RNA的多种功能和感染性相关,例如翻译、复制、定位或/和衣壳化。这些信号可存在于病毒RNA基因组的5’和3’非翻译区和编码序列区(CDS)中。在一个实施方式中,这些靶破坏结构可位于IRES元件(内部核糖体进入位点)内,IRES元件负责捕捉宿主核糖体以启动翻译一些病毒RNA基因组的病毒蛋白。
与现有技术相比,作用机制不仅仅是人工RNA的杂交区域和靶破坏结构的靶杂交区域之间的杂交,还有在该杂交时触发的结构变化将使RNA片段的二级结构改变,最终影响其功能。例如,病毒RNA基因组和病毒mRNA包含对其功能必需的高度结构化区域。所设计的人工RNA与病毒RNA基因组中的靶破坏结构的结合将导致结构展开(二级结构的变化),并因此导致感染性的降低或甚至抑制(参见,例如图3)。
例如,RNA病毒基因组中靶破坏结构的破坏可干扰RNA结合蛋白(RBP)对其靶病毒RNA序列的可及性。注意,该策略与目的在于杂交RBP的确切结合位点的策略不同(参见,例如图4)。
有时,RBP结合需要见于在特定结构环境中的单链RNA区域,例如,紧接茎环的3’(参见,例如图4,左侧)。在我们的情况下,与不同的靶杂交区域杂交可能破坏该茎环(其是靶破坏结构的一部分),并且尽管结合位点保持可接近,但是结构变化使得RBP结合无效(参见,例如图4,右侧)。
结构
所设计的人工RNA(如circRNA)的结构非常有柔性。通常,它包含至少一个、优选几个杂交区域,所述杂交区域与存在于(优选病毒基因组中的)一个或多个RNA片段的至少一个、优选几个靶破坏结构中的至少一个、优选几个靶杂交区域杂交。人工RNA中的这些优选几个杂交区域优选地被准随机的序列分隔,从某种意义上说,所述准随机的序列不执行除分隔优选几个杂交区域之外的任何功能,并且允许遵守设计约束条件(低二级结构,特定GC含量等)。例如,图5中描述了人工RNA的示例,该人工RNA是靶向病毒基因组的3个不同靶破坏结构的circRNA,其中每个靶破坏结构有2个杂交区域。具有相同颜色的区域代表它们靶向同一病毒靶破坏结构的事实。
然而,如上所述,优选地将靶向特定靶破坏结构内同一靶杂交区域的人工RNA(如circRNA)的这些优选几个杂交区域设计为彼此不同。这是可能的,因为RNA允许三种不同的碱基配对类型:G-C、A-U和G-U。该事实还导致,例如病毒从人工RNA逃逸(通过突变)更困难,因为它需要同时逃逸所有不同的杂交区域。注意杂交要求不同于反义或互补性要求。
还可设计单个人工RNA的杂交区域,以靶向不同RNA片段的不同区域,例如不同RNA病毒的不同区域,从而获得广谱人工RNA。
第六方面,本发明提供了筛查人工环状RNA的方法,所述人工环状RNA包含两个或更多个杂交区域,所述杂交区域能够通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构,其中所述靶破坏结构被定义为包含:
i.第一区域,所述第一区域具有在未配对核苷酸的第二区域之后或之前的至少发夹环;和
ii.至少一个靶杂交区域,所述靶杂交区域包含在至少5个核苷酸、优选10个或更多个核苷酸的双链区之后或之前的至少2个核苷酸、优选3个或更多个核苷酸的单链区,并且
其中所述方法包括以下步骤:
a)鉴别作为总长度为7至100个核苷酸、优选10至50个核苷酸的那些区域的人工环状RNA的两个或更多个杂交区域,当与所述至少一个靶杂交区域杂交时,所述两个或更多个杂交区域与所述至少一个靶杂交区域之间的杂交能量比所述靶破坏结构的能量更负,从而破坏所述一个或多个靶破坏结构,其中包含于所述人工环状RNA中的所述两个或更多个杂交区域中的每一个通过RNA反向折叠工具如NUPACK、RNAifold或MoiRNAiFold来鉴别,
b)将步骤a)中鉴别的人工环状RNA设计为包含能够破坏所述一个或多个靶破坏结构的两个或更多个杂交区域,其中所述人工环状RNA的长度为150至800个核苷酸,优选200至600个核苷酸,
c)任选地选择步骤b)中所设计的能够通过杂交破坏所述一个或多个靶破坏结构的人工环状RNA,以及任选地将所述人工环状RNA包装成产品。
如上所述,在根据第六方面或其任何实施方式的方法的步骤a)中,根据步骤a)的两个或更多个杂交区域的鉴别通过RNA反向折叠工具如NUPACK、RNAifold、MoiRNAiFold进行,其中所述方法优选包括以下步骤:
a)通过向RNA反向折叠工具提供至少靶破坏结构/序列和靶杂交区域两者,产生与包含于一个或多个RNA片段的一个或多个靶破坏结构中的至少一个靶杂交区域完全杂交的可能的两个或更多个杂交区域的一个或多个核苷酸序列,
b)获得那些杂交区域的仅一个或多个核苷酸序列作为输出,当与靶杂交区域杂交时,两个或更多个杂交区域与至少一个靶杂交区域之间的杂交能量比靶破坏区域的能量更负,从而破坏靶破坏结构。
在根据第六方面或其任何实施方式的方法的优选实施方式中,所述方法包括将一个或多个RNA片段中包含以下的那些结构鉴别为靶破坏结构的预步骤:
a)第一区域,所述第一区域具有在未配对核苷酸的第二区域之后或之前的至少发夹环;和
b)至少一个靶杂交区域,所述靶杂交区域包含在至少5个核苷酸、优选10个或更多个核苷酸的双链区之后或之前的至少2个核苷酸、优选3个或更多个核苷酸的单链区。
在根据第六方面或其任何实施方式的方法的优选实施方式中,所述方法包括进一步的步骤d),该步骤在生物环境中确认步骤b)中所设计的人工环状RNA通过杂交破坏一个或多个RNA片段的一个或多个靶破坏结构的能力。
在优选实施方式中,根据第六方面或其任何实施方式的方法包括使用以上表1至表7中所定义的靶破坏结构和靶杂交区域。
现在通过参考以下实施例进一步阐述本发明,所述实施例并不打算限制本发明范围。
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
实施例
实施例1
如先前在circRNA的产生部分中所公开,已设计并测试了针对HCV基因组(参见SEQID NO.:1)的两个不同区域的4种circRNA(circ_hcv_ires1(SEQ ID NO.:2)、circ_hcv_cds1(SEQ ID NO.:4)、circ_hcv_cds2(SEQ ID NO.:6)y circ_hcv_combo1(SEQ ID NO.:5))。circ_hcv_ires1含有靶向IRES元件中的序列的7个长度为33个核苷酸的杂交位点。circ_hcv_cds1包含靶向编码区中的序列的8个长度为28个核苷酸的杂交位点。circ_hcv_cds2包含靶向编码区中的序列的12个杂交位点。circ_hcv_combo1每个靶区域(IRES1(SEQID NO.:25)、IRES2(SEQ ID NO.:26)和cHP(CDS1)(SEQ ID NO.:27))包含3个杂交位点。HCV基因组中的靶区域描述于图6和SEQ ID NO.:1中。IRES1(结构域IV)和IRES2(结构域V)靶破坏结构在[C Romero-López et al.2007(Cell Mol Life Sci.2007Nov;64(22):2994-3006.doi:10.1007/s00018-007-7345-y)https://pubmed.ncbi.nlm.nih.gov/17938858/]中被认为是SRVVLC。CDS1(cHP)和CDS2(SL427)靶破坏结构在[Pirakitikulr et al.2016(Molecular Cell 62,111–120April 7,2016a2016 Elsevier Inc.http://dx.doi.org/10.1016/j.molcel.2016.01.024)]中被认为是SRVVLC。
细胞培养和转染
在Huh7细胞中转染每种circRNA,转染后24小时,用表达荧光素酶的HCV衍生物感染细胞。感染后48小时后,测量荧光素酶水平,并将这些值与对照(模拟转染)进行比较。对每个条件进行5个生物学重复。所有测试的circRNA均显著抑制HCV感染。如图7所示,所有设计的circRNA都能够以相对于对照的30%至70%范围的不同效率降低感染。
实施例2
如先前在circRNA的产生部分中所公开,已设计并测试了针对DENV(登革病毒)基因组(参见SEQ ID NO.:7)的两个不同区域的3种circRNA(circ_dv_3utr(SEQ ID NO.:8),circ_dv_cHP_v1(SEQ ID NO.:9)和circ_dv_cHP_v2(SEQ ID NO.:10))。先前已描述了产生携带海肾荧光素酶报告基因的DENV的质粒(pFK-DVs-R2A)(Scaturro,P.etal.Characterization of the mode of action of a potent dengue virus capsidinhibitor.J.Virol.88,11540–55(2014))。所有三种circRNA都包含靶向DENV基因组中相应区域的7个杂交区域(参见图8和SEQ ID NO.:7)。对应于circ_dv_3utr(sHP)的靶破坏结构在[Huber et al.2019(Nature Communications volume 10,Article number:1408(2019)),and the target disruption structure cHP was considered SRVVLC in Fernández-Sanlés et al.2017(Front Microbiol.2017;8:546,doi:10.3389/fmicb.2017.00546)]中被认为是SRVVLC。
细胞培养和转染
将人胚胎肾细胞系HEK293维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5%CO
结果示于图9中,并描述了DENV感染相对于对照(空质粒)的显著抑制。
实施例3
如先前在circRNA的产生部分中所公开,已设计并测试了针对CHIKV(基孔肯亚病毒)基因组(SEQ ID NO.:11)的三个不同区域的5种circRNA(chikv_5utr1(SEQ ID NO.:12)、chikv_5utr2(SEQ ID NO.:13)、chikv_RSE1(SEQ ID NO.:14)、chikv_RSE2(SEQ IDNO.:15)和chikv_RE(SEQ ID NO.:39)。产生携带高斯荧光素酶的报告基因的CHIKV的质粒(由Dr.Merits,University of Tartu,Estonia馈赠)直接基于从来自La Reunion的人患者(分离株LR2006_OPY1(DQ443544)中分离的病毒序列。所有5种circRNA都包含靶向CHIKV基因组(SEQ ID NO:11)中相应区域的6个杂交区域。CHIKV的5’UTR的靶破坏结构和靶破坏结构“重复序列结构”在[Hossain Khan et al.,2002(Journal of General Virology(2002),83,3075–3084)]中都被认为是SRVVLC。重编码元件,即CHIKV的第三靶破坏结构,在[A.Kendra et al.,2018(J.Biol.Chem.(2018)293(45)17536–17545)]中被认为是SRVVLC。
细胞培养和转染
将人胚胎肾细胞系HEK293维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5%CO
结果显示在图10中,并描述了CHIKV感染相对于对照(空质粒)的显著抑制。
实施例4
如先前在circRNA的产生部分中所公开,已设计并测试了针对DENV(登革病毒)基因组(SEQ ID NO.:7)的一个区域和HCV基因组(SEQ ID NO.:1)的一个区域的3种circRNA(DENV1 cHP_HCV CDS2_1(SEQ ID NO.:17)、DENV cHP_HCV CDS2_2(circ_dv_cHP_v1_circ_hcv_cds2_2,SEQ ID NO.:32)和(DENV1 cHP_HCV CDS2_T(SEQ ID NO.:16)。先前已描述了产生携带萤火虫荧光素酶报告基因的HCV的质粒(pFK-Luc-Jc1)(Wakita T et al.,Production of infectious hepatitis C virus in tissue culture from a clonedviral genome,Nat Med,2005;11:791-796)。先前已经描述了产生携带海肾荧光素酶报告基因的DENV的质粒(pFK-DVs-R2A)(Scaturro,P.et al.,Characterization of the modeof action of a potent dengue virus capsid inhibitor,J.Virol.88,11540-55(2014))。
前两种circRNA包含靶向DENV基因组和HCV基因组中相应区域的6个杂交区域,最后一种circRNA包含针对DENV的7个杂交区域和针对HCV的12个杂交区域。
细胞培养和转染
将人胚胎肾细胞系HEK293维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5%CO
将人肝癌细胞系Huh7/Scr维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5%CO
过夜温育后,除去DMEM培养基,细胞用1X PBS洗涤。细胞于37℃用不同的病毒接种4小时。最后,用新鲜培养基更换含有病毒的培养基。感染后48小时,测定荧光素酶活性。细胞用1X PBS洗涤,于150μl Passive裂解缓冲液中裂解并冷冻。解冻后,将裂解物通过吹打重悬。将50μl裂解物与25μl荧光素酶测定试剂(Promega)混合,并于室温下温育5分钟。过后,在光度计中测量荧光素酶活性2秒。以相对于对照感染(用circoVIR质粒转染的细胞)的百分比绘制平均相对光单位(RLU)。
图11、图12、图13、图14和图24中显示针对DENV和HCV的各circRNA以及来自实施例1和2的相应阳性和阴性对照的结果。所有circRNA在两种病毒感染中都显示抑制,表明设计circRNA的广谱能力。
实施例5
如先前在circRNA的产生部分中所公开,已设计并测试了针对WMV(西尼罗病毒)基因组(SEQ ID NO.:20)的一个区域的2种circRNA(circ_wnv_slII_1(SEQ ID NO.:24),circ_wnv_slII_2(SEQ ID NO.:19))。产生携带Nanoluc荧光素酶报告基因的WNV的质粒由University of Tartu,Estonia的Dr.Merits馈赠。两种circRNA都包含靶向WNV基因组中相应区域的7个杂交区域。靶破坏结构SLII在[Fernández-Sanlés et al.,2017(FrontMicrobiol.2017;8:546doi:10.3389/fmicb.2017.00546)]中被认为是SRVVLC。
细胞培养和转染
将人胚胎肾细胞系HEK293维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5%CO
结果示于图15中,并描述了WNV感染相对于对照(空质粒)的显著抑制。
实施例6
如先前在circRNA的产生部分中所公开,已设计并测试了针对DENV(登革病毒)基因组(SEQ ID NO.:7)的一个区域和WNV基因组(SEQ ID NO.:20)的一个区域的3种circRNA(dchp_wsIlI_A(SEQ ID NO.:21)、dchp_wsIlI_B(SEQ ID NO.:22)和dchp_wsIlI_C(SEQ IDNO.:23)。先前已经描述了产生携带海肾荧光素酶报告基因的DENV的质粒(pFK-DVs-R2A)(Scaturro,P.et al.,Characterization of the mode of action of a potent denguevirus capsid inhibitor,J.Virol.88,11540–55(2014))。产生携带Nanoluc荧光素酶报告基因的WNV的质粒由University of Tartu,Estonia的Dr.Merits馈赠。circRNA包含靶向DENV基因组和WNV基因组中相应区域的6个杂交区域。
细胞培养和转染
将人胚胎肾细胞系HEK293维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5%CO
图16和图17显示针对DENV和WNV的各circRNA以及来自实施例2和5的相应阳性和阴性对照的结果。circRNA在两种病毒感染中都显示抑制,表明设计circRNA的广谱能力。
实施例7
测试circ_hcv_cds2(SEQ ID NO.:6)是否可抑制已建立的感染。为此,Huh7/Scr细胞用HCVJC1-luc感染,48小时后用circ_hcv_cds2(SEQ ID NO.:6)转染。转染后24小时测量荧光素酶值。重要地,circ_hcv_cds2以与感染前细胞表达circ_hcv_cds2时相似的效率抑制感染性(图18)。
实施例8
接着,检验circ_hcv_cds2(SEQ ID NO.:6)是否确实抑制所描述的靶序列功能,病毒RNA复制。为此,使用携带荧光素酶报道基因但不携带衣壳化所需的病毒结构基因的HCVRNA复制子。因此,这些复制子允许病毒RNA基因组的有效翻译和复制,但不允许病毒体产生。Huh7/Scr细胞用circ_hcv_cds2(SEQ IDNO.:6)或相应的空质粒转染,第二天用HCV复制子转染。在HCV复制子转染后4小时和48小时测量荧光素酶值。选择这些时间是因为已建立的动力学证明产生的荧光素酶在4小时时仅来自HCV RNA翻译,且在48小时时来自翻译和复制。确实,在转染后48小时观测到病毒感染性降低,而4小时未观测到(图19),表明circ_hcv_cds2损害病毒RNA复制。
实施例9
按照与实施例8相同的步骤,设计circ_dv_3utr(SEQ ID NO.:8)和circ_dv_cHP_v1(SEQ ID NO.:9)靶向DENV RNA基因组中指导RNA复制的结构,并测试它们是否抑制DENVRNA复制。结果表明,circ_dv_3utr和circ_dv_cHP_v1在48小时(RNA基因组被翻译和复制时)抑制荧光素酶表达水平,但在8小时(RNA基因组仅被翻译时)未抑制荧光素酶表达水平(图20)。
实施例10
将人胚胎肾细胞系HEK293和肝癌细胞系Huh7维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5% CO
实施例11
如先前在circRNA的产生部分中所公开,已设计并测试了针对SARS-CoV-2基因组(SEQ ID NO:58;SEQ ID NO:59;SEQ ID NO:60;SEQ ID NO:62)的四个区域的12种circRNA(circ_SARS_tA_4(SEQ ID NO.:49)、circ_SARS_tA_5(SEQ ID NO.:68)、circ_SARS_tA_6(SEQ ID NO.:69)、circ_SARS_tA_7(SEQ ID NO.:70)、circ_SARS_tC_3(SEQ ID NO.:52)、circ_SARS_tC_5(SEQ ID NO.:71)、circ_SARS_tC_6(SEQ ID NO.:72)、circ_SARS_3utr_6(SEQ ID NO.:63)、circ_SARS_3utr_7(SEQ ID NO.:89)、circ_SARS_3utr_8(SEQ ID NO.:65)、circ_SARS_5utr_4(SEQ ID NO.:66)、circ_SARS_5utr_5(SEQ ID NO.:67)。SARS-CoV-2株hCoV-19/Spain/VH000001133/2020(EPI_ISL_418860)由Universitat Autonoma deBarcelona的Miguel Chillon馈赠。所设计的circRNA包含靶向SARS-CoV-2基因组中相应区域的6至7个杂交区域。靶破坏结构复制位点在[J.Goebel et al.,2004(American Societyfor Microbiology.Journal of VirologyVolume 78,Issue14,Pages 7846-7851https://doi.org/10.1128/JVI.78.14.7846-7851.2004)]中被认为是SRVVLC。靶破坏结构SL-2在[Chen et al.,2010(Virology 401(2010)29–41doi:10.1016/j.virol.2010.02.007)]中被认为是SRVVLC。按照以上[Rangan et al.,2020(doi:https://doi.org/10.1101/2020.03.27.012906)]所提及的RNAz方法,其余的靶破坏结构(A、C和D)被认为是病毒保守结构。
实施例12细胞培养和转染
将绿色猴肾上皮细胞系VERO E6维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5% CO
图33中显示针对SARS-CoV-2的各circRNA的结果。circRNA在SARS-CoV-2病毒感染中显示抑制,表明所设计的circRNA的抗病毒作用。
实施例13
将绿色猴肾上皮细胞系VERO E6维持在补充有10%热灭活胎牛血清(FBS)和10%非必需氨基酸的杜氏改良伊格尔培养基(DMEM,Invitrogen,Carlsbad,CA)中。使细胞在37℃,具有5% CO
实施例14
在本实施例中,提供了一些RNAiFold的输入文件,以查验靶破坏结构是否确实可能通过杂交破坏(然后立即设计具有杂交区域的circRNA来破坏它)。
对于HCV靶破坏结构cHP和相应的靶杂交区域(SEQ ID NO.:27,以粗体突出显示):
#RNAscdstr
((((((((((((((((((((((((((((&)))))))))))))))))))))))))))),,,,,,,,,,,,,,,,,,,,,,,,
#RNAseqcon
NNNNNNNNNNNNNNNNNNNNNNNNNNNN&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
输入“((((((((((((((((((((((((((((&)))))))))))))))))))))))))))),,,,,,,,,,,,,,,,,,,,,,,,”意味着软件应当寻找长度为k个核苷酸的核苷酸序列,其中在最小自由能结构中位于对应于左括号“(”位置的核苷酸应当与位于对应右括号“)”位置的核苷酸碱基配对。逗号“,”意味着最小自由能结构中的对应位置的核苷酸可以是配对的碱基,也可以不是。&符号将两条链分开。请注意,在第二条链上的碱基配对的核苷酸(靶破坏结构)对应于靶杂交区域。
进一步地,输入:
“NNNNNNNNNNNNNNNNNNNNNNNNNNNN&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
意指固定可供选择的核苷酸,其中N表示任意核苷酸(A、C、G或U)。在这种情况下,靶破坏结构对应的核苷酸是固定的,并且杂交区域对应的所有核苷酸都是不固定的(N-任意核苷酸)。结果如下所示:
UCUGGGCGACGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UUUGGGCGACGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UCUGGGUGACGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UUUGGGUGACGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UCUGGGCGGCGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UUUGGGCGGCGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UCUGGGUGGCGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UUUGGGUGGCGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UCUGGGCGAUGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
UUUGGGCGAUGGUUGGUGUUUCUUUUGG&CCAAAAGAAACACCAACCGUCGCCCAGAAGACGUUAAGUUCCCGGGCGGCGG
对于CHIKV靶破坏结构RSE和相应的靶杂交区域(SEQ ID NO 35):
#RNAscdstr
(((((((((((((((((((((((((((((((((&)))))))))))))))))))))))))))))))))........
#RNAseqcon
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAAC
结果如下所示:
UUGCGUGGCCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGUGUGGCCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGCGUAGCCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGUGUAGCCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGCGUGGUCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGUGUGGUCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGCGUAGUCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGUGUAGUCCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGCGUAGCUCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
UUGUGUAGCUCUUUGGUCUGUAGGUUGUUUGCU&AGCAAAUAAUCUAUAGAUCAAAGGGCUACGCAACCCCUGAA
对于WNV靶破坏结构SLII和相应的靶杂交区域(SEQ ID NO 37):
#RNAscdstr
(((((((((((((((((((((((((((((&))))))))))))))))))))))))))))),,,,,,,,,,,,,,,,,,
#RNAseqcon
NNNNNNNNNNNNNNNNNNNNNNNNNNNNN&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
结果如下所示:
GAUUUUCUGGCCUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
AAUUUUCUGGCCUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
GAUUUUUUGGCCUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
AAUUUUUUGGCCUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
GAUUUUCUGGCUUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
AAUUUUCUGGCUUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
GAUUUUUUGGCUUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
AAUUUUUUGGCUUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
GACUUUCUGGCCUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
AACUUUCUGGCCUGGCUUUUUCCUCAAGA&UUUUGAGGAGAAAGUCAGGCCGGGAAGUUCCCGCCACC
对于SARS-Cov2靶破坏结构复制位点和相应的靶杂交区域(SEQ ID NO 62):
#RNAscdstr
(((((((((((((((((((((((((((((((((((&)))))))))))))))))))))))))))))))))))......
#RNAseqcon
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
结果如下所示:
CUACUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
UUACUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
CUGCUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
UUGCUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
CUAUUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
UUAUUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
CUGUUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
UUGUUUGUGCUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
CUGCUUGUGUUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
UUGCUUGUGUUAUGUAGUUACGGGAGUUCGUUCUG&CAGAAUGAAUUCUCGUAACUACAUAGCACAAG
在所有情况下,包括上述未描述的情况,RNAiFold返回多个解决方案,即对应于潜在的应通过杂交破坏相应的靶破坏结构的杂交区域的序列。
最后,我们在此描述了本发明中使用的公认点括号表示法,以获得每种病毒的靶杂交区域(在此以粗体突出显示):
HCV
靶1:IRES 1(结构域II)
SEQ ID NO.:76,Romero et al.2007(Cell Mol Life Sci.2007Nov;64(22):2994-3006.doi:10.1007/s00018-007-7345-y.)中的图1a:
...................((((.((((.....(((((..(((.((...((((((.......))))))....)).)))..).))))))))))))...
靶2:IRES 2(结构域IV)
SEQ ID NO.:77,Romero et al.(Cell Mol Life Sci.2007Nov;64(22):2994-3006.doi:10.1007/s00018-007-7345-y.)中的图1a:
...........((((.......)))).......
靶3:CDS1(cHP)
SEQ ID NO.:78;Pirakitikulr et al.2016(Pirakitikulr et al.,2016,Molecular Cell 62,111–120April 7,2016Elsevier Inc)中的图S1 B::
................(((((((((.(..(((.....)))..))))))))))
靶4:CDS2(SL427)
SEQ ID NO.:79;Pirakitikulr et al.2016(Pirakitikulr et al.,2016,Molecular Cell 62,111–120April 7,2016Elsevier Inc)中的图2a:.
(((((((..((((((...(((((((..(((((...((((......)))).))))).)))))))....)))))).)))))))
DNV
靶1:sHP
SEQ ID NO.:29;Huber et al.,2019(Huber et al.Nature Communicationsvolume 10,Article number:1408,2019)中的图1c:
...............((((......))))...
靶2:cHP
SEQ ID NO.:30;Sanlés et al.2017(Sanlés et al.2017.
......(((((.(........))))))......
CHIKV
靶1:5’UTR:
SEQ ID NO.:80,Khan et al.2002(Khan et al.Journal Of General VirologyVolume 83,Issue 12)中的图3A:
..(((((((((.....)))))))))....((((((((.((((........))..)).)))))))).....
靶2:重复序列元件:
SEQ ID NO.:81,Khan et al.2002(Khan et al.Journal Of General VirologyVolume 83,Issue 12)中的图4B:
.................(((..(((........))).))).
靶3:重编码元件:
SEQ ID NO.:82,Kendra et al.2018(Kendra et al.2018Protein SynthesisAnd Degradation|Volume 293,Issue 45,P17536-17545,November 09,2018)中的图4D:
.((((((..(((((((..(((....))).......((((.....)))))))).)))..)))).)).
...
WNV
靶1:SLII:
SEQ ID NO.:83,Sanlés et al.2017(Sanlés et al.2017.Front Microbiol.;8:546.)中的图3:
.............(.((((((((((...))))).(((((.............)))))..))))))..
SARS-CoV-2
靶1:复制位点在pangolin和RatG13中完全保守
SEQ ID NO.:84,Goebel et al.2004(Goebel et al..Journal Of Virology,July 2004,p.7846–7851)中的图1:
...............(((((((((.((....))..)))))))))....
靶2:SL-2,
SEQ ID NO.:58,Cheng Chen et al.2010(Cheng chen et al.Virology,Volume401,Issue 1,25May 2010,Pages29-41)中的图3b:
...........(((((.....))))).
靶3:靶A
SEQ ID NO.:85,Rangan et al.2020(Rangan et al.bioRxiv preprint doi:https://doi.org/10.1101/2020.03.27.012906)中的图2:
...............(((((.((((.(((((.......))))).)))).)))))..
靶4:靶C
SEQ ID NO.:86,Rangan et al.2020(Rangan et al.bioRxiv preprint doi:https://doi.org/10.1101/2020.03.27.012906)中的图2:
................((((((((((((...)))).))))))))..
靶5:靶D
SEQ ID NO.:87,Rangan et al.2020(Rangan et al.bioRxiv preprint doi:https://doi.org/10.1101/2020.03.27.012906)中的图2:
.................(((((.(((((......)))))..)))))...
本发明的其他项
本发明还包括以下项:
1.一种200至600个核苷酸的人工环状RNA,其具有针对一个或多个RNA病毒基因组的一个或多个结构化区域的大小为10至50个核苷酸的6至20个杂交区域,其中所述杂交区域具有彼此不同的序列,其中所述一个或多个结构化区域是在单链区之后的对病毒生命周期至关重要的结构化区域(SRVVLC),并且其中所述人工环状RNA能够破坏一个或多个SRVVLC的结构,从而使病毒的感染性降低。
2.项1的人工环状RNA,其中所述杂交区域:
a)由大小至多20个核苷酸的非杂交区域分隔;或
b)未由非杂交区分隔;或
c)是重叠的。
3.根据前述项中任一项所述的人工环状RNA,其中所述杂交区域的靶标是所述病毒基因组的IRES元件的SRVVLC。
4.根据项1或2中任一项所述的人工环状RNA,其中所述杂交区域的靶标是所述病毒基因组的5’UTR的SRVVLC。
5.根据项1或2中任一项所述的人工环状RNA,其中所述杂交区域的靶标是所述病毒基因组的CDS的SRVVLC。
6.根据项1或2所述的人工环状RNA,其中所述杂交区域的靶标是所述病毒基因组的3’UTR的SRVVLC。
7.根据项1至6中任一项所述的人工环状RNA,其中所述杂交区域的靶标是所述病毒基因组的IRES元件、5’UTR区、CDS区或3’UTR区中的两个或更多个的组合。
8.根据项3或7所述的人工环状RNA,其中所述病毒基因组选自HCV、HAV、脊髓灰质炎病毒、柯萨奇病毒B和鼻病毒(普通感冒)。
9.根据项4至7中任一项所述的人工环状RNA,其中所述病毒基因组选自HCV、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒和黄热病毒。
10.根据项1至9中任一项所述的人工环状RNA,其中所述环状RNA具有针对两种或更多种RNA病毒基因组的广谱活性。
11.一种组合物,其包含根据前述项中任一项所述的人工环状RNA。
12.一种试剂盒,其包括根据项1至10中任一项所述的人工环状RNA或根据项11所述的组合物、以及用于使用所述环状RNA或组合物的说明书。
13.根据项1至10中任一项所述的人工环状RNA、或根据项11所述的组合物、或根据项12所述的试剂盒,其中所述环状RNA包含选自以下的序列:SEQ ID NO.:2、4、5、6(针对HCV);8、9、10(针对登革病毒);12、13、14、15、39(针对基孔肯亚病毒);16和17(针对HCV和登革病毒的广谱活性);24和19(针对西尼罗病毒);21、22和23(针对登革病毒和西尼罗病毒的广谱活性);32(针对HCV和登革病毒的广谱活性),其中6至20个杂交区域的一个或多个靶标包含于SEQ ID NO.:1、SEQ ID No.:7、SEQ IDNo.:11和/或SEQ ID NO.:20中。
14.用于预防和/或治疗病毒感染的根据项1至10或13中任一项所述的人工环状RNA、或根据项11或13所述的组合物、或根据项12或13所述的试剂盒。
15.根据项14的用途的所述人工环状RNA或组合物或试剂盒,其中所述病毒感染由HCV、HAV、脊髓灰质炎病毒、柯萨奇病毒B、鼻病毒(普通感冒)、登革病毒、寨卡病毒、基孔肯亚病毒、西尼罗病毒或黄热病毒引起。
