用于RNA建库中快速去除靶RNA的逆转录阻碍探针及其应用
文献发布时间:2023-06-19 10:33:45
技术领域
本发明专利涉及用于RNA建库中快速去除靶RNA的逆转录阻碍探针及其应用,属于生物技术领域。
背景技术
近年来,随着二代测序技术的快速发展,各类RNA建库技术和测序技术层出不穷并且迭代升级,大大地降低了RNA建库和测序的难度和成本,这也使得RNA二代测序技术成为生命进展和疾病诊断等领域的重要研究手段。但是,不同来源的样本中的RNA种类占比具有极大的不均一性,如正常细胞中核糖体rRNA约占总RNA的90%-95%,血液中球蛋白的mRNA约占总mRNA的76%以上,这些RNA的存在占据了大部分的测序数据,严重影响到低丰度RNA的检测,提高了测序的成本。
现在主要有两类策略来去除这些在RNA建库过程中不需要的高丰度RNA。第一类策略为磁珠捕获法。比如polyA富集法,利用oligo(dT)磁珠来捕获富集含有polyA的mRNA,这种方法只适用于完整性较好的RNA,能够捕获的RNA种类有限,大大降低了测序的多样性,而且这种方法对polyA长度越长的RNA存在捕获效率越高,会造成RNA建库的偏好性;或者靶RNA捕获去除法,利用带有biotin修饰的反义DNA探针与靶RNA杂交后,用链亲和霉素磁珠对靶RNA进行去除。另一类种策略为核酸酶降解法。比如RNase H切割去除法,利用RNase H特异性识别并降解DNA:RNA杂交链中RNA的活性对靶RNA进行降解;或者CRISPR/Cas9切割法,利用sgRNA-Cas9系统对构建好的双链DNA文库中靶RNA来源的双链DNA进行切割。这些方法存在成本高、操作复杂(约20-40步)、耗时长(约2h)、对样本来源和RNA丰度要求高、试剂难以储存等缺陷,严重阻碍了RNA二代测序建库技术的工业自动化发展和其在疾病快速诊断领域的应用。
发明内容
本发明的目的是提供一种用于RNA建库中快速去除靶RNA的逆转录阻碍探针,可以快速去除RNA建库中的rRNA。
本发明采用的技术方案为:
一种用于RNA建库中快速去除靶RNA的逆转录阻碍探针,所述探针长度为20-30nt,与靶RNA序列严格互补配对,且与其他非靶RNA不存在连续超过15 nt 的互补配对,探针的Tm值不低于80℃,自身互补(Self complementarity)值小于5,探针的3’-OH利用修饰的脱氧核糖核苷酸进行封闭;所述探针有多个,探针覆盖靶RNA区域不低于全长RNA的1/6,探针在靶RNA上分布均匀,探针之间的距离不超过100 nt。
优选的,所述探针的脱氧核糖核苷酸包括一种或多种修饰,每种修饰的数量为1个或多个,修饰种类包括但不限于肽核酸(PNA)、锁核酸(LNA)、脱氧尿嘧啶核苷酸(dU)、MGB(核酸小沟嵌合物)、2-O-甲基核糖、5-羟基丁酸酰基-脱氧尿苷、2-氨基脱氧腺苷和5-甲基脱氧胞苷修饰,使得探针的Tm值在80℃-95 ℃之间。
优选的,所述修饰的位置靠近 探针的5’端,至少50%所述修饰的位置应处在探针5’端的前三分之一区域。
优选的,探针长度为20-30 nt,且与其他非靶RNA不存在连续超过10 nt的互补配对,自身互补值小于3。
优选的,探针覆盖靶RNA区域不低于全长RNA的1/3,探针之间的距离不超过30 nt。
所述靶RNA为5.8S rRNA,探针为表1所示的5.8S rRNA probe的混合物。
所述靶RNA为18S rRNA,探针为表1所示的18S rRNA probe的混合物。
所述靶RNA为28S rRNA,探针为表1所示的28S rRNA probe的混合物。
本发明还公开了上述的逆转录阻碍探针在RNA建库中快速去除靶RNA的应用。
优选的,其步骤包括:
(1)提取样本中的总RNA,在总RNA中加入逆转录阻碍探针和buffer,进行梯度退火反应;
(2)在反应产物中加入链特异性合成buffer和一链cDNA合成酶混合物,进行逆转录反应合成cDNA;
(3)二链cDNA合成及末端修复、接头连接、文库扩增及测序。
优选的,所述步骤(1)中梯度退火条件为:95℃ 5 min,75℃ 1 min, 55℃ 1 min,4 ℃ 保存。
优选的,所述步骤(2)中合成反应条件为:25℃ 10 min, 42℃ 15 min,70℃ 15min,4℃ 保存。
本发明利用逆转录阻碍探针法在一链cDNA合成时实现对靶RNA的逆转录进行高效率高特异性的抑制,从而阻碍靶RNA参与下游建库过程。本发明具有操作简单(一步操作)、耗时极短(2 min)、对非靶RNA保留完整、特异性强、效率高、成本低和基因检出数高等优点,非常适合用于工业自动化建库和疾病快速诊断领域。
附图说明
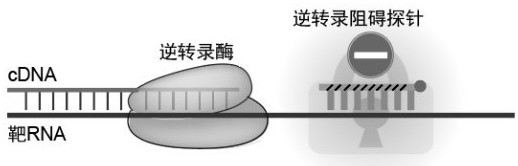
图1为逆转录阻碍探针法示意图。
图2为逆转录阻碍探针对5.8S rRNA逆转录合成的抑制示意图。
图3为qPCR法鉴定不同修饰种类探针对逆转录的抑制作用。
图4为qPCR法鉴定探针中LNA的分布位置对靶RNA逆转录的抑制效果。
图5为qPCR法鉴定不同长度LNA修饰探针对靶RNA逆转录的抑制效果。
图6为qPCR法鉴定不同浓度LNA修饰探针对靶RNA逆转录的抑制效果。
图7为梯度退火下全覆盖逆转录阻碍探针竞争结合靶RNA作用原理示意图。
图8为全覆盖NGS 5.8S rRNA probe mix 作用位点示意图(阴影代表探针靶位置,斜体加粗代表LNA修饰碱基对于的靶位点)。
图9为qPCR法鉴定全覆盖逆转录阻碍探针对5.8S rRNA逆转录合成的抑制作用。
图10为qPCR法鉴定全覆盖逆转录阻碍探针对18S rRNA逆转录合成的抑制作用。
图11为 qPCR法鉴定全覆盖逆转录阻碍探针对28S rRNA逆转录合成的抑制作用。
图12为 逆转录阻碍探针法在NGS建库过程中快速去除靶RNA原理示意图(仅需2min)。
图13为NGS RNA建库过程中三种去除rRNA方法的示意图。
图14为qPCR验证三种去除rRNA方法的效果。
图15为RNA-seq比较三种去除rRNA方法的效果。
图16为四种建库方法测序相关性。
图17为同等测序深度下四种建库方法基因检出数。
具体实施方式
下面结合附图对本发明的具体实施方式做进一步说明。
实施例1
通过以下详细说明结合附图可以进一步理解本发明的特点和优点。所提供的实施例仅是对本发明方法的说明,而不以任何方式限制本发明揭示的其余内容。本实施例所使用的探针和引物序列及修饰如表1所示,N为随机碱基,即A、T、C、G中任意一种碱基,RandomHexamers为用于逆转录的通用引物。
图1和图2展示了本发明的反应机理:利用强结合能力的碱基修饰逆转录阻碍探针(如LNA,PNA等,并且探针的3’端被NH2C6封闭),使得逆转录阻碍探针能够优先锁定靶RNA,阻止逆转录酶通过逆转录阻碍探针锁定的靶RNA位点,从而达到在逆转录过程中去除靶RNA的目的。
表1 探针及引物序列
其中序列117-126为RT-qPCR的定量引物。序号为1-112的探针是根据人/小鼠/大鼠的rRNA设计的,可适用于人、小鼠或大鼠的RNA建库。
实施例1:不同修饰种类探针对靶RNA逆转录的抑制作用
参照图2,本实施例设计带有不同修饰的5.8S特异性探针(表1序号1-5),实现对5.8S rRNA逆转录过程的抑制。实施例中采用的Total RNA系按常规方法从培养的Human293FT cell(GM-070006H,购自吉满生物科技(上海)有限公司)细胞提取,以下同。具体实施方式如下:
表2
混匀后瞬离。85 °C 5 min,立即置于冰上5 min。
表3
混匀后瞬离。25 °C 10 min, 42 °C 15 min,70 °C 15 min,4 °C hold;
稀释1000倍后,用Hieff UNICON® qPCR SYBR Green Master Mix (11200ES03)对5.8S cDNA进行定量,定量引物见表1。定量结果见图3。
从定量结果可以看出,肽核酸(PNA)、锁核酸(LNA)、脱氧尿嘧啶(dU)、DNA小沟结合物(MGB)和2-氧甲基(2’-O-Me)修饰探针都能够有效抑制靶RNA的逆转录,其中LNA探针抑制逆转录的效果最好(见表4)。
表4:不同5.8s rRNA探针投入量对靶RNA逆转录的抑制效率
实施例2: 探针中LNA的分布位置对靶RNA逆转录的抑制效果
本实施例设计不同LNA修饰位置的5.8S特异性探针(表1序号6-8),实现对5.8SrRNA 逆转录过程的抑制。具体实施方式参照例1,定量结果见图4。
从定量结果可以看出,当探针中LNA的位置靠近探针的左端(即5’端),对靶RNA逆转录的阻碍效果要高于探针中LNA的位置靠近探针的右端(即3’端)。
表5:探针中LNA分布位置对靶RNA逆转录的抑制效率
实施例3: 不同长度LNA修饰探针对靶RNA逆转录的抑制效果
本实施例设计不同长度的带有LNA修饰的5.8S特异性探针(表1序号9-14),实现对5.8S rRNA 逆转录过程的抑制。具体实施方式参照例1,定量结果见图5。
从定量结果可以看出,当LNA探针长度在20-25 nt时,对靶RNA逆转录过程的抑制效果最好。
表6:不同长度LNA修饰探针对靶RNA逆转录的抑制效率
实施例4:不同浓度LNA探针对靶RNA逆转录的抑制效果
参照图2,本实施例利用不同浓度的带有LNA修饰的5.8S特异性探针(表1序号16),实现对5.8S rRNA 逆转录过程的抑制。具体实施方式参照例1,定量结果见图6。
从定量结果可以看出,本方法使用的锁核酸修饰探针能够有效抑制靶RNA的逆转录,当探针投入量达到4 μM时,5.8S rRNA的逆转录抑制效率能达到99.81%(见表4)。
表7:不同浓度LNA探针对靶RNA逆转录的抑制效率
实施例5:5.8S全覆盖逆转录阻碍探针对随机引物结合的竞争作用和一链cDNA合成的抑制作用
参照图7和图8,本实施例利用5.8S特异性LNA修饰探针(表1序号15-17混合物)高Tm值高结合能力的特点,通过梯度退火的方式保证探针比随机引物优先结合到靶RNA上,实现对5.8S rRNA逆转录高效特异性的抑制。具体实施方式如下:
表8
混匀后瞬离。85 °C 5 min,75 °C 2 min,70 °C 2 min,65 °C 2 min,60 °C 2min,55 °C 2 min,37 °C 2 min,25 °C 5 min,4 °C hold;
表9
混匀后瞬离。25 °C 10 min, 42 °C 15 min,70 °C 15 min,4 °C hold;
稀释1000倍后,用Hieff UNICON® qPCR SYBR Green Master Mix (11200) 对5.8S、18S、28S、Actin和Gapdh cDNA进行定量,定量引物见表1,定量结果见图9。
从定量结果可以看出,本方法中的梯度退火能够有效保证高Tm值的逆转录阻碍探针优先于随机引物结合到靶RNA上,进而抑制靶RNA的逆转录,且对其他非靶RNA的逆转录影响很小。当探针投入量达到4 μM时,5.8S rRNA的逆转录抑制效率能达到99.75%(见表10)。
表10:不同投入量5.8S逆转录阻碍探针竞争结合靶RNA和抑制逆转录的效率
实施例6:18S和28S全覆盖逆转录阻碍探针对随机引物结合的竞争作用和一链cDNA合成的抑制作用
按照实施例5的方法,本实施例利用18S(表1序号序列18-48混合物)或28S(表1序号序列49-112混合物)特异性修饰探针高Tm值高结合能力的特点,通过梯度退火的方式保证探针比随机引物优先结合到靶RNA上,实现对18S 或28S rRNA逆转录高效特异性的抑制。探针序列和引物见表1,定量结果见图10和图11。
从定量结果可以看出,本方法中设计的逆转录阻碍探针能够高效地阻碍18S和28SrRNA的逆转录。当探针混合物(Probe Mix)投入量达到4 μM时,18S rRNA的逆转录抑制效率能达到99.99%,28S rRNA的逆转录抑制效率能达到99.97%。由于18S rRNA和28S rRNA在总RNA占比太大,抑制其逆转录后,其他RNA的逆转录效率有轻微升高(见表11和表12)。
表11:不同投入量18S逆转录阻碍探针竞争结合靶RNA和抑制逆转录的效率
表12:不同投入量28S逆转录阻碍探针竞争结合靶RNA和抑制逆转录的效率
实施例7:三种rRNA探针混合物不同退火条件对随机引物结合的竞争作用和一链cDNA合成的抑制作用
本实施例验证了不同退火条件下探针对靶RNA的一链cDNA合成的抑制效果,具体实施方式如下:
表13
吹打混匀后瞬离。
条件1:85 °C 5 min,75 °C 2 min,70 °C 2 min,65 °C 2 min,60 °C 2 min,55°C 2 min,37 °C 2 min,25 °C 5 min,4 °C hold。
条件2:85 °C 5 min,75 °C 2 min,65 °C 2 min, 55 °C 2 min,37 °C 2 min,25°C 5 min,4 °C hold。
条件3: 75 °C 2 min,55 °C 2 min,25 °C 5 min,4 °C hold。
条件4: 75 °C 2 min,55 °C 2 min,4 °C hold。
条件5: 75 °C 1 min,55 °C 1 min,4 °C hold。
条件6: 75 °C 1 min,4 °C hold。
表14
混匀后瞬离。25 °C 10 min, 42 °C 15 min,70 °C 15 min,4 °C hold;
稀释1000倍后,用Hieff UNICON® qPCR SYBR Green Master Mix (11200) 对5.8S、18S、28S、Actin和Gapdh cDNA进行定量,定量引物见表1,RNA的抑制效率见表15。
表15:不同退火条件下逆转录阻碍探针竞争结合靶RNA和抑制逆转录的效率
由表15的定量结果可以看出,在1uL 4 μM NGS 5.8S/18S/28S rRNA probe mix使用量下不同退火条件都能够有效抑制靶RNA的逆转录,其中条件5(75 °C 1 min,55 °C 1min,4 °C hold)操作简单,并不降低抑制效率,三条靶RNA的抑制效率都能达到99.8%以上,是最合适的处理条件(如图12)。
实施例8:逆转录阻碍探针法与传统法在一链合成时的去除效果对比
参照图13,本实施例系统性比较polyA RNA富集法、RNase H切割去除法和逆转录阻碍探针法对靶RNA的去除效果。具体实施方式如下:
1)polyA富集法。使用Hieff NGS® MaxUp II Dual-mode mRNA Library PrepKit for Illumina (Yeasen, 12300)对mRNA进行富集。
取1 μg总RNA,用Nuclease free水将体积补至50 μL。吸取50 μL mRNA CaptureBeads加入至50 μL总RNA样品中,吹打混匀。65 °C 5 min;室温孵育5 min。将样品置于磁力架中,室温静置5 min,小心移除上清。将样品从磁力架上取出,用200 μL Beads WashBuffer重悬磁珠,反复吹打混匀。将样品置于磁力架中,室温静置5 min,小心移除上清。重复洗涤磁珠一次。加入50 μL Tris Buffer重悬磁珠,吹打混匀。80 °C 2 min;25 °C hold。将样品从PCR仪中取出,加入50 μL Beads Binding Buffer,反复吹打混匀。室温放置5min。将样品置于磁力架中,室温静置5 min,小心移除上清。用200 μL Beads Wash Buffer重悬磁珠,吹打混匀,将样品重新放回至磁力架中,室温静置5 min,吸掉全部上清。将样品从磁力架上取出,用18.5 μL Frag/Prime buffer重悬磁珠,吹打混匀。将样品置于PCR仪中, 94 °C 5 min,立即放在磁力架上。吸取17 μL 片段化mRNA,加入6 μL StrandSpecificity Reagent和2 μL 1st Strand Enzyme Mix混匀后瞬离。25 °C 10 min, 42 °C15 min,70 °C 15 min,4 °C hold。取0.5 μL产物稀释100倍后,用Hieff UNICON® qPCRSYBR Green Master Mix (11200) 对5.8S、18S、28S和Actin cDNA进行定量。
2)RNase H切割去除法。使用Hieff NGS® MaxUp rRNA Depletion Kit (Human/Mouse/Rat) (Yeasen, 12253)对rRNA进行去除。
表16
吹打混匀后瞬离。95 °C 2 min,95 °C-22 °C -0.1 °C/s,22 °C 5 min,4 °Chold。
表17
吹打混匀后瞬离,37 °C 30 min,4 °C hold。
表18
吹打混匀后瞬离,37 °C 30 min,4 °C hold。加入110 μL Hieff NGS® RNACleaner (Yeasen, 12602),充分吹打混匀,室温孵育5 min。将PCR管置于磁力架中分离磁珠和液体,待溶液澄清后(约3 min),小心移除上清。保持PCR管始终置于磁力架中,加入200 μL Nuclease free H2O新鲜配制的80%乙醇漂洗磁珠,室温孵育 30 sec 后,小心移除上清。重复漂洗一次。用 10 μL移液器吸干净残留液体。保持 PCR 管始终置于磁力架中,室温下开盖干燥磁珠(5~10 min)。用18.5 μL Frag/Prime buffer重悬磁珠,吹打混匀,室温静置5 min。将PCR管置于磁力架中分离磁珠和液体,待溶液澄清后(约3 min),吸取17 μLmRNA于新PCR管中。将样品置于PCR仪中, 94 °C 5 min,立即冰浴5 min。
表19
混匀后瞬离。25 °C 10 min, 42 °C 15 min,70 °C 15 min,4 °C hold。取0.5 μL产物稀释100倍后,用Hieff UNICON® qPCR SYBR Green Master Mix (11200) 对5.8S、18S、28S和Actin cDNA进行定量。
3)逆转录阻碍探针法。
表20
吹打混匀后瞬离。95 °C 5 min,75 °C 1 min,55 °C 1 min,4 °C hold。
表21
加入6 μL Strand Specificity Reagent和2 μL 1st Strand Enzyme Mix混匀后瞬离。25 °C 10 min, 42 °C 15 min,70 °C 15 min,4 °C hold。取0.5 μL产物稀释100倍后,用Hieff UNICON® qPCR SYBR Green Master Mix (11200) 对5.8S、18S、28S和ActincDNA进行定量。
三种方法的定量结果见图14。从定量结果可以看出,逆转录阻碍探针达到0.16 μM终浓度时,在NGS RNA建库过程中,rRNA的一链合成效率抑制率达到99.96%以上,已经显著超过RNase H切割去除法的去除效率,与polyA富集法的去除效率相当(99.94%),且对其他非靶RNA的一链合成效率有更大的促进作用(见表22)。
表22:三种不同靶RNA去除方法在NGS RNA建库中靶RNA一链合成抑制效率的比较
实施例9:逆转录阻碍探针法与传统法在RNA NGS建库中的性能对比
参照图13,本实施例利用基于illumina的二代测序平台,系统性比较polyA RNA富集法、RNase H切割去除法和逆转录阻碍探针法在RNA建库过程中对靶RNA的去除效果。实施例3中剩下的1st strand cDNA 产物,通过Hieff NGS® MaxUp II Dual-mode mRNALibrary Prep Kit for Illumina (Yeasen, 12300)进行建库,流程包括:
1)二链合成
表23
吹打混匀后瞬离。16 °C 30 min,72 °C 15 min,4 °C hold。
2)接头连接
表24
吹打混匀后瞬离。20 °C 15 min, 4 °C hold。
3)两轮磁珠回收
加入60 μL Hieff NGS® DNA Selection Beads (Yeasen, 12601),充分吹打混匀,室温孵育5 min。将PCR管置于磁力架中分离磁珠和液体,待溶液澄清后(约3 min),小心移除上清。保持PCR管始终置于磁力架中,加入 200 μL Nuclease free H2O新鲜配制的80%乙醇漂洗磁珠,室温孵育 30 sec 后,小心移除上清。重复漂洗一次。用 10 μL移液器吸干净残留液体。保持 PCR 管始终置于磁力架中,室温下开盖干燥磁珠(5 min)。加入53 μLddH2O,吹打至充分混匀,室温静置5 min。将PCR管短暂离心并置于磁力架中静置,待溶液澄清后(约5 min),小心移取50 μL上清至新PCR管中。
加入80 μL Hieff NGS® DNA Selection Beads (Yeasen, 12601),重复上述回收操作。加入22 μL ddH2O,吹打至充分混匀,室温静置5 min。将PCR管短暂离心并置于磁力架中静置,待溶液澄清后(约5 min),小心移取20 μL上清至新PCR管中。
3)文库扩增、回收、定量及测序
表25
吹打混匀后瞬离。按照以下反应程序进行文库扩增。
表26
加入45 μL Hieff NGS® DNA Selection Beads (Yeasen, 12601),重复上述回收操作。加入22 μL ddH2O,吹打至充分混匀,室温静置5 min。将PCR管短暂离心并置于磁力架中静置,待溶液澄清后(约5 min),小心移取20 μL上清至新PCR管中。用dsDNA HS AssayKit for Qubit (Yeasen, 12640)对文库进行定量,并利用Illumina的NovaSeq 6000平台进行文库测序。rRNA去除效果见图15。
表27 四种建库方式文库产量
表28: 四种建库方式rRNA残留率测序数据中rRNA残留率
由表27、表28和图15可知,逆转录阻碍探针法能够显著提高RNA建库的产量,且文库测序中核糖体RNA的残留显著低于polyA富集法和RNase H切割去除法,这说明逆转录阻碍探针法能够高效的去除建库过程中的靶RNA。此外,相关性分析(见图16)结果显示逆转录阻碍探针法与RNA直接建库具有更好的相关性,这说明逆转录阻碍探针法具有更小的建库偏好性,更能反映原始RNA的真实信息。同时,在同等测序深度下,逆转录探针法的基因检出数显著高于其他三种建库方式(见图17),这表明结合逆转录探针法的RNA测序技术获得的信息更加完整、全面。
实施例10:不同探针投入比例在RNA NGS建库过程中的去除靶RNA的效果
由于总RNA中三条rRNA的含量并不相同(约为1:6:10),所以本实施例验证了三条探针不同比例在RNA NGS建库过程中对rRNA的去除效果。具体实施方式如下:
表29
吹打混匀后瞬离。95 °C 5 min,75 °C 1 min,55 °C 1 min,4 °C hold。
表30
表31
加入6 μL Strand Specificity Reagent和2 μL 1st Strand Enzyme Mix混匀后瞬离。25 °C 10 min, 42 °C 15 min,70 °C 15 min,4 °C hold。后续RNA NGS建库参照实施例9进行。
表32 不同探针投入比例文库产量
表33: 四种建库方式rRNA残留率测序数据中rRNA残留率
由表32、表33可知,探针投入量越少,建库产量越高。当使用1 ug总RNA进行RNA建库时,1 uLNGS rRNA逆转录阻碍探针混合物中每条5.8S NGS rRNA探针掺入浓度应为0.1-8uM,每条18S NGS rRNA探针掺入浓度应为1-8 uM,每条28S NGS rRNA探针掺入浓度应为2-8uM,在这个探针投入浓度范围可以使得RNA NGS测序数据中每条rRNA的残留测序数据均不超过总数据的1%,符合常规RNA测序中对rRNA的去除效率要求。出于节省的原则,我们下游实施例中使用的NGS 5.8S/18S/28S rRNA probe mix每条5.8S NGS rRNA探针掺入浓度为0.1 uM,每条18S NGS rRNA探针掺入浓度为1 uM,每条28S NGS rRNA探针掺入浓度为2 uM。
实施例11:逆转录阻碍探针对不同RNA投入量样本中靶RNA的去除效果
本实施例验证了逆转录阻碍探针对不同RNA投入量样本NGS建库过程中对rRNA的去除效果。具体实施方式如下:
表34
吹打混匀后瞬离。95 °C 5 min,75 °C 1 min,55 °C 1 min,4 °C hold。
表35
表36
加入6 μL Strand Specificity Reagent和2 μL 1st Strand Enzyme Mix混匀后瞬离。25 °C 10 min, 42 °C 15 min,70 °C 15 min,4 °C hold。后续RNA NGS建库参照实施例9进行,PCR扩增循环数见表35。
表37 不同RNA投入量文库产量
表38: 四种建库方式rRNA残留率测序数据中rRNA残留率
由表37、表38可知,逆转录阻碍探针法可以有效去除不同RNA投入量的NGS建库过程中的靶RNA。当RNA投入量为100-1000 ng时,NGS 5.8S/18S/28S rRNA probe mix的最佳使用量为1 μl;当RNA投入量为10-100 ng时,NGS 5.8S/18S/28S rRNA probe mix的的最佳使用量为0.1 μl;当RNA投入量为10 pg(单细胞)-10 ng时,NGS 5.8S/18S/28S rRNA probemix的的最佳使用量为0.01 μl。总rRNA的残留率低于1%。从基因检出数上看,逆转录阻碍探针法比其他传统的rRNA去除法具有更大的优势。
综上,我们发明了一种用于RNA建库中快速去除靶RNA的逆转录阻碍探针及其方法流程。本发明具有操作简单(一步操作)、耗时极短(2 min)、对非靶RNA保留完整、特异性强、效率高、成本低和基因检出数高等优点,非常适合用于工业自动化建库和疾病快速诊断领域。任何在逆转录过程中利用逆转录阻碍探针混合物去除靶RNA的方法都应属于本专利的保护范围。
序列表
<110> 翌圣生物科技(上海)有限公司
<120> 用于RNA建库中快速去除靶RNA的逆转录阻碍探针及其应用
<141> 2020-12-30
<160> 126
<170> SIPOSequenceListing 1.0
<210> 1
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 1
tgatcaatgt gtcct 15
<210> 2
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 2
tgatcaatgt gtcct 15
<210> 3
<211> 15
<212> DNA/RNA
<213> Artificial Sequence
<400> 3
ugaucaaugu guccu 15
<210> 4
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 4
tgatcaatgt gtcct 15
<210> 5
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 5
tgatcaatgt gtcct 15
<210> 6
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 6
tgatcaatgt gtcct 15
<210> 7
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 7
tgatcaatgt gtcct 15
<210> 8
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 8
tgatcaatgt gtcct 15
<210> 9
<211> 15
<212> DNA
<213> Artificial Sequence
<400> 9
tgatcaatgt gtcct 15
<210> 10
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 10
tgatcaatgt gtcctgcaat 20
<210> 11
<211> 25
<212> DNA
<213> Artificial Sequence
<400> 11
tgatcaatgt gtcctgcaat tcaca 25
<210> 12
<211> 30
<212> DNA
<213> Artificial Sequence
<400> 12
tgatcaatgt gtcctgcaat tcacattaat 30
<210> 13
<211> 35
<212> DNA
<213> Artificial Sequence
<400> 13
tgatcaatgt gtcctgcaat tcacattaat tctcg 35
<210> 14
<211> 40
<212> DNA
<213> Artificial Sequence
<400> 14
tgatcaatgt gtcctgcaat tcacattaat tctcgcagct 40
<210> 15
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 15
tcatcgacgc acgagccgag 20
<210> 16
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 16
cgatgatcaa tgtgtcctgc aa 22
<210> 17
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 17
caggcgtagc cccgggagga ac 22
<210> 18
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 18
tgcatggctt aatctttgag aca 23
<210> 19
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 19
ataactgatt taatgagcca tt 22
<210> 20
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 20
agctctagaa ttaccacagt t 21
<210> 21
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 21
gatctgataa atgcacgcat c 21
<210> 22
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 22
caaagccgcc ggcgcccgcc 20
<210> 23
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 23
tcgaatgggt cgtcgccgcc a 21
<210> 24
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 24
gtcacccgtg gtcaccatgg t 21
<210> 25
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 25
ccttggatgt ggtagccgtt t 21
<210> 26
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 26
ttatttttcg tcactacctc c 21
<210> 27
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 27
gttaaaggat ttaaagtgga ctc 23
<210> 28
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 28
ttaatatacg ctattggagc 20
<210> 29
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 29
gaccgcccgc ccgctcccaa 20
<210> 30
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 30
cagctaagag catcgagggg 20
<210> 31
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 31
ttgaacactc taattttttc a 21
<210> 32
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 32
aaatagaacc gcggtcctat t 21
<210> 33
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 33
gcggcgcaat acgaatgccc cc 22
<210> 34
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 34
ggcaaatgct ttcgctctgg t 21
<210> 35
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 35
gaactacgac ggtatctgat c 21
<210> 36
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 36
gcccggcggg tcatgggaat a 21
<210> 37
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 37
tttaagtttc agctttgcaa 20
<210> 38
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 38
gttgagtcaa attaagccgc 20
<210> 39
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 39
agaaagagct atcaatctgt ca 22
<210> 40
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 40
cggaattaac cagacaaatc 20
<210> 41
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 41
acgccgaccg ctcgggggtc 20
<210> 42
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 42
acagacctgt tattgctcaa t 21
<210> 43
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 43
acacgctgag ccagtcagtg t 21
<210> 44
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 44
tccccgatcc ccatcacgaa 20
<210> 45
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 45
cgcaagctta tgacccgcac tta 23
<210> 46
<211> 24
<212> DNA
<213> Artificial Sequence
<400> 46
catccaatcg gtagtagcga cggg 24
<210> 47
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 47
gtcttctcag cgctccgcca gg 22
<210> 48
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 48
cctacggaaa ccttgttacg ac 22
<210> 49
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 49
ccgttactga gggaatcctg gtt 23
<210> 50
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 50
catgtcccgc gccccgccgc gg 22
<210> 51
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 51
agaaggactt gggcccccca 20
<210> 52
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 52
cgggcccggc gcgccggggg cc 22
<210> 53
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 53
ggagtttacc acccgctttg g 21
<210> 54
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 54
tcttttcaac tttcccttac ggt 23
<210> 55
<211> 24
<212> DNA
<213> Artificial Sequence
<400> 55
accccacccg tttacctctt aacg 24
<210> 56
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 56
gggccgccga cacggccgga 20
<210> 57
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 57
gggggaaggg agggcgggtg 20
<210> 58
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 58
ggccccgccc gcccaccccc gca 23
<210> 59
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 59
atgcgcccgg cggcggccgg tc 22
<210> 60
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 60
ggccttccca gccgtcccgg ag 22
<210> 61
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 61
ccgagggagg acgcggggcc 20
<210> 62
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 62
ccgccgccgc cgccccgacc cgc 23
<210> 63
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 63
ggggggctgt aacactcggg 20
<210> 64
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 64
ggggagagcg cggcgacggg 20
<210> 65
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 65
gggggagacc cccctcgcgg g 21
<210> 66
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 66
cgccgtggga ggggtggccc gg 22
<210> 67
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 67
ccgacccgcg cgcggcaccc 20
<210> 68
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 68
ccccgggccc gacggcgcga c 21
<210> 69
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 69
cgaccccgtg cgctcgctcc gc 22
<210> 70
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 70
tgttagactc cttggtccgt g 21
<210> 71
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 71
ggcgagcgcg ccggccttca c 21
<210> 72
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 72
gccggtggtg cgccctcggc g 21
<210> 73
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 73
ttcaccatct ttcgggtcct aa 22
<210> 74
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 74
ccagagtttc ctctggcttc gc 22
<210> 75
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 75
gccagctatc ctgagggaaa 20
<210> 76
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 76
tgagatcgtt tcggccccaa g 21
<210> 77
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 77
aagtggccca ctaggcactc gc 22
<210> 78
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 78
tcaaccaaca ccttttctgg gg 22
<210> 79
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 79
agggctagtt gattcggcag gt 22
<210> 80
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 80
ccacacgcgc gcgcgcgcgc gc 22
<210> 81
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 81
gcgggaggag gggaggaggc gt 22
<210> 82
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 82
gcgccctagg cttcaaggct c 21
<210> 83
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 83
cttcggcctt caaagttctc gtt 23
<210> 84
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 84
gggcaacgga ggccatcgcc c 21
<210> 85
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 85
accgcactgg acgcctcgcg g 21
<210> 86
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 86
ctctctcggg gcgaacccat tcc 23
<210> 87
<211> 24
<212> DNA
<213> Artificial Sequence
<400> 87
ctctcccccg gattttcaag ggcc 24
<210> 88
<211> 25
<212> DNA
<213> Artificial Sequence
<400> 88
cttgccgact tcccttacct acatt 25
<210> 89
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 89
cgcccagccc cgcttcgcgc c 21
<210> 90
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 90
gggagggagc gagcggcgcg c 21
<210> 91
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 91
ccgcccccac gcggcgctcc 20
<210> 92
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 92
cgggtgcccg ggcccccctc 20
<210> 93
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 93
cggccgcgac gcccgccgca gc 22
<210> 94
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 94
gccgcccgac cgctccccgc c 21
<210> 95
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 95
aacggggggc ggacggggcc 20
<210> 96
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 96
cgcggaaccg cggccccggg 20
<210> 97
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 97
ttaattaaac agtcggattc c 21
<210> 98
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 98
cgtttacccg cgcttcattg a 21
<210> 99
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 99
cgttcatcca ttcatgcgcg t 21
<210> 100
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 100
actagagtca agctcaacag ggt 23
<210> 101
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 101
cccctcgcgg ggacaccggg 20
<210> 102
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 102
gcgagagccc ctcggggctc gc 22
<210> 103
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 103
aactccccac ctggcactgt c 21
<210> 104
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 104
aagcgagctt ttgcccttct 20
<210> 105
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 105
cctcctgctt aaaacccaaa a 21
<210> 106
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 106
caatgatagg aagagccgac a 21
<210> 107
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 107
cctgtctcac gacggtctaa 20
<210> 108
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 108
cctcagccaa gcacatacac ca 22
<210> 109
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 109
tgccgtatcg ttccgcctgg gc 22
<210> 110
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 110
tcccgcgcgc gcggggcgcg t 21
<210> 111
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 111
gccgcgcccc gtttcccagg a 21
<210> 112
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 112
acgtacgaaa ccccgaccca g 21
<210> 113
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 113
aagcgacgct cagacaggcg tag 23
<210> 114
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 114
tacctggttg atcctgccag 20
<210> 115
<211> 24
<212> DNA
<213> Artificial Sequence
<400> 115
gacaaaccct tgtgtcgagg gctg 24
<210> 116
<211> 6
<212> DNA/RNA
<213> Artificial Sequence
<400> 116
nnnnnn 6
<210> 117
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 117
ctcttagcgg tggatcactc g 21
<210> 118
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 118
gcaagtgcgt tcgaagtgtc 20
<210> 119
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 119
gttttcggaa ctgaggccat g 21
<210> 120
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 120
tcggaactac gacggtatct g 21
<210> 121
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 121
cctagtgggc cacttttggt 20
<210> 122
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 122
ggctagttga ttcggcaggt 20
<210> 123
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 123
ccttccagca gatgtggatc a 21
<210> 124
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 124
ttctgcgcaa gttaggtttt gtc 23
<210> 125
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 125
ggagcgagat ccctccaaaa t 21
<210> 126
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 126
ggctgttgtc atacttctca tgg 23
- 用于RNA建库中快速去除靶RNA的逆转录阻碍探针及其应用
- 用于RNA建库中快速去除靶RNA的逆转录阻碍探针及其应用
