一种脑转移相关基因的CRISPR/Cas9文库高通量筛选方法
文献发布时间:2023-06-19 11:19:16
技术领域
本发明属于癌症诊断技术领域,涉及一种脑转移相关基因的CRISPR/Cas9文库高通量筛选方法。
背景技术
CRISPR/Cas9系统由Cas9蛋白和sgRNA(single guide RNA)这两个组分组成。sgRNA可以识别带有PAM(protospacer adjacent motif)序列的互补序列,Cas9蛋白可以对靶DNA序列进行切割,因此Cas9/sgRNA系统可以通过sgRNA引导Cas9蛋白识别并剪切带有sgRNA靶点的特异性双链DNA,导致基因序列的插入或缺失,最终实现基因的定向编辑。CRISPR/Cas9文库是针对每个基因设计3-10条的sgRNA,利用芯片一次性合成针对数百甚至数万基因的sgRNA库,经过慢病毒包装后以低感染复数感染稳定表达Cas9蛋白的宿主细胞,在适当的筛选条件下测试筛选前后sgRNA的丰度变化,进而找出关键基因。CRISPR/Cas9文库筛选技术可以针对多个基因实现高通量的筛选,高效、快捷地筛选出研究者所需的关键基因。
目前,CRISPR/Cas9文库筛选技术多应用于:(1)体外加药筛选。将sgRNA文库包装成慢病毒后感染稳定表达Cas9的宿主细胞,而后将细胞培养于含有不同药物或不同药物浓度的培养基内,加药筛选培养一定时间后,测试筛选前后sgRNA的丰度变化。体外加药筛选可以筛选耐药/增敏基因,验证药物与基因所在信号通路之间的相互影响;(2)小鼠皮下瘤模型筛选肿瘤发生发展的相关基因。将sgRNA文库包装成慢病毒后感染稳定表达Cas9的宿主细胞,而后将细胞接种于免疫缺陷小鼠的皮下,待接种的细胞形成皮下瘤后,将皮下瘤取下并提取DNA,测试接种前后sgRNA的丰度变化。
肺癌、乳腺癌、黑色素瘤发生脑转移(Brain Metastasis,BM)的患者,由于缺少有效的治疗手段,预后极差。面对肿瘤的脑转移,目前手术、立体定向放疗、全脑放疗等局部治疗为脑转移的一线治疗方法,但由于其对影像学上不可见的潜在病灶的控制率差,局部治疗后颅内进展的发生率高。而全身系统治疗,如卡培他滨、替莫唑胺等,在脑转移中的治疗地位并不明确。临床医师面对颅内进展往往“束手无策”,颅内进展所致的死亡率高达37%。这提示脑转移问题——阐明可能机制、找到潜在干预措施是目前乳腺癌研究的重中之重。
肿瘤细胞的转移需经历从原发灶到转移灶的侵袭-转移级联过程。首先肿瘤细胞突破基膜侵入周围基质,而后进入脉管系统到达转移部位,并最终增殖形成临床可检测到的转移克隆灶。其中克隆灶形成是一个极端低效的过程,绝大部份细胞不能很好地适应转移灶的组织微环境。在其脑转移发生过程中细胞是如何适应颅内微环境的,此机制至今未明,研究也极少。显而易见,脑转移灶与乳腺原发灶的代谢微环境截然不同,转移瘤细胞要在新的微环境存活,首先需要代偿机制以适应新的微环境。
但目前并未有研究者提出如何将CRISPR/Cas9文库筛选技术应用于肿瘤的脑转移的研究中。
发明内容
为了解决上述技术问题,本发明提供了一种通过CRISPR/Cas9文库筛选技术获得脑转移相关候选基因的方法。该方法建立了脑转移相关基因高通量筛选技术,拓宽了CRISPR/Cas9文库的使用范围,能够准确、高效地挖掘调控脑转移的关键基因,为脑转移预防和治疗提供新靶点、新途径。
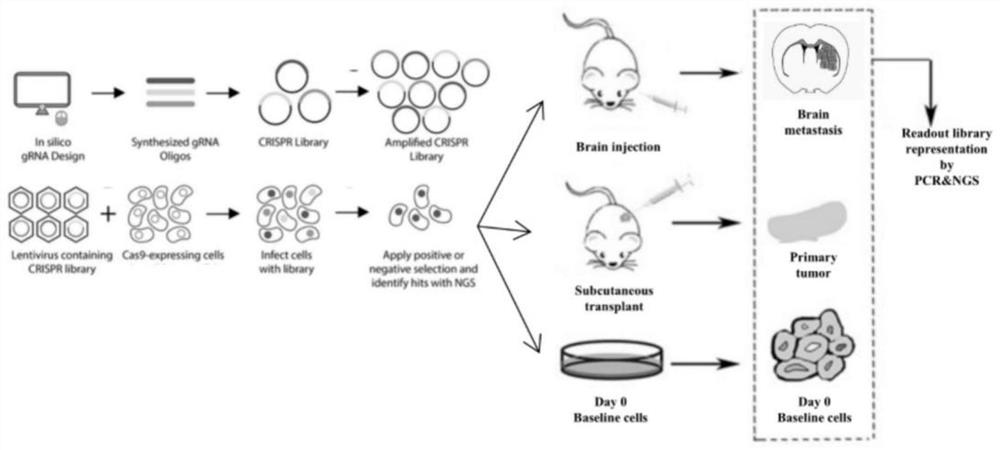
本发明提供了一种脑转移相关候选基因的CRISPR/Cas9文库高通量筛选方法,包括以下步骤,
步骤S1、构建脑转移相关基因的sgRNA文库:
步骤S2、将sgRNA文库进行慢病毒包装,感染稳定表达CAS9蛋白的宿主细胞,并筛选以获得稳定表达sgRNA文库的细胞;
步骤S3、将慢病毒感染后的细胞接种于免疫缺陷的小鼠颅内及脂肪垫;
步骤S4、将颅内成瘤及皮下成瘤的肿瘤组织取出,提取基因组DNA后,对sgRNA的靶向区进行二代测序建库,通过判断前后sgRNA的丰度变化,筛选出脑转移相关候选基因。
在某些实施例中,所述脑转移相关基因为通过检测确定的脑转移灶与肿瘤原发灶中差异表达的基因,如表1中所示。
在某些实施例中,所述步骤S1包括以下步骤,
(a)提供核酸构建物Z1,所述的核酸构建物Z1具有以下结构:
L-N20-R
其中,
L为位于脑转移相关基因的sgRNA的编码序列上游的左同源臂序列,
N20为脑转移相关基因的sgRNA编码序列,
R为位于脑转移相关基因的sgRNA的编码序列下游的右同源臂序列;
(b)以所述的核酸构建物Z1为模板,进行PCR扩增,从而获得扩增产物;
(c)将所述扩增产物与经线性化的表达载体混合,并用Gibson assembly法进行连接,从而形成连接产物;
(d)将所述连接产物转化大肠杆菌,从而获得转化子,并从所述转化子中抽提质粒,从而获得所述的脑转移相关基因的sgRNA文库。
在某些实施例中,所述左同源臂L的核苷酸序列为SEQ ID NO:1所示,右同源臂R的核苷酸序列为SEQ ID NO:2所示。
与现有技术相比本发明具有的技术效果:
1)本发明针对脑转移相关基因高通量筛选这一空白领域,原创性地建立了脑转移相关基因高通量筛选技术,第一次将感染了脑转移相关基因sgRNA文库的细胞通过脑立体定位仪进行颅内成瘤,以筛选促使肿瘤细胞适应颅内微环境的关键基因,这样拓宽了CRISPR/Cas9文库的使用范围,能够准确、高效地挖掘调控脑转移的关键基因,为脑转移预防和治疗提供新靶点、新途径。
2)通过本发明提供的方法,对不同癌种(如非小细胞肺癌、黑色素瘤、乳腺癌)的脑转移灶及原发灶标本进行检测,将得到差异表达基因按本发明所示的方法构建脑转移相关基因文库,并进行高通量筛选,寻找不同癌种中调控脑转移的关键基因。
3)对同一癌种的不同亚型(如乳腺癌的Luminal型、HER2阳性型、三阴型)的脑转移灶及原发灶标本进行检测,将得到的差异表达基因按本发明所示的方法构建脑转移相关基因文库,并进行高通量筛选,寻找不同乳腺癌亚型中调控脑转移的关键基因。
附图说明
为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式描述中所需要使用的附图作简单地介绍。
图1为本发明高通量筛选方法的流程示意图。
图2为实施例2中所述sgRNA涉及原理示例图。
图3为实施例9中小鼠颅内成瘤IVIS成像图。
图4为小鼠颅内获得的文库中靶向基因在压力筛选后的排序。
图5为小鼠皮下获得的文库中靶向基因在压力筛选后的排序。
具体实施方式
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。除非特别说明,本发明采用的试剂、方法和设备如无特别说明,均为常规方法,所使用的试验材料如无特别说明,均可从商业公司获取。
实施例1
收集5位三阴性乳腺癌单纯脑转移患者的乳腺癌原发灶与脑转移灶的配对样本,共5对10个样本。使用RNA-Quick Purification Kit(ES Science#RN001)试剂盒提取样本RNA,并进行RNA-Seq检测。对RNA-seq的检测结果进行分析,以Fold change值同时大于1.5作为筛选条件,筛选差异表达基因,共得到597个脑转移灶高表达基因(如表1所示)。
表1 三阴性乳腺癌脑转移相关基因
实施例2
根据sgRNA在线设计软件(http://crispr.mit.edu/),对实施例1中的每个基因设计6个sgRNA。每个sgRNA为20bp,在sgRNA的左右两侧加上27bp的同源臂(左侧同源臂的序列为ATATCTTGTGGAAAGGACGAAACACCG(SEQ ID NO:1),右侧同源臂的序列为GTTTTAGAGCTAGAAATAGCAAGTTAA(SEQ ID NO:2)),示例如图2所示。在高通量芯片上进行原位合成针对多个基因的寡核苷酸探针池。
实施例3
1、扩增寡核苷酸探针池
向寡核苷酸探针池的粉末中加入TE Buffer重悬,并使用如下的反应体系进行聚合酶链反应(polymerase chain reaction,PCR):
PCR扩增程序为:
PCR扩增体系为:
引物为:
A.正向引物:
GTAACTTGAAAGTATTTCGATTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACC(SEQ IDNO:3)
B.反向引物:
ACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC(SEQ IDNO:4)
2、酶切lentiGuide-puro载体
酶切体系:
酶切体系温度设置:
Step1:55℃90分钟
Step2:4℃°
将酶切所得产物用2%琼脂糖凝胶进行DNA电泳,切下10000bp的条带用Fastpuregel DNA Extraction Mini Kit(诺唯赞#DC301)进行胶回收。
3、sgRNA文库质粒的组装
将步骤1及步骤2所得的片段利用Gibson assembly反应进行片段组装,得到质粒文库。
Gibson assembly反应体系:
Gibson assembly反应体系温度设置:
Step1:50℃60分钟
Step2:4℃。
4、扩增sgRNA文库质粒
取50ul E.coli DH5αElectro-Cells(TAKARA#9027)于冰上融化,在融化的细胞中加入1~2μl步骤3所得的sgRNA文库。而后将混合液注入到冰中预冷的0.1cm电转杯内(Cuvette)。电压冲击后(BIO-RAD MicroPulser,1.5kV),迅速置于冰中冷却,加入1ml冰中预冷的SOC培养基(TAKARA#9027),37℃振荡培养1小时(160~250rpm)后取适量涂布氨苄抗性的LB平板上,并于37℃过夜培养。确保获得的单克隆菌落的数量>300*sgRNA数,以确保每个sgRNA都得到有效扩增。最后使用无菌的细胞推刮器将所有的单克隆菌落刮下,并使用无内毒素质粒大抽提试剂盒(天根,#DP117)提取质粒DNA,得到扩增的sgRNA文库。
实施例4 Cas9蛋白稳转株的构建
将293T细胞提前种板,当细胞达到70%-80%汇合度时进行慢病毒包装:在600ul的生理盐水中依次加入6ug lentiCas9-Blast(Addgene#52962)质粒,1.8ug包装质粒AGP090,4.5ug包装质粒AGP091,36.9ulPEI溶液(1mg/mL),涡旋震荡8秒使溶液混合均匀,室温静置10min后缓慢均匀地滴入到293T细胞培养基中,而后放于37°培箱中孵育过夜。12小时后更换培养基,48小时后用10ml注射剂收集培养基上清,用0.45μm滤器进行过滤。将宿主细胞细胞(如MDA-MB-231)提前种在六孔板,当细胞汇合度达到50%时,每孔加入500ulCas9病毒液,2ul polybrene(10mg/ml),并用培养基补足至2ml,置于37°含5%CO
实施例5 Luciferase稳转株的构建
将293T细胞提前种板,当细胞达到70%-80%汇合度时进行慢病毒包装:在600ul的生理盐水中依次加入6ug pWPXL-GFP-Luc质粒,1.8ug包装质粒AGP090,4.5ug包装质粒AGP091,36.9ul PEI溶液(1mg/mL),涡旋震荡8秒使溶液混合均匀,而后室温静置10min,并缓慢均匀地滴入到293T细胞培养基中,而后放于37℃培箱中孵育过夜。12小时后更换培养基,48小时后用10ml注射剂收集培养基上清,用0.45μm滤器进行过滤。将实施例4所得的细胞提前种在六孔板,当细胞汇合度达到50%时,每孔加入500ul Luciferase病毒液,2ulpolybrene(10mg/ml),并用培养基补足至2ml,置于37℃含5%CO
实施例6 sgRNA文库的慢病毒包装
将293T细胞提前种板,当细胞达到70%-80%汇合度时进行慢病毒包装:在600ul的生理盐水中依次加入6ug实施例3所得的sgRNA文库质粒,1.8ug包装质粒AGP090,4.5ug包装质粒AGP091,36.9ul PEI溶液(1mg/mL),涡旋震荡8秒使溶液混合均匀,而后室温静置10min,并缓慢均匀地滴入到293T细胞培养基中,而后放于37℃培箱中孵育过夜。12小时后更换培养基,48小时后用10ml注射剂收集培养基上清,并用0.45μm滤器进行过滤,按1ml进行病毒液的分装,并保存于-80°。
实施例7 sgRNA文库病毒滴度检测
将实施例5所得细胞按照每孔10
MOI=加嘌呤霉素筛选孔的细胞数/未加嘌呤霉素筛选的对照孔的细胞数。
实施例8 sgRNA文库稳转株的构建
将实施例5所得细胞按照每孔10
实施例9颅内微环境阴性筛选
将实施例8所得的细胞胰酶消化后用PBS重悬,而后按下述所示的技术方法接种于免疫缺陷的小鼠(BALB/C裸鼠,雌性,6周)的颅内及脂肪垫。
--颅内注射:使用眼科剪剪开小鼠头皮,暴露前囟。并尽量刮开头颅表面的筋膜。在脑立体定位仪的辅助下,在前囟右侧2.5mm,前1mm处用halmiton微量注射器(#7653-01)进行颅内注射,注射深度为3mm,每只小鼠注射3ul细胞悬液(10万细胞/ul),在5min内缓慢注射完。最后用3M组织胶封闭皮肤伤口。
--皮下注射:将500ul细胞悬液(10*106细胞/mL)与500ul基质胶(BD,#356234)1:1混匀后,用1ml注射针进行注射,注射部位为脂肪垫。针头在皮下穿行1.5cm,注射后轻轻按压拔针点有利于防止漏液。
小鼠颅内成瘤IVIS成像如图3所示,稳定表达sgRNA文库的稳转株在小鼠左侧大脑半球成瘤。
实施例10高通量测序与生信分析
1、基因组DNA的提取
使用QIAamp DNA Mini Kit试剂盒(Qiagen,#51304)对肿瘤组织进行DNA提取。
2、二代测序建库
通过巣式PCR进行二代测序建库。巢式PCR分为两轮。
第一轮PCR扩增程序为:
第一轮PCR扩增体系为:
Next High-Fidelity 2X PCR Master Mix(NEB,#M0541S) 25μl
10μM Forward Primer 2.5μl
10μM Reverse Primer 2.5μl
基因组DNA 10ng,
加ddH2O至50μl
第一轮PCR引物为:
A.正向引物:
AATGGACTATCATATGCTTACCGTAACTTGAAAGTATTTCG(SEQ ID NO:5)
B.反向引物:
CTTTAGTTTGTATGTCTGTTGCTATTATGTCTACTATTCTTTCC(SEQ ID NO:6)
对第一轮PCR的产物使用胶回收试剂盒进行纯化,并进行核酸定量。而后对第一轮的产物进行第二轮的PCR扩增,以使得第一轮的片段两端加上二代测序上机时所需的Illumina接头和区分样品所需的barcode片段。
第二轮PCR扩增程序为:
PCR扩增体系为:
Next High-Fidelity 2X PCR Master Mix(NEB,#M0541S) 25μl
10μM Forward Primer 2.5μl
10μM Reverse Primer 2.5μl
第一轮PCR纯化产物1ng,
加ddH2O至50μl
引物为:
A.正向引物:
AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCTTCTTGTGGAAAGGACGAAACACCG(SEQ ID NO:7)
B.反向引物:
CAAGCAGAAGACGGCATACGAGAT(8bp的barcode片段)GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTTCTACTATTCTTTCCCCTGCACTGT(SEQ ID NO:8)
将第二轮PCR产物使用AMPure XP磁珠进行纯化,而后送测序公司对样品进行二代测序,测序平台为Illumina Hiseq X Ten。
3、二代测序数据分析
将sgRNA的序列导入Geneious9(http://www.geneious.com/)中,而后将测序所得的测序结果(.fastq文件)导入Geneious9软件中,确定每条sgRNA在筛选前及筛选后的counts数变化,counts数变化明显的基因排名靠前。
分析结果如图4、5所示,经过颅内微环境筛选后,SLC3A2基因的counts数变化明显,排名靠前,说明敲除SLC3A2基因的细胞不能很好地适应颅内微环境,SLC3A2基因对于肿瘤细胞适应颅内微环境具有重要作用。而经过皮下微环境筛选后,SLC3A2基因的counts数变化不明显,排名靠后,说明SLC3A2基因对于肿瘤细胞适应皮下微环境的作用不显著。进一步说明SLC3A2基因是肿瘤细胞适应颅内微环境的特异性的基因。
以上显示和描述了本发明的基本原理、主要特征和本发明的优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内。
SEQUENCE LISTING
<110> 复旦大学附属肿瘤医院
<120> 一种脑转移相关基因的CRISPR/Cas9文库高通量筛选方法
<130> 2021
<160> 8
<170> PatentIn version 3.3
<210> 1
<211> 27
<212> DNA
<213> 人工序列(artificial sequence)
<400> 1
atatcttgtg gaaaggacga aacaccg 27
<210> 2
<211> 27
<212> DNA
<213> 人工序列(artificial sequence)
<400> 2
gttttagagc tagaaatagc aagttaa 27
<210> 3
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 3
gtaacttgaa agtatttcga tttcttggct ttatatatct tgtggaaagg acgaaacacc 60
<210> 4
<211> 60
<212> DNA
<213> 人工序列(artificial sequence)
<400> 4
actttttcaa gttgataacg gactagcctt attttaactt gctatttcta gctctaaaac 60
<210> 5
<211> 41
<212> DNA
<213> 人工序列(artificial sequence)
<400> 5
aatggactat catatgctta ccgtaacttg aaagtatttc g 41
<210> 6
<211> 44
<212> DNA
<213> 人工序列(artificial sequence)
<400> 6
ctttagtttg tatgtctgtt gctattatgt ctactattct ttcc 44
<210> 7
<211> 82
<212> DNA
<213> 人工序列(artificial sequence)
<400> 7
aatgatacgg cgaccaccga gatctacact ctttccctac acgacgctct tccgatcttc 60
ttgtggaaag gacgaaacac cg 82
<210> 8
<211> 91
<212> DNA
<213> 人工序列(artificial sequence)
<220>
<221> misc_feature
<223> n为 A/T/C/G
<220>
<221> misc_feature
<222> (25)..(32)
<223> n is a, c, g, or t
<400> 8
caagcagaag acggcatacg agatnnnnnn nngtgactgg agttcagacg tgtgctcttc 60
cgatcttcta ctattctttc ccctgcactg t 91
- 一种脑转移相关基因的CRISPR/Cas9文库高通量筛选方法
- 一种设计和构建猪全基因组CRISPR/Cas9敲除文库的方法
