一种基于深度学习网络的细菌显微图像分割方法
文献发布时间:2023-06-19 11:22:42
技术领域
本发明涉及细菌显微镜图像分割领域,特别是指一种基于深度学习网络的细菌显微图像分割方法。
背景技术
在对图像的研究和处理中,往往图像中包含的信息不是我们都感兴趣的,我们会根据需求,大脑会自动来判断哪些是我们需要的图像信息,并且往往这种特定的包含我们想要信息的图像部分对应于图像中具有特殊性质(边缘,形状,颜色等),在生物图像处理中我们常称为前景,相应的其他图像部分为背景。在生物领域中,培育的细菌在显微镜下成像,这些成像的图片往往受到显微镜对焦面改变、培养基中的杂志以及环境亮度等因素影响,这些包含噪音的图像往往使得研究人员很难将细菌和背景很清楚的分离开,也就影响后续的一些判断。
目前,生物图像处理领域中的图像分割方法主要分为:1.利用数字图像处理、拓扑学、统计学、数学等方面来进行图像分割的传统方法。2.近几年随着算力的增加以及深度学习的井喷式发展,基于深度学习的方法对于图像分割领域越来越占有不可取代的地位。
传统的方法主要包括:
1.基于阈值的分割方法:它的主要思想是根据图像的灰度特征来计算单个或多个灰度阈值,然后遍历图像中所有像素,将其灰度值和设置的阈值经行比较,大于阈值的像素设置为前景,小于阈值的像素设置为背景。它的优点是计算简单,效率高,缺点也很明显:根本没有考虑空间的特征,对图像中的噪声极其敏感,鲁棒性差。
2.基于边缘检测的分割算法:它是通过聚焦检测不同区域的边缘像素来解决分割问题的。比较有代表性的有梯度算法、Roberts算法、Sobel算法、Laplacian算法等,它的优点是速度快和边缘检测准确,缺点是他只能检测边缘像素,不能保证边缘的连续性以及完整性。
3.基于小波变换的图像分割方法:它是数字图像处理技术中比较重要的部分:首先由二进小波变换将图像的直方图分解为不同层次的小波系数,然后依据给定的分割准则和小波系数选择阈值门限,最后利用阈值标出图像分割的区域。他的优点是空域和频域的局域变换,能进行多尺度分析,并且由于是对频域操作,所以对噪声不敏感。缺点是很难选取合适的滤波器。
细菌图像由于其形状各异,并且多以菌落的形式生长,数量繁多且重叠严重,所以更加大了对其分割的难度。对于细菌分割领域,有公开利用超像素来进行图像分割的方法(例如CN103914841A),这种方法还是基于传统的图像分割方法(如计算超像素、以及计算每个超像素区域的颜色、形状、尺寸特征,利用先验知识对每个超像素区域进行初步滤波,并进行分割以确定候选细菌区域)来分割的,过程较为繁琐且效果不佳。
深度学习方法:完全区别于传统图像分割方法,它基于算力的支持,对大量带有标签的数据集进行训练,让机器自主学习到图像的内在规律及特征。主要包括:
1.基于CNN的图像分割:主要代表为2017年何凯明等人[He Kaiming,GkioxariGeorgia,DollarPiotr,Girshick Ross.Mask R-CNN.[J].IEEE transactions onpattern analysis and machine intelligence,2020,42(2).]提出的 MaskR-CNN,主要完成了图像的像素级分割。
2.基于RNN的图像分割:主要代表为2015年Visin等人[Visin F,Kastner K,Courville A,et al.ReSeg:A Recurrent Neural Network for Object Segmentation[J].Computer ence,2015.]提出的ReSeg模型,他的核心是利用多个(Recurrent neuralnetworks)RNN 组合在一起,充分考虑二零图像上下文的信息关系。
3.基于上采样的图像分割方法:主要代表为2015年JonathanLong等人[Long J,Shelhamer E,Darrell T.Fully Convolutional Networks for Semantic Segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence,2015,39(4):640-651.] 提出的FCN模型,它在图像分割领域已然成为了业界标杆,其中的许多分割思想都具有开创性的意义。并且之后一些著名的如SegNet、Unet、Deeplab等网络或多或少都借鉴了其思想。
目前的许多基于深度学习的方法,广泛应用于自然图像,医学图像、生物图像的处理中,特别在对于生物图像中细菌图像的识别还有许多分割问题需要解决。
现有公开了一种基于U-Net网络对细胞进行图像分割的方法,它是主要利用U-Net网络对宫颈细胞核进行图像分割。主要包括三个步骤:
1.对图像进行图像预处理,包括利用Canny算子和Otsu算法结合数学形态学操作,完成细胞核对比度增强。
2.利用U-Net网络进行图像的分割,在单一的U-Net网络中加入了空洞卷积和残差网络,提高网络的感知域。
3.利用聚类的检测方法对细胞的边缘进行检测。
其对于图像的预处理做了很大部分的工作,较为繁琐,若是不进行这些对比度增强,则会造成分割效果不佳。
发明内容
本发明的主要目的在于克服现有技术采用的深度学习网络进行分割时,前期的图像预处理繁琐复杂,提出一种基于深度学习网络的细菌显微图像分割方法,能快速而又精准的对细菌显微图像进行自动分割,省去前期过多复杂的图像预处理环节,节省时间。
本发明采用如下技术方案:
一种基于深度学习网络的细菌显微图像分割方法,其特征在于,包括如下步骤:
1)培养细菌,并在显微镜下按照固定时间间隔拍摄一组细菌生长图片,进行图像预处理,构建彼此之间没有交集的训练集、验证集和测试集,且训练集包括原始图像和相对应的标签图像,验证集和测试集分别只包含原始图像;
2)构建U-Net++模型,其具有编码器模块和解码器模块,编码器模块进行特征提取,解码器模块进行特征还原解码到原图的尺寸,将训练集输入U-Net++模型进行训练,再将验证集输入训练后的U-Net++模型进行验证,得到训练好的U-Net++模型;
3)将测试集输入训练好的U-Net++模型,输出二值化分割图像。
优选的,步骤1)中所述图像预处理包括,先采用ImageJ图像处理软件调整图像的亮度和对比度,再对图像进行批量裁剪操作。
优选的,步骤1)中,采用开源软件Labelme来对细菌生长图片中的细菌进行人工打标签操作得到所述标签图像,并保证原始图像和标签图像一一对应。
优选的,步骤1)中,还包括对所述训练集、所述验证集和所述测试集中的图像进行反转、扭曲、镜像、裁剪操作实现数据集的扩增。
优选的,所述编码器模块包含多个相同结构的编码器子模块,编码器子模块包括两次卷积操作加上一次池化操作。
优选的,所述卷积操作采用3*3的卷积核,激活函数为leaky Relu,填充方式为samepadding,初始化权重的方法为he_normal;池化操作采用最大池化方案,使用的池化卷积核的大小为2*2。
优选的,所述解码器模块包括多个相同结构的解码器子模块,每个解码器子模块包括一次反卷积操作,一次跳连接参数融合操作和两次卷积操作。
优选的,所述反卷积操作采用2*2的卷积核,激活函数为leaky Relu,步长为2,填充的方式为samepadding;跳连接参数融合操作是将具有同层相同特征的图像参数进行融合,融合后只增加参数的深度;卷积操作采用3*3的卷积核,激活函数为leaky Relu,填充方式为samepadding,初始化权重的方法为he_normal。
优选的,所述U-Net++模型采用Adam作为优化器,学习率为1*10
优选的,还包括步骤4)将所述U-Net++模型得到的二值化分割图像进行计算细菌的占比,以此得到细菌按时间为横轴的生长情况图。
由上述对本发明的描述可知,与现有技术相比,本发明具有如下有益效果:
1、本发明的方法,采用了U-Ne++网络结构,能够自主学习选择最合适的网络层数,并且结合了浅层次网络和深层次网络获取到的不同特征,将其融合,更具鲁棒性,大大减少繁琐的图像预处理操作的耗时,并且达到更优秀的图像分割效果,且一旦网络学习完成,后续图像处理速度也将又飞速的提升。
2、本发明的方法,进行深度学习前,对图像进行简单调整图像的亮度和对比度,再对图像进行批量裁剪操作,省去了前期过多复杂的图像预处理环节,节省时间。
3、本发明的方法,在编码器模块中,每进行一次编码器子模块,就会提取出图像中包含的一定信息,比如边界和颜色,卷积次数越多,就能抓取到图像的一些抽象特征,可以增加对输入图像的一些小扰动的鲁棒性,比如图像平移,旋转等,减少过拟合的风险,降低运算量,和增加感受野的大小。
4、本发明的方法,其解码器模块包括多个解码器子模块,把抽象的特征还原解码到原图的尺寸,并最终得到分割的结果,且涉及的跳连接参数融合操作是将具有同层相同特征的图像参数进行融合,融合后只增加参数的深度,它联系了输入图像的很多信息,有助于还原降采样所带来的信息损失。
5、本发明的方法,能快速而又精准的对细菌显微图像进行自动分割,并可根据分割后的一组图像定量的表示细菌的生长情况,不干扰细菌的生长状态。
附图说明
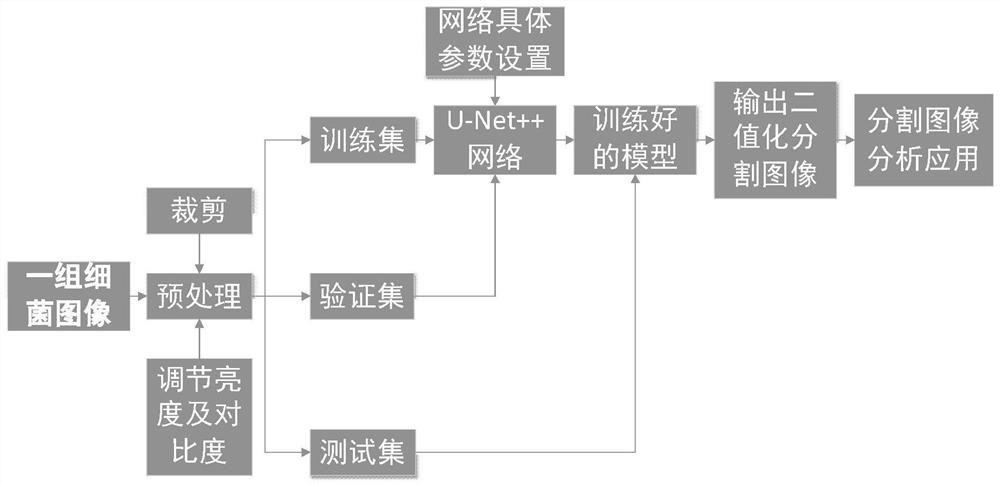
图1为本发明方法流程图;
图2a为训练集中的原始图像;
图2b为标签图像;
图3为本发明U-Net++模型的总体框架图;
图4a为测试集中的原始图像;
图4b为输出的二值化分割图像;
图5为大肠杆菌生长曲线图。
以下结合附图和具体实施例对本发明作进一步详述。
具体实施方式
以下通过具体实施方式对本发明作进一步的描述。
参见图1,一种基于深度学习网络的细菌显微图像分割方法,包括如下步骤:
1)培养细菌,并在显微镜下按照固定时间间隔拍摄一组细菌生长图片,进行图像预处理,构建彼此之间没有交集的训练集、验证集和测试集,且训练集包括原始图像和相对应的标签图像,验证集和测试集分别只包含原始图像。
由于细菌在显微镜下拍下的图像像素值一般都很大,并且图像的质量不一,所以在进行深度学习前有必要对图像进行简单的裁剪、亮度及对比度增强预处理过程。具体包括如下:
1.1)培养细菌,并在显微镜下按照固定时间间隔拍摄一组细菌生长图片。培养细菌时要尽可能的减少杂质的引入。例如在培养大肠杆菌的时候,固体培养基需要倒置以减少细菌呼吸产生的水蒸气将培养皿中的杂志引入,从而影响后续图像的质量。
同时,要选取合适的显微镜观察倍数,合适的显微镜观察倍数能让成像更加清晰。还要选取合适的拍摄间隔时间。
以大肠杆菌为例:大肠杆菌在光学显微镜下采用40倍镜以固定时间间隔5分钟拍一次图像,生成一组100张2048*2048的生长图像。在培养大肠杆菌的时候,固体培养基需要倒置以减少细菌呼吸产生的水蒸气将培养皿中的杂质引入,避免影响后续图像的质量。
1.2)打开ImageJ图像处理软件合适参数调整图像的亮度和对比度。ImageJ软件是一款开源的图像处理软件,是目前对图像进行处理操作的首选软件。对于上述步骤A1拍摄好的图像,可以直接将一组图像由ImageJ打开,再通过软件的Image下拉框,选择Adjust操作,并打开其中的Brightness/Contrast选项,在打开的操作框中可直接选择Auto按钮,软件会自动选择合适的亮度以及对比度,当然效果不理想也可自己手动调节亮度Brightness和Contrast 的值,最后点击Apply选项完成对图像的批量预处理。采用ImageJ软件最快速对图像进行简单的基本操作,软件的自带的批量化操作也让操作人员省去了不少时间。
例如:打开ImageJ图像处理软件将图像转换为单通道8bit的图像后并选择合适参数调整图像的亮度和对比度。
1.3)对上一步处理后的高像素图像进行裁剪操作。若不进行适当的裁剪,输入到深度学习网络中单次处理的数据量就会非常大,对计算机的算力要求也会线性变大。裁剪的大小根据深度学习网络的输入大小决定,一般裁剪后每张图像的大小不大于512*512像素,如果训练算力很高,可以适当加大像素值。裁剪的过程采用滑动窗口的方式进行,2048*2048像素的图像处理后变为16张512*512像素的图像,一组100张2048*2048的图片最终裁剪为1600 张512*512像素的图像。
对于构建数据集,保证训练集包括原始图像和相对应的标签图像用来输入到神经网络中进行训练,验证集和测试集只包含原始图像。例如:1600张512*512像素的图像对应每张图片的16个互不交涉的位置,取出其中10个位置的图像,得到1000张间隔5分钟的512*512 的图像作为训练集图像,剩下600张间隔5分钟的512*512的图像分别对应图像剩余两个位置,取出两个位置的200张作图片为验证集,四个位置的400张图片作为测试集。训练集:验证集:测试集=5:1:2,并且彼此之间没有交集。
在训练集中,可采用开源软件Labelme来对细菌图像中的细菌经行人工打标签操作,保证原始图像和标签图像一一对应,参见图2a、图2b;再对训练集、验证集和测试集中的图像进行反转、扭曲、镜像、裁剪等操作实现数据集的扩增。
进一步的,在对图像进行手工打标签中,采用多边形标注,需要尽可能地贴合细菌,多边形边数越多,标签图像也就越精准。标注好的图像会生成json文件,利用json转png图像格式操作后,可最终生成对应地标签文件,其中闭合多边形内的是标注的细菌,内部像素用像素值0来表示(即白色),其余的为背景用像素值255来填充(即黑色)。
其中,扩增数据集可使用Tensorflow深度学习框架中已经写好的函数ImageDataGenerator 来快速生成,包括旋转|反射变换、翻转变换、缩放变换、平移变换、尺度变换等。继承父类 ImageDataGenerator的方法后重写函数,将训练集中的图像和对应标签图像打包,同时生成增强数据的,确保原图和对应标签图像进行同样的操作转换。
2)构建U-Net++模型,其具有编码器模块和解码器模块,编码器模块(下采样)进行特征提取,解码器模块(上采样)进行特征还原解码到原图的尺寸,将训练集输入U-Net++模型进行训练,再将验证集输入训练后的U-Net++模型进行验证,得到训练好的U-Net++模型。
本发明的U-Net++模型,每一次的编码器操作后都有相应的解码器来还原,优势是可以抓取不同层次的特征,并将学习到的浅层次特征和深层次特征整合起来,并且网络可以自己去学习不同深度特征的重要性,具有很好的鲁棒性。
编码器模块也称为特征提取阶段,包含多个相同结构的编码器子模块,每个编码器子模块包括两次卷积操作加上一次池化操作。每进行一次编码器子模块,就会提取出图像中包含的一定信息,比如边界和颜色,卷积次数越多,就能抓取到图像的一些抽象特征,它可以增加对输入图像的一些小扰动的鲁棒性,比如图像平移,旋转等,减少过拟合的风险,降低运算量,和增加感受野的大小。
该模块中,卷积操作的参数为:采用3*3的卷积核,激活函数为leaky Relu,填充方式为“samepadding”,初始化权重的方法为“he_normal”;池化操作采用最大池化方案,使用的池化卷积核的大小为2*2。
实际应用中,下采样部分可以采用经典的BottleNeckblock,Residualblock或者Denseblock 替换。
解码器模块也称为特征还原模块,它的作用是把抽象的特征还原解码到原图的尺寸,并最终得到分割的结果。其包括多个相同结构的解码器子模块,每个解码器子模块包括一次反卷积操作,一次跳连接参数融合操作和两次卷积操作。
该模块中,反卷积操作的参数为:采用2*2的卷积核,激活函数为leaky Relu,步长为2,填充的方式为“samepadding”;跳连接参数融合操作是将具有同层相同特征的图像参数进行融合,融合后只增加参数的深度,它联系了输入图像的很多信息,有助于还原降采样所带来的信息损失;卷积操作的参数为:采用3*3的卷积核,激活函数为leaky Relu,填充方式为“samepadding”,初始化权重的方法为“he_normal”。
本发明模型结构如图3所示,经过四次降采样,并在每一层都进行了长短跳连接,十次上采样操作,具体U-Net++模型网络结构参数如下表所示:
优选的,本发明网络层数为4层,这里的网络层数也可以采用任意的数值来替换,分割难度大的任务往往采用更深的网络层数,反之亦然。构建好U-Net++模型后,采用Adam(Adaptive Moment Estimation)作为优化器,学习率为1*10
本发明中U-Net++网络参数例如卷积核大小、数量,学习率,优化器等都可以根据具体情况来设置合适的参数。
3)将测试集输入训练好的U-Net++模型,输出二值化分割图像。即训练好的U-Net++模型可用于细菌显微图像分割。
例如:采用硬件平台为GeForce GTX 1080Ti高能性显卡的服务器,选用Tensorflow深度学习框架,使用的数据集即为上述处理好的1600张图片加上数据增广后的图片。在长达5个小时的20个周期的训练后,U-Net++网络在验证集上的准确率达到了91.2%。训练完成后对测试集400张图像进行测试输出。
另外,传统的度量细菌生长曲线的方法是:将细菌接种到一只具有侧壁试管的三角烧瓶内的培养液中,在适宜的培养温度和良好的通气状态下,定时取出此三角烧瓶,用分光光度计测定菌浓度(光密度值,OD值),并将所得结果与相对应的培养时间绘制出坐标图,就可获得该菌的生长曲线。这种方法需要定时取出含有细菌的培养液,耗时耗力。
本发明还可包括步骤4)将通过U-Net++模型得到的二值化分割图像进行计算细菌的占比,以此得到细菌按时间为横轴的生长情况图。采用深度学习方法直接对固定间隔拍摄的细菌图像进行分割,再将分割后的二值化图像进行计算细菌的占比,以此得到细菌按时间为横轴的生长情况图。此方法完全不干扰细菌的生长状态,利用深度学习进行图像分割也更快更好的分离出细菌的前景,以便后期直接通过分割后的图像定量表示细菌的生长情况。
例如:测试时输出的图像是前景为大肠杆菌的二值化黑白图像,背景像素为0表示黑色,前景细菌像素为255表示白色,如图4a所示为测试集中某一张大肠杆菌图像,图4b为其对应预测分割输出图。对测试集输出的400张图片选择其中一组100张间隔5分钟的图片用来定量表示细菌的生长情况。按顺序读取每张图像的像素值,计算出大肠杆菌面积占整张图像的百分比,以此作为纵轴,以5分钟的时间间隔作为横轴画出大肠杆菌生长曲线,如图5所示。
本发明采用了U-Ne++网络结构,能够自主学习选择最合适的网络层数,并且结合了浅层次网络和深层次网络获取到的不同特征,将其融合,更具鲁棒性,大大减少繁琐的图像预处理操作的耗时,并且达到更优秀的图像分割效果,且一旦网络学习完成,后续图像处理速度也将又飞速的提升。
上述仅为本发明的具体实施方式,但本发明的设计构思并不局限于此,凡利用此构思对本发明进行非实质性的改动,均应属于侵犯本发明保护范围的行为。
- 一种基于深度学习网络的细菌显微图像分割方法
- 一种基于深度学习网络的病理图像分割方法
