甘蓝型油菜BnMAPK1基因在提高植物耐荫性中的应用及方法
文献发布时间:2023-06-19 11:37:30
技术领域
本发明涉及植物基因工程技术领域,具体涉及甘蓝型油菜BnMAPK1基因在提高植物耐荫性中的应用,还涉及甘蓝型油菜BnMAPK1基因提高植物耐荫性的方法。
背景技术
油菜是世界上重要的农作物之一,在种植面积、产量、植物油供给、蛋白来源等方面位于我国油料作物之首,在农业生产中占据举足轻重的地位。随着人口增长、生活水平提高和养殖业发展,食用植物油和蛋白饲料需求量不断增加。而我国耕地面积持续减少,农作物种植面积扩大空间相对有限,粮油用地矛盾突出,水土资源未得到充分利用,使得油料生产能力增长缓慢,产需缺口不断扩大,进口持续增加,对外依存度大幅度上升,食用植物油自给率不到40%。因此,提高油菜的生产能力并挖掘其增产潜力成为了遗传育种研究的核心问题。
油菜在生长过程中,受到湿害、冻害、病害和光胁迫的机率较大,其中,光照是影响油菜生长发育、产量及品质的关键因素之一。我国油菜总种植面积的70%左右在长江流域,在油菜生长的苗期及蕾薹期,长江流域正处在多雨时节,光照不足,严重制约着油菜的生产及油料行业发展。因而,挖掘油菜耐荫性相关的基因并解析其调控机理对油菜的增产及品种选育具有重要意义。
促分裂原活化蛋白激酶(Mitogen-activated protein kinases,MAPK)级联是真核生物信号传递网络中的重要途径之一,主要在基因的表达调控和细胞质功能活动中发挥着关键作用,尤其在植物的生物和非生物胁迫应答中起着重要的作用。MAPK级联途径由上至下分别由MAPKKK(MAPK kinase kinase)、MAPKK(MAPK kinase)以及MAPK组成。其中,最下游的信号模块MAPKs参与调控生长发育、细胞凋亡、响应多种生物及非生物胁迫,如病原菌、干旱、低温、高温、氧化胁迫等。拟南芥MAPKs有20个成员,分为A~D族:Kiegerl等报道A族MAPK3/6响应盐胁迫、损伤、JA、SA;Kosetsu等研究发现B族MAPK4响应渗透胁迫,参与细胞板膨胀及胞质分裂、SA及JA途径,调控病原菌的防御;Danquah等研究表明C族MAPK1参与生长素诱导的细胞扩张,响应损伤、激素、ROS、H2O2、盐胁迫等。这些研究表明,MAPKs基因在协调植物的生长发育及抗逆过程中扮演着重要角色。然而,甘蓝型油菜BnMAPK1基因在植物光响应过程中的作用尚未见报道。
发明内容
有鉴于此,本发明的目的之一在于提供甘蓝型油菜BnMAPK1基因在提高植物耐荫性中的应用;本发明的目的之二在于提供甘蓝型油菜BnMAPK1基因提高植物耐荫性的方法。
为达到上述目的,本发明提供如下技术方案:
1、甘蓝型油菜BnMAPK1基因在提高植物耐荫性中的应用,所述BnMAPK1基因的核苷酸序列如SEQ ID NO.1所示;所述植物为云薹科芸薹属植物。
优选的,所述植物为甘蓝型油菜。
本发明优选的,所述甘蓝型油菜BnMAPK1基因在弱光下提高植物的叶绿素相对含量中的应用。
本发明优选的,甘蓝型油菜BnMAPK1基因在弱光下提高植物的净光合速率中的应用。
本发明优选的,甘蓝型油菜BnMAPK1基因在弱光下提高植物的胞间CO
本发明优选的,甘蓝型油菜BnMAPK1基因在弱光下提高植物的蒸腾速率中的应用。
本发明优选的,甘蓝型油菜BnMAPK1基因在弱光下提高植物的PSⅡ光化学反应中心活性中的应用。
本发明优选的,甘蓝型油菜BnMAPK1基因在弱光下提高植物的RuBP羧化酶活性中的应用。
本发明优选的,所述弱光为光合有效辐射低于800μmol·m
2、甘蓝型油菜BnMAPK1基因提高植物耐荫性的方法,包含如下步骤:将SEQ IDNO.1所示序列构建到植物表达载体上,通过农杆菌介导,筛选转基因植株,获得耐荫性增强的植株。
本发明的有益效果在于:本发明公开了在甘蓝型油菜超量表达BnMAPK1基因,能提高弱光胁迫下油菜叶片的叶绿素相对含量,同时能够缓解长期弱光照胁迫引起的净光合速率、胞间CO
BnMAPK1受到弱光胁迫的诱导表达,qPCR相对表达水平以及RNA-Seq差异表达基因的分析表明,BnMAPK1通过光合作用-天线蛋白pathway对光系统Ⅱ复合体LHCB基因的调控作用,BnMAPK1通过负调控光系统Ⅱ中LHCB1.3、LHCB3和LHCB4.2基因的表达,进而正向调控甘蓝型油菜耐荫性的分子机理,为MAPKs级联的功能研究提供了参考资料,同时为耐荫性分子标记的开发及其优良品种的选育奠定了理论基础。
附图说明
为了使本发明的目的、技术方案和有益效果更加清楚,本发明提供如下附图进行说明:
图1为甘蓝型油菜(中油821DH)叶片的光响应趋势图;
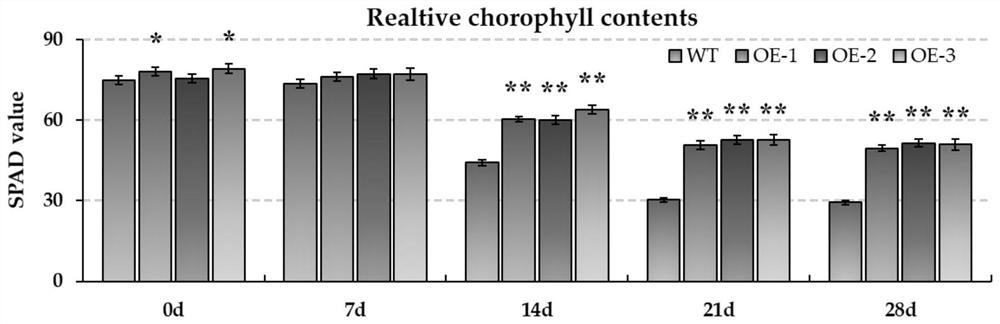
图2为BnMAPK1超量表达对弱光胁迫下油菜叶绿素相对含量的影响;
图3为BnMAPK1超量表达对弱光甘蓝型油菜光合特性的影响(A:净光合速率测定;B:气孔导度测定;C:胞间CO
图4为BnMAPK1超量表达对弱光甘蓝型油菜叶绿素荧光特性的影响(A:初始荧光测定;B:光化学淬灭系数测定;C:非光化学淬灭系数测定;D:最大光化学量子产量测定;Control:正常光照条件下,Shading:弱光处理28天);
图5为BnMAPK1超量表达对弱光甘蓝型油菜RUBP羧化酶活性的影响;
图6为弱光胁迫条件下野生型油菜BnMAPK1的基因表达分析;
图7为弱光胁迫前后的OE与WT植株的差异表达基因分析(A:弱光处理前后OE和WT各组的差异表达基因统计;B:弱光处理前后OE和WT各组差异表达基因上下调统计;C:12h_OE vs WT组显著差异表达基因的热图分析(|Log
图8为弱光胁迫条件下OE和WT植株RNA-Seq差异基因的GO富集分析(部分结果)(其中GO:0016701表示氧化还原酶活性,通过结合分子氧作用于单个供体;GO:0016702表示氧化还原酶活性,通过结合分子氧、两个氧原子而作用于单个供体);
图9为弱光胁迫条件下12h_OEvs WT组光合相关的GO-BP富集和DEGs分析(A:12h_OEvs WT组的光合相关GO-BP分析;B:12h_OEvs WT组光合相关显著差异表达基因的热图分析(|Log
图10为弱光胁迫条件下OE和WT植株RNA-Seq差异基因的KEGG富集分析(部分结果);
图11为弱光胁迫条件下BnMAPK1调控光合作用-天线蛋白pathway(A:光合作用-天线蛋白pathway模型;B:光合作用-天线蛋白pathway差异表达基因的热图分析;C:差异基因的RNA-Seq和qPCR趋势分析);
图12为弱光胁迫条件下OE和WT植株RNA-Seq数据的qPCR验证。
具体实施方式
下面结合附图和具体实施例对本发明作进一步说明,以使本领域的技术人员可以更好的理解本发明并能予以实施,但所举实施例不作为对本发明的限定。
本试验所用的甘蓝型油菜中油821DH系野生型材料由重庆市油菜工程技术研究中心提供,BnMAPK1超量表达转基因材料(T
本试验所用材料均种植于植物光照培养箱(Conviron,PGR15,Canada),生长条件为25℃(16h光照)/22℃(8h黑暗),光照强度800μmol·m
实施例1、BnMAPK1启动子的克隆及顺式作用元件分析
选取3-4周甘蓝型油菜中油821幼苗,分别提取根、茎和叶片的基因组DNA(gDNA),等质量混合,制备混合gDNA模板备用。参考GENOSCOPE数据库中甘蓝型油菜序列,设计特异引物ProBnMAPK1-F、ProBnMAPK1-R,以中油821植株混合gDNA为模板,克隆BnMAPK1(BnaC05g07560D)基因的启动子。获得了一个长度为1389bp的启动子,命名为ProBnMAPK1。克隆引物序列如下:
ProBnMAPK1-F SEQ:5′-ATACTATTGTTTAAATGCATGTACTG-3′(SEQ ID NO.2);
ProBnMAPK1-R SEQ:5′-TAGTTTCCTCTCCTTCTTCATCACA-3′(SEQ ID NO.3);
克隆得到的启动子序列如SEQ ID NO.4所示。
利用PlantCARE在线工具对ProBnMAPK1进行顺式作用元件分析,结果如表1所示,ProBnMAPK1中存在大量的光响应元件,包括Box4、G-Box、GAG-motif、Sp1、TCT-motif、As-2-Box等,表明BnMAPK1在光响应过程中具有重要的调控作用。
表1 ProBnMAPK1的主要顺式作用元件分析.
实施例2、甘蓝型油菜中油821的光响应曲线测定
为了探究BnMAPK1如何调控光响应过程,首先对甘蓝型油菜中油821DH系植株进行光合曲线的测定。待幼苗生长至3-4周时,取3株长势基本一致的健康植株,利用便携式光合仪(LICOR,LI-6400,USA)测量各植株相同部位的3片成熟叶片的净光合速率Pn(Netphotosynthetic rate)。将CO
表2甘蓝型油菜光响应拟合和实测特征参数分析.
结果如图1所示,PAR在0~800μmol·m
实施例3、BnMAPK1超量表达对弱光环境下甘蓝型油菜光合特性的影响
以甘蓝型油菜BnMAPK1超量表达(OE)和中油821DH野生型(WT)植株为试验材料,并进行300μmol·m
3.1 BnMAPK1超量表达对弱光环境下叶绿素相对含量的影响
选取3个稳定表达T
结果如图2所示,弱光处理前,与WT植株(74.86)相比,OE-1和OE-3株系的叶片SPAD值具有显著差异(p value<0.05),分别为78.17和79.23,而OE-2(75.67)与WT无显著差异。在弱光处理7d时,OE各株系的SPAD值与WT植株无显著差异。弱光处理14d、24d和28d时,OE与WT植株的SPAD值均表现为下降趋势。与WT植株相比,OE植株在14d、24d和28d时的SPAD值均极显著上调(p value<0.01),OE的平均SPAD值(61.52、52.01、50.66)分别高于WT(44.2、30.35、29.33)39.19%、71.38%和72.71%。表明,弱光胁迫可降低油菜叶片的叶绿素相对含量,而BnMAPK1能够缓解这种现象,使叶绿素相对含量维持在较高水平,通过正向调控光响应过程维持植株的光能利用能力。
3.2 BnMAPK1超量表达对弱光环境下光合参数的影响
选取OE株系(OE-1,OE-2,OE-3)和WT长势一致的健康植株,分别于弱光处理0d和28d时,采用LI-6400光合仪对各植株未被遮挡且完全展开叶片的相同部位进行光合特性的测定,包括净光合速率Pn、气孔导度Gs(Stomatal conductance)、胞间CO
结果如图3所示,正常光照条件下(0d),OE各株系与WT植株的Pn、Gs和Ci均无显著差异;但OE-1(6.91)和OE-3(6.99)株系的Tr显著高于WT(6.19),OE-2(6.77)与WT无显著差异。弱光处理28d后,OE各株系和WT植株的Pn、Gs、Ci和Tr均显示出下降趋势,但WT下降幅度更大。OE株系中,平均Pn(13.38)极显著高于WT(10.29)30.06%(图3,A);平均Gs(0.41)极显著高于WT(0.22)86.36%(图3,B);平均Ci(269.40)极显著高于WT(200.79)34.17%(图3,C);平均Tr(4.09)极显著高于WT(1.88)117.55%(图3,D)。表明,BnMAPK1超量表达能够缓解长期弱光照胁迫引起的光合速率、胞间CO
3.3 BnMAPK1超量表达对弱光环境下叶绿素荧光参数的影响
选取弱光处理0d和28d的OE和WT株系,采用LI-6400光合仪测定未被遮挡且完全展开叶片相同部位的叶绿素荧光参数。测定前,需先关闭培养箱内光源黑暗处理12h。测定参数包括初始荧光F
结果如图4所示,弱光胁迫前(0d),OE与WT植株的F
3.4 BnMAPK1超量表达对弱光环境下RuBP羧化酶活性的影响
分别测定弱光处理0、7、14、21、28d的OE和WT株系RuBP羧化酶活性。每个时间段各株系选取3株长势一致的健康植株叶片取样,置于-20℃保存备用。称取0.5g叶片加入6mL预冷的提取液(100mmol·L
结果如图5所示,弱光处理前(0d),OE平均RuBP羧化酶活性(3.69U·g
这些研究证实了BnMAPK1正向调控光合作用,提高甘蓝型油菜耐荫性,与启动子顺式作用元件分析结果一致。
实施例4、BnMAPK1正向调控甘蓝型油菜耐荫性的分子机理
为进一步检测BnMAPK1的耐荫性转录调控机理,首先对甘蓝型油菜中油821DH系植株进行弱光胁迫处理,分别于0、1.5、3、6、9、12、15h取样,并迅速置于液氮冷冻。采用RNAprep Pure Plant Kit提取植物总RNA,利用Takara PrimeScript RT reagent Kit进行RNA纯化及反转录,并参照Takara SYBR Premix Ex Taq II Kit对BnMAPK1基因的相对表达量进行qPCR检测引物为BnMAPK1-qF、BnMAPK1-qR,引物序列如下:
BnMAPK1-qF SEQ:5′-GCTCAAGCTTCTACGCCATC-3′(SEQ ID NO.5);
BnMAPK1-qR SEQ:5′-TCGAAGCAACTGGAACAAGA-3′(SEQ ID NO.6);
BnMAPK1表达趋势如图6所示,在0~3h内BnMAPK1对弱光响应缓慢;3h后表达上调,至12h达到峰值;随后表达逐渐降低;表明BnMAPK1受到弱光胁迫的诱导表达。
4.1 RNA-Seq差异表达基因分析
为了探究BnMAPK1调控光合作用的分子机理,采用Illumina平台Paired-end RNA-Seq测定弱光处理前后(0h和12h)的OE及WT植株的转录组数据,每个样本设三个生物学重复。分别比较分析弱光处理前的OE与WT组(0h_OE vs WT)、弱光处理12h的OE与WT组(12h_OEvs WT)、弱光处理前后的WT组(WT_12h vs 0h)以及弱光处理前后的OE组(OE_12h vs 0h)的差异表达基因DEGs(Differentially expressed genes)。去除FDR(False discoveryrate)>0.05的基因、同种样本三个生物学重复的FPKM(Fragments per kilobase of exonmodel per million mapped fragments)均<1以及|Log
4.2 RNA-Seq差异表达基因的GO和KEGG富集分析
对12h_OE vs WT组中的3000个DEGs进行GO分析,去除p.adjust>0.05的pathway,分别富集到分子功能MF(Molecular function)、细胞组分CC(Cellular component)和生物学过程BP(Biological process)pathway19、83和468条。其中,MF主要富集到与氧化还原酶活性、钴离子结合、核糖体结构组成、结构分子活性、四砒咯结合等分子功能;CC主要富集到细胞质、质体、叶绿体、细胞壁、胞内细胞器等细胞组分;BP主要富集到小分子代谢过程、草酸代谢过程、有机酸代谢过程、响应刺激、响应化学物质等生物学过程(图8)。
对12h_OE vs WT的468条GO-BPpathway进一步分析,共获得12条与光合相关的生物学过程,主要包括响应红光/远红光/蓝光、响应光刺激、参与光合作用中的光反应/光捕获过程、响应光强度等(图9,A)。对上述12条GO-BPpathway进一步分析,发现参与光合作用的DEGs共276个,显著表达的DEGs共19个,显著上调和下调的基因数分别为12、7个(图9,B)。
有研究表明,PSAF(PhotosystemⅠsubunit F)是光系统Ⅰ反应中心蛋白的F亚基,是光系统Ⅰ复合体的重要组成部分,介导细胞色素C6与质体蓝素中的的电子传递过程。PSBO1(PhotosystemⅡoxygen-evolving complex 1)和PSBR(PhotosystemⅡsubunit R)均能够结合光系统Ⅱ的核心区域,保持光系统Ⅱ的稳定性,前者的内源蛋白含量与植物光合效率呈正相关,后者参与调控PSBP和PSBQ亚基形成。LHCB4.2(Light-harvesting complexphotosystemⅡ4.2)是光捕获天线复合体的组分之一,响应光刺激,参与调控光系统Ⅱ中的光捕获过程。我们的研究发现,与弱光胁迫WT植株相比,弱光胁迫OE植株光系统Ⅰ基因PSAF上调11.7倍,光系统Ⅱ基因PSBO1和PSBR分别上调286.5、8.8倍,而光系统Ⅱ中LHCB4.2(Light-harvesting complex photosystemⅡ4.2)基因下调862.0倍。这些结果表明,BnMAPK1可能通过调节光系统复合体重要亚基的表达参与光合作用过程,进而提高甘蓝型油菜植株的耐荫性。
对12h_OE vs WT组中的3000个DEGs进行KEGG分析,去除Qvalue>0.05的pathway,共富集到18条pathway,包括过氧化物酶体、脂肪酸降解、碳代谢、光合作用-天线蛋白、次生代谢产物的生物合成等(图10)。这些结果进一步证实了BnMAPK1在甘蓝型油菜光合作用的重要功能。
4.3 BnMAPK1调控光捕获复合体(LHCⅡ)的形成
对KEGG富集到的光合作用-天线蛋白pathway进行分析,发现光系统Ⅰ相关基因的表达无显著变化,而光系统Ⅱ中LHCB1-4和LHCB6基因的表达明显受到BnMAPK1的调控(图11,A);其中,编码LHCB1-4和LHCB6亚基的13个基因的转录水平显著下调(图11,B)。在此基础上,我们对设计了9个基因的qPCR特异引物(表3)对RNA-Seq数据进行验证,结果显示qPCR相对表达水平与RNA-Seq趋势一致(图11,C),表明RNA-Seq数据可靠,进一步证实了BnMAPK1通过光合作用-天线蛋白pathway对光系统Ⅱ复合体LHCB基因的调控作用。
表3 RNA-Seq差异基因的qPCR检测引物
弱光胁迫后的OE和WT植株的qPCR验证结果如图12所示,LHCB1.4(BnaC03g18980D/BnaA05g09410D/BnaA03g15830D)在OE中的表达(2496.7±767.3、1422.2±139.1、1202.6±342.4)分别为WT(7381.9±1198.0、3463.1±261.9、4486.6±470.7)的0.34、0.41、0.27倍;然而LHCB1.4(BnaA05g09380D)拷贝在OE(0.02)和WT(0.10)植株中的表达均较低。LHCB2.3(BnaC09g01520D)和LHCB6(BnaC08g38660D)基因在OE中的表达(1783.2±232.8、1427.1±198.7)为WT(3060.8±594.1、2103.7±505.8)的0.58、0.68倍,相对差异较低。LHCB1.3(BnaA04g20150D)、LHCB3(BnaA10g07350D)和LHCB4.2(BnaA05g29390D)基因在OE(2298.1±473.8、428.3±194.9、356.2±108.6)中表达较WT(10415.1±949.6、1879.6±203.6、4317.2±531.0)分别为0.22、0.23、0.08倍,相对差异较高。这些结果表明,BnMAPK1可能通过负调控LHCB1.3(BnaA04g20150D)、LHCB3(BnaA10g07350D)和LHCB4.2(BnaA05g29390D)基因的表达,影响光系统Ⅱ复合体的稳定性及光合效率,进而提高甘蓝型油菜的耐荫性。有趣的是,我们前期的BnMAPK1酵母双杂交文库筛选、点对点酵母双杂交及双分支荧光素酶互补试验发现,BnaA02g09700D拷贝编码的LHCB3蛋白能够与BnMAPK1发生相互作用,但其转录水平并不受到BnMAPK1的影响。因此,我们推测BnMAPK1可能通过不同的分子机制调控LHCB3不同的拷贝,影响LHCB3亚基的稳定性,进而通过光系统Ⅱ复合体提高甘蓝型油菜的光合效率,增强植株的耐荫性,使其在弱光环境能够维持正常的生长发育。
综上所述,我们的研究阐述了BnMAPK1通过负调控光系统Ⅱ中LHCB1.3、LHCB3和LHCB4.2基因的表达,进而正向调控甘蓝型油菜耐荫性的分子机理,为MAPKs级联的功能研究提供了参考资料,同时为耐荫性分子标记的开发及其优良品种的选育奠定了理论基础。
以上所述实施例仅是为充分说明本发明而所举的较佳的实施例,本发明的保护范围不限于此。本技术领域的技术人员在本发明基础上所作的等同替代或变换,均在本发明的保护范围之内。本发明的保护范围以权利要求书为准。
序列表
<110> 西南大学
<120> 甘蓝型油菜BnMAPK1基因在提高植物耐荫性中的应用及方法
<160> 24
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1107
<212> DNA
<213> 甘蓝型油菜(Brassica napus L.)
<400> 1
atggcgacac cagttgatcc tcctaatggt gttaggaacc aagggaagca ttacttcacc 60
atgtggcaaa acctattcga gatcgacacc aagtacatgc caatcaaacc cattggccgt 120
ggtgcatacg gtgtcgtctg ctcttcggtt aacactgata acaacgagaa agttgctatc 180
aagaagattc acaatgtcta tgagaatagg atcgatgcat tgaggactct acgtgagctc 240
aagcttctac gccatcttag acatgaaaat gtcattgctt tgaaagatgt catgatgcct 300
attcataaga ggagcttcaa ggatgtgtat cttgtttatg agctcatgga tactgatctc 360
caccagatta tcaagtcttc tcaagttctt agtaatgatc actgccaata cttcttgttc 420
cagttgcttc gagggctcaa gtatatacac tcagccaaca tactccaccg agatttgaaa 480
ccaggtaacc tccttatcaa cgcgaactgc gatctgaaga tatgtgactt cggccttgcg 540
agaacgagca acaccaaggg acagctcatg actgaatatg tagtgactcg ttggtacaga 600
gctcctgagc ttctcctctg ctgcgacaac tacggaacat ccattgatgt ttggtccgtt 660
ggttgcattt tcgccgagct tctcggtaga aaaccgattt tccaaggaac tgaatgctta 720
aaccagctta aactcattgt caacattctt ggtagccaaa gagacgaaga tcttgagttc 780
atagataacc ccaaagccaa acgttacatc agatcgcttc cttattcacc tgggatgtct 840
ttatctaggc tttacccggg agctcatgtt ttggccatcg accttcttca gaaaatgctt 900
gtttttgatc cgtccaagag gattagtgtt actgaagcgc ttcagcatcc ttacatggcg 960
cctctgtatg atccgaatgc aaaccctccg gctcaagtcc ctattgatct cgatgttgat 1020
gaggagttgg gagaggagat gataagagag atgatgtgga atgagatgct tcattaccat 1080
cctcaagctt caccatctga gctgctc 1107
<210> 2
<211> 26
<212> DNA
<213> Artificial Sequence
<400> 2
atactattgt ttaaatgcat gtactg 26
<210> 3
<211> 25
<212> DNA
<213> Artificial Sequence
<400> 3
tagtttcctc tccttcttca tcaca 25
<210> 4
<211> 1389
<212> DNA
<213> 甘蓝型油菜(Brassica napus L.)
<400> 4
atactattgt ttaaatgcat gtactgtaaa gattaatttt actctggtta agctaaggag 60
cacccagaaa aagggtaaac aaacaactgg aaagaatctc tgtctggttt tctataatga 120
ttaaaagctg tattgcttat ggtgaattag ttggaaatct gtaaatatac tttttatatt 180
atatggtgtg tgtgtgttta tgtttcaaaa tatctgagac atgctttacc ataaaggctt 240
tctggctgag ctgtaatatt gacataattg gttgtaagag aatgagtgat cataatgata 300
cctaacattt ttgtttaatc aaacaccaaa ccttatttta tttgtttata cttaaatgtt 360
acaaaccctt tcattccata ctgtttttta gatttttgcc caccccctag atttatgggc 420
cttccctttt ttgcctgcat gggcctactt tctcttatta atggaaactt ttgactattt 480
tatcttttat tctgtgatca tctcccattt tcccctagat gtaacagaat ttcaatttag 540
acttagactt agagtgactg tattttgata tatttttttc agttctcaaa cgttgttacc 600
tttcattata tttaacatgt cttttggtgt taaaaattag agttttccta gagggaaaaa 660
aatcgtagag taagatttaa tattagcccg tttcacatat gacaagatga tgatgatgca 720
tagtttggtg aacaatcacc actaacagaa aaataattta ataatgacgt gaatcttttt 780
gttaactggt ttgggcttta atttaagccc atataataag acaagatcat gtaggcatca 840
cccactttcc tccaaaagat aacgtacgaa caagcaagta gagagtcaac tgtccgaacg 900
ttgaccaaat ctctcacttc cttccacggt tccatcttct ctctccggcc aattctaaga 960
tctcaccctc atcgttgatc gtcggcgtac accaactgtt tgtaactcat ccctacgttc 1020
tccggtacac atttttcaca ttctgatctc acaagttcct ttaaaattcg actaagattg 1080
tcaagcttaa gatttgtatc ttttcatttt cacttttgat agaacaatca ttttgcgcaa 1140
gaaattcact gaaatacagt ttaaattcta aagtttgtga tattacaagt aaatcaattc 1200
tttagttttt cttttttaaa atctttaatt ttgtgttctt gaggtgttag ttgatgttcg 1260
tgtgatcaaa gtgttatgaa aaaattatat attttttgga tgatagttat gaaaaaatta 1320
tatatttttt tctgtgtatc aatttgattt cttgattggt gaattgtgat gaagaaggag 1380
aggaaacta 1389
<210> 5
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 5
gctcaagctt ctacgccatc 20
<210> 6
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 6
tcgaagcaac tggaacaaga 20
<210> 7
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 7
ttctccttca gcatcagaag tcc 23
<210> 8
<211> 24
<212> DNA
<213> Artificial Sequence
<400> 8
taggtacttg actctttcgg aacc 24
<210> 9
<211> 26
<212> DNA
<213> Artificial Sequence
<400> 9
ttgaaggtta cagagtcgcc ggagaa 26
<210> 10
<211> 28
<212> DNA
<213> Artificial Sequence
<400> 10
tgggtcggta gcaagaccca acgggtca 28
<210> 11
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 11
aaccgcccgt gtcactatgc gc 22
<210> 12
<211> 25
<212> DNA
<213> Artificial Sequence
<400> 12
aacttctagc tcacggtttc ttgca 25
<210> 13
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 13
cgaggtcttt ggaaccgggc 20
<210> 14
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 14
tgaggtagct cgggggctct cca 23
<210> 15
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 15
agccatttgg gctactcagc c 21
<210> 16
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 16
cctttaactc tgcgaaggcc 20
<210> 17
<211> 28
<212> DNA
<213> Artificial Sequence
<400> 17
acttcagcta tccaacactc ctctttta 28
<210> 18
<211> 30
<212> DNA
<213> Artificial Sequence
<400> 18
tatgacggtg tgttctctga gaatggacct 30
<210> 19
<211> 26
<212> DNA
<213> Artificial Sequence
<400> 19
attcatgagc tcaagcagtg ttttga 26
<210> 20
<211> 26
<212> DNA
<213> Artificial Sequence
<400> 20
atctcccaag tacactatgg gaaatg 26
<210> 21
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 21
ggctccagag gttcagagag tgt 23
<210> 22
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 22
ccatccacta gctctacctt gcc 23
<210> 23
<211> 24
<212> DNA
<213> Artificial Sequence
<400> 23
tttgggaacc ggcgtaggca ctgg 24
<210> 24
<211> 26
<212> DNA
<213> Artificial Sequence
<400> 24
gtcttttccc aaacccaacg ggtcga 26
- 甘蓝型油菜BnMAPK1基因在提高植物耐荫性中的应用及方法
- 甘蓝型油菜BnMAPK1基因在提高植物耐荫性中的应用及方法
