一种定性和定量测定蛋白质的方法及应用
文献发布时间:2023-06-19 11:47:31
技术领域
本发明属于蛋白质检测技术领域,具体涉及一种定性和定量测定蛋白质的方法及应用。
背景技术
目前常用的蛋白质检测方法,一般为采用放射免疫测定法(RIA)或酶联免疫吸附分析法(ELISA),以定量定性分析样品中含有的蛋白质,但需要造价昂贵的检测设备,且操作过程复杂,因此不能随时随地的进行检测分析。
除了上述检测方法外,生化实验室中还常采用双缩脲法、BCA法、考马斯亮蓝法、Lowry法和紫外吸收法等生化检测方法来测定蛋白含量。但是上述方法还存在如下缺点:例如,考马斯亮蓝法对测试样品中的蛋白分子大小有更多限制;紫外吸收法基于蛋白中的芳香族氨基酸残基在280nm下的光吸收值进行检测,由于不同蛋白中的芳香族氨基酸的含量不同,该方法的检测准确性和专一性不好;同时,对于传统的双缩脲法、BCA法和Lowry法而言,并不适用于对含有溶解性差的醇溶蛋白和谷蛋白的谷物样品以及大部分动、植物工业衍生品中的蛋白含量进行检测。而且,待测样品体系中存在的淀粉、脂类以及纤维等成分也会影响上述实验室方法的检测准确度。
发明内容
有鉴于此,本发明的目的在于提供一种定性和定量测定蛋白质的方法及应用,所述测定方法基于三元络合物体系检测蛋白质,是一种快速准确的蛋白质定性定量的测定方法,还能用于体外高通量筛选细胞株。
为了实现上述发明目的,本发明提供以下技术方案:
本发明提供了一种定性和定量测定蛋白质的方法,包括以下步骤:(1)根据待测蛋白质种类,筛选分别与待测蛋白质的不同表位特异性结合的第一蛋白和第二蛋白,第一蛋白和第二蛋白间无相互作用;
(2)将第一蛋白的编码基因与报告酶的一段基因片段融合后,表达纯化得第一融合蛋白;将第二蛋白的编码基因与报告酶的另一段基因片段融合后,表达纯化得第二融合蛋白;
(3)将包含待测蛋白质的溶液与第一融合蛋白和第二融合蛋白混合孵育,将孵育产物和所述报告酶的底物混合后,检测光强度,从而检测所述溶液中所述待测蛋白质存在与否和所述待测蛋白质的含量。
优选的,当步骤(1)中所述待测蛋白质为免疫球蛋白G时,第一蛋白的种类包括Protein G或Protein L,第二蛋白的种类包括Protein L或Protein G。
优选的,步骤(2)所述报告酶包括荧光素酶NanoLuc@、分裂辣根过氧化物酶、分裂过氧化物酶APEX2或分裂荧光素酶。
优选的,所述荧光素酶NanoLuc@的一段基因片段的核苷酸序列包含SEQ ID NO.1所示的序列,另一段基因片段的核苷酸序列包含SEQ ID NO.2所示的序列。
优选的,所述分裂辣根过氧化物酶的一段基因片段的核苷酸序列包含SEQ IDNO.3所示的序列,另一段基因片段的核苷酸序列包含SEQ ID NO.4所示的序列。
优选的,所述分裂过氧化物酶APEX2的一段基因片段的核苷酸序列包含SEQ IDNO.5所示的序列,另一段基因片段的核苷酸序列包含SEQ ID NO.6所示的序列。
优选的,所述分裂荧光素酶包括海肾荧光素酶、萤火虫荧光素酶、发红光叩甲虫荧光素酶或发绿光叩甲虫荧光素酶。
优选的,步骤(3)所述包含待测蛋白质的溶液、第一融合蛋白和第二融合蛋白混合溶液、报告酶的底物的体积比为98:1:1。
本发明还提供了上述方法在体外筛选高表达细胞株中的应用。
本发明提供了一种定性和定量测定蛋白质的方法,所述方法基于三元络合物体系检测蛋白质,将两个分别与待测溶液中不同表位特异性结合的没有相互作用的蛋白,依次与报告酶的不同片段组合成融合基因,并通过表达纯化后,获得两种融合蛋白,所述两个融合蛋白内包含的报告酶的蛋白片段在相互靠近时会发生重组,当加入报告酶底物时,会释放光信号,同时,被测蛋白的参考标准物质(或者参考材料)的发光程度与其浓度成正相关,且通过发光读数的检测数据即可高通量筛选出高表达的细胞株。
附图说明
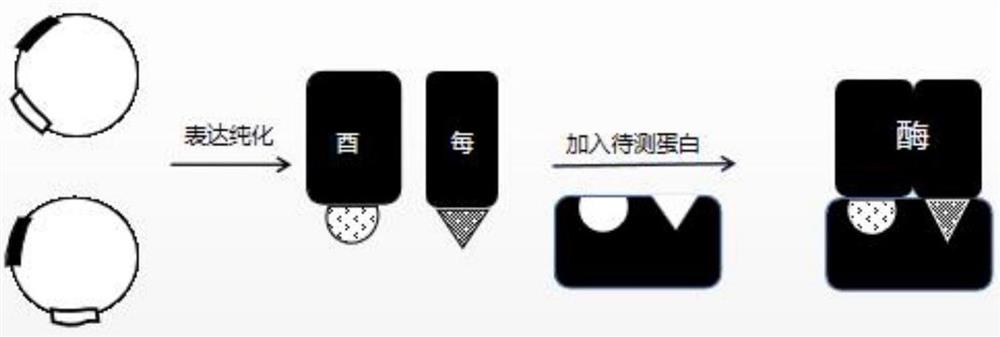
图1为本发明所述定性和定量测定蛋白质的方法的实验流程图;
图2为以荧光素酶NanoLuc@为基础定量蛋白质;
图3为分裂辣根过氧化物酶为基础定量蛋白质;
图4为分裂过氧化物酶APEX2为基础定量蛋白质。
具体实施方式
本发明提供了一种定性和定量测定蛋白质的方法,具体流程如图1所示,包括以下步骤:(1)根据待测蛋白质种类,筛选分别与待测蛋白质的不同表位特异性结合的第一蛋白和第二蛋白,第一蛋白和第二蛋白间无相互作用;
(2)将第一蛋白的编码基因与报告酶的一段基因片段融合后,表达纯化得第一融合蛋白;将第二蛋白的编码基因与报告酶的另一段基因片段融合后,表达纯化得第二融合蛋白;
(3)将包含待测蛋白质的溶液与第一融合蛋白和第二融合蛋白混合孵育,将孵育产物和所述报告酶的底物混合后,检测光强度,从而检测所述溶液中所述待测蛋白质存在与否和所述待测蛋白质的含量。
本发明根据待测蛋白质种类,筛选分别与待测蛋白质的不同表位特异性结合的第一蛋白和第二蛋白,第一蛋白和第二蛋白间无相互作用。当所述待测溶液中蛋白质为免疫球蛋白G时,第一蛋白的种类包括Protein G(SEQ ID NO.7)或Protein L(SEQ ID NO.8),第二蛋白的种类包括Protein L或Protein G。本发明对所述Protein G和Protein L的来源并没有特殊限定,优选为本领域的常规市售产品。
本发明将第一蛋白的编码基因与报告酶的一段基因片段融合后,表达纯化得第一融合蛋白;将第二蛋白的编码基因与报告酶的另一段基因片段融合后,表达纯化得第二融合蛋白。本发明所述报告酶优选包括荧光素酶NanoLuc@、分裂辣根过氧化物酶、分裂过氧化物酶APEX2或分裂荧光素酶,且所述荧光素酶NanoLuc@的一段基因片段(LgBit)的核苷酸序列优选包含SEQ ID NO.1所示的序列,另一段基因片段(SmBit)的核苷酸序列优选包含SEQID NO.2所示的序列;所述分裂辣根过氧化物酶的一段基因片段(HPR L)的核苷酸序列优选包含SEQ ID NO.3所示的序列,另一段基因片段(HPR R)的核苷酸序列优选包含SEQ IDNO.4所示的序列;所述分裂过氧化物酶APEX2的一段基因片段(AP)的核苷酸序列优选包含SEQ ID NO.5所示的序列,另一段基因片段(Ex2)的核苷酸序列优选包含SEQ ID NO.6所示的序列。本发明所述分裂荧光素酶优选包括海肾荧光素酶、萤火虫荧光素酶、发红光叩甲虫荧光素酶或发绿光叩甲虫荧光素酶。本发明对所述表达纯化的方法并没有特殊限定,优选包括真核表达后纯化或原核表达后纯化。本发明实施例中,包含第一蛋白的基因序列、荧光素酶基因片段(LgBit)的表达载体可以为pET28a-Protein G-LgBit,包含第二蛋白的基因序列、荧光素酶互补基因片段(SmBit)的表达载体可以为pET28a-Protein L-SmBit,经表达纯化后获得的融合蛋白分别为第一融合蛋白Protein G-LgBit和第二融合蛋白Protein L-SmBit。
得第一融合蛋白和第二融合蛋白后,本发明将包含待测蛋白质的溶液与第一融合蛋白和第二融合蛋白混合孵育,将孵育产物和所述报告酶的底物混合后,检测光强度,从而检测所述溶液中所述待测蛋白质存在与否和所述待测蛋白质的含量。本发明所述第一融合蛋白和第二融合蛋白中分别包含的第一蛋白和第二蛋白之间没有相互作用,且其内报告酶的两个片段也不能相互靠近并重组,即便是加入报告酶底物后,检测仅包含两种融合蛋白的混合物时发光强度很弱;当将上述两种融合蛋白中的第一蛋白和第二蛋白,同时特异性结合到待测样本中的蛋白质的不同表位,形成三元络合物,报告酶的两个片段相互靠近并重组,加入报告酶底物后,检测包含待测样本与两种融合蛋白的三元络合物时,会出现明显的发光现象,同时,被测蛋白的参考标准物质(或者参考材料)的发光程度与其浓度成正相关关系。
本发明进行混合孵育时,所述包含待测蛋白质的溶液、第一融合蛋白和第二融合蛋白混合溶液、报告酶的底物的体积比为98:1:1。在本发明实施例中,利用免疫球蛋白G进行实验时,包含Protein G的融合蛋白与包含Protein L的融合蛋白的体积比为1:2。本发明所述混合孵育的温度优选为室温,时间为优选2~5min。
本发明还提供了上述方法在体外筛选高表达细胞株中的应用。
本发明所述方法优选与上述定性和定量测定蛋白质的具体操作方法相同,在此不再赘述。
下面结合实施例对本发明提供的一种定性和定量测定蛋白质的方法及应用进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。
实施例1
以荧光素酶NanoLuc@为报告酶,构建分别包含Protein G-LgBit基因以及ProteinL-SmBit基因的载体,纯化获得以上2种融合蛋白,检测已知浓度的待测物的发光值,并以待测物的浓度为横坐标,发光值为纵坐标绘制标准曲线。
1、将ProteinG-LgBit、ProteinL-Smbit基因构建至pET-28a(+)原核表达载体,并进行测序验证
(1)人工合成ProteinG-LgBit序列(SEQ ID NO.9),并连接入pET28a(+)载体的HindIII和NotI酶切位点间,转化Stbl3感受态细胞,筛选阳性菌落进行测序,对测序正确的菌落小提质粒,并酶切验证。
(2)人工合成ProteinL-Smbit序列(SEQ ID NO.10),并连接入pET28a(+)载体的HindIII和NotI酶切位点间,转化Stbl3感受态细胞,筛选阳性菌落进行测序,对测序正确的菌落小提质粒,并酶切验证。
2、两种融合蛋白表达纯化
1)将质粒转化到BL21大肠杆菌细胞中。
2)挑选菌落接种到含有50μg/ml卡那霉素20ml LB培养基中,并于37℃摇动过夜。
3)将培养物转移到1L的新鲜LB培养基中,该培养基包含50μg/ml卡那霉素。
4)当培养物在摇动(250rpm)下于37℃下生长至OD
5)将培养物进一步在37℃下孵育3~4h或在30℃下孵育6~8h。
6)表达结束后,5000rpm,离心15min,收集细胞,并在纯化前-20℃保存。
7)将来自2L培养物的细胞沉淀重悬于含有1mM苯甲基磺酰氟(PMSF)的20ml缓冲液I(200mM NaCl,50mM Na
8)离心细胞裂解液以除去不溶的细胞碎片,并过滤上清液(孔径为0.22μm,Corning),并保持在冰上直至装载到纯化柱上。
9)装柱:将HisSep Ni-NTAAgarose Resin借助重力的作用装入合适的纯化柱中。
10)清洗:3-5倍柱体积去离子水冲洗色谱柱。
11)平衡:至少5倍柱体积的Lysis Buffer平衡色谱柱。
12)上样。
13)平衡:Wash Buffer平衡柱子,直到紫外吸收达到一个稳定的基线(一般至少10-15个柱体积)。注意收集流出液。
14)洗脱:使用5-10倍柱体积Elution Buffer洗脱,收集洗脱液即目的蛋白溶液。
3、上机检测
1)标准溶液:将已知浓度的兔IgG分别用新鲜培养基配制成浓度为0、5、10、15、20、25、30、40、50ng/μl的检测液。
2)融合蛋白混合液:分别取100ng ProteinL-smbit融合蛋白、50ng ProteinG-Lgbit融合蛋白,加入100μl上清液中充分混合。
3)程序:分别取98μl的每个标准溶液,空白溶液,并依次加入1μl的融合蛋白混合液,1μl的发光底物,混匀;室温下孵育2~5min,荧光照度计检测荧光值。
结果如图2显示,二种融合蛋白能很好的结合IgG蛋白;且随着IgG蛋白浓度的升高,荧光值越高,且呈良好的线性关系。
实施例2
以分裂辣根过氧化物酶为报告酶,构建分别包含ProteinG-HRP-L基因以及ProteinL-HRP-R基因的载体,纯化获得以上2种融合蛋白,检测已知浓度的待测物的发光值,并以待测物的浓度为横坐标,荧光值为纵坐标绘制标准曲线。
1、将ProteinG-HRP-L、ProteinL-HRP-R基因构建至pET-28a(+)原核表达载体,并进行测序验证
(1)人工合成如SEQ ID NO.11所示的ProteinG-HRP-L的基因序列,连接入pET28a(+)的HindIII和XhoI酶切位点间,连接产物转化Stbl3感受态细胞,筛选阳性菌落进行测序,对测序正确的菌落小提质粒,并酶切验证。
(2)人工合成如SEQ ID NO.12所示的ProteinL-HRP-R的基因序列,连接入pET28a(+)的NdeI和XhoI酶切位点间,连接产物转化Stbl3感受态细胞,筛选阳性菌落进行测序,对测序正确的菌落小提质粒,并酶切验证。
2、两种融合蛋白表达纯化
融合蛋白提取纯化同实施案例1。
3、上机检测
1)标准溶液:将已知浓度的兔IgG分别用新鲜培养基配制成浓度为0、1、3、6、9、12、15、18、21、24、27、30ng/μl的检测液。
2)融合蛋白混合液:分别取100ng ProteinL-HRP-R融合蛋白、50ng ProteinG-HRP-L融合蛋白,加入100μl上清液中充分混合。
3)程序:分别取98μl的每个标准溶液,空白溶液,并依次加入1μl的融合蛋白混合液,1μl的发光底物,混匀;室温下孵育2~5min,荧光照度计检测荧光值。
结果如图3显示,二种融合蛋白能很好的结合IgG蛋白;且随着IgG蛋白浓度的升高,荧光值越高,且呈良好的线性关系。
实施例3
以分裂过氧化物酶APEX2为报告酶,构建分别包含ProteinG-AP基因以及ProteinL-EX2基因的载体,纯化获得以上2种融合蛋白,检测已知浓度的待测物的发光值,并以待测物的浓度为横坐标,发光值为纵坐标绘制标准曲线。
1、将ProteinG-AP(SEQ ID NO.13)、ProteinL-EX2(SEQ ID NO.14)基因构建至pET-28a(+)原核表达载体,将测序正确的序列分别插入pET28a(+)载体的HindIII/XhoI和NdeI/XhoI酶切位点间,连接产物转化Stbl3感受态细胞,筛选阳性菌落进行测序,对测序正确的菌落小提质粒。
2、两种融合蛋白表达纯化
融合蛋白提取纯化同实施案例1。
3、上机检测
1)标准溶液:将已知浓度的兔IgG分别用新鲜培养基配制成浓度为0、1、3、6、9、12、15、18、21、24、27、30ng/μl的检测液。
2)融合蛋白混合液:分别取100ng ProteinL-EX2融合蛋白、50ng ProteinG-AP融合蛋白,加入100μl上清液中充分混合。
3)程序:分别取98μl的每个标准溶液,空白溶液,并依次加入1μl的融合蛋白混合液,1μl的发光底物,混匀;室温下孵育2~5min,荧光照度计检测荧光值。
结果如图4显示,二种融合蛋白能很好的结合IgG蛋白;且随着IgG蛋白浓度的升高,荧光值越高,且呈良好的线性关系。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110> 广州辉骏生物科技股份有限公司
<120> 一种定性和定量测定蛋白质的方法及应用
<160> 14
<170> SIPOSequenceListing 1.0
<210> 1
<211> 477
<212> DNA
<213> 人工序列(artifical sequence)
<400> 1
atggtgttca ccctggagga cttcgtgggc gattgggagc agacagccgc ctacaacctg 60
gatcaggtgc tggaacaggg aggagtgtcc agcctgctgc agaatctggc agtgtccgtg 120
acccccatcc agagaatcgt gagaagcggc gagaacgccc tgaagatcga catccacgtg 180
atcatcccct acgagggact gagcgccgat cagatggccc agatcgagga ggtgttcaag 240
gtggtgtacc ccgtggacga tcaccacttc aaggtcatcc tgccctacgg cacactggtc 300
atcgacggag tgacccccaa catgctcaac tacttcggca ggccttacga aggcatcgcc 360
gtgttcgacg gcaagaagat caccgtgacc ggcaccctct ggaacggcaa caagatcatc 420
gacgagagac tgatcacccc agacggctct atgctgttcc gcgtgaccat caacagc 477
<210> 2
<211> 36
<212> DNA
<213> 人工序列(artifical sequence)
<400> 2
agcgtgacgg ggtatcgctt atttgaggag attctg 36
<210> 3
<211> 636
<212> DNA
<213> 人工序列(artifical sequence)
<400> 3
atgcagttga cccctacgtt ttacgacaac tcctgcccaa acgtatcgaa tatagtgaga 60
gacacaattg tgaacgagct aagatctgac ccgagaattg cggcttctat cctaagacta 120
cacttccatg actgctttgt caatgggtgt gacgccagta tcttgctcga caacaccacc 180
tcatttcgca ccgaaaagga cgcctttggg aacgcaaact cagcgcgggg gttcccggtg 240
atcgatcgta tgaaggcagc ggtagagtca gcttgcccaa gaacagtctc ctgcgcagat 300
ctattgacaa ttgctgctca gcagtcggta acgctggccg gaggaccgtc gtggcgcgtc 360
cctttgggcc gacgcgactc tcttcaagca ttcctggacc tagcaaatgc taatcttccg 420
gccccctttt ttaccttgcc ccagttgaag gactcattcc ggaacgtggg tcttaaccga 480
tcaagcgact tggtagcctt atcaggcgga cacaccttcg gcaagaacca gtgcaggttt 540
atcatggaca gactatataa tttctctaac acggggctac ctgacccgac tcttaatacc 600
acgtatttac agacactcag gggcttatgt ccgtta 636
<210> 4
<211> 294
<212> DNA
<213> 人工序列(artifical sequence)
<400> 4
atgaatggta acctttctgc actggtagac ttcgatttga gaactccaac cattttcgat 60
aacaagtact acgtcaatct agaagaacaa aaagggctaa tacagtccga ccaggagctg 120
ttttcatcgc cgaacgcaac cgatacaatc ccactagtta ggtccttcgc aaactcaact 180
cagacctttt ttaatgcatt tgtggaagcg atggatcgta tgggtaacat tacgccccta 240
accgggaccc aaggtcaaat ccgacttaat tgtagagtcg ttaattctaa ctca 294
<210> 5
<211> 600
<212> DNA
<213> 人工序列(artifical sequence)
<400> 5
atgggaaagt cttacccaac cgtgagcgct gattaccaga aggccgttga gaaggcgaag 60
aagaggctcg gaggcttcat cgctgagaag agatgcgctc ctctaatgct ccgtttggca 120
tggcactctg ctggaactta cgacgtgcgc tcgaagaccg gtggtccctt cggaaccata 180
agatacccct ccgaactcgc tcacggcgct aacagcggtc ttgacatcgc tgttaggctt 240
ttggagccac tcaaagcgga gtttcctatt ttgagctacg ccgatttcta ccagttggct 300
ggcgttgttg ccgttgaggt caccggtgga cctgaagttc ccttccatcc tggaagagag 360
gacaaacctg agctaccacc agagggtcgc ttgcccgatg ccactaaggg ttctgaccat 420
ttgagagatg tgtttggcaa agctatgggg cttagtgacc gagatatcgt tgctctgtct 480
gggggtcaca ctcttggagc tgcgcacaag gagcgttctg gattcgaggg tccctggacc 540
tctaatcctc ttgttttcga caactcatac ttcaaggagt tgttgagtgg tgagaaagaa 600
<210> 6
<211> 150
<212> DNA
<213> 人工序列(artifical sequence)
<400> 6
ggcctccttc agctaccttc tgacaaggca cttttgtctg accctgtgtt ccgccctctt 60
gttgagaaat atgcatcgga cgaagatgcc ttcttcgctg attacgctga ggctcaccaa 120
aagctttctg agcttgggtt tgctgaagcc 150
<210> 7
<211> 618
<212> DNA
<213> 人工序列(artifical sequence)
<400> 7
catcatcatc atcaccatca tcatggtatg acctataaac tgattctgaa tggcaaaacc 60
ctgaaaggcg aaaccaccac cgaagcagtt gatgcagcaa ccgcagaaaa agtttttaaa 120
cagtatgcca atgacaacgg tgttgatggt gaatggacct atgatgatgc aaccaaaacc 180
tttaccgtta ccgaaaaacc ggaagtgatt gatgcaagcg aactgacccc ggcagttacc 240
acctataaat tagtgattaa tggtaaaacc ctgaagggcg aaaccacaac cgaagcagtg 300
gatgccgcaa ccgcagagaa agtgtttaaa cagtacgcca atgataatgg cgttgatggc 360
gaatggacct acgatgatgc aacaaaaacc tttacagtta ccgaaaagcc ggaagtgatc 420
gatgccagtg aactgacccc tgccgtgacc acctataagc tggttattaa tggtaaaaca 480
ctgaaaggtg aaaccaccac aaaagcagtt gatgcggaaa ccgcagaaaa ggcctttaaa 540
cagtatgcga atgataatgg tgttgatggc gtttggacct atgacgatgc caccaaaacc 600
ttcaccgtta ccgagagt 618
<210> 8
<211> 1098
<212> DNA
<213> 人工序列(artifical sequence)
<400> 8
agcatgaaag aagaaacccc ggaaactccg gagacagata gcgaagagga agtaacaatt 60
aaagcaaatc ttatcttcgc gaacggcagc acccaaacgg cggaatttaa aggaaccttc 120
gaaaaagcga cgagcgaagc gtatgcgtat gcggatacct taaaaaaaga taacggcgag 180
tacaccgtgg acgtggcgga caaaggctac accctgaaca tcaagtttgc cgggaaagag 240
aagacacctg aagagcctaa ggaagaagtg accatcaaag ccaatttaat ctacgcagat 300
ggcaaaaccc aaacggctga gtttaaagga acctttgagg aggctactgc ggaggcgtat 360
cgttatgccg atgcgcttaa aaaagataat ggcgagtata ccgtggatgt agccgacaag 420
ggctatacac tcaacattaa attcgcgggc aaggaaaaga cccctgagga gccgaaagaa 480
gaagttacaa tcaaggcgaa cttgatctac gcagacggca aaacgcagac ggctgaattt 540
aaggggacgt tcgaagaggc cacagcggaa gcgtatcgct atgcagatct tctggcgaaa 600
gaaaacggca agtatacagt tgacgtggcg gataaagggt atacgctgaa cataaaattt 660
gcgggcaagg aaaagactcc ggaggagccg aaggaagagg tgactatcaa agcaaactta 720
atttatgcgg atggcaagac gcagacagcg gaatttaagg gcacctttgc cgaagcgaca 780
gccgaggcct atcgttatgc tgacctatta gcaaaagaga acggtaagta taccgcagac 840
ctggaagacg gcggctacac cattaacatt agattcgccg ggaaaaaggt ggatgaaaaa 900
ccggaagaaa aagaacaagt gaccataaaa gaaaacattt attttgaaga tggcaccgtg 960
cagaccgcga cctttaaggg tacctttgcg gaagcgacgg cagaggcgta tcgctatgct 1020
gatctgctga gcaaagaaca tgggaaatat accgcagatc tggaggatgg cggctatact 1080
attaacatac gcttcgcg 1098
<210> 9
<211> 1110
<212> DNA
<213> 人工序列(artificial sequence)
<400> 9
tataaactga ttctgaatgg caaaaccctg aaaggcgaaa ccaccaccga agcagttgat 60
gcagcaaccg cagaaaaagt ttttaaacag tatgccaatg acaacggtgt tgatggtgaa 120
tggacctatg atgatgcaac caaaaccttt accgttaccg aaaaaccgga agtgattgat 180
gcaagcgaac tgaccccggc agttaccacc tataaattag tgattaatgg taaaaccctg 240
aagggcgaaa ccacaaccga agcagtggat gccgcaaccg cagagaaagt gtttaaacag 300
tacgccaatg ataatggcgt tgatggcgaa tggacctacg atgatgcaac aaaaaccttt 360
acagttaccg aaaagccgga agtgatcgat gccagtgaac tgacccctgc cgtgaccacc 420
tataagctgg ttattaatgg taaaacactg aaaggtgaaa ccaccacaaa agcagttgat 480
gcggaaaccg cagaaaaggc ctttaaacag tatgcgaatg ataatggtgt tgatggcgtt 540
tggacctatg acgatgccac caaaaccttc accgttaccg agagttcggg tggagggtcg 600
gtgttcaccc tggaggactt cgtgggcgat tgggagcaga cagccgccta caacctggat 660
caggtgctgg aacagggagg agtgtccagc ctgctgcaga atctggcagt gtccgtgacc 720
cccatccaga gaatcgtgag aagcggcgag aacgccctga agatcgacat ccacgtgatc 780
atcccctacg agggactgag cgccgatcag atggcccaga tcgaggaggt gttcaaggtg 840
gtgtaccccg tggacgatca ccacttcaag gtcatcctgc cctacggcac actggtcatc 900
gacggagtga cccccaacat gctcaactac ttcggcaggc cttacgaagg catcgccgtg 960
ttcgacggca agaagatcac cgtgaccggc accctctgga acggcaacaa gatcatcgac 1020
gagagactga tcaccccaga cggctctatg ctgttccgcg tgaccatcaa cagcgcggcc 1080
gcactcgagc accaccacca ccaccactga 1110
<210> 10
<211> 1170
<212> DNA
<213> 人工序列(artificial sequence)
<400> 10
agcatgaaag aagaaacccc ggaaactccg gagacagata gcgaagagga agtaacaatt 60
aaagcaaatc ttatcttcgc gaacggcagc acccaaacgg cggaatttaa aggaaccttc 120
gaaaaagcga cgagcgaagc gtatgcgtat gcggatacct taaaaaaaga taacggcgag 180
tacaccgtgg acgtggcgga caaaggctac accctgaaca tcaagtttgc cgggaaagag 240
aagacacctg aagagcctaa ggaagaagtg accatcaaag ccaatttaat ctacgcagat 300
ggcaaaaccc aaacggctga gtttaaagga acctttgagg aggctactgc ggaggcgtat 360
cgttatgccg atgcgcttaa aaaagataat ggcgagtata ccgtggatgt agccgacaag 420
ggctatacac tcaacattaa attcgcgggc aaggaaaaga cccctgagga gccgaaagaa 480
gaagttacaa tcaaggcgaa cttgatctac gcagacggca aaacgcagac ggctgaattt 540
aaggggacgt tcgaagaggc cacagcggaa gcgtatcgct atgcagatct tctggcgaaa 600
gaaaacggca agtatacagt tgacgtggcg gataaagggt atacgctgaa cataaaattt 660
gcgggcaagg aaaagactcc ggaggagccg aaggaagagg tgactatcaa agcaaactta 720
atttatgcgg atggcaagac gcagacagcg gaatttaagg gcacctttgc cgaagcgaca 780
gccgaggcct atcgttatgc tgacctatta gcaaaagaga acggtaagta taccgcagac 840
ctggaagacg gcggctacac cattaacatt agattcgccg ggaaaaaggt ggatgaaaaa 900
ccggaagaaa aagaacaagt gaccataaaa gaaaacattt attttgaaga tggcaccgtg 960
cagaccgcga cctttaaggg tacctttgcg gaagcgacgg cagaggcgta tcgctatgct 1020
gatctgctga gcaaagaaca tgggaaatat accgcagatc tggaggatgg cggctatact 1080
attaacatac gcttcgcggg cggcggcagc gtgacggggt atcgcttatt tgaggagatt 1140
ctgctcgagc accaccacca ccaccactga 1170
<210> 11
<211> 1307
<212> DNA
<213> 人工序列(artificial sequence)
<400> 11
atgcatcatc atcatcacca tcatcatggt atgacctata aactgattct gaatggcaaa 60
accctgaaag gcgaaaccac caccgaagca gttgatgcag caaccgcaga aaaagttttt 120
aaacagtatg ccaatgacaa cggtgttgat ggtgaatgga cctatgatga tgcaaccaaa 180
acctttaccg ttaccgaaaa accggaagtg attgatgcaa gcgaactgac cccggcagtt 240
accacctata aattagtgat taatggtaaa accctgaagg gcgaaaccac aaccgaagca 300
gtggatgccg caaccgcaga gaaagtgttt aaacagtacg ccaatgataa tggcgttgat 360
ggcgaatgga cctacgatga tgcaacaaaa acctttacag ttaccgaaaa gccggaagtg 420
atcgatgcca gtgaactgac ccctgccgtg accacctata agctggttat taatggtaaa 480
acactgaaag gtgaaaccac cacaaaagca gttgatgcgg aaaccgcaga aaaggccttt 540
aaacagtatg cgaatgataa tggtgttgat ggcgtttgga cctatgacga tgccaccaaa 600
accttcaccg ttaccgagag ttcgggtgga gggtcatgca gttgacccct acgttttacg 660
acaactcctg cccaaacgta tcgaatatag tgagagacac aattgtgaac gagctaagat 720
ctgacccgag aattgcggct tctatcctaa gactacactt ccatgactgc tttgtcaatg 780
ggtgtgacgc cagtatcttg ctcgacaaca ccacctcatt tcgcaccgaa aaggacgcct 840
ttgggaacgc aaactcagcg cgggggttcc cggtgatcga tcgtatgaag gcagcggtag 900
agtcagcttg cccaagaaca gtctcctgcg cagatctatt gacaattgct gctcagcagt 960
cggtaacgct ggccggagga ccgtcgtggc gcgtcccttt gggccgacgc gactctcttc 1020
aagcattcct ggacctagca aatgctaatc ttccggcccc cttttttacc ttgccccagt 1080
tgaaggactc attccggaac gtgggtctta accgatcaag cgacttggta gccttatcag 1140
gcggacacac cttcggcaag aaccagtgca ggtttatcat ggacagacta tataatttct 1200
ctaacacggg gctacctgac ccgactctta ataccacgta tttacagaca ctcaggggct 1260
tatgtccgtt agcggccgca ctcgagcacc accaccacca ccactga 1307
<210> 12
<211> 1428
<212> DNA
<213> 人工序列(artificial sequence)
<400> 12
agcatgaaag aagaaacccc ggaaactccg gagacagata gcgaagagga agtaacaatt 60
aaagcaaatc ttatcttcgc gaacggcagc acccaaacgg cggaatttaa aggaaccttc 120
gaaaaagcga cgagcgaagc gtatgcgtat gcggatacct taaaaaaaga taacggcgag 180
tacaccgtgg acgtggcgga caaaggctac accctgaaca tcaagtttgc cgggaaagag 240
aagacacctg aagagcctaa ggaagaagtg accatcaaag ccaatttaat ctacgcagat 300
ggcaaaaccc aaacggctga gtttaaagga acctttgagg aggctactgc ggaggcgtat 360
cgttatgccg atgcgcttaa aaaagataat ggcgagtata ccgtggatgt agccgacaag 420
ggctatacac tcaacattaa attcgcgggc aaggaaaaga cccctgagga gccgaaagaa 480
gaagttacaa tcaaggcgaa cttgatctac gcagacggca aaacgcagac ggctgaattt 540
aaggggacgt tcgaagaggc cacagcggaa gcgtatcgct atgcagatct tctggcgaaa 600
gaaaacggca agtatacagt tgacgtggcg gataaagggt atacgctgaa cataaaattt 660
gcgggcaagg aaaagactcc ggaggagccg aaggaagagg tgactatcaa agcaaactta 720
atttatgcgg atggcaagac gcagacagcg gaatttaagg gcacctttgc cgaagcgaca 780
gccgaggcct atcgttatgc tgacctatta gcaaaagaga acggtaagta taccgcagac 840
ctggaagacg gcggctacac cattaacatt agattcgccg ggaaaaaggt ggatgaaaaa 900
ccggaagaaa aagaacaagt gaccataaaa gaaaacattt attttgaaga tggcaccgtg 960
cagaccgcga cctttaaggg tacctttgcg gaagcgacgg cagaggcgta tcgctatgct 1020
gatctgctga gcaaagaaca tgggaaatat accgcagatc tggaggatgg cggctatact 1080
attaacatac gcttcgcggg cggcggcatg aatggtaacc tttctgcact ggtagacttc 1140
gatttgagaa ctccaaccat tttcgataac aagtactacg tcaatctaga agaacaaaaa 1200
gggctaatac agtccgacca ggagctgttt tcatcgccga acgcaaccga tacaatccca 1260
ctagttaggt ccttcgcaaa ctcaactcag acctttttta atgcatttgt ggaagcgatg 1320
gatcgtatgg gtaacattac gcccctaacc gggacccaag gtcaaatccg acttaattgt 1380
agagtcgtta attctaactc actcgagcac caccaccacc accactga 1428
<210> 13
<211> 1271
<212> DNA
<213> 人工序列(artificial sequence)
<400> 13
atgcatcatc atcatcacca tcatcatggt atgacctata aactgattct gaatggcaaa 60
accctgaaag gcgaaaccac caccgaagca gttgatgcag caaccgcaga aaaagttttt 120
aaacagtatg ccaatgacaa cggtgttgat ggtgaatgga cctatgatga tgcaaccaaa 180
acctttaccg ttaccgaaaa accggaagtg attgatgcaa gcgaactgac cccggcagtt 240
accacctata aattagtgat taatggtaaa accctgaagg gcgaaaccac aaccgaagca 300
gtggatgccg caaccgcaga gaaagtgttt aaacagtacg ccaatgataa tggcgttgat 360
ggcgaatgga cctacgatga tgcaacaaaa acctttacag ttaccgaaaa gccggaagtg 420
atcgatgcca gtgaactgac ccctgccgtg accacctata agctggttat taatggtaaa 480
acactgaaag gtgaaaccac cacaaaagca gttgatgcgg aaaccgcaga aaaggccttt 540
aaacagtatg cgaatgataa tggtgttgat ggcgtttgga cctatgacga tgccaccaaa 600
accttcaccg ttaccgagag ttcgggtgga gggtcatggg aaagtcttac ccaaccgtga 660
gcgctgatta ccagaaggcc gttgagaagg cgaagaagag gctcggaggc ttcatcgctg 720
agaagagatg cgctcctcta atgctccgtt tggcatggca ctctgctgga acttacgacg 780
tgcgctcgaa gaccggtggt cccttcggaa ccataagata cccctccgaa ctcgctcacg 840
gcgctaacag cggtcttgac atcgctgtta ggcttttgga gccactcaaa gcggagtttc 900
ctattttgag ctacgccgat ttctaccagt tggctggcgt tgttgccgtt gaggtcaccg 960
gtggacctga agttcccttc catcctggaa gagaggacaa acctgagcta ccaccagagg 1020
gtcgcttgcc cgatgccact aagggttctg accatttgag agatgtgttt ggcaaagcta 1080
tggggcttag tgaccgagat atcgttgctc tgtctggggg tcacactctt ggagctgcgc 1140
acaaggagcg ttctggattc gagggtccct ggacctctaa tcctcttgtt ttcgacaact 1200
catacttcaa ggagttgttg agtggtgaga aagaagcggc cgcactcgag caccaccacc 1260
accaccactg a 1271
<210> 14
<211> 1284
<212> DNA
<213> 人工序列(artificial sequence)
<400> 14
agcatgaaag aagaaacccc ggaaactccg gagacagata gcgaagagga agtaacaatt 60
aaagcaaatc ttatcttcgc gaacggcagc acccaaacgg cggaatttaa aggaaccttc 120
gaaaaagcga cgagcgaagc gtatgcgtat gcggatacct taaaaaaaga taacggcgag 180
tacaccgtgg acgtggcgga caaaggctac accctgaaca tcaagtttgc cgggaaagag 240
aagacacctg aagagcctaa ggaagaagtg accatcaaag ccaatttaat ctacgcagat 300
ggcaaaaccc aaacggctga gtttaaagga acctttgagg aggctactgc ggaggcgtat 360
cgttatgccg atgcgcttaa aaaagataat ggcgagtata ccgtggatgt agccgacaag 420
ggctatacac tcaacattaa attcgcgggc aaggaaaaga cccctgagga gccgaaagaa 480
gaagttacaa tcaaggcgaa cttgatctac gcagacggca aaacgcagac ggctgaattt 540
aaggggacgt tcgaagaggc cacagcggaa gcgtatcgct atgcagatct tctggcgaaa 600
gaaaacggca agtatacagt tgacgtggcg gataaagggt atacgctgaa cataaaattt 660
gcgggcaagg aaaagactcc ggaggagccg aaggaagagg tgactatcaa agcaaactta 720
atttatgcgg atggcaagac gcagacagcg gaatttaagg gcacctttgc cgaagcgaca 780
gccgaggcct atcgttatgc tgacctatta gcaaaagaga acggtaagta taccgcagac 840
ctggaagacg gcggctacac cattaacatt agattcgccg ggaaaaaggt ggatgaaaaa 900
ccggaagaaa aagaacaagt gaccataaaa gaaaacattt attttgaaga tggcaccgtg 960
cagaccgcga cctttaaggg tacctttgcg gaagcgacgg cagaggcgta tcgctatgct 1020
gatctgctga gcaaagaaca tgggaaatat accgcagatc tggaggatgg cggctatact 1080
attaacatac gcttcgcggg cggcggcggc ctccttcagc taccttctga caaggcactt 1140
ttgtctgacc ctgtgttccg ccctcttgtt gagaaatatg catcggacga agatgccttc 1200
ttcgctgatt acgctgaggc tcaccaaaag ctttctgagc ttgggtttgc tgaagccctc 1260
gagcaccacc accaccacca ctga 1284
- 一种定性和定量测定蛋白质的方法及应用
- 一种目标多肽和蛋白质定量测定方法
