基于单分子测序检测核苷酸变异方法与装置
文献发布时间:2023-06-19 12:04:09
技术领域
本发明涉及基因测序领域,具体涉及一种基于单分子测序检测核苷酸变异方法与装置。
背景技术
SNP一般指单核苷酸多态性。单核苷酸多态性主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性。它是人类可遗传的变异中最常见的一种,占所有已知多态性的90%以上。SNP在人类基因组中广泛存在,平均每300个碱基对中就有1个,估计其总数可达300万。这些SNP与人类生活息息相关,比如乙醇脱氢酶和乙醛脱氢酶某些位点的不同基因型决定这个人是否能饮酒,以及产生酒精肝;不同的基因型也会影响基本的发生概率,比如BRCA基因的某些基因型,会使女性患乳腺癌和卵巢癌的概率提高几倍甚至十几倍。基因型也可以用来做亲子鉴定。对于其他物质同样适用,比如宠物狗的血统溯源。SNV可在体细胞突变,是后天产生的(非遗传得来的),很多疾病的发生发展都是由于一些SNV突变引起的。比如肺癌中的EGFR基因L858R的突变。目前,SNP/SNV的检测都是基于高深度测序平台和复杂的核酸测序数据算法突变检测,但普遍存在成本过高,不适于大样本筛查,不适于大样本筛查SNV/SNP与疾病关联性等问题。
由此,开发一种成本低,精准度高的确定核酸样本中单核苷酸变异的方法很有必要。
发明内容
本发明旨在至少在一定程度上解决相关技术中的技术问题之一。
本发明的发明人在研究中发现目前采用的确定核苷酸变异的方法成本较高,算法复杂,不适于在大样本中进行新的单核苷酸变异的筛查,更不适于在大样本中进行单核苷酸变异与各类型疾病的关联性分析,例如乳腺癌或卵巢癌的大样本人群中单核苷酸变异与上述疾病的关联性。
为此,发明人提供了一种确定核酸样本中单核苷酸变异的方法、确定核酸样本中单核苷酸变异的装置、计算机可读介质和电子设备。所提供的方法基于低深度测序技术获取核酸样本,并利用简单的算法对所获取的数据进行分析,最终获取精准度极大降低了检测成本,简化了算法的同时,保证了检测结果的精准度,利用该方法获取单核苷酸变异的精准度可达到百分之八十以上。
本发明的一个目的在于提供一种确定核酸样本中单核苷酸变异的方法及确定核酸样本中单核苷酸变异的装置。
具体而言,本发明提供了如下技术方案:
在本发明的第一方面,本发明提供了一种确定核酸样本中单核苷酸变异的方法。根据本发明的实施例,包括:对所述核酸样本进行双向低深度测序,以获取所述核酸样本的测序数据,所述测序数据包括所述核酸样本的正向测序读段和反向测序读段;基于所述测序数据,所述正向测序读段和所述反向测序读段各自独立地与参考序列进行第一比对处理,以便获得测序读段在所述参考序列的位置,和所述测序读段的重叠区域;基于所述重叠区域,获得所述单核苷酸变异。根据本发明的实施例,利用低深度测序获取双向测序数据,并对所获取的双向测序数据、参考序列进行比对分析,即可获得准确性高达80%以上的单核苷酸变异,同时极大地降低了测序成本和分析成本,利用简化的分析算法和低成本的低深度测序可以达到高精准度的单核苷酸变异的检测。
根据本发明的实施例,以上所述的方法可以进一步包括如下技术特征:
根据本发明的实施例,基于所述正向测序读段或所述反向测序读段的长度,所述重叠区域的核酸数目不小于10bp。根据本发明的实施例,将正向测序数据读段和反向测序数据读段进行匹配,重叠区域为两个测序读段在参考染色体上的重叠区域。在重叠区域中,可以存在两个测序读段在同一位点上出现不同核苷酸类型的情况,也可以重叠区域内的各位点中,两条测序读段的核苷酸各自独立地相同的情况。当重叠区域的核酸数目不小于10bp时,可以作为目标重叠区域。
根据本发明的实施例,基于所述重叠区域,所述正向测序读段和所述反向测序读段中的核苷酸各自独立地相同。根据本发明的实施例,仅保留所述正向测序读段和所述反向测序读段中的核苷酸各自独立地相同的重叠区域及测序读段,并进行单核苷酸变异分析。发明人经过大量的研究发现,在大样本量单核苷酸变异分析中,基于低深度测序所获取的测序数据,对于重叠区域中某位点的核苷酸类型在正向测序读段和反向测序读段中不同的情况,不需要采用更为复杂的算法进行处理,例如常规分析中使用的贝叶斯模型等。发明人经过大量的研究发现,上述位点存在真实的单核苷酸变异的可能性很低,可以直接舍弃,简化运算,但并不会在很大程度上影响检测单核苷酸变异的真实性。
根据本发明的实施例,所述方法进一步包括:基于所述正向测序读段和所述反向测序读段,将所述重叠区域的至少部分与所述参考序列进行比对,以便获得所述单核苷酸变异。在将正向测序读段与反向测序读段分别与参考序列进行比对,能够与参考序列比对上的测序读段部分为重叠区域。将所述正向测序读段和反向测序读段上的重叠区域的核苷酸与所述参考序列比对,可以是部分核苷酸,也可以是重叠部分的全部核苷酸,测序读段和参考序列上的相同位点具有不同核苷酸时,该位点可能具有单核苷酸变异,需要进行进一步筛选判断。
根据本发明的实施例,所述比对是通过如下方式进行的:
确定所述正向测序读段和所述反向测序读段重叠区域的至少部分上的待定单核苷酸变异,所述待定单核苷酸变异是所述重叠区域的至少部分上的核苷酸与所述参考序列上的核苷酸不同的核苷酸;所述待定单核苷酸变异与预知单核苷酸变异不在同一位点,是所述待定单核苷酸变异为目标单核苷酸变异的指示。根据本发明的实施例,将重叠区域与参考序列进行比对,在重叠区域的读段上与参考序列出现不同核苷酸类型的位点为可能的单核苷酸变异位点,重叠区域的读段上该位点的核苷酸类型为变异核苷酸类型。查询并获取在在该位点上是否具有已知核苷酸变异类型,如果其具有已知核苷酸变异类型,并与重叠区域上的核苷酸类型相同,则该位点及核苷酸变异类型被判定为已知单核苷酸变异;如果其具有已知核苷酸变异类型,并与重叠区域上的核苷酸类型不同,则将重叠区域上该位点的核苷酸类型判定为非单核苷酸变异。发明人经过大量的研究发现,在某位点上已经具有已知核苷酸变异的情况下,重叠区域所检出的新的核苷酸变异类型可直接判定为非单核苷酸变异,并不需要使用复杂算法进行进一步分析。其为非单核苷酸变异的概率很高,将其直接判定为非单核苷酸变异并不会在很大程度上影响检测的精准度,反而可以大幅降低算法复杂度。
根据本发明的实施例,所述待定单核苷变异与所述正向测序读段和所述反向测序读段末端距离不小于9bp。发明人经过大量的研究发现,待定核苷酸变异位点在正向测序读段中的位置,距离正向测序读段的末端(上游末端和下游末端)小于9bp时,该核苷酸变异位点不被判定为单核苷酸变异。同理,待定核苷酸变异位点在反向测序读段中的位置,距离反向测序读段的末端(上游末端和下游末端)小于9bp时,该核苷酸变异位点不被判定为单核苷酸变异。依据上述方法确定的单核苷酸变异精准度较高,并且判定直接、简单。
根据本发明的实施例,所述方法进一步包括:将所述待定核苷酸变异所在的所述正向测序读段和所述反向测序读段各自独立地与所述参考序列进行第二比对处理,以便获得第一测序读段和第二测序读段与所述参考序列的重叠区域,所述第二比对处理所使用的比对软件与所述第一比对处理所使用的比对软件不同,所述第二比对处理的所述重叠区域与所述第一比对处理的所述重叠区域相同,是所述待定单核苷酸变异为目标单核苷酸变异的指示。根据本发明的实施例,例如,第一比对使用BWA软件进行,则第二比对可以选用SOAP2、Bowtie、GATK Indel Realignment的任意一个。此外,还可以进行第三比对、第四比对等,各比对使用不同比对软件进行。对比对软件没有特殊限制,可以进行序列比对即可。对使用比对软件的顺序没有特别限制。
根据本发明的实施例,所述重叠区域中,所述正向测序读段与所述反向测序读段在同一位点具有不同核苷酸类型,是所述测序数据不作后续处理的指示。根据本发明的实施例,所述不做后续处理为舍弃该正向测序读段和该反向测序读段,不以上述两个读段的测序结果为依据进行后续的单核苷酸变异分析。
在本发明的第二方面,本发明提出了一种确定核酸样本中单核苷酸变异的装置。根据本发明的实施例,所述装置包括:测序数据模块,所述测序数据获取模块被配置为对所述核酸样本进行双向低深度测序,以获取所述核酸样本的测序数据,所述测序数据包括所述核酸样本的正向测序读段和反向测序读段;重叠区域获取模块,所述重叠区域获取模块被配置为基于所述测序数据,所述正向测序读段和所述反向测序读段各自独立地与参考序列进行第一比对处理,以便获得所述测序读段在所述参考序列上的位置和所述测序读段的重叠区域;单核苷酸变异获取模块,所述单核苷酸变异获取模块被配置为基于所述重叠区域,获得所述单核苷酸变异。根据本发明的实施例,所述装置根据上述方法进行装置各部分设置,采用该装置可以快速准确定核酸样本中的单核苷酸变异。
根据本发明的实施例,所述装置可以进一步包括如下技术特征:
根据本发明的实施例,基于所述正向测序读段或所述反向测序读段的长度,所述重叠区域的核酸数目不小于10bp。
根据本发明的实施例,基于所述重叠区域,所述正向测序读段和所述反向测序读段中的核苷酸各自独立地相同。
根据本发明的实施例,所述装置进一步包括:参考序列比对单元,所述参考序列比对单元被配置为基于所述正向测序读段和所述反向测序读段,将所述重叠区域的至少部分与所述参考序列进行比对,以便获得所述单核苷酸变异。
根据本发明的实施例,所述装置进一步包括:待定单核苷酸变异确定单元,所述待定核苷酸变异确定单元被配置为确定所述正向测序读段和所述反向测序读段重叠区域的至少部分上的待定单核苷酸变异,所述待定单核苷酸变异是所述重叠区域的至少部分上的核苷酸与所述参考序列上的核苷酸不同的核苷酸;所述待定单核苷酸变异与预知单核苷酸变异不在同一位点,是所述待定单核苷酸变异为目标单核苷酸变异的指示。
根据本发明的实施例,所述待定单核苷变异与所述正向测序读段和所述反向测序读段末端距离不小于9bp。
根据本发明的实施例,所述装置进一步包括:第二比对处理单元,所述第二比对处理单元被配置为将所述待定核苷酸变异所在的所述正向测序读段和所述反向测序读段各自独立地与所述参考序列进行第二比对处理,以便获得第一测序读段和第二测序读段与所述参考序列的重叠区域,所述第二比对处理所使用的比对软件与所述第一比对处理所使用的比对软件不同,所述第二比对处理的所述重叠区域与所述第一比对处理的所述重叠区域相同,是所述待定单核苷酸变异为目标单核苷酸变异的指示。
根据本发明的实施例,所述重叠区域中,所述正向测序读段与所述反向测序读段在同一位点具有不同核苷酸类型,是所述测序数据不作后续处理的指示。
在本发明的第三方面,本发明提出了一种确定待测样本基因型的方法。根据本发明的实施例,所述方法包括:获取所述待测样本的单核苷酸变异,所述单核苷酸变异是通过上述方法或上述装置获得的;利用所述单核苷酸变异,获取所述待检测样本的基因型。根据本发明的实施例,根据待检测样本的单核苷酸变异可以对待检测样本的基因型进行检测,利用该方法确定的基因型,可以用于1)全基因组关联研究:发现和某些疾病和表型(比如升高)相关的,新的基因型和位点;2)基于已有的发现,进行疾病风险,表型预估,比如:乙醇脱氢酶和乙醛脱氢酶某些位点的不同基因型决定这个人是否能饮酒,以及是否容易患酒精肝;BRCA基因的某些基因型,会使女性患乳腺癌和卵巢癌的概率提高几倍甚至十几倍;3)亲子鉴定等;4)对于其他物种同样适用,比如宠物狗的血统溯源。
在本发明的第四方面,本发明提出了一种确定待测样本基因型的装置。根据本发明的实施例,所述装置包括:单核苷酸变异获取设备,所述设备包括上述本发明第二方面所提出的装置;和基因型获取设备,所述设备被配置为利用所获得的单核苷酸变异获取所述待测样本的基因型。
在本发明的第五方面,本发明提供了一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现本发明第一方面或本发明第三方面所述方法的步骤。
在本发明的第六方面,本发明提供了一种电子设备,包括:本发明第五方面所述的计算机可读存储介质;以及一个或者多个处理器,用于执行所述计算机可读存储介质中的程序。
本发明的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
附图说明
本发明的上述和/或附加的方面和优点从结合下面附图对实施例的描述中将变得明显和容易理解,其中:
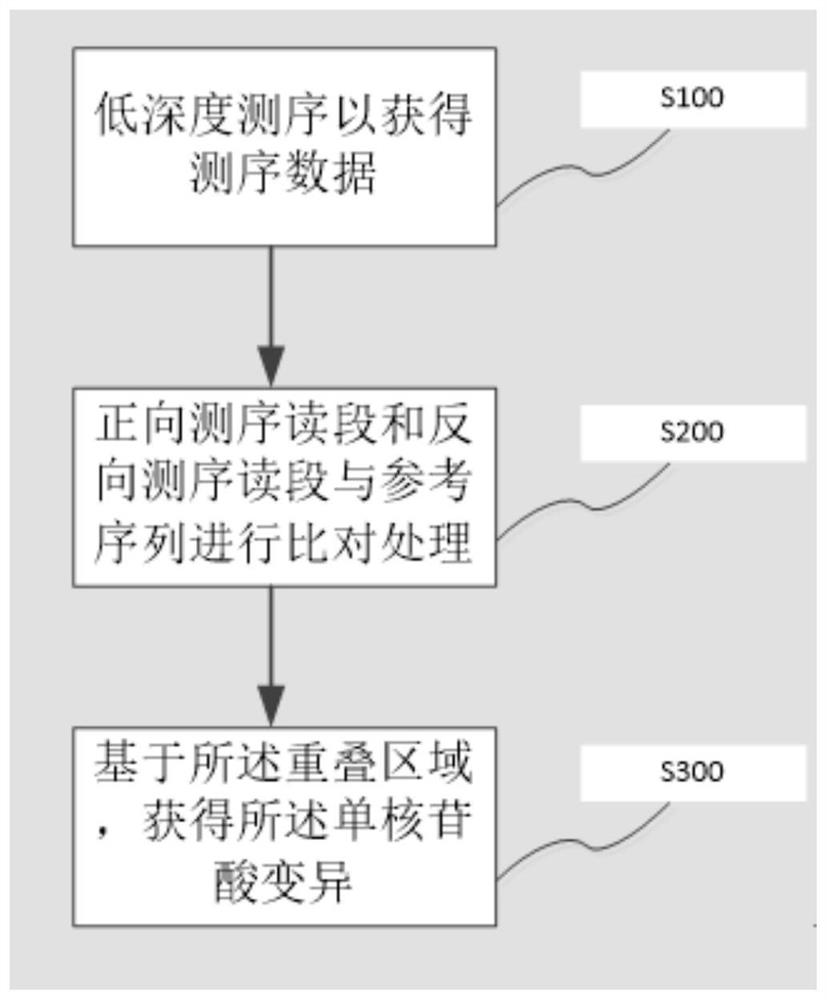
图1是根据本发明实施例的确定核酸样本中单核苷酸变异的方法示意图;
图2是根据本发明实施例的确定核酸样本中单核苷酸变异的装置示意图;
图3是根据本发明实施例的当核酸的正义链和反义链被测序时的不同场景的示意图,其中反向互补序列与正义链应为反向互补,为使附图更清楚,将反向互补序列进行了反向互补转换,使其与正向链序列相同。
具体实施方式
下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,旨在用于解释本发明,而不能理解为对本发明的限制。
此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括至少一个该特征。在本发明的描述中,“多个”的含义是至少两个,例如两个,三个等,除非另有明确具体的限定。
在本发明中,除非另有明确的规定和限定,术语“安装”、“相连”、“连接”、“固定”等术语应做广义理解,例如,可以是固定连接,也可以是可拆卸连接,或成一体;可以是机械连接,也可以是电连接;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通或两个元件的相互作用关系,除非另有明确的限定。对于本领域的普通技术人员而言,可以根据具体情况理解上述术语在本发明中的具体含义。
需要注意的是,本文中,“低深度测序”是指测序深度在小于5X。
需要注意的是,本文中,“高深度测序”是指测序深度大于30X。
需要注意的是,本文中,核酸样本可以取自组织、细胞、器官、血液、体液等。
为了降低单核苷酸变异检测的成本及简化单核苷酸变异的算法,能够实现针对数量较大的种群进行单核苷酸变异的检测、筛选以及单核苷酸变异与疾病的相关性分析,发明人创造性地使用低深度测序的方式进行单核苷酸变异的检测与确定,有利于新的单核苷酸变异的发现与利用。
由此,在本发明的一个方面,本发明提出了一种确定核酸样本中单核苷酸变异的方法。根据本发明的实施例,参考图1,包括:S100,对所述核酸样本进行双向低深度测序,以获取所述核酸样本的测序数据,所述测序数据包括所述核酸样本的正向测序读段和反向测序读段;S200,基于所述测序数据,所述正向测序读段和所述反向测序读段各自独立地与参考序列进行第一比对处理,以便获得测序读段和所述参考序列的重叠区域;S300,基于所述重叠区域,获得所述单核苷酸变异。根据本发明的实施例,利用低深度测序获取双向测序数据,并对所获取的双向测序数据、参考序列进行比对分析,即可获得精准度高达80%以上的单核苷酸变异,同时极大地降低了测序成本和分析成本,利用简化的分析算法和低成本的第深度测序可以达到高精准度的单核苷酸变异的检测。
根据本发明的实施例,基于所述正向测序读段或所述反向测序读段的长度,所述重叠区域的核酸数目不小于10bp。根据本发明的实施例,将正向测序数据读段和反向测序数据读段进行匹配,重叠区域为两个测序读段在参考染色体上的重叠区域。在重叠区域中,可以存在两个测序读段在同一位点上出现不同核苷酸类型的情况,也可以重叠区域内的各位点中,两条测序读段的核苷酸各自独立地相同的情况。当重叠区域的核酸数目不小于10bp时,可以作为目标重叠区域。
根据本发明具体的实施例,一个位点存在SNV突变,参考碱基是A,已知突变是C。根据该位点在正向测序读段和反向测序读段中的碱基类型,读段可分为五个不同类别:
a)来自一个片段的读段1和读段2可以是一致的,并且与参考碱基一致,均为A。
b)来自一个片段的读段1和读段2可以是一致的,并且与已知突变一致,均为C。
c)读段1和读段2也可以是一致的,但是与参考碱基或已知突变不一致,均为T。
d)读段1和读段2在相同位点具有不同的碱基,一个为T,一个为A。
e)读段1和读段2在相同位置一个与参考碱基一致,为A,一个与参考碱基不一致,为G。
针对上述五种类型,c、d、e三种类型不做后续处理,即,在所述重叠区域中,所述正向测序读段与所述反向测序读段在同一位点具有不同核苷酸类型,是所述测序数据不作后续处理的指示。根据本发明的实施例,所述不做后续处理为舍弃该正向测序读段和该反向测序读段,不以上述两个读段的测序结果为依据进行后续的单核苷酸变异分析。
根据本发明的实施例,基于所述重叠区域,所述正向测序读段和所述反向测序读段中的核苷酸各自独立地相同。根据本发明的实施例,仅保留所述正向测序读段和所述反向测序读段中的核苷酸各自独立地相同的重叠区域及测序读段,并进行单核苷酸变异分析。发明人经过大量的研究发现,在大样本量单核苷酸变异分析中,基于低深度测序所获取的测序数据,对于重叠区域中某位点的核苷酸类型在正向测序读段和反向测序读段中不同的情况,不需要采用更为复杂的算法进行处理,例如常规分析中使用的贝叶斯模型等。发明人经过大量的研究发现,上述位点存在真实的单核苷酸变异的可能性很低,可以直接舍弃,简化运算,但并不会在很大程度上影响检测单核苷酸变异的真实性。
根据本发明的实施例,所述方法进一步包括:
基于所述正向测序读段和所述反向测序读段,将所述重叠区域的至少部分与所述参考序列进行比对,以便获得所述单核苷酸变异。
根据本发明的实施例,所述比对是通过如下方式进行的:
确定所述正向测序读段和所述反向测序读段重叠区域的至少部分上的待定单核苷酸变异,所述待定单核苷酸变异是所述重叠区域的至少部分上的核苷酸与所述参考序列上的核苷酸不同的核苷酸;所述待定单核苷酸变异与预知单核苷酸变异不在同一位点,是所述待定单核苷酸变异为目标单核苷酸变异的指示。根据本发明的实施例,将重叠区域与参考序列进行比对,在重叠区域的读段上与参考序列出现不同核苷酸类型的位点为可能的单核苷酸变异位点,重叠区域的读段上该位点的核苷酸类型为变异核苷酸类型。查询并获取在在该位点上是否具有已知核苷酸变异类型,如果其具有已知核苷酸变异类型,并与重叠区域上的核苷酸类型相同,则该位点及核苷酸变异类型被判定为已知单核苷酸变异;如果其具有已知核苷酸变异类型,并与重叠区域上的核苷酸类型不同,则将重叠区域上该位点的核苷酸类型判定为非单核苷酸变异。发明人经过大量的研究发现,在某位点上已经具有已知核苷酸变异的情况下,重叠区域所检出的新的核苷酸变异类型可直接判定为非单核苷酸变异,并不需要使用复杂算法进行进一步分析。其为非单核苷酸变异的概率很高,将其直接判定为非单核苷酸变异并不会在很大程度上影响检测的精准度,反而可以大幅降低算法复杂度。
根据本发明的实施例,所述待定单核苷变异与所述正向测序读段和所述反向测序读段末端距离不小于9bp。发明人经过大量的研究发现,待定核苷酸变异位点在正向测序读段中的位置,距离正向测序读段的末端(上游末端和下游末端)小于9bp时,该核苷酸变异位点不被判定为单核苷酸变异。同理,待定核苷酸变异位点在反向测序读段中的位置,距离反向测序读段的末端(上游末端和下游末端)小于9bp时,该核苷酸变异位点不被判定为单核苷酸变异。依据上述方法确定的单核苷酸变异精准度较高,并且判定直接、简单。
根据本发明的实施例,基于上述方法获得单核苷酸变异后,获得的两条reads同时支持一个突变的单核苷酸变异。同时得到支持单核苷酸变异的读段reads,重新提取测序读段序列,转换成fastq格式。使用其他比对软件(bowtie)和GATK重比对软件重新比对到人的参考基因组上,过滤掉不同比对软件,比对起始位置存在差异的单核苷突变;同时使用下面的方法进一步过滤单核苷酸变异:
1)单核苷酸变异的位置,在读段的两端(9bp内);
2)同一位点存在已知突变和新的突变;
4)单核苷酸变异位置的碱基质量任意一条小于30;
5)过滤掉突变位于基因组上的简单重复区域,black region。
根据本发明的实施例,在进行测序数据分析之前,对获取的测序数据进行质量控制,对下列情形的读段不做保留:
a)任意一条读段比对质量值<30,
b)重复的reads,以及supplementary alignment reads;
c)比对结果包含插入和缺失(InDel),reads的经过5’或3’端剪接(softclipping/hard clipping)比对上,以及reads中间包含gap(skipped region from thereference),比对结果中包含的碱基错配数>2。
在本发明的第二方面,本发明提出了一种确定核酸样本中单核苷酸变异的装置。根据本发明的实施例,参考图2,所述装置包括:测序数据模块100,所述测序数据获取模块被配置为对所述核酸样本进行双向低深度测序,以获取所述核酸样本的测序数据,所述测序数据包括所述核酸样本的正向测序读段和反向测序读段;重叠区域获取模块200,所述重叠区域获取模块200与所述测序数据模块100相连,所述重叠区域获取模块被配置为基于所述测序数据,所述正向测序读段和所述反向测序读段各自独立地与参考序列进行第一比对处理,以便获得所述测序读段和所述参考序列的重叠区域;单核苷酸变异获取模块300,所述单核苷酸变异获取模块300与所述重叠区域获取模块200相连,所述单核苷酸变异获取模块被配置为基于所述重叠区域,获得所述单核苷酸变异。根据本发明的实施例,所述装置根据上述方法进行装置各部分设置,采用该装置可以快速准确定确定核酸样本中的单核苷酸变异。
根据本发明的实施例,基于所述正向测序读段或所述反向测序读段的长度,所述重叠区域的核酸数目不小于10bp。
根据本发明的实施例,基于所述重叠区域,所述正向测序读段和所述反向测序读段中的核苷酸各自独立地相同。
根据本发明的实施例,所述装置进一步包括:参考序列比对单元,所述参考序列比对单元被配置为基于所述正向测序读段和所述反向测序读段,将所述重叠区域的至少部分与所述参考序列进行比对,以便获得所述单核苷酸变异。
根据本发明的实施例,所述装置进一步包括:
待定单核苷酸变异确定单元,所述待定核苷酸变异确定单元被配置为确定所述正向测序读段和所述反向测序读段重叠区域的至少部分上的待定单核苷酸变异,所述待定单核苷酸变异是所述重叠区域的至少部分上的核苷酸与所述参考序列上的核苷酸不同的核苷酸;
所述待定单核苷酸变异与预知单核苷酸变异不在同一位点,是所述待定单核苷酸变异为目标单核苷酸变异的指示。根据本发明的实施例,所述待定单核苷变异与所述正向测序读段和所述反向测序读段末端距离不小于9bp。
根据本发明的实施例,所述装置进一步包括:第二比对处理单元,所述第二比对处理单元被配置为将所述待定核苷酸变异所在的所述正向测序读段和所述反向测序读段各自独立地与所述参考序列进行第二比对处理,以便获得第一测序读段和第二测序读段与所述参考序列的重叠区域,所述第二比对处理所使用的比对软件与所述第一比对处理所使用的比对软件不同,所述第二比对处理的所述重叠区域与所述第一比对处理的所述重叠区域相同,是所述待定单核苷酸变异为目标单核苷酸变异的指示。
根据本发明的实施例,所述重叠区域中,所述正向测序读段与所述反向测序读段在同一位点具有不同核苷酸类型,是所述测序数据不作后续处理的指示。
在本发明的第三方面,本发明提出了一种确定待测样本基因型的方法。根据本发明的实施例,所述方法包括:获取所述待测样本的单核苷酸变异,所述单核苷酸变异是通过上述方法或上述装置获得的;利用所述单核苷酸变异,获取所述待检测样本的基因型。根据本发明的实施例,根据待检测样本的单核苷酸变异可以对待检测样本的基因型进行检测,利用该方法确定的基因型,可以用于1)全基因组关联研究:发现和某些疾病和表型(比如升高)相关的,新的基因型和位点;2)基于已有的发现,进行疾病风险,表型预估:BRCA基因的某些基因型,会使女性患乳腺癌和卵巢癌的概率提高几倍甚至十几倍;3)亲子鉴定等;4)对于其他物种同样适用,比如宠物狗的血统溯源。
在本发明的第四方面,本发明提出了一种确定待测样本基因型的装置。根据本发明的实施例,所述装置包括:单核苷酸变异获取设备,所述设备包括上述本发明第二方面所提出的装置;和基因型获取设备,所述设备被配置为利用所获得的单核苷酸变异获取所述待测样本的基因型。
在本发明的第五方面,本发明提供了一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现本发明第一方面或本发明第三方面所述方法的步骤。
在本发明的第六方面,本发明提供了一种电子设备,包括:本发明第五方面所述的计算机可读存储介质;以及一个或者多个处理器,用于执行所述计算机可读存储介质中的程序。
下面参考具体实施例,对本发明进行描述,需要说明的是,这些实施例仅仅是描述性的,而不以任何方式限制本发明。
实施例1:
样本的处理和建库测序。样本可以是组织,血浆,唾液等任何生物样本,使用常规试剂盒进行DNA提取和建库,测序。下面仅给出的是一个基于血液样本的提取,建库,测序实施例,
1.血浆分离
a)准备好实验所需的仪器、试剂、耗材,高速冷冻离心机应提前预冷至4℃。
b)如果外周血样本是用EDTA抗凝管采集的,抽血之后立马放进4℃冰箱,并在2小时内进行血浆分离。如果外周血样本是用Streck管等游离核酸保存管采集的,则可在常温放置,并在采血管说明书规定的时间内进行血浆分离分离。
c)记录样本信息,将采血管配平,将高速冷冻离心机换成水平转子,并设定参数:温度4℃,离心力1600g,时间10min。将采血管配平之后放置在离心机中,进行离心。
d)离心完成之后,将采血管放置在生物安全柜的离心管架上。将离心后采血管中的上清收集至新的15mL离心管中,在管壁标记样本编号以及操作时间。注意在收集上清时需要仔细操作,避免吸入白细胞。
e)将高速冷冻离心机换成角转子,并设定参数:温度4℃,离心力16000g,时间10min。
将装有上清的15mL离心管配平之后放置在离心机中,进行离心。
f)离心完成之后,将装有上清的15mL离心管放置在生物安全柜的离心管架上。将离心后离心管中的上清收集至新的15mL离心管中。并取出500ul上清保存在1.5mL离心管中,并用于后续肿瘤标志物检测。注意在收集上清时需要仔细操作,避免吸入沉淀。
这一步的目的是去除血浆当中的细胞碎片等杂质。
g)将血浆以及血细胞放置于-80℃冰箱保存,备用。
h)实验完成后,将所有物品归位,并清洁实验台面,将生物安全柜紫外灯打开,照射30min后关闭。记录详细的实验记录。
2.cfDNA提取
i)准备好实验所需的仪器、试剂、耗材。打开水浴锅,并调节温度至60℃。打开金属浴,并调节温度至56℃。确认试剂盒有效期,buffer ACB是否加有合适量的异丙醇,bufferACW1以及buffer ACW1是否加有合适量的无水乙醇。
j)记录样本编号等信息。
k)若是分离的新鲜血浆,则直接进行cfDNA提取。若血浆冻存在-80℃条件下,需将血浆样本解冻后,在16,000x g[固定角转头]的离心力以及4℃的温度条件下离心5min以去除冷冻沉淀。
l)按照表1配置所需量的ACL混合液。
表1:处理4ml样本所需的Buffel ACL以及carrier RNA(溶解于Buffer AVE)体积用量
m)转移400μl Proteinase K至装有4ml血浆的50ml离心管中。间断涡旋30s以充分混匀。
n)加入3.2ml的Buffer ACL(含有1.0μg carrier RNA)。剧烈涡旋混匀15秒。确保离心管经剧烈涡旋,以保证样本和Buffer ACL的重复混匀,从而实现高效的裂解。
o)注意:此步完成后请不要中断实验并立即进行下步的裂解孵育步骤。
p)将离心管接着60℃水浴30分钟。
q)向上述反应液中加入7.2ml的Buffer ACB。盖上管盖,间断涡旋15s以充分混匀。
r)将含有Buffer ACB的裂解液至于冰上孵育或冷藏孵育5min。
s)组装抽滤装置:把VacValve插在24孔底上,再把VacConnectors插入VacValve中,再将QIAamp Mini硅胶膜柱连接到VacConnectors上,最后把20ml扩容管插入到硅胶膜柱上。确保扩容管插入紧实以防止样本泄露。注意:将2ml收集管留下至后续空转时才使用。并在硅胶膜柱上做好样本编号的标记。VacValve可调节流速,VacConnectors可以防止污染,QIAamp Mini硅胶膜柱用于吸附DNA,扩容管用于装大体积血浆。
t)把孵育完的混合物转移至扩容管中,打开真空泵,待离心柱中的裂解液完全抽干后,关闭真空泵,打开24孔底座一侧的排气阀将压力释放到0兆帕。小心地将扩容管拆下并丢弃。
u)向QIAamp Mini硅胶膜柱中加入600μl的Buffer ACW1,关闭排气阀,并打开真空泵,进行抽滤液体。当离心柱中Buffer ACW1被抽干后,关闭真空泵,打开24孔底座一侧的排气阀将压力释放到0兆帕。
v)向QIAamp Mini硅胶膜柱中加入750μl的Buffer ACW2,关闭排气阀,并打开真空泵,进行抽滤液体。当离心柱中Buffer ACW2被抽干后,关闭真空泵,打开24孔底座一侧的排气阀将压力释放到0兆帕。
w)向QIAamp Mini硅胶膜柱中加入750μl的无水乙醇溶液,关闭排气阀,并打开真空泵,进行抽滤液体。当离心柱中无水乙醇被抽干后,关闭真空泵,打开24孔底座一侧的排气阀将压力释放到0兆帕。关闭真空泵电源。
x)盖上QIAamp Mini硅胶膜柱并从真空支管上取下后放置到干净的2ml收集管中,将VacConnector丢弃。收集管在全速条件(20,000x g;14,000rpm)下离心3min。
y)将QIAamp Mini硅胶膜柱放置到新的2ml收集管中,开盖并置于56℃条件下的金属浴上干燥10min至硅胶膜彻底干燥。
z)将QIAamp Mini硅胶膜柱取出后放置到干净的1.5ml洗脱管(试剂盒自带)中,并将使用过的2ml的收集管丢弃。
aa)向QIAamp Mini硅胶膜柱中硅胶膜的中央小心加入Nuclease-free water进行洗脱:20~60ul)。盖上管盖后在室温孵育3min。
bb)将洗脱管置于小型离心机中全速(20,000x g;14,000rpm)离心1min来洗脱cfDNA。
cc)质量标准与评估
Qubit HS定量:取1μL cfDNA利用Qubit 4.0(Thermo Fisher Scientific,Q33226)结合Qubit dsDNA HS Assay Kits(Thermo Fisher Scientific,Q32854)进行定量测定,记录浓度ng/uL。
Agilent 2100检测:取1μL cfDNA利用Agilent 2100生物分析仪(Agilent,G29939BA)结合Agilent High Sensitivity DNA Kit(Agilent,5067-4626)进行cfDNA峰图检测,测定cfDNA片段分布。
dd)实验完成后,将所有物品归位,并清洁实验台面,将生物安全柜紫外灯打开,照射30min后关闭。记录详细的实验记录。
3.cfDNA文库构建
a)建库前准备
i.从4℃冰箱取出纯化DNA所用的磁珠(AMPureXP beads,Beckman),室温平衡30min再使用。
ii.从-20℃冰箱内取出End Repair&A-Tailing Buffer和End Repair&A-TailingBuffer enzyme mix试剂,置于冰盒上解冻,待用。
iii.将要建库的cfDNA样本名称、取样日期、DNA浓度记录在实验记录本上,并编写好编号,方便之后操作。
iv.取相应数量的200μL PCR管,写好编号(管盖和管壁都标注编号)。
v.按cfDNA建库起始量10ng≤X≤100ng标准计算每个cfDNA样本所需要的DNA溶液体积,记录在实验记录本上,并取相应的体积置于对应的200μL PCR管内。
vi.向每个200μL PCR管内加入适量的Nuclease-Free water,使终体积达到50μL。
vii.注:在建库过程中配制所有反应体系应遵循如下规则:若样本少于四个,不需配制混合体系,每个样本独立加入反应体系中的每种成分溶液;若超过四个样本,则将反应体系中每个成分溶液按所需用量的105%配制混合体系,然后逐一加入各个样本中。
b)末端修复&加A
i.按照表2所示,配制末端修复&加A反应体系。
表2:
ii.向每个200μl PCR管内加入10μL上述末端修复反应体系,混匀后低速离心,设定PCR仪,程序如下表3。
表3:
iii.将反应体系从PCR仪中取出,放置在小黄板上,并进行接头连接反应。
c)接头连接反应体系
i.按照表4所示,配制接头连接反应体系。
表4:
ii.向每个反应管中加入45μL上述反应体系,温和混合均匀,低速离心。
iii.根据input DNA量加入适量的adapter,具体DNA:adapter如下表5,每个反应管各加入5μL adapter。另外根据测序要求,每个样本加入不同的adapter,使得同一个lane中不会出现两个样本使用同一个adapter的情况,记录好每个样本使用的adapter信息。
表5:
iv.混合均匀,并放入PCR仪中,设定温度20℃,反应15min。
d)DNA纯化
i.配制80%乙醇(例如配置50mL 80%乙醇:40mL无水乙醇+10mL Nuclease-freeWater),80%乙醇应现用现配。
ii.准备相应数量的1.5mL样本管,并做好相应的标记。
iii.将事先在室温平衡好的磁珠充分震荡混匀,并向每个管中分装88μL。
iv.将上述加了adapter的DNA与磁珠混匀。室温静置10min。
v.将1.5mL样本管置于磁力架上,进行磁珠吸附,直至溶液澄清。
vi.小心移除上清液,再加入200μL 80%乙醇,将样本管水平旋转360度,静置30s后弃上清液。(此过程,离心管一直保持在磁力架上。)
vii.重复上述步骤一次。
viii.应将所有残留的酒精溶液移除。打开管盖,常温下干燥磁珠,挥发乙醇,以免过多乙醇影响后续反应体系中酶的效果。注意:不可过分干燥磁珠,否则会导致DNA不容易从磁珠上洗脱下来,造成产量损失。当磁珠表面不再有光泽时即为干燥完成。
ix.每个样本管内加入21μL Nuclease-Free water,重悬浮磁珠,充分混匀后室温静置5min。
x.准备一批新的200μL PCR管,管盖管壁标注对应的样本编号。
xi.将样本管置于磁力架,进行磁珠吸附,直至溶液澄清后,将上清液转移至对应编号的PCR管中,作为PCR实验的模板。
e)文库扩增
i.按照表6所示,配制文库扩增反应体系。
表6:
ii.每个0.2mL样本管内加入30μL Pre-PCR扩增反应体系,温和混匀并低速离心,放入PCR仪中反应。
iii.将PCR仪设定如下程序,PCR cycle应根据input DNA量适当调整,见表7。
表7:
iv.循环数选择参考表格8。
表8:
v.Pre-PCR反应结束后,开始进行文库纯化。
f)文库纯化
i.准备相应数量的1.5mL样本管,并做好相应的标记。
ii.将事先在室温平衡好的磁珠充分震荡混匀,并向每个管中分装50μL。
iii.将上述加了adapter的DNA与磁珠混匀。室温静置10min。
iv.将1.5mL样本管置于磁力架上,进行磁珠吸附,直至溶液澄清。
v.小心移除上清液,再加入200μL 80%乙醇,将样本管水平旋转360度,静置30s后弃上清液。(此过程,离心管一直保持在磁力架上。)
vi.重复上述步骤一次。
vii.应将所有残留的酒精溶液移除。打开管盖,常温下干燥磁珠,挥发乙醇,以免过多乙醇影响后续反应体系中酶的效果。注意:不可过分干燥磁珠,否则会导致DNA不容易从磁珠上洗脱下来,造成产量损失。当磁珠表面不再有光泽时即为干燥完成。
viii.每个样本管内加入35μL Nuclease-Free water,重悬浮磁珠,充分混匀后室温静置5min。
ix.准备一批新的离心管,管盖上标注所属项目,取样日期,样本名称;管壁上标注接头信息,建库日期,浓度。
x.将1.5mL样本管置于磁力架上,进行磁珠吸附,直至溶液澄清后,将上清液转移至对应的新的写有样本信息的1.5mL离心管。
xi.取1ul文库使用Qubit进行定量,1ul文库使用Agilent 2100测定文库片段大小,并记录相应信息。
xii.样本放入相对应项目的冻存盒内,置于-20℃保存。
xiii.实验完成后,将所有物品归位,并清洁实验台面,将超净工作台紫外灯打开,照射30min后关闭。记录详细的实验信息。
4.文库pooling
g)准备好实验所需的仪器、试剂、耗材。
h)按照测定的浓度以及所需要测定的数据量,计算各个样本需要pooling体积。
i)取一个新的1.5ml离心管,做好标记。将各个样本按照计算的pooling体积进行pooling在同一个1.5ml离心管中。
j)混合均匀之后,测定浓度,并记录信息。
k)实验完成后,将所有物品归位,并清洁实验台面。
5.上机测序
将上述pooling好的文库用Tris-HCl以及NaOH进行稀释变性,然后进行上机测序。
实施例2:
(1)过滤掉低质量等reads后,使用比对软件(bwa)将这些测序reads比对到人的参考基因组上(hg19)。
(2)过滤比对结果,首先挑选正常配对,比对上的reads,在进一步过滤掉掉:
a)任意一条reads比对质量值<30,
b)重复的reads,以及supplementary alignment reads;
c)比对结果包含插入和缺失(InDel),reads的经过5’或3’端剪接(softclipping/hard clipping)比对上,以及reads中间包含gap(skipped region from thereference),比对结果中包含的碱基错配数>2。
(3)同时取出配对reads的比对结果;对于配对reads重叠区域,如图1:readsA_1与readsA_2的重叠区域,按照下面的方法进行统计,寻找单核苷酸变异;
假设一个位点存在SNV突变,参考碱基是A,突变是C。来自不同片段的读段可分为五个不同类别(图3):
a)来自一个片段的读段1和读段2可以是一致的,并且与参考序列(“Ref_base_PE”)一致。
b)来自一个片段的读段1和读段2可以是一致的,并且与突变(“Alt_base_PE”)一致。
c)读段1和读段2也可以是一致的,但是与参考碱基或已知突变(“Other_PE”)不一致的碱基。
d)读段1和读段2在相同位点具有不同的核苷酸(“Diff_base_PE”)。
e)读段1和读段2在相同位置一个与参考一样,一个与参考不一样(“Diff_base_SE”)
(4)基于上面的算法,获得的两条reads同时支持一个突变的单核苷酸变异。同时得到支持单核苷酸变异的读段reads,重新提取测序读段序列,转换成fastq格式。使用其他比对软件(bowtie)和GATK重比对软件重新比对到人的参考基因组上,过滤掉不同比对软件,比对起始位置存在差异的单核苷突变;同时使用下面的方法进一步过滤单核苷酸变异:
1)单核苷酸变异的位置,在读段的两端(9bp内);
2)同一位点存在Alt_base_PE和Other_PE的突变;
4)单核苷酸变异位置的碱基质量任意一条小于30;
5)过滤掉突变位于基因组上的简单重复区域,black region。
(3)统计过滤比对结果后的测序数据量,读段1和读段2的重叠的总的测序数据量;实施例样本1的结果如表9。
表9:
(4)实施例样本1同时进行了高深度测序(~50X),将该样本使用Varscan2检测到的变异作为参考集,计算单核苷酸变异检测准确率:86.12%。
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必须针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。
尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。
- 基于单分子测序检测核苷酸变异方法与装置
- 一种基于甲基化测序数据进行变异检测的方法及装置
