基于转录组学和影像组学的生存预测方法及装置
文献发布时间:2024-04-18 19:58:21
技术领域
本发明涉及医学图像处理的技术领域,尤其涉及一种基于转录组学和影像组学的生存预测方法,以及基于转录组学和影像组学的生存预测装置。
背景技术
生存预测是医学领域中重要的研究方向之一,其目的是对患者的生存时间进行预测。传统的生存预测方法主要基于临床病历和生化指标等临床数据,但这些数据往往无法全面反映疾病的复杂性和异质性。转录组学是研究全基因组范围内RNA表达总和,能够全面反映基因表达的差异性和多样性。影像组学则是将医学影像数据与计算机科学相结合的交叉学科,可以通过分析影像数据中的各种特征,来揭示疾病的生物学特征,从而提高生存预测的准确性。基于转录组学和影像组学的生存预测方法和装置,能够更全面、更准确地预测患者的生存时间,为临床医学提供更精准的诊断和治疗指导。
发明内容
为克服现有技术的缺陷,本发明要解决的技术问题是提供了一种基于转录组学和影像组学的生存预测方法,其能更准确、更全面地预测患者的生存时间,且可以应用于多种类型的疾病中,具有很高的实用性和应用价值。尤其适用于胶质母细胞瘤(GBM)等恶性肿瘤。
本发明的技术方案是:这种基于转录组学和影像组学的生存预测方法,其包括以下步骤:
(1)获取肿瘤患者和部分正常被试的表达谱数据,通过差异表达分析,获得差异表达的mRNA和lncRNA;
(2)从影像数据中分割出各患者的肿瘤区域,提取影像组学特征;
(3)对差异表达的mRNA、lncRNA和影像组学特征进行单因素Cox回归,选取P值小于0.05的因素;然后进一步通过LASSO回归筛选与生存显著相关的特征,构建转录组学和影像组学预后标签;
(4)整合转录组学标签、影像组学标签和临床数据建立多组学预测模型,绘制列线图及生存曲线;
(5)与临床因素预测模型进行区分度、校准度及临床有效性方面的对比,评估多组学预测模型的性能。
本发明通过对研究对象获得转录组学数据和影像组学数据;然后进一步通过LASSO回归筛选与生存显著相关的特征,构建转录组学和影像组学预后标签;整合转录组学标签、影像组学标签和临床数据建立多组学预测模型,与临床因素预测模型进行区分度、校准度及临床有效性方面的对比,评估多组学预测模型的性能。非常适合于GBM等恶性肿瘤生存预测方面的研究。
还提供了一种基于转录组学和影像组学的生存预测装置,其包括:
转录组学数据处理模块,其配置来处理被试的转录组学数据,使用R语言中的limma包对数据进行标准化后,然后通过对患者与正常组的mRNA和lncRNA表达数据分别进行差异表达分析,筛选差异表达的mRNA和lncRNA,筛选标准均为校正后P值(FDR)≤0.01且|FoldChange|≥2;
影像组学数据处理模块,包括图像分割及影像组学特征选择;
LASSO数据处理模块,将转录组学数据处理模块和影像组学数据处理模块得到的数据进行处理,得到影像组学和转录组学预后标签;
预测模型建立模块,构建两组不同的预测模型,一是基于临床候选预后危险因素构建的临床因素预测模型,二是基于临床候选预后危险因素、影像组学标签、转录组学标签的多组学预测模型,对构建的临床预测模型,在训练集中通过反复抽取,采用10折交叉验证进行内部验证,然后通过验证数据集进行外部验证。
附图说明
图1示出了差异mRNA和差异lncRNA的火山图。
图2 示出了LASSO回归筛选转录组学及影像组学特征的系数收敛图。
图3示出了高低风险组的生存曲线和多组学列线图。
图4示出了训练集和验证集不同预测模型的ROC曲线、校准曲线和DCA决策曲线。
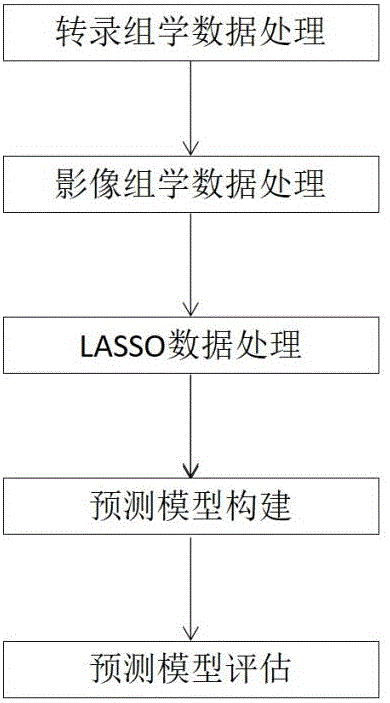
图5 示出了根据本发明的基于转录组学和影像组学的生存预测方法的流程图。
具体实施方式
如图5所示,这种转录组学和影像组学的生存预测方法,其包括以下步骤:
(1)获取肿瘤患者和部分正常被试的表达谱数据,通过差异表达分析,获得差异表达的mRNA和lncRNA;
(2)从影像数据中分割出各患者的肿瘤区域,提取影像组学特征;
(3)对差异表达的mRNA、lncRNA和影像组学特征进行单因素Cox回归,选取P值小于0.05的因素;然后进一步通过LASSO回归筛选与生存显著相关的特征,构建转录组学和影像组学预后标签;
(4)整合转录组学标签、影像组学标签和临床数据建立多组学预测模型,绘制列线图及生存曲线;
(5)与临床因素预测模型进行区分度、校准度及临床有效性方面的对比,评估多组学预测模型的性能。
本发明通过对研究对象获得转录组学数据和影像组学数据;然后进一步通过LASSO回归筛选与生存显著相关的特征,构建转录组学和影像组学预后标签;整合转录组学标签、影像组学标签和临床数据建立多组学预测模型,与临床因素预测模型进行区分度、校准度及临床有效性方面的对比,评估多组学预测模型的性能。非常适合于GBM等恶性肿瘤生存预测方面的研究。
优选地,该方法还包括步骤(6),统计分析均采用R 3.6.0,所用R软件包如下:glmnet包用于LASSO回归。survival包用于生存分析,ROC曲线和曲线下面积(AUC)的结果由timeROC包得到。DCA用“stdca.R”的功能进行。用Kaplan-Meier方法绘制生存曲线,并通过log-rank检验进行比较。训练集及验证集之间患者的比较对连续性变量进行t检验或方差分析列出P值,对分类型变量,做卡方检验,如有单元格理论频数小于10,自动增加Fish精确检验。所有统计结果均为双尾,P值小于0.05认为具有显著统计学差异。
优选地,所述步骤(1)中,使用R语言中的limma包对数据进行标准化后,然后通过对肿瘤患者与正常组的mRNA和lncRNA表达数据分别进行差异表达分析,筛选差异表达的mRNA和lncRNA。筛选标准均为校正后P值(FDR)≤0.01且|FoldChange|≥2。
优选地,所述步骤(2)中,使用ITK-SNAP(www.itk-snap.org)软件对患者的FLAIR图像进行肿瘤三维分割。需要统计的扫描参数包括层厚(thickness)、TR/TE,层间距(slicegap)、翻转角(flip angle)等。感兴趣区需要覆盖整个肿瘤及水肿区域。所有影像组学特征均使用python 3.7中的pyradiomics插件(https://pyradiomics.readthedocs.io/en/latest/)提取。为了确认特征的可重复性,由两位放射科医生对随机选择的30例患者的感兴趣区(Region Of Interest, ROI)进行分割,计算两个ROI的组内相关系数(Intraclasscorrelation efficient, ICC),当ICC达到0.8时将每个特征用于进一步提取。使用高斯和拉普拉斯(Laplacian of Gaussian, LoG)滤波器、小波(wavelet)滤波器对图像进行预处理。两种滤波器所计算的特征均包含一阶统计特征和基于统计的纹理特征。
优选地,所述步骤(3)中,对差异表达mRNA、lncRNA和影像组学特征分别进行单因素Cox分析,选取P值小于0.05的因素,然后使用R语言中的glmnet包,用Lasso Cox回归方法筛选与预后显著相关的因素,将其作为转录组学和影像组学预后标签。
优选地,所述步骤(4)中,根据影像组学风险评分的中位数将患者分为高危组或低危组。分别绘制影像组学和转录组学的生存曲线以显示高危和低危患者的生存状况。综合转录组学标签、影像组学标签和临床参数构建了多组学列线图以便临床医生方便准确的预测患者的生存率。
优选地,所述步骤(5)中,构建两组不同的预测模型,一是基于临床候选预后危险因素构建的临床因素预测模型,二是基于临床候选预后危险因素、影像组学标签、转录组学标签的多组学预测模型。对构建的临床预测模型,在训练集中通过反复抽取,采用10折交叉验证(Cross-validation)进行内部验证。然后通过验证数据集进行外部验证。依据预测及预测模型国际指南与标准,从区分度(Discrimination)、校准度(Calibration)及临床有效性(Clinical validity)三个方面来评价预测模型的预测效能。通过AUC、C指数、综合判别改善指数(IDI)、重分类改善指标(NRI)来对模型的区分度进行评价。通过校准曲线进行模型的校准度评价。通过决策曲线分析(DCA)量化不同概率下的净获益,以确定临床因素预测模型和多组学预测模型的临床有效性。
本领域普通技术人员可以理解,实现上述实施例方法中的全部或部分步骤是可以通过程序来指令相关的硬件来完成,所述的程序可以存储于一计算机可读取存储介质中,该程序在执行时,包括上述实施例方法的各步骤,而所述的存储介质可以是:ROM/RAM、磁碟、光盘、存储卡等。因此,与本发明的方法相对应的,本发明还同时包括一种基于转录组学和影像组学的生存预测装置,该装置通常以与方法各步骤相对应的功能模块的形式表示。该装置包括:
转录组学数据处理模块,其配置来对处理被试的转录组学数据,使用R语言中的limma包对数据进行标准化后,然后通过对患者与正常组的mRNA和lncRNA表达数据分别进行差异表达分析,筛选差异表达的mRNA和lncRNA,筛选标准均为校正后P值(FDR)≤0.01且|FoldChange|≥2;
影像组学数据处理模块,包括图像分割及影像组学特征选择;
LASSO数据处理模块,将转录组学数据处理模块和影像组学数据处理模块得到的数据进行处理,得到影像组学和转录组学预后标签;
预测模型建立模块,构建两组不同的模型,一是基于临床候选预后危险因素构建的临床因素预测模型,二是基于临床候选预后危险因素、影像组学标签、转录组学标签的多组学预测模型;对构建的临床预测模型,在训练集中通过反复抽取,采用10折交叉验证进行内部验证,然后通过验证数据集进行外部验证。
优选地,该装置还包括预测模型评价模块,依据预测及预测模型国际指南与标准,从区分度、校准度及临床有效性三个方面来评价预测模型的预测效能,通过AUC、C指数、综合判别改善指数、重分类改善指标来对模型的区分度进行评价,通过校准曲线进行模型的校准度评价,通过决策曲线分析量化不同概率下的净获益,以确定临床因素预测模型和多组学预测模型的临床有效性。
以下更详细地说明本发明,具体包括:
本研究在公共数据库收集胶质母细胞瘤的62例样本(57例GBM患者,5例对照),他们具有完整转录组学信息、磁共振影像信息和临床信息,随后,将57个患者随机分成训练集(n=35)和验证集(n =22),比例为6:4。
随后,使用R语言中的limma包对数据进行标准化后,然后通过对GBM患者与正常组的mRNA和lncRNA表达数据分别进行差异表达分析,筛选差异表达的mRNA和lncRNA。筛选标准均为校正后P值(FDR)≤0.01且|FoldChange|≥2。
使用ITK-SNAP(www.itk-snap.org)软件对患者的FLAIR图像进行肿瘤三维分割。需要统计的扫描参数包括层厚(thickness)、TR/TE,层间距(slice gap)、翻转角(flipangle)等。感兴趣区需要覆盖整个肿瘤及水肿区域。所有影像组学特征均使用python 3.7中的pyradiomics插件(https://pyradiomics.readthedocs.io/en/latest/)提取。为了确认特征的可重复性,由两位放射科医生对随机选择的30例患者的感兴趣区(Region OfInterest, ROI)进行分割,计算两个ROI的组内相关系数(Intraclass correlationefficient, ICC),当ICC达到0.8时将每个特征用于进一步提取。使用高斯和拉普拉斯(Laplacian of Gaussian, LoG)滤波器、小波(wavelet)滤波器对图像进行预处理。两种滤波器所计算的特征均包含一阶统计特征和基于统计的纹理特征。
对差异表达mRNA、lncRNA和影像组学特征分别进行单因素Cox分析,选取P值小于0.05的因素,然后使用R语言中的glmnet包,用Lasso Cox回归方法筛选与预后显著相关的因素,将其作为转录组学和影像组学预后标签。
根据影像组学风险评分的中位数将患者分为高危组或低危组。分别绘制影像组学和转录组学的生存曲线以显示高危和低危患者的生存状况。综合转录组学标签、影像组学标签和临床参数构建了多组学列线图以便临床医生方便准确的预测GBM患者的生存率。
构建两组不同的预测模型,一是基于临床候选预后危险因素构建的临床因素预测模型,二是基于临床候选预后危险因素、影像组学标签、转录组学标签的多组学预测模型。对构建的临床预测模型,在训练集中通过反复抽取,采用10折交叉验证(Cross-validation)进行内部验证。然后通过验证数据集进行外部验证。依据预测及预测模型国际指南与标准,从区分度(Discrimination)、校准度(Calibration)及临床有效性(Clinicalvalidity)三个方面来评价预测模型的预测效能。通过AUC、C指数、综合判别改善指数(IDI)、重分类改善指标(NRI)来对模型的区分度进行评价。通过校准曲线进行模型的校准度评价。通过决策曲线分析(DCA)量化不同概率下的净获益,以确定临床因素预测模型和多组学预测模型的临床有效性。
训练集和验证集患者的临床特征如表1。根据年龄是否大于60岁分为高龄组和低龄组,在训练集和验证集,患者的中位生存时间分别为1.21年和1.67年。患者年龄、性别、种族、KPS评分、肿瘤分型、CpG岛甲基化表型(CIMP)、IDH分型、是否接受放疗、是否接受药物治疗、以及总生存期均无统计学差异(P = 0.187-1.000)。
表1 训练集、验证集入组患者的人口统计学资料
选择校正后P值(FDR)≤0.01且|FoldChange|≥2的mRNA和lncRNA为差异表达mRNA和lncRNA。与正常人相比,GBM患者有3129个差异mRNA、1132个差异lncRNA(图1)。对差异mRNA和lncRNA做单因素生存分析,其中P值小于0.05的差异mRNA有102个,差异lncRNA有38个。
通过pyradiomics插件得到了851个影像组学特征,其中小波滤波器得到的特征744个,LoG滤波器得到的特征93个,基于形状和大小的特征14个。对影像组学特征做单因素Cox生存分析,P值小于0.05的特征有6个(表2)。
表2 与预后有关的影像组学特征
将单因素生存分析P值小于0.05的差异mRNA102个、差异lncRNA38个、影像组学特征6个(共计146个)纳入LASSO Cox回归模型,使用十倍交叉验证筛选最优的与预后相关的特征(图2a),通过调节不同参数的lambda值获得最小的偏差,筛选出最佳的特征(图2b)。LASSO Cox回归筛选出21个特征,其中影像组学特征2个,转录组学特征19个(mRNA14个,lncRNA5个),将其作为转录组学和影像组学预后标签(表3)。转录组学和影像组学风险评分的中位数为1.405和1.185,可分别将GBM患者分为高危组和低危组。
表3 影像组学和转录组学标签
单因素Cox分析表明,没有接受放疗(HR = 3.099,P = 0.011)、G-CIMP阴性(HR =9.440,P = 0.027)、IDH野生型(HR = 10.404,P = 0.002)、没有接受药物治疗(HR =2.491,P = 0.021)、影像组学高风险(HR = 1.946,P = 0.022)和转录组学高风险(HR =19.339,P < 0.05)是GBM总生存期的预后因素(表4);多因素Cox分析表明,没有接受药物治疗(HR = 7.069,P < 0.05)、影像组学高风险(HR = 2.622,P < 0.05)和转录组学高风险(HR = 25.000,P < 0.05)是GBM总生存期的预后因素(表4)。应用Kaplan-Meier生存曲线显示高危和低危患者的生存状况,结果二者均有统计学差异,Log-rank检验P值<0.05(图3a和图3b)。将单因素Cox回归P值<0.05的预后因素纳入多组学列线图(图3c)。
表4 GBM患者总生存期的单因素和多因素Cox分析
在训练集和验证集中,多组学预测模型的AUC分别为0.964和0.907,C-index分别为0.869和0.891;临床预测模型的AUC分别为0.830和0.787,C-index分别为0.784和0.751;多组学预测模型相对于临床因素预测模型的IDI分别为0.119和0.222(图4a和图4b)。多组学预测模型在训练集中的NRI值是0.302,95%可信区间为(-0.417,1.000),在验证集中的NRI值是0.727,95%可信区间为(-0.324,1.597)。说明在训练集和验证集中,多组学预测模型的区分度高于临床因素预测模型。
采用校准曲线对多组学预测模型进行校准度评价,多组学预测模型的校准曲线距离45度对角线更加接近,模型的校准度较高(图4c)。使用DCA决策曲线评估多组学预测模型和临床因素预测模型的临床有用性,与临床因素预测模型相比,多组学预测模型曲线更加靠近右上角,其净获益增加更多(图4d)。
以上所述,仅是本发明的较佳实施例,并非对本发明作任何形式上的限制,凡是依据本发明的技术实质对以上实施例所作的任何简单修改、等同变化与修饰,均仍属本发明技术方案的保护范围。
- 一种基于影像组学的生存期预测方法及装置
- 一种基于影像组学的生存期预测方法及装置
