一种肉鸡SNP分子标记组合及其应用
文献发布时间:2024-04-18 19:58:30
技术领域
本发明属于生物技术领域,具体涉及一种肉鸡SNP分子标记组合及其应用。
背景技术
中国是全球禽类消费第一大国,鸡肉是我国的第二大肉类消费品种。鸡肉不仅具有“高蛋白、低脂肪、低热量”的营养特点,而且肉鸡的饲料转化率更高,消耗的资源更少,相比牛肉、羊肉、猪肉,鸡肉生产更加低碳,更有利于可持续发展。然而我国肉鸡育种起步较晚,高生产性能遗传资源积累基础薄弱,分子育种等新技术应用不够,导致我国在优质地方种质资源保护、质量提升和新品种选育等方面进展缓慢。
分子育种准确率高,可有效提高育种工作效率,已经成为现代家禽育种的主要方式。分子育种技术包括基因组育种技术和基因转移育种技术,基因组育种的前提是高通量、高密度的分子标记开发。单核苷酸多态性(SingleNucleotidePolymorphism,SNP)是第三代分子标记技术,指在基因组水平上由于单个核苷酸的转换、颠换、缺失和插入等4种变异而引起的基因组水平的多态性表现。SNP作为基因组中分布更为广泛的遗传标记,具有密度高、遗传稳定性高和易于自动化分析等特点,在动植物分子育种方面具有广泛的应用。
因此,提供一种高密度、高准确率、低成本、实用性强的基于SNP的肉鸡高通量液相芯片非常重要。
发明内容
本发明旨在至少解决上述现有技术中存在的技术问题之一。为此,本发明提出一种肉鸡SNP分子标记组合。
本发明还提出一种用于检测上述肉鸡SNP分子标记组合的基因芯片。
本发明还提出一种用于检测上述肉鸡SNP分子标记组合的试剂盒。
本发明还提出一种上述肉鸡SNP分子标记组合、基因芯片或试剂盒的应用。
本发明还提出一种肉鸡育种方法。
根据本发明的一个方面,提出了一种肉鸡SNP分子标记组合,包括60784个SNP分子标记中的至少一种,所述60784个SNP分子标记的物理位置是基于鸡的参考基因组GRCg7b进行序列比对确定的,位点信息具体如下:
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
/>
在本发明的一些实施方式中,所述SNP位点的位置信息采用染色体编号:物理位置的形式进行表示。
在本发明的一些实施方式中,所述60784个SNP分子标记如SNP00001-60784所示;SNP00001-60784的具体位点信息如上表从上至下,从左至右依次排序。
在本发明的第二方面,提出了一种肉鸡SNP芯片,所述肉鸡SNP芯片包括用于检测上述的肉鸡SNP分子标记组合的引物组和/或探针。
在本发明的一些实施方式中,所述肉鸡SNP芯片对样本中目标区域进行捕获的方式为液相探针捕获。
在本发明的第三方面,提出了一种试剂盒,所述试剂盒包含用于检测肉鸡SNP分子标记组合的引物组和/或探针。
在本发明的第四方面,提出了上述肉鸡SNP分子标记组合、肉鸡SNP芯片和试剂盒中的至少一种的应用。
在本发明的一些实施方式中,所述应用具体可以通过以下方法实现:
S1、采用上述肉鸡SNP分子标记组合、肉鸡SNP芯片和试剂盒中的至少一种对待测试的肉鸡样本进行基因型分型,获得基因型分型结果;
S2、对步骤S1获得的基因型分型结果进行分析。
在本发明的一些实施方式中,所述步骤S2中,所述分析的方法包括但不限于PCA分析。
在本发明的一些实施方式中,所述应用为在肉鸡基因分型中的应用。
在本发明的一些实施方式中,所述应用为在肉鸡全基因组关联分析或全基因组选择或遗传多样性评估或遗传图谱构建及基因定位中的应用。
在本发明的一些实施方式中,所述应用为在肉鸡聚类分析和/或亲缘关系鉴定中的应用。
在本发明的一些实施方式中,所述应用为在肉鸡品种鉴定中的应用。
在本发明的一些实施方式中,所述亲缘关系鉴定和/或肉鸡品种鉴定为湖南省内肉鸡品种和湖南省外肉鸡品种的亲缘关系鉴定和/或肉鸡品种鉴定。所述湖南省内肉鸡品种包括桃源鸡、东安鸡、黄郎鸡、雪峰乌骨鸡、石门黑鸡、湘佳黑凤鸡中的至少一种。湖南省外肉鸡品种包括狼山鸡、江汉鸡、五华黄鸡和浙杨黄鸡中的至少一种。
在本发明的一些实施方式中,所述应用为在肉鸡育种或辅助育种中的应用。
在本发明的一些实施方式中,所述育种或辅助育种包括辅助的主效基因选择、回交育种及其背景选择、分子辅助育种、QTL分析、物种进化分析和种质资源鉴定中的至少一种。
在本发明的第五方面,提出了一种肉鸡育种方法,包括如下步骤:利用上述肉鸡SNP分子标记组合、肉鸡SNP芯片和试剂盒中的至少一种对待测肉鸡基因组DNA进行检测、基因型分型,选择合适肉鸡进行后续育种。
在本发明的一些实施方式中,所述检测基于液相探针捕获测序分型技术进行。
本发明至少具有以下有益效果:
本发明从湖南省地方品种桃源鸡、东安鸡、黄郎鸡、雪峰乌骨鸡和商业肉鸡品种等遗传资源材料中选择具有代表性的296份肉鸡材料,从中筛选出均匀覆盖全基因组、多态性高、特异性强、通用性好的60784个SNP标记(MAF值均≥0.15),将其制备得到肉鸡60K SNP液相芯片,填补了湖南地方肉鸡育种芯片的空白;利用实施例提供的肉鸡SNP分子标记组合、用于检测上述60784个SNP标记组合的SNP芯片或试剂盒在位点选择和样本检测数量上具有灵活性强、高自主可控等多项优势;可实现高通量SNP基因分型,分型成本低,检出率高,稳定性好,针对特定基因组目标区域的位点检出率、重复样本基因分型一致性均在99%以上,分型结果准确可靠,能够极大地提升肉鸡的育种准确性和效率,为肉鸡全基因组关联分析、全基因组选择、品种鉴定及选育、遗传多样性评估、种质资源和亲缘关系鉴定、遗传图谱构建及基因定位、分子标记辅助育种等提供了重要的工具;可实现湖南省地方品种和省外肉鸡品种的品种鉴定和亲缘关系鉴定,能够科学引导肉鸡杂交改良工作,加速肉鸡育种进程;基于目标区间基因组序列液相捕获的精准定位测序分型技术,不仅可对目标位点进行分型,而且可检测目标区间及其周围一定范围内的所有遗传变异位点,输出结果信息量大。
附图说明
下面结合附图和实施例对本发明做进一步的说明,其中:
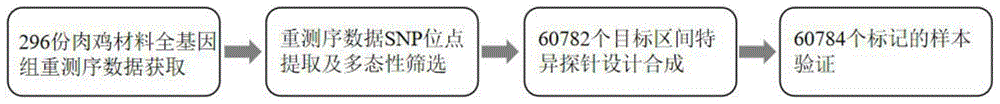
图1为本发明实施例1中的位点开发流程图;
图2为本发明实施例1中的SNP位点在不同染色体上的分布图;
图3为本发明实施例1中的SNP位点在不同染色体上的密度分布图;
图4为本发明测试例中的基于液相探针捕获测序分型技术的原理图;
图5为本发明测试例中对56份肉鸡样本位点的检出率统计结果;
图6为本发明测试例中的重复样本基因型一致率检测结果图;
图7为本发明测试例中的60K SNP液相芯片区分5个鸡品种(江汉鸡、狼山鸡、石门黑鸡、五华黄鸡和浙杨黄鸡)PCA图。
具体实施方式
以下将结合实施例对本发明的构思及产生的技术效果进行清楚、完整地描述,以充分地理解本发明的目的、特征和效果。显然,所描述的实施例只是本发明的一部分实施例,而不是全部实施例,基于本发明的实施例,本领域的技术人员在不付出创造性劳动的前提下所获得的其他实施例,均属于本发明保护的范围。
实施例1
本实施例提供了一种肉鸡SNP分子标记组合,包括60784个SNP位点,每个SNP位点包含两个不同碱基变异位点,用于检测该位点的等位基因变化,所述60784个SNP位点的物理位置是基于鸡的参考基因组(GRCg7b)序列比对确定的,所述60784个SNP分子标记如发明内容部分所示(具体信息如发明内容表格中所示)。其筛选流程如图1所示,具体如下:
(1)从湖南省地方品种桃源鸡、东安鸡、黄郎鸡、雪峰乌骨鸡和商业肉鸡品种等遗传资源材料中选择具有代表性的296份肉鸡材料。采集296份肉鸡的血液样本,提取基因组DNA,用质量分数为1%的琼脂糖凝胶电泳检测,保证基因组完整性,并测定DNA浓度,将DNA置于-20℃冰箱保存备用。采用酶切打断法构建基因组DNA文库,并进行全基因组重测序。
(2)将测序获得的原始数据进行质控,去掉被接头污染的reads和低质量reads,得到高质量的clean reads。再将其与鸡的参考基因组GRCg7b进行比对,获得各样本的比对结果。对比对结果进行SNP位点的提取,通过数据分析获得SNP位点的染色体位置、最小等位基因频率、缺失率、杂合率等指标。将所有材料作为一个群体计算指标,按照最小等位基因频率MAF≥0.15、缺失率<0.05、杂合率≤0.35、测序深度≥10的过滤指标对全基因组SNP位点进行初步筛选,获得1317443个SNP位点。再将10个品系分开计算指标,按照MAF≥0.1的过滤指标对初筛得到的SNP位点进行第二次筛选,以确保各品系内位点多态性,获得179406个SNP位点。
将上述所有候选位点用于探针设计。探针设计方法是:针对每一个SNP位点前后100bp范围内设计探针,探针长度一般为100bp,GC含量在20%~80%之间。根据位点筛选要求和探针设计结果,去除在基因组上无法唯一比对、侧翼序列中包含重复序列的探针。根据物理位置均匀分布挑选位点,保留2851个功能位点和61个候选基因位点,最终筛选获得高质量、高多态SNP位点60784个,位点均匀覆盖全基因组各染色体(如图2、图3所示)。
实施例2
本实施例提供一种肉鸡全基因组液相基因芯片。
基于优化的热力学稳定性算法模型,针对实施例1提供的60784个用于芯片开发的SNP标记设计、合成60782条特异性探针,得到肉鸡60K SNP液相芯片。
实施例3
本实施例提供应用实施例2中的肉鸡全基因组液相基因芯片用于对肉鸡进行基因分型的方法。步骤如下:
(1)基因组DNA的提取及质控
提取待测肉鸡样本的基因组DNA,对DNA样品进行质量检测。质量检测包括用Qubit对DNA浓度进行测定,用1%琼脂糖凝胶电泳检测DNA的完整性。检测合格的样品于-20℃保存备用。
(2)构建文库、文库杂交捕获
使用酶切打断法构建样本基因组DNA文库,利用实施例2中的肉鸡全基因组液相基因芯片中的探针对位于不同基因组位置的多个不同目标序列进行液相杂交捕获,对捕获的目标基因组序列进行扩增富集、捕获文库构建,其原理如图4所示。
使用Qubit检测捕获文库浓度,采用Agilent 2100/2200Bioanalyzer检测捕获文库片段大小是否在250bp~500bp之间。采用华大测序平台对质检合格的捕获文库进行PE150测序。
(3)数据分析
高通量测序后的原始数据经过质控过滤等处理,使用fastp软件去除含有接头污染和低质量的reads。利用BWA软件将获得的clean reads与鸡参考基因组GRCg7b进行比对,再用GATK软件对测序结果进行变异位点分析,获得变异位点的基因型数据。
测试例1
本测试例采用实施例2提供的肉鸡60K SNP液相芯片对56份肉鸡样本(8份黄土二号、8份邵伯鸡、24份石门黑鸡、8份湘佳黑凤鸡、8份黄狼鸡)进行基因分型检测,具体使用方法如实施例3所示。
经测序与数据分析,56份肉鸡样本的位点平均检出率为99.57%;杂合率在31.61%~34.34%;技术重复样本基因型平均一致率为99.53%。检测结果如图5和6所示。
这表明使用实施例2提供的肉鸡60K SNP液相芯片对待检测材料进行基因分型时,目标位点检出率高,稳定性好,分型结果准确可靠,可应用于分子标记辅助育种。
测试例2
本测试例采用实施例2提供的肉鸡60K SNP液相芯片对455份湘佳黑凤鸡样本进行基因分型检测,具体使用方法如实施例3所示。同时以“京芯一号”鸡55K商业化芯片作为对照,对836份湘佳黑凤鸡样本进行基因分型检测。
对于样本的基因分型数据,过滤掉最小等位基因频率(MAF)<0.05、位点检出率<0.9、HWE检验(p<0.00001)的SNP位点。结果显示,实施例2提供的肉鸡60K SNP液相芯片的位点利用率达91.44%,样本位点平均检出率为99.63%,平均杂合率为0.35,位点平均MAF为0.27;“京芯一号”芯片位点利用率76.53%,样本位点平均检出率99.01%,平均杂合率为0.30,位点平均MAF为0.25。
结果表明与“京芯一号”鸡55K商业化芯片相比,实施例2提供的肉鸡60K SNP液相芯片的位点利用率、检出率更高,位点多态性更好,各方面性能均优于“京芯一号”鸡55K商业化芯片。
测试例3
本测试例采用实施例2提供的肉鸡60K SNP液相芯片对湖南省地方品种(石门黑鸡)和湖南省外品种(江汉鸡、狼山鸡、五华黄鸡和浙杨黄鸡)进行基因分型检测,具体使用方法如实施例3所示。
对样本的基因型分型结果进行主成分分析(PCA分析),计算各品种主成分,选取品种特征贡献度前二的主成分进行分析。结果显示,实施例2提供的肉鸡60K SNP液相芯片可以对石门黑鸡、江汉鸡、狼山鸡、五华黄鸡和浙杨黄鸡5个品种进行区分;其中,湖南省地方品种(石门黑鸡)和湖南省外品种明显区分。结果如图7所示。
这表明,实施例2提供的肉鸡60K SNP液相芯片能够很好的应用于肉鸡的品种鉴定和亲缘关系鉴定,科学指导肉鸡品种改良和种质资源保护。
上面结合附图对本发明实施例作了详细说明,但是本发明不限于上述实施例,在所属技术领域普通技术人员所具备的知识范围内,还可以在不脱离本发明宗旨的前提下作出各种变化。此外,在不冲突的情况下,本发明的实施例及实施例中的特征可以相互组合。
- 一种与绵羊多羔性状相关的SNP分子标记及其检测试剂盒和应用
- 一种与鸡生长肉用性状相关的SNP分子标记及其应用
- 一种与鸡屠体性状相关的SNP分子标记及其应用
- 一种与瓦氏黄颡鱼生长性状相关的SNP分子标记及应用
- 一种鉴定瓦氏黄颡鱼低溶氧耐受性和肥满度的SNP分子标记及其应用
- 一种鉴定仙居鸡品种的SNP分子标记、SNP分子标记组合及其应用和鉴定方法
- 一种鉴定白耳黄鸡品种的SNP分子标记、SNP分子标记组合及其应用和鉴定方法
