一种棉纤维强度相关基因的发现方法及应用
文献发布时间:2024-04-18 19:58:53
技术领域
本发明属于植物遗传技术领域,涉及一种棉纤维强度相关基因的发现方法及应用。
背景技术
棉花是世界上重要的经济作物,也是一种重要的油料作物;棉纤维是纺织工业的重要资源,也是细胞生物学研究的优良模型。国内主推的棉花品种缺少纤维长度、纤维强度、纤维细度搭配协调的特优质陆地棉品种,因此,在继续维持棉花高产量的同时,进一步提升棉花纤维品质是重中之重,而挖掘并鉴定出优良纤维品质的候选基因,培育出纤维品质优良的棉花品种显得极为重要。纤维品质差异显著的近等基因系和高通量测序技术的应用有助于鉴定纤维发育相关的关键基因及其调控机制。长链非编码RNA(long non-codingRNA,lncRNA)是基因组转录的重要组成部分,在植物发育和应激反应等广泛的生物过程中发挥着作用。前人研究表明lncRNA在棉纤维发育过程中存在着重要影响,因此,对其进行深入的研究,有望发掘棉纤维发育关键基因从而应用于棉花纤维品质分子育种。
发明内容
有鉴于此,本发明目的在于提供一种棉纤维强度相关基因的发现方法及应用。
本发明采用的技术方案如下:
一种棉纤维强度相关基因的发现方法,包括:选择黄褐棉渐渗系IL9及其陆地棉轮回亲本PD94042构成的一对近等基因系为材料,选择开花后17和21天的棉纤维,提取RNA,反转录为cDNA,建库开展lncRNA测序分析,然后进行靶基因的预测与筛选,得到棉纤维强度相关基因;
所述棉纤维强度相关基因包括Gomus.D05G015100、Gomus.A05G281300和Gomus.A10G226800;
其中,Gomus.D05G015100的编码序列如SEQ ID NO.1所示;
Gomus.A05G281300的编码序列如SEQ ID NO.2所示;
Gomus.A10G226800的编码序列如SEQ ID NO.3所示。
本发明还提供了一种棉纤维强度相关基因在改良棉花棉纤维强度中的应用,所述棉纤维强度相关基因为Gomus.D05G015100、Gomus.A05G281300和Gomus.A10G226800中的一种或几种;
其中,Gomus.D05G015100的编码序列如SEQ ID NO.1所示;
Gomus.A05G281300的编码序列如SEQ ID NO.2所示;
Gomus.A10G226800的编码序列如SEQ ID NO.3所示。
本发明还提供了一种棉纤维强度相关基因在分子标记辅助选育棉花品种中的应用,所述棉纤维强度相关基因为Gomus.D05G015100、Gomus.A05G281300和Gomus.A10G226800中的一种或几种;
其中,Gomus.D05G015100的编码序列如SEQ ID NO.1所示;
Gomus.A05G281300的编码序列如SEQ ID NO.2所示;
Gomus.A10G226800的编码序列如SEQ ID NO.3所示。
G.mustelinum v1.1|Gomus.D05G015100.1CDS
>ATGGGGGCTTCTATTTTATCAGATCTCGGAACCGAGATCTTAATTCCAGTTTGCGCCGTGATTGGAATT GCCTTTTCTCTCTTACAATGGGTTCTCGTTTCCAAGGTGAAGCTCTCTCCTGGTCGAGACCCGGGTTCACCTAGAAACAACGGTGCGGGTGGAAAAAATGACTGCGCCGATTACCTCATTGAAGAAGAAGAAGGACTTAATGATCATAATGTTGTTCTTAAATGTGCTGAAATTCAAAGCGCCATCTCTGAAGGGGCAACCTCATTTCTTTTCACCGAGTACCAGTATGTTGGCATTTTCATGGTTGCTTTTGCAATCTTGATTTTTGTCTTCCTTGGTTCGGTAGAGGGTTTCAGCACAAAGAGTCAGCCTTGCACATATGACCAATCTAAGATTTGCAAGCCTGCTCTTGCCACTGCTGCATTCAGCACAATATCTTTCTTACTTGGTGCTGTCACTTCAGTTGTTTCTGGCTTTCTTGGAATGAAGATTGCTACCTATGCAAATGCTAGAACCACCCTGGAGGCAAGAAAGGGAGTTGGAAAGGCATTTATCACTGCATTTAGATCTGGTGCTGTCATGGGTTTTCTTCTTGCTGCAAATGGCCTTCTCGTGCTCTACATTACCATCAACTTATTCAAGATCTACTATGGTGATGATTGGGGTGGTCTTTTTGAAGCGATCACTGGTTATGGGCTTGGAGGTTCATCCATGGCACTTTTTGGTAGAGTTGGTGGAGGCATCTACACCAAAGCTGCTGATGTGGGTGCTGATCTTGTTGGCAAGGTGGAAAGGAACATTCCTGAGGATGACCCTAGAAACCCAGCTGTTATTGCTGACAATGTTGGAGATAATGTTGGTGATATAGCTGGAATGGGATCTGATCTTTTTGGTTCCTATGCTGAATCATCTTGTGCTGCACTTGTTGTTGCTTCCATCTCTTCTTTTGGCATCAATCATGAGTTCACTCCAATGTTATATCCTCTCATCATAAGTTCTGTTGGTATCATTGTTTGTTTAATCACAACCTTATTTGCAACTGATTTCTTTGAGATCAAGGCTGTTAAGGAAATTGAGCCATCATTGAAGAGGCAACTTATCATATCCACTGTTCTCATGACTATTGGAATTGGGGTTGTTAGTTGGGTAGCTCTTCCATCTTCCTTTACCATTTTCAACTTTGGAGATCAGAAAGTTGTCAAGAACTGGCAACTATTCTTATGTGTTGCTGTTGGTCTTTGGGCTGGTCTAATTATTGGTTTTGTAACCGAGTACTATACCAGTAACGCATACAGCCCTGTACAAGATGTTGCTGATTCCTGCAGAACTGGAGCAGCAACTAATGTTATTTTCGGCCTTGCATTGGGTTACAAGTCTTGCATTATTCCTATATTTGCTATTGCAGTCAGTATTTTTGTTAGTTTTAGCTTTGCTGCTATGTATGGCATTGCTGTTGCTGCCCTTGGAATGCTGAGCACCATAGCTACTGGACTGGCTATTGATGCTTATGGTCCTATCAGTGACAATGCTGGAGGTATTGCTGAGATGGCTGGCATGAGCCACAGAATACGAGAGAGAACGGATGCTCTGGATGCTGCTGGAAACACCACTGCTGCTATTGGAAAGGGTTTCGCCATTGGTTCAGCTGCCCTTGTGTCCCTTGCCCTCTTTGGTGCTTTTGTGAGCCGTGCTGCTATTTCAACAGTTGATGTATTGACCCCTAAAGTTTTTATTGGTTTGCTTGTCGGGGCAATGCTTCCTTACTGGTTTTCTGCTATGACCATGAAGAGTGTGGGAAGTGCAGCTTTGAAGATGGTTGAGGAAGTGCGCAGGCAATTCAACACAATCCCAGGTCTCATGGAGGGTACTGCTAAACCTGACTATGCTACCTGTGTTAAGATCTCTACTGATGCTTCAATCAAAGAAATGATCCCACCTGGTGCCCTTGTTATGCTCACACCCCTTGTTGTTGGGATCTTTTTTGGCGTGGAAACTCTCTCTGGTGTCCTCGCTGGATCTCTTGTCTCTGGTGTCCAGATTGCTATCTCTGCATCCAACACAGGAGGTGCTTGGGATAATGCCAAGAAGTATATTGAGGCTGGTGCTTCTGAGCATGCAAAAACTCTTGGTCCCAAAGGTTCAGATCCACATAAGGCTGCCGTTATTGGTGACACCATTGGTGACCCTTTAAAGGATACATCTGGGCCATCACTGAACATCCTCATCAAGCTAATGGCTGTTGAATCTCTTGTCTTTGCTCCCTTCTTTGCTACACATGGTGGGCTGCTTTTTAAGATATTTTAA
G.mustelinum v1.1|Gomus.A05G281300.1 CDS
>ATGTATCAACCTTCCAGTCGAAAAGAAGCTGACATGAGGCTTGATAGCCAAGTTCTAGATCTAGAAACGGCTGTAAAAGATGGGATTTTGGGAGGAAATTGTGGGGTGATATCCACTGGTTTTGGTTTAAAGAAATTGGATCTGAAGTTAATGGTCGAAGAGCTTGAGTCCATTGATATGCCTACGGTTTTCATATGTCCAATTTCTTTGGAGCCAATGCGAGACCCTGTGACCCTTTGCACTGGTCAAACCTACGAGAGATCCAACATTGTTAAATGGTTCTCATTAGGCCATTACACATGTCCCACCACAATGCAAGAGCTTTGGGATGATTCGGTGACTCCAAATAAAACCCTTCAGCAGCTAATTTATAGTTGGTTTTCACAGAAGTATTTGGCTATGAAGAAGAGGTCAGAGGATGTGCAAGGAAGGGTCAAAGAGGTTTTGGAGAATTTGAAGAAAGTTAAGGGTCAAGCTAGAGTTCAAGCACTTAAAGAGCTTAGGCAAGTGGTTCAAGCTCATGGGACAGCTTTGAAGACTGTAGCGGAAAATGGAGGCATTGCTTTCATTTCATCTTTGCTTGGTCCTTTTACAACCCATGCTGTTGGCTCAGAGGTGATTGGTATTCTTGTGAACTTCAATCTTGATTTAGATTCCAAGTCTGATTTGTTGCAACCTGCTAAAATATCTTTGATAGTTGATATTTTAAATGAAGGGTCGATTGAGACCAAGATTAACTGTACTAGGTTGATTGGAATGTTGATGGGAGGGAATGATTTTGCATCCCAAAATGTTGCAAGTTTGAGTCTTTTAGTTGGTTTGTTGAGGTTAGTGAAAGATAAAAAGCACCAAAGTGGGGTTTTATCTGGACTCATTTTACTCAAAACAGTTTGTTCTAATGAATCAGTTAGGAACTCTTTTGTCAATGTTGGGGCAGTCCCTCAATTGGTTGAATTGATGCCTGGTTTGAACAATGAATGTTTGGAATTAGTCCTTTGTATATTGGAACTTTTGTCAAGTATTCCAGAAGGAAGATTGGCTCTAAAAGATTGTCCAAATACCATACCAAATGTGGTGAAATTATTGATGAAAGCTTCTGAAAACTGTACTCAGCTTGCATTGTCAATACTCTGGTCCATATGCAAGTTTGCACCAGAGGAATGCGCTTCACTTGCTGTTGATGCTGGTCTAGCTGCAAAGCTCCTTCTAGTTATACAGAGTGGTTATGATCCCGTGTTGAAGCAACGGTCGGCCGAGTTATTGAAACTGTGTAGCCTAAATTTCACTGATAACATCTTTATTTCCAAGTGTAAGCTTACACGAACTATTGAATGA
G.mustelinum v1.1|Gomus.A10G226800.1 CDS
>ATGATAACAGTCATGGATTCTAGGAAAGAAGTTGTGAAAAATTCAGAAAAATGCTTAGATCCTCAGCTATGGCATGCTTGTGCTGGTGGTATGGTACAAATGCCATCAGTGAACTCTAAGGTGTTCTATTTCCCTCAGGGTCATGCTGAGCATGCCAATGGGAATGTAGACTTTGGGAATCTCCCGATTCCGTCGCTCGTCTTGTGTCGGGTTTCCGCCGTTAGGTTTATGGCGGATCCCGAAACTGATGAGGTTTATGCTAAAATCATGTTGATTCCTTTGAGAGAGAATAGTTTTGGGGTTGAAGATGATGGTTTTGATGGGAATGTTGGGGTGGAGAATCCTGAAAAATCTGCTTCCTTTGCTAAGACATTGACTCAGTCCGATGCTAACAACGGTGGGGGGTTCTCGGTTCCACGGTATTGCGCGGAGACTATATTTCCTAGGTTGGATTATAATGCTGAACCCCCTGTTCAGACTATTCTTGCTAAGGATGTTCATGGTGAGGTTTGGAAATTTAGGCATATATATAGGGGGACGCCTCGTCGACATCTTTTGACGACGGGGTGGAGTAATTTCGTCAACCACAAGAAGCTTGTGGCTGGTGATTCGATTGTGTTTCTTAGAGCTGATAATGGAGATCTTTGTGTGGGGATCCGTAGGGCGAAAAGAGGACTTGGGGGTGGACATGAATTTCCTGGATGGAACTCTGTGAGTGGAACCTCTGGTTCACAAGTTGGGAGTTATTCTCCGTTTTTGAGGGAGGGGGAGAGTAAATTGATGAGGAAGGATTGTAATGGGGATCCAAGGGGAAGAATAAGGGCCGACTCCGTGATTGAAGCTGCAAGTCATGCAGCTAGTGGGCAGCCCTTTGAGGTCGTTTACTATCCACGAACTAGCACTCCTGAGTTCTGCGTGAAGGCCTCATCCGTTAGAGCTGCAATGCAAATCCAGTGGTATCCTGGGATGAGGTTCAAAATGCCTTTTGAAACTGAGGACTCTTCACGGATTAGCTGGTTCATGGGAACGATATCTACTGCTCAGGTTGTCGATCCCATCCGTTGGCCTAATTCTCCATGGCGACTACTGCAGGTAGCATGGGATGAGCCAGACTTACTCCACAATGTGAAGCGTGTTAGCCCATGGTTGGTTGAATTGGTCACAAACATACCAGCTATTAATCTTAATCCTTTCTCGCCCCCAAGGAAAAAGATGCGGCTTCCACAACACCCAGACATTTCCTTTCTTAACCAAATTCCAATGCCATCATTTTCCGGGAACTCATTCAGATCCAGCAGCCCCATGCGTTGCATTACGGACAACATTCCTGGAGGCATACAGGGAGCCAGGCATGAACCGTTCGGATTATCTTCATCAGATCTCCGCTCCAACAAGCTGCATTCAGGTCTGTTTCCATCTGGTTTTCATCAACTTGATCGTACTGCCCCACCTACTAGACTTTCCGGCAACAACTTTTGTAGCGACAACGCCAACAATACAAATATATCTTCCTTGTTGACAATAGGAAACCCAACCCAGAGTTTGAAACAAAGCAATGATAGCAAGACACCCCATATTGTATTGTTTGGTCAACTCATTTTCTGTGAGCAGCGGGCTTCTCAGAGCTGCTCAGGTGATACGGTTGGAAACAGTTCATCAGATGGGAATACAGAGAAGACTGCTATTTCCTCTGATGGAACTGGATCTGTGTTACATCAAAATGTTCGGGAAAATTCTTCGGATGAAGGGTTTCCTTGGTGCAAAGAGGATCAAAAAACTGATCTAGGATTGGAGACCGGTCATTGCAAAGTGTTTATGGAATCAGAGAATGTAGGTAGAACCCTTGATCTTTCCGTTCTCGGATCATATGAAGAGCTGTATGGGAAGCTGGCCAACATGTTTGGCATAGAAAGTTCAGAGACGCTGAGCAGTGTGCTCTACCGTGATGCTGCCGGTTCAGTTAAACACACTGGAGATGAGCCCTTCAGCGAGTTTATGAAAACAGCAAGGAGGCTAACGATTCTCATGGATTCAAGCAGTGACAACTTAGAAAGATAG
与现有技术相比,本发明选择黄褐棉渐渗系IL9及其陆地棉轮回亲本PD94042构成的一对近等基因系为材料,选择开花后17和21天的棉纤维,通过lncRNA测序分析,然后进行靶基因的预测与筛选,得到棉纤维强度相关基因,表达验证和功能验证均表明本发明提供的纤维强度功能基因发现方法准确高效,这些基因可用于改良棉花的纤维品质,也可用于作为选种育种的分子标记。
附图说明
图1为lncRNA差异表达图;
图2为纤维强度相关lncRNA靶基因的表达分析;注:柱形图上方的星号统计显著差异(***P<0.001,****P<0.0001);
图3为纤维强度相关靶基因在棉花中的VIGS功能验证实验中各植株苗期叶色表型图;其中,图中第一列为阴性对照(空载体pCLCrV::00),第二列为阳性对照(沉默了八氢番茄红素脱氢酶基因的植株pCLCrV::GhPDS),第三列为实验组(pCLCrV:Target gene);A-C组目的基因分别为Gomus.A05G281300、Gomus.D05G015100、Gomus.A10G226800;pCLCrV:Target gene代表分别沉默3个目的基因实验组;
图4为VIGS沉默植株的基因表达验证;A:叶片;B:17dpa-棉纤维;C:21dpa-棉纤维。柱形图上方的星号表示统计显著差异(***P<0.01,***P<0.001,****P<0.0001)。
具体实施方式
本发明提供了一种棉纤维强度相关基因的发现方法,包括:
步骤1、lncRNA测序分析
利用纤维品质综合表现突出的黄褐棉渐渗系IL9及其陆地棉轮回亲本PD94042构成的一对近等基因系为材料,选择开花后17和21天(days post anthesis,dpa)的棉纤维,提取RNA,反转录为cDNA,建库开展lncRNA测序分析。lncRNA是作为长链非编码RNA的一类,其长度通常>200bp,依据与编码序列的位置关系不同可将其分为intergenic lncRNA(简称lincRNA),intronic lncRNA,anti-sense lncRNA,sense lncRNA,bidirectional lncRNA等类型。其中lincRNA所占比例最高,本实验主要是对lincRNA,intronic lncRNA,anti-sense lncRNA这三种类型的转录本进行筛选。
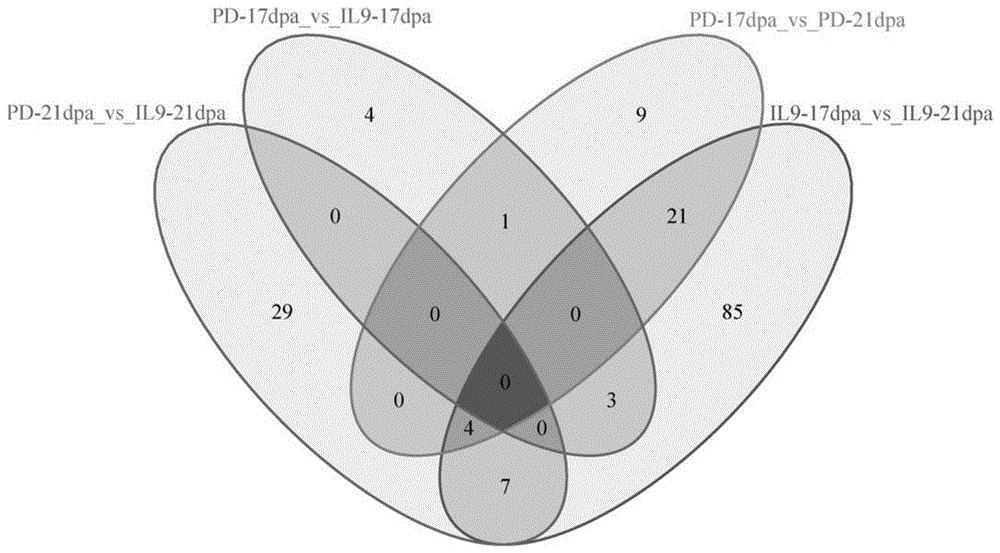
对鉴定到的lncRNA进行差异表达分析,共获得差异lncRNA数量为203个,与mRNA中结果趋势一致,在IL9-17dpa与IL9-21dpa之间差异数量最多,共120个,在PD94042-17dpa与IL9-17dpa中差异数最少,仅有8个,将其统计结果如图1所示。
步骤2、lncRNA靶基因的预测
由于lncRNA本身不具有编码功能的这一特征,因此它发挥作用的原理通常是通过两种方式,即cis和trans方式作用于蛋白编码靶基因来实现;另外一种是基于lncRNA与mRNA碱基互补配对而产生作用。在此原理的基础上,利用靶基因预测工具LncTar对我们的lncRNA进行靶基因的预测。其中cis作用靶基因预测基本原理认为lncRNA的功能与其坐标临近的蛋白编码基因相关,于是在位于lncRNA上下游100kb左右位置的蛋白编码基因挑选出来,作为其靶基因。然而trans作用对于其相关靶基因预测与cis作用不同,其预测的基本原理是认为lncRNA的功能与本身和编码基因的位置关系无关,而与其共表达的蛋白编码基因相关,可以通过样本间lncRNA与蛋白编码基因的表达量相关性分析或共表达分析方法来预测其靶基因。进而根据(来源)相关注释信息,筛选与纤维强度相关的靶基因。
根据相关注释信息,本研究筛选了其中3个与纤维强度相关的靶基因,其功能注释如表1所示。
表1靶基因功能注释信息
步骤3、纤维强度相关靶基因的表达验证
提取及纯化植物材料总RNA,RNA反转录为cDNA,开展qRT-PCR实验。利用PrimerPremier5软件通过目的基因序列进行筛选设计出合适的引物,将设计得到的引物序列送往苏州泓迅生物科技有限公司进行引物合成,开展靶基因的表达验证。对于相关靶基因利用其黄褐棉同源基因序列,进行qRT-PCR验证,这3个靶基因的表达量在不同程度上都有明显的上调或下调(图2),且转录组数据结果表达趋势一致。
步骤4、纤维强度相关靶基因在棉花中的VIGS功能验证
在棉花中开展候选基因的基因克隆与基于病毒介导沉默(virus-induced genesilencing,VIGS)的功能验证。用VIGS技术获得了靶基因Gomus.D05G015100、Gomus.A05G281300、Gomus.A10G226800的基因沉默植株,沉默植株出现纤维强度显著降低的表型;认为这3个基因对棉纤维强度有着重要的作用。
图3为纤维强度相关靶基因在棉花中的VIGS功能验证实验中各植株苗期叶色表型图,图中第一列为阴性对照,第二列为阳性对照,第三列为实验组;A-C组目的基因分别为Gomus.A05G281300、Gomus.D05G015100、Gomus.A10G226800。pCLCrV::00为空载体,代表阴性对照;pCLCrV::GhPDS为沉默了八氢番茄红素脱氢酶(Phytoene desaturase,PDS)基因的植株,该基因沉默使得保护作用缺失,叶绿素的合成受到光漂白作用,导致其真叶出现漂白现象;因此,将沉默GhPDS基因的植株作为阳性对照。pCLCrV:Target gene代表分别沉默了3个目的基因实验组。
黄褐棉导入系IL9幼苗VIGS处理15天后发病,在图3中可以看到,阴性对照仅有轻微的萎蔫状态;阳性对照中沉默了八氢番茄红素脱氢酶(Phytoene desaturase,PDS)基因的植株,使得保护作用缺失,叶绿素的合成受到光漂白作用,导致其真叶出现漂白现象(图3),证明此次的VIGS菌液注射成功。确定菌液注射成功后,为确保棉花后续正常生长发育不受影响,将其从小盆移入直径21cm大盆中。
在pCLCrV::GhPDS出现白化表型后,取pCLCrV::Gomus.D05G015100、pCLCrV::Gomus.A05G281300、pCLCrV::Gomus.A10G226800沉默植株的叶片以及17dpa和21dpa的纤维,提取总RNA,反转录后进行了qRT-PCR实验(图4)。
图4VIGS沉默植株的基因表达验证;注:A:叶片;B:17dpa-棉纤维;C:21dpa-棉纤维。柱形图上方的星号表示统计显著差异(***P<0.01,***P<0.001,****P<0.0001)。
由图4可知目的基因的表达量无论是在叶片,还是在纤维中相比阴性对照都表现出现显著的下降。说明VIGS侵染是成功且正确的,目的基因被有效沉默了。
VIGS植株生长5-6个月后,取自然状态下成熟的棉纤维,每个候选基因各收取三个生物学重复,从纤维长度、纤维强度和纤维整齐度等方面进行相关表型的测量。
表2VIGS植株纤维表型测定值
(*均为与pCLCrV::00比较的差异显著性;*P<0.05,**P<0.01)
通过对VIGS植株相关表型测定(表2)后发现,与阴性对照相比,全部3个基因沉默植株的纤维强度值均显著低于阴性对照组。
- 棉纤维颜色相关基因及其在调控棉纤维颜色中的应用
- 与口腔癌脂质代谢相关基因的发现方法及基因的应用
