诊断胰腺癌的检测试剂和试剂盒及应用
文献发布时间:2024-04-18 20:00:50
技术领域
本申请涉及生物医学技术领域,具体而言,涉及诊断胰腺癌的检测试剂和试剂盒及其应用。
背景技术
胰腺癌是起源于胰腺导管上皮及腺泡细胞的恶性肿瘤,其起病隐匿,进展迅速,恶性程度极高,是预后最差的恶性肿瘤之一,胰腺导管腺癌约占胰腺癌的90%以上。近年来,胰腺癌的发病率和死亡率在全球范围内呈上升趋势。由于早期无症状及缺乏有效的诊断方法,胰腺癌在早期很难被诊断出来,绝大多数患者在初次发现癌症时其已发生转移,从而错失最佳的治疗时机。近40年来,胰腺癌的低存活率都没有得到显著的改善。因此,若能够在早期发现胰腺癌,提高手术率,有助于提高患者的生存率和生活质量。
目前,胰腺癌的诊断主要依赖于影像学检查,如计算机断层扫描(CT)、磁共振成像(MRI)、正电子发射计算机断层扫描(PET)、内镜超声(EUS)等。其中,计算机断层扫描(CT)是最常用的对胰腺癌进行诊断和分期的方法,而在内镜超声下对胰腺占位性病变进行穿刺活检,有利于鉴定病变的良恶性。考虑到上述方法或因碘造影剂而具有肾毒性风险,或具有辐射性,或为有创操作,因此,急需便捷的、无辐射性的、高灵敏度、高特异性的、无创的胰腺癌筛查和诊断方法。一些血液肿瘤标志物的诊断方法可以部分满足便捷、无辐射性的、微创的检测的要求,如临床上常用的与胰腺癌诊断相关的肿瘤标志物如CA19-9、CEA、CA125等。其中血清标志物CA19-9可用于胰腺癌的辅助诊断、复发监测等,具有一定的临床应用价值。但是,约有10%的胰腺癌患者不表达CA19-9蛋白,而且一些胆道感染或胆道梗阻等患者也会出现CA19-9的升高,因此,利用血液CA19-9的水平诊断或辅助诊断胰腺癌具有较大的局限性,其灵敏度和特异性较低。
因此,急需一种灵敏度高的、特异性高的血液标记物用于胰腺癌诊断或辅助诊断。
发明内容
基于此,有必要提供一种灵敏度高和特异性高的诊断胰腺癌的检测试剂和试剂盒,用于诊断胰腺癌,特别是胰腺癌的早期诊断。
具体技术方案如下:
本申请的第一方面,提供了一种诊断胰腺癌的检测试剂,所述检测试剂包括能够特异性检测受试者样本中目标区域的甲基化水平的试剂,以GRCh38.p14为参考,所述目标区域包括Chr6:30171742~30172465的全长或部分区域。
在其中一个实施例中,所述目标区域包括区域1和/或区域2:
所述区域1为Chr6:30171779-30172465,正链;
所述区域2为Chr6:30171742-30172444,负链。
在其中一个实施例中,所述目标区域包括区域3~区域12中的一个或多个:
所述区域3为Chr6:30171780-30171930,正链;
所述区域4为Chr6:30171845-30172007,正链;
所述区域5为Chr6:30172016-30172123,正链;
所述区域6为Chr6:30172107-30172226,正链;
所述区域7为Chr6:30172340-30172463,正链;
所述区域8为Chr6:30172289-30172444,负链;
所述区域9为Chr6:30172197-30172309,负链;
所述区域10为Chr6:30172090-30172190,负链;
所述区域11为Chr6:30171924-30172044,负链;
所述区域12为Chr6:30171783-30171879,负链。
在其中一个实施例中,所述试剂通过如下方法中的一种或多种实现对所述目标区的甲基化水平的检测:
甲基化特异性PCR、亚硫酸氢盐测序法、甲基化特异性微阵列法、全基因组甲基化测序法、焦磷酸测序法、甲基化特异性高效液相层析法、数字PCR法、甲基化特异性高分辨率溶解曲线法、甲基化敏感性限制性内切酶法和甲基化荧光定量PCR法。
在其中一个实施例中,所述试剂包括检测所述目标区域的甲基化水平的引物对;或还包括检测目标区域的检测探针。
在其中一个实施例中,所述试剂包括检测所述区域1和/或所述区域2的甲基化水平的甲基化引物对和非甲基化引物对:
检测区域1甲基化水平的甲基化引物对的核苷酸序列如SEQ ID NO.7和SEQ IDNO.8所示,非甲基化引物对的核苷酸序列如SEQ ID NO.9和SEQ ID NO.10所示;
检测区域2甲基化水平的甲基化引物对的核苷酸序列如SEQ ID NO.11和SEQ IDNO.12所示,非甲基化引物对的核苷酸序列如SEQ ID NO.13和SEQ ID NO.14所示。
在其中一个实施例中,所述试剂包括检测区域3~区域12中的一个或多个区域的甲基化水平的引物对及其对应的检测探针:
检测区域3的甲基化水平的引物对的核苷酸序列如SEQ ID NO.35和SEQ ID NO.36所示,检测探针的核苷酸序列如SEQ ID NO.37所示;
检测区域4的甲基化水平的引物对的核苷酸序列如SEQ ID NO.38和SEQ ID NO.39所示,检测探针的核苷酸序列如SEQ ID NO.40所示;
检测区域5的甲基化水平的引物对的核苷酸序列如SEQ ID NO.41和SEQ ID NO.42所示,检测探针的核苷酸序列如SEQ ID NO.43所示;
检测区域6的甲基化水平的引物对的核苷酸序列如SEQ ID NO.44和SEQ ID NO.45所示,检测探针的核苷酸序列如SEQ ID NO.46所示;
检测区域7的甲基化水平的引物对的核苷酸序列如SEQ ID NO.47和SEQ ID NO.48所示,检测探针的核苷酸序列如SEQ ID NO.49所示;
检测区域8的甲基化水平的引物对的核苷酸序列如SEQ ID NO.50和SEQ ID NO.51所示,检测探针的核苷酸序列如SEQ ID NO.52所示;
检测区域9的甲基化水平的引物对的核苷酸序列如SEQ ID NO.53和SEQ ID NO.54所示,检测探针的核苷酸序列如SEQ ID NO.55所示;
检测区域10的甲基化水平的引物对的核苷酸序列如SEQ ID NO.56和SEQ IDNO.57所示,检测探针的核苷酸序列如SEQ ID NO.58所示;
检测区域11的甲基化水平的引物对的核苷酸序列如SEQ ID NO.59和SEQ IDNO.60所示,检测探针的核苷酸序列如SEQ ID NO.61所示;
检测区域12的甲基化水平的引物对的核苷酸序列如SEQ ID NO.62和SEQ IDNO.63所示,检测探针的核苷酸序列如SEQ ID NO.64所示。
在其中一个实施例中,所述样本包括血液样本、组织样本或细胞样本。
本申请的第二方面,提供了一种诊断胰腺癌的试剂盒,所述试剂盒包括上述任一项所述的检测试剂。
在其中一个实施例中,所述试剂盒还包括核酸提取试剂、甲基化转化试剂、核酸纯化试剂、PCR反应试剂、测序试剂、质控品中的至少一种。
本申请的第三方面,提供了能够特异性检测受试者样本中目标区域的甲基化水平的试剂在制备胰腺癌的诊断产品中的应用,以GRCh38.p14为参考,所述目标区域包括Chr6:30171742-30172465的全长或部分区域。
相对于现有技术,本申请具备如下有益效果:
本申请提供用于诊断胰腺癌的检测试剂和试剂盒,对Chr6:30171742~30172465的全长或部分区域的甲基化水平进行检测,可用于胰腺癌的诊断或辅助诊断,其检测血液样本的灵敏度范围为73.53%~86.76%,特异性范围为92.05%~97.73%,具有较高的灵敏度和特异性,对于胰腺癌的早期治疗和提高患者生存率具有重要意义。
附图说明
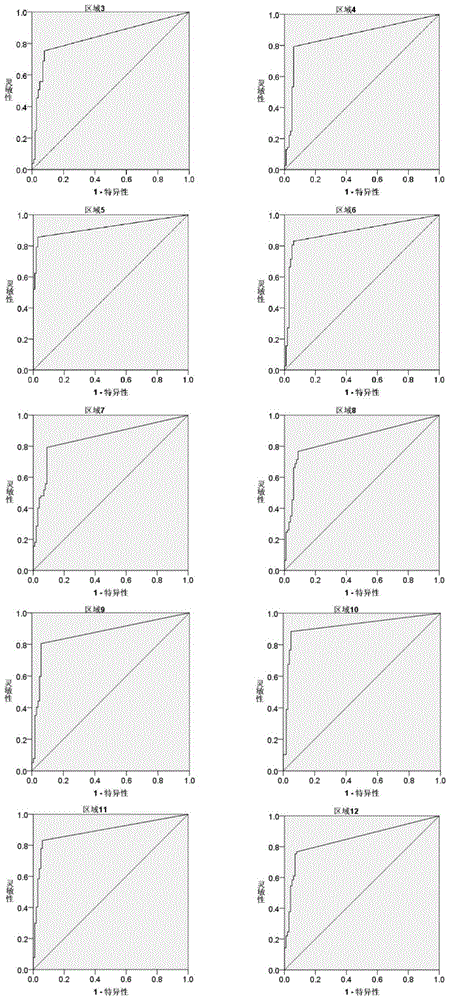
图1为区域3~区域12诊断胰腺癌血液样本和健康人血液样本的ROC曲线。
具体实施方式
为了便于理解本发明,下面将对本发明进行更全面的描述。但是,本发明可以以许多不同的形式来实现,并不限于本文所描述的实施例。相反地,提供这些实施例的目的是使本发明公开内容更加透彻全面。
除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。
术语解释
术语“及/或”或“和/或”均是指包括一个或多个相关的所列项目的任意的和所有的组合。
术语“多个”指两个以上;“多种”指两种以上;“以上”与数字结合表示数量时包括本数,例如“两个以上”包括两个。
术语“诊断”包括辅助诊断、复发风险评估、癌变风险和癌变程度的评估、预后判断等方面。
术语“基因”是指编码产生氨基酸多肽链的DNA区段,它包括参与基因转录/翻译和转录/翻译调控的位于编码区和非编码区的序列,以及外显子和内含子序列。
术语“寡核苷酸”或“多核苷酸”或“核苷酸”或“核酸”是指具有两个或更多个脱氧核糖核苷酸或核糖核苷酸的分子,优选多于三个,并且通常多于十个。确切的大小将取决于许多因素,而所述因素又取决于寡核苷酸的最终功能或用途。寡核苷酸可以任何方式产生,包括化学合成、DNA复制、逆转录或其组合。DNA的典型脱氧核糖核苷酸是胸腺嘧啶、腺嘌呤、胞嘧啶和鸟嘌呤。RNA的典型核糖核苷酸是尿嘧啶、腺嘌呤、胞嘧啶和鸟嘌呤。
术语“甲基化”为DNA化学修饰的一种形式,能够在不改变DNA序列的前提下,改变遗传表现。DNA甲基化是指在DNA甲基转移酶的作用下,在基因组CpG二核苷酸的胞嘧啶第5号碳位共价结合一个甲基基团。DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达。
术语“甲基化水平”指的是一段DNA序列中一个或多个CpG二核苷酸中的胞嘧啶是否发生甲基化、或发生甲基化的频率/比例/百分数,既代表定性的概念又代表定量的概念。在实际应用中,可根据实际情况采用不同的检测指标比较DNA甲基化水平。如在一些情况下,可根据样本检测的Ct值进行比较;在一些情况下,可计算样本中基因甲基化的比例,即甲基化分子数/(甲基化分子数+非甲基化分子数)×100,然后再进行比较;在一些情况下,还需要对各个指标进行统计学上的分析整合,得出最终的判定指标。
术语“引物”是指可以用在扩增方法(例如聚合酶链式反应PCR)中,基于与目标基因或其一部分区域相对应的多核苷酸序列来扩增目的序列的寡核苷酸。通常,用于扩增多核苷酸序列的PCR引物中的至少一个对于该多核苷酸序列是序列特异性的。引物的确切长度取决于很多因素,包括温度、引物来源以及所用方法等。例如,对于诊断和预后应用,根据靶序列的复杂度,寡核苷酸引物通常含有至少10、15、20、25或更多个核苷酸,但是也可以含有更少的核苷酸。在本公开内容中,术语“引物”是指能与目标DNA分子的双链杂交或能与目标DNA分子中位于待扩增核苷酸序列两翼的区域杂交的一对引物。
术语“Taqman探针”是指包含5’荧光基团和3’淬灭基团的一段寡核苷酸序列。当探针与DNA上的相应位点结合时,因为荧光基团附近存在淬灭基团,探针不会发出荧光。在扩增过程中,如果探针与被扩增的链结合,DNA聚合酶(如Taq酶)的5’-3’核酸外切酶活性会消化探针,荧光基团远离淬灭基团,其能量不被吸收,即产生荧光信号。每经过一个PCR循环,荧光信号也和目的片段一样,有一个同步的指数增长的过程。
术语“桑格尔测序”即一代测序,其反应系统包含:目标片段、四种脱氧核糖核苷酸(dNTP)、DNA聚合酶,引物等,还需要加入不同荧光基团标记的4种双脱氧核糖核苷酸(ddNTP)。由于ddNTP缺乏延伸所需要的3’-OH基团,使延长的寡聚核苷酸选择性地在G、A、T或C处终止,通过光激发将四种光波长信号转化为电脑可识别的电信号,根据反应管中最后掺入的ddNTP的荧光信号判断目的DNA序列。
术语“甲基化特异性PCR”是目前研究甲基化最敏感的实验技术之一,能发现最低约50pg DNA的甲基化。单链DNA经亚硫酸氢盐转化后,所有未甲基化的胞嘧啶脱氨转变为尿嘧啶,而CpG位点中甲基化的胞嘧啶保持不变,因此分别设计两对针对甲基化和非甲基化序列的引物,通过PCR扩增即可将甲基化与非甲基化的DNA序列区分开来。在本公开内容中,进行实时定量甲基化特异性PCR时加入甲基化引物,若Ct值满足所述要求(例如在组织样本中Ct≤38),则表明目标序列是甲基化的。
术语“AUC”是“曲线下面积”的缩写。具体地,其是指接受者操作特征(ROC)曲线下面积。ROC曲线是诊断测试的不同可能切割点的真阳性率相比于假阳性率的图。其取决于所选择切割点的灵敏度与特异性之间的权衡(灵敏度的任何增加将伴随着特异性的降低)。ROC曲线下面积(AUC)是诊断测试准确度的度量(面积越大越好;最佳值是1;随机测试将具有位于对角线上的ROC曲线,面积为0.5)。
本申请通过亚硫酸氢盐测序方法或荧光定量甲基化特异性PCR法检测受试者样本中TRIM15基因位于Chr6:30171742-30172465区域的DNA的甲基化水平,用于胰腺癌的无创或微创诊断,且该方法灵敏度和特异性高。
因此,本申请一实施方式提供了一种诊断胰腺癌的检测试剂,包括能够特异性检测生物样本中目标区域的甲基化水平的试剂,目标区域包括Chr6:30171742-30172465的全长或部分区域。
在一个具体示例中,目标区域包括区域1和/或区域2;具体的,区域1为Chr6:30171779-30172465(正链),区域2为Chr6:30171742-30172444(负链)。
在一个具体示例中,可采用亚硫酸氢盐测序方法检测目标区域的甲基化水平,其中,区域1诊断胰腺癌的灵敏度为84.42%、特异性为94.57%;区域2诊断胰腺癌的灵敏度为81.08%、特异性为93.41%。在一个具体示例中,目标区域包括区域3~区域12中的一个或多个;具体的,区域3为Chr6:30171780-30171930,正链;区域4为Chr6:30171845-30172007,正链;区域5为Chr6:30172016-30172123,正链;区域6为Chr6:30172107-30172226,正链;区域7为Chr6:30172340-30172463,正链;区域8为Chr6:30172289-30172444,负链;区域9为Chr6:30172197-30172309,负链;区域10为Chr6:30172090-30172190,负链;区域11为Chr6:30171924-30172044,负链;区域12为Chr6:30171783-30171879,负链。
在一个具体示例中,可采用甲基化荧光定量PCR的方法检测目标区域的甲基化水平,其中,区域3-12诊断胰腺癌的灵敏度均高于75%,其诊断的特异性均高于91%,其ROC曲线下面积(AUC)值均大于0.83。
优选地,区域5和区域10的诊断性能最佳,其ROC曲线的AUC值都大于0.9,其中区域5诊断胰腺癌血液样本的灵敏度为85.7%,特异性为97.1%;区域10诊断胰腺癌血液样本的灵敏度为88.3%,特异性为95.1%。
需要说明的是,在本文中,如未特殊说明,染色体上的位置均是以GRCh38.p14为参考;此外,因为染色体上的DNA是由正链和负链组成的双链结构,对于用染色体位置表示的区域,如果未指明是DNA的正链还是负链,则表示既可以是该区域的DNA的正链,也可以是该区域的DNA的负链,还可以是该区域的DNA的正负两条链。例如,若区域Chr6:30171742~30172465被描述为“Chr6:30171742~30172465”,则表示可以是Chr6:30171742~30172465区域内的DNA的正链,也可以是Chr6:30171742~30172465区域内的DNA的负链,还可以是Chr6:30171742~30172465区域内的DNA的正、负两条链。
在一个具体示例中,样本可选自血液样本、组织样本或细胞样本,也可以为其他生物样本如尿液样本、唾液样本等。
在一个具体示例中,该试剂通过以下方法中的一种或多种实现对目标区的甲基化水平的检测:甲基化特异性PCR、亚硫酸氢盐测序法、甲基化特异性微阵列法、全基因组甲基化测序法、焦磷酸测序法、甲基化特异性高效液相层析法、数字PCR法、甲基化特异性高分辨率溶解曲线法、甲基化敏感性限制性内切酶法和甲基化荧光定量PCR法。
在一个具体示例中,该试剂包括检测目标区域的甲基化水平的引物对;还可以包括检测目标区域的检测探针。
在一个具体示例中,该试剂包括检测区域1和/或区域2甲基化水平的甲基化引物对和非甲基化引物对:检测区域1的甲基化引物对的核苷酸序列如SEQ ID NO.7和SEQ IDNO.8所示,非甲基化引物对的核苷酸序列如SEQ ID NO.9和SEQ ID NO.10所示;检测区域2甲基化水平的甲基化引物对的核苷酸序列如SEQ ID NO.11和SEQ ID NO.12所示,非甲基化引物对的核苷酸序列如SEQ ID NO.13和SEQ ID NO.14所示。
在另一个具体示例中,该试剂包括检测区域3~区域12中的一个或多个区域的甲基化水平的引物对及其对应的检测探针;具体的,检测区域3的甲基化水平的引物对的核苷酸序列如SEQ ID NO.35和SEQ ID NO.36所示,检测探针的核苷酸序列如SEQ ID NO.37所示;检测区域4的甲基化水平的引物对的核苷酸序列如SEQ ID NO.38和SEQ ID NO.39所示,检测探针的核苷酸序列如SEQ ID NO.40所示;检测区域5的甲基化水平的引物对的核苷酸序列如SEQ ID NO.41和SEQ ID NO.42所示,检测探针的核苷酸序列如SEQ ID NO.43所示;检测区域6的甲基化水平的引物对的核苷酸序列如SEQ ID NO.44和SEQ ID NO.45所示,检测探针的核苷酸序列如SEQ ID NO.46所示;检测区域7的甲基化水平的引物对的核苷酸序列如SEQ ID NO.47和SEQ ID NO.48所示,检测探针的核苷酸序列如SEQ ID NO.49所示;检测区域8的甲基化水平的引物对的核苷酸序列如SEQ ID NO.50和SEQ ID NO.51所示,检测探针的核苷酸序列如SEQ ID NO.52所示;检测区域9的甲基化水平的引物对的核苷酸序列如SEQ ID NO.53和SEQ ID NO.54所示,检测探针的核苷酸序列如SEQ ID NO.55所示;检测区域10的甲基化水平的引物对的核苷酸序列如SEQ ID NO.56和SEQ ID NO.57所示,检测探针的核苷酸序列如SEQ ID NO.58所示;检测区域11的甲基化水平的引物对的核苷酸序列如SEQ ID NO.59和SEQ ID NO.60所示,检测探针的核苷酸序列如SEQ ID NO.61所示;检测区域12的甲基化水平的引物对的核苷酸序列如SEQ ID NO.62和SEQ ID NO.63所示,检测探针的核苷酸序列如SEQ ID NO.64所示。
进一步,本申请又一实施方式提供了一种诊断胰腺癌的试剂盒,所述试剂盒包括上述任一项的检测试剂。
在一个具体示例中,试剂盒还包括核酸提取试剂、甲基化转化试剂、核酸纯化试剂、PCR反应试剂、测序试剂、质控品中的至少一种。核酸提取试剂用于提取样本中的核酸分子。甲基化转化试剂用于将DNA中未发生甲基化的胞嘧啶脱氨基转变成尿嘧啶,而甲基化的胞嘧啶保持不变;可选地,甲基化转化试剂包括亚硫酸氢盐。核酸纯化试剂用于核酸分子的纯化。PCR反应试剂可包括扩增缓冲液、dNTPs、DNA聚合酶和Mg
此外,本申请一实施方式还提供了一种胰腺癌的诊断方法,该方法通过检测受试者样本中目标区域的甲基化水平进行胰腺癌的诊断。具体地,目标区域如上文所述。
在一个具体示例中,检测受试者样本(受试者的样本如血液样本、细胞样本或组织样本)中目标区域的甲基化水平,若受试者样本中目标区域的甲基化水平显著高于健康人的样本,则认为受试者患有胰腺癌,该方法诊断胰腺癌具有高的灵敏度和特异性。
在一个具体示例中,检测受试者样本(受试者的样本如血液样本、细胞样本或组织样本)中目标区域的甲基化水平,计算扩增所有的样本目标区域的ΔCt值,根据ΔCt值判断样本在该目标区域的甲基化状态。具体地,目标区域如上文所述。
具体的判断标准为:若某一样本的ΔCt值小于或等于截断值,则该样本在此区域为甲基化阳性即判定为胰腺癌阳性;若某一样本的ΔCt值大于截断值,则该样本为甲基化阴性即判定为胰腺癌阴性。其中,截断值可通过对胰腺癌的训练集样本进行ROC曲线分析来测定。优选地,各个区域的截断值如表11所示。该方法诊断同样具有高的灵敏度和特异性。
更进一步,本申请一实施方式还提供了能够特异性检测受试者样本中目标区域的甲基化水平的试剂在制备胰腺癌的诊断产品中的应用,以GRCh38.p14为参考,目标区域包括Chr6:30171742-30172465的全长或部分区域。
在一个具体示例中,诊断产品包括但不限于诊断胰腺癌的试剂、试剂盒、试纸、诊断系统等。其中,诊断系统包括检测部分和分析部分。检测部分检测目标区域的甲基化水平,分析部分分析甲基化水平,并判断是否患有胰腺癌。
本申请中所涉及的胰腺癌均指胰腺导管腺癌。
具体实施例
下面将结合实施例对本申请的实施方案进行详细描述。应理解,这些实施例仅用于说明本申请而不用于限制本申请的范围。下列实施例中未注明具体条件的实验方法,优先参考本申请中给出的指引,还可以按照本领域的实验手册或常规条件,还可以按照制造厂商所建议的条件,或者参考本领域已知的实验方法。
下述的具体实施例中,涉及原料组分的量度参数,如无特别说明,可能存在称量精度范围内的细微偏差。涉及温度和时间参数,允许仪器测试精度或操作精度导致的可接受的偏差。
实施例1
通过亚硫酸氢盐测序法分析胰腺癌组织样本中TRIM15基因位于Chr6:30171742-30172465区域的DNA的甲基化水平及其诊断胰腺癌的性能。
1.样本的收集
共收集了86例经病理活检确诊为胰腺癌的组织样本和102例进行常规体检的健康人抗凝血血液样本。胰腺组织样本均为福尔马林浸泡、石蜡包埋的组织。所有样本的收集过程经过伦理委员会的审批,所有志愿者都签署了知情同意书。
2.样本DNA的提取
对于胰腺组织样本,采用QIAamp DNA FFPE Tissue Kit(Cat:56404)提取组织DNA,具体操作参见试剂盒说明书。
对于抗凝血血液样本,经离心后,取白细胞层用于基因组DNA的提取。采用天根生化科技(北京)有限公司的离心柱型血液/细胞/组织基因组DNA提取试剂盒(DP304)进行白细胞基因组DNA提取,具体操作参见试剂盒说明书。
3.样本DNA的转化和纯化
样本DNA的转化和纯化所用试剂盒均为武汉艾米森生命科技有限公司核酸转化试剂(鄂汉械备20200843号),具体操作步骤参见试剂盒说明书。
4.PCR扩增及测序
以TRIM15基因位于Chr6:30171742-30172465区域的DNA分子作为参考序列,分别以该区域DNA分子经亚硫酸氢盐转化后的正链、负链作为模板,设计测序引物。所述测序引物包括甲基化测序引物和非甲基化测序引物,分别用来扩增甲基化的模板和非甲基化的模板。筛选甲基化引物对和非甲基化引物对的配比,以保证在一个PCR反应体系中同时加入甲基化引物对和非甲基化引物对时,当模板中存在大于等于1%的甲基化序列时,即可扩增得到甲基化的产物,仅当模板为非甲基化序列时,扩增产物为非甲基化产物。TRIM15基因位于Chr6:30171779-30172465区域(区域1)正链DNA序列如SEQ ID NO.1所示,该区域完全甲基化的序列经亚硫酸氢盐转化后如SEQ ID NO.2所示,该区域完全未甲基化的序列经亚硫酸氢盐转化后如SEQ ID NO.3所示。TRIM15基因位于Chr6:30171742-30172444区域(区域2)负链DNA序列如SEQ ID NO.4所示,该区域完全甲基化的序列经亚硫酸氢盐转化后如SEQ IDNO.5所示,该区域完全未甲基化的序列经亚硫酸氢盐转化后如SEQ ID NO.6所示。
SEQ ID NO.1:
GGAACATCTGTGGTTGCCGCCCGCTGTTGATGTCTGCGCGCTCCTCCCTCTAGGGGTCATCACTCTGGACCCTCAGACCGCCAGCCGGAGCCTGGTTCTCTCGGAAGACAGGAAGTCAGTGAGGTACACCCGGCAGAAGAAGAGCCTGCCAGACAGCCCCCTGCGCTTCGACGGCCTCCCGGCGGTTCTGGGCTTCCCGGGCTTCTCCTCCGGGCGCCACCGCTGGCAGGTTGACCTGCAGCTGGGCGACGGCGGCGGCTGCACGGTGGGGGTGGCCGGGGAGGGGGTGAGGAGGAAGGGAGAGATGGGACTCAGCGCCGAGGACGGCGTCTGGGCCGTGATCATCTCGCACCAGCAGTGCTGGGCCAGCACCTCCCCGGGCACCGACCTGCCGCTGAGCGAGATCCCGCGCGGCGTGAGAGTCGCCCTGGACTACGAGGCGGGGCAGGTGACCCTCCACAACGCCCAGACCCAGGAGCCCATCTTCACCTTCACTGCCTCTTTCTCCGGCAAAGTCTTCCCTTTCTTTGCCGTCTGGAAAAAAGGTTCCTGCCTTACGCTGAAAGGCTGAAGTGGGGCGCGCGAAGGGCGGCGAAGCGGAGACGGCGGCTCTCCGGGATCCAGCTCCGCCCCTGGCCAGTGTGCGGCCCGGGGGCTCCCTGTGCCCGCGTGAGGCGAGAGAACAGG。
SEQ ID NO.2:
GGAATATTTGTGGTTGTCGTTCGTTGTTGATGTTTGCGCGTTTTTTTTTTTAGGGGTTATTATTTTGGATTTTTAGATCGTTAGTCGGAGTTTGGTTTTTTCGGAAGATAGGAAGTTAGTGAGGTATATTCGGTAGAAGAAGAGTTTGTTAGATAGTTTTTTGCGTTTCGACGGTTTTTCGGCGGTTTTGGGTTTTTCGGGTTTTTTTTTCGGGCGTTATCGTTGGTAGGTTGATTTGTAGTTGGGCGACGGCGGCGGTTGTACGGTGGGGGTGGTCGGGGAGGGGGTGAGGAGGAAGGGAGAGATGGGATTTAGCGTCGAGGACGGCGTTTGGGTCGTGATTATTTCGTATTAGTAGTGTTGGGTTAGTATTTTTTCGGGTATCGATTTGTCGTTGAGCGAGATTTCGCGCGGCGTGAGAGTCGTTTTGGATTACGAGGCGGGGTAGGTGATTTTTTATAACGTTTAGATTTAGGAGTTTATTTTTATTTTTATTGTTTTTTTTTTCGGTAAAGTTTTTTTTTTTTTTGTCGTTTGGAAAAAAGGTTTTTGTTTTACGTTGAAAGGTTGAAGTGGGGCGCGCGAAGGGCGGCGAAGCGGAGACGGCGGTTTTTCGGGATTTAGTTTCGTTTTTGGTTAGTGTGCGGTTCGGGGGTTTTTTGTGTTCGCGTGAGGCGAGAGAATAGG。
SEQ ID NO.3:
GGAATATTTGTGGTTGTTGTTTGTTGTTGATGTTTGTGTGTTTTTTTTTTTAGGGGTTATTATTTTGGATTTTTAGATTGTTAGTTGGAGTTTGGTTTTTTTGGAAGATAGGAAGTTAGTGAGGTATATTTGGTAGAAGAAGAGTTTGTTAGATAGTTTTTTGTGTTTTGATGGTTTTTTGGTGGTTTTGGGTTTTTTGGGTTTTTTTTTTGGGTGTTATTGTTGGTAGGTTGATTTGTAGTTGGGTGATGGTGGTGGTTGTATGGTGGGGGTGGTTGGGGAGGGGGTGAGGAGGAAGGGAGAGATGGGATTTAGTGTTGAGGATGGTGTTTGGGTTGTGATTATTTTGTATTAGTAGTGTTGGGTTAGTATTTTTTTGGGTATTGATTTGTTGTTGAGTGAGATTTTGTGTGGTGTGAGAGTTGTTTTGGATTATGAGGTGGGGTAGGTGATTTTTTATAATGTTTAGATTTAGGAGTTTATTTTTATTTTTATTGTTTTTTTTTTTGGTAAAGTTTTTTTTTTTTTTGTTGTTTGGAAAAAAGGTTTTTGTTTTATGTTGAAAGGTTGAAGTGGGGTGTGTGAAGGGTGGTGAAGTGGAGATGGTGGTTTTTTGGGATTTAGTTTTGTTTTTGGTTAGTGTGTGGTTTGGGGGTTTTTTGTGTTTGTGTGAGGTGAGAGAATAGG。
SEQ ID NO.4:
GGCACAGGGAGCCCCCGGGCCGCACACTGGCCAGGGGCGGAGCTGGATCCCGGAGAGCCGCCGTCTCCGCTTCGCCGCCCTTCGCGCGCCCCACTTCAGCCTTTCAGCGTAAGGCAGGAACCTTTTTTCCAGACGGCAAAGAAAGGGAAGACTTTGCCGGAGAAAGAGGCAGTGAAGGTGAAGATGGGCTCCTGGGTCTGGGCGTTGTGGAGGGTCACCTGCCCCGCCTCGTAGTCCAGGGCGACTCTCACGCCGCGCGGGATCTCGCTCAGCGGCAGGTCGGTGCCCGGGGAGGTGCTGGCCCAGCACTGCTGGTGCGAGATGATCACGGCCCAGACGCCGTCCTCGGCGCTGAGTCCCATCTCTCCCTTCCTCCTCACCCCCTCCCCGGCCACCCCCACCGTGCAGCCGCCGCCGTCGCCCAGCTGCAGGTCAACCTGCCAGCGGTGGCGCCCGGAGGAGAAGCCCGGGAAGCCCAGAACCGCCGGGAGGCCGTCGAAGCGCAGGGGGCTGTCTGGCAGGCTCTTCTTCTGCCGGGTGTACCTCACTGACTTCCTGTCTTCCGAGAGAACCAGGCTCCGGCTGGCGGTCTGAGGGTCCAGAGTGATGACCCCTAGAGGGAGGAGCGCGCAGACATCAACAGCGGGCGGCAACCACAGATGTTCCTAGAGCAGGCAACGCACGTGGGAGGCAGAAGGAGTAC。
SEQ ID NO.5:
GGTATAGGGAGTTTTCGGGTCGTATATTGGTTAGGGGCGGAGTTGGATTTCGGAGAGTCGTCGTTTTCGTTTCGTCGTTTTTCGCGCGTTTTATTTTAGTTTTTTAGCGTAAGGTAGGAATTTTTTTTTTAGACGGTAAAGAAAGGGAAGATTTTGTCGGAGAAAGAGGTAGTGAAGGTGAAGATGGGTTTTTGGGTTTGGGCGTTGTGGAGGGTTATTTGTTTCGTTTCGTAGTTTAGGGCGATTTTTACGTCGCGCGGGATTTCGTTTAGCGGTAGGTCGGTGTTCGGGGAGGTGTTGGTTTAGTATTGTTGGTGCGAGATGATTACGGTTTAGACGTCGTTTTCGGCGTTGAGTTTTATTTTTTTTTTTTTTTTTATTTTTTTTTCGGTTATTTTTATCGTGTAGTCGTCGTCGTCGTTTAGTTGTAGGTTAATTTGTTAGCGGTGGCGTTCGGAGGAGAAGTTCGGGAAGTTTAGAATCGTCGGGAGGTCGTCGAAGCGTAGGGGGTTGTTTGGTAGGTTTTTTTTTTGTCGGGTGTATTTTATTGATTTTTTGTTTTTCGAGAGAATTAGGTTTCGGTTGGCGGTTTGAGGGTTTAGAGTGATGATTTTTAGAGGGAGGAGCGCGTAGATATTAATAGCGGGCGGTAATTATAGATGTTTTTAGAGTAGGTAACGTACGTGGGAGGTAGAAGGAGTAT。
SEQ ID NO.6:
GGTATAGGGAGTTTTTGGGTTGTATATTGGTTAGGGGTGGAGTTGGATTTTGGAGAGTTGTTGTTTTTGTTTTGTTGTTTTTTGTGTGTTTTATTTTAGTTTTTTAGTGTAAGGTAGGAATTTTTTTTTTAGATGGTAAAGAAAGGGAAGATTTTGTTGGAGAAAGAGGTAGTGAAGGTGAAGATGGGTTTTTGGGTTTGGGTGTTGTGGAGGGTTATTTGTTTTGTTTTGTAGTTTAGGGTGATTTTTATGTTGTGTGGGATTTTGTTTAGTGGTAGGTTGGTGTTTGGGGAGGTGTTGGTTTAGTATTGTTGGTGTGAGATGATTATGGTTTAGATGTTGTTTTTGGTGTTGAGTTTTATTTTTTTTTTTTTTTTTATTTTTTTTTTGGTTATTTTTATTGTGTAGTTGTTGTTGTTGTTTAGTTGTAGGTTAATTTGTTAGTGGTGGTGTTTGGAGGAGAAGTTTGGGAAGTTTAGAATTGTTGGGAGGTTGTTGAAGTGTAGGGGGTTGTTTGGTAGGTTTTTTTTTTGTTGGGTGTATTTTATTGATTTTTTGTTTTTTGAGAGAATTAGGTTTTGGTTGGTGGTTTGAGGGTTTAGAGTGATGATTTTTAGAGGGAGGAGTGTGTAGATATTAATAGTGGGTGGTAATTATAGATGTTTTTAGAGTAGGTAATGTATGTGGGAGGTAGAAGGAGTAT。
分别以SEQ ID NO.2、SEQ ID NO.3和SEQ ID NO.5、SEQ ID NO.6为模板,设计TRIM15基因DNA正、负链对应的甲基化测序引物对和非甲基化测序引物对,测序引物对的核苷酸序列如表1所示。
表1、测序引物的核苷酸序列
随后进行PCR反应,PCR体系的配方如表2所示,反应体系中的用到的引物对(表1)由上海生工生物合成,用到的模板为从组织样本或白细胞样本中提取的且经亚硫酸氢盐转化的DNA,其它组分均购自TAKARA(Cat:R010A)。PCR体系在冰上配置,配置结束后按照表3所示的反应程序进行扩增。将扩增产物送公司进行桑格尔测序,测序引物同扩增引物,包括甲基化测序引物对和非甲基化测序引物对。
表2、PCR反应体系
表3、PCR扩增程序
5.结果分析
对测序成功的样本,分析每个样本各个扩增子中CpG二核苷酸中胞嘧啶的甲基化情况。若测序结果中CpG二核苷酸中胞嘧啶显示为胸腺嘧啶,则其为非甲基化的,若测序结果中CpG二核苷酸中胞嘧啶仍显示为胞嘧啶,则其为完全甲基化化的,若测序结果中CpG二核苷酸中胞嘧啶为双峰,既有胞嘧啶又有胸腺嘧啶,则其为部分甲基化的。如果一个扩增子中95%以上的CpG二核苷酸中胞嘧啶是甲基化的(包括完全甲基化和部分甲基化),则认为此样本在该扩增区域是甲基化阳性的;否则认为此样本在该扩增区域为甲基化阴性。
86例组织样本和102例健康人白细胞样本的测序结果及甲基化状态如表4所示。在86例组织样本中,对于区域1,共有77例样本测序成功;对于区域2,共有74例样本测序成功。在102例健康人白细胞样本中,对于区域1,共有92例样本测序成功;对于区域2,共有91例样本测序成功。
表4、组织样本和白细胞样本中CpG位点在区域1和2中的甲基化比例
/>
/>
桑格尔测序结果显示,胰腺癌组织样本中TRIM15基因位于Chr6:30171742-30172465区域的DNA的甲基化水平显著高于其在健康人白细胞中的甲基化水平。
根据表4中的数据分别统计利用区域1、区域2的甲基化水平检测胰腺癌组织样本和健康人白细胞样本的灵敏度和特异性。性能分析所用的样本均为测序成功的样本。灵敏度为病理结果为阳性的样本中甲基化阳性的比例,特异性为病理结果为阴性的样本中甲基化阴性的比例。利用桑格尔测序的方法,通过检测区域1和区域2的甲基化水平诊断胰腺癌组织样本、健康人白细胞样本的灵敏度和特异性如表5所示。
表5、亚硫酸氢盐测序法检测胰腺癌组织样本的性能
由表5可以看出,利用桑格尔测序的方法,通过检测TRIM15基因位于Chr6:30171742-30172465区域的DNA正、负链的甲基化水平,可以有效区分胰腺癌组织样本和健康人白细胞样本。具体地,区域1检测胰腺癌组织样本的灵敏度为84.42%,其检测健康人白细胞样本的特异性为94.57%;区域2检测胰腺癌组织样本的灵敏度为81.08%,其检测健康人白细胞样本的特异性为93.41%。
实施例2
通过荧光定量甲基化特异性PCR法分析血浆样本中TRIM15基因位于Chr6:30171742-30172465的区域诊断胰腺癌的性能。
本实施例提供了一种基于荧光定量甲基化特异性PCR(qMSP)的方法检测血液样本中Chr6:30171742-30172465区域的子区域的甲基化水平的方法,可以根据Ct值来判断血液样本的甲基化状态,进而判断样本是否为癌症样本,该方法简单、便捷、易于操作、可以在半个工作日内出示检测报告。具体的实验流程及检测效果如下所示:
以本实施例中所用到的数据作为训练集。
1.样本的收集
胰腺癌患者血液样本的收集:共收集了77例经组织活检确诊为胰腺癌的患者的血液样本,每人收集约10mL。所有血液样本的收集过程经过伦理委员会的审批,所有志愿者都签署了知情同意书,所有样本均采用匿名化处理。
使用实施例1中收集的健康人血浆样本作为胰腺癌患者血浆样本的对照,健康人抗凝血血液样本的收集同实施例1。
2.样本DNA的提取
实施例1中收集的抗凝血样本经离心后,收集血浆层,用于提取血浆游离DNA。使用天根生化科技(北京)有限公司的磁珠法血清/血浆游离DNA(cfDNA)提取试剂盒(DP709)进行血浆cfDNA提取,具体操作按照试剂盒说明书进行。
3.样本DNA的转化和纯化步骤同实施例1。
4.荧光定量甲基化特异性PCR
由于血浆cfDNA片段较短,且在170bp处富集,又考虑到荧光定量PCR的扩增效率,因此在本实施例中,难以在血浆样本中直接扩增TRIM15基因位于Chr6:30171742-30172465位置的全长区域。因此,将TRIM15基因位于Chr6:30171742-30172465的区域分为若干个子区域(子区域的DNA片段长度均不超过170bp),分别检测各个样本在各个子区域中的甲基化状态,进而区分癌变和健康样本。
对于各个子区域,以经亚硫酸氢盐转化后的序列作为模板,分别设计用于扩增各个子区域的引物对和检测探针,利用qMSP法检测各样本在各个子区域中的甲基化状态。TRIM15基因位于Chr6:30171742-30172465的子区域及其经亚硫酸氢盐转化后的序列如表6所示。可以理解的是,TRIM15基因位于Chr6:30171742-30172465的子区域也可以是未列举的其它位于该区域的子区域。用于扩增和检测各个子区域的引物对和探针的核苷酸序列如表7所示,检测引物对和探针可识别的甲基化的胞嘧啶位点如表8所示。
表6、TRIM15基因位于Chr6:30171742-30172465的子区域序列
/>
/>
表7、扩增和检测各个子区域的引物对和检测探针的核苷酸序列
表8、子区域的检测引物对及检测探针可识别的甲基化的胞嘧啶位点
/>
在进行qMSP反应时,PCR反应体系的配方如表9所示,反应体系中用到的引物对和检测探针由上海生工生物合成,用到的模板为从血浆样本中提取的且经亚硫酸氢盐转化的DNA,其它组分均购自Invitrogen(Cat:14966005)。从表9可以看出,在每个PCR管中,除加入目标区域的引物对和检测探针以外,还需加入内参基因ACTB的引物对和检测探针。检测探针均为TaqMan探针,目标区域的检测探针5’端的荧光报告基团为FAM,3’端的荧光淬灭基团均为MGB,ACTB基因的检测探针5’端的荧光报告基团为VIC,3’端的荧光淬灭基团为BHQ-1。PCR反应体系配置结束后按照表10所示的反应程序进行扩增。
表9、qMSP反应体系
表10、qMSP反应程序
阴性对照管:对于每个子区域,按照表9的配方配置qMSP反应体系,但是模板为TE缓冲液。
阳性对照管:对于每个子区域,按照表9的配方配置qMSP反应体系,但是模板为含ACTB(转化后序列)和目标区域的人工合成的质粒。阳性对照模板的制备方法:将ACTB基因经亚硫酸氢盐转化后的序列和SEQ ID NO.25~SEQ ID NO.34所对应的序列分别克隆至pUC57上,形成人工合成质粒,将各个质粒均稀释到10
Ct值读取:qPCR反应结束后,可手动调整基线,将基线荧光值标准差的10倍高度对应的荧光强度值设置为阈值,阈值线为穿过阈值的与X轴平行的直线,其必须位于指数扩增期内,阈值线与扩增曲线的交点所对应的循环数即为Ct值。
质量控制:阴性对照要无扩增,阳性对照要有明显的指数增长期,且阳性对照的Ct值应在26-30之间。待检样本的内参基因的Ct值应≤33,阴性对照、阳性对照及内参基因均满足上述要求后,表明本次实验有效,可进行下一步样本结果的判定。否则,当次实验无效,须重新进行检测。
5.qMSP结果分析
定义ΔCt=Ct(目的区域)-Ct(ACTB),计算扩增所有的样本各个子区域的ΔCt值,然后使用SPSS 22.0软件进行受试者操作曲线特征(ROC)分析。具体地,将胰腺癌血浆样本的状态变量定为“1”,将健康人血浆样本的状态变量定为“0”,点击“分析”-“ROC曲线图”,指定检验变量和状态变量的值,将状态变量的值定为1,并选择“较小的检验结果表示更明确的检验”,得出ROC分析结果,选择约登指数(灵敏度+特异性-1)最大时的ΔCt为截断值,并得到平均AUC值、灵敏度和特异性数值,如表11所示,各区域的ROC曲线如图1所示。
表11、区域3~区域12检测胰腺癌血液样本的性能
由表11可以看出,区域3~区域7诊断胰腺癌血液样本的性能良好。总体来看,所有子区域区分胰腺癌血液样本的灵敏度均高于75%,其诊断的特异性均高于91%,其ROC曲线下面积(AUC)值均大于0.83。具体来看,区域5和区域10的诊断性能最佳,其ROC曲线的AUC值都大于0.9,其中区域5诊断胰腺癌血液样本的灵敏度为85.7%,特异性为97.1%;区域10诊断胰腺癌血液样本的灵敏度为88.3%,特异性为95.1%。可见,利用TRIM15基因位于Chr6:30171742-30172465区域的甲基化水平可以有效区分胰腺癌血液样本和正常样本。
实施例3
以本实施例中所用到的数据作为测试集。
1.样本的收集
收集68例经组织活检确诊为胰腺癌的患者的血液样本,此外还收集了健康人的血液样本88例,每份血样样本的体积大于8mL。所有血液样本的收集过程经过伦理委员会的审批,所有志愿者都签署了知情同意书,所有样本均采用匿名化处理。
2.样本DNA的提取、转化和纯化同实施例2。
3.qMSP反应及结果分析
按实施例2中所述方法,以经亚硫酸氢盐转化后的样本DNA作为模板,进行qMSP反应。计算每个样本在各个子区域的ΔCt值,根据ΔCt值判断样本在该子区域的甲基化状态。具体的判断标准为:若某一样本的ΔCt值小于等于截断值(如表11所述各个区域的截断值),则该样本在此区域为甲基化阳性即判定胰腺癌阳性;若某一样本的ΔCt值大于截断值,则该样本为甲基化阴性即判定胰腺癌阴性。然后根据样本在某一子区域的甲基化状态计算利用该子区域诊断胰腺癌血液样本的灵敏度和特异性。灵敏度为癌症患者样本中甲基化阳性样本的比例,特异性为健康人样本中甲基化阴性样本的比例。
表12、利用区域3~区域12的截断值诊断胰腺癌血液样本的性能
由表12的数据可以看出,在测试样本中,区域3~区域12检测胰腺癌患者血液样本的灵敏度范围为73.53%~86.76%,其检测健康人血液样本的特异性范围为92.05%~97.73%,可见区域3~区域12在测试样本中的诊断性能与其在训练样本中的诊断性能相似。综上,TRIM15基因子区域(区域3~区域12)的甲基化水平可以有效诊断胰腺癌。
以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各个技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
以上所述实施例仅表达了本申请的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本申请构思的前提下,还可以做出若干变形和改进,这些都属于本申请的保护范围。因此,本申请专利的保护范围应以所附权利要求为准,说明书及附图可以用于解释权利要求的内容。
