一种快速高效的基因型与表型数据监督降维方法
文献发布时间:2024-04-18 20:00:50
技术领域
本发明属于生物信息学技术领域,具体涉及一种快速高效的基因型与表型数据的监督降维方法。
背景技术
随着科学技术的不断发展,高通量测序技术已经广泛应用于生物学研究中,并使其获得强大的数据产出能力,包括基因组学、转录组学、蛋白质组学等生物学大数据。这些数据的应用将为疾病的诊断分型、基因组预测等领域提供巨量的信息。然而生物大数据中特征变量的数目往往远大于样本量,这容易造成模型的过拟合从而影响预测;而当预测变量之间有中等或者高相关性,即存在多重共线性时,传统的回归方法估计的系数并不稳定。为了解决这一问题,研究者们提出了许多降维方法,而常用的降维方法如主成分分析(PCA)属于无监督降维,尽管速度较快,但在预测领域表现不佳。在诸多监督学习中,JayMagidson提出了一种被称为相关成分回归(Correlated Component Regression,CCR)的方法,该方法从预测变量中按顺序提取相关分量,每个新的相关分量通过从先前捕获的相关分量中剔除无关变异来捕获抑制因子的影响,从而有效降低预测变量数目。相比其他方法,CCR具有尺度不变性的优势,预测变量是否标准化对分量结果并不影响,这使其适用于更多种类的预测器。然而CCR将每次提取的分量都作为新的协变量加入回归,这降低了CCR的计算效率,尤其在数据量较大时表现更为明显;另外其缺乏确定分量数目的条件,只能根据经验和后验信息进行筛选,这限制了CCR在实际中的应用。
发明内容
本发明要解决的技术问题是在基因型与表型数据的降维过程中,针对CCR的不足,提出一种快速高效的监督降维方法,在CCR的基础上结合梯度提升决策树(GradientBoosted Decision Tree)的思想,并以线性模型进行迭代加快降维速度,称之为BoostedCCR。
为解决上述技术问题,本发明所采取的技术方案是:融合梯度提升决策树策略与相关分量回归方法,通过对损失函数的拟合减少协变量的逐层累加,在不损失更多信息、保留CCR尺度不变性优势的同时以线性模式提升降维效率,优化了确定压缩分量的标准,实现了最终压缩分量的可自动确定。具体包括以下步骤:
S1)初始表型数据归一化处理,归一化后的初始表型向量用Y表示;归一化处理的方法为每个样本的表型值与表型值中的最小值的差除以表型值中的最大值与表型值中的最小值的差,以向量化形式记为公式(1),其中min(Y
S2)根据公式(2),基于归一化后的初始表型向量Y与基因型矩阵X
S3)基于基因型矩阵X
S4)根据公式(4),基于所述步骤S3)中的S
ΔY
S5)基于步骤S4)中的ΔY
S6)重复步骤S1)至S5),通过不断拟合损失函数得到第二轮的压缩分量S
与现有技术相比,本发明具有以下优点及有益效果:1.本发明采用线性算法,计算复杂度低,计算速度在现有基因组降维技术中最快;2.本发明通过结合梯度提升策略,以及采用相关的收敛方法,使降维后的数据具备原始数据近似或更佳的预测性能。
附图说明
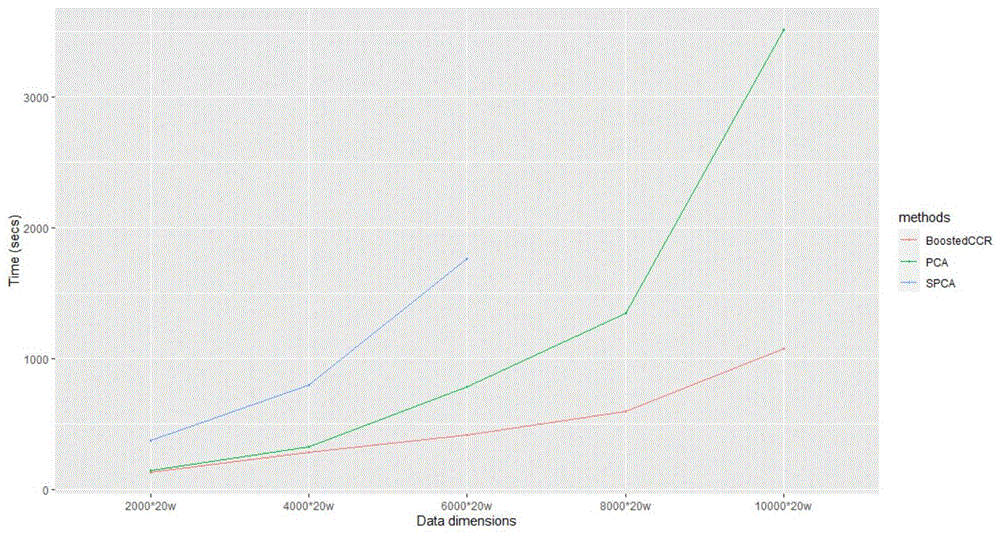
图1为实施例1中比较本发明所述方法(BoostedCCR)与PCA、SPCA在五个维度下大型基因型矩阵降维中的耗时。
图2为实施例2中基因组预测领域标准方法GBLUP和本发明所述方法(BoostedCCR)结合多种预测模型应用的预测准确性比较。
具体实施方式
实施例1:BoostedCCR在基因型与表型数据快速降维中的应用
以基因组数据——SNP基因分型矩阵为例,我们使用R包simer随机模拟了维度为2000×200000、4000×200000、6000×200000、8000×200000、10000×200000的大型基因型矩阵和对应的表型值。以维度为2000×200000的基因型矩阵(表1)和对应的表型数据(表2)示例,其中ID为样本编号,SNP为位点标记编号,主等位基因纯合编码为0,杂合基因编码为1,次等位基因纯合编码为2;Phenotype表示基于基因型矩阵由simer模拟出的表型值。
表1维度为2000×200000的基因型矩阵(示例)
表2维度为2000×200000的基因型对应的表型数据(示例)
将本发明所述的监督降维方法(BoostedCCR)与常见降维方法如主成分分析(PCA)、监督主成分分析(SPCA)、线性判别分析(LDA)及独立成分分析(ICA)在降维时间上进行比较。其中PCA由R基础包中的prcomp()函数实现,SPCA由R包MXM中的supervised.pca()函数实现,LDA由R包MASS中的lda()函数实现,ICA由R包fastICA中的fastICA()函数实现,以上方法均采用函数默认参数。本实施例中BoostedCCR具体步骤如下:
S1)根据公式(1)对示例表型进行归一化;初始示例表型向量记为Y
S2)根据公式(2),基于归一化后的表型向量Y与基因型矩阵X
S3)基于基因型矩阵X
S4)根据公式(4),基于所述步骤S3)中的S
ΔY
S5)基于步骤S4)中的ΔY
S6)重复步骤S1)至S5),通过不断拟合损失函数得到第二轮的压缩分量S
LDA与ICA无法对本实施例中的大型数据进行降维处理,表现为内存消耗过高难以有效运算,图1中未列出这两种方法。如图1所示,BoostedCCR、PCA、SPCA均实现了降维,其中SPCA在8000×200000的维度及以上无法正常工作,据随着维度的增大,BoostedCCR时间优势更加明显。
实施例2:BoostedCCR在基因组预测中的应用
基因组预测的准确性用真实表型与预测表型的皮尔森相关系数计算,以基因组最佳线性无偏预测(GBLUP)为衡量标准。由于GBLUP难以对大型基因型矩阵运算,因此我们在真实的非大数据中比较降维后基因型矩阵在基因组预测中的效果。接下来以模式生物拟南芥样本数据对本发明所述降维方法在基因组预测中的应用进行说明,并以经典的GBLUP方法作为基准进行比较。
S1)从公共数据集(http://arabidopsis.usc.edu)中获取拟南芥基因型数据(示例:表3)和表型数据(示例:表4)。表4中三种表型分别为拟南芥的FLC基因表达量(FLC)、10℃下的平均开花时间(FT10)以及10℃下的开花叶数(LN10)。
表3:拟南芥基因型数据(示例)
表4:拟南芥表型数据(示例)
S2)以表型FLC为例,随机将个体顺序打乱等比例分成5组,将其中4组表型数据及对应的基因型矩阵作为训练集,剩余1组作为测试集。
S3)在表型FLC中,利用本文所述方法(BoostedCCR)对拟南芥基因型数据进行降维,得到降维后的新数据。
对训练集基因型数据和表型数据作如下处理:
S3.1)根据公式(1)对训练集表型进行归一化;初始表型向量记为Y
S3.2)根据公式(2),基于S3.1)归一化后的表型向量Y与训练集基因型矩阵X
S3.3)基于训练集基因型矩阵X
S3.4)根据公式(4),基于所述步骤S3.3)中的S
ΔY
S3.5)基于S3.4)中的ΔY
S3.6)重复步骤S3.1)至S3.5),通过不断拟合损失函数得到第二轮的压缩分量S
基于训练集数据的降维过程,同时对测试集基因型数据作如下处理:
S3.7)基于测试集基因型数据与S3.2)所得每列SNP的回归系数的集合
S3.8)在训练集中重复步骤S3.1)-S3.5)的过程中,同时进行测试集的处理,即在最终得到训练集压缩分量S
S3.9)将降维后的训练集基因型数据S
S4)基于拟合完成的预测模型,通过测试集压缩分量S
S5)模型的预测准确性用预测表型和原始测试集表型的皮尔森相关系数计算,经过5次重复的五折交叉验证,取平均值衡量预测效果。结果如图2所示,相比于GBLUP的预测准确性,在拟南芥的三个性状中,经过本文所述方法降维后的数据结合三种预测模型均取得了良好的预测效果;其中在拟南芥LN10性状中LM、EN和SVR的预测准确性明显高于GBLUP,这说明本文所述方法(BoostedCCR)在基因组降维领域有较好的表现,并且经过BoostedCCR降维后的数据具有灵活的应用空间。
以上实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述实施例对本发明进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述实施例所记载的技术方案进行修改,或者对其中部分或者全部技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明权利要求所限定的范围。
- 一种驱动转向单元、模块化车辆及模块化运输系统
- 一种采用齿轮传动的翻转式转向与导向的RGV驱动角模块
- 采用电推杆驱动的翻转式转向与导向功能的RGV驱动角模块
