一种检测待测样本所属个体是否患有遗传病的试剂盒
文献发布时间:2024-04-18 20:01:23
技术领域
本发明涉及基因检测技术领域,具体涉及一种检测待测样本所属个体是否患有单基因遗传病的试剂盒。
背景技术
地中海贫血是一种单基因遗传性疾病,主要携带者在热带和亚热带地区。中国南方沿海地区是地贫高发区,部分省份高达25%的人携带地贫变异。根据基因变异的分子生物学特征,地贫分为α-地贫、β-地贫和δ-地贫。我国α-地贫携带者总数超过其他类型地贫携带者,其中广西α-地贫携带率高达17%。
α-地贫可以分为缺失型和非缺失型两大基因类型。95%以上为缺失型,是由于α珠蛋白基因大片段缺失所致。α珠蛋白基因位于16号染色体上,每条16号染色体上有2个α珠蛋白基因,一对16号染色体上有4个珠蛋白基因。地贫具有高度的遗传异质性。一条染色体上缺失或缺陷1个α基因(α珠蛋白基因),为α
胚胎植入前遗传学检测(preimplantation genetic testing,PGT)又称为第三代试管婴儿。是指在辅助生殖过程中对植入前的胚胎进行基因突变或染色体检测,选择正常的胚胎植入宫腔,以降低家族遗传性疾病、染色体异常及反复流产等风险的遗传学检测技术。
PGT从源头上真正实现了优生优育。PGT技术的最大难点在于样本量极少,仅为一至数个细胞,容易发生等位基因脱扣(allele drop-out,ADO),从而造成误诊。经研究表明,ADO是造成PGT误诊的主要原因。为了提高诊断准确性、降低误诊率,可以加入致病基因上下游多态位点(SNP)连锁分析来进行遗传学诊断。目前,基于二代测序的多重PCR扩增与单体型分析技术的结合是α-地中海贫血胚胎植入前遗传学检测诊断中最常见的检测手段,不仅可以对α-地中海贫血突变的直接检测结果进一步确认,还能预防等位基因脱扣(ADO)导致的PGT-M误诊。但是无论一个位点多态性有多高,一旦脱扣,就没有任何诊断价值。所以,需要选择较多的多态性位点数量,即使一个或几个位点脱扣,还有其他的位点能够提供遗传信息,不影响最终诊断。而且,在配子进行减数分裂的过程中,SNP位点和致病基因之间可能发生重组,两者距离越远,发生重组的可能性越大。所以在α-地中海贫血的检测中,需要提供一套更好更具有特异性的SNP位点组合并设计一套特异性引物组合,助力α-地中海贫血突变的检出。
目前应用于α-地中海贫血的胚胎植入前遗传学检测诊断方法是发育至第5天或者第6天的囊胚行活检,对活检细胞进行全基因组扩增后,再利用相应技术进行诊断。目前常用的实验技术包括gap-PCR、巢式PCR以及多重PCR。gap-PCR具有灵敏度高的优点,但是gap-PCR需要对缺失片段设计特异性探针,探针设计难度大,合成工艺要求及成本高。巢式PCR有内外两对引物,RCR反应分为两步,相对普通PCR,巢式PCR提高了PCR技术的灵敏度、特异性和精确度,但是巢式PCR需要两次扩增,操作复杂,交叉污染几率大,且对引物的要求更高。多重PCR捕获目标序列具有操作简单、成本低,对样本起始量要求低,大大缩短了检测流程和检测时间。然而,多重PCR拟捕获目标区域越多,则扩增的特异性、准确性、均一性就会受到影响,多重PCR捕获技术的实现难度越大。
与此同时,现有技术中使用“SEA缺失区域+下游区域”进行单体型分析,会造成一旦下游大片区域发生重组,从而造成对个体的单体型遗传自哪一个亲本进行判断时出现错误的结果。
发明内容
根据第一方面,在一实施例中,提供一种检测SNP位点的试剂在制备用于检测待测样本所属个体是否患有遗传疾病或患有遗传疾病的风险度的试剂盒中的用途,所述SNP位点位于人类基因组第16号染色体的SEA缺失区域的上游和/或下游1Mb的连锁内。
根据第二方面,在一实施例中,提供一种检测待测样本所属个体是否患有遗传疾病或患有遗传疾病的风险度的试剂盒,所述试剂盒包含用于检测人类基因组第16号染色体的SEA缺失区域的上游和/或下游的SNP位点的引物。
根据第三方面,在一实施例中,提供一种检测待测样本所属个体是否患有遗传疾病或患有遗传疾病的风险度的方法,所述方法包含对人类基因组第16号染色体的SEA缺失区域的上游和/或下游的SNP位点进行单体型分析检测。
发明效果
在一个具体的实施方式中,本发明的检验α-地贫突变构建单体型的SNP位点来自--
在一个具体的实施方式中,本发明的检验α-地贫突变构建单体型的SNP位点来自α
在一个具体的实施方式中,本发明的检验α-地贫突变构建单体型的SNP位点来自--
在一个具体的实施方式中,由于本发明检测的SNP位点不仅仅只是--
在一个具体的实施方式中,本发明在下游基础上增加了上游区域的协同判断,即便下游或上游某一区域发生了重组,另外一区域也可防止单体型判断失误,判断的准确性比仅评估下游区域显著提高。
在一个具体的实施方式中,本发明可仅通过上下游所筛选的SNP位点进行分型,无需借助α-地贫区域SNP位点。
在一个具体的实施方式中,本发明结合所筛选的SNP位点与靶向扩增区间内相关的其他SNP位点,一起进行单体型分析,增加有效位点数目,帮助单体型分型,从而能够帮助α-地贫检测更好的判定。
在一个具体的实施方式中,本发明在多重PCR引物设计难度加大的情况下,使用重数更多的引物进行检测,所设计的引物组合能够将靶向缺失区间及连锁位点同时扩增,靶向位点结果与连锁位点单体型结果相互验证,准确率更高,且节约成本。
在一个具体的实施方式中,本发明通用性好,由于多个SNP位点同时检测,可以用于多种α-地贫的家系组合胚胎移植前检测,也可用于胎儿检测、流产组织检测。
附图说明
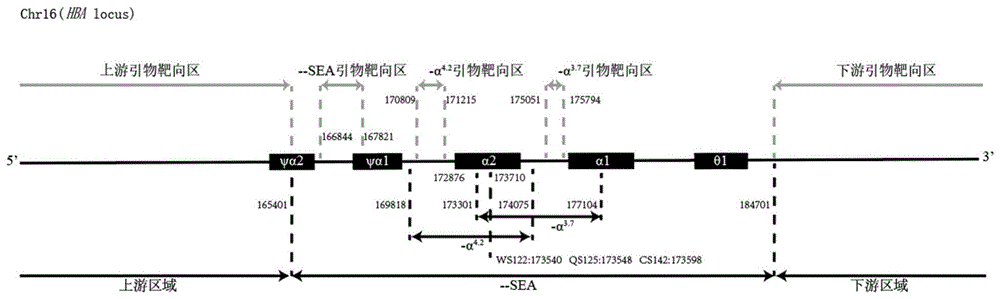
图1为人类基因组第16号染色体的α-地中海贫血相关位置示意图。
具体实施方式
下面通过具体实施方式结合附图对本发明作进一步详细说明。在以下的实施方式中,很多细节描述是为了使得本申请能被更好的理解。然而,本领域技术人员可以毫不费力的认识到,其中部分特征在不同情况下是可以省略的,或者可以由其他材料、方法所替代。在某些情况下,本申请相关的一些操作并没有在说明书中显示或者描述,这是为了避免本申请的核心部分被过多的描述所淹没,而对于本领域技术人员而言,详细描述这些相关操作并不是必要的,他们根据说明书中的描述以及本领域的一般技术知识即可完整了解相关操作。
另外,说明书中所描述的特点、操作或者特征可以以任意适当的方式结合形成各种实施方式。同时,方法描述中的各步骤或者动作也可以按照本领域技术人员所能显而易见的方式进行顺序调换或调整。因此,说明书和附图中的各种顺序只是为了清楚描述某一个实施例,并不意味着是必须的顺序,除非另有说明其中某个顺序是必须遵循的。
本文中为部件所编序号本身,例如“第一”、“第二”等,仅用于区分所描述的对象,不具有任何顺序或技术含义。
术语解释
在本申请中,单体型是指位于一条染色体特定区域的一组相互关联,并倾向于整体遗传给后代的单核苷酸多态性的组合。
在本申请中,上游是指一段基因位于靠近染色体表达起点(起始子)的位置,在一个具体的实施方式中,本发明的检验α-地贫突变构建单体型的SNP位点来自--
在本申请中,下游是指一段基因位于靠近染色体表达终点(终止子)的位置,在一个具体的实施方式中,本发明的检验α-地贫突变构建单体型的SNP位点来自--
在本申请中,等位基因脱扣是扩增样本量极低时的常见现象,特指应用PCR进行扩增时,仅扩增出2个等位基因中的1个。
在本申请中,有效位点是指对于构建家系单体型提供有效SNP信息的位点,一般其中一方为杂合的位点,如在处理母亲单体型时选择的母亲杂合,父亲纯合的位点,处理父亲单体型时选择的父亲杂合,母亲纯合的位点,并判断这些位点是否位于携带致病突变的单体型上。
在一实施例中,本发明在多重PCR引物设计难度加大的情况下,使用重数更多的引物进行检测,有效提高检测准确性。
根据第一方面,在一实施例中,提供一种检测SNP位点的试剂在制备用于检测待测样本所属个体是否患有遗传疾病或患有遗传疾病的风险度的试剂盒中的用途,所述SNP位点位于人类基因组第16号染色体的SEA缺失区域的上游和/或下游1Mb的连锁内。
除非特别说明,否则本申请中SNP位点所对应的基因组均为人类GRCh38版本基因组。
在一实施例中,所述SNP位点位于所述SEA缺失区域上游的如下位置中的至少一个碱基对,或至少两个碱基对:
39279、42391、44919、47354、47610、51263、51277、51304、53009、53423、53923、55320、55325、55444、56777、57162、57275、58586、59673、60167、60927、62114、62378、62515、63495、63926、63972、64537、65043、65074、65659、65819、68659、68811、72212、72784、72850、72884、73222、73898、74142、74163、74473、75343、75463、75758、77231、77578、77825、77828、77906、78180、78955、80198、80854、80894、81110、81554、81566、82225、82247、82511、83947、84442、84458、84746、86143、86259、86889、88020、88033、89088、89369、89603、91707、92827、93505、95327、95360、96489、96963、96967、98354、102222、103235、105322、105510、105891、105968、107143、111939、112999、113599、113668、120045、120329、123184、124170、125727、125728、126158、126744、130407、135103、135124、137430、137510、138304、138607、140238、142315、143587、148094、149541、151184、155036、157060、157612、157910、159817、161106、161107、162329、163800、163804、165380。
在一实施例中,所述SNP位点位于所述SEA缺失区域上游的如下位置中的至少一个碱基对,或至少两个碱基对:
51263、55444、60927、65043、86889、89369、95327、126744、130407、135103、135124、137510、138304、138607、157612、163800、163804。
在一实施例中,所述SNP位点位于所述SEA缺失区域下游的如下位置中的至少一个碱基对,或至少两个碱基对:
190281、197889、198358、198419、206279、211867、225998、227459、235314、239695、243563、250389、254804、261854、262254、263407、266891、266901、272935、274052、276826、286660、299293、306689、308161、309953、310303、310888、311873、312638、315024、319413、320484、327623、330425、340780、342461、346264、354750、355416、361092、363747、364374、364608、366663、369092、370678、373420、373423、376432、377309、377349、377479、377516、377657、377784、377820、377866、379129、379951、382751、389550、389618、391710、397416、399162、399345、399934、400165、400309、407226、411762、427619、440645、440835、442529、445538、464407、466922、472672、484765、484954、487979、489140、489429、489882、490137、491671、491679、492148、492727、493357、520623、529900、534772、538054、551632、551892、552313、555481、556913、562698、563168、565326、572234、572325、573082、574114、574713、575384、577920、578130、578302、580367、580405、580665、580710、582115、582138、582180、582198、582225、582233、582728、582736、582767、583125、583353、583354、583764、583843、583851、584260、585794、587212、592249、592610、595968、596421、596571、597006、601279、601517、611142、611335、611451、621682。
在一实施例中,所述SNP位点位于所述SEA缺失区域下游的如下位置中的至少一个碱基对,或至少两个碱基对:
190281、198358、198419、211867、235314、239695、243563、250389、261854、266891、369092、389618、391710、397416、399345、400165、411762、442529、445662、464407、466922、484954、487979、489140、489429、489882、490137、492727、520623、534772、538054、555481、556913、562698、565326、582138、582736、587212、596421、611142。
在一实施例中,用于检测所述SEA缺失区域上游的SNP位点的扩增子起始位置和终止位置包含如下表1所示扩增子起始位置和终止位置中的至少一组(每一行的扩增子起始位置和终止位置为一组):
表1
/>
/>
在一实施例中,用于检测所述SEA缺失区域下游的SNP位点的扩增子起始位置和终止位置包含如下表2所示扩增子起始位置和终止位置中的至少一组(每一行的扩增子起始位置和终止位置为一组):
表2
/>
/>
在一实施例中,用于检测所述SEA缺失区域上游的SNP位点的引物包含如SEQ IDNo.1~216所示核苷酸序列中的至少一种。
在一实施例中,用于检测所述SEA缺失区域下游的SNP位点的引物包含如SEQ IDNo.277~532所示核苷酸序列中的至少一种。
在一实施例中,所述SNP位点还包含位于SEA缺失区域的如下位置中的至少一个碱基对,或至少两个碱基对:
165856、166191、166352、166421、166594、167089、167775、168533、168561、171058、171127、175160、175387、175654、178479、178571、181168、182006、183232、183819、184365、184429。
在一实施例中,所示SNP位点还包含位于SEA缺失区域地如下位置中的至少一个碱基对,或至少两个碱基对:
171058、171127。
在一实施例中,用于检测所述SEA缺失区域的SNP位点的扩增子起始位置和终止位置包含如下表3所示扩增子起始位置和终止位置中的至少一组(每一行的扩增子起始位置和终止位置为一组):
表3
在一实施例中,用于检测所述SEA缺失区域的SNP位点的引物包含如SEQ IDNo.217~276所示核苷酸序列中的至少一种。
在一实施例中,所述遗传疾病包含地中海贫血。
在一实施例中,所述地中海贫血包含α-地中海贫血。
在一实施例中,所述人类基因组可以选自人类GRCh38版本基因组。
在一实施例中,所述待测样本所属个体包括但不限于胚胎或胎儿。
在一实施例中,所述待测样本的来源包括但不限于胚胎囊胚期滋养层细胞。
在一实施例中,所述待测样本包括但不限于全基因组扩增产物。
根据第二方面,在一实施例中,提供一种检测待测样本所属个体是否患有遗传疾病或患有遗传疾病的风险度的试剂盒,所述试剂盒包含用于检测人类基因组第16号染色体的SEA缺失区域的上游和/或下游的SNP位点的引物。
在一实施例中,用于检测所述SEA缺失区域上游的SNP位点的引物包含如SEQ IDNo.1~216所示核苷酸序列中的至少一种。
在一实施例中,用于检测所述SEA缺失区域下游的SNP位点的引物包含如SEQ IDNo.277~532所示核苷酸序列中的至少一种。
在一实施例中,所述试剂盒还包含用于检测SEA缺失区域的引物。
在一实施例中,用于检测所述SEA缺失区域的SNP位点的引物包含如SEQ IDNo.217~276所示核苷酸序列中的至少一种。
在一实施例中,所述遗传疾病包含单基因遗传病。
在一实施例中,所述遗传疾病包含地中海贫血。
在一实施例中,所述地中海贫血包含α-地中海贫血。
在一实施例中,所述人类基因组可以选自人类GRCh38版本基因组。
在一实施例中,所述待测样本所属个体包括但不限于胚胎或胎儿。
在一实施例中,所述待测样本的来源包括但不限于胚胎囊胚期滋养层细胞。
在一实施例中,所述待测样本包括但不限于全基因组扩增产物。
在一实施例中,本发明提供了一组用于检测六种α-地贫突变的SNP有效位点,一共295个位点,如下表4所示。其中上游126个,--
表4
/>
/>
/>
/>
注:下划直线标示的位点上方为上游SNP扩增子,下划直线标示的位点与下划波浪线标示的位点之间(包括下划直线标示的位点以及下划波浪线标示的位点)为SEA区域SNP扩增子,下划波浪线标示的位点下方为下游SNP扩增子。
在一实施例中,本发明基于多重PCR扩增技术,针对所筛选SNP有效位点设计了266对特异性引物序列组合,如表5所示,其中上游包含108对引物,六种突变的特异性区域或位点包含30对引物,下游包含128对引物。这些引物特点包括:1)在目标染色体上序列唯一;2)具有相同的退火温度;3)上下游靶向扩增区间至少包含一个所筛选的SNP位点。
表5
/>
/>
/>
/>
/>
/>
/>
/>
在一实施例中,本发明基于所设计的266对引物对应的扩增区间,进一步筛选了靶向扩增区间内的其它SNP有效位点一共6665个,与上述有效位点组合,共同构建单体型,本发明囊括这6665个位点不仅能够增加单体型分型的有效位点数目,且由于这些SNP位点中很多位点突变概率低,一旦发生突变则能更好的帮助地贫突变的检出。
根据第三方面,本发明提供一种检测待测样本所属个体是否患有遗传疾病或患有遗传疾病的风险度的方法,所述方法包含对人类基因组第16号染色体的SEA缺失区域的上游和/或下游的SNP位点进行单体型分析检测。
在一个具体的实施方式中,本发明中的SNP位点为本发明说明书中记载的具体SNP位点。
在一实施例中,单体型分析方法的操作步骤如下:
(1)获取亲代双方和胚胎待测样本的DNA;其中亲代双方及其先证者采用全血样本,胚胎待测样本是获取胚胎发育至囊胚期,取出3~5个滋养层细胞,运用单细胞全基因组扩增(whole genome amplification,WGA)方法对细胞中的基因组DNA进行富集。
(2)如图1所示,选择--
(3)将二代测序结果进行过滤,得到过滤后的测序结果,过滤的条件包括序列中含有N的比例,以及含有低质量碱基数的比例,所述N为测序得到的碱基无法判断,过滤一条序列中N的比例大于5%的序列,以及一条序列中质量值小于20的比例大于40%的序列。
(4)将过滤后的测序结果与参考人类基因组序列进行比对,获得经过比对的测序数据集合;所述比对的软件采用BWA或SOAP;所述参考人类基因组为GRCh38,确定目标区域多态性位点的基因型,筛选有效位点,最后分析单体型。
实施例1:通过上下游SNP位点确定单体型
一、材料
本实施例样本来源于9例胚胎囊胚期滋养层细胞全基因组扩增产物、父母及其女儿的外周血细胞DNA样本,其中男方基因检测为携带--
二、方法
(1)使用本发明设计的引物组(共266对SNP引物),按照Fapon Single Cellgenome-Ampli Kit(NK023)标准建库流程进行建库,并在Ion Torrent PGM上测序,得到片段长度分布在100~150bp的目标区域DNA片段序列。
(2)利用fastp-0.21.0软件对原始数据进行过滤,利用BWA软件比对到人类GRCh38参考基因组,去除低质量序列,统计目标位点的测序深度,并去除深度小于100的位点、确定目标区域SNP位点的基因型。
(3)利用父母和胚胎目标区域SNP位点的基因型,筛选有效位点构建单体型。利用已经建立好的单体型对胚胎是否携带突变进行判断。
三、结果
选取目标区域的92个有效位点构建单体型,结果如表6所示。
表6上下游SNP位点判断单体型结果数据
/>
注:下划直线标示的数据上侧为上游SNP有效位点,下划直线标示的数据及下侧为下游SNP有效位点,加粗的数据为携带致病缺失基因的染色单体。
续表6上下游SNP位点判断单体型结果数据右侧续表
/>
结果表明:
表6中借助至少任意上游2个SNP位点和任意下游2个SNP位点的组合即可完成单体型分析。例如通过上游SNP位点39279、59667可确定胚胎1的第一条单体39279处的C及59667处的G来源于母亲的chrA,第二条39279处的C及59667处的A来源于父亲的chrC。通过下游SNP位点430096、445538可确定胚胎1的第一条单体430096处的T和445538处的T来源于母亲的chrA,第二条430096处的C和445538处的C来源于父亲的chrC。
男方染色单体分为chrC、chrD,chrD为携带致病突变染色单体,chrC为正常染色体单体;女方染色单体分为chrA、chrB,chrA携带致病突变染色单体,chrB为正常染色体单体;女儿为正常。pgd2022070701为母源缺失,pgd2022070704为纯合缺失,pgd2022070705为母源缺失,pgd2022070706为父源缺失,pgd2022070707为父源缺失,pgd2022070708为母源缺失,pgd2022070709为父源缺失,pgd2022070710为纯合缺失,pgd2022070711为纯合缺失。
实施例2
一、材料
本实施例样本来源于6例胚胎囊胚期滋养层细胞全基因组扩增产物、父母及女方母亲的外周血细胞DNA样本,其中女方基因检测为携带--
二、方法
采用与实施例1相同的方法进行分析。
三、结果
选取目标区域的75个有效位点构建单体型,结果如表7所示。
表7上下游SNP位点判断单体型结果数据
/>
/>
注:下划直线标示的数据上侧为上游SNP有效位点,下划直线标示的数据及下侧为下游SNP有效位点,加粗的数据为携带致病缺失基因的染色单体。
续表7上下游SNP位点判断单体型结果数据右侧续表
/>
结果表明:
基于和实施例1相类似的原理,表7中借助至少任意上游2个SNP位点和下游2个SNP位点的组合即可完成单体型分析。
女方染色单体分为chrA、chrB,chrA携带致病突变染色单体,chrB为正常染色体单体;男方及女方母亲为正常。pgd20220915003为正常,pgd20220915004为母源缺失,pgd20220915005为母源缺失,pgd20220915006为正常,pgd20220915007为母源缺失,pgd2022070708为正常。
实施例3
一、材料
本实施例样本来源于11例胚胎囊胚期滋养层细胞全基因组扩增产物、父母及女儿的外周血细胞DNA样本,其中女方及女儿基因检测为携带-α
二、方法
采用与实施例1相同的方法进行分析。
三、结果
选取目标区域的150个有效位点构建单体型,结果如表8所示。
表8上下游SNP位点判断单体型结果数据
/>
/>
/>
注:下划直线标示的数据上侧为上游SNP有效位点,下划直线标示的数据及下侧为下游SNP有效位点,加粗的数据为携带致病缺失基因的染色单体。
续表8上下游SNP位点判断单体型结果数据右侧续表
/>
/>
/>
结果表明:
基于和实施例1相类似的原理,表8中借助至少任意上游2个SNP位点和下游2个SNP位点的组合即可完成单体型分析。
女方染色单体分为chrA、chrB,chrA携带致病突变染色单体,chrB为正常染色体单体;男方为正常,女儿为母源缺失。pgd20220525001、pgd20220525002、pgd20220525004、pgd20220525008、pgd20220525011等样本为正常,pgd20220525003、pgd20220525006、pgd20220525007、pgd20220525009、pgd20220525010、pgd20220525013等样本为正常。
实施例4
一、材料
本实施例样本来源于5例胚胎囊胚期滋养层细胞全基因组扩增产物、父母及女儿的外周血细胞DNA样本,其中男方基因检测为携带--
二、方法
采用与实施例1相同的方法进行分析。
三、结果
选取目标区域的113个有效位点构建单体型,结果如表9所示。
表9上下游SNP位点判断单体型结果数据
/>
/>
注:下划直线标示的数据及上侧为上游SNP有效位点,下划直线标示与下划波浪线标志之间的数据为SEA缺失区域SNP有效位点,下划波浪线标示的数据及下侧为下游SNP有效位点,加粗的数据为携带致病缺失基因的染色单体。
续表9上下游SNP位点判断单体型结果数据右侧续表
/>
/>
结果表明:
基于和实施例1相类似的原理,表9中借助至少任意上游2个SNP位点和下游2个SNP位点的组合即可完成单体型分析。
男方染色单体分为chrA、chrB,chrB携带致病突变染色单体,chrA为正常染色体单体;女方及女儿为正常。pgd20220820001为正常,pgd20220820002为父源缺失,pgd20220820003为正常,pgd20220820004为父源缺失,pgd20220820005为父源缺失。
实施例5
一、材料
本实施例样本来源于8例胚胎囊胚期滋养层细胞全基因组扩增产物、父母及儿子的外周血细胞DNA样本,其中男方和女方基因检测均为携带--
二、方法
采用与实施例1相同的方法进行分析。
三、结果
选取目标区域的109个有效位点构建单体型,结果如表10所示。
表10上下游SNP位点判断单体型结果数据
/>
/>
注:下划直线标示的数据及上侧为上游SNP有效位点,下划直线标示与下划波浪线标志之间的数据为SEA缺失区域SNP有效位点,下划波浪线标示的数据及下侧为下游SNP有效位点,加粗的数据为携带致病缺失基因的染色单体。
续表10上下游SNP位点判断单体型结果数据右侧续表
/>
/>
结果表明:
基于和实施例1相类似的原理,表10中借助至少任意上游2个SNP位点和下游2个SNP位点的组合即可完成单体型分析。
男方染色单体分为chrA、chrB,chrA携带致病突变染色单体,chrB为正常染色体单体;女方染色单体分为chrC、chrD,chrD携带致病突变染色单体,chrC为正常染色体单体;儿子为正常。pgd20220613001为父源缺失,pgd20220613002为纯合缺失,pgd20220613003为正常,pgd20220613004为母源缺失,pgd20220613005为纯合缺失,pgd20220613006为母源缺失,pgd20220613007为父源缺失,pgd20220613008为父源缺失。
实施例6:panel捕获性能指标检测
本实施例使用与实施例1相同的9例胚胎囊胚期滋养层细胞全基因组扩增产物、父母及其女儿的外周血细胞DNA样本,使用本发明设计的引物组(共266对SNP引物),按照Fapon Single Cell genome-Ampli Kit(NK023)标准建库流程进行建库,并在Ion TorrentPGM上测序,得到片段长度分布在100-150bp的目标区域DNA片段序列。对获得的测序下机数据进行常规参数的质控和分析。
检测结果:
对实施例1中的样本进行测试,能够获得靶向区间全覆盖,目标全覆盖,最低测序深度为100x,mapping rate≥99%,ontarget rate≥95%,100x depth rate≥95%。具体信息如下表所示。
表11
/>
注:“Mapping rate”代表重测序时比对率;“Ontarget rate”代表中靶率,表示测序数据比对到靶向扩增子的比例;“>0.2x Average Depth Rate”代表大于0.2x平均测序深度位点数目的比例,该值越高,扩增均一性越好;“>100x Depth Rate”代表目标位点的覆盖率,该值越高,说明目标位点覆盖度越高。
从表11可见,使用本发明的引物扩增后的各样本测序数据显示,具有优异的比对率、扩增均一性以及目标位点覆盖率。
以上应用了具体个例对本发明进行阐述,只是用于帮助理解本发明,并不用以限制本发明。对于本发明所属技术领域的技术人员,依据本发明的思想,还可以做出若干简单推演、变形或替换。
- 一种自动供给一次性水杯存取盒
- 一种小型开口物料盒自动存取机器人及其操作方法
- 一种小型开口物料盒自动存取机器人
