程序性细胞死亡1(PD1)特异性核酸酶
文献发布时间:2023-06-19 11:32:36
本申请要求2018年9月18日提交的美国临时申请号62/732,674的权益,将其公开内容通过引用以其整体特此并入。
不适用
[0002.1]本申请含有以ASCII格式电子提交并且通过引用以其整体特此并入的序列表。在2019年9月9日创建的所述ASCII副本命名为8325018140SL.txt并且大小为15,792字节。
技术领域
本公开文本涉及多肽和基因组工程以及同源重组领域。
背景技术
人工核酸酶,如工程化锌指核酸酶(ZFN)、转录激活因子样效应核酸酶(TALEN)、具有工程化crRNA/tracr RNA(“单指导RNA”)的CRISPR/Cas系统(也称为RNA指导核酸酶)和/或基于Argonaute系统的核酸酶(例如,来自嗜热栖热菌(T.thermophilus),称为‘TtAgo',(Swarts等人(2014)Nature 507(7491):258-261)包含与切割结构域相关或可操作地连接的DNA结合结构域(核苷酸或多肽),并已用于基因组序列的靶向改变。例如,核酸酶已被用于插入外源序列、使一个或多个内源基因失活、产生具有改变的基因表达模式的生物体(例如,作物)和细胞系等。参见例如,美国专利号9,255,250;9,200,266;9,045,763;9,005,973;8,956,828;8,945,868;8,703,489;8,586,526;6,534,261;6,599,692;6,503,717;6,689,558;7,067,317;7,262,054;7,888,121;7,972,854;7,914,796;7,951,925;8,110,379;8,409,861;美国专利公开20030232410;20050208489;20050026157;20050064474;20060063231;20080159996;201000218264;20120017290;20110265198;20130137104;20130122591;20130177983以及20130177960和20150056705。例如,一对核酸酶(例如,锌指核酸酶、TALEN、dCas-Fok融合物)可以用于切割基因组序列。所述对中的每个成员通常包括与核酸酶的一个或多个切割结构域(或半结构域)连接的工程化(非天然存在的)DNA结合蛋白。当DNA结合蛋白结合至其靶位点时,与那些DNA结合蛋白连接的切割结构域被定位成使得可以发生二聚化和随后对基因组的切割。
已经描述了许多用于增强DNA切割特异性(减少靶外切割)的策略。这些方法的细节随其应用的核酸酶的功能特征和局限性而改变。对于全蛋白质系统(例如TALEN(Miller2011)、ZFN(Kim 1996)和大范围核酸酶(Grizot 2009)),用于改善特异性的简单而直接的方法是重新工程化碱基感应界面(Miller 2015;Rebar 1994;Jarjour 2009)。其他更普遍的策略包括破坏二聚对称性以抑制非预期核酸酶种类的形成(Miller 2007),以及去除非特异性DNA接触(Guilinger 2014)。对于特征为较短靶标的CRISPR-Cas系统,许多研究集中于扩展切割所需的识别事件,以补救复杂基因组中唯一寻址的局限性(Ran 2013、Tsai2014、Guilinger 2014a、Bolukbasi 2015)。鉴于Watson-Crick相互作用在靶标识别中的主导作用,用于工程化CRISPR-Cas碱基感应界面的机会比较有限,但最近的研究已经开始检查PAM界面和合成指南(Yin 2018;Ryan 2018)。Cas9的特异性也通过去除非特异性接触而得到改善(Kleinstiver 2016;Slaymaker 2016;Chen 2017),但其代价是降低了至少一子集的所得变体的活性(Kulcsar 2017;DeWitt 2016;Zhang 2017)。最后,最近的两项研究使用基于选择的方法来鉴定具有更大靶上偏好的Cas9变体(Hu 2018;Casini 2018)。这些方法中的每一种均产生了相对于亲本起始框架具有改善的特异性的试剂。
对于解释和基本原理,这些策略的共同特征是它们几乎完全专注于初始靶标结合。少有例外地,每个策略均公开或隐含地将靶标结合视为DNA切割特异性的唯一决定因素,因此,通过重新工程化下游步骤来改善切割偏好的潜力在很大程度上仍未得到探索。鉴于用于加强酶特异性的动力学调节的生物学的优先性以及基因组编辑过程的最低催化要求(成功的编辑可能每个细胞仅需要一次切割事件),这似乎为提高特异性提供了一个重要的未开发维度。尽管最近对化脓性链球菌(S.pyogenes)Cas9的研究已经开始表征中间结构(Singh 2016、Jiang 2016、Jiang 2017)和切割过程的动力学(Sternberg 2015、Dagdas2017、Raper 2018),但利用这些见解的努力迄今为止还很有限,并且在高度修饰的细胞中未展现出独特的特异性(Chen2017)。此外,以动力学为重点的框架尚未应用于如TALEN和ZFN的核酸酶,其较长的靶标提供在靶上与靶外位点之间固有的较宽能量间隙,其中动力学优化可能特别有益。
美国公开号20180087072公开了包含在切割结构域(例如,FokI)和/或ZFP骨架中的突变的高度特异性核酸酶以及制备和使用此类高度特异性核酸酶的方法。
程序性死亡受体(PD1,也称为PDCD1)是一种通过以下两种机制防范自身免疫的免疫检查点:(1)通过促进淋巴结中抗原特异性T细胞的凋亡(程序性细胞死亡);以及(2)通过减少调节性T细胞(抗炎抑制性T细胞)的凋亡。Francisco等人(2010)ImmunologicalReviews 236:219-42;Fife等人(2011)Annals New York Academy of Sciences 1217:45-59。PD-1抑制剂是一类阻断PD-1的新型药物,其激活免疫系统来攻击肿瘤,并用于治疗各种类型的癌症。参见例如,Jelinek等人(2017)Immunology 152(3):357-371。
因此,仍然需要表现出非常低或没有靶外切割活性的靶向PD1的改进的核酸酶。
发明内容
本公开文本提供了高度特异性的PD1核酸酶,即人工核酸酶(例如,锌指核酸酶(ZFN)、TALEN、CRISPR/Cas核酸酶),其包含一个或多个DNA结合结构域区域(例如,锌指蛋白或TALE的骨架)中的突变和/或FokI核酸酶切割结构域或切割半结构域中的一个或多个突变。
因此,在一个方面,本文描述的是靶向PD1的工程化(人工)核酸酶,所述核酸酶包含至少一个与PD1中的靶位点结合的DNA结合结构域(例如,ZFP)和至少一个切割半结构域,所述切割半结构域与衍生出这些突变体的亲本(例如,野生型)切割结构域相比包含一个或多个突变。在某些实施方案中,所述核酸酶是锌指核酸酶(ZFN),其包含一对ZFN,也称为左和右(或第一和第二)ZFN,其中所述对中的每个ZFN均包含ZFP PD1 DNA结合结构域和切割结构域(或切割半结构域)。在某些方面,PD1核酸酶是ZFN,其包含指定为12942(SEQ ID NO:3)和25029(SEQ ID NO:5)的ZFP,以及其中包含一个或多个突变的FokI切割结构域。在某些实施方案中,所述一个或多个突变是任何所附表格和图中所示的突变中的一个或多个,包括这些突变体彼此之间以及与其他突变体(如二聚化和/或催化结构域突变体以及切口酶突变)的任何组合。如本文所述的突变包括但不限于改变切割结构域的电荷的突变,例如带正电残基向非带正电残基的突变(例如,K和R残基的突变(例如,突变为S);N残基的突变(例如,突变为D)和Q残基的突变(例如,突变为E);被预测为接近DNA骨架(例如,位置(-5)、(-3)等)的残基的突变;和/或其他残基处(例如,在二聚化结构域中)的突变。
在某些实施方案中,工程化切割半结构域源自FokI或FokI同源物,并且包含氨基酸残基414-426、443-450、467-488、501-502和/或521-531中的一个或多个,包括387、393、394、398、400、416、418、422、427、434、439、441、442、444、446、448、472、473、476、478、479、480、481、487、495、497、506、516、523、525、527、529、534、559、569、570和/或571中的一个或多个中的突变。在某些实施方案中,ZFN对的左ZFN包含一个或多个突变。在其他实施方案中,ZFN对的右ZFN包含一个或多个突变。在其他实施方案中,ZFN的右ZFN和左ZFN两者均包含一个或多个突变。突变可能包括在相应位置处与FokI同源的天然限制酶中发现的残基的突变。在某些实施方案中,所述突变是取代,例如用任何不同的氨基酸(例如丙氨酸(A)、半胱氨酸(C)、天冬氨酸(D)、谷氨酸(E)、组氨酸(H)、苯丙氨酸(F)、甘氨酸(G)、天冬酰胺(N)、丝氨酸(S)、缬氨酸(V)、精氨酸(R)、谷氨酰胺(Q)或苏氨酸(T))取代野生型残基。考虑了突变体的任何组合,包括但不限于所附表格和图中所示的那些。在某些实施方案中,FokI核酸酶(切割)结构域包含416、418、422、476、479、481、525和/或531中的一处或多处(优选416、422、476、481和/或525处并且甚至更优选416、481和/或525处)的突变(单独或与其他突变如ELD、KKR等组合)。在某些实施方案中,核酸酶的至少一个FokI结构域包含(i)单个突变,例如在位置416处(例如,其中野生型R残基被E、F或N残基取代)、位置418处(例如,其中野生型S残基被D或E残基取代)、位置422处(例如,其中野生型R残基被H残基取代)、位置476处(例如,其中野生型N残基被D、E、G或T残基取代)、位置481处(例如,其中野生型Q残基被D、E或H残基取代)、位置525处(例如,其中野生型K残基被A、S、T或V残基取代)、位置527处(例如,其中野生型N残基被D残基取代)或位置531处(例如,其中野生型Q残基被R残基取代);或(ii)416处(例如,其中野生型R残基被E、F、或N残基取代)、位置418处(例如,其中野生型S残基被D或E残基取代)、位置422(例如,其中野生型R残基被H残基取代)、位置476处(例如,其中野生型N残基被D、E、G或T残基取代)、位置481处(例如,其中野生型Q残基被D、E或H残基取代)、位置525处(例如,其中野生型K残基被A、S、T或V残基取代)、位置527处(例如,其中野生型N残基被D残基取代)或位置531处(例如,其中野生型Q残基被R残基取代)中之一处的突变与一个或多个另外的残基(例如,二聚化突变如ELD、KKR等)的突变组合。参见例如,图1-图3。在某些实施方案中,所述突变包含选自以下的单个突变:R416E、R416F、R416N、S418D、S418E、R422H、N476D、N476E、N476G、N476T、I479T、I479Q、Q481A、Q481D、Q481E、Q481H、K525A、K525S、K525T、K525V、N527D和/或Q531R突变。取代突变的非限制性例子在图1-图3中示出。核酸酶对的一个或两个组分的核酸酶(切割)结构域也可以包含位置418、432、441、448、476、481、483、486、487、490、496、499、523、527、537、538和559处的一个或多个突变,包括但不限于ELD、KKR、ELE、KKS。参见例如,美国专利号8,623,618。
因此,本文描述的是切割程序性细胞死亡1(PD1)基因的锌指核酸酶(ZFN)或TALEN,所述ZFN或TALEN包含第一和第二(也称为左和右)ZFN和TALEN,每个ZFN包含与PD1基因中的靶位点结合的ZFP DNA结合结构域和FokI切割结构域,每个TALEN包含与PD1基因中的靶位点结合的TAL效应子DNA结合结构域和FokI切割结构域,其中ZFN或TALEN的FokI切割结构域中的至少一个进一步包含FokI切割结构域中相对于野生型FokI编号的416、418、422、476、479、481、525、527或531中的一处或多处的取代突变。在某些实施方案中,第一和/或第二FokI切割结构域中的取代突变如下:R416E、R416F、R416N、S418D、S418E、R422H、N476D、N476E、N476G、N476T、I479T、I479Q、Q481A、Q481D、Q481E、Q481H、K525A、K525S、K525T、K525V、N527D和/或Q531R。在某些实施方案中,根据前述权利要求中任一项所述的ZFN或TALEN,其中第一和/或第二FokI切割结构域中的取代突变如下:R416E、R416F、R416N、R422H、N476G、N476T、Q481D、Q481H、K525A、K525S、K525T或K525V。在某些实施方案中,所述核酸酶包含具有如SEQ ID NO:3所示的氨基酸序列的第一ZFN,并且第二ZFP DNA结合结构域包含具有如SEQ ID NO:5所示的氨基酸序列的ZFP。
本文描述的人工核酸酶还可以包括在DNA结合结构域内、在识别靶序列的核苷酸的残基之外的一个或多个氨基酸的突变(例如,‘ZFP骨架’(在DNA识别螺旋区之外)或‘TALE骨架’(在RVD之外)的一个或多个突变),所述一个或多个氨基酸可以与DNA骨架上的磷酸非特异性地相互作用。因此,在某些实施方案中,本发明包括ZFP骨架中并非核苷酸靶标特异性所需的阳离子型氨基酸残基的突变。在一些实施方案中,ZFP骨架中的这些突变包含使阳离子型氨基酸残基突变为中性或阴离子型氨基酸残基。在一些实施方案中,ZFP骨架中的这些突变包含使极性氨基酸残基突变至中性或非极性氨基酸残基。在优选实施方案中,突变是在相对于DNA结合螺旋的位置(-5)、(-9)和/或位置(-14)进行。在一些实施方案中,锌指可包含在(-5)、(-9)和/或(-14)的一个或多个突变。在另外的实施方案中,多指锌指蛋白中的一个或多个锌指可以包含(-5)、(-9)和/或(-14)中的突变。在某些实施方案中,锌指蛋白的1、2、3、4、5或6个指状物包含一个或多个骨架突变(例如,在1、2、3、4、5或6个指状物中的(-5))。在一些实施方案中,在(-5)、(-9)和/或(-14)的氨基酸(例如精氨酸(R)或赖氨酸(K))突变为丙氨酸(A)、亮氨酸(L)、Ser(S)、Asp(N)、Glu(E)、Tyr(Y)和/或谷氨酰胺(Q)。在一些实施方案中,位置-5处的Arg(R)改变为Tyr(Y)、Asp(N)、Glu(E)、Leu(L)、Gln(Q)或Ala(A)。在其他实施方案中,位置(-9)处的Arg(R)被Ser(S)、Asp(N)或Glu(E)替代。在另外的实施方案中,位置(-14)处的Arg(R)被Ser(S)或Gln(Q)替代。在其他实施方案中,融合多肽可以包含锌指DNA结合结构域中的突变,其中(-5)、(-9)和/或(-14)位置处的氨基酸在一个或多个指状物(例如,ZFP的3个指状物、4个指状物、5个指状物或6个指状物)中改变为任何组合中的任何上述列出的氨基酸。
本文描述的突变(单独或以任何组合)提供具有增加特异性的PD1核酸酶。
在另一个方面,提供了编码如本文所述的任何工程化切割半结构域或融合蛋白的多核苷酸。因此,还提供了编码一个或多个ZFN或TALEN的一种或多种多核苷酸。
在又另一方面,还提供了包含如本文所述的任何核酸酶、多肽(例如,融合分子或融合多肽)和/或多核苷酸的细胞,包括包含如本文所述的一种或多种ZFN、一种或多种TALEN和/或一种或多种多核苷酸的分离的细胞(或细胞群体)。还提供了由如本文所述的分离的细胞或方法产生的经遗传修饰的细胞的分离群体,其中PD1基因被核酸酶特异性修饰。在某些实施方案中,在分离的细胞群体中,PD1的遗传修饰的靶上与靶外比率大于200。还描述了从如本文所述的经遗传修饰的细胞的分离群体遗传的部分或完全分化的细胞。如本文所述的经遗传修饰的细胞的分离群体可以进一步包含一种或多种另外的遗传修饰,任选地包括使除PD1之外的一种或多种基因(如T细胞受体基因、B2M基因和/或CTLA-4基因)失活,和/或转基因(如CAR转基因)的整合。
在一个实施方案中,所述细胞包含一对融合多肽,一个或两个融合多肽除如本文所述的一个或多个突变之外,还包含残基处的一个或多个另外的突变,例如美国专利8,962,281中所述的工程化切割半结构域。
本文还提供了已经被本发明的多肽和/或多核苷酸修饰的细胞。在一些实施方案中,所述细胞包含核酸酶介导的转基因插入,或核酸酶介导的PD1基因敲除。经修饰的细胞和源自所述经修饰的细胞的任何细胞不一定较长久地包含本发明的核酸酶,但保留由此类核酸酶介导的基因组修饰。
在又另一方面,提供了靶向切割PD1基因的方法;使同源重组在细胞中发生的方法;治疗感染的方法;和/或治疗疾病的方法。这些方法可以在体外、离体或体内或以其组合实践。所述方法涉及通过表达如本文所述的一对融合多肽(即,一对融合多肽,其中一个或两个融合多肽包含如本文所述的工程化切割半结构域)来切割细胞中预定目标区域处的细胞染色质。在某些实施方案中,与包含没有如本文所述突变的切割结构域的核酸酶相比,对靶上位点(例如,PD1基因)相对于对靶外位点(例如,非PD1基因)的靶向切割偏好增加了至少50%至200%(或其间的任何值)或更多,包括50%-60%(或其间的任何值)、60%-70%(或其间的任何值)、70%-80%(或其间的任何值)、80%-90%(或其间的任何值)、90%至200%(或其间的任何值)或>200%的任何值。类似地,使用如本文所述的方法和组合物,靶外位点切割减少了1-100倍或更多倍,包括但不限于1-50倍(或其间的任何值)。在其他方面,如本文所述的成对PD1核酸酶的组分(左和右)以任何比率单独施用。在一些实施方案中,所述左组分和右组分以相等的量施用。在一些实施方案中,工程化核酸酶复合物的工程化切割半结构域配偶体用于接触细胞,其中所述复合物的每个配偶体以1:1的比率或以不等于一比一的与其他配偶体的比率来给予。在一些实施方案中,两个配偶体(半切割结构域)的比率以1:1、1:2、1:3、1:4、1:5、1:6、1:8、1:9、1:10或1:20比率或其间的任何值给出。在其他实施方案中,两个配偶体的比率大于1:30。在其他实施方案中,两个配偶体以被选择为不等于1:1的比率部署。在一些方面,每个配偶体作为mRNA被递送至细胞,或者在病毒或非病毒载体中被递送,其中递送等量或非等量的编码每个配偶体的mRNA或载体。在另外的实施方案中,核酸酶复合物的每个配偶体均可以包含在单个病毒或非病毒载体上,但被有意表达,使得两个配偶体被同等地表达,或者一个配偶体以比另一个更高或更低的值表达,最终向细胞递送的切割半结构域的比率不等于一比一。在一些实施方案中,使用具有不同表达效率的不同启动子来表达每个切割半结构域。在其他实施方案中,使用病毒或非病毒载体将两个切割结构域递送至细胞,其中两者均从相同的开放式阅读框表达,但编码两个配偶体的基因被序列(例如自切割2A序列或IRES)分开,从而导致3'配偶体以较低速率表达,使得两个配偶体的比率为1:2、1:3、1:4、1:5、1:6、1:8、1:9、1:10或1:20比率,或其间的任何值。在其他实施方案中,两个配偶体以相等比率,或者以被选择为不等于1:1的比率部署。
还提供了改变内源PD1基因的方法,例如以引入靶向突变。在某些实施方案中,遗传修饰PD1基因的方法包括向细胞中引入一种或多种靶向核酸酶以在预定位点处的细胞染色质中产生双链断裂,并引入与断裂区域中的细胞染色质的核苷酸序列具有同源性的供体多核苷酸。细胞DNA修复过程通过双链断裂的存在而被激活,并且供体多核苷酸被用作修复断裂的模板,从而导致将供体的全部或部分核苷酸序列引入细胞染色质。因此,可以改变细胞染色质中的PD1序列,并且在某些实施方案中,可以将所述PD1序列转化为供体多核苷酸中存在的序列。
靶向改变包括但不限于点突变(即,单个碱基对向不同碱基对的转化)、取代(即,多个碱基对向相同长度的不同序列的转化)、插入一个或多个碱基对、缺失一个或多个碱基对以及前述序列改变的任何组合。改变还可以包括作为编码序列的一部分的碱基对的转化,使得经编码的氨基酸被改变。
供体多核苷酸可以是DNA或RNA,可以是线型或环状,并且可以是单链或双链。它可以作为裸核酸、作为与一种或多种递送剂(例如,脂质体、纳米颗粒、泊洛沙姆)的复合物或包含在病毒递送媒介物(例如像腺病毒、慢病毒或腺相关病毒(AAV))中被递送至细胞。供体序列的长度范围可以为10至5,000个核苷酸(或其间核苷酸的任何整数值)或更长。在一些实施方案中,供体包含侧接有与所靶向切割位点具有同源性的区域的全长基因。在一些实施方案中,供体缺少同源性区域并通过非同源性依赖性机制(即,NHEJ)被整合至靶基因座中。在其他实施方案中,供体包含更小的核酸片段,其侧翼为同源区域以用于细胞中(即用于基因校正)。在一些实施方案中,供体包含编码功能或结构组分(如shRNA、RNAi、miRNA等)的基因。在其他实施方案中,供体包含编码结合至目标基因和/或调节其表达的调节元件的序列。在其他实施方案中,供体是结合至目标基因和/或调节其表达的目标调节蛋白(例如,ZFP TF、TALE TF或CRISPR/Cas TF)。
在又另一方面,还提供了包含如本文所述的任何多肽(例如,融合分子)和/或多核苷酸的细胞。在一个实施方案中,所述细胞包含一对融合分子,每个融合分子均包含如本文所公开的切割结构域。本文还提供了使用如本文所述的一种或多种核酸酶产生的经遗传修饰的细胞(例如,T细胞)的分离群体,其中PD1基因被所述核酸酶特异性修饰(如与其他基因相比)。在这些细胞中,这些细胞中的PD1基因是被一种或多种核酸酶遗传修饰的(例如,通过插入和/或缺失(“插入缺失(indel)”)而突变),但与没有本文描述的一个或多个FokI突变的PD1核酸酶相比,使用这些核酸酶进行的PD1基因之外的遗传修饰减少了1-100倍或更多倍,包括但不限于1-50倍(或其间的任何值)。在某些实施方案中,在分离的细胞群体中,由一种或多种核酸酶进行的遗传修饰的少于1%(例如,少于0.5%)在PD1基因之外。在某些方面,由核酸酶产生的群体的至少40%细胞包括对PD1的修饰(插入缺失),而少于0.05%的细胞包括由核酸酶进行的靶外(非PD1)遗传修饰。在仍其他实施方案中,与使用没有本文描述的FokI突变的PD1靶向核酸酶的遗传修饰的靶上/靶外比相比,使用本文描述的核酸酶产生的经遗传修饰细胞的分离群体具有遗传修饰的更大相对靶上/靶外(PD1/非PD1)比,任选地其中由本发明的核酸酶制备的细胞中的靶上/靶外遗传修饰比是100或更大、或150或更大、或200或更大,如附图所示。细胞包括经培养的细胞、生物体中的细胞以及已经从生物体中移除以进行处理的细胞,在所述情况下将在处理后将细胞和/或其后代返回生物体中。细胞染色质中的目标区域可以是例如基因组序列或其部分。
包含如本文所述的一种或多种ZFN或TALEN、一种或多种多核苷酸或细胞的分离群体的组合物要求用于治疗疾病或障碍如癌症。还提供了一种治疗受试者的疾病或障碍(例如,癌症)的方法,所述方法包括切割PD1基因,向有需要的受试者施用如本文所述的一种或多种ZFN或TALEN、一种或多种多核苷酸或细胞的分离群体。
在另一个方面,本文描述了一种试剂盒,其包含如本文所述的融合蛋白或编码如本文所述的一种或多种锌指蛋白、切割结构域和/或融合蛋白的多核苷酸;辅助试剂;以及任选的说明书和适宜的容器。所述试剂盒还可以包括一种或多种核酸酶或编码此类核酸酶的多核苷酸。
从整体而言鉴于本公开文本,这些和其他方面对于熟练技术人员来说都是易于清楚的。
附图说明
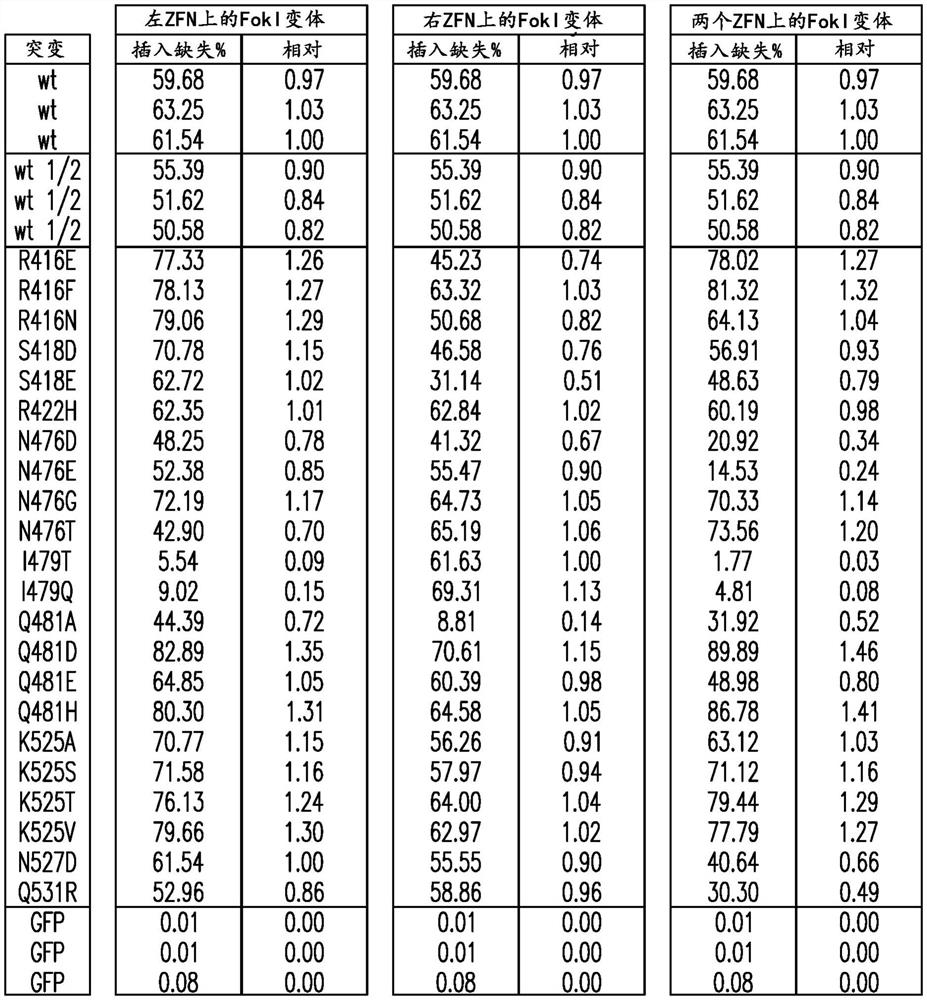
图1A和图1B示出了针对所指示PD1 ZFN对的FokI变体筛选数据的结果。图1A示出了所指示变体的结果。对于所述对中的每个ZFN,从具有500ng mRNA的剂量的单个复制品中确定值。“wt”指示先前报道的PD1ZFN对(例如,12942/25029)。“wt1/2”指示先前报道的PD1ZFN对,其在250ng的每种ZFN单体的剂量下测试。相对指示作为“wt”样品的三次重复测量的平均值的分数的插入缺失%。左侧两列值是用与先前报道的右ZFN组合的左ZFN中指示的FokI确定的。中间列的值类似,但指示的FokI变体位于右ZFN上,且左ZFN未修饰。最右列的值是用左ZFN和右ZFN两者上指示的FokI变体确定的。图1B示出了如所指示在其FokI结构域内带有单残基取代的PD1 ZFN二聚体的变体的靶上活性。“半剂量”亲本样品使用250ng RNA进行递送。为了突出显示相对信号强度,表格值被嵌入灰色热图中。箭头突出显示了表现为完全保留高水平靶上活性的变体,这些变体在后续研究中得到了进一步表征。
图2A和图2B是示出在多个剂量下测试的PD1 ZFN对的所指示FokI变体的靶上(“PD1”)和靶外(“OT1”、“OT2”和“OT3”)切割活性(插入缺失%)的表格。图2A示出了R416E、R416F、R416N、R422H、N476G、N476T、Q481D、Q481H、K525A、K525S、K525T、K525V(和GFP对照)的结果。图2B示出了图2A中所示数据的子集(R416E、R46N、Q481D、Q481H、K525T和K525V)。值是三个生物复制品的平均值。“wt”指示先前报道的PD1 ZFN对(12942/25029)。对于其他样品,在左ZFN和右ZFN两者上均使用了左列中所指示的FokI变体。所述对中每个ZFN的mRNA剂量在第二列中指示。还示出了靶上位点与靶外位点之间的相对切割活性(最后一列“靶上/靶外”)。
图3示出了在其FokI结构域内带有单残基取代的PD1 ZFN二聚体的变体的靶上和靶外活性。每个变体均作为二聚体进行测试,其中两个ZFN均带有所指示的取代(第1列,“亲本”指示在进行所指示的FokI突变之前的对(例如,12942/25029))。使用指示量的每种ZFN单体的mRNA(第2列),通过核转染将ZFN递送至人K562细胞,随后在第3天进行基因组DNA分离,并对预期靶标处的插入缺失进行深度测序分析。第3列提供了在预期靶标处测量的插入缺失%,且第4-6列指示在三个先前已知的靶外位点处的插入缺失%。值是三个生物复制品的平均值。第7列列出了靶外插入缺失之和,且第8列示出了靶上:靶外插入缺失比率(=第3列/第7列)。
图4示出了在指示剂量下,在靶上和靶外位点处使用所指示TALEN对(靶向CCR5)切割的结果。
每个变体均作为二聚体进行测试,其中两个TALEN均带有所指示的取代(第1列,“亲本”指示没有所指示FokI突变的CCR5 TALEN对)。使用指示量的每种ZFN单体的mRNA(第2列),通过核转染将TALEN递送至人K562细胞,随后在第3天进行基因组DNA分离,并对预期靶标处的插入缺失进行深度测序分析。第3列提供了在预期靶标处测量的插入缺失%,且第4-6列指示在三个先前已知的靶外位点处的插入缺失%。值是三个生物复制品的平均值。第7列列出了靶外插入缺失之和,且第8列给出了靶上:靶外插入缺失比率(=第3列/第7列)。为了帮助突出显示相对信号强度,表格值被嵌入灰色热图中。
具体实施方式
本文公开了用于通过差异减少靶外切割来增加PD1基因的靶上工程化核酸酶切割的特异性的方法和组合物。
概述
除非另有指示,否则方法的实践以及本文所公开的组合物的制备和使用采用分子生物学、生物化学、染色质结构和分析、计算化学、细胞培养、重组DNA和相关领域的常规技术,这些技术是本领域所熟知的。这些技术在文献中有充分解释。参见例如,Sambrook等人MOLECULAR CLONING:A LABORATORY MANUAL,第二版,Cold Spring Harbor LaboratoryPress,1989和第三版,2001;Ausubel等人,CURRENT PROTOCOLS IN MOLECULAR BIOLOGY,John Wiley&Sons,纽约,1987和定期更新;丛书METHODS IN ENZYMOLOGY,Academic Press,圣地亚哥;Wolffe,CHROMATIN STRUCTURE AND FUNCTION,第三版,Academic Press,圣地亚哥,1998;METHODS IN ENZYMOLOGY,第304卷,“Chromatin”(P.M.Wassarman和A.P.Wolffe编辑),Academic Press,圣地亚哥,1999;和METHODS IN MOLECULAR BIOLOGY,第119卷,“Chromatin Protocols”(P.B.Becker编辑)Humana Press,托托瓦,1999。
定义
术语“核酸”、“多核苷酸”和“寡核苷酸”可互换使用,并且是指呈线性或环状构象并且呈单链或双链形式的脱氧核糖核苷酸或核糖核苷酸聚合物。出于本公开文本的目的,这些术语不应被解释为关于聚合物的长度为限制性的。这些术语可以涵盖天然核苷酸的已知类似物,以及在碱基、糖和/或磷酸部分(例如,硫代磷酸酯骨架)中被修饰的核苷酸。通常,特定核苷酸的类似物具有相同的碱基配对特异性;即,A的类似物将与T进行碱基配对。
术语“多肽”、“肽”和“蛋白质”可互换使用以指代氨基酸残基的聚合物。所述术语还适用于如下氨基酸聚合物,其中一个或多个氨基酸是相应天然存在的氨基酸的化学类似物或经修饰衍生物。
“结合”是指高分子之间(例如,蛋白质与核酸之间)的序列特异性非共价相互作用。并非结合相互作用的所有组分都需要是序列特异性的(例如,与DNA骨架中的磷酸残基接触),只要所述相互作用作为整体是序列特异性的即可。此类相互作用的特征通常是解离常数(K
“结合蛋白”是能够非共价结合至另一分子的蛋白质。结合蛋白可以结合至例如DNA分子(DNA结合蛋白)、RNA分子(RNA结合蛋白)和/或蛋白质分子(蛋白质结合蛋白)。在蛋白质结合蛋白的情况下,其可以结合至自身(以形成同二聚体、同三聚体等),和/或其可以结合至一种或多种不同蛋白质的一个或多个分子。结合蛋白可以具有多于一种类型的结合活性。例如,锌指蛋白具有DNA结合、RNA结合和蛋白质结合活性。在RNA指导的核酸酶系统的情况下,RNA指导对于核酸酶组分(Cas9或Cfp1)是异源的,并且两者均可以被工程化。
“DNA结合分子”是可以与DNA结合的分子。这种DNA结合分子可以是多肽、蛋白质的结构域、较大蛋白质内的结构域或多核苷酸。在一些实施方案中,所述多核苷酸是DNA,而在其他实施方案中,所述多核苷酸是RNA。在一些实施方案中,所述DNA结合分子是核酸酶的蛋白质结构域(例如FokI结构域),而在其他实施方案中,所述DNA结合分子是RNA指导的核酸酶(例如Cas9或Cfp1)的指导RNA组分。
“DNA结合蛋白”(或结合结构域)是蛋白质或较大蛋白质内的结构域,其以序列特异性方式(例如通过一个或多个锌指或者通过分别与锌指蛋白或TALE中的一个或多个RVD相互作用)结合DNA。术语锌指DNA结合蛋白通常缩写为锌指蛋白或ZFP。
“锌指DNA结合蛋白”(或结合结构域)是蛋白质或者较大蛋白质内的结构域,其以序列特异性方式通过一个或多个锌指结合DNA,所述锌指是结合结构域内通过锌离子配位而稳定其结构的氨基酸序列的区域。术语锌指DNA结合蛋白通常缩写为锌指蛋白或ZFP。术语“锌指核酸酶”包括一种ZFN以及二聚化以切割靶基因的一对ZFN(所述对中的成员被称为“左和右”或“第一和第二”或“对”)。
“TALE DNA结合结构域”或“TALE”是包含一个或多个TALE重复结构域/单元的多肽。重复结构域参与TALE与其同源靶DNA序列的结合。单个“重复单元”(也称为“重复”)的长度通常为33-35个氨基酸,并且表现出与天然存在的TALE蛋白中的其他TALE重复序列具有至少一些序列同源性。参见例如,美国专利号8,586,526,将其通过引用以其整体并入本文。术语“TALEN”包括一种TALEN以及二聚化以切割靶基因的一对TALEN(所述对中的成员被称为“左和右”或“第一和第二”或“对”)。
锌指和TALE DNA结合结构域可以经“工程化”以结合到预定的核苷酸序列,例如通过对天然存在的锌指蛋白的识别螺旋区工程化(改变一个或多个氨基酸)或通过对DNA结合中涉及的氨基酸(“重复可变双残基”或RVD区域)工程化。因此,工程化锌指蛋白或TALE蛋白是非天然存在的蛋白质。用于工程化锌指蛋白和TALE的方法的非限制性例子是设计和选择。经设计的蛋白是在自然界中不存在的蛋白质,其设计/组成主要源自合理标准。设计的合理标准包括应用替换规则和计算机化算法,以用于处理存储现有ZFP或TALE设计和结合数据的信息的数据库中的信息。参见例如,美国专利号8,586,526;6,140,081;6,453,242;和6,534,261;还参见WO 98/53058;WO 98/53059;WO 98/53060;WO 02/016536和WO 03/016496。
“所选择的”锌指蛋白、TALE蛋白或CRISPR/Cas系统并非在自然界中发现,其产生主要来自经验过程,如噬菌体展示、相互作用陷阱、合理设计或杂交体选择。参见例如,US5,789,538;US 5,925,523;US 6,007,988;US 6,013,453;US 6,200,759;WO 95/19431;WO96/06166;WO 98/53057;WO 98/54311;WO 00/27878;WO 01/60970;WO 01/88197和WO 02/099084。
“TtAgo”是原核Argonaute蛋白,被认为参与基因沉默。TtAgo源自细菌嗜热栖热菌(Thermus thermophilus)。参见例如Swarts等人,同上;G.Sheng等人,(2013)Proc.Natl.Acad.Sci.U.S.A.111,652)。“TtAgo系统”是通过TtAgo酶切割所需的所有组分,包括例如指导DNA。
“重组”是指两个多核苷酸之间交换遗传信息的过程,包括但不限于通过非同源末端接合(NHEJ)进行的捕捉以及同源重组。出于本公开文本的目的,“同源重组(HR)”是指这种交换的特化形式,其发生于例如细胞中的双链断裂通过同源定向修复机制修复期间。这个过程需要核苷酸序列同源性,使用“供体”分子为模板来修复“靶”分子(即,经历双链断裂的分子),并且因为其导致遗传信息从供体转移至靶标,被不同地称为“非交换型基因转化”或“短束基因转化”。不希望受任何特定理论约束,这种转移可以涉及在断裂的靶标与供体之间形成的异源双链体DNA的错配修正,和/或“合成依赖性链退火”(其中使用供体重合成将成为靶标的一部分的遗传信息),和/或相关过程。这种特化的HR通常导致靶分子序列的改变,使得供体多核苷酸的部分或全部序列掺入靶多核苷酸中。
在本公开文本的某些方法中,如本文所述的一种或多种靶向核酸酶在预定位点(例如,目标基因或基因座)处的靶序列(例如,细胞染色质)中产生双链断裂(DSB)。DSB介导如本文所述构建体(例如供体)的整合。任选地,所述构建体与断裂区域中的核苷酸序列具有同源性。表达构建体可以是物理整合的,或者可替代地,表达盒用作通过同源重组修复断裂的模板,从而导致将表达盒中的全部或部分核苷酸序列引入细胞染色质中。因此,可以改变细胞染色质中的第一序列,并且在某些实施方案中,可以将所述第一序列转化为表达盒中存在的序列。因此,术语“替代”(“replace”或“replacement”)的使用可以理解为代表一个核苷酸序列被另一个核苷酸序列替代(即,在信息意义上的序列替代),并且不一定需要一个多核苷酸被另一个多核苷酸物理地或化学地替代。
在本文描述的任何方法和组合物(例如,核酸酶、使用这些核酸酶制备的细胞等)中,另外的工程化核酸酶可以用于细胞内另外的靶位点的另外的双链切割。
在用于细胞染色质的目标区域中的序列的靶向重组和/或替代和/或改变的方法的某些实施方案中,通过与外源“供体”核苷酸序列的同源重组来改变染色体序列。如果存在与断裂区同源的序列,则通过细胞染色质中存在双链断裂来刺激这种同源重组。
在本文描述的任何方法和组合物(例如,核酸酶、使用这些核酸酶制备的细胞等)中,第一核苷酸序列(“供体序列”)可以包含与目标区域中的基因组序列同源但不相同的序列,从而刺激同源重组以在目标区域中插入不同序列。因此,在某些实施方案中,供体序列中与目标区域中的序列同源的部分展现出与被替代的基因组序列约80%至99%(或其之间的任何整数)的序列同一性。在其他实施方案中,例如,如果超过100个连续碱基对的供体与基因组序列之间只有1个核苷酸不同,则供体与基因组序列之间的同源性高于99%。在某些情况下,供体序列的非同源部分可以包含在目标区域中不存在的序列,从而将新序列引入目标区域中。在这些情况下,非同源序列的侧翼通常为与目标区域中的序列同源或相同的50-1,000个碱基对(或其间的任何整数值)或大于1,000的任意数量的碱基对的序列。在其他实施方案中,供体序列与第一序列非同源,并通过非同源重组机制插入基因组中。
本文描述的任何方法可以用于通过靶向整合供体序列或通过切割一个或多个靶序列随后进行易错NHEJ介导的修复来破坏一个或多个目标基因的表达而使细胞中的一种或多种靶序列部分或完全失活。还提供了具有部分或完全失活的基因的细胞系。
此外,本文描述的靶向整合的方法也可用于整合一种或多种外源序列。外源核酸序列可以包含例如一种或多种基因或cDNA分子、或任何类型的编码或非编码序列、以及一种或多种控制元件(例如启动子)。另外,外源核酸序列可产生一种或多种RNA分子(例如,小发夹RNA(shRNA)、抑制性RNA(RNAi)、微小RNA(miRNA)等)。
“切割”是指DNA分子的共价骨架的断裂。切割可通过多种方法来引发,包括但不限于磷酸二酯键的酶水解或化学水解。单链切割和双链切割都是可能的,并且双链切割可以作为两个不同的单链切割事件的结果而发生。DNA切割可导致产生平头末端或交错末端。在某些实施方案中,融合多肽用于靶向的双链DNA切割。
“切割半结构域”是与第二多肽(相同或不同)形成具有切割活性(优选双链切割活性)的复合物的多肽序列。术语“第一和第二切割半结构域”、“+和-切割半结构域”以及“右和左切割半结构域”可互换使用,以指代二聚化的切割半结构域对。术语“切割结构域”与术语“切割半结构域”可互换使用。术语“FokI切割结构域”包括如本文所示的FokI序列以及任何FokI同源物。
“工程化切割半结构域”是已经被修饰以便与另一个切割半结构域(例如,另一个工程化切割半结构域)形成专性异二聚体的切割半结构域。
术语“序列”是指任何长度的核苷酸序列,其可以是DNA或RNA;可以是线性、环状或分支的,并且可以是单链或双链的。术语“转基因”是指被插入基因组中的核苷酸序列。转基因可以具有任何长度,例如长度介于2与100,000,000个核苷酸之间(或其间或高于其的任何整数值),优选地长度介于约100与100,000个核苷酸之间(或其间的任何整数),更优选地长度介于约2000与20,000个核苷酸之间(或其间的任何值)并且甚至更优选,介于约5与15kb之间(或其间的任何值)。
“染色体”是包含细胞基因组的全部或一部分的染色质复合物。细胞的基因组通常以其核型为特征,所述核型是构成所述细胞基因组的所有染色体的集合。细胞的基因组可以包含一条或多条染色体。
“游离体”是复制型核酸,核蛋白复合物或包含不属于细胞染色体核型的一部分的核酸的其他结构。游离体的例子包括质粒、微环和某些病毒基因组。本文描述的肝特异性构建体可以维持游离状态,或者可替代地,可以稳定地整合到细胞中。
“外源”分子是并非在细胞中正常存在,但可以通过一种或多种遗传、生物化学或其他方法引入细胞中的分子。“在细胞中正常存在”是关于细胞的特定发育阶段和环境条件来确定。因此,例如,仅在肌肉的胚胎发育期间存在的分子关于成体肌肉细胞是外源分子。类似地,通过热休克引入的分子关于非热休克细胞是外源分子。外源分子可以包含例如功能失常的内源分子或功能正常的内源分子的功能失常形式的功能形式。
外源分子尤其可以是小分子,如通过组合化学工艺产生的小分子,或高分子,如蛋白质、核酸、碳水化合物、脂质、糖蛋白、脂蛋白、多糖、上述分子的任何经修饰衍生物、或者包含一种或多种上述分子的任何复合物。核酸包括DNA和RNA,可以是单链或双链的;可以是线性、分支或环状的;并且可以是任何长度。核酸包括能够形成双螺旋的那些,以及形成三螺旋的核酸。参见例如,美国专利号5,176,996和5,422,251。蛋白质包括但不限于DNA结合蛋白、转录因子、染色质重塑因子、甲基化的DNA结合蛋白、聚合酶、甲基化酶、脱甲基酶、乙酰基酯酶、脱乙酰酶、激酶、磷酸酶、连接酶、去泛素化酶(deubiquitinases)、整合酶、重组酶、连接酶、拓扑异构酶、促旋酶和解螺旋酶。
外源分子可以是与内源分子类型相同的分子,例如,外源蛋白质或核酸。例如,外源核酸可以包含引入细胞中的感染性病毒基因组、质粒或游离体,或在细胞中并非正常存在的染色体。将外源分子引入细胞中的方法是本领域技术人员已知的,并且包括但不限于脂质介导的转移(即,脂质体,包括中性和阳离子型脂质)、电穿孔、直接注射、细胞融合、粒子轰击、磷酸钙共沉淀、DEAE-葡聚糖介导的转移和病毒载体介导的转移。外源分子还可以是与内源分子类型相同的分子,但源自与细胞来源不同的物种。例如,可将人类核酸序列引入最初源自小鼠或仓鼠的细胞系中。将外源分子引入植物细胞中的方法是本领域技术人员已知的,并且包括但不限于原生质体转化、碳化硅(例如,WHISKERS
相比之下,“内源”分子是在特定发育阶段在特定环境条件下在特定细胞中正常存在的分子。例如,内源核酸可以包含染色体、线粒体、叶绿体或其他细胞器的基因组、或天然存在的游离型核酸。另外的内源分子可以包括蛋白质,例如转录因子和酶。
如本文所用,术语“外源核酸的产物”包括多核苷酸和多肽产物两者,例如,转录产物(多核苷酸,如RNA)和翻译产物(多肽)。
“融合”分子是其中两个或更多个亚基分子被连接(优选地共价连接)的分子。亚基分子可以是相同化学类型的分子,或者可以是不同化学类型的分子。融合分子的例子包括但不限于融合蛋白(例如,蛋白质DNA结合结构域与切割结构域之间的融合物)、与切割结构域可操作地缔合的多核苷酸DNA结合结构域(例如,sgRNA)之间的融合物,以及融合核酸(例如,编码融合蛋白的核酸)。
细胞中融合蛋白的表达可以源自将融合蛋白递送至细胞或通过将编码融合蛋白的多核苷酸递送至细胞来获得,其中多核苷酸发生转录,并且转录物发生翻译,以产生融合蛋白。反式剪接、多肽切割和多肽连接也可以参与蛋白质在细胞中的表达。将多核苷酸和多肽递送至细胞的方法呈现于本公开文本中的其他地方。
出于本公开文本的目的,“基因”包括编码基因产物(参见下文)的DNA区域,以及所有调节基因产物的产生的DNA区域,不论此类调节序列是否与编码序列和/或所转录序列相邻。因此,基因包括但不一定限于启动子序列、终止子、翻译调节序列如核糖体结合位点和内部核糖体进入位点、增强子、沉默子、隔离子、边界元件、复制起点、基质附着位点以及基因座控制区。
“基因表达”是指将基因中所含的信息转化为基因产物。基因产物可以是基因的直接转录产物(例如,mRNA、tRNA、rRNA、反义RNA、核酶、结构RNA或任何其他类型的RNA)或通过mRNA的翻译产生的蛋白质。基因产物还包括通过如加帽、聚腺苷酸化、甲基化和编辑的过程修饰的RNA,以及通过例如甲基化、乙酰化、磷酸化、泛素化、ADP核糖基化、豆蔻酰化(myristilation)和糖基化修饰的蛋白质。
对基因表达的“调节”是指基因活性的变化。对表达的调节可以包括但不限于基因激活和基因阻遏。基因组编辑(例如,切割、改变、失活、随机突变)可以用于调节表达。基因失活是指如与不包括如本文所述的ZFP、TALE或CRISPR/Cas系统的细胞相比,基因表达的任何减少。因此,基因失活可以是部分失活或完全失活。
“目标区域”是细胞染色质的任何区域,如例如基因或者基因内或与基因相邻的非编码序列,其中可期望所述区域结合外源分子。结合可以用于靶向DNA切割和/或靶向重组的目的。例如,目标区域可以存在于染色体、游离体、细胞器基因组(例如,线粒体、叶绿体)或感染性病毒基因组中。目标区域可以位于基因的编码区内,位于经转录非编码区(例如前导序列、尾随序列或内含子)内,或位于非转录区内在编码区的上游或下游。目标区域的长度可以小至单一核苷酸对或多达2,000个核苷酸对,或任何整数值的核苷酸对。
“安全港”基因座是基因组内的基因座,在其中可以插入基因而对宿主细胞没有任何有害作用。最有益的是如下安全港基因座:其中插入的基因序列的表达不受邻近基因的任何通读表达的干扰。由一种或多种核酸酶靶向的安全港基因座的非限制性例子包括CCR5、HPRT、AAVS1、Rosa和白蛋白。参见例如,美国专利号7,951,925;8,771,985;8,110,379;7,951,925;美国公开号20100218264;20110265198;20130137104;20130122591;20130177983;20130177960;20150056705和20150159172。
“报告基因”或“报告序列”是指产生蛋白质产物的任何序列,所述蛋白质产物在常规测定中优选地但不一定易于测量。适宜报告基因包括但不限于编码介导抗生素抗性(例如,氨苄青霉素抗性、新霉素抗性、G418抗性、嘌呤霉素抗性)的蛋白质的序列、编码有色或荧光或发光蛋白(例如,绿色荧光蛋白、增强绿色荧光蛋白、红色荧光蛋白、萤光素酶)和介导增强的细胞生长和/或基因扩增的蛋白质(例如,二氢叶酸还原酶)的序列。表位标签包括例如一个或多个拷贝的FLAG、His、myc、Tap、HA或任何可检测的氨基酸序列。“表达标签”包括编码报道物的序列,其可与所需基因序列可操作连接以监测目标基因的表达。
“真核”细胞包括但不限于真菌细胞(如酵母)、植物细胞、动物细胞、哺乳动物细胞和人细胞(例如,T细胞),包括干细胞(多能和多潜能)。
关于两个或更多个组分(如序列元件)的并列,术语“操作性连接”和“操作性地连接”(或“可操作连接”)可互换使用,其中所述组分经排列使得两个组分正常发挥功能并允许以下可能性:至少一个所述组分可以介导对至少一个其他组分发挥的功能。通过说明,如果转录调节序列响应于一种或多种转录调节因子的存在或不存在而控制编码序列的转录水平,则转录调节序列如启动子操作性地连接至编码序列。转录调节序列通常与编码序列顺式操作性连接,但无需与其直接相邻。例如,增强子是与编码序列操作性连接的转录调节序列,即使它们不邻接。
蛋白质、多肽或核酸的“功能性片段”是如下蛋白质、多肽或核酸:其序列与全长蛋白质、多肽或核酸不同,但保留与全长蛋白质、多肽或核酸相同的功能。功能性片段可以具有与相应的天然分子相比更多、更少或相同的残基数,和/或可以含有一个或多个氨基酸或核苷酸取代。确定核酸或蛋白质功能(例如,编码功能、与另一核酸杂交的能力、酶活性测定)的方法是本领域所熟知的。
多核苷酸“载体”或“构建体”能够将基因序列转移至靶细胞。通常,“载体构建体”、“表达载体”、“表达构建体”、“表达盒”和“基因转移载体”意指能够引导目标基因表达并且可以将基因序列转移至靶细胞的任何核酸构建体。因此,所述术语包括克隆和表达媒介物,以及整合载体。
术语“受试者”和“患者”可互换使用,并且是指哺乳动物如人患者和非人灵长类动物,以及实验动物如兔子、狗、猫、大鼠、小鼠和其他动物。因此,如本文所用的术语“受试者”或“患者”意指可以向其施用本发明的表达盒的任何哺乳动物患者或受试者。本发明的受试者包括患有障碍的受试者。
如本文所用的术语“治疗”(“treating”和“treatment”)是指减轻症状的严重性和/或频率,消除症状和/或根本原因,预防症状和/或其根本原因的发生,以及改善或补救损伤。癌症、单基因病和移植物抗宿主疾病是可以使用本文描述的组合物和方法治疗的病症的非限制性例子。
“染色质”是包含细胞基因组的核蛋白结构。细胞染色质包含核酸(主要是DNA)和蛋白质(包括组蛋白和非组蛋白染色体蛋白)。大多数的真核细胞染色质以核小体的形式存在,其中核小体核心包含大约150个碱基对的DNA,所述DNA与八聚体相缔合,所述八聚体包含组蛋白H2A、H2B、H3和H4各两个;并且接头DNA(长度可变,取决于生物体)在核小体核心之间延伸。组蛋白H1分子通常与接头DNA相缔合。出于本公开文本的目的,术语“染色质”意为涵盖原核和真核的所有类型的细胞核蛋白。细胞染色质包含染色体和游离体染色质。
“可及区域”是细胞染色质中的位点,其中存在于核酸中的靶位点可以被识别靶位点的外源分子结合。不希望受任何特定理论约束,据信可及区域是未被包装到核小体结构中的区域。可及区域的独特结构通常可以通过其对化学和酶探针(例如,核酸酶)的敏感性来检测。
“靶位点”或“靶序列”是定义核酸的一部分的核酸序列,结合分子将结合至所述部分,前提是存在用于结合的充分条件。例如,序列5'-GAATTC-3'是Eco RI限制性核酸内切酶的靶位点。“预期的”或“靶上”序列是结合分子意图结合的序列,并且“非预期的”或“靶外”序列包括由并非预期靶标的结合分子结合的任何序列。
DNA结合分子/结构域
本文描述了包含与任何目标基因或基因座中的靶位点特异性地结合的DNA结合分子/结构域的组合物。任何DNA结合分子/结构域均可以用于本文公开的组合物和方法,包括但不限于锌指DNA结合结构域、TALE DNA结合结构域、CRISPR/Cas核酸酶的DNA结合部分(指导或sgRNA)、或来自大范围核酸酶的DNA结合结构域。
在某些实施方案中,DNA结合结构域包含锌指蛋白。优选地,锌指蛋白是非天然存在的,因为其被工程化为结合至所选的靶位点。参见例如,Beerli等人(2002)NatureBiotechnol.20:135-141;Pabo等人(2001)Ann.Rev.Biochem.70:313-340;Isalan等人(2001)Nature Biotechnol.19:656-660;Segal等人(2001)Curr.Opin.Biotechnol.12:632-637;Choo等人(2000)Curr.Opin.Struct.Biol.10:411-416;美国专利号6,453,242;6,534,261;6,599,692;6,503,717;6,689,558;7,030,215;6,794,136;7,067,317;7,262,054;7,070,934;7,361,635;7,253,273;和美国专利公开号2005/0064474;2007/0218528;2005/0267061,上述文献全部通过引用以其整体并入本文。
在某些实施方案中,DNA结合结构域包含与PD1基因中的靶位点结合的锌指蛋白,例如,如美国专利公开号2012/0060230(例如,表1)中所公开的,所述专利通过引用以其整体并入本文。适宜ZFP的非限制性例子包括指定为12942和25029的ZFP,其具有美国专利号8,563,314的表2和表3中所示的识别螺旋区域。在某些实施方案中,ZFP DNA结合结构域具有如SEQ ID NO:3或SEQ ID NO:5所示的氨基酸序列。
与天然存在的锌指蛋白相比,工程化的锌指结合结构域可以具有新型结合特异性。工程化方法包括但不限于合理设计和各种类型的选择。合理设计包括例如使用数据库,所述数据库包含三联体(或四联体)核苷酸序列和单独锌指氨基酸序列,其中每个三联体或四联体核苷酸序列与结合特定三联体或四联体序列的锌指的一个或多个氨基酸序列缔合。参见例如,美国专利6,453,242和6,534,261,将其通过引用以其整体并入本文。
包括噬菌体展示和双杂交系统的示例性选择方法披露于美国专利5,789,538;5,925,523;6,007,988;6,013,453;6,410,248;6,140,466;6,200,759;和6,242,568;以及WO98/37186;WO 98/53057;WO 00/27878;WO 01/88197和GB 2,338,237中。另外,例如在美国专利号6,794,136中已经描述了对锌指结合结构域的结合特异性的增强。
另外,如这些和其他参考文献中所披露,锌指结构域和/或多指锌指蛋白可使用任何适宜的接头序列(包括例如,长度为5个或更多个氨基酸的接头)连接在一起。对于长度为6个或更多个氨基酸的示例性接头序列,还参见美国专利号6,479,626;6,903,185;和7,153,949。本文描述的蛋白质可包括蛋白质的单个锌指之间的适宜接头的任何组合。另外,例如在美国专利号6,794,136中已经描述了对锌指结合结构域的结合特异性的增强。
靶位点的选择;用于设计和构建融合蛋白(和编码所述融合蛋白的多核苷酸)的ZFP和方法是本领域技术人员已知的并且详细描述于以下文献中:美国专利号6,140,081;5,789,538;6,453,242;6,534,261;5,925,523;6,007,988;6,013,453;6,200,759;WO 95/19431;WO 96/06166;WO 98/53057;WO 98/54311;WO 00/27878;WO 01/60970WO 01/88197;WO 02/099084;WO 98/53058;WO 98/53059;WO 98/53060;WO 02/016536和WO 03/016496。
另外,如这些和其他参考文献中所披露,锌指结构域和/或多指锌指蛋白可使用任何适宜的接头序列(包括例如,长度为5个或更多个氨基酸的接头)连接在一起。对于长度为6个或更多个氨基酸的示例性接头序列,还参见美国专利号6,479,626;6,903,185;和7,153,949。本文描述的蛋白质可包括蛋白质的单个锌指之间的适宜接头的任何组合。
通常,ZFP包含至少三个指状物。某些ZFP包含四个、五个或六个或更多个指状物。包括三个指状物的ZFP通常识别包括9或10个核苷酸的靶位点;包括4个指状物的ZFP通常识别包括12至14个核苷酸的靶位点;而具有6个指状物的ZFP可以识别包括18至21个核苷酸的靶位点。ZFP还可以是融合蛋白,其包括一个或多个调节结构域,所述结构域可以是转录激活或阻遏结构域。
在一些实施方案中,DNA结合结构域可能源自核酸酶。例如,归巢内切核酸酶和大范围核酸酶的识别序列,如I-SceI、I-CeuI、PI-PspI、PI-Sce、I-SceIV、I-CsmI、I-PanI、I-SceII、I-PpoI、I-SceIII、I-CreI、I-TevI、I-TevII和I-TevIIII是已知的。也参见美国专利号5,420,032;美国专利号6,833,252;Belfort等人(1997)Nucleic Acids Res.25:3379-3388;Dujon等人(1989)Gene 82:115-118;Perler等人(1994)Nucleic Acids Res.22,1125-1127;Jasin(1996)Trends Genet.12:224-228;Gimble等人(1996)J.Mol.Biol.263:163-180;Argast等人(1998)J.Mol.Biol.280:345-353和New England Biolabs目录。另外,可以对归巢核酸内切酶和大范围核酸酶的DNA结合特异性进行工程化以结合非天然靶位点。参见例如,Chevalier等人(2002)Molec.Cell 10:895-905;Epinat等人(2003)NucleicAcids Res.31:2952-2962;Ashworth等人(2006)Nature 441:656-659;Paques等人(2007)Current Gene Therapy7:49-66;美国专利公开号20070117128。
在某些实施方案中,与本文描述的突变体切割结构域一起使用的锌指蛋白包含骨架区域(例如,编号为-1至6的7个氨基酸识别螺旋区域之外的区域)的一个或多个突变(取代、缺失和/或插入),例如在位置-14、-9和/或-5中的一处或多处。一个或多个这些位置处的野生型残基可以缺失、被替代为任何氨基酸残基和/或包括一个或多个另外的残基。在一些实施方案中,位置-5处的Arg(R)改变为Tyr(Y)、Asp(N)、Glu(E)、Leu(L)、Gln(Q)或Ala(A)。在其他实施方案中,位置(-9)处的Arg(R)被Ser(S)、Asp(N)或Glu(E)替代。在另外的实施方案中,位置(-14)处的Arg(R)被Ser(S)或Gln(Q)替代。在其他实施方案中,融合多肽可以包含锌指DNA结合结构域中的突变,其中在一个或多个指状物中(-5)、(-9)和/或(-14)位置处的氨基酸以任何组合改变为任何以上列出的氨基酸,例如锌指蛋白的1、2、3、4、5或6个指状物的骨架突变(例如,(-5)突变)。
在其他实施方案中,DNA结合结构域包含来自转录激活因子样(TAL)效应子(TALE)的工程化结构域,其类似于源自植物病原体黄单胞菌属(Xanthomonas)(参见Boch等人,(2009)Science 326:1509-1512和Moscou和Bogdanove,(2009)Science 326:1501)和罗尔斯通氏菌属(Ralstonia)(参见Heuer等人(2007)Applied and EnvironmentalMicrobiology73(13):4379-4384);美国专利公开号20110301073和20110145940的那些工程化结构域。已知黄单胞菌属的植物病原性细菌会在重要的作物植物中引起许多种疾病。黄单胞菌属的致病性取决于保守的III型分泌(T3S)系统,其将多于25种不同的效应子蛋白注入植物细胞中。所注入的这些蛋白质包括转录激活因子样效应子(TALE)其模拟植物转录激活因子并操纵植物转录组(参见Kay等人(2007)Science318:648-651)。这些蛋白质含有DNA结合结构域和转录激活结构域。最充分表征的TALE之一是来自野油菜黄单胞菌辣椒斑点致病变种(Xanthomonas campestgris pv.Vesicatoria)的AvrBs3(参见Bonas等人(1989)Mol Gen Genet 218:127-136和WO 2010079430)。TALE含有串联重复序列的中心化结构域,每个重复序列含有大约34个氨基酸,所述重复序列是这些蛋白质的DNA结合特异性的关键。另外,它们含有核定位序列和酸性转录激活结构域(综述参见Schornack S等人(2006)J Plant Physiol 163(3):256-272)。另外,在植物致病性细菌青枯雷尔氏菌(Ralstonia solanacearum)中,已经发现名为brg11和hpx17的两种基因与青枯雷尔氏菌生物变型1菌株GMI1000中和生物变型4菌株RS1000中黄单胞菌属的AvrBs3家族是同源的(参见Heuer等人(2007)Appl and Envir Micro 73(13):4379-4384)。这些基因的核苷酸序列彼此98.9%相同,但在hpx17的重复结构域中相差1,575个碱基对的缺失。然而,两种基因产物具有与黄单胞菌属的AvrBs3家族蛋白小于40%序列同一性。
这些TAL效应子的特异性取决于在串联重复序列中发现的序列。重复序列包含大约102个碱基对,并且这些重复序列通常彼此91%-100%同源(Bonas等人,同上)。重复序列的多态性通常位于位置12和13处,并且在位置12和13的高变双残基(重复可变双残基或RVD区域)的身份与TAL效应子的靶序列中连续核苷酸的身份之间似乎存在一一对应关系(参见Moscou和Bogdanove,(2009)Science 326:1501以及Boch,等人(2009)Science326:1509-1512)。通过实验,已经确定这些TAL效应子的DNA识别的天然代码,使得位置12和13的HD序列(重复可变双残基或RVD)导致结合至胞嘧啶(C),NG结合至T,NI结合至A、C、G或T,NN结合至A或G,并且ING结合至T。已经将这些DNA结合重复序列以重复序列的新的组合和数量装配为蛋白质,以制造能与新序列相互作用的人工转录因子,并激活非内源报告基因在植物细胞中的表达(Boch等人,同上)。工程化TAL蛋白已与FokI切割半结构域相连,以产生TAL效应子结构域核酸酶融合物(TALEN),包括带有非典型RVD的TALEN。参见例如,美国专利号8,586,526。
在一些实施方案中,TALEN包含核酸内切酶(例如,FokI)切割结构域或切割半结构域。在其他实施方案中,TALE-核酸酶是大范围TAL。这些大范围TAL核酸酶是包含TALE DNA结合结构域和大范围核酸酶切割结构域的融合蛋白。大范围核酸酶切割结构域作为单体具有活性,并且其活性不需要二聚化。(参见Boissel等人,(2013)Nucl Acid Res:1-13,doi:10.1093/nar/gkt1224)。
在仍其他实施方案中,核酸酶包含紧凑TALEN。这些是将TALE DNA结合结构域连接至TevI核酸酶结构域的单链融合蛋白。融合蛋白可以用作通过TALE区定位的切口酶,或者可以产生双链断裂,取决于TALE DNA结合结构域关于TevI核酸酶结构域定位的位置(参见Beurdeley等人(2013)Nat Comm:1-8DOI:10.1038/ncomms2782)。另外,核酸酶结构域还可展现DNA结合功能性。任何TALEN可以与另外的TALEN(例如,具有一个或多个大范围TALE的一个或多个TALEN(cTALEN或FokI-TALEN))组合使用。
另外,如这些和其他参考文献中所披露,锌指结构域和/或多指锌指蛋白或TALE可使用任何适宜的接头序列(包括例如,长度为5个或更多个氨基酸的接头)连接在一起。对于长度为6个或更多个氨基酸的示例性接头序列,还参见美国专利号6,479,626;6,903,185;和7,153,949。本文描述的蛋白质可包括蛋白质的单个锌指之间的适宜接头的任何组合。另外,例如在美国专利号6,794,136中已经描述了对锌指结合结构域的结合特异性的增强。在某些实施方案中,DNA结合结构域是CRISPR/Cas核酸酶系统的一部分,包括与DNA结合的单一指导RNA(sgRNA)DNA结合分子。参见例如,美国专利号8,697,359和美国专利公开号20150056705和20150159172。编码系统的RNA组分的CRISPR(规律间隔成簇短回文重复序列)基因座和编码蛋白质的cas(CRISPR相关)基因座(Jansen等人,2002.Mol.Microbiol.43:1565-1575;Makarova等人,2002.Nucleic Acids Res.30:482-496;Makarova等人,2006.Biol.Direct 1:7;Haft等人,2005.PLoS Comput.Biol.1:e60)组成CRISPR/Cas核酸酶系统的基因序列。微生物宿主中的CRISPR基因座含有CRISPR相关(Cas)基因以及能够对CRISPR介导的核酸切割的特异性进行编程的非编码RNA元件的组合。
在一些实施方案中,DNA结合结构域是TtAgo系统的一部分(参见Swarts等人,同上;Sheng等人,同上)。在真核生物中,基因沉默是由蛋白质的Argonaute(Ago)家族介导的。在这个范例中,Ago与小(19-31nt)RNA结合。此蛋白质-RNA沉默复合物通过所述小RNA与靶标之间的Watson-Crick碱基配对来识别靶RNA,并且以核酸内切方式切割靶RNA(Vogel(2014)Science 344:972-973)。相比之下,原核Ago蛋白与小的单链DNA片段结合,并可能起检测和去除外来(通常是病毒)DNA的作用(Yuan等人,(2005)Mol.Cell 19,405;Olovnikov等人(2013)Mol.Cell 51,594;Swarts等人,同上)。示例性的原核Ago蛋白包括来自风产液菌(Aquifex aeolicus)、类球红细菌(Rhodobacter sphaeroides)、和嗜热栖热菌(Thermusthermophilus)的那些。
最充分表征的原核Ago蛋白之一是来自嗜热栖热菌的蛋白(TtAgo;Swarts等人,同上)。TtAgo与具有5'磷酸基团的15nt或13-25nt单链DNA片段缔合。TtAgo结合的这种“指导DNA”用于引导蛋白质-DNA复合物结合第三方DNA分子中的Watson-Crick互补DNA序列。一旦这些指导DNA中的序列信息允许鉴定靶DNA,TtAgo-指导DNA复合物就会切割靶DNA。TtAgo-指导DNA复合物与其靶DNA结合时的结构也支持这种机制(G.Sheng等人,同上)。来自类球红细菌的Ago(RsAgo)具有相似的特性(Olivnikov等人,同上)。
可以将任意DNA序列的外源指导DNA加载到TtAgo蛋白上(Swarts等人,同上)。由于TtAgo切割的特异性是由指导DNA引导的,因此,与研究者指定的外源指导DNA形成的TtAgo-DNA复合物将TtAgo靶DNA切割引导至研究者指定的互补靶DNA。以此方式,可以在DNA中产生靶向双链断裂。使用TtAgo-指导DNA系统(或来自其他生物的直系同源Ago-指导DNA系统)允许在细胞内靶向切割基因组DNA。这种切割可以是单链或双链的。为了切割哺乳动物基因组DNA,优选使用为在哺乳动物细胞中表达而优化的TtAgo密码子形式。另外,可能优选的是用体外形成的TtAgo-DNA复合物处理细胞,其中TtAgo蛋白与细胞穿透肽融合。另外,可能优选的是使用已经通过诱变而改变以在37℃下具有改善活性的TtAgo蛋白形式。使用本领域中利用DNA断裂的标准技术,可以使用Ago-RNA介导的DNA切割来影响一系列结果,包括基因敲除、靶向基因添加、基因校正、靶向基因缺失。
因此,可以使用任何DNA结合分子/结构域。
融合分子
还提供了包含如本文所述的DNA结合结构域(例如,ZFP或TALE、CRISPR/Cas组分(如单一指导RNA))和异源调节(功能)结构域(或其功能片段)的融合分子。常见结构域包括,例如转录因子结构域(激活因子、阻遏物、共激活因子、共阻遏物)、沉默子、癌基因(例如,myc、jun、fos、myb、max、mad、rel、ets、bcl、myb、mos家族成员等);DNA修复酶和其相关因子和修饰剂;DNA重排酶和其相关因子和修饰剂;染色质相关蛋白和其修饰剂(例如激酶、乙酰基酯酶和脱乙酰酶);和DNA修饰酶(例如,甲基转移酶、拓扑异构酶、解螺旋酶、连接酶、激酶、磷酸酶、聚合酶、核酸内切酶)和其相关因子和修饰剂。美国专利公开号20050064474;20060188987和2007/0218528有关于DNA结合结构域和核酸酶切割结构域的融合物的细节,所述专利通过引用以其整体并入本文。
用于实现激活的适宜结构域包括HSV VP16激活结构域(参见例如,Hagmann等人,J.Virol.71,5952-5962(1997));核激素受体(参见例如,Torchia等人,Curr.Opin.Cell.Biol.10:373-383(1998));核因子κB的p65亚基(Bitko和Barik,J.Virol.72:5610-5618(1998)以及Doyle和Hunt,Neuroreport8:2937-2942(1997));Liu等人,Cancer Gene Ther.5:3-28(1998));或人工嵌合功能结域,如VP64(Beerli等人,(1998)Proc.Natl.Acad.Sci.USA95:14623-33);以及降解决定子(Molinari等人,(1999)EMBOJ.18,6439-6447)。另外的示例性激活结构域包括Oct 1、Oct-2A、Sp1、AP-2和CTF1(Seipel等人,EMBO J.11,4961-4968(1992))以及p300、CBP、PCAF、SRC1PvALF、AtHD2A和ERF-2。参见例如,Robyr等人(2000)Mol.Endocrinol.14:329-347;Collingwood等人(1999)J.Mol.Endocrinol.23:255-275;Leo等人(2000)Gene 245:1-11;Manteuffel-Cymborowska(1999)Acta Biochim.Pol.46:77-89;McKenna等人(1999)J.SteroidBiochem.Mol.Biol.69:3-12;Malik等人(2000)Trends Biochem.Sci.25:277-283;和Lemon等人(1999)Curr.Opin.Genet.Dev.9:499-504。另外的示例性激活结构域包括但不限于OsGAI、HALF-1、C1、AP1、ARF-5、ARF-6、ARF-7和ARF-8、CPRF1、CPRF4、MYC-RP/GP和TRAB1。参见例如,Ogawa等人(2000)Gene 245:21-29;Okanami等人(1996)Genes Cells 1:87-99;Goff等人(1991)Genes Dev.5:298-309;Cho等人(1999)Plant Mol.Biol.40:419-429;Ulmason等人(1999)Proc.Natl.Acad.Sci.USA 96:5844-5849;Sprenger-Haussels等人(2000)Plant J.22:1-8;Gong等人(1999)Plant Mol.Biol.41:33-44;和Hobo等人(1999)Proc.Natl.Acad.Sci.USA 96:15,348-15,353。
本领域技术人员将清楚,在DNA结合结构域与功能结构域之间形成融合蛋白(或编码所述融合蛋白的核酸)时,激活结构域或与激活结构域相互作用的分子都适合作为功能结构域。本质上能够将激活复合物和/或激活活性(例如,组蛋白乙酰化)募集到靶基因的任何分子都可用作融合蛋白的激活结构域。例如在美国专利公开2002/0115215和2003/0082552以及在WO 02/44376中描述了适用于融合分子中的功能结构域的隔离子结构域、定位结构域、和染色质重塑蛋白,例如含ISWI的结构域和/或甲基结合结构域蛋白。
示例性阻遏结构域包括但不限于KRAB A/B、KOX、TGF-β诱导型早期基因(TIEG)、v-erbA、SID、MBD2、MBD3、DNMT家族的成员(例如,DNMT1、DNMT3A、DNMT3B)、Rb和MeCP2。参见例如,Bird等人(1999)Cell 99:451-454;Tyler等人(1999)Cell 99:443-446;Knoepfler等人(1999)Cell 99:447-450;和Robertson等人(2000)Nature Genet.25:338-342。另外的示例性阻遏结构域包括但不限于ROM2和AtHD2A。参见例如,Chem等人(1996)Plant Cell 8:305-321;和Wu等人(2000)Plant J.22:19-27。
融合分子是通过克隆和生化缀合方法来构建的,所述方法是本领域技术人员所熟知的。融合分子包含DNA结合结构域和功能结构域(例如,转录激活或阻遏结构域)。融合分子还任选地包含核定位信号(例如,来自SV40中型T抗原)和表位标签(例如,FLAG和血凝素)。融合蛋白(和编码所述融合蛋白的核酸)设计为使得在融合物的组分之间保存翻译阅读框。
一方面功能结构域(或其功能性片段)的多肽组分与另一方面非蛋白质DNA结合结构域(例如,抗生素、嵌入剂、小沟结合物、核酸)之间的融合物是通过本领域技术人员已知的生化缀合方法来构建。参见例如,Pierce Chemical Company(Rockford,IL)目录。已经描述用于制造小沟结合物与多肽之间的融合物的方法和组合物。Mapp等人(2000)Proc.Natl.Acad.Sci.USA 97:3930-3935。此外,CRISPR/Cas系统的单一指导RNA与功能结构域缔合形成活性转录调节因子和核酸酶。
在某些实施方案中,靶位点存在于细胞染色质的可及区域。可以按照例如在美国专利号7,217,509和7,923,542中描述的那样确定可及区域。如果在细胞染色质的可及区域中不存在靶位点,则可以产生一个或多个可及区域,如美国专利号7,785,792和8,071,370中所述。在另外的实施方案中,融合分子的DNA结合结构域不论其靶位点是否在可及区域中都能够结合细胞染色质。例如,此类DNA结合结构域能够与接头DNA和/或核小体DNA结合。这种类型的“先锋”DNA结合结构域的例子见于某些类固醇受体和肝细胞核因子3(HNF3)中(Cordingley等人(1987)Cell 48:261-270;Pina等人(1990)Cell 60:719-731;和Cirillo等人(1998)EMBO J.17:244-254)。
融合分子可以用药学上可接受的载体来配制,如本领域技术人员已知。参见例如,Remington's Pharmaceutical Sciences,第17版,1985;以及美国专利号6,453,242和6,534,261。
融合分子的功能性组分/结构域可以选自多种不同组分中的任一种,所述组分能在融合分子通过其DNA结合结构域结合至靶序列后影响基因转录。因此,功能性组分可以包括但不限于各种转录因子结构域,如激活因子、阻遏物、共激活因子、共阻遏物和沉默子。
另外的示例性功能结构域披露于例如美国专利号6,534,261和6,933,113。
也可以选择由外源小分子或配体调节的功能结构域。例如,可以使用
核酸酶
在某些实施方案中,融合蛋白包含DNA结合结构域和切割(核酸酶)结构域。因此,可以使用核酸酶,例如工程化核酸酶来实现基因修饰。工程化核酸酶技术是基于天然存在的DNA结合蛋白的工程化。例如,已经描述了具有调整的DNA结合特异性的归巢核酸内切酶的工程化。Chames等人(2005)Nucleic Acids Res 33(20):e178;Arnould等人(2006)J.Mol.Biol.355:443-458。此外,还描述了ZFP的工程化。参见例如,美国专利号6,534,261;6,607,882;6,824,978;6,979,539;6,933,113;7,163,824;和7,013,219。
此外,ZFP和/或TALE已与核酸酶结构域融合形成ZFN和TALEN,这是功能实体,所述功能实体能够通过其工程化(ZFP或TALE)DNA结合结构域识别其预期核酸靶标,并导致DNA通过核酸酶活性在DNA结合位点附近被切割。参见例如,Kim等人(1996)Proc Nat'l AcadSci USA93(3):1156-1160。最近,此类核酸酶已用于多种生物的基因组修饰。参见例如,美国专利公开20030232410;20050208489;20050026157;20050064474;20060188987;20060063231;和国际公开WO 07/014275。
因此,本文描述的方法和组合物是广泛适用的,并且可包括任何目标核酸酶。核酸酶的非限制性例子包括大范围核酸酶、TALEN和锌指核酸酶。核酸酶可以包含异源DNA结合和切割结构域(例如,锌指核酸酶;大范围核酸酶DNA结合结构域和异源切割结构域),或者可替代地,天然存在的核酸酶的DNA结合结构域可发生改变以结合至所选靶位点(例如,已经工程化以结合至与同源结合位点不同的位点的大范围核酸酶)。
在本文描述的任何核酸酶中,所述核酸酶可以包含工程化TALE DNA结合结构域和核酸酶结构域(例如,核酸内切酶和/或大范围核酸酶结构域),也称为TALEN。工程化这些TALEN蛋白以与用户选择的靶序列进行稳健的、位点特异性的相互作用的方法和组合物已经公布(参见美国专利号8,586,526)。在一些实施方案中,TALEN包含核酸内切酶(例如,FokI)切割结构域或切割半结构域。在其他实施方案中,TALE-核酸酶是大范围TAL。这些大范围TAL核酸酶是包含TALE DNA结合结构域和大范围核酸酶切割结构域的融合蛋白。大范围核酸酶切割结构域作为单体具有活性,并且其活性不需要二聚化。(参见Boissel等人,(2013)Nucl Acid Res:1-13,doi:10.1093/nar/gkt1224)。另外,核酸酶结构域还可展现DNA结合功能性。
在仍其他实施方案中,核酸酶包含紧凑TALEN(cTALEN)。这些是将TALE DNA结合结构域连接至TevI核酸酶结构域的单链融合蛋白。融合蛋白可以用作通过TALE区定位的切口酶,或者可以产生双链断裂,取决于TALE DNA结合结构域关于TevI核酸酶结构域定位的位置(参见Beurdeley等人(2013)Nat Comm:1-8DOI:10.1038/ncomms2782)。任何TALEN可以与另外的TALEN(例如,具有一个或多个大范围TAL的一个或多个TALEN(cTALEN或FokI-TALEN))或另外的DNA切割酶组合使用。
在某些实施方案中,核酸酶包含展现切割活性的大范围核酸酶(归巢核酸内切酶)或其一部分。天然存在的大范围核酸酶识别15-40个碱基对的切割位点,并且通常分为四个家族:LAGLIDADG家族(“LAGLIDADG”公开为SEQ ID NO:6)、GIY-YIG家族、His-Cyst盒家族和HNH家族。示例性归巢核酸内切酶包括I-SceI、I-CeuI、PI-PspI、I-Sce、I-SceIV、I-CsmI、I-PanI、I-SceII、I-PpoI、I-SceIII、I-CreI、I-TevI、I-TevII和I-TevIII。上述示例性归巢核酸内切酶的识别序列是已知的。也参见美国专利号5,420,032;美国专利号6,833,252;Belfort等人(1997)Nucleic Acids Res.25:3379-3388;Dujon等人(1989)Gene 82:115-118;Perler等人(1994)Nucleic Acids Res.22,1125-1127;Jasin(1996)TrendsGenet.12:224-228;Gimble等人(1996)J.Mol.Biol.263:163-180;Argast等人(1998)J.Mol.Biol.280:345-353和New England Biolabs目录。
来自天然存在的大范围核酸酶(主要来自LAGLIDADG家族(“LAGLIDADG”,公开为SEQ ID NO:6))的DNA结合结构域已用于促进植物、酵母、果蝇、哺乳动物细胞和小鼠中的位点特异性基因组修饰,但这种方法仅限于修饰保留大范围核酸酶识别序列的任何同源基因(Monet等人(1999),Biochem.Biophysics.Res.Common.255:88-93)或已引入识别序列的预工程化基因组(Route等人(1994),Mol.Cell.Biol.14:8096-106;Chilton等人(2003),Plant Physiology.133:956-65;Puchta等人(1996),Proc.Natl.Acad.Sci.USA 93:5055-60;Rong等人(2002),Genes Dev.16:1568-81;Gouble等人(2006),J.Gene Med.8(5):616-622)。因此,已尝试将大范围核酸酶工程化以表现出在医学或生物技术相关位点处的新型结合特异性(Porteus等人(2005),Nat.Biotechnol.23:967-73;Sussman等人(2004),J.Mol.Biol.342:31-41;Epinat等人(2003),Nucleic Acids Res.31:2952-62;Chevalier等人(2002)Molec.Cell 10:895-905;Epinat等人(2003)Nucleic Acids Res.31:2952-2962;Ashworth等人(2006)Nature 441:656-659;Paques等人(2007)Current GeneTherapy7:49-66;美国专利公开号20070117128;20060206949;20060153826;20060078552;和20040002092)。另外,来自大范围核酸酶的天然存在的或工程化DNA结合结构域可与来自异源核酸酶(例如,FokI)的切割结构域可操作地连接,和/或来自大范围核酸酶的切割结构域可与异源DNA结合结构域(例如,ZFP或TALE)可操作地连接。
在其他实施方案中,核酸酶是锌指核酸酶(ZFN)或TALE DNA结合结构域-核酸酶融合蛋白(TALEN)。ZFN和TALEN包含DNA结合结构域(锌指蛋白或TALE DNA结合结构域),其已被工程化为可结合至选择基因中的靶位点;以及切割结构域或切割半结构域(例如,来自本文描述的限制性核酸酶和/或大范围核酸酶)。
如上所详述,锌指结合结构域和TALE DNA结合结构域可以被工程化以结合选择的序列。参见例如,Beerli等人(2002)Nature Biotechnol.20:135-141;Pabo等人(2001)Ann.Rev.Biochem.70:313-340;Isalan等人(2001)Nature Biotechnol.19:656-660;Segal等人(2001)Curr.Opin.Biotechnol.12:632-637;Choo等人(2000)Curr.Opin.Struct.Biol.10:411-416。与天然存在的蛋白质相比,工程化的锌指结合结构域或TALE蛋白可以具有新型结合特异性。工程化方法包括但不限于合理设计和各种类型的选择。合理设计包括例如使用数据库,所述数据库包含三联体(或四联体)核苷酸序列和单独锌指或TALE氨基酸序列,其中每个三联体或四联体核苷酸序列与结合特定三联体或四联体序列的锌指或TALE重复单元的一个或多个氨基酸序列缔合。参见例如,美国专利6,453,242和6,534,261,将其通过引用以其整体并入本文。
靶位点的选择;以及设计和构建融合蛋白(和编码其的多核苷酸)的方法是本领域技术人员已知的,并且在美国专利号7,888,121和8,409,861中进行了详细描述,通过引用以其整体并入本文。
另外,如这些和其他参考文献中所披露,锌指结构域、TALE和/或多指锌指蛋白可使用任何适宜的接头序列(包括例如,长度为5个或更多个氨基酸的接头)连接在一起。关于示例性6个或更多个氨基酸长度的接头序列,参见例如,美国专利号6,479,626;6,903,185;和7,153,949。本文描述的蛋白质可包括蛋白质的单个锌指之间的适宜接头的任何组合。还参见美国专利号8,772,453。
因此,核酸酶例如ZFN、TALEN和/或大范围核酸酶可包含任何DNA结合结构域和任何核酸酶(切割)结构域(切割结构域,切割半结构域)。如上所述,切割结构域可以与DNA结合结构域异源,例如锌指或TAL效应子DNA结合结构域和来自核酸酶的切割结构域、或大范围核酸酶DNA结合结构域和来自不同核酸酶的切割结构域。异源切割结构域可以从任何核酸内切酶或核酸外切酶获得。可以衍生出切割结构域的示例性核酸内切酶包括但不限于限制性核酸内切酶和归巢核酸内切酶。参见例如,2002-2003目录,New England Biolabs,马萨诸塞州贝弗利;和Belfort等人(1997)Nucleic Acids Res.25:3379-3388。切割DNA的另外的酶是已知的(例如,S1核酸酶;绿豆核酸酶;胰腺DNA酶I;微球菌核酸酶;酵母HO核酸内切酶;还参见Linn等人(编辑)Nucleases,Cold Spring Harbor Laboratory Press,1993)。这些酶(或其功能片段)中的一种或多种可以用作切割结构域和切割半结构域的来源。
类似地,切割半结构域可以源自如上所述的需要二聚化以实现切割活性的任何核酸酶或其部分。通常,如果融合蛋白包含切割半结构域,则切割需要两个融合蛋白。可替代地,可以使用包含两个切割半结构域的单一蛋白质。两个切割半结构域可以源自相同核酸内切酶(或其功能性片段),或者每个切割半结构域可以源自不同核酸内切酶(或其功能性片段)。另外,两个融合蛋白的靶位点优选地关于彼此来布置,使得两个融合蛋白与其各自的靶位点的结合将切割半结构域置于针对彼此的空间定向中,此允许切割半结构域例如通过二聚化形成功能性切割结构域。因此,在某些实施方案中,靶位点的近边缘被5-10个核苷酸或15-18个核苷酸隔开。然而,任何整数个核苷酸或核苷酸对(例如2至50个核苷酸对或更多)都可以插入两个靶位点之间。通常,切割位点位于靶位点之间。
限制性核酸内切酶(限制酶)存在于许多物种中,并且能够与DNA(在识别位点)进行序列特异性结合,并能够在结合位点处或结合位点附近切割DNA。某些限制酶(例如,IIS型)在远离识别位点的位点处切割DNA,并且具有可分离的结合结构域和切割结构域。例如,IIS型FokI酶催化DNA的双链切割,在一条链上距其识别位点9个核苷酸处,并且在另一条链上距其识别位点13个核苷酸处进行切割。参见例如,美国专利5,356,802;5,436,150;和5,487,994;以及Li等人(1992)Proc.Natl.Acad.Sci.USA89:4275-4279;Li等人(1993)Proc.Natl.Acad.Sci.USA 90:2764-2768;Kim等人(1994a)Proc.Natl.Acad.Sci.USA 91:883-887;Kim等人(1994b)J.Biol.Chem.269:31,978-31,982。因此,在一个实施方案中,融合蛋白包含来自至少一种IIS型限制酶的切割结构域(或切割半结构域)和一个或多个锌指结合结构域(其可以工程化或可以不经工程化)。
其切割结构域可与结合结构域分离的示例性IIS型限制酶是FokI。此特定酶作为二聚体具有活性。Bitinaite等人(1998)Proc.Natl.Acad.Sci.USA 95:10,570-10,575。因此,出于本公开文本的目的,在公开的融合蛋白中使用的FokI酶的部分被认为是切割半结构域。因此,对于使用锌指-FokI融合物对细胞序列进行的靶向双链切割和/或靶向替代,可以使用各自包含FokI切割半结构域的两种融合蛋白来重构催化活性切割结构域。可替代地,也可以使用包含锌指结合结构域和两个Fok I切割半结构域的单一多肽分子。在本公开文本的其他地方提供了使用锌指-FokI融合物进行靶向切割和靶向序列改变的参数。
切割结构域或切割半结构域可以是蛋白质的任一部分,其保留切割活性,或者保留多聚化(例如,二聚化)以形成功能性切割结构域的能力。
示例性的IIS型限制酶描述于国际公开WO 07/014275中,以其整体并入本文。另外的限制酶也包含可分离的结合结构域和切割结构域,并且本公开文本考虑了这些。参见例如,Roberts等人(2003)Nucleic AcidsRes.31:418-420。
在某些实施方案中,所述切割结构域包含来自FokI核酸内切酶的切割结构域。全长FokI如下所示。本文描述的核酸酶中使用的切割结构域以斜体和下划线(全长蛋白的位置384至579)显示,其中全蛋白序列描述如下(SEQ ID NO:1):
源自FokI的切割半结构域可能在一个或多个氨基酸残基中包含突变。突变包括取代(将野生型氨基酸残基取代为不同残基)、插入(一个或多个氨基酸残基)和/或缺失(一个或多个氨基酸残基)。在某些实施方案中,残基414-426、443-450、467-488、501-502和/或521-531(相对于上述全长序列编号)中的一个或多个发生突变,因为这些残基在Miller等人((2007)Nat Biotechnol 25:778-784)描述的与其靶位点结合的ZFN分子模型中位于DNA骨架附近。在某些实施方案中,位置416、422、447、448和/或525处的一个或多个残基发生突变。在某些实施方案中,所述突变包括用任何不同的残基(例如丙氨酸(A)残基、半胱氨酸(C)残基、天冬氨酸(D)残基、谷氨酸(E)残基、组氨酸(H)残基,苯丙氨酸(F)残基,甘氨酸(G)残基,天冬酰胺(N)残基,丝氨酸(S)残基或苏氨酸(T)残基)取代野生型残基。在其他实施方案中,位置416、418、422、446、448、476、479、480、481、525、527和/或531中的一处或多处的野生型残基被任何其他残基替代,包括但不限于R416E、R416F、R416N、S418D、S418E、R422H、N476D、N476E、N476G、N476T、I479T、I479Q、Q481A、Q481D、Q481E、Q481H、K525A、K525S、K525T、K525V、N527D和/或Q531R。
在某些实施方案中,所述切割结构域包含最小化或防止同二聚化的一个或多个工程化切割半结构域(也称为二聚化结构域突变体),例如如在美国专利号7,914,796;8,034,598;和8,623,618;以及美国专利公开号20110201055所述,将所有上述专利的公开内容通过引用以其整体并入本文。FokI的位置446、447、479、483、484、486、487、490、491、496、498、499、500、531、534、537和538(相对全长FokI序列编号)处的氨基酸残基都是影响FokI切割半结构域的二聚化的靶标。突变可能包括在与FokI同源的天然限制酶中发现的残基的突变。在优选的实施方案中,位置416、422、447、448和/或525处的突变包括用不带电荷或带负电荷的氨基酸替代带正电荷的氨基酸。在另一个实施方案中,除了一个或多个氨基酸残基416、422、447、448或525中的突变之外,工程化切割半结构域还包含氨基酸残基499、496和486中的突变。
在某些实施方案中,本文描述的组合物包括FokI的工程化切割半结构域,其形成专性异二聚体,如例如美国专利号7,914,796;8,034,598;8,961,281和8,623,618;美国专利公开号20080131962和20120040398中所述。因此,在一个优选的实施方案中,本发明提供了融合蛋白,其中工程化切割半结构域包含这样的多肽:其中除了位置416、422、447、448或525(相对于本文所示的野生型FokI编号)处的一个或多个突变之外,位置486处的野生型Gln(Q)残基被Glu(E)残基替代、位置499处的野生型Ile(I)残基被Leu(L)残基替代,并且位置496处的野生型Asn(N)残基被Asp(D)或Glu(E)残基(“ELD”或“ELE”)替代。在另一个实施方案中,工程化切割半结构域源自野生型FokI切割半结构域,并且除了氨基酸残基416、422、447、448或525处的一个或多个突变之外,还包含相对于野生型FokI编号的氨基酸残基490、538和537中的突变。在优选的实施方案中,本发明提供了一种融合蛋白,其中工程化切割半结构域包含这样的多肽:其中除了位置416、422、447、448或525处的一个或多个突变之外,位置490处的野生型Glu(E)残基被Lys(K)残基替代、位置538处的野生型Ile(I)残基被Lys(K)残基替代,并且位置537处的野生型His(H)残基被Lys(K)残基或Arg(R)残基(“KKK”或“KKR”)替代(参见U.S.8,962,281,通过引用并入本文)。参见例如,美国专利号7,914,796;8,034,598和8,623,618;出于所有目的,将其公开内容通过引用以其整体并入。在其他实施方案中,工程化切割半结构域包含“Sharkey”和/或“Sharkey”突变(参见Guo等人,(2010)J.Mol.Biol.400(1):96-107)。
在另一个实施方案中,工程化切割半结构域源自野生型FokI切割半结构域,并且除了氨基酸残基416、422、447、448或525处的一个或多个突变之外,还包含相对于野生型FokI或FokI同源物编号的氨基酸残基490和538中的突变。在优选的实施方案中,本发明提供了一种融合蛋白,其中工程化切割半结构域包含这样的多肽:其中除了位置416、422、447、448或525处的一个或多个突变之外,位置490处的野生型Glu(E)残基被Lys(K)残基替代,并且位置538处的野生型Ile(I)残基被Lys(K)残基(“KK”)替代。在优选的实施方案中,本发明提供了一种融合蛋白,其中工程化切割半结构域包含这样的多肽:其中除了位置416、422、447、448或525处的一个或多个突变之外,位置486处的野生型Gln(Q)残基被Glu(E)残基替代,并且位置499处的野生型Ile(I)残基被Leu(L)残基(“EL”)替代(参见U.S.8,034,598,通过引用并入本文)。
在一个方面,本发明提供了一种融合蛋白,其中工程化切割半结构域包含这样的多肽:其中FokI催化结构域中位置387、393、394、398、400、402、416、422、427、434、439、441、447、448、469、487、495、497、506、516、525、529、534、559、569、570、571中的一处或多处的野生型氨基酸残基发生突变。提供了包含如任何所附表格和图中所示的一个或多个突变的核酸酶结构域。在一些实施方案中,所述一个或多个突变将野生型氨基酸从带正电荷的残基改变为中性残基或带负电荷的残基。在任何这些实施方案中,所述突变体还可以在包含一个或多个另外的突变的FokI结构域中制备。在优选的实施方案中,这些另外的突变在二聚化结构域中,例如在位置418、432、441、481、483、486、487、490、496、499、523、527、537、538和/或559处。突变的非限制性例子包括任何切割结构域(例如,FokI或FokI的同源物)在位置393、394、398、416、421、422、442、444、472、473、478、480、525或530处的野生型残基突变(例如,取代)为任何氨基酸残基(例如,K393X、K394X、R398X、R416S、D421X、R422X、K444X、S472X、G473X、S472、P478X、G480X、K525X和A530X,其中第一个残基描绘野生型,并且X是指取代野生型残基的任何氨基酸)。在一些实施方案中,X是E、D、H、A、K、S、T、D或N。其他示例性突变包括S418E、S418D、S446D、S446R、K448A、I479Q、I479T、Q481A、Q481N、Q481E、A530E和/或A530K,其中氨基酸残基相对于全长FokI野生型切割结构域及其同源物编号。在某些实施方案中,组合可以包括416和422、位置416处的突变和K448A、K448A和I479Q、K448A和Q481A和/或K448A和位置525处的突变。在一个实施方案中,位置416处的野生残基可以被Glu(E)残基替代(R416E),位置422处的野生型残基被His(H)残基替代(R422H),并且位置525处的野生型残基被Ala(A)残基替代。如本文所述的切割结构域可以进一步包括另外的突变,包括但不限于在位置432、441、483、486、487、490、496、499、527、537、538和/或559处,例如二聚化结构域突变体(例如,ELD、KKR)和或切口酶突变体(催化结构域的突变)。具有本文描述突变的切割半结构域形成本领域已知的异二聚体。
因此,本文提供了切割PD1基因的核酸酶。在某些实施方案中,所述核酸酶是包含左ZFN和右ZFN的ZFN核酸酶对的ZFN,所述左ZFN和右ZFN各自包含切割结构域(例如,FokI切割结构域)和PD1结合ZFP,其中所述左ZFN和右ZFN的切割结构域二聚化并且PD1基因被切割。在某些方面,PD1结合ZFP包含指定为12942和25029的ZFP,其完整氨基酸序列如下所示(SEQ ID NO:3和SEQ ID NO:5)。在其他实施方案中,所述核酸酶是包含左TALEN和右TALEN的TALEN核酸酶对的TALEN,所述左TALEN和右TALEN各自包含切割结构域(例如,FokI切割结构域)和PD1结合TAL效应子结构域,其中所述左TALEN和右TALEN的切割结构域二聚化并且PD1基因被切割。
所述对的PD1基因结合ZFN中的一个或两个进一步包括在FokI切割结构域中相对于野生型FokI(SEQ ID NO:1)编号的416、418、422、476、479、481、525和/或531,优选416、422、476、481和/或525并且甚至更优选416、481和/或525中的至少一个或多个的一个或多个突变。核酸酶对的一个或两个组分的核酸酶(切割)结构域也可以包含位置418、432、441、448、476、481、483、486、487、490、496、499、523、527、537、538和/或559处的一个或多个突变,包括但不限于ELD、KKR、ELE、KKS。参见例如,美国专利号8,623,618。在某些实施方案中,PD1核酸酶对中的一个或两个包括单个突变,例如在位置416处(例如,其中野生型R残基被E、F或N残基取代)、在位置418处(例如,其中野生型S残基被D或E残基取代)、在位置422处(例如,其中野生型R残基被H残基取代)、在位置476处(例如,其中野生型N残基被D、E、G或T残基取代)、在位置481处(例如,其中野生型Q残基被D、E或H残基取代)、在位置525处(例如,其中野生型K残基被A、S、T或V残基取代)、在位置527处(例如,其中野生型N残基被D残基取代)或在位置531处(例如,其中野生型Q残基被R残基取代)。参见例如,图1-图3。在某些实施方案中,所述突变包含选自以下的单个突变:R416E、R416F、R416N、S418D、S418E、R422H、N476D、N476E、N476G、N476T、I479T、I479Q、Q481A、Q481D、Q481E、Q481H、K525A、K525S、K525T、K525V、N527D、Q531R突变。取代突变的非限制性例子在图1-图3中示出。在仍其他实施方案中,核酸酶对(ZFN或TALEN)中的一个或两个核酸酶包含单个突变,例如在位置416处(例如,其中野生型R残基被E、F或N残基取代)、在位置418处(例如,其中野生型S残基被D或E残基取代)、在位置422处(例如,其中野生型R残基被H残基取代)、在位置476处(例如,其中野生型N残基被D、E、G或T残基取代)、在位置481处(例如,其中野生型Q残基被D、E或H残基取代)、在位置525处(例如,其中野生型K残基被A、S、T或V残基取代)、在位置527处(例如,其中野生型N残基被D残基取代)或在位置531处(例如,其中野生型Q残基被R残基取代),以及所述单个突变之外的另外的突变,例如ELE、ELD、KKR等一个或多个突变。因此,核酸酶对的一个或两个成员包括如本文所述的一个或多个突变。
可替代地,核酸酶可使用所谓的“分裂酶(split-enzyme)”技术在体内装配于核酸靶位点处(参见例如美国专利公开号20090068164)。此类分裂酶的组分可在单独的表达构建体上表达,或者可以连接于一个开放式阅读框中,其中单独组分例如由自切割的2A肽或IRES序列隔开。组分可以是单独锌指结合结构域或大范围核酸酶核酸结合结构域的结构域。
可以在使用前例如在基于酵母的染色体系统中针对活性来筛选核酸酶(例如,ZFN和/或TALEN),如美国专利号9,506,120和8,563,314中所述。
在某些实施方案中,核酸酶包含CRISPR/Cas系统。编码系统的RNA组分的CRISPR(规律间隔成簇短回文重复序列)基因座和编码蛋白质的Cas(CRISPR相关)基因座(Jansen等人,2002.Mol.Microbiol.43:1565-1575;Makarova等人,2002.Nucleic Acids Res.30:482-496;Makarova等人,2006.Biol.Direct 1:7;Haft等人,2005.PLoS Comput.Biol.1:e60)组成CRISPR/Cas核酸酶系统的基因序列。微生物宿主中的CRISPR基因座含有CRISPR相关(Cas)基因以及能够对CRISPR介导的核酸切割的特异性进行编程的非编码RNA元件的组合。
II型CRISPR是最充分表征的系统之一,并在四个连续步骤中进行靶向DNA双链断裂。第一步,从CRISPR基因座转录两个非编码RNA:前crRNA阵列和tracrRNA。第二步,tracrRNA与前crRNA的重复区杂交,并且介导将前crRNA加工成包含单独的间隔子序列的成熟crRNA。第三步,成熟的crRNA:tracrRNA复合物通过crRNA上的间隔子与靶DNA上靠近原间隔子邻近基序(PAM)的原间隔子之间的Watson-Crick碱基配对将Cas9引导至靶DNA,这是靶标识别的附加要求。最后,Cas9介导靶DNA的切割,以在原间隔子内产生双链断裂。CRISPR/Cas系统的活动包括三个步骤:(i)在称为‘适应’的过程中将外来DNA序列插入CRISPR阵列中以防止后来的攻击,(ii)表达相关蛋白质,以及表达并加工所述阵列,之后(iii)RNA介导干扰外来核酸。因此,在细菌细胞中,若干种所谓的‘Cas’蛋白参与CRISPR/Cas系统的天然功能,并在如插入外来DNA等功能中发挥作用。
在一些实施方案中,使用CRISPR-Cpf1系统。在弗朗西斯氏菌属物种(Francisellaspp)中鉴定的CRISPR-Cpf1系统是一种2类CRISPR-Cas系统,可在人细胞中介导稳健的DNA干扰。尽管Cpf1和Cas9在功能上是保守的,但它们在许多方面(包括Cpf1和Cas9的指导RNA和底物特异性)都存在差异(参见Fagerlund等人,(2015)Genom Bio 16:251)。Cas9与Cpf1蛋白之间的主要区别是Cpf1不利用tracrRNA,并且因此仅需要crRNA。FnCpf1 crRNA的长度为42-44个核苷酸(19个核苷酸的重复序列和23-25个核苷酸的间隔子),并且包含单个茎-环,其可以耐受保留二级结构的序列变化。此外,Cpf1crRNA明显短于Cas9所需的约100个核苷酸的工程化sgRNA,并且FnCpfl的PAM要求是在置换链上的5'-TTN-3'和5'-CTA-3'。尽管Cas9和Cpf1都在靶DNA中产生双链断裂,但是Cas9使用其RuvC和HNH样结构域在指导RNA的种子序列内进行平头末端切割,而Cpf1使用RuvC样结构域在种子序列外产生交错切割。由于Cpf1远离关键种子区域进行交错切割,因此NHEJ不会破坏靶位点,因此确保Cpf1可以继续切割同一位点,直到发生所需的HDR重组事件为止。因此,在本文描述的方法和组合物中,应理解术语“Cas”包括Cas9和Cfp1蛋白二者。因此,如本文所用,“CRISPR/Cas系统”是指CRISPR/Cas和/或CRISPR/Cfp1系统,包括核酸酶和/或转录因子系统二者。
在某些实施方案中,Cas蛋白可以是天然存在的Cas蛋白的“功能衍生物”。天然序列多肽的“功能衍生物”是具有与天然序列多肽共同的定性生物特性的化合物。“功能衍生物”包括但不限于天然序列的片段和天然序列多肽及其片段的衍生物,条件是它们具有与相应的天然序列多肽共同的生物活性。本文所预期的生物活性是功能衍生物将DNA底物水解为片段的能力。术语“衍生物”包括多肽的氨基酸序列变体、其共价修饰和融合物,如衍生的Cas蛋白。Cas多肽或其片段的适宜的衍生物包括但不限于Cas蛋白或其片段的突变体、融合物、共价修饰。Cas蛋白,包括Cas蛋白或其片段,以及Cas蛋白或其片段的衍生物,可以从细胞中获得或化学合成或通过这两种程序的组合获得。细胞可以是天然产生Cas蛋白的细胞,或天然产生Cas蛋白并且经基因工程化以更高表达水平产生内源Cas蛋白或从外源引入的核酸(所述核酸编码与内源Cas相同或不同的Cas)产生Cas蛋白的细胞。在一些情况下,细胞不会天然产生Cas蛋白,而是经基因工程化以产生Cas蛋白。在一些实施方案中,Cas蛋白是一种小的Cas9直系同源物,用于通过AAV载体进行递送(Ran等人(2015)Nature 510,第186页)。
一种或多种核酸酶可以在靶位点进行一个或多个双链和/或单链切割。在某些实施方案中,核酸酶包含无催化活性的切割结构域(例如,FokI和/或Cas蛋白)。参见例如,美国专利号9,200,266;8,703,489以及Guilli nger等人(2014)Nature Biotech.32(6):577-582。无催化活性的切割结构域可以与催化活性结构域组合充当切口酶以进行单链切割。因此,可以组合使用两种切口酶以在特定区域中进行双链切割。在本领域还已知另外的切口酶,例如,McCaffery等人(2016)Nucleic Acids Res.44(2):e11.doi:10.1093/nar/gkv878.Epub 2015年10月19日。
本文还提供了包含如本文所述的一种或多种ZFN、TALEN和/或多核苷酸的细胞,以及由包含本发明核酸酶的细胞产生的经遗传修饰的细胞。因此,本发明提供了使用如本文所述的一种或多种核酸酶产生的经遗传修饰细胞(例如,T细胞)的分离群体,在所述细胞群体中PD1基因通过所述一种或多种核酸酶进行遗传修饰(例如,通过插入和/或缺失(插入缺失)而突变),且所述核酸酶的特异性与不包含本文描述的一个或多个FokI突变的PD1核酸酶相比有所增加。可以以多种方式确定特异性,包括但不限于:比较由核酸酶进行的靶上(PD1)遗传修饰和靶外(非PD1)遗传修饰;确定靶上与靶外遗传修饰之间的倍数(或百分比)差异和/或;确定靶外修饰的实际百分比(任选地与靶上修饰相比)。也参见附图。
在某些方面,使用核酸酶产生的经遗传修饰的细胞的分离群体的相对靶上/靶外(PD1/非PD1)比大于使用没有如本文所述FokI突变的PD1核酸酶的遗传修饰的靶上/靶外比。参见例如,图2A和图2B。在某些实施方案中,由核酸酶产生的细胞中的遗传修饰的靶上/靶外比大于100,优选大于150并且甚至更优选大于200。因此,与没有本文描述的一个或多个FokI突变的PD1核酸酶相比,这些细胞中由核酸酶产生的PD1基因外的遗传修饰(靶外突变)可以减少1-100倍或更多倍,包括但不限于1-50倍(或其间的任何值)。在某些实施方案中,在分离的细胞群体中,由一种或多种核酸酶进行的遗传修饰的少于1%(例如,少于0.5%)在PD1基因之外。在仍其他方面中,由如本文所述的一种或多种核酸酶产生的细胞群的至少40%(例如,至少40%、至少45%、至少50%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%或至少90%)的细胞包括对PD1的修饰(插入缺失),并且少于0.05%的细胞包括由所述核酸酶进行的靶外(非PD1)遗传修饰。在仍其他方面中,使用本文描述核酸酶产生的经遗传修饰的细胞的分离群体具有150或更大的相对靶上/靶外(PD1/非PD1)比。因此,本文提供了其中PD1被特异性改变的经遗传修饰的细胞。本文描述的经遗传修饰的细胞可以包括进一步的修饰,包括例如对一个或多个另外的基因(TCR、B2M、CTLA-4、安全港基因)进行的遗传修饰(插入和/或缺失)。还提供了从经PD1遗传修饰细胞的分离群体
递送
可以通过任何适宜的手段将本文描述的蛋白质(例如,核酸酶)、多核苷酸和/或包含所述蛋白质和/或多核苷酸的组合物递送至靶细胞,所述任何适宜的手段包括例如,通过注射蛋白质和/或mRNA组分。
适宜的细胞包括但不限于真核和原核细胞和/或细胞系。此类细胞或由此类细胞生成的细胞系的非限制性例子包括T细胞、COS、CHO(例如CHO-S、CHO-K1、CHO-DG44、CHO-DUXB11、CHO-DUKX、CHOK1SV)、VERO、MDCK、WI38、V79、B14AF28-G3、BHK、HaK、NS0、SP2/0-Ag14、HeLa、HEK293(例如HEK293-F、HEK293-H、HEK293-T)、和perC6细胞以及昆虫细胞(如草地贪夜蛾(Spodoptera fugiperda(Sf)))或真菌细胞(如酵母属(Saccharomyces)、毕赤酵母属(Pichia)和裂殖酵母属(Schizosaccharomyces))。在某些实施方案中,细胞系是CHO-K1细胞系、MDCK细胞系或HEK293细胞系。适宜的细胞还包括干细胞,举例来说,如为胚胎干细胞、诱导多能干细胞(iPS细胞)、造血干细胞、神经元干细胞和间充质干细胞。
递送如本文所述的包含DNA结合结构域的蛋白质的方法描述于例如以下文献中:美国专利号6,453,242;6,503,717;6,534,261;6,599,692;6,607,882;6,689,558;6,824,978;6,933,113;6,979,539;7,013,219;和7,163,824,上述所有文献的公开内容都是通过引用以其整体并入本文。
如本文所述的DNA结合结构域和包含这些DNA结合结构域的融合蛋白也可以使用包含编码一种或多种DNA结合蛋白的序列的载体来递送。另外,还可以通过这些载体递送另外的核酸(例如,供体)。可以使用任何载体系统,包括但不限于质粒载体、逆转录病毒载体、慢病毒载体、腺病毒载体、痘病毒载体、疱疹病毒载体和腺相关病毒载体等。还参见,美国专利号6,534,261;6,607,882;6,824,978;6,933,113;6,979,539;7,013,219和7,163,824,将它们通过引用以其全文并入本文。此外,将清楚的是,这些载体中的任何一种都可以包含一种或多种DNA结合蛋白编码序列和/或视需要另外的核酸。因此,当将如本文所述的一种或多种DNA结合蛋白和视需要另外的DNA引入细胞中时,它们可以被携带在同一载体上或不同载体上。当使用多个载体时,每个载体可以包含编码一种或多种DNA结合蛋白的序列和根据需要另外的核酸。
常规的基于病毒和非病毒的基因转移方法可以用于将编码工程化DNA结合蛋白的核酸引入细胞(例如,哺乳动物细胞)和靶组织中,并根据需要共引入另外的核苷酸序列。此类方法还可以用于在体外向细胞施用核酸(例如,其编码DNA结合蛋白和/或供体)。在某些实施方案中,施用核酸以用于体内或离体基因疗法用途。非病毒载体递送系统包括DNA质粒、裸核酸和与递送媒介物(如脂质体或泊洛沙姆)复合的核酸。病毒载体递送系统包括DNA和RNA病毒,其在递送至细胞后具有游离或整合的基因组。对于基因疗法程序的综述,参见Anderson,Science 256:808-813(1992);Nabel和Felgner,TIBTECH 11:211-217(1993);Mitani和Caskey,TIBTECH 11:162-166(1993);Dillon,TIBTECH 11:167-175(1993);Miller,Nature 357:455-460(1992);Van Brunt,Biotechnology 6(10):1149-1154(1988);Vigne,Restorative Neurology and Neuroscience 8:35-36(1995);Kremer和Perricaudet,British Medical Bulletin 51(1):31-44(1995);Haddada等人,CurrentTopics in Microbiology and Immunology Doerfler和
非病毒递送核酸的方法包括电穿孔、脂转染、显微注射、基因枪、病毒体、脂质体、免疫脂质体、聚阳离子或脂质:核酸缀合物、裸DNA、mRNA、人工病毒粒子、和药剂增强的DNA摄取。还可以使用采用例如Sonitron 2000系统(Rich-Mar)的声致穿孔(sonoporation)来递送核酸。在优选实施方案中,一种或多种核酸是作为mRNA来递送。同样优选的是使用加帽的mRNA来增加翻译效率和/或mRNA稳定性。尤其优选的是ARCA(抗反向帽类似物)帽或其变体。参见美国专利号7,074,596和8,153,773,其是通过引用并入本文。
另外的示例性核酸递送系统包括由以下各项提供的那些:Amaxa Biosystems(科隆,德国)、Maxcyte,Inc.(罗克维尔,马里兰)、BTX Molecular Delivery Systems(霍利斯顿,MA)和Copernicus Therapeutics Inc(参见例如US6008336)。脂转染描述于例如,US 5,049,386、US 4,946,787;和US 4,897,355中,并且脂转染试剂在商业上出售(例如,Transfectam
脂质:核酸复合物(包括靶向脂质体如免疫脂质复合物)的制备是本领域技术人员熟知的(参见例如,Crystal,Science 270:404-410(1995);Blaese等人,Cancer GeneTher.2:291-297(1995);Behr等人,Bioconjugate Chem.5:382-389(1994);Remy等人,Bioconjugate Chem.5:647-654(1994);Gao等人,Gene Therapy 2:710-722(1995);Ahmad等人,Cancer Res.52:4817-4820(1992);美国专利号4,186,183、4,217,344、4,235,871、4,261,975、4,485,054、4,501,728、4,774,085、4,837,028和4,946,787)。
另外的递送方法包括使用将要递送的核酸包装至EnGeneIC递送媒介物(EDV)中。这些EDV被使用双特异性抗体特异性地递送至靶组织中,其中所述抗体的一个臂对所述靶组织具有特异性,并且另一个臂对EDV具有特异性。抗体将EDV携带至靶细胞表面,之后EDV通过胞吞作用被引入细胞中。进入细胞后,内容物被释放(参见MacDiarmid等人(2009)Nature Biotechnology 27(7)第643页)。
根据需要使用基于RNA或DNA病毒的系统来递送编码工程化DNA结合蛋白和/或供体(例如CAR或ACTR)的核酸利用了高度进化的过程,所述过程将病毒靶向至体内的特定细胞并将病毒有效载荷运输至细胞核。可以将病毒载体直接施用至患者(体内),或其可以用于在体外处理细胞,以及将经修饰细胞施用至患者(离体)。常规的递送核酸的基于病毒的系统包括但不限于用于基因转移的逆转录病毒、慢病毒、腺病毒、腺相关病毒、痘苗病毒和单纯疱疹病毒载体。使用逆转录病毒、慢病毒和腺相关病毒基因转移方法时,在宿主基因组中整合是可能的,通常导致所插入转基因的长期表达。另外,已经在许多不同细胞类型和靶组织中观察到高转导效率。
逆转录病毒的向性可以通过并入外来包膜蛋白、扩大靶细胞的潜在靶群体来改变。慢病毒载体是能够转导或感染非分裂细胞并且通常产生高病毒滴度的逆转录病毒载体。逆转录病毒基因转移系统的选择取决于靶组织。逆转录病毒载体由顺式作用的长末端重复序列构成,其具有对多达6-10kb的外来序列的包装能力。最小顺式作用LTR对载体的复制和包装而言是足够的,所述载体随后用于将治疗性基因整合至靶细胞中以提供持久的转基因表达。广泛使用的逆转录病毒载体包括基于鼠类白血病病毒(MuLV)、长臂猿白血病病毒(GaLV)、猿猴免疫缺陷病毒(SIV)、人免疫缺陷病毒(HIV)及其组合的那些载体(参见例如,Buchscher等人,J.Virol.66:2731-2739(1992);Johann等人,J.Virol.66:1635-1640(1992);Sommerfelt等人,Virol.176:58-59(1990);Wilson等人,J.Virol.63:2374-2378(1989);Miller等人,J.Virol.65:2220-2224(1991);PCT/US94/05700)。
在其中瞬时表达是优选的应用中,可以使用基于腺病毒的系统。基于腺病毒的载体能够在许多细胞类型中具有极高的转导效率,并且不需要细胞分裂。使用此类载体,已经获得高滴度和高表达水平。这种载体可以在相对简单的系统中大量生产。腺相关病毒(“AAV”)载体还用于用靶核酸转导细胞,例如在核酸和肽的体外生产中,以及用于体内和离体基因疗法程序中(参见例如,West等人,Virology 160:38-47(1987);美国专利号4,797,368;WO 93/24641;Kotin,Human Gene Therapy 5:793-801(1994);Muzyczka,J.Clin.Invest.94:1351(1994))。重组AAV载体的构建描述于许多出版物中,包括美国专利号5,173,414;Tratschin等人,Mol.Cell.Biol.5:3251-3260(1985);Tratschin等人,Mol.Cell.Biol.4:2072-2081(1984);Hermonat和Muzyczka,PNAS USA 81:6466-6470(1984);以及Samulski等人,J.Virol.63:03822-3828(1989)。
至少六种病毒载体方法目前可用于临床试验中的基因转移,所述试验利用涉及通过插入辅助细胞系中的基因与缺陷性载体互补以产生转导剂的方法。
pLASN和MFG-S是已经用于临床试验中的逆转录病毒载体的例子(Dunbar等人,Blood 85:3048-305(1995);Kohn等人,Nat.Med.1:1017-102(1995);Malech等人,PNAS USA94:22 12133-12138(1997))。PA317/pLASN是第一个用于基因疗法试验中的治疗性载体。(Blaese等人,Science270:475-480(1995))。已经观察到MFG-S包装的载体的转导效率为50%或更高。(Ellem等人,Immunol Immunother.44(1):10-20(1997);Dranoff等人,Hum.Gene Ther.1:111-2(1997)。
重组腺相关病毒载体(rAAV)是基于缺陷性和非病原性细小病毒腺相关2型病毒的有前景的替代性基因递送系统。所有载体都源自仅保留侧接转基因表达盒的AAV 145bp反向末端重复序列的质粒。由于整合至经转导细胞基因组中所致的有效基因转移和稳定转基因递送是这种载体系统的关键特征。(Wagner等人,Lancet 351:9117 1702-3(1998),Kearns等人,Gene Ther.9:748-55(1996))。根据本发明,也可以使用其他AAV血清型,包括AAV1、AAV3、AAV4、AAV5、AAV6、AAV8、AAV8.2、AAV9和AAVrh10以及假型化AAV,例如AAV2/8、AAV2/5和AAV2/6。
复制缺陷型重组腺病毒载体(Ad)可以以高滴度产生,并且易于感染多种不同的细胞类型。大多数腺病毒载体被工程化使得转基因替代Ad E1a、E1b和/或E3基因;随后使复制缺陷性载体在反式提供缺失的基因功能的人293细胞中增殖。Ad载体可以在体内转导多种类型的组织,包括非分裂的已分化细胞,如在肝、肾和肌肉中发现的那些。常规Ad载体具有较大运载能力。在临床试验中使用Ad载体的例子涉及使用肌内注射进行抗肿瘤免疫的多核苷酸疗法(Sterman等人,Hum.Gene Ther.7:1083-9(1998))。在临床试验中使用腺病毒载体进行基因转移的另外的例子包括Rosenecker等人,Infection24:1 5-10(1996);Sterman等人,Hum.Gene Ther.9:7 1083-1089(1998);Welsh等人,Hum.Gene Ther.2:205-18(1995);Alvarez等人,Hum.Gene Ther.5:597-613(1997);Topf等人,Gene Ther.5:507-513(1998);Sterman等人,Hum.Gene Ther.7:1083-1089(1998)。
包装细胞用于形成能感染宿主细胞的病毒颗粒。此类细胞包括包装腺病毒的293细胞以及包装逆转录病毒的ψ2细胞或PA317细胞。用于基因疗法中的病毒载体通常是由将核酸载体包装至病毒颗粒中的生产细胞系产生。载体通常含有包装和随后整合至宿主中(如果适用)所需的最小病毒序列,其他病毒序列由编码要表达的蛋白质的表达盒替代。失去的病毒功能由包装细胞系反式供应。例如,用于基因疗法中的AAV载体通常仅具有来自AAV基因组的反向末端重复(ITR)序列,所述序列是包装和整合至宿主基因组中所需的。将病毒DNA包装于细胞系中,所述细胞系含有编码其他AAV基因(即,rep和cap)但缺少ITR序列的辅助质粒。所述细胞系还被作为辅助者的腺病毒感染。辅助病毒促进AAV载体的复制和从辅助质粒表达AAV基因。由于缺少ITR序列,辅助质粒并未被大量包装。腺病毒的污染可通过例如热处理来减少,腺病毒对所述热处理比AAV更敏感。
在许多基因疗法应用中,可期望以高度特异性将基因疗法载体递送至特定组织类型。因此,可以修饰病毒载体,以通过将配体表达为与病毒外表面上的病毒外壳蛋白的融合蛋白而具有对给定细胞类型的特异性。配体被选择以具有对已知存在于目标细胞类型上的受体的亲和力。例如,Han等人(Proc.Natl.Acad.Sci.USA 92:9747-9751(1995))报道,莫洛尼(Moloney)鼠白血病病毒可以被修饰以表达与gp70融合的人神经生长因子,并且重组病毒感染某些表达人表皮生长因子受体的人乳腺癌细胞。这个原则可以扩展至其他病毒-靶细胞对,其中靶细胞表达受体并且病毒表达包含针对细胞表面受体的配体的融合蛋白。例如,丝状噬菌体可以被工程化以展示对实际上任何所选细胞受体具有特异性结合亲和力的抗体片段(例如,FAB或Fv)。虽然上述说明主要适用于病毒载体,但是相同原则可以适用于非病毒载体。此类载体可以被工程化以含有特定摄取序列,所述摄取序列有利于被特定靶细胞摄取。
用于CRISPR/Cas系统的递送方法可以包括上述那些方法。例如,在动物模型中,可以使用玻璃针将体外转录的编码Cas的mRNA或重组Cas蛋白直接注射到单细胞阶段胚胎中,以得到经基因组编辑的动物。为了在体外细胞中表达Cas和指导RNA,通常通过脂质转染或电穿孔将编码它们的质粒转染到细胞中。此外,重组Cas蛋白可以与体外转录的指导RNA复合,其中Cas-指导RNA核糖核蛋白被目标细胞摄取(Kim等人(2014)Genome Res24(6):1012)。出于治疗目的,可以通过病毒和非病毒技术的组合来递送Cas和指导RNA。例如,编码Cas的mRNA可以通过纳米颗粒递送来递送,而指导RNA和任何所需的转基因或修复模板通过AAV来递送(Yin等人(2016)Nat Biotechnol 34(3)第328页)。
如下所述,基因治疗载体通常可以通过全身施用(例如,静脉内、腹膜内、肌内、皮下或颅内输注)或局部施用,通过施用至单独患者来进行体内递送。可替代地,可以将载体离体递送至细胞中,如从单独患者外植的细胞(例如,淋巴细胞、骨髓抽取物、组织活检)或全适供血者造血干细胞,之后将所述细胞再植入患者体内,通常在选择已经并入所述载体的细胞后再植入。
用于诊断学、研究、移植或用于基因疗法的离体细胞转染(例如,通过将经转染细胞再输注至宿主生物体中)是本领域技术人员所熟知的。在优选的实施方案中,从受试者生物体中分离细胞,用DNA结合蛋白核酸(基因或cDNA)转染,然后再输注回受试者生物体(例如,患者)体内。适于离体转染的多种细胞类型是本领域技术人员所熟知的(参见例如,Freshney等人,
在一个实施方案中,在离体程序中使用干细胞进行细胞转染和基因疗法。使用干细胞的优点在于,其可以在体外分化为其他细胞类型,或者可以被引入哺乳动物(如细胞供体)中,其中其将移入骨髓中。在体外使用细胞因子(如GM-CSF、IFN-γ和TNF-α)将CD34+细胞分化为临床上重要的免疫细胞类型的方法是已知的(参见Inaba等人,J.Exp.Med.176:1693-1702(1992))。
使用已知方法分离干细胞以供转导和分化。例如,通过用结合不期望细胞(如CD4+和CD8+(T细胞)、CD45+(全B细胞)、GR-1(粒细胞)和Iad(已分化的抗原呈递细胞))的抗体淘选骨髓细胞来从骨髓细胞分离干细胞(参见Inaba等人,J.Exp.Med.176:1693-1702(1992))。
已经被修饰的干细胞也可以用于一些实施方案中。例如,已经变得可抵抗细胞凋亡的神经元干细胞可用作治疗性组合物,其中所述干细胞还含有本发明的ZFP TF。对细胞凋亡的抵抗可以例如通过在干细胞中使用BAX或BAK特异性ZFN敲除BAX和/或BAK来实现(参见,美国专利号8,597,912),或者例如在那些半胱天冬酶被破坏的干细胞中,也使用半胱天冬酶-6特异性ZFN来进行。
也可以将含有治疗性DNA结合蛋白(或编码这些蛋白的核酸)的载体(例如,逆转录病毒、腺病毒、脂质体等)直接施用至生物体以用于在体内转导细胞。可替代地,可以施用裸DNA。通过通常用于引入分子以与血液或组织细胞最终接触的任何途径来施用,所述途径包括但不限于注射、输注、局部施加和电穿孔。施用此类核酸的合适方法是本领域技术人员可获得的并且熟知的,并且尽管可以使用多于一种途径来施用特定组合物,但是特定途径通常可以提供比另一条途径更直接且更有效的反应。
将DNA引入造血干细胞中的方法披露于例如美国专利号5,928,638中。可用于将转基因引入造血干细胞(例如,CD34+细胞)中的载体包括腺病毒35型。
适于将转基因引入免疫细胞(例如,T细胞)中的载体包括非整合性慢病毒载体。参见例如,Ory等人(1996)Proc.Natl.Acad.Sci.USA93:11382-11388;Dull等人(1998)J.Virol.72:8463-8471;Zuffery等人(1998)J.Virol.72:9873-9880;Follenzi等人(2000)Nature Genetics 25:217-222。
药学上可接受的载体部分取决于所施用的特定组合物,以及取决于用于施用所述组合物的特定方法。因此,存在众多种可使用的药物组合物的适宜配制品,如下文所述(参见例如,Remington's Pharmaceutical Sciences,第17版,1989)。
如上所述,所公开的方法和组合物可以用于任何类型的细胞中,包括但不限于原核细胞、真菌细胞、古细菌细胞、植物细胞、昆虫细胞、动物细胞、脊椎动物细胞、哺乳动物细胞和人细胞,包括任何类型的T细胞和干细胞。用于蛋白质表达的适宜细胞系是本领域技术人员已知的,并且包括但不限于COS、CHO(例如,CHO-S、CHO-K1、CHO-DG44、CHO-DUXB11)、VERO、MDCK、WI38、V79、B14AF28-G3、BHK、HaK、NS0、SP2/0-Ag14、HeLa、HEK293(例如,HEK293-F、HEK293-H、HEK293-T)、perC6、昆虫细胞(如草地贪夜蛾)和真菌细胞(如酵母菌属、毕赤酵母属和裂殖酵母属)。还可以使用这些细胞系的后代、变体和衍生物。
应用
工程化PD1核酸酶在疾病的治疗和预防中的应用,有望在多个领域中有用。PD1参与多种疾病和障碍,包括但不限于癌症和自身免疫性疾病。参见例如,Chamoto等人(2017)Curr Top Microbiol Immunol.410:75-97。例如,已显示PD1参与调节响应于慢性抗原的T细胞激活与T细胞耐受之间的平衡。在HIV1感染期间,已发现CD4+T细胞中的PD1表达增加。人们认为,当在存在慢性抗原暴露的情况下(如在HIV感染的病例中)观察到T细胞功能障碍时,PD1上调在某种程度上与T细胞消耗(定义为关键效应子功能的进行性丧失)有关。PD1上调也可能与慢性病毒感染期间这些相同组细胞的凋亡增加相关(参见Petrovas等人,(2009)J Immunol.183(2):1120-32)。PD1也可能在肿瘤特异性逃避免疫监视方面发挥作用。已证明在慢性髓性白血病(CML)和急性髓性白血病(AML)两者中,PD1在肿瘤特异性细胞毒性T淋巴细胞(CTL)中高表达。PD1在黑色素瘤浸润性T淋巴细胞(TIL)中也上调(参见Dotti(2009)Blood 114(8):1457-58)。已发现肿瘤表达PD1配体(PDL),所述PD1配体在与CTL中PD1的上调组合时可能是T细胞功能丧失和CTL无法介导有效抗肿瘤反应的促成因素。因此,本文描述的方法和组合物用于增加这些新型工具的特异性,以确保所需的PD1靶位点(例如,用于治疗障碍,如癌症和自身免疫性障碍)将是主要(或唯一)的切割位置。对于所有体外、体内和离体应用,最小化或消除如本文所述的靶外切割增加这种技术的潜力。
核酸酶介导的PD1的遗传修饰可用于治疗多种遗传疾病和其他疾病,包括但不限于癌症。例如,经遗传修饰的T细胞(例如,CAR+细胞)可以通过如本文所述的PD1的遗传修饰进一步修饰,以提供用于癌症的治疗组合物。
如上所述,本文描述的组合物和方法可以用于基因修饰、基因校正和基因破坏。基因修饰的非限制性例子包括基于同源定向修复(HDR)的靶向整合;基于HDR的基因校正;基于HDR的基因修饰;基于HDR的基因破坏;基于NHEJ的基因破坏和/或HDR、NHEJ和/或单链退火(SSA)的组合。单链退火(SSA)是指通过用5'-3'核酸外切酶切除DSB以暴露2个互补区域来修复两个重复序列之间以相同取向发生的双链断裂。然后,编码2个同向重复序列的单链彼此退火,并且经退火的中间体可以被加工,使得单链尾部(单链DNA中未与任何序列退火的部分)被消化掉、间隙通过DNA聚合酶被填充,并且DNA末端被重新连接。这导致位于同向重复序列之间的序列缺失。
所述组合物和方法还可以用于体细胞疗法,从而允许产生已被修饰以增强其生物学特性的细胞储备。此类细胞可以被输注给各种患者,而与细胞的供体来源及其与受体的组织相容性无关。
所描述的工程化切割半结构域也可以用于如下基因修饰方案中:所述方案需要在多个靶标处同时切割,以使中间区域缺失或一次改变两个特定基因座。在两个靶标处切割将需要四个ZFN或TALEN的细胞表达,这可能潜在地产生十种不同的活性ZFN或TALEN组合。对于此类应用,用这些新型变体取代野生型核酸酶结构域将消除不需要的组合的活性,并减少靶外切割的机会。如果在某个所需DNA靶标处的切割需要核酸酶对A+B的活性,并且在第二个所需DNA靶标处的同时切割需要核酸酶对X+Y的活性,则使用本文描述的突变可以防止A与A配对、A与X配对、A与Y配对等。
引用的示例性参考文献
Beane JD,Lee G,Zheng Z,Mendel M,Abate-Daga D,Bharathan M,Black M,Gandhi N,Yu Z,Chandran S,Giedlin M,Ando D,Miller J,Paschon D,Guschin D,RebarEJ,Reik A,Holmes MC,Gregory PD,Restifo NP,Rosenberg SA,Morgan RA,Feldman SA(2015).Clinical Scale Zinc Finger Nuclease-mediated Gene Editing of PD-1inTumor Infiltrating Lymphocytes for the Treatment of Metastatic Melanoma.MolTher.23:1380-1390.doi:10.1038/mt.2015.71.
Bibikova M,Beumer K,Trautman JK,and Carroll,D(2003).Enhancing genetargeting with designed zinc finger nucleases.Science,300:764.
Bibikova M,Golic M,Golic KG and Carroll D(2002).Targeted chromosomalcleavage and mutagenesis in Drosophila using zinc-finger nucleases.Genetics,161:1169-1175.
Briggs GE,Haldane JBS(1925).A Note on the Kinetics of EnzymeAction.Biochem J.19:338–339.
Bolukbasi MF,Gupta A,Oikemus S,Derr AG,Garber M,Brodsky MH,Zhu LJ,Wolfe SA(2015).DNA-binding-domain fusions enhance the targeting range andprecision of Cas9.Nat Methods.12:1150-6.doi:10.1038/nmeth.3624.
Casini A,Olivieri M,Petris G,Montagna C,Reginato G,Maule G,LorenzinF,Prandi D,Romanel A,Demichelis F,Inga A,Cereseto A(2018).A highly specificSpCas9 variant is identified by in vivo screening in yeast.Nat Biotechnol.36:265-271.doi:10.1038/nbt.4066.
Chen JS,Dagdas YS,Kleinstiver BP,Welch MM,Sousa AA,Harrington LB,Sternberg SH,Joung JK,Yildiz A,Doudna JA(2017).Enhanced proofreading governsCRISPR-Cas9 targeting accuracy.Nature.550:407-410.doi:10.1038/nature24268.
Dagdas YS,Chen JS,Sternberg SH,Doudna JA,Yildiz A(2017).Aconformational checkpoint between DNA binding and cleavage by CRISPR-Cas9.SciAdv.3:eaao0027.doi:10.1126/sciadv.aao0027.
De Ravin SS,Reik A,Liu PQ,Li L,Wu X,Su L,Raley C,Theobald N,Choi U,Song AH,Chan A,Pearl JR,Paschon DE,Lee J,Newcombe H,Koontz S,Sweeney C,ShivakDA,Zarember KA,Peshwa MV,Gregory PD,Urnov FD,Malech HL(2016).Targeted geneaddition in human CD34(+)hematopoietic cells for correction of X-linkedchronic granulomatous disease.Nat Biotechnol.34:424-9.doi:10.1038/nbt.3513.
DeWitt MA,Magis W,Bray NL,Wang T,Berman JR,Urbinati F,Heo SJ,MitrosT,
DiCarlo JE,Mahajan VB,Tsang SH(2018).Gene therapy and genome surgeryin the retina.J Clin Invest.128:2177-2188.doi:10.1172/JCI120429.
Donsante A,Miller DG,Li Y,Vogler C,Brunt EM,Russell DW,Sands MS(2007).AAV vector integration sites in mouse hepatocellularcarcinoma.Science.317:477.
Doyon Y,Vo TD,Mendel MC,Greenberg SG,Wang J,Xia DF,Miller JC,UrnovFD,Gregory PD,Holmes MC(2011).Enhancing zinc-finger-nuclease activity withimproved obligate heterodimeric architectures.Nat Methods.8:74-9.doi:10.1038/nmeth.1539.
Fersht AR,Dingwall C(1979).Evidence for the double-sieve editingmechanism in protein synthesis.Steric exclusion of isoleucine by valyl-tRNAsynthetases.Biochemistry.18(12):2627-31.
Gao X,Tao Y,Lamas V,Huang M,Yeh WH,Pan B,Hu YJ,Hu JH,Thompson DB,ShuY,Li Y,Wang H,Yang S,Xu Q,Polley DB,Liberman MC,Kong WJ,Holt JR,Chen ZY,LiuDR(2018).Treatment of autosomal dominanthearing loss by in vivo delivery ofgenome editing agents.Nature.553(7687):217-221.doi:10.1038/nature25164.
Grizot S,Smith J,Daboussi F,Prieto J,Redondo P,Merino N,Villate M,Thomas S,Lemaire L,Montoya G,Blanco FJ,
Guilinger JP,David B Thompson&David R Liu(2014a).Fusion ofcatalytically inactive Cas9 to FokI nuclease improves the specificity ofgenome modification.Nature Biotechnol.32:577–582.
Guilinger JP,Pattanayak V,Reyon D,Tsai SQ,Sander JD,Joung JK,Liu DR(2014b).Broad specificity profiling of TALENs results in engineered nucleaseswith improved DNA-cleavage specificity.Nat Methods.11:429-35.doi:10.1038/nmeth.2845.
Hopfield JJ(1974).Kinetic proofreading:a new mechanism for reducingerrors in biosynthetic processes requiring high specificity.Proc.Natl.Acad.Sci.U.S.A.71:4135–9.
Hu JH,Miller SM1,2,3,Geurts MH1,2,3,Tang W1,2,3,Chen L1,2,3,Sun N1,2,3,Zeina CM1,2,3,Gao X1,2,3,Rees HA1,2,3,Lin Z1,2,3,Liu DR(2018).Evolved Cas9variants with broad PAM compatibility and high DNA specificity.Nature.556:57-63.doi:10.1038/nature26155.
Huang CH,Lee KC,Doudna JA(2018).Applications of CRISPR-Cas Enzymes inCancer Therapeutics and Detection.Trends Cancer.20184:499-512.doi:10.1016/j.trecan.2018.05.006.
Kim JS,Pabo CO(1998).Getting a handhold on DNA:design of poly-zincfinger proteins with femtomolar dissociation constants.Proc Natl Acad Sci U SA.95:2812-7.
Kleinstiver BP,Pattanayak V,Prew MS,Tsai SQ,Nguyen NT,Zheng Z,JoungJK(2016).High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wideoff-target effects.Nature.529:490-5.doi:10.1038/nature16526..
Jarjour J,West-Foyle H,Certo MT,Hubert CG,Doyle L,Getz MM,StoddardBL,Scharenberg AM(2009).High-resolution profiling of homing endonucleasebinding and catalytic specificity using yeast surface display.Nucleic AcidsRes.37:6871-80.doi:10.1093/nar/gkp726.
Jiang F,Taylor DW,Chen JS,Kornfeld JE,Zhou K,Thompson AJ,Nogales E,Doudna JA (2016).Structures of a CRISPR-Cas9 R-loop complex primed for DNAcleavage.Science.351:867-71.doi:10.1126/science.aad8282.
Jiang F,Doudna JA(2017).CRISPR-Cas9 Structures and Mechanisms.AnnuRev Biophys.46:505-529.doi:10.1146/annurev-biophys-062215-010822.
Kim YG,Cha J,Chandrasegaran S(1996).Hybrid restriction enzymes:zincfinger fusions to FokI cleavage domain.Proc Natl Acad Sci U S A.93:1156-60
Kulcsár PI,Tálas A,Huszár K,Ligeti Z,Tóth E,Weinhardt N,Fodor E,Welker E(2017).Crossing enhanced and high fidelity SpCas9 nucleases tooptimize specificity and cleavage.Genome Biol.18:190.doi:10.1186/s13059-017-1318-8.
Michaelis L,Menten ML(1913).Die Kinetik der Invertinwirkung,Biochemische Zeitschrift 49:333–369
Miller JC,Holmes MC,Wang J,Guschin DY,Lee YL,Rupniewski I,BeausejourCM,Waite AJ,Wang NS,Kim KA,Gregory PD,Pabo CO,Rebar EJ(2007).An improvedzinc-finger nuclease architecture for highly specific genome editing.NatBiotechnol.25:778-85.DOI:10.1038/nbt1319
Miller JC,Zhang L,Xia DF,Campo JJ,Ankoudinova IV,Guschin DY,BabiarzJE,Meng X,Hinkley SJ,Lam SC,Paschon DE,Vincent AI,Dulay GP,Barlow KA,ShivakDA,Leung E,Kim JD,Amora R,Urnov FD,Gregory PD,Rebar EJ(2015).Improvedspecificity of TALE-based genome editing using an expanded RVD repertoire.NatMethods.12:465-71.doi:10.1038/nmeth.3330.
Miller JC,Tan S,Qiao G,Barlow KA,Wang J,Xia DF,Meng X,Paschon DE,Leung E,Hinkley SJ,Dulay GP,Hua KL,Ankoudinova I,Cost GJ,Urnov FD,Zhang HS,Holmes MC,Zhang L,Gregory PD,Rebar EJ(2011).A TALE nuclease architecture forefficient genome editing.Nat Biotechnol.29:143-8.doi:10.1038/nbt.1755.
Pattanayak V,Ramirez CL,Joung JK,Liu,DR(2011).Revealing off-targetcleavage specificities of zinc-finger nucleases by in vitro selection.Naturemethods,8:765-770.
Pavletich NP,Pabo CO(1991).Zinc finger-DNA recognition:crystalstructure of a Zif268-DNA complex at 2.1 A.Science.252:809-17.
Pernstich C,Halford SE(2012).Illuminating the reaction pathway of theFokI restriction endonuclease by fluorescence resonance energytransfer.Nucleic Acids Res.40:1203-13.doi:10.1093/nar/gkr809.
Porteus MH,Baltimore D(2003).Chimeric Nucleases Stimulate GeneTargeting in Human Cells.Science 300:763 DOI:10.1126/science.1078395
Ran FA,Cong L,Yan WX,Scott DA,Gootenberg JS,Kriz AJ,Zetsche B,ShalemO,Wu X,Makarova KS,Koonin EV,Sharp PA,Zhang F(2015).In vivo genome editingusing Staphylococcus aureus Cas9.Nature.520:186-91.doi:10.1038/nature14299.
Ran FA,Hsu PD,Lin CY,Gootenberg JS,Konermann S,Trevino AE,Scott DA,Inoue A,Matoba S,Zhang Y,Zhang F(2013).Double nicking by RNA-guided CRISPRCas9 for enhanced genome editing specificity.Cell.154:1380-9.doi:10.1016/j.cell.2013.08.021.
Raper AT,Anthony A.Stephenson,Suo Z(2018).Functional InsightsRevealed by the Kinetic Mechanism of CRISPR/Cas9 J.Am.Chem.Soc.140:2971–2984DOI:10.1021/jacs.7b13047
Ryan DE,Taussig D,Steinfeld I,Phadnis SM,Lunstad BD,Singh M,Vuong X,Okochi KD,McCaffrey R,Olesiak M,Roy S,Yung CW,Curry B,Sampson JR,Bruhn L,Dellinger DJ(2018).Improving CRISPR-Cas specificity with chemicalmodifications in single-guide RNAs.Nucleic Acids Res.46:792-803.doi:10.1093/nar/gkx1199.
Seki A and Rutz S(2018).Optimized RNP transfection for highlyefficient CRISPR/Cas9-mediated gene knockout in primary T cells.J ExpMed.215:985–997.doi:10.1084/jem.20171626
Singh D,Sternberg SH,Fei J,Doudna JA,Ha T(2016).Real-time observationof DNA recognition and rejection by the RNA-guided endonuclease Cas9.NatCommun.7:12778.doi:10.1038/ncomms12778.
Slaymaker IM,Gao L,Zetsche B,Scott DA,Yan WX,Zhang F(2016).Rationallyengineered Cas9 nucleases with improved specificity.Science.351:84-8.doi:10.1126/science.aad5227.
Sternberg SH,LaFrance B,Kaplan M,Doudna JA(2015).Conformationalcontrol of DNA target cleavage by CRISPR-Cas9.Nature.527:110-3.doi:10.1038/nature15544.
Tsai SQ,Nicolas Wyvekens,Cyd Khayter,Jennifer A Foden,Vishal Thapar,Deepak Reyon,Mathew J Goodwin,Martin J Aryee,Joung JK(2014).Dimeric CRISPRRNA-guided FokI nucleases for highly specific genome editing.NatureBiotechnol.32:569–576
Tsai SQ,Zheng Z,Nguyen NT,Liebers M,Topkar VV,Thapar V,Wyvekens N,Khayter C,Iafrate AJ,Le LP,Aryee MJ,Joung JK(2015).GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases.NatBiotechnol.33:187-197.doi:10.1038/nbt.3117.
Tsai SQ and Joung JK(2016).Defining and improving the genome-widespecificities of CRISPR–Cas9 nucleases.Nature Reviews Genetics.17,300–312.
Urnov FD,Miller,JC,Lee YL,Beausejour CM,Rock JM,Augustus S,JamiesonAC,Porteus MH,Gregory PD and Holmes MC(2005).Highly efficient endogenoushuman gene correction using designed zinc-finger nucleases.Nature,435,646-651.
Yin H,Song CQ,Suresh S,Kwan SY,Wu Q,Walsh S,Ding J,Bogorad RL,Zhu LJ,Wolfe SA,Koteliansky V,Xue W,Langer R,Anderson DG(2018).Partial DNA-guidedCas9 enables genome editing with reduced off-target activity.Nat ChemBiol.14:311-316.doi:10.1038/nchembio.2559.
Zhang D,Zhang H,Li T,Chen K,Qiu JL,Gao C(2017).Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases.Genome Biol.18:191.doi:10.1186/s13059-017-1325-9.
实施例1:特定PD1特异性核酸酶的产生
如美国专利公开号20180087072中所述,产生具有低水平靶外效应的PD1特异性核酸酶。原始(亲本)ZFN包含ZFP 12942和25029,如美国专利号8,563,314(例如,表2和表3)中所述。
亲本ZFN对的核苷酸和氨基酸序列如下所示(识别螺旋区域的氨基酸序列加下划线,靶向的FokI残基以小写显示,选择用于分析的FokI位置为粗体加下划线):
12942(左PD1 ZFN)
核酸序列:
ATGGACTACAAAGACCATGACGGTGATTATAAAGATCATGACATCGATTACAAGGATGACGATGACAAGATGGCCCCCAAGAAGAAGAGGAAGGTGGGCATTCACGGGGTACCCGCCGCTATGGCTGAGAGGCCCTTCCAGTGTCGAATCTGCATGCGTAAGTTTGCCCAGTCCGGCCACCTGTCCCGCCATACCAAGATACACACGGGCGAGAAGCCCTTCCAGTGTCGAATCTGCATGCGTAACTTCAGTCGTAGTGACAGCCTGAGCGTACACATCCGCACCCACACAGGCGAGAAGCCTTTTGCCTGTGACATTTGTGGGAGGAAATTTGCCCACAACGACAGCCGCAAAAACCATACCAAGATACACACGGGATCTCAGAAGCCCTTCCAGTGTCGAATCTGCATGCGTAACTTCAGTCGCTCCGACGACCTGACCCGCCACATCCGCACCCACACAGGCGAGAAGCCTTTTGCCTGTGACATTTGTGGGAGGAAGTTTGCCCGCTCCGACCACCTGACCCAGCATACCAAGATACACCTGCGGGGATCCCAGCTGGTGAAGAGCGAGCTGGAGGAGAAGAAGTCCGAGCTGCGGCACAAGCTGAAGTACGTGCCCCACGAGTACATCGAGCTGATCGAGATCGCCAGGAACAGCACCCAGGACCGCATCCTGGAGATGAAGGTGATGGAGTTCTTCATGAAGGTGTACGGCTACAGGGGAAAGCACCTGGGCGGAAGCAGAAAGCCTGACGGCGCCATCTATACAGTGGGCAGCCCCATCGATTACGGCGTGATCGTGGACACAAAGGCCTACAGCGGCGGCTACAATCTGCCTATCGGCCAGGCCGACGAGATGCAGAGATACGTGGAGGAGAACCAGACCCGGAATAAGCACATCAACCCCAACGAGTGGTGGAAGGTGTACCCTAGCAGCGTGACCGAGTTCAAGTTCCTGTTCGTGAGCGGCCACTTCAAGGGCAACTACAAGGCCCAGCTGACCAGGCTGAACCACATCACCAACTGCAATGGCGCCGTGCTGAGCGTGGAGGAGCTGCTGATCGGCGGCGAGATGATCAAAGCCGGCACCCTGACACTGGAGGAGGTGCGGCGCAAGTTCAACAACGGCGAGATCAACTTCAGATCTTGATAA(SEQ ID NO:2)。
氨基酸序列:
25029(右PD1 ZFN)
核酸序列:
ATGGACTACAAAGACCATGACGGTGATTATAAAGATCATGACATCGATTACAAGGATGACGATGACAAGATGGCCCCCAAGAAGAAGAGGAAGGTGGGCATTCACGGGGTACCCGCCGCTATGGCTGAGAGGCCCTTCCAGTGTCGAATCTGCATGTGTAAGTTTGCCCGCAACGCCGCCCTGACCCGCCATACCAAGATACACACGGGCGAGAAGCCGTTCCAGTGTCGCATCTGCATGCGTAACTTCAGTCGCTCCGACGAGCTGACCCGCCACATCCGCACCCACACAGGCGAGAAGCCTTTTGCTTGCGACATTTGTGGGAGGAAGTTTGCCCGGCACCACCACCTGGCCGCCCATACCAAGATACACACGGGATCTCAGAAGCCCTTCCAGTGTCGAATCTGCATGCGTAACTTCAGTACCCGCCCGGTGCTGAAGCGCCACATCCGCACCCACACAGGCGAGAAGCCTTTTGCTTGCGACATTTGTGGGAGGAAGTTTGCCGACCGCTCCGCCCTGGCCCGCCATACCAAGATACACCTGCGGGGATCCCAGCTGGTGAAGAGCGAGCTGGAGGAGAAGAAGTCCGAGCTGCGGCACAAGCTGAAGTACGTGCCCCACGAGTACATCGAGCTGATCGAGATCGCCAGGAACAGCACCCAGGACCGCATCCTGGAGATGAAGGTGATGGAGTTCTTCATGAAGGTGTACGGCTACAGGGGAAAGCACCTGGGCGGAAGCAGAAAGCCTGACGGCGCCATCTATACAGTGGGCAGCCCCATCGATTACGGCGTGATCGTGGACACAAAGGCCTACAGCGGCGGCTACAATCTGCCTATCGGCCAGGCCGACGAGATGCAGAGATACGTGGAGGAGAACCAGACCCGGAATAAGCACATCAACCCCAACGAGTGGTGGAAGGTGTACCCTAGCAGCGTGACCGAGTTCAAGTTCCTGTTCGTGAGCGGCCACTTCAAGGGCAACTACAAGGCCCAGCTGACCAGGCTGAACCACATCACCAACTGCAATGGCGCCGTGCTGAGCGTGGAGGAGCTGCTGATCGGCGGCGAGATGATCAAAGCCGGCACCCTGACACTGGAGGAGGTGCGGCGCAAGTTCAACAACGGCGAGATCAACTTCAGATCTTGATAA(SEQ ID NO:4)
氨基酸序列:
使用这些PD1 ZFN,筛选了一组22个FokI变体(在图1中列出,包括R416E、R416F、R416N、S418D、S418E、R422H、N476D、K448A、N476E、N476G、N476T、I479T、I479Q、Q481A、Q481D、Q481E、Q481H、K525A、K525S、K525T、K525V、N527D、Q531R)。分析了在其FokI结构域中带有单残基取代的PD1 ZFN二聚体的变体的靶上活性。将每个变体均作为二聚体进行测试,其中两个ZFN均带有指示的取代ZFN,并通过mRNA核转染(500ng的每种单体)递送至人K562细胞,随后在第3天进行基因组DNA分离,并对预期靶标处的插入缺失进行深度测序分析。“半剂量”亲本样品使用250ng RNA进行递送。为了突出显示相对信号强度,对表格值加阴影。箭头突出显示了表现为完全保留高水平靶上活性的示例性变体。
对从靶上筛选中鉴定出的FokI变体(包括R416E、R416F、R416N、R422H、N476G、N476T、Q481D、Q481H、K525A、K525S、K525T和K525V)进一步表征在靶上位点和靶外位点二者(包括3种先前描述的最高修饰的靶外位点)处的活性(Beane 2015)。简而言之,使用指示量的每种ZFN单体的mRNA,通过核转染将所述对的两个ZFN中具有相同FokI变体的ZFN递送至人K562细胞。在第3天分离基因组DNA,并在预期的靶标和靶外位点处确定针对插入缺失的深度测序分析。
如图2A至图3所示,靶外活性显著降低(10至100倍),其中最具选择性的取代(Q481D)表现为靶上切割偏好增加>200倍(参见图3,其中亲本二聚体为75%PD1插入缺失/6.4%OT插入缺失,相比之下变体为81%/0.03%)。
如本文所示,通过修饰FokI结构域产生特异性高度增强的PD1ZFN。
实施例2:TALEN FokI变体
如美国专利公开号20180087072中所述,设计和测试TALEN对。所使用的TALEN对(“样品”)都是如美国专利号8,586,526中所述包含101041和101047的DNA结合结构域的TAL效应子的FokI变体,其基于所包括的FokI变体如图4中所示被重命名。
如图4所示,几种FokI变体构建体更具活性,包括含有S446R突变、Q5531R或Q481H突变(在二聚体的一个或两个TALEN中)的TALEN。在30℃下,在右TALEN上使用FokI变体Q531R,并且在左TALEN和右TALEN两者上使用FokI变体Q481H,使靶外活性降低了超过4倍,同时保持完全的靶上活性。此外,大多数TALEN表现出特异性增加了至少2至8倍(如通过相对靶上/靶外水平测量)。
本文提及的所有专利、专利申请和出版物均通过引用以其整体特此并入。
虽然已经出于清晰理解的目的通过说明和实施例详细提供了公开文本,但是本领域技术人员将了解,可以在不背离本公开文本的精神或范围的情况下进行各种改变和修改。因此,前面的描述和实施例不应理解为具有限制性。
序列表
<110> 桑格摩生物治疗股份有限公司
<120> 程序性细胞死亡1(PD1)特异性核酸酶
<130> 8325-0181.40
<140>
<141>
<150> 62/732,674
<151> 2018-09-18
<160> 6
<170> PatentIn版本3.5
<210> 1
<211> 579
<212> PRT
<213> 未知
<220>
<223> 未知的描述:
FokI蛋白序列
<400> 1
Met Val Ser Lys Ile Arg Thr Phe Gly Trp Val Gln Asn Pro Gly Lys
1 5 10 15
Phe Glu Asn Leu Lys Arg Val Val Gln Val Phe Asp Arg Asn Ser Lys
20 25 30
Val His Asn Glu Val Lys Asn Ile Lys Ile Pro Thr Leu Val Lys Glu
35 40 45
Ser Lys Ile Gln Lys Glu Leu Val Ala Ile Met Asn Gln His Asp Leu
50 55 60
Ile Tyr Thr Tyr Lys Glu Leu Val Gly Thr Gly Thr Ser Ile Arg Ser
65 70 75 80
Glu Ala Pro Cys Asp Ala Ile Ile Gln Ala Thr Ile Ala Asp Gln Gly
85 90 95
Asn Lys Lys Gly Tyr Ile Asp Asn Trp Ser Ser Asp Gly Phe Leu Arg
100 105 110
Trp Ala His Ala Leu Gly Phe Ile Glu Tyr Ile Asn Lys Ser Asp Ser
115 120 125
Phe Val Ile Thr Asp Val Gly Leu Ala Tyr Ser Lys Ser Ala Asp Gly
130 135 140
Ser Ala Ile Glu Lys Glu Ile Leu Ile Glu Ala Ile Ser Ser Tyr Pro
145 150 155 160
Pro Ala Ile Arg Ile Leu Thr Leu Leu Glu Asp Gly Gln His Leu Thr
165 170 175
Lys Phe Asp Leu Gly Lys Asn Leu Gly Phe Ser Gly Glu Ser Gly Phe
180 185 190
Thr Ser Leu Pro Glu Gly Ile Leu Leu Asp Thr Leu Ala Asn Ala Met
195 200 205
Pro Lys Asp Lys Gly Glu Ile Arg Asn Asn Trp Glu Gly Ser Ser Asp
210 215 220
Lys Tyr Ala Arg Met Ile Gly Gly Trp Leu Asp Lys Leu Gly Leu Val
225 230 235 240
Lys Gln Gly Lys Lys Glu Phe Ile Ile Pro Thr Leu Gly Lys Pro Asp
245 250 255
Asn Lys Glu Phe Ile Ser His Ala Phe Lys Ile Thr Gly Glu Gly Leu
260 265 270
Lys Val Leu Arg Arg Ala Lys Gly Ser Thr Lys Phe Thr Arg Val Pro
275 280 285
Lys Arg Val Tyr Trp Glu Met Leu Ala Thr Asn Leu Thr Asp Lys Glu
290 295 300
Tyr Val Arg Thr Arg Arg Ala Leu Ile Leu Glu Ile Leu Ile Lys Ala
305 310 315 320
Gly Ser Leu Lys Ile Glu Gln Ile Gln Asp Asn Leu Lys Lys Leu Gly
325 330 335
Phe Asp Glu Val Ile Glu Thr Ile Glu Asn Asp Ile Lys Gly Leu Ile
340 345 350
Asn Thr Gly Ile Phe Ile Glu Ile Lys Gly Arg Phe Tyr Gln Leu Lys
355 360 365
Asp His Ile Leu Gln Phe Val Ile Pro Asn Arg Gly Val Thr Lys Gln
370 375 380
Leu Val Lys Ser Glu Leu Glu Glu Lys Lys Ser Glu Leu Arg His Lys
385 390 395 400
Leu Lys Tyr Val Pro His Glu Tyr Ile Glu Leu Ile Glu Ile Ala Arg
405 410 415
Asn Ser Thr Gln Asp Arg Ile Leu Glu Met Lys Val Met Glu Phe Phe
420 425 430
Met Lys Val Tyr Gly Tyr Arg Gly Lys His Leu Gly Gly Ser Arg Lys
435 440 445
Pro Asp Gly Ala Ile Tyr Thr Val Gly Ser Pro Ile Asp Tyr Gly Val
450 455 460
Ile Val Asp Thr Lys Ala Tyr Ser Gly Gly Tyr Asn Leu Pro Ile Gly
465 470 475 480
Gln Ala Asp Glu Met Gln Arg Tyr Val Glu Glu Asn Gln Thr Arg Asn
485 490 495
Lys His Ile Asn Pro Asn Glu Trp Trp Lys Val Tyr Pro Ser Ser Val
500 505 510
Thr Glu Phe Lys Phe Leu Phe Val Ser Gly His Phe Lys Gly Asn Tyr
515 520 525
Lys Ala Gln Leu Thr Arg Leu Asn His Ile Thr Asn Cys Asn Gly Ala
530 535 540
Val Leu Ser Val Glu Glu Leu Leu Ile Gly Gly Glu Met Ile Lys Ala
545 550 555 560
Gly Thr Leu Thr Leu Glu Glu Val Arg Arg Lys Phe Asn Asn Gly Glu
565 570 575
Ile Asn Phe
<210> 2
<211> 1155
<212> DNA
<213> 人工序列
<220>
<223> 人工序列的描述:合成
多核苷酸
<400> 2
atggactaca aagaccatga cggtgattat aaagatcatg acatcgatta caaggatgac 60
gatgacaaga tggcccccaa gaagaagagg aaggtgggca ttcacggggt acccgccgct 120
atggctgaga ggcccttcca gtgtcgaatc tgcatgcgta agtttgccca gtccggccac 180
ctgtcccgcc ataccaagat acacacgggc gagaagccct tccagtgtcg aatctgcatg 240
cgtaacttca gtcgtagtga cagcctgagc gtacacatcc gcacccacac aggcgagaag 300
ccttttgcct gtgacatttg tgggaggaaa tttgcccaca acgacagccg caaaaaccat 360
accaagatac acacgggatc tcagaagccc ttccagtgtc gaatctgcat gcgtaacttc 420
agtcgctccg acgacctgac ccgccacatc cgcacccaca caggcgagaa gccttttgcc 480
tgtgacattt gtgggaggaa gtttgcccgc tccgaccacc tgacccagca taccaagata 540
cacctgcggg gatcccagct ggtgaagagc gagctggagg agaagaagtc cgagctgcgg 600
cacaagctga agtacgtgcc ccacgagtac atcgagctga tcgagatcgc caggaacagc 660
acccaggacc gcatcctgga gatgaaggtg atggagttct tcatgaaggt gtacggctac 720
aggggaaagc acctgggcgg aagcagaaag cctgacggcg ccatctatac agtgggcagc 780
cccatcgatt acggcgtgat cgtggacaca aaggcctaca gcggcggcta caatctgcct 840
atcggccagg ccgacgagat gcagagatac gtggaggaga accagacccg gaataagcac 900
atcaacccca acgagtggtg gaaggtgtac cctagcagcg tgaccgagtt caagttcctg 960
ttcgtgagcg gccacttcaa gggcaactac aaggcccagc tgaccaggct gaaccacatc 1020
accaactgca atggcgccgt gctgagcgtg gaggagctgc tgatcggcgg cgagatgatc 1080
aaagccggca ccctgacact ggaggaggtg cggcgcaagt tcaacaacgg cgagatcaac 1140
ttcagatctt gataa 1155
<210> 3
<211> 383
<212> PRT
<213> 人工序列
<220>
<223> 人工序列的描述:合成
多肽
<400> 3
Met Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp
1 5 10 15
Tyr Lys Asp Asp Asp Asp Lys Met Ala Pro Lys Lys Lys Arg Lys Val
20 25 30
Gly Ile His Gly Val Pro Ala Ala Met Ala Glu Arg Pro Phe Gln Cys
35 40 45
Arg Ile Cys Met Arg Lys Phe Ala Gln Ser Gly His Leu Ser Arg His
50 55 60
Thr Lys Ile His Thr Gly Glu Lys Pro Phe Gln Cys Arg Ile Cys Met
65 70 75 80
Arg Asn Phe Ser Arg Ser Asp Ser Leu Ser Val His Ile Arg Thr His
85 90 95
Thr Gly Glu Lys Pro Phe Ala Cys Asp Ile Cys Gly Arg Lys Phe Ala
100 105 110
His Asn Asp Ser Arg Lys Asn His Thr Lys Ile His Thr Gly Ser Gln
115 120 125
Lys Pro Phe Gln Cys Arg Ile Cys Met Arg Asn Phe Ser Arg Ser Asp
130 135 140
Asp Leu Thr Arg His Ile Arg Thr His Thr Gly Glu Lys Pro Phe Ala
145 150 155 160
Cys Asp Ile Cys Gly Arg Lys Phe Ala Arg Ser Asp His Leu Thr Gln
165 170 175
His Thr Lys Ile His Leu Arg Gly Ser Gln Leu Val Lys Ser Glu Leu
180 185 190
Glu Glu Lys Lys Ser Glu Leu Arg His Lys Leu Lys Tyr Val Pro His
195 200 205
Glu Tyr Ile Glu Leu Ile Glu Ile Ala Arg Asn Ser Thr Gln Asp Arg
210 215 220
Ile Leu Glu Met Lys Val Met Glu Phe Phe Met Lys Val Tyr Gly Tyr
225 230 235 240
Arg Gly Lys His Leu Gly Gly Ser Arg Lys Pro Asp Gly Ala Ile Tyr
245 250 255
Thr Val Gly Ser Pro Ile Asp Tyr Gly Val Ile Val Asp Thr Lys Ala
260 265 270
Tyr Ser Gly Gly Tyr Asn Leu Pro Ile Gly Gln Ala Asp Glu Met Gln
275 280 285
Arg Tyr Val Glu Glu Asn Gln Thr Arg Asn Lys His Ile Asn Pro Asn
290 295 300
Glu Trp Trp Lys Val Tyr Pro Ser Ser Val Thr Glu Phe Lys Phe Leu
305 310 315 320
Phe Val Ser Gly His Phe Lys Gly Asn Tyr Lys Ala Gln Leu Thr Arg
325 330 335
Leu Asn His Ile Thr Asn Cys Asn Gly Ala Val Leu Ser Val Glu Glu
340 345 350
Leu Leu Ile Gly Gly Glu Met Ile Lys Ala Gly Thr Leu Thr Leu Glu
355 360 365
Glu Val Arg Arg Lys Phe Asn Asn Gly Glu Ile Asn Phe Arg Ser
370 375 380
<210> 4
<211> 1155
<212> DNA
<213>人工序列
<220>
<223> 人工序列的描述:合成
多核苷酸
<400> 4
atggactaca aagaccatga cggtgattat aaagatcatg acatcgatta caaggatgac 60
gatgacaaga tggcccccaa gaagaagagg aaggtgggca ttcacggggt acccgccgct 120
atggctgaga ggcccttcca gtgtcgaatc tgcatgtgta agtttgcccg caacgccgcc 180
ctgacccgcc ataccaagat acacacgggc gagaagccgt tccagtgtcg catctgcatg 240
cgtaacttca gtcgctccga cgagctgacc cgccacatcc gcacccacac aggcgagaag 300
ccttttgctt gcgacatttg tgggaggaag tttgcccggc accaccacct ggccgcccat 360
accaagatac acacgggatc tcagaagccc ttccagtgtc gaatctgcat gcgtaacttc 420
agtacccgcc cggtgctgaa gcgccacatc cgcacccaca caggcgagaa gccttttgct 480
tgcgacattt gtgggaggaa gtttgccgac cgctccgccc tggcccgcca taccaagata 540
cacctgcggg gatcccagct ggtgaagagc gagctggagg agaagaagtc cgagctgcgg 600
cacaagctga agtacgtgcc ccacgagtac atcgagctga tcgagatcgc caggaacagc 660
acccaggacc gcatcctgga gatgaaggtg atggagttct tcatgaaggt gtacggctac 720
aggggaaagc acctgggcgg aagcagaaag cctgacggcg ccatctatac agtgggcagc 780
cccatcgatt acggcgtgat cgtggacaca aaggcctaca gcggcggcta caatctgcct 840
atcggccagg ccgacgagat gcagagatac gtggaggaga accagacccg gaataagcac 900
atcaacccca acgagtggtg gaaggtgtac cctagcagcg tgaccgagtt caagttcctg 960
ttcgtgagcg gccacttcaa gggcaactac aaggcccagc tgaccaggct gaaccacatc 1020
accaactgca atggcgccgt gctgagcgtg gaggagctgc tgatcggcgg cgagatgatc 1080
aaagccggca ccctgacact ggaggaggtg cggcgcaagt tcaacaacgg cgagatcaac 1140
ttcagatctt gataa 1155
<210> 5
<211> 383
<212> PRT
<213> 人工序列
<220>
<223> 人工序列的描述:合成
多肽
<400> 5
Met Asp Tyr Lys Asp His Asp Gly Asp Tyr Lys Asp His Asp Ile Asp
1 5 10 15
Tyr Lys Asp Asp Asp Asp Lys Met Ala Pro Lys Lys Lys Arg Lys Val
20 25 30
Gly Ile His Gly Val Pro Ala Ala Met Ala Glu Arg Pro Phe Gln Cys
35 40 45
Arg Ile Cys Met Cys Lys Phe Ala Arg Asn Ala Ala Leu Thr Arg His
50 55 60
Thr Lys Ile His Thr Gly Glu Lys Pro Phe Gln Cys Arg Ile Cys Met
65 70 75 80
Arg Asn Phe Ser Arg Ser Asp Glu Leu Thr Arg His Ile Arg Thr His
85 90 95
Thr Gly Glu Lys Pro Phe Ala Cys Asp Ile Cys Gly Arg Lys Phe Ala
100 105 110
Arg His His His Leu Ala Ala His Thr Lys Ile His Thr Gly Ser Gln
115 120 125
Lys Pro Phe Gln Cys Arg Ile Cys Met Arg Asn Phe Ser Thr Arg Pro
130 135 140
Val Leu Lys Arg His Ile Arg Thr His Thr Gly Glu Lys Pro Phe Ala
145 150 155 160
Cys Asp Ile Cys Gly Arg Lys Phe Ala Asp Arg Ser Ala Leu Ala Arg
165 170 175
His Thr Lys Ile His Leu Arg Gly Ser Gln Leu Val Lys Ser Glu Leu
180 185 190
Glu Glu Lys Lys Ser Glu Leu Arg His Lys Leu Lys Tyr Val Pro His
195 200 205
Glu Tyr Ile Glu Leu Ile Glu Ile Ala Arg Asn Ser Thr Gln Asp Arg
210 215 220
Ile Leu Glu Met Lys Val Met Glu Phe Phe Met Lys Val Tyr Gly Tyr
225 230 235 240
Arg Gly Lys His Leu Gly Gly Ser Arg Lys Pro Asp Gly Ala Ile Tyr
245 250 255
Thr Val Gly Ser Pro Ile Asp Tyr Gly Val Ile Val Asp Thr Lys Ala
260 265 270
Tyr Ser Gly Gly Tyr Asn Leu Pro Ile Gly Gln Ala Asp Glu Met Gln
275 280 285
Arg Tyr Val Glu Glu Asn Gln Thr Arg Asn Lys His Ile Asn Pro Asn
290 295 300
Glu Trp Trp Lys Val Tyr Pro Ser Ser Val Thr Glu Phe Lys Phe Leu
305 310 315 320
Phe Val Ser Gly His Phe Lys Gly Asn Tyr Lys Ala Gln Leu Thr Arg
325 330 335
Leu Asn His Ile Thr Asn Cys Asn Gly Ala Val Leu Ser Val Glu Glu
340 345 350
Leu Leu Ile Gly Gly Glu Met Ile Lys Ala Gly Thr Leu Thr Leu Glu
355 360 365
Glu Val Arg Arg Lys Phe Asn Asn Gly Glu Ile Asn Phe Arg Ser
370 375 380
<210> 6
<211> 9
<212> PRT
<213> 未知
<220>
<223> 未知的描述:
“LAGLIDADG”家族肽基序序列
<400> 6
Leu Ala Gly Leu Ile Asp Ala Asp Gly
1 5
- 程序性细胞死亡1(PD1)特异性核酸酶
- CRISPR-Cas9特异性敲除人PD1基因的方法以及用于特异性靶向PD1基因的sgRNA
