一种基于增强子和启动子数据预测关键转录因子的方法
文献发布时间:2023-06-19 19:37:02
技术领域
本发明涉及生物信息技术领域,具体而言,涉及一种基于增强子和启动子数据预测关键转录因子的方法。
背景技术
随着高通量测序技术的发展和表观遗传学的兴起,染色质免疫共沉淀技术及转录组等测序技术应用十分广泛,不仅有力地推动了基础研究的发展,也逐渐应用于临床医学的转化。通常,我们聚焦高通量测序分析的基本套路是通过一系列分析找到目标靶基因,而往往忽略了上游机制中起着关键作用的转录因子。转录因子可识别并结合特异的顺式调控元件调控基因转录,直接控制基因表达的时间、地点和程度,在多种疾病的发生发展中均起着至关重要的作用。然而目前仍缺少基于准确数据预测关键转录因子的方法。
增强子和启动子均属于调控基因表达的重要转录调控元件,在基因的上游交错分布。通常情况下,转录因子可以通过其DNA结构域结合基因的启动子区域,实现对基因转录的调控。但值得注意的是,哺乳动物的染色体结构是通过调控增强子和启动子之间的三维相互作用来调控基因转录的。转录因子不单与基因的启动子结合,同样受基因的增强子的显著影响,在大多数情况下,激活的增强子会招募转录因子与启动子发生互作,通过增强子-启动子的相互作用实现对基因的非线性转录调控。即增强子和启动子共同决定着基因的转录水平。而现阶段预测基因关键转录因子的方法多是仅基于基因的启动子数据,而忽略了转录因子通过上游增强子与启动子相互作用而实现对基因表达的非线性调控。因此,亟需全面利用调控元件数据更新转录因子预测技术,从而解决目前方法预测结果不准确的问题。
发明内容
有鉴于此,本发明的目的在于提供一种基于增强子和启动子数据预测关键转录因子的方法,本发明的方法能够快速预测出介导增强子和启动子调控基因表达的关键转录因子。
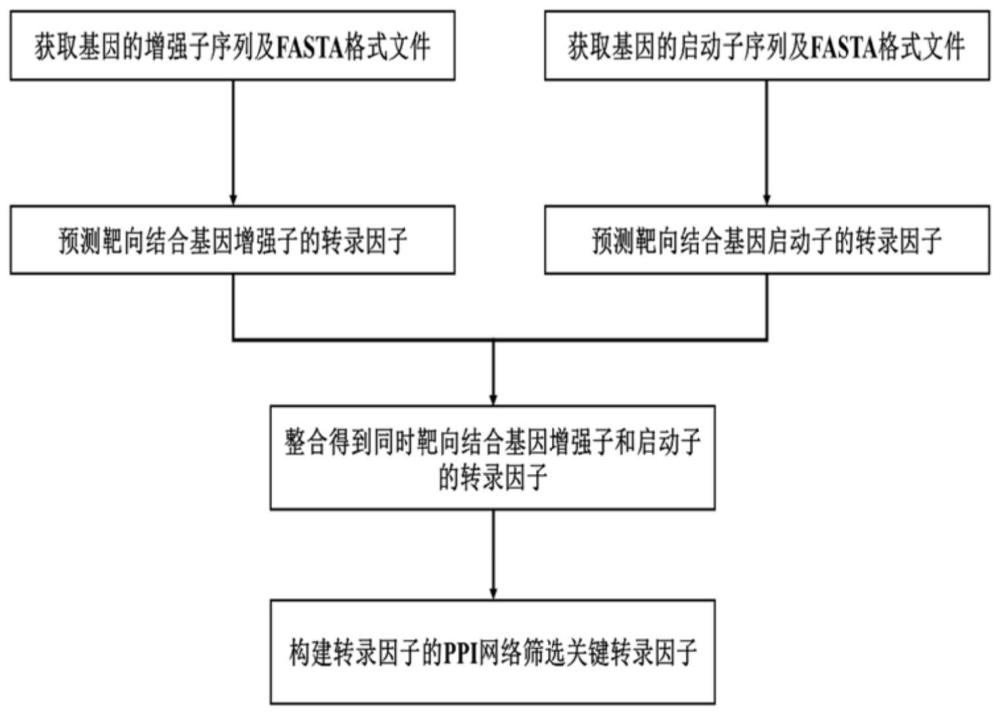
本发明提供了一种基于增强子和启动子数据预测关键转录因子的方法,包括以下步骤:
1)通过染色质免疫共沉淀测序(ChIP-Seq)或公开数据库获得基因增强子序列(具体的,人或小鼠的基因增强子序列可获取自VISTA Enhancer Browser(https://enhancer.lbl.gov/),猪的基因增强子序列可获取自Pig Genome Regulatory ElementBrowser(http://segtp.jxau.edu.cn/pencode/?genome=susScr11)),并通过公开数据库UCSC(https://genome.ucsc.edu/)获取基因增强子序列的FASTA格式文件;
2)根据公开数据库UCSC(https://genome.ucsc.edu/)中的基因转录起始位点位置信息,获取基因启动子序列的FASTA格式文件;
3)将基因增强子序列的FASTA格式文件导入转录因子在线预测系统PROMO(https://alggen.lsi.upc.es/),得到靶向结合增强子的转录因子;
4)将基因启动子序列的FASTA格式文件导入转录因子在线预测系统PROMO(https://alggen.lsi.upc.es/),得到靶向结合基因启动子的转录因子;
5)筛选同时靶向结合基因增强子和启动子的转录因子;
6)将步骤5得到的转录因子导入STRING数据库(http://string-db.org/),构建转录因子的蛋白质相互作用(PPI)网络;
7)筛选在蛋白质相互作用网络中处于核心位置的转录因子作为调控基因表达的关键转录因子。
优选的,步骤2)中选择基因的Upstream 2,000bases,CDS Exons和Downstream100bases序列生成FASTA格式文件。
优选的,步骤3)和步骤4)中预测转录因子时,将Maximum matrix dissimilarityrate设置为5%。
本发明中基因增强子序列、基因增强子序列的FASTA格式文件、基因启动子序列的FASTA格式文件等均是已知的,能通过相关的公开数据库直接查询获得。
本发明的优点在于:
1、本发明创新性地构建了整合基因增强子和启动子数据筛选关键转录因子的核心技术;
2、与现有仅基于基因启动子数据预测转录因子的方法相比,本发明通过引入增强子数据全面利用调控元件数据进行转录因子预测,有效解决了现有转录因子预测技术预测效率低,预测结果不准确的问题。
3、本发明中所述转录因子预测步骤均可通过在线数据系统可视化操作实现,操作上简单易行,降低了研究人员预测关键转录因子的技术门槛,具有更高的普适性;
4、本发明能够准确筛选出调控基因表达的关键转录因子,降低了研究人员的实验工作量及分析成本;
5、通过本发明所预测出的调控基因表达的关键转录因子可加深研究人员对于基因转录调控机制的探索,有利于研究人员基于关键转录因子进一步阐明疾病发生发展与转归的机制,并将进一步拓展转录因子在疾病治疗中的潜在应用价值;
附图说明
图1为本发明的流程图,
图2是本发明实施例2预测到的调控大鼠SCD基因表达的关键转录因子C/EBPβ;
图3是本发明实施例2的预测效果评价示意图。
具体实施方法
下面结合实施例及附图对本发明做进一步解释,但不限定于本发明。
实施例1:
本发明提供了一种基于增强子和启动子数据预测关键转录因子的方法,如图1所示包括以下步骤:
1)通过染色质免疫共沉淀后测序(ChIP-Seq)或公开数据库获得基因增强子序列,并通过公开数据库UCSC(https://genome.ucsc.edu/)获取基因增强子序列的FASTA格式文件;其中,基因增强子序列的获取具体为:人或小鼠的基因增强子序列可获取自VISTAEnhancer Browser(https://enhancer.lbl.gov/);猪的基因增强子序列可获取自PigGenome Regulatory Element Browser
(http://segtp.jxau.edu.cn/pencode/?genome=susScr11);
2)根据公开数据库UCSC(https://genome.ucsc.edu/)中的基因转录起始位点位置信息,获取基因启动子序列的FASTA格式文件;
3)将基因增强子序列的FASTA格式文件导入转录因子在线预测系统PROMO(https://alggen.lsi.upc.es/),得到靶向结合基因增强子的转录因子;
4)将基因启动子序列的FASTA格式文件导入转录因子在线预测系统PROMO(https://alggen.lsi.upc.es/),得到靶向结合基因启动子的转录因子;
5)筛选同时靶向结合基因增强子和启动子的整合转录因子;
6)将步骤5得到的整合转录因子导入STRING数据库(http://string-db.org/),构建整合转录因子的蛋白质相互作用(PPI)网络;
7)筛选在蛋白质相互作用网络中处于核心位置的转录因子作为调控基因表达的关键转录因子。
实施例2
预测调控大鼠SCD基因表达的关键转录因子:
其步骤如下:
1)通过ChIP-Seq获得大鼠SCD基因的增强子序列(Chr1:264,150,872-264,175,682),并通过公开数据库UCSC(https://genome.ucsc.edu/)获取大鼠SCD基因增强子序列的FASTA格式文件(>rn6_dna range=chr1:264150872-264175682 5'pad=0 3'pad=0strand=+repeatMasking=none);
2)根据公开数据库UCSC(https://genome.ucsc.edu/)中的大鼠SCD基因转录起始位点位置信息,获取大鼠SCD基因启动子序列(Chr1:264,172,730-264,174,729)的FASTA格式文件。具体的,所选择的序列为大鼠SCD基因Upstream 2,000bases,CDS Exons和Downstream 100bases生成FASTA格式文件(>rn6_refGene_NM_139192range=chr1:264172730-264174729 5'pad=0 3'pad=0strand=-repeatMasking=none);
3)将大鼠SCD基因增强子序列的FASTA格式文件导入转录因子在线预测系统PROMO(https://alggen.lsi.upc.es/),设置Maximum matrix dissimilarity rate为5%,得到靶向结合大鼠SCD基因增强子的转录因子;
4)将大鼠SCD基因启动子序列的FASTA格式文件导入转录因子在线预测系统PROMO(https://alggen.lsi.upc.es/),设置Maximum matrix dissimilarity rate为5%,得到靶向结合大鼠SCD基因启动子的转录因子;
5)筛选同时靶向结合大鼠SCD基因增强子和启动子的整合转录因子;
6)将步骤5得到的整合转录因子导入STRING数据库(http://string-db.org/),构建整合转录因子的蛋白质相互作用(PPI)网络;
7)筛选在蛋白质相互作用网络中处于核心位置的转录因子C/EBPβ作为调控大鼠SCD基因表达的关键转录因子,参见图2,其关键转录因子为C/EBPβ。
实施例3:验证实验
为验证实施例2中“C/EBPβ作为调控大鼠SCD基因表达的关键转录因子”结论的真实性与有效性,我们分别进行了双荧光素酶报告基因实验和实时荧光定量聚合酶链式反应(RT-qPCR)实验。
其中,双荧光素酶报告基因实验流程简述如下:
(1)质粒构建:对于双荧光素酶报告基因实验,所用质粒分别为:①报告基因质粒:带有大鼠SCD基因启动子(其正义链为:5’-GGGTCAGAGCATCTCAGGGACC-3’,反义链为:5’-TTCACCCAGCAGCAGGCGAAAG-3’)的表达萤火虫荧光素酶的报告基因质粒pGL3-SCD;②内参质粒:表达海参荧光素酶的内参质粒pRL-TK;③转录因子C/EBPβ的表达质粒pcDNA3.1-C/EBPβ;④pcDNA3.1空白对照质粒。
(2)细胞转染:待细胞铺板密度达约70%时,将细胞随机分为C/EBPβ组和空载组。使用①报告基因质粒,②内参质粒和③转录因子C/EBPβ的表达质粒pcDNA3.1-C/EBPβ共转染C/EBPβ组细胞;使用等量①报告基因质粒,②内参质粒和④pcDNA3.1空白对照质粒在同等环境下共转染空载组细胞。
(3)荧光素酶活性测定与数据处理:充分裂解细胞,离心后取上清,按顺序分别测定萤火虫萤光素酶和海肾萤光素酶的相对光单位(relative light unit,RLU)。而后用萤火虫萤光素酶测定得到的RLU值除以海肾萤光素酶测定得到的RLU值作为荧光素酶活性。而后根据得到的荧光素酶活性来比较C/EBPβ组和空载组间大鼠SCD基因的激活程度。如图3(a)所示,实验发现C/EBPβ组的荧光素酶活性显著高于空载组(组间比较采用t检验,***:P<0.001),即转录因子C/EBPβ具有促进大鼠SCD基因表达的作用,印证了本发明中基于增强子和启动子数据预测关键转录因子的方法的预测准确性。
其中,实时荧光定量聚合酶链式反应(RT-qPCR)实验流程简述如下:
(1)待细胞生长适宜时,将细胞随机分为C/EBPβ组和空载组。使用C/EBPβ基因过表达质粒转染C/EBPβ组细胞,使用空白质粒转染空载组细胞;
(2)使用异硫氰酸胍-苯酚法(TRIzol法)提取细胞总RNA并测定RNA浓度;
(3)去除基因组DNA,其中PCR程序为42℃2min,4℃∞;
(4)反转录得cDNA,其中PCR程序为37℃15min,85℃5s,4℃∞;
(5)使用SYBR GreenⅠ荧光染料法进行RT-qPCR,其中RT-qPCR扩增条件为:cDNA预变性95℃10min,然后变性—退火—延伸循环95℃30s,60℃1min,40个循环。随后根据2
综上,双荧光素酶报告基因实验结果和RT-qPCR实验结果共同说明C/EBPβ是参与大鼠SCD基因表达调控的关键转录因子,使用该发明方法基于增强子和启动子数据预测关键转录因子结果准确可靠。
由以上实例可知,本发明提供了一种基于增强子和启动子数据预测关键转录因子的方法,该方法能够通过整合增强子和启动子数据准确、快速地预测出调控基因表达的关键转录因子。
上述对实施例的描述是为便于该技术领域的普通技术人员能理解和应用本发明。熟悉本领域技术的人员显然可以容易地对实施案例做出各种修改,并把在此说明的一般原理应用到其他实施例中而不必经过创造性的劳动。因此,本发明不限于这里的实施案例,本领域技术人员根据本发明的揭示,不脱离本发明范畴所做出的改进和修改都应该在本发明的保护范围之内。
- 一种高效高通量启动子与增强子文库构建方法
- 一种融合多数据特征预测关键蛋白质的计算方法
- 一种基于神经网络的经营数据预测方法、可读存储介质和预测系统
- 一种基于堆叠式集成策略的用于高效识别特定细胞系增强子-启动子相互作用的预测方法
- 基于Hilbert编码的增强子-启动子相互作用预测方法及装置
