基于卷积神经网络预测蛋白和配体分子结合自由能的方法
文献发布时间:2023-06-19 09:27:35
技术领域
本发明属于基于人工智能的药物研发领域,具体为基于卷积神经网络预测蛋白和配体分子结合自由能的方法,实现了应用卷积神经网络(CNN)精准预测受体-药物分子的结合自由能,以应用于新药设计与开发。
背景技术
新药设计与开发是一个创造性和探索性的研究工作。药物分子设计是以理性的策略和科学的规划,构建具有预期药理活性的新分子实体。分子设计是将具有活性的化合物经过逐步优化,转化为在人体内安全有效且可控易得的化合物,在变换和修饰结构过程中满足对药物多维属性的要求。
先导物优化过程,包括其他基于结构的药物设计方法,比如从头设计等,最终往往都面临一个同样的问题,就是配体分子和受体分子之间相互作用的评价。许多药物分子的活性都是通过与受体大分子之间的相互作用表现出来的,所以受体和配体之间结合自由能的预测是基于结构的计算机辅助药物分子设计的核心问题。精确的自由能预测方法能够大大提高药物设计的效率。
目前的结合自由能预测主要是基于计算化学的模拟方法,包括基于分子力学和连续介质模型的MM/GBSA、MM/PBSA 方法,以及自由能微扰(FEP)和热力学积分(TI)方法。其中,由于在先导物优化过程中涉及到大量骨架结构相似的同系物结构,所以基于FEP的相对结合自由能计算应用较广。另一方面,也有许多基于经验和知识的打分函数来预测受体-药物分子的亲和力。
已有技术问题及缺陷描述:
当前的评价受体-药物分子相互作用强度的方法,包括基于计算化学的模拟方法,以及基于经验或知识的打分函数方法,都存在一定缺陷。如FEP方法,面临着模拟体系准备、溶剂模型的处理、力场选择以及计算成本等问题,使得在实践中大规模应用较为困难。而打分函数方法,主要是在一些化学结构非常不同的数据集上训练拟合得到,难以泛化到具有很小结构差异的同系物配体上,所以在先导物优化场景中应用有限。
发明内容
为解决以上问题,本发明提出一种基于卷积神经网络的方法预测靶点和药物分子的结合自由能。
为实现上述目的,本发明采用以下方案:
基于卷积神经网络预测蛋白和配体分子结合自由能的方法,包括以下步骤:蛋白和小分子结构数据收集、蛋白和小分子描述符计算和构象生成以及卷积神经网络架构处理和数据分析模块。
具体包括以下步骤:
(1)从数据库获取高质量的蛋白-配体复合物晶体结构,以及从文献中收集小分子结构并进行3D构象准备;
从几个公共结构数据库收集蛋白-配体复合物结构数据,并从近年发表文献中提取结构数据,以及几个药物靶点的公共数据集。所有分子通过内部分子准备流程进行配体准备,包括加氢、质子化、三维构象生成等,再以共晶配体分子为参考结构,将所有分子进行分子对接。
(2)对蛋白和小分子进行编码和描述符计算,将蛋白处理为3D图像;
为利用卷积神经网络,需要对配体和蛋白分子进行编码。和计算机视觉问题相似,将蛋白结构处理为3D图像。与2D图像(由像素组成)类似,3D图像由体素组成。将这个3D图像的坐标定义在一个盒子内,并将3D图像离散到密度为1*1*1Å
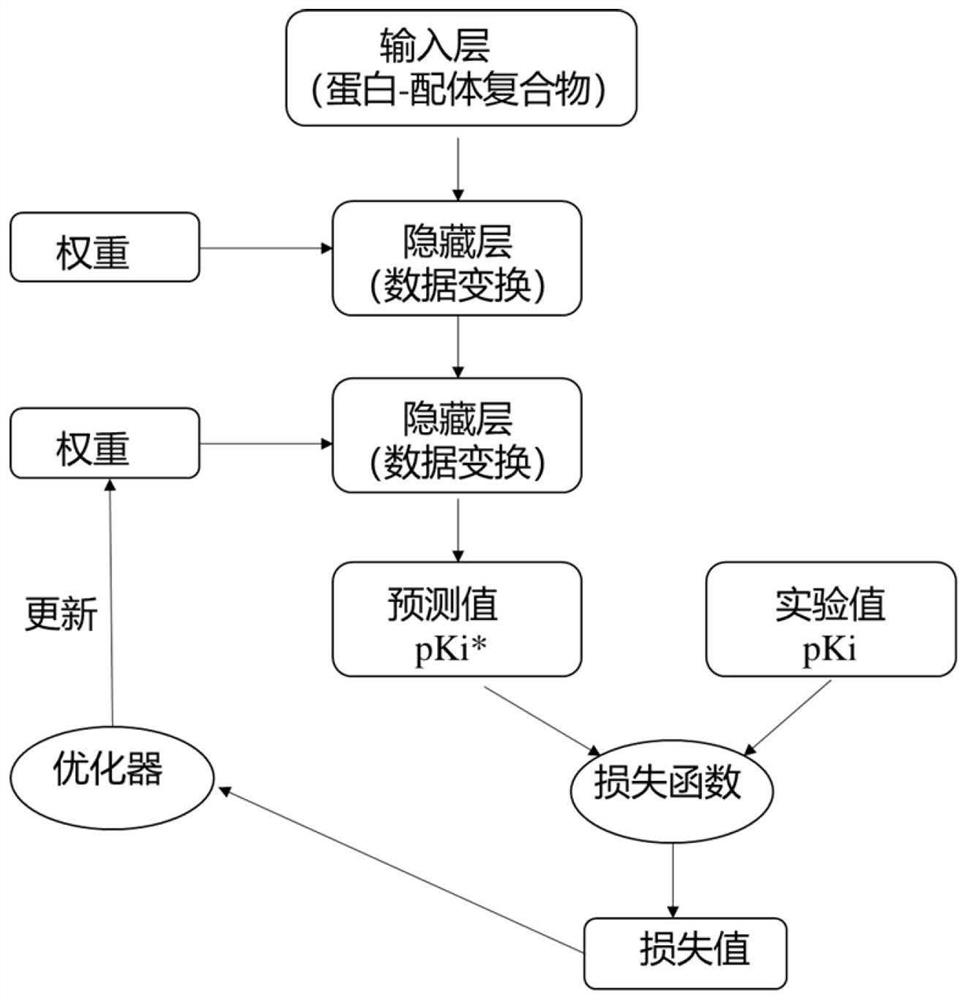
(3)用蛋白的3D图像作为输入,设计卷积神经网络的架构,包含输入层、隐藏层和输出层;
卷积神经网络主要包括输入层、隐藏层、输出层,其中隐藏层包含卷积层、池化层和全连接层。卷积层在输入特征空间学到的是局部模式。这个特性使得卷积神经网络学到的模式具有平移不变性。当网络在蛋白质的3D图像某个位置学到某个模式之后,它可以在任何地方识别这个模式。这使得卷积神经网络在处理蛋白质图像时可以高效利用数据,所以只需要更少的训练样本就可以学到具有泛化能力的数据表示。除此之外,该网络还可以学到模式的空间层次结构。第一个卷积层学习较小的局部模式。第二个卷积层将学习由第一层特征组成的更大的模式,以此类推。这使得网络可以有效地学习蛋白质图像中越来越复杂、越来越抽象的视觉概念。
(4)对预训练的模型进行特征提取和微调,以应用在新数据集上;
利用特征提取,可以很容易将现有的卷积神经网络复用于新的数据集。将模型在大型数据集上预训练保存好,预训练网络学到的特征的空间层次结构可以有效地作为视觉世界的通用模型,因此这些特征可用于其他靶点的结合自由能预测。
(5)待所有任务完成后,调用Pandas和Matplotlib数据分析库,直接在jupyter中绘制分析结果曲线。
其中,所述的数据库为PDB、scPDB、PDBbind数据库。
进一步的,所述步骤(2)中,将蛋白处理为3D图像,并用Keras的IamgeDataGenerator实例读取的图像执行多次随机变换来实现数据增强。
步骤(3)中所述的隐藏层包含4个卷积层和2个最大池化层;输出层为密集连接层,并在前面添加一个Dropout层以防止过拟合。
本发明的隐藏层中使用了4个卷积层和2个最大池化层。对所有层使用ReLU激活函数。为防止过拟合,本发明在模型的密集连接层之前添加一个Dropout层。本发明中权重的初始化使用Xavier初始化,优化器使用Adam随机梯度下降。
除此之外,为防止过拟合,本发明通过对IamgeDataGenerator实例读取的图像执行多次随机变换来实现数据增强,这样从现有的训练样本中生成更多的训练数据。这样,模型在训练时不会两次查看完全相同的图像,这让模型能够观察到数据的更多内容,从而具有更好的泛化能力。
本发明中训练好的卷积神经网络包含两部分:首先是一系列池化层和卷积层,最后是一个密集连接层。第一部分是模型的卷积基。本发明的特征提取过程,取出之前训练好的网络的卷积基,在上面运行新数据。避免重复使用密集连接层,因为卷积基学到的表示更加通用,因此更适合重复使用。而密集连接层的表示不再包含物体在输入图像中的位置信息,它舍弃了空间的概念。
卷积层提取的表示的通用性及可复用性取决于该层在模型中的深度。模型中更靠近底部的是局部的、高度通用的特征图,而更靠近顶部的层提取的是更加抽象的概念,所以本发明只使用模型的前几层来做特征提取,而不使用整个卷积基。
另一种模型复用方法是模型微调(fine-tuning)。本发明将模型顶部的几层“解冻”,并将解冻的这几层和新增加的部分联合训练。它略微调整了所复用模型中更加抽象的表示,以让这些表示与研究问题更相关。本发明中微调的步骤如下:
(a)在已训练好的基网络上添加自定义网络;
(b)冻结基网络;
(c)训练新添加的部分;
(d)解冻基网络的一些层;
(e)联合训练解冻的这些层和添加的部分;
本发明采用以上技术方案,其优点在于:在训练完大型数据集之后,指定具体靶点和小分子初始结构,可以自动计算其结合自由能及数据分析。用户可以在Jupyter notebook中随时查看和修改任务的执行状态,也能通过图像界面的方式可视化已有的计算和分析结果。
本发明带来了如下了效果:
1. 实现了蛋白质和小分子的空间层次结构表示,将其表示为计算机视觉问题中的3D图像,以作为卷积神经网络的输入;
2.实现了快速、精准的蛋白-配体结合自由能计算,与实验值相比,误差较小,并可以将结果自动进行数据分析和可视化;
3. 学习到的蛋白-配体空间层次结构可为下一步药物设计提供洞见,实现新的从头分子设计。
附图说明
图1是本发明应用卷积神经网络计算蛋白-配体结合自由能的原理图;
图2是本发明所用卷积神经网络的架构图;
图3是本发明中卷积神经网络模型的交叉验证示意图;
图4是本发明中卷积神经网络模型训练过程中验证集的精度示意图;
图5是本发明中卷积神经网络模型训练过程的平滑示意图;
图6是本发明中对小分子化合物的结构特征描述;
图7是本发明中基于已有实验数据的模型预测示意图;
图8是本发明中的从头设计实验结果预测示意图。
具体实施方式
下面结合附图,对本发明的较优的实施例作进一步的详细说明:
以下实施例采用如图1所示的原理,所用卷积神经网络的架构如图2所示。
实施例1
针对一个激酶靶点进行结合自由能预测:
首先收集该靶点的小分子抑制剂,进行3D构象准备和分子对接计算,再调用分子描述符模块对这些分子和蛋白进行处理,输入到卷积神经网络模型中。如图3所示,将所有分子数据分为5份,每次选取其中一份作为验证集,其他四份作为训练集,这样总共将模型训练5轮,即5折交叉验证,最后将所有的验证分数取平均值作为最终的验证打分。图4、图5表示模型训练过程中在验证集上的表现,从图4中可以看出,模型起始阶段误差较大,但经过50轮左右训练后,误差降到2.3左右,后续维持在这个水平。图5是图4的平滑版。由于图4中纵轴的范围较大,且数据方差也相对较大,所以较难看清模型训练过程中的规律。所以图5删除图4中前10个数据点,因为它们的取值范围较大,与曲线上其他点不同;同时将每个数据点表示为前面数据点的指数移动平均值,以得到平滑的曲线。从图5可以看出,在80轮训练后,模型的验证打分最好,后续趋向于过拟合,所以只需选取此时的模型作为最终模型。
分子描述结果如图6。通过这些分子描述,卷积神经网络识别到分子的空间结构特征,分别用不同球体表示,如浅色球体表示疏水特征,深色球体表示氢键供体特征,更深色的球体表示氢键受体特征等。
通过识别这些不同的空间结构特征,模型学习到蛋白-配体结合过程中的关键细节,并通过卷积层和最大池化层的数据变换,不断学习、迭代,最后计算完成后,调用数据分析模块分析计算结果。对比计算预测值和实验测试值,如图7所示,可以看出,计算结果和实验结果高度吻合,均匀分布在一条直线上,相关性系数达到0.87,表明模型学习到了该靶点和小分子结合过程中的匹配细节,从而有较好的预测能力。
实施例2
针对一个靶点进行先导化合物结构优化:
通过虚拟筛选得到一些初步活性不错的苗头化合物,利用该卷积神经网络模型对此批苗头化合物进行结构优化,以得到先导化合物。
首先将苗头化合物与靶点进行分子对接,再将它们进行编码和计算分子描述符。将其输入卷积神经网络模型,预测化合物与靶点的结合自由能。根据苗头化合物和靶点之间的结合模式,以及模型预测的结合自由能值和空间层次结构信息,对苗头化合物进行结构改造和优化,在苗头化合物上添加和靶点空间层次结构匹配的基团,与靶点形成更好的结构互补,以设计新的化合物。随后再将新化合物与靶点进行分子对接和编码计算,输入模型并给出结合自由能预测值。再次分析新化合物和靶点的结合模式和自由能预测值,并于上一批次结果比较,获得新的洞见,并反馈给团队成员。团队成员根据计算结果进行评估反馈,挑选分子进行化学合成和生物活性测试,并将实验结果反馈给计算团队。计算团队再根据实验值和计算值进行比较,分析结构和数据关系,进行新一轮的药物设计。如图8所示,横轴表示通过设计并合成出的化合物实验测试生物活性,纵轴表示通过模型预测的活性,可以看出,两者较均匀地分布在一条直线附近,表示有较好的相关性,相关性系数达到0.77。在这个实际药物发现的案例中,本发明中的模型起到积极推进的作用。经过几轮的迭代和测试,加速找到了合理的先导化合物。过程中所有的数据、方法和结构信息都会自动保存,便于日后溯源。
本方法和其他传统的药物设计方法比较,流程化的操作提升了工作效率,以计算结果与实验结果的高度吻合表明了模型的精准性。本案例中还保存了计算和实验的一切结构和信息数据,使得计算分析过程更加方便,进一步提升了效率。
