基于L1范数图的抗病毒药物筛选方法
文献发布时间:2023-06-19 19:28:50
技术领域
本发明属于生物信息学、计算生物学与人工智能交叉领域,具体涉及一种基于L1范数图的抗病毒药物筛选方法。
背景技术
冠状病毒SARS-CoV-2引发全球性疫情,成为了世界公共卫生的严重威胁,寻找有效的抗病毒药成为极为紧迫的任务。然而,按常规方法研发药物可能需要耗时十多年、耗资数十亿美元,在短时间内开发出一种有效抗病毒药物是极为困难的。考虑到已成熟的药品其有效性、安全性和毒性都是经过测试的,于是“老药新用”从已经应用的药品中寻找有效方案是应对突发疫情应对药物的一种高效的解决方法。
抗病毒药物筛选方法已有报道,其中一类是基于结构的虚拟筛选方法,如使用动力学模拟技术计算潜在药物和靶标间的结合能力,通过分子动力学模拟计算药物的吸收、分布、代谢、排泄和毒性等。此类方法通常存在模拟过程复杂、对使用者经验要求高等不足。国防科技大学天河超算团队提出了基于自由能微扰-绝对结合自由能方法的新冠药物虚拟筛选方法,但这种基于自由能的大规模筛选方法对算力要求较高,需要借助超级计算机平台,且耗时以周计算。
发明内容
为了克服现有技术的上述缺陷,本发明提供一种基于L1范数图的抗病毒药物筛选方法(Virus-Drug Association prediction based on L
本发明提供的技术方案具体包括以下步骤:
步骤一,输入已知的病毒-药物关联对,构建邻接矩阵A,若为已知关联对则对应位置为1,否则为0,此矩阵的行数为病毒数量nv,列数为药物数量nd;
步骤二,分别计算药物间高斯距离相似性和病毒间高斯距离相似性:若药物d(i)与某个病毒之间存在关联,则对应位置记为1否则记为0,形成一个1×nv大小的0或1构成的向量,记之为药物d(i)的向量谱IP(d(i)),然后计算药物d(i)和d(j)之间的高斯距离相似性
/>
上式中,参数γ
以类似的方式定义病毒v(i)和v(j)之间的高斯距离相似性,1×nd大小的0或1构成的向量,记之为病毒v(i)的向量谱IP(v(i));
以上γ’
步骤三,输入病毒基因组序列,使用既有较为成熟的多序列比方法计算病毒序列相似性,输入药物的化学结构得到药物MACCS指纹,采用谷本系数(Tanimoto Coefficient,即Jaccard相似度)计算药物化学结构相似性;
步骤四,使用快速核学习方法整合病毒基因组序列相似矩阵和病毒高斯距离相似矩阵,具体是通过求解下面的半正定规划式:
式中,第一项为范数项表示相似矩阵的整合误差大小,第二项为为正则化项,作用是避免过拟合,其中A为病毒-药物关联邻接矩阵,S
步骤五,基于半监督学习方法,从病毒空间视角构造目标函数,具体为:
其中S
步骤六,使用L1范数重写上述目标函数得到:
步骤七,求算数平均值,得到最终的预测结果
本发明的技术效果和优点:
1、通过引入稀疏范数约束项使目标函数生成稀疏解,能有效减轻训练数据集中存在的内在噪声的影响,进而使得病毒-药物关联预测结果更具有鲁棒性、更准确;
2、整合的病毒相似性以及整合的药物相似性都可以在每轮迭代期间根据标签矩阵自适应地改变,具有较好的可扩展性与健壮性,使本方法能获得较佳预测结果;
3、通过集成了拉普拉斯项融合了流形学习理论,属于半监督模型,能够高效利用阴性样本信息,降低了模型构建的难度,提升了预测性能。
附图说明
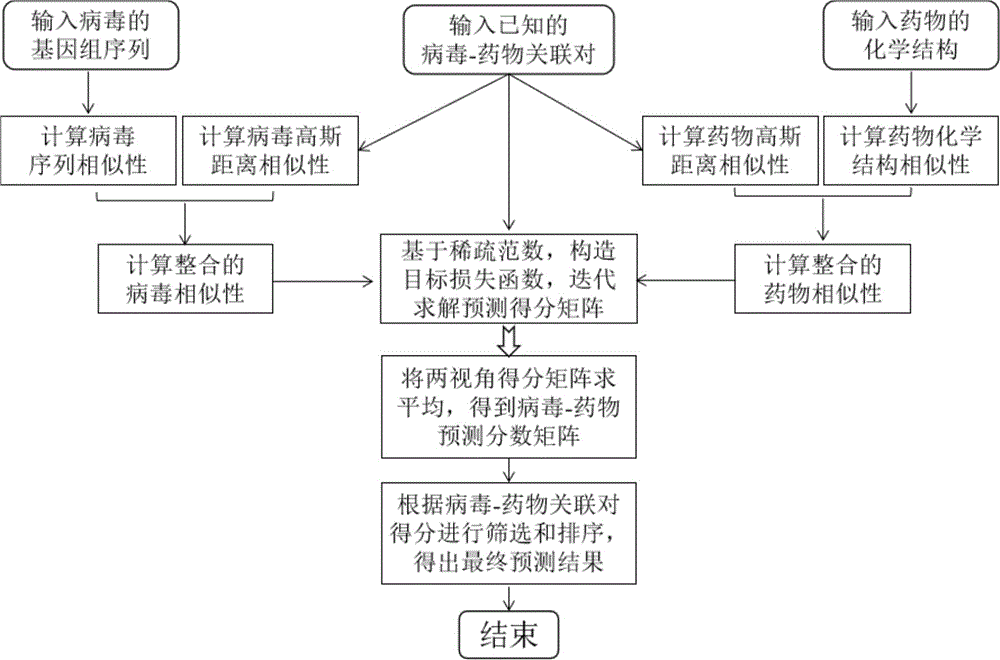
图1为本发明的总体流程图。
图2为本发明与几种已报道方法在同一数据集上五倍交叉验证的结果图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行完整地描述。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
本发明的实施例中所使用的已知人类药物-病毒关联数据是从有关文献中收集的,先使用文本挖掘技术对文献报道的经过实验验证的药物-病毒相互作用对进行整理后,获得455个已证实的人类病毒-药物相互作用,涉及34种病毒与219种药物(文献DOI:10.1016/j.asoc.2021.107135);药物化学结构从DrugBank数据库下载,病毒基因组核苷酸序列从美国国家生物技术信息中心NCBI数据库获得,然后执行如图1所示的一种基于L1范数图的抗病毒药物筛选方法,具体包括如下步骤:
步骤一,输入已知的药物-病毒关联对,构建邻接矩阵A:
得到的邻接矩阵A元素为0或1,大小为34行×219列,i与j的取值范围满足1≤i≤34,1≤j≤219;
步骤二,分别计算病毒高斯距离相似性和药物高斯距离相似性:
若某一个病毒v(i)与某药物之间存在关联,则对应位置记为1否则记为0,形成一个1×219大小的0或1构成的行向量,记之为病毒v(i)的向量谱IP(v(i)),然后计算病毒v(i)和v(j)之间的高斯距离相似性:
上式中,参数γ
以类似的方式定义药物d(i)和d(j)之间的高斯距离相似性:
取
其中nv表示病毒的数量,此例中为34,nd表示药物的数量,此例中为219,此步计算后得到大小为34×34的对称矩阵S
步骤三,输入病毒基因组序列,使用多序列比对工具MAFFT计算得到病毒序列相似矩阵S
步骤四,使用快速核学习方法整合病毒基因组序列相似矩阵和病毒高斯距离相似矩阵,具体通过求解下面的半正定规划式:
式中,第一项为范数项表示相似矩阵的整合误差大小,第二项为为正则化项,作用是避免过拟合,其中A为病毒-药物关联邻接矩阵,S
步骤五,基于半监督学习方法,从病毒空间视角构造目标函数,具体为:
其中S
步骤六,使用L1范数重写上述目标函数得到
步骤七,求算数平均值,得到最终的预测结果
具体使用R语言编程实现上述算法时,输入矩阵:病毒-药物关联对矩阵A、病毒整合相似矩阵S
本发明的有效性验证:
如图1所示的一种基于L1范数图的抗病毒药物筛选方法,采用五重交叉验证进行预测性能评估,具体实施方式为:先将所有已知的药物-病毒关联随机平均分成5组,再将5组中的每一组依次设为测试样本,其他组作为训练样本(测试样本选取情况不同时,依赖测试样本计算所得的高斯距离相似性矩阵亦随之改变)。使用训练样本作为本方法的输入得到预测结果,最后将该组中每个测试样本的预测分数与候选样本的分数进行比较。为了减少生成测试样本的过程中随机划分对结果造成的影响,进行了100次五折交叉验证。
使用R语言编程计算后获得了如下数据,如图2所示为本方法L1norm-VDA与现已报道的的几种病毒-药物筛选模型之间的AUROC(ROC曲线下面积)值比较。本方法在5折交叉验证中取得了0.7897±0.0017的AUROC值,表现出了比几种经典模型更加出色的预测性能。
另外一方面,对具体某种病毒,如新型冠状病毒(SARS-CoV-2)使用本方法来做预测,筛选评分矩阵Q
下表展示了预测结果前20个药物名称和支持的文献PMID、引文格式或DOI号。
最后:以上所述仅为本发明的优选实施例之一,并不用于限制本发明,凡在本发明的精神和原则之内,所作的细微修改、等同替换、简单改进等,均应包含在本发明的保护范围之内。
