一种多模态药物-蛋白质靶点相互作用预测方法及系统
文献发布时间:2023-06-19 19:30:30
技术领域
本发明属于生物基因和人工智能药物研究技术领域,尤其涉及一种多模态药物-蛋白质靶点相互作用预测方法及系统。
背景技术
在药物研发过程中,一款新药的研发一般需要经历临床前研发和临床试验两个阶段,其中在临床前研发阶段又需要经历苗头化合物(Hit)发现、先导化合物(Lead)优化和药效试验等过程,通过在研发早期通过预测方式快速发现潜在药物-蛋白质靶点相互作用(Drug Protein Interactions,DPIs),从而显著缩小潜在药物分子的搜索空间,实现高效准确的药物研发流程。随着医药数据的不断积累,使用基于数据驱动的机器学习方法预测DPI受到了越来越多的关注,较于传统的基于药化专家经验或实验室测定来发现有效化合物的方法,使用机器学习方法预测药物-蛋白质靶点相互作用关系能够对海量化合物进行高通量筛选,进而在早期发现具有较高活性和价值的候选化合物。目前基于机器学习的DPI预测方法主要可以分为基于相似度/距离的方法、基于特征的方法和基于深度学习的方法,其中基于深度学习的方法能够利用深度学习方法强大的特征提取和学习能力有效的挖掘大规模药化数据中存在的潜在关联,从而为DPI的准确预测提供了坚实的基础。
DPI预测主要由药物分子和蛋白质靶点的特征表示和特征交互两部分组成。通常情况下,药物分子可由一维SMILES序列进行表示,也可由二维的分子图结构进行描述;蛋白质靶点可由一维的氨基酸序列进行表示,也可由三维的空间结构来表示,但是对于蛋白质靶点而言,其三维空间结构的获取与有效的特征编码通常难以实现,因此在实际应用中通常基于一维氨基酸序列获取蛋白质靶点的语义特征作为其特征表示。而目前已有研究提出利用二维距离矩阵对蛋白质的空间结构进行表征,其中矩阵的每个元素表示对应的氨基酸之间的距离,这为蛋白质的结构表征提供了新的解决思路。尽管如此,在目前众多的DPI预测方法中仍然存在着许多难点问题亟待解决:1)为了实现DPI的准确预测,需要对药物分子和蛋白质靶点进行充分且有效的特征表示,利用不同模态的信息增强模型的学习能力;2)药物分子与蛋白质靶点的结合与相互作用实质上是在三维空间上发生的,因此结构信息在DPI预测问题中具有重要的作用,但是现有的DPI预测方法缺乏有效的手段对药物分子和蛋白质靶点的结构信息进行提取与表征;3)药物分子和蛋白质靶点特征的有效融合对于DPI的准确预测至关重要,因此需要设计合适的特征融合网络结构。
因此,上述的现有技术确实有待提出更佳解决方案的必要性。
发明内容
本发明的目的是提供一种多模态药物-蛋白质靶点相互作用预测方法及系统,该方法首先分别对药物分子和蛋白质靶点进行信息编码;在药物分子的特征编码方面,使用图神经网络对分子图进行特征提取与编码获取分子的结构特征,同时利用预训练模型Mol2vec获取分子的语义特征;在蛋白质靶点特征编码方面,基于蛋白质靶点的预测亲和度矩阵和序列匹配特征构建蛋白质图,并利用图神经网络和卷积神经网络提取蛋白质靶点的结构特征,同时基于预训练BERT模型以氨基酸序列作为输入获取到蛋白质靶点的语义特征。在获取到药物分子和蛋白质靶点的结构、语义特征后,进一步利用Transformer网络对药物分子和蛋白质靶点的特征进行融合以进行DPI的预测。通过对结构、语义多模态信息的表征与融合,该方法可以有效提取到药物分子和蛋白质靶点的相互作用信息从而实现准确的DPI预测,提高药物-蛋白质靶点相互作用的预测准确度。
本发明一方面提供了一种多模态药物-蛋白质靶点相互作用预测方法,包括:
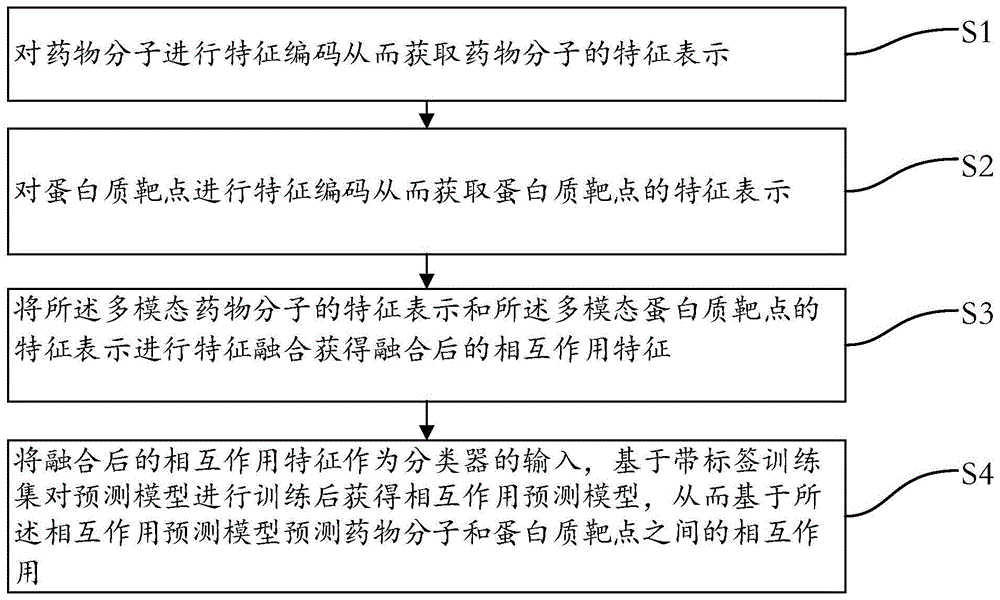
S1,对药物分子进行特征编码从而获取药物分子的特征表示;
S2,对蛋白质靶点进行特征编码从而获取蛋白质靶点的特征表示;
S3,将所述多模态药物分子的特征表示和所述多模态蛋白质靶点的特征表示进行特征融合获得融合后的相互作用特征;
S4,将融合后的相互作用特征作为分类器的输入,基于带标签训练集对预测模型进行训练后获得相互作用预测模型,从而基于所述相互作用预测模型预测药物分子和蛋白质靶点之间的相互作用。
优选的,所述S1包括:
S11,利用RDKit工具将分子的SMILES序列表示转换为图结构表示,并基于图结构表示得到相应的分子图特征;
S12,将分子图特征作为输入,通过图神经网络编码得到药物分子的结构特征;
S13,将分子的SMILES序列作为输入,通过第一预训练模型得到药物分子的语义特征;
S14,将所述药物分子的结构特征和所述药物分子的语义特征进行整合编码,得到相应的多模态药物分子的特征表示。
优选的,所述第一预训练模型为预训练Mol2vec模型。
优选的,所述S2包括:
S21,基于蛋白质靶点的预测氨基酸亲和度矩阵和基于氨基酸序列匹配的特征向量构建蛋白质图,所述蛋白质图的结构特征表征蛋白质中氨基酸残基之间的空间结构关系;
S22,将蛋白图特征作为输入,通过图神经网络和卷积神经网络编码得到蛋白质靶点的结构特征;
S23,将所述蛋白质靶点的一维氨基酸序列作为输入,通过第二预训练模型得到蛋白质靶点的语义特征;
S24,将所述蛋白质靶点的结构特征和所述蛋白质靶点的语义特征进行整合编码,得到相应的多模态蛋白质靶点的特征表示。
优选的,所述第二预训练模型为预训练BERT模型。
优选的,所述S3包括:
S31,将药物分子的多模态特征M
S32,对交互特征X在节点维度进行聚合获得融合后的相互作用特征。
优选的,所述S32所述聚合包括:
从而得到可以描述药物-蛋白质靶点相互作用的特征向量h;x
优选的,所述S4包括:
S41,将药物-蛋白质靶点相互作用特征向量h输入到包含多个全连接层的DPI分类器中;
S42,定义损失函数L如下:
其中
S43,基于带标签的训练集和预定义的损失函数对所述DPI分类器进行训练得到表现良好的相互作用预测模型。
本发明的第二方面提供一种多模态药物-蛋白质靶点相互作用预测系统,包括:
第一特征编码模块,用于对药物分子进行特征编码从而获取药物分子的特征表示;
第二特征编码模块,用于对蛋白质靶点进行特征编码从而获取蛋白质靶点的特征表示;
特征融合模块,用于将所述多模态药物分子的特征表示和所述多模态蛋白质靶点的特征表示进行特征融合获得融合后的相互作用特征;
相互作用预测模块,用于将融合后的相互作用特征作为分类器的输入,基于带标签训练集对预测模型进行训练后获得相互作用预测模型,从而基于所述相互作用预测模型预测药物分子和蛋白质靶点之间的相互作用。
本发明的第三方面在于提供多模态药物-蛋白质靶点相互作用预测方法在潜在药物分子的搜索中的应用。
本发明提供的方法、系统以及应用,具有如下有益的技术效果:
本发明创新性地提出了一种基于结构的多模态药物-蛋白质靶点相互作用预测方法,通过融合药物分子和蛋白质靶点的结构与语义多模态特征,能够对药物分子和蛋白质靶点实现完整充分的描述,同时利用Transformer网络能够对药物分子和蛋白质靶点的相互作用进行有效建模,从而实现更加准确地DPI预测。该发明不仅能够提高DPI预测的准确度,而且利用Transformer网络中存在的注意力机制能够对药物分子和蛋白质靶点的相互作用机制进行定性解释,对于模型在实际项目中的应用具有良好的促进作用。
附图说明
图1为根据本发明优选实施例示出的多模态药物-蛋白质靶点相互作用预测方法流程示意图;
图2为根据本发明优选实施例示出的多模态药物-蛋白质靶点相互作用预测方法原理示意图;
图3为根据本发明优选实施例示出的多模态药物-蛋白质靶点相互作用预测方法系统架构图。
具体实施方式
下面结合附图和实施例,对本发明的具体实施方式作进一步详细描述。以下实施例用于说明本发明,但不用来限制本发明的范围。
实施例中使用的术语如下:
SMILES:简化分子线性输入规范
DPI:药物-蛋白质靶点相互作用
RDKit:开源的化学工具包
Transformer:一种基于注意力机制的深度学习模型
实施例一
如图1-2所示,提供了一种多模态药物-蛋白质靶点相互作用预测方法,包括:
S1,对药物分子进行特征编码从而获取药物分子的特征表示;
作为优选的实施方式,所述S1基于图神经网络对分子图进行特征提取与编码获取分子的结构特征,同时利用预训练模型Mol2vec获取分子的语义特征实现,包括:
S11,利用RDKit工具将分子的SMILES序列表示转换为图结构表示,并基于图结构表示得到相应的分子图特征;
S12,将分子图特征作为输入,通过图神经网络编码得到药物分子的结构特征(如图2所示的步骤1.1);
S13,将分子的SMILES序列作为输入,通过预训练Mol2vec模型得到药物分子的语义特征(如图2所示的步骤1.2);
S14,将所述药物分子的结构特征和所述药物分子的语义特征进行整合编码,得到相应的多模态药物分子的特征表示(如图2所示的步骤1.3)。
S2,对蛋白质靶点进行特征编码从而获取蛋白质靶点的特征表示;
作为优选的实施方式,所述S2基于蛋白质靶点的预测氨基酸亲和度矩阵和基于氨基酸序列匹配的特征向量构建蛋白质图,并利用图神经网络和卷积神经网络提取蛋白质靶点的结构特征,同时基于预训练BERT模型以氨基酸序列作为输入获取到蛋白质靶点的语义特征实现。包括:
S21,基于蛋白质靶点的预测氨基酸亲和度矩阵和基于氨基酸序列匹配的特征向量构建蛋白质图,所述蛋白质图的结构特征表征蛋白质中氨基酸残基之间的空间结构关系;
S22,将蛋白图特征作为输入,通过图神经网络和卷积神经网络编码得到蛋白质靶点的结构特征(如图2所示的步骤2.1);
S23,将所述蛋白质靶点的一维氨基酸序列作为输入,通过预训练BERT模型得到蛋白质靶点的语义特征(如图2所示的步骤2.2);
S24,将所述蛋白质靶点的结构特征和所述蛋白质靶点的语义特征进行整合编码,得到相应的多模态蛋白质靶点的特征表示(如图2所示的步骤2.3)。
S3,将所述多模态药物分子的特征表示和所述多模态蛋白质靶点的特征表示进行特征融合获得融合后的相互作用特征;
作为优选的实施方式,基于Transformer解码网络对所述多模态药物分子的特征表示和所述多模态蛋白质靶点的特征表示进行特征融合,利用注意力机制模拟药物分子和靶点蛋白的相互作用得到相互作用特征以进行DPI的预测(如图2所示的步骤3)。包括:
S31,将药物分子的多模态特征M
S32,对交互特征X在节点维度进行聚合获得融合后的相互作用特征:
从而得到可以描述药物-蛋白质靶点相互作用的特征向量h;x
S4,将融合后的相互作用特征作为分类器的输入,基于带标签训练集对预测模型进行训练后获得相互作用预测模型(如图2所示的步骤4),从而基于所述相互作用预测模型预测药物分子和蛋白质靶点之间的相互作用。
作为优选的实施方式,所述S4包括:
S41,将药物-蛋白质靶点相互作用特征向量h输入到包含多个全连接层的DPI分类器中;
S42,定义损失函数L如下:
其中
S43,基于带标签的训练集和预定义的损失函数对所述DPI分类器进行训练得到表现良好的相互作用预测模型。
通过以上步骤即可预测药物分子和蛋白质靶点之间的相互作用,在此基础上利用Transformer中存在的注意力机制可以进一步对药物分子和蛋白质靶点之间的相互作用机制进行定性解释。
实施例二
参见图3,一种多模态药物-蛋白质靶点相互作用预测系统,包括:
第一特征编码模块101,用于对药物分子进行特征编码从而获取药物分子的特征表示;
第二特征编码模块102,用于对蛋白质靶点进行特征编码从而获取蛋白质靶点的特征表示;
特征融合模块103,用于将所述多模态药物分子的特征表示和所述多模态蛋白质靶点的特征表示进行特征融合获得融合后的相互作用特征;
相互作用预测模块104,用于将融合后的相互作用特征作为分类器的输入,基于带标签训练集对预测模型进行训练后获得相互作用预测模型,从而基于所述相互作用预测模型预测药物分子和蛋白质靶点之间的相互作用。
实施例三
提供多模态药物-蛋白质靶点相互作用预测方法在潜在药物分子的搜索中的应用。
对于给定药物-蛋白质靶点相互作用训练集条件下构建DPI预测模型的具体步骤如下:
基于大量的分子SMILES和氨基酸序列数据集,分别对Mol2vec模型和BERT模型进行预训练;
获取药物分子的多模态特征表示。
以药物分子E1E(Cc1cc(nc(n1)SCC(=O)…)为例:
(1)利用RDKit工具包将分子的SMILES序列M转换为由34维节点特征V
(2)将图数据G
(3)将分子序列M输入预训练模型Mol2vec得到维度为300的预训练特征,然后使用全连接层进行编码变换得到300维的药物分子语义特征M
(4)将得到的药物分子结构特征M
获取蛋白质的多模态特征表示,以CA13蛋白受体(MMSRLSWGYREHN…)为例:
基于氨基酸序列P分别计算构成64维节点特征V
将蛋白质图G
将氨基酸序列P输入预训练模型BERT得到维度为768的预训练特征,然后使用全连接层进行特征编码得到256维的蛋白质靶点语义特征P
将蛋白质靶点的结构特征P
药物分子-蛋白质的特征融合:
(1)将药物分子的多模态特征M
(2)对交互特征X在节点维度进行聚合:
从而得到可以描述药物-蛋白质靶点相互作用的特征向量h;
DPI分类器:
(1)将药物-蛋白质靶点相互作用特征向量h输入到包含多个全连接层的分类器中,损失函数L如下:
其中
尽管已描述了本发明的优选实施例,但本领域内的技术人员一旦得知了基本创造性概念,则可对这些实施例作出另外的变更和修改。所以,所附权利要求意欲解释为包括优选实施例以及落入本发明范围的所有变更和修改。显然,本领域的技术人员可以对本发明进行各种改动和变型而不脱离本发明的精神和范围。这样,倘若本发明的这些修改和变型属于本发明权利要求及其等同技术的范围之内,则本发明也意图包含这些改动和变型在内。
