一种用于药敏预测模型样本构建的方法、装置及设备
文献发布时间:2023-06-19 19:30:30
技术领域
本申请涉及机器学习技术领域,尤其涉及一种用于药敏预测模型样本构建的技术。
背景技术
随着人工智能的不断发展,机器学习模型被应用到各个领域。在医学基础临床领域,已有各种基于机器学习模型的预测模型,比如,用于预测药物敏感性的药敏预测模型。
现有基于机器学习模型的药敏预测,通常使用PCA(Principal ComponentAnalysis,主成分分析)分析方法,直接采用基因表达数据或人为选择相关数据作为预测特征的样本,输入基于预先选择的单一机器学习模型训练后得到药敏预测模型,将大量算力集中于模型超参数的选取,获得药敏预测数据。由于使用了单一数学逻辑、相同特征来进行所有药物的敏感性预测,预测准确性不高。
而且现有基于机器学习模型的药敏预测模型往往是选择单一、套用模拟人脑的机器学习模型、或者主观选择一个机器学习模型,没有考虑到贴合实际问题,而且将大量的算力用于模型超参数的选取,容易导致过拟合情况。由于超参数的选取需要大量重复训练,最终获得一个可解释性差的黑盒模型,而在最终获得药敏预测模型的过程中会丢弃多数训练后效果不好的模型,造成训练资源的浪费。
发明内容
本申请的目的是提供一种用于药敏预测模型样本构建的方法、装置及设备,以将基于该样本训练得到的药敏预测模型用于药敏预测,至少部分解决现有技术中基于单一机器学习模型的药敏预测模型的预测准确性不高的技术问题。
根据本申请的一个方面,提供了一种用于药敏预测模型样本构建的方法,其中,所述方法包括:
获取细胞的基因表达数据及其药敏数据;
基于所述细胞的基因表达数据,获得所述细胞在基因集数据库的每个基因集上的得分;
将所述每个基因集上的得分进行归一化处理,获得所述细胞的第一特征数据,并基于所述第一特征数据,获得所述细胞的第二特征数据;
将所述第一特征数据和所述第二特征数据合集处理,得到所述细胞的第三特征数据,并将所述细胞和其对应的第三特征数据、药敏数据作为样本数据,以构建药敏预测模型的样本。
可选地,其中,在获得所述细胞在基因集数据库的每个基因集上的得分前,若细胞在基因集数据库的某个基因集上的某个或者某些基因无对应基因表达数据,则补全所述基因的基因表达数据。
可选地,其中,所述基于所述细胞的基因表达数据,获得所述细胞在基因集数据库的每个基因集上的得分包括:
对所述细胞的基因表达数据,采用单样本基因集富集分析方法,获得所述细胞在基因集数据库的每个基因集上的得分。
可选地,其中,所述基于所述第一特征数据,获得所述细胞的第二特征数据包括:
对所述第一特征数据进行数学处理,获得所述细胞的第二特征数据,其中,所述数学处理包括以下至少一项:
平方运算;
交叉运算;
立方运算;
自然对数运算;
box-cox变换。
可选地,所述一种用于药敏预测模型样本构建的方法还包括:
获得若干不同细胞的样本数据,以构建第一样本数据集,并将所述第一样本数据集划分成第一训练样本数据集和第一测试样本数据集;
基于所述第一训练样本数据集,训练若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第一测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;
将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。
可选地,其中,所述将第一样本数据集划分成第一训练样本数据集和第一测试样本数据集包括:
采用KS检验,将第一样本数据集划分成第一训练样本数据集和第一测试样本数据集。
可选地,其中,所述基于所述第一训练样本数据集,训练若干预设机器学习模型包括:
采用交叉检验和bootstrap采样,从所述第一训练样本数据集中获取训练样本数据,训练若干预设机器学习模型;
重复训练预设次数。
可选地,其中,所述将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型包括:
采用贪心前向选择法,将完成预训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。
可选地,所述一种用于药敏预测模型样本构建的方法还包括:
置换所述第一样本数据集中不同样本的第三特征数据的一个特征的特征值,得到第二样本数据集;
集进行预测,并根据预测结果准确度的变化,得到所述特征的重要性得分;
遍历所述第一样本数据集中样本的第三特征数据的每一个特征,重复上述步骤,得到每个特征的重要性得分;
基于所述第一样本数据集,选择预设数量个重要性得分最高的特征,构建第三样本数据集,其中,所述第三样本数据集中每个样本数据包括细胞和其对应的第三特征数据中对应所述预设数量个重要性得分最高的特征数据。
可选地,所述一种用于药敏预测模型样本构建的方法还包括:
将所述第三样本数据集划分成第三训练样本数据集和第三测试样本数据集;
基于所述第三训练样本数据集,训练所述若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第三测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;
将完成训练的每个预设机器学习模型进行融合,得到第二药敏预测模型。
可选地,所述一种用于药敏预测模型样本构建的方法还包括:
获取待测细胞的基因表达数据,并基于所述待测细胞的基因表达数据,获得所述待测细胞在基因集数据库的每个基因集上的得分;
将所述每个基因集上的得分进行归一化处理,获得所述待测细胞的第一特征数据,并基于所述第一特征数据,获得所述待测细胞的第二特征数据;
将所述第一特征数据和所述第二特征数据合集处理,得到所述待测细胞的第三特征数据;
将所述待测细胞的第三特征数据输入所述第二药敏预测模型,以预测所述待测细胞的药敏数据。
根据本申请的另一个方面,提供了一种用于药敏预测模型样本构建的装置,其中,所述装置包括:
第一模块,用于获取细胞的基因表达数据及其药敏数据;
第二模块,用于基于所述细胞的基因表达数据,获得所述细胞在基因集数据库的每个基因集上的得分;
第三模块,用于将所述每个基因集上的得分进行归一化处理,获得所述细胞的第一特征数据,并基于所述第一特征数据,获得所述细胞的第二特征数据;
第四模块,用于将所述第一特征数据和所述第二特征数据合集处理,得到所述细胞的第三特征数据,并将所述细胞和其对应的第三特征数据、药敏数据作为样本数据,以构建药敏预测模型的样本。
可选地,所述一种用于药敏预测模型样本构建的装置还包括:
第五模块,用于获得若干不同细胞的样本数据,以构建第一样本数据集,并将所述第一样本数据集划分成第一训练样本数据集和第一测试样本数据集;
第六模块,用于基于所述第一训练样本数据集,训练若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第一测试样本数据集进行测试,若MSE误差满足预设阈值时,完成每个预设机器学习模型的训练;
第七模块,用于将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。
可选地,所述一种用于药敏预测模型样本构建的装置还包括:
第八模块,用于置换所述第一样本数据集中不同样本的第三特征数据的一个特征的特征值,得到第二样本数据集;
第九模块,用于采用所述第一药敏预测模型对所述第一样本数据集和所述第二样本数据集进行预测,并根据预测结果准确度的变化,得到所述特征的重要性得分;
第十模块,用于遍历所述第一样本数据集中样本的第三特征数据的每一个特征,重复上述步骤,得到每个特征的重要性得分;
第十一模块,用于基于所述第一样本数据集,选择预设数量个重要性得分最高的特征,构建第三样本数据集,其中,所述第三样本数据集中每个样本数据包括细胞和其对应的第三特征数据中对应所述预设数量个重要性得分最高的特征数据。
可选地,所述一种用于药敏预测模型样本构建的装置还包括:
第十二模块,用于将所述第三样本数据集划分成第三训练样本数据集和第三测试样本数据集;
第十三模块,用于基于所述第三训练样本数据集,训练所述若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第三测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;
第十四模块,用于将完成训练的每个预设机器学习模型进行融合,得到第二药敏预测模型。
可选地,所述一种用于药敏预测模型样本构建的装置还包括:
第十五模块,用于获取待测细胞的基因表达数据,并基于所述待测细胞的基因表达数据,获得所述待测细胞在基因集数据库的每个基因集上的得分;
第十六模块,用于将所述每个基因集上的得分进行归一化处理,获得所述待测细胞的第一特征数据,并基于所述第一特征数据,获得所述待测细胞的第二特征数据;
第十七模块,用于将所述第一特征数据和所述第二特征数据合集处理,得到所述待测细胞的第三特征数据;
第十八模块,用于将所述待测细胞的第三特征数据输入所述第二药敏预测模型,以预测所述待测细胞的药敏数据。
与现有技术相比,本申请提供了一种用于药敏预测模型样本构建的方法、装置及设备。其方法包括:获取细胞的基因表达数据及其药敏数据;基于细胞的基因表达数据,获得该细胞在基因集数据库的每个基因集上的得分;将每个基因集上的得分进行归一化处理,获得该细胞的第一特征数据,并基于该第一特征数据,获得该细胞的第二特征数据;将该第一特征数据和第二特征数据合集处理,得到该细胞的第三特征数据,并将该细胞和其对应的第三特征数据、药敏数据作为样本数据。可选地,其方法还包括:获得若干不同细胞的样本数据,以构建第一样本数据集,并将所述第一样本数据集划分成第一训练样本数据集和第一测试样本数据集;基于第一训练样本数据集,训练若干预设机器学习模型;当所述预设机器模型的MSE误差满足预设阈值时,基于第一测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。可选地,其方法还包括:置换第一样本数据集中不同样本的第三特征数据的一个特征的特征值,得到第二样本数据集;采用第一药敏预测模型对第一样本数据集和第二样本数据集进行预测,并根据预测结果准确度的变化,得到该特征的重要性得分;遍历第一样本数据集中样本的第三特征数据的每一个特征,重复上述步骤,得到每个特征的重要性得分;基于第一样本数据集,选择预设数量个重要性得分最高的特征,构建第三样本数据集,其中,第三样本数据集中每个样本数据包括细胞和其对应的第三特征数据中对应该预设数量个重要性得分最高的特征数据。可选地,其方法还包括:将第三样本数据集划分成第三训练样本数据集和第三测试样本数据集;基于第三训练样本数据集,训练上述若干预设机器学习模型;当预设机器模型的MSE误差满足预设阈值时,基于第三测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;将完成训练的每个预设机器学习模型进行融合,得到第二药敏预测模型。
本申请提供的一种用于药敏预测模型样本构建的方法、装置及设备可带来的技术效果:
将基于细胞原始的基因表达数据转化为具有生物学意义的基因集得分,并进行数学处理,得到非单一的特征数据,使得包括此特征数据的样本可体现药物的不同的作用机理的关联性。可选地,采用前述样本构建的样本数据集训练得到的若干机器学习模型,经融合得到药敏预测模型,可提升训练效率及预测准确度,还可根据需要调整参与融合的机器学习模型的数量,提供了可扩展性和可适用性。可选地,通过特征置换,可确定不同特征的重要性,选择重要性高的若干特征构建新的样本数据集,训练得到新的若干机器学习模型,经融合得到新的药敏预测模型,可降低训练数据量、提升训练效率,且不降低预测准确度。
附图说明
通过阅读参照以下附图所作的对非限制性实施例所作的详细描述,本申请的其它特征、目的和优点将会变得更明显:
图1示出根据本申请一个方面的一种用于药敏预测模型样本构建的方法流程图;
图2示出根据本申请一个方面的一种用于药敏预测模型构建的方法流程图;
图3示出根据本申请另一个方面的一种用于药敏预测模型样本数据集构建的方法流程图;
图4示出根据本申请另一个方面的一种用于药敏预测模型构建的方法流程图;
图5示出根据本申请的又一方面的一种药敏预测的方法流程图;
图6示出根据本申请的再一个方面的一种用于药敏预测模型样本构建的装置示意图;
附图中相同或相似的附图标记代表相同或相似的部件。
具体实施方式
下面结合附图对本申请作进一步详细描述。
在本申请的各实施例的一个典型的配置中,方法的执行主体、系统各可信方和/或装置各模块均可以包括一个或多个处理器(CPU)、输入/输出接口、网络接口和内存。
内存可能包括计算机可读介质中的非永久性存储器,随机存取存储器(RAM)和/或非易失性内存等形式,如只读存储器(ROM)或闪存(flash RAM)。内存是计算机可读介质的示例。
计算机可读介质包括永久性和非永久性、可移动和非可移动媒体可以由任何方法或技术来实现信息存储。信息可以是计算机可读指令、数据结构、程序的模块或其他数据。计算机的存储介质的例子包括,但不限于相变内存(PRAM)、静态随机存取存储器(SRAM)、动态随机存取存储器(DRAM)、其他类型的随机存取存储器(RAM)、只读存储器(ROM)、电可擦除可编程只读存储器(EEPROM)、快闪记忆体或其他内存技术、只读光盘只读存储器(CD-ROM)、数字多功能光盘(DVD)或其他光学存储、磁盒式磁带,磁带磁盘存储或其他磁性存储设备或任何其他非传输介质,可用于存储可以被计算设备访问的信息。按照本文中的界定,计算机可读介质不包括非暂存电脑可读媒体(transitory media),如调制的数据信号和载波。
为更进一步阐述本申请所采取的技术手段及取得的效果,下面结合附图及各实施例,对本申请的技术方案,进行清楚和完整的描述。
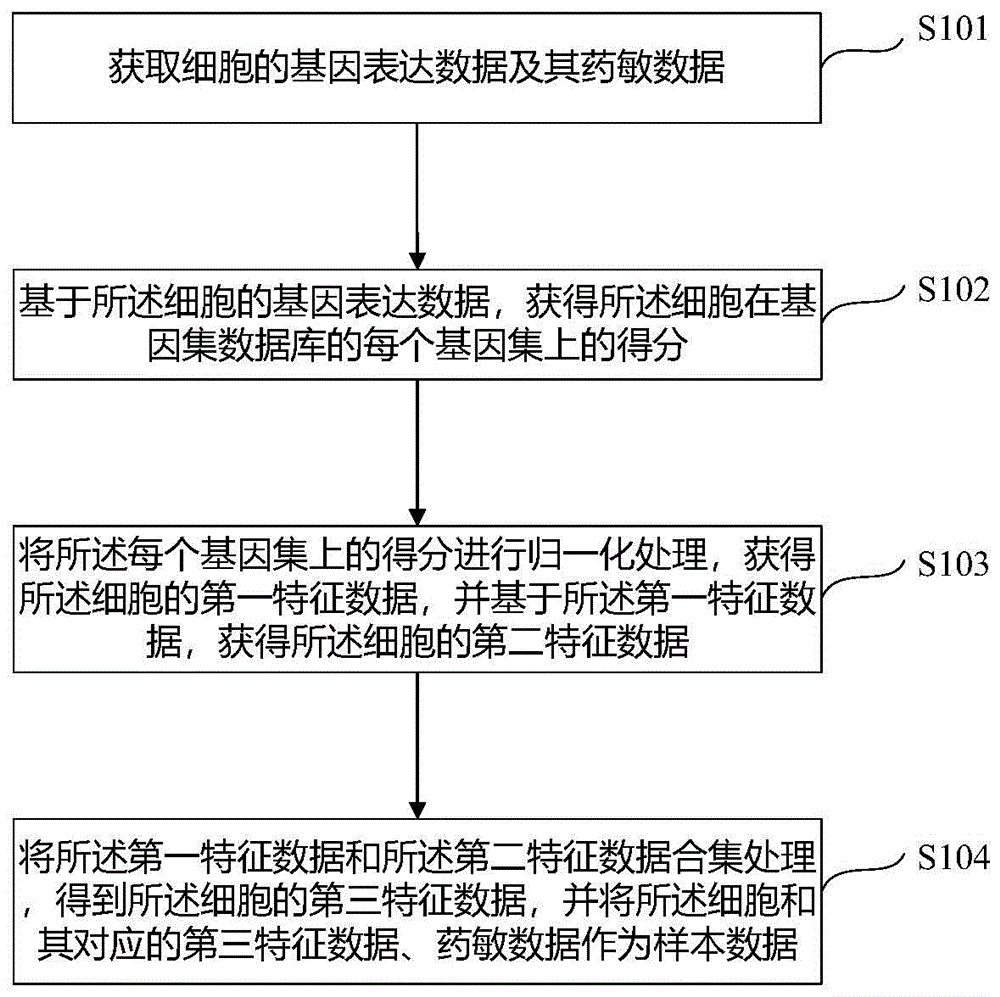
图1示出根据本申请一个方面的一种用于药敏预测模型样本构建的方法,其中,一个实施例的方法包括:
S101获取细胞的基因表达数据及其药敏数据;
S102基于所述细胞的基因表达数据,获得所述细胞在基因集数据库的每个基因集上的得分;
S103将所述每个基因集上的得分进行归一化处理,获得所述细胞的第一特征数据,并基于所述第一特征数据,获得所述细胞的第二特征数据;
S104将所述第一特征数据和所述第二特征数据合集处理,得到所述细胞的第三特征数据,并将所述细胞和其对应的第三特征数据、药敏数据作为样本数据。
其中,本申请的方法实施例可通过设备100实施,所述设备100为安装有相关软硬件、具有一定算力的计算机设备和/或云。其中,所述计算机设备包括但不限于个人计算机、笔记本电脑、工业计算机、网络主机、单个网络服务器或多个网络服务器集;所述云由基于云计算(Cloud Computing)的大量计算机或网络服务器构成,其中,云计算是分布式计算的一种,由一群松散耦合的计算机集组成的一个虚拟超级计算机。
在此,所述计算机和/或云仅为举例,其他现有的或者今后可能出现的设备和/或资源共享平台如适用于本申请也应包含在本申请的保护范围内,在此,以引用的方式包含于此。
在该实施例中,在步骤S101中,设备100获取到某一种细胞的基因表达数据及其药敏数据。
其中,某一种细胞的基因表达数据可采用公开数据集中的基因表达数据,例如,可采用CCLE(Cancer Cell Line Encyclopedia,癌细胞系百科全书)数据库或者CellMiner数据库中的该细胞的基因表达数据。CCLE数据库是由隶属于美国麻省理工学院和哈佛大学的博德研究所(Broad Institute)牵头的一项肿瘤基因组学研究项目,涵盖了三十多种组织来源上千种细胞系基因表达情况、突变、拷贝数、甲基化等。CellMiner数据库主要是以美国国家癌症研究所癌症研究中心列出的60种癌细胞为基础而建立基因表达数据库,其中包含了转录谱测序获得的基因表达数据。
其中,该细胞的药敏数据可采用CTRP(Cancer Therapeutics Response Portal,癌症治疗反应门户)数据库或者GDSC(Genomics of Drug Sensitivity in Cancer,癌症药物敏感性基因组学)数据库等公开的药敏数据库中该细胞的药敏数据。CTRP数据库是将有癌细胞系的遗传、谱系和其他细胞特征与小分子敏感性联系起来,目的是加速发现与患者匹配的癌症治疗分子药物。GDSC数据库是由英国剑桥大学维康桑格(Wellcome Sanger)研究所开发的,其中收集了肿瘤细胞对药物的敏感度和反应。这些公开数据集中包含了多种肿瘤细胞对化合物、靶向药的药敏实验数据。
其中,也可在实验中获得某一种细胞,对该细胞进行转录谱测序,以获得该细胞的基因表达数据;进行药敏实验,以获得该细胞的药敏数据。
其中,若不能同时得到某一种细胞的基因表达数据和其对应的药敏数据,则不选择该细胞。
继续在该实施例中,在步骤S102中,设备100基于该细胞的基因表达数据,获得该细胞在基因集数据库的每个基因集上的得分。
其中,设备100根据该细胞的基因表达数据,与基因集数据库中的每个基因集进行基因数据对照,获得该细胞在基因集数据库的每个基因集上的得分。
示例性的,可根据该细胞的基因表达数据,与MsigDB(Molecular SignaturesDatabase,分子特征数据库)数据库中的每个基因集进行基因数据对照,获得该细胞在MsigDB数据库的每个基因集上的得分。其中,MsigDB数据库当前最主流的公开基因集数据库之一,汇总了许多常用的其他基因集数据库的数据,而且持续更新完善,并对公众免费开放使用。目前MsigDB数据库的2022年版本共收录了9个主要集合:C1~C8,外加一个Hallmark集合,共有33000+个基因集,这些基因集具有良好注释、含有生物学意义。
也可获取该细胞在其他基因集数据库中的每个基因集上的得分,其他基因集数据库可以是其他比较有名的基因集数据库,比如,KEGG数据库、Reactome数据库、Wikipath数据库、GO数据库等。这些基因集数据库中的数据也被部分收录在MsigDB数据库中,例如,KEGG数据库、Reactome数据库、Wikipath数据库分别为MsigDB数据库C2基因集合的一部分,GO数据库收录在MsigDB数据库C5基因集合中。其中,KEGG数据库、Reactome数据库、Wikipath数据库中的数据侧重点在信号通路,而GO数据库包含了3个子类:分子功能、生物学过程及细胞组件。
在此,不限定具体基因集数据库,所述MsigDB数据库或其他基因集数据库等仅为举例,其他现有的或者今后可能出现的基因集数据库如适用于本申请也应包含在本申请的保护范围内,在此,以引用的方式包含于此。
由于组成细胞的基因不可能包括所有已知基因,某一种细胞包括的基因可能不包含基因集数据库的某个基因集上的一个或多个基因。为了使得该细胞可在基因集数据库的每个基因集上都能获得得分,可选地,在步骤S102中,在获得所述细胞在基因集数据库的每个基因集上的得分前,若该细胞在基因集数据库的某个基因集上的某个或者某些基因无对应基因表达数据,则在该细胞的基因表达数据中补全缺失基因的基因表达数据。
其中,在将该细胞的基因表达数据与基因集数据库中的每个基因集进行基因数据进行对照时,如果该细胞的基因表达数据中不包含某个基因集上的某个或者某些基因的数据,则在该细胞的基因表达数据中补全这个或这些基因的数据,以得到该细胞的结构完整的基因表达数据,另外,还有助于基于后续步骤构建的多样性的样本数据,用于机器学习模型的训练。其中,考虑到缺失数据往往是表达量过少导致的,可以将该细胞的基因表达数据中补全的这个或这些基因的数据设置为0,以进行补全。
可选地,其中,所述步骤S102包括:
对所述细胞的基因表达数据,采用单样本基因集富集分析方法,获得所述细胞在基因集数据库的每个基因集上的得分。
其中,ssGSEA(single sample Gene Set Enrichment Analysis,单样本基因集富集分析)方法是以某一种细胞的基因表达数据为样本进行差异基因分析的一种差异基因分析方法。差异基因分析通常会获得大量基因,但海量的基因不好系统分析、找到相似规律,需要进行注释并分析与哪些与疾病相关的通路相关,以便进一步挖掘有用信息。一般的差异基因分析(例如基于GO数据库或基于Pathway通路级别)侧重于比较两组间的基因表达差异,集中关注少数几个显著上调或下调的基因,对于差异基因检出的阈值,异常的敏感,需要客户给出差异基因的一个明确的定义(阈值),例如abs(logFC)≧2.0&FDR≦0.05,这容易遗漏部分差异表达不显著却有重要生物学意义的基因,忽略了一些基因的生物特性、基因调控网络之间的关系以及基因功能和意义等有价值的信息。而采用ssGSEA方法进行差异基因分析,不需要指定明确的差异基因阈值,而是根据实际数据的整体趋势,即使在没有预先定义阈值的情况下也能在表达谱整体层次上对基因进行差异分析,从而从数理统计上把基因表达数据与生物学意义很好地衔接起来,能够更轻松、更合理地解读结果。具体地,根据该细胞的基因表达数据,可以获得其对应基因集数据库的某条已知通路中的基因,对感兴趣的基因进行打分,获得在该条已知通路中的基因集上的得分。
在步骤S102中,示例性的,采用ssGSEA方法对某一种细胞的基因表达数据进行分析,可获得该细胞在MsigDB数据库的每个基因集上的得分。
继续在该实施例中,在步骤S103中,设备100将基因集数据库的每个基因集上的得分进行归一化处理,获得该细胞的第一特征数据,并基于所述第一特征数据,获得所述细胞的第二特征数据。
其中,为了便于后续数据处理,可将获得的基因集数据库的每个基因集上的得分进行归一化处理,得到该细胞的第一特征数据,实现数据的无量纲化,降低具体细胞和/或获取流程对得分的影响。其中,若基因集数据库包含n个基因集,或选用基因集数据库的n个基因集,获取该细胞的基因表达数据在这n个基因集上的得分,则该细胞的第一特征数据可以包括:n个特征,即该细胞的基因表达数据在n个基因集上的归一化得分,每个归一化得分的具体数值即为每个特征的特征值。
例如,采用ssGSEA方法获得ACHN细胞(人肾细胞腺癌细胞)在MsigDB数据库的部分基因集上的得分,对得分值进行归一化处理,得到第一特征数据中各特征的对应特征值如下表1。
表1
注:归一化得分实际上是对各基因集上的得分进行归一化后再加1,以方便后续的数学处理。
可选地,在步骤S103中,所述基于所述第一特征数据,获得所述细胞的第二特征数据包括:
对所述第一特征数据进行数学处理,获得所述细胞的第二特征数据,其中,所述数学处理包括以下至少一项:
平方运算;
交叉运算;
立方运算;
自然对数运算;
box-cox变换。
其中,为了获得便于后续机器学习模型的训练,设备100还对获得的第一特征数据进行数学处理,获得该细胞的第二特征数据,其中,数学处理可以包括以下至少一项:
对第一特征数据进行平方运算,和/或对第一特征数据进行交叉运算,和/或对第一特征数据进行立方运算,和/或对第一特征数据进行自然对数运算,和/或对第一特征数据进行box-cox变换。
例如,对上述表1中的ACHN细胞的第一特征数据各特征值进行平方运算、立方运算、自然对数运算、box-cox变换处理后如下表2。
表2
例如,对上述表1中的ACHN细胞的第一特征数据各特征值进行交叉运算处理后如下表3。
表3
为了便于描述,以上表1~表3中仅列出了6个基因集。
继续在该实施例中,在步骤S104中,设备100将所述第一特征数据和所述第二特征数据合集处理,得到所述细胞的第三特征数据,并将所述细胞和其对应的第三特征数据、药敏数据作为样本数据,以构建药敏预测模型的样本。
其中,设备100将该细胞的第一特征数据和第二特征数据进行汇总合集处理,组成数据集合,得到该细胞的第三特征数据。将该细胞和其对应的第三特征数据、药敏数据作为样本数据,以构建药敏预测模型的样本,用于训练机器学习模型,以得到药敏预测模型。
根据上述方法实施例可获得不同细胞的样本数据,构建样本数据集,可用来训练机器学习模型,以获得药敏预测模型。其中,该样本数据包括:细胞标识,比如细胞的名称;第三特征数据;该细胞对应的药敏数据。示例性的,在采用ssGSEA方法获得的ACHN细胞(人肾细胞腺癌细胞)在MsigDB数据库的每个基因集上的得分基础上得到ACHN细胞的第一特征数据,进行平方运算、立方运算、自然对数运算、box-cox变换以及交叉运算得到ACHN细胞的第二特征数据,再将ACHN细胞的第一特征数据和第二特征数据汇总合集,得到ACHN细胞的第三特征数据(若选用MsigDB数据库的n个基因集的每个基因集上的得分,则第一特征数据包括n个特征,每个特征为每个基因集上的归一化得分,特征值为其对应的得分值。第二特征数据包括(4n+n(n-1)/2)个特征,其中,对应平方运算的n个特征,每个特征的特征值为对第一特征数据中的每个特征的特征值进行平方运算得到的值;对应立方运算的n个特征,每个特征的特征值为对第一特征数据中的每个特征的特征值进行立方运算得到的值;对应自然对数运算的n个特征,每个特征的特征值为对第一特征数据中的每个特征的特征值进行自然对数运算得到的值;对应box-cox变换的n个特征,每个特征的特征值为对第一特征数据中的每个特征的特征值进行box-cox变换得到的值;对应交叉运算的(n(n-1)/2)个特征,每个特征的特征值为对第一特征数据中的相应两个特征的特征值进行运算得到的值。而第三特征数据是第一特征数据与第二特征数据汇总合集,包括(5n+n(n-1)/2)个特征及其特征值。若采用如CTRP数据库或其它公开数据库中的ACHN细胞的AUC(Area Under theCurve,曲线下面积)数据作为药敏数据(CTRP数据库中ACHN细胞的部分药物的AUC药敏数据如下表4),则ACHN细胞对应的样本数据包括:ACHN细胞名称,ACHN细胞的第三特征数据,ACHN细胞的AUC值。
表4
可选地,图2示出根据本申请一个方面的一种用于药敏预测模型构建的方法流程图,其中,该方法包括:
S201获得若干不同细胞的样本数据,以构建第一样本数据集,并将所述第一样本数据集划分成第一训练样本数据集和第一测试样本数据集;
S202基于所述第一训练样本数据集,训练若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第一测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;
S203将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。
其中,该方法实施例也可通过设备100实施。
在该实施例中,在步骤S201中,设备100可获得采用如前述图1所示方法实施例获得的不同细胞的样本数据,包括每个细胞和其对应的第三特征数据、药敏数据,以构建第一样本数据集,并对第一样本数据集中的样本数据进行划分,得到第一训练样本数据集和第一测试样本数据集。比如,按8:2比例对第一样本数据集中的样本数据进行划分,其中80%的样本划分到第一训练样本数据集中,用于机器学习模型的训练,20%的样本划分到第一测试样本数据集中,用于机器学习模型训练效果测试。
可选地,其中,所述将所述第一样本数据集划分成第一训练样本数据集和第一测试样本数据集包括:
采用KS检验,将第一样本数据集划分成第一训练样本数据集和第一测试样本数据集。
其中,可以采用随机划分的方法将第一样本数据集划分成第一训练样本数据集和第一测试样本数据集,划分后进行KS检验,若没有通过,则再次随机划分后再进行KS检验,重复随机划分,直至通过KS检验为止。采用KS检验将第一样本数据集划分成第一训练样本数据集和第一测试样本数据集,以使第一训练样本数据集和第一测试样本数据集中的样本数据服从相同分布,可以确保避免出现数据迁移的问题,保证了样本数据划分的随机性,同时增加了模型效果检查的可靠性。
继续在该实施例中,在步骤S202中,设备100基于所述第一训练样本数据集,训练若干预设机器学习模型,当某个预设机器模型在训练后的MSE误差满足预设阈值时,则设备100采用第一测试样本数据集对该训练后的模型进行测试,若MSE误差满足预设阈值,则完成该预设机器学习模型的训练。遍历每个预设机器模型,直至完成每个预设机器学习模型的训练,得到若干个训练后的机器学习模型。
其中,针对现有药敏预测模型往往是针对单一机器学习模型,使得某些不适合该机器学习模型的特征信息被忽略,导致预测准确性不高。在该实施例中,选择若干已预设超参数组的机器学习模型,采用第一训练样本数据集对每个机器学习模型进行训练,然后采用第一测试样本数据集对训练效果进行测试。
示例性的,所述若干预设机器学习模型可以选择现有经典的、经过检验在某一或某些应用范围内有有益效果的基础机器学习模型,可以包括如下7大类、13个具体的机器学习模型,其中,同一大类模型的不同名字代表了选择的不同的经典超参数组:
1、KNN(K-NearestNeighbor,K最邻近)模型中的KNeighborsUnif和KNeighborsDist
2、LightGBM(light Gradient Boosting Machine,轻梯度提升机)模型中的LightGBM、LightGBMXT和LightGBMLarge
3、随机森林模型中的RandomForestGini和RandomForestEntr
4、ExtraTree(极限树)模型中的ExtraTreesGini,ExtraTreesEntr
5、CatBoost
6、XGBoost
7、人工神经网络中的NeuralNetFastAI和NeuralNetTorch
可选地,在步骤S202中,所述基于所述第一训练样本数据集,训练若干预设机器学习模型包括:
采用交叉检验和bootstrap采样,从所述第一训练样本数据集中获取训练样本数据,训练若干预设机器学习模型;
重复训练预设次数。
其中,在从第一训练样本数据集中获取训练样本数据,训练每个预设机器学习模型时,可采用交叉检验方法。示例性的,采用交叉检验方法,将第一训练样本数据集划分为k个子集,每次采用(k-1)个子集进行训练,在剩下的一个子集上评测。
其中,还可再对(k-1)个子集采用bootstrap采样方法。示例性的,采用bootstrap采样方法,从一个子集中采样训练样本来构建用于机器学习模型的训练样本集,若要抽取m轮,则每轮抽取若干个训练样本数据作为一个训练样本子集(每次抽取后放回,因此,在被抽取的子集中,有些样本数据可能被多次抽取到,而有些样本数据可能一次都没有被抽中),共进行m轮抽取,得到m个相互独立的训练样本子集。每次采用1个训练样本子集来训练预设机器学习模型,可得到该机器学习模型的一个训练模型,m个训练样本数据子集共得到该机器学习模型的m个训练模型。计算这m个训练模型的均值作为该机器学习模型的训练后的模型。
在预设机器学习模型的训练过程中,还可以重复预设次数,采用交叉检验和bootstrap采样方法,向着MSE(Mean Square Error,均方误差)递减的方向训练,或者重复训练,直到MSE不再降低。
示例性的,采用交叉检验和bootstrap采样,从所述第一训练样本数据集中获取训练样本数据,训练上述7大类的13个预设机器学习模型,重复训练预设次数,当训练后的每个预设机器学习模型的MSE满足预设阈值,且经过采用第一测试样本数据集对训练后的每个预设机器学习模型测试通过后,则完成上述7大类的13个预设机器学习模型的训练,得到13个训练后的机器学习模型。
继续在该实施例中,在步骤S203中,设备100将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。
其中,得到的每个训练后的机器学习模型可能对不同药物的药敏程度不同,基于单个训练后的机器学习模型,对不同药物药敏预测准确度不高,可将该若干个训练后的机器学习模型融合成一个模型,针对不同药物的预测,融合模型中各个机器学习模型的权重(贡献度)可能不同,以提升融合后的模型的药敏预测准确度。在融合过程中,若某个机器学习模型对融合后的模型没有贡献,则其权重为0。
可选地,在步骤S203中,所述将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型包括:
采用贪心前向选择法,将完成预训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。
其中,采用贪心前向选择法,将完成训练的每个预设机器学习模型进行线性融合,确定每个训练后的机器学习模型的权重,得到融合后的第一药敏预测模型。
样本数据中采用的特征数据若过多,往往会占用设备100过多的硬盘、内存等资源,而且模型的训练时间也可能会过长,还可能会出现过拟合,训练后的融合模型的药敏预测准确率并没有显著提高,甚至会因为低质量的特征数据导致药敏预测准确率下降,因此,其实际应用价值可能并不高。在药敏预测中,往往是少量基因相关特征的贡献更大,可考虑对样本数据中的特征数据进行筛选,将最重要的基因相关特征数据筛选出来,以最重要的特征数据构建样本数据。
可选地,图3示出根据本申请另一个方面的一种用于药敏预测模型样本数据集构建的方法流程图,其中,该方法包括:
S301置换所述第一样本数据集中不同样本的第三特征数据的一个特征的特征值,得到第二样本数据集;
S302采用所述第一药敏预测模型对所述第一样本数据集和所述第二样本数据集进行预测,并根据预测结果准确度的变化,得到所述特征的重要性得分;
S303遍历所述第一样本数据集中样本的第三特征数据的每一个特征,重复上述步骤,得到每个特征的重要性得分;
S304基于所述第一样本数据集,选择预设数量个重要性得分最高的特征,构建第三样本数据集,其中,所述第三样本数据集中每个样本数据包括细胞和其对应的第三特征数据中对应所述预设数量个重要性得分最高的特征数据。
其中,该方法实施例也可通过设备100实施。通过随机打乱样本数据中某个特征在样本数据中的位置,从而使得每个样本数据中该特征对应的特征数据发生变化,其它特征数据不变,从而可通过比较第一药敏预测模型对打乱后的样本数据集的预测结果与对原样本数据集的预测结果,得到预测结果准确度的变化,可确定该特征的重要程度。
在该实施例中,在步骤S301中,设备100可置换如前述图2所示方法实施例得到的第一样本数据集中不同样本的第三特征数据的一个特征的特征值,得到第二样本数据集。
其中,可以是随机置换第一样本数据集中每个样本的第三特征数据的一个特征的特征值,得到第二样本数据集。
继续在该实施例中,在步骤S302中,设备100采用如前述图2所示方法实施例得到的第一药敏预测模型对所述第一样本数据集和所述第二样本数据集进行预测,并根据预测结果准确度的变化,得到所述特征的重要性得分。
其中,设备100采用第一药敏预测模型,对如前述图2所示方法实施例得到的第一样本数据集中的每个细胞的第三特征数据进行预测,并将预测结果与对应的药敏数据进行比较,得到每个细胞的药敏预测偏差,计算所有细胞的药敏预测偏差的MSE,作为第一样本数据集的预测结果准确度。设备100还采用第一药敏预测模型,对得到的第二样本数据集中的每个细胞的第三特征数据进行预测,并将预测结果与对应的药敏数据进行比较,得到每个细胞的药敏预测偏差,计算所有细胞的药敏预测偏差的MSE,作为第二样本数据集的预测结果准确度。比较第一样本数据集和第二样本数据集的预测结果准确度,将两者的差值作为该特征的重要性得分。
继续在该实施例中,在步骤S303中,设备100遍历第一样本数据集中样本的第三特征数据的每一个特征,重复步骤S301和S302,可得到第一样本数据集的样本中的第三特征数据的每个特征的重要性得分。
继续在该实施例中,在步骤S304中,根据步骤S303中得到的第一样本数据集的样本中的第三特征数据的每个特征的重要性得分,选择预设数量个重要性得分最高的特征,保留第三特征数据中这预设数量个特征及其数据,删除每个样本中的第三特征数据的其它特征及其数据,得到每个细胞的新的特征数据,将细胞和其对应的新的特征数据以及其对应的药敏数据作为新的样本数据,与第一样本数据集的样本数量相同,构建第三样本数据集,其中,所述第三样本数据集中每个样本数据包括细胞和其对应的第三特征数据中对应所述预设数量个重要性得分最高的特征数据,以及对应的药敏数据。
采用第三样本数据集训练如前述图2所示方法实施例的若干预设机器学习模型,可获得预测准确度达到预期,且资源开销可接受的药敏预测模型,具有实际应用价值。
可选地,图4示出根据本申请另一个方面的一种用于药敏预测模型构建的方法流程图,其中,该方法包括:
S401将所述第三样本数据集划分成第三训练样本数据集和第三测试样本数据集;
S402基于所述第三训练样本数据集,训练所述若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第三测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;
S403将完成训练的每个预设机器学习模型进行融合,得到第二药敏预测模型。
其中,该方法实施例也可通过设备100实施。在该方法实施例中,步骤S401~S403的操作实施与如前述图2所示方法实施例和/或可选实施例中的步骤S201~S203中的相应操作类似或相同,在此不再赘述。通过该方法实施例,可得到第二药敏预测模型。
可将得到的第二药敏预测模型用于待测细胞的药敏预测。
可选地,图5示出根据本申请的又一方面的一种药敏预测的方法流程图,其中,该方法包括:
S501获取待测细胞的基因表达数据,并基于所述待测细胞的基因表达数据,获得所述待测细胞在基因集数据库的每个基因集上的得分;
S502将所述每个基因集上的得分进行归一化处理,获得所述待测细胞的第一特征数据,并基于所述第一特征数据,获得所述待测细胞的第二特征数据;
S503将所述第一特征数据和所述第二特征数据合集处理,得到所述待测细胞的第三特征数据;
S504将所述待测细胞的第三特征数据输入所述第二药敏预测模型,以预测所述待测细胞的药敏数据。
其中,该方法实施例也可通过设备100实施。
在该实施例中,在步骤S501中,设备100获取待测细胞的基因表达数据,并基于所述待测细胞的基因表达数据,获得所述待测细胞在基因集数据库的每个基因集上的得分。
其中,待测细胞可能为生物实验中不同培养状态下的细胞系,或来源于患者的细胞,患者细胞可通过手术、活检等临床手段获取,该待测细胞收集后进行转录谱测序,得到该待测细胞的基因表达数据。设备100根据该待测细胞的基因表达数据,与基因集数据库中的每个基因集进行基因数据对照,获得该待测细胞在基因集数据库的每个基因集上的得分。示例性的,可根据该待测细胞的基因表达数据,采用ssGSEA方法,与MsigDB数据库中的每个基因集进行基因数据对照,获得该待测细胞在MsigDB数据库的每个基因集上的得分。在将该待测细胞的基因表达数据与MsigDB数据库中的每个基因集进行基因数据进行对照时,如果该待测细胞的基因表达数据中不包含某个基因集上的某个或者某些基因的数据,则在该待测细胞的基因表达数据中补全这个或这些基因的数据,以得到该待测细胞的结构完整的基因表达数据。
继续在该实施例中,在步骤S502中,设备100将该待测细胞对应的基因集数据库的每个基因集上的得分进行归一化处理,获得该待测细胞的第一特征数据,并基于所述第一特征数据,获得所述待测细胞的第二特征数据。
其中,为了便于后续数据处理,将该待测细胞对应的基因集数据库的每个基因集上的得分进行归一化处理,得到该待测细胞的第一特征数据。示例性的,若基因集数据库有n个基因集,或选用基因集数据库的n个基因集,获取该待测细胞的基因表达数据在这n个基因集上的得分,则待测细胞的第一特征数据可以包括:n个特征,即待测细胞的基因表达数据在n个基因集上的归一化得分,每个归一化得分的具体数值即为每个特征的特征值。还可对该待测细胞的第一特征数据进行数学处理,获得该待测细胞的第二特征数据。示例性的,该数学处理可以包括以下至少一项:平方运算、交叉运算、立方运算、自然对数运算和box-cox变换。
继续在该实施例中,在步骤S503中,设备100将该待测细胞的第一特征数据和第二特征数据合集处理,得到该待测细胞的第三特征数据。
其中,设备100将该待测细胞的第一特征数据和第二特征数据进行汇总合集处理,组成数据集合,得到该待测细胞的第三特征数据。
继续在该实施例中,在步骤S504中,设备100将该待测细胞的第三特征数据输入如前述图4所示方法实施例的第二药敏预测模型,以预测该待测细胞的药敏数据。
其中,可将得到的该待测细胞的第三特征数据输入如前述图4所示方法实施例的第二药敏预测模型,以预测该待测细胞的药敏数据。还可以先从该待测细胞的第三特征数据中筛选出与前述图3所示方法实施例中相同的预设数量个重要性得分最高的特征数据,然后将这预设数量个重要性得分最高的特征数据输入如前述图4所示方法实施例的第二药敏预测模型,以预测该待测细胞的药敏数据。
图6示出根据本申请再一个方面的一种用于药敏预测模型样本构建的装置示意图,其中,一个实施例的装置包括:
第一模块601,用于获取细胞的基因表达数据及其药敏数据;
第二模块602,用于基于所述细胞的基因表达数据,获得所述细胞在基因集数据库的每个基因集上的得分;
第三模块603,用于将所述每个基因集上的得分进行归一化处理,获得所述细胞的第一特征数据,并基于所述第一特征数据,获得所述细胞的第二特征数据;
第四模块604,用于将所述第一特征数据和所述第二特征数据合集处理,得到所述细胞的第三特征数据,并将所述细胞和其对应的第三特征数据、药敏数据作为样本数据,以构建药敏预测模型的样本。
其中,所述装置部署在实施上述方法实施例和/或可选实施例的设备100中,所述装置可以是软件装置或软硬组合装置。
在该实施例中,该装置的第一模块601获取到某一种细胞的基因表达数据及其药敏数据。其中,某一种细胞的基因表达数据可采用公开数据集中的基因表达数据,例如,可采用CCLE数据库或者CellMiner数据库中的该细胞的基因表达数据。该细胞的药敏数据可采用CTRP数据库或者GDSC数据库等公开的药敏数据库中该细胞的药敏数据。其中,也可在实验中获得某一种细胞,通过临床实验,对该细胞进行转录谱测序,以获得该细胞的基因表达数据;进行药敏实验,以获得该细胞的药敏数据。其中,若不能同时得到某一种细胞的基因表达数据和其对应的药敏数据,则不选择该细胞。
继续在该实施例中,该装置的第二模块602根据该细胞的基因表达数据,与基因集数据库中的每个基因集进行基因数据对照,获得该细胞在基因集数据库的每个基因集上的得分。示例性的,可根据该细胞的基因表达数据,采用ssGSEA方法,与MsigDB数据库中的每个基因集进行基因数据对照,获得该细胞在MsigDB数据库的每个基因集上的得分。
继续在该实施例中,该装置的第三模块603将该细胞在基因集数据库的每个基因集上的得分进行归一化处理,获得该细胞的第一特征数据,并基于所述第一特征数据,获得所述细胞的第二特征数据。
其中,为了便于后续数据处理,可将获得的基因集数据库的每个基因集上的得分进行归一化处理,得到该细胞的第一特征数据,实现数据的无量纲化,降低具体细胞和/或获取流程对得分的影响。示例性的,若基因集数据库包含n个基因集,或选用基因集数据库的n个基因集,获取该细胞的基因表达数据在这n个基因集上的得分,则该细胞的第一特征数据可以包括:n个特征,即该细胞的基因表达数据在n个基因集上的归一化得分,每个归一化得分的具体数值即为每个特征的特征值。其中,还可对该待测细胞的第一特征数据进行数学处理,获得该待测细胞的第二特征数据。示例性的,该数学处理可以包括以下至少一项:平方运算、交叉运算、立方运算、自然对数运算和box-cox变换。
继续在该实施例中,该装置的第四模块604将所述第一特征数据和所述第二特征数据合集处理,得到所述细胞的第三特征数据,并将所述细胞和其对应的第三特征数据、药敏数据作为样本数据,以构建药敏预测模型的样本。其中,第四模块604将该细胞的第一特征数据和第二特征数据进行汇总合集处理,组成数据集合,得到该细胞的第三特征数据。将该细胞和其对应的第三特征数据、药敏数据作为样本数据,以构建药敏预测模型的样本,用于训练机器学习模型,以得到药敏预测模型。
通过该装置,可获得不同细胞的样本数据,构建样本数据集,可用来训练机器学习模型,以获得药敏预测模型。其中,该样本数据包括:细胞标识,比如细胞的名称;第三特征数据;该细胞对应的药敏数据。
可选地,该装置还包括:
第五模块,用于获得若干不同细胞的样本数据,以构建第一样本数据集,并将所述第一样本数据集划分成第一训练样本数据集和第一测试样本数据集;
第六模块,用于基于所述第一训练样本数据集,训练若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第一测试样本数据集进行测试,若MSE误差满足预设阈值时,完成每个预设机器学习模型的训练;
第七模块,用于将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。
其中,该装置的第五模块可获得采用采用如前述图1方法实施例获得的不同细胞的样本数据,包括每个细胞和其对应的第三特征数据、药敏数据,以构建第一样本数据集,并对第一样本数据集中的样本数据进行划分,得到第一训练样本数据集和第一测试样本数据集。
其中,针对现有药敏预测模型往往是针对单一机器学习模型,使得某些不适合该机器学习模型的特征信息被忽略,导致预测准确性不高,该装置的第六模块用第一训练样本数据集中的训练样本训练若干已预设超参数组的机器学习模型,当某个预设机器模型在训练后的MSE误差满足预设阈值时,采用第一测试样本数据集对该训练后的模型进行测试,若MSE误差满足预设阈值,则完成该预设机器学习模型的训练。遍历每个预设机器模型,直至完成每个预设机器学习模型的训练,得到若干个训练后的机器学习模型。示例性的,所述若干预设机器学习模型可以选择现有经典的、经过检验在某一或某些应用范围内有有益效果的基础机器学习模型,可以包括如下7大类、13个具体的机器学习模型,其中,同一大类模型的不同名字代表了选择的不同的经典超参数组:
1、KNN(K-NearestNeighbor,K最邻近)模型中的KNeighborsUnif和KNeighborsDist
2、LightGBM(light Gradient Boosting Machine,轻梯度提升机)模型中的LightGBM、LightGBMXT和LightGBMLarge
3、随机森林模型中的RandomForestGini和RandomForestEntr
4、ExtraTree(极限树)模型中的ExtraTreesGini,ExtraTreesEntr
5、CatBoost
6、XGBoost
7、人工神经网络中的NeuralNetFastAI和NeuralNetTorch
示例性的,通过第六模块,采用交叉检验和bootstrap采样,从第一训练样本数据集中获取训练样本数据,训练上述7大类的13个预设机器学习模型,重复训练预设次数,当训练后的每个预设机器学习模型的MSE满足预设阈值,且经过采用第一测试样本数据集对训练后的每个预设机器学习模型测试通过后,则完成上述7大类的13个预设机器学习模型的训练,得到13个训练后的机器学习模型。
其中,该装置的第七模块将完成训练的每个预设机器学习模型进行融合,得到第一药敏预测模型。示例性的,采用贪心前向选择法,将完成训练的每个预设机器学习模型进行线性融合,确定每个训练后的机器学习模型在融合后的第一药敏预测模型中的权重。
样本数据中采用的特征数据若过多,往往会占用设备100过多的硬盘、内存等资源,而且模型的训练时间也可能会过长,还可能会出现过拟合,训练后的融合模型的药敏预测准确率并没有显著提高,因此,其实际应用价值可能并不高。在药敏预测中,往往是少量基因相关特征的贡献度,可考虑对样本数据中的特征数据进行筛选,将最重要的基因相关特征数据筛选出来,以最重要的特征数据构建样本数据。
可选地,该装置还包括:
第八模块,用于置换所述第一样本数据集中不同样本的第三特征数据的一个特征的特征值,得到第二样本数据集;
第九模块,用于采用所述第一药敏预测模型对所述第一样本数据集和所述第二样本数据集进行预测,并根据预测结果准确度的变化,得到所述特征的重要性得分;
第十模块,用于遍历所述第一样本数据集中样本的第三特征数据的每一个特征,重复上述步骤,得到每个特征的重要性得分;
第十一模块,用于基于所述第一样本数据集,选择预设数量个重要性得分最高的特征,构建第三样本数据集,其中,所述第三样本数据集中每个样本数据包括细胞和其对应的第三特征数据中对应所述预设数量个重要性得分最高的特征数据。
其中,该装置的第八模块可置换如前述图2所示方法实施例得到的第一样本数据集中不同样本的第三特征数据的一个特征的特征值,得到第二样本数据集。其中,可以是随机置换第一样本数据集中每个样本的第三特征数据的一个特征的特征值,得到第二样本数据集。
其中,该装置的第九模块可采用如前述图2所示实施例得到的第一药敏预测模型对所述第一样本数据集和所述第二样本数据集进行预测,并根据预测结果准确度的变化,得到所述特征的重要性得分。其中,采用第一药敏预测模型,对如前述图2所示方法实施例得到的第一样本数据集中的每个细胞的第三特征数据进行预测,并将预测结果与对应的药敏数据进行比较,得到每个细胞的药敏预测偏差,计算所有细胞的药敏预测偏差的MSE,作为第一样本数据集的预测结果准确度。设备100还采用第一药敏预测模型,对得到的第二样本数据集中的每个细胞的第三特征数据进行预测,并将预测结果与对应的药敏数据进行比较,得到每个细胞的药敏预测偏差,计算所有细胞的药敏预测偏差的MSE,作为第二样本数据集的预测结果准确度。比较第一样本数据集和第二样本数据集的预测结果准确度,将两者的差值作为该特征的重要性得分。
其中,该装置的第十模块遍历第一样本数据集中样本的第三特征数据的每一个特征,重复通过第八模块和第九模块的操作步骤,可得到第一样本数据集的样本中的第三特征数据的每个特征的重要性得分。
其中,该装置的第十一模块根据第十模块得到的第一样本数据集的样本中的第三特征数据的每个特征的重要性得分,选择预设数量个重要性得分最高的特征,保留第三特征数据中这预设数量个特征及其数据,删除每个样本中的第三特征数据的其它特征及其数据,得到每个细胞的新的特征数据,将细胞和其对应的新的特征数据以及其对应的药敏数据作为新的样本数据,与第一样本数据集的样本数量相同,构建第三样本数据集,其中,所述第三样本数据集中每个样本数据包括细胞和其对应的第三特征数据中对应所述预设数量个重要性得分最高的特征数据,以及对应的药敏数据。
可选地,该装置还包括:
第十二模块,用于将所述第三样本数据集划分成第三训练样本数据集和第三测试样本数据集;
第十三模块,用于基于所述第三训练样本数据集,训练所述若干预设机器学习模型,当所述预设机器模型的MSE误差满足预设阈值时,基于所述第三测试样本数据集进行测试,若MSE误差满足预设阈值,完成每个预设机器学习模型的训练;
第十四模块,用于将完成训练的每个预设机器学习模型进行融合,得到第二药敏预测模型。
其中,该装置的第十二模块对第三样本数据集中的样本数据进行划分,得到第三训练样本数据集和第三测试样本数据集。示例性的,可以采用随机划分的方法将第三样本数据集划分成第三训练样本数据集和第三测试样本数据集,划分后进行KS检验,若没有通过,则再次随机划分后再进行KS检验,重复随机划分,直至通过KS检验为止。
其中,该装置的第十三模块采用第三训练样本数据集,训练若干预设机器学习模型,当某个预设机器模型在某一轮次训练后的MSE误差满足预设阈值时,则采用第三测试样本数据集对该训练后的模型进行测试,若MSE误差满足预设阈值,则完成该预设机器学习模型的训练。遍历每个预设机器模型,直至完成每个预设机器学习模型的训练,得到若干个训练后的机器学习模型。示例性的,所述若干预设机器学习模型可以选择现有经典的、经过检验在某一或某些应用范围内有有益效果的基础机器学习模型,可以包括如下7大类、13个具体的机器学习模型,其中,同一大类模型的不同名字代表了选择的不同的经典超参数组:
1、KNN(K-NearestNeighbor,K最邻近)模型中的KNeighborsUnif和KNeighborsDist
2、LightGBM(light Gradient Boosting Machine,轻梯度提升机)模型中的LightGBM、LightGBMXT和LightGBMLarge
3、随机森林模型中的RandomForestGini和RandomForestEntr
4、ExtraTree(极限树)模型中的ExtraTreesGini,ExtraTreesEntr
5、CatBoost
6、XGBoost
7、人工神经网络中的NeuralNetFastAI和NeuralNetTorch
示例性的,通过第十三模块,采用交叉检验和bootstrap采样,从第三训练样本数据集中获取训练样本数据,训练上述7大类的13个预设机器学习模型,重复训练预设次数,当训练后的每个预设机器学习模型的MSE满足预设阈值,且经过采用第三测试样本数据集对训练后的每个预设机器学习模型测试通过后,则完成上述7大类的13个预设机器学习模型的训练,得到13个训练后的机器学习模型。
其中,该装置的第十四模块将完成训练的每个预设机器学习模型进行融合,得到第二药敏预测模型。示例性的,可采用贪心前向选择法,将完成训练的每个预设机器学习模型进行线性融合,确定每个训练后的机器学习模型的权重,得到融合后的第二药敏预测模型。
可选地,该装置还包括:
第十五模块,用于获取待测细胞的基因表达数据,并基于所述待测细胞的基因表达数据,获得所述待测细胞在基因集数据库的每个基因集上的得分;
第十六模块,用于将所述每个基因集上的得分进行归一化处理,获得所述待测细胞的第一特征数据,并基于所述第一特征数据,获得所述待测细胞的第二特征数据;
第十七模块,用于将所述第一特征数据和所述第二特征数据合集处理,得到所述待测细胞的第三特征数据;
第十八模块,用于将所述待测细胞的第三特征数据输入所述第二药敏预测模型,以预测所述待测细胞的药敏数据。
其中,该装置的第十五模块获取待测细胞的基因表达数据,并根据该待测细胞的基因表达数据,与基因集数据库中的每个基因集进行基因数据对照,获得该待测细胞在基因集数据库的每个基因集上的得分。示例性的,可根据该待测细胞的基因表达数据,采用ssGSEA方法,与MsigDB数据库中的每个基因集进行基因数据对照,获得该待测细胞在MsigDB数据库的每个基因集上的得分。在将该待测细胞的基因表达数据与MsigDB数据库中的每个基因集进行基因数据进行对照时,如果该待测细胞的基因表达数据中不包含某个基因集上的某个或者某些基因的数据,则在该待测细胞的基因表达数据中补全这个或这些基因的数据,以得到该待测细胞的结构完整的基因表达数据。
其中,该装置的第十六模块将该待测细胞对应的基因集数据库的每个基因集上的得分进行归一化处理,得到该待测细胞的第一特征数据。示例性的,若基因集数据库有n个基因集,或选用基因集数据库的n个基因集,获取该待测细胞的基因表达数据在这n个基因集上的得分,则待测细胞的第一特征数据可以包括:n个特征,即待测细胞的基因表达数据在n个基因集上的归一化得分,每个归一化得分的具体数值即为每个特征的特征值。还可对该待测细胞的第一特征数据进行数学处理,获得该待测细胞的第二特征数据。示例性的,该数学处理可以包括以下至少一项:平方运算、交叉运算、立方运算、自然对数运算和box-cox变换。
其中,该装置的第十七模块将该待测细胞的第一特征数据和第二特征数据进行汇总合集处理,组成数据集合,得到该待测细胞的第三特征数据。
其中,该装置的第十八模块将该待测细胞的第三特征数据输入如前述图4所示方法实施例的第二药敏预测模型,以预测该待测细胞的药敏数据。其中,还可以先从该待测细胞的第三特征数据中筛选出与前述图3所示方法实施例中相同的预设数量个重要性得分最高的特征数据,然后将这预设数量个重要性得分最高的特征数据输入如前述图4所示方法实施例的第二药敏预测模型,以预测该待测细胞的药敏数据。
本申请的一个可选实施例中,还提供了一种计算机可读介质,所述计算机可读介质存储有计算机可读指令,所述计算机可读指令可被处理器执行以实现前述各方法实施例或可选实施例。
需要注意的是,本申请中各方法实施例或可选实施例可在软件和/或软件与硬件的组合体中被实施。本申请中涉及的软件程序可以通过处理器执行以实现上述各实施例的步骤或功能。同样地,本申请的软件程序(包括相关的数据结构)可以被存储到计算机可读记录介质中。
另外,本申请的一部分可被应用为计算机程序产品,例如计算机程序指令,当其被计算机执行时,通过该计算机的操作,可以调用或提供根据本申请的方法和/或技术方案。而调用本申请的方法的程序指令,可能被存储在固定的或可移动的记录介质中,和/或通过广播或其他信号承载媒体中的数据流而被传输,和/或被存储在根据所述程序指令运行的计算机设备的工作存储器中。
本申请的又一个可选实施例中,还提供了一种用于药敏预测模型样本构建的设备,该设备包括:存储计算机程序指令的存储器和用于执行程序指令的处理器,其中,当该计算机程序指令被该处理器执行时,触发该设备执行如前述各方法实施例和/或可选实施例,和/或技术方案的操作。
对于本领域技术人员而言,显然本申请不限于上述示范性实施例的细节,而且在不背离本申请的精神或基本特征的情况下,能够以其他的具体形式实现本申请。因此,无论从哪一点来看,均应将实施例看作是示范性的,而且是非限制性的,本申请的范围由所附权利要求而不是上述说明限定,因此旨在将落在权利要求的等同要件的含义和范围内的所有变化涵括在本申请内。不应将权利要求中的任何附图标记视为限制所涉及的权利要求。此外,显然“包括”一词不排除其他单元或步骤,单数不排除复数。装置权利要求中陈述的多个单元或装置也可以由一个单元或装置通过软件和/或者硬件来实现。第一,第二等词语用来表示名称,而并不表示任何特定的顺序。
