一种基于双路径并行集成决策的DHSs识别预测方法
文献发布时间:2023-06-19 19:30:30
技术领域
本发明涉及位点预测技术领域,尤其涉及一种基于双路径并行集成决策的DHSs识别预测方法。
背景技术
遗传学中,对DNase I核酸内切酶的切割过敏的特定染色质区域被称为DNase I超敏位点(DHSs),DHSs能够提供哺乳动物中转录调控元件和染色质状态的重要信息,多年来,DHSs为确定启动子、增强子、抑制子和基因组控制区等转录调控元件的精确位置担任了可靠的路标;此外,全基因组关联研究(GWAS)确定了许多与癌症、心血管疾病等疾病和性状相关的非编码变体。而这些变体集中在DHSs标记的调节DNA中,因此,准确识别DHSs有助于进一步探究基因的调控与常见疾病的致病机制;
DNase I超敏位点(DHS)是对DNase I酶的切割表现出超敏反应的染色质区域,它是调节DNA的通用标记,并且与多种疾病和表型性状的遗传变异相关,然而,常规的实验方法识别DHSs耗时且昂贵,而在小鼠不同发育阶段的基因上的DHSs是动态变化的,不同组织的序列也有很大的差异性,单一模型往往难以实现对多类型数据的准确识别,因此,本发明提出一种基于双路径并行集成决策的DHSs识别预测方法以解决现有技术中存在的问题。
发明内容
针对上述问题,本发明的目的在于提出一种基于双路径并行集成决策的DHSs识别预测方法,该基于双路径并行集成决策的DHSs识别预测方法通过BiGRU-Attention模型和MSFRN模型的双模型训练的方式可以捕获到不同层次的重要信息,减少信息丢失,构成的双路径iDHS-DPPE模型识别预测,能够有效提升泛化能力和鲁棒性,可以适应小鼠不同组织和发育阶段DHS的变化,同时能够有效、准确的识别DHSs。
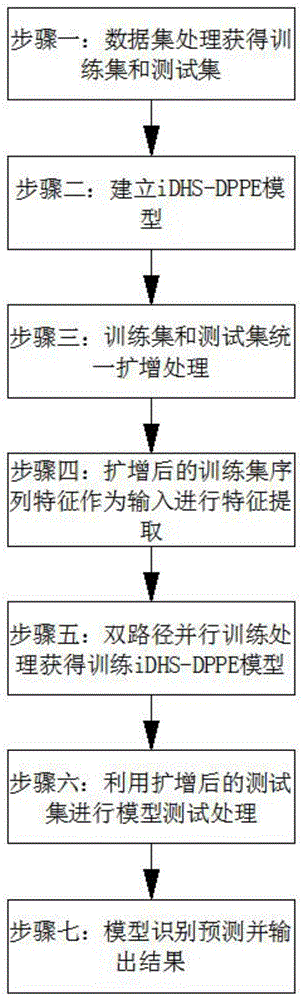
为实现本发明的目的,本发明通过以下技术方案实现:一种基于双路径并行集成决策的DHSs识别预测方法,包括以下步骤:
步骤一、数据集处理,获取iDHS-Deep基准数据集中小鼠基因组的DHS序列,再利用CD-HIT对其进行处理后划分获得训练集和测试集;
步骤二、模型建立,建立包含特征提取模块、双路径集成模块和输出模块的iDHS-DPPE模型,其中双路径集成模块包含BiGRU-Attention模型和MSFRN模型;
步骤三、训练集和测试集统一化处理,将训练集和测试集中的序列长度进行扩充处理,获得统一长度的序列并按照滑动窗口拆分为2-mers,获得扩增后的训练集和测试集;
步骤四、特征提取,将扩增后的训练集序列特征作为输入经过iDHS-DPPE模型的特征提取模块进行特征提取,获得提取特征;
步骤五、双路径并行训练,将获得的提取特征作为输入分别导入包含BiGRU-Attention模型和MSFRN模型的双路径集成模块中进行训练,并结合特征提取模块和输出模块获得训练后的iDHS-DPPE模型;
步骤六、模型测试,将扩增后的测试集同样经过步骤四的特征提取处理,并将获得的提取特征输入训练后的iDHS-DPPE模型进行模型测试,获得测试后的iDHS-DPPE模型;
步骤七、模型识别预测,将待测数据经过步骤三和步骤四的处理后输入测试后的iDHS-DPPE模型,并由其输出识别预测结果。
进一步改进在于:所述步骤一中获取的小鼠基因组的DHS序列包含小鼠不同组织和发育阶段的DHS综合图谱,选择50~301bp长度的DHS序列作为阳性样本,选择特定的DNA片段作为阴性样本,然后利用CD-HIT消除相似度阈值为0.8的序列,最后将不同细胞类型和发育阶段的样本中70%作为训练集,30%作为独立的测试集。
进一步改进在于:所述步骤二中特征提取模块包含嵌入层、卷积层和池化层,所述输出模块将双路径集成模块输出的决策集成得到综合评估的结果输出并进行DHSs的识别。
进一步改进在于:所述步骤二中BiGRU-Attention模型包含BiGRU和多头注意力层,利用BiGRU捕获远程依赖,利用多头注意力层选择关键特征信息,所述MSFRN模型包含进行级联的六个卷积核为64的不同尺度的卷积层以及一个卷积核为192的卷积层进行特征的残差连接融合。
进一步改进在于:所述步骤三中扩充处理具体为当序列长度不足301bp时,在序列尾部添加“N”,将长度不足的序列统一扩充至最大长度301bp,然后进行拆分获得2-mers,然后再将不同的核苷酸映射为0~20内范围不同的值,则扩充序列被编码为长度300的数字向量,作为特征提取模块的输入。
进一步改进在于:所述步骤五中模型训练的输出利用G值来衡量置信度,G值的计算公式如下
G=abs(2cp-1),cp∈[0,1]
其中cp为置信概率,则当置信概率cp越高,G至越大,则判定模型的判断可靠,当cp≥0.5时,模型判断输入序列为DHSs,相反则输入序列不是DHSs。
本发明的有益效果为:本发明通过BiGRU-Attention模型和MSFRN模型的双模型训练的方式可以捕获到不同层次的重要信息,减少信息丢失,构成的双路径iDHS-DPPE模型识别预测,能够有效提升泛化能力和鲁棒性,可以适应小鼠不同组织和发育阶段DHS的变化,同时能够有效、准确的识别DHSs,为DHSs识别提供了一种高效可靠的方法。
附图说明
图1为本发明实施例1方法流程图。
图2为本发明实施例1中iDHS-DPPE模型总体架构图。
图3为本发明实施例1中BiGRU-Attention模型架构图。
图4为本发明实施例1中MSFRN模型架构图。
图5为本发明实施例2不同特征编码方式的性能比较折线图。
图6为本发明实施例2与不同机器学习分类器的性能比较结果图。
图7为本发明实施例2多种神经网络的比较结果图。
图8为本发明实施例2与现有多种方法的比较结果图。
具体实施方式
为了加深对本发明的理解,下面将结合实施例对本发明做进一步详述,本实施例仅用于解释本发明,并不构成对本发明保护范围的限定。
实施例1
根据图1-图4所示,本实施例提供了一种基于双路径并行集成决策的DHSs识别预测方法,包括以下步骤:
步骤一、数据集处理,获取iDHS-Deep基准数据集中小鼠基因组的DHS序列,再利用CD-HIT对其进行处理后划分获得训练集和测试集;
获取的小鼠基因组的DHS序列包含小鼠不同组织和发育阶段的DHS综合图谱,选择50~301bp长度的DHS序列作为阳性样本,选择特定的DNA片段作为阴性样本,然后利用CD-HIT消除相似度阈值为0.8的序列,最后将不同细胞类型和发育阶段的样本中70%作为训练集,30%作为独立的测试集,具体如下表1。
表1数据集详情
/>
步骤二、模型建立,建立包含特征提取模块、双路径集成模块和输出模块的iDHS-DPPE模型,其中双路径集成模块包含BiGRU-Attention模型和MSFRN模型,如说明书附图2所示;
其中特征提取模块包含嵌入层、卷积层和池化层,所述输出模块将双路径集成模块输出的决策集成得到综合评估的结果输出并进行DHSs的识别;
具体为将DNA序列由2-mers编码方案映射到嵌入层、卷积层和池化层进一步提取特征信息;通过双路径的BiGRU-Attention模型和MSFRN模型分别捕获局部信息和远程依赖,获得概率表示;最后,将这两个模型的决策集成得到综合评估的输出结果,识别是否为DHSs;
BiGRU-Attention模型包含BiGRU和多头注意力层,利用BiGRU捕获远程依赖,BiGRU将生成的正向序列和反向序列进行拼接,结果输出传递到下游网络,能够充分地学习序列中的上下文信息,BiGRU的输出设置为35维,最后两个相反方向的输出拼接为70维;
利用多头注意力层选择关键特征信息,多头注意力层使模型训练过程中更加关注重要特征,忽略不重要的信息,为BiGRU的输出分配不同的权重,在每个头部内获得一个中间表示,然后将每个头的结果连接起来并映射回原始维度,如说明书附图3所示;
所述MSFRN模型包含进行级联的六个卷积核为64的不同尺度的卷积层以及一个卷积核为192的卷积层进行特征的残差连接融合,跨通道组织信息,如说明书附图4所示,进行级联的六个卷积核为64的不同尺度的卷积层可以得到序列不同尺度的信息,减少信息的丢失,实现分层特征的融合;
MSFRN模型通过融合不同尺度的特征,提取了相邻特征之间的局部关系,提升模型表达能力。
步骤三、训练集和测试集统一化处理,由于卷积神经网络需要固定大小的输入,而数据集分成的训练集和测试集中序列长度不等,因此将训练集和测试集中的序列长度进行扩充处理,获得统一长度的序列并按照滑动窗口拆分为2-mers,获得扩增后的训练集和测试集;
扩充处理具体为当序列长度不足301bp时,在序列尾部添加“N”,将长度不足的序列统一扩充至最大长度301bp,然后进行拆分获得2-mers,然后再将不同的核苷酸映射为0~20内范围不同的值,则扩充序列被编码为长度300的数字向量,作为特征提取模块的输入,将生成的序列特征导入嵌入层矩阵经过卷积层和最大池化层进一步进行特征提取。
步骤四、特征提取,将扩增后的训练集序列特征作为输入经过iDHS-DPPE模型的特征提取模块进行特征提取,获得提取特征。
步骤五、双路径并行训练,将获得的提取特征作为输入分别导入包含BiGRU-Attention模型和MSFRN模型的双路径集成模块中进行训练,并结合特征提取模块和输出模块获得训练后的iDHS-DPPE模型;
模型训练的输出利用G值来衡量置信度,G值的计算公式如下
G=abs(2cp-1),cp∈[0,1]
其中cp为置信概率,则当置信概率cp越高,G至越大,则判定模型的判断可靠,当cp≥0.5时,模型判断输入序列为DHSs,相反则输入序列不是DHSs。
步骤六、模型测试,将扩增后的测试集同样经过步骤四的特征提取处理,并将获得的提取特征输入训练后的iDHS-DPPE模型进行模型测试,获得测试后的iDHS-DPPE模型。
步骤七、模型识别预测,将待测数据经过步骤三和步骤四的处理后输入测试后的iDHS-DPPE模型,并由其输出识别预测结果,输出结果与步骤五中判断方法相同,当cp≥0.5时,模型判断输入序列为DHSs,相反则输入序列不是DHSs。
实施例2
根据图5-图8所示,本实施例提供了一种基于双路径并行集成决策的DHSs识别预测方法的比较和讨论。
一、评估标准
使用包含包括灵敏度(Sn)、特异性(Sp)、准确度(Acc)和马修相关系数(Mcc)四种通用指标来评价模型的预测性能,计算公式如下:
其中TP、TN、FP和FN分别表示真阳性、真阴性、假阳性和假阴性的数量,同时曲线下面积(AUROC)和精确召回曲线下面积(AUPR)也被用来评估模型。
二、与不同特征表示的比较
初始特征会对深度学习模型的训练结果产生重要影响。为了确定最佳的核苷酸映射的长度,进行了实验。由于序列长度的限制,对于k-mers中k,只考虑k=1,2,3的情况。另外,生物领域常见的onehot编码方案也被添加。所有数据集5折交叉验证得到的AUROC值显示在说明书附图5中。
1-mers和onehot都是提取单个碱基的信息,但可能忽视了碱基与碱基之间的联系。而2-mers将两个碱基看作一个整体,更能充分利用相邻核苷酸之间的长期依赖性。3-mers也可以捕获多个碱基对的顺序信息,所以能够得到很好的预测性能。但更高阶的编码往往意味着更高的计算成本,容易出现过拟合的情况。然而在部分数据量较小的数据集中,2-mers会比3-mers更合适。
结果证明了2-mers编码方案能够充分地提取序列信息,对于识别DHSs是有效的。
三、与不同机器学习分类器的比较
对iDHS-DPPE和SVM、KNN、RF、XGBoost等四种常见机器学习分类器进行比较。其中,SVM、KNN、RF和XGBoost均采用默认的参数设置。公平地说,这些方法与iDHS-DPPE同样都是以2-mers编码的特征向量作为输入。AUROC结果如说明书附图6所示。iDHS-DPPE明显最优,说明了构建集成深度学习模型对预测DHSs是有优势的、可行的。
四、与不同的神经网络结构的比较
将特征向量输入到多种不同的神经网络中以验证双路径并行集成策略的效果,与3种结构进行比较:CNN-LSTM、BiGRU-Attention模型和MSFRN模型;其中CNN-LSTM网络结构与iDHS-Deep中相同。
CNN-LSTM、BiGRU-Attention、MSFRN和iDHS-DPPE的AUROC值平均值分别为0.910、0.926、0.928和0.931,如说明书附图7所示。可以发现iDHS-DPPE在所有方法中取得了最好的结果;与CNN-LSTM的比较说明了iDHS-DPPE的网络结构能够更加准确地表达特征;与BiGRU-Attention、MSFRN的比较验证了双路径并行集成策略的有效性,增强了模型的预测性能。
五、与现有方法的比较
为了进一步证明实施例1中iDHS-DPPE模型的优越性,在神经管基准数据集上将其与以下已经发表的方法进行比较,包括SVM-RevcKmer、iDHS-EL、iDHSs-PseTNC、iDHS-DSAMS、iDHS-DXG、iDHS-Deep。其中iDHS-Deep是专门用于小鼠基因组中的DHSs的预测工具,而其他方法则是用于预测人类的。实验结果如说明书附图8所示,可以看到,iDHS-DPPE在所有评估指标中都是最优的。
另外,iDHS-Deep和iDHS-DPPE在所有独立测试集上得到的平均结果件下表2。具体来说,iDHS-DPPE的Sn、Sp、Acc、Mcc和AUROC的平均值分别比最先进的方法iDHS-Deep高0.9%、3.8%、2.1%、4.3%和2%。显然,iDHS-DPPE在预测区分小鼠DHSs方面更加强大稳健。
表2与iDHS-Deep独立测试的平均结果比较
以上显示和描述了本发明的基本原理、主要特征和优点。本行业的技术人员应该了解,本发明不受上述实施例的限制,上述实施例和说明书中描述的只是说明本发明的原理,在不脱离本发明精神和范围的前提下,本发明还会有各种变化和改进,这些变化和改进都落入要求保护的本发明范围内。本发明要求保护范围由所附的权利要求书及其等效物界定。
